Method Article
Çekirdek Miktarını Kolaylaştırmak için Bilgisayarla Görme Kitaplıklarını Kullanma
Bu Makalede
Özet
Bu makalede, bir dizi hücre yoğunluğunda doğrulanmış açık kaynaklı bir yürütülebilir program kullanarak görüntü tabanlı çekirdek nicelemeyi otomatikleştirmek için adım adım yöntemler açıklanmaktadır. Bu program, maliyetle ilgili engelleri, sınırlı teknolojik becerilere sahip kullanıcılar için erişilebilirliği ve mevcut teknolojilerin kullanımını sınırlayabilecek uygulamaya özel doğrulamayı ele alan bir alternatif sunar.
Özet
Canlı hücre tahlilleri ve görüntü tabanlı hücre analizleri, doğru yorumlama için veri normalizasyonu gerektirir. Yaygın olarak kullanılan bir yöntem, çekirdekleri boyamak ve nicelleştirmek, ardından çekirdek sayısına veri normalizasyonudur. Bu çekirdek sayısı genellikle tek çekirdekli hücreler için hücre sayısı olarak ifade edilir. Manuel niceleme zahmetli ve zaman alıcı olabilse de, mevcut otomatik yöntemler tüm kullanıcılar tarafından tercih edilmeyebilir, bu özel uygulama için doğrulama olmayabilir veya maliyet açısından engelleyici olabilir. Burada, floresan DNA lekeleriyle boyanmış çekirdeklerin ölçülebilir görüntülerini yakalamak ve ardından Python bilgisayarla görme kitaplıkları kullanılarak geliştirilen otomatik bir nesne sayma yazılım programı kullanarak çekirdekleri ölçmek için adım adım talimatlar sunuyoruz. Ayrıca bu programı bir dizi hücre yoğunluğunda doğruluyoruz. Programın yürütülmesi için tam süre, görüntü sayısına ve bilgisayar donanımına bağlı olarak değişse de, bu program, programın çalışması için çekirdekleri sayma saatlerini saniyelere birleştirir. Bu protokol sabit, lekeli hücrelerin görüntüleri kullanılarak geliştirilmiş olsa da, canlı hücrelerdeki lekeli çekirdeklerin görüntüleri ve immünofloresan uygulamaları da bu program kullanılarak ölçülebilir. Sonuç olarak, bu program, yüksek derecede teknolojik beceri gerektirmeyen bir seçenek sunar ve hücre ve moleküler biyologların iş akışlarını düzene sokmalarına, sıkıcı ve zaman alıcı çekirdek niceleme görevini otomatikleştirmelerine yardımcı olmak için doğrulanmış, açık kaynaklı bir alternatiftir.
Giriş
Fonksiyonel ve görüntü tabanlı deneyler, deneysel tedavilerin tüm hücre biyokimyası ve fizyolojisi üzerindeki etkilerini anlamak için kritik öneme sahiptir. Hücre biyolojisi deneylerinden elde edilen verilerin geçerli bir şekilde yorumlanması, veri normalizasyonu da dahil olmak üzere deneysel protokolün doğruluğuna ve tekrarlanabilirliğine bağlıdır. Örneğin, başlangıçta ve spesifik ilaçlarla tedaviden sonra canlı hücrelerde oksijen tüketimi ve hücre dışı asitleşme oranlarının analizleri, enerji metabolizmasının çeşitli yönlerinin değerlendirilmesine izin verir 1,2. Hücre kültürlerinin süpernatanında laktat dehidrojenaz gibi enzimlerin aktivitesinin ölçülmesi, hücre zarı bütünlüğününölçülmesine yardımcı olabilir 3. Kültürlenmiş hücrelerin fiksasyondan önce annexin V ve propidium iyodür ile boyanması, apoptotik ve nekrotik hücrelerindeğerlendirilmesine izin verir 4. Bununla birlikte, kuyular arasındaki hücre yoğunluğundaki farklılıklar, bu tahlillerin her birinin sonuçlarını etkiler. Yalnızca tohumlama yoğunluğuna güvenmek, tohumlama için hücre sayımındaki hatalar, kaplama sırasında ortamdaki hücre yoğunluğundaki değişiklikler veya deneyler boyunca numuneler veya tedaviler arasındaki farklı hücre büyümesi oranları nedeniyle yanıltıcı sonuçlar sağlayabilir. Bu nedenle, deneysel sonuçların normalleştirilmesi gereklidir.
İşlevsel ve görüntü tabanlı hücre verileri için mevcut normalleştirme yöntemleri, protein konsantrasyonu5 veya çekirdek veya hücre sayısı6'yı içerir. Verileri protein konsantrasyonuna normalleştirmek için, tahlil çalıştırıldıktan sonra hücrelerin proteaz inhibitörü ile eşit hacimlerde lizis tamponunda ekstrakte edilmesi ve protein miktar tayini için ek bir tahlil (örneğin, bikinkoninik asit tahlili, Bradford tahlili, vb.) yapılması gerekir. Bu yöntemin doğruluğu tam hücre ekstraksiyonuna dayanır; Herhangi bir proteini geride bırakmak, miktar belirleme hatalarına katkıda bulunur. Çekirdekleri veya hücreleri saymak, üzerinde büyüdükleri yüzeyden hiçbir hücrenin çıkarılmasının gerekmediği bir alternatif sağlar. Bunun yerine, canlı veya sabit hücreler, kolorimetrik veya floresan boyalar kullanılarak boyanır veya karşı boyanır ve sayım, tüm hücrelerin veya sadece çekirdeklerin görüntülerine dayanarak yapılabilir. Floresan mikroskobu uygulamaları için tüm hücreler yerine çekirdeğin ölçülmesi ve kaynaşmış hücrelerdeki (örneğin miyotüpler) veya dokulardaki verileri normalleştirmek için tercih edilir. Bir floresan yaklaşımı kullanılarak çekirdeklerin miktar tayini için, floresan DNA boyaları (örneğin, 4',6-diamidino-2-fenilindol diklorür (DAPI), Hoechst 33342, Hoechst 33358, DRAQ5, vb.) canlı veya sabit hücrelere uygulanır ve floresan mikroskobu kullanılarak görüntülenir. DAPI, Hoechst ve DRAQ5 tercihen DNA 7,8,9'un adenin-timin açısından zengin bölgelerine bağlanarak çekirdekleri tespit etmek için bir araç sağlar. Boya konsantrasyonunun optimizasyonu, hedef dışı bağlanmayı önler (yani, mitokondriyal DNA'ya, punktat sitozolik floresansa neden olur)10. Optimizasyon süreci, sabit hücreler için DAPI içeren ticari olarak temin edilebilen montaj ortamı (örneğin, DAPI'li VECTASHIELD Antifade Montaj Ortamı) ve canlı hücreler için yayınlanmış leke konsantrasyonları (örneğin, Hoechst) kullanılarak kolaylaştırılabilir6,11,12. Boyandıktan sonra, canlı veya sabit hücrelerdeki çekirdekler, uygun uyarma filtresi (örneğin, ultraviyole [DAPI ve Hoechst] veya uzak kırmızı [DRAQ5]) kullanılarak görselleştirilir. DAPI, Hoechst ve DRAQ5 için uyarma maksimumları, floresan mikroskobu için spektrumun her iki ucundadır. Bu özellik ve bu boyaların DNA özgüllüğü ve optimum konsantrasyonlarda kullanımları, hedef dışı floresansı en aza indirir. Elde edilen görüntüler, siyah bir arka plana karşı parlak çekirdekleri gösterir ve bu çekirdekler, tek çekirdekli hücreler için hücre sayısının bir ölçüsü olarak ölçülür. Histolojik boyalar ve parlak alan mikroskobu13 kullanılarak diğer doğru leke bazlı çekirdek niceleme yöntemleri mevcut olsa da, bu yaklaşımları kullanarak otomasyon mümkün olsa da daha zordur.
Altın standart olmaya devam etse de, manuel çekirdek ölçümü zahmetli, zaman alıcıdır ve uzun süreli sayımla potansiyel insan hatasına karşı hassastır. Otomatik hücre sayma programları mevcut olmasına rağmen, bunlar tüm kullanıcılar tarafından tercih edilmez, maliyet açısından engelleyici olabilir ve belirli uygulamalar için doğrulama minimum düzeyde olabilir. Python, son yıllarda biyologlar için değerli ve erişilebilir bir kaynak haline geldi. Python bilgisayarla görme kitaplıkları özellikle görüntü analiziiçin kullanışlıdır 14. Burada, floresan boyalarla boyanmış ve floresan mikroskobu kullanılarak görüntülenen çekirdekleri ölçmek için Python kodu kullanılarak geliştirilen yürütülebilir programımızı kullanmak için adım adım talimatlar sunuyoruz. Burada açıklanan programı kullanmak için kodlama becerileri gerekli değildir. Bu iş akışı, floresan DNA boyalarının uygulandığı hücrelere veya dokulara özgüdür. Parlak alanlı görüntülerle kullanılmak üzere tasarlanmamıştır. Bölüm 1'de, floresan mikroskobu kullanılarak toplanan görüntülerin bu iş akışıyla uyumlu bir şekilde nasıl yakalanacağı ve kaydedileceği açıklanmaktadır. Bölüm 2, çekirdek niceleme programını çalıştırılabilir ve getirilebilir bir çıktı olarak çalıştırmak için talimatlar sağlar. Yürütülebilir program, sağlanan .exe dosyasından doğrudan çalıştırılabilir ve kaliteli görüntülerdeki çekirdekleri ölçmek için herhangi bir değişiklik gerektirmez. Bu yürütülebilir dosya Windows tabanlı bir bilgisayar gerektirir. Bölüm 3, programı yürütülebilir dosya yerine bir Python betiği olarak çalıştırmak için talimatlar sağlar (Mac veya Linux tabanlı sistemler için gereklidir, Windows tabanlı sistemler için isteğe bağlıdır) ve istenirse kodun ayarlanmasına izin verir. Bölüm 4'te sonuçların normalleştirme için nasıl kullanılacağı açıklanmaktadır. Bu protokolleri, laboratuvarımızdan alınan doğrulama verileri de dahil olmak üzere temsili sonuçlar takip eder.
Protokol
NOT: Ek dosyalar aşağıdaki bağlantıda bulunabilir https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. Floresan mikroskobu kullanarak görüntü yakalama ve kaydetme
- İstenilen DNA boyası ile boyama da dahil olmak üzere görüntülenecek hücre veya doku örneklerini hazırlayın. Burada kullanılan görüntüleri elde etmek için, C2C12 miyoblastları (CRL-1772, Amerikan Tipi Kültür Koleksiyonu), standart kültür koşulları altında (% 5 CO2, 37 ° C, nemlendirilmiş), 50 mM EtOH 6 ile veya 50 mM EtOH6 olmadan 48-72 saat boyunca 6 oyuklu plakalarda büyütüldü ve daha önce tarif edildiği gibi buz gibi soğuk metanol içinde sabitlendi13. Sabit hücreler, DAPI içeren bir montaj ortamı15 kullanılarak monte edildi ve görüntülemeden önce karanlıkta kurutuldu.
- Floresan mikroskobunun ve bilgisayarın tüm bileşenlerini yerleşik standart çalıştırma prosedürlerine göre açın. Mikroskopla ilişkili yazılımı açın.
- Örnekleri göz merceğinden görselleştirmek için mikroskobu ayarlayın. Hedefi kontrol edin. Gerekirse, burunluğu istenen hedefe çevirin. Görüntü alanının doğru bir şekilde gösterilmesi için yazılım programının istenen hedefin seçildiğinden emin olun. Burada, görüntüler 10x'lik bir objektif kullanılarak yakalandı.
- Işıklar kapalıyken, numuneyi (slayt, tabak veya tabak) sahneye yerleştirin. Uygun filtreyle canlı görüntülemeyi açın ve pozlama süresini önceden belirlenmiş bir süreye veya otomatik pozlamaya ayarlayın. Burada DAPI filtresi ve 61-62 ms pozlama süresi kullanılmıştır. Özellikle yüksek hücre yoğunluğuna sahip görüntüler için pozlama süresini ayarlama konusunda rehberlik için sorun giderme tablosuna bakın.
- Göz merceğini kullanarak, lekeli çekirdekler görünene kadar brüt ayar düğmesini kullanarak odağı yavaşça ayarlayın, ardından çekirdekler net ve net görünene kadar odağı yavaşça ayarlamak için ince ayar düğmesini kullanın.
NOT: Uygun görüntü örnekleri Şekil 1'de gösterilmiştir.- Arka planda aşırı lekelenme görülürse veya çekirdekler çok sönük görünürse, maruz kalma süresini manuel olarak ayarlayın. Tek bir görüntüde, bazı çekirdekler için sinyal yoğunluğunda gözle görülür farklılıklar olabilir; Bununla birlikte, yuvarlak şekil, minimum sinyal paraziti ile tüm çekirdekler için görünür olmalıdır.
NOT: Alt uygulamalarda kullanılan program, farklı yoğunluklara karşı dayanıklıdır. Çekirdek niceleme programının geliştirilmesi ve doğrulanması için kullanılan ham görüntüler, bir dizi sinyal yoğunluğuna sahip görüntüleri içerir ve Açık Bilim Çerçevesi deposu aracılığıyla edinilebilir (yukarıda belirtilen bağlantıya bakın).
- Arka planda aşırı lekelenme görülürse veya çekirdekler çok sönük görünürse, maruz kalma süresini manuel olarak ayarlayın. Tek bir görüntüde, bazı çekirdekler için sinyal yoğunluğunda gözle görülür farklılıklar olabilir; Bununla birlikte, yuvarlak şekil, minimum sinyal paraziti ile tüm çekirdekler için görünür olmalıdır.
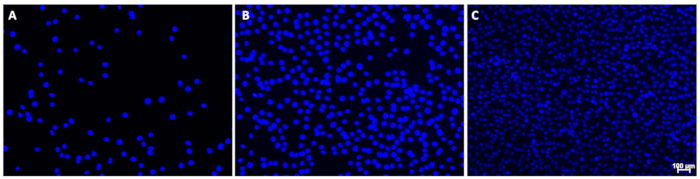
Şekil 1: DAPI ile boyanmış çekirdeklerin örnek görüntüleri. Bu görüntüler, minimum arka plana sahip (A) düşük, (B) orta veya (C) yüksek hücre yoğunluklarındaki çekirdekleri göstermektedir. Görüntü C'deki çekirdeklerin değişen sinyal yoğunluğuna dikkat edin; Bu, nicelleştirmeye müdahale etmedi. Ölçek çubuğu (C) 100 μm'yi gösterir; Çekirdek ölçümü için kullanılan görüntülerden ölçek çubukları çıkarılmalıdır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- Çekirdekler göz merceğinden odaklandıktan sonra, algılama modunu kameraya geçirin. İnce ayar düğmesini kullanarak odağı ayarlayın.
- Gerekirse pozlama süresini ayarlayın. Sonraki örnekler ve görüntülerle kullanım için son pozlama süresine dikkat edin.
NOT: Tüm görüntülerde aynı pozlama süresini kullanmak ideal olsa da, floresan yoğunluğu numuneler arasında ölçülmediği veya karşılaştırılmadığı için pozlama süresindeki farklılıklar bu uygulama için kabul edilebilir. - Yazılımdaki uygun görüntü yakalama düğmesini kullanarak görüntüyü yakalayın. Numunelere birden fazla leke uygulanmışsa, filtreyi değiştirin (örneğin, FITC, TRITC veya Cy5'e) ve birleştirme amacıyla aynı alanı görüntüleyin.
- Örneğin/numunenin ışığa maruz kalmasını en aza indirmek için deklanşörü uygulayın. Ölçek çubuğuna yazmadan, görüntüyü evrensel bir şekil dosyası biçiminde (.tif, .png veya .jpg) özel bir klasöre kaydedin. Her görüntü dosyası adının uygun şekilde açıklayıcı olduğundan veya dosya adları için kullanılan herhangi bir kodlamanın uygun bağlantılı belgelere sahip olduğundan emin olun.
NOT: Bu, niceleme programı için Görüntü Dizini olacaktır. Çok renkli görüntüleme kullanılmışsa, yalnızca çekirdeklerin görüntülerini ayrı, ayrılmış bir klasöre kopyalayın. Bu, görüntüleme seansının sonunda da yapılabilir. Çoğu görüntüleme yazılımında varsayılan dışa aktarma ayarları kullanılarak dışa aktarılan görüntüler, kaliteli görüntülerin yakalanmış olması koşuluyla, genellikle bu programla kullanım için yeterli çözünürlüktedir. Bununla birlikte, çekirdeklerin morfolojisinin net olduğundan emin olmak için görüntüler dışa aktarıldıktan sonra görsel olarak kontrol edilmelidir. Görüntüler .jpg biçiminde kaydedilirse, görüntülerin aşırı sıkıştırılmamasına ve kalitenin korunmasına dikkat edilmelidir. - Deklanşörü açın, canlı görüntülemeye dönün ve görüntüleme için örnek üzerinde başka bir konuma tarayın. 1.8-1.9 adımlarını gerektiği kadar tekrarlayın. 10x büyütmede yakalanan 6 oyuklu plakalara ekilen hücrelerin görüntüleri için, oyuk başına 10-15 görüntü yakalayın (numune alanı boyunca hücre yoğunluğunun tutarlılığına bağlı olarak).
- Tek bir örnek için tüm görüntüler alındıktan sonra, aşağıdaki seçeneklerden birini gerçekleştirin.
- Sonraki örneğe geçin ve 1.4-1.10 arasındaki adımları tekrarlayın.
- Protokolü duraklatın ve başka bir zaman geri dönün. Numuneler ışıktan korunmalıdır. Canlı hücrelerle çalışıyorsanız bu seçenek önerilmez.
- Görüntüleme tamamlandıktan sonra, oda ışıklarını açmadan önce tüm numunelerin ışık korumalı olduğundan emin olun. Görüntüleri içeren klasör yerel olarak kaydedilmişse, dosyaları bir flash sürücüye, bulut depolama konumuna veya niceleme programının çalıştırılacağı bilgisayarın erişebileceği başka bir ağ sürücüsüne aktarın. Özel bir klasöre kaydedin.
- Mikroskobun ve bilgisayarın tüm parçalarını yerleşik standart çalıştırma prosedürlerine göre kapatın.
- Görüntüleme işlemi tamamlanmıştır. Bölüm 2'de açıklandığı gibi otomatik niceleme programını kullanarak veya alternatif olarak programı Bölüm 3'te açıklandığı gibi bir Python betiği olarak çalıştırarak dosyaları analiz edin.
2. Otomatik çekirdek niceleme programını yürütülebilir bir dosya olarak çalıştırmak
NOT: Programı yürütülebilir dosya olarak çalıştıran bu yöntem, Windows tabanlı bilgisayarlarla uyumludur ve Windows işletim sistemi kullanıcıları için önerilen yöntemdir. Mac OS veya diğer Windows olmayan kullanıcılar programı bir Python betiği olarak çalıştırmalıdır (bkz. Bölüm 3).
- Bilgisayarda Python'un kurulu olduğundan emin olun. Gerekirse Python'u python.org/downloads'dan (tercih edilen) veya Anaconda ya da Miniconda kullanarak yükleyin. Yayınlandığı tarih itibariyle, bu program Python 3.10-3.12 sürümleri ile test edilmiştir.
- Tarayıcıyı açın ve https://github.com/rbudnar/nuclei_counter/releases gidin. count_nuclei.zip adlı dosyanın en son sürümünü indirin.
- İndirilenler klasöründen, Zip dosyasına sağ tıklayın ve Tüm dosyaları yerel bilgisayarda istediğiniz konuma ayıklayın (bkz. Şekil 2A). Bu dosya konumunu açık tutun; bu dosya yolu adım 2.5'te kullanılacaktır (bkz. Şekil 2B).
NOT: Count_nuclei artık bir uygulama dosyası olarak görünmelidir. Bu yürütülebilir dosyadır. - Arama çubuğunda CMD veya Komut İstemi'ni arayarak bir komut istemi açın. Dizini yürütülebilir dosyanın dosya yoluna değiştirmek için cd komutunu kullanın (bkz. Şekil 2C) ve Enter tuşuna basın. Örnek komut satırı: C:\Users\janedoe>cd [cd'den sonra boşluk, ardından dosya yolunu kopyala/yapıştır]
- Sonraki komut satırına aşağıdakini yazın ve Enter tuşuna basın.
count_nuclei.exe --img_dir "yol/için/görüntüler" --output_dir "yol/hedef/çıktı" --save_images --output_count_filename "results.csv"
burada path/to/images, analiz edilecek görüntüleri içeren klasörün dosya yoludur (bkz. adım 1.14); path/to/output, .csv dosyasının kaydedilmesi gereken klasörün dosya yoludur (sağlanmadığında varsayılan olarak geçerli çalışma dizinine geçer); ve results.csv, elde edilen sayımların kaydedileceği dosya adıdır (sağlanmazsa, çıktı dosyası adı varsayılan olarak count_results.csv'dir).
Not: Komut satırında tırnak işaretleri gereklidir. Dosya yolundan önce tırnak işaretlerinden sonra boşluk olmadığından emin olun (bkz. Şekil 2C). - Bir sonraki komut satırı göründüğünde, işlem tamamlanmıştır. Konturlar ve sonuçlar elektronik tablosu artık adım 2.5'te çıktı dizini olarak belirtilen konumda mevcuttur (yani, yol/çıkış/çıktı; bkz. Şekil 2C).
- Sayım kalitesini doğrulamak için konturları görsel olarak inceleyin. Sonuçlar artık Bölüm 4'te açıklandığı gibi verileri normalleştirmek için kullanılabilir.
NOT: Konturlarda eksik çekirdekler var gibi görünüyorsa (örneğin, çekirdekler çok soluk görünüyor) veya çekirdek gruplarını tek bir öğede kümeliyorsa (örneğin, aşırı pozlanmış görüntü, çok fazla arka plan, artefaktlar), kullanıcı sorun giderme tablosuna başvurmalıdır ve Bölüm 4'e geçmeden önce görüntü alımını optimize etmek için Bölüm 1'e dönmesi gerekebilir.
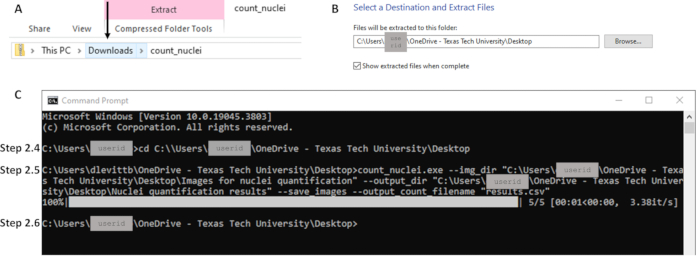
Şekil 2: Bölüm 2 için iş akışının önemli bölümlerine örnekler. (A) İndirilenler klasöründen dosyaları ayıklayın, adım 2.3'te istenen hedefe ayıklamayı (B) etkinleştirmek için okla gösterildiği gibi doğru klasöre tıklamaya dikkat edin. (C) Adım 2.4 ve 2.5 için komut örnekleri ve adım 2.6'da tamamlandığını gösteren bir sonraki komut satırı. Adım 2.4 (C)'deki dosya yolunun, adım 2.3 (B)'deki ayıklama konumuyla eşleştiğine dikkat edin. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
3. Otomatik çekirdek niceleme programını Python komut dosyası olarak çalıştırma
- Bilgisayarda Python'un kurulu olduğundan emin olun. Gerekirse Python'u python.org/downloads'dan (tercih edilen) veya Anaconda ya da Miniconda kullanarak yükleyin. Yayınlandığı tarih itibariyle, bu program Python 3.10-3.12 sürümleri ile test edilmiştir.
- Tarayıcıyı açın ve https://github.com/rbudnar/nuclei_counter gidin. Yeşil Kod düğmesine tıklayın, ardından kod deposunu indirmek için ZIP İndir'e tıklayın (bkz. Şekil 3A).
- Windows işletim sistemi için aşağıdaki talimatları kullanın. Mac OS tabanlı makineler için adım 3.4'e bakın.
- İndirilenler klasöründen, Dosya'ya sağ tıklayın ve dosyaları yerel bir bilgisayara ayıklamak için Tümünü Ayıkla'yı seçin (bkz. Şekil 3B-C).
- Ayıklanan dosya konumunda nuclei_counter-main adlı klasörü açın. Bu kod deposudur.
NOT: Bunun yalnızca bu program ilk kez kullanıldığında indirilmesi gerekir; Ayıklanan dosyaları erişilebilir bir ağ konumuna taşıyın ve bu konumu not edin. - Arama çubuğunda CMD veya Komut İstemi'ni arayarak bir komut istemi açın. Python, Anaconda veya Miniconda tarafından yüklendiyse, standart bir komut istemi yerine Anaconda İstemi'ni açın. Bu, arama çubuğuna Anaconda Prompt yazarak bulunabilir.
- Dizini kod deposu olarak değiştirmek için cd komutunu kullanın ve Enter tuşuna basın. Büyük/küçük harfe duyarlı olan aşağıdaki kod satırına bakın. Dosya yolunu kopyalarken nuclei_counter-main adlı klasörün açık olduğundan emin olun; pyproject veya pyproject.toml adlı dosya görünür olmalıdır (bkz. Şekil 3D). Örnek komut satırı: C:\Users\janedoe>cd [cd'den sonra boşluk, ardından dosya yolunu kopyala/yapıştır]
- Sonraki komut satırında, boşluk ve nokta dahil olmak üzere aşağıdakini yazın (bkz. Şekil 3D:
pip install -e .
NOT: Adım 3.3.5 ve 3.3.6'nın yalnızca bu program herhangi bir bilgisayarda ilk kez çalıştırıldığında tamamlanması gerekir. Bu zaten tamamlanmışsa, adım 3.3.7'ye geçin. Yükleme işleminin başlatılması birkaç dakika sürebilir. - Enter tuşuna basın. Bu komutu çalıştırmak, bu komut dosyasını çalıştırmak için gerekli kitaplıkları yükler ve kod ayarlamalarının gerekli olduğu durumlarda depoyu düzenlenebilir bir modda yükler (bkz. Şekil 3D).
- Dizini ana kaynak kodu dizinine değiştirmek için sonraki komut satırına aşağıdakileri yazın (bkz. Şekil 3E):
cd "nuclei_counter" - Sonraki komut satırına aşağıdakini yazın ve Enter tuşuna basın (bkz. Şekil 3E):
python count_nuclei.py --img_dir "yol/için/görüntüler" --output_dir "yol/çıkış" --save_images --output_count_filename "results.csv"
Burada path/to/images, analiz edilecek görüntüleri içeren klasörün dosya yoludur (adım 1.14); path/to/output, .csv dosyasının kaydedilmesi gereken klasörün dosya yoludur (sağlanmadığında varsayılan olarak geçerli çalışma dizinine geçer); ve results.csv, elde edilen sayımların kaydedileceği dosya adıdır (sağlanmazsa, çıktı dosyası adı varsayılan olarak count_results.csv'dir).
Not: Komut satırında tırnak işaretleri gereklidir. Dosya yolundan önce tırnak işaretlerinden sonra boşluk olmadığından emin olun. Python betiği, sağlanan dosya yolundaki dosyaları okuyacak ve dizindeki tüm görüntüler için çekirdek sayma programını yürütecektir (bkz. Şekil 3E). Elde edilen sayımlar, belirtilen konumdaki sonuç .csv dosyasına kaydedilecektir. - Bir sonraki komut satırı göründüğünde, işlem tamamlanmıştır (bkz. Şekil 3E). Konturlar ve sonuçlar elektronik tablosu artık adım 3.3.8'de çıktı dizini olarak belirtilen konumda (yani, yol/çıkış/çıktı) mevcuttur.
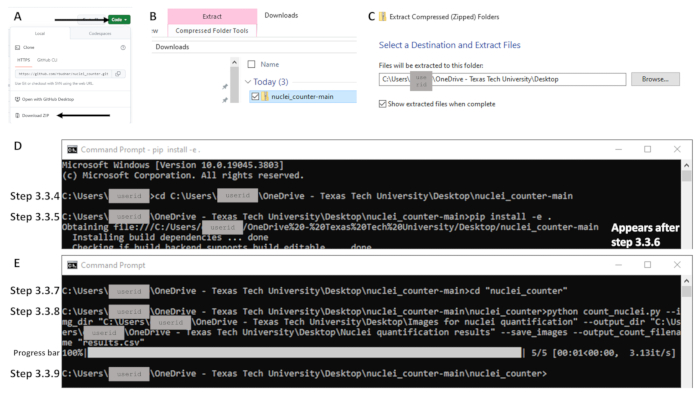
Şekil 3: Windows işletim sistemi kullanan bölüm 3 için iş akışının önemli bölümlerine örnekler. Koda erişmek için Kod etiketli yeşil düğmeye tıklayın ve (A) Zip dosyasını indirin (oklarla gösterilir). (B) Zip dosyasına sağ tıklayarak (mavi renkle gösterilir) ve (C) istenen hedefi ayarlayarak ayıklayın. (D) Komut istemini açtıktan sonra, 3.3.4 ve 3.3.5 adımları için komut örnekleri. Adım 3.3.5 komutunun tümünün tek bir satırda olduğunu unutmayın; enter tuşuna bastıktan sonra bir sonraki satırda "Dosya alınıyor" ile başlayan metin görünür (adım 3.3.6; yani, komutu çalıştırmak). (E) 3.3.7 ve 3.3.8 adımları için komut örnekleri, 3.3.8 adımının yürütülmesini takiben bir ilerleme çubuğu ve 3.3.9 adımının tamamlandığını gösteren komut satırı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- Mac OS için aşağıdaki yönergeleri kullanın.
- İndirilenler klasöründen, Dosya menüsüne tıklayın ve dosyaları yerel bilgisayara çıkarmak için Aç'ı seçin (bkz. Şekil 4A).
- Ayıklanan dosya konumunda nuclei_counter-main adlı klasörü açın. Bu kod deposudur.
NOT: Bunun yalnızca bu program ilk kez kullanıldığında indirilmesi gerekir; Ayıklanan dosyaları erişilebilir bir ağ konumuna taşıyın ve bu konumu not edin. - Spot ışığını açan command + boşluk çubuğuna basarak bir terminal açın; Ardından, "Terminal" i arayın.
- Dizini kod deposu olarak değiştirmek için cd komutunu kullanın ve Enter tuşuna basın. Büyük/küçük harfe duyarlı olan aşağıdaki kod satırına bakın. Dosya yolunu bir metin belgesine yazın ve terminale kopyalayın. Dosya yolunun cd'den hemen önceki metinle başladığından emin olun (örneğin, Şekil 4B'de /Users/userid). Örnek komut satırı: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- Bir sonraki komut satırında, Kullanıcı Kimliği'nden sonra bir dolar işareti görünecektir (bkz. Şekil 4B). Dolar işaretinden sonra bir boşluk olduğundan emin olun ve komutun sonundaki boşluk ve nokta da dahil olmak üzere aşağıdakini yazın. Pip'ten hemen sonra boşluk bırakmadan uygun Python sürümünü ekleyin: pip3 install -e .
NOT: Adım 3.4.5 ve 3.4.6'nın yalnızca bu program herhangi bir bilgisayarda ilk kez çalıştırıldığında tamamlanması gerekir. Bu işlem zaten tamamlanmışsa, adım 3.4.7'ye geçin. Yükleme işleminin başlatılması birkaç dakika sürebilir. - Enter tuşuna basın. Bu komutu çalıştırmak, bu komut dosyasını çalıştırmak için gerekli kitaplıkları yükler ve olay kodu ayarlamalarının gerekli olduğu durumlarda depoyu düzenlenebilir bir modda yükler.
- Bir sonraki komut satırında, dolar işaretinden sonra bir boşluk olduğundan emin olun, ardından dizini ana kaynak kodu dizinine değiştirmek için aşağıdakileri yazın (bkz. Şekil 4C):
cd "nuclei_counter" - Sonraki komut satırında, aşağıdaki komut satırını yazın. 3 dışında bir sürüm kullanıyorsanız Python'dan sonraki numarayı doğru sürüm numarasıyla değiştirin. Ardından Enter tuşuna basın (bkz. Şekil 4C):
python3 count_nuclei.py --img_dir "yol/için/görüntüler" --output_dir " yol/çıkış" --save_images --output_count_filename "results.csv"
burada path/to/images, analiz edilecek görüntüleri içeren klasörün dosya yoludur (adım 1.14); path/to/output, .csv dosyasının kaydedilmesi gereken klasörün dosya yoludur (sağlanmadığında varsayılan olarak geçerli çalışma dizinine geçer); ve results.csv, elde edilen sayımların kaydedileceği dosya adıdır (sağlanmazsa, çıktı dosyası adı varsayılan olarak count_results.csv'dir).
Not: Komut satırında tırnak işaretleri gereklidir. Dosya yolundan önce tırnak işaretlerinden sonra boşluk olmadığından emin olun. Python betiği, sağlanan dosya yolundaki dosyaları okuyacak ve dizindeki tüm görüntüler için çekirdek sayma programını yürütecektir (bkz. Şekil 4C). Elde edilen sayımlar, belirtilen konumdaki sonuç .csv dosyasına kaydedilecektir. - Bir sonraki komut satırı göründüğünde, işlem tamamlanmıştır (bkz. Şekil 4C). Konturlar ve sonuç elektronik tablosu artık adım 3.4.8'de çıktı dizini olarak belirtilen konumda (yani, yol/çıkış/çıktı) mevcuttur.
- Sayım kalitesini doğrulamak için konturları görsel olarak inceleyin. Sonuçlar artık Bölüm 4'te açıklandığı gibi verileri normalleştirmek için kullanılabilir.
NOT: Konturlarda eksik çekirdekler var gibi görünüyorsa (örneğin, çekirdekler çok soluk görünüyor) veya çekirdek gruplarını tek bir öğede kümeliyorsa (örneğin, aşırı pozlanmış görüntü, çok fazla arka plan, artefaktlar), kullanıcı Tablo 1'e başvurmalıdır ve Bölüm 4'e geçmeden önce görüntü alımını optimize etmek için Bölüm 1'e dönmesi gerekebilir.
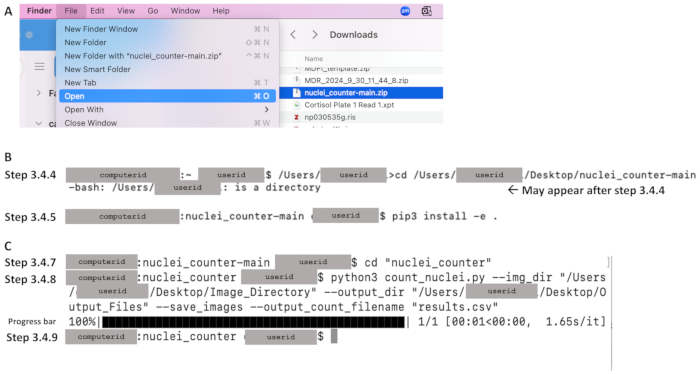
Şekil 4: Mac OS kullanarak bölüm 3 için iş akışının önemli bölümlerine örnekler. Kodu indirdikten sonra (bkz. Şekil 3A), (A) Zip dosyasına (mavi renkle gösterilir) tıklayarak ve istenen hedefi ayarlayarak ayıklayın. (B) Terminali açtıktan sonra, 3.4.4 (dizini kod deposuna değiştirme) ve 3.4.5 (pip yükleme) adımları için komut örnekleri. (C) 3.4.7 ve 3.4.8 adımları için komut örnekleri, 3.4.8 adımının yürütülmesini takiben bir ilerleme çubuğu ve 3.4.9 adımının tamamlandığını gösteren komut satırı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
4. Deneysel verileri normalleştirmek için sayımları kullanma
- Her görüntüyü otomatik program kullanılarak belirlenen çekirdek nicelemesi ile bağlayan results.csv dosyasını açın.
- Normalleştirme yöntemine karar verin: 1) görüntü bazında (örneğin, çok renkli floresan mikroskobu ise), 2) verileri, numune başına ortalama çekirdek sayısının bir referans numuneye (veya duruma) oranı olarak normalleştirin, 3) numune başına çekirdek sayısına normalleştirin (örneğin, toplam çekirdek sayısını numune alanına göre tahmin edin). Normalleştirme yöntemi için öneriler Tartışma bölümündedir.
- Normalleştirme görüntü bazında gerçekleştirilecekse, her görüntü için deneysel verileri o görüntü için çekirdek sayısına bölmeniz yeterlidir.
- Verileri oran olarak normalleştirmek için aşağıda açıklanan adımları izleyin.
- Tek bir kopya için birden fazla görüntünün toplandığı herhangi bir örnek için (örneğin, 6 oyuklu bir plakada tek bir kuyucuktan yakalanan 10-15 görüntü), görüntü başına ortalama çekirdek sayısını hesaplayın. Bu çekirdek sayımları, tek çekirdekli hücreler veya çok çekirdekli hücrelerde (miyotüpler gibi) birbirine kaynaşmış bir dizi hücre varsayılarak hücre sayısı olarak kabul edilir.
- Bir referans numunesi veya bir referans koşulu seçin (örneğin, kontrol koşulundaki tüm numunelerdeki ortalama sayım) ve sayımı not edin.
- Tüm örneklerin sayısını bu referans sayısına bölün ve bir normalleştirme değeri elde edin. Bir referans numune kullanılmışsa, referans numune için normalleştirme değerini 1 olarak kullanın.
NOT: Bir referans koşulu kullanılmışsa, koşul genelinde ortalama normalleştirme değeri artık 1 olacaktır. Referanstan daha az çekirdeğe sahip numuneler (yani daha az yoğun) 1'den küçük normalizasyon değerlerine sahip olacaktır ve referanstan daha fazla çekirdeğe sahip numuneler (yani daha yoğun) 1'den büyük normalizasyon değerlerine sahip olacaktır. - Her bir kuyucuk için deneysel verileri karşılık gelen hesaplanan normalizasyon değerine bölün.
- Verileri numune başına çekirdek sayısına normalleştirmek için aşağıda açıklanan adımları izleyin.
- Tek bir kopya için birden fazla görüntünün toplandığı herhangi bir örnek için (örneğin, 6 oyuklu bir plakada tek bir kuyucuktan yakalanan 10-15 görüntü), görüntü başına ortalama çekirdek sayısını hesaplayın. Bu çekirdek sayımları, tek çekirdekli hücreler veya çok çekirdekli hücrelerde (miyotüpler gibi) birbirine kaynaşmış hücre sayısı varsayılarak hücre sayısı olarak kabul edilir.
- Verilerin türetildiği slaytın (lamelli), kuyunun veya çanağın yüzey alanını belirleyin. Bu bilgiler, ürün web sitelerindeki özellikler bölümünde bulunabilir.
- Yakalanan her görüntünün alanını belirleyin. Bu bilgiler genellikle görüntüleme veya analiz yazılımında meta veri olarak bulunur.
- Numune yüzey alanının ve görüntü alanının aynı birimlerle ifade edildiğinden emin olun (örn. mm2). Değilse, istediğiniz birime dönüştürün.
- Aşağıdaki denklemi kullanarak her örnek için toplam sayıyı hesaplayın:
TC = AC • (SA/IA)
burada TC toplam sayım, AC ortalama sayı, SA yüzey alanı, IA görüntü alanıdır. - Deneysel verileri, numune için karşılık gelen TC'ye bölün.
- Deneysel verileri normalleştirin ve uygun şekilde istatistiksel analizler için kullanın.
Sonuçlar
Her toplu görüntü çalıştırması şunları üretir: 1) tanımlanan çekirdeklerin ana hatlarını gösteren konturların uygulandığı bir dizi görüntü dosyası (Şekil 5) ve 2) görüntü dosyası adlarını ve ilişkili sayıları birbirine bağlayan bir .csv dosyası (elektronik tablo). Konturları görüntülemek, kullanıcının sayım kalitesini görsel olarak değerlendirmesine olanak tanır. Spesifik olarak, bölüm 1'e göre elde edilen görüntüler, çekirdeğin program tarafından sayıldığını gösteren düz yeşil bir çizgi ile çevrili tüm (veya neredeyse tüm) çekirdeklere sahip olmalıdır. Bu konturlar burada programı ayarlamak için kullanıldı.
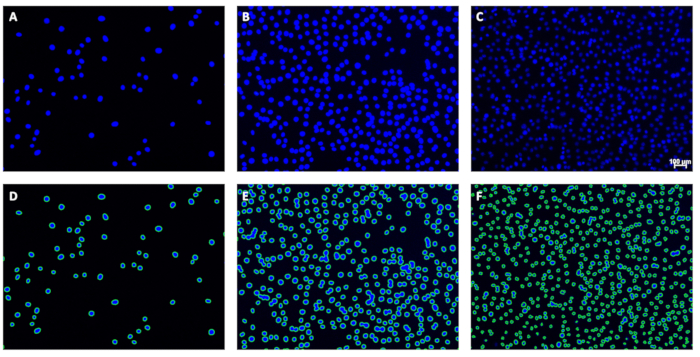
Şekil 5: Orijinal görüntü örnekleri ve karşılık gelen konturlar. (A-C) Analizden önce orijinal görüntülerin örnekleri ve analizden sonra (DF) karşılık gelen konturlar hemen aşağıdadır. Ölçek çubuğu (C) 100 μm'yi gösterir; Çekirdek ölçümü için kullanılan görüntülerden ölçek çubukları çıkarılmalıdır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
İlk sonuçları kullanarak doğrulama
Değişen hücre yoğunluklarındaki C2C12 miyoblastları metanolle sabitlendi, montaj ortamında DAPI ile karşı boyandı ve .jpg dosya formatında 10x büyütmede 120 görüntü elde edildi. Otomatik program, Görüntü Dizini bu görüntüleri içeren klasöre ayarlanmış olarak çalıştırıldı ve elde edilen .csv dosyası kaydedildi. Ayrı olarak, her görüntü, otomatik sayımlara ve birbirlerinin sayımlarına kör olan iki eğitimli araştırmacı tarafından manuel olarak ölçüldü. Ham görüntüler, otomatik program tarafından oluşturulan konturlar ve manuel ve otomatik sayımları içeren bir sonuç elektronik tablosu Açık Bilim Çerçevesi (OSF) deposunda mevcuttur (bölüm 1'deki bağlantıya bakın). Sınıf içi korelasyon katsayıları (ICC'ler; ortalama ölçümler), 1) iki manuel sayım ve 2) otomatik program kullanılarak elde edilen iki manuel sayım ve sayımların ortalaması arasındaki değerlendiriciler arası güvenilirliği belirlemek için SPSS Statistics kullanılarak hesaplanmıştır. Floresan mikroskobu 16,17,18 kullanılarak elde edilen görüntülerdeki nesneleri ölçmek için tasarlanmış diğer programları doğrulamak için benzer yaklaşımlar kullanılmıştır. ICC'ler ayrıca, tüm veri setinin değerlendirilmesinin hücre yoğunluğuna dayalı güvenilirlikteki herhangi bir potansiyel değişikliği maskelemediğinden emin olmak için her çeyrek için ortalama manuel sayımlar ve otomatik sayımlar arasında hesaplandı. Çeyrekler, ortalama manuel sayıma dayalı olarak 25., 50. ve 75. yüzdelik kesme noktaları kullanılarak tanımlandı ve çeyrek 1 (Q1; en düşük hücre yoğunluğu) ile Q4 (en yüksek hücre yoğunluğu) arasında değişti. ATÇ'ler ortalama ölçümlere dayalı olarak raporlanır ve aşağıdaki tanımlara göre yorumlanır: zayıf (ATK <0,50), orta (0,50 ≤EKA <0,75), iyi (0,75 ≤EKA <0,90) veya mükemmel (EKS ≥0,90)19.
Görüntü başına çekirdek, manuel sayım ortalamalarına göre 44,5 ile 1160 arasında değişiyordu. İki manuel sayım arasında değerlendirici güvenilirliği mükemmeldi (ICC > 0.999, p < 0.0001; Şekil 6A) ve ortalama manuel ve otomatik sayımlar arasında (ICC = 0.993, p < 0.0001; Şekil 6B). Güvenilirlik çeyrekler arasında mükemmel kaldı (Q1: ICC = 0.996, p < 0.0001; 2. Çeyrek: ICC= 0.997, p < 0.0001; Q3: ICC = 0.998, p < 0.0001; 4. Çeyrek: ICC = 0.986, p < 0.0001; Şekil 6C-F). Şekil 6B'deki en uygun çizgiden uzak olan tek veri noktası, Şekil 7'de gösterilen görüntüyü temsil eder. Görüntü, bilgisayarla görme programının her bir çekirdeği tanımasını engelleyen, birlikte gruplandırılmış birden çok çekirdeğin birkaç alanına sahiptir. Bu sorun, daha kısa bir maruz kalma süresi kullanılarak düzeltilebilir. Tablo 1'de böyle bir görüntünün nasıl işleneceği açıklanmaktadır.
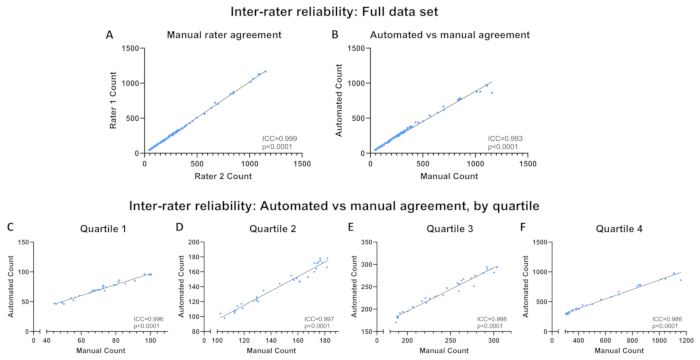
Şekil 6: Değerlendiriciler arası güvenilirlik. (A) manuel değerlendirici sayımları arasında ve (B) otomatik sayımlar ile ortalama manuel sayımlar arasında tam veri kümesinde değerlendiriciler arası güvenilirlik. (C-F) Otomatik sayımlar ve çeyreklere göre ortalama manuel sayımlar arasında değerlendiriciler arası güvenilirlik. Güvenirlik sınıf içi korelasyon katsayısı (ICC) olarak ifade edilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 7: Doğru çekirdek nicelemesi için iki engeli olan bir görüntü örneği. (A) Orijinal görüntü ve (B) ilişkili konturlar. Ok 1, birden fazla çekirdeğin bir araya toplandığı bir alanı gösterir ve yuvarlak nükleer şekli, birden fazla çekirdeğin yazılım tarafından algılanmasını engelleyecek kadar bozar. Bu, bu görüntü boyunca çeşitli yerlerde meydana gelir. Ok 2, birden fazla çekirdeğin arkasında görünen soluk bir haleyi gösterir; Bu hale, yazılımın onu bir çekirdek olarak içermesi için yeterince parlak göründü ve böylece çevrelediği çoklu çekirdeğin sayısını kaybetti. Bu görüntü, Şekil 6B'deki en uygun çizgiden çok uzaktaki tek veri noktasını temsil eder ve bu engellerle ilgili olası sorunları vurgular. Ölçek çubuğu (A) 100 μm'yi gösterir; Çekirdek ölçümü için kullanılan görüntülerden ölçek çubukları çıkarılmalıdır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Çekirdek niceleme programımızın mevcut seçeneklere göre çeşitli avantajları vardır: yalnızca minimum teknolojik beceri gerektirir, çekirdek nicelemesinin özel görevi için doğrulanmıştır ve açık kaynaklıdır; ikincisi, maliyetle ilgili engellerin üstesinden gelir. Sonuç olarak, bu program, hücre ve moleküler biyologlara, floresan mikroskobu kullanılarak yakalanan görüntülerdeki çekirdekleri hızlı ve doğru bir şekilde ölçmek için ek bir seçenek sunar. Şu anda mevcut olan otomatik çekirdek veya hücre sayma programları tüm kullanıcılar tarafından tercih edilmez, bazıları maliyet açısından engelleyicidir ve belirli uygulamalar için doğrulama minimum düzeyde olabilir veya hiç olmayabilir. BioTek Cytation hattı gibi özel ekipmanlara, çok çeşitli ek uygulamalara ek olarak nükleer boyama20 kullanarak yerinde hücre sayımını doğrulayan teknik bültenler eşlik eder. Doğrulama verileri mevcut olsa da ve Cytation ürün serisi, özellikle canlı hücre verilerini kolay ve verimli bir şekilde normalleştirmek için Seahorse Hücre Dışı Akı Analizörleri ile arayüzlendiğinde yaygın olarak kullanılsa da, bu ürün, sınırlı ekipman finansmanı olan laboratuvarlar için maliyet açısından engelleyici olabilir. Örneğin, floresan mikroskobu için dört objektif ve dört filtre küpü ile donatılmış temel modelin liste fiyatı 2024 takvim yılında 100.000 ABD dolarını aştı. Özel ekipman satın alma imkânı olmayanlar için açık kaynaklı yazılım, görüntü analizi için ücretsiz alternatifler sunar. Çekirdek ve hücre ölçümü için yararlı olan açık kaynaklı yazılım, ImageJ'yi (Ulusal Sağlık Enstitüleri, Bethesda, MD, ABD; 21. ImageJ platformunu kullanarak görüntü işleme otomasyonu için makro komut dosyaları yazmak isteyenler için eklentiler artık mevcut. Tüm hücre görüntüleri ile faz kontrastı veya parlak alan mikroskobu kullanan ImageJ tabanlı hücre sayımı otomasyonu daha önce tanımlanmıştı 22,23. CellProfiler, floresan boyalar veya standart histolojik lekelerle lekelenmiş hücre görüntülerinin analizi için yaygın olarak yayınlanan bir uygulamadır ve bir dizi biyolojik uygulamada faydasını açıklayan erken bir makale genellikle doğrulama24 olarak anılır. Bununla birlikte, bu el yazması herhangi bir özel biyolojik bulguyu doğrulamaz, bu da çekirdek sayımı25 gibi kullanılan çeşitli otomasyon boru hatları için ek doğrulama ve şeffaflık gerektirir. Ayrıca, CellProfiler, hücre analizi için verimli bir yazılım paketi olsa da, tüm bilim adamları onu kullanıcı dostu bulmuyor. Bu nedenle, açık kaynaklı yazılım kullanarak çekirdekleri ölçmek için alternatif bir otomatik program geliştirmeyi ve doğrulamayı amaçladık. Özellikle, bu iş akışını geliştirmek için Python'daki mevcut görüntü işleme kitaplıklarını kullandık.
Bu yöntem, son kullanıcının DAPI26 gibi DNA boyaları kullanılarak boyandığı hücreler veya dokularla başlar. Görüntüleri otomasyon yazılımıyla uyumlu bir şekilde yakalamak ve kaydetmek için genel yönergeler sağlanmıştır. Ayrıntılar farklı mikroskoplara ve görüntüleme yazılımlarına göre farklılık gösterebilse de, ölçülebilir görüntüler elde etmek için bölüm 1 için genel iş akışı aynıdır. Canlı numuneler kullanılıyorsa, uygun kişisel koruyucu ekipmanın giyildiğinden ve görüntüleme ortamının bu numuneler için uygun olduğundan emin olmak için özen gösterilmelidir (örn. sıcaklık,CO2 yüzdesi, tampon ilavesi, vb.)27. Bu protokol yalnızca DNA boyalarının görüntülenmesini tanımlarken, kullanıcılar birleştirme amacıyla aynı alanın diğer renkleriniyakalayabilir 15 ve nükleer görüntüleri miktar belirleme programıyla kullanmak üzere ayrı bir klasöre kopyalayabilir. Görüntüleme, canlı numuneler için uygun KKD ile karanlık bir odada yapılmalı ve gerektiğinde uygun dezenfeksiyon kullanılmalıdır (örn.% 70 etanol ile). Işıklar açılmadan önce lekeli numuneler ışıktan korunmalıdır. Kaliteli görüntü yakalama, doğru çekirdek ölçümü için kritik öneme sahiptir. Uygun pozlama süresine ve parlak çekirdekler ile siyah bir arka plan arasında güçlü kontrasta sahip görüntülerde bile, hücre gruplarının arkasında aşırı kümelenmiş hücrelerin veya halelerin varlığı (bkz. Şekil 7) niceleme doğruluğunu azaltabilir. Bu durumlarda, sinyallerin yoğunluğunu azaltmak, çekirdek sinyallerinin minimum düzeyde birleşmesini ve halelerin en aza indirilmesini sağlarken, çekirdeklerin yuvarlak şekillerini tespit etmek için yeterince güçlü bir sinyali korumak için maruz kalma sürelerinin değiştirilmesi gerekebilir. Bu adımlar yetersizse, numunenin bitişik bir bölümünün görüntülenmesi gerekebilir. Tek bir deney için tüm görüntüler aynı büyütmede yakalanmalı ve kullanılan hedefe karşılık gelen büyütme, görüntüleme yazılımında uygun şekilde ayarlanmalıdır. Görüntüleme için kullanılan hedefin, çekirdek sayısı görüntülenen alandan tüm kuyu (veya slayt) alanına tahmin edileceğinde, görüntüleme yazılımındaki ayarlarla eşleşmesi özellikle önemlidir. Bu adım, hedef her değiştirildiğinde gerçekleşmelidir, ancak mikroskop ve yazılım kurulurken kolayca gözden kaçabilir. Bölüm 1 için gereken süre, bir dizi faktöre bağlı olarak değişir (örneğin, floresan mikroskobu ile kullanıcı deneyimi, görüntü ve numune sayısı, mikroskop vb.). İki adet 6 oyuklu plakada oyuk başına 10-15 lekeli çekirdek görüntüsü yakalamak için, kullanıcı yaklaşık 1-2 saat zaman ayırmalıdır.
Görüntüler yakalandıktan ve özel bir klasöre kaydedildikten sonra, nicelleştirilmeye hazırdırlar. Niceleme için en basit yöntem, Python veya herhangi bir kitaplık yüklemeye gerek kalmadan bu önceden paketlenmiş otomatik programı çalıştırmaktır (bölüm 2). Programın çalıştırılabilir olarak hazırlandığı ve Pyinstaller kullanılarak paketlendiği unutulmamalıdır. Pyinstaller kullanılarak oluşturulan yazılımlar genellikle virüsten koruma yazılımı tarafından işaretlenir. Virüsten koruma yazılımı dosyanın açılmasını engelliyorsa, kullanıcının devam etmek için dosyayı güvenli olarak işaretlemesi gerekir. Bu, bölüm 2'yi başarılı bir şekilde gerçekleştirmek için kritik öneme sahiptir. Alternatif olarak, herhangi bir kullanıcı, istenirse otomatik niceleme programını yürütülebilir dosya yerine bir Python betiği (bölüm 3) olarak çalıştırmayı seçebilir. Kullanıcı programı bir Mac veya Linux işletim sistemi kullanarak çalıştırmak isterse, programın Python üzerinden çalıştırılması gerekir. Bu yöntem Pyinstaller gerektirmez ancak teknik olarak daha zor olabilir. Bölüm 3'te ayrıntılı olarak açıklanan örneklerle birlikte adım adım protokol, bu programı bir komut dosyası olarak çalıştırmanın önündeki teknik engelleri kaldırmayı amaçlamaktadır. Programı çalıştırmak için bölüm 2 veya bölüm 3 kullanılıp kullanılmadığı, kodun komut istemlerine doğru şekilde girildiğinden emin olmak çok önemlidir. Protokol boyunca örnek kod satırları dahil edilmiştir ve kullanıcı boşluklara, tırnak işaretlerine ve diğer noktalama işaretlerine çok dikkat etmelidir. Kullanıcı ayrıca dosya yollarının doğru olduğundan emin olmalıdır. Görüntüler ve çıktılar için dosya yollarının bir Word belgesinde bulunması yararlı olur. Komutlar ayrıca bu Word belgesine yazılabilir ve gerektiğinde kopyalanıp yapıştırılabilir.
Bölüm 4'te açıklandığı gibi, bu otomatik çekirdek niceleme programından elde edilen sayıları kullanarak deneysel verileri normalleştirmek için birden fazla yaklaşım vardır. Deneysel verilerin her görüntüde bir dizi olay içerdiği ve bir oranın istendiği görüntü tabanlı deneyler için (örneğin, eklin V-pozitif hücrelerin oranı28, ACE2 reseptörlerini eksprese eden hücrelerin oranı29, lekeli parçacıkları alan hücre sayısı30, vb.), her görüntü için deneysel veriler basitçe elde edilen sayıya bölünebilir (0 ile 1 arasında bir oran ile sonuçlanır). Ardından, aynı slayttan, tabaktan veya kuyudan çoğaltılan görüntüler arasında ortalama oran hesaplanabilir. Bir yüzde isteniyorsa, son oranları 100 ile çarpmanız yeterlidir. Son olarak, bu ortalama oranlar veya yüzdeler, aşağı yönlü istatistiksel analizlerde kullanılabilir. Normalleştirilecek deneysel veriler, canlı hücrelerdeki fonksiyonel deneylerden (örneğin, oksijen tüketim oranları 6,11,12) veya hücre kültürü süpernatantından (örneğin, süpernatant31'deki laktat dehidrojenaz aktivitesi veya spesifik bir salgılanan sitokin32 konsantrasyonu, vb.) olduğunda, çekirdek sayımlarının ortalaması alınmalıdır. Daha sonra, çekirdek sayımı, lamın (lamel ), kuyunun veya tabağın yüzey alanına tahmin edilebilir veya sayım, bir referans numuneye veya koşula karşı bir oran olarak ifade edilebilir. Slaytın, kuyunun veya çanağın yüzey alanına ekstrapolasyon, verilerin türetildiği hücre sayısına (örneğin, 103 hücre başına dakikada tüketilen oksijen) karşı deneysel verileri ifade etmek için yararlıdır. Bu yöntemi kullanmak için, slaytın (lamel ), kuyunun veya çanak yüzeyin ve her görüntüde yakalanan alanın bilinmesi gerekir. Numune mevcut tüm yüzey alanını kaplamıyorsa, deneysel veriler az miktarda süpernatanttan geliyorsa (yani, birim hacim başına aktivite veya konsantrasyon olarak ifade edilir) veya görüntü ve/veya yüzey alanı bilinmiyorsa, o zaman en iyi seçenek, tek bir numune için tüm görüntülerdeki çekirdek sayılarının ortalamasını almak ve çekirdek sayısını bir referans numuneye veya koşula göre ifade etmektir. Elde edilen değer, çekirdek yoğunluğu veya hücre yoğunluğu (tek çekirdekli hücreler kullanılıyorsa) için deneysel verileri düzeltmek için kullanılır.
Bu yöntemin yürütülmesi basit olsa da, hangi kullanıcıların bilmesi gerektiği konusunda birkaç olası sorun vardır. Bu olası sorunlar, olası nedenler ve sorunları çözmek için sorun giderme adımlarıyla birlikte Tablo 1'de listelenmiştir. Burada açıklanan program, mevcut yazılıma kıyasla önemli avantajlara sahiptir; Bununla birlikte, birkaç sınırlama kabul edilmelidir. Ana dezavantaj, niceleme programının parlak alan görüntüleriyle değil, yalnızca floresan mikroskobu kullanılarak yakalanan görüntülerle kullanılabilmesidir. Programı oluşturmak için 10x büyütmede yakalanan DAPI lekeli görüntüler kullanılırken, program renk veya boyuttan ziyade çekirdeğin morfolojisini tanımak için oluşturulduğundan, program diğer DNA boyaları ve büyütmeleri ile birlikte kullanılabilir. Programın doğruluğunu çeşitli hücre yoğunlukları içeren görüntülerde doğrulamış olsak da, son kullanıcı aralığın dışında kalan hücre yoğunluklarına sahip olabilir. Bu durumda, kaliteli görüntülerin elde edilmesi koşuluyla yine de doğru sayımlar bekliyoruz; Bununla birlikte, kullanıcı doğruluğu sağlamak için konturları ve çıktıyı dikkatlice kontrol etmelidir. Hücre kültürü uygulamaları için, görüntülerdeki çekirdeklerin miktar tayini, tek katmanlı hücreler için en uygun olanıdır. Kültürlenmiş hücreler üst üste gelirse, çekirdekler maskelenebilir ve bu da çekirdek miktarının hafife alınmasına neden olur. Görüntülerdeki çekirdeklerin nicelleştirilmesinin iki boyutlu doğası, süspansiyon, kalın doku blokları veya diğer üç boyutlu uygulamalardaki hücrelerden toplam çekirdek sayısını belirlemek için bu programın kullanılmasını engeller. Bununla birlikte, bu program, kalın doku blokları gibi üç boyutlu modellerden yakalanan iki boyutlu görüntülerde floresan DNA boyaları ile etiketlenmiş çekirdekleri hala ölçebilir. İstenen uygulama için görüntülerdeki çekirdeklerin miktarının belirlenmesinin faydası, son kullanıcı tarafından belirlenmelidir.
| Sorun | Olası nedenler | Muhtemel çözümler |
| Görüntülerde aşırı arka plan | Maruz kalma süresi çok yüksek | Maruz kalma süresini azaltın |
| Çekirdekler görüntülerde birbirleriyle birleşiyor gibi görünüyor | Floresan DNA boya konsantrasyonu çok yüksek veya çok fazla DNA boya montaj ortamı | Optimizasyon sırasında boya konsantrasyonunu azaltın; Mümkün olan en az miktarda montaj ortamı kullanın |
| Çekirdekler çok hafif görünüyor veya bazı çekirdekler görünmüyor | Maruz kalma süresi çok düşük | Pozlama süresini artırın |
| Foto beyazlatma (foto ağartma | Lekeli numunelerin karanlıkta saklanmasını sağlayarak ve numunelerin aydınlatmaya maruz kalmasını en aza indirerek (örneğin, gerekli minimum maruz kalma süresini kullanarak ve bir görüntü çekildikten sonra veya not alırken olduğu gibi gerekmediğinde aydınlatmayı kapatarak) foto ağartmayı önleyin | |
| Halo efektleri veya çekirdeğe özgü olmayan şekillerin ortaya çıkması | Maruz kalma süresi çok yüksek | Maruz kalma süresini azaltın |
| Canlı hücrelerde veya sabit hücrelere yapışan hassas görev sileceklerinden veya bazı tezgah üstü koruyuculardan gelen tiftik | Tüy bırakmayan bir ortam sağlayın: numuneler maruz kaldığında hassas görev sileceklerinin kullanımını en aza indirin (örneğin, canlı hücreler için biyogüvenlik kabininde, sabit hücrelerin sabitlenmesi ve kurutulması sırasında veya boyama işlemi sırasında); Tezgah üstü koruyucuların boyama sırasında slaytlar veya plakalar üzerinde tüy bırakmadığından emin olun | |
| Sabit hücrelerin / dokuların yüzeyindeki diğer anormallikler | Numuneleri son derece dikkatli kullanın ve sabitleme ve boyama sırasında uygun önlemleri ve en iyi uygulamaları izleyin | |
| Işık yolundaki bozukluklar | Mikroskobun tüm parçalarının temiz olduğundan ve tüy ve toz içermediğinden emin olun; Görüntülemeden önce çevresel tozu ve tüyleri gidermek için slaytların/tabakların/tabakların dışını %70 etanol ile temizleyin | |
| Yürütülebilir dosya açılmıyor | Virüsten koruma yazılımı dosyayı engelledi | Dosyayı güvenli olarak işaretleme |
| Komut istemlerindeki hatalar | Komut satırındaki boşlukla ilgili sorunlar | Komutun başlangıcının komut satırındaki ">" sembolünden hemen sonra boşluk olmadan yazıldığını, dosya yolları için açık tırnak işaretlerinden sonra boşluk bulunmadığını ve verilen kod ve örneklerle boşluk ve eşleşme olduğunu bir kez daha kontrol edin. |
| Komut satırında kullanılan uygunsuz durumlar | Kod büyük/küçük harfe duyarlıdır; Uygun şekilde büyük ve küçük harfler kullandığınızdan emin olun | |
| Dosya yolu ile ilgili sorunlar | Dosya yolunun ilişkili dosya klasörünün içini gösterdiğinden emin olun (yani, dosya yolu kopyalandığında dosya klasörü açıktır). | |
| Yukarıdaki sorun giderme adımları kullanılarak çözülmeyen programla ilgili sorunlar | Yukarıda belirtilen yazılım veya komut satırı sorunları. | GitHub aracılığıyla bir sorun gönderin. https://github.com/rbudnar/nuclei_counter'a gidin. Üst kısımdaki menü çubuğunda "Sorunlar"ı seçin ("Kod"un sağındaki ikinci seçenek). Göndermek için sağ taraftaki "Yeni Sayı" yazan yeşil düğmeyi tıklayın. Sorunu göndermeden önce GitHub'da oturum açmanız için bir istem olacaktır. Gerekirse ücretsiz bir hesap oluşturma seçeneği de olacaktır. |
Tablo 1: Kaliteli görüntüler yakalamak ve otomatik çekirdek niceleme iş akışını çalıştırmak için sorun giderme.
Genel olarak, burada açıklanan yöntemler, bilgisayarla görme kitaplıklarını kullanarak geliştirdiğimiz ve birden fazla değerlendiriciden gelen manuel sayımlara göre doğruladığımız otomatik çekirdek niceleme programını kullanmak için adım adım ayrıntılar sağlar. Bu program, kodlamayla ilgili çeşitli düzeylerde teknolojik becerilere sahip bilim adamları tarafından kullanılabilirliği sağlamak için geliştirilmiştir, ücretsiz olarak kullanılabilir ve özellikle çekirdek niceleme görevi için doğrulanmıştır. Manuel sayımla karşılaştırıldığında, bu program son kullanıcıya değerli zaman kazandıracak ve veri normalizasyonu için doğru ve güvenilir çekirdek sayımları sağlayacaktır.
Açıklamalar
Yazarlar herhangi bir çıkar çatışması beyan etmemektedir.
Teşekkürler
Bu çalışmanın finansmanı Ulusal Sağlık Enstitüleri / Ulusal Yaşlanma Enstitüsü (R01AG084597; DEL ve HYL) ve Texas Tech Üniversitesi'nden (DEL) başlangıç fonları ile. Yazarlar, bu çalışmaya katkıda bulunan lisans araştırmacılarına (REH, MRD, CJM, AKW) finansal destek sağladıkları için Texas Tech University Undergraduate Research Scholars ve TrUE Scholars programlarına teşekkür eder. Ayrıca Dr. Lauren S. Gollahon ve Michael P. Bassett'e laboratuvar alanlarını ve ekipmanlarını nezaketle paylaştıkları için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
Referanslar
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır