Method Article
Использование библиотек компьютерного зрения для упрощения количественного определения ядер
В этой статье
Резюме
В этой статье описываются пошаговые методы автоматизации количественного определения ядер на основе изображений с помощью исполняемой программы с открытым исходным кодом, проверенной для диапазона плотности клеток. Эта программа предоставляет альтернативу, которая устраняет барьеры, связанные со стоимостью, доступностью для пользователей с ограниченными технологическими навыками и проверкой специфичных приложений, которая может ограничивать полезность существующих технологий.
Аннотация
Анализ живых клеток и анализ клеток на основе изображений требуют нормализации данных для точной интерпретации. Обычно используемым методом является окрашивание и количественное определение ядер с последующей нормализацией данных до количества ядер. Это количество ядер часто выражается в виде количества клеток для одноядерных клеток. В то время как ручная количественная оценка может быть трудоемкой и отнимать много времени, доступные автоматизированные методы могут быть предпочтительными не для всех пользователей, могут не иметь проверки для этого конкретного приложения или могут быть непомерно дорогими. Здесь мы предоставляем пошаговые инструкции по получению количественных изображений ядер, окрашенных флуоресцентными красителями ДНК, и последующей количественной оценке ядер с помощью автоматизированного программного обеспечения для подсчета объектов, разработанного с использованием библиотек компьютерного зрения Python. Мы также проверяем эту программу в диапазоне плотностей клеток. Хотя точное время выполнения программы зависит от количества изображений и компьютерного оборудования, эта программа объединяет часы работы, подсчитывая ядра в секунды для выполнения программы. Хотя этот протокол был разработан с использованием изображений неподвижных, окрашенных клеток, изображения окрашенных ядер в живых клетках и иммунофлуоресцентные приложения также могут быть количественно оценены с помощью этой программы. В конечном счете, эта программа предоставляет вариант, который не требует высокой степени технологических навыков и является проверенной альтернативой с открытым исходным кодом, помогающей клеточным и молекулярным биологам оптимизировать свои рабочие процессы, автоматизируя утомительную и трудоемкую задачу количественного определения ядер.
Введение
Функциональные эксперименты и эксперименты на основе изображений имеют решающее значение для понимания влияния экспериментальных методов лечения на биохимию и физиологию всей клетки. Корректная интерпретация данных экспериментов по клеточной биологии зависит от точности и воспроизводимости протокола эксперимента, включая нормализацию данных. Например, анализ потребления кислорода и скорости внеклеточного закисления в живых клетках на исходном уровне и после лечения специфическими препаратами позволяет оценить различные аспекты энергетического обмена 1,2. Измерение активности ферментов, таких как лактатдегидрогеназа, в надосадочной жидкости клеточных культур может помочь в количественнойоценке целостности клеточной мембраны. Окрашивание культивируемых клеток аннексином V и йодидом пропидия перед фиксацией позволяет оценить апоптотические и некротические клетки4. Тем не менее, различия в плотности клеток в разных лунках влияют на результаты каждого из этих анализов. Полагаясь исключительно на плотность посева, можно получить вводящие в заблуждение результаты из-за ошибок при подсчете клеток для посева, изменений плотности клеток в средах во время нанесения покрытий или разной скорости роста клеток между образцами или обработками в ходе экспериментов. Поэтому требуется нормализация экспериментальных результатов.
Современные методы нормализации функциональных и визуальных данных клеток включают концентрацию белка5 или ядер или количество клеток6. Чтобы нормализовать данные по концентрации белка, клетки должны быть экстрагированы в равных объемах буфера для лизиса с ингибитором протеазы после проведения анализа, а также должен быть проведен дополнительный анализ (например, анализ бицинхониновой кислоты, анализ Брэдфорда и т.д.) для количественного определения белка. Точность этого метода зависит от полной экстракции клеток; Отказ от любого белка приводит к ошибкам в количественном определении. Подсчет ядер или клеток является альтернативой, когда нет необходимости удалять клетки с поверхности, на которой они выращиваются. Вместо этого живые или неподвижные клетки окрашиваются или контрокрашиваются с помощью колориметрических или флуоресцентных красителей, а подсчет может быть выполнен на основе изображений целых клеток или только ядер. Количественное определение ядер предпочтительнее цельных клеток для флуоресцентной микроскопии и нормализации данных в слитых клетках (например, миотрубках) или тканях. Для количественного определения ядер с использованием флуоресцентного подхода флуоресцентные ДНК-красители (например, 4',6-диамидино-2-фенилиндола дихлорид (DAPI), Hoechst 33342, Hoechst 33358, DRAQ5 и т.д.) наносят на живые или фиксированные клетки и визуализируют с помощью флуоресцентной микроскопии. DAPI, Hoechst и DRAQ5 преимущественно связываются с богатыми аденин-тиминами областями ДНК 7,8,9, обеспечивая средства для обнаружения ядер. Оптимизация концентрации красителя предотвращает нецелевое связывание (т.е. с митохондриальной ДНК, вызывая точечную цитозольную флуоресценцию)10. Процесс оптимизации может быть оптимизирован за счет использования коммерчески доступных монтажных сред, содержащих DAPI для фиксированных элементов (например, VECTASHIELD Antifade Mounting Medium with DAPI) и опубликованных концентраций красителей для живых клеток (например, Hoechst)6,11,12. После окрашивания ядра живых или фиксированных клеток визуализируются с помощью соответствующего фильтра возбуждения (например, ультрафиолетового [DAPI и Hoechst] или дальнего красного [DRAQ5]). Максимумы возбуждения для DAPI, Hoechst и DRAQ5 находятся на обоих концах спектра для флуоресцентной микроскопии. Этот атрибут, а также специфичность ДНК этих красителей и их использование в оптимальных концентрациях, сводит к минимуму флуоресценцию за пределами мишени. Полученные изображения показывают яркие ядра на черном фоне, и эти ядра количественно измеряются как мера количества клеток для одноядерных клеток. В то время как существуют и другие точные методы количественного определения ядер на основе окрашивания с использованием гистологических красителей и светлопольной микроскопии13, автоматизация с использованием этих подходов более сложна, хотя и возможна.
Несмотря на то, что это остается золотым стандартом, ручное количественное определение ядер является трудоемким, трудоемким и подверженным потенциальной человеческой ошибке при длительном подсчете. Несмотря на то, что существуют программы автоматического подсчета ячеек, они не являются предпочтительными для всех пользователей, могут быть непомерно дорогими, а валидация для конкретных приложений может быть минимальной. В последние годы Python стал ценным и доступным ресурсом для биологов. Библиотеки компьютерного зрения Python особенно полезны для анализа изображений14. Здесь мы предоставляем пошаговые инструкции по использованию нашей исполняемой программы, разработанной с использованием кода Python, для количественного определения ядер, окрашенных флуоресцентными красителями и визуализированных с помощью флуоресцентной микроскопии. Навыки программирования не являются обязательными для использования описанной здесь программы. Этот рабочий процесс специфичен для клеток или тканей с применением флуоресцентных ДНК-красителей. Он не предназначен для использования со светлыми изображениями. В разделе 1 описывается, как захватывать и сохранять изображения, собранные с помощью флуоресцентной микроскопии, способом, совместимым с этим рабочим процессом. В разделе 2 приведены инструкции по запуску программы количественного определения ядер в качестве исполняемого и извлекаемого выхода. Исполняемая программа может быть запущена непосредственно из предоставленного файла .exe и не требует модификаций для количественного определения ядер в качественных изображениях. Для этого исполняемого файла требуется ПК под управлением Windows. Раздел 3 содержит инструкции по запуску программы в виде скрипта Python, а не исполняемого файла (требуется для систем на базе Mac или Linux, необязательно для систем на базе Windows) и позволяет при желании настроить код. В разделе 4 описывается, как использовать результаты для нормализации. За этими протоколами следуют репрезентативные результаты, в том числе данные валидации из нашей лаборатории.
протокол
ПРИМЕЧАНИЕ: Дополнительные файлы можно найти по следующей ссылке https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. Захват и сохранение изображений с помощью флуоресцентной микроскопии
- Подготовьте образцы клеток или тканей для визуализации, включая окрашивание желаемым ДНК-красителем. Для получения использованных здесь изображений миобласты C2C12 (CRL-1772, American Type Culture Collection) выращивали в 6-луночных планшетах в течение 48-72 ч в стандартных условиях культивирования (5%CO2, 37 °C, увлажненные), с 50 мМ EtOH6 или без него, и фиксировали в ледяном метаноле, как описано ранее13. Фиксированные элементы монтировали с помощью DAPI-содержащего монтажного носителя15 и сушили в темноте перед визуализацией.
- Включите все компоненты флуоресцентного микроскопа и компьютера в соответствии с установленными стандартными рабочими процедурами. Откройте программное обеспечение, связанное с микроскопом.
- Установите микроскоп для визуализации образцов через окуляр. Проверьте цель. При необходимости поверните револьвер в нужное положение. Убедитесь, что в программе выбран желаемый объектив для точного отображения области изображения. Здесь изображения были сняты с использованием 10-кратного объектива.
- Выключив свет, положите образец (слайд, тарелку или блюдо) на сцену. Включите изображение в реальном времени с помощью соответствующего фильтра и установите время экспозиции либо на заданную продолжительность, либо на автоматическую экспозицию. Здесь был использован фильтр DAPI и время экспозиции 61-62 мс. В таблице устранения неполадок приведены рекомендации по настройке времени экспозиции, особенно для изображений с высокой плотностью ячеек.
- С помощью окуляра медленно отрегулируйте фокус с помощью ручки грубой регулировки, пока не станут видны окрашенные ядра, затем используйте ручку точной регулировки, чтобы медленно отрегулировать фокус, пока ядра не станут четкими и четкими.
ПРИМЕЧАНИЕ: Примеры подходящих изображений показаны на рисунке 1.- Если появляется чрезмерное окрашивание фона или если ядра кажутся очень тусклыми, отрегулируйте время экспозиции вручную. На одном изображении могут быть видимые различия в интенсивности сигнала для некоторых ядер; Тем не менее, круглая форма должна быть видна для всех ядер с минимальными помехами сигнала.
ПРИМЕЧАНИЕ: Программа, используемая в последующих приложениях, устойчива к различным интенсивностям. Необработанные изображения, используемые для разработки и валидации программы количественной оценки ядер, включают изображения с диапазоном интенсивностей сигнала и доступны через репозиторий Open Science Framework (см. ссылку, упомянутую выше).
- Если появляется чрезмерное окрашивание фона или если ядра кажутся очень тусклыми, отрегулируйте время экспозиции вручную. На одном изображении могут быть видимые различия в интенсивности сигнала для некоторых ядер; Тем не менее, круглая форма должна быть видна для всех ядер с минимальными помехами сигнала.
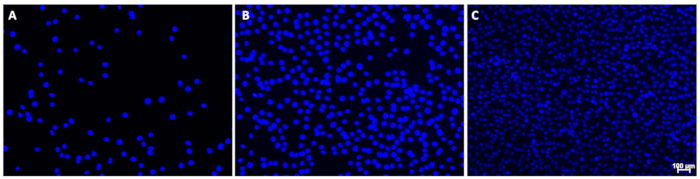
Рисунок 1: Примеры изображений ядер, окрашенных DAPI. На этих изображениях изображены ядра с (А) низкой, (В) средней или (С) высокой плотностью клеток с минимальным фоном. Обратите внимание на изменяющуюся интенсивность сигнала ядер на изображении C; Это не мешало количественной оценке. Масштабная линейка (C) показывает 100 μм; Масштабные линейки должны быть опущены на изображениях, используемых для количественного определения ядер. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
- Как только ядра окажутся в фокусе через окуляр, переключите режим обнаружения на камеру. Настройте фокусировку с помощью ручки точной регулировки.
- При необходимости отрегулируйте время экспозиции. Запишите окончательное время экспозиции для использования с последующими образцами и изображениями.
ПРИМЕЧАНИЕ: Несмотря на то, что использование одного и того же времени экспозиции для всех изображений является идеальным, вариации времени экспозиции приемлемы для этого приложения, поскольку интенсивность флуоресценции не измеряется количественно и не сравнивается между образцами. - Сделайте снимок с помощью соответствующей кнопки захвата изображения в программном обеспечении. Если на образцы нанесено несколько окрашиваний, переключите фильтр (например, на FITC, TRITC или Cy5) и сделайте изображение одной и той же области для объединения.
- Установите затвор, чтобы свести к минимуму воздействие света на образец. Не записывая в масштабную линейку, сохраните изображение в универсальном формате файла фигур (.tif, .png или .jpg) в специальной папке. Убедитесь, что имя файла каждого изображения является надлежащим образом описательным или что любой код, используемый для имен файлов, имеет соответствующую связанную документацию.
ПРИМЕЧАНИЕ: Он станет каталогом изображений для программы количественной оценки. Если использовалась многоцветная визуализация, скопируйте только изображения ядер в отдельную папку. Это также можно сделать в конце сеанса визуализации. Изображения, экспортируемые с использованием стандартных настроек экспорта в большинстве программ обработки изображений, обычно имеют разрешение, достаточное для использования с этой программой, при условии, что были получены качественные изображения. Тем не менее, изображения должны быть визуально проверены после экспорта, чтобы убедиться, что морфология ядер ясна. Если изображения сохраняются в .jpg формате, следует позаботиться о том, чтобы изображения не были чрезмерно сжаты, а качество было сохранено. - Откройте затвор, вернитесь к визуализации в реальном времени и отсканируйте образец в другое место для визуализации. При необходимости повторяйте шаги 1.8-1.9. Для изображений клеток, засеянных в 6-луночные планшеты, снятых с 10-кратным увеличением, сделайте 10-15 изображений на лунку (в зависимости от плотности клеток на площади образца).
- После получения всех изображений для одного образца выполните один из следующих вариантов.
- Переходим к следующему образцу и повторяем шаги 1.4-1.10.
- Поставьте протокол на паузу и вернитесь в другое время. Образцы должны оставаться защищенными от света. Этот вариант не рекомендуется при работе с живыми клетками.
- После завершения визуализации убедитесь, что все образцы защищены от света, прежде чем включать свет в комнате. Если папка с изображениями сохранена локально, перенесите файлы на флэш-накопитель, в облачное хранилище или на другой сетевой диск, доступный компьютеру, на котором будет запущена программа количественной оценки. Сохраните в специальной папке.
- Выключите все части микроскопа и компьютера в соответствии с установленными стандартными рабочими процедурами.
- Теперь визуализация завершена. Проанализируйте файлы с помощью программы автоматической количественной оценки, как описано в разделе 2, или, в качестве альтернативы, запустив программу в виде скрипта Python, как описано в разделе 3.
2. Запуск автоматизированной программы количественного определения ядер в качестве исполняемого файла
ПРИМЕЧАНИЕ: Этот метод, запускающий программу в качестве исполняемого файла, совместим с ПК на базе Windows и является рекомендуемым для пользователей ОС Windows. Пользователи Mac OS или другие пользователи, отличные от Windows, должны запускать программу как скрипт Python (см. раздел 3).
- Убедитесь, что на компьютере установлен Python. При необходимости установите Python из python.org/downloads (предпочтительно) или с помощью Anaconda или Miniconda. На дату публикации данная программа была протестирована с Python версий 3.10-3.12.
- Откройте браузер и перейдите в раздел https://github.com/rbudnar/nuclei_counter/releases. Загрузите последнюю версию файла с именем count_nuclei.zip.
- В папке Downloads щелкните правой кнопкой мыши Zip-файл и выберите команду Extract All files в нужное место на локальном компьютере (см. рисунок 2A). Держите это местоположение файла открытым; этот путь к файлу будет использоваться на шаге 2.5 (см. рисунок 2B).
ПРИМЕЧАНИЕ: Count_nuclei теперь должен отображаться как файл приложения. Это исполняемый файл. - Откройте командную строку, выполнив поиск CMD или Командная строка в строке поиска. Используйте команду cd, чтобы изменить каталог на путь к исполняемому файлу (см. рисунок 2C) и нажмите Enter. Пример командной строки: C:\Users\janedoe>cd [пробел после cd, затем копирование/вставка пути к файлу]
- В следующей командной строке введите следующую команду и нажмите клавишу Enter.
count_nuclei.exe --img_dir "путь/к/изображениям" --output_dir "путь/к/выводу" --save_images --output_count_filename "results.csv"
где path/to/images – путь к файлу к папке, содержащей анализируемые изображения (см. шаг 1.14); path/to/output - это путь к папке, в которую должен быть сохранен .csv файл (по умолчанию текущий рабочий каталог, если он не указан); и results.csv — это имя файла, в котором будут сохранены результирующие счетчики (если оно не указано, имя выходного файла по умолчанию — count_results.csv).
ПРИМЕЧАНИЕ: Кавычки нужны в командной строке. Убедитесь, что перед путем к файлу нет пробелов после кавычек (см. рис. 2C). - Когда появится следующая командная строка, обработка будет завершена. Контуры и таблица результатов теперь доступны в месте, указанном в качестве выходного каталога на шаге 2.5 (т.е. путь/к/выходу; см. рисунок 2C).
- Визуально осмотрите контуры, чтобы убедиться в качестве подсчета. Теперь результаты можно использовать для нормализации данных, как описано в разделе 4.
ПРИМЕЧАНИЕ: Если в контурах отсутствуют ядра (например, ядра кажутся слишком слабыми) или группируются группы ядер в один элемент (например, переэкспонированное изображение, слишком много фона, артефакты), пользователю следует обратиться к таблице устранения неполадок и, возможно, потребуется вернуться к разделу 1 для оптимизации получения изображения, прежде чем переходить к разделу 4.
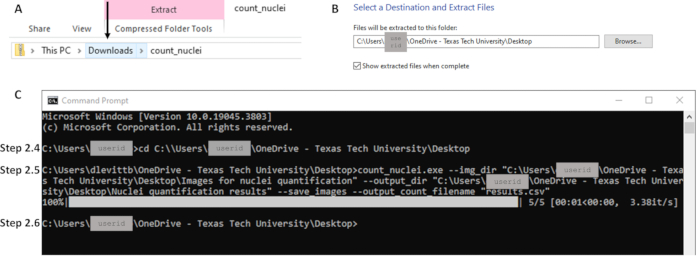
Рисунок 2: Примеры ключевых частей рабочего процесса для раздела 2. (A) Извлекайте файлы из папки Downloads, стараясь нажать на нужную папку, как указано стрелкой, чтобы включить извлечение (B) в нужное место назначения на шаге 2.3. (C) Примеры команд для шагов 2.4 и 2.5 и следующей командной строки, указывающей на завершение на шаге 2.6. Обратите внимание, что путь к файлу на шаге 2.4 (C) совпадает с расположением для извлечения на шаге 2.3 (B). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
3. Запуск программы автоматической количественной оценки ядер в виде скрипта Python
- Убедитесь, что на компьютере установлен Python. При необходимости установите Python из python.org/downloads (предпочтительно) или с помощью Anaconda или Miniconda. На дату публикации данная программа была протестирована с Python версий 3.10-3.12.
- Откройте браузер и перейдите в раздел https://github.com/rbudnar/nuclei_counter. Нажмите кнопку «Зеленый код », затем нажмите «Загрузить ZIP », чтобы загрузить репозиторий кода (см. рисунок 3A).
- Используйте следующие инструкции для ОС Windows. Смотрите шаг 3.4 для компьютеров на базе Mac OS.
- В папке «Загрузки» щелкните правой кнопкой мыши файл и выберите «Извлечь все», чтобы извлечь файлы на локальный компьютер (см. рисунок 3B-C).
- В папке извлеченного файла откройте папку с именем nuclei_counter-main. Это репозиторий кода.
ПРИМЕЧАНИЕ: Это необходимо загрузить только при первом использовании этой программы; Просто переместите извлеченные файлы в доступное сетевое расположение и запишите это местоположение. - Откройте командную строку, выполнив поиск CMD или Командная строка в строке поиска. Если Python был установлен с помощью Anaconda или Miniconda, откройте приглашение Anaconda вместо стандартной командной строки. Это можно найти, набрав Anaconda Prompt в строке поиска.
- Используйте команду cd для изменения каталога на репозиторий кода и нажмите Enter. Смотрите строку кода ниже, которая чувствительна к регистру. Убедитесь, что папка с именем nuclei_counter-main открыта при копировании пути к файлу; файл с именем pyproject или pyproject.toml должен быть виден (см. Рисунок 3D). Пример командной строки: C:\Users\janedoe>cd [пробел после cd, затем копирование/вставка пути к файлу]
- В следующей командной строке введите следующее, включая пробел и точку (см. Рисунок 3D):
pip install -e .
ПРИМЕЧАНИЕ: Шаги 3.3.5 и 3.3.6 должны быть выполнены только при первом запуске этой программы на любом компьютере. Если это уже завершено, перейдите к шагу 3.3.7. Процесс установки может занять несколько секунд для инициализации. - Нажмите Enter. Выполнение этой команды устанавливает необходимые библиотеки для запуска этого скрипта и устанавливает репозиторий в редактируемом режиме в случае, если требуется корректировка кода (см. Рисунок 3D).
- Введите следующее в следующую командную строку, чтобы изменить каталог на основной каталог исходного кода (см. рисунок 3E):
CD "nuclei_counter" - В следующей командной строке введите следующее и нажмите Enter (см. Рисунок 3E):
Python count_nuclei.py --img_dir "путь/к/изображениям" --output_dir "путь/к/выводу" --save_images --output_count_filename "results.csv"
Где path/to/images — путь к файлу к папке, содержащей анализируемые изображения (шаг 1.14); path/to/output - это путь к папке, в которую должен быть сохранен .csv файл (по умолчанию текущий рабочий каталог, если он не указан); и results.csv — это имя файла, в котором будут сохранены результирующие счетчики (если оно не указано, имя выходного файла по умолчанию — count_results.csv).
ПРИМЕЧАНИЕ: Кавычки нужны в командной строке. Убедитесь, что перед путем к файлу нет пробелов после кавычек. Скрипт Python прочитает файлы по указанному пути к файлу и выполнит программу подсчета ядер для всех изображений в каталоге (см. рисунок 3E). Полученные подсчеты будут сохранены в файле результатов .csv указанном расположении. - Когда появляется следующая командная строка, обработка завершается (см. рисунок 3E). Контуры и таблица результатов теперь доступны в месте, указанном в качестве выходного каталога на шаге 3.3.8 (т.е. путь/к/выходу).
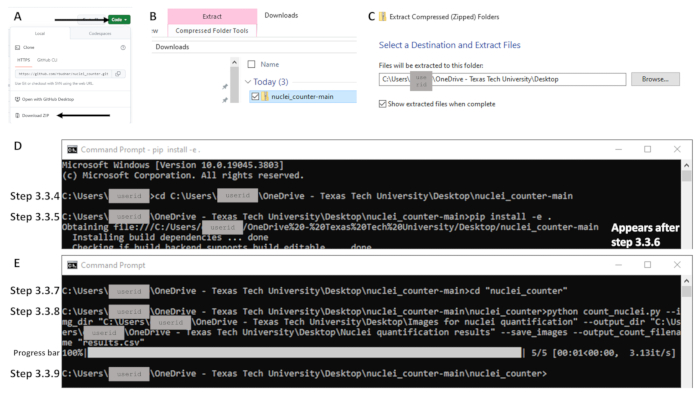
Рисунок 3: Примеры ключевых частей рабочего процесса для раздела 3 с использованием ОС Windows. Чтобы получить доступ к коду, нажмите зеленую кнопку с надписью « Код » и (A) загрузите Zip-файл (обозначен стрелками). (B) Распакуйте, щелкнув правой кнопкой мыши по Zip-файлу (показан синим цветом) и (C), установив желаемое место назначения. (D) После открытия командной строки примеры команд для шагов 3.3.4 и 3.3.5. Обратите внимание, что все команды для шага 3.3.5 находятся в одной строке; текст, начинающийся с "Получение файла" на следующей строке, появляется после нажатия клавиши Enter (шаг 3.3.6; т.е. выполнение команды). (E) Примеры команд для шагов 3.3.7 и 3.3.8, индикатор выполнения после выполнения шага 3.3.8 и командная строка, показывающая завершение на шаге 3.3.9. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
- Используйте следующие инструкции для Mac OS.
- В папке «Загрузки» нажмите на меню «Файл » и выберите «Открыть », чтобы извлечь файлы на локальный компьютер (см. рисунок 4A).
- В папке извлеченного файла откройте папку с именем nuclei_counter-main. Это репозиторий кода.
ПРИМЕЧАНИЕ: Это необходимо загрузить только при первом использовании этой программы; Просто переместите извлеченные файлы в доступное сетевое расположение и запишите это местоположение. - Откройте терминал нажатием клавиш Command+пробел , после чего откроется прожектор; Затем выполните поиск по запросу «Терминал».
- Используйте команду cd для изменения каталога на репозиторий кода и нажмите Enter. Смотрите строку кода ниже, которая чувствительна к регистру. Введите путь к файлу в текстовом документе и скопируйте его в терминал. Убедитесь, что путь к файлу начинается с текста, непосредственно предшествующего cd (например, /Users/userid на рисунке 4B). Пример командной строки: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- В следующей командной строке после идентификатора пользователя появится знак доллара (см. рисунок 4B). Убедитесь, что после знака доллара есть пробел, и введите следующее, включая пробел и точку в конце команды. Сразу после pip без пробела укажите соответствующую версию Python: pip3 install -e .
ПРИМЕЧАНИЕ: Шаги 3.4.5 и 3.4.6 должны быть выполнены только при первом запуске этой программы на любом компьютере. Если это уже завершено, переходим к шагу 3.4.7. Процесс установки может занять несколько секунд для инициализации. - Нажмите Enter. Выполнение этой команды устанавливает необходимые библиотеки для запуска этого скрипта и устанавливает репозиторий в редактируемом режиме, если требуется корректировка кода.
- В следующей командной строке убедитесь, что после знака доллара есть пробел, затем введите следующее, чтобы изменить каталог на основной каталог исходного кода (см. рисунок 4C):
CD "nuclei_counter" - В следующей командной строке введите командную строку ниже. Замените номер после Python на правильный номер версии, если вы используете версию, отличную от 3. Затем нажмите Enter (см. Рисунок 4C):
python3 count_nuclei.py --img_dir "путь/к/изображениям" --output_dir " путь/к/выводу" --save_images --output_count_filename "results.csv"
где path/to/images – путь к файлу к папке, содержащей анализируемые изображения (шаг 1.14); path/to/output - это путь к папке, в которую должен быть сохранен .csv файл (по умолчанию текущий рабочий каталог, если он не указан); и results.csv — это имя файла, в котором будут сохранены результирующие счетчики (если оно не указано, имя выходного файла по умолчанию — count_results.csv).
ПРИМЕЧАНИЕ: Кавычки нужны в командной строке. Убедитесь, что перед путем к файлу нет пробелов после кавычек. Скрипт Python прочитает файлы по указанному пути к файлу и выполнит программу подсчета ядер для всех изображений в каталоге (см. рисунок 4C). Полученные подсчеты будут сохранены в файле результатов .csv указанном месте. - Когда появляется следующая командная строка, обработка завершается (см. рис. 4C). Контуры и таблица результатов теперь доступны в месте, указанном в качестве выходного каталога на шаге 3.4.8 (т.е. путь/к/выходу).
- Визуально осмотрите контуры, чтобы убедиться в качестве подсчета. Теперь результаты можно использовать для нормализации данных, как описано в разделе 4.
ПРИМЕЧАНИЕ: Если в контурах отсутствуют ядра (например, ядра кажутся слишком слабыми) или группируются группы ядер в один элемент (например, переэкспонированное изображение, слишком много фона, артефакты), пользователю следует обратиться к таблице 1 и, возможно, потребуется вернуться к разделу 1 для оптимизации получения изображения, прежде чем переходить к разделу 4.
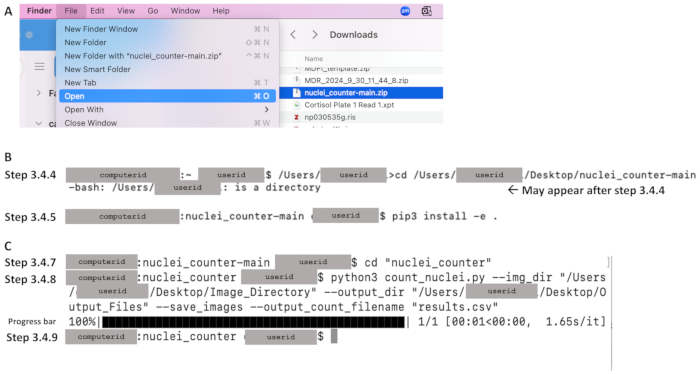
Рисунок 4: Примеры ключевых частей рабочего процесса для раздела 3 с использованием Mac OS. После загрузки кода (см. рис. 3A) (A) извлеките, щелкнув по Zip-файлу (показан синим цветом) и установив желаемое место назначения. (B) После открытия терминала примеры команд для шагов 3.4.4 (смена директории на репозиторий кода) и 3.4.5 (установка pip). (C) Примеры команд для шагов 3.4.7 и 3.4.8, индикатор выполнения после выполнения шага 3.4.8 и командная строка, показывающая завершение на шаге 3.4.9. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
4. Использование счетчиков для нормализации экспериментальных данных
- Откройте файл results.csv, связывающий каждое изображение с количественной оценкой ядер, определенной с помощью автоматизированной программы.
- Определитесь с методом нормализации: 1) на основе изображения за изображением (например, при многоцветной флуоресцентной микроскопии), 2) нормализуйте данные как отношение среднего количества ядер на образец к сравнению с эталонным образцом (или условием), 3) нормализуйте до количества ядер на образец (например, экстраполируйте общее количество ядер на площадь образца). Рекомендации по методу нормализации находятся в разделе Обсуждение.
- Если нормализация будет выполняться для каждого изображения, просто разделите экспериментальные данные для каждого изображения на количество ядер для этого изображения.
- Чтобы нормализовать данные в виде соотношения, выполните действия, описанные ниже.
- Для любых образцов, в которых было собрано несколько изображений для одной реплики (например, 10-15 изображений, полученных из одной лунки в 6-луночном планшете), рассчитайте среднее количество ядер на изображение. Эти подсчеты ядер рассматриваются как количество клеток, предполагая, что одноядерные клетки или несколько клеток слились вместе в многоядерные клетки (например, в миотрубках).
- Выберите эталонную выборку или эталонное условие (например, среднее количество по всем выборкам из контрольного условия) и запишите количество.
- Разделите количество для всех выборок на это эталонное количество, в результате чего получится значение нормализации. Если использовалась эталонная выборка, используйте значение нормализации для эталонной выборки как 1.
ПРИМЕЧАНИЕ: Если бы использовалось эталонное условие, среднее значение нормализации по условию теперь было бы равно 1. Образцы с меньшим числом ядер, чем эталонный (т.е. менее плотные), будут иметь значения нормализации меньше 1, а образцы с большим количеством ядер, чем эталонный (т.е. более плотные), будут иметь значения нормализации больше 1. - Разделите экспериментальные данные по каждой скважине на соответствующее рассчитанное значение нормализации.
- Чтобы нормализовать данные по количеству ядер в образце, выполните действия, описанные ниже.
- Для любых образцов, в которых было собрано несколько изображений для одной реплики (например, 10-15 изображений, полученных из одной лунки в 6-луночном планшете), рассчитайте среднее количество ядер на изображение. Эти подсчеты ядер рассматриваются как количество клеток, предполагающих одноядерные клетки, или количество клеток, слитых вместе в многоядерных клетках (например, в миотрубках).
- Определите площадь поверхности предметного стекла (покровного стекла), колодца или чашки, из которой были получены данные. Эту информацию можно найти в разделе технических характеристик на веб-сайтах продуктов.
- Определите площадь каждого захваченного изображения. Эта информация часто доступна в виде метаданных в программном обеспечении для визуализации или анализа.
- Убедитесь, что площадь поверхности образца и площадь изображения выражены в одних и тех же единицах измерения (например,мм2). Если нет, преобразуйте в нужную единицу измерения.
- Рассчитайте общее количество для каждой выборки с помощью следующего уравнения:
TC = AC • (SA/IA)
где TC — общее количество, AC — среднее количество, SA — площадь поверхности, IA — площадь изображения. - Разделите экспериментальные данные на соответствующее ТС для образца.
- Нормализовать экспериментальные данные и использовать их для статистического анализа по мере необходимости.
Результаты
При каждом пакетном прогоне изображений создается: 1) набор файлов изображений с нанесенными контурами, показывающими контуры идентифицированных ядер (рисунок 5), и 2) файл .csv (электронная таблица), связывающий имена файлов изображений и связанные с ними счетчики. Просмотр контуров позволит пользователю визуально оценить качество подсчета. В частности, изображения, полученные в соответствии с разделом 1, должны иметь все (или почти все) ядра, окруженные сплошной зеленой линией, указывающей на то, что ядро было подсчитано программой. Эти контуры были использованы для настройки программы здесь.
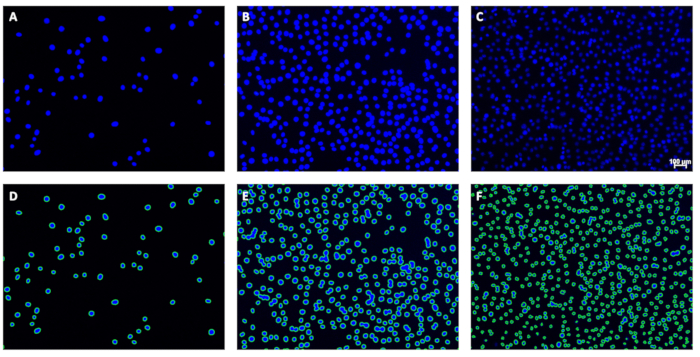
Рисунок 5: Примеры исходных изображений и соответствующих контуров. (А-С) Ниже приведены примеры исходных изображений до анализа и (D-F) соответствующих контуров после анализа. Масштабная линейка (C) показывает 100 μм; Масштабные линейки должны быть опущены на изображениях, используемых для количественного определения ядер. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Валидация с использованием первоначальных результатов
Миобласты C2C12 при различной плотности клеток фиксировали метанолом, окрашивали DAPI в монтажных средах, и получали 120 изображений при 10-кратном увеличении в .jpg формате файла. Автоматическая программа была запущена с Image Directory, установленной на папку, содержащую эти изображения, и полученный файл .csv был сохранен. Отдельно каждое изображение было вручную количественно определено двумя обученными исследователями, которые не обращали внимания на автоматические подсчеты и подсчеты друг друга. Необработанные изображения, контуры, сгенерированные автоматизированной программой, и таблица результатов, включающая ручные и автоматические подсчеты, доступны в репозитории Open Science Framework (OSF) (см. ссылку в разделе 1). Внутриклассовые коэффициенты корреляции (ICCs; средние меры) рассчитывались с помощью SPSS Statistics для определения между: надежностью между: 1) двумя ручными подсчетами и 2) средним значением двух ручных подсчетов и подсчетов, полученных с помощью автоматизированной программы. Аналогичные подходы были использованы для валидации других программ, предназначенных для количественной оценки объектов на изображениях, полученных с помощью флуоресцентной микроскопии 16,17,18. ICC также рассчитывались между средним ручным подсчетом и автоматическим подсчетом для каждого квартиля, чтобы гарантировать, что оценка всего набора данных не скрывает никаких потенциальных изменений надежности на основе плотности клеток. Квартили были определены с использованием25-го,50-го и75-го процентилей на основе среднего ручного подсчета и варьируются от квартили 1 (Q1; самая низкая плотность ячеек) до Q4 (самая высокая плотность ячеек). ICC представляются на основе средних показателей и интерпретируются на основе следующих определений: плохой (ICC <0,50), средний (0,50 ≤ICC <0,75), хороший (0,75 ≤ICC <0,90) или отличный (ICC ≥0,90)19.
Количество ядер на изображение варьировалось от 44,5 до 1160 на основе ручных средних значений. Надежность интеррейтера была отличной между двумя ручными подсчетами (ICC > 0,999, p < 0,0001; Рисунок 6А) и между средними ручными и автоматическими подсчетами (ICC = 0,993, p < 0,0001; Рисунок 6B). Надежность оставалась отличной по всем квартилям (Q1: ICC = 0,996, p < 0,0001; Q2: ICC= 0,997, p < 0,0001; Q3: ICC = 0,998, p < 0,0001; Q4: ICC = 0,986, p < 0,0001; Рисунок 6C-F). Единственная точка данных, удаленная от линии наилучшего соответствия на рисунке 6B, представляет изображение, показанное на рисунке 7. Изображение имеет несколько областей с несколькими ядрами, сгруппированными вместе, что не позволяет программе компьютерного зрения распознать каждое ядро. Эту проблему можно исправить, используя более короткое время экспозиции. В таблице 1 описано, как работать с таким изображением.
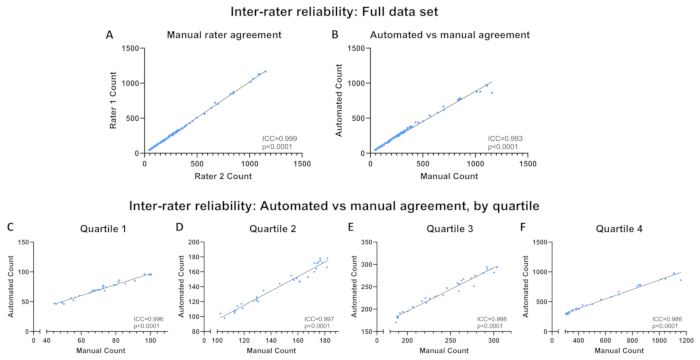
Рисунок 6: Надежность между оценщиками. Надежность между оценщиками по всему набору данных (A) между ручными подсчетами и (B) между автоматическими подсчетами и средними ручными подсчетами. (К-Ж) Межрейтинговая надежность между автоматическим подсчетом и средним ручным подсчетом по квартилям. Надежность выражается в виде внутриклассового коэффициента корреляции (ICC). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 7: Пример изображения с двумя барьерами для точного количественного определения ядер. (A) Исходное изображение и (B) связанные контуры. Стрелка 1 указывает на область с несколькими ядрами, слипшимися вместе, искажающими круглую форму ядра настолько, что это затрудняет обнаружение нескольких ядер программным обеспечением. Это происходит в нескольких местах на этом изображении. Стрелка 2 указывает на блеклый ореол, появляющийся за несколькими ядрами; Это гало выглядело достаточно ярким, чтобы программное обеспечение включило его в качестве ядра, тем самым потеряв количество нескольких ядер, которые оно окружает. На этом изображении показана единственная точка данных, удаленная от линии наилучшего соответствия на рисунке 6B, что подчеркивает потенциальные проблемы, связанные с этими барьерами. Масштабная линейка (А) показывает 100 мкм; Масштабные линейки должны быть опущены на изображениях, используемых для количественного определения ядер. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Обсуждение
Наша программа количественной оценки ядер имеет ряд преимуществ по сравнению с существующими вариантами: она требует лишь минимальных технологических навыков, проверена для конкретной задачи количественной оценки ядер и имеет открытый исходный код; Последнее позволяет преодолеть барьеры, связанные с затратами. В конечном счете, эта программа предоставляет клеточным и молекулярным биологам дополнительную возможность быстро и точно количественно определять ядра на изображениях, полученных с помощью флуоресцентной микроскопии. Доступные в настоящее время автоматизированные программы подсчета ядер или клеток не являются предпочтительными для всех пользователей, некоторые из них непомерно дороги, а валидация может быть минимальной или отсутствовать для конкретных приложений. Специализированное оборудование, такое как линия BioTek Cytation, сопровождается техническими бюллетенями, подтверждающими валидацию подсчета клеток in situ с использованием ядерного окрашивания20, в дополнение к широкому спектру дополнительных применений. Несмотря на то, что данные валидации существуют, а линейка продуктов Cytation широко используется, особенно при сопряжении с анализаторами внеклеточного потока Seahorse для простой и эффективной нормализации данных живых клеток, этот продукт может быть непомерно дорогим для лабораторий с ограниченным финансированием оборудования. Например, прейскурантная цена на базовую модель, оснащенную четырьмя объективами и четырьмя фильтрующими кубами для флуоресцентной микроскопии, превысила 100 000 долларов США в 2024 календарном году. Для тех, у кого нет средств на покупку специализированного оборудования, программное обеспечение с открытым исходным кодом предоставляет бесплатные альтернативы для анализа изображений. Программное обеспечение с открытым исходным кодом, полезное для количественного определения ядер и клеток, включает ImageJ (National Institutes of Health, Bethesda, MD, США; доступно по адресу 21. Теперь доступны плагины для тех, кто желает писать макроскрипты для автоматизации обработки изображений с помощью платформы ImageJ. Автоматизация подсчета клеток на основе ImageJ с использованием фазового контраста или светлопольной микроскопии с изображениями целых клеток была описана ранее22,23. CellProfiler является широко публикуемым приложением для анализа изображений клеток, окрашенных флуоресцентными красителями или стандартными гистологическими красителями, и ранняя статья, описывающая его полезность в ряде биологических приложений, часто упоминается как валидация24. Тем не менее, данная рукопись не подтверждает каких-либо конкретных биологических открытий, что требует дополнительной валидации и прозрачности для различных используемых конвейеров автоматизации, таких как подсчет ядер25. Более того, несмотря на то, что CellProfiler является эффективным программным пакетом для анализа клеток, не все ученые находят его удобным для пользователя. Поэтому мы поставили перед собой цель разработать и проверить альтернативную автоматизированную программу для количественной оценки ядер с использованием программного обеспечения с открытым исходным кодом. В частности, мы использовали существующие библиотеки компьютерного зрения на Python для разработки этого рабочего процесса.
Этот метод начинается с клеток или тканей, которые конечный пользователь окрашивает с помощью красителей ДНК, таких как DAPI26. Приведены общие рекомендации по захвату и сохранению изображений способом, совместимым с программным обеспечением для автоматизации. Несмотря на то, что детали могут отличаться в зависимости от микроскопа и программного обеспечения для визуализации, общий рабочий процесс для раздела 1 для получения количественных изображений одинаков. При использовании живых образцов необходимо позаботиться о том, чтобы на них были надеты соответствующие средства индивидуальной защиты и условия визуализации соответствовали этим образцам (например, температура, процентное содержаниеCO2, добавление буфера и т. д.).27. В то время как данный протокол описывает визуализацию только красителей ДНК, пользователи могут захватывать другие цвета той же области для целей объединения15 и просто копировать ядерные изображения в отдельную папку для использования с программой количественной оценки. Визуализацию следует проводить в темной комнате с использованием соответствующих СИЗ для живых образцов, а при необходимости следует использовать надлежащую дезинфекцию (например, с использованием 70% этанола). Окрашенные образцы следует беречь от света перед включением света. Качественный захват изображений имеет решающее значение для точного количественного определения ядер. Даже на изображениях с подходящим временем экспозиции и сильным контрастом между яркими ядрами и черным фоном присутствие чрезмерно сгруппированных клеток или ореолов за группами клеток (см. рис. 7) может снизить точность количественной оценки. В этих случаях может потребоваться изменение времени экспозиции для снижения интенсивности сигналов, обеспечения минимального слияния сигналов ядер и минимизации ореолов при сохранении достаточно сильного сигнала для обнаружения округлых форм ядер. Если этих шагов недостаточно, может потребоваться визуализация соседнего участка образца. Все изображения для одного эксперимента должны быть получены с одинаковым увеличением, а увеличение, соответствующее используемому объективу, должно быть правильно установлено в программном обеспечении для обработки изображений. Особенно важно, чтобы объектив, используемый для визуализации, соответствовал настройкам программного обеспечения для визуализации, когда количество ядер будет экстраполировано из области визуализации на всю область лунки (или предметного стекла). Этот шаг должен выполняться при каждом переключении объектива, но его легко упустить из виду при настройке микроскопа и программного обеспечения. Время, необходимое для работы с разделом 1, зависит от ряда факторов (например, от пользовательского опыта работы с флуоресцентной микроскопией, количества изображений и образцов, микроскопа и т. д.). Чтобы получить 10-15 изображений окрашенных ядер в лунке в двух 6-луночных планшетах, пользователь должен зарезервировать примерно 1-2 часа времени.
После того, как изображения были сняты и сохранены в специальной папке, они готовы к количественной оценке. Самым простым методом количественной оценки является запуск этой предварительно упакованной автоматизированной программы без необходимости установки Python или каких-либо библиотек (раздел 2). Следует отметить, что программа подготовлена в виде исполняемого файла и была упакована с помощью Pyinstaller. Программное обеспечение, созданное с помощью Pyinstaller, обычно помечается антивирусным программным обеспечением. Если антивирусное программное обеспечение блокирует открытие файла, пользователь должен пометить файл как безопасный, чтобы продолжить. Это имеет решающее значение для успешного выполнения раздела 2. В качестве альтернативы, любой пользователь может запустить программу автоматической количественной оценки в виде скрипта Python (раздел 3), а не в виде исполняемого файла, если это необходимо. Если пользователь хочет запустить программу с помощью операционной системы Mac или Linux, программа должна быть запущена через Python. Этот метод не требует Pyinstaller, но может быть более технически сложным. Пошаговый протокол вместе с примерами, подробно описанными в разделе 3, направлен на устранение технических барьеров для запуска этой программы в виде скрипта. Независимо от того, используется ли раздел 2 или раздел 3 для запуска программы, крайне важно убедиться, что код правильно введен в командную строку. Примеры строк кода включены во весь протокол, и пользователь должен уделять особое внимание пробелам, кавычкам и другим знакам препинания. Пользователь также должен убедиться в правильности путей к файлам. Полезно иметь пути к файлам для изображений и вывода, доступные в документе Word. Команды также могут быть введены в этот документ Word и скопированы/вставлены по мере необходимости.
Как описано в разделе 4, существует несколько подходов к нормализации экспериментальных данных с использованием подсчетов, полученных с помощью этой автоматизированной программы количественного определения ядер. Для экспериментов на основе изображений, в которых экспериментальные данные включают количество событий в каждом изображении и требуется соответствующая пропорция (например, доля аннексин V-положительных клеток28, доля клеток, экспрессирующих рецепторы ACE229, количество клеток, поглощающих окрашенные частицы30 и т.д.), экспериментальные данные для каждого изображения можно просто разделить на полученное количество (в результате чего получается пропорция от 0 до 1). Затем можно рассчитать среднюю пропорцию для повторяющихся изображений с одного и того же слайда, чашки или лунного колодца. Если вы хотите получить процентное соотношение, просто умножьте окончательные пропорции на 100. Наконец, эти средние пропорции или проценты могут быть использованы в последующем статистическом анализе. Когда нормализуемые экспериментальные данные получены в результате функциональных экспериментов на живых клетках (например, нормы потребления кислорода 6,11,12) или супернатанта клеточной культуры (например, активность лактатдегидрогеназы в надосадочной жидкости31 или концентрация специфического секретируемого цитокина32 и т.д.), количество ядер должно быть сначала усреднено по всем изображениям, полученным из одной и той же лунки. Затем количество ядер может быть экстраполировано на площадь поверхности предметного стекла (покровного стекла), колодца или чашки, или количество может быть выражено в виде отношения к эталонному образцу или состоянию. Экстраполяция на площадь поверхности предметного стекла, колодца или чашки полезна для выражения экспериментальных данных в зависимости от количества клеток (при использовании одноядерных клеток), из которых были получены данные (например, потребление кислорода в минуту на10-3 клетки). Чтобы использовать этот метод, необходимо знать площадь поверхности предметного стекла (покровного стекла), колодца или чашки и площадь, захваченную на каждом изображении. Если образец не покрывает всю доступную площадь поверхности, экспериментальные данные получены от небольшого количества надосадочной жидкости (т.е. выражены как активность или концентрация на единицу объема) или изображение и/или площадь поверхности неизвестны, то наилучшим вариантом является усреднение количества ядер по всем изображениям для одного образца и выражение количества ядер относительно эталонного образца или условия. Полученное значение используется для коррекции экспериментальных данных на плотность ядер или плотность клеток (при использовании одноядерных клеток).
Несмотря на то, что этот метод прост в исполнении, существует несколько потенциальных проблем, о которых пользователи должны знать. Эти потенциальные проблемы, а также возможные причины и шаги по их устранению перечислены в таблице 1. Описанная здесь программа имеет ключевые преимущества по сравнению с существующим программным обеспечением; Тем не менее, следует признать некоторые ограничения. Основным недостатком является то, что программу количественной оценки можно использовать только с изображениями, полученными с помощью флуоресцентной микроскопии, а не с изображениями в светлом поле. В то время как изображения, окрашенные DAPI, снятые при 10-кратном увеличении, были использованы для создания программы, программу можно использовать с другими красителями ДНК и увеличениями, поскольку программа построена таким образом, чтобы распознавать морфологию ядер, а не цвет или размер. Несмотря на то, что мы проверили точность программы на изображениях, содержащих различные плотности ячеек, у конечного пользователя плотность ячеек может выходить за пределы диапазона. В этом случае мы по-прежнему ожидаем точных подсчетов при условии получения качественных изображений; Тем не менее, пользователь должен тщательно проверять контуры и вывод, чтобы убедиться в точности. Для применения в клеточных культурах количественная оценка ядер на изображениях наиболее подходит для клеток в монослое. Если культивируемые клетки перекрываются, ядра могут быть замаскированы, что приводит к недооценке количества ядер. Двумерный характер количественной оценки ядер на изображениях исключает использование этой программы для определения общего числа ядер из клеток в суспензии, толстых тканевых блоках или других трехмерных приложениях. Тем не менее, эта программа все еще может количественно определять ядра, помеченные флуоресцентными красителями ДНК, на двумерных изображениях, полученных из трехмерных моделей, таких как толстые блоки тканей. Полезность количественного определения ядер в изображениях для желаемого приложения должна быть определена конечным пользователем.
| Проблема | Возможные причины | Возможные решения |
| Избыток фона на изображениях | Слишком большое время экспозиции | Сокращение времени воздействия |
| На изображениях ядра кажутся сливающимися друг с другом | Слишком высокая концентрация флуоресцентного ДНК-красителя или слишком большое количество среды для монтажа ДНК-красителя | Снижение концентрации красителя при оптимизации; Используйте минимально возможное количество монтажных носителей |
| Ядра кажутся слишком светлыми или некоторые ядра не видны | Слишком малое время экспозиции | Увеличьте время экспозиции |
| Фотоотбеливание | Предотвратите фотообесцвечивание, обеспечив хранение окрашенных образцов в темноте и сведя к минимуму воздействие на образцы света (например, используя минимально необходимое время экспозиции и отключая освещение, когда оно не требуется, например, после съемки изображения или во время заметок) | |
| Появление гало-эффектов или неспецифических для ядра форм | Слишком большое время экспозиции | Сокращение времени воздействия |
| Ворсинки от деликатных рабочих дворников или некоторых настольных протекторов в живых ячейках или при прикреплении к неподвижным ячейкам | Обеспечьте безворсовую среду: сведите к минимуму использование чувствительных рабочих стеклоочистителей при воздействии образцов (например, в шкафу биобезопасности для живых клеток, во время фиксации и сушки неподвижных клеток или в процессе окрашивания); Убедитесь, что настольные протекторы не оставляют ворсинок на горках или пластинах во время окрашивания | |
| Другие аномалии на поверхности неподвижных клеток/тканей | Обращайтесь с образцами с особой осторожностью и соблюдайте надлежащие меры предосторожности и передовые методы во время фиксации и окрашивания | |
| Помехи в световом тракте | Убедитесь, что все части микроскопа чисты и не содержат ворса и пыли; Перед визуализацией очистите слайды/тарелки/тарелки с 70% этанолом для удаления пыли и ворса из окружающей среды. | |
| Исполняемый файл не открывается | Антивирусное программное обеспечение заблокировало файл | Пометьте файл как безопасный |
| Ошибки в командной строке | Проблемы с интервалами в командной строке | Убедитесь, что начало команды вводится сразу после символа «>» в командной строке без пробела, что нет пробелов после открытых кавычек для путей к файлам, а также что интервалы и их соответствие приведенному коду и примерам. |
| Неправильные регистры, используемые в командной строке | Код чувствителен к регистру; Обязательно используйте заглавные и строчные буквы по мере необходимости | |
| Проблемы с путем к файлу | Убедитесь, что путь к файлу указывает на внутреннюю часть связанной папки с файлами (т. е. папка с файлами открыта при копировании пути к файлу). | |
| Проблемы с программой, которые не были решены с помощью описанных выше действий по устранению неполадок | Проблемы с программным обеспечением или командной строкой, отмеченные выше. | Отправьте сообщение о проблеме через GitHub. Перейдите в раздел https://github.com/rbudnar/nuclei_counter. В строке меню вверху выберите «Проблемы» (второй вариант, справа от «Код»). Нажмите зеленую кнопку справа с надписью «Новый выпуск», чтобы отправить заявку. Перед отправкой проблемы будет предложено войти в GitHub. Также будет возможность создать бесплатную учетную запись, если это необходимо. |
Таблица 1: Устранение неполадок для получения качественных изображений и выполнения автоматизированного рабочего процесса количественного определения ядер.
В целом, описанные здесь методы содержат пошаговые инструкции по использованию автоматизированной программы количественного определения ядер, которую мы разработали с использованием библиотек компьютерного зрения и проверили на основе ручных подсчетов от нескольких оценщиков. Эта программа была разработана для обеспечения удобства использования учеными с различными уровнями технологических навыков, связанных с программированием, находится в свободном доступе и была проверена специально для задачи количественного определения ядер. По сравнению с ручным подсчетом, эта программа сэкономит ценное время конечного пользователя и обеспечит точный и надежный подсчет ядер для нормализации данных.
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
Финансирование этой работы было предоставлено Национальными институтами здравоохранения/Национальным институтом по проблемам старения (R01AG084597; DEL и HYL) и за счет стартовых фондов Техасского технического университета (DEL). Авторы хотели бы поблагодарить программы Техасского технологического университета Undergraduate Research Scholars и TrUE Scholars за оказание финансовой поддержки студентам-исследователям, которые внесли свой вклад в эту работу (REH, MRD, CJM, AKW). Мы также благодарим докторов Лорен С. Голлахон и Майкла. Массетта за любезное предоставление их лабораторного пространства и оборудования.
Материалы
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
Ссылки
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены