Method Article
使用计算机视觉库简化细胞核定量
摘要
本文介绍了使用开源可执行程序自动进行基于图像的细胞核定量的分步方法,该程序在一系列细胞密度中进行了验证。该计划提供了一种替代方案,可以解决与成本相关的障碍、技术技能有限的用户的可访问性以及可能限制现有技术效用的特定应用程序验证。
摘要
活细胞检测和基于图像的细胞分析需要数据归一化才能准确解释。一种常用的方法是对细胞核进行染色和定量,然后将数据归一化为细胞核计数。该细胞核计数通常表示为单核细胞的细胞计数。虽然手动定量可能费力且耗时,但可用的自动化方法可能并非所有用户都喜欢,可能缺乏针对此特定应用的验证,或者可能成本高昂。在这里,我们提供了捕获用荧光 DNA 染料染色的细胞核的可量化图像的分步说明,然后使用使用 Python 计算机视觉库开发的自动对象计数软件程序对细胞核进行定量。我们还在一系列细胞密度中验证了该程序。尽管程序执行的确切时间因图像数量和计算机硬件而异,但该程序将数小时的细胞核计数工作合并为几秒钟,以便程序运行。虽然该方案是使用固定的染色细胞的图像开发的,但也可以使用该方案对活细胞和免疫荧光应用中染色细胞核的图像进行量化。最终,该程序提供了一个不需要高度技术技能的选项,并且是一种经过验证的开源替代方案,可帮助细胞和分子生物学家简化他们的工作流程,自动化繁琐且耗时的细胞核定量任务。
引言
功能和基于图像的实验对于了解实验性治疗对全细胞生物化学和生理学的影响至关重要。对细胞生物学实验数据的有效解释取决于实验方案的准确性和可重复性,包括数据归一化。例如,在基线和用特定药物治疗后分析活细胞的耗氧量和细胞外酸化率可以评估能量代谢的各个方面 1,2。测量细胞培养物上清液中乳酸脱氢酶等酶的活性有助于量化细胞膜完整性3。固定前用膜联蛋白 V 和碘化丙啶对培养的细胞进行染色,可以评估凋亡细胞和坏死细胞4。然而,孔间细胞密度的差异会影响这些检测的结果。由于接种细胞计数错误、接种时培养基中细胞密度的变化或整个实验过程中样品或处理之间的细胞生长速率不同,仅依赖接种密度可能会提供误导性结果。因此,需要对实验结果进行标准化。
目前功能和基于图像的细胞数据的归一化方法包括蛋白质浓度5 (protein concentration) 或细胞核或细胞计数6 (nuclei or cell count)。为了将数据标准化为蛋白质浓度,在检测运行后,必须在等体积的裂解缓冲液和蛋白酶抑制剂中提取细胞,并且必须进行额外的检测(例如,二辛可宁酸测定、Bradford 测定等)以进行蛋白质定量。这种方法的准确性取决于完整的细胞提取;遗漏任何蛋白质都会导致定量错误。细胞核或细胞计数提供了一种替代方案,无需从细胞生长的表面去除细胞。相反,使用比色或荧光染色剂对活细胞或固定细胞进行染色或复染,并且可以根据整个细胞或仅细胞核的图像进行计数。对于荧光显微镜应用以及标准化融合细胞(例如肌管)或组织中的数据,定量细胞核优于全细胞。为了使用荧光方法定量细胞核,将荧光 DNA 染料(例如,4′,6-二脒基-2-苯吲哚二氯化物 (DAPI)、Hoechst 33342、Hoechst 33358、DRAQ5 等)应用于活细胞或固定细胞,并使用荧光显微镜成像。DAPI、Hoechst 和 DRAQ5 优先与 DNA 中富含腺嘌呤-胸腺嘧啶的区域结合 7,8,9,提供了一种检测细胞核的方法。优化染料浓度可防止脱靶结合(即与线粒体 DNA 结合,导致点状胞质荧光)10。通过使用含有固定细胞 DAPI 的市售封片剂(例如,含 DAPI 的 VECTASHIELD 抗淬灭封片剂)和已公布的活细胞染色剂浓度(例如 Hoechst)6,11,12,可以简化优化过程。染色后,使用适当的激发滤光片(例如,紫外 [DAPI 和 Hoechst] 或远红 [DRAQ5])观察活细胞或固定细胞中的细胞核。DAPI、Hoechst 和 DRAQ5 的最大激发波长位于荧光显微镜光谱的任一端。这一特性,加上这些染料的 DNA 特异性及其在最佳浓度下的使用,可最大限度地减少脱靶荧光。所得图像在黑色背景下显示明亮的细胞核,这些细胞核被量化为单核细胞细胞数量的量度。虽然存在其他使用组织学染色和明场显微镜的基于染色的准确细胞核定量方法13,但使用这些方法实现自动化更加困难,尽管有可能。
虽然手动细胞核定量仍然是金标准,但手动细胞核定量费力、耗时,并且长时间计数容易出现潜在的人为错误。尽管存在自动细胞计数程序,但并非所有用户都喜欢它们,它们可能成本高昂,并且对特定应用的验证可能很少。近年来,Python 已成为生物学家宝贵且可访问的资源。Python 计算机视觉库对于图像分析特别有用14。在这里,我们提供了使用使用 Python 代码开发的可执行程序来量化用荧光染料染色并使用荧光显微镜成像的细胞核的分步说明。使用此处描述的程序不需要编码技能。该工作流程特定于应用了荧光 DNA 染料的细胞或组织。它不适用于明场图像。第 1 节描述了如何以与此工作流程兼容的方式捕获和保存使用荧光显微镜收集的图像。第 2 节提供了将细胞核定量程序作为可执行文件运行并获取输出的说明。可执行程序可以直接从提供的 .exe 文件运行,无需修改即可量化高质量图像中的细胞核。此可执行文件需要基于 Windows 的 PC。第 3 节提供了将程序作为 Python 脚本而不是可执行文件运行的说明(基于 Mac 或 Linux 的系统需要,基于 Windows 的系统可选),并允许根据需要调整代码。第 4 节介绍如何使用结果进行规范化。这些方案之后是具有代表性的结果,包括来自我们实验室的验证数据。
研究方案
注意:补充文件可在以下链接中找到 https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. 使用荧光显微镜捕获和保存图像
- 准备要成像的细胞或组织样品,包括用所需的 DNA 染料染色。为了获得此处使用的图像,将 C2C12 成肌细胞(CRL-1772,美国典型培养物收藏中心)在标准培养条件(5% CO 2,37°C,加湿)下在 6 孔板中生长 48-72 小时,有或没有 50 mM EtOH6,并如前所述固定在冰冷的甲醇中13。使用含有 DAPI 的封固剂15 封固固定细胞,并在成像前避光干燥。
- 根据既定的标准作程序打开荧光显微镜和计算机的所有组件。打开与显微镜关联的软件。
- 设置显微镜以通过目镜观察样品。检查目标。如果需要,将物镜转盘转到所需的物镜。确保软件程序选择了所需的物镜,以准确指示图像区域。在这里,图像是使用 10 倍物镜拍摄的。
- 关灯,将样品(载玻片、板或培养皿)放在载物台上。使用适当的滤镜打开实时成像,并将曝光时间设置为预先确定的持续时间或自动曝光。这里使用 DAPI 滤波器和 61-62 毫秒的曝光时间。有关设置曝光时间的指导,尤其是对于单元格密度较高的图像,请参阅故障诊断表。
- 使用目镜使用粗略调节旋钮缓慢调整焦距,直到可以看到染色的细胞核,然后使用微调旋钮缓慢调整焦距,直到细胞核清晰锐利。
注意: 图 1 显示了适当图像的示例。- 如果出现过多的背景染色或细胞核看起来非常暗淡,请手动调整曝光时间。在单个图像中,某些细胞核的信号强度可能存在明显的差异;但是,圆形应该对所有原子核都可见,并且信号干扰最小。
注:下游应用中使用的程序对不同的强度都很稳健。用于开发和验证细胞核定量程序的原始图像包括具有一系列信号强度的图像,可通过开放科学框架存储库获得(参见上述链接)。
- 如果出现过多的背景染色或细胞核看起来非常暗淡,请手动调整曝光时间。在单个图像中,某些细胞核的信号强度可能存在明显的差异;但是,圆形应该对所有原子核都可见,并且信号干扰最小。
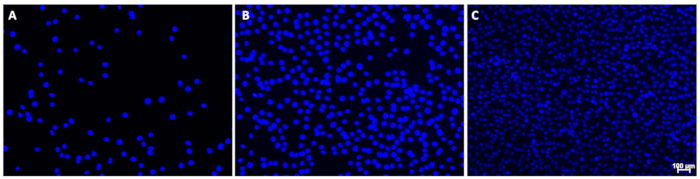
图 1:DAPI 染色细胞核的示例图像。 这些图像描绘了 (A) 低、(B) 中或 (C) 高细胞密度的细胞核,背景最小。请注意图像 C 上细胞核的信号强度变化;这不会影响定量。比例尺 (C) 表示 100 μm;用于细胞核定量的图像中应省略比例尺。 请单击此处查看此图的较大版本。
- 一旦细胞核通过目镜聚焦,将检测模式切换到相机。使用微调旋钮调整焦距。
- 如有需要,请调整曝光时间。记下用于后续样本和图像的最终曝光时间。
注意:尽管在所有图像中使用相同的曝光时间是理想的,但对于此应用程序,曝光时间的变化是可以接受的,因为荧光强度无法量化或比较样品之间的荧光强度。 - 使用软件上的相应图像捕获按钮捕获图像。如果样品应用了多个染色剂,请切换过滤器(例如,切换到 FITC、TRITC 或 Cy5)并对同一区域进行成像以进行合并。
- 应用快门以尽量减少样品暴露在光线下。无需在比例尺中刻录,而是将图像以通用图形文件格式(.tif、.png 或 .jpg)存储在专用文件夹中。确保每个图像文件名都具有适当的描述性,或者用于文件名的任何编码都有适当的链接文档。
注意:这将成为量化程序的图像目录。如果使用多色成像,请仅将细胞核的图像复制到单独的专用文件夹中。这也可以在成像会话结束时完成。在大多数成像软件上使用默认导出设置导出的图像通常具有足够的分辨率,可用于此程序,前提是已捕获高质量的图像。但是,导出后应目视检查图像,以确保细胞核的形态清晰。如果图像以 .jpg 格式保存,则应注意确保图像不会过度压缩,并保持质量。 - 打开快门,返回实时成像,然后扫描样品到另一个位置进行成像。根据需要重复步骤 1.8-1.9。对于以 10 倍放大倍率捕获的 6 孔板中接种的细胞图像,每孔捕获 10-15 张图像(取决于整个样品区域细胞密度的一致性)。
- 采集单个样品的所有图像后,执行以下选项之一。
- 继续下一个示例并重复步骤 1.4-1.10。
- 暂停协议并在其他时间返回。样品应避光保存。如果处理活细胞,则不建议使用此选项。
- 成像完成后,在打开房间灯之前,请确保所有样品都受到遮光保护。如果包含图像的文件夹保存在本地,请将文件传输到 U 盘、云存储位置或将运行量化程序的计算机可访问的其他网络驱动器。保存在专用文件夹中。
- 根据既定的标准作程序关闭显微镜和计算机的所有部件。
- 成像现已完成。如第 2 节所述,使用自动定量程序分析文件,或者,如第 3 节所述,将程序作为 Python 脚本运行。
2. 将自动细胞核定量程序作为可执行文件运行
注意:此方法将程序作为可执行文件运行,与基于 Windows 的 PC 兼容,并且是 Windows作系统用户的推荐方法。Mac OS 或其他非 Windows 用户应将程序作为 Python 脚本运行(请参阅第 3 节)。
- 确保计算机上安装了 Python。如果需要,请从 python.org/downloads(首选)或使用 Anaconda 或 Miniconda 安装 Python。截至发布之日,该程序已使用 Python 版本 3.10-3.12 进行了测试。
- 打开浏览器并导航到 https://github.com/rbudnar/nuclei_counter/releases。下载名为 count_nuclei.zip 的文件的最新版本。
- 从 Downloads 文件夹中,右键单击 Zip 文件 并将 Extract All files 解压到本地计算机上的所需位置(参见 图 2A)。保持此文件位置处于打开状态;此文件路径将在步骤 2.5 中使用(参见 图 2B)。
注意:Count_nuclei现在应显示为应用程序文件。这是可执行文件。 - 通过搜索 CMD 或搜索栏中的命令提示符打开命令提示符。使用 cd 命令将目录更改为可执行文件的文件路径(参见 图 2C),然后按 Enter。示例命令行:C:\Users\janedoe>cd [cd 后面的空格,然后复制/粘贴文件路径]
- 在下一个命令行中,键入以下内容,然后按 Enter。
count_nuclei.exe --img_dir “路径/到/图像” --output_dir “路径/到/输出” --save_images --output_count_filename “results.csv”
其中 path/to/images 是包含要分析的图像的文件夹的文件路径(请参阅步骤 1.14);path/to/output 是应保存.csv文件的文件夹的文件路径(如果未提供,则默认为当前工作目录);results.csv 是结果计数将保存到的文件名(如果未提供,则输出文件名默认为 count_results.csv)。
注意:命令行中需要引号。确保文件路径前的引号后没有空格(参见 图 2C)。 - 当下一个命令行出现时,处理完成。轮廓和结果电子表格现在位于步骤 2.5 中指定为输出目录的位置(即,path/to/output;参见 图 2C)。
- 目视检查轮廓以验证计数质量。结果现在可用于标准化数据,如第 4 节所述。
注意:如果等值线似乎遗漏了细胞核(例如,细胞核看起来太暗)或将细胞核组聚集到一个项目中(例如,图像曝光过度、背景过多、伪影),则用户应参考故障排除表,并且可能需要返回第 1 节以优化图像采集,然后再继续第 4 节。
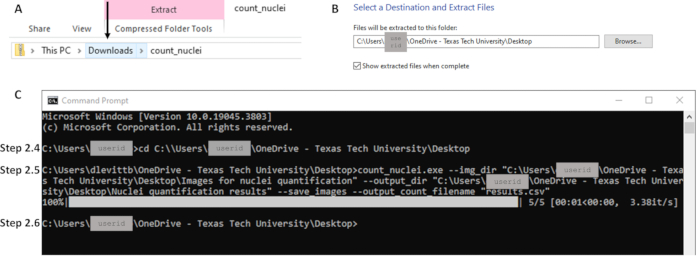
图 2:第 2 节工作流程的关键部分示例。 (A) 从 Downloads 文件夹中提取文件,注意单击箭头指示的正确文件夹,以在步骤 2.3 中将 (B) 提取到所需的目的地。(C) 步骤 2.4 和 2.5 的命令示例,以及指示步骤 2.6 中的完成的下一个命令行。请注意,步骤 2.4 (C) 中的文件路径与步骤 2.3 (B) 中的提取位置匹配。 请单击此处查看此图的较大版本。
3. 以 Python 脚本形式运行自动细胞核定量程序
- 确保计算机上安装了 Python。如果需要,请从 python.org/downloads(首选)或使用 Anaconda 或 Miniconda 安装 Python。截至发布之日,该程序已使用 Python 版本 3.10-3.12 进行了测试。
- 打开浏览器并导航到 https://github.com/rbudnar/nuclei_counter。单击 Green Code 按钮,然后单击 Download ZIP 以下载代码存储库(参见 图 3A)。
- 对于 Windows作系统,请按照以下说明进行作。请参阅步骤 3.4 对于基于 Mac OS 的计算机。
- 从 Downloads 文件夹中,右键单击 File 并选择 Extract All 将文件提取到本地计算机(参见图 3B-C)。
- 在解压缩的文件位置,打开名为 nuclei_counter-main 的文件夹。这是代码仓库。
注意:这只需要在第一次使用此程序时下载;只需将提取的文件移动到可访问的网络位置并记下该位置即可。 - 通过搜索 CMD 或搜索栏中的命令提示符打开命令提示符。如果 Python 已由 Anaconda 或 Miniconda 安装,请打开 Anaconda 提示符,而不是标准命令提示符。这可以通过在搜索栏中键入 Anaconda Prompt 来找到。
- 使用 cd 命令将目录更改为代码存储库,然后按 Enter。请参阅下面的代码行,该代码行区分大小写。确保在复制文件路径时打开 nuclei_counter-main 的文件夹;名为 pyproject 或 pyproject.toml 的文件应该是可见的(参见 图 3D)。示例命令行:C:\Users\janedoe>cd [cd 后面的空格,然后复制/粘贴文件路径]
- 在下一个命令行中,键入以下内容,包括空格和句点(参见 图 3D:
pip install -e 的 .
注意:步骤 3.3.5 和 3.3.6 只需在任何计算机上首次运行此程序时完成。如果已完成此作,请继续执行步骤 3.3.7。初始化安装过程可能需要一些时间。 - 按 Enter 键。运行此命令会安装运行此脚本所需的库,并在需要调整代码的情况下以可编辑模式安装存储库(参见 图 3D)。
- 在下一个命令行中键入以下内容,将目录更改为主源代码目录(参见 图 3E):
cd “nuclei_counter” - 在下一个命令行中,键入以下内容并按 Enter (参见 图 3E):
python count_nuclei.py --img_dir “路径/到/图像” --output_dir “路径/到/输出” --save_images --output_count_filename “results.csv”
其中 path/to/images 是包含要分析的图像的文件夹的文件路径(步骤 1.14);path/to/output 是应保存.csv文件的文件夹的文件路径(如果未提供,则默认为当前工作目录);results.csv 是结果计数将保存到的文件名(如果未提供,则输出文件名默认为 count_results.csv)。
注意:命令行中需要引号。确保文件路径前的引号后没有空格。python 脚本将读取提供的文件路径中的文件,并为目录中的所有图像执行原子核计数程序(参见 图 3E)。结果计数将保存到指定位置的结果.csv文件中。 - 当下一个命令行出现时,处理完成(参见 图 3E)。轮廓和结果电子表格现在位于步骤 3.3.8 中指定为输出目录的位置(即 path/to/output)。
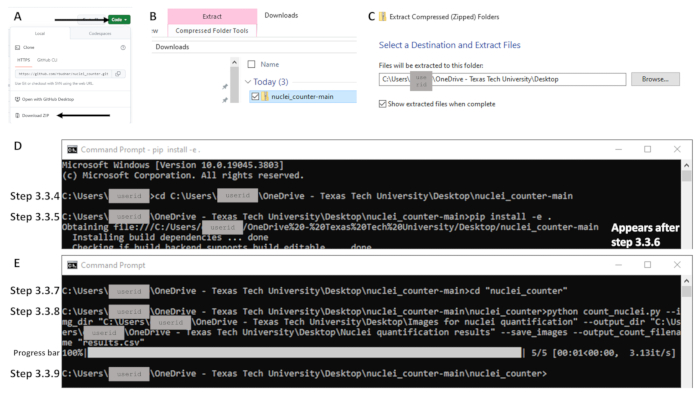
图 3:使用 Windows作系统的第 3 部分工作流程的关键部分示例。 要访问代码,请单击标有 Code 的绿色按钮,然后 (A) 下载 Zip 文件(由箭头指示)。(B) 右键单击 Zip 文件(以蓝色显示)进行解压,并 (C) 设置所需的目标。(D) 打开命令提示符后,步骤 3.3.4 和 3.3.5 的命令示例。请注意,步骤 3.3.5 的命令都在一行上;按下 Enter 键后,下一行开始的文本 “Obtaining file” 出现(步骤 3.3.6;即运行命令)。(E) 步骤 3.3.7 和 3.3.8 的命令示例、执行步骤 3.3.8 后的进度条以及显示步骤 3.3.9 中完成的命令行。 请单击此处查看此图的较大版本。
- 对于 Mac OS,请按照以下说明进行作。
- 从 Downloads 文件夹中,单击 File 菜单并选择 Open 将文件提取到本地计算机(参见 图 4A)。
- 在解压缩的文件位置,打开名为 nuclei_counter-main 的文件夹。这是代码仓库。
注意:这只需要在第一次使用此程序时下载;只需将提取的文件移动到可访问的网络位置并记下该位置即可。 - 按 command+space bar 打开终端,打开 Spotlight;然后,搜索 “Terminal”。
- 使用 cd 命令将目录更改为代码存储库,然后按 Enter。请参阅下面的代码行,该代码行区分大小写。在文本文档中键入文件路径并将其复制到终端中。确保文件路径以 cd 前面的文本开头(例如, 图 4B 中的 /Users/userid)。命令行示例:/Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- 在下一个命令行中,User ID 后面将出现一个美元符号(参见 图 4B)。确保美元符号后有一个空格,然后键入以下内容,包括命令末尾的空格和句点。在 pip 后面立即包含适当的 Python 版本,不带空格: pip3 install -e 。
注意:步骤 3.4.5 和 3.4.6 只需在任何计算机上首次运行此程序时完成。如果已完成此作,请继续执行步骤 3.4.7。初始化安装过程可能需要一些时间。 - 按 Enter 键。运行此命令会安装运行此脚本所需的库,并在需要调整代码的情况下以可编辑模式安装存储库。
- 在下一个命令行中,确保美元符号后面有一个空格,然后键入以下内容以将目录更改为主源代码目录(参见 图 4C):
cd “nuclei_counter” - 在下一个命令行中,键入下面的命令行。如果使用 3 以外的版本,请将 Python 后面的数字替换为正确的版本号。然后,按 Enter (参见 图 4C):
python3 count_nuclei.py --img_dir “路径/到/图像” --output_dir “路径/到/输出” --save_images --output_count_filename “results.csv”
其中 path/to/images 是包含要分析的图像的文件夹的文件路径(步骤 1.14);path/to/output 是应保存.csv文件的文件夹的文件路径(如果未提供,则默认为当前工作目录);results.csv 是结果计数将保存到的文件名(如果未提供,则输出文件名默认为 count_results.csv)。
注意:命令行中需要引号。确保文件路径前的引号后没有空格。Python 脚本将读取提供的文件路径中的文件,并为目录中的所有图像执行细胞核计数程序(参见 图 4C)。结果计数将保存到指定位置的结果.csv文件中。 - 当下一个命令行出现时,处理完成(参见 图 4C)。轮廓和结果电子表格现在位于步骤 3.4.8 中指定为输出目录的位置(即 path/to/output)。
- 目视检查轮廓以验证计数质量。结果现在可用于标准化数据,如第 4 节所述。
注意:如果等值线似乎遗漏了细胞核(例如,细胞核看起来太暗)或将细胞核组聚集成一个项目(例如,图像曝光过度、背景过多、伪影),用户应参考 表 1 ,并且可能需要返回第 1 节以优化图像采集,然后再继续第 4 节。
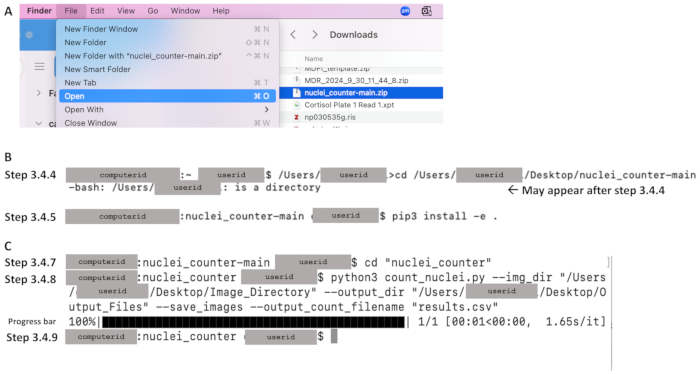
图 4:使用 Mac OS 的第 3 部分工作流程的关键部分示例。 下载代码后(参见 图 3A),(A) 通过单击 Zip 文件(以蓝色显示)并设置所需的目标进行提取。(B) 打开终端后,步骤 3.4.4(将目录更改为代码仓库)和 3.4.5(安装 pip)的命令示例。(C) 步骤 3.4.7 和 3.4.8 的命令示例、执行步骤 3.4.8 后的进度条以及显示步骤 3.4.9 中的完成的命令行。 请单击此处查看此图的较大版本。
4. 使用计数对实验数据进行归一化
- 打开 results.csv 文件,将每个图像与使用自动程序确定的细胞核定量链接起来。
- 确定归一化方法:1) 逐个图像基础(例如,如果是多色荧光显微镜),2) 将数据归一化为每个样品的平均细胞核数与参考样品(或条件)的比率,3) 归一化为每个样品的细胞核数(例如,将总细胞核数外推到样品区域)。有关规范化方法的建议,请参见 Discussion 部分。
- 如果要逐张图像进行归一化,只需将每张图像的实验数据除以该图像的细胞核数即可。
- 要按比率规范化数据,请按照下面描述的步骤作。
- 对于为单个重复收集多个图像的任何样品(例如,从 6 孔板中的单个孔捕获的 10-15 张图像),计算每个图像的平均细胞核数。这些细胞核计数被认为是假设单核细胞或在多核细胞(例如肌管)中融合在一起的细胞计数。
- 选择参比样品或参比条件(例如,对照条件中所有样品的平均计数)并记下计数。
- 将所有样本的计数除以该引用计数,得到一个标准化值。如果使用了参考样本,则使用参考样本的标准化值为 1。
注意:如果使用了参考条件,则该条件的平均规范化值现在为 1。细胞核数少于参比值(即密度较低)的样品的归一化值将小于 1,而细胞核数多于参比细胞核(即密度更大)的样品的归一化值将大于 1。 - 将每个孔的实验数据除以相应的计算归一化值。
- 要将数据归一化为每个样品的细胞核计数,请按照以下步骤作。
- 对于为单个重复收集多个图像的任何样品(例如,从 6 孔板中的单个孔捕获的 10-15 张图像),计算每个图像的平均细胞核数。这些细胞核计数被认为是假设单核细胞的细胞计数,或在多核细胞(例如肌管)中融合在一起的细胞数。
- 确定从中获取数据的载玻片(盖玻片)、孔或培养皿的表面积。此信息可在商品网站的规格部分找到。
- 确定每个捕获的图像的面积。此信息通常作为成像或分析软件中的元数据提供。
- 确保样品表面积和图像面积以相同的单位表示(例如,mm2)。如果没有,请转换为所需的单位。
- 使用以下公式计算每个样本的总数:
TC = 交流 • (SA/IA)
其中 TC 是总数,AC 是平均计数,SA 是表面积,IA 是图像面积。 - 将实验数据除以样品的相应 TC。
- 对实验数据进行标准化,并酌情用于统计分析。
结果
每次批量图像运行都会产生:1) 一组应用了轮廓的图像文件,显示已识别细胞核的轮廓(图 5),以及 2) 一个链接图像文件名和相关计数的.csv文件(电子表格)。查看等值线将允许用户直观地评估计数质量。具体来说,根据第 1 节获得的图像应具有所有(或几乎所有)细胞核被一条绿色实线包围,表明程序对细胞核进行了计数。这些等值线用于调整此处的程序。
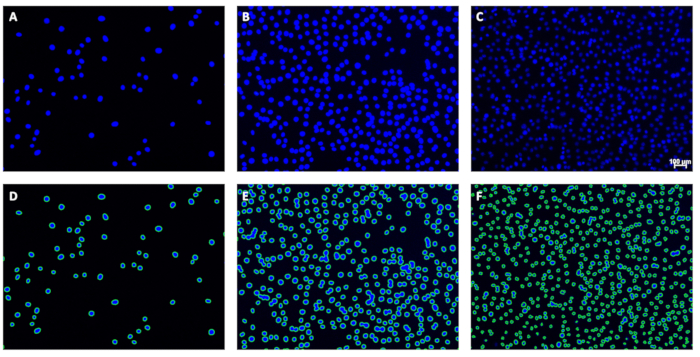
图 5:原始图像和相应轮廓的示例。 (A-C)分析前的原始图像示例和分析后 (D-F) 相应的等值线示例。比例尺 (C) 表示 100 μm;用于细胞核定量的图像中应省略比例尺。 请单击此处查看此图的较大版本。
使用初始结果进行验证
将不同细胞密度的 C2C12 成肌细胞用甲醇固定,在封固剂中用 DAPI 复染,并以 10 倍放大倍率获得 .jpg 文件格式的 120 张图像。运行自动化程序,将 Image Directory 设置为包含这些图像的文件夹,并保存生成的 .csv 文件。另外,每张图像都由两名训练有素的研究人员手动量化,他们对自动计数和彼此的计数视而不见。开放科学框架 (OSF) 存储库中提供了原始图像、由自动程序生成的等高线以及包括手动和自动计数的结果电子表格(参见第 1 节中的链接)。使用 SPSS Statistics 计算类内相关系数 (ICC;平均测量),以确定 1) 两个手动计数和 2) 两个手动计数的平均值和使用自动程序获得的计数之间的评分者间可靠性。类似的方法已被用于验证其他旨在量化使用荧光显微镜获得的图像中的对象的程序 16,17,18。还在每个四分位数的平均手动计数和自动计数之间计算 ICC,以确保评估整个数据集不会掩盖基于细胞密度的可靠性的任何潜在变化。根据平均手动计数,使用第 25、 50和第 75个百分位分割点定义四分位数,范围从四分位数 1 (Q1;最低细胞密度) 到 Q4 (最高细胞密度)。ICC 根据平均测量值报告,并根据以下定义进行解释:差 (ICC <0.50)、中度 (0.50 ≤ICC <0.75)、良好 (0.75 ≤ICC <0.90) 或极好 (ICC ≥0.90)19。
根据手动计数平均值,每张图像的细胞核数范围为 44.5 至 1160。两个手动计数之间的评分者间可靠性非常好 (ICC > 0.999,p < 0.0001;图 6A)以及平均手动和自动计数之间 (ICC = 0.993,p < 0.0001;图 6B)。四分位数的可靠性仍然非常出色(Q1:ICC = 0.996,p < 0.0001;Q2:ICC= 0.997,p < 0.0001;Q3:ICC = 0.998,p < 0.0001;Q4:ICC = 0.986,p < 0.0001;图 6C-F)。远离图 6B 中最佳拟合线的单个数据点表示图 7 中所示的图像。图像包含多个细胞核的多个区域,这些区域组合在一起,从而阻止计算机视觉程序识别每个细胞核。这个问题可以通过使用较短的曝光时间来解决。表 1 描述了如何处理此类镜像。
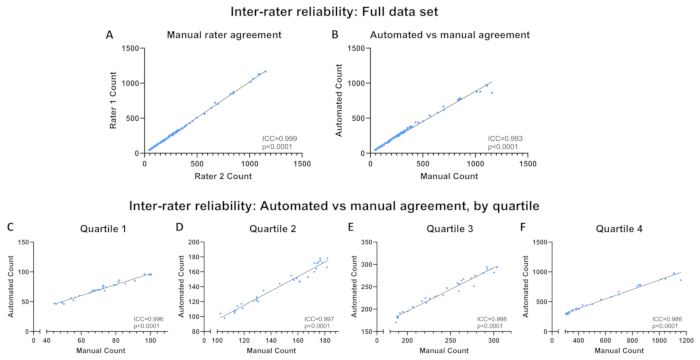
图 6:评分者间可靠性。整个数据集 (A) 之间的手动评分者计数和 (B) 自动计数和平均手动计数之间的评分者间可靠性。(C-F)自动计数与按四分位数的平均手动计数之间的评分者间可靠性。可靠性表示为类内相关系数 (ICC)。请单击此处查看此图的较大版本。

图 7:准确细胞核定量存在两个障碍的图像示例。 (A) 原始图像和 (B) 相关轮廓。箭头 1 表示多个细胞核聚集在一起的区域,圆形细胞核形状扭曲到足以阻碍软件对多个细胞核的检测。这发生在此图像中的多个位置。箭头 2 表示出现在多个原子核后面的褪色光晕;这个光晕看起来足够亮,以至于软件可以将其作为一个原子核包含进来,从而失去了它周围的多个原子核的数量。该图表示与 图 6B 中的最佳拟合线相去甚远的单个数据点,强调了这些障碍的潜在问题。比例尺 (A) 表示 100 μm;用于细胞核定量的图像中应省略比例尺。 请单击此处查看此图的较大版本。
讨论
与现有选项相比,我们的细胞核定量程序具有几个优势:它只需要最少的技术技能,针对细胞核定量的特定任务进行了验证,并且是开源的;后者克服了与成本相关的障碍。最终,该程序为细胞和分子生物学家提供了另一种选择,可以快速准确地量化使用荧光显微镜捕获的图像中的细胞核。目前可用的自动细胞核或细胞计数程序并非所有用户都喜欢,有些程序成本高昂,并且对于特定应用可能很少或没有验证。BioTek Cytation 系列等专用设备随附技术公告,可验证使用细胞核染色20 进行原位细胞计数,以及各种其他应用。虽然存在验证数据并且 Cytation 产品线被广泛使用,尤其是与 Seahorse 细胞外通量分析仪连接以轻松有效地标准化活细胞数据时,该产品对于可用设备资金有限的实验室来说可能成本高昂。例如,在 2024 日历年,配备四个物镜和四个荧光显微镜滤光片立方体的基本型号的标价超过 100,000 美元。对于那些无法购买专用设备的用户,开源软件为图像分析提供了免费的替代方案。可用于细胞核和细胞定量的开源软件包括 ImageJ(美国国立卫生研究院,美国马里兰州贝塞斯达;可在 21.插件现在可供希望使用 ImageJ 平台为图像处理自动化编写宏脚本的用户使用。之前已经描述了使用相差或明场显微镜和全细胞图像的基于 ImageJ 的细胞计数自动化22,23。CellProfiler 是一种广泛发表的应用程序,用于分析用荧光染料或标准组织学染色剂染色的细胞图像,描述其在一系列生物学应用中的效用的早期文章通常被称为验证24。然而,这份手稿并未验证任何特定的生物学发现,因此需要对使用的各种自动化管道进行额外的验证和透明度,例如用于细胞核计数25。此外,虽然 CellProfiler 是一个高效的细胞分析软件包,但并非所有科学家都认为它对用户友好。因此,我们的目标是开发和验证一种替代的自动化程序,以使用开源软件量化细胞核。具体来说,我们利用了 Python 中现有的计算机视觉库来开发此工作流程。
该方法从最终用户使用 DNA 染料(如 DAPI26)染色的细胞或组织开始。提供了以与自动化软件兼容的方式捕获和保存图像的一般准则。尽管不同的显微镜和成像软件的细节可能有所不同,但第 1 部分的一般工作流程是相同的,以获得可量化的图像。如果使用活体样品,则必须注意确保佩戴适当的个人防护装备,并且成像环境适合这些样品(例如,温度、CO2 百分比、添加缓冲液等)27. 虽然该协议仅描述了 DNA 染料的成像,但用户可以捕获同一区域的其他颜色以进行合并15,只需将细胞核图像复制到单独的文件夹中即可用于定量程序。应在暗室中进行成像,并为活体样本提供适当的 PPE,并应根据需要进行适当的消毒(例如,使用 70% 乙醇)。在打开灯之前,应避光处理染色的样品。高质量的图像捕获对于准确的细胞核定量至关重要。即使在具有适当曝光时间且明亮细胞核与黑色背景之间形成强烈对比的图像中,过度聚集的细胞或细胞群后面的光晕(参见 图 7)也可能会降低定量准确性。在这些情况下,可能需要改变曝光时间以降低信号强度,确保最小地减少细胞核信号的合并并最大限度地减少光晕,同时保持足够强的信号来检测细胞核的圆形。如果这些步骤不足,则可能需要对样品的相邻部分进行成像。单个实验的所有图像必须以相同的放大倍率捕获,并且必须在成像软件中正确设置与所用物镜相对应的放大倍率。当细胞核计数将从成像区域外推到整个孔(或载玻片)区域时,用于成像的物镜必须与成像软件上的设置相匹配尤为重要。每次切换物镜时都应执行此步骤,但在设置显微镜和软件时很容易忽略。第 1 部分所需的时间因多种因素而异(例如,荧光显微镜的用户体验、图像和样品的数量、显微镜等)。要在两个 6 孔板中每孔捕获 10-15 张染色细胞核的图像,用户应保留大约 1-2 小时的时间。
一旦图像被捕获并保存在专用文件夹中,就可以进行量化了。最简单的量化方法是运行这个预打包的自动化程序,而无需安装 Python 或任何库(第 2 节)。应该注意的是,该程序是作为可执行文件准备的,并使用 Pyinstaller 打包。使用 Pyinstaller 构建的软件通常由防病毒软件标记。如果防病毒软件阻止打开文件,则用户应将该文件标记为可以安全继续。这对于成功执行第 2 部分至关重要。或者,如果需要,任何用户都可以选择将自动定量程序作为 Python 脚本(第 3 节)而不是可执行文件运行。如果用户希望使用 Mac 或 Linux作系统运行程序,则必须通过 Python 运行该程序。这种方法不需要 Pyinstaller,但可能在技术上更具挑战性。第 3 节中详述的分步协议和示例旨在消除将此程序作为脚本运行的技术障碍。无论使用 section 2 还是 section 3 来运行程序,确保将代码正确输入到命令提示符中都至关重要。整个协议中都包含示例代码行,用户应特别注意间距、引号和其他标点符号。用户还应确保文件路径准确无误。在 Word 文档中提供图像和输出的文件路径会很有帮助。命令也可以键入到此 Word 文档中,并根据需要复制/粘贴。
如第 4 节所述,有多种方法可以使用从该自动细胞核定量程序获得的计数来标准化实验数据。对于基于图像的实验,其中实验数据包括每个图像中的多个事件并且需要一个比例(例如,膜联蛋白 V 阳性细胞的比例28、表达 ACE2 受体的细胞比例29、摄取染色颗粒的细胞数量30 等),每个图像的实验数据可以简单地除以结果计数(产生从 0 到 1 的比例)。然后,可以计算来自同一载玻片、培养皿或孔的复制图像的平均比例。如果需要百分比,只需将最终比例乘以 100 即可。最后,这些平均比例或百分比可用于下游统计分析。当要归一化的实验数据来自活细胞中的功能实验(例如,耗氧率 6,11,12)或来自细胞培养上清液(例如,上清液31 中的乳酸脱氢酶活性或特定分泌的细胞因子32 的浓度等)时,应首先对从同一孔捕获的所有图像的细胞核计数进行平均。然后,可以将细胞核计数外推到载玻片(盖玻片)、孔或培养皿的表面积,或者可以将计数表示为与参考样品或条件的比率。外推到载玻片、孔或培养皿的表面积有助于表达实验数据与得出数据的细胞数量(如果使用单核细胞)的关系(例如,每 103 个细胞每分钟消耗的氧气)。要使用这种方法,必须知道载玻片(盖玻片)、孔或培养皿的表面积以及每个图像中捕获的面积。如果样品未覆盖整个可用表面积,实验数据来自少量上清液(即,表示为每单位体积的活性或浓度),或者图像和/或表面积未知,则最佳选择是平均单个样品所有图像的细胞核计数,并表示相对于参考样品或条件的细胞核计数。结果值用于校正细胞核密度或细胞密度的实验数据(如果使用单核细胞)。
虽然这种方法易于执行,但对于哪些用户应该认识到,存在几个潜在问题。 表 1 列出了这些潜在问题以及可能的原因和解决问题的故障排除步骤。与现有软件相比,此处描述的程序具有关键优势;但是,必须承认一些限制。主要缺点是定量程序只能用于使用荧光显微镜捕获的图像,而不能用于明场图像。虽然使用以 10 倍放大倍率捕获的 DAPI 染色图像来构建该程序,但该程序可以与其他 DNA 染料和放大倍数一起使用,因为该程序旨在识别细胞核的形态,而不是颜色或大小。虽然我们在包含各种细胞密度的图像上验证了该程序的准确性,但最终用户的细胞密度可能超出范围。在这种情况下,如果获得高质量的图像,我们仍然预计会有准确的计数;但是,用户应仔细检查轮廓和输出以确保准确性。对于细胞培养应用,图像中细胞核的定量最适合单层细胞。如果培养的细胞重叠,细胞核可能会被掩盖,从而导致细胞核数量被低估。量化图像中细胞核的二维性质排除了使用该程序来确定悬浮液、厚组织块或其他三维应用中细胞核总数的可能性。然而,该程序仍然可以量化从 3D 模型(如厚组织块)捕获的二维图像中用荧光 DNA 染料标记的细胞核。对于所需应用,对图像中的细胞核进行定量的效用应由最终用户决定。
| 问题 | 可能原因 | 可能的解决方案 |
| 图像中的背景过多 | 曝光时间过长 | 减少曝光时间 |
| 细胞核在图像中似乎彼此融合 | DNA 染料荧光浓度过高或过多DNA 染料封片剂 | 优化过程中降低染料浓度;使用尽可能少的封固剂 |
| 细胞核看起来太亮或某些细胞核不可见 | 曝光时间过短 | 增加曝光时间 |
| 光漂白 | 通过确保染色样品保持黑暗中储存并尽量减少样品暴露在照明下(例如,使用必要的最短曝光时间,并在不需要时关闭照明,例如在捕获图像后或做笔记时)来防止光漂白 | |
| 出现光晕效果或非原子核特定形状 | 曝光时间过长 | 减少曝光时间 |
| 活细胞中或粘附在固定细胞上的精密任务刮水器或某些台式保护装置产生的棉绒 | 确保无绒环境:样品暴露时(例如,在活细胞的生物安全柜中、固定细胞的固定和干燥期间或染色过程中),尽量减少使用精密的任务擦拭布;确保台式保护膜在染色过程中不会在载玻片或板上留下棉绒 | |
| 固定细胞/组织表面的其他异常 | 在固定和染色过程中,请极其小心地处理样品,并遵循适当的预防措施和最佳实践 | |
| 光路中的干扰 | 确保显微镜的所有部件都干净且没有棉绒和灰尘;成像前用 70% 乙醇清洁载玻片/板/培养皿的外部,以去除环境灰尘和棉绒 | |
| 可执行文件无法打开 | 防病毒软件阻止了该文件 | 将文件标记为安全 |
| 命令提示符中的错误 | 命令行中的间距问题 | 仔细检查命令的开头是否紧跟在命令行中 “>” 符号之后键入,没有空格,文件路径的左引号后没有空格,以及空格是否与提供的代码和示例匹配。 |
| 命令行中使用的不正确的情况 | 代码区分大小写;请务必酌情使用大写和小写字母 | |
| 文件路径问题 | 确保文件路径指示关联文件夹的内部(即,复制文件路径时文件夹处于打开状态)。 | |
| 使用上述故障排除步骤无法解决的程序问题 | 上述软件或命令行问题。 | 通过 GitHub 提交问题。导航到 https://github.com/rbudnar/nuclei_counter。在顶部的菜单栏中,选择 “Issues” (第二个选项,位于 “Code” 右侧)。点击右侧显示 “New Issue” 的绿色按钮提交。在提交问题之前,将提示登录 GitHub。如果需要,还可以选择创建免费帐户。 |
表 1: 用于捕获高质量图像和运行自动细胞核定量工作流程的故障排除。
总体而言,本文描述的方法提供了使用自动细胞核定量程序的分步详细信息,该程序我们使用计算机视觉库开发,并根据来自多个评分者的手动计数进行了验证。该程序的开发是为了确保具有不同水平编码相关技术技能的科学家的可用性,可以免费获得,并且专门针对细胞核定量任务进行了验证。与手动计数相比,该程序将为最终用户节省宝贵的时间,并为数据归一化提供准确可靠的细胞核计数。
披露声明
作者声明没有利益冲突。
致谢
这项工作的资金由美国国立卫生研究院/国家老龄化研究所 (R01AG084597;DEL 和 HYL)以及德克萨斯理工大学 (DEL) 的启动资金。作者要感谢德克萨斯理工大学本科生研究学者和 TrUE 学者计划为为这项工作做出贡献的本科研究人员(REH、MRD、CJM、AKW)提供财政支持。我们还感谢 Lauren S. Gollahon 博士和 Michael P. Massett 博士慷慨地分享他们的实验室空间和设备。
材料
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
参考文献
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。