Method Article
Utilizzo delle librerie di visione artificiale per semplificare la quantificazione dei nuclei
In questo articolo
Riepilogo
Questo articolo descrive i metodi passo dopo passo per automatizzare la quantificazione dei nuclei basata su immagini utilizzando un programma eseguibile open source convalidato in una gamma di densità cellulari. Questo programma fornisce un'alternativa che affronta le barriere relative ai costi, all'accessibilità per gli utenti con competenze tecnologiche limitate e alla convalida specifica dell'applicazione che può limitare l'utilità delle tecnologie esistenti.
Abstract
I saggi su cellule vive e le analisi cellulari basate su immagini richiedono la normalizzazione dei dati per un'interpretazione accurata. Un metodo comunemente usato consiste nel colorare e quantificare i nuclei, seguito dalla normalizzazione dei dati per il conteggio dei nuclei. Questo conteggio dei nuclei è spesso espresso come conteggio delle cellule per le cellule uninucleate. Sebbene la quantificazione manuale possa essere laboriosa e dispendiosa in termini di tempo, i metodi automatizzati disponibili potrebbero non essere preferiti da tutti gli utenti, potrebbero non essere convalidati per questa specifica applicazione o potrebbero essere proibitivi in termini di costi. Qui, forniamo istruzioni dettagliate per acquisire immagini quantificabili di nuclei colorati con colorazioni di DNA fluorescenti e successivamente quantificare i nuclei utilizzando un programma software di conteggio automatico degli oggetti sviluppato utilizzando librerie di visione artificiale Python. Convalidiamo anche questo programma in una gamma di densità cellulari. Sebbene il tempo esatto per l'esecuzione del programma vari in base al numero di immagini e all'hardware del computer, questo programma consolida le ore di lavoro contando i nuclei in secondi per l'esecuzione del programma. Sebbene questo protocollo sia stato sviluppato utilizzando immagini di cellule fissate e colorate, anche le immagini di nuclei colorati in cellule vive e le applicazioni di immunofluorescenza possono essere quantificate utilizzando questo programma. In definitiva, questo programma fornisce un'opzione che non richiede un alto grado di competenza tecnologica ed è un'alternativa convalidata e open source per aiutare i biologi cellulari e molecolari a semplificare i loro flussi di lavoro, automatizzando il compito noioso e dispendioso in termini di tempo della quantificazione dei nuclei.
Introduzione
Gli esperimenti funzionali e basati su immagini sono fondamentali per comprendere l'impatto dei trattamenti sperimentali sulla biochimica e sulla fisiologia dell'intera cellula. Una valida interpretazione dei dati provenienti da esperimenti di biologia cellulare dipende dall'accuratezza e dalla riproducibilità del protocollo sperimentale, compresa la normalizzazione dei dati. Ad esempio, le analisi del consumo di ossigeno e dei tassi di acidificazione extracellulare in cellule vive al basale e dopo il trattamento con farmaci specifici consentono di valutare vari aspetti del metabolismo energetico 1,2. La misurazione dell'attività di enzimi come la lattato deidrogenasi nel surnatante delle colture cellulari può aiutare a quantificare l'integrità della membrana cellulare3. La colorazione delle cellule in coltura con annessina V e ioduro di propidio prima della fissazione consente la valutazione delle cellule apoptotiche e necrotiche4. Tuttavia, le differenze nella densità cellulare tra i pozzetti influenzano i risultati di ciascuno di questi saggi. Affidarsi esclusivamente alla densità di semina potrebbe fornire risultati fuorvianti a causa di errori nel conteggio delle cellule per la semina, variazioni nella densità cellulare nei terreni durante la piastratura o diversi tassi di crescita cellulare tra campioni o trattamenti durante gli esperimenti. Pertanto, è necessaria la normalizzazione dei risultati sperimentali.
Gli attuali metodi di normalizzazione per i dati cellulari funzionali e basati su immagini includono la concentrazione di proteine5 o i nuclei o la conta cellulare6. Per normalizzare i dati sulla concentrazione proteica, le cellule devono essere estratte in volumi uguali di tampone di lisi con l'inibitore della proteasi dopo l'esecuzione del test e deve essere eseguito un test aggiuntivo (ad esempio, test dell'acido bicinconninico, test di Bradford, ecc.) per la quantificazione delle proteine. L'accuratezza di questo metodo si basa sull'estrazione completa delle cellule; Lasciare indietro qualsiasi proteina contribuisce a errori di quantificazione. Il conteggio dei nuclei o delle cellule fornisce un'alternativa in cui non è necessario rimuovere le cellule dalla superficie su cui vengono coltivate. Invece, le cellule vive o fissate vengono colorate o controcolorate utilizzando colorazioni colorimetriche o fluorescenti e il conteggio può essere eseguito sulla base di immagini di cellule intere o solo di nuclei. La quantificazione dei nuclei è preferita rispetto alle cellule intere per applicazioni di microscopia a fluorescenza e per normalizzare i dati in cellule fuse (ad esempio, miotubi) o tessuti. Per la quantificazione dei nuclei utilizzando un approccio a fluorescenza, i coloranti fluorescenti del DNA (ad esempio, 4',6-diamidino-2-fenilindolo dicloruro (DAPI), Hoechst 33342, Hoechst 33358, DRAQ5, ecc.) vengono applicati a cellule vive o fissate e visualizzati mediante microscopia a fluorescenza. DAPI, Hoechst e DRAQ5 si legano preferenzialmente alle regioni ricche di adenina timina del DNA 7,8,9, fornendo un mezzo per rilevare i nuclei. L'ottimizzazione della concentrazione del colorante previene il legame fuori bersaglio (cioè al DNA mitocondriale, causando la fluorescenza citosolica puntata)10. Il processo di ottimizzazione può essere semplificato utilizzando terreni di montaggio disponibili in commercio contenenti DAPI per celle fisse (ad esempio, VECTASHIELD Antifade Mounting Medium con DAPI) e concentrazioni pubblicate di coloranti per cellule vive (ad esempio, Hoechst)6,11,12. Una volta colorati, i nuclei nelle cellule vive o fissate vengono visualizzati utilizzando l'appropriato filtro di eccitazione (ad esempio, ultravioletto [DAPI e Hoechst] o rosso lontano [DRAQ5]). I massimi di eccitazione per DAPI, Hoechst e DRAQ5 si trovano alle due estremità dello spettro per la microscopia a fluorescenza. Questo attributo, oltre alla specificità del DNA di questi coloranti e al loro uso a concentrazioni ottimali, riduce al minimo la fluorescenza fuori bersaglio. Le immagini risultanti mostrano nuclei luminosi su uno sfondo nero e questi nuclei sono quantificati come misura del numero di cellule per le cellule uninucleate. Mentre esistono altri metodi accurati di quantificazione dei nuclei basati sulle colorazioni che utilizzano le colorazioni istologiche e la microscopia in campo chiaro13, l'automazione è più difficile, anche se possibile, utilizzando questi approcci.
Sebbene rimanga il gold standard, la quantificazione manuale dei nuclei è laboriosa, dispendiosa in termini di tempo e suscettibile di potenziali errori umani con un conteggio prolungato. Sebbene esistano programmi di conteggio automatico delle cellule, non sono preferiti da tutti gli utenti, possono essere proibitivi in termini di costi e la convalida per applicazioni specifiche può essere minima. Python è diventato una risorsa preziosa e accessibile per i biologi negli ultimi anni. Le librerie di visione artificiale Python sono particolarmente utili per l'analisi delle immagini14. Qui, forniamo istruzioni dettagliate per l'utilizzo del nostro programma eseguibile sviluppato utilizzando il codice Python per quantificare i nuclei colorati con coloranti fluorescenti e visualizzati utilizzando la microscopia a fluorescenza. Le competenze di codifica non sono necessarie per utilizzare il programma qui descritto. Questo flusso di lavoro è specifico per cellule o tessuti con coloranti di DNA fluorescenti applicati. Non è destinato all'uso con immagini in campo chiaro. La sezione 1 descrive come acquisire e salvare le immagini raccolte utilizzando la microscopia a fluorescenza in modo compatibile con questo flusso di lavoro. La sezione 2 fornisce le istruzioni per l'esecuzione del programma di quantificazione dei nuclei come eseguibile e per il recupero dell'output. Il programma eseguibile può essere eseguito direttamente dal file .exe fornito e non richiede modifiche per quantificare i nuclei in immagini di qualità. Questo eseguibile richiede un PC basato su Windows. La sezione 3 fornisce istruzioni per l'esecuzione del programma come script Python piuttosto che come eseguibile (richiesto per i sistemi basati su Mac o Linux, opzionale per i sistemi basati su Windows) e consente di ottimizzare il codice se lo si desidera. Nella sezione 4 viene descritto come utilizzare i risultati per la normalizzazione. Questi protocolli sono seguiti da risultati rappresentativi, compresi i dati di convalida del nostro laboratorio.
Protocollo
NOTA: i file supplementari sono disponibili al seguente link https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. Acquisizione e salvataggio di immagini mediante microscopia a fluorescenza
- Preparare campioni di cellule o tessuti da sottoporre a visualizzazioni, compresa la colorazione con il colorante di DNA desiderato. Per ottenere le immagini qui utilizzate, i mioblasti C2C12 (CRL-1772, American Type Culture Collection) sono stati coltivati in piastre a 6 pozzetti per 48-72 ore in condizioni di coltura standard (5% CO2, 37 °C, umidificate), con o senza 50 mM di EtOH6, e fissate in metanolo ghiacciato come precedentemente descritto13. Le celle fisse sono state montate utilizzando un mezzo di montaggio contenente DAPI15 e asciugate al buio prima dell'imaging.
- Accendere tutti i componenti del microscopio a fluorescenza e del computer secondo le procedure operative standard stabilite. Aprire il software associato al microscopio.
- Impostare il microscopio per visualizzare i campioni attraverso l'oculare. Controlla l'obiettivo. Se necessario, ruotare il portaobiettivi sull'obiettivo desiderato. Assicurarsi che il programma software abbia selezionato l'obiettivo desiderato per un'indicazione accurata dell'area dell'immagine. Qui, le immagini sono state catturate utilizzando un obiettivo 10x.
- Con le luci spente, posizionare il campione (vetrino, piatto o piatto) sul tavolino. Attiva l'imaging dal vivo con il filtro appropriato e imposta il tempo di esposizione su una durata predeterminata o sull'esposizione automatica. In questo caso, è stato utilizzato il filtro DAPI e il tempo di esposizione di 61-62 ms. Vedere la tabella di risoluzione dei problemi per indicazioni sull'impostazione del tempo di esposizione, in particolare per le immagini con un'elevata densità di celle.
- Utilizzando l'oculare, regolare lentamente la messa a fuoco utilizzando la manopola di regolazione lorda fino a quando non sono visibili i nuclei macchiati, quindi utilizzare la manopola di regolazione fine per regolare lentamente la messa a fuoco fino a quando i nuclei non appaiono chiari e nitidi.
NOTA: Esempi di immagini appropriate sono mostrati nella Figura 1.- Se appare un'eccessiva colorazione dello sfondo o se i nuclei appaiono molto scuri, regolare manualmente il tempo di esposizione. In una singola immagine, ci possono essere differenze visibili nell'intensità del segnale per alcuni nuclei; Tuttavia, la forma rotonda dovrebbe essere visibile per tutti i nuclei con un'interferenza minima del segnale.
NOTA: Il programma utilizzato nelle applicazioni downstream è robusto a diverse intensità. Le immagini grezze utilizzate per lo sviluppo e la convalida del programma di quantificazione dei nuclei includono immagini con una gamma di intensità del segnale e sono disponibili attraverso l'Open Science Framework repository (vedi link sopra menzionato).
- Se appare un'eccessiva colorazione dello sfondo o se i nuclei appaiono molto scuri, regolare manualmente il tempo di esposizione. In una singola immagine, ci possono essere differenze visibili nell'intensità del segnale per alcuni nuclei; Tuttavia, la forma rotonda dovrebbe essere visibile per tutti i nuclei con un'interferenza minima del segnale.
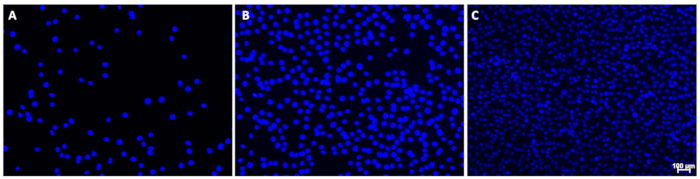
Figura 1: Immagini di esempio di nuclei colorati con DAPI. Queste immagini raffigurano nuclei a (A) bassa, (B) media o (C) ad alta densità cellulare con uno sfondo minimo. Si noti la variazione dell'intensità del segnale dei nuclei nell'immagine C; Ciò non ha interferito con la quantificazione. La barra della scala (C) indica 100 μm; Le barre della scala devono essere omesse dalle immagini utilizzate per la quantificazione dei nuclei. Clicca qui per visualizzare una versione più grande di questa figura.
- Una volta che i nuclei sono a fuoco attraverso l'oculare, passare alla modalità di rilevamento sulla fotocamera. Regolare la messa a fuoco utilizzando la manopola di regolazione fine.
- Regolare il tempo di esposizione, se necessario. Annotare il tempo di esposizione finale per l'uso con campioni e immagini successivi.
NOTA: Sebbene l'utilizzo dello stesso tempo di esposizione su tutte le immagini sia l'ideale, le variazioni del tempo di esposizione sono accettabili per questa applicazione perché l'intensità della fluorescenza non viene quantificata o confrontata tra i campioni. - Acquisire l'immagine utilizzando l'apposito pulsante di acquisizione delle immagini sul software. Se ai campioni sono applicati più coloranti, cambiare il filtro (ad esempio, su FITC, TRITC o Cy5) e acquisire l'immagine della stessa area a scopo di unione.
- Applicare l'otturatore per ridurre al minimo l'esposizione del campione alla luce. Senza masterizzare in una barra di scala, salvare l'immagine in un formato di file di figura universale (.tif, .png o .jpg) in una cartella dedicata. Assicurarsi che ogni nome di file immagine sia descrittivo in modo appropriato o che qualsiasi codice utilizzato per i nomi dei file disponga di una documentazione collegata appropriata.
NOTA: Questa diventerà la directory delle immagini per il programma di quantificazione. Se è stata utilizzata l'imaging multicolore, copiare solo le immagini dei nuclei in una cartella separata e dedicata. Questo può essere fatto anche alla fine della sessione di imaging. Le immagini esportate utilizzando le impostazioni di esportazione predefinite sulla maggior parte dei software di imaging hanno in genere una risoluzione sufficiente per l'uso con questo programma, a condizione che siano state acquisite immagini di qualità. Tuttavia, le immagini devono essere controllate visivamente dopo l'esportazione per assicurarsi che la morfologia dei nuclei sia chiara. Se le immagini vengono salvate in .jpg formato, è necessario prestare attenzione per garantire che le immagini non siano eccessivamente compresse e che la qualità sia mantenuta. - Aprire l'otturatore, tornare all'imaging dal vivo ed eseguire la scansione del campione in un'altra posizione per l'imaging. Ripetere i passaggi 1.8-1.9 secondo necessità. Per le immagini di cellule seminate in piastre a 6 pozzetti acquisite con un ingrandimento 10x, acquisire 10-15 immagini per pozzetto (a seconda della consistenza della densità cellulare nell'area del campione).
- Dopo aver acquisito tutte le immagini per un singolo campione, eseguire una delle seguenti opzioni.
- Passate all'esempio successivo e ripetete i passaggi 1.4-1.10.
- Metti in pausa il protocollo e torna in un altro momento. I campioni devono rimanere protetti dalla luce. Questa opzione non è consigliata se si lavora con cellule vive.
- Una volta completata l'imaging, assicurarsi che tutti i campioni siano protetti dalla luce prima di accendere le luci della stanza. Se la cartella contenente le immagini viene salvata localmente, trasferire i file su una chiavetta USB, una posizione di archiviazione cloud o un'altra unità di rete accessibile al computer in cui verrà eseguito il programma di quantificazione. Salva in una cartella dedicata.
- Spegnere tutte le parti del microscopio e del computer secondo le procedure operative standard stabilite.
- L'imaging è ora completo. Analizzare i file utilizzando il programma di quantificazione automatica come descritto nella Sezione 2 o, in alternativa, eseguendo il programma come script Python come descritto nella Sezione 3.
2. Esecuzione del programma di quantificazione automatica dei nuclei come eseguibile
NOTA: Questo metodo, che esegue il programma come eseguibile, è compatibile con i PC basati su Windows ed è il metodo consigliato per gli utenti del sistema operativo Windows. Mac OS o altri utenti non Windows dovrebbero eseguire il programma come script Python (vedere la Sezione 3).
- Assicurarsi che Python sia installato nel computer. Se necessario, installare Python da python.org/downloads (preferito) o utilizzando Anaconda o Miniconda. Alla data di pubblicazione, questo programma è stato testato con le versioni Python 3.10-3.12.
- Apri il browser e vai a https://github.com/rbudnar/nuclei_counter/releases. Scarica l'ultima versione del file denominato count_nuclei.zip.
- Dalla cartella Download, fare clic con il pulsante destro del mouse sul file Zip ed estrarre tutti i file nella posizione desiderata sul computer locale (vedere la Figura 2A). Mantieni aperta questa posizione del file; questo percorso di file verrà utilizzato nel passaggio 2.5 (vedere la Figura 2B).
NOTA: Count_nuclei dovrebbe ora apparire come file dell'applicazione. Questo è il file eseguibile. - Apri un prompt dei comandi cercando CMD o Prompt dei comandi nella barra di ricerca. Utilizzare il comando cd per modificare la directory nel percorso del file eseguibile (vedere la Figura 2C) e premere Invio. Esempio di riga di comando: C:\Users\janedoe>cd [spazio dopo cd, quindi copia/incolla il percorso del file]
- Nella riga di comando successiva, digitare quanto segue e premere INVIO.
count_nuclei.exe --img_dir "percorso/delle/immagini" --output_dir "percorso/della/uscita" --save_images --output_count_filename "results.csv"
dove percorso/a/immagini è il percorso del file della cartella contenente le immagini da analizzare (vedi passaggio 1.14); path/to/output è il percorso del file della cartella in cui il file .csv deve essere salvato (il valore predefinito è la directory di lavoro corrente quando non viene fornito); e results.csv è il nome del file in cui verranno salvati i conteggi risultanti (se non viene fornito, il nome del file di output predefinito è count_results.csv).
NOTA: le virgolette sono necessarie nella riga di comando. Assicurarsi che non vi siano spazi dopo le virgolette prima del percorso del file (vedere la Figura 2C). - Quando viene visualizzata la riga di comando successiva, l'elaborazione è completa. I contorni e il foglio di calcolo dei risultati sono ora disponibili nella posizione specificata come directory di output nel passaggio 2.5 (ad esempio, percorso/a/output; vedere la Figura 2C).
- Ispezionare visivamente i contorni per verificare la qualità del conteggio. I risultati possono ora essere utilizzati per normalizzare i dati come descritto nella Sezione 4.
NOTA: Se sembra che i contorni abbiano perso nuclei (ad esempio, i nuclei appaiono troppo deboli) o raggruppato gruppi di nuclei in un unico elemento (ad esempio, immagine sovraesposta, sfondo eccessivo, artefatti), l'utente deve fare riferimento alla tabella di risoluzione dei problemi e potrebbe essere necessario tornare alla Sezione 1 per ottimizzare l'acquisizione dell'immagine prima di passare alla Sezione 4.
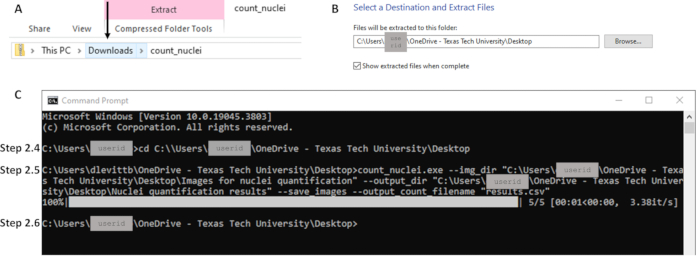
Figura 2: Esempi di parti chiave del flusso di lavoro per la sezione 2. (A) Estrarre i file dalla cartella Download, facendo attenzione a fare clic sulla cartella corretta come indicato dalla freccia per abilitare l'estrazione (B) nella destinazione desiderata nel passaggio 2.3. (C) Esempi di comandi per i passaggi 2.4 e 2.5 e la riga di comando successiva che indica il completamento nel passaggio 2.6. Si noti che il percorso del file nel passaggio 2.4 (C) corrisponde alla posizione per l'estrazione nel passaggio 2.3 (B). Clicca qui per visualizzare una versione più grande di questa figura.
3. Esecuzione del programma di quantificazione automatica dei nuclei come script Python
- Assicurarsi che Python sia installato nel computer. Se necessario, installare Python da python.org/downloads (preferito) o utilizzando Anaconda o Miniconda. Alla data di pubblicazione, questo programma è stato testato con le versioni Python 3.10-3.12.
- Apri il browser e vai a https://github.com/rbudnar/nuclei_counter. Fare clic sul pulsante Codice verde , quindi fare clic su Scarica ZIP per scaricare il repository del codice (vedere la Figura 3A).
- Utilizzare le seguenti istruzioni per il sistema operativo Windows. Vedere il passaggio 3.4 per i computer basati su Mac OS.
- Dalla cartella Download, fare clic con il pulsante destro del mouse su File e selezionare Estrai tutto per estrarre i file su un computer locale (vedere la Figura 3B-C).
- Nel percorso del file estratto, apri la cartella denominata nuclei_counter-main. Questo è il repository del codice.
NOTA: Questo deve essere scaricato solo la prima volta che questo programma viene utilizzato; È sufficiente spostare i file estratti in un percorso di rete accessibile e prendere nota di tale percorso. - Apri un prompt dei comandi cercando CMD o Prompt dei comandi nella barra di ricerca. Se Python è stato installato da Anaconda o Miniconda, apri il prompt di Anaconda invece di un prompt dei comandi standard. Questo può essere trovato digitando Anaconda Prompt nella barra di ricerca.
- Utilizzare il comando cd per modificare la directory nel repository del codice e premere Invio. Vedere la riga di codice seguente, che fa distinzione tra maiuscole e minuscole. Assicurarsi che la cartella denominata nuclei_counter-main sia aperta quando si copia il percorso del file; il file denominato pyproject o pyproject.toml dovrebbe essere visibile (vedere la Figura 3D). Esempio di riga di comando: C:\Users\janedoe>cd [spazio dopo cd, quindi copia/incolla il percorso del file]
- Nella riga di comando successiva, digitare quanto segue, inclusi lo spazio e il punto (vedere la Figura 3D):
pip install -e .
NOTA: i passaggi 3.3.5 e 3.3.6 devono essere completati solo la prima volta che questo programma viene eseguito su qualsiasi computer. Se questa operazione è già stata completata, procedere al passaggio 3.3.7. L'inizializzazione del processo di installazione potrebbe richiedere alcuni istanti. - Premere Invio. L'esecuzione di questo comando installa le librerie necessarie per eseguire questo script e installa il repository in modalità modificabile nel caso in cui siano necessarie modifiche al codice (vedere la Figura 3D).
- Digitare quanto segue nella riga di comando successiva per modificare la directory nella directory principale del codice sorgente (vedere la Figura 3E):
CD "nuclei_counter" - Nella riga di comando successiva, digitare quanto segue e premere Invio (vedere la Figura 3E):
python count_nuclei.py --img_dir "percorso/delle/immagini" --output_dir "percorso/della/uscita" --save_images --output_count_filename "results.csv"
Dove percorso/a/immagini è il percorso del file della cartella contenente le immagini da analizzare (passaggio 1.14); path/to/output è il percorso del file della cartella in cui il file .csv deve essere salvato (il valore predefinito è la directory di lavoro corrente quando non viene fornito); e results.csv è il nome del file in cui verranno salvati i conteggi risultanti (se non viene fornito, il nome del file di output predefinito è count_results.csv).
NOTA: le virgolette sono necessarie nella riga di comando. Assicurarsi che non vi siano spazi dopo le virgolette prima del percorso del file. Lo script python leggerà i file nel percorso fornito ed eseguirà il programma di conteggio dei nuclei per tutte le immagini nella directory (vedi Figura 3E). I conteggi risultanti verranno salvati nel file di .csv risultati nella posizione specificata. - Quando viene visualizzata la riga di comando successiva, l'elaborazione è completa (vedere la Figura 3E). Le curve di livello e il foglio di calcolo dei risultati sono ora disponibili nella posizione specificata come directory di output nel passaggio 3.3.8 (ad esempio, percorso/a/output).
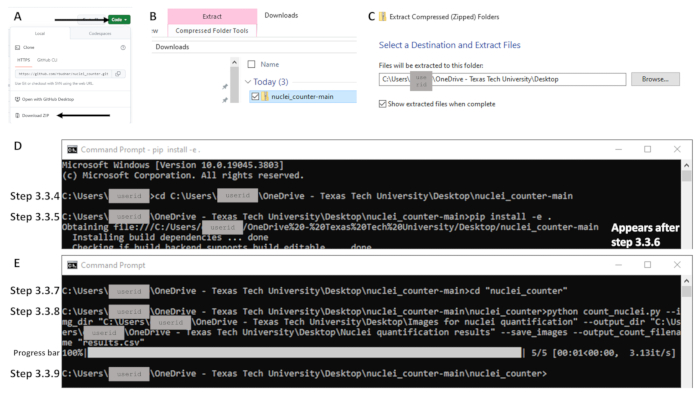
Figura 3: Esempi di parti chiave del flusso di lavoro per la sezione 3 con il sistema operativo Windows. Per accedere al codice, fare clic sul pulsante verde con l'etichetta Codice e (A) scaricare il file Zip (indicato da frecce). (B) Estrarre facendo clic con il pulsante destro del mouse sul file Zip (mostrato in blu) e (C) impostando la destinazione desiderata. (D) Dopo aver aperto il prompt dei comandi, vengono visualizzati esempi di comandi per i passaggi 3.3.4 e 3.3.5. Si noti che il comando per il passaggio 3.3.5 è tutto su una riga; il testo che inizia con "Ottenimento del file" nella riga successiva viene visualizzato dopo aver premuto Invio (passaggio 3.3.6, ovvero l'esecuzione del comando). (E) Esempi di comandi per i passaggi 3.3.7 e 3.3.8, una barra di avanzamento dopo l'esecuzione del passaggio 3.3.8 e la riga di comando che mostra il completamento nel passaggio 3.3.9. Clicca qui per visualizzare una versione più grande di questa figura.
- Utilizzate le seguenti istruzioni per Mac OS.
- Dalla cartella Download, fare clic sul menu File e selezionare Apri per estrarre i file sul computer locale (vedere la Figura 4A).
- Nel percorso del file estratto, apri la cartella denominata nuclei_counter-main. Questo è il repository del codice.
NOTA: Questo deve essere scaricato solo la prima volta che questo programma viene utilizzato; È sufficiente spostare i file estratti in un percorso di rete accessibile e prendere nota di tale percorso. - Apri un terminale premendo comando+barra spaziatrice che apre spotlight; Quindi, cerca "terminale".
- Utilizzare il comando cd per modificare la directory nel repository del codice e premere Invio. Vedere la riga di codice seguente, che fa distinzione tra maiuscole e minuscole. Digita il percorso del file in un documento di testo e copialo nel terminale. Assicurarsi che il percorso del file inizi con il testo immediatamente precedente cd (ad esempio, /Users/userid nella Figura 4B). Esempio di riga di comando: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- Sulla riga di comando successiva, dopo l'ID utente viene visualizzato il simbolo del dollaro (vedere la Figura 4B). Assicurati che ci sia uno spazio dopo il simbolo del dollaro e digita quanto segue, inclusi lo spazio e il punto alla fine del comando. Includi la versione di Python appropriata immediatamente dopo pip senza uno spazio: pip3 install -e .
NOTA: i passaggi 3.4.5 e 3.4.6 devono essere completati solo la prima volta che questo programma viene eseguito su qualsiasi computer. Se questa operazione è già stata completata, procedere al passaggio 3.4.7. L'inizializzazione del processo di installazione potrebbe richiedere alcuni istanti. - Premere Invio. L'esecuzione di questo comando installa le librerie necessarie per eseguire questo script e installa il repository in modalità modificabile nel caso in cui siano necessarie modifiche al codice.
- Nella riga di comando successiva, assicurarsi che ci sia uno spazio dopo il simbolo del dollaro, quindi digitare quanto segue per modificare la directory nella directory principale del codice sorgente (vedere la Figura 4C):
CD "nuclei_counter" - Nella riga di comando successiva, digitare la riga di comando seguente. Sostituisci il numero dopo Python con il numero di versione corretto se utilizzi una versione diversa da 3. Quindi, premere Invio (vedere la Figura 4C):
python3 count_nuclei.py --img_dir "percorso/delle/immagini" --output_dir "percorso/della/uscita" --save_images --output_count_filename "results.csv"
dove percorso/a/immagini è il percorso del file della cartella contenente le immagini da analizzare (passaggio 1.14); path/to/output è il percorso del file della cartella in cui il file .csv deve essere salvato (il valore predefinito è la directory di lavoro corrente quando non viene fornito); e results.csv è il nome del file in cui verranno salvati i conteggi risultanti (se non viene fornito, il nome del file di output predefinito è count_results.csv).
NOTA: le virgolette sono necessarie nella riga di comando. Assicurarsi che non vi siano spazi dopo le virgolette prima del percorso del file. Lo script Python leggerà i file nel percorso fornito ed eseguirà il programma di conteggio dei nuclei per tutte le immagini nella directory (vedere la Figura 4C). I conteggi risultanti verranno salvati nel file di .csv risultati nella posizione specificata. - Quando viene visualizzata la riga di comando successiva, l'elaborazione è completa (vedere la Figura 4C). I contorni e il foglio di calcolo dei risultati sono ora disponibili nella posizione specificata come directory di output nel passaggio 3.4.8 (ad esempio, percorso/della/uscita).
- Ispezionare visivamente i contorni per verificare la qualità del conteggio. I risultati possono ora essere utilizzati per normalizzare i dati come descritto nella Sezione 4.
NOTA: Se sembra che i contorni abbiano nuclei mancanti (ad esempio, i nuclei appaiono troppo deboli) o raggruppano gruppi di nuclei in un unico elemento (ad esempio, immagine sovraesposta, sfondo eccessivo, artefatti), l'utente deve fare riferimento alla Tabella 1 e potrebbe essere necessario tornare alla Sezione 1 per ottimizzare l'acquisizione dell'immagine prima di passare alla Sezione 4.
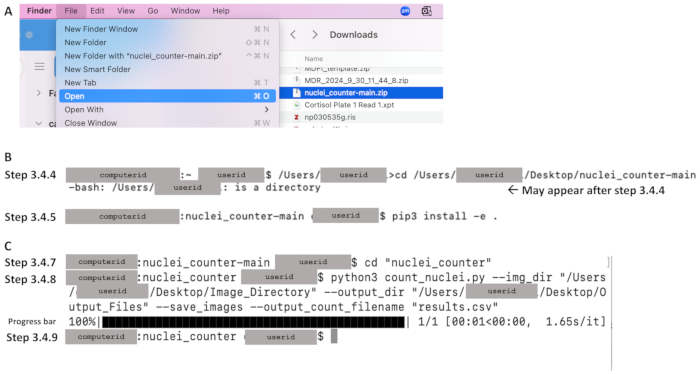
Figura 4: Esempi di parti chiave del flusso di lavoro per la sezione 3 con Mac OS. Dopo aver scaricato il codice (vedi Figura 3A), (A) estrarre cliccando sul file Zip (mostrato in blu) e impostando la destinazione desiderata. (B) Dopo aver aperto il terminale, esempi di comandi per i passaggi 3.4.4 (modifica della directory nel repository di codice) e 3.4.5 (installazione di pip). (C) Esempi di comandi per i passaggi 3.4.7 e 3.4.8, una barra di avanzamento dopo l'esecuzione del passaggio 3.4.8 e la riga di comando che mostra il completamento nel passaggio 3.4.9. Clicca qui per visualizzare una versione più grande di questa figura.
4. Utilizzo dei conteggi per normalizzare i dati sperimentali
- Apri il file results.csv che collega ogni immagine con la quantificazione dei nuclei determinata utilizzando il programma automatizzato.
- Decidere il metodo di normalizzazione: 1) immagine per immagine (ad esempio, se si utilizza la microscopia a fluorescenza multicolore), 2) normalizzare i dati come rapporto tra il numero medio di nuclei per campione rispetto a un campione di riferimento (o condizione), 3) normalizzare il conteggio dei nuclei per campione (ad esempio, estrapolare il conteggio totale dei nuclei nell'area del campione). Le raccomandazioni per il metodo di normalizzazione sono nella sezione Discussione.
- Se la normalizzazione verrà eseguita immagine per immagine, è sufficiente dividere i dati sperimentali per ogni immagine per il numero di nuclei per quell'immagine.
- Per normalizzare i dati come rapporto, seguire la procedura descritta di seguito.
- Per tutti i campioni in cui sono state raccolte più immagini per una singola replica (ad esempio, 10-15 immagini acquisite da un singolo pozzetto in una piastra a 6 pozzetti), calcolare il numero medio di nuclei per immagine. Questi conteggi dei nuclei sono considerati come il conteggio delle cellule assumendo cellule uninucleate o un numero di cellule fuse insieme in cellule multinucleate (come con i miotubi).
- Selezionare un campione di riferimento o una condizione di riferimento (ad esempio, il conteggio medio di tutti i campioni della condizione di controllo) e annotare il conteggio.
- Dividere il conteggio per tutti i campioni per il conteggio di riferimento, ottenendo un valore di normalizzazione. Se è stato utilizzato un campione di riferimento, utilizzare il valore di normalizzazione per il campione di riferimento come 1.
NOTA: se è stata utilizzata una condizione di riferimento, il valore medio di normalizzazione in tutta la condizione sarebbe ora 1. I campioni con meno nuclei rispetto al riferimento (cioè meno densi) avranno valori di normalizzazione inferiori a 1 e i campioni con più nuclei rispetto al riferimento (cioè più densi) avranno valori di normalizzazione maggiori di 1. - Dividi i dati sperimentali per ciascun pozzetto per il corrispondente valore di normalizzazione calcolato.
- Per normalizzare i dati in base al numero di nuclei per campione, seguire i passaggi descritti di seguito.
- Per tutti i campioni in cui sono state raccolte più immagini per una singola replica (ad esempio, 10-15 immagini acquisite da un singolo pozzetto in una piastra a 6 pozzetti), calcolare il numero medio di nuclei per immagine. Questi conteggi dei nuclei sono considerati come il conteggio delle cellule che assumono cellule uninucleate o il numero di cellule fuse insieme in cellule multinucleate (come con i miotubi).
- Determinare la superficie del vetrino (vetrino coprioggetti), del pozzetto o del piatto da cui sono stati derivati i dati. Queste informazioni sono disponibili nella sezione delle specifiche sui siti Web dei prodotti.
- Determinare l'area di ogni immagine acquisita. Queste informazioni sono spesso disponibili come metadati nel software di imaging o analisi.
- Assicurarsi che l'area della superficie del campione e l'area dell'immagine siano espresse nelle stesse unità (ad esempio, mm2). In caso contrario, convertire nell'unità desiderata.
- Calcolare il conteggio totale per ciascun campione utilizzando la seguente equazione:
TC = AC • (SA/IA)
dove TC è il conteggio totale, AC è il conteggio medio, SA è la superficie, IA è l'area dell'immagine. - Dividi i dati sperimentali per il TC corrispondente per il campione.
- Normalizzare i dati sperimentali e utilizzarli per le analisi statistiche, se necessario.
Risultati
Ogni esecuzione di immagini batch produce: 1) un insieme di file immagine con contorni applicati che mostrano i contorni dei nuclei identificati (Figura 5) e 2) un file di .csv (foglio di calcolo) che collega i nomi dei file immagine e i conteggi associati. La visualizzazione dei contorni consentirà all'utente di valutare visivamente la qualità del conteggio. In particolare, le immagini ottenute secondo la sezione 1 dovrebbero avere tutti (o quasi tutti) i nuclei circondati da una linea verde continua che indica che il nucleo è stato contato dal programma. Questi contorni sono stati utilizzati per regolare il programma qui.
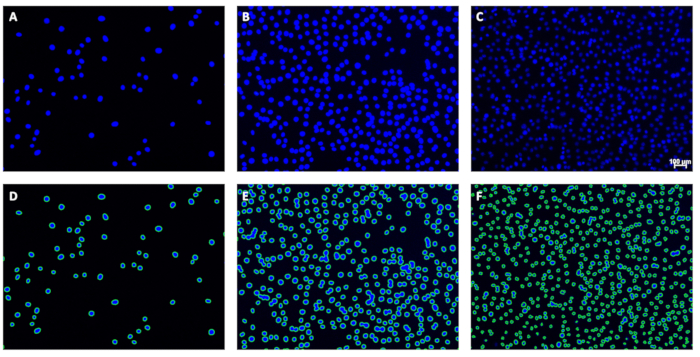
Figura 5: Esempi di immagini originali e contorni corrispondenti. (A-C) Esempi di immagini originali prima dell'analisi e (D-F) contorni corrispondenti dopo l'analisi direttamente sotto. La barra della scala (C) indica 100 μm; Le barre della scala devono essere omesse dalle immagini utilizzate per la quantificazione dei nuclei. Clicca qui per visualizzare una versione più grande di questa figura.
Convalida utilizzando i risultati iniziali
I mioblasti C2C12 a densità cellulare variabili sono stati fissati con metanolo, controcolorati con DAPI in mezzi di montaggio e sono state ottenute 120 immagini con ingrandimento 10x in formato .jpg file. Il programma automatizzato è stato eseguito con la directory delle immagini impostata sulla cartella contenente queste immagini e il file .csv risultante è stato salvato. Separatamente, ogni immagine è stata quantificata manualmente da due ricercatori addestrati che erano all'oscuro dei conteggi automatici e dei conteggi reciproci. Le immagini grezze, i contorni generati dal programma automatizzato e un foglio di calcolo dei risultati che include conteggi manuali e automatizzati sono disponibili nell'archivio Open Science Framework (OSF) (vedi link nella sezione 1). I coefficienti di correlazione intraclasse (ICC; misure medie) sono stati calcolati utilizzando le statistiche SPSS per determinare l'affidabilità dell'interclasse tra 1) i due conteggi manuali e 2) la media dei due conteggi manuali e dei conteggi ottenuti utilizzando il programma automatizzato. Approcci simili sono stati utilizzati per convalidare altri programmi progettati per quantificare oggetti in immagini ottenute utilizzando la microscopia a fluorescenza 16,17,18. Gli ICC sono stati anche calcolati tra i conteggi manuali medi e i conteggi automatizzati per ciascun quartile per garantire che la valutazione dell'intero set di dati non mascherasse potenziali cambiamenti nell'affidabilità in base alla densità delle celle. I quartili sono stati definiti utilizzando i punti di taglio del 25°, 50° e 75° percentile in base al conteggio manuale medio e vanno dal quartile 1 (Q1; densità cellulare più bassa) a Q4 (densità cellulare più alta). Gli ICC sono riportati in base a misure medie e interpretati in base alle seguenti definizioni: scarso (ICC <0,50), moderato (0,50 ≤ICC <0,75), buono (0,75 ≤ICC <0,90) o eccellente (ICC ≥0,90)19.
I nuclei per immagine variavano da 44,5 a 1160 in base alle medie di conteggio manuale. L'affidabilità dell'interrater è risultata eccellente tra i due conteggi manuali (ICC > 0,999, p < 0,0001; Figura 6A) e tra i conteggi medi manuali e automatizzati (ICC = 0,993, p < 0,0001; Figura 6B). L'affidabilità è rimasta eccellente in tutti i quartili (Q1: ICC = 0,996, p < 0,0001; Q2: ICC= 0,997, p < 0,0001; Q3: ICC = 0,998, p < 0,0001; Q4: ICC = 0,986, p < 0,0001; Figura 6C-F). Il singolo punto dati lontano dalla linea di adattamento migliore nella Figura 6B rappresenta l'immagine mostrata nella Figura 7. L'immagine ha diverse aree di più nuclei raggruppati insieme, impedendo al programma di visione artificiale di riconoscere ogni nucleo. Questo problema può essere risolto utilizzando un tempo di esposizione più breve. La tabella 1 descrive come gestire un'immagine di questo tipo.
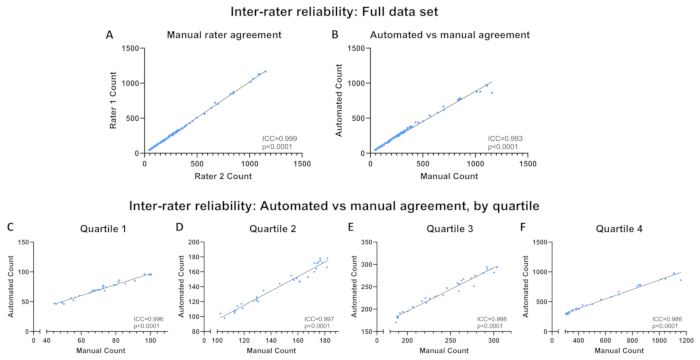
Figura 6: Affidabilità tra valutatori. Affidabilità tra valutatori nell'intero set di dati (A) tra i conteggi manuali dei valutatori e (B) tra i conteggi automatizzati e i conteggi manuali medi. (C-F) Affidabilità tra valutatori tra conteggi automatizzati e conteggi manuali medi per quartile. Affidabilità espressa come coefficiente di correlazione intraclasse (ICC). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 7: Esempio di un'immagine con due barriere alla quantificazione accurata dei nuclei. (A) Immagine originale e (B) contorni associati. La freccia 1 indica un'area con più nuclei raggruppati insieme, distorcendo la forma nucleare rotonda abbastanza da impedire il rilevamento software di più nuclei. Ciò si verifica in diverse posizioni in questa immagine. La freccia 2 indica un alone sbiadito che appare dietro più nuclei; Questo alone è apparso abbastanza luminoso da permettere al software di includerlo come nucleo, perdendo così il conteggio dei nuclei multipli che lo circonda. Questa immagine rappresenta il singolo punto dati lontano dalla linea di adattamento migliore nella Figura 6B, sottolineando i potenziali problemi con queste barriere. La barra della scala (A) indica 100 μm; Le barre della scala devono essere omesse dalle immagini utilizzate per la quantificazione dei nuclei. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Il nostro programma di quantificazione dei nuclei presenta diversi vantaggi rispetto alle opzioni esistenti: richiede solo competenze tecnologiche minime, è convalidato per il compito specifico di quantificazione dei nuclei ed è open-source; Quest'ultimo supera gli ostacoli legati ai costi. In definitiva, questo programma fornisce ai biologi cellulari e molecolari un'opzione aggiuntiva per quantificare in modo rapido e accurato i nuclei nelle immagini acquisite utilizzando la microscopia a fluorescenza. I programmi di conteggio automatizzato dei nuclei o delle cellule attualmente disponibili non sono preferiti da tutti gli utenti, alcuni sono proibitivi in termini di costi e la convalida può essere minima o assente per applicazioni specifiche. Apparecchiature specializzate, come la linea di certificazione BioTek, sono accompagnate da bollettini tecnici che convalidano la conta cellulare in situ utilizzando la colorazione nucleare20, oltre a un'ampia varietà di applicazioni aggiuntive. Sebbene esistano dati di convalida e la linea di prodotti Cytation sia ampiamente utilizzata, soprattutto se interfacciata con gli analizzatori di flusso extracellulare Seahorse per normalizzare i dati delle cellule vive in modo semplice ed efficiente, questo prodotto può essere proibitivo in termini di costi per i laboratori con un finanziamento limitato delle attrezzature. Ad esempio, il prezzo di listino per il modello base dotato di quattro obiettivi e quattro cubi filtranti per la microscopia a fluorescenza ha superato i 100.000 dollari USA nell'anno solare 2024. Per coloro che non hanno i mezzi per acquistare apparecchiature specializzate, il software open source fornisce alternative gratuite per l'analisi delle immagini. Il software open source utile per la quantificazione di nuclei e cellule include ImageJ (National Institutes of Health, Bethesda, MD, USA; disponibile al 21 agosto). I plug-in sono ora disponibili per coloro che desiderano scrivere script macro per l'automazione dell'elaborazione delle immagini utilizzando la piattaforma ImageJ. L'automazione del conteggio delle cellule basata su ImageJ mediante contrasto di fase o microscopia in campo chiaro con immagini di cellule intere è stata precedentemente descritta22,23. CellProfiler è un'applicazione ampiamente pubblicata per l'analisi di immagini cellulari colorate con coloranti fluorescenti o coloranti istologici standard, e un primo articolo che ne descrive l'utilità in una vasta gamma di applicazioni biologiche è spesso citato come validazione24. Tuttavia, questo manoscritto non convalida alcun particolare risultato biologico, il che richiede un'ulteriore convalida e trasparenza per le varie pipeline di automazione utilizzate, come per il conteggio dei nuclei25. Inoltre, sebbene CellProfiler sia un pacchetto software efficiente per l'analisi cellulare, non tutti gli scienziati lo trovano facile da usare. Pertanto, abbiamo mirato a sviluppare e convalidare un programma automatizzato alternativo per quantificare i nuclei utilizzando un software open source. In particolare, abbiamo utilizzato le librerie di visione artificiale esistenti in Python per sviluppare questo flusso di lavoro.
Questo metodo inizia con cellule o tessuti che l'utente finale ha colorato utilizzando coloranti di DNA come DAPI26. Vengono fornite linee guida generali per l'acquisizione e il salvataggio delle immagini in modo compatibile con il software di automazione. Sebbene i dettagli possano differire a seconda dei microscopi e dei software di imaging, il flusso di lavoro generale per la sezione 1 è lo stesso per ottenere immagini quantificabili. Se si utilizzano campioni vivi, è necessario prestare attenzione per assicurarsi che siano indossati dispositivi di protezione individuale adeguati e che l'ambiente di imaging sia appropriato per tali campioni (ad esempio, temperatura, percentuale di CO2, aggiunta di tampone, ecc.)27. Sebbene questo protocollo descriva l'imaging solo dei coloranti al DNA, gli utenti possono catturare altri colori della stessa area per scopi di fusione15 e semplicemente copiare le immagini nucleari in una cartella separata per l'uso con il programma di quantificazione. L'imaging deve essere eseguito in una stanza buia con DPI appropriati per campioni vivi e deve essere utilizzata un'adeguata disinfezione secondo necessità (ad esempio, con etanolo al 70%). I campioni colorati devono essere protetti dalla luce prima di accendere le luci. L'acquisizione di immagini di qualità è fondamentale per una quantificazione accurata dei nuclei. Anche nelle immagini con un tempo di esposizione appropriato e un forte contrasto tra nuclei luminosi e uno sfondo nero, la presenza di celle eccessivamente raggruppate o aloni dietro gruppi di celle (vedere la Figura 7) può ridurre l'accuratezza della quantificazione. In questi casi, potrebbe essere necessario modificare i tempi di esposizione per diminuire l'intensità dei segnali, garantendo una fusione minima dei segnali dei nuclei e la minimizzazione degli aloni, pur mantenendo un segnale abbastanza forte da rilevare le forme rotonde dei nuclei. Se questi passaggi non sono sufficienti, potrebbe essere necessario l'imaging di una sezione adiacente del campione. Tutte le immagini di un singolo esperimento devono essere acquisite con lo stesso ingrandimento e l'ingrandimento corrispondente all'obiettivo utilizzato deve essere impostato correttamente nel software di imaging. È particolarmente importante che l'obiettivo utilizzato per l'imaging corrisponda alle impostazioni del software di imaging quando il conteggio dei nuclei verrà estrapolato dall'area dell'immagine all'intera area del pozzetto (o del vetrino). Questo passaggio dovrebbe essere eseguito ogni volta che si cambia l'obiettivo, ma viene facilmente trascurato durante l'impostazione del microscopio e del software. Il tempo richiesto per la sezione 1 varia in base a una serie di fattori (ad esempio, l'esperienza dell'utente con la microscopia a fluorescenza, il numero di immagini e campioni, il microscopio, ecc.). Per acquisire 10-15 immagini di nuclei colorati per pozzetto in due piastre a 6 pozzetti, l'utente deve riservare circa 1-2 ore di tempo.
Una volta che le immagini sono state catturate e salvate in una cartella dedicata, sono pronte per essere quantificate. Il metodo più semplice per la quantificazione consiste nell'eseguire questo programma automatizzato preconfezionato senza la necessità di installare Python o alcuna libreria (sezione 2). Va notato che il programma è preparato come eseguibile ed è stato impacchettato utilizzando Pyinstaller. Il software creato utilizzando Pyinstaller è comunemente segnalato dal software antivirus. Se il software antivirus blocca l'apertura del file, l'utente deve contrassegnare il file come sicuro per procedere. Questo è fondamentale per eseguire con successo la sezione 2. In alternativa, qualsiasi utente può scegliere di eseguire il programma di quantificazione automatica come script Python (sezione 3) piuttosto che come eseguibile, se lo desidera. Se l'utente desidera eseguire il programma utilizzando un sistema operativo Mac o Linux, il programma deve essere eseguito tramite Python. Questo metodo non richiede Pyinstaller, ma può essere tecnicamente più impegnativo. Il protocollo passo-passo insieme agli esempi, descritti in dettaglio nella sezione 3, mira a rimuovere le barriere tecniche all'esecuzione di questo programma come script. Indipendentemente dal fatto che la sezione 2 o la sezione 3 venga utilizzata per eseguire il programma, è fondamentale assicurarsi che il codice venga immesso correttamente nei prompt dei comandi. Le righe di codice di esempio sono incluse in tutto il protocollo e l'utente deve prestare particolare attenzione alla spaziatura, alle virgolette e ad altri segni di punteggiatura. L'utente deve inoltre assicurarsi che i percorsi dei file siano accurati. È utile avere i percorsi dei file per le immagini e l'output disponibili in un documento di Word. I comandi possono anche essere digitati in questo documento Word e copiati/incollati secondo necessità.
Come descritto nella sezione 4, esistono diversi approcci per normalizzare i dati sperimentali utilizzando i conteggi ottenuti da questo programma di quantificazione automatizzata dei nuclei. Per gli esperimenti basati su immagini in cui i dati sperimentali includono un numero di eventi in ciascuna immagine e si desidera una proporzione (ad esempio, la proporzione di cellule V-positive all'annessina28, la proporzione di cellule che esprimono i recettori ACE229, il numero di cellule che assorbono particelle colorate30, ecc.), i dati sperimentali per ciascuna immagine possono essere semplicemente divisi per il conteggio risultante (risultante in una proporzione da 0 a 1). Quindi, la proporzione media può essere calcolata tra le immagini replicate dalla stessa diapositiva, piatto o pozzetto. Se si desidera una percentuale, è sufficiente moltiplicare le proporzioni finali per 100. Infine, queste proporzioni medie o percentuali possono essere utilizzate nelle analisi statistiche a valle. Quando i dati sperimentali da normalizzare provengono da esperimenti funzionali in cellule vive (ad esempio, tassi di consumo di ossigeno 6,11,12) o da surnatante di colture cellulari (ad esempio, attività della lattato deidrogenasi nel surnatante31 o concentrazione di una specifica citochina secreta32, ecc.), la conta dei nuclei deve prima essere mediata tra tutte le immagini catturate dallo stesso pozzetto. Quindi, la conta dei nuclei può essere estrapolata all'area superficiale del vetrino (vetrino coprioggetti), del pozzetto o del piatto, oppure la conta può essere espressa come rapporto rispetto a un campione o a una condizione di riferimento. L'estrapolazione alla superficie del vetrino, del pozzetto o del piatto è utile per esprimere i dati sperimentali rispetto al numero di cellule (se si utilizzano cellule uninucleate) da cui sono stati derivati i dati (ad esempio, ossigeno consumato al minuto per 103 cellule). Per utilizzare questo metodo, è necessario conoscere la superficie del vetrino (vetrino coprioggetti), del pozzetto o del piatto e l'area catturata in ciascuna immagine. Se il campione non copre l'intera superficie disponibile, i dati sperimentali provengono da una piccola quantità di surnatante (cioè espressi come attività o concentrazione per unità di volume), o l'immagine e/o l'area superficiale non sono note, allora l'opzione migliore è quella di calcolare la media dei nuclei in tutte le immagini per un singolo campione ed esprimere il conteggio dei nuclei rispetto a un campione di riferimento o a una condizione. Il valore risultante viene utilizzato per correggere i dati sperimentali per la densità dei nuclei o la densità delle cellule (se si utilizzano cellule uninucleate).
Sebbene questo metodo sia semplice da eseguire, esistono diversi potenziali problemi di cui gli utenti dovrebbero essere consapevoli. Questi potenziali problemi, insieme alle possibili cause e ai passaggi per la risoluzione dei problemi, sono elencati nella Tabella 1. Il programma qui descritto presenta vantaggi chiave rispetto ai software esistenti; Tuttavia, è necessario riconoscere diverse limitazioni. Lo svantaggio principale è che il programma di quantificazione può essere utilizzato solo con immagini acquisite con la microscopia a fluorescenza, non con immagini in campo chiaro. Mentre per costruire il programma sono state utilizzate immagini colorate con DAPI, catturate con un ingrandimento di 10x, il programma può essere utilizzato con altri coloranti e ingrandimenti di DNA poiché il programma è costruito per riconoscere la morfologia dei nuclei piuttosto che il colore o le dimensioni. Sebbene sia stata convalidata l'accuratezza del programma su immagini contenenti una varietà di densità di celle, l'utente finale potrebbe avere densità di celle che non rientrano nell'intervallo. In questo caso, prevediamo comunque conteggi accurati a condizione che si ottengano immagini di qualità; Tuttavia, l'utente deve controllare attentamente i contorni e l'output per garantire la precisione. Per le applicazioni di coltura cellulare, la quantificazione dei nuclei nelle immagini è più appropriata per le cellule in un monostrato. Se le cellule in coltura si sovrappongono, i nuclei possono essere mascherati, con conseguente sottostima della quantità di nuclei. La natura bidimensionale della quantificazione dei nuclei nelle immagini preclude l'uso di questo programma per determinare il numero totale di nuclei da cellule in sospensione, blocchi di tessuto spesso o altre applicazioni tridimensionali. Tuttavia, questo programma può ancora quantificare i nuclei marcati con coloranti di DNA fluorescenti in immagini bidimensionali catturate da modelli tridimensionali come blocchi di tessuto spessi. L'utilità di quantificare i nuclei nelle immagini per l'applicazione desiderata dovrebbe essere determinata dall'utente finale.
| Problema | Possibili cause | Possibili soluzioni |
| Sfondo eccessivo nelle immagini | Tempo di esposizione troppo alto | Ridurre il tempo di esposizione |
| I nuclei sembrano fondersi tra loro nelle immagini | Concentrazione di colorante di DNA fluorescente troppo alta o troppo mezzo di montaggio del colorante di DNA | Ridurre la concentrazione di colorante durante l'ottimizzazione; Utilizzare la minor quantità possibile di supporti di montaggio |
| I nuclei appaiono troppo chiari o alcuni nuclei non sono visibili | Tempo di esposizione troppo basso | Aumenta il tempo di esposizione |
| Fotosbiancamento | Prevenire il fotobleaching assicurandosi che i campioni colorati rimangano conservati al buio e riducendo al minimo l'esposizione dei campioni all'illuminazione (ad esempio, utilizzando il tempo di esposizione minimo necessario e disattivando l'illuminazione quando non è necessaria, ad esempio dopo l'acquisizione di un'immagine o durante lo scatto di appunti) | |
| Effetti alone o comparsa di forme non specifiche del nucleo | Tempo di esposizione troppo alto | Ridurre il tempo di esposizione |
| Lanugine da tergicristalli delicati o da alcune protezioni da banco in celle vive o aderenti a celle fisse | Garantire un ambiente privo di lanugine: ridurre al minimo l'uso di tergicristalli delicati quando i campioni sono esposti (ad esempio, in una cabina di biosicurezza per cellule vive, durante la fissazione e l'essiccazione di cellule fissate o durante il processo di colorazione); Assicurarsi che le protezioni da banco non lascino lanugine su vetrini o piastre durante la colorazione | |
| Altre anomalie sulla superficie di cellule/tessuti fissati | Maneggiare i campioni con estrema cura e seguire le precauzioni e le migliori pratiche adeguate durante il fissaggio e la colorazione | |
| Disturbi nel percorso della luce | Assicurarsi che tutte le parti del microscopio siano pulite e prive di lanugine e polvere; Pulire l'esterno di vetrini/piastre/piastre con etanolo al 70% per rimuovere polvere e lanugine ambientali prima dell'imaging | |
| Il file eseguibile non si apre | Il software antivirus ha bloccato il file | Contrassegna il file come sicuro |
| Errori nei prompt dei comandi | Problemi con la spaziatura nella riga di comando | Verificare che l'inizio del comando sia digitato immediatamente dopo il simbolo ">" nella riga di comando senza spazi, che non siano presenti spazi dopo le virgolette aperte per i percorsi dei file e che la spaziatura corrisponda al codice fornito e agli esempi. |
| Casi impropri utilizzati nella riga di comando | Il codice fa distinzione tra maiuscole e minuscole. Assicurati di utilizzare lettere maiuscole e minuscole a seconda dei casi | |
| Problemi con il percorso del file | Assicurarsi che il percorso del file indichi l'interno della cartella del file associata (ad esempio, la cartella del file è aperta quando viene copiato il percorso del file). | |
| Problemi con il programma che non vengono risolti utilizzando i passaggi per la risoluzione dei problemi sopra | Problemi relativi al software o alla riga di comando indicati in precedenza. | Inviare un problema tramite GitHub. Vai a https://github.com/rbudnar/nuclei_counter. Nella barra dei menu in alto, seleziona "Problemi" (seconda opzione, a destra di "Codice"). Fai clic sul pulsante verde sul lato destro che dice "Nuovo numero" per inviare. Ci sarà una richiesta per accedere a GitHub prima di inviare il problema. Ci sarà anche un'opzione per creare un account gratuito, se necessario. |
Tabella 1: Risoluzione dei problemi per l'acquisizione di immagini di qualità e l'esecuzione del flusso di lavoro automatizzato di quantificazione dei nuclei.
Nel complesso, i metodi qui descritti forniscono dettagli passo dopo passo per utilizzare il programma di quantificazione automatizzata dei nuclei che abbiamo sviluppato utilizzando librerie di visione artificiale e convalidato rispetto ai conteggi manuali di più valutatori. Questo programma è stato sviluppato per garantire l'usabilità da parte di scienziati con vari livelli di competenze tecnologiche legate alla codifica, è disponibile gratuitamente ed è stato convalidato specificamente per il compito di quantificazione dei nuclei. Rispetto al conteggio manuale, questo programma farà risparmiare tempo prezioso all'utente finale e fornirà conteggi dei nuclei accurati e affidabili per la normalizzazione dei dati.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Il finanziamento per questo lavoro è stato fornito dal National Institutes of Health/National Institute on Aging (R01AG084597; DEL e HYL) e da fondi di avviamento della Texas Tech University (DEL). Gli autori desiderano ringraziare i programmi Texas Tech University Undergraduate Research Scholars e TrUE Scholars per aver fornito sostegno finanziario ai ricercatori universitari che hanno contribuito a questo lavoro (REH, MRD, CJM, AKW). Ringraziamo anche i dottori Lauren S. Gollahon e Michael P. Massett per aver gentilmente condiviso lo spazio e le attrezzature del loro laboratorio.
Materiali
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
Riferimenti
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon