Method Article
Usando bibliotecas de visão computacional para simplificar a quantificação de núcleos
Neste Artigo
Resumo
Este artigo descreve métodos passo a passo para automatizar a quantificação de núcleos baseada em imagem usando um programa executável de código aberto validado em uma variedade de densidades de células. Este programa fornece uma alternativa que aborda as barreiras relacionadas ao custo, acessibilidade para usuários com habilidades tecnológicas limitadas e validação específica do aplicativo que pode limitar a utilidade das tecnologias existentes.
Resumo
Ensaios de células vivas e análises de células baseadas em imagem requerem normalização de dados para uma interpretação precisa. Um método comumente usado é corar e quantificar núcleos, seguido de normalização de dados para contagem de núcleos. Essa contagem de núcleos é frequentemente expressa como contagem de células para células uninucleadas. Embora a quantificação manual possa ser trabalhosa e demorada, os métodos automatizados disponíveis podem não ser preferidos por todos os usuários, podem não ter validação para essa aplicação específica ou podem ter um custo proibitivo. Aqui, fornecemos instruções passo a passo para capturar imagens quantificáveis de núcleos corados com colorações de DNA fluorescente e, posteriormente, quantificar os núcleos usando um programa de software automatizado de contagem de objetos desenvolvido usando bibliotecas de visão computacional Python. Também validamos este programa em uma variedade de densidades de células. Embora o tempo exato para a execução do programa varie de acordo com o número de imagens e hardware do computador, este programa consolida horas de trabalho contando núcleos em segundos para que o programa seja executado. Embora este protocolo tenha sido desenvolvido usando imagens de células fixas e coradas, imagens de núcleos corados em células vivas e aplicações de imunofluorescência também podem ser quantificadas usando este programa. Em última análise, este programa oferece uma opção que não requer um alto grau de habilidade tecnológica e é uma alternativa validada e de código aberto para ajudar os biólogos celulares e moleculares a simplificar seus fluxos de trabalho, automatizando a tarefa tediosa e demorada de quantificação de núcleos.
Introdução
Experimentos funcionais e baseados em imagens são essenciais para a compreensão dos impactos dos tratamentos experimentais na bioquímica e fisiologia de células inteiras. A interpretação válida dos dados de experimentos de biologia celular depende da precisão e reprodutibilidade do protocolo experimental, incluindo a normalização dos dados. Por exemplo, análises do consumo de oxigênio e das taxas de acidificação extracelular em células vivas no início e após o tratamento com drogas específicas permitem avaliar vários aspectos do metabolismo energético 1,2. Medir a atividade de enzimas como a lactato desidrogenase no sobrenadante de culturas de células pode ajudar a quantificar a integridade da membrana celular3. A coloração de células cultivadas com anexina V e iodeto de propídio antes da fixação permite a avaliação de células apoptóticas e necróticas4. No entanto, as diferenças na densidade celular entre os poços afetam os resultados de cada um desses ensaios. Confiar apenas na densidade de semeadura pode fornecer resultados enganosos devido a erros na contagem de células para semeadura, variações na densidade celular no meio durante o plaqueamento ou diferentes taxas de crescimento celular entre amostras ou tratamentos ao longo dos experimentos. Portanto, a normalização dos resultados experimentais é necessária.
Os métodos atuais de normalização para dados celulares funcionais e baseados em imagem incluem concentração de proteína5 ou núcleos ou contagem de células6. Para normalizar os dados para a concentração de proteínas, as células devem ser extraídas em volumes iguais de tampão de lise com inibidor de protease após a execução do ensaio, e um ensaio adicional (por exemplo, ensaio de ácido bicinconínico, ensaio de Bradford, etc.) deve ser realizado para quantificação de proteínas. A precisão deste método depende da extração completa da célula; deixar qualquer proteína para trás contribui para erros na quantificação. A contagem de núcleos ou células fornece uma alternativa em que nenhuma célula precisa ser removida da superfície em que são cultivadas. Em vez disso, as células vivas ou fixas são coradas ou contra-coradas usando colorações colorimétricas ou fluorescentes, e a contagem pode ser realizada com base em imagens de células inteiras ou apenas núcleos. A quantificação de núcleos é preferível a células inteiras para aplicações de microscopia de fluorescência e para normalizar dados em células fundidas (por exemplo, miotubos) ou tecidos. Para quantificação de núcleos usando uma abordagem de fluorescência, corantes de DNA fluorescente (por exemplo, dicloreto de 4′,6-diamidino-2-fenilindol (DAPI), Hoechst 33342, Hoechst 33358, DRAQ5, etc.) são aplicados a células vivas ou fixas e fotografados usando microscopia de fluorescência. DAPI, Hoechst e DRAQ5 ligam-se preferencialmente a regiões ricas em adenina-timina do DNA 7,8,9, fornecendo um meio para detectar núcleos. A otimização da concentração do corante evita a ligação fora do alvo (ou seja, ao DNA mitocondrial, causando fluorescência citosólica pontilhada)10. O processo de otimização pode ser simplificado usando meios de montagem disponíveis comercialmente contendo DAPI para células fixas (por exemplo, VECTASHIELD Antifade Mounting Medium com DAPI) e concentrações publicadas de corantes para células vivas (por exemplo, Hoechst)6,11,12. Uma vez corados, os núcleos em células vivas ou fixas são visualizados usando o filtro de excitação apropriado (por exemplo, ultravioleta [DAPI e Hoechst] ou vermelho distante [DRAQ5]). Os máximos de excitação para DAPI, Hoechst e DRAQ5 estão em cada extremidade do espectro para microscopia de fluorescência. Esse atributo, mais a especificidade do DNA desses corantes e seu uso em concentrações ideais, minimiza a fluorescência fora do alvo. As imagens resultantes mostram núcleos brilhantes contra um fundo preto, e esses núcleos são quantificados como uma medida do número de células para células uninucleadas. Embora existam outros métodos precisos de quantificação de núcleos baseados em coloração usando colorações histológicas e microscopia de campo claro13, a automação é mais difícil, embora possível, usando essas abordagens.
Embora continue sendo o padrão-ouro, a quantificação manual de núcleos é trabalhosa, demorada e suscetível a possíveis erros humanos com contagem prolongada. Embora existam programas automatizados de contagem de células, eles não são preferidos por todos os usuários, podem ter um custo proibitivo e a validação para aplicações específicas pode ser mínima. Python tornou-se um recurso valioso e acessível para biólogos nos últimos anos. As bibliotecas de visão computacional Python são particularmente úteis para análise de imagens14. Aqui, fornecemos instruções passo a passo para usar nosso programa executável desenvolvido usando código Python para quantificar núcleos corados com corantes fluorescentes e imagens usando microscopia de fluorescência. Habilidades de codificação não são necessárias para usar o programa descrito aqui. Esse fluxo de trabalho é específico para células ou tecidos com corantes de DNA fluorescente aplicados. Não se destina ao uso com imagens de campo claro. A Seção 1 descreve como capturar e salvar imagens coletadas usando microscopia de fluorescência de maneira compatível com esse fluxo de trabalho. A Seção 2 fornece instruções para executar o programa de quantificação de núcleos como uma saída executável e de busca. O programa executável pode ser executado diretamente do arquivo .exe fornecido e não requer modificações para quantificar núcleos em imagens de qualidade. Este executável requer um PC baseado em Windows. A Seção 3 fornece instruções para executar o programa como um script Python em vez de um executável (necessário para sistemas baseados em Mac ou Linux, opcional para sistemas baseados em Windows) e permite ajustar o código, se desejado. A seção 4 descreve como usar os resultados para normalização. Esses protocolos são seguidos por resultados representativos, incluindo dados de validação de nosso laboratório.
Protocolo
NOTA: Arquivos suplementares podem ser encontrados no seguinte link https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. Captura e salvamento de imagens usando microscopia de fluorescência
- Prepare amostras de células ou tecidos a serem visualizadas, incluindo coloração com o corante de DNA desejado. Para obter as imagens aqui utilizadas, mioblastos C2C12 (CRL-1772, American Type Culture Collection) foram cultivados em placas de 6 poços por 48-72 h sob condições de cultura padrão (5% CO2, 37 °C, umidificado), com ou sem 50 mM EtOH6, e fixados em metanol gelado conforme descrito anteriormente13. As células fixas foram montadas usando um meio de montagem contendo DAPI15 e secas no escuro antes da imagem.
- Ligue todos os componentes do microscópio de fluorescência e do computador de acordo com os procedimentos operacionais padrão estabelecidos. Abra o software associado ao microscópio.
- Defina o microscópio para visualizar amostras através da ocular. Verifique o objetivo. Se necessário, gire o revólver para o objetivo desejado. Certifique-se de que o programa de software tenha o objetivo desejado selecionado para uma indicação precisa da área da imagem. Aqui, as imagens foram capturadas usando uma objetiva de 10x.
- Com as luzes apagadas, coloque a amostra (slide, prato ou prato) no palco. Ligue a imagem ao vivo com o filtro apropriado e defina o tempo de exposição para uma duração predeterminada ou para a exposição automática. Aqui, filtro DAPI e tempo de exposição de 61-62 ms foram usados. Consulte a tabela de resolução de problemas para obter orientações sobre como definir o tempo de exposição, especialmente para imagens com densidades de células elevadas.
- Usando a ocular, ajuste lentamente o foco usando o botão de ajuste bruto até que os núcleos manchados fiquem visíveis e, em seguida, use o botão de ajuste fino para ajustar lentamente o foco até que os núcleos pareçam claros e nítidos.
NOTA: Exemplos de imagens apropriadas são mostrados na Figura 1.- Se aparecer coloração de fundo excessiva ou se os núcleos parecerem muito escuros, ajuste o tempo de exposição manualmente. Em uma única imagem, pode haver diferenças visíveis na intensidade do sinal para alguns núcleos; no entanto, a forma redonda deve ser visível para todos os núcleos com interferência mínima de sinal.
NOTA: O programa usado em aplicações downstream é robusto para diferentes intensidades. As imagens brutas usadas para o desenvolvimento e validação do programa de quantificação de núcleos incluem imagens com uma variedade de intensidades de sinal e estão disponíveis através do repositório Open Science Framework (veja o link mencionado acima).
- Se aparecer coloração de fundo excessiva ou se os núcleos parecerem muito escuros, ajuste o tempo de exposição manualmente. Em uma única imagem, pode haver diferenças visíveis na intensidade do sinal para alguns núcleos; no entanto, a forma redonda deve ser visível para todos os núcleos com interferência mínima de sinal.
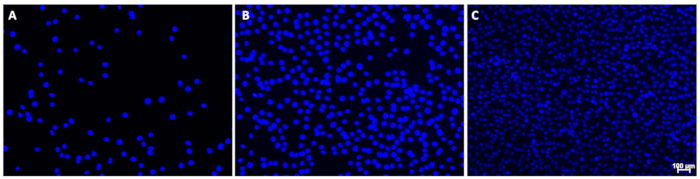
Figura 1: Exemplos de imagens de núcleos corados com DAPI. Essas imagens mostram núcleos em (A) baixa, (B) média ou (C) alta densidade celular com fundo mínimo. Observe a intensidade variável do sinal dos núcleos na imagem C; Isso não interferiu na quantificação. A barra de escala (C) indica 100 μm; As barras de escala devem ser omitidas das imagens usadas para quantificação de núcleos. Clique aqui para ver uma versão maior desta figura.
- Quando os núcleos estiverem focados através da ocular, mude o modo de detecção para a câmera. Ajuste o foco usando o botão de ajuste fino.
- Ajuste o tempo de exposição, se necessário. Observe o tempo de exposição final para uso com amostras e imagens subsequentes.
NOTA: Embora o uso do mesmo tempo de exposição em todas as imagens seja ideal, variações no tempo de exposição são aceitáveis para esta aplicação porque a intensidade da fluorescência não é quantificada ou comparada entre as amostras. - Capture a imagem usando o botão de captura de imagem apropriado no software. Se as amostras tiverem várias colorações aplicadas, troque o filtro (por exemplo, para FITC, TRITC ou Cy5) e crie imagens da mesma área para fins de mesclagem.
- Aplique o obturador para minimizar a exposição da amostra à luz. Sem gravar em uma barra de escala, salve a imagem em um formato de arquivo de figura universal (.tif, .png ou .jpg) em uma pasta dedicada. Certifique-se de que cada nome de arquivo de imagem seja adequadamente descritivo ou que qualquer codificação usada para nomes de arquivo tenha a documentação vinculada apropriada.
NOTA: Este se tornará o diretório de imagens para o programa de quantificação. Se a imagem multicolorida foi usada, copie apenas as imagens dos núcleos para uma pasta separada e dedicada. Isso também pode ser feito no final da sessão de imagem. As imagens exportadas usando as configurações de exportação padrão na maioria dos softwares de imagem geralmente têm resolução suficiente para uso com este programa, desde que imagens de qualidade tenham sido capturadas. No entanto, as imagens devem ser verificadas visualmente após a exportação para garantir que a morfologia dos núcleos esteja clara. Se as imagens forem salvas em .jpg formato, deve-se tomar cuidado para garantir que as imagens não sejam excessivamente compactadas e a qualidade seja mantida. - Abra o obturador, retorne à imagem ao vivo e digitalize a amostra para outro local para obter imagens. Repita as etapas 1.8 a 1.9 conforme necessário. Para imagens de células semeadas em placas de 6 poços capturadas com ampliação de 10x, capture de 10 a 15 imagens por poço (dependendo da consistência da densidade celular em toda a área da amostra).
- Depois que todas as imagens de uma única amostra forem adquiridas, execute uma das opções a seguir.
- Passe para o próximo exemplo e repita as etapas 1.4-1.10.
- Pause o protocolo e retorne em outro momento. As amostras devem permanecer protegidas da luz. Esta opção não é recomendada se estiver trabalhando com células vivas.
- Quando a imagem estiver concluída, certifique-se de que todas as amostras estejam protegidas contra luz antes de acender as luzes da sala. Se a pasta que contém as imagens for salva localmente, transfira os arquivos para um pen drive, local de armazenamento em nuvem ou outra unidade de rede acessível ao computador onde o programa de quantificação será executado. Salve em uma pasta dedicada.
- Desligue todas as partes do microscópio e do computador de acordo com os procedimentos operacionais padrão estabelecidos.
- A imagem agora está completa. Analise os arquivos usando o programa de quantificação automatizada, conforme descrito na Seção 2 ou, alternativamente, executando o programa como um script Python, conforme descrito na Seção 3.
2. Executando o programa automatizado de quantificação de núcleos como um executável
NOTA: Este método, executando o programa como um executável, é compatível com PCs baseados em Windows e é o método recomendado para usuários do sistema operacional Windows. Mac OS ou outros usuários não Windows devem executar o programa como um script Python (consulte a Seção 3).
- Verifique se o Python está instalado no computador. Se necessário, instale o Python a partir do python.org/downloads (preferencial) ou usando o Anaconda ou o Miniconda. Na data de publicação, este programa foi testado com as versões 3.10-3.12 do Python.
- Abra o navegador e navegue até https://github.com/rbudnar/nuclei_counter/releases. Baixe a versão mais recente do arquivo chamado count_nuclei.zip.
- Na pasta Downloads, clique com o botão direito do mouse no arquivo Zip e em Extract All files para o local desejado no computador local (consulte a Figura 2A). Mantenha este local de arquivo aberto; esse caminho de arquivo será usado na etapa 2.5 (consulte a Figura 2B).
NOTA: Count_nuclei agora deve aparecer como um arquivo de aplicativo. Este é o arquivo executável. - Abra um prompt de comando pesquisando CMD ou Prompt de Comando na barra de pesquisa. Use o comando cd para alterar o diretório para o caminho do arquivo executável (consulte a Figura 2C) e pressione Enter. Exemplo de linha de comando: C:\Users\janedoe>cd [espaço após cd e, em seguida, copie/cole o caminho do arquivo]
- Na próxima linha de comando, digite o seguinte e pressione Enter.
count_nuclei.exe --img_dir "caminho/para/imagens" --output_dir "caminho/para/saída" --save_images --output_count_filename "results.csv"
onde caminho/para/imagens é o caminho do arquivo para a pasta que contém as imagens a serem analisadas (consulte a etapa 1.14); path/to/output é o caminho do arquivo para a pasta onde o arquivo .csv deve ser salvo (o padrão é o diretório de trabalho atual quando não fornecido); e results.csv é o nome do arquivo no qual as contagens resultantes serão salvas (se não for fornecido, o nome do arquivo de saída será padronizado para count_results.csv).
NOTA: As aspas são necessárias na linha de comando. Certifique-se de que não haja espaços após aspas antes do caminho do arquivo (consulte a Figura 2C). - Quando a próxima linha de comando for exibida, o processamento estará concluído. Os contornos e a planilha de resultados agora estão disponíveis no local especificado como o diretório de saída na etapa 2.5 (ou seja, caminho/para/saída; consulte a Figura 2C).
- Inspecione visualmente os contornos para verificar a qualidade da contagem. Os resultados agora podem ser usados para normalizar os dados, conforme descrito na Seção 4.
NOTA: Se os contornos parecerem ter perdido núcleos (por exemplo, núcleos parecem muito fracos) ou agrupar grupos de núcleos em um único item (por exemplo, imagem superexposta, muito fundo, artefatos), o usuário deve consultar a tabela de solução de problemas e pode precisar retornar à Seção 1 para otimizar a aquisição de imagens antes de passar para a Seção 4.
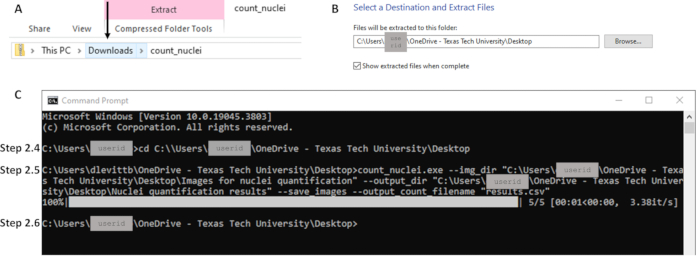
Figura 2: Exemplos de partes importantes do fluxo de trabalho para a seção 2. (A) Extraia arquivos da pasta Downloads, tendo o cuidado de clicar na pasta correta, conforme indicado pela seta, para habilitar a extração (B) para o destino desejado na etapa 2.3. (C) Exemplos de comandos para as etapas 2.4 e 2.5 e a próxima linha de comando indicando a conclusão na etapa 2.6. Observe que o caminho do arquivo na etapa 2.4 (C) corresponde ao local para extração na etapa 2.3 (B). Clique aqui para ver uma versão maior desta figura.
3. Executando o programa automatizado de quantificação de núcleos como script Python
- Verifique se o Python está instalado no computador. Se necessário, instale o Python a partir do python.org/downloads (preferencial) ou usando o Anaconda ou o Miniconda. Na data de publicação, este programa foi testado com as versões 3.10-3.12 do Python.
- Abra o navegador e navegue até https://github.com/rbudnar/nuclei_counter. Clique no botão Green Code e, em seguida, clique em Download ZIP para fazer download do repositório de código (consulte a Figura 3A).
- Use as instruções a seguir para o sistema operacional Windows. Consulte a etapa 3.4 para máquinas baseadas em Mac OS.
- Na pasta Downloads, clique com o botão direito do mouse no arquivo e selecione Extrair tudo para extrair os arquivos para um computador local (consulte a Figura 3B-C).
- No local do arquivo extraído, abra a pasta chamada nuclei_counter-main. Este é o repositório de código.
NOTA: Isso só precisa ser baixado na primeira vez que este programa for usado; Basta mover os arquivos extraídos para um local de rede acessível e anotar esse local. - Abra um prompt de comando pesquisando CMD ou Prompt de Comando na barra de pesquisa. Se o Python tiver sido instalado pelo Anaconda ou Miniconda, abra o prompt do Anaconda em vez de um prompt de comando padrão. Isso pode ser encontrado digitando Anaconda Prompt na barra de pesquisa.
- Use o comando cd para alterar o diretório para o repositório de código e pressione Enter. Consulte a linha de código abaixo, que diferencia maiúsculas de minúsculas. Certifique-se de que a pasta chamada nuclei_counter-main esteja aberta ao copiar o caminho do arquivo; o arquivo chamado pyproject ou pyproject.toml deve estar visível (consulte a Figura 3D). Exemplo de linha de comando: C:\Users\janedoe>cd [espaço após cd e, em seguida, copie/cole o caminho do arquivo]
- Na próxima linha de comando, digite o seguinte, incluindo o espaço e o ponto (consulte a Figura 3D:
pip instalar -e .
NOTA: As etapas 3.3.5 e 3.3.6 só precisam ser concluídas na primeira vez que este programa for executado em qualquer computador. Se isso já tiver sido concluído, vá para a etapa 3.3.7. O processo de instalação pode levar alguns instantes para inicializar. - Pressione Enter. A execução desse comando instala as bibliotecas necessárias para executar esse script e instala o repositório em um modo editável nos ajustes de código de evento necessários (consulte a Figura 3D).
- Digite o seguinte na próxima linha de comando para alterar o diretório para o diretório principal do código-fonte (consulte a Figura 3E):
CD "nuclei_counter" - Na próxima linha de comando, digite o seguinte e pressione Enter (consulte a Figura 3E):
python count_nuclei.py --img_dir "caminho/para/imagens" --output_dir "caminho/para/saída" --save_images --output_count_filename "results.csv"
Onde caminho/para/imagens é o caminho do arquivo para a pasta que contém as imagens a serem analisadas (etapa 1.14); path/to/output é o caminho do arquivo para a pasta onde o arquivo .csv deve ser salvo (o padrão é o diretório de trabalho atual quando não fornecido); e results.csv é o nome do arquivo no qual as contagens resultantes serão salvas (se não for fornecido, o nome do arquivo de saída será padronizado para count_results.csv).
NOTA: As aspas são necessárias na linha de comando. Certifique-se de que não haja espaços após aspas antes do caminho do arquivo. O script python lerá os arquivos no caminho do arquivo fornecido e executará o programa de contagem de núcleos para todas as imagens no diretório (consulte a Figura 3E). As contagens resultantes serão salvas no arquivo de .csv de resultados no local especificado. - Quando a próxima linha de comando for exibida, o processamento estará concluído (consulte a Figura 3E). Os contornos e a planilha de resultados agora estão disponíveis no local especificado como o diretório de saída na etapa 3.3.8 (ou seja, caminho/para/saída).
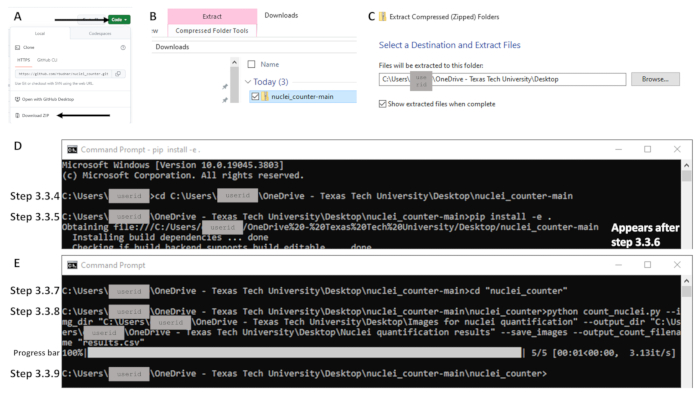
Figura 3: Exemplos de partes importantes do fluxo de trabalho para a seção 3 usando o sistema operacional Windows. Para acessar o código, clique no botão verde Código e (A) baixe o arquivo Zip (indicado por setas). (B) Extraia clicando com o botão direito do mouse no arquivo Zip (mostrado em azul) e (C) definindo o destino desejado. (D) Depois de abrir o prompt de comando, exemplos de comandos para as etapas 3.3.4 e 3.3.5. Observe que o comando para a etapa 3.3.5 está todo em uma linha; o texto que começa com "Obtendo arquivo" na próxima linha aparece após pressionar enter (etapa 3.3.6; ou seja, executar o comando). (E) Exemplos de comandos para as etapas 3.3.7 e 3.3.8, uma barra de progresso após a execução da etapa 3.3.8 e a linha de comando mostrando a conclusão na etapa 3.3.9. Clique aqui para ver uma versão maior desta figura.
- Use as instruções a seguir para Mac OS.
- Na pasta Downloads, clique no menu File e selecione Open para extrair os arquivos para o computador local (consulte a Figura 4A).
- No local do arquivo extraído, abra a pasta chamada nuclei_counter-main. Este é o repositório de código.
NOTA: Isso só precisa ser baixado na primeira vez que este programa for usado; Basta mover os arquivos extraídos para um local de rede acessível e anotar esse local. - Abra um terminal pressionando command + barra de espaço que abre o holofote; Em seguida, pesquise "terminal".
- Use o comando cd para alterar o diretório para o repositório de código e pressione Enter. Consulte a linha de código abaixo, que diferencia maiúsculas de minúsculas. Digite o caminho do arquivo em um documento de texto e copie-o para o terminal. Certifique-se de que o caminho do arquivo comece com o texto imediatamente anterior ao cd (por exemplo, /Users/userid na Figura 4B). Exemplo de linha de comando: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- Na próxima linha de comando, um cifrão aparecerá após o ID do usuário (consulte a Figura 4B). Certifique-se de que haja um espaço após o cifrão e digite o seguinte, incluindo o espaço e o ponto no final do comando. Inclua a versão apropriada do Python imediatamente após pip sem um espaço: pip3 install -e .
NOTA: As etapas 3.4.5 e 3.4.6 só precisam ser concluídas na primeira vez que este programa for executado em qualquer computador. Se isso já tiver sido concluído, vá para a etapa 3.4.7. O processo de instalação pode levar alguns instantes para inicializar. - Pressione Enter. A execução desse comando instala as bibliotecas necessárias para executar esse script e instala o repositório em um modo editável nos ajustes de código de evento necessários.
- Na próxima linha de comando, certifique-se de que haja um espaço após o cifrão e digite o seguinte para alterar o diretório para o diretório principal do código-fonte (consulte a Figura 4C):
CD "nuclei_counter" - Na próxima linha de comando, digite a linha de comando abaixo. Substitua o número após Python pelo número de versão correto se estiver usando uma versão diferente de 3. Em seguida, pressione Enter (consulte a Figura 4C):
python3 count_nuclei.py --img_dir "caminho/para/imagens" --output_dir " caminho/para/saída" --save_images --output_count_filename "results.csv"
onde caminho/para/imagens é o caminho do arquivo para a pasta que contém as imagens a serem analisadas (etapa 1.14); path/to/output é o caminho do arquivo para a pasta onde o arquivo .csv deve ser salvo (o padrão é o diretório de trabalho atual quando não fornecido); e results.csv é o nome do arquivo no qual as contagens resultantes serão salvas (se não for fornecido, o nome do arquivo de saída será padronizado para count_results.csv).
NOTA: As aspas são necessárias na linha de comando. Certifique-se de que não haja espaços após aspas antes do caminho do arquivo. O script Python lerá os arquivos no caminho do arquivo fornecido e executará o programa de contagem de núcleos para todas as imagens no diretório (consulte a Figura 4C). As contagens resultantes serão salvas nos resultados .csv arquivo no local especificado. - Quando a próxima linha de comando for exibida, o processamento estará concluído (consulte a Figura 4C). Os contornos e a planilha de resultados agora estão disponíveis no local especificado como o diretório de saída na etapa 3.4.8 (ou seja, caminho/para/saída).
- Inspecione visualmente os contornos para verificar a qualidade da contagem. Os resultados agora podem ser usados para normalizar os dados, conforme descrito na Seção 4.
NOTA: Se os contornos parecerem ter núcleos perdidos (por exemplo, núcleos parecem muito fracos) ou agrupar grupos de núcleos em um único item (por exemplo, imagem superexposta, muito fundo, artefatos), o usuário deve consultar a Tabela 1 e pode precisar retornar à Seção 1 para otimizar a aquisição da imagem antes de passar para a Seção 4.
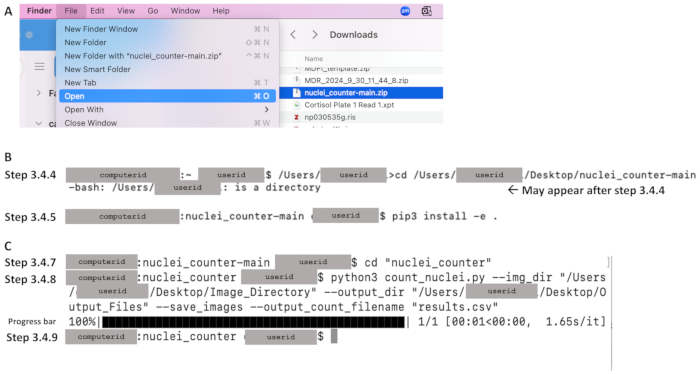
Figura 4: Exemplos de partes importantes do fluxo de trabalho para a seção 3 usando o Mac OS. Depois de baixar o código (ver Figura 3A), (A) extraia clicando no arquivo Zip (mostrado em azul) e definindo o destino desejado. (B) Depois de abrir o terminal, exemplos de comandos para as etapas 3.4.4 (alterar o diretório para o repositório de código) e 3.4.5 (instalar o pip). (C) Exemplos de comandos para as etapas 3.4.7 e 3.4.8, uma barra de progresso após a execução da etapa 3.4.8 e a linha de comando mostrando a conclusão na etapa 3.4.9. Clique aqui para ver uma versão maior desta figura.
4. Usando contagens para normalizar dados experimentais
- Abra o arquivo results.csv que vincula cada imagem com a quantificação de núcleos determinada usando o programa automatizado.
- Decida sobre o método de normalização: 1) imagem por imagem (por exemplo, se microscopia de fluorescência multicolorida), 2) normalizar os dados como uma proporção da contagem média de núcleos por amostra versus uma amostra de referência (ou condição), 3) normalizar para a contagem de núcleos por amostra (por exemplo, extrapolar a contagem total de núcleos para a área da amostra). As recomendações para o método de normalização estão na seção Discussão.
- Se a normalização for realizada imagem por imagem, basta dividir os dados experimentais de cada imagem pelo número de núcleos dessa imagem.
- Para normalizar os dados como uma proporção, siga as etapas descritas abaixo.
- Para quaisquer amostras em que várias imagens foram coletadas para uma única réplica (por exemplo, 10-15 imagens capturadas de um único poço em uma placa de 6 poços), calcule o número médio de núcleos por imagem. Essas contagens de núcleos são consideradas como a contagem de células assumindo células uninucleadas ou um número de células fundidas em células multinucleadas (como com miotubos).
- Selecione uma amostra de referência ou uma condição de referência (por exemplo, contagem média em todas as amostras da condição de controle) e anote a contagem.
- Divida a contagem de todas as amostras por essa contagem de referência, resultando em um valor de normalização. Se uma amostra de referência foi usada, use o valor de normalização para a amostra de referência como 1.
NOTA: Se uma condição de referência fosse usada, o valor médio de normalização na condição agora seria 1. Amostras com menos núcleos do que a referência (ou seja, menos densas) terão valores de normalização menores que 1, e amostras com mais núcleos do que a referência (ou seja, mais densas) terão valores de normalização maiores que 1. - Divida os dados experimentais de cada poço pelo valor de normalização calculado correspondente.
- Para normalizar os dados para a contagem de núcleos por amostra, siga as etapas descritas abaixo.
- Para quaisquer amostras em que várias imagens foram coletadas para uma única réplica (por exemplo, 10-15 imagens capturadas de um único poço em uma placa de 6 poços), calcule o número médio de núcleos por imagem. Essas contagens de núcleos são consideradas como a contagem de células assumindo células uninucleadas, ou número de células fundidas em células multinucleadas (como com miotubos).
- Determine a área de superfície da lâmina (lamínula), poço ou prato do qual os dados foram derivados. Essas informações podem ser encontradas na seção de especificações nos sites dos produtos.
- Determine a área de cada imagem capturada. Essas informações geralmente estão disponíveis como metadados no software de imagem ou análise.
- Certifique-se de que a área da superfície da amostra e a área da imagem sejam expressas nas mesmas unidades (por exemplo, mm2). Caso contrário, converta para a unidade desejada.
- Calcule a contagem total para cada amostra usando a seguinte equação:
TC = AC • (SA/IA)
onde TC é a contagem total, AC é a contagem média, SA é a área de superfície, IA é a área da imagem. - Divida os dados experimentais pelo TC correspondente para a amostra.
- Normalize os dados experimentais e use para análises estatísticas, conforme apropriado.
Resultados
Cada execução de imagem em lote produz: 1) um conjunto de arquivos de imagem com contornos aplicados mostrando os contornos dos núcleos identificados (Figura 5) e 2) um arquivo de .csv (planilha) vinculando os nomes dos arquivos de imagem e as contagens associadas. A visualização dos contornos permitirá que o usuário avalie visualmente a qualidade da contagem. Especificamente, as imagens obtidas de acordo com a seção 1 devem ter todos (ou quase todos) os núcleos cercados por uma linha verde sólida indicando que o núcleo foi contado pelo programa. Esses contornos foram usados para ajustar o programa aqui.
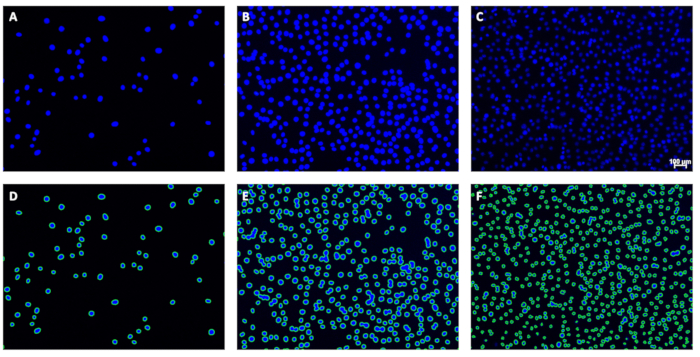
Figura 5: Exemplos de imagens originais e contornos correspondentes. (AC) Exemplos de imagens originais antes da análise e (D-F) contornos correspondentes após a análise diretamente abaixo. A barra de escala (C) indica 100 μm; As barras de escala devem ser omitidas das imagens usadas para quantificação de núcleos. Clique aqui para ver uma versão maior desta figura.
Validação usando resultados iniciais
Os mioblastos C2C12 em densidades celulares variadas foram fixados em metanol, contracorados com DAPI em meios de montagem e 120 imagens foram obtidas com aumento de 10x em .jpg formato de arquivo. O programa automatizado foi executado com o Diretório de imagens definido para a pasta que contém essas imagens e o arquivo .csv resultante foi salvo. Separadamente, cada imagem foi quantificada manualmente por dois pesquisadores treinados que estavam cegos para as contagens automatizadas e para as contagens uns dos outros. Imagens brutas, contornos gerados pelo programa automatizado e uma planilha de resultados, incluindo contagens manuais e automatizadas, estão disponíveis no repositório Open Science Framework (OSF) (consulte o link na seção 1). Os coeficientes de correlação intraclasse (ICCs; medidas médias) foram calculados usando o SPSS Statistics para determinar a confiabilidade entre os avaliadores entre 1) as duas contagens manuais e 2) a média das duas contagens manuais e contagens obtidas usando o programa automatizado. Abordagens semelhantes têm sido utilizadas para validar outros programas destinados a quantificar objetos em imagens obtidas por microscopia de fluorescência 16,17,18. Os ICCs também foram calculados entre contagens manuais médias e contagens automatizadas para cada quartil para garantir que a avaliação de todo o conjunto de dados não mascarasse nenhuma mudança potencial na confiabilidade com base na densidade celular. Os quartis foram definidos usando os pontos de cortedos percentis 25, 50 e 75 com base na contagem manual média e variam do quartil 1 (Q1; menor densidade celular) a Q4 (maior densidade celular). Os CCIs são relatados com base em medidas médias e interpretados com base nas seguintes definições: ruim (CCI <0,50), moderado (0,50 ≤CCI <0,75), bom (0,75 ≤CCI <0,90) ou excelente (CCI ≥0,90)19.
Os núcleos por imagem variaram de 44,5 a 1160 com base nas médias de contagem manual. A confiabilidade interavaliadores foi excelente entre as duas contagens manuais (ICC > 0,999, p < 0,0001; Figura 6A) e entre as contagens manuais e automatizadas médias (CCI = 0,993, p < 0,0001; Figura 6B). A confiabilidade permaneceu excelente em todos os quartis (Q1: ICC = 0,996, p < 0,0001; Q2: CCI= 0,997, p < 0,0001; Q3: CCI = 0,998, p < 0,0001; Q4: CCI = 0,986, p < 0,0001; Figura 6C-F). O único ponto de dados longe da linha de melhor ajuste na Figura 6B representa a imagem mostrada na Figura 7. A imagem possui várias áreas de múltiplos núcleos agrupados, impedindo que o programa de visão computacional reconheça cada núcleo. Esse problema pode ser corrigido usando um tempo de exposição mais curto. A Tabela 1 descreve como lidar com essa imagem.
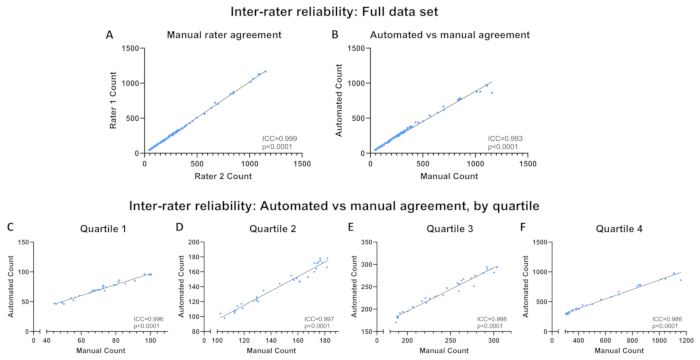
Figura 6: Confiabilidade entre avaliadores. Confiabilidade entre avaliadores em todo o conjunto de dados (A) entre contagens manuais de avaliadores e (B) entre contagens automatizadas e contagens manuais médias. (C-F) Confiabilidade entre avaliadores entre contagens automatizadas e contagens manuais médias por quartil. Confiabilidade expressa como coeficiente de correlação intraclasse (CCI). Clique aqui para ver uma versão maior desta figura.

Figura 7: Exemplo de uma imagem com duas barreiras para a quantificação precisa de núcleos. (A) Imagem original e (B) contornos associados. A seta 1 indica uma área com vários núcleos agrupados, distorcendo a forma nuclear redonda o suficiente para impedir a detecção de vários núcleos pelo software. Isso ocorre em vários locais ao longo desta imagem. A seta 2 indica um halo desbotado aparecendo atrás de vários núcleos; Esse halo parecia brilhante o suficiente para que o software o incluísse como um núcleo, perdendo assim a contagem dos múltiplos núcleos que o cerca. Esta imagem representa o único ponto de dados longe da linha de melhor ajuste na Figura 6B, enfatizando os problemas potenciais com essas barreiras. A barra de escala (A) indica 100 μm; As barras de escala devem ser omitidas das imagens usadas para quantificação de núcleos. Clique aqui para ver uma versão maior desta figura.
Discussão
Nosso programa de quantificação de núcleos tem várias vantagens sobre as opções existentes: requer apenas habilidades tecnológicas mínimas, é validado para a tarefa específica de quantificação de núcleos e é de código aberto; o último supera as barreiras relacionadas aos custos. Em última análise, este programa fornece aos biólogos celulares e moleculares uma opção adicional para quantificar núcleos com rapidez e precisão em imagens capturadas usando microscopia de fluorescência. Os programas automatizados de contagem de núcleos ou células atualmente disponíveis não são preferidos por todos os usuários, alguns têm um custo proibitivo e a validação pode ser mínima ou ausente para aplicações específicas. Equipamentos especializados, como a linha BioTek Cytation, são acompanhados por boletins técnicos que validam a contagem de células in situ usando coloração nuclear20, além de uma ampla variedade de aplicações adicionais. Embora existam dados de validação e a linha de produtos Cytation seja amplamente utilizada, especialmente quando conectada com analisadores de fluxo extracelular Seahorse para normalizar dados de células vivas com facilidade e eficiência, este produto pode ter um custo proibitivo para laboratórios com financiamento limitado de equipamentos disponíveis. Por exemplo, o preço de tabela para o modelo básico equipado com quatro objetivas e quatro cubos de filtro para microscopia de fluorescência ultrapassou US$ 100.000 dólares americanos no ano civil de 2024. Para aqueles sem meios para adquirir equipamentos especializados, o software de código aberto oferece alternativas gratuitas para análise de imagens. O software de código aberto útil para quantificação de núcleos e células inclui o ImageJ (National Institutes of Health, Bethesda, MD, EUA; disponível em 21. Os plug-ins agora estão disponíveis para aqueles que desejam escrever scripts de macro para automação de processamento de imagem usando a plataforma ImageJ. A automação da contagem de células baseada em ImageJ usando contraste de fase ou microscopia de campo claro com imagens de células inteiras foi descrita anteriormente22,23. O CellProfiler é um aplicativo amplamente publicado para análise de imagens celulares coradas com corantes fluorescentes ou colorações histológicas padrão, e um artigo inicial descrevendo sua utilidade em uma variedade de aplicações biológicas é frequentemente citado como validação24. No entanto, este manuscrito não valida nenhum achado biológico específico, necessitando de validação e transparência adicionais para vários pipelines de automação usados, como para contagem de núcleos25. Além disso, embora o CellProfiler seja um pacote de software eficiente para análise celular, nem todos os cientistas o consideram fácil de usar. Portanto, objetivamos desenvolver e validar um programa automatizado alternativo para quantificar núcleos usando software de código aberto. Especificamente, empregamos bibliotecas de visão computacional existentes em Python para desenvolver esse fluxo de trabalho.
Este método começa com células ou tecidos que o usuário final corou usando corantes de DNA, como DAPI26. São fornecidas diretrizes gerais para capturar e salvar imagens de maneira compatível com o software de automação. Embora os detalhes possam diferir com diferentes microscópios e softwares de imagem, o fluxo de trabalho geral para a seção 1 é o mesmo para obter imagens quantificáveis. Se estiver usando amostras vivas, deve-se tomar cuidado para garantir que o equipamento de proteção individual apropriado seja usado e o ambiente de imagem seja apropriado para essas amostras (por exemplo, temperatura, porcentagem de CO2, adição de tampão, etc.)27. Embora este protocolo descreva imagens apenas dos corantes de DNA, os usuários podem capturar outras cores da mesma área para fins de fusão15 e simplesmente copiar as imagens nucleares em uma pasta separada para uso com o programa de quantificação. Os exames de imagem devem ser realizados em uma sala escura com EPI apropriado para amostras vivas e a desinfecção adequada deve ser usada conforme necessário (por exemplo, com etanol a 70%). As amostras coradas devem ser protegidas da luz antes de acender as luzes. A captura de imagens de qualidade é fundamental para a quantificação precisa dos núcleos. Mesmo em imagens com tempo de exposição adequado e forte contraste entre núcleos brilhantes e um fundo preto, a presença de células excessivamente aglomeradas ou halos atrás de grupos de células (ver Figura 7) pode diminuir a precisão da quantificação. Nesses casos, os tempos de exposição podem precisar ser alterados para diminuir a intensidade dos sinais, garantindo a fusão mínima dos sinais dos núcleos e a minimização dos halos, mantendo um sinal forte o suficiente para detectar as formas redondas dos núcleos. Se essas etapas forem insuficientes, pode ser necessário obter imagens de uma seção adjacente da amostra. Todas as imagens de um único experimento devem ser capturadas com a mesma ampliação, e a ampliação correspondente à objetiva utilizada deve ser definida corretamente no software de imagem. É especialmente crítico que a objetiva usada para imagens corresponda às configurações do software de imagem quando a contagem de núcleos for extrapolada da área de imagem para toda a área do poço (ou lâmina). Esta etapa deve ocorrer sempre que a objetiva for trocada, mas é facilmente esquecida ao configurar o microscópio e o software. O tempo necessário para a seção 1 varia de acordo com vários fatores (por exemplo, experiência do usuário com microscopia de fluorescência, número de imagens e amostras, microscópio, etc.). Para capturar 10-15 imagens de núcleos corados por poço em duas placas de 6 poços, o usuário deve reservar aproximadamente 1-2 h de tempo.
Depois que as imagens são capturadas e salvas em uma pasta dedicada, elas estão prontas para serem quantificadas. O método mais simples de quantificação é executar este programa automatizado pré-empacotado sem a necessidade de instalar o Python ou qualquer biblioteca (seção 2). Deve-se notar que o programa é preparado como um executável e foi empacotado usando o Pyinstaller. O software criado usando o Pyinstaller é comumente sinalizado pelo software antivírus. Se o software antivírus bloquear a abertura do arquivo, o usuário deverá sinalizar o arquivo como seguro para prosseguir. Isso é fundamental para executar com sucesso a seção 2. Como alternativa, qualquer usuário pode optar por executar o programa de quantificação automatizado como um script Python (seção 3) em vez de um executável, se desejar. Se o usuário deseja executar o programa usando um sistema operacional Mac ou Linux, o programa deve ser executado por meio do Python. Este método não requer Pyinstaller, mas pode ser mais desafiador tecnicamente. O protocolo passo a passo, juntamente com exemplos, detalhados na seção 3, visa remover as barreiras técnicas para executar este programa como um script. Se a seção 2 ou a seção 3 é usada para executar o programa, é fundamental garantir que o código seja inserido corretamente nos prompts de comando. Exemplos de linhas de código são incluídos em todo o protocolo, e o usuário deve prestar muita atenção ao espaçamento, aspas e outras pontuações. O usuário também deve garantir que os caminhos do arquivo sejam precisos. É útil ter os caminhos de arquivo para as imagens e a saída disponíveis em um documento do Word. Os comandos também podem ser digitados neste documento do Word e copiados/colados conforme necessário.
Conforme descrito na seção 4, existem várias abordagens para normalizar dados experimentais usando contagens obtidas neste programa automatizado de quantificação de núcleos. Para experimentos baseados em imagens em que os dados experimentais incluem um número de eventos em cada imagem e uma proporção é desejada (por exemplo, proporção de células positivas para anexina V28, a proporção de células que expressam receptores ACE229, número de células que absorvem partículas coradas30, etc.), os dados experimentais para cada imagem podem simplesmente ser divididos pela contagem resultante (resultando em uma proporção de 0 a 1). Em seguida, a proporção média pode ser calculada em imagens replicadas da mesma lâmina, prato ou poço. Se uma porcentagem for desejada, basta multiplicar as proporções finais por 100. Finalmente, essas proporções ou porcentagens médias podem ser usadas em análises estatísticas a jusante. Quando os dados experimentais a serem normalizados são de experimentos funcionais em células vivas (por exemplo, taxas de consumo de oxigênio 6,11,12) ou de sobrenadante de cultura de células (por exemplo, atividade da lactato desidrogenase no sobrenadante31 ou concentração de uma citocina secretada específica32, etc.), as contagens de núcleos devem primeiro ser calculadas em todas as imagens capturadas do mesmo poço. Em seguida, a contagem de núcleos pode ser extrapolada para a área de superfície da lâmina (lamínula), poço ou prato, ou a contagem pode ser expressa como uma razão versus uma amostra ou condição de referência. A extrapolação para a área de superfície da lâmina, poço ou prato é útil para expressar dados experimentais versus o número de células (se estiver usando células uninucleadas) das quais os dados foram derivados (por exemplo, oxigênio consumido por minuto por 103 células). Para usar esse método, a área de superfície da lâmina (lamínula), poço ou prato e a área capturada em cada imagem devem ser conhecidas. Se a amostra não cobrir toda a área de superfície disponível, os dados experimentais forem de uma pequena quantidade de sobrenadante (ou seja, expressos como atividade ou concentração por unidade de volume) ou a imagem e/ou área de superfície não forem conhecidas, então a melhor opção é calcular a média das contagens de núcleos em todas as imagens para uma única amostra e expressar a contagem de núcleos em relação a uma amostra ou condição de referência. O valor resultante é usado para corrigir dados experimentais para densidade de núcleos ou densidade celular (se estiver usando células uninucleadas).
Embora esse método seja simples de executar, existem vários problemas potenciais sobre os quais os usuários devem estar cientes. Esses problemas potenciais, juntamente com as possíveis causas e etapas de solução de problemas para resolver os problemas, estão listados na Tabela 1. O programa descrito aqui tem vantagens importantes em comparação com o software existente; no entanto, várias limitações devem ser reconhecidas. A principal desvantagem é que o programa de quantificação só pode ser usado com imagens capturadas usando microscopia de fluorescência, não imagens de campo claro. Embora as imagens coradas com DAPI capturadas com ampliação de 10x tenham sido usadas para construir o programa, o programa pode ser usado com outros corantes e ampliações de DNA, uma vez que o programa é construído para reconhecer a morfologia dos núcleos em vez de cor ou tamanho. Embora tenhamos validado a precisão do programa em imagens contendo uma variedade de densidades celulares, o usuário final pode ter densidades celulares que estão fora do intervalo. Nesse caso, ainda prevemos contagens precisas, desde que imagens de qualidade sejam obtidas; no entanto, o usuário deve verificar cuidadosamente os contornos e a saída para garantir a precisão. Para aplicações de cultura de células, a quantificação de núcleos em imagens é mais apropriada para células em monocamada. Se as células cultivadas se sobrepõem, os núcleos podem ser mascarados, resultando em uma subestimação da quantidade de núcleos. A natureza bidimensional da quantificação de núcleos em imagens impede o uso deste programa para determinar o número total de núcleos de células em suspensão, blocos de tecido espesso ou outras aplicações tridimensionais. No entanto, este programa ainda pode quantificar núcleos marcados com corantes de DNA fluorescente em imagens bidimensionais capturadas de modelos tridimensionais, como blocos de tecido espesso. A utilidade de quantificar núcleos em imagens para a aplicação desejada deve ser determinada pelo usuário final.
| Problema | Possíveis causas | Soluções possíveis |
| Fundo excessivo nas imagens | Tempo de exposição muito alto | Reduza o tempo de exposição |
| Os núcleos parecem se fundir uns com os outros em imagens | Concentração de corante de DNA fluorescente muito alta ou muita mídia de montagem de corante de DNA | Reduzir a concentração de corante durante a otimização; Use a menor quantidade possível de mídia de montagem |
| Os núcleos parecem muito claros ou alguns núcleos não são visíveis | Tempo de exposição muito baixo | Aumente o tempo de exposição |
| Fotobranqueamento | Evite o fotobranqueamento, garantindo que as amostras manchadas permaneçam armazenadas no escuro e minimizando a exposição das amostras à iluminação (por exemplo, usando o tempo mínimo de exposição necessário e desligando a iluminação quando não for necessária, como depois que uma imagem é capturada ou ao fazer anotações) | |
| Efeitos de halo ou formas não específicas do núcleo aparecendo | Tempo de exposição muito alto | Reduza o tempo de exposição |
| Fiapos de limpadores de tarefas delicadas ou alguns protetores de bancada em células vivas ou aderidos a células fixas | Garanta um ambiente livre de fiapos: minimize o uso de limpadores de tarefas delicadas quando as amostras são expostas (por exemplo, em cabines de biossegurança para células vivas, durante a fixação e secagem de células fixas ou durante o processo de coloração); Certifique-se de que os protetores de bancada não deixem fiapos nas lâminas ou placas durante a coloração | |
| Outras anormalidades na superfície de células/tecidos fixos | Manuseie as amostras com extremo cuidado e siga as devidas precauções e práticas recomendadas durante a fixação e coloração | |
| Distúrbios no caminho da luz | Certifique-se de que todas as partes do microscópio estejam limpas e livres de fiapos e poeira; Limpe a parte externa das lâminas/pratos/pratos com etanol a 70% para remover poeira e fiapos ambientais antes da imagem | |
| O arquivo executável não abre | O software antivírus bloqueou o arquivo | Sinalize o arquivo como seguro |
| Erros em prompts de comando | Problemas com espaçamento na linha de comando | Verifique se o início do comando é digitado imediatamente após o símbolo ">" na linha de comando sem espaço, se não há espaços presentes após as aspas abertas para caminhos de arquivo e se o espaçamento corresponde ao código fornecido e aos exemplos. |
| Casos impróprios usados na linha de comando | O código diferencia maiúsculas de minúsculas; Certifique-se de usar letras maiúsculas e minúsculas conforme apropriado | |
| Problemas com o caminho do arquivo | Certifique-se de que o caminho do arquivo indique o interior da pasta de arquivos associada (ou seja, a pasta de arquivos está aberta quando o caminho do arquivo é copiado). | |
| Problemas com o programa que não são resolvidos usando as etapas de solução de problemas acima | Problemas de software ou linha de comando mencionados acima. | Envie um problema via GitHub. Navegue até https://github.com/rbudnar/nuclei_counter. Na barra de menu na parte superior, selecione "Problemas" (segunda opção, à direita de "Código"). Clique no botão verde no lado direito que diz "Novo problema" para enviar. Haverá um prompt para fazer login no GitHub antes de enviar o problema. Haverá também a opção de criar uma conta gratuita, se necessário. |
Tabela 1: Solução de problemas para capturar imagens de qualidade e executar o fluxo de trabalho automatizado de quantificação de núcleos.
No geral, os métodos descritos aqui fornecem detalhes passo a passo para usar o programa automatizado de quantificação de núcleos que desenvolvemos usando bibliotecas de visão computacional e validados em relação a contagens manuais de vários avaliadores. Este programa foi desenvolvido para garantir a usabilidade por cientistas com vários níveis de habilidades tecnológicas relacionadas à codificação, está disponível gratuitamente e foi validado especificamente para a tarefa de quantificação de núcleos. Comparado com a contagem manual, este programa economizará um tempo valioso do usuário final e fornecerá contagens de núcleos precisas e confiáveis para normalização de dados.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
O financiamento para este trabalho foi fornecido pelos Institutos Nacionais de Saúde/Instituto Nacional do Envelhecimento (R01AG084597; DEL e HYL) e por fundos iniciais da Texas Tech University (DEL). Os autores gostariam de agradecer aos programas Texas Tech University Undergraduate Research Scholars e TrUE Scholars por fornecer apoio financeiro aos pesquisadores de graduação que contribuíram para este trabalho (REH, MRD, CJM, AKW). Também agradecemos aos Drs. Lauren S. Gollahon e Michael P. Massett por compartilharem graciosamente seu espaço e equipamentos de laboratório.
Materiais
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
Referências
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados