Method Article
Uso de bibliotecas de visión artificial para optimizar la cuantificación de núcleos
En este artículo
Resumen
Este artículo describe métodos paso a paso para automatizar la cuantificación de núcleos basada en imágenes utilizando un programa ejecutable de código abierto validado en una variedad de densidades celulares. Este programa proporciona una alternativa que aborda las barreras relacionadas con el costo, la accesibilidad para los usuarios con habilidades tecnológicas limitadas y la validación específica de la aplicación que puede limitar la utilidad de las tecnologías existentes.
Resumen
Los ensayos de células vivas y los análisis de células basadas en imágenes requieren la normalización de los datos para una interpretación precisa. Un método comúnmente utilizado es la tinción y cuantificación de núcleos, seguida de la normalización de datos para el recuento de núcleos. Este recuento de núcleos a menudo se expresa como recuento de células para células uninucleadas. Si bien la cuantificación manual puede ser laboriosa y llevar mucho tiempo, es posible que todos los usuarios no prefieran los métodos automatizados disponibles, que carezcan de validación para esta aplicación específica o que tengan un costo prohibitivo. Aquí, proporcionamos instrucciones paso a paso para capturar imágenes cuantificables de núcleos teñidos con tinciones de ADN fluorescente y, posteriormente, cuantificar los núcleos utilizando un programa de software de conteo de objetos automatizado desarrollado con bibliotecas de visión artificial de Python. También validamos este programa en un rango de densidades celulares. Aunque el tiempo exacto de ejecución del programa varía en función del número de imágenes y del hardware del ordenador, este programa consolida horas de trabajo contando los núcleos en segundos para que el programa se ejecute. Si bien este protocolo se desarrolló utilizando imágenes de células fijas y teñidas, las imágenes de núcleos teñidos en células vivas y las aplicaciones de inmunofluorescencia también se pueden cuantificar con este programa. En última instancia, este programa proporciona una opción que no requiere un alto grado de habilidad tecnológica y es una alternativa validada y de código abierto para ayudar a los biólogos celulares y moleculares a optimizar sus flujos de trabajo, automatizando la tediosa y lenta tarea de cuantificación de núcleos.
Introducción
Los experimentos funcionales y basados en imágenes son fundamentales para comprender los impactos de los tratamientos experimentales en la bioquímica y la fisiología de las células completas. La interpretación válida de los datos de los experimentos de biología celular depende de la precisión y reproducibilidad del protocolo experimental, incluida la normalización de los datos. Por ejemplo, los análisis del consumo de oxígeno y de las tasas de acidificación extracelular en células vivas al inicio y después del tratamiento con fármacos específicos permiten evaluar diversos aspectos del metabolismo energético 1,2. La medición de la actividad de enzimas como la lactato deshidrogenasa en el sobrenadante de los cultivos celulares puede ayudar a cuantificar la integridad de la membrana celular3. La tinción de las células cultivadas con anexina V y yoduro de propidio antes de la fijación permite la evaluación de las células apoptóticas y necróticas4. Sin embargo, las diferencias en la densidad celular entre los pocillos afectan los resultados de cada uno de estos ensayos. Confiar únicamente en la densidad de siembra podría proporcionar resultados engañosos debido a errores en el recuento de células para la siembra, variaciones en la densidad celular en el medio durante la siembra o diferentes tasas de crecimiento celular entre muestras o tratamientos a lo largo de los experimentos. Por lo tanto, se requiere la normalización de los resultados experimentales.
Los métodos actuales de normalización para datos celulares funcionales y basados en imágenes incluyen la concentración de proteínas5 o núcleos o recuento de células6. Para normalizar los datos a la concentración de proteínas, las células deben extraerse en volúmenes iguales de tampón de lisis con inhibidor de proteasa después de ejecutar el ensayo, y se debe realizar un ensayo adicional (por ejemplo, ensayo de ácido bicinconínico, ensayo de Bradford, etc.) para la cuantificación de proteínas. La precisión de este método se basa en la extracción completa de células; Dejar atrás cualquier proteína contribuye a errores en la cuantificación. El recuento de núcleos o células proporciona una alternativa en la que no es necesario extraer células de la superficie en la que se cultivan. En su lugar, las células vivas o fijas se tiñen o contratiñen mediante tinciones colorimétricas o fluorescentes, y el recuento se puede realizar basándose en imágenes de células enteras o solo de núcleos. Se prefiere la cuantificación de núcleos en lugar de células enteras para aplicaciones de microscopía de fluorescencia y para normalizar datos en células fusionadas (por ejemplo, miotubos) o tejidos. Para la cuantificación de núcleos mediante un enfoque de fluorescencia, se aplican colorantes fluorescentes de ADN (por ejemplo, dicloruro de 4′,6-diamidino-2-fenilindol (DAPI), Hoechst 33342, Hoechst 33358, DRAQ5, etc.) a células vivas o fijas y se obtienen imágenes mediante microscopía de fluorescencia. DAPI, Hoechst y DRAQ5 se unen preferentemente a las regiones ricas en adenina-timina del ADN 7,8,9, proporcionando un medio para detectar núcleos. La optimización de la concentración de colorante evita la unión fuera del objetivo (es decir, al ADN mitocondrial, lo que provoca fluorescencia citosólica punteada)10. El proceso de optimización puede simplificarse mediante el uso de medios de montaje disponibles en el mercado que contengan DAPI para celdas fijas (p. ej., VECTASHIELD Antifade Mounting Medium con DAPI) y concentraciones publicadas de tinciones para celdas vivas (p. ej., Hoechst)6,11,12. Una vez teñidos, los núcleos de las células vivas o fijas se visualizan utilizando el filtro de excitación adecuado (por ejemplo, ultravioleta [DAPI y Hoechst] o rojo lejano [DRAQ5]). Los máximos de excitación para DAPI, Hoechst y DRAQ5 se encuentran en ambos extremos del espectro para la microscopía de fluorescencia. Este atributo, además de la especificidad del ADN de estos colorantes y su uso en concentraciones óptimas, minimiza la fluorescencia fuera del objetivo. Las imágenes resultantes muestran núcleos brillantes sobre un fondo negro, y estos núcleos se cuantifican como una medida del número de células para células uninucleadas. Si bien existen otros métodos precisos de cuantificación de núcleos basados en tinciones que utilizan tinciones histológicas y microscopía de campo claro13, la automatización es más difícil, aunque posible, utilizando esos enfoques.
Si bien sigue siendo el estándar de oro, la cuantificación manual de núcleos es laboriosa, requiere mucho tiempo y es susceptible a posibles errores humanos con un conteo prolongado. Aunque existen programas de conteo automatizado de células, no son los preferidos por todos los usuarios, pueden tener un costo prohibitivo y la validación para aplicaciones específicas puede ser mínima. Python se ha convertido en un recurso valioso y accesible para los biólogos en los últimos años. Las bibliotecas de visión artificial de Python son particularmente útiles para el análisis de imágenes14. Aquí, proporcionamos instrucciones paso a paso para usar nuestro programa ejecutable desarrollado con código Python para cuantificar núcleos teñidos con tintes fluorescentes y fotografiados con microscopía de fluorescencia. No es necesario tener conocimientos de codificación para utilizar el programa descrito aquí. Este flujo de trabajo es específico de células o tejidos a los que se les han aplicado tintes fluorescentes de ADN. No está diseñado para su uso con imágenes de campo claro. En la sección 1 se describe cómo capturar y guardar las imágenes recogidas mediante microscopía de fluorescencia de forma compatible con este flujo de trabajo. La sección 2 proporciona instrucciones para ejecutar el programa de cuantificación de núcleos como un ejecutable y obtener salida. El programa ejecutable se puede ejecutar directamente desde el archivo de .exe proporcionado y no requiere modificaciones para cuantificar los núcleos en imágenes de calidad. Este ejecutable requiere una PC basada en Windows. La sección 3 proporciona instrucciones para ejecutar el programa como un script de Python en lugar de un ejecutable (necesario para sistemas basados en Mac o Linux, opcional para sistemas basados en Windows) y permite ajustar el código si se desea. En la sección 4 se describe cómo utilizar los resultados para la normalización. A estos protocolos les siguen resultados representativos, incluidos los datos de validación de nuestro laboratorio.
Protocolo
NOTA: Los archivos complementarios se pueden encontrar en el siguiente enlace https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. Captura y almacenamiento de imágenes mediante microscopía de fluorescencia
- Prepare las muestras de células o tejidos para obtener imágenes, incluida la tinción con el tinte de ADN deseado. Para obtener las imágenes aquí utilizadas, los mioblastos C2C12 (CRL-1772, American Type Culture Collection) se cultivaron en placas de 6 pocillos durante 48-72 h en condiciones de cultivo estándar (5% CO2, 37 °C, humidificado), con o sin 50 mM de EtOH6, y se fijaron en metanol helado como se describió anteriormente13. Las células fijas se montaron utilizando un medio de montaje15 que contenía DAPI y se secaron en la oscuridad antes de la obtención de imágenes.
- Encienda todos los componentes del microscopio de fluorescencia y la computadora de acuerdo con los procedimientos operativos estándar establecidos. Abra el software asociado con el microscopio.
- Configure el microscopio para visualizar las muestras a través del ocular. Revisa el objetivo. Si es necesario, gire el revólver hacia el objetivo deseado. Asegúrese de que el programa de software tenga seleccionado el objetivo deseado para obtener una indicación precisa del área de la imagen. Aquí, las imágenes se capturaron con un objetivo 10x.
- Con las luces apagadas, coloque la muestra (portaobjetos, plato o plato) en el escenario. Activa las imágenes en directo con el filtro adecuado y ajusta el tiempo de exposición a una duración predeterminada o a la exposición automática. En este caso, se utilizó un filtro DAPI y un tiempo de exposición de 61-62 ms. Consulte la tabla de solución de problemas para obtener orientación sobre cómo configurar el tiempo de exposición, especialmente para imágenes con densidades de celdas altas.
- Con el ocular, ajuste lentamente el enfoque con la perilla de ajuste grueso hasta que los núcleos manchados sean visibles, luego use la perilla de ajuste fino para ajustar lentamente el enfoque hasta que los núcleos aparezcan claros y nítidos.
NOTA: En la Figura 1 se muestran ejemplos de imágenes apropiadas.- Si aparece una tinción de fondo excesiva o si los núcleos parecen muy tenues, ajuste el tiempo de exposición manualmente. En una sola imagen, puede haber diferencias visibles en la intensidad de la señal para algunos núcleos; Sin embargo, la forma redonda debe ser visible para todos los núcleos con una interferencia de señal mínima.
NOTA: El programa utilizado en las aplicaciones posteriores es robusto a diferentes intensidades. Las imágenes en bruto utilizadas para el desarrollo y la validación del programa de cuantificación de núcleos incluyen imágenes con un rango de intensidades de señal y están disponibles a través del repositorio Open Science Framework (ver enlace mencionado anteriormente).
- Si aparece una tinción de fondo excesiva o si los núcleos parecen muy tenues, ajuste el tiempo de exposición manualmente. En una sola imagen, puede haber diferencias visibles en la intensidad de la señal para algunos núcleos; Sin embargo, la forma redonda debe ser visible para todos los núcleos con una interferencia de señal mínima.
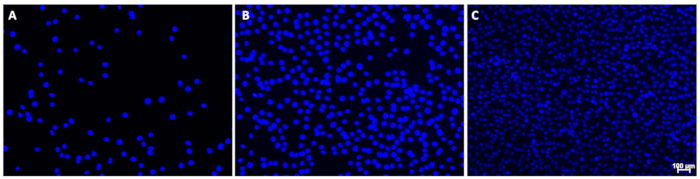
Figura 1: Imágenes de ejemplo de núcleos teñidos con DAPI. Estas imágenes muestran núcleos con densidades celulares (A) bajas, (B) medias o (C) altas con un fondo mínimo. Obsérvese la variación de la intensidad de la señal de los núcleos a través de la imagen C; Esto no interfirió con la cuantificación. La barra de escala (C) indica 100 μm; Las barras de escala deben omitirse de las imágenes utilizadas para la cuantificación de núcleos. Haga clic aquí para ver una versión más grande de esta figura.
- Una vez que los núcleos estén enfocados a través del ocular, cambie el modo de detección a la cámara. Ajuste el enfoque con la perilla de ajuste fino.
- Ajuste el tiempo de exposición si es necesario. Tenga en cuenta el tiempo de exposición final para su uso con muestras e imágenes posteriores.
NOTA: Aunque lo ideal es utilizar el mismo tiempo de exposición en todas las imágenes, las variaciones en el tiempo de exposición son aceptables para esta aplicación porque la intensidad de la fluorescencia no se cuantifica ni se compara entre muestras. - Capture la imagen utilizando el botón de captura de imagen apropiado en el software. Si las muestras tienen varias tinciones aplicadas, cambie el filtro (por ejemplo, a FITC, Tritc o Cy5) y obtenga una imagen de la misma área para fusionar.
- Aplique el obturador para minimizar la exposición de la muestra a la luz. Sin grabar en una barra de escala, guarde la imagen en un formato de archivo de figura universal (.tif, .png o .jpg) en una carpeta dedicada. Asegúrese de que cada nombre de archivo de imagen sea adecuadamente descriptivo o que cualquier codificación utilizada para los nombres de archivo tenga la documentación vinculada adecuada.
NOTA: Este se convertirá en el directorio de imágenes para el programa de cuantificación. Si se utilizaron imágenes multicolor, copie solo las imágenes de los núcleos en una carpeta separada y dedicada. Esto también se puede hacer al final de la sesión de imágenes. Las imágenes exportadas con la configuración de exportación predeterminada en la mayoría de los programas de procesamiento de imágenes suelen tener una resolución suficiente para su uso con este programa, siempre que se hayan capturado imágenes de calidad. Sin embargo, las imágenes deben verificarse visualmente después de la exportación para asegurarse de que la morfología de los núcleos sea clara. Si las imágenes se guardan en .jpg formato, se debe tener cuidado de que las imágenes no se compriman excesivamente y se mantenga la calidad. - Abra el obturador, vuelva a las imágenes en vivo y escanee la muestra a otra ubicación para obtener imágenes. Repita los pasos 1.8 y 1.9 según sea necesario. En el caso de imágenes de células sembradas en placas de 6 pocillos con un aumento de 10x, capture de 10 a 15 imágenes por pocillo (dependiendo de la consistencia de la densidad de células en el área de la muestra).
- Una vez adquiridas todas las imágenes de una sola muestra, realice una de las siguientes opciones.
- Pase a la siguiente muestra y repita los pasos 1.4-1.10.
- Pausa el protocolo y vuelve en otro momento. Las muestras deben permanecer protegidas de la luz. Esta opción no se recomienda si se trabaja con células vivas.
- Una vez completadas las imágenes, asegúrese de que todas las muestras estén protegidas contra la luz antes de encender las luces de la habitación. Si la carpeta que contiene las imágenes se guarda localmente, transfiera los archivos a una unidad de memoria USB, una ubicación de almacenamiento en la nube u otra unidad de red accesible para el equipo donde se ejecutará el programa de cuantificación. Guarde en una carpeta dedicada.
- Apague todas las partes del microscopio y la computadora de acuerdo con los procedimientos operativos estándar establecidos.
- Las imágenes ya están completas. Analice los archivos utilizando el programa de cuantificación automatizada como se describe en la Sección 2 o, alternativamente, ejecutando el programa como un script de Python como se describe en la Sección 3.
2. Ejecución del programa automatizado de cuantificación de núcleos como ejecutable
NOTA: Este método, que ejecuta el programa como un ejecutable, es compatible con PC basadas en Windows y es el método recomendado para los usuarios del sistema operativo Windows. Mac OS u otros usuarios que no sean de Windows deben ejecutar el programa como un script de Python (consulte la Sección 3).
- Asegúrese de que Python esté instalado en la computadora. Si es necesario, instale Python desde python.org/downloads (preferido) o mediante Anaconda o Miniconda. A la fecha de publicación, este programa ha sido probado con las versiones 3.10-3.12 de Python.
- Abra el navegador y navegue hasta https://github.com/rbudnar/nuclei_counter/releases. Descargue la última versión del archivo denominado count_nuclei.zip.
- En la carpeta Descargas, haga clic con el botón derecho en el archivo Zip y extraiga todos los archivos a la ubicación deseada en el equipo local (consulte la Figura 2A). Mantenga abierta esta ubicación de archivo; esta ruta de archivo se utilizará en el paso 2.5 (consulte la Figura 2B).
NOTA: Count_nuclei debería aparecer ahora como un archivo de aplicación. Este es el archivo ejecutable. - Abra un símbolo del sistema buscando CMD o Símbolo del sistema en la barra de búsqueda. Utilice el comando cd para cambiar el directorio a la ruta de acceso del archivo ejecutable (consulte la figura 2C) y pulse Intro. Ejemplo de línea de comandos: C:\Users\janedoe>cd [espacio después del cd, luego copiar/pegar la ruta del archivo]
- En la siguiente línea de comandos, escriba lo siguiente y presione Entrar.
count_nuclei.exe --img_dir "ruta/a/imágenes" --output_dir "ruta/a/salida" --save_images --output_count_filename "results.csv"
donde ruta/a/imágenes es la ruta del archivo a la carpeta que contiene las imágenes que se van a analizar (consulte el paso 1.14); path/to/output es la ruta del archivo a la carpeta donde se debe guardar el archivo .csv (el valor predeterminado es el directorio de trabajo actual cuando no se proporciona); y results.csv es el nombre de archivo en el que se guardarán los recuentos resultantes (si no se proporciona, el nombre del archivo de salida se establece de forma predeterminada en count_results.csv).
NOTA: Las comillas son necesarias en la línea de comandos. Asegúrese de que no haya espacios después de las comillas antes de la ruta del archivo (consulte la figura 2C). - Cuando aparece la siguiente línea de comandos, el procesamiento se ha completado. Los contornos y la hoja de cálculo de resultados ahora están disponibles en la ubicación especificada como directorio de salida en el paso 2.5 (es decir, ruta/hacia/salida; consulte la Figura 2C).
- Inspeccione visualmente los contornos para verificar la calidad del recuento. Los resultados ahora se pueden utilizar para normalizar los datos, como se describe en la Sección 4.
NOTA: Si los contornos parecen tener núcleos perdidos (por ejemplo, los núcleos parecen demasiado débiles) o agrupan grupos de núcleos en un solo elemento (por ejemplo, imagen sobreexpuesta, demasiado fondo, artefactos), el usuario debe consultar la tabla de solución de problemas y es posible que deba volver a la Sección 1 para optimizar la adquisición de imágenes antes de pasar a la Sección 4.
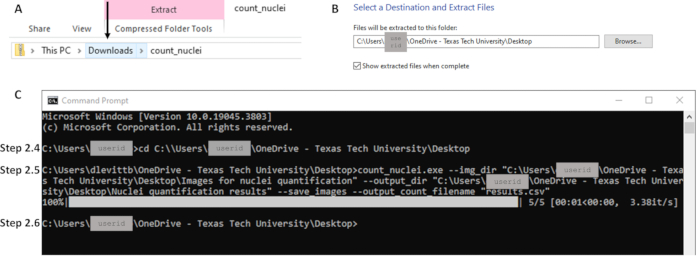
Figura 2: Ejemplos de partes clave del flujo de trabajo para la sección 2. (A) Extraiga los archivos de la carpeta Descargas, teniendo cuidado de hacer clic en la carpeta correcta como lo indica la flecha para permitir la extracción (B) al destino deseado en el paso 2.3. (C) Ejemplos de comandos para los pasos 2.4 y 2.5 y la siguiente línea de comandos que indica la finalización en el paso 2.6. Tenga en cuenta que la ruta del archivo en el paso 2.4 (C) coincide con la ubicación de extracción en el paso 2.3 (B). Haga clic aquí para ver una versión más grande de esta figura.
3. Ejecución del programa automatizado de cuantificación de núcleos como script de Python
- Asegúrese de que Python esté instalado en la computadora. Si es necesario, instale Python desde python.org/downloads (preferido) o mediante Anaconda o Miniconda. A la fecha de publicación, este programa ha sido probado con las versiones 3.10-3.12 de Python.
- Abra el navegador y navegue hasta https://github.com/rbudnar/nuclei_counter. Haga clic en el botón Código verde y, a continuación, haga clic en Descargar ZIP para descargar el repositorio de código (consulte la Figura 3A).
- Utilice las siguientes instrucciones para el sistema operativo Windows. Consulte el paso 3.4 para equipos basados en Mac OS.
- En la carpeta Descargas, haga clic con el botón derecho en el archivo y seleccione Extraer todo para extraer los archivos a un equipo local (consulte la figura 3B-C).
- En la ubicación del archivo extraído, abra la carpeta denominada nuclei_counter-main. Este es el repositorio de código.
NOTA: Esto solo necesita ser descargado la primera vez que se utiliza este programa; Simplemente mueva los archivos extraídos a una ubicación de red accesible y tome nota de esa ubicación. - Abra un símbolo del sistema buscando CMD o Símbolo del sistema en la barra de búsqueda. Si Anaconda o Miniconda han instalado Python, abra el símbolo del sistema de Anaconda en lugar de un símbolo del sistema estándar. Esto se puede encontrar escribiendo Anaconda Prompt en la barra de búsqueda.
- Utilice el comando cd para cambiar el directorio al repositorio de código y pulse Intro. Consulte la línea de código a continuación, que distingue entre mayúsculas y minúsculas. Asegúrese de que la carpeta denominada nuclei_counter-main esté abierta al copiar la ruta del archivo; el archivo denominado pyproject o pyproject.toml debe estar visible (consulte la figura 3D). Ejemplo de línea de comandos: C:\Users\janedoe>cd [espacio después del cd, luego copiar/pegar la ruta del archivo]
- En la siguiente línea de comandos, escriba lo siguiente, incluidos el espacio y el punto (consulte la figura 3D:
pip install -e .
NOTA: Los pasos 3.3.5 y 3.3.6 solo deben completarse la primera vez que se ejecuta este programa en cualquier computadora. Si esto ya se ha completado, continúe con el paso 3.3.7. El proceso de instalación puede tardar unos minutos en inicializarse. - Presione Entrar. Al ejecutar este comando, se instalan las bibliotecas necesarias para ejecutar este script e instala el repositorio en un modo editable en caso de que sea necesario realizar ajustes en el código (consulte la figura 3D).
- Escriba lo siguiente en la siguiente línea de comandos para cambiar el directorio al directorio de código fuente principal (consulte la figura 3E):
CD "nuclei_counter" - En la siguiente línea de comandos, escriba lo siguiente y presione Entrar (consulte la figura 3E):
python count_nuclei.py --img_dir "ruta/a/imágenes" --output_dir "ruta/a/salida" --save_images --output_count_filename "results.csv"
Donde ruta/a/imágenes es la ruta del archivo a la carpeta que contiene las imágenes que se van a analizar (paso 1.14); path/to/output es la ruta del archivo a la carpeta donde se debe guardar el archivo .csv (el valor predeterminado es el directorio de trabajo actual cuando no se proporciona); y results.csv es el nombre de archivo en el que se guardarán los recuentos resultantes (si no se proporciona, el nombre del archivo de salida se establece de forma predeterminada en count_results.csv).
NOTA: Las comillas son necesarias en la línea de comandos. Asegúrese de que no haya espacios después de las comillas antes de la ruta del archivo. El script de python leerá los archivos en la ruta de archivo proporcionada y ejecutará el programa de conteo de núcleos para todas las imágenes en el directorio (consulte la Figura 3E). Los recuentos resultantes se guardarán en el archivo de .csv de resultados en la ubicación especificada. - Cuando aparece la siguiente línea de comandos, el procesamiento se ha completado (consulte la figura 3E). Los contornos y la hoja de cálculo de resultados ahora están disponibles en la ubicación especificada como directorio de salida en el paso 3.3.8 (es decir, ruta/hacia/salida).
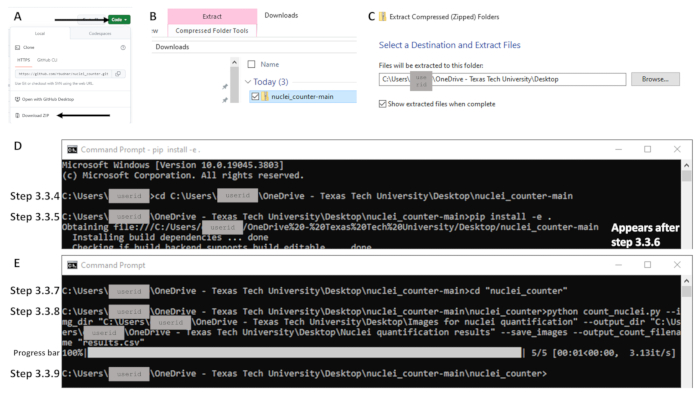
Figura 3: Ejemplos de partes clave del flujo de trabajo para la sección 3 con el sistema operativo Windows. Para acceder al código, haga clic en el botón verde con la etiqueta Código y (A) descargue el archivo Zip (indicado por flechas). (B) Extraiga haciendo clic con el botón derecho en el archivo Zip (que se muestra en azul) y (C) configurando el destino deseado. (D) Después de abrir el símbolo del sistema, ejemplos de comandos para los pasos 3.3.4 y 3.3.5. Tenga en cuenta que el comando para el paso 3.3.5 está todo en una línea; el texto que comienza con "Obteniendo archivo" en la siguiente línea aparece después de presionar enter (paso 3.3.6; es decir, ejecutar el comando). (E) Ejemplos de comandos para los pasos 3.3.7 y 3.3.8, una barra de progreso después de la ejecución del paso 3.3.8 y la línea de comandos que muestra la finalización en el paso 3.3.9. Haga clic aquí para ver una versión más grande de esta figura.
- Utilice las siguientes instrucciones para Mac OS.
- En la carpeta Descargas, haga clic en el menú Archivo y seleccione Abrir para extraer los archivos en el equipo local (consulte la Figura 4A).
- En la ubicación del archivo extraído, abra la carpeta denominada nuclei_counter-main. Este es el repositorio de código.
NOTA: Esto solo necesita ser descargado la primera vez que se utiliza este programa; Simplemente mueva los archivos extraídos a una ubicación de red accesible y tome nota de esa ubicación. - Abra un terminal presionando comando + barra espaciadora que abre el foco; A continuación, busca "terminal".
- Utilice el comando cd para cambiar el directorio al repositorio de código y pulse Intro. Consulte la línea de código a continuación, que distingue entre mayúsculas y minúsculas. Escriba la ruta del archivo en un documento de texto y cópielo en el terminal. Asegúrese de que la ruta del archivo comience con el texto inmediatamente anterior al cd (por ejemplo, /Users/userid en la Figura 4B). Ejemplo de línea de comandos: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- En la siguiente línea de comandos, aparecerá un signo de dólar después del ID de usuario (consulte la figura 4B). Asegúrese de que haya un espacio después del signo de dólar y escriba lo siguiente, incluidos el espacio y el punto al final del comando. Incluya la versión apropiada de Python inmediatamente después de pip sin un espacio: pip3 install -e .
NOTA: Los pasos 3.4.5 y 3.4.6 solo deben completarse la primera vez que se ejecuta este programa en cualquier computadora. Si esto ya se ha completado, continúe con el paso 3.4.7. El proceso de instalación puede tardar unos minutos en inicializarse. - Presione Entrar. Al ejecutar este comando, se instalan las bibliotecas necesarias para ejecutar este script e instala el repositorio en modo editable en caso de que sea necesario realizar ajustes en el código.
- En la siguiente línea de comandos, asegúrese de que haya un espacio después del signo de dólar y, a continuación, escriba lo siguiente para cambiar el directorio al directorio principal del código fuente (consulte la figura 4C):
CD "nuclei_counter" - En la siguiente línea de comandos, escriba la línea de comandos a continuación. Reemplace el número después de Python por el número de versión correcto si usa una versión que no sea 3. A continuación, pulse Intro (véase la figura 4C):
python3 count_nuclei.py --img_dir "ruta/a/imágenes" --output_dir "ruta/a/salida" --save_images --output_count_filename "results.csv"
donde ruta/a/imágenes es la ruta del archivo a la carpeta que contiene las imágenes que se van a analizar (paso 1.14); path/to/output es la ruta del archivo a la carpeta donde se debe guardar el archivo .csv (el valor predeterminado es el directorio de trabajo actual cuando no se proporciona); y results.csv es el nombre de archivo en el que se guardarán los recuentos resultantes (si no se proporciona, el nombre del archivo de salida se establece de forma predeterminada en count_results.csv).
NOTA: Las comillas son necesarias en la línea de comandos. Asegúrese de que no haya espacios después de las comillas antes de la ruta del archivo. El script de Python leerá los archivos en la ruta de archivo proporcionada y ejecutará el programa de conteo de núcleos para todas las imágenes en el directorio (consulte la Figura 4C). Los recuentos resultantes se guardarán en el archivo de .csv de resultados en la ubicación especificada. - Cuando aparece la siguiente línea de comandos, el procesamiento se ha completado (consulte la figura 4C). Los contornos y la hoja de cálculo de resultados ahora están disponibles en la ubicación especificada como directorio de salida en el paso 3.4.8 (es decir, ruta/hacia/salida).
- Inspeccione visualmente los contornos para verificar la calidad del recuento. Los resultados ahora se pueden utilizar para normalizar los datos, como se describe en la Sección 4.
NOTA: Si los contornos parecen tener núcleos perdidos (por ejemplo, los núcleos parecen demasiado débiles) o agrupan grupos de núcleos en un solo elemento (por ejemplo, imagen sobreexpuesta, demasiado fondo, artefactos), el usuario debe consultar la Tabla 1 y es posible que deba volver a la Sección 1 para optimizar la adquisición de imágenes antes de pasar a la Sección 4.
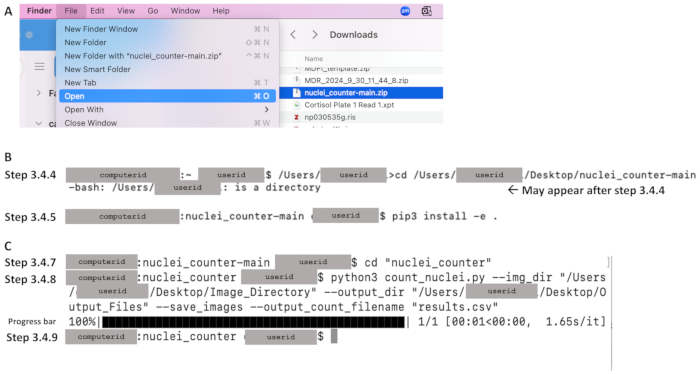
Figura 4: Ejemplos de partes clave del flujo de trabajo para la sección 3 utilizando Mac OS. Después de descargar el código (consulte la Figura 3A), (A) extraiga haciendo clic en el archivo Zip (que se muestra en azul) y establezca el destino deseado. (B) Después de abrir el terminal, ejemplos de comandos para los pasos 3.4.4 (cambiar el directorio al repositorio de código) y 3.4.5 (instalar pip). (C) Ejemplos de comandos para los pasos 3.4.7 y 3.4.8, una barra de progreso después de la ejecución del paso 3.4.8 y la línea de comandos que muestra la finalización en el paso 3.4.9. Haga clic aquí para ver una versión más grande de esta figura.
4. Uso de recuentos para normalizar datos experimentales
- Abra el archivo results.csv que vincula cada imagen con la cuantificación de núcleos determinada mediante el programa automatizado.
- Decida el método de normalización: 1) imagen por imagen (p. ej., si se trata de microscopía de fluorescencia multicolor), 2) normalice los datos como una relación entre el recuento medio de núcleos por muestra frente a una muestra (o condición) de referencia, 3) normalice el recuento de núcleos por muestra (p. ej., extrapole el recuento total de núcleos al área de la muestra). Las recomendaciones para el método de normalización se encuentran en la sección Discusión.
- Si la normalización se va a realizar imagen por imagen, simplemente divida los datos experimentales de cada imagen por el número de núcleos de esa imagen.
- Para normalizar los datos como una proporción, siga los pasos que se describen a continuación.
- Para cualquier muestra en la que se hayan recogido varias imágenes para una sola réplica (por ejemplo, de 10 a 15 imágenes capturadas de un solo pocillo en una placa de 6 pocillos), calcule el número medio de núcleos por imagen. Estos recuentos de núcleos se consideran como el recuento de células, suponiendo células uninucleadas o un número de células fusionadas en células multinucleadas (como con miotubos).
- Seleccione una muestra de referencia o una condición de referencia (por ejemplo, el recuento promedio de todas las muestras de la condición de control) y anote el recuento.
- Divida el recuento de todas las muestras por ese recuento de referencia, lo que da como resultado un valor de normalización. Si se utilizó una muestra de referencia, utilice el valor de normalización de la muestra de referencia como 1.
NOTA: Si se utilizara una condición de referencia, el valor medio de normalización en toda la condición sería ahora 1. Las muestras con menos núcleos que la referencia (es decir, menos densas) tendrán valores de normalización inferiores a 1, y las muestras con más núcleos que la referencia (es decir, más densas) tendrán valores de normalización superiores a 1. - Divida los datos experimentales de cada pozo por el valor de normalización calculado correspondiente.
- Para normalizar los datos en el recuento de núcleos por muestra, siga los pasos que se describen a continuación.
- Para cualquier muestra en la que se hayan recogido varias imágenes para una sola réplica (por ejemplo, de 10 a 15 imágenes capturadas de un solo pocillo en una placa de 6 pocillos), calcule el número medio de núcleos por imagen. Estos recuentos de núcleos se consideran como el recuento de células suponiendo células uninucleadas, o el número de células fusionadas en células multinucleadas (como con miotubos).
- Determine el área de superficie del portaobjetos (cubreobjetos), pozo o plato del que se derivaron los datos. Esta información se puede encontrar en la sección de especificaciones en los sitios web de los productos.
- Determine el área de cada imagen capturada. Esta información suele estar disponible como metadatos en el software de imágenes o análisis.
- Asegúrese de que el área de la superficie de la muestra y el área de la imagen se expresen en las mismas unidades (por ejemplo, mm2). Si no es así, conviértalo a la unidad deseada.
- Calcule el recuento total de cada muestra utilizando la siguiente ecuación:
TC = AC • (SA/IA)
donde TC es el recuento total, AC es el recuento promedio, SA es el área de superficie, IA es el área de la imagen. - Divida los datos experimentales por el TC correspondiente a la muestra.
- Normalizar los datos experimentales y utilizarlos para análisis estadísticos, según corresponda.
Resultados
Cada ejecución de imagen por lotes produce: 1) un conjunto de archivos de imagen con contornos aplicados que muestran los contornos de los núcleos identificados (Figura 5) y 2) un archivo de .csv (hoja de cálculo) que vincula los nombres de los archivos de imagen y los recuentos asociados. La visualización de los contornos permitirá al usuario evaluar visualmente la calidad del recuento. Específicamente, las imágenes obtenidas de acuerdo con la sección 1 deben tener todos (o casi todos) los núcleos rodeados por una línea verde continua que indique que el núcleo fue contado por el programa. Estos contornos se utilizaron para afinar el programa aquí.
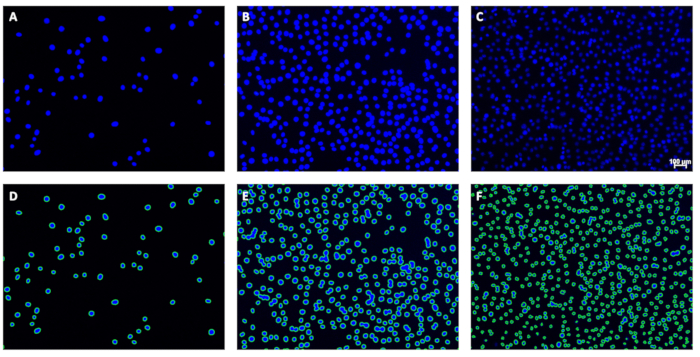
Figura 5: Ejemplos de imágenes originales y contornos correspondientes. (A-C) Ejemplos de imágenes originales antes del análisis y (D-F) contornos correspondientes después del análisis directamente debajo. La barra de escala (C) indica 100 μm; Las barras de escala deben omitirse de las imágenes utilizadas para la cuantificación de núcleos. Haga clic aquí para ver una versión más grande de esta figura.
Validación mediante resultados iniciales
Los mioblastos C2C12 a diferentes densidades celulares se fijaron con metanol, se contratiñeron con DAPI en medios de montaje y se obtuvieron 120 imágenes con un aumento de 10x en formato de archivo .jpg. El programa automatizado se ejecutó con el directorio de imágenes establecido en la carpeta que contiene estas imágenes y se guardó el archivo .csv resultante. Por separado, cada imagen fue cuantificada manualmente por dos investigadores capacitados que estaban ciegos a los recuentos automatizados y a los recuentos de los demás. Las imágenes sin procesar, los contornos generados por el programa automatizado y una hoja de cálculo de resultados que incluye recuentos manuales y automatizados están disponibles en el repositorio de Open Science Framework (OSF) (ver enlace en la sección 1). Los coeficientes de correlación intraclase (CCI; medidas promedio) se calcularon utilizando SPSS Statistics para determinar la confiabilidad entre evaluadores entre 1) los dos recuentos manuales y 2) el promedio de los dos recuentos manuales y los recuentos obtenidos utilizando el programa automatizado. Enfoques similares se han utilizado para validar otros programas diseñados para cuantificar objetos en imágenes obtenidas mediante microscopía de fluorescencia 16,17,18. También se calcularon los ICC entre los recuentos manuales promedio y los recuentos automatizados para cada cuartil para garantizar que la evaluación de todo el conjunto de datos no enmascarara ningún cambio potencial en la confiabilidad en función de la densidad de celdas. Los cuartiles se definieron utilizando los puntos de corte delos percentiles 25, 50 y 75 según el recuento manual promedio, y oscilaron entre el cuartil 1 (Q1; densidad de celdas más baja) y Q4 (densidad de celdas más alta). Los CCI se reportan con base en medidas promedio y se interpretan con base en las siguientes definiciones: deficiente (CCI <0,50), moderado (0,50 ≤CCI <0,75), bueno (0,75 ≤CCI <0,90) o excelente (CCI ≥0,90)19.
Los núcleos por imagen oscilaron entre 44,5 y 1160 según los promedios de recuento manual. La confiabilidad entre evaluadores fue excelente entre los dos conteos manuales (ICC > 0,999, p < 0,0001; Figura 6A) y entre los recuentos manuales y automatizados promedio (ICC = 0,993, p < 0,0001; Figura 6B). La fiabilidad se mantuvo excelente en todos los cuartiles (Q1: ICC = 0,996, p < 0,0001; Q2: CCI= 0,997, p < 0,0001; Q3: CCI = 0,998, p < 0,0001; Q4: ICC = 0,986, p < 0,0001; Figura 6C-F). El único punto de datos lejos de la línea de mejor ajuste en la Figura 6B representa la imagen que se muestra en la Figura 7. La imagen tiene varias áreas de múltiples núcleos agrupados, lo que impide que el programa de visión artificial reconozca cada núcleo. Este problema se puede solucionar utilizando un tiempo de exposición más corto. En la tabla 1 se describe cómo manejar una imagen de este tipo.
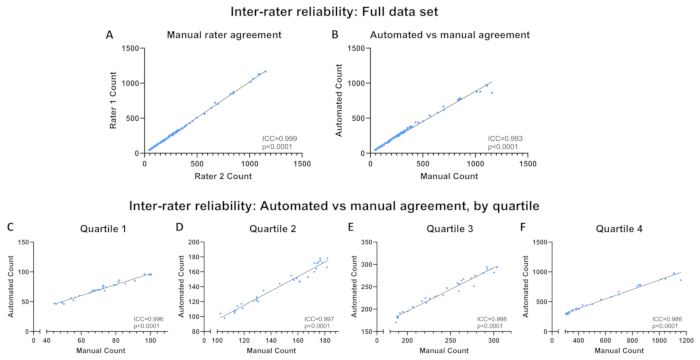
Figura 6: Fiabilidad entre evaluadores. Fiabilidad entre evaluadores en todo el conjunto de datos (A) entre los recuentos manuales de evaluadores y (B) entre los recuentos automatizados y los recuentos manuales promedio. (C-F) Fiabilidad entre evaluadores entre los recuentos automatizados y los recuentos manuales promedio por cuartil. Fiabilidad expresada como coeficiente de correlación intraclase (CCI). Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: Ejemplo de una imagen con dos barreras para la cuantificación precisa de los núcleos. (A) Imagen original y (B) contornos asociados. La flecha 1 indica un área con múltiples núcleos agrupados, distorsionando la forma nuclear redonda lo suficiente como para impedir la detección de software de múltiples núcleos. Esto ocurre en varios lugares a lo largo de esta imagen. La flecha 2 indica un halo desvaído que aparece detrás de múltiples núcleos; Este halo apareció lo suficientemente brillante como para que el software lo incluyera como núcleo, perdiendo así los recuentos de los múltiples núcleos que rodea. Esta imagen representa el único punto de datos lejos de la línea de mejor ajuste en la Figura 6B, lo que enfatiza los problemas potenciales con estas barreras. La barra de escala (A) indica 100 μm; Las barras de escala deben omitirse de las imágenes utilizadas para la cuantificación de núcleos. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Nuestro programa de cuantificación de núcleos tiene varias ventajas sobre las opciones existentes: requiere solo habilidades tecnológicas mínimas, está validado para la tarea específica de cuantificación de núcleos y es de código abierto; Este último supera las barreras relacionadas con los costos. En última instancia, este programa proporciona a los biólogos celulares y moleculares una opción adicional para cuantificar núcleos de forma rápida y precisa en imágenes capturadas con microscopía de fluorescencia. Los programas de recuento automatizado de núcleos o células disponibles actualmente no son los preferidos por todos los usuarios, algunos tienen un costo prohibitivo y la validación puede ser mínima o inexistente para aplicaciones específicas. Los equipos especializados, como la línea BioTek Cytation, van acompañados de boletines técnicos que validan el recuento de células in situ mediante tinción nuclear20, además de una amplia variedad de aplicaciones adicionales. Si bien existen datos de validación y la línea de productos Cytation se usa ampliamente, especialmente cuando se interconecta con los analizadores de flujo extracelular Seahorse para normalizar datos de células vivas de manera fácil y eficiente, este producto puede tener un costo prohibitivo para los laboratorios con fondos limitados para equipos disponibles. Por ejemplo, el precio de lista del modelo base equipado con cuatro objetivos y cuatro cubos de filtro para microscopía de fluorescencia superó los 100.000 dólares estadounidenses en el año natural 2024. Para aquellos que no tienen los medios para comprar equipos especializados, el software de código abierto ofrece alternativas gratuitas para el análisis de imágenes. El software de código abierto útil para la cuantificación de núcleos y células incluye ImageJ (National Institutes of Health, Bethesda, MD, EE. UU.; disponible en 21. Los complementos ahora están disponibles para aquellos que desean escribir scripts de macros para la automatización del procesamiento de imágenes utilizando la plataforma ImageJ. La automatización del recuento de células basada en ImageJ mediante contraste de fase o microscopía de campo claro con imágenes de células completas ha sido descrita previamente22,23. CellProfiler es una aplicación ampliamente publicada para el análisis de imágenes celulares teñidas con colorantes fluorescentes o tinciones histológicas estándar, y un artículo temprano que describe su utilidad en una variedad de aplicaciones biológicas a menudo se cita como validación24. Sin embargo, este manuscrito no valida ningún hallazgo biológico en particular, lo que requiere una validación y transparencia adicionales para varias tuberías de automatización utilizadas, como para el recuento de núcleos25. Además, aunque CellProfiler es un paquete de software eficiente para el análisis de células, no todos los científicos lo encuentran fácil de usar. Por lo tanto, nos propusimos desarrollar y validar un programa automatizado alternativo para cuantificar núcleos utilizando software de código abierto. Específicamente, empleamos bibliotecas de visión artificial existentes en Python para desarrollar este flujo de trabajo.
Este método comienza con células o tejidos que el usuario final ha teñido utilizando tinciones de ADN como DAPI26. Se proporcionan pautas generales para capturar y guardar imágenes de una manera compatible con el software de automatización. Aunque los detalles pueden diferir con los diferentes microscopios y software de imágenes, el flujo de trabajo general para la sección 1 es el mismo para obtener imágenes cuantificables. Si se utilizan muestras vivas, se debe tener cuidado para asegurarse de que se use el equipo de protección personal adecuado y que el entorno de imágenes sea apropiado para esas muestras (p. ej., temperatura, porcentaje de CO2, adición de tampón, etc.)27. Si bien este protocolo describe la obtención de imágenes de los tintes de ADN, los usuarios pueden capturar otros colores de la misma área para fines de fusión15, y simplemente copiar las imágenes nucleares en una carpeta separada para su uso con el programa de cuantificación. Las imágenes deben realizarse en una habitación oscura con el EPP adecuado para muestras vivas y se debe usar una desinfección adecuada según sea necesario (p. ej., con etanol al 70%). Las muestras teñidas deben protegerse de la luz antes de encender las luces. La captura de imágenes de calidad es fundamental para una cuantificación precisa de los núcleos. Incluso en imágenes con un tiempo de exposición adecuado y un fuerte contraste entre núcleos brillantes y un fondo negro, la presencia de células excesivamente agrupadas o halos detrás de grupos de células (véase la Figura 7) puede disminuir la precisión de la cuantificación. En estos casos, puede ser necesario alterar los tiempos de exposición para disminuir la intensidad de las señales, asegurando una fusión mínima de las señales de los núcleos y la minimización de los halos, al tiempo que se mantiene una señal lo suficientemente fuerte como para detectar las formas redondas de los núcleos. Si esos pasos son insuficientes, puede ser necesario tomar imágenes de una sección adyacente de la muestra. Todas las imágenes de un solo experimento deben capturarse con el mismo aumento, y el aumento correspondiente al objetivo utilizado debe configurarse correctamente en el software de imágenes. Es especialmente crítico que el objetivo utilizado para la obtención de imágenes coincida con la configuración del software de imágenes cuando el recuento de núcleos se extrapolará del área de la imagen a toda el área del pocillo (o portaobjetos). Este paso debe tener lugar cada vez que se cambia el objetivo, pero se pasa por alto fácilmente al configurar el microscopio y el software. El tiempo requerido para la sección 1 varía en función de una serie de factores (por ejemplo, la experiencia del usuario con la microscopía de fluorescencia, el número de imágenes y muestras, el microscopio, etc.). Para capturar de 10 a 15 imágenes de núcleos teñidos por pocillo en dos placas de 6 pocillos, el usuario debe reservar aproximadamente 1-2 h de tiempo.
Una vez que las imágenes se han capturado y guardado en una carpeta dedicada, están listas para ser cuantificadas. El método más simple para la cuantificación es ejecutar este programa automatizado preempaquetado sin necesidad de instalar Python ni ninguna biblioteca (sección 2). Cabe señalar que el programa está preparado como un ejecutable y se empaquetó usando Pyinstaller. El software creado con Pyinstaller suele ser marcado por el software antivirus. Si el software antivirus bloquea la apertura del archivo, el usuario debe marcar el archivo como seguro para continuar. Esto es fundamental para realizar con éxito la sección 2. Alternativamente, cualquier usuario puede optar por ejecutar el programa de cuantificación automatizada como un script de Python (sección 3) en lugar de un ejecutable, si lo desea. Si el usuario desea ejecutar el programa utilizando un sistema operativo Mac o Linux, el programa debe ejecutarse a través de Python. Este método no requiere Pyinstaller, pero puede ser más desafiante técnicamente. El protocolo paso a paso, junto con los ejemplos, detallados en la sección 3, tiene como objetivo eliminar las barreras técnicas para ejecutar este programa como un script. Ya sea que se use la sección 2 o la sección 3 para ejecutar el programa, es fundamental asegurarse de que el código se ingrese correctamente en los símbolos del sistema. Las líneas de código de ejemplo se incluyen en todo el protocolo, y el usuario debe prestar mucha atención al espaciado, las comillas y otros signos de puntuación. El usuario también debe asegurarse de que las rutas de los archivos sean precisas. Es útil tener las rutas de archivo para las imágenes y la salida disponibles en un documento de Word. Los comandos también se pueden escribir en este documento de Word y copiar/pegar según sea necesario.
Como se describe en la sección 4, existen múltiples enfoques para normalizar los datos experimentales utilizando recuentos obtenidos de este programa automatizado de cuantificación de núcleos. En el caso de los experimentos basados en imágenes en los que los datos experimentales incluyen un número de eventos en cada imagen y se desea una proporción (por ejemplo, la proporción de células positivas para la anexina V28, la proporción de células que expresan receptores ACE229, el número de células que absorben partículas teñidas30, etc.), los datos experimentales de cada imagen pueden dividirse simplemente por el recuento resultante (lo que da como resultado una proporción de 0 a 1). A continuación, se puede calcular la proporción media entre las imágenes replicadas de la misma diapositiva, plato o pozo. Si se desea un porcentaje, simplemente multiplique las proporciones finales por 100. Por último, estas proporciones o porcentajes medios pueden utilizarse en los análisis estadísticos posteriores. Cuando los datos experimentales que se van a normalizar proceden de experimentos funcionales en células vivas (por ejemplo, tasas de consumo de oxígeno 6,11,12) o de sobrenadante de cultivo celular (por ejemplo, actividad de lactato deshidrogenasa en el sobrenadante31 o concentración de una citocina32 específica secretada, etc.), los recuentos de núcleos deben promediarse primero en todas las imágenes capturadas desde el mismo pocillo. Luego, el recuento de núcleos se puede extrapolar al área de la superficie del portaobjetos (cubreobjetos), pocillo o plato, o el recuento se puede expresar como una relación frente a una muestra o condición de referencia. La extrapolación a la superficie del portaobjetos, pocillo o plato es útil para expresar datos experimentales en comparación con el número de células (si se utilizan células uninucleadas) de las que se derivaron los datos (por ejemplo, oxígeno consumido por minuto por 103 células). Para utilizar este método, se debe conocer el área de la superficie del portaobjetos (cubreobjetos), pocillo o plato y el área capturada en cada imagen. Si la muestra no cubre toda el área de superficie disponible, los datos experimentales provienen de una pequeña cantidad de sobrenadante (es decir, expresados como actividad o concentración por unidad de volumen), o no se conoce la imagen y/o el área de superficie, entonces la mejor opción es promediar los recuentos de núcleos en todas las imágenes para una sola muestra y expresar el recuento de núcleos en relación con una muestra o condición de referencia. El valor resultante se utiliza para corregir los datos experimentales de la densidad de núcleos o la densidad celular (si se utilizan células uninucleadas).
Si bien este método es fácil de ejecutar, existen varios problemas potenciales que los usuarios deben conocer. Estos posibles problemas, junto con las posibles causas y los pasos de solución de problemas para resolverlos, se enumeran en la Tabla 1. El programa descrito aquí tiene ventajas clave en comparación con el software existente; Sin embargo, hay que reconocer varias limitaciones. La principal desventaja es que el programa de cuantificación solo se puede utilizar con imágenes capturadas con microscopía de fluorescencia, no con imágenes de campo claro. Si bien se utilizaron imágenes teñidas con DAPI capturadas con un aumento de 10x para construir el programa, el programa se puede usar con otros tintes y aumentos de ADN, ya que el programa está diseñado para reconocer la morfología de los núcleos en lugar del color o el tamaño. Si bien validamos la precisión del programa en imágenes que contienen una variedad de densidades de celdas, el usuario final puede tener densidades de celdas que caen fuera del rango. En este caso, seguimos anticipando recuentos precisos siempre que se obtengan imágenes de calidad; Sin embargo, el usuario debe verificar cuidadosamente los contornos y la salida para garantizar la precisión. Para aplicaciones de cultivo celular, la cuantificación de núcleos en imágenes es más apropiada para células en una monocapa. Si las células cultivadas se superponen, los núcleos pueden enmascararse, lo que resulta en una subestimación de la cantidad de núcleos. La naturaleza bidimensional de la cuantificación de núcleos en imágenes impide el uso de este programa para determinar el número total de núcleos de células en suspensión, bloques de tejido grueso u otras aplicaciones tridimensionales. Sin embargo, este programa aún puede cuantificar núcleos marcados con tintes de ADN fluorescentes en imágenes bidimensionales capturadas a partir de modelos tridimensionales, como bloques de tejido gruesos. La utilidad de cuantificar núcleos en imágenes para la aplicación deseada debe ser determinada por el usuario final.
| Problema | Posibles causas | Posibles soluciones |
| Fondo excesivo en las imágenes | Tiempo de exposición demasiado alto | Reducir el tiempo de exposición |
| Los núcleos parecen fusionarse entre sí en imágenes | Concentración de tinte de ADN fluorescente demasiado alta o demasiado medio de montaje de tinte de ADN | Reducir la concentración de colorante durante la optimización; Utilice la menor cantidad posible de medios de montaje |
| Los núcleos parecen demasiado ligeros o algunos núcleos no son visibles | Tiempo de exposición demasiado bajo | Aumentar el tiempo de exposición |
| Fotoblanqueo | Evite el fotoblanqueo asegurándose de que las muestras manchadas permanezcan almacenadas en la oscuridad y minimizando la exposición de las muestras a la iluminación (por ejemplo, utilizando el tiempo de exposición mínimo necesario y apagando la iluminación cuando no sea necesaria, como después de capturar una imagen o mientras se toman notas) | |
| Efectos de halo o formas no específicas del núcleo que aparecen | Tiempo de exposición demasiado alto | Reducir el tiempo de exposición |
| Pelusas de limpiaparabrisas delicados o algunos protectores de sobremesa en celdas vivas o adheridas a celdas fijas | Garantice un entorno libre de pelusas: minimice el uso de toallitas delicadas cuando las muestras estén expuestas (por ejemplo, en un gabinete de bioseguridad para células vivas, durante la fijación y el secado de células fijas o durante el proceso de tinción); Asegúrese de que los protectores de sobremesa no dejen pelusas en los portaobjetos o placas durante la tinción | |
| Otras anomalías en la superficie de las células/tejidos fijados | Manipule las muestras con extremo cuidado y siga las precauciones adecuadas y las mejores prácticas durante la fijación y la tinción. | |
| Perturbaciones en la trayectoria de la luz | Asegúrese de que todas las partes del microscopio estén limpias y libres de pelusas y polvo; Limpie el exterior de los portaobjetos/placas/platos con etanol al 70% para eliminar el polvo ambiental y las pelusas antes de la toma de imágenes | |
| El archivo ejecutable no se abre | El software antivirus bloqueó el archivo | Marcar el archivo como seguro |
| Errores en los símbolos del sistema | Problemas con el espaciado en la línea de comandos | Compruebe que el comienzo del comando se escriba inmediatamente después del símbolo ">" en la línea de comandos sin un espacio, que no haya espacios después de las comillas abiertas para las rutas de archivo y que el espaciado y coincida con el código proporcionado y los ejemplos. |
| Casos incorrectos utilizados en la línea de comandos | El código distingue entre mayúsculas y minúsculas; Asegúrese de usar letras mayúsculas y minúsculas según corresponda | |
| Problemas con la ruta del archivo | Asegúrese de que la ruta del archivo indique el interior de la carpeta de archivos asociada (es decir, la carpeta de archivos está abierta cuando se copia la ruta del archivo). | |
| Problemas con el programa que no se resuelven siguiendo los pasos de solución de problemas anteriores | Problemas de software o de línea de comandos mencionados anteriormente. | Envíe una propuesta a través de GitHub. Vaya a https://github.com/rbudnar/nuclei_counter. En la barra de menú de la parte superior, seleccione "Problemas" (segunda opción, a la derecha de "Código"). Haga clic en el botón verde en el lado derecho que dice "Nuevo número" para enviar. Aparecerá un mensaje para iniciar sesión en GitHub antes de enviar el problema. También habrá una opción para crear una cuenta gratuita si es necesario. |
Tabla 1: Solución de problemas para capturar imágenes de calidad y ejecutar el flujo de trabajo automatizado de cuantificación de núcleos.
En general, los métodos descritos en este documento proporcionan detalles paso a paso para utilizar el programa de cuantificación de núcleos automatizado que desarrollamos utilizando bibliotecas de visión artificial y validamos con recuentos manuales de múltiples evaluadores. Este programa fue desarrollado para garantizar la usabilidad por parte de científicos con varios niveles de habilidades tecnológicas relacionadas con la codificación, está disponible de forma gratuita y fue validado específicamente para la tarea de cuantificación de núcleos. En comparación con el conteo manual, este programa ahorrará al usuario final un tiempo valioso y proporcionará recuentos de núcleos precisos y confiables para la normalización de datos.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Los fondos para este trabajo fueron proporcionados por los Institutos Nacionales de Salud/Instituto Nacional sobre el Envejecimiento (R01AG084597; DEL y HYL) y por fondos de puesta en marcha de la Universidad Tecnológica de Texas (DEL). Los autores desean agradecer a los programas de Becarios de Investigación de Pregrado y Becarios TrUE de la Universidad Tecnológica de Texas por brindar apoyo financiero a los investigadores de pregrado que contribuyeron a este trabajo (REH, MRD, CJM, AKW). También agradecemos a los Dres. Lauren S. Gollahon y Michael P. Massett por compartir gentilmente su espacio y equipo de laboratorio.
Materiales
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
Referencias
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados