Method Article
Computer Vision 라이브러리를 사용하여 핵 정량 분석 간소화
요약
이 문서에서는 다양한 세포 밀도에서 검증된 오픈 소스 실행 프로그램을 사용하여 이미지 기반 핵 정량화를 자동화하는 단계별 방법을 설명합니다. 이 프로그램은 비용, 제한된 기술 역량을 가진 사용자의 접근성 및 기존 기술의 유용성을 제한할 수 있는 애플리케이션별 검증과 관련된 장벽을 해결하는 대안을 제공합니다.
초록
라이브 셀 분석 및 이미지 기반 세포 분석에는 정확한 해석을 위해 데이터 정규화가 필요합니다. 일반적으로 사용되는 방법은 핵을 염색하고 정량화한 다음 핵 수에 대한 데이터 정규화입니다. 이 핵 수는 종종 단핵 세포의 세포 수로 표현됩니다. 수동 정량화는 힘들고 시간이 많이 소요될 수 있지만, 사용 가능한 자동화 방법은 모든 사용자가 선호하지 않을 수 있으며, 이 특정 응용 분야에 대한 검증이 부족하거나 비용이 많이 들 수 있습니다. 여기에서는 형광 DNA 염색으로 염색된 핵의 정량화 가능한 이미지를 캡처한 후 Python 컴퓨터 비전 라이브러리를 사용하여 개발된 자동 객체 계수 소프트웨어 프로그램을 사용하여 핵을 정량화하기 위한 단계별 지침을 제공합니다. 또한 다양한 세포 밀도에서 이 프로그램을 검증합니다. 프로그램 실행의 정확한 시간은 이미지 수와 컴퓨터 하드웨어에 따라 다르지만 이 프로그램은 프로그램 실행을 위해 핵을 초 단위로 계산하는 작업 시간을 통합합니다. 이 프로토콜은 고정된 염색된 세포의 이미지를 사용하여 개발되었지만, 살아있는 세포 및 면역 형광 응용 분야에서 염색된 핵의 이미지도 이 프로그램을 사용하여 정량화할 수 있습니다. 궁극적으로 이 프로그램은 높은 수준의 기술력을 필요로 하지 않는 옵션을 제공하며, 세포 및 분자 생물학자가 워크플로우를 간소화하고 지루하고 시간이 많이 소요되는 핵 정량 작업을 자동화하는 데 도움이 되는 검증된 오픈 소스 대안입니다.
서문
기능 및 이미지 기반 실험은 실험적 치료가 전세포 생화학 및 생리학에 미치는 영향을 이해하는 데 매우 중요합니다. 세포 생물학 실험 데이터의 유효한 해석은 데이터 정규화를 포함한 실험 프로토콜의 정확성과 재현성에 달려 있습니다. 예를 들어, 기준선에서 그리고 특정 약물 처리 후 살아있는 세포의 산소 소비량 및 세포 외 산성화 속도를 분석하면 에너지 대사의 다양한 측면을 평가할 수 있습니다 1,2. 세포 배양의 상층액에서 젖산 탈수소효소와 같은 효소의 활성을 측정하면 세포막 무결성을 정량화하는 데 도움이 될 수 있습니다3. 고정 전에 배양된 세포를 Annexin V 및 propidium iodide로 염색하면 세포사멸 및 괴사 세포를 평가할 수 있습니다4. 그러나 웰 간의 세포 밀도 차이는 이러한 각 분석의 결과에 영향을 미칩니다. 시딩 밀도에만 전적으로 의존하면 파종을 위한 세포 계수 오류, 플레이팅 중 배지의 세포 밀도 변화 또는 실험 전반에 걸쳐 샘플 또는 처리 간의 세포 성장 속도 차이로 인해 잘못된 결과를 제공할 수 있습니다. 따라서 실험 결과의 정규화가 필요합니다.
기능 및 이미지 기반 세포 데이터에 대한 현재 정규화 방법에는 단백질 농도5 또는 핵 또는 세포 수6가 포함됩니다. 데이터를 단백질 농도로 정규화하려면 분석을 실행한 후 프로테아제 억제제와 동일한 부피의 용해 완충액에서 세포를 추출해야 하며, 단백질 정량화를 위해 추가 분석(예: bicinchoninic acid assay, Bradford assay 등)을 수행해야 합니다. 이 방법의 정확도는 완전한 세포 추출에 달려 있습니다. 단백질을 남기면 정량 분석의 오류가 발생합니다. 핵 또는 세포를 계수하는 것은 세포가 자라는 표면에서 세포를 제거할 필요가 없는 대안을 제공합니다. 대신, 살아 있거나 고정된 세포는 비색 또는 형광 염색을 사용하여 염색 또는 역염색되며, 전체 세포 또는 핵의 이미지를 기반으로 계수를 수행할 수 있습니다. 핵 정량화는 형광 현미경 검사 응용 분야를 위해 전체 세포보다 선호되며 융합 세포(예: 근관) 또는 조직의 데이터를 정규화합니다. 형광 접근법을 사용한 핵 정량화를 위해 형광 DNA 염료(예: 4',6-diamidino-2-phenylindole dichloride(DAPI), Hoechst 33342, Hoechst 33358, DRAQ5 등)를 살아 있거나 고정된 세포에 적용하고 형광 현미경을 사용하여 이미지화합니다. DAPI, Hoechst 및 DRAQ5는 DNA 7,8,9의 아데닌-티민이 풍부한 영역에 우선적으로 결합하여 핵을 검출하는 수단을 제공합니다. 염료 농도의 최적화는 off-target 결합(즉, 미토콘드리아 DNA에 대한 반점 세포질 형광 유발)을 방지합니다10. 최적화 프로세스는 고정 세포에 대한 DAPI(예: DAPI를 사용한 VECTASHIELD Antifade Mounting Medium)를 포함하는 상업적으로 이용 가능한 마운팅 미디어와 라이브 셀에 대한 공개된 염색 농도(예: Hoechst)를 사용하여 간소화할 수 있습니다6,11,12. 염색이 완료되면 적절한 여기 필터(예: 자외선[DAPI 및 Hoechst] 또는 원적외[DRAQ5])를 사용하여 살아 있거나 고정된 세포의 핵을 시각화합니다. DAPI, Hoechst 및 DRAQ5의 여기 최대값은 형광 현미경 검사의 경우 스펙트럼의 양쪽 끝에 있습니다. 이러한 특성과 함께 이러한 염료의 DNA 특이성 및 최적 농도에서의 사용은 off-target 형광을 최소화합니다. 결과 이미지는 검은색 배경에 밝은 핵을 보여주며, 이러한 핵은 단핵 세포의 세포 수를 측정하기 위해 정량화됩니다. 조직학적 염색 및 명시야 현미경13을 사용하여 핵을 정량화하는 다른 정확한 염색 기반 방법이 존재하지만, 이러한 접근 방식을 사용하는 자동화는 가능하더라도, 더 어렵습니다.
여전히 황금 표준으로 남아 있지만, 수동 핵 정량화는 힘들고 시간이 많이 걸리며 장시간 계수로 인해 잠재적인 인적 오류에 취약합니다. 자동화된 세포 계수 프로그램이 존재하지만 모든 사용자가 선호하지 않고 비용이 많이 들 수 있으며 특정 응용 프로그램에 대한 검증이 최소화될 수 있습니다. Python은 최근 몇 년 동안 생물학자들에게 유용하고 접근하기 쉬운 리소스가 되었습니다. Python 컴퓨터 비전 라이브러리는 이미지 분석에 특히 유용합니다14. 여기에서는 Python 코드를 사용하여 개발된 실행 프로그램을 사용하여 형광 염료로 염색되고 형광 현미경을 사용하여 이미지화된 핵을 정량화하는 데 대한 단계별 지침을 제공합니다. 여기에 설명된 프로그램을 사용하는 데 코딩 기술이 필요하지 않습니다. 이 워크플로우는 형광 DNA 염료가 적용된 세포 또는 조직에 특이적입니다. 명시야 이미지에는 사용할 수 없습니다. 섹션 1에서는 이 워크플로우와 호환되는 방식으로 형광 현미경 검사를 사용하여 수집된 이미지를 캡처하고 저장하는 방법을 설명합니다. 섹션 2에서는 핵 정량화 프로그램을 실행 파일로 실행하고 출력을 가져오는 방법에 대한 지침을 제공합니다. 실행 프로그램은 제공된 .exe 파일에서 직접 실행할 수 있으며 고품질 이미지에서 핵을 정량화하기 위해 수정할 필요가 없습니다. 이 실행 파일을 사용하려면 Windows 기반 PC가 필요합니다. 섹션 3에서는 프로그램을 실행 파일이 아닌 Python 스크립트로 실행하는 방법에 대한 지침을 제공하며(Mac 또는 Linux 기반 시스템의 경우 필수, Windows 기반 시스템의 경우 선택 사항) 원하는 경우 코드를 조정할 수 있습니다. 섹션 4에서는 정규화에 결과를 사용하는 방법을 설명합니다. 이러한 프로토콜 다음에는 당사 실험실의 검증 데이터를 포함한 대표적인 결과가 나옵니다.
프로토콜
참고: 추가 파일은 다음 링크에서 찾을 수 있습니다 https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. 형광 현미경을 사용한 이미지 캡처 및 저장
- 원하는 DNA 염료로 염색하는 것을 포함하여 이미징할 세포 또는 조직 샘플을 준비합니다. 여기에 사용된 이미지를 얻기 위해, C2C12 근아세포(CRL-1772, American Type Culture Collection)를 50mMEtOH6의 유무에 관계없이 표준 배양 조건(5%CO2, 37°C, 가습)하에서 48-72시간 동안 6-웰 플레이트에서 성장시키고, 앞서 설명한 바와 같이 얼음-차가운 메탄올에 고정시켰다13. 고정 세포는 DAPI-함유 장착 매체(15)를 사용하여 장착하고 이미징 전에 암흑 속에서 건조시켰습니다.
- 확립된 표준 작동 절차에 따라 형광 현미경과 컴퓨터의 모든 구성 요소를 켭니다. 현미경과 연결된 소프트웨어를 엽니다.
- 현미경을 설정하여 s를 시각화합니다.amp접안렌즈를 통해 les. 목표를 확인합니다. 필요한 경우 노즈피스를 원하는 목표로 돌립니다. 이미지 영역을 정확하게 표시하기 위해 소프트웨어 프로그램이 원하는 대물렌즈를 선택했는지 확인하십시오. 여기에서 이미지는 10x 대물렌즈를 사용하여 캡처되었습니다.
- 불을 끈 상태에서 샘플(슬라이드, 접시 또는 접시)을 무대에 놓습니다. 적절한 필터로 라이브 이미징을 켜고 노출 시간을 미리 결정된 지속 시간 또는 자동 노출로 설정합니다. 여기에서는 61-62ms의 DAPI 필터와 노출 시간을 사용했습니다. 특히 세포 밀도가 높은 이미지의 경우 노출 시간 설정에 대한 지침은 문제 해결 표를 참조하십시오.
- 접안렌즈를 사용하여 얼룩진 핵이 보일 때까지 총 조정 손잡이를 사용하여 초점을 천천히 조정한 다음 미세 조정 손잡이를 사용하여 핵이 깨끗하고 선명하게 나타날 때까지 초점을 천천히 조정합니다.
참고: 적절한 이미지의 예가 그림 1에 나와 있습니다.- 과도한 배경 염색이 나타나거나 핵이 매우 어둡게 나타나면 노출 시간을 수동으로 조정하십시오. 단일 이미지에서 일부 핵에 대한 신호 강도에 눈에 띄는 차이가 있을 수 있습니다. 그러나 둥근 모양은 신호 간섭을 최소화하면서 모든 핵에서 볼 수 있어야 합니다.
참고: 다운스트림 응용 프로그램에 사용되는 프로그램은 다양한 강도에 강합니다. 핵 정량화 프로그램의 개발 및 검증에 사용되는 원시 이미지에는 다양한 신호 강도를 가진 이미지가 포함되며 Open Science Framework 리포지토리를 통해 사용할 수 있습니다(위에서 언급한 링크 참조).
- 과도한 배경 염색이 나타나거나 핵이 매우 어둡게 나타나면 노출 시간을 수동으로 조정하십시오. 단일 이미지에서 일부 핵에 대한 신호 강도에 눈에 띄는 차이가 있을 수 있습니다. 그러나 둥근 모양은 신호 간섭을 최소화하면서 모든 핵에서 볼 수 있어야 합니다.
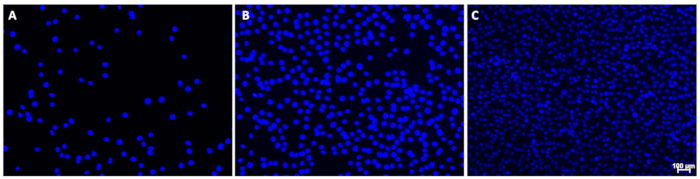
그림 1: DAPI 염색 핵의 예시 이미지. 이 이미지는 최소한의 배경으로 (A) 낮음, (B) 중간 또는 (C) 높은 세포 밀도의 핵을 묘사합니다. 이미지 C에서 핵의 신호 강도가 다양하다는 점에 주목하십시오. 이것은 정량화를 방해하지 않았습니다. 눈금 막대 (C)는 100 μm를 나타냅니다. 핵 정량화에 사용되는 이미지에서 스케일 바는 생략해야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 접안렌즈를 통해 핵에 초점이 맞춰지면 감지 모드를 카메라로 전환합니다. 미세 조정 손잡이를 사용하여 초점을 조정합니다.
- 필요한 경우 노출 시간을 조정합니다. 후속 샘플 및 이미지에 사용할 최종 노출 시간을 기록해 둡니다.
참고: 모든 이미지에서 동일한 노출 시간을 사용하는 것이 이상적이지만, 형광 강도가 샘플 간에 정량화되거나 비교되지 않기 때문에 이 응용 분야에서는 노출 시간의 변동이 허용됩니다. - 소프트웨어의 적절한 이미지 캡처 버튼을 사용하여 이미지를 캡처합니다. 샘플에 여러 염색이 적용된 경우 필터를 전환하고(예: FITC, TRITC 또는 Cy5로) 병합을 위해 동일한 영역을 이미지화합니다.
- 빛에 대한 샘플 노출을 최소화하기 위해 셔터를 적용합니다. 축척 막대에서 굽지 않고 전용 폴더에 범용 그림 파일 형식(.tif, .png 또는 .jpg)으로 이미지를 저장합니다. 각 이미지 파일 이름에 적절한 설명이 포함되어 있는지 또는 파일 이름에 사용되는 모든 코딩에 적절한 링크 설명서가 있는지 확인합니다.
참고: 이것은 수량화 프로그램의 이미지 디렉토리가 됩니다. 멀티컬러 이미징을 사용한 경우 핵 이미지만 별도의 전용 폴더에 복사합니다. 이 작업은 이미징 세션이 끝날 때도 수행할 수 있습니다. 대부분의 이미징 소프트웨어에서 기본 내보내기 설정을 사용하여 내보낸 이미지는 고품질 이미지가 캡처된 경우 일반적으로 이 프로그램에서 사용하기에 충분한 해상도입니다. 그러나 핵의 형태가 명확한지 확인하기 위해 내보내기 후 이미지를 육안으로 확인해야 합니다. 이미지를 .jpg 형식으로 저장하는 경우 이미지가 과도하게 압축되지 않고 품질이 유지되도록 주의해야 합니다. - 셔터를 열고 라이브 이미징으로 돌아간 다음 이미징을 위해 샘플을 다른 위치로 스캔합니다. 필요에 따라 1.8-1.9단계를 반복합니다. 10배 배율로 캡처한 6-well plate에 seeded 세포 이미지의 경우 well당 10-15개의 이미지를 캡처합니다(샘플 영역 전체의 세포 밀도 일관성에 따라 다름).
- 단일 샘플에 대한 모든 이미지를 획득한 후 다음 옵션 중 하나를 수행합니다.
- 다음 샘플로 이동하여 1.4-1.10단계를 반복합니다.
- 프로토콜을 일시 중지하고 다른 시간에 다시 반환합니다. 샘플은 빛으로부터 보호되어야 합니다. 이 옵션은 라이브 셀로 작업하는 경우에는 권장되지 않습니다.
- 이미징이 완료되면 실내 조명을 켜기 전에 모든 샘플이 빛으로부터 보호되어 있는지 확인하십시오. 이미지가 포함된 폴더가 로컬에 저장된 경우 썸 드라이브, 클라우드 스토리지 위치 또는 정량화 프로그램이 실행될 컴퓨터에서 액세스할 수 있는 기타 네트워크 드라이브로 파일을 전송합니다. 전용 폴더에 저장합니다.
- 확립된 표준 작동 절차에 따라 현미경과 컴퓨터의 모든 부품을 차단하십시오.
- 이제 이미징이 완료되었습니다. 섹션 2에 설명된 대로 자동화된 수량화 프로그램을 사용하거나 섹션 3에 설명된 대로 프로그램을 Python 스크립트로 실행하여 파일을 분석합니다.
2. 자동화된 핵 정량화 프로그램을 실행 파일로 실행 가능
참고: 프로그램을 실행 파일로 실행하는 이 방법은 Windows 기반 PC와 호환되며 Windows OS 사용자에게 권장되는 방법입니다. Mac OS 또는 기타 비 Windows 사용자는 프로그램을 Python 스크립트로 실행해야 합니다(섹션 3 참조).
- Python이 컴퓨터에 설치되어 있는지 확인합니다. 필요한 경우 python.org/downloads(선호)에서 Python을 설치하거나 Anaconda 또는 Miniconda를 사용하여 Python을 설치합니다. 발행일 현재 이 프로그램은 Python 버전 3.10-3.12에서 테스트되었습니다.
- 브라우저를 열고 https://github.com/rbudnar/nuclei_counter/releases 로 이동합니다. count_nuclei.zip라는 파일의 최신 버전을 다운로드합니다.
- Downloads(다운로드) 폴더에서 Zip 파일을 마우스 오른쪽 버튼으로 클릭하고 로컬 컴퓨터의 원하는 위치에 모든 파일의 압축을 풉니다( 그림 2A 참조). 이 파일 위치를 열어 두십시오. 이 파일 경로는 2.5단계에서 사용됩니다( 그림 2B 참조).
참고: 이제 Count_nuclei 응용 프로그램 파일로 나타납니다. 이것은 실행 파일입니다. - 검색 창에서 CMD 또는 명령 프롬프트를 검색하여 명령 프롬프트를 엽니다. cd 명령을 사용하여 디렉토리를 실행 파일의 파일 경로로 변경하고( 그림 2C 참조) Enter 키를 누릅니다. 예제 명령 줄: C:\Users\janedoe>cd [cd 뒤에 공백을 둔 다음 파일 경로 복사/붙여넣기]
- 다음 명령줄에서 다음을 입력하고 Enter 키를 누릅니다.
count_nuclei.exe --img_dir "경로/대상/이미지" --output_dir "경로/대상/출력" --save_images --output_count_filename "results.csv"
여기서 path/to/images는 분석할 이미지가 포함된 폴더의 파일 경로입니다(1.14단계 참조). path/to/output은 .csv 파일을 저장해야 하는 폴더의 파일 경로입니다(제공되지 않은 경우 기본값은 현재 작업 디렉토리입니다). results.csv는 결과 카운트가 저장될 파일 이름입니다(제공되지 않은 경우 출력 파일 이름은 기본적으로 count_results.csv로 설정됨).
참고: 명령줄에는 따옴표가 필요합니다. 파일 경로 앞에 따옴표 뒤에 공백이 없는지 확인합니다( 그림 2C 참조). - 다음 명령줄이 나타나면 처리가 완료된 것입니다. 이제 등고선 및 결과 스프레드시트를 2.5단계에서 출력 디렉터리로 지정된 위치(즉, path/to/output, 그림 2C 참조)에서 사용할 수 있습니다.
- 윤곽을 육안으로 검사하여 계수 품질을 확인합니다. 이제 섹션 4에 설명된 대로 결과를 사용하여 데이터를 정규화할 수 있습니다.
참고: 윤곽이 핵을 놓친 것으로 보이거나(예: 핵이 너무 희미하게 보임) 핵 그룹을 단일 항목으로 군집화하는 경우(예: 과다 노출된 이미지, 너무 많은 배경, 아티팩트) 사용자는 문제 해결 표를 참조해야 하며 섹션 4로 이동하기 전에 이미지 획득을 최적화하기 위해 섹션 1로 돌아가야 할 수 있습니다.
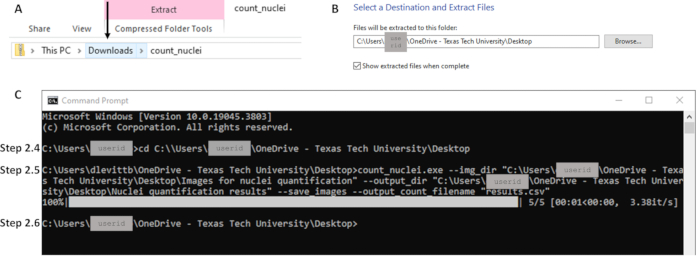
그림 2: 섹션 2에 대한 워크플로우의 주요 부분의 예. (A) 다운로드 폴더에서 파일을 추출하고, 화살표로 표시된 대로 올바른 폴더를 클릭하도록 주의하여 2.3단계에서 원하는 대상으로 추출(B)을 활성화합니다. (C) 2.4단계 및 2.5단계에 대한 명령의 예와 2.6단계의 완료를 나타내는 다음 명령줄. 2.4단계(C)의 파일 경로는 2.3단계(B)의 추출 위치와 일치합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 자동화된 핵 정량화 프로그램을 Python 스크립트로 실행
- Python이 컴퓨터에 설치되어 있는지 확인합니다. 필요한 경우 python.org/downloads(선호)에서 Python을 설치하거나 Anaconda 또는 Miniconda를 사용하여 Python을 설치합니다. 발행일 현재 이 프로그램은 Python 버전 3.10-3.12에서 테스트되었습니다.
- 브라우저를 열고 https://github.com/rbudnar/nuclei_counter 로 이동합니다. Green Code 버튼을 클릭한 다음 Download ZIP 을 클릭하여 코드 저장소를 다운로드합니다( 그림 3A 참조).
- Windows OS에 대한 다음 지침을 사용합니다. Mac OS 기반 컴퓨터의 경우 3.4단계를 참조하십시오.
- Downloads(다운로드) 폴더에서 File(파일)을 마우스 오른쪽 버튼으로 클릭하고 Extract All(모두 추출)을 선택하여 로컬 컴퓨터에 파일 압축을 풉니다(그림 3B-C 참조).
- 추출된 파일 위치에서 nuclei_counter-main이라는 폴더를 엽니다. 이것은 코드 저장소입니다.
참고: 이 프로그램을 처음 사용할 때만 다운로드하면 됩니다. 압축을 푼 파일을 액세스 가능한 네트워크 위치로 이동하고 해당 위치를 기록해 두기만 하면 됩니다. - 검색 창에서 CMD 또는 명령 프롬프트를 검색하여 명령 프롬프트를 엽니다. Python이 Anaconda 또는 Miniconda에 의해 설치된 경우 표준 명령 프롬프트 대신 Anaconda 프롬프트를 엽니다. 검색 창에 Anaconda Prompt를 입력하여 찾을 수 있습니다.
- cd 명령을 사용하여 디렉터리를 코드 저장소로 변경하고 Enter 키를 누릅니다. 대/소문자를 구분하는 아래 코드 줄을 참조하십시오. 파일 경로를 복사할 때 nuclei_counter-main이라는 폴더가 열려 있는지 확인합니다. pyproject 또는 pyproject.toml이라는 파일이 표시되어야 합니다( 그림 3D 참조). 예제 명령 줄: C:\Users\janedoe>cd [cd 뒤에 공백을 둔 다음 파일 경로 복사/붙여넣기]
- 다음 명령줄에서 공백과 마침표를 포함하여 다음을 입력합니다( 그림 3D 참조).
pip install -e 입니다.
참고: 3.3.5 및 3.3.6 단계는 이 프로그램을 컴퓨터에서 처음 실행할 때만 완료하면 됩니다. 이 작업이 이미 완료된 경우 3.3.7단계로 진행합니다. 설치 프로세스를 초기화하는 데 몇 분 정도 걸릴 수 있습니다. - 엔터 키를 치시오. 이 명령을 실행하면 이 스크립트를 실행하는 데 필요한 라이브러리가 설치되고 코드 조정이 필요한 이벤트에서 편집 가능한 모드로 리포지토리를 설치합니다(그림 3D 참조).
- 다음 명령줄에 다음을 입력하여 디렉터리를 기본 소스 코드 디렉터리로 변경합니다( 그림 3E 참조).
CD "nuclei_counter" - 다음 명령줄에서 다음을 입력하고 Enter 키를 누릅니다( 그림 3E 참조).
python count_nuclei.py --img_dir "경로/to/이미지" --output_dir "경로/to/output" --save_images --output_count_filename "results.csv"
여기서 path/to/images는 분석할 이미지가 포함된 폴더의 파일 경로입니다(1.14단계). path/to/output은 .csv 파일을 저장해야 하는 폴더의 파일 경로입니다(제공되지 않은 경우 기본값은 현재 작업 디렉토리입니다). results.csv는 결과 카운트가 저장될 파일 이름입니다(제공되지 않은 경우 출력 파일 이름은 기본적으로 count_results.csv로 설정됨).
참고: 명령줄에는 따옴표가 필요합니다. 파일 경로 앞에 따옴표 뒤에 공백이 없는지 확인합니다. python 스크립트는 제공된 파일 경로의 파일을 읽고 디렉토리의 모든 이미지에 대해 핵 계수 프로그램을 실행합니다( 그림 3E 참조). 결과 개수는 지정된 위치의 결과 .csv 파일에 저장됩니다. - 다음 명령줄이 나타나면 처리가 완료된 것입니다( 그림 3E 참조). 이제 윤곽선 및 결과 스프레드시트를 3.3.8단계에서 출력 디렉터리로 지정된 위치(즉, path/to/output)에서 사용할 수 있습니다.
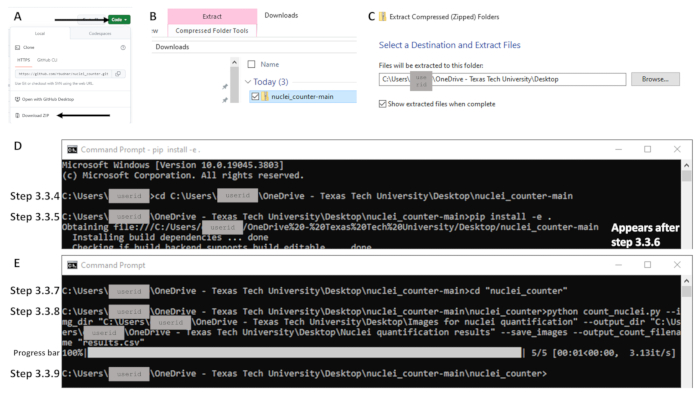
그림 3: Windows OS를 사용하는 섹션 3에 대한 워크플로의 주요 부분의 예. 코드에 액세스하려면 코드 라고 표시된 녹색 버튼을 클릭하고 (A) Zip 파일(화살표로 표시)을 다운로드합니다. (B) Zip 파일(파란색으로 표시)을 마우스 오른쪽 버튼으로 클릭하고 (C) 원하는 대상을 설정하여 압축을 풉니다. (D) 명령 프롬프트를 연 후 3.3.4 및 3.3.5 단계에 대한 명령의 예. 3.3.5 단계의 명령은 모두 한 줄에 있습니다. 다음 줄에서 "Obtaining file"로 시작하는 텍스트는 Enter 키를 누른 후에 나타납니다(3.3.6단계, 즉, 명령 실행). (E) 3.3.7 및 3.3.8 단계에 대한 명령의 예, 3.3.8 단계 실행 후 진행 표시줄 및 3.3.9 단계의 완료를 보여주는 명령 줄. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- Mac OS의 경우 다음 지침을 사용하십시오.
- Downloads(다운로드) 폴더에서 File(파일 ) 메뉴를 클릭하고 Open( 열기 )을 선택하여 로컬 컴퓨터에 파일의 압축을 풉니다( 그림 4A 참조).
- 추출된 파일 위치에서 nuclei_counter-main이라는 폴더를 엽니다. 이것은 코드 저장소입니다.
참고: 이 프로그램을 처음 사용할 때만 다운로드하면 됩니다. 압축을 푼 파일을 액세스 가능한 네트워크 위치로 이동하고 해당 위치를 기록해 두기만 하면 됩니다. - 스포트라이트를 여는 command+스페이스 바 를 눌러 터미널을 엽니다. 그런 다음 "터미널"을 검색합니다.
- cd 명령을 사용하여 디렉터리를 코드 저장소로 변경하고 Enter 키를 누릅니다. 대/소문자를 구분하는 아래 코드 줄을 참조하십시오. 텍스트 문서에 파일 경로를 입력하고 터미널에 복사합니다. 파일 경로가 cd 바로 앞의 텍스트로 시작하는지 확인합니다(예: 그림 4B의 /Users/userid). 예제 명령 줄: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- 다음 명령줄에서 사용자 ID 뒤에 달러 기호가 나타납니다( 그림 4B 참조). 달러 기호 뒤에 공백이 있는지 확인하고 명령 끝에 공백과 마침표를 포함하여 다음을 입력합니다. 공백 없이 pip 바로 뒤에 적절한 Python 버전을 포함합니다(예: pip3 install -e ).
참고: 3.4.5 및 3.4.6 단계는 컴퓨터에서 이 프로그램을 처음 실행할 때만 완료하면 됩니다. 이 작업이 이미 완료된 경우 3.4.7단계로 진행합니다. 설치 프로세스를 초기화하는 데 몇 분 정도 걸릴 수 있습니다. - 엔터 키를 치시오. 이 명령을 실행하면 이 스크립트를 실행하는 데 필요한 라이브러리가 설치되고 코드 조정이 필요한 이벤트에서 편집 가능한 모드로 리포지토리를 설치합니다.
- 다음 명령줄에서 달러 기호 뒤에 공백이 있는지 확인한 후 다음을 입력하여 디렉토리를 기본 소스 코드 디렉토리로 변경합니다( 그림 4C 참조).
CD "nuclei_counter" - 다음 명령줄에서 아래 명령줄을 입력합니다. 3 이외의 버전을 사용하는 경우 Python 뒤의 숫자를 올바른 버전 번호로 바꿉니다. 그런 다음 Enter 키를 누릅니다( 그림 4C 참조).
python3 count_nuclei.py --img_dir "경로/대상/이미지" --output_dir "경로/대상/출력" --save_images --output_count_filename "results.csv"
여기서 path/to/images는 분석할 이미지가 포함된 폴더의 파일 경로입니다(1.14단계). path/to/output은 .csv 파일을 저장해야 하는 폴더의 파일 경로입니다(제공되지 않은 경우 기본값은 현재 작업 디렉토리입니다). results.csv는 결과 카운트가 저장될 파일 이름입니다(제공되지 않은 경우 출력 파일 이름은 기본적으로 count_results.csv로 설정됨).
참고: 명령줄에는 따옴표가 필요합니다. 파일 경로 앞에 따옴표 뒤에 공백이 없는지 확인합니다. Python 스크립트는 제공된 파일 경로의 파일을 읽고 디렉토리의 모든 이미지에 대해 핵 계수 프로그램을 실행합니다( 그림 4C 참조). 결과 개수는 지정된 위치의 결과 .csv 파일에 저장됩니다. - 다음 명령줄이 나타나면 처리가 완료된 것입니다( 그림 4C 참조). 이제 윤곽선 및 결과 스프레드시트를 3.4.8단계에서 출력 디렉터리로 지정된 위치(즉, path/to/output)에서 사용할 수 있습니다.
- 윤곽을 육안으로 검사하여 계수 품질을 확인합니다. 이제 섹션 4에 설명된 대로 결과를 사용하여 데이터를 정규화할 수 있습니다.
참고: 윤곽이 핵을 놓친 것으로 보이거나(예: 핵이 너무 희미하게 보임) 핵 그룹을 단일 항목(예: 과다 노출된 이미지, 너무 많은 배경, 아티팩트)으로 클러스터링하는 경우, 사용자는 표 1 을 참조해야 하며 섹션 4로 이동하기 전에 이미지 획득을 최적화하기 위해 섹션 1로 돌아가야 할 수 있습니다.
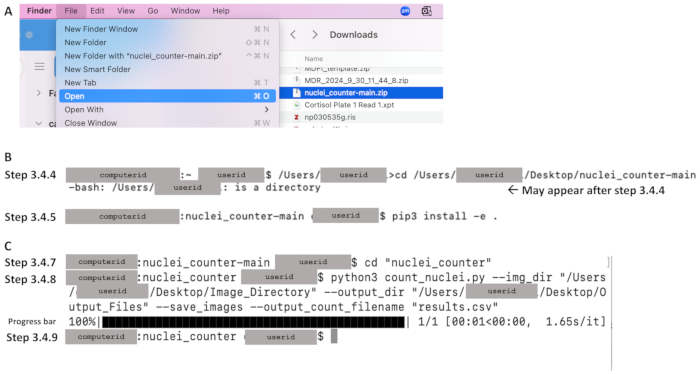
그림 4: Mac OS를 사용하는 섹션 3에 대한 워크플로우의 주요 부분의 예. 코드를 다운로드한 후( 그림 3A 참조) (A) Zip 파일(파란색으로 표시)을 클릭하고 원하는 대상을 설정하여 압축을 풉니다. (B) 터미널을 연 후 3.4.4단계(디렉토리를 코드 저장소로 변경) 및 3.4.5단계(pip 설치)에 대한 명령의 예. (C) 3.4.7 및 3.4.8 단계에 대한 명령의 예, 3.4.8 단계 실행 후 진행 표시줄 및 3.4.9 단계의 완료를 보여주는 명령 줄. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. 카운트를 사용하여 실험 데이터 정규화
- 자동화된 프로그램을 사용하여 결정된 핵 정량화와 각 이미지를 연결하는 results.csv 파일을 엽니다.
- 정규화 방법을 결정합니다: 1) 이미지별 기준(예: 다색 형광 현미경 검사의 경우), 2) 참조 샘플(또는 조건)에 대한 샘플당 평균 핵 수의 비율로 데이터 정규화, 3) 샘플당 핵 수로 정규화(예: 총 핵 수를 샘플 면적에 외삽). 정규화 방법에 대한 권장 사항은 토론 섹션에 있습니다.
- 정규화가 이미지별로 수행되는 경우 각 이미지에 대한 실험 데이터를 해당 이미지의 핵 수로 나누기만 하면 됩니다.
- 데이터를 비율로 정규화하려면 아래 설명된 단계를 수행합니다.
- 단일 복제에 대해 여러 이미지가 수집된 샘플(예: 6-웰 플레이트의 단일 웰에서 캡처된 10-15개의 이미지)의 경우 이미지당 평균 핵 수를 계산합니다. 이러한 핵 수는 단핵 세포 또는 다핵 세포(예: 근관)에서 함께 융합된 여러 세포를 가정하는 세포 수로 간주됩니다.
- 기준 표본 또는 기준 조건(예: 제어 조건의 모든 표본에 대한 평균 개수)을 선택하고 개수를 기록합니다.
- 모든 샘플에 대한 개수를 해당 참조 개수로 나누어 정규화 값을 생성합니다. 참조 샘플이 사용된 경우 참조 샘플의 정규화 값을 1로 사용합니다.
참고: 참조 조건이 사용된 경우 조건 전체의 평균 정규화 값은 이제 1이 됩니다. 기준보다 핵이 적은 샘플(즉, 밀도가 낮음)은 정규화 값이 1보다 작고, 참조보다 핵이 많은 샘플(즉, 밀도가 낮음)은 정규화 값이 1보다 큽니다. - 각 웰에 대한 실험 데이터를 해당 계산된 정규화 값으로 나눕니다.
- 데이터를 샘플당 핵 수로 정규화하려면 아래에 설명된 단계를 따르십시오.
- 단일 복제에 대해 여러 이미지가 수집된 샘플(예: 6-웰 플레이트의 단일 웰에서 캡처된 10-15개의 이미지)의 경우 이미지당 평균 핵 수를 계산합니다. 이러한 핵 수는 단핵 세포 또는 다핵 세포(예: 근관)에서 함께 융합된 세포의 수를 가정하는 세포 수로 간주됩니다.
- 슬라이드(커버슬립), 웰 또는 데이터가 파생된 접시의 표면적을 결정합니다. 이 정보는 제품 웹 사이트의 사양 섹션에서 찾을 수 있습니다.
- 캡처된 각 이미지의 영역을 결정합니다. 이 정보는 이미징 또는 분석 소프트웨어에서 메타데이터로 사용할 수 있는 경우가 많습니다.
- 샘플 표면적과 이미지 영역이 동일한 단위(예: mm2)로 표현되는지 확인합니다. 그렇지 않은 경우 원하는 단위로 변환하십시오.
- 다음 방정식을 사용하여 각 샘플의 총 개수를 계산합니다.
TC = AC • (SA/IA)
여기서 TC는 총 수, AC는 평균 수, SA는 표면적, IA는 이미지 영역입니다. - 표본에 해당하는 TC로 실험 데이터를 나눕니다.
- 실험 데이터를 정규화하고 적절하게 통계 분석에 사용합니다.
결과
각 배치 이미지 실행은 1) 식별된 핵의 윤곽을 보여주는 윤곽이 적용된 이미지 파일 세트(그림 5) 및 2) 이미지 파일 이름과 관련 개수를 연결하는 .csv 파일(스프레드시트)을 생성합니다. 윤곽을 보면 사용자가 계수 품질을 시각적으로 평가할 수 있습니다. 구체적으로, 섹션 1에 따라 얻어진 이미지는 핵이 프로그램에 의해 계수되었음을 나타내는 실선 녹색 선으로 둘러싸인 모든(또는 거의 모든) 핵을 가져야 합니다. 이 윤곽선은 여기에서 프로그램을 조정하는 데 사용되었습니다.
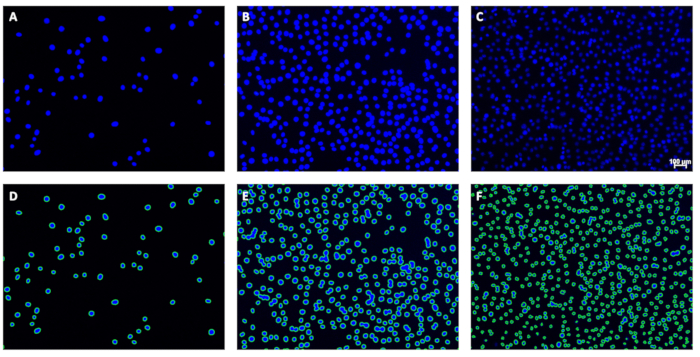
그림 5: 원본 이미지와 해당 윤곽선의 예. (에이씨) 분석 전 원본 이미지와 분석 후 (D-F) 해당 윤곽의 예가 바로 아래에 있습니다. 눈금 막대 (C)는 100 μm를 나타냅니다. 핵 정량화에 사용되는 이미지에서 스케일 바는 생략해야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
초기 결과를 사용한 검증
다양한 세포 밀도의 C2C12 근아세포를 메탄올 고정하고 장착 매체에서 DAPI로 대조염색했으며 .jpg 파일 형식으로 10배 배율로 120개의 이미지를 얻었습니다. 이러한 이미지가 포함된 폴더로 설정된 이미지 디렉토리로 설정된 상태에서 자동화된 프로그램을 실행하고 결과 .csv 파일을 저장했습니다. 이와는 별개로, 각 이미지는 자동화된 계수와 서로의 계수에 눈이 먼 두 명의 훈련된 연구원에 의해 수동으로 정량화되었습니다. 원시 이미지, 자동화된 프로그램에 의해 생성된 윤곽선, 수동 및 자동 계수를 포함한 결과 스프레드시트는 Open Science Framework(OSF) 저장소에서 사용할 수 있습니다(섹션 1의 링크 참조). 클래스 내 상관 계수(ICC, 평균 측정값)는 1) 두 개의 수동 계수와 2) 두 개의 수동 계수 및 자동화된 프로그램을 사용하여 얻은 카운트 간의 평가자 간 신뢰성을 결정하기 위해 SPSS Statistics를 사용하여 계산되었습니다. 형광 현미경 16,17,18을 사용하여 얻은 이미지에서 물체를 정량화하도록 설계된 다른 프로그램을 검증하기 위해 유사한 접근 방식이 사용되었습니다. 또한 ICC는 전체 데이터 세트를 평가할 때 셀 밀도에 따른 신뢰성의 잠재적 변화를 가리지 않도록 하기 위해 각 사분위수에 대한 평균 수동 계수와 자동 계수 사이에서 계산되었습니다. 사분위수는 평균 수동 계수를 기반으로 25번째, 50번째 및 75번째 백분위수 절단 지점을 사용하여 정의되었으며 범위는 사분위수 1(Q1, 가장 낮은 세포 밀도)에서 Q4(가장 높은 세포 밀도)까지입니다. ICC는 평균 측정치를 기반으로 보고되며 나쁨(ICC <0.50), 보통(0.50 ≤ICC <0.75), 양호(0.75 ≤ICC <0.90) 또는 우수(ICC ≥0.90)19의 정의에 따라 해석됩니다.
이미지당 핵 범위는 수동 계수 평균을 기준으로 44.5에서 1160 사이였습니다. 두 개의 수동 계수 간에 평가자 간 신뢰도가 우수했다(ICC > 0.999, p < 0.0001; 그림 6A) 평균 수동 계수와 자동 계수 사이(ICC = 0.993, p < 0.0001; 그림 6B). 신뢰도는 사분위수 전반에 걸쳐 우수하게 유지되었다(Q1: ICC = 0.996, p < 0.0001; Q2: ICC= 0.997, p < 0.0001; Q3: ICC = 0.998, p < 0.0001; Q4: ICC = 0.986, p < 0.0001; 그림 6C-F). 그림 6B의 최적선에서 멀리 떨어진 단일 데이터 포인트는 그림 7에 표시된 이미지를 나타냅니다. 이미지에는 여러 핵이 그룹화된 여러 영역이 있어 컴퓨터 비전 프로그램이 각 핵을 인식하지 못합니다. 이 문제는 더 짧은 노출 시간을 사용하여 해결할 수 있습니다. 표 1에서는 이러한 이미지를 처리하는 방법을 설명합니다.
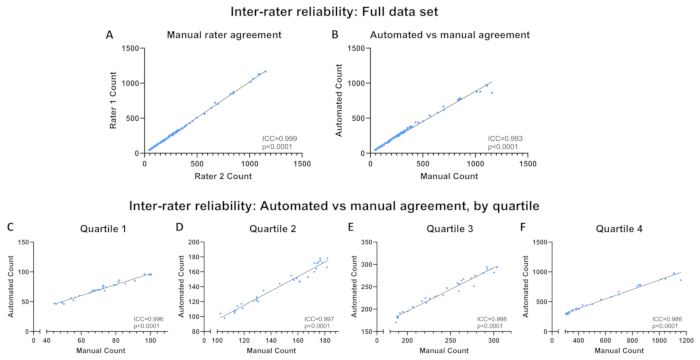
그림 6: 평가자 간 신뢰도. 전체 데이터 세트에 대한 평가자 간 신뢰도: (A) 수동 평가자 카운트 사이 및 (B) 자동 계수와 평균 수동 계수 사이. (씨-에프) 자동 계수와 사분위수별 평균 수동 계수 사이의 평가자 간 신뢰성. ICC(intraclass correlation coefficient)로 표현되는 신뢰도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 7: 정확한 핵 정량화를 방해하는 두 개의 장벽이 있는 이미지의 예. (A) 원본 이미지 및 (B) 관련 윤곽선. 화살표 1은 여러 개의 핵이 뭉쳐 있는 영역을 나타내며, 여러 핵의 소프트웨어 감지를 방해할 만큼 둥근 핵 모양이 왜곡되어 있습니다. 이 문제는 이 이미지의 여러 위치에서 발생합니다. 화살표 2는 여러 핵 뒤에 나타나는 희미한 후광을 나타냅니다. 이 후광은 소프트웨어가 그것을 핵으로 포함할 수 있을 만큼 충분히 밝게 보였고, 그로 인해 그것을 둘러싼 여러 핵의 수를 잃게 되었습니다. 이 이미지는 그림 6B의 최적선에서 멀리 떨어진 단일 데이터 점을 나타내며, 이러한 장벽의 잠재적인 문제를 강조합니다. 눈금 막대 (A)는 100 μm를 나타냅니다. 핵 정량화에 사용되는 이미지에서 스케일 바는 생략해야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
당사의 핵 정량화 프로그램은 기존 옵션에 비해 몇 가지 장점이 있습니다: 최소한의 기술만 필요하고, 핵 정량화의 특정 작업에 대해 검증되었으며, 오픈 소스입니다. 후자는 비용 관련 장벽을 극복합니다. 궁극적으로 이 프로그램은 세포 및 분자 생물학자에게 형광 현미경을 사용하여 캡처한 이미지에서 핵을 빠르고 정확하게 정량화할 수 있는 추가 옵션을 제공합니다. 현재 사용 가능한 자동 핵 또는 세포 계수 프로그램은 모든 사용자가 선호하지 않으며, 일부는 비용이 많이 들고, 특정 응용 분야에 대한 검증이 최소한이거나 아예 없을 수 있습니다. BioTek Cytation 라인과 같은 특수 장비에는 다양한 추가 응용 분야 외에도 핵 염색20을 사용하여 현장 세포 계수를 검증하는 기술 게시판이 함께 제공됩니다. 검증 데이터가 존재하고 Cytation 제품 라인이 널리 사용되지만, 특히 살아있는 세포 데이터를 쉽고 효율적으로 정규화하기 위해 Seahorse Extracellular Flux Analyzer와 인터페이스할 때 이 제품은 장비 자금이 제한된 실험실에서 비용을 많이 부담할 수 있습니다. 예를 들어, 형광 현미경 검사를 위한 4개의 대물렌즈와 4개의 필터 큐브가 장착된 기본 모델의 정가는 2024년에 미화 100,000달러를 초과했습니다. 특수 장비를 구입할 수단이 없는 사람들을 위해 오픈 소스 소프트웨어는 이미지 분석을 위한 무료 대안을 제공합니다. 핵 및 세포 정량화에 유용한 오픈 소스 소프트웨어로는 ImageJ(National Institutes of Health, Bethesda, MD, USA; 21에서 사용 가능)가 있습니다. 이제 ImageJ 플랫폼을 사용하여 이미지 처리 자동화를 위한 매크로 스크립트를 작성하려는 사용자를 위해 플러그인을 사용할 수 있습니다. 전체 세포 이미지와 함께 위상차 또는 명시야 현미경을 사용하는 ImageJ 기반 세포 계수 자동화는 이전에 설명되었습니다22,23. CellProfiler는 형광 염료 또는 표준 조직학적 염색으로 염색된 세포 이미지를 분석하기 위해 널리 발표된 응용 프로그램이며, 다양한 생물학적 응용 분야에서 CellProfiler의 유용성을 설명하는 초기 논문은 종종 검증24로 인용됩니다. 그러나 이 원고는 특정 생물학적 발견을 검증하지 않으므로 핵 계수25와 같이 사용되는 다양한 자동화 파이프라인에 대한 추가 검증 및 투명성이 필요합니다. 또한 CellProfiler는 세포 분석을 위한 효율적인 소프트웨어 패키지이지만 모든 과학자가 사용자 친화적이라고 생각하는 것은 아닙니다. 따라서 우리는 오픈 소스 소프트웨어를 사용하여 핵을 정량화하는 대체 자동화 프로그램을 개발하고 검증하는 것을 목표로 했습니다. 구체적으로 말하자면, 이 워크플로우를 개발하기 위해 Python의 기존 컴퓨터 비전 라이브러리를 사용했습니다.
이 방법은 최종 사용자가 DAPI26과 같은 DNA 염색을 사용하여 염색한 세포 또는 조직으로 시작합니다. 자동화 소프트웨어와 호환되는 방식으로 이미지를 캡처하고 저장하기 위한 일반적인 지침이 제공됩니다. 세부 사항은 현미경 및 이미징 소프트웨어마다 다를 수 있지만 섹션 1의 일반적인 작업 흐름은 정량화 가능한 이미지를 얻기 위해 동일합니다. 살아있는 샘플을 사용하는 경우 적절한 개인 보호 장비를 착용하고 이미징 환경이 해당 샘플에 적합한지 확인하기 위해 주의를 기울여야 합니다(예: 온도, CO2 비율, 완충액 추가 등).27. 이 프로토콜은 DNA 염료의 이미징만을 설명하지만, 사용자는 병합 목적15을 위해 동일한 영역의 다른 색상을 캡처할 수 있으며, 정량화 프로그램과 함께 사용하기 위해 핵 이미지를 별도의 폴더에 복사하기만 하면 됩니다. 이미징은 살아있는 샘플에 적합한 PPE가 있는 암실에서 수행해야 하며 필요에 따라 적절한 소독(예: 70% 에탄올)을 사용해야 합니다. 얼룩진 샘플은 조명을 켜기 전에 빛으로부터 보호해야 합니다. 고품질 이미지 캡처는 정확한 핵 정량화를 위해 매우 중요합니다. 적절한 노출 시간과 밝은 핵과 검은색 배경 사이의 강한 대비가 있는 이미지에서도 세포 그룹 뒤에 과도하게 응집된 세포나 후광이 존재하면( 그림 7 참조) 정량화 정확도가 떨어질 수 있습니다. 이러한 경우, 신호의 강도를 줄이기 위해 노출 시간을 변경해야 할 수 있으며, 핵 신호의 병합을 최소화하고 헤일로를 최소화하는 동시에 핵의 둥근 모양을 감지할 수 있을 만큼 충분히 강한 신호를 유지해야 합니다. 이러한 단계가 충분하지 않은 경우 샘플의 인접한 부분을 이미징해야 할 수 있습니다. 단일 실험을 위한 모든 이미지는 동일한 배율로 캡처되어야 하며, 사용된 대물렌즈에 해당하는 배율은 이미징 소프트웨어에서 적절하게 설정되어야 합니다. 이미징에 사용되는 대물렌즈가 이미징 영역에서 전체 웰(또는 슬라이드) 영역으로 핵 개수를 외삽할 때 이미징 소프트웨어의 설정과 일치하는 것이 특히 중요합니다. 이 단계는 대물렌즈가 전환될 때마다 수행되어야 하지만 현미경과 소프트웨어를 설정할 때 쉽게 간과됩니다. 섹션 1에 필요한 시간은 여러 요인(예: 형광 현미경 검사에 대한 사용자 경험, 이미지 및 샘플 수, 현미경 등)에 따라 다릅니다. 2개의 6-well 플레이트에서 well당 10-15개의 염색된 핵 이미지를 캡처하려면 사용자는 약 1-2시간의 시간을 예약해야 합니다.
이미지를 캡처하여 전용 폴더에 저장하면 정량화할 준비가 된 것입니다. 정량화를 위한 가장 간단한 방법은 파이썬이나 라이브러리를 설치할 필요 없이 사전 패키징된 자동화 프로그램을 실행하는 것입니다(섹션 2). 프로그램은 실행 파일로 준비되었으며 Pyinstaller를 사용하여 패키지화되었다는 점에 유의해야 합니다. Pyinstaller를 사용하여 빌드된 소프트웨어는 일반적으로 바이러스 백신 소프트웨어에 의해 플래그가 지정됩니다. 바이러스 백신 소프트웨어가 파일 열기를 차단하는 경우 사용자는 계속 진행하려면 파일을 안전한 것으로 표시해야 합니다. 이는 섹션 2를 성공적으로 수행하는 데 중요합니다. 또는 원하는 경우 모든 사용자가 자동화된 수량화 프로그램을 실행 파일이 아닌 Python 스크립트(섹션 3)로 실행하도록 선택할 수 있습니다. 사용자가 Mac 또는 Linux 운영 체제를 사용하여 프로그램을 실행하려면 Python을 통해 프로그램을 실행해야 합니다. 이 방법은 Pyinstaller가 필요하지 않지만 기술적으로 더 어려울 수 있습니다. 섹션 3에 자세히 설명된 예제와 함께 단계별 프로토콜은 이 프로그램을 스크립트로 실행하는 데 대한 기술적 장벽을 제거하는 것을 목표로 합니다. 섹션 2 또는 섹션 3을 사용하여 프로그램을 실행하는지 여부에 관계없이 코드가 명령 프롬프트에 올바르게 입력되었는지 확인하는 것이 중요합니다. 예제 코드 줄은 프로토콜 전체에 포함되어 있으며 사용자는 간격, 따옴표 및 기타 문장 부호에 주의해야 합니다. 또한 사용자는 파일 경로가 정확한지 확인해야 합니다. Word 문서에서 사용할 수 있는 이미지 및 출력의 파일 경로를 사용하는 것이 도움이 됩니다. 이 Word 문서에 명령을 입력하고 필요에 따라 복사/붙여넣기할 수도 있습니다.
섹션 4에서 설명한 바와 같이, 이 자동화된 핵 정량화 프로그램에서 얻은 계수를 사용하여 실험 데이터를 정규화하는 여러 가지 접근 방식이 있습니다. 실험 데이터가 각 이미지에 다수의 이벤트를 포함하고 비율(예: 부속 V-양성 세포(28)의 비율, ACE2 수용체(29)를 발현하는 세포의 비율, 염색된 입자를 흡수하는 세포의 수30 등)가 필요한 이미지 기반 실험의 경우, 각 이미지에 대한 실험 데이터를 결과 수(0에서 1의 비율로 나눕니다)로 간단히 나눌 수 있습니다. 그런 다음 동일한 슬라이드, 접시 또는 웰의 복제 이미지에서 평균 비율을 계산할 수 있습니다. 백분율이 필요한 경우 최종 비율에 100을 곱하기만 하면 됩니다. 마지막으로, 이러한 평균 비율 또는 백분율은 다운스트림 통계 분석에 사용할 수 있습니다. 정규화할 실험 데이터가 살아있는 세포의 기능 실험(예: 산소 소비율 6,11,12) 또는 세포 배양 상등액(예: 상등액31 내 젖산 탈수소효소 활성 또는 특정 분비 사이토카인32의 농도 등)에서 얻은 경우, 먼저 동일한 웰에서 캡처된 모든 이미지에서 핵 수를 평균화해야 합니다. 그런 다음, 핵 계수는 슬라이드(커버슬립), 웰 또는 접시의 표면적에 외삽될 수 있으며, 또는 계수는 기준 샘플 또는 조건에 대한 비율로 표현될 수 있습니다. 슬라이드, 웰 또는 접시의 표면적에 대한 외삽은 데이터가 파생된 세포의 수(단핵 세포를 사용하는 경우)에 대한 실험 데이터를 표현하는 데 유용합니다(예: 103 세포당 분당 산소 소비). 이 방법을 사용하려면 슬라이드(커버슬립), 웰 또는 접시의 표면적과 각 이미지에 캡처된 영역을 알아야 합니다. 샘플이 사용 가능한 전체 표면적을 커버하지 않거나, 실험 데이터가 소량의 상등액에서 추출되었거나(즉, 단위 부피당 활성 또는 농도로 표현됨), 이미지 및/또는 표면적을 알 수 없는 경우, 가장 좋은 옵션은 단일 샘플에 대한 모든 이미지에서 핵 수를 평균화하고 참조 샘플 또는 조건을 기준으로 핵 수를 표현하는 것입니다. 결과 값은 핵 밀도 또는 세포 밀도(단핵 세포를 사용하는 경우)에 대한 실험 데이터를 수정하는 데 사용됩니다.
이 방법은 실행이 간단하지만 사용자가 알고 있어야 하는 몇 가지 잠재적인 문제가 있습니다. 이러한 잠재적 문제와 가능한 원인 및 문제 해결을 위한 문제 해결 단계가 표 1에 나열되어 있습니다. 여기에 설명된 프로그램은 기존 소프트웨어에 비해 주요 이점이 있습니다. 그러나 몇 가지 제한 사항을 인정해야 합니다. 가장 큰 단점은 정량화 프로그램은 명시야 이미지가 아닌 형광 현미경을 사용하여 캡처한 이미지에만 사용할 수 있다는 것입니다. 10배 배율로 캡처한 DAPI 염색 이미지가 프로그램을 구축하는 데 사용되었지만, 이 프로그램은 색상이나 크기가 아닌 핵의 형태를 인식하도록 구축되었기 때문에 다른 DNA 염료 및 배율과 함께 사용할 수 있습니다. 다양한 세포 밀도를 포함하는 이미지에서 프로그램의 정확성을 검증했지만 최종 사용자는 범위를 벗어난 세포 밀도를 가질 수 있습니다. 이 경우, 고품질 이미지를 얻을 수 있다면 정확한 계수를 예상할 수 있습니다. 그러나 사용자는 정확성을 보장하기 위해 윤곽과 출력을 주의 깊게 확인해야 합니다. 세포 배양 응용 분야의 경우, 이미지 내 핵의 정량화는 단층의 세포에 가장 적합합니다. 배양된 세포가 겹치면 핵이 가려져 핵 수가 과소평가될 수 있습니다. 이미지에서 핵을 정량화하는 2차원 특성으로 인해 현탁액, 두꺼운 조직 블록 또는 기타 3차원 응용 분야에 있는 세포의 총 핵 수를 결정하기 위해 이 프로그램을 사용할 수 없습니다. 그러나 이 프로그램은 두꺼운 조직 블록과 같은 3차원 모델에서 캡처한 2차원 이미지에서 형광 DNA 염료로 표지된 핵을 여전히 정량화할 수 있습니다. 원하는 응용 분야를 위해 이미지에서 핵을 정량화하는 유용성은 최종 사용자가 결정해야 합니다.
| 문제 | 가능한 원인 | 가능한 해결책 |
| 이미지의 과도한 배경 | 노출 시간이 너무 높음 | 노출 시간 단축 |
| 핵은 이미지에서 서로 합쳐지는 것처럼 보입니다. | 형광 DNA 염료 농도가 너무 높거나 너무 많은 DNA 염료 장착 매체 | 최적화하는 동안 염료 농도를 줄입니다. 가능한 한 최소한의 장착 매체를 사용하십시오. |
| 핵이 너무 밝게 보이거나 일부 핵이 보이지 않음 | 노출 시간이 너무 짧음 | 노출 시간 늘리기 |
| 광표백 | 염색된 샘플을 어두운 곳에 보관하고 샘플이 조명에 노출되는 것을 최소화하여(예: 필요한 최소 노출 시간을 사용하고 이미지를 캡처한 후 또는 메모하는 동안과 같이 필요하지 않을 때 조명을 끄음) 광표백을 방지합니다. | |
| 후광 효과 또는 핵 특이적이지 않은 모양이 나타나는 경우 | 노출 시간이 너무 높음 | 노출 시간 단축 |
| 살아있는 세포의 섬세한 작업용 와이퍼 또는 일부 탁상용 보호대에서 보푸라기 또는 고정된 세포에 부착 | 보푸라기가 없는 환경 보장: 샘플이 노출될 때(예: 살아있는 세포를 위한 생물 안전 캐비닛, 고정 세포의 고정 및 건조 중 또는 염색 공정 중) 섬세한 작업용 와이퍼의 사용을 최소화합니다. 벤치탑 프로텍터가 염색하는 동안 슬라이드나 플레이트에 보푸라기를 남기지 않는지 확인하십시오. | |
| 고정된 세포/조직 표면의 기타 이상 | 샘플을 각별히 주의하여 다루고 고정 및 염색하는 동안 적절한 예방 조치와 모범 사례를 따르십시오. | |
| 광 경로의 방해 | 현미경의 모든 부분이 깨끗하고 보푸라기와 먼지가 없는지 확인하십시오. 이미징 전에 환경 먼지와 보푸라기를 제거하기 위해 70% 에탄올로 슬라이드/플레이트/접시 외부를 청소합니다. | |
| 실행 파일이 열리지 않습니다. | 바이러스 백신 소프트웨어가 파일을 차단했습니다. | 파일을 안전한 것으로 플래그 지정 |
| 명령 프롬프트의 오류 | 명령 줄의 간격 문제 | 명령 줄에서 명령의 시작 부분이 공백 없이 ">" 기호 바로 뒤에 입력되었는지, 파일 경로에 대한 열린 따옴표 뒤에 공백이 없는지, 제공된 코드 및 예제의 간격과 일치하는지 다시 확인합니다. |
| 명령 줄에서 부적절한 대소 문자가 사용됩니다. | 코드는 대/소문자를 구분합니다. 대문자와 소문자를 적절하게 사용하고 사용하십시오 | |
| 파일 경로 문제 | 파일 경로가 연결된 파일 폴더의 내부를 나타내는지 확인합니다(즉, 파일 경로를 복사할 때 파일 폴더가 열려 있음). | |
| 위의 문제 해결 단계를 사용하여 해결되지 않은 프로그램 문제 | 위에서 언급한 소프트웨어 또는 명령줄 문제. | GitHub를 통해 문제를 제출합니다. https://github.com/rbudnar/nuclei_counter 로 이동합니다. 상단의 메뉴 표시줄에서 "문제"("코드" 오른쪽에 있는 두 번째 옵션)를 선택합니다. 오른쪽에 있는 "New Issue"라고 표시된 녹색 버튼을 클릭하여 제출합니다. 문제를 제출하기 전에 GitHub에 로그인하라는 메시지가 표시됩니다. 필요한 경우 무료 계정을 만들 수 있는 옵션도 있습니다. |
표 1: 고품질 이미지를 캡처하고 자동화된 핵 정량 분석 워크플로우를 실행하기 위한 문제 해결.
전반적으로, 본 명세서에 설명된 방법은 컴퓨터 비전 라이브러리를 사용하여 개발하고 여러 평가자의 수동 계수에 대해 검증한 자동 핵 정량화 프로그램을 사용하기 위한 단계별 세부 정보를 제공합니다. 이 프로그램은 다양한 수준의 코딩 관련 기술을 가진 과학자들이 유용성을 보장하기 위해 개발되었으며, 무료로 사용할 수 있으며, 핵 정량화 작업을 위해 특별히 검증되었습니다. 수동 계수와 비교하여 이 프로그램은 최종 사용자의 귀중한 시간을 절약하고 데이터 정규화를 위한 정확하고 신뢰할 수 있는 핵 계수를 제공합니다.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
이 연구를 위한 자금은 미국 국립보건원(National Institutes of Health)/국립노화연구소(National Institute on Aging, R01AG084597; DEL 및 HYL) 및 텍사스 공과 대학(DEL)의 창업 자금으로 지원되었습니다. 저자는 이 작업에 기여한 학부 연구자(REH, MRD, CJM, AKW)에게 재정적 지원을 제공한 Texas Tech University Undergraduate Research Scholars 및 TrUE Scholars 프로그램에 감사를 표합니다. 또한 실험실 공간과 장비를 친절하게 공유해 주신 Lauren S. Gollahon 박사와 Michael P. Massett 박사에게도 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
참고문헌
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유