Method Article
שימוש בספריות ראייה ממוחשבת לייעול כימות הגרעינים
In This Article
Summary
מאמר זה מתאר שיטות צעד-אחר-צעד לאוטומציה של כימות גרעינים מבוססי תמונה באמצעות תוכנית הפעלה בקוד פתוח המאומתת על פני מגוון צפיפויות תאים. תוכנית זו מספקת אלטרנטיבה המטפלת בחסמים הקשורים לעלות, נגישות למשתמשים בעלי כישורים טכנולוגיים מוגבלים ואימות ספציפי ליישום שעלול להגביל את התועלת של טכנולוגיות קיימות.
Abstract
מבחני תאים חיים וניתוחי תאים מבוססי תמונה דורשים נורמליזציה של נתונים לפרשנות מדויקת. שיטה נפוצה היא צביעה וכימות גרעינים, ולאחר מכן נורמליזציה של נתונים לספירת גרעינים. ספירת גרעינים זו מתבטאת לעתים קרובות כספירת תאים עבור תאים חד-גרעיניים. בעוד שכימות ידני יכול להיות מייגע וגוזל זמן, שיטות אוטומטיות זמינות עשויות שלא להיות מועדפות על ידי כל המשתמשים, עשויות להיות חסרות אימות עבור יישום ספציפי זה, או עשויות להיות חסכוניות. כאן, אנו מספקים הוראות שלב אחר שלב ללכידת תמונות ניתנות לכימות של גרעינים מוכתמים בכתמי DNA פלואורסצנטיים ולאחר מכן לכימות הגרעינים באמצעות תוכנת ספירת אובייקטים אוטומטית שפותחה באמצעות ספריות ראייה ממוחשבת של Python. אנו גם מאמתים תוכנית זו על פני מגוון צפיפויות תאים. למרות שהזמן המדויק לביצוע התוכנית משתנה בהתאם למספר התמונות וחומרת המחשב, תוכנית זו מאחדת שעות עבודה בספירת גרעינים לשניות להפעלת התוכנית. בעוד שפרוטוקול זה פותח באמצעות תמונות של תאים קבועים ומוכתמים, ניתן לכמת גם תמונות של גרעינים מוכתמים בתאים חיים ויישומי אימונופלואורסצנציה באמצעות תוכנה זו. בסופו של דבר, תוכנית זו מספקת אפשרות שאינה דורשת רמה גבוהה של מיומנות טכנולוגית ומהווה חלופה מאומתת בקוד פתוח כדי לסייע לביולוגים תאיים ומולקולריים לייעל את זרימות העבודה שלהם, תוך אוטומציה של המשימה המייגעת וגוזלת הזמן של כימות גרעינים.
Introduction
ניסויים פונקציונליים ומבוססי תמונה הם קריטיים להבנת ההשפעות של טיפולים ניסיוניים על ביוכימיה ופיזיולוגיה של תאים שלמים. פרשנות תקפה של נתונים מניסויים בביולוגיה של התא תלויה בדיוק וביכולת השחזור של פרוטוקול הניסוי, כולל נורמליזציה של נתונים. לדוגמה, ניתוחים של צריכת חמצן ושיעורי החמצה חוץ-תאית בתאים חיים בנקודת ההתחלה ולאחר טיפול בתרופות ספציפיות מאפשרים הערכה של היבטים שונים של חילוף חומרים אנרגטי 1,2. מדידת פעילותם של אנזימים כגון לקטט דהידרוגנאז בסופרנטנט של תרביות תאים יכולה לסייע בכימות שלמות קרום התא3. צביעת תאים מתורבתים עם נספח V ופרופידיום יודיד לפני הקיבוע מאפשרת הערכה של תאים אפופטוטיים ונמקיים4. עם זאת, הבדלים בצפיפות התאים בין בארות משפיעים על התוצאות של כל אחת מהבדיקות הללו. הסתמכות אך ורק על צפיפות הזריעה עלולה לספק תוצאות מטעות עקב שגיאות בספירת תאים לזריעה, שינויים בצפיפות התאים במדיה בזמן הציפוי, או שיעורים שונים של צמיחת תאים בין דגימות או טיפולים במהלך הניסויים. לכן, נדרשת נורמליזציה של תוצאות הניסוי.
שיטות הנורמליזציה הנוכחיות לנתוני תאים פונקציונליים ומבוססי תמונה כוללות ריכוז חלבון5 או גרעינים או ספירת תאים6. כדי לנרמל נתונים לריכוז החלבון, יש לחלץ תאים בנפחים שווים של מאגר ליזה עם מעכב פרוטאז לאחר הפעלת הבדיקה, ויש לבצע בדיקה נוספת (למשל, בדיקת חומצה ביצינצ'ונינית, בדיקת ברדפורד וכו') לכימות חלבון. הדיוק של שיטה זו מסתמך על מיצוי תאים מלא; השארת כל חלבון מאחור תורמת לטעויות בכימות. ספירת גרעינים או תאים מספקת אלטרנטיבה שבה אין צורך להסיר תאים מהמשטח עליו הם גדלים. במקום זאת, תאים חיים או קבועים מוכתמים או מוכתמים נגדית באמצעות כתמים קולורימטריים או פלואורסצנטיים, וניתן לבצע ספירה על סמך תמונות של תאים שלמים או גרעינים בלבד. כימות גרעינים עדיף על פני תאים שלמים ליישומי מיקרוסקופיה פלואורסצנטית ולנורמליזציה של נתונים בתאים מאוחים (למשל, צינורות) או רקמות. לצורך כימות גרעינים בגישה פלואורסצנטית, צבעי DNA פלואורסצנטיים (למשל, 4′,6-diamidino-2-phenylindole dichloride (DAPI), Hoechst 33342, Hoechst 33358, DRAQ5 וכו') מיושמים על תאים חיים או קבועים ומצולמים באמצעות מיקרוסקופיה פלואורסצנטית. DAPI, Hoechst ו-DRAQ5 נקשרים באופן מועדף לאזורים עשירים באדנין-תימין של DNA 7,8,9, ומספקים אמצעי לזיהוי גרעינים. אופטימיזציה של ריכוז הצבע מונעת קשירה מחוץ למטרה (כלומר, ל-DNA המיטוכונדריאלי, הגורמת לפלואורסצנציה ציטוזולית מנוקבת)10. ניתן לייעל את תהליך האופטימיזציה על ידי שימוש באמצעי הרכבה זמינים מסחרית המכילים DAPI עבור תאים קבועים (למשל, VECTASHIELD Antifade Mounting Medium עם DAPI) וריכוזים מפורסמים של כתמים לתאים חיים (למשל, Hoechst)6,11,12. לאחר הצביעה, גרעינים בתאים חיים או קבועים מוצגים באמצעות מסנן העירור המתאים (למשל, אולטרה סגול [DAPI ו-Hoechst] או אדום רחוק [DRAQ5]). מקסימום עירור עבור DAPI, Hoechst ו-DRAQ5 נמצאים בשני קצות הספקטרום עבור מיקרוסקופיה פלואורסצנטית. תכונה זו, בתוספת ספציפיות ה-DNA של צבעים אלה והשימוש בהם בריכוזים אופטימליים, ממזערת את הקרינה מחוץ למטרה. התמונות המתקבלות מראות גרעינים בהירים על רקע שחור, וגרעינים אלה מכומתים כמדד למספר התאים עבור תאים חד-גרעיניים. בעוד שקיימות שיטות מדויקות אחרות לכימות גרעינים מבוססות כתמים באמצעות כתמים היסטולוגיים ומיקרוסקופ שדה בהיר13, אוטומציה קשה יותר, אם כי אפשרית, באמצעות גישות אלה.
למרות שזה נשאר תקן הזהב, כימות גרעינים ידני הוא מייגע, גוזל זמן ורגיש לטעויות אנוש פוטנציאליות עם ספירה ממושכת. למרות שקיימות תוכניות אוטומטיות לספירת תאים, הן אינן מועדפות על ידי כל המשתמשים, עשויות להיות חסכוניות ואימות עבור יישומים ספציפיים עשוי להיות מינימלי. פייתון הפכה בשנים האחרונות למשאב יקר ערך ונגיש עבור ביולוגים. ספריות ראייה ממוחשבת של Python שימושיות במיוחד לניתוח תמונות14. כאן, אנו מספקים הוראות שלב אחר שלב לשימוש בתוכנית ההפעלה שלנו שפותחה באמצעות קוד Python כדי לכמת גרעינים מוכתמים בצבעים פלואורסצנטיים ומצולמים באמצעות מיקרוסקופיה פלואורסצנטית. כישורי קידוד אינם נחוצים כדי להשתמש בתוכנית המתוארת כאן. זרימת עבודה זו ספציפית לתאים או רקמות עם צבעי DNA פלואורסצנטיים. הוא אינו מיועד לשימוש עם תמונות בעלות שדה בהיר. סעיף 1 מתאר כיצד ללכוד ולשמור תמונות שנאספו באמצעות מיקרוסקופ פלואורסצנטי באופן התואם לזרימת עבודה זו. סעיף 2 מספק הוראות להפעלת תוכנית כימות הגרעינים כפלט הפעלה ואחזור. ניתן להפעיל את תוכנית ההפעלה ישירות מקובץ .exe שסופק ואינה דורשת שינויים כדי לכמת גרעינים בתמונות איכותיות. קובץ הפעלה זה דורש מחשב מבוסס Windows. סעיף 3 מספק הוראות להפעלת התוכנית כסקריפט Python ולא כקובץ הפעלה (נדרש למערכות מבוססות Mac או Linux, אופציונלי למערכות מבוססות Windows) ומאפשר לכוונן את הקוד אם רוצים. סעיף 4 מתאר כיצד להשתמש בתוצאות לנורמליזציה. לאחר פרוטוקולים אלה מופיעות תוצאות מייצגות, כולל נתוני אימות מהמעבדה שלנו.
Protocol
הערה: ניתן למצוא קבצים משלימים בקישור הבא https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. צילום ושמירת תמונות באמצעות מיקרוסקופ פלואורסצנטי
- הכן דגימות תאים או רקמות להדמיה, כולל צביעה בצבע DNA רצוי. כדי להשיג את התמונות המשמשות כאן, מיובלסטים C2C12 (CRL-1772, American Type Culture Collection) גודלו בצלחות של 6 בארות במשך 48-72 שעות בתנאי תרבית סטנדרטיים (5% CO2, 37 מעלות צלזיוס, לחות), עם או בלי 50 מ"מ EtOH6, וקובעו במתנול קר כקרח כפי שתואר קודם לכן13. תאים קבועים הותקנו באמצעות מדיוםהרכבה 15 המכיל DAPI ויובשו בחושך לפני ההדמיה.
- הפעל את כל רכיבי המיקרוסקופ הקרינה והמחשב על פי נהלי הפעלה סטנדרטיים שנקבעו. פתח את התוכנה המשויכת למיקרוסקופ.
- הגדר את המיקרוסקופ כדי לדמיין דגימות דרך העינית. בדוק את המטרה. במידת הצורך, סובב את האף למטרה הרצויה. ודא שלתוכנה נבחרה המטרה הרצויה לאינדיקציה מדויקת של אזור התמונה. כאן, התמונות צולמו באמצעות מטרה של פי 10.
- כשהאורות כבויים, הניחו את הדגימה (שקופית, צלחת או צלחת) על הבמה. הפעל הדמיה חיה עם המסנן המתאים והגדר את זמן החשיפה למשך זמן קבוע מראש או לחשיפה אוטומטית. כאן נעשה שימוש במסנן DAPI וזמן חשיפה של 61-62 אלפיות השנייה. עיין בטבלת פתרון הבעיות לקבלת הנחיות להגדרת זמן חשיפה, במיוחד עבור תמונות עם צפיפות תאים גבוהה.
- בעזרת העינית כוונן לאט את המיקוד באמצעות כפתור הכוונון הגס עד שנראים גרעינים מוכתמים, ולאחר מכן השתמש בכפתור הכוונון העדין כדי לכוונן לאט את המיקוד עד שהגרעינים נראים ברורים וחדים.
הערה: דוגמאות לתמונות מתאימות מוצגות באיור 1.- אם מופיע כתמי רקע מוגזמים או אם הגרעינים נראים עמומים מאוד, התאם את זמן החשיפה באופן ידני. בתמונה בודדת, עשויים להיות הבדלים נראים לעין בעוצמת האות עבור גרעינים מסוימים; עם זאת, הצורה העגולה צריכה להיות גלויה עבור כל הגרעינים עם הפרעות אות מינימליות.
הערה: התוכנית המשמשת ביישומים במורד הזרם חזקה לעוצמות שונות. תמונות גולמיות המשמשות לפיתוח ואימות של תוכנית כימות הגרעינים כוללות תמונות עם מגוון עוצמות אותות וזמינות דרך מאגר מסגרת המדע הפתוח (ראה קישור שהוזכר לעיל).
- אם מופיע כתמי רקע מוגזמים או אם הגרעינים נראים עמומים מאוד, התאם את זמן החשיפה באופן ידני. בתמונה בודדת, עשויים להיות הבדלים נראים לעין בעוצמת האות עבור גרעינים מסוימים; עם זאת, הצורה העגולה צריכה להיות גלויה עבור כל הגרעינים עם הפרעות אות מינימליות.
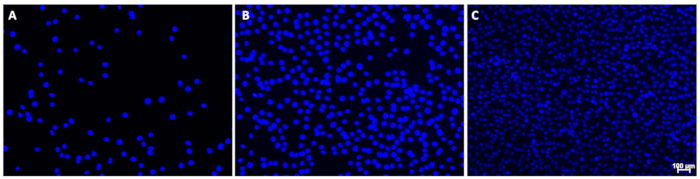
איור 1: תמונות לדוגמה של גרעינים מוכתמים ב-DAPI. תמונות אלה מתארות גרעינים בצפיפות תאים נמוכה (A), (B) בינונית או (C) גבוהה עם רקע מינימלי. שימו לב לעוצמת האות המשתנה של הגרעינים על פני תמונה C; זה לא הפריע לכימות. סרגל קנה המידה (C) מציין 100 מיקרומטר; יש להשמיט פסי קנה מידה מתמונות המשמשות לכימות גרעינים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
- לאחר שהגרעינים נמצאים בפוקוס דרך העינית, העבר את מצב הזיהוי למצלמה. כוונן את המיקוד באמצעות כפתור הכוונון העדין.
- התאם את זמן החשיפה במידת הצורך. שים לב לזמן החשיפה הסופי לשימוש עם דגימות ותמונות עוקבות.
הערה: למרות ששימוש באותו זמן חשיפה בכל התמונות הוא אידיאלי, וריאציות בזמן החשיפה מקובלות עבור יישום זה מכיוון שעוצמת הקרינה אינה מכומתת או מושווה בין דגימות. - צלם את התמונה באמצעות כפתור לכידת התמונה המתאים בתוכנה. אם הוחלו על דגימות מספר כתמים, החלף את המסנן (למשל, ל-FITC, TRITC או Cy5) וצלם את אותו אזור למטרות מיזוג.
- החל את התריס כדי למזער את חשיפת הדגימה לאור. מבלי לצרוב בסרגל קנה מידה, שמור את התמונה בפורמט קובץ איור אוניברסלי (.tif, .png או .jpg) בתיקייה ייעודית. ודא שכל שם קובץ תמונה הוא תיאורי כראוי או שלכל קידוד המשמש לשמות קבצים יש תיעוד מקושר מתאים.
הערה: זה יהפוך לספריית התמונות עבור תוכנית הכימות. אם נעשה שימוש בהדמיה רב-צבעונית, העתק רק תמונות של גרעינים לתיקייה ייעודית נפרדת. ניתן לעשות זאת גם בסוף מפגש ההדמיה. תמונות המיוצאות באמצעות הגדרות ייצוא המוגדרות כברירת מחדל ברוב תוכנות ההדמיה הן בדרך כלל ברזולוציה מספיקה לשימוש עם תוכנית זו, בתנאי שתמונות איכותיות נלכדו. עם זאת, יש לבדוק את התמונות לאחר הייצוא כדי לוודא שהמורפולוגיה של הגרעינים ברורה. אם התמונות נשמרות בפורמט .jpg, יש להקפיד שהתמונות לא יידחסו יתר על המידה ושהאיכות תישמר. - פתח את התריס, חזור להדמיה חיה וסרוק על פני הדגימה למיקום אחר לצורך הדמיה. חזור על שלבים 1.8-1.9 לפי הצורך. עבור תמונות של תאים שנזרעו בלוחות של 6 בארות שצולמו בהגדלה של פי 10, צלם 10-15 תמונות לכל באר (תלוי בעקביות צפיפות התאים על פני שטח הדגימה).
- לאחר רכישת כל התמונות עבור דגימה בודדת, בצע אחת מהאפשרויות הבאות.
- עבור לדוגמא הבאה וחזור על שלבים 1.4-1.10.
- השהה את הפרוטוקול וחזור במועד אחר. הדגימות צריכות להישאר מוגנות מפני אור. אפשרות זו אינה מומלצת אם עובדים עם תאים חיים.
- לאחר השלמת ההדמיה, ודא שכל הדגימות מוגנות מפני אור לפני הדלקת אורות החדר. אם התיקיה המכילה תמונות נשמרת באופן מקומי, העבר קבצים לכונן אצבע, למיקום אחסון בענן או לכונן רשת אחר הנגיש למחשב שבו תופעל תוכנית הכימות. שמור בתיקיה ייעודית.
- כבה את כל חלקי המיקרוסקופ והמחשב על פי נהלי הפעלה סטנדרטיים שנקבעו.
- ההדמיה הושלמה כעת. נתח את הקבצים באמצעות תוכנית הכימות האוטומטית כמתואר בסעיף 2 או, לחילופין, על ידי הפעלת התוכנית כסקריפט Python כמתואר בסעיף 3.
2. הרצת תוכנית כימות הגרעינים האוטומטית כקובץ הפעלה
הערה: שיטה זו, המפעילה את התוכנית כקובץ הפעלה, תואמת למחשבים מבוססי Windows והיא השיטה המומלצת למשתמשי מערכת ההפעלה Windows. Mac OS או משתמשים אחרים שאינם Windows צריכים להריץ את התוכנית כסקריפט Python (ראה סעיף 3).
- ודא ש-Python מותקן במחשב. במידת הצורך, התקן את Python מ-python.org/downloads (מועדף) או באמצעות Anaconda או Miniconda. נכון למועד הפרסום, תוכנית זו נבדקה עם גרסאות Python 3.10-3.12.
- פתח את הדפדפן ונווט אל https://github.com/rbudnar/nuclei_counter/releases. הורד את הגירסה העדכנית ביותר של הקובץ בשם count_nuclei.zip.
- מהתיקייה הורדות, לחץ לחיצה ימנית על קובץ ה-Zip וחלץ את כל הקבצים למיקום הרצוי במחשב המקומי (ראה איור 2א). השאר מיקום קובץ זה פתוח; נתיב קובץ זה ישמש בשלב 2.5 (ראה איור 2ב).
הערה: Count_nuclei אמור להופיע כעת כקובץ יישום. זהו קובץ ההפעלה. - פתח שורת פקודה על ידי חיפוש CMD או שורת פקודה בשורת החיפוש. השתמש בפקודה cd כדי לשנות את הספרייה ל file נתיב עבור קובץ ההפעלה file (ראה איור 2C) והקש Enter. שורת פקודה לדוגמה: C:\Users\janedoe>cd [רווח אחרי cd, ואז העתק/הדבק נתיב קובץ]
- בשורת הפקודה הבאה, הקלד את הטקסט הבא והקש Enter.
count_nuclei.exe --img_dir "נתיב/אל/תמונות" --output_dir "נתיב/אל/פלט" --save_images --output_count_filename "results.csv"
כאשר path/to/images הוא נתיב הקובץ לתיקייה המכילה תמונות לניתוח (ראה שלב 1.14); path/to/output הוא נתיב הקובץ לתיקייה שבה יש לשמור את קובץ .csv (ברירת המחדל היא ספריית העבודה הנוכחית כאשר אינה מסופקת); ו-results.csv הוא שם הקובץ שבו יישמרו הספירות המתקבלות (אם לא סופק, שם קובץ הפלט מוגדר כברירת מחדל ל-count_results.csv).
הערה: יש צורך במרכאות בשורת הפקודה. ודא שאין רווחים אחרי מרכאות לפני נתיב הקובץ (ראה איור 2C). - כאשר שורת הפקודה הבאה מופיעה, העיבוד הושלם. קווי המתאר והגיליון האלקטרוני של התוצאות זמינים כעת במיקום שצוין כספריית הפלט בשלב 2.5 (כלומר, נתיב/אל/פלט; ראה איור 2C).
- בדוק חזותית את קווי המתאר כדי לוודא את איכות הספירה. כעת ניתן להשתמש בתוצאות כדי לנרמל נתונים כמתואר בסעיף 4.
הערה: אם נראה שקווי המתאר החמיצו גרעינים (למשל, גרעינים נראים חלשים מדי) או אשכול קבוצות של גרעינים לפריט יחיד (למשל, תמונה עם חשיפת יתר, יותר מדי רקע, חפצים), על המשתמש לעיין בטבלת פתרון הבעיות וייתכן שיצטרך לחזור לסעיף 1 כדי לייעל את רכישת התמונה לפני המעבר לסעיף 4.
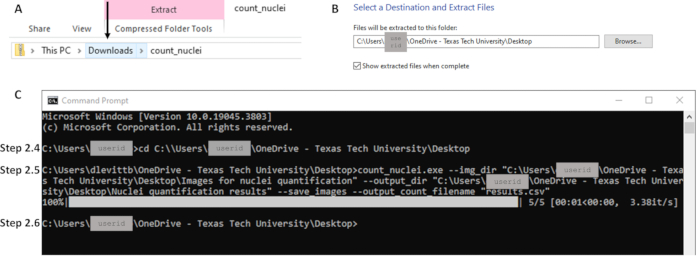
איור 2: דוגמאות לחלקים מרכזיים של זרימת העבודה עבור סעיף 2. (A) חלץ קבצים מתיקיית ההורדות, תוך הקפדה ללחוץ על התיקייה הנכונה כפי שמצוין על ידי החץ כדי לאפשר חילוץ (B) ליעד הרצוי בשלב 2.3. (ג) דוגמאות לפקודות עבור שלבים 2.4 ו-2.5 ושורת הפקודה הבאה המציינת השלמה בשלב 2.6. שים לב שנתיב הקובץ בשלב 2.4 (C) תואם למיקום החילוץ בשלב 2.3 (B). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
3. הרצת תוכנת כימות הגרעינים האוטומטית כסקריפט Python
- ודא ש-Python מותקן במחשב. במידת הצורך, התקן את Python מ-python.org/downloads (מועדף) או באמצעות Anaconda או Miniconda. נכון למועד הפרסום, תוכנית זו נבדקה עם גרסאות Python 3.10-3.12.
- פתח את הדפדפן ונווט אל https://github.com/rbudnar/nuclei_counter. לחץ על לחצן הקוד הירוק ולאחר מכן לחץ על הורד ZIP כדי להוריד את מאגר הקוד (ראה איור 3A).
- השתמש בהוראות הבאות עבור מערכת ההפעלה Windows. ראה שלב 3.4 עבור מחשבים מבוססי Mac OS.
- מהתיקייה הורדות, לחץ לחיצה ימנית על הקובץ ובחר חלץ הכל כדי לחלץ את הקבצים למחשב מקומי (ראה איור 3B-C).
- במיקום הקובץ שחולץ, פתח את התיקיה בשם nuclei_counter-main. זהו מאגר הקוד.
הערה: יש להוריד אפשרות זו רק בפעם הראשונה שבה נעשה שימוש בתוכנית זו; כל שעליך לעשות הוא להעביר את הקבצים שחולצו למיקום רשת נגיש ולרשום את המיקום הזה. - פתח שורת פקודה על ידי חיפוש CMD או שורת פקודה בשורת החיפוש. אם Python הותקן על ידי Anaconda או Miniconda, פתח את שורת Anaconda במקום שורת פקודה רגילה. ניתן למצוא זאת על ידי הקלדת Anaconda Prompt בשורת החיפוש.
- השתמש בפקודה cd כדי לשנות את הספרייה למאגר הקוד והקש Enter. ראה את שורת הקוד למטה, שהיא תלוית רישיות. ודא שהתיקייה בשם nuclei_counter-main פתוחה בעת העתקת נתיב הקובץ; הקובץ בשם pyproject או pyproject.toml אמור להיות גלוי (ראה איור 3D). שורת פקודה לדוגמה: C:\Users\janedoe>cd [רווח אחרי cd, ואז העתק/הדבק נתיב קובץ]
- בשורת הפקודה הבאה, הקלד את הפרטים הבאים, כולל הרווח והנקודה (ראה איור תלת-ממד:
pip install -e .
הערה: יש להשלים את שלבים 3.3.5 ו-3.3.6 רק בפעם הראשונה שתוכנית זו מופעלת במחשב כלשהו. אם זה כבר הושלם, המשך לשלב 3.3.7. תהליך ההתקנה עשוי להימשך מספר רגעים לאתחול. - הקש Enter. הפעלת פקודה זו מתקינה את הספריות הנדרשות להפעלת סקריפט זה ומתקינה את המאגר במצב ניתן לעריכה במקרה שיש צורך בהתאמות קוד (ראה איור 3D).
- הקלד את הטקסט הבא בשורת הפקודה הבאה כדי לשנות את הספרייה לספריית קוד המקור הראשית (ראה איור 3E):
תקליטור "nuclei_counter" - בשורת הפקודה הבאה, הקלד את הטקסט הבא והקש Enter (ראה איור 3E):
Python count_nuclei.py --img_dir "נתיב/אל/תמונות" --output_dir "נתיב/אל/פלט" --save_images --output_count_filename "results.csv"
כאשר path/to/images הוא נתיב הקובץ לתיקייה המכילה תמונות לניתוח (שלב 1.14); path/to/output הוא נתיב הקובץ לתיקייה שבה יש לשמור את קובץ .csv (ברירת המחדל היא ספריית העבודה הנוכחית כאשר אינה מסופקת); ו-results.csv הוא שם הקובץ שבו יישמרו הספירות המתקבלות (אם לא סופק, שם קובץ הפלט מוגדר כברירת מחדל ל-count_results.csv).
הערה: יש צורך במרכאות בשורת הפקודה. ודא שאין רווחים אחרי מרכאות לפני נתיב הקובץ. סקריפט ה-python יקרא את הקבצים בנתיב הקובץ שסופק ויפעיל את תוכנית ספירת הגרעינים עבור כל התמונות בספרייה (ראה איור 3E). הספירות המתקבלות יישמרו בקובץ .csv התוצאות במיקום שצוין. - כאשר שורת הפקודה הבאה מופיעה, העיבוד הושלם (ראה איור 3E). קווי המתאר והגיליון האלקטרוני של התוצאות זמינים כעת במיקום שצוין כספריית הפלט בשלב 3.3.8 (כלומר, נתיב/אל/פלט).
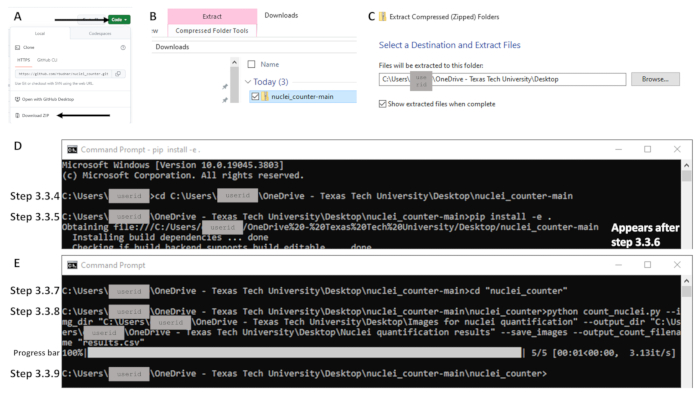
איור 3: דוגמאות לחלקים עיקריים של זרימת העבודה עבור סעיף 3 באמצעות מערכת ההפעלה Windows. כדי לגשת לקוד, לחץ על הלחצן הירוק שכותרתו קוד ו- (A) הורד את קובץ ה- Zip (מסומן בחצים). (B) חלץ על ידי לחיצה ימנית על קובץ ה-Zip (מוצג בכחול) ו-(C) הגדרת היעד הרצוי. (ד) לאחר פתיחת שורת הפקודה, דוגמאות לפקודות עבור שלבים 3.3.4 ו-3.3.5. שים לב שהפקודה עבור שלב 3.3.5 נמצאת כולה בשורה אחת; הטקסט המתחיל "השגת קובץ" בשורה הבאה מופיע לאחר לחיצה על Enter (שלב 3.3.6; כלומר, הפעלת הפקודה). (ה) דוגמאות לפקודות עבור שלבים 3.3.7 ו-3.3.8, מד התקדמות לאחר ביצוע שלב 3.3.8 ושורת הפקודה המציגה השלמה בשלב 3.3.9. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
- השתמש בהוראות הבאות עבור Mac OS.
- מהתיקייה הורדות, לחץ על התפריט קובץ ובחר פתח כדי לחלץ את הקבצים למחשב המקומי (ראה איור 4א).
- במיקום הקובץ שחולץ, פתח את התיקיה בשם nuclei_counter-main. זהו מאגר הקוד.
הערה: יש להוריד אפשרות זו רק בפעם הראשונה שבה נעשה שימוש בתוכנית זו; כל שעליך לעשות הוא להעביר את הקבצים שחולצו למיקום רשת נגיש ולרשום את המיקום הזה. - פתח מסוף על ידי לחיצה על command+מקש רווח הפותח זרקור; לאחר מכן, חפש "טרמינל".
- השתמש בפקודה cd כדי לשנות את הספרייה למאגר הקוד והקש Enter. ראה את שורת הקוד למטה, שהיא תלוית רישיות. הקלד את נתיב הקובץ במסמך טקסט והעתק אותו למסוף. ודא ש-file נתיב מתחיל בטקסט שלפני התקליטור (למשל, /Users/userid באיור 4B). שורת פקודה לדוגמה: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- בשורת הפקודה הבאה, יופיע סימן דולר אחרי מזהה המשתמש (ראה איור 4B). ודא שיש רווח אחרי סימן הדולר והקלד את הפרטים הבאים, כולל הרווח והנקודה בסוף הפקודה. כלול את גרסת Python המתאימה מיד לאחר pip ללא רווח: pip3 install -e .
הערה: יש להשלים את שלבים 3.4.5 ו-3.4.6 רק בפעם הראשונה שתוכנית זו מופעלת במחשב כלשהו. אם זה כבר הושלם, המשך לשלב 3.4.7. תהליך ההתקנה עשוי להימשך מספר רגעים לאתחול. - הקש Enter. הפעלת פקודה זו מתקינה ספריות נדרשות להפעלת סקריפט זה ומתקינה את המאגר במצב ניתן לעריכה במקרה שיש צורך בהתאמות קוד.
- בשורת הפקודה הבאה, ודא שיש רווח אחרי סימן הדולר ולאחר מכן הקלד את הטקסט הבא כדי לשנות את הספרייה לספריית קוד המקור הראשית (ראה איור 4C):
תקליטור "nuclei_counter" - בשורת הפקודה הבאה, הקלד את שורת הפקודה שלהלן. החלף את המספר אחרי Python במספר הגרסה הנכון אם אתה משתמש בגרסה שאינה 3. לאחר מכן, הקש Enter (ראה איור 4C):
python3 count_nuclei.py --img_dir "נתיב/אל/תמונות" --output_dir "נתיב/אל/פלט" --save_images --output_count_filename "results.csv"
כאשר path/to/images הוא נתיב הקובץ לתיקייה המכילה תמונות לניתוח (שלב 1.14); path/to/output הוא נתיב הקובץ לתיקייה שבה יש לשמור את קובץ .csv (ברירת המחדל היא ספריית העבודה הנוכחית כאשר אינה מסופקת); ו-results.csv הוא שם הקובץ שבו יישמרו הספירות המתקבלות (אם לא סופק, שם קובץ הפלט מוגדר כברירת מחדל ל-count_results.csv).
הערה: יש צורך במרכאות בשורת הפקודה. ודא שאין רווחים אחרי מרכאות לפני נתיב הקובץ. סקריפט Python יקרא את הקבצים בנתיב הקובץ שסופק ויפעיל את תוכנית ספירת הגרעינים עבור כל התמונות בספרייה (ראה איור 4C). הספירות המתקבלות יישמרו בקובץ התוצאות .csv במיקום שצוין. - כאשר שורת הפקודה הבאה מופיעה, העיבוד הושלם (ראה איור 4C). קווי המתאר והגיליון האלקטרוני של התוצאות זמינים כעת במיקום שצוין כספריית הפלט בשלב 3.4.8 (כלומר, נתיב/אל/פלט).
- בדוק חזותית את קווי המתאר כדי לוודא את איכות הספירה. כעת ניתן להשתמש בתוצאות כדי לנרמל נתונים כמתואר בסעיף 4.
הערה: אם נראה שקווי המתאר החמיצו גרעינים (למשל, גרעינים נראים חלשים מדי) או קבוצות של גרעינים לפריט יחיד (למשל, תמונה עם חשיפת יתר, יותר מדי רקע, חפצים), המשתמש צריך לעיין בטבלה 1 וייתכן שיצטרך לחזור לסעיף 1 כדי לייעל את רכישת התמונה לפני המעבר לסעיף 4.
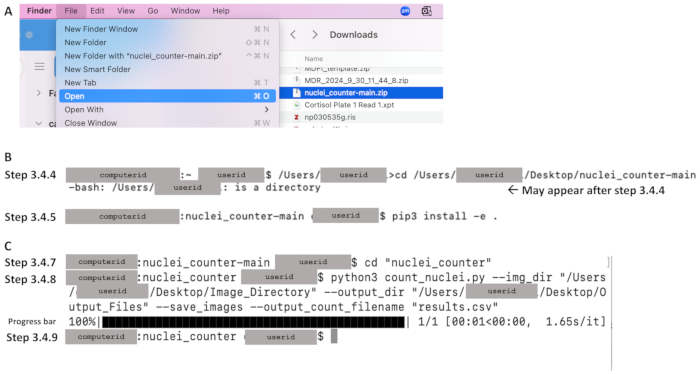
איור 4: דוגמאות לחלקים מרכזיים של תהליך העבודה עבור סעיף 3 באמצעות Mac OS. לאחר הורדת הקוד (ראה איור 3A), (A) חלץ על ידי לחיצה על ה-Zip file (מוצג בכחול) והגדרת היעד הרצוי. (ב) לאחר פתיחת הטרמינל, דוגמאות לפקודות עבור שלבים 3.4.4 (שינוי הספרייה למאגר הקוד) ו-3.4.5 (התקנת pip). (ג) דוגמאות לפקודות עבור שלבים 3.4.7 ו- 3.4.8, מד התקדמות לאחר ביצוע שלב 3.4.8 ושורת הפקודה המציגה השלמה בשלב 3.4.9. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
4. שימוש בספירות כדי לנרמל נתוני ניסוי
- פתח את קובץ results.csv המקשר כל תמונה עם כימות גרעינים שנקבע באמצעות התוכנית האוטומטית.
- החליטו על שיטת הנורמליזציה: 1) בסיס תמונה אחר תמונה (למשל, אם מיקרוסקופיה פלואורסצנטית רב-צבעונית), 2) נרמל נתונים כיחס בין ספירת הגרעינים הממוצעת לדגימה לעומת דגימת ייחוס (או תנאי), 3) לנרמל לספירת גרעינים לדגימה (למשל, אקסטרפולציה של ספירת הגרעינים הכוללת לשטח הדגימה). המלצות לשיטת הנורמליזציה נמצאות בסעיף דיון.
- אם הנורמליזציה תבוצע על בסיס תמונה אחר תמונה, פשוט חלקו את נתוני הניסוי עבור כל תמונה במספר הגרעינים של אותה תמונה.
- כדי לנרמל נתונים כיחס, בצע את השלבים המתוארים להלן.
- עבור כל דגימות שבהן נאספו תמונות מרובות עבור שכפול יחיד (למשל, 10-15 תמונות שצולמו מבאר אחת בצלחת של 6 בארות), חשב את המספר הממוצע של גרעינים לתמונה. ספירת גרעינים זו נחשבת לספירת התאים בהנחה של תאים חד-גרעיניים או מספר תאים שהתמזגו יחד בתאים מרובי גרעינים (כגון עם שרירי שריר).
- בחר מדגם ייחוס או תנאי ייחוס (למשל, ספירה ממוצעת בכל הדגימות מתנאי הבקרה) ורשום את הספירה.
- חלקו את הספירה של כל הדגימות בספירת הייחוס הזו, וכתוצאה מכך נוצר ערך נורמליזציה. אם נעשה שימוש בדגימת ייחוס, השתמש בערך הנורמליזציה עבור מדגם הייחוס כ-1.
הערה: אם נעשה שימוש בתנאי ייחוס, ערך הנורמליזציה הממוצע בתנאי יהיה כעת 1. לדגימות עם פחות גרעינים מההתייחסות (כלומר, פחות צפופות) יהיו ערכי נורמליזציה קטנים מ-1, ולדגימות עם יותר גרעינים מההתייחסות (כלומר, צפופות יותר) יהיו ערכי נורמליזציה גדולים מ-1. - חלקו את נתוני הניסוי עבור כל באר בערך הנורמליזציה המחושב המתאים.
- כדי לנרמל נתונים לספירת גרעינים לכל דגימה, בצע את השלבים המתוארים להלן.
- עבור כל דגימות שבהן נאספו תמונות מרובות עבור שכפול יחיד (למשל, 10-15 תמונות שצולמו מבאר אחת בצלחת של 6 בארות), חשב את המספר הממוצע של גרעינים לתמונה. ספירת גרעינים זו נחשבת לספירת התאים בהנחה של תאים חד-גרעיניים, או מספר התאים שהתמזגו יחד בתאים מרובי גרעינים (כגון עם צינורות).
- קבע את שטח הפנים של המגלשה (כיסוי), הבאר או הצלחת שממנה נגזרו הנתונים. מידע זה ניתן למצוא בסעיף המפרטים באתרי המוצרים.
- קבע את השטח של כל תמונה שצולמה. מידע זה זמין לעתים קרובות כמטא-נתונים בתוכנת ההדמיה או הניתוח.
- ודא ששטח הפנים של המדגם ושטח התמונה באים לידי ביטוי באותן יחידות (למשל, מ"מ2). אם לא, המר ליחידה הרצויה.
- חשב את הספירה הכוללת עבור כל דגימה באמצעות המשוואה הבאה:
TC = AC • (SA/IA)
כאשר TC הוא הספירה הכוללת, AC הוא הספירה הממוצעת, SA הוא שטח הפנים, IA הוא שטח התמונה. - חלקו את נתוני הניסוי ב-TC המתאים עבור המדגם.
- לנרמל נתוני ניסוי ולהשתמש בניתוחים סטטיסטיים לפי הצורך.
תוצאות
כל ריצת תמונת אצווה מייצרת: 1) קבוצה של קבצי תמונה עם קווי מתאר מוחלים המציגים את קווי המתאר של גרעינים מזוהים (איור 5), ו-2) קובץ .csv (גיליון אלקטרוני) המקשר בין שמות קבצי תמונה והספירות המשויכות. צפייה בקווי המתאר תאפשר למשתמש להעריך חזותית את איכות הספירה. באופן ספציפי, תמונות המתקבלות על פי סעיף 1 צריכות להכיל את כל הגרעינים (או כמעט את כולם) מוקפים בקו ירוק מוצק המציין שהגרעין נספר על ידי התוכנית. קווי המתאר האלה שימשו לכוונון התוכנית כאן.
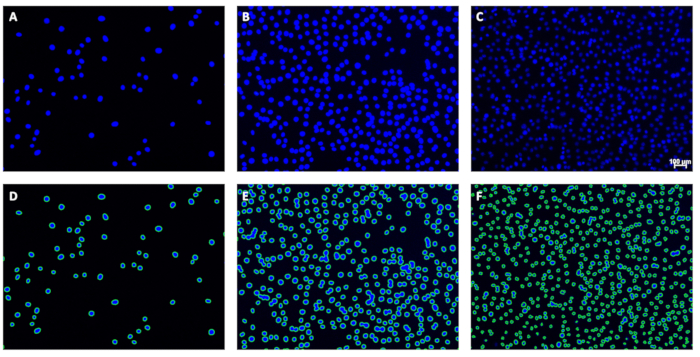
איור 5: דוגמאות של תמונות מקוריות וקווי מתאר תואמים. (א-ג) דוגמאות לתמונות מקוריות לפני הניתוח וקווי מתאר תואמים (D-F) לאחר הניתוח ישירות למטה. סרגל קנה המידה (C) מציין 100 מיקרומטר; יש להשמיט פסי קנה מידה מתמונות המשמשות לכימות גרעינים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
ולידציה באמצעות תוצאות ראשוניות
מיובלסטים C2C12 בצפיפות תאים משתנה היו מקובעים במתנול, מוכתמים ב-DAPI במדיית הרכבה, ו-120 תמונות התקבלו בהגדלה של פי 10 בפורמט קובץ .jpg. התוכנית האוטומטית הופעלה כאשר ספריית התמונות מוגדרת לתיקיה המכילה תמונות אלה, וקובץ .csv שנוצר נשמר. בנפרד, כל תמונה כומתה ידנית על ידי שני חוקרים מיומנים שהיו עיוורים לספירות האוטומטיות ולספירות זה של זה. תמונות גולמיות, קווי מתאר שנוצרו על ידי התוכנית האוטומטית וגיליון אלקטרוני של תוצאות הכולל ספירה ידנית ואוטומטית זמינים במאגר Open Science Framework (OSF) (ראה קישור בסעיף 1). מקדמי מתאם תוך-מחלקתיים (ICCs; מדדים ממוצעים) חושבו באמצעות סטטיסטיקת SPSS כדי לקבוע את מהימנות המעריכים בין 1) שתי הספירות הידניות ו-2) הממוצע של שתי הספירות והספירות הידניות שהתקבלו באמצעות התוכנית האוטומטית. גישות דומות שימשו לאימות תוכניות אחרות שנועדו לכמת אובייקטים בתמונות שהתקבלו באמצעות מיקרוסקופ פלואורסצנטי 16,17,18. ICCs חושבו גם בין ספירות ידניות ממוצעות וספירות אוטומטיות עבור כל רבעון כדי להבטיח שהערכת מערך הנתונים כולו לא תסוות שינויים פוטנציאליים באמינות על סמך צפיפות התאים. הרבעים הוגדרו באמצעות נקודות החיתוךשל האחוזון ה-25,ה-50 וה-75 בהתבסס על ספירה ידנית ממוצעת, ונעים בין רבעון 1 (Q1; צפיפות התאים הנמוכה ביותר) ל-Q4 (צפיפות התאים הגבוהה ביותר). ICCs מדווחים על סמך מדדים ממוצעים ומפורשים על סמך ההגדרות הבאות: גרוע (ICC <0.50), בינוני (0.50 ≤ICC <0.75), טוב (0.75 ≤ICC <0.90) או מצוין (ICC ≥0.90)19.
הגרעינים לתמונה נעו בין 44.5 ל-1160 בהתבסס על ממוצעי ספירה ידנית. מהימנות הבין-מעריכים הייתה מצוינת בין שתי הספירות הידניות (ICC > 0.999, p < 0.0001; איור 6A) ובין ספירות ידניות ואוטומטיות ממוצעות (ICC = 0.993, p < 0.0001; איור 6B). האמינות נותרה מצוינת בכל הרבעים (Q1: ICC = 0.996, p < 0.0001; Q2: ICC= 0.997, עמ' < 0.0001; Q3: ICC = 0.998, עמ' < 0.0001; Q4: ICC = 0.986, p < 0.0001; איור 6C-F). נקודת הנתונים היחידה הרחוקה מקו ההתאמה הטוב ביותר באיור 6B מייצגת את התמונה המוצגת באיור 7. בתמונה יש כמה אזורים של מספר גרעינים מקובצים יחד, מה שמונע מתוכנת הראייה הממוחשבת לזהות כל גרעין. ניתן לתקן בעיה זו באמצעות זמן חשיפה קצר יותר. טבלה 1 מתארת כיצד לטפל בתמונה כזו.
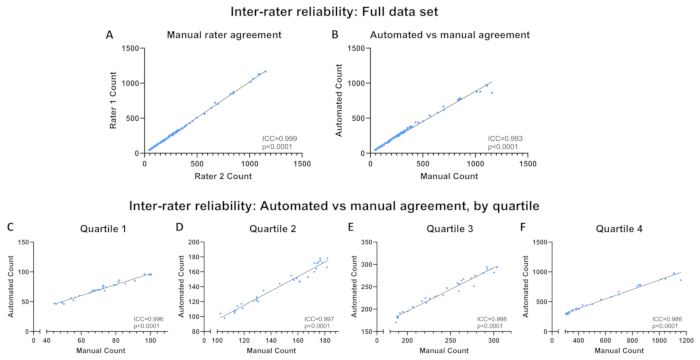
איור 6: אמינות בין מדרגים. אמינות בין מעריכים על פני מערך הנתונים המלא (A) בין ספירות מדרגים ידניות ו-(B) בין ספירות אוטומטיות לספירה ידנית ממוצעת. (ג-ו) אמינות בין מעריכים בין ספירה אוטומטית לספירה ידנית ממוצעת לפי רבעון. מהימנות מבוטאת כמקדם מתאם תוך-מחלקה (ICC). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 7: דוגמה לתמונה עם שני מחסומים לכימות מדויק של גרעינים. (A) התמונה המקורית ו-(B) קווי המתאר המשויכים. חץ 1 מציין אזור עם מספר גרעינים מקובצים יחד, ומעוות את הצורה הגרעינית העגולה מספיק כדי לעכב זיהוי תוכנה של גרעינים מרובים. זה קורה בכמה מקומות לאורך התמונה הזו. חץ 2 מציין הילה דהויה המופיעה מאחורי גרעינים מרובים; הילה זו נראתה בהירה מספיק כדי שהתוכנה תכלול אותה כגרעין, ובכך תאבד את ספירת הגרעינים המרובים שהיא מקיפה. תמונה זו מייצגת את נקודת הנתונים היחידה הרחוקה מהקו המתאים ביותר באיור 6B, תוך הדגשת הבעיות הפוטנציאליות עם חסמים אלה. סרגל קנה המידה (A) מציין 100 מיקרומטר; יש להשמיט פסי קנה מידה מתמונות המשמשות לכימות גרעינים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Discussion
לתוכנית כימות הגרעינים שלנו יש מספר יתרונות על פני האפשרויות הקיימות: היא דורשת מיומנויות טכנולוגיות מינימליות בלבד, מאומתת למשימה הספציפית של כימות גרעינים והיא קוד פתוח; האחרון מתגבר על חסמים הקשורים לעלות. בסופו של דבר, תוכנית זו מספקת לביולוגים תאיים ומולקולריים אפשרות נוספת לכמת במהירות ובדייקנות גרעינים בתמונות שצולמו באמצעות מיקרוסקופיה פלואורסצנטית. תוכניות ספירת גרעינים או תאים אוטומטיות הזמינות כיום אינן מועדפות על ידי כל המשתמשים, חלקן יקרות, והאימות עשוי להיות מינימלי או נעדר עבור יישומים ספציפיים. ציוד מיוחד, כגון קו BioTek Cytation, מלווה בעלונים טכניים המאמתים ספירת תאים באתרם באמצעות צביעה גרעינית20 בנוסף למגוון רחב של יישומים נוספים. בעוד שקיימים נתוני אימות וקו המוצרים של Cytation נמצא בשימוש נרחב, במיוחד כאשר הוא מתממשק עם מנתחי שטף חוץ-תאיים של סוסון ים כדי לנרמל נתוני תאים חיים בקלות וביעילות, מוצר זה יכול להיות חסכוני עבור מעבדות עם מימון ציוד מוגבל זמין. לדוגמה, מחיר המחירון של דגם הבסיס המצויד בארבע מטרות וארבע קוביות פילטר למיקרוסקופיה פלואורסצנטית עלה על 100,000 דולר אמריקאי בשנה הקלנדרית 2024. למי שאין לו אמצעים לרכוש ציוד מיוחד, תוכנת קוד פתוח מספקת חלופות ללא עלות לניתוח תמונות. תוכנת קוד פתוח שימושית לכימות גרעינים ותאים כוללת את ImageJ (המכונים הלאומיים לבריאות, בת'סדה, מרילנד, ארה"ב; זמין ב-21. תוספים זמינים כעת למי שרוצה לכתוב סקריפטים של מאקרו לאוטומציה של עיבוד תמונה באמצעות פלטפורמת ImageJ. אוטומציה של ספירת תאים מבוססת ImageJ באמצעות ניגודיות פאזה או מיקרוסקופ שדה בהיר עם תמונות תא שלם תוארה בעבר22,23. CellProfiler הוא יישום שפורסם בהרחבה לניתוח תמונות תאים מוכתמות בצבעים פלואורסצנטיים או כתמים היסטולוגיים סטנדרטיים, ומאמר מוקדם המתאר את התועלת שלו במגוון יישומים ביולוגיים מצוטט לעתים קרובות כאימות24. עם זאת, כתב יד זה אינו מאמת שום ממצא ביולוגי מסוים, מה שמחייב אימות ושקיפות נוספים עבור צינורות אוטומציה שונים המשמשים, כגון עבור ספירת גרעינים25. יתר על כן, בעוד ש-CellProfiler היא חבילת תוכנה יעילה לניתוח תאים, לא כל המדענים מוצאים אותה ידידותית למשתמש. לכן, שאפנו לפתח ולתקף תוכנה אוטומטית חלופית לכימות גרעינים באמצעות תוכנת קוד פתוח. באופן ספציפי, השתמשנו בספריות ראייה ממוחשבת קיימות ב-Python כדי לפתח את זרימת העבודה הזו.
שיטה זו מתחילה בתאים או רקמות שמשתמש הקצה הכתים באמצעות כתמי DNA כגון DAPI26. ניתנות הנחיות כלליות לצילום ושמירת תמונות באופן התואם לתוכנת אוטומציה. למרות שהפרטים עשויים להיות שונים עם מיקרוסקופים ותוכנות הדמיה שונות, זרימת העבודה הכללית עבור סעיף 1 זהה להשגת תמונות הניתנות לכימות. אם משתמשים בדגימות חיות, יש להקפיד על לבישת ציוד מגן אישי מתאים וסביבת ההדמיה מתאימה לדגימות אלה (למשל, טמפרטורה, אחוזCO2, תוספת מאגר וכו')27. בעוד שפרוטוקול זה מתאר הדמיה של צבעי ה-DNA בלבד, משתמשים יכולים ללכוד צבעים אחרים של אותו אזור למטרות מיזוג15, ופשוט להעתיק את התמונות הגרעיניות לתיקייה נפרדת לשימוש עם תוכנית הכמות. יש לבצע הדמיה בחדר חשוך עם PPE מתאים לדגימות חיות ולהשתמש בחיטוי מתאים לפי הצורך (למשל, עם 70% אתנול). יש להגן על דגימות מוכתמות מפני אור לפני הדלקת האורות. לכידת תמונה איכותית היא קריטית לכימות מדויק של גרעינים. אפילו בתמונות עם זמן חשיפה מתאים וניגודיות חזקה בין גרעינים בהירים לרקע שחור, נוכחותם של תאים או הילות גושים מדי מאחורי קבוצות תאים (ראה איור 7) עשויה להפחית את דיוק הכימות. במקרים אלה, ייתכן שיהיה צורך לשנות את זמני החשיפה כדי להפחית את עוצמת האותות, להבטיח מיזוג מינימלי של אותות גרעינים ומזעור הילות תוך שמירה על אות חזק מספיק כדי לזהות את הצורות העגולות של הגרעינים אם שלבים אלה אינם מספיקים, ייתכן שיהיה צורך בהדמיה של קטע סמוך של הדגימה. יש לצלם את כל התמונות לניסוי בודד באותה הגדלה, וההגדלה המתאימה למטרה בה נעשה שימוש חייבת להיות מוגדרת כראוי בתוכנת ההדמיה. זה קריטי במיוחד שהמטרה המשמשת להדמיה תתאים להגדרות בתוכנת ההדמיה כאשר ספירת הגרעינים תועבר אקסטרפולציה מהאזור המצולם לכל אזור הבאר (או המגלשה). שלב זה צריך להתרחש בכל פעם שהמטרה מתחלפת, אך קל להתעלם ממנו בעת הגדרת המיקרוסקופ והתוכנה. הזמן הנדרש לסעיף 1 משתנה בהתאם למספר גורמים (למשל, חווית משתמש במיקרוסקופיה פלואורסצנטית, מספר תמונות ודגימות, מיקרוסקופ וכו'). כדי לצלם 10-15 תמונות של גרעינים מוכתמים לבאר בשתי צלחות של 6 בארות, על המשתמש לשמור כ-1-2 שעות זמן.
לאחר שהתמונות נלכדו ונשמרו בתיקייה ייעודית, הן מוכנות לכימות. השיטה הפשוטה ביותר לכימות היא הפעלת תוכנית אוטומטית ארוזה מראש זו ללא צורך בהתקנת Python או ספריות כלשהן (סעיף 2). יש לציין כי התוכנית מוכנה כקובץ הפעלה ונארזה באמצעות Pyinstaller. תוכנה שנבנתה באמצעות Pyinstaller מסומנת בדרך כלל על ידי תוכנת אנטי-וירוס. אם תוכנת אנטי-וירוס חוסמת את פתיחת הקובץ, על המשתמש לסמן את הקובץ כבטוח להמשיך. זה קריטי כדי לבצע בהצלחה את סעיף 2. לחלופין, כל משתמש יכול לבחור להריץ את תוכנית הכימות האוטומטית כסקריפט Python (סעיף 3) ולא כקובץ הפעלה, אם תרצה. אם המשתמש מעוניין להפעיל את התוכנית באמצעות מערכת הפעלה Mac או Linux, יש להפעיל את התוכנית דרך Python. שיטה זו אינה דורשת Pyinstaller אך עשויה להיות מאתגרת יותר מבחינה טכנית. הפרוטוקול שלב אחר שלב יחד עם דוגמאות, המפורטות בסעיף 3, נועד להסיר את המחסומים הטכניים להפעלת תוכנית זו כסקריפט. בין אם סעיף 2 או סעיף 3 משמשים להפעלת התוכנית, חשוב לוודא שהקוד מוזן כהלכה לשורות הפקודה. שורות קוד לדוגמה כלולות לאורך הפרוטוקול, והמשתמש צריך לשים לב היטב למרווחים, מרכאות וסימני פיסוק אחרים. על המשתמש גם לוודא שנתיבי הקבצים מדויקים. זה מועיל שנתיבי הקבצים עבור התמונות והפלט יהיו זמינים במסמך Word. ניתן גם להקליד פקודות במסמך Word זה ולהעתיק/להדביק לפי הצורך.
כפי שמתואר בסעיף 4, ישנן גישות מרובות לנרמל נתונים ניסיוניים באמצעות ספירות המתקבלות מתוכנית כימות גרעינים אוטומטית זו. עבור ניסויים מבוססי תמונה שבהם נתוני הניסוי כוללים מספר אירועים בכל תמונה ורצוי פרופורציה (למשל, שיעור התאים החיוביים ל-V28, שיעור התאים המבטאים קולטני ACE229, מספר התאים הקולטים חלקיקים מוכתמים30 וכו'), ניתן פשוט לחלק את נתוני הניסוי עבור כל תמונה בספירה המתקבלת (וכתוצאה מכך פרופורציה מ-0 ל-1). לאחר מכן, ניתן לחשב את הפרופורציה הממוצעת על פני תמונות משוכפלות מאותה שקופית, צלחת או באר. אם רוצים אחוזים, פשוט הכפילו את הפרופורציות הסופיות ב-100. לבסוף, ניתן להשתמש בפרופורציות או באחוזים ממוצעים אלה בניתוחים סטטיסטיים במורד הזרם. כאשר נתוני הניסוי שיש לנרמל הם מניסויים פונקציונליים בתאים חיים (למשל, שיעורי צריכת חמצן 6,11,12) או מסופרנטנט תרבית תאים (למשל, פעילות לקטט דהידרוגנאז בסופרנטנט31 או ריכוז של ציטוקין מופרש ספציפי32 וכו'), תחילה יש לחשב ממוצע של ספירת הגרעינים על פני כל התמונות שצולמו מאותה באר. לאחר מכן, ניתן להעריך את ספירת הגרעינים לשטח הפנים של השקופית (כיסוי), באר או צלחת, או שניתן לבטא את הספירה כיחס לעומת דגימת ייחוס או תנאי. אקסטרפולציה לשטח הפנים של השקופית, הבאר או הצלחת שימושית לביטוי נתוני ניסוי לעומת מספר התאים (אם משתמשים בתאים חד-גרעיניים) שמהם נגזרו הנתונים (למשל, חמצן נצרך לדקה לכל 103 תאים). כדי להשתמש בשיטה זו, יש לדעת את שטח הפנים של השקופית (כיסוי), הבאר או הצלחת ואת השטח שצולם בכל תמונה. אם הדגימה אינה מכסה את כל שטח הפנים הזמין, נתוני הניסוי הם מכמות קטנה של סופרנטנט (כלומר, מבוטא כפעילות או ריכוז ליחידת נפח), או שהתמונה ו/או שטח הפנים אינם ידועים, אז האפשרות הטובה ביותר היא ממוצע ספירת הגרעינים על פני כל התמונות עבור דגימה בודדת ולבטא את ספירת הגרעינים ביחס לדגימת ייחוס או מצב. הערך המתקבל משמש לתיקון נתוני ניסוי עבור צפיפות גרעינים או צפיפות תאים (אם משתמשים בתאים חד-גרעיניים).
למרות ששיטה זו פשוטה לביצוע, ישנן מספר בעיות פוטנציאליות שמשתמשים צריכים להיות מודעים להן. בעיות פוטנציאליות אלה, יחד עם סיבות אפשריות ושלבי פתרון בעיות לפתרון הבעיות, מפורטות בטבלה 1. לתוכנית המתוארת כאן יתרונות מרכזיים בהשוואה לתוכנה קיימת; עם זאת, יש להכיר במספר מגבלות. החיסרון העיקרי הוא שניתן להשתמש בתוכנית הכימות רק עם תמונות שצולמו באמצעות מיקרוסקופ פלואורסצנטי, ולא תמונות שדה בהיר. בעוד שתמונות מוכתמות ב-DAPI שצולמו בהגדלה של פי 10 שימשו לבניית התוכנית, ניתן להשתמש בתוכנה עם צבעי DNA והגדלות אחרות מכיוון שהתוכנה בנויה לזהות את המורפולוגיה של הגרעינים ולא את הצבע או הגודל. בעוד שאימתנו את דיוק התוכנית בתמונות המכילות מגוון צפיפויות תאים, למשתמש הקצה עשויה להיות צפיפות תאים מחוץ לטווח. במקרה זה, אנו עדיין מצפים לספירה מדויקת בתנאי שמתקבלות תמונות איכותיות; עם זאת, על המשתמש לבדוק היטב את קווי המתאר והפלט כדי להבטיח דיוק. עבור יישומי תרבית תאים, כימות גרעינים בתמונות הוא המתאים ביותר לתאים בשכבה אחת. אם תאים מתורבתים חופפים, הגרעינים עשויים להיות מוסווים, וכתוצאה מכך הערכת חסר של כמות הגרעינים. האופי הדו-ממדי של כימות גרעינים בתמונות מונע את השימוש בתוכנית זו לקביעת המספר הכולל של גרעינים מתאים בתרחיף, גושי רקמות עבות או יישומים תלת מימדיים אחרים. עם זאת, תוכנית זו עדיין יכולה לכמת גרעינים המסומנים בצבעי DNA פלואורסצנטיים בתמונות דו-ממדיות שצולמו ממודלים תלת מימדיים כגון גושי רקמות עבות. התועלת של כימות גרעינים בתמונות עבור היישום הרצוי צריכה להיקבע על ידי משתמש הקצה.
| בעיה | סיבות אפשריות | פתרונות אפשריים |
| רקע מוגזם בתמונות | זמן חשיפה גבוה מדי | צמצם את זמן החשיפה |
| נראה כי גרעינים מתמזגים זה עם זה בתמונות | ריכוז צבע DNA פלואורסצנטי גבוה מדי או יותר מדי מדיה להרכבת צבע DNA | הפחת את ריכוז הצבע במהלך האופטימיזציה; השתמש בכמות הקטנה ביותר האפשרית של מדיית הרכבה |
| גרעינים נראים קלים מדי או שחלק מהגרעינים אינם נראים | זמן החשיפה נמוך מדי | הגדלת זמן החשיפה |
| פוטוהלבנה | מנע פוטו-הלבנה על ידי הבטחת שדגימות מוכתמות יישארו מאוחסנות בחושך ועל ידי מזעור החשיפה של הדגימות לתאורה (למשל, על ידי שימוש בזמן החשיפה המינימלי הדרוש ועל ידי כיבוי התאורה כאשר אין בה צורך, כגון לאחר צילום תמונה או תוך כדי רישום הערות) | |
| אפקטי הילה או צורות שאינן ספציפיות לגרעין מופיעות | זמן חשיפה גבוה מדי | צמצם את זמן החשיפה |
| מוך ממגבי משימות עדינים או כמה מגיני ספסל בתאים חיים או נצמדים לתאים קבועים | הקפידו על סביבה נטולת מוך: צמצמו את השימוש במגבי משימות עדינים כאשר הדגימות נחשפות (למשל, בארון בטיחות ביולוגית לתאים חיים, במהלך קיבוע וייבוש של תאים קבועים, או במהלך תהליך הצביעה); ודא שמגני השולחן אינם משאירים מוך על שקופיות או צלחות במהלך הצביעה | |
| חריגות אחרות על פני השטח של תאים/רקמות קבועות | טפל בדגימות בזהירות רבה ופעל לפי אמצעי זהירות נאותים ושיטות עבודה מומלצות במהלך תיקון וצביעה | |
| הפרעות בנתיב האור | ודא שכל חלקי המיקרוסקופ נקיים וללא מוך ואבק; נקו את החלק החיצוני של שקופיות/צלחות/כלים עם 70% אתנול כדי להסיר אבק סביבתי ומוך לפני ההדמיה | |
| קובץ ההפעלה לא ייפתח | תוכנת אנטי-וירוס חסמה את הקובץ | סימון הקובץ בדגל כבטוח |
| שגיאות בהנחיות פקודה | בעיות בריווח בשורת הפקודה | בדוק שוב שתחילת הפקודה מוקלדת מיד לאחר הסמל ">" בשורת הפקודה ללא רווח, שאין רווחים לאחר המרכאות הפתוחות עבור נתיבי קבצים, ושהמרווח ותואם את הקוד שסופק ואת הדוגמאות. |
| מקרים לא נאותים המשמשים בשורת הפקודה | הקוד רגיש לאותיות גדולות וקטנות; הקפד להשתמש באותיות רישיות וקטנות לפי הצורך | |
| בעיות בנתיב קובץ | ודא שנתיב הקובץ מציין את החלק הפנימי של תיקיית הקבצים המשויכת (כלומר, תיקיית הקבצים פתוחה בעת העתקת נתיב הקובץ). | |
| בעיות בתוכנית שלא נפתרו באמצעות שלבי פתרון הבעיות שלעיל | בעיות תוכנה או שורת פקודה שצוינו לעיל. | שלח בעיה דרך GitHub. נווט אל https://github.com/rbudnar/nuclei_counter. בשורת התפריטים בחלק העליון, בחר "בעיות" (אפשרות שנייה, מימין ל"קוד"). לחץ על הכפתור הירוק בצד ימין שאומר "גיליון חדש" כדי לשלוח. תופיע הנחיה להיכנס ל-GitHub לפני שליחת הבעיה. תהיה גם אפשרות ליצור חשבון בחינם במידת הצורך. |
טבלה 1: פתרון בעיות לצילום תמונות איכותיות והפעלת זרימת העבודה האוטומטית של כימות גרעין.
בסך הכל, השיטות המתוארות כאן מספקות פרטים שלב אחר שלב לשימוש בתוכנית כימות הגרעינים האוטומטית שפיתחנו באמצעות ספריות ראייה ממוחשבת ואומתה מול ספירות ידניות ממספר מדרגים. תוכנה זו פותחה כדי להבטיח שימושיות על ידי מדענים בעלי רמות שונות של מיומנויות טכנולוגיות הקשורות לתכנות, זמינה בחינם ואושרה במיוחד למשימת כימות גרעינים. בהשוואה לספירה ידנית, תוכנה זו תחסוך למשתמש הקצה זמן יקר ותספק ספירת גרעינים מדויקת ואמינה לנורמליזציה של נתונים.
Disclosures
המחברים מצהירים שאין ניגודי אינטרסים.
Acknowledgements
המימון לעבודה זו ניתן על ידי המכונים הלאומיים לבריאות/המכון הלאומי להזדקנות (R01AG084597; DEL ו-HYL) ועל ידי קרנות סטארט-אפ מאוניברסיטת טקסס טק (DEL). המחברים רוצים להודות לחוקרי המחקר לתואר ראשון של אוניברסיטת טקסס טק ולתוכניות TrUE Scholars על מתן תמיכה כספית לחוקרים לתואר ראשון שתרמו לעבודה זו (REH, MRD, CJM, AKW). אנו מודים גם לד"ר לורן ס. גולהון ומייקל פ. מאסט על שיתוף אדיב בשטח המעבדה ובציוד שלהם.
Materials
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
References
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved