Method Article
Utilisation des bibliothèques de vision par ordinateur pour rationaliser la quantification des noyaux
Dans cet article
Résumé
Cet article décrit des méthodes étape par étape pour automatiser la quantification de noyaux basée sur des images à l’aide d’un programme exécutable open source validé sur une gamme de densités cellulaires. Ce programme offre une solution de rechange qui s’attaque aux obstacles liés au coût, à l’accessibilité pour les utilisateurs ayant des compétences technologiques limitées et à la validation propre à l’application qui peut limiter l’utilité des technologies existantes.
Résumé
Les tests sur cellules vivantes et les analyses de cellules basées sur l’image nécessitent une normalisation des données pour une interprétation précise. Une méthode couramment utilisée consiste à colorer et à quantifier les noyaux, puis à normaliser les données en fonction du nombre de noyaux. Ce nombre de noyaux est souvent exprimé en nombre de cellules uninucléées. Bien que la quantification manuelle puisse être laborieuse et chronophage, les méthodes automatisées disponibles peuvent ne pas être préférées par tous les utilisateurs, peuvent manquer de validation pour cette application spécifique ou peuvent être prohibitives. Ici, nous fournissons des instructions étape par étape pour capturer des images quantifiables de noyaux colorés avec des colorants d’ADN fluorescents, puis quantifier les noyaux à l’aide d’un logiciel de comptage d’objets automatisé développé à l’aide de bibliothèques de vision par ordinateur Python. Nous validons également ce programme sur une gamme de densités cellulaires. Bien que le temps exact d’exécution du programme varie en fonction du nombre d’images et de matériel informatique, ce programme consolide les heures de travail en comptant les noyaux en secondes pour que le programme s’exécute. Bien que ce protocole ait été développé à l’aide d’images de cellules fixes et colorées, les images de noyaux colorés dans des cellules vivantes et des applications d’immunofluorescence peuvent également être quantifiées à l’aide de ce programme. En fin de compte, ce programme offre une option qui ne nécessite pas un haut degré de compétence technologique et constitue une alternative validée et open-source pour aider les biologistes cellulaires et moléculaires à rationaliser leurs flux de travail, en automatisant la tâche fastidieuse et chronophage de quantification des noyaux.
Introduction
Les expériences fonctionnelles et basées sur l’image sont essentielles pour comprendre les impacts des traitements expérimentaux sur la biochimie et la physiologie des cellules entières. L’interprétation valide des données des expériences de biologie cellulaire dépend de la précision et de la reproductibilité du protocole expérimental, y compris la normalisation des données. Par exemple, l’analyse de la consommation d’oxygène et des taux d’acidification extracellulaire dans les cellules vivantes au départ et après le traitement avec des médicaments spécifiques permet d’évaluer divers aspects du métabolisme énergétique 1,2. La mesure de l’activité d’enzymes telles que la lactate déshydrogénase dans le surnageant des cultures cellulaires peut aider à quantifier l’intégrité de la membrane cellulaire3. La coloration des cellules cultivées avec de l’annexine V et de l’iodure de propidium avant la fixation permet d’évaluer les cellules apoptotiques et nécrotiques4. Cependant, les différences de densité cellulaire entre les puits affectent les résultats de chacun de ces tests. Se fier uniquement à la densité d’ensemencement pourrait fournir des résultats trompeurs en raison d’erreurs dans le comptage des cellules pour l’ensemencement, de variations de la densité cellulaire dans les milieux pendant le placage ou de différents taux de croissance cellulaire entre les échantillons ou les traitements tout au long des expériences. Par conséquent, la normalisation des résultats expérimentaux est nécessaire.
Les méthodes actuelles de normalisation des données cellulaires fonctionnelles et basées sur l’image comprennent la concentration en protéines5 ou les noyaux ou le nombre de cellules6. Pour normaliser les données sur la concentration en protéines, les cellules doivent être extraites dans des volumes égaux de tampon de lyse avec un inhibiteur de protéase après l’exécution de l’essai, et un essai supplémentaire (p. ex., essai d’acide bicinchoninique, essai de Bradford, etc.) doit être effectué pour la quantification des protéines. La précision de cette méthode repose sur l’extraction complète des cellules ; L’abandon de toute protéine contribue aux erreurs de quantification. Le comptage des noyaux ou des cellules offre une alternative lorsqu’il n’est pas nécessaire de retirer les cellules de la surface sur laquelle elles sont cultivées. Au lieu de cela, les cellules vivantes ou fixes sont colorées ou contre-colorées à l’aide de colorants colorimétriques ou fluorescents, et le comptage peut être effectué sur la base d’images de cellules entières ou uniquement de noyaux. La quantification des noyaux est préférée à celle des cellules entières pour les applications de microscopie à fluorescence et pour normaliser les données dans les cellules fusionnées (par exemple, les myotubes) ou les tissus. Pour la quantification des noyaux à l’aide d’une approche de fluorescence, des colorants d’ADN fluorescents (par exemple, le dichlorure de 4′,6-diamidino-2-phénylindole (DAPI), Hoechst 33342, Hoechst 33358, DRAQ5, etc.) sont appliqués sur des cellules vivantes ou fixes et imagés à l’aide de la microscopie à fluorescence. DAPI, Hoechst et DRAQ5 se lient préférentiellement aux régions riches en adénine-thymine de l’ADN7, 8, 9, fournissant un moyen de détecter les noyaux. L’optimisation de la concentration du colorant empêche la liaison hors cible (c’est-à-dire à l’ADN mitochondrial, provoquant une fluorescence cytosolique ponctuée)10. Le processus d’optimisation peut être rationalisé en utilisant des supports de montage disponibles dans le commerce contenant du DAPI pour les cellules fixes (par exemple, VECTASHIELD Antifade Mounting Medium avec DAPI) et des concentrations publiées de colorants pour les cellules vivantes (par exemple, Hoechst)6,11,12. Une fois colorés, les noyaux des cellules vivantes ou fixes sont visualisés à l’aide du filtre d’excitation approprié (par exemple, ultraviolet [DAPI et Hoechst] ou rouge lointain [DRAQ5]). Les maxima d’excitation pour DAPI, Hoechst et DRAQ5 se situent aux deux extrémités du spectre pour la microscopie à fluorescence. Cet attribut, ainsi que la spécificité de l’ADN de ces colorants et leur utilisation à des concentrations optimales, minimisent la fluorescence hors cible. Les images résultantes montrent des noyaux brillants sur un fond noir, et ces noyaux sont quantifiés comme une mesure du nombre de cellules uninucléées. Bien qu’il existe d’autres méthodes précises de quantification des noyaux basées sur les colorants à l’aide des colorants histologiques et de la microscopie à fond clair13, l’automatisation est plus difficile, bien que possible, avec ces approches.
Bien qu’elle reste la référence absolue, la quantification manuelle des noyaux est laborieuse, prend du temps et est susceptible d’être commise par l’homme en cas de comptage prolongé. Bien qu’il existe des programmes de comptage automatisé de cellules, ils ne sont pas préférés par tous les utilisateurs, peuvent être prohibitifs et la validation pour des applications spécifiques peut être minimale. Python est devenu une ressource précieuse et accessible pour les biologistes ces dernières années. Les bibliothèques de vision par ordinateur Python sont particulièrement utiles pour l’analyse d’images14. Ici, nous fournissons des instructions étape par étape pour l’utilisation de notre programme exécutable développé à l’aide de code Python pour quantifier les noyaux colorés avec des colorants fluorescents et imagés à l’aide de la microscopie à fluorescence. Il n’est pas nécessaire d’avoir des compétences en codage pour utiliser le programme décrit ici. Ce flux de travail est spécifique aux cellules ou aux tissus auxquels des colorants d’ADN fluorescents sont appliqués. Il n’est pas destiné à être utilisé avec des images en fond clair. La section 1 décrit comment capturer et enregistrer des images collectées à l’aide de la microscopie à fluorescence d’une manière compatible avec ce flux de travail. La section 2 fournit des instructions pour exécuter le programme de quantification des noyaux en tant qu’exécutable et récupérer la sortie. Le programme exécutable peut être exécuté directement à partir du fichier .exe fourni et ne nécessite aucune modification pour quantifier les noyaux dans des images de qualité. Cet exécutable nécessite un PC Windows. La section 3 fournit des instructions pour exécuter le programme en tant que script Python plutôt qu’en tant qu’exécutable (requis pour les systèmes basés sur Mac ou Linux, facultatif pour les systèmes basés sur Windows) et permet d’ajuster le code si vous le souhaitez. La section 4 décrit comment utiliser les résultats pour la normalisation. Ces protocoles sont suivis de résultats représentatifs, y compris des données de validation de notre laboratoire.
Protocole
REMARQUE : Les fichiers supplémentaires se trouvent au lien suivant https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. Capture et enregistrement d’images à l’aide de la microscopie à fluorescence
- Préparez les échantillons de cellules ou de tissus à imager, y compris la coloration avec le colorant d’ADN souhaité. Pour obtenir les images utilisées ici, des myoblastes C2C12 (CRL-1772, American Type Culture Collection) ont été cultivés dans des plaques à 6 puits pendant 48 à 72 h dans des conditions de culture standard (5 % de CO2, 37 °C, humidifiés), avec ou sans 50 mM d’EtOH6, et fixés dans du méthanol glacé comme décrit précédemment13. Les cellules fixes ont été montées à l’aide d’un support de montage contenant du DAPI15 et séchées dans l’obscurité avant l’imagerie.
- Allumez tous les composants du microscope à fluorescence et de l’ordinateur conformément aux procédures de fonctionnement standard établies. Ouvrez le logiciel associé au microscope.
- Réglez le microscope pour visualiser les échantillons à travers l’oculaire. Vérifiez l’objectif. Si nécessaire, tournez le nez vers l’objectif souhaité. Assurez-vous que le logiciel a sélectionné l’objectif souhaité pour une indication précise de la zone de l’image. Ici, les images ont été capturées à l’aide d’un objectif 10x.
- Avec les lumières éteintes, placez l’échantillon (lame, assiette ou plat) sur la scène. Activez l’imagerie en direct avec le filtre approprié et réglez le temps d’exposition sur une durée prédéterminée ou sur l’exposition automatique. Ici, un filtre DAPI et un temps d’exposition de 61-62 ms ont été utilisés. Consultez le tableau de dépannage pour obtenir des conseils sur la définition du temps d’exposition, en particulier pour les images à haute densité de cellules.
- À l’aide de l’oculaire, ajustez lentement la mise au point à l’aide du bouton de réglage grossier jusqu’à ce que les noyaux colorés soient visibles, puis utilisez le bouton de réglage fin pour ajuster lentement la mise au point jusqu’à ce que les noyaux apparaissent clairs et nets.
REMARQUE : Des exemples d’images appropriées sont présentés à la Figure 1.- Si une coloration de fond excessive apparaît ou si les noyaux semblent très sombres, ajustez manuellement le temps d’exposition. Dans une seule image, il peut y avoir des différences visibles dans l’intensité du signal pour certains noyaux ; Cependant, la forme ronde doit être visible pour tous les noyaux avec un minimum d’interférences de signal.
REMARQUE : Le programme utilisé dans les applications en aval est robuste à différentes intensités. Les images brutes utilisées pour le développement et la validation du programme de quantification des noyaux comprennent des images avec une gamme d’intensités de signal et sont disponibles dans le référentiel du Cadre de science ouverte (voir lien mentionné ci-dessus).
- Si une coloration de fond excessive apparaît ou si les noyaux semblent très sombres, ajustez manuellement le temps d’exposition. Dans une seule image, il peut y avoir des différences visibles dans l’intensité du signal pour certains noyaux ; Cependant, la forme ronde doit être visible pour tous les noyaux avec un minimum d’interférences de signal.
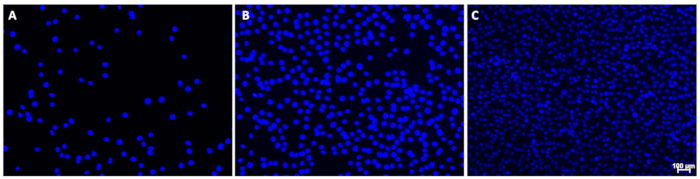
Figure 1 : Exemples d’images de noyaux colorés au DAPI. Ces images représentent des noyaux à (A) faibles, (B) moyennes ou (C) hautes densités cellulaires avec un arrière-plan minimal. Notez l’intensité variable du signal des noyaux sur l’image C ; Cela n’a pas interféré avec la quantification. La barre d’échelle (C) indique 100 μm ; Les barres d’échelle doivent être omises des images utilisées pour la quantification des noyaux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Une fois que les noyaux sont mis au point à travers l’oculaire, basculez le mode de détection sur l’appareil photo. Réglez la mise au point à l’aide du bouton de réglage fin.
- Ajustez le temps d’exposition si nécessaire. Notez le temps d’exposition final à utiliser avec les échantillons et les images suivants.
REMARQUE : Bien que l’utilisation du même temps d’exposition sur toutes les images soit idéale, les variations du temps d’exposition sont acceptables pour cette application car l’intensité de fluorescence n’est pas quantifiée ou comparée entre les échantillons. - Capturez l’image à l’aide du bouton de capture d’image approprié du logiciel. Si plusieurs colorants ont été appliqués sur des échantillons, changez le filtre (par exemple, sur FITC, TRITC ou Cy5) et imagez la même zone à des fins de fusion.
- Appliquez l’obturateur pour minimiser l’exposition de l’échantillon à la lumière. Sans graver dans une barre d’échelle, enregistrez l’image dans un format de fichier de figure universel (.tif, .png ou .jpg) dans un dossier dédié. Assurez-vous que chaque nom de fichier image est correctement descriptif ou que tout codage utilisé pour les noms de fichiers comporte une documentation liée appropriée.
REMARQUE : Cela deviendra le répertoire d’images pour le programme de quantification. Si l’imagerie multicolore a été utilisée, copiez uniquement les images des noyaux dans un dossier séparé et dédié. Cela peut également être fait à la fin de la séance d’imagerie. Les images exportées à l’aide des paramètres d’exportation par défaut de la plupart des logiciels d’imagerie ont généralement une résolution suffisante pour être utilisées avec ce programme, à condition que des images de qualité aient été capturées. Cependant, les images doivent être vérifiées visuellement après l’exportation pour s’assurer que la morphologie des noyaux est claire. Si les images sont enregistrées dans .jpg format, veillez à ce que les images ne soient pas excessivement compressées et que la qualité soit maintenue. - Ouvrez l’obturateur, revenez à l’imagerie en direct et balayez l’échantillon jusqu’à un autre emplacement pour l’imagerie. Répétez les étapes 1.8-1.9 si nécessaire. Pour obtenir des images de cellules ensemencées dans des plaques à 6 puits capturées à un grossissement de 10x, capturez 10 à 15 images par puits (en fonction de la cohérence de la densité cellulaire dans la zone de l’échantillon).
- Une fois toutes les images d’un seul échantillon acquises, effectuez l’une des options suivantes.
- Passez à l’échantillon suivant et répétez les étapes 1.4 à 1.10.
- Mettez le protocole en pause et revenez à un autre moment. Les échantillons doivent rester à l’abri de la lumière. Cette option n’est pas recommandée si vous travaillez avec des cellules vivantes.
- Une fois l’imagerie terminée, assurez-vous que tous les échantillons sont protégés de la lumière avant d’allumer les lumières de la pièce. Si le dossier contenant les images est enregistré localement, transférez les fichiers vers une clé USB, un emplacement de stockage en nuage ou un autre lecteur réseau accessible à l’ordinateur sur lequel le programme de quantification sera exécuté. Enregistrez-le dans un dossier dédié.
- Éteignez toutes les parties du microscope et de l’ordinateur conformément aux procédures d’utilisation standard établies.
- L’imagerie est maintenant terminée. Analysez les fichiers à l’aide du programme de quantification automatique décrit à la section 2 ou, alternativement, en exécutant le programme en tant que script Python comme décrit à la section 3.
2. Exécution du programme de quantification automatisée des noyaux en tant qu’exécutable
REMARQUE : Cette méthode, qui exécute le programme en tant qu’exécutable, est compatible avec les PC Windows et est recommandée pour les utilisateurs du système d’exploitation Windows. Mac OS ou d’autres utilisateurs non Windows doivent exécuter le programme en tant que script Python (voir Section 3).
- Assurez-vous que Python est installé sur l’ordinateur. Si nécessaire, installez Python à partir de python.org/downloads (de préférence) ou à l’aide d’Anaconda ou de Miniconda. À la date de publication, ce programme a été testé avec les versions 3.10-3.12 de Python.
- Ouvrez le navigateur et accédez à https://github.com/rbudnar/nuclei_counter/releases. Téléchargez la dernière version du fichier nommé count_nuclei.zip.
- Dans le dossier Téléchargements, cliquez avec le bouton droit de la souris sur le fichier Zip et extrayez tous les fichiers à l’emplacement souhaité sur l’ordinateur local (voir Figure 2A). Gardez cet emplacement de fichier ouvert ; ce chemin d’accès au fichier sera utilisé à l’étape 2.5 (voir Figure 2B).
REMARQUE : Count_nuclei devrait maintenant apparaître sous forme de fichier de candidature. Il s’agit du fichier exécutable. - Ouvrez une invite de commande en recherchant CMD ou Invite de commande dans la barre de recherche. Utilisez la commande cd pour modifier le répertoire en chemin d’accès au fichier exécutable (voir Figure 2C) et appuyez sur Entrée. Exemple de ligne de commande : C :\Users\janedoe>cd [espace après cd, puis copier/coller le chemin du fichier]
- Sur la ligne de commande suivante, tapez ce qui suit et appuyez sur Entrée.
count_nuclei.exe --img_dir « chemin/vers/images » --output_dir « chemin/vers/sortie » --save_images --output_count_filename « results.csv »
où chemin/vers/images est le chemin d’accès au dossier contenant les images à analyser (voir étape 1.14) ; path/to/output est le chemin d’accès au dossier où le fichier .csv doit être enregistré (par défaut le répertoire de travail actuel lorsqu’il n’est pas fourni) ; et results.csv est le nom de fichier dans lequel les comptes résultants seront enregistrés (s’il n’est pas fourni, le nom du fichier de sortie est par défaut count_results.csv).
REMARQUE : Les guillemets sont nécessaires dans la ligne de commande. Assurez-vous qu’il n’y a pas d’espace après les guillemets avant le chemin d’accès au fichier (voir Figure 2C). - Lorsque la ligne de commande suivante apparaît, le traitement est terminé. Les contours et la feuille de calcul des résultats sont maintenant disponibles à l’emplacement spécifié comme répertoire de sortie à l’étape 2.5 (c’est-à-dire chemin/vers/sortie ; voir la figure 2C).
- Inspectez visuellement les contours pour vérifier la qualité du comptage. Les résultats peuvent maintenant être utilisés pour normaliser les données, comme décrit à la section 4.
REMARQUE : Si les contours semblent avoir des noyaux manqués (par exemple, les noyaux semblent trop faibles) ou regrouper des groupes de noyaux en un seul élément (par exemple, image surexposée, trop d’arrière-plan, artefacts), l’utilisateur doit se référer au tableau de dépannage et peut avoir besoin de revenir à la section 1 pour optimiser l’acquisition de l’image avant de passer à la section 4.
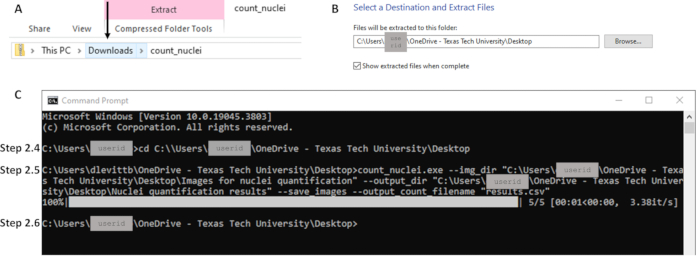
Figure 2 : Exemples d’éléments clés du flux de travail pour la section 2. (A) Extrayez les fichiers du dossier Téléchargements en prenant soin de cliquer sur le bon dossier comme indiqué par la flèche pour activer l’extraction (B) vers la destination souhaitée à l’étape 2.3. (C) Exemples de commandes pour les étapes 2.4 et 2.5 et la ligne de commande suivante indiquant l’achèvement à l’étape 2.6. Notez que le chemin d’accès au fichier à l’étape 2.4 (C) correspond à l’emplacement d’extraction à l’étape 2.3 (B). Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Exécution du programme de quantification automatisée des noyaux en tant que script Python
- Assurez-vous que Python est installé sur l’ordinateur. Si nécessaire, installez Python à partir de python.org/downloads (de préférence) ou à l’aide d’Anaconda ou de Miniconda. À la date de publication, ce programme a été testé avec les versions 3.10-3.12 de Python.
- Ouvrez le navigateur et accédez à https://github.com/rbudnar/nuclei_counter. Cliquez sur le bouton Code vert , puis sur Télécharger le fichier ZIP pour télécharger le référentiel de code (voir Figure 3A).
- Utilisez les instructions suivantes pour le système d’exploitation Windows. Voir l’étape 3.4 pour les machines basées sur Mac OS.
- Dans le dossier Téléchargements, cliquez avec le bouton droit de la souris sur le fichier et sélectionnez Extraire tout pour extraire les fichiers sur un ordinateur local (voir Figure 3B-C).
- Dans l’emplacement du fichier extrait, ouvrez le dossier nommé nuclei_counter-main. Il s’agit du référentiel de code.
REMARQUE : Il ne doit être téléchargé que la première fois que ce programme est utilisé ; Il suffit de déplacer les fichiers extraits vers un emplacement réseau accessible et de noter cet emplacement. - Ouvrez une invite de commande en recherchant CMD ou Invite de commande dans la barre de recherche. Si Python a été installé par Anaconda ou Miniconda, ouvrez l’invite Anaconda au lieu d’une invite de commande standard. Vous pouvez le trouver en tapant Anaconda Prompt dans la barre de recherche.
- Utilisez la commande cd pour changer le répertoire en référentiel de code et appuyez sur Entrée. Voir la ligne de code ci-dessous, qui est sensible à la casse. Assurez-vous que le dossier nommé nuclei_counter-main est ouvert lors de la copie du chemin d’accès au fichier ; le fichier nommé pyproject ou pyproject.toml doit être visible (voir Figure 3D). Exemple de ligne de commande : C :\Users\janedoe>cd [espace après cd, puis copier/coller le chemin du fichier]
- Sur la ligne de commande suivante, tapez ce qui suit, y compris l’espace et le point (voir Figure 3D :
pip install -e .
REMARQUE : Les étapes 3.3.5 et 3.3.6 ne doivent être terminées que la première fois que ce programme est exécuté sur un ordinateur. Si cela a déjà été fait, passez à l’étape 3.3.7. L’initialisation du processus d’installation peut prendre quelques instants. - Appuyez sur Entrée. L’exécution de cette commande installe les bibliothèques requises pour exécuter ce script et installe le référentiel en mode modifiable dans le cas où des ajustements de code seraient nécessaires (voir Figure 3D).
- Tapez ce qui suit dans la ligne de commande suivante pour remplacer le répertoire par le répertoire principal du code source (voir Figure 3E) :
cd « nuclei_counter » - Sur la ligne de commande suivante, tapez ce qui suit et appuyez sur Entrée (voir Figure 3E) :
python count_nuclei.py --img_dir « chemin/vers/images » --output_dir « chemin/vers/sortie » --save_images --output_count_filename « results.csv »
où chemin/vers/images est le chemin d’accès au dossier contenant les images à analyser (étape 1.14) ; path/to/output est le chemin d’accès au dossier où le fichier .csv doit être enregistré (par défaut le répertoire de travail actuel lorsqu’il n’est pas fourni) ; et results.csv est le nom de fichier dans lequel les comptes résultants seront enregistrés (s’il n’est pas fourni, le nom du fichier de sortie est par défaut count_results.csv).
REMARQUE : Les guillemets sont nécessaires dans la ligne de commande. Assurez-vous qu’il n’y a pas d’espaces après les guillemets avant le chemin d’accès au fichier. Le script python lira les fichiers dans le chemin d’accès fourni et exécutera le programme de comptage de noyaux pour toutes les images du répertoire (voir Figure 3E). Les décomptes résultants seront enregistrés dans le fichier .csv résultats à l’emplacement spécifié. - Lorsque la ligne de commande suivante apparaît, le traitement est terminé (voir Figure 3E). Les contours et la feuille de calcul des résultats sont maintenant disponibles à l’emplacement spécifié comme répertoire de sortie à l’étape 3.3.8 (c’est-à-dire chemin/vers/sortie).
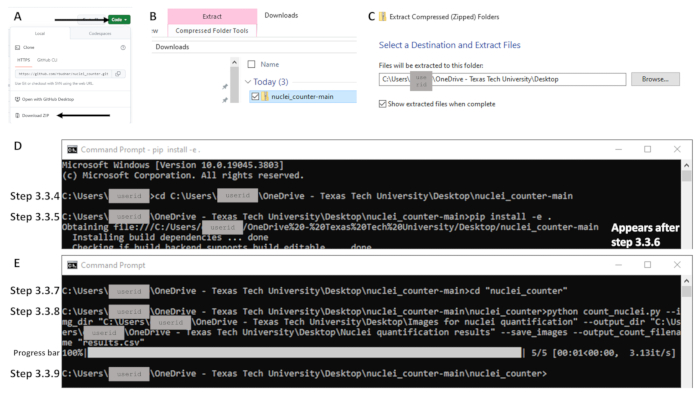
Figure 3 : Exemples d’éléments clés du flux de travail de la section 3 à l’aide du système d’exploitation Windows. Pour accéder au code, cliquez sur le bouton vert intitulé Code et (A) téléchargez le fichier Zip (indiqué par des flèches). (B) Extrayez en faisant un clic droit sur le fichier Zip (en bleu) et (C) en définissant la destination souhaitée. (D) Après avoir ouvert l’invite de commande, exemples de commandes pour les étapes 3.3.4 et 3.3.5. Notez que la commande de l’étape 3.3.5 est sur une seule ligne ; le texte commençant par « Obtention du fichier » sur la ligne suivante apparaît après avoir appuyé sur Entrée (étape 3.3.6 ; c’est-à-dire exécution de la commande). (E) Exemples de commandes pour les étapes 3.3.7 et 3.3.8, une barre de progression après l’exécution de l’étape 3.3.8 et la ligne de commande indiquant l’achèvement de l’étape 3.3.9. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Utilisez les instructions suivantes pour Mac OS.
- Dans le dossier Téléchargements, cliquez sur le menu Fichier et sélectionnez Ouvrir pour extraire les fichiers sur l’ordinateur local (voir Figure 4A).
- Dans l’emplacement du fichier extrait, ouvrez le dossier nommé nuclei_counter-main. Il s’agit du référentiel de code.
REMARQUE : Il ne doit être téléchargé que la première fois que ce programme est utilisé ; Il suffit de déplacer les fichiers extraits vers un emplacement réseau accessible et de noter cet emplacement. - Ouvrez un terminal en appuyant sur commande + barre d’espace qui ouvre le projecteur ; Ensuite, recherchez « Terminal ».
- Utilisez la commande cd pour changer le répertoire en référentiel de code et appuyez sur Entrée. Voir la ligne de code ci-dessous, qui est sensible à la casse. Tapez le chemin d’accès au fichier dans un document texte et copiez-le dans le terminal. Assurez-vous que le chemin d’accès au fichier commence par le texte qui précède immédiatement le cd (par exemple, /Users/userid dans la Figure 4B). Exemple de ligne de commande : /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- Sur la ligne de commande suivante, un signe dollar apparaît après l’ID utilisateur (voir Figure 4B). Assurez-vous qu’il y a un espace après le signe dollar et tapez ce qui suit, y compris l’espace et le point à la fin de la commande. Incluez la version appropriée de Python immédiatement après pip sans espace : pip3 install -e .
REMARQUE : Les étapes 3.4.5 et 3.4.6 ne doivent être effectuées que la première fois que ce programme est exécuté sur un ordinateur. Si cela a déjà été fait, passez à l’étape 3.4.7. L’initialisation du processus d’installation peut prendre quelques instants. - Appuyez sur Entrée. L’exécution de cette commande installe les bibliothèques requises pour exécuter ce script et installe le référentiel en mode modifiable si des ajustements de code sont nécessaires.
- Sur la ligne de commande suivante, assurez-vous qu’il y a un espace après le signe dollar, puis tapez ce qui suit pour remplacer le répertoire par le répertoire principal du code source (voir la figure 4C) :
cd « nuclei_counter » - Sur la ligne de commande suivante, tapez la ligne de commande ci-dessous. Remplacez le numéro après Python par le numéro de version correct si vous utilisez une version autre que 3. Appuyez ensuite sur Entrée (voir Figure 4C) :
python3 count_nuclei.py --img_dir « chemin/vers/images » --output_dir « chemin/vers/sortie » --save_images --output_count_filename « results.csv »
où chemin/vers/images est le chemin d’accès au dossier contenant les images à analyser (étape 1.14) ; path/to/output est le chemin d’accès au dossier où le fichier .csv doit être enregistré (par défaut le répertoire de travail actuel lorsqu’il n’est pas fourni) ; et results.csv est le nom de fichier dans lequel les comptes résultants seront enregistrés (s’il n’est pas fourni, le nom du fichier de sortie est par défaut count_results.csv).
REMARQUE : Les guillemets sont nécessaires dans la ligne de commande. Assurez-vous qu’il n’y a pas d’espaces après les guillemets avant le chemin d’accès au fichier. Le script Python lira les fichiers dans le chemin d’accès fourni et exécutera le programme de comptage de noyaux pour toutes les images du répertoire (voir Figure 4C). Les décomptes résultants seront enregistrés dans le fichier .csv résultats à l’emplacement spécifié. - Lorsque la ligne de commande suivante apparaît, le traitement est terminé (voir Figure 4C). Les contours et la feuille de calcul des résultats sont maintenant disponibles à l’emplacement spécifié comme répertoire de sortie à l’étape 3.4.8 (c’est-à-dire chemin/vers/sortie).
- Inspectez visuellement les contours pour vérifier la qualité du comptage. Les résultats peuvent maintenant être utilisés pour normaliser les données, comme décrit à la section 4.
REMARQUE : Si les contours semblent avoir des noyaux manquants (p. ex., les noyaux semblent trop faibles) ou regrouper des groupes de noyaux en un seul élément (p. ex., image surexposée, trop d’arrière-plan, artefacts), l’utilisateur doit se référer au tableau 1 et peut avoir besoin de revenir à la section 1 pour optimiser l’acquisition d’images avant de passer à la section 4.
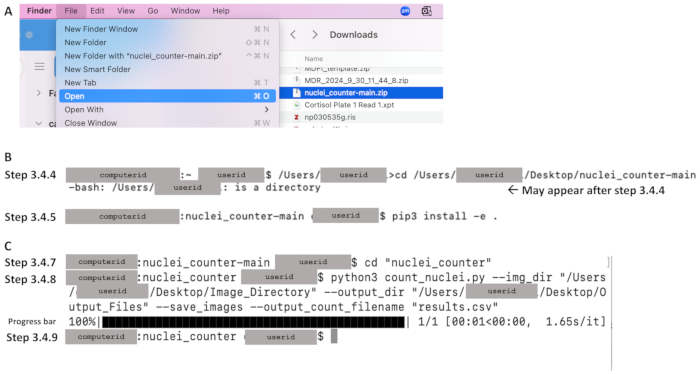
Figure 4 : Exemples d’éléments clés du flux de travail de la section 3 sous Mac OS. Après avoir téléchargé le code (voir Figure 3A), (A) extrayez-le en cliquant sur le fichier Zip (en bleu) et en définissant la destination souhaitée. (B) Après l’ouverture du terminal, exemples de commandes pour les étapes 3.4.4 (changement du répertoire en dépôt de code) et 3.4.5 (installation de pip). (C) Exemples de commandes pour les étapes 3.4.7 et 3.4.8, une barre de progression après l’exécution de l’étape 3.4.8 et la ligne de commande indiquant l’achèvement à l’étape 3.4.9. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Utilisation des dénombrements pour normaliser les données expérimentales
- Ouvrez le fichier results.csv reliant chaque image avec la quantification des noyaux déterminée à l’aide du programme automatisé.
- Décider de la méthode de normalisation : 1) image par image (par exemple, si la microscopie à fluorescence multicolore), 2) normaliser les données en fonction du nombre moyen de noyaux par échantillon par rapport à un échantillon de référence (ou condition), 3) normaliser en fonction du nombre de noyaux par échantillon (par exemple, extrapoler le nombre total de noyaux à la surface de l’échantillon). Les recommandations pour la méthode de normalisation se trouvent dans la section Discussion.
- Si la normalisation doit être effectuée image par image, il suffit de diviser les données expérimentales de chaque image par le nombre de noyaux de cette image.
- Pour normaliser les données sous forme de ratio, suivez les étapes décrites ci-dessous.
- Pour tous les échantillons où plusieurs images ont été collectées pour une seule répétition (par exemple, 10 à 15 images capturées à partir d’un seul puits dans une plaque à 6 puits), calculez le nombre moyen de noyaux par image. Ce nombre de noyaux est considéré comme le nombre de cellules en supposant des cellules uninucléées ou un certain nombre de cellules fusionnées dans des cellules multinucléées (comme avec des myotubes).
- Sélectionnez soit un échantillon de référence, soit une condition de référence (p. ex., dénombrement moyen de tous les échantillons à partir de la condition de contrôle) et notez le dénombrement.
- Divisez le nombre de tous les échantillons par ce nombre de référence, ce qui donne une valeur de normalisation. Si un échantillon de référence a été utilisé, utilisez la valeur de normalisation de l’échantillon de référence comme 1.
REMARQUE : Si une condition de référence a été utilisée, la valeur moyenne de normalisation pour l’ensemble de la condition serait désormais de 1. Les échantillons contenant moins de noyaux que la référence (c’est-à-dire moins denses) auront des valeurs de normalisation inférieures à 1, et les échantillons avec plus de noyaux que la référence (c’est-à-dire plus denses) auront des valeurs de normalisation supérieures à 1. - Divisez les données expérimentales de chaque puits par la valeur de normalisation calculée correspondante.
- Pour normaliser les données en fonction du nombre de noyaux par échantillon, suivez les étapes décrites ci-dessous.
- Pour tous les échantillons où plusieurs images ont été collectées pour une seule répétition (par exemple, 10 à 15 images capturées à partir d’un seul puits dans une plaque à 6 puits), calculez le nombre moyen de noyaux par image. Ce nombre de noyaux est considéré comme le nombre de cellules en supposant des cellules uninucléées, ou le nombre de cellules fusionnées dans des cellules multinucléées (comme avec des myotubes).
- Déterminez la surface de la lame (lamelle), du puits ou de la parabole à partir de laquelle les données ont été dérivées. Ces informations se trouvent dans la section des spécifications sur les sites Web des produits.
- Déterminez la zone de chaque image capturée. Ces informations sont souvent disponibles sous forme de métadonnées dans le logiciel d’imagerie ou d’analyse.
- Assurez-vous que la surface de l’échantillon et la surface de l’image sont exprimées dans les mêmes unités (p. ex., mm2). Si ce n’est pas le cas, convertissez dans l’unité souhaitée.
- Calculez le dénombrement total pour chaque échantillon à l’aide de l’équation suivante :
TC = AC • (SA/IA)
où TC est le dénombrement total, AC est le dénombrement moyen, SA est la surface, IA est la surface de l’image. - Divisez les données expérimentales par le TC correspondant pour l’échantillon.
- Normaliser les données expérimentales et les utiliser pour des analyses statistiques, le cas échéant.
Résultats
Chaque série d’images par lots produit : 1) un ensemble de fichiers d’images avec des contours appliqués montrant les contours des noyaux identifiés (Figure 5), et 2) un fichier .csv (tableur) reliant les noms des fichiers d’images et les comptages associés. La visualisation des contours permettra à l’utilisateur d’évaluer visuellement la qualité du comptage. Plus précisément, les images obtenues selon la section 1 doivent avoir tous les noyaux (ou presque tous) entourés d’une ligne verte continue indiquant que le noyau a été compté par le programme. Ces contours ont été utilisés pour régler le programme ici.
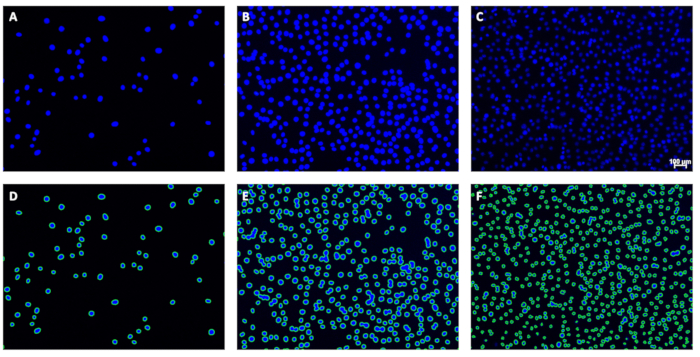
Figure 5 : Exemples d’images originales et de contours correspondants. (A-C) Exemples d’images originales avant l’analyse et (D-F) de contours correspondants après analyse directement ci-dessous. La barre d’échelle (C) indique 100 μm ; Les barres d’échelle doivent être omises des images utilisées pour la quantification des noyaux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Validation à l’aide des premiers résultats
Des myoblastes C2C12 à différentes densités cellulaires ont été fixés au méthanol, contre-colorés avec du DAPI dans des supports de montage, et 120 images ont été obtenues à un grossissement de 10x dans .jpg format de fichier. Le programme automatisé a été exécuté avec le répertoire d’images défini dans le dossier contenant ces images, et le fichier .csv résultant a été enregistré. Séparément, chaque image a été quantifiée manuellement par deux chercheurs formés qui étaient aveugles aux comptages automatisés et aux comptages de l’autre. Des images brutes, des contours générés par le programme automatisé et une feuille de calcul des résultats comprenant des comptages manuels et automatisés sont disponibles dans le référentiel du Cadre de science ouverte (OSF) (voir le lien à la section 1). Les coefficients de corrélation intraclasse (ICC ; mesures moyennes) ont été calculés à l’aide des statistiques SPSS pour déterminer la fiabilité inter-juges entre 1) les deux comptages manuels et 2) la moyenne des deux comptages manuels et des dénombrements obtenus à l’aide du programme automatisé. Des approches similaires ont été utilisées pour valider d’autres programmes conçus pour quantifier des objets dans des images obtenues à l’aide de la microscopie à fluorescence 16,17,18. Les ICC ont également été calculés entre les comptages manuels moyens et les comptages automatisés pour chaque quartile afin de s’assurer que l’évaluation de l’ensemble des données ne masquait aucun changement potentiel de fiabilité en fonction de la densité cellulaire. Les quartiles ont été définis à l’aide des points de coupure des 25e, 50e et 75centiles basés sur le dénombrement manuel moyen, et vont du quartile 1 (Q1 ; densité cellulaire la plus faible) à Q4 (densité cellulaire la plus élevée). Les ICC sont déclarées sur la base de mesures moyennes et interprétées selon les définitions suivantes : faible (ICC <0,50), modérée (0,50 ≤ICC <0,75), bonne (0,75 ≤ICC <0,90) ou excellente (ICC ≥0,90)19.
Les noyaux par image variaient de 44,5 à 1160 sur la base des moyennes de comptage manuel. La fiabilité inter-juges était excellente entre les deux comptages manuels (ICC > 0,999, p < 0,0001 ; Graphique 6A) et entre les comptages manuels et automatisés moyens (ICC = 0,993, p < 0,0001 ; Figure 6B). La fiabilité est demeurée excellente dans tous les quartiles (Q1 : ICC = 0,996, p < 0,0001 ; Q2 : ICC = 0,997, p < 0,0001 ; Q3 : CCI = 0,998, p < 0,0001 ; Q4 : CCI = 0,986, p < 0,0001 ; Figures 6C-F). Le point de données unique loin de la ligne d’ajustement optimale de la figure 6B représente l’image illustrée à la figure 7. L’image comporte plusieurs zones de plusieurs noyaux regroupés, empêchant le programme de vision par ordinateur de reconnaître chaque noyau. Ce problème peut être résolu en utilisant un temps d’exposition plus court. Le tableau 1 décrit comment gérer une telle image.
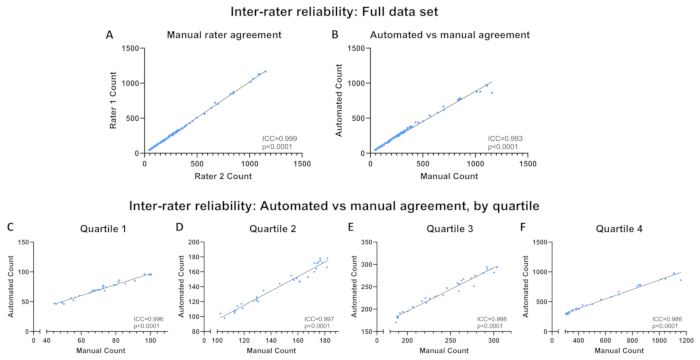
Figure 6 : Fiabilité inter-juges. Fiabilité inter-juges sur l’ensemble complet des données (A) entre les comptages manuels des évaluateurs et (B) entre les comptages automatisés et les comptages manuels moyens. (C-F) Fiabilité inter-juges entre les comptages automatisés et les comptages manuels moyens par quartile. Fiabilité exprimée en coefficient de corrélation intraclasse (ICC). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 7 : Exemple d’une image avec deux barrières pour une quantification précise des noyaux. (A) l’image originale et (B) les contours associés. La flèche 1 indique une zone avec plusieurs noyaux agglomérés, déformant suffisamment la forme nucléaire ronde pour empêcher la détection logicielle de plusieurs noyaux. Cela se produit à plusieurs endroits sur cette image. La flèche 2 indique un halo fané apparaissant derrière plusieurs noyaux ; Ce halo est apparu suffisamment brillant pour que le logiciel l’inclue comme noyau, perdant ainsi le nombre de noyaux multiples qu’il entoure. Cette image représente le seul point de données loin de la ligne la mieux ajustée de la figure 6B, mettant l’accent sur les problèmes potentiels liés à ces obstacles. La barre d’échelle (A) indique 100 μm ; Les barres d’échelle doivent être omises des images utilisées pour la quantification des noyaux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Notre programme de quantification des noyaux présente plusieurs avantages par rapport aux options existantes : il ne nécessite que des compétences technologiques minimales, est validé pour la tâche spécifique de quantification des noyaux et est open-source ; Ce dernier permet de surmonter les obstacles liés aux coûts. En fin de compte, ce programme offre aux biologistes cellulaires et moléculaires une option supplémentaire pour quantifier rapidement et précisément les noyaux dans des images capturées à l’aide de la microscopie à fluorescence. Les programmes automatisés de comptage de noyaux ou de cellules actuellement disponibles ne sont pas préférés par tous les utilisateurs, certains sont d’un coût prohibitif et la validation peut être minime ou absente pour des applications spécifiques. Des équipements spécialisés, tels que la ligne de cytation BioTek, sont accompagnés de bulletins techniques validant le comptage cellulaire in situ à l’aide de la coloration nucléaire20 en plus d’une grande variété d’applications supplémentaires. Bien qu’il existe des données de validation et que la gamme de produits Cytation soit largement utilisée, en particulier lorsqu’elle est interfacée avec les analyseurs de flux extracellulaires Seahorse pour normaliser facilement et efficacement les données de cellules vivantes, ce produit peut être d’un coût prohibitif pour les laboratoires disposant d’un financement limité en matière d’équipement. Par exemple, le prix catalogue du modèle de base équipé de quatre objectifs et de quatre cubes de filtre pour la microscopie à fluorescence a dépassé 100 000 dollars américains au cours de l’année civile 2024. Pour ceux qui n’ont pas les moyens d’acheter de l’équipement spécialisé, les logiciels open source offrent des alternatives gratuites pour l’analyse d’images. Parmi les logiciels open source utiles pour la quantification des noyaux et des cellules, citons ImageJ (National Institutes of Health, Bethesda, MD, États-Unis ; disponible à la p. 21. Des plugins sont maintenant disponibles pour ceux qui souhaitent écrire des scripts de macro pour l’automatisation du traitement d’images à l’aide de la plate-forme ImageJ. L’automatisation du comptage cellulaire basée sur imageJ à l’aide de la microscopie à contraste de phase ou à fond clair avec des images de cellules entières a déjà été décrite22,23. CellProfiler est une application largement publiée pour l’analyse d’images cellulaires colorées avec des colorants fluorescents ou des colorants histologiques standard, et un premier article décrivant son utilité dans une gamme d’applications biologiques est souvent cité comme validation24. Cependant, ce manuscrit ne valide aucune découverte biologique particulière, ce qui nécessite une validation et une transparence supplémentaires pour les différents pipelines d’automatisation utilisés, tels que pour les noyaux au comptant25. De plus, bien que CellProfiler soit un progiciel efficace pour l’analyse cellulaire, tous les scientifiques ne le trouvent pas convivial. Par conséquent, nous avons cherché à développer et à valider un programme automatisé alternatif pour quantifier les noyaux à l’aide d’un logiciel open source. Plus précisément, nous avons utilisé des bibliothèques de vision par ordinateur existantes en Python pour développer ce flux de travail.
Cette méthode commence par des cellules ou des tissus que l’utilisateur final a colorés à l’aide de colorants d’ADN tels que le DAPI26. Des directives générales pour la capture et l’enregistrement d’images d’une manière compatible avec les logiciels d’automatisation sont fournies. Bien que les détails puissent différer selon les microscopes et les logiciels d’imagerie, le flux de travail général de la section 1 est le même pour obtenir des images quantifiables. Si vous utilisez des échantillons vivants, il faut s’assurer que l’équipement de protection individuelle approprié est porté et que l’environnement d’imagerie est approprié pour ces échantillons (p. ex., température, pourcentage de CO2, ajout d’un tampon, etc.)27. Bien que ce protocole ne décrit l’imagerie que des colorants d’ADN, les utilisateurs peuvent capturer d’autres couleurs de la même zone à des fins de fusion15, et simplement copier les images nucléaires dans un dossier séparé pour les utiliser avec le programme de quantification. L’imagerie doit être effectuée dans une pièce sombre avec un EPI approprié pour les échantillons vivants et une désinfection appropriée doit être utilisée au besoin (p. ex., avec de l’éthanol à 70 %). Les échantillons colorés doivent être protégés de la lumière avant d’allumer les lumières. Une capture d’image de qualité est essentielle pour une quantification précise des noyaux. Même dans les images avec un temps d’exposition approprié et un fort contraste entre des noyaux brillants et un fond noir, la présence de cellules ou de halos excessivement agglomérés derrière des groupes de cellules (voir figure 7) peut diminuer la précision de la quantification. Dans ces cas, il peut être nécessaire de modifier les temps d’exposition pour diminuer l’intensité des signaux, ce qui garantit une fusion minimale des signaux des noyaux et une minimisation des halos tout en maintenant un signal suffisamment fort pour détecter les formes rondes des noyaux. Si ces étapes sont insuffisantes, il peut être nécessaire d’imager une section adjacente de l’échantillon. Toutes les images d’une même expérience doivent être capturées avec le même grossissement, et le grossissement correspondant à l’objectif utilisé doit être correctement réglé dans le logiciel d’imagerie. Il est particulièrement important que l’objectif utilisé pour l’imagerie corresponde aux paramètres du logiciel d’imagerie lorsque le nombre de noyaux sera extrapolé de la zone imagée à l’ensemble de la zone du puits (ou de la lame). Cette étape doit avoir lieu à chaque changement d’objectif, mais elle est facilement négligée lors de la configuration du microscope et du logiciel. Le temps requis pour la section 1 varie en fonction d’un certain nombre de facteurs (p. ex., expérience de l’utilisateur avec la microscopie à fluorescence, nombre d’images et d’échantillons, microscope, etc.). Pour capturer 10 à 15 images de noyaux colorés par puits dans deux plaques de 6 puits, l’utilisateur doit réserver environ 1 à 2 h de temps.
Une fois les images capturées et enregistrées dans un dossier dédié, elles sont prêtes à être quantifiées. La méthode la plus simple de quantification consiste à exécuter ce programme automatisé pré-packagé sans avoir besoin d’installer Python ou des bibliothèques (section 2). Il convient de noter que le programme est préparé en tant qu’exécutable et a été empaqueté à l’aide de Pyinstaller. Les logiciels construits à l’aide de Pyinstaller sont généralement signalés par un logiciel antivirus. Si un logiciel antivirus bloque l’ouverture du fichier, l’utilisateur doit marquer le fichier comme sûr pour continuer. Ceci est essentiel pour mener à bien la section 2. Alternativement, n’importe quel utilisateur peut choisir d’exécuter le programme de quantification automatique comme un script Python (section 3) plutôt que comme un exécutable, s’il le souhaite. Si l’utilisateur souhaite exécuter le programme à l’aide d’un système d’exploitation Mac ou Linux, le programme doit être exécuté via Python. Cette méthode ne nécessite pas Pyinstaller, mais peut être plus difficile techniquement. Le protocole étape par étape ainsi que des exemples, détaillés dans la section 3, visent à supprimer les obstacles techniques à l’exécution de ce programme en tant que script. Que la section 2 ou la section 3 soit utilisée pour exécuter le programme, il est essentiel de s’assurer que le code est correctement entré dans les invites de commande. Des exemples de lignes de code sont inclus dans l’ensemble du protocole, et l’utilisateur doit prêter une attention particulière à l’espacement, aux guillemets et à d’autres signes de ponctuation. L’utilisateur doit également s’assurer que les chemins d’accès aux fichiers sont exacts. Il est utile d’avoir les chemins d’accès aux fichiers pour les images et la sortie disponibles dans un document Word. Les commandes peuvent également être saisies dans ce document Word et copiées/collées si nécessaire.
Comme décrit à la section 4, il existe plusieurs approches pour normaliser les données expérimentales à l’aide des dénombrements obtenus à partir de ce programme automatisé de quantification des noyaux. Pour les expériences basées sur l’image où les données expérimentales incluent un certain nombre d’événements dans chaque image et où une proportion est souhaitée (par exemple, la proportion de cellules V-positives à l’annexine28, la proportion de cellules exprimant les récepteurs ACE229, le nombre de cellules qui absorbent les particules colorées30, etc.), les données expérimentales de chaque image peuvent simplement être divisées par le nombre résultant (ce qui donne une proportion de 0 à 1). Ensuite, la proportion moyenne peut être calculée sur des images répliquées à partir de la même diapositive, du même plat ou du même puits. Si un pourcentage est souhaité, il suffit de multiplier les proportions finales par 100. Enfin, ces proportions ou pourcentages moyens peuvent être utilisés dans les analyses statistiques en aval. Lorsque les données expérimentales à normaliser proviennent d’expériences fonctionnelles sur des cellules vivantes (par exemple, taux de consommation d’oxygène6, 11, 12) ou d’un surnageant de culture cellulaire (par exemple, l’activité de la lactate déshydrogénase dans le surnageant31 ou la concentration d’une cytokine32 sécrétée spécifique, etc.), le nombre de noyaux doit d’abord être moyenné sur toutes les images capturées à partir du même puits. Ensuite, le nombre de noyaux peut être extrapolé à la surface de la lame (lamelle), du puits ou de la boîte, ou le nombre peut être exprimé sous forme de rapport par rapport à un échantillon ou à une condition de référence. L’extrapolation à la surface de la lame, du puits ou de la boîte est utile pour exprimer les données expérimentales en fonction du nombre de cellules (si l’on utilise des cellules uninucléées) à partir desquelles les données ont été dérivées (par exemple, oxygène consommé par minute pour 103 cellules). Pour utiliser cette méthode, la surface de la diapositive (lamelle), du puits ou de la parabole et la surface capturée dans chaque image doivent être connues. Si l’échantillon ne couvre pas toute la surface disponible, si les données expérimentales proviennent d’une petite quantité de surnageant (c’est-à-dire exprimées en activité ou en concentration par unité de volume), ou si l’image et/ou la surface ne sont pas connues, la meilleure option consiste à faire la moyenne du nombre de noyaux sur toutes les images d’un seul échantillon et à exprimer le nombre de noyaux par rapport à un échantillon ou à une condition de référence. La valeur résultante est utilisée pour corriger les données expérimentales pour la densité des noyaux ou la densité cellulaire (si vous utilisez des cellules uninucléées).
Bien que cette méthode soit simple à exécuter, il existe plusieurs problèmes potentiels dont les utilisateurs doivent être conscients. Ces problèmes potentiels, ainsi que les causes possibles et les étapes de dépannage pour les résoudre, sont répertoriés dans le Tableau 1. Le programme décrit ici présente des avantages clés par rapport aux logiciels existants ; Cependant, plusieurs limites doivent être reconnues. Le principal inconvénient est que le programme de quantification ne peut être utilisé qu’avec des images capturées à l’aide de la microscopie à fluorescence, et non des images en fond clair. Alors que des images colorées au DAPI capturées à un grossissement de 10x ont été utilisées pour construire le programme, le programme peut être utilisé avec d’autres colorants et grossissements d’ADN puisque le programme est conçu pour reconnaître la morphologie des noyaux plutôt que la couleur ou la taille. Bien que nous ayons validé l’exactitude du programme sur des images contenant diverses densités de cellules, l’utilisateur final peut avoir des densités de cellules qui se situent en dehors de la plage. Dans ce cas, nous prévoyons toujours des comptages précis à condition d’obtenir des images de qualité ; Cependant, l’utilisateur doit vérifier soigneusement les contours et la sortie pour s’assurer de leur exactitude. Pour les applications de culture cellulaire, la quantification des noyaux dans les images est la plus appropriée pour les cellules dans une monocouche. Si les cellules en culture se chevauchent, les noyaux peuvent être masqués, ce qui entraîne une sous-estimation de la quantité de noyaux. La nature bidimensionnelle de la quantification des noyaux dans les images exclut l’utilisation de ce programme pour déterminer le nombre total de noyaux à partir de cellules en suspension, de blocs de tissus épais ou d’autres applications tridimensionnelles. Cependant, ce programme peut toujours quantifier des noyaux marqués avec des colorants d’ADN fluorescents dans des images bidimensionnelles capturées à partir de modèles tridimensionnels tels que des blocs de tissus épais. L’utilité de quantifier les noyaux dans les images pour l’application souhaitée doit être déterminée par l’utilisateur final.
| Problème | Causes possibles | Solutions possibles |
| Arrière-plan excessif dans les images | Temps d’exposition trop élevé | Réduire le temps d’exposition |
| Les noyaux semblent fusionner les uns avec les autres dans les images | Concentration de colorant d’ADN fluorescent trop élevée ou trop de support de montage de colorant d’ADN | Réduire la concentration de colorant lors de l’optimisation ; Utilisez le moins de support de montage possible |
| Les noyaux semblent trop légers ou certains noyaux ne sont pas visibles | Temps d’exposition trop faible | Augmenter le temps d’exposition |
| Photoblanchiment | Prévenir le photoblanchiment en veillant à ce que les échantillons colorés restent stockés dans l’obscurité et en minimisant l’exposition des échantillons à l’éclairage (p. ex., en utilisant le temps d’exposition minimal nécessaire et en éteignant l’éclairage lorsqu’il n’est pas nécessaire, par exemple après la capture d’une image ou pendant la prise de notes) | |
| Effets de halo ou formes non spécifiques au noyau apparaissant | Temps d’exposition trop élevé | Réduire le temps d’exposition |
| Peluches provenant d’essuie-glaces délicats ou de certains protecteurs de paillasse dans des cellules vivantes ou adhérant à des cellules fixes | Assurer un environnement non pelucheux : réduire au minimum l’utilisation d’essuie-glaces délicats lorsque les échantillons sont exposés (p. ex., dans une enceinte de biosécurité pour les cellules vivantes, pendant la fixation et le séchage des cellules fixes, ou pendant le processus de coloration) ; Assurez-vous que les protecteurs de paillasse ne laissent pas de peluches sur les lames ou les plaques pendant la coloration | |
| Autres anomalies à la surface des cellules/tissus fixés | Manipulez les échantillons avec le plus grand soin et suivez les précautions appropriées et les meilleures pratiques lors de la fixation et de la coloration | |
| Perturbations du trajet de la lumière | Assurez-vous que toutes les parties du microscope sont propres et exemptes de peluches et de poussière ; Nettoyez l’extérieur des lames/plaques/plats avec de l’éthanol à 70 % pour éliminer la poussière et les peluches environnementales avant l’imagerie | |
| Le fichier exécutable ne s’ouvre pas | Un logiciel antivirus a bloqué le fichier | Marquer le fichier comme sûr |
| Erreurs dans les invites de commande | Problèmes d’espacement en ligne de commande | Vérifiez que le début de la commande est tapé immédiatement après le symbole « > » dans la ligne de commande sans espace, qu’il n’y a pas d’espace après les guillemets ouverts pour les chemins d’accès aux fichiers, et que l’espacement et la correspondance correspondent au code fourni et aux exemples. |
| Cas inappropriés utilisés en ligne de commande | Le code est sensible à la casse ; Assurez-vous d’utiliser des majuscules et des minuscules le cas échéant | |
| Problèmes avec le chemin d’accès au fichier | Assurez-vous que le chemin d’accès au fichier indique l’intérieur du dossier de fichiers associé (c’est-à-dire que le dossier de fichiers est ouvert lorsque le chemin d’accès au fichier est copié). | |
| Problèmes avec le programme qui ne sont pas résolus à l’aide des étapes de dépannage ci-dessus | Problèmes de logiciel ou de ligne de commande mentionnés ci-dessus. | Soumettez un problème via GitHub. Accédez à https://github.com/rbudnar/nuclei_counter. Dans la barre de menu en haut, sélectionnez « Problèmes » (deuxième option, à droite de « Code »). Cliquez sur le bouton vert sur le côté droit qui indique « Nouveau problème » à soumettre. Il y aura une invite à se connecter à GitHub avant de soumettre le problème. Il y aura également une option pour créer un compte gratuit si nécessaire. |
Tableau 1 : Dépannage pour la capture d’images de qualité et l’exécution du flux de travail de quantification automatisée des noyaux.
Dans l’ensemble, les méthodes décrites ici fournissent des détails étape par étape pour utiliser le programme automatisé de quantification des noyaux que nous avons développé à l’aide de bibliothèques de vision par ordinateur et validé par rapport aux comptages manuels de plusieurs évaluateurs. Ce programme a été développé pour être utilisé par des scientifiques ayant différents niveaux de compétences technologiques liées au codage, est disponible gratuitement et a été validé spécifiquement pour la tâche de quantification des noyaux. Par rapport au comptage manuel, ce programme fera gagner un temps précieux à l’utilisateur final et fournira des comptages de noyaux précis et fiables pour la normalisation des données.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Le financement de ces travaux a été fourni par les National Institutes of Health/National Institute on Aging (R01AG084597 ; DEL et HYL) et par des fonds de démarrage de la Texas Tech University (DEL). Les auteurs tiennent à remercier les programmes Undergraduate Research Scholars et TrUE Scholars de l’Université Texas Tech pour leur soutien financier aux chercheurs de premier cycle qui ont contribué à ce travail (REH, MRD, CJM, AKW). Nous remercions également les Drs Lauren S. Gollahon et Michael P. Massett d’avoir gracieusement partagé leur espace et leur équipement de laboratoire.
matériels
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
Références
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon