Method Article
استخدام مكتبات رؤية الكمبيوتر لتبسيط القياس الكمي للنوى
In This Article
Summary
توضح هذه المقالة طرقا خطوة بخطوة لأتمتة القياس الكمي للنوى المستندة إلى الصور باستخدام برنامج قابل للتنفيذ مفتوح المصدر تم التحقق من صحته عبر مجموعة من كثافات الخلايا. يوفر هذا البرنامج بديلا يعالج الحواجز المتعلقة بالتكلفة ، وإمكانية الوصول للمستخدمين ذوي المهارات التكنولوجية المحدودة ، والتحقق من الصحة الخاصة بالتطبيق التي قد تحد من فائدة التقنيات الحالية.
Abstract
تتطلب فحوصات الخلايا الحية وتحليلات الخلايا المستندة إلى الصور تطبيع البيانات للتفسير الدقيق. الطريقة الشائعة الاستخدام هي تلطيخ النوى وتحديدها كميا ، متبوعا بتطبيع البيانات لعدد النوى. غالبا ما يتم التعبير عن عدد النوى هذا على أنه عدد خلايا للخلايا أحادية النواة. في حين أن القياس الكمي اليدوي يمكن أن يكون شاقا ويستغرق وقتا طويلا ، فقد لا يفضل جميع المستخدمين الطرق المؤتمتة المتاحة ، أو قد تفتقر إلى التحقق من صحة هذا التطبيق المحدد ، أو قد تكون باهظة التكلفة. هنا ، نقدم إرشادات خطوة بخطوة لالتقاط صور قابلة للقياس الكمي للنوى الملطخة ببقع الحمض النووي الفلورية وبالتالي تحديد النوى باستخدام برنامج آلي لعد الكائنات تم تطويره باستخدام مكتبات رؤية الكمبيوتر Python. نقوم أيضا بالتحقق من صحة هذا البرنامج عبر مجموعة من كثافات الخلايا. على الرغم من أن الوقت المحدد لتنفيذ البرنامج يختلف بناء على عدد الصور وأجهزة الكمبيوتر ، إلا أن هذا البرنامج يدمج ساعات العمل لعد النوى في ثوان لتشغيل البرنامج. بينما تم تطوير هذا البروتوكول باستخدام صور الخلايا الثابتة والملطخة ، يمكن أيضا قياس صور النوى الملطخة في الخلايا الحية وتطبيقات التألق المناعي باستخدام هذا البرنامج. في النهاية ، يوفر هذا البرنامج خيارا لا يتطلب درجة عالية من المهارة التكنولوجية وهو بديل مفتوح المصدر تم التحقق من صحته لمساعدة علماء الأحياء الخلوية والجزيئية في تبسيط سير عملهم ، وأتمتة المهمة الشاقة والمستهلكة للوقت المتمثلة في قياس النوى.
Introduction
تعد التجارب الوظيفية والقائمة على الصور أمرا بالغ الأهمية لفهم تأثيرات العلاجات التجريبية على الكيمياء الحيوية وعلم وظائف الأعضاء للخلية بأكملها. يعتمد التفسير الصحيح للبيانات من تجارب بيولوجيا الخلية على دقة وقابلية تكرار البروتوكول التجريبي ، بما في ذلك تطبيع البيانات. على سبيل المثال ، تسمح تحليلات استهلاك الأكسجين ومعدلات التحمض خارج الخلية في الخلايا الحية عند خط الأساس وبعد العلاج بأدوية معينة بتقييم الجوانب المختلفة لعملية التمثيل الغذائي للطاقة1،2. يمكن أن يساعد قياس نشاط الإنزيمات مثل نازعة هيدروجين اللاكتات في المادة الطافية لمزارع الخلايا في تحديد سلامة غشاء الخلية3. يسمح تلطيخ الخلايا المستنبتة بالملحق V ويوديد البروبيديوم قبل التثبيت بتقييم الخلايا المبرمجة والميتة4. ومع ذلك ، فإن الاختلافات في كثافة الخلايا عبر الآبار تؤثر على نتائج كل من هذه المقايسات. يمكن أن يؤدي الاعتماد فقط على كثافة البذر إلى نتائج مضللة بسبب الأخطاء في عد الخلايا للبذر ، أو الاختلافات في كثافة الخلايا في الوسائط أثناء الطلاء ، أو معدلات مختلفة لنمو الخلايا بين العينات أو العلاجات خلال التجارب. لذلك ، يلزم تطبيع النتائج التجريبية.
تتضمن طرق التطبيع الحالية لبيانات الخلية الوظيفية والقائمة على الصور تركيز البروتين5 أو عدد النوى أوالخلايا 6. لتطبيع البيانات إلى تركيز البروتين ، يجب استخراج الخلايا بكميات متساوية من المخزن المؤقت للتحلل باستخدام مثبط الإنزيم البروتيني بعد تشغيل الفحص ، ويجب إجراء فحص إضافي (على سبيل المثال ، مقايسة حمض البيسينشونيك ، ومقايسة برادفورد ، وما إلى ذلك) لقياس البروتين. تعتمد دقة هذه الطريقة على استخراج الخلايا بالكامل. ترك أي بروتين وراءه يساهم في حدوث أخطاء في القياس الكمي. يوفر عد النوى أو الخلايا بديلا حيث لا يلزم إزالة الخلايا من السطح الذي تنمو عليه. بدلا من ذلك ، يتم تلطيخ الخلايا الحية أو الثابتة أو تلطيخها باستخدام البقع اللونية أو الفلورية ، ويمكن إجراء العد بناء على صور الخلايا الكاملة أو النوى فقط. يفضل تحديد النوى على الخلايا الكاملة لتطبيقات الفحص المجهري الفلوري ولتطبيع البيانات في الخلايا المنصهرة (مثل الأنابيب العضلية) أو الأنسجة. لتقدير النوى باستخدام نهج التألق ، يتم تطبيق أصباغ الحمض النووي الفلورية (على سبيل المثال ، 4 ، 6-diamidino-2-phenylindole dichloride (DAPI) ، Hoechst 33342 ، Hoechst 33358 ، DRAQ5 ، إلخ) على الخلايا الحية أو الثابتة وتصويرها باستخدام المجهر الفلوري. ترتبط DAPI و Hoechst و DRAQ5 بشكل تفضيلي بالمناطق الغنية بالأدينين الثايمين من الحمض النووي7،8،9 ، مما يوفر وسيلة للكشف عن النوى. يمنع تحسين تركيز الصبغة الارتباط خارج الهدف (أي بالحمض النووي للميتوكوندريا ، مما يتسبب في التألق العصاري الخلوي المنقط)10. يمكن تبسيط عملية التحسين باستخدام وسائط التثبيت المتاحة تجاريا التي تحتوي على DAPI للخلايا الثابتة (على سبيل المثال ، وسيط التركيب المضاد للتلاشي VECTASHIELD مع DAPI) والتركيزات المنشورة من البقع للخلايا الحية (على سبيل المثال ، Hoechst) 6،11،12. بمجرد تلطيخها ، يتم تصور النوى في الخلايا الحية أو الثابتة باستخدام مرشح الإثارة المناسب (على سبيل المثال ، الأشعة فوق البنفسجية [DAPI و Hoechst] أو الأحمر البعيد [DRAQ5]). الحد الأقصى للإثارة ل DAPI و Hoechst و DRAQ5 في أي من طرفي الطيف للفحص المجهري الفلوري. هذه السمة ، بالإضافة إلى خصوصية الحمض النووي لهذه الأصباغ واستخدامها بتركيزات مثالية ، تقلل من التألق خارج الهدف. تظهر الصور الناتجة نوى ساطعة على خلفية سوداء ، ويتم قياس هذه النوى كميا كمقياس لعدد الخلايا للخلايا أحادية النواة. في حين أن هناك طرق أخرى دقيقة قائمة على البقع لقياس النوى باستخدام البقع النسيجية والفحص المجهري الساطع13 ، فإن الأتمتة أكثر صعوبة ، وإن كانت ممكنة ، باستخدام هذه الأساليب.
في حين أنه لا يزال المعيار الذهبي ، فإن القياس الكمي اليدوي للنوى شاق ويستغرق وقتا طويلا وعرضة للخطأ البشري المحتمل مع العد المطول. على الرغم من وجود برامج عد الخلايا الآلي ، إلا أنها لا يفضلها جميع المستخدمين ، وقد تكون باهظة التكلفة ، وقد يكون التحقق من صحة تطبيقات معينة ضئيلا. أصبحت Python موردا قيما ويمكن الوصول إليه لعلماء الأحياء في السنوات الأخيرة. تعد مكتبات رؤية الكمبيوتر Python مفيدة بشكل خاص لتحليل الصور14. هنا ، نقدم إرشادات خطوة بخطوة لاستخدام برنامجنا القابل للتنفيذ الذي تم تطويره باستخدام كود Python لتحديد النوى الملطخة بأصباغ الفلورسنت وتصويرها باستخدام المجهر الفلوري. مهارات الترميز ليست ضرورية لاستخدام البرنامج الموضح هنا. سير العمل هذا خاص بالخلايا أو الأنسجة التي يتم تطبيق أصباغ الحمض النووي الفلورسنت. إنه غير مخصص للاستخدام مع صور المجال الساطع. يصف القسم 1 كيفية التقاط الصور التي تم جمعها وحفظها باستخدام الفحص المجهري الفلوري بطريقة متوافقة مع سير العمل هذا. يوفر القسم 2 تعليمات لتشغيل برنامج القياس الكمي للنوى كإخراج قابل للتنفيذ والجلب. يمكن تشغيل البرنامج القابل للتنفيذ مباشرة من الملف .exe المقدم ولا يتطلب أي تعديلات لتحديد النوى في الصور عالية الجودة. يتطلب هذا الملف القابل للتنفيذ جهاز كمبيوتر يعمل بنظام Windows. يوفر القسم 3 إرشادات لتشغيل البرنامج كبرنامج نصي Python بدلا من ملف قابل للتنفيذ (مطلوب للأنظمة المستندة إلى Mac أو Linux ، اختياري للأنظمة المستندة إلى Windows) ويسمح بضبط الكود إذا رغبت في ذلك. يصف القسم 4 كيفية استخدام النتائج للتطبيع. تتبع هذه البروتوكولات نتائج تمثيلية ، بما في ذلك بيانات التحقق من الصحة من مختبرنا.
Protocol
ملاحظة: يمكن العثور على الملفات التكميلية على الرابط التالي https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. التقاط الصور وحفظها باستخدام المجهر الفلوري
- قم بإعداد عينات الخلايا أو الأنسجة ليتم تصويرها ، بما في ذلك تلطيخ صبغة الحمض النووي المرغوبة. للحصول على الصور المستخدمة هنا ، نمت الخلايا العضلية C2C12 (CRL-1772 ، مجموعة الثقافة الأمريكية من النوع الأمريكي) في ألواح مكونة من 6 آبار لمدة 48-72 ساعة في ظل ظروف الاستزراع القياسية (5٪ ثاني أكسيد الكربون2 ، 37 درجة مئوية ، مرببة) ، مع أو بدون 50 ملي مولار EtOH6 ، ومثبتة في الميثانول المثلج كما هو موضح سابقا13. تم تركيب الخلايا الثابتة باستخدام وسيط تركيب يحتوي على DAPI15 وتجفيفها في الظلام قبل التصوير.
- قم بتشغيل جميع مكونات المجهر الفلوري والكمبيوتر وفقا لإجراءات التشغيل القياسية المعمول بها. افتح البرنامج المرتبط بالمجهر.
- اضبط المجهر لتصوير العينات من خلال العدسة. تحقق من الهدف. إذا لزم الأمر ، أدر قطعة الأنف إلى الهدف المطلوب. تأكد من أن البرنامج لديه الهدف المطلوب المحدد للحصول على إشارة دقيقة لمنطقة الصورة. هنا ، تم التقاط الصور باستخدام هدف 10x.
- مع إطفاء الأنوار ، ضع العينة (شريحة أو طبق أو طبق) على المسرح. قم بتشغيل التصوير المباشر باستخدام الفلتر المناسب واضبط وقت التعريض إما على مدة محددة مسبقا أو على التعريض الضوئي التلقائي. هنا ، تم استخدام مرشح DAPI ووقت التعرض من 61-62 مللي ثانية. راجع جدول استكشاف الأخطاء وإصلاحها للحصول على إرشادات حول تعيين وقت التعريض الضوئي، خاصة للصور ذات الكثافة العالية للخلايا.
- باستخدام العدسة ، اضبط التركيز ببطء باستخدام مقبض الضبط الإجمالي حتى تظهر النوى الملطخة ، ثم استخدم مقبض الضبط الدقيق لضبط التركيز ببطء حتى تظهر النوى واضحة ونقية.
ملاحظة: يتم عرض أمثلة على الصور المناسبة في الشكل 1.- إذا ظهرت تلطيخ مفرط في الخلفية أو إذا بدت النوى باهتة جدا، فاضبط وقت التعريض الضوئي يدويا. في صورة واحدة ، قد تكون هناك اختلافات واضحة في شدة الإشارة لبعض النوى. ومع ذلك ، يجب أن يكون الشكل الدائري مرئيا لجميع النوى مع الحد الأدنى من تداخل الإشارة.
ملاحظة: البرنامج المستخدم في تطبيقات المصب قوي بدرجات شدة مختلفة. تتضمن الصور الأولية المستخدمة لتطوير برنامج القياس الكمي للنوى والتحقق من صحته صورا ذات مجموعة من شدة الإشارة وهي متاحة من خلال مستودع Open Science Framework (انظر الرابط المذكور أعلاه).
- إذا ظهرت تلطيخ مفرط في الخلفية أو إذا بدت النوى باهتة جدا، فاضبط وقت التعريض الضوئي يدويا. في صورة واحدة ، قد تكون هناك اختلافات واضحة في شدة الإشارة لبعض النوى. ومع ذلك ، يجب أن يكون الشكل الدائري مرئيا لجميع النوى مع الحد الأدنى من تداخل الإشارة.
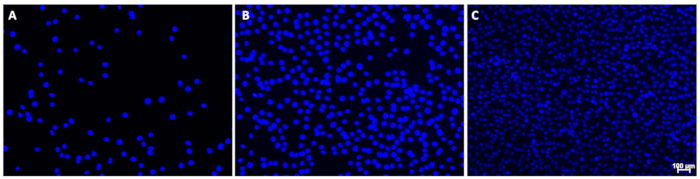
الشكل 1: أمثلة على صور النوى الملطخة ب DAPI. تصور هذه الصور النوى عند كثافة الخلايا (A) منخفضة أو (B) متوسطة أو (C) عالية مع الحد الأدنى من الخلفية. لاحظ كثافة الإشارة المتغيرة للنوى عبر الصورة C. هذا لم يتعارض مع القياس الكمي. يشير شريط المقياس (C) إلى 100 ميكرومتر. يجب حذف أشرطة المقياس من الصور المستخدمة لقياس النوى. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- بمجرد أن تصبح النوى في نطاق التركيز البؤري من خلال العدسة العينية ، قم بتبديل وضع الكشف إلى الكاميرا. اضبط التركيز باستخدام مقبض الضبط الدقيق.
- اضبط وقت التعرض إذا لزم الأمر. لاحظ وقت التعريض النهائي للاستخدام مع العينات والصور اللاحقة.
ملاحظة: على الرغم من أن استخدام نفس وقت التعريض الضوئي عبر جميع الصور يعد مثاليا، إلا أن الاختلافات في وقت التعريض مقبولة لهذا التطبيق لأن شدة التألق لا يتم قياسها كميا أو مقارنتها عبر العينات. - التقط الصورة باستخدام زر التقاط الصورة المناسب في البرنامج. إذا كانت العينات تحتوي على بقع متعددة مطبقة، فقم بتبديل الفلتر (على سبيل المثال، إلى FITC أو TRITC أو Cy5) وقم بتصوير نفس المنطقة لأغراض الدمج.
- ضع الغالق لتقليل تعرض العينة للضوء. بدون النسخ في شريط المقياس، احفظ الصورة بتنسيق ملف شكل عالمي (.tif أو .png أو .jpg) في مجلد مخصص. تأكد من أن كل اسم ملف صورة وصفي بشكل مناسب أو أن أي ترميز مستخدم لأسماء الملفات يحتوي على وثائق مرتبطة مناسبة.
ملاحظة: سيصبح هذا دليل الصور لبرنامج القياس الكمي. إذا تم استخدام التصوير متعدد الألوان، فقم بنسخ صور النوى فقط إلى مجلد منفصل ومخصص. يمكن القيام بذلك أيضا في نهاية جلسة التصوير. عادة ما تكون الصور التي يتم تصديرها باستخدام إعدادات التصدير الافتراضية في معظم برامج التصوير ذات دقة كافية للاستخدام مع هذا البرنامج، بشرط التقاط صور عالية الجودة. ومع ذلك ، يجب فحص الصور بصريا بعد التصدير للتأكد من أن مورفولوجيا النوى واضحة. إذا تم حفظ الصور بتنسيق .jpg ، فيجب توخي الحذر لضمان عدم ضغط الصور بشكل مفرط والحفاظ على الجودة. - افتح الغالق وارجع إلى التصوير المباشر وامسح ضوئيا عبر العينة إلى موقع آخر للتصوير. كرر الخطوات 1.8-1.9 حسب الحاجة. بالنسبة لصور الخلايا المصنفة في ألواح ذات 6 آبار تم التقاطها بتكبير 10x ، التقط 10-15 صورة لكل بئر (اعتمادا على تناسق كثافة الخلية عبر منطقة العينة).
- بعد الحصول على جميع الصور لعينة واحدة، قم بتنفيذ أحد الخيارات التالية.
- انتقل إلى العينة التالية وكرر الخطوات 1.4-1.10.
- أوقف البروتوكول مؤقتا والعودة في وقت آخر. يجب أن تظل العينات محمية من الضوء. لا ينصح بهذا الخيار في حالة العمل مع الخلايا الحية.
- بمجرد اكتمال التصوير ، تأكد من أن جميع العينات محمية من الضوء قبل تشغيل أضواء الغرفة. إذا تم حفظ المجلد الذي يحتوي على صور محليا، فقم بنقل الملفات إلى محرك أقراص مصغر أو موقع تخزين سحابي أو محرك أقراص شبكة آخر يمكن الوصول إليه من قبل الكمبيوتر حيث سيتم تشغيل برنامج القياس الكمي. حفظ في مجلد مخصص.
- أغلق جميع أجزاء المجهر والكمبيوتر وفقا لإجراءات التشغيل القياسية المعمول بها.
- اكتمل التصوير الآن. قم بتحليل الملفات باستخدام برنامج القياس الكمي التلقائي كما هو موضح في القسم 2 أو، بدلا من ذلك، عن طريق تشغيل البرنامج كبرنامج نصي Python كما هو موضح في القسم 3.
2. تشغيل برنامج القياس الكمي الآلي للنوى كملف قابل للتنفيذ
ملاحظة: هذه الطريقة، التي تقوم بتشغيل البرنامج كملف قابل للتنفيذ، متوافقة مع أجهزة الكمبيوتر التي تعمل بنظام التشغيل Windows وهي الطريقة الموصى بها لمستخدمي نظام التشغيل Windows. يجب على نظام التشغيل Mac OS أو غيرهم من المستخدمين غير التابعين لنظام التشغيل Windows تشغيل البرنامج كبرنامج نصي Python (انظر القسم 3).
- تأكد من تثبيت Python على الكمبيوتر. إذا لزم الأمر ، قم بتثبيت Python من python.org/downloads (مفضل) أو باستخدام Anaconda أو Miniconda. اعتبارا من تاريخ النشر ، تم اختبار هذا البرنامج باستخدام إصدارات Python 3.10-3.12.
- افتح المتصفح وانتقل إلى https://github.com/rbudnar/nuclei_counter/releases. قم بتنزيل أحدث إصدار من الملف المسمى count_nuclei.zip.
- من مجلد التنزيلات ، انقر بزر الماوس الأيمن فوق ملف Zip واستخراج جميع الملفات إلى الموقع المطلوب على الكمبيوتر المحلي (انظر الشكل 2 أ). احتفظ بموقع الملف هذا مفتوحا. سيتم استخدام مسار الملف هذا في الخطوة 2.5 (انظر الشكل 2 ب).
ملاحظة: يجب أن يظهر Count_nuclei الآن كملف تطبيق. هذا هو الملف القابل للتنفيذ. - افتح موجه الأوامر عن طريق البحث في CMD أو موجه الأوامر في شريط البحث. استخدم الأمر cd لتغيير الدليل إلى مسار الملف للملف القابل للتنفيذ (انظر الشكل 2C) واضغط على Enter. مثال على سطر الأوامر: C:\Users\janedoe>cd [مسافة بعد القرص المضغوط ، ثم نسخ/لصق مسار الملف]
- في سطر الأوامر التالي، اكتب ما يلي واضغط على Enter.
count_nuclei.exe - img_dir "المسار / إلى / الصور" - output_dir "المسار / إلى / الإخراج" - save_images - output_count_filename "results.csv"
حيث يكون path/to/images هو مسار الملف إلى المجلد الذي يحتوي على صور المراد تحليلها (انظر الخطوة 1.14)؛ path/to/output هو مسار الملف إلى المجلد حيث يجب حفظ الملف .csv (الإعدادات الافتراضية لدليل العمل الحالي عند عدم توفيرها) ؛ و results.csv هو اسم الملف الذي سيتم حفظ الأعداد الناتجة فيه (إذا لم يتم توفيره ، فإن اسم ملف الإخراج افتراضيا هو count_results.csv).
ملاحظة: هناك حاجة إلى علامات اقتباس في سطر الأوامر. تأكد من عدم وجود مسافات بعد علامات الاقتباس قبل مسار الملف (انظر الشكل 2C). - عند ظهور سطر الأوامر التالي، تكتمل المعالجة. تتوفر الآن الخطوط وجدول بيانات النتائج في الموقع المحدد كدليل الإخراج في الخطوة 2.5 (أي المسار / إلى/ الإخراج ؛ انظر الشكل 2 ج).
- افحص الخطوط بصريا للتحقق من جودة العد. يمكن الآن استخدام النتائج لتطبيع البيانات كما هو موضح في القسم 4.
ملاحظة: إذا بدا أن الخطوط قد فاتتها نوى (على سبيل المثال، تبدو النوى باهتة جدا) أو مجموعات عنقودية من النوى في عنصر واحد (على سبيل المثال، الصورة المعرضة للضوء المفرط، والكثير من الخلفية، والقطع الأثرية)، فيجب على المستخدم الرجوع إلى جدول استكشاف الأخطاء وإصلاحها وقد يحتاج إلى العودة إلى القسم 1 لتحسين الحصول على الصورة قبل الانتقال إلى القسم 4.
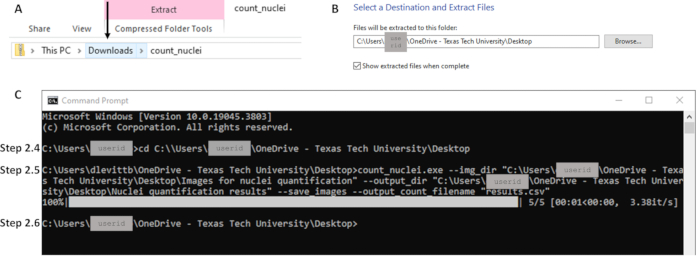
الشكل 2: أمثلة على الأجزاء الرئيسية لسير العمل للقسم 2. (أ) استخراج الملفات من مجلد التنزيلات، مع الحرص على النقر فوق المجلد الصحيح كما هو موضح بالسهم لتمكين الاستخراج (B) إلى الوجهة المطلوبة في الخطوة 2.3. (ج) أمثلة على أوامر الخطوتين 2.4 و 2.5 وسطر الأوامر التالي الذي يشير إلى الانتهاء في الخطوة 2.6. لاحظ أن مسار الملف في الخطوة 2.4 (C) يطابق موقع الاستخراج في الخطوة 2.3 (B). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
3. تشغيل برنامج القياس الكمي للنوى الآلي كبرنامج نصي Python
- تأكد من تثبيت Python على الكمبيوتر. إذا لزم الأمر ، قم بتثبيت Python من python.org/downloads (مفضل) أو باستخدام Anaconda أو Miniconda. اعتبارا من تاريخ النشر ، تم اختبار هذا البرنامج باستخدام إصدارات Python 3.10-3.12.
- افتح المتصفح وانتقل إلى https://github.com/rbudnar/nuclei_counter. انقر فوق الزر Green Code ، ثم انقر فوق تنزيل ZIP لتنزيل مستودع التعليمات البرمجية (انظر الشكل 3 أ).
- استخدم الإرشادات التالية لنظام التشغيل Windows. انظر الخطوة 3.4 للأجهزة التي تعمل بنظام التشغيل Mac OS.
- من مجلد التنزيلات ، انقر بزر الماوس الأيمن فوق ملف وحدد استخراج الكل لاستخراج الملفات إلى كمبيوتر محلي (انظر الشكل 3B-C).
- في موقع الملف المستخرج ، افتح المجلد المسمى nuclei_counter-main. هذا هو مستودع التعليمات البرمجية.
ملاحظة: يجب تنزيل هذا فقط في المرة الأولى التي يتم فيها استخدام هذا البرنامج ؛ ما عليك سوى نقل الملفات المستخرجة إلى موقع شبكة يمكن الوصول إليه وتدوين هذا الموقع. - افتح موجه الأوامر عن طريق البحث في CMD أو موجه الأوامر في شريط البحث. إذا تم تثبيت Python بواسطة Anaconda أو Miniconda ، فافتح موجه Anaconda بدلا من موجه الأوامر القياسي. يمكن العثور على ذلك عن طريق كتابة Anaconda Prompt في شريط البحث.
- استخدم الأمر cd لتغيير الدليل إلى مستودع التعليمات البرمجية واضغط على Enter. انظر سطر التعليمات البرمجية أدناه ، وهو حساس لحالة الأحرف. تأكد من فتح المجلد المسمى nuclei_counter-main عند نسخ مسار الملف. يجب أن يكون الملف المسمى pyproject أو pyproject.toml مرئيا (انظر الشكل 3D). مثال على سطر الأوامر: C:\Users\janedoe>cd [مسافة بعد القرص المضغوط ، ثم نسخ/لصق مسار الملف]
- في سطر الأوامر التالي ، اكتب ما يلي ، بما في ذلك المسافة والنقطة (انظر الشكل 3D:
تثبيت النقطة -ه .
ملاحظة: يجب إكمال الخطوتين 3.3.5 و 3.3.6 فقط في المرة الأولى التي يتم فيها تشغيل هذا البرنامج على أي جهاز كمبيوتر. إذا تم الانتهاء من ذلك بالفعل، فانتقل إلى الخطوة 3.3.7. قد تستغرق عملية التثبيت بضع لحظات للتهيئة. - اضغط على Enter. يؤدي تشغيل هذا الأمر إلى تثبيت المكتبات المطلوبة لتشغيل هذا البرنامج النصي وتثبيت المستودع في وضع قابل للتحرير في الحدث ، وهناك حاجة إلى تعديلات التعليمات البرمجية (انظر الشكل 3 د).
- اكتب ما يلي في سطر الأوامر التالي لتغيير الدليل إلى دليل التعليمات البرمجية المصدر الرئيسي (انظر الشكل 3E):
القرص المضغوط "nuclei_counter" - في سطر الأوامر التالي ، اكتب ما يلي واضغط على Enter (انظر الشكل 3E):
python count_nuclei.py --img_dir "المسار / إلى / الصور" - output_dir "المسار / إلى / الإخراج" - save_images - output_count_filename "results.csv"
حيث يكون path/to/images هو مسار الملف إلى المجلد الذي يحتوي على صور المراد تحليلها (الخطوة 1.14)؛ path/to/output هو مسار الملف إلى المجلد حيث يجب حفظ الملف .csv (الإعدادات الافتراضية لدليل العمل الحالي عند عدم توفيرها) ؛ و results.csv هو اسم الملف الذي سيتم حفظ الأعداد الناتجة فيه (إذا لم يتم توفيره ، فإن اسم ملف الإخراج افتراضيا هو count_results.csv).
ملاحظة: هناك حاجة إلى علامات اقتباس في سطر الأوامر. تأكد من عدم وجود مسافات بعد علامات الاقتباس قبل مسار الملف. سيقرأ البرنامج النصي python الملفات الموجودة في مسار الملف المقدم وينفذ برنامج عد النوى لجميع الصور الموجودة في الدليل (انظر الشكل 3E). سيتم حفظ الأعداد الناتجة في ملف النتائج .csv في الموقع المحدد. - عندما يظهر سطر الأوامر التالي ، تكتمل المعالجة (انظر الشكل 3E). تتوفر الآن الخطوط العريضة وجدول بيانات النتائج في الموقع المحدد كدليل الإخراج في الخطوة 3.3.8 (أي المسار / إلى/ الإخراج).
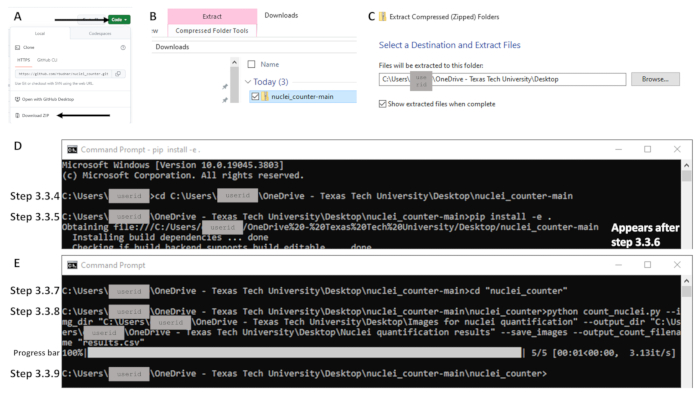
الشكل 3: أمثلة على الأجزاء الرئيسية لسير العمل للقسم 3 باستخدام نظام التشغيل Windows. للوصول إلى الرمز ، انقر فوق الزر الأخضر المسمى Code و (A) قم بتنزيل ملف Zip (المشار إليه بالأسهم). (ب) قم بالاستخراج بالنقر بزر الماوس الأيمن على ملف Zip (الموضح باللون الأزرق) و (C) وتعيين الوجهة المطلوبة. (د) بعد فتح موجه الأوامر، أمثلة على الأوامر الخاصة بالخطوتين 3-3-4 و3-3-5. لاحظ أن الأمر الخاص بالخطوة 3.3.5 هو كل شيء في سطر واحد. يظهر النص الذي يبدأ "الحصول على ملف" في السطر التالي بعد الضغط على Enter (الخطوة 3.3.6 ؛ أي تشغيل الأمر). (ه) أمثلة على أوامر الخطوتين 3.3.7 و 3.3.8 ، وشريط التقدم بعد تنفيذ الخطوة 3.3.8 ، وسطر الأوامر الذي يوضح الإكمال في الخطوة 3.3.9. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- استخدم الإرشادات التالية لنظام التشغيل Mac OS.
- من مجلد التنزيلات ، انقر فوق قائمة ملف وحدد فتح لاستخراج الملفات إلى الكمبيوتر المحلي (انظر الشكل 4 أ).
- في موقع الملف المستخرج ، افتح المجلد المسمى nuclei_counter-main. هذا هو مستودع التعليمات البرمجية.
ملاحظة: يجب تنزيل هذا فقط في المرة الأولى التي يتم فيها استخدام هذا البرنامج ؛ ما عليك سوى نقل الملفات المستخرجة إلى موقع شبكة يمكن الوصول إليه وتدوين هذا الموقع. - افتح محطة بالضغط على command + مفتاح المسافة الذي يفتح الأضواء ؛ ثم ابحث عن "terminal".
- استخدم الأمر cd لتغيير الدليل إلى مستودع التعليمات البرمجية واضغط على Enter. انظر سطر التعليمات البرمجية أدناه ، وهو حساس لحالة الأحرف. اكتب مسار الملف في مستند نصي وانسخه إلى المحطة الطرفية. تأكد من أن مسار الملف يبدأ بالنص الذي يسبق القرص المضغوط مباشرة (على سبيل المثال ، /Users/userid في الشكل 4 ب). مثال على سطر الأوامر: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- في سطر الأوامر التالي ، ستظهر علامة الدولار بعد معرف المستخدم (انظر الشكل 4 ب). تأكد من وجود مسافة بعد علامة الدولار، واكتب ما يلي، بما في ذلك المسافة والنقطة في نهاية الأمر. قم بتضمين إصدار Python المناسب مباشرة بعد النقطة بدون مسافة: pip3 install -e .
ملاحظة: يجب إكمال الخطوتين 3.4.5 و 3.4.6 فقط في المرة الأولى التي يتم فيها تشغيل هذا البرنامج على أي جهاز كمبيوتر. إذا تم إكمال ذلك بالفعل، فانتقل إلى الخطوة 3.4.7. قد تستغرق عملية التثبيت بضع لحظات للتهيئة. - اضغط على Enter. يؤدي تشغيل هذا الأمر إلى تثبيت المكتبات المطلوبة لتشغيل هذا البرنامج النصي وتثبيت المستودع في وضع قابل للتحرير في التعليمات البرمجية للحدث ، وهناك حاجة إلى تعديلات.
- في سطر الأوامر التالي ، تأكد من وجود مسافة بعد علامة الدولار ، ثم اكتب ما يلي لتغيير الدليل إلى دليل التعليمات البرمجية المصدر الرئيسي (انظر الشكل 4C):
القرص المضغوط "nuclei_counter" - في سطر الأوامر التالي، اكتب سطر الأوامر أدناه. استبدل الرقم بعد Python برقم الإصدار الصحيح إذا كنت تستخدم إصدارا آخر غير 3. ثم اضغط على Enter (انظر الشكل 4C):
python3 count_nuclei.py --img_dir "المسار / إلى / الصور" - output_dir "المسار / إلى / الإخراج" - save_images - output_count_filename "results.csv"
حيث يكون path/to/images هو مسار الملف إلى المجلد الذي يحتوي على صور المراد تحليلها (الخطوة 1.14)؛ path/to/output هو مسار الملف إلى المجلد حيث يجب حفظ الملف .csv (الإعدادات الافتراضية لدليل العمل الحالي عند عدم توفيرها) ؛ و results.csv هو اسم الملف الذي سيتم حفظ الأعداد الناتجة فيه (إذا لم يتم توفيره ، فإن اسم ملف الإخراج افتراضيا هو count_results.csv).
ملاحظة: هناك حاجة إلى علامات اقتباس في سطر الأوامر. تأكد من عدم وجود مسافات بعد علامات الاقتباس قبل مسار الملف. سيقرأ البرنامج النصي Python الملفات الموجودة في مسار الملف المقدم وينفذ برنامج عد النوى لجميع الصور الموجودة في الدليل (انظر الشكل 4C). سيتم حفظ الأعداد الناتجة في ملف النتائج .csv في الموقع المحدد. - عندما يظهر سطر الأوامر التالي ، تكتمل المعالجة (انظر الشكل 4C). تتوفر الآن الخطوط وجدول بيانات النتائج في الموقع المحدد كدليل الإخراج في الخطوة 3.4.8 (أي path/to/output).
- افحص الخطوط بصريا للتحقق من جودة العد. يمكن الآن استخدام النتائج لتطبيع البيانات كما هو موضح في القسم 4.
ملاحظة: إذا بدا أن الخطوط قد فاتتها نوى (على سبيل المثال ، تبدو النوى باهتة جدا) أو مجموعات عنقودية من النوى في عنصر واحد (على سبيل المثال ، الصورة المعرضة بشكل مفرط ، والكثير من الخلفية ، والقطع الأثرية) ، فيجب على المستخدم الرجوع إلى الجدول 1 وقد يحتاج إلى العودة إلى القسم 1 لتحسين الحصول على الصور قبل الانتقال إلى القسم 4.
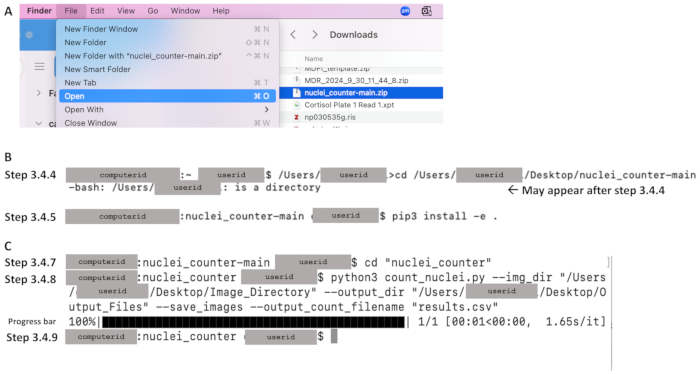
الشكل 4: أمثلة على الأجزاء الرئيسية من سير العمل للقسم 3 باستخدام نظام التشغيل Mac OS. بعد تنزيل الكود (انظر الشكل 3 أ) ، (أ) استخرج بالنقر فوق ملف Zip (الموضح باللون الأزرق) وتعيين الوجهة المطلوبة. (ب) بعد فتح المحطة الطرفية ، أمثلة على أوامر الخطوتين 3.4.4 (تغيير الدليل إلى مستودع التعليمات البرمجية) و 3.4.5 (تثبيت النقطة). (ج) أمثلة على أوامر الخطوتين 3.4.7 و 3.4.8 ، وشريط التقدم بعد تنفيذ الخطوة 3.4.8 ، وسطر الأوامر الذي يوضح الإكمال في الخطوة 3.4.9. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
4. استخدام التهم لتطبيع البيانات التجريبية
- افتح ملف results.csv الذي يربط كل صورة بقياس النوى المحدد باستخدام البرنامج الآلي.
- حدد طريقة التطبيع: 1) أساس كل صورة على حدة (على سبيل المثال ، إذا كان المجهر الفلوري متعدد الألوان) ، 2) تطبيع البيانات كنسبة من متوسط عدد النوى لكل عينة مقابل عينة مرجعية (أو شرط) ، 3) تطبيع عدد النوى لكل عينة (على سبيل المثال ، استقراء إجمالي عدد النوى إلى منطقة العينة). وترد التوصيات الخاصة بطريقة التطبيع في قسم المناقشة.
- إذا كان سيتم إجراء التطبيع على أساس كل صورة على حدة ، فما عليك سوى تقسيم البيانات التجريبية لكل صورة على عدد النوى لتلك الصورة.
- لتطبيع البيانات كنسبة، اتبع الخطوات الموضحة أدناه.
- بالنسبة لأي عينات تم فيها جمع صور متعددة لتكرار واحد (على سبيل المثال ، 10-15 صورة تم التقاطها من بئر واحد في صفيحة مكونة من 6 آبار) ، احسب متوسط عدد النوى لكل صورة. تعتبر أعداد النوى هذه على أنها عدد الخلايا بافتراض الخلايا أحادية النواة أو عدد من الخلايا المدمجة معا في خلايا متعددة النوى (مثل الأنابيب العضلية).
- حدد إما عينة مرجعية أو حالة مرجعية (على سبيل المثال، متوسط العد عبر جميع العينات من شرط التحكم) ولاحظ العدد.
- اقسم عدد جميع العينات على هذا العدد المرجعي، مما ينتج عنه قيمة التطبيع. إذا تم إستخدام عينة مرجعية، فاستخدم قيمة التسوية للعينة المرجعية ك 1.
ملاحظة: إذا تم إستخدام شرط مرجعي، فسيكون متوسط قيمة التسوية عبر الشرط الآن 1. العينات التي تحتوي على نوى أقل من المرجع (أي أقل كثافة) سيكون لها قيم تطبيع أقل من 1 ، والعينات التي تحتوي على نوى أكثر من المرجع (أي أكثر كثافة) سيكون لها قيم تطبيع أكبر من 1. - اقسم البيانات التجريبية لكل بئر على قيمة التسوية المحسوبة المقابلة.
- لتطبيع البيانات إلى عدد النوى لكل عينة، اتبع الخطوات الموضحة أدناه.
- بالنسبة لأي عينات تم فيها جمع صور متعددة لتكرار واحد (على سبيل المثال ، 10-15 صورة تم التقاطها من بئر واحد في صفيحة مكونة من 6 آبار) ، احسب متوسط عدد النوى لكل صورة. تعتبر أعداد النوى هذه على أنها عدد الخلايا بافتراض الخلايا أحادية النواة ، أو عدد الخلايا المدمجة معا في خلايا متعددة النوى (مثل الأنابيب العضلية).
- حدد مساحة سطح الشريحة (الغطاء) أو البئر أو الطبق الذي اشتقت منه البيانات. يمكن العثور على هذه المعلومات في قسم المواصفات على مواقع الويب الخاصة بالمنتجات.
- تحديد مساحة كل صورة تم التقاطها. غالبا ما تتوفر هذه المعلومات كبيانات وصفية في برنامج التصوير أو التحليل.
- تأكد من التعبير عن مساحة سطح العينة ومساحة الصورة بنفس الوحدات (على سبيل المثال ، مم2). إذا لم يكن كذلك ، فقم بالتحويل إلى الوحدة المطلوبة.
- احسب العدد الإجمالي لكل عينة باستخدام المعادلة التالية:
TC = AC • (SA / IA)
حيث TC هو العدد الإجمالي ، AC هو متوسط العدد ، SA هي مساحة السطح ، IA هي منطقة الصورة. - اقسم البيانات التجريبية على TC المقابل للعينة.
- تطبيع البيانات التجريبية واستخدامها في التحليلات الإحصائية حسب الاقتضاء.
النتائج
ينتج كل تشغيل للصور الدفعية: 1) مجموعة من ملفات الصور مع الخطوط المطبقة التي توضح الخطوط العريضة للنوى المحددة (الشكل 5) ، و 2) ملف .csv (جدول بيانات) يربط أسماء ملفات الصور والأعداد المرتبطة بها. سيسمح عرض الخطوط للمستخدم بتقييم جودة العد بصريا. على وجه التحديد ، يجب أن تحتوي الصور التي تم الحصول عليها وفقا للقسم 1 على جميع (أو كل تقريبا) النوى المحاطة بخط أخضر صلب يشير إلى أن النواة قد تم عدها بواسطة البرنامج. تم استخدام هذه الخطوط لضبط البرنامج هنا.
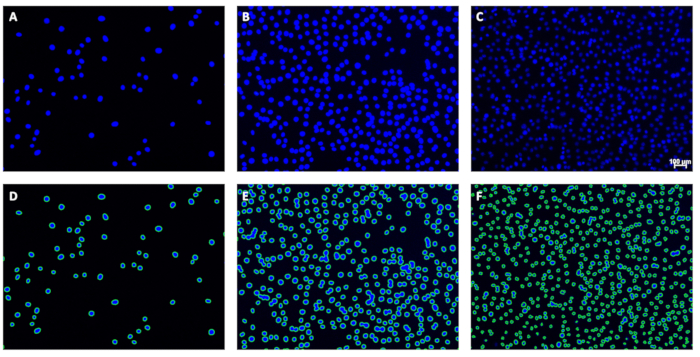
الشكل 5: أمثلة على الصور الأصلية والخطوط المقابلة. (أ-ج) أمثلة على الصور الأصلية قبل التحليل و (D-F) الخطوط المقابلة بعد التحليل أدناه مباشرة. يشير شريط المقياس (C) إلى 100 ميكرومتر. يجب حذف أشرطة المقياس من الصور المستخدمة لقياس النوى. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
التحقق من الصحة باستخدام النتائج الأولية
كانت الأرومات العضلية C2C12 بكثافات الخلايا المختلفة مثبتة بالميثانول ، وتم تلبطيخها باستخدام DAPI في وسائط التركيب ، وتم الحصول على 120 صورة بتكبير 10x بتنسيق ملف .jpg. تم تشغيل البرنامج التلقائي مع تعيين دليل الصور إلى المجلد الذي يحتوي على هذه الصور ، وتم حفظ ملف .csv الناتج. بشكل منفصل ، تم تحديد كل صورة يدويا من قبل باحثين مدربين كانا أعمى عن التعداد الآلي وتعداد بعضهما البعض. تتوفر الصور الأولية والخطوط التي تم إنشاؤها بواسطة البرنامج الآلي وجدول بيانات النتائج بما في ذلك التعداد اليدوي والآلي في مستودع إطار العلوم المفتوحة (OSF) (انظر الرابط في القسم 1). تم حساب معاملات الارتباط داخل الفئة (ICCs ؛ متوسط المقاييس) باستخدام إحصائيات SPSS لتحديد موثوقية المقيم بين 1) العددين اليدويين و 2) متوسط العددين اليدويين والعددين اللذين تم الحصول عليهما باستخدام البرنامج الآلي. تم استخدام مناهج مماثلة للتحقق من صحة البرامج الأخرى المصممة لتحديد الكائنات في الصور التي تم الحصول عليها باستخدام الفحص المجهري الفلوري16،17،18. تم حساب ICCs أيضا بين متوسط العد اليدوي والتعداد الآلي لكل ربع للتأكد من أن تقييم مجموعة البيانات بأكملها لا يخفي أي تغييرات محتملة في الموثوقية بناء على كثافة الخلايا. تم تحديد الأرباع باستخدام نقاط القطع المئوية 25و 50 و 75 بناء على متوسط العد اليدوي ، وتتراوح من الربع 1 (Q1 ؛ أقل كثافة للخلايا) إلى Q4 (أعلى كثافة للخلايا). يتم الإبلاغ عن المؤتمرات الدولية بناء على متوسط المقاييس وتفسيرها بناء على التعاريف التالية: ضعيف (ICC <0.50) ، متوسط (0.50 ≤ICC <0.75) ، جيد (0.75 ≤ICC <0.90) ، أو ممتاز (ICC ≥0.90)19.
تراوحت النوى لكل صورة من 44.5 إلى 1160 بناء على متوسطات العد اليدوي. وكانت موثوقية المقيم ممتازا بين العددين اليدويين (ICC > 0,999, p < 0,0001; الشكل 6 أ) وبين متوسط التعداد اليدوي والآلي (ICC = 0.993 ، ص < 0.0001 ؛ الشكل 6 ب). ظلت الموثوقية ممتازة عبر الأرباع (Q1: ICC = 0.996 ، ص < 0.0001 ؛ س 2: ICC= 0.997 ، ص < 0.0001 ؛ س 3: غرفة التجارة الدولية = 0.998 ، ص < 0.0001 ؛ س 4: غرفة التجارة الدولية = 0.986 ، ص < 0.0001 ؛ الشكل 6C-F). تمثل نقطة البيانات الفردية البعيدة عن أفضل خط ملاءمة في الشكل 6 ب الصورة الموضحة في الشكل 7. تحتوي الصورة على عدة مناطق من نوى متعددة مجمعة معا ، مما يمنع برنامج رؤية الكمبيوتر من التعرف على كل نواة. يمكن إصلاح هذه المشكلة باستخدام وقت تعرض أقصر. يصف الجدول 1 كيفية التعامل مع مثل هذه الصورة.
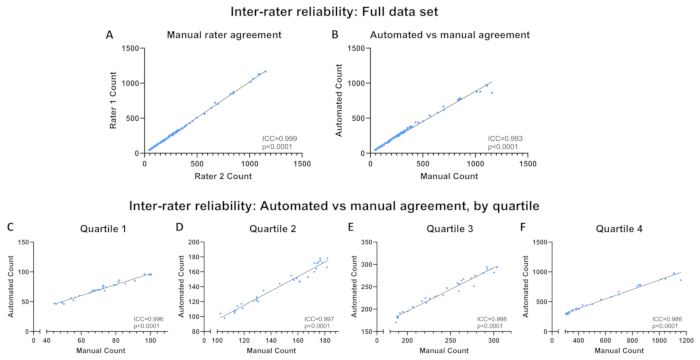
الشكل 6: الموثوقية بين المقيمين. الموثوقية بين المقيمين عبر مجموعة البيانات الكاملة (A) بين عدد المقيمين اليدوي و (B) بين الجرد الآلي ومتوسط العد اليدوي. (C-F) الموثوقية بين المقيمين بين العد الآلي ومتوسط العد اليدوي حسب الربع. الموثوقية معبرا عنها كمعامل ارتباط داخل الفئة (ICC). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 7: مثال على صورة بها حاجزان أمام القياس الكمي الدقيق للنوى. (أ) الصورة الأصلية و (ب) الخطوط المرتبطة بها. يشير السهم 1 إلى منطقة بها نوى متعددة متجمعة معا ، مما يشوه الشكل النووي الدائري بما يكفي لإعاقة اكتشاف البرامج للنوى المتعددة. يحدث هذا في عدة مواقع في جميع أنحاء هذه الصورة. يشير السهم 2 إلى هالة باهتة تظهر خلف نوى متعددة. بدت هذه الهالة ساطعة بما يكفي لتضمينها كنواة ، وبالتالي فقدان عدد النوى المتعددة التي يحيط بها. تمثل هذه الصورة نقطة البيانات الفردية البعيدة عن الخط الأنسب في الشكل 6 ب ، مع التركيز على المشاكل المحتملة مع هذه الحواجز. يشير شريط المقياس (A) إلى 100 ميكرومتر ؛ يجب حذف أشرطة المقياس من الصور المستخدمة لقياس النوى. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Discussion
يتمتع برنامج قياس النوى الخاص بنا بالعديد من المزايا مقارنة بالخيارات الحالية: فهو لا يتطلب سوى الحد الأدنى من المهارات التكنولوجية ، ويتم التحقق من صحته للمهمة المحددة المتمثلة في تحديد كمية النوى ، وهو مفتوح المصدر. هذا الأخير يتغلب على الحواجز المتعلقة بالتكلفة. في النهاية ، يوفر هذا البرنامج لعلماء الأحياء الخلوية والجزيئية خيارا إضافيا لتحديد النوى بسرعة ودقة في الصور الملتقطة باستخدام الفحص المجهري الفلوري. لا يفضل جميع المستخدمين برامج عد الخلايا أو النوى الآلية المتاحة حاليا ، وبعضها باهظ التكلفة ، وقد يكون التحقق ضئيلا أو غائبا لتطبيقات محددة. المعدات المتخصصة ، مثل خط BioTek Cytation ، مصحوبة بنشرات فنية تتحقق من صحة عد الخلايا في الموقع باستخدام التلوين النووي20 بالإضافة إلى مجموعة متنوعة من التطبيقات الإضافية. في حين أن بيانات التحقق من الصحة موجودة ويستخدم خط إنتاج Cytation على نطاق واسع ، خاصة عند التفاعل مع Seahorse Extracell Flux Analyzers لتطبيع بيانات الخلايا الحية بسهولة وكفاءة ، يمكن أن يكون هذا المنتج باهظ التكلفة للمختبرات ذات التمويل المحدود للمعدات المتاحة. على سبيل المثال ، تجاوزت قائمة الأسعار للنموذج الأساسي المجهز بأربعة أهداف وأربعة مكعبات ترشيح للفحص المجهري الفلوري 100,000 دولار أمريكي في السنة التقويمية 2024. بالنسبة لأولئك الذين ليس لديهم وسائل لشراء معدات متخصصة ، توفر البرامج مفتوحة المصدر بدائل مجانية لتحليل الصور. تتضمن البرامج مفتوحة المصدر المفيدة للنوى والخلايا ImageJ (المعاهد الوطنية للصحة ، بيثيسدا ، دكتوراه في الطب ، الولايات المتحدة الأمريكية ؛ متوفرة في 21. المكونات الإضافية متاحة الآن لأولئك الذين يرغبون في كتابة نصوص ماكرو لأتمتة معالجة الصور باستخدام منصة ImageJ. تم وصف أتمتة عد الخلايا المستندة إلى ImageJ باستخدام تباين الطور أو الفحص المجهري للحقل الساطع مع صور الخلية الكاملةسابقا 22،23. CellProfiler هو تطبيق منشور على نطاق واسع لتحليل صور الخلايا الملطخة بأصباغ الفلورسنت أو البقع النسيجية القياسية ، وغالبا ما يتم الاستشهاد بمقال مبكر يصف فائدته عبر مجموعة من التطبيقات البيولوجية على أنه التحققمن الصحة 24. ومع ذلك ، فإن هذه المخطوطة لا تتحقق من صحة أي اكتشاف بيولوجي معين ، مما يستلزم مزيدا من التحقق من الصحة والشفافية لخطوط أنابيب الأتمتة المختلفة المستخدمة ، مثل عد النوى25. علاوة على ذلك ، في حين أن CellProfiler عبارة عن حزمة برامج فعالة لتحليل الخلايا ، إلا أن جميع العلماء لا يجدونها سهلة الاستخدام. لذلك ، كنا نهدف إلى تطوير والتحقق من صحة برنامج آلي بديل لتحديد النوى باستخدام برامج مفتوحة المصدر. على وجه التحديد ، استخدمنا مكتبات رؤية الكمبيوتر الحالية في Python لتطوير سير العمل هذا.
تبدأ هذه الطريقة بالخلايا أو الأنسجة التي قام المستخدم النهائي بتلطيخها باستخدام بقع الحمض النووي مثل DAPI26. يتم توفير إرشادات عامة لالتقاط الصور وحفظها بطريقة متوافقة مع برامج الأتمتة. على الرغم من أن التفاصيل قد تختلف باختلاف المجاهر وبرامج التصوير، إلا أن سير العمل العام للقسم 1 هو نفسه للحصول على صور قابلة للقياس الكمي. في حالة استخدام عينات حية ، يجب توخي الحذر لضمان ارتداء معدات الحماية الشخصية المناسبة وبيئة التصوير المناسبة لتلك العينات (على سبيل المثال ، درجة الحرارة ، النسبة المئوية لثاني أكسيد الكربون2 ، إضافة المخزن المؤقت ، إلخ.)27. بينما يصف هذا البروتوكول تصوير أصباغ الحمض النووي فقط ، يمكن للمستخدمين التقاط ألوان أخرى من نفس المنطقة لأغراضالدمج 15 ، ونسخ الصور النووية ببساطة إلى مجلد منفصل لاستخدامها مع برنامج القياس الكمي. يجب إجراء التصوير في غرفة مظلمة مع معدات الوقاية الشخصية المناسبة للعينات الحية ويجب استخدام التطهير المناسب حسب الحاجة (على سبيل المثال ، مع 70٪ إيثانول). يجب حماية العينات الملطخة من الضوء قبل تشغيل الأضواء. يعد التقاط الصور عالي الجودة أمرا بالغ الأهمية لقياس النوى بدقة. حتى في الصور ذات وقت التعرض المناسب والتباين القوي بين النوى الساطعة والخلفية السوداء ، فإن وجود خلايا أو هالات متكتلة بشكل مفرط خلف مجموعات من الخلايا (انظر الشكل 7) قد يقلل من دقة القياس الكمي. في هذه الحالات ، قد تحتاج أوقات التعرض إلى التغيير لتقليل شدة الإشارات ، مما يضمن الحد الأدنى من دمج إشارات النوى وتقليل الهالات مع الحفاظ على إشارة قوية بما يكفي لاكتشاف الأشكال المستديرة للنوى. إذا كانت هذه الخطوات غير كافية، فقد يكون من الضروري تصوير قسم مجاور من العينة. يجب التقاط جميع الصور لتجربة واحدة بنفس التكبير ، ويجب ضبط التكبير المقابل للهدف المستخدم بشكل صحيح في برنامج التصوير. من الأهمية بمكان أن يتطابق الهدف المستخدم للتصوير مع الإعدادات الموجودة على برنامج التصوير عندما يتم استقراء عدد النوى من المنطقة المصورة إلى منطقة البئر (أو الشريحة) بأكملها. يجب أن تتم هذه الخطوة في كل مرة يتم فيها تبديل الهدف ولكن يمكن التغاضي عنها بسهولة عند إعداد المجهر والبرنامج. يختلف الوقت اللازم للقسم 1 بناء على عدد من العوامل (على سبيل المثال ، تجربة المستخدم مع الفحص المجهري الفلوري ، وعدد الصور والعينات ، والمجهر ، وما إلى ذلك). لالتقاط 10-15 صورة للنوى الملطخة لكل بئر في لوحين من 6 آبار ، يجب على المستخدم حجز ما يقرب من 1-2 ساعة من الوقت.
بمجرد التقاط الصور وحفظها في مجلد مخصص ، تكون جاهزة للقياس الكمي. إن أبسط طريقة للقياس الكمي هي تشغيل هذا البرنامج الآلي المعبأ مسبقا دون الحاجة إلى تثبيت Python أو أي مكتبات (القسم 2). وتجدر الإشارة إلى أن البرنامج تم إعداده كملف قابل للتنفيذ وتم تعبئته باستخدام Pyinstaller. عادة ما يتم وضع علامة على البرامج التي تم إنشاؤها باستخدام Pyinstaller بواسطة برنامج مكافحة الفيروسات. إذا قام برنامج مكافحة الفيروسات بحظر فتح الملف، فيجب على المستخدم وضع علامة على الملف على أنه آمن للمتابعة. هذا أمر بالغ الأهمية لتنفيذ القسم 2 بنجاح. بدلا من ذلك ، يمكن لأي مستخدم اختيار تشغيل برنامج القياس الكمي الآلي كبرنامج نصي Python (القسم 3) بدلا من ملف قابل للتنفيذ ، إذا رغبت في ذلك. إذا رغب المستخدم في تشغيل البرنامج باستخدام نظام تشغيل Mac أو Linux ، فيجب تشغيل البرنامج من خلال Python. لا تتطلب هذه الطريقة Pyinstaller ولكنها قد تكون أكثر صعوبة من الناحية الفنية. يهدف البروتوكول خطوة بخطوة إلى جانب الأمثلة ، المفصلة في القسم 3 ، إلى إزالة الحواجز التقنية التي تحول دون تشغيل هذا البرنامج كبرنامج نصي. سواء تم استخدام القسم 2 أو القسم 3 لتشغيل البرنامج ، فمن الأهمية بمكان التأكد من إدخال التعليمات البرمجية بشكل صحيح في موجهات الأوامر. يتم تضمين أمثلة على أسطر التعليمات البرمجية في جميع أنحاء البروتوكول ، ويجب على المستخدم الانتباه جيدا إلى التباعد وعلامات الاقتباس وعلامات الترقيم الأخرى. يجب على المستخدم أيضا التأكد من دقة مسارات الملفات. من المفيد أن تكون مسارات الملفات للصور والمخرجات متوفرة في مستند Word. يمكن أيضا كتابة الأوامر في مستند Word هذا ونسخها / لصقها حسب الحاجة.
كما هو موضح في القسم 4 ، هناك طرق متعددة لتطبيع البيانات التجريبية باستخدام الأعداد التي تم الحصول عليها من برنامج القياس الكمي للنوى الآلي هذا. بالنسبة للتجارب القائمة على الصور حيث تتضمن البيانات التجريبية عددا من الأحداث في كل صورة وتكون النسبة مطلوبة (على سبيل المثال ، نسبة الخلايا الإيجابية على شكل حرف V28 ، ونسبة الخلايا التي تعبر عن مستقبلات ACE229 ، وعدد الخلايا التي تمتص الجسيمات الملطخة30 ، وما إلى ذلك) ، يمكن ببساطة تقسيم البيانات التجريبية لكل صورة على العدد الناتج (مما يؤدي إلى نسبة من 0 إلى 1). بعد ذلك ، يمكن حساب متوسط النسبة عبر نسخ الصور من نفس الشريحة أو الطبق أو البئر. إذا كانت النسبة المئوية مطلوبة ، فما عليك سوى ضرب النسب النهائية في 100. أخيرا ، يمكن استخدام هذه النسب أو النسب المئوية المتوسطة في التحليلات الإحصائية النهائية. عندما تكون البيانات التجريبية المراد تطبيعها من التجارب الوظيفية في الخلايا الحية (على سبيل المثال ، معدلات استهلاك الأكسجين6،11،12) أو من طافي لزراعة الخلايا (على سبيل المثال ، نشاط نازعة هيدروجين اللاكتات في الطاف31 أو تركيز سيتوكين مفرزمعين 32 ، وما إلى ذلك) ، يجب أولا حساب متوسط عدد النوى عبر جميع الصور الملتقطة من نفس البئر. بعد ذلك ، يمكن استقراء عدد النوى إلى مساحة سطح الشريحة (غطاء الغطاء) ، البئر ، أو الطبق ، أو يمكن التعبير عن العدد كنسبة مقابل عينة أو حالة مرجعية. يعد الاستقراء إلى مساحة سطح الشريحة أو البئر أو الطبق مفيدا للتعبير عن البيانات التجريبية مقابل عدد الخلايا (في حالة استخدام خلايا أحادية النواة) التي اشتقت منها البيانات (على سبيل المثال ، الأكسجين المستهلك في الدقيقة لكل 103 خلايا). لاستخدام هذه الطريقة ، يجب معرفة مساحة سطح الشريحة (غطاء الغطاء) أو البئر أو الطبق والمنطقة الملتقطة في كل صورة. إذا كانت العينة لا تغطي مساحة السطح المتاحة بالكامل ، فإن البيانات التجريبية من كمية صغيرة من المادة الطافية (أي معبرا عنها كنشاط أو تركيز لكل وحدة حجم) ، أو الصورة و / أو مساحة السطح غير معروفة ، فإن الخيار الأفضل هو متوسط عدد النوى عبر جميع الصور لعينة واحدة والتعبير عن عدد النوى بالنسبة لعينة مرجعية أو حالة. يتم استخدام القيمة الناتجة لتصحيح البيانات التجريبية لكثافة النوى أو كثافة الخلية (في حالة استخدام خلايا أحادية النواة).
في حين أن هذه الطريقة سهلة التنفيذ ، إلا أن هناك العديد من المشكلات المحتملة التي يجب أن يكون المستخدمون على دراية بها. هذه المشكلات المحتملة ، جنبا إلى جنب مع الأسباب المحتملة وخطوات استكشاف الأخطاء وإصلاحها لحل المشكلات ، مدرجة في الجدول 1. البرنامج الموصوف هنا له مزايا رئيسية مقارنة بالبرامج الحالية. ومع ذلك ، يجب الاعتراف بالعديد من القيود. العيب الرئيسي هو أنه لا يمكن استخدام برنامج القياس الكمي إلا مع الصور الملتقطة باستخدام المجهر الفلوري ، وليس صور المجال الساطع. بينما تم استخدام الصور الملطخة ب DAPI التي تم التقاطها بتكبير 10x لبناء البرنامج ، يمكن استخدام البرنامج مع أصباغ وتكبيرات الحمض النووي الأخرى نظرا لأن البرنامج مصمم للتعرف على مورفولوجيا النوى بدلا من اللون أو الحجم. بينما تحققنا من دقة البرنامج على الصور التي تحتوي على مجموعة متنوعة من كثافات الخلايا ، قد يكون لدى المستخدم النهائي كثافة خلايا تقع خارج النطاق. في هذه الحالة ، ما زلنا نتوقع عمليات دقية بشرط الحصول على صور عالية الجودة ؛ ومع ذلك ، يجب على المستخدم التحقق بعناية من الخطوط والمخرجات لضمان الدقة. بالنسبة لتطبيقات زراعة الخلايا ، فإن القياس الكمي للنوى في الصور هو الأنسب للخلايا في طبقة واحدة. إذا تداخلت الخلايا المستنبتة ، فقد يتم إخفاء النوى ، مما يؤدي إلى التقليل من كمية النوى. تمنع الطبيعة ثنائية الأبعاد لقياس النوى في الصور استخدام هذا البرنامج لتحديد العدد الإجمالي للنوى من الخلايا المعلقة أو كتل الأنسجة السميكة أو غيرها من التطبيقات ثلاثية الأبعاد. ومع ذلك ، لا يزال بإمكان هذا البرنامج تحديد النوى المسماة بأصباغ الحمض النووي الفلورية في صور ثنائية الأبعاد تم التقاطها من نماذج ثلاثية الأبعاد مثل كتل الأنسجة السميكة. يجب أن يحدد المستخدم النهائي فائدة القياس الكمي للنوى في الصور للتطبيق المطلوب.
| مشكلة | الأسباب المحتملة | الحلول الممكنة |
| خلفية زائدة في الصور | وقت التعرض مرتفع جدا | تقليل وقت التعرض |
| يبدو أن النوى تندمج مع بعضها البعض في الصور | تركيز صبغة الحمض النووي الفلوري مرتفع جدا أو الكثير من وسائط تركيب صبغة الحمض النووي | تقليل تركيز الصبغة أثناء التحسين ؛ استخدم أقل قدر ممكن من وسائط التركيب |
| تبدو النوى خفيفة جدا أو بعض النوى غير مرئية | وقت التعرض منخفض جدا | زيادة وقت التعرض |
| التبييض الضوئي | منع التبييض الضوئي عن طريق ضمان بقاء العينات الملطخة مخزنة في الظلام وتقليل تعرض العينات للإضاءة (على سبيل المثال ، باستخدام الحد الأدنى من وقت التعرض اللازم وإيقاف الإضاءة عندما لا تكون هناك حاجة إليها ، مثل بعد التقاط الصورة أو أثناء تدوين الملاحظات) | |
| تأثيرات الهالة أو الأشكال غير الخاصة بالنواة التي تظهر | وقت التعرض مرتفع جدا | تقليل وقت التعرض |
| نسالة من مساحات المهام الحساسة أو بعض واقيات الطاولة في الخلايا الحية أو الالتصاق بالخلايا الثابتة | ضمان بيئة خالية من النسالة: تقليل استخدام مساحات المهام الدقيقة عند تعرض العينات (على سبيل المثال ، في خزانة السلامة البيولوجية للخلايا الحية ، أو أثناء تثبيت الخلايا الثابتة وتجفيفها ، أو أثناء عملية التلوين) ؛ تأكد من أن واقيات الطاولة لا تترك الوبر على الشرائح أو الأطباق أثناء التلوين | |
| تشوهات أخرى على سطح الخلايا / الأنسجة الثابتة | تعامل مع العينات بعناية فائقة واتبع الاحتياطات المناسبة وأفضل الممارسات أثناء التثبيت والتلطيخ | |
| اضطرابات في مسار الضوء | تأكد من أن جميع أجزاء المجهر نظيفة وخالية من الوبر والغبار ؛ نظف الجزء الخارجي من الشرائح / الأطباق / الأطباق باستخدام 70٪ من الإيثانول لإزالة الغبار البيئي والوبر قبل التصوير | |
| لن يتم فتح الملف القابل للتنفيذ | حظر برنامج مكافحة الفيروسات الملف | وضع علامة على الملف على أنه آمن |
| أخطاء في موجهات الأوامر | مشاكل في التباعد في سطر الأوامر | تحقق مرة أخرى من كتابة بداية الأمر مباشرة بعد الرمز ">" في سطر الأوامر بدون مسافة، وعدم وجود مسافات بعد علامات الاقتباس المفتوحة لمسارات الملفات، وأن التباعد ويتطابق مع التعليمات البرمجية المقدمة والأمثلة. |
| الحالات غير الصحيحة المستخدمة في سطر الأوامر | الشفرة حساسة لحالة الأحرف. تأكد من استخدام الأحرف الكبيرة والصغيرة حسب الاقتضاء | |
| مشاكل في مسار الملف | تأكد من أن مسار الملف يشير إلى داخل مجلد الملف المقترن (على سبيل المثال ، يكون مجلد الملف مفتوحا عند نسخ مسار الملف). | |
| مشاكل البرنامج التي لم يتم حلها باستخدام خطوات استكشاف الأخطاء وإصلاحها أعلاه | مشاكل البرامج أو سطر الأوامر المذكورة أعلاه. | أرسل مشكلة عبر GitHub. انتقل إلى https://github.com/rbudnar/nuclei_counter. في شريط القائمة في الجزء العلوي ، حدد "المشكلات" (الخيار الثاني ، على يمين "الرمز"). انقر فوق الزر الأخضر على الجانب الأيسر الذي يشير إلى "مشكلة جديدة" لإرسالها. ستكون هناك مطالبة بتسجيل الدخول إلى GitHub قبل إرسال المشكلة. سيكون هناك أيضا خيار لإنشاء حساب مجاني إذا لزم الأمر. |
الجدول 1: استكشاف الأخطاء وإصلاحها لالتقاط صور عالية الجودة وتشغيل سير عمل القياس الكمي للنوى التلقائي.
بشكل عام ، توفر الطرق الموضحة هنا تفاصيل خطوة بخطوة لاستخدام برنامج القياس الكمي الآلي للنوى الذي طورناه باستخدام مكتبات رؤية الكمبيوتر والتحقق من صحته مقابل العد اليدوي من مقيمين متعددين. تم تطوير هذا البرنامج لضمان قابلية الاستخدام من قبل العلماء بمستويات مختلفة من المهارات التكنولوجية المتعلقة بالترميز ، وهو متاح مجانا ، وتم التحقق من صحته خصيصا لمهمة القياس الكمي للنوى. بالمقارنة مع العد اليدوي ، سيوفر هذا البرنامج وقتا ثمينا للمستخدم النهائي ويوفر عددا دقيقا وموثوقا به للنوى لتطبيع البيانات.
Disclosures
يعلن أصحاب البلاغ عدم وجود تضارب في المصالح.
Acknowledgements
تم توفير التمويل لهذا العمل من قبل المعاهد الوطنية للصحة / المعهد الوطني للشيخوخة (R01AG084597; DEL و HYL) ومن خلال الصناديق الناشئة من جامعة تكساس للتكنولوجيا (DEL). يود المؤلفون أن يشكروا برامج الباحثين الجامعيين بجامعة تكساس للتكنولوجيا وعلماء TrUE على تقديم الدعم المالي للباحثين الجامعيين الذين ساهموا في هذا العمل (REH ، MRD ، CJM ، AKW). كما نشكر الدكتورين لورين إس جواهون ومايكل ب. ماسيت على تقاسم مساحة المختبر والمعدات الخاصة بهما.
Materials
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
References
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved