Method Article
Verwendung von Computer-Vision-Bibliotheken zur Optimierung der Zellkernquantifizierung
In diesem Artikel
Zusammenfassung
In diesem Artikel werden Schritt-für-Schritt-Methoden zur Automatisierung der bildbasierten Zellkernquantifizierung mithilfe eines ausführbaren Open-Source-Programms beschrieben, das über eine Reihe von Zelldichten validiert wurde. Dieses Programm bietet eine Alternative, die Barrieren in Bezug auf Kosten, Zugänglichkeit für Benutzer mit begrenzten technologischen Fähigkeiten und anwendungsspezifische Validierung beseitigt, die den Nutzen bestehender Technologien einschränken können.
Zusammenfassung
Lebendzell-Assays und bildbasierte Zellanalysen erfordern eine Datennormalisierung für eine genaue Interpretation. Eine häufig verwendete Methode ist die Färbung und Quantifizierung von Zellkernen, gefolgt von einer Datennormalisierung auf die Zellkernzahl. Diese Zellzahl wird oft als Zellzahl für einkernige Zellen ausgedrückt. Während die manuelle Quantifizierung mühsam und zeitaufwändig sein kann, werden die verfügbaren automatisierten Methoden möglicherweise nicht von allen Anwendern bevorzugt, sind möglicherweise nicht für diese spezielle Anwendung validiert oder können unerschwinglich sein. Hier stellen wir Schritt-für-Schritt-Anleitungen für die Erfassung quantifizierbarer Bilder von Zellkernen bereit, die mit fluoreszierenden DNA-Färbungen gefärbt wurden, und die anschließende Quantifizierung der Zellkerne mit einem automatisierten Softwareprogramm zur Objektzählung, das unter Verwendung von Python-Computer-Vision-Bibliotheken entwickelt wurde. Wir validieren dieses Programm auch für eine Reihe von Zelldichten. Obwohl die genaue Zeit für die Programmausführung je nach Anzahl der Bilder und der Computerhardware variiert, konsolidiert dieses Programm die Stunden der Arbeit zum Zählen von Kernen in Sekunden, damit das Programm ausgeführt werden kann. Während dieses Protokoll unter Verwendung von Bildern von fixierten, gefärbten Zellen entwickelt wurde, können mit diesem Programm auch Bilder von gefärbten Zellkernen in lebenden Zellen und Immunfluoreszenzanwendungen quantifiziert werden. Letztendlich bietet dieses Programm eine Option, die kein hohes Maß an technologischem Know-how erfordert und eine validierte Open-Source-Alternative darstellt, um Zell- und Molekularbiologen bei der Rationalisierung ihrer Arbeitsabläufe zu unterstützen und die mühsame und zeitaufwändige Aufgabe der Zellkernquantifizierung zu automatisieren.
Einleitung
Funktionelle und bildbasierte Experimente sind entscheidend für das Verständnis der Auswirkungen experimenteller Behandlungen auf die Biochemie und Physiologie der gesamten Zelle. Die valide Interpretation von Daten aus zellbiologischen Experimenten hängt von der Genauigkeit und Reproduzierbarkeit des Versuchsprotokolls ab, einschließlich der Datennormalisierung. So ermöglichen Analysen des Sauerstoffverbrauchs und der extrazellulären Versauerungsraten in lebenden Zellen zu Studienbeginn und nach der Behandlung mit spezifischen Arzneimitteln die Beurteilung verschiedener Aspekte des Energiestoffwechsels 1,2. Die Messung der Aktivität von Enzymen wie Laktatdehydrogenase im Überstand von Zellkulturen kann bei der Quantifizierung der Integrität der Zellmembran helfen3. Die Färbung kultivierter Zellen mit Annexin V und Propidiumiodid vor der Fixierung ermöglicht die Beurteilung apoptotischer und nekrotischer Zellen4. Unterschiede in der Zelldichte zwischen den Wells beeinflussen jedoch die Ergebnisse der einzelnen Assays. Sich ausschließlich auf die Aussaatdichte zu verlassen, könnte zu irreführenden Ergebnissen führen, da Fehler beim Zählen von Zellen für die Aussaat, Schwankungen der Zelldichte in Medien während der Beschichtung oder unterschiedliche Zellwachstumsraten zwischen Proben oder Behandlungen während der Experimente verursacht werden. Daher ist eine Normalisierung der experimentellen Ergebnisse erforderlich.
Zu den aktuellen Normalisierungsmethoden für funktionelle und bildbasierte Zelldaten gehören die Proteinkonzentration5 oder die Zellkern- oder Zellzahl6. Um die Daten auf die Proteinkonzentration zu normalisieren, müssen die Zellen nach dem Assay in gleichen Volumina Lysepuffer mit Proteaseinhibitor extrahiert werden, und ein zusätzlicher Assay (z. B. Bicinchoninsäure-Assay, Bradford-Assay usw.) muss zur Proteinquantifizierung durchgeführt werden. Die Genauigkeit dieser Methode beruht auf einer vollständigen Zellextraktion; Das Zurücklassen von Proteinen trägt zu Fehlern bei der Quantifizierung bei. Das Zählen von Zellkernen oder Zellen bietet eine Alternative, bei der keine Zellen von der Oberfläche entfernt werden müssen, auf der sie gezüchtet werden. Stattdessen werden lebende oder fixierte Zellen mit kolorimetrischen oder fluoreszierenden Färbungen gefärbt oder gegengefärbt, und die Zählung kann auf der Grundlage von Bildern ganzer Zellen oder nur von Zellkernen durchgeführt werden. Die Quantifizierung von Zellkernen wird für Anwendungen der Fluoreszenzmikroskopie und zur Normalisierung von Daten in fusionierten Zellen (z. B. Myotuben) oder Geweben gegenüber ganzen Zellen bevorzugt. Zur Quantifizierung von Zellkernen unter Verwendung eines Fluoreszenzansatzes werden fluoreszierende DNA-Farbstoffe (z. B. 4′,6-Diamidino-2-phenylindoldichlorid (DAPI), Hoechst 33342, Hoechst 33358, DRAQ5 usw.) auf lebende oder fixierte Zellen aufgebracht und mittels Fluoreszenzmikroskopie abgebildet. DAPI, Hoechst und DRAQ5 binden bevorzugt an adenin-thyminreiche Regionen der DNA 7,8,9 und bieten so ein Mittel zum Nachweis von Zellkernen. Die Optimierung der Farbstoffkonzentration verhindert eine Off-Target-Bindung (d. h. an mitochondriale DNA, die zu punktförmiger zytosolischer Fluoreszenz führt)10. Der Optimierungsprozess kann durch die Verwendung von kommerziell erhältlichen Einbettmedien, die DAPI enthalten, für fixierte Zellen (z. B. VECTASHIELD Antifade Eindeckmedium mit DAPI) und veröffentlichten Färbekonzentrationen für lebende Zellen (z. B. Hoechst) (z. B. Hoechst) vereinfacht werden6,11,12. Nach der Färbung werden Zellkerne in lebenden oder fixierten Zellen mit dem entsprechenden Anregungsfilter (z. B. Ultraviolett [DAPI und Hoechst] oder dunkelrot [DRAQ5]) sichtbar gemacht. Anregungsmaxima für DAPI, Hoechst und DRAQ5 liegen an beiden Enden des Spektrums für die Fluoreszenzmikroskopie. Dieses Attribut sowie die DNA-Spezifität dieser Farbstoffe und ihre Verwendung in optimalen Konzentrationen minimieren die Off-Target-Fluoreszenz. Die resultierenden Bilder zeigen helle Zellkerne vor schwarzem Hintergrund, und diese Zellkerne werden als Maß für die Zellzahl für einkernige Zellen quantifiziert. Während es andere genaue färbebasierte Methoden zur Quantifizierung von Zellkernen gibt, die histologische Färbungen und Hellfeldmikroskopie13 verwenden, ist die Automatisierung mit diesen Ansätzen schwieriger, wenn auch möglich.
Die manuelle Quantifizierung von Kernen ist zwar nach wie vor der Goldstandard, aber mühsam, zeitaufwändig und anfällig für potenzielle menschliche Fehler bei längerer Zählung. Obwohl es automatisierte Zellzählprogramme gibt, werden sie nicht von allen Benutzern bevorzugt, können unerschwinglich sein und die Validierung für bestimmte Anwendungen kann minimal sein. Python hat sich in den letzten Jahren zu einer wertvollen und zugänglichen Ressource für Biologen entwickelt. Python-Computer-Vision-Bibliotheken sind besonders nützlich für die Bildanalyse14. Hier finden Sie eine Schritt-für-Schritt-Anleitung für die Verwendung unseres ausführbaren Programms, das mit Python-Code entwickelt wurde, um Kerne zu quantifizieren, die mit Fluoreszenzfarbstoffen gefärbt und mit Fluoreszenzmikroskopie abgebildet wurden. Programmierkenntnisse sind nicht notwendig, um das hier beschriebene Programm zu nutzen. Dieser Arbeitsablauf ist spezifisch für Zellen oder Gewebe, auf die fluoreszierende DNA-Farbstoffe aufgetragen werden. Es ist nicht für die Verwendung mit Hellfeldbildern vorgesehen. In Abschnitt 1 wird beschrieben, wie mit der Fluoreszenzmikroskopie aufgenommene Bilder in einer Weise aufgenommen und gespeichert werden, die mit diesem Arbeitsablauf kompatibel ist. Abschnitt 2 enthält Anweisungen zum Ausführen des Kernquantifizierungsprogramms als ausführbare Datei und zum Abrufen der Ausgabe. Das ausführbare Programm kann direkt aus der bereitgestellten .exe Datei ausgeführt werden und erfordert keine Änderungen, um Kerne in qualitativ hochwertigen Bildern zu quantifizieren. Für diese ausführbare Datei ist ein Windows-basierter PC erforderlich. Abschnitt 3 enthält Anweisungen zum Ausführen des Programms als Python-Skript und nicht als ausführbare Datei (erforderlich für Mac- oder Linux-basierte Systeme, optional für Windows-basierte Systeme) und ermöglicht bei Bedarf die Optimierung des Codes. In Abschnitt 4 wird beschrieben, wie die Ergebnisse für die Normalisierung verwendet werden. Diesen Protokollen folgen repräsentative Ergebnisse, einschließlich Validierungsdaten aus unserem Labor.
Protokoll
HINWEIS: Ergänzende Dateien finden Sie unter folgendem Link https://osf.io/a2s4d/?view_only=2d1042eb8f7c4c4a84579fe4e84fb03c
1. Aufnehmen und Speichern von Bildern mit der Fluoreszenzmikroskopie
- Bereiten Sie Zell- oder Gewebeproben für die Abbildung vor, einschließlich der Färbung mit dem gewünschten DNA-Farbstoff. Um die hier verwendeten Bilder zu erhalten, wurden C2C12-Myoblasten (CRL-1772, American Type Culture Collection) in 6-Well-Platten für 48-72 h unter Standardkulturbedingungen (5% CO2, 37 °C, befeuchtet), mit oder ohne 50 mM EtOH6 gezüchtet und in eiskaltem Methanol fixiert, wie zuvor beschrieben13. Fixierte Zellen wurden unter Verwendung eines DAPI-haltigen Eindeckmediums15 eingehängt und vor der Bildgebung im Dunkeln getrocknet.
- Schalten Sie alle Komponenten des Fluoreszenzmikroskops und des Computers gemäß den festgelegten Standardarbeitsanweisungen ein. Öffnen Sie die mit dem Mikroskop verknüpfte Software.
- Stellen Sie das Mikroskop so ein, dass Proben durch das Okular sichtbar werden. Überprüfen Sie das Ziel. Drehen Sie den Objektivrevolver bei Bedarf auf das gewünschte Objektiv. Stellen Sie sicher, dass das Softwareprogramm das gewünschte Objektiv ausgewählt hat, um eine genaue Anzeige des Bildbereichs zu erhalten. Hier wurden Bilder mit einem 10-fach-Objektiv aufgenommen.
- Legen Sie die Probe (Objektträger, Teller oder Schale) bei ausgeschaltetem Licht auf den Tisch. Schalten Sie die Live-Bildgebung mit dem entsprechenden Filter ein und stellen Sie die Belichtungszeit entweder auf eine vorher festgelegte Dauer oder auf automatische Belichtung ein. Hier wurde ein DAPI-Filter und eine Belichtungszeit von 61-62 ms verwendet. In der Tabelle zur Fehlerbehebung finden Sie eine Anleitung zum Einstellen der Belichtungszeit, insbesondere bei Bildern mit hoher Zelldichte.
- Stellen Sie mit dem Okular den Fokus langsam mit dem Brutto-Einstellknopf ein, bis die gefärbten Kerne sichtbar sind, und stellen Sie dann mit dem Feineinstellknopf den Fokus langsam ein, bis die Kerne klar und scharf erscheinen.
HINWEIS: Beispiele für geeignete Bilder sind in Abbildung 1 dargestellt.- Wenn eine übermäßige Hintergrundfärbung auftritt oder wenn die Kerne sehr dunkel erscheinen, passen Sie die Belichtungszeit manuell an. In einem einzigen Bild kann es sichtbare Unterschiede in der Signalintensität für einige Kerne geben; Die runde Form sollte jedoch für alle Kerne mit minimalen Signalstörungen sichtbar sein.
HINWEIS: Das Programm, das in Downstream-Anwendungen verwendet wird, ist robust gegenüber unterschiedlichen Intensitäten. Rohbilder, die für die Entwicklung und Validierung des Kernquantifizierungsprogramms verwendet werden, umfassen Bilder mit einer Reihe von Signalintensitäten und sind über das Open Science Framework Repository verfügbar (siehe oben erwähnter Link).
- Wenn eine übermäßige Hintergrundfärbung auftritt oder wenn die Kerne sehr dunkel erscheinen, passen Sie die Belichtungszeit manuell an. In einem einzigen Bild kann es sichtbare Unterschiede in der Signalintensität für einige Kerne geben; Die runde Form sollte jedoch für alle Kerne mit minimalen Signalstörungen sichtbar sein.
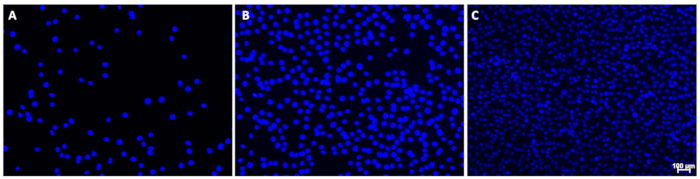
Abbildung 1: Beispielbilder von DAPI-gefärbten Zellkernen. Diese Bilder zeigen Zellkerne mit (A) niedriger, (B) mittlerer oder (C) hoher Zelldichte mit minimalem Hintergrund. Beachten Sie die unterschiedliche Signalintensität der Kerne in Bild C; Dies beeinträchtigte die Quantifizierung nicht. Der Maßstabsbalken (C) zeigt 100 μm an; Maßstabsbalken sollten bei Bildern, die für die Quantifizierung von Kernen verwendet werden, weggelassen werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Sobald die Kerne durch das Okular scharf gestellt sind, schalten Sie den Detektionsmodus auf die Kamera. Stellen Sie den Fokus mit dem Feineinstellknopf ein.
- Passen Sie die Belichtungszeit bei Bedarf an. Notieren Sie sich die endgültige Belichtungszeit für die Verwendung mit nachfolgenden Proben und Bildern.
HINWEIS: Obwohl die Verwendung der gleichen Belichtungszeit für alle Bilder ideal ist, sind Schwankungen der Belichtungszeit für diese Anwendung akzeptabel, da die Fluoreszenzintensität nicht quantifiziert oder über Proben hinweg verglichen wird. - Nehmen Sie das Bild mit der entsprechenden Bildaufnahmetaste in der Software auf. Wenn auf Proben mehrere Färbungen angewendet wurden, schalten Sie den Filter um (z. B. auf FITC, TRITC oder Cy5) und beleuchten Sie denselben Bereich zum Zusammenführen.
- Betätigen Sie den Verschluss, um die Lichteinwirkung der Probe zu minimieren. Speichern Sie das Bild in einem universellen Figurendateiformat (.tif, .png oder .jpg, ohne eine Maßstabsleiste einzubrennen, in einem speziellen Ordner. Stellen Sie sicher, dass jeder Bilddateiname angemessen beschreibend ist oder dass die für Dateinamen verwendete Codierung über eine entsprechende verknüpfte Dokumentation verfügt.
HINWEIS: Dies wird zum Bildverzeichnis für das Quantifizierungsprogramm. Wenn mehrfarbiges Imaging verwendet wurde, kopieren Sie nur Bilder von Zellkernen in einen separaten, dedizierten Ordner. Dies kann auch am Ende der Bildgebungssitzung erfolgen. Bilder, die mit den Standardexporteinstellungen der meisten Imaging-Software exportiert werden, haben in der Regel eine ausreichende Auflösung für die Verwendung mit diesem Programm, vorausgesetzt, es wurden qualitativ hochwertige Bilder aufgenommen. Die Bilder sollten jedoch nach dem Export visuell überprüft werden, um sicherzustellen, dass die Morphologie der Zellkerne klar ist. Wenn Bilder in .jpg Format gespeichert werden, sollte darauf geachtet werden, dass die Bilder nicht übermäßig komprimiert werden und die Qualität erhalten bleibt. - Öffnen Sie den Verschluss, kehren Sie zur Live-Bildgebung zurück und scannen Sie die Probe zur Bildgebung an eine andere Stelle. Wiederholen Sie die Schritte 1.8 bis 1.9 nach Bedarf. Für Bilder von Zellen, die in 6-Well-Platten ausgesät wurden, die bei 10-facher Vergrößerung aufgenommen wurden, nehmen Sie 10-15 Bilder pro Well auf (abhängig von der Konsistenz der Zelldichte im gesamten Probenbereich).
- Nachdem alle Bilder für ein einzelnes Beispiel erfasst wurden, führen Sie eine der folgenden Optionen aus.
- Fahren Sie mit dem nächsten Beispiel fort und wiederholen Sie die Schritte 1.4-1.10.
- Pausieren Sie das Protokoll und kehren Sie zu einem anderen Zeitpunkt zurück. Die Proben sollten vor Licht geschützt bleiben. Diese Option wird nicht empfohlen, wenn mit lebenden Zellen gearbeitet wird.
- Stellen Sie nach Abschluss der Bildgebung sicher, dass alle Proben lichtgeschützt sind, bevor Sie die Raumbeleuchtung einschalten. Wenn der Ordner mit den Bildern lokal gespeichert ist, übertragen Sie die Dateien auf einen USB-Stick, einen Cloud-Speicherort oder ein anderes Netzlaufwerk, auf das der Computer zugreifen kann, auf dem das Quantifizierungsprogramm ausgeführt wird. Speichern Sie in einem speziellen Ordner.
- Schalten Sie alle Teile des Mikroskops und des Computers gemäß den festgelegten Standardarbeitsanweisungen aus.
- Die Bildgebung ist nun abgeschlossen. Analysieren Sie die Dateien mit dem automatisierten Quantifizierungsprogramm, wie in Abschnitt 2 beschrieben, oder indem Sie das Programm als Python-Skript ausführen, wie in Abschnitt 3 beschrieben.
2. Ausführen des automatisierten Kernquantifizierungsprogramms als ausführbare Datei
HINWEIS: Diese Methode, bei der das Programm als ausführbare Datei ausgeführt wird, ist mit Windows-basierten PCs kompatibel und wird für Benutzer des Windows-Betriebssystems empfohlen. Mac OS oder andere Nicht-Windows-Benutzer sollten das Programm als Python-Skript ausführen (siehe Abschnitt 3).
- Stellen Sie sicher, dass Python auf dem Computer installiert ist. Installieren Sie bei Bedarf Python von python.org/downloads (bevorzugt) oder mit Anaconda oder Miniconda. Zum Zeitpunkt der Veröffentlichung wurde dieses Programm mit den Python-Versionen 3.10-3.12 getestet.
- Öffnen Sie den Browser und navigieren Sie zu https://github.com/rbudnar/nuclei_counter/releases. Laden Sie die neueste Version der Datei mit dem Namen count_nuclei.zip herunter.
- Klicken Sie im Ordner Downloads mit der rechten Maustaste auf die Zip-Datei , und extrahieren Sie alle Dateien an den gewünschten Speicherort auf dem lokalen Computer (siehe Abbildung 2A). Lassen Sie diesen Dateispeicherort geöffnet. Dieser Dateipfad wird in Schritt 2.5 verwendet (siehe Abbildung 2B).
HINWEIS: Count_nuclei sollte jetzt als Anwendungsdatei angezeigt werden. Dies ist die ausführbare Datei. - Öffnen Sie eine Eingabeaufforderung, indem Sie CMD oder Eingabeaufforderung in der Suchleiste suchen. Verwenden Sie den Befehl cd, um das Verzeichnis in den Dateipfad für die ausführbare Datei zu ändern (siehe Abbildung 2C), und drücken Sie die Eingabetaste. Beispiel für eine Befehlszeile: C:\Users\janedoe>cd [Leerzeichen nach cd, dann Dateipfad kopieren/einfügen]
- Geben Sie in der nächsten Befehlszeile Folgendes ein, und drücken Sie die Eingabetaste.
count_nuclei.exe --img_dir "Pfad/zu/Bildern" --output_dir "Pfad/zu/Ausgabe" --save_images --output_count_filename "results.csv"
wobei path/to/images der Dateipfad zu dem Ordner ist, der die zu analysierenden Bilder enthält (siehe Schritt 1.14); path/to/output ist der Dateipfad zu dem Ordner, in dem die .csv Datei gespeichert werden soll (standardmäßig das aktuelle Arbeitsverzeichnis, wenn nicht angegeben); und results.csv ist der Dateiname, unter dem die resultierenden Zählungen gespeichert werden (wenn nicht angegeben, wird der Name der Ausgabedatei standardmäßig auf count_results.csv gesetzt).
HINWEIS: In der Befehlszeile sind Anführungszeichen erforderlich. Stellen Sie sicher, dass nach Anführungszeichen vor dem Dateipfad keine Leerzeichen stehen (siehe Abbildung 2C). - Wenn die nächste Befehlszeile angezeigt wird, ist die Verarbeitung abgeschlossen. Die Konturlinien und die Ergebnistabelle sind nun an dem Ort verfügbar, der in Schritt 2.5 als Ausgabeverzeichnis angegeben wurde (d.h. Pfad/zum/Ausgabe; siehe Abbildung 2C).
- Überprüfen Sie die Konturen visuell, um die Zählqualität zu überprüfen. Die Ergebnisse können nun verwendet werden, um Daten zu normalisieren, wie in Abschnitt 4 beschrieben.
HINWEIS: Wenn Konturen fehlende Kerne zu enthalten scheinen (z. B. scheinen Kerne zu schwach zu sein) oder Gruppen von Kernen in einem einzigen Element zu gruppieren (z. B. überbelichtetes Bild, zu viel Hintergrund, Artefakte), sollte der Benutzer die Fehlerbehebungstabelle lesen und muss möglicherweise zu Abschnitt 1 zurückkehren, um die Bildaufnahme zu optimieren, bevor er mit Abschnitt 4 fortfährt.
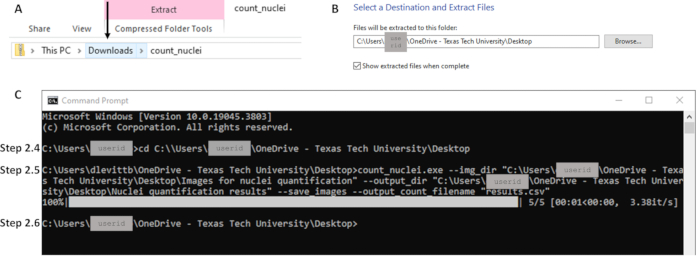
Abbildung 2: Beispiele für wichtige Teile des Workflows für Abschnitt 2. (A) Extrahieren Sie Dateien aus dem Downloads-Ordner, wobei Sie darauf achten, auf den richtigen Ordner zu klicken, wie durch den Pfeil angezeigt, um die Extraktion (B) zum gewünschten Ziel in Schritt 2.3 zu ermöglichen. (C) Beispiele für Befehle für die Schritte 2.4 und 2.5 und die nächste Befehlszeile, die den Abschluss in Schritt 2.6 anzeigt. Beachten Sie, dass der Dateipfad in Schritt 2.4 (C) mit dem Speicherort für die Extraktion in Schritt 2.3 (B) übereinstimmt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Ausführen des automatisierten Programms zur Quantifizierung von Kernen als Python-Skript
- Stellen Sie sicher, dass Python auf dem Computer installiert ist. Installieren Sie bei Bedarf Python von python.org/downloads (bevorzugt) oder mit Anaconda oder Miniconda. Zum Zeitpunkt der Veröffentlichung wurde dieses Programm mit den Python-Versionen 3.10-3.12 getestet.
- Öffnen Sie den Browser und navigieren Sie zu https://github.com/rbudnar/nuclei_counter. Klicken Sie auf die Schaltfläche "Grüner Code" und dann auf "ZIP herunterladen ", um das Code-Repository herunterzuladen (siehe Abbildung 3A).
- Verwenden Sie die folgenden Anweisungen für das Windows-Betriebssystem. Weitere Informationen finden Sie in Schritt 3.4 für Mac OS-basierte Computer.
- Klicken Sie im Ordner Downloads mit der rechten Maustaste auf die Datei und wählen Sie Alle extrahieren aus, um die Dateien auf einen lokalen Computer zu extrahieren (siehe Abbildung 3B-C).
- Öffnen Sie am Speicherort der extrahierten Datei den Ordner mit dem Namen nuclei_counter-main. Dies ist das Code-Repository.
HINWEIS: Dies muss nur heruntergeladen werden, wenn dieses Programm zum ersten Mal verwendet wird. Verschieben Sie einfach die extrahierten Dateien an einen zugänglichen Netzwerkspeicherort und notieren Sie sich diesen Speicherort. - Öffnen Sie eine Eingabeaufforderung, indem Sie CMD oder Eingabeaufforderung in der Suchleiste suchen. Wenn Python von Anaconda oder Miniconda installiert wurde, öffnen Sie die Anaconda-Eingabeaufforderung anstelle einer Standard-Eingabeaufforderung. Dies finden Sie, indem Sie Anaconda Prompt in die Suchleiste eingeben.
- Verwenden Sie den Befehl cd, um das Verzeichnis in das Code-Repository zu ändern, und drücken Sie die Eingabetaste. Weitere Informationen finden Sie in der folgenden Codezeile, in der zwischen Groß- und Kleinschreibung unterschieden wird. Stellen Sie sicher, dass der Ordner mit dem Namen nuclei_counter-main geöffnet ist, wenn Sie den Dateipfad kopieren. Die Datei mit dem Namen pyproject oder pyproject.toml sollte sichtbar sein (siehe Abbildung 3D). Beispiel für eine Befehlszeile: C:\Users\janedoe>cd [Leerzeichen nach cd, dann Dateipfad kopieren/einfügen]
- Geben Sie in der nächsten Befehlszeile Folgendes ein, einschließlich des Leerzeichens und des Punktes (siehe Abbildung 3D):
pip install -e .
HINWEIS: Die Schritte 3.3.5 und 3.3.6 müssen nur ausgeführt werden, wenn dieses Programm zum ersten Mal auf einem Computer ausgeführt wird. Wenn dies bereits abgeschlossen ist, fahren Sie mit Schritt 3.3.7 fort. Die Initialisierung des Installationsvorgangs kann einige Augenblicke dauern. - Drücken Sie die Eingabetaste. Durch das Ausführen dieses Befehls werden die erforderlichen Bibliotheken zum Ausführen dieses Skripts installiert und das Repository in einem bearbeitbaren Modus installiert, falls Codeanpassungen erforderlich sind (siehe Abbildung 3D).
- Geben Sie Folgendes in die nächste Befehlszeile ein, um das Verzeichnis in das Hauptverzeichnis des Quellcodes zu ändern (siehe Abbildung 3E):
CD "nuclei_counter" - Geben Sie in der nächsten Befehlszeile Folgendes ein, und drücken Sie die Eingabetaste (siehe Abbildung 3E):
python count_nuclei.py --img_dir "Pfad/zu/Bildern" --output_dir "Pfad/zu/Ausgabe" --save_images --output_count_filename "results.csv"
Dabei ist path/to/images der Dateipfad zu dem Ordner, der die zu analysierenden Bilder enthält (Schritt 1.14); path/to/output ist der Dateipfad zu dem Ordner, in dem die .csv Datei gespeichert werden soll (standardmäßig das aktuelle Arbeitsverzeichnis, wenn nicht angegeben); und results.csv ist der Dateiname, unter dem die resultierenden Zählungen gespeichert werden (wenn nicht angegeben, wird der Name der Ausgabedatei standardmäßig auf count_results.csv gesetzt).
HINWEIS: In der Befehlszeile sind Anführungszeichen erforderlich. Stellen Sie sicher, dass nach Anführungszeichen vor dem Dateipfad keine Leerzeichen stehen. Das Python-Skript liest die Dateien im angegebenen Dateipfad und führt das Kernzählprogramm für alle Bilder im Verzeichnis aus (siehe Abbildung 3E). Die resultierenden Zählungen werden in den Ergebnissen .csv Datei am angegebenen Speicherort gespeichert. - Wenn die nächste Befehlszeile angezeigt wird, ist die Verarbeitung abgeschlossen (siehe Abbildung 3E). Die Konturlinien und die Ergebnistabelle sind nun an dem Speicherort verfügbar, der in Schritt 3.3.8 als Ausgabeverzeichnis angegeben wurde (d. h. Pfad/zu/Ausgabe).
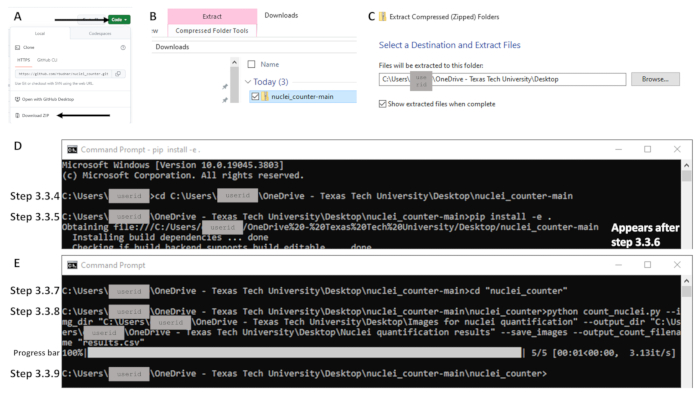
Abbildung 3: Beispiele für wichtige Teile des Workflows für Abschnitt 3 unter Verwendung des Windows-Betriebssystems. Um auf den Code zuzugreifen, klicken Sie auf die grüne Schaltfläche Code und (A) laden Sie die Zip-Datei herunter (gekennzeichnet durch Pfeile). (B) Extrahieren Sie, indem Sie mit der rechten Maustaste auf die Zip-Datei (blau dargestellt) klicken und (C) das gewünschte Ziel festlegen. (D) Nach dem Öffnen der Eingabeaufforderung Beispiele für Befehle für die Schritte 3.3.4 und 3.3.5. Beachten Sie, dass sich der Befehl für Schritt 3.3.5 in einer Zeile befindet; Der Text, der mit "Datei abrufen" in der nächsten Zeile beginnt, erscheint nach dem Drücken der Eingabetaste (Schritt 3.3.6; d.h. Ausführen des Befehls). (E) Beispiele für Befehle für die Schritte 3.3.7 und 3.3.8, einen Fortschrittsbalken nach der Ausführung von Schritt 3.3.8 und die Befehlszeile, die den Abschluss in Schritt 3.3.9 anzeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Verwenden Sie die folgenden Anweisungen für Mac OS.
- Klicken Sie im Ordner Downloads auf das Menü Datei und wählen Sie Öffnen , um die Dateien auf den lokalen Computer zu extrahieren (siehe Abbildung 4A).
- Öffnen Sie am Speicherort der extrahierten Datei den Ordner mit dem Namen nuclei_counter-main. Dies ist das Code-Repository.
HINWEIS: Dies muss nur heruntergeladen werden, wenn dieses Programm zum ersten Mal verwendet wird. Verschieben Sie einfach die extrahierten Dateien an einen zugänglichen Netzwerkspeicherort und notieren Sie sich diesen Speicherort. - Öffnen Sie ein Terminal, indem Sie Befehl + Leertaste drücken, um das Spotlight zu öffnen. Suchen Sie dann nach "Terminal".
- Verwenden Sie den Befehl cd, um das Verzeichnis in das Code-Repository zu ändern, und drücken Sie die Eingabetaste. Weitere Informationen finden Sie in der folgenden Codezeile, in der zwischen Groß- und Kleinschreibung unterschieden wird. Geben Sie den Dateipfad in ein Textdokument ein und kopieren Sie ihn in das Terminal. Stellen Sie sicher, dass der Dateipfad mit dem Text beginnt, der unmittelbar vor cd steht (z. B. /Users/userid in Abbildung 4B). Beispiel für eine Befehlszeile: /Users/janedoe›cd /Users/janedoe/Desktop/nuclei_counter-main
- In der nächsten Befehlszeile wird nach der Benutzer-ID ein Dollarzeichen angezeigt (siehe Abbildung 4B). Stellen Sie sicher, dass nach dem Dollarzeichen ein Leerzeichen steht, und geben Sie Folgendes ein, einschließlich des Leerzeichens und des Punktes am Ende des Befehls. Fügen Sie die entsprechende Python-Version unmittelbar nach pip ohne Leerzeichen ein: pip3 install -e .
HINWEIS: Die Schritte 3.4.5 und 3.4.6 müssen nur ausgeführt werden, wenn dieses Programm zum ersten Mal auf einem Computer ausgeführt wird. Wenn dies bereits abgeschlossen ist, fahren Sie mit Schritt 3.4.7 fort. Die Initialisierung des Installationsvorgangs kann einige Augenblicke dauern. - Drücken Sie die Eingabetaste. Durch Ausführen dieses Befehls werden die erforderlichen Bibliotheken zum Ausführen dieses Skripts installiert und das Repository in einem bearbeitbaren Modus installiert, falls Codeanpassungen erforderlich sind.
- Stellen Sie in der nächsten Befehlszeile sicher, dass nach dem Dollarzeichen ein Leerzeichen steht, und geben Sie dann Folgendes ein, um das Verzeichnis in das Hauptverzeichnis des Quellcodes zu ändern (siehe Abbildung 4C):
CD "nuclei_counter" - Geben Sie in der nächsten Befehlszeile die folgende Befehlszeile ein. Ersetzen Sie die Nummer nach Python durch die richtige Versionsnummer, wenn Sie eine andere Version als 3 verwenden. Drücken Sie dann die Eingabetaste (siehe Abbildung 4C):
python3 count_nuclei.py --img_dir "Pfad/zu/Bilder" --output_dir " Pfad/zu/Ausgabe" --save_images --output_count_filename "results.csv"
wobei path/to/images der Dateipfad zu dem Ordner ist, der die zu analysierenden Bilder enthält (Schritt 1.14); path/to/output ist der Dateipfad zu dem Ordner, in dem die .csv Datei gespeichert werden soll (standardmäßig das aktuelle Arbeitsverzeichnis, wenn nicht angegeben); und results.csv ist der Dateiname, unter dem die resultierenden Zählungen gespeichert werden (wenn nicht angegeben, wird der Name der Ausgabedatei standardmäßig auf count_results.csv gesetzt).
HINWEIS: In der Befehlszeile sind Anführungszeichen erforderlich. Stellen Sie sicher, dass nach Anführungszeichen vor dem Dateipfad keine Leerzeichen stehen. Das Python-Skript liest die Dateien im angegebenen Dateipfad und führt das Kernzählprogramm für alle Bilder im Verzeichnis aus (siehe Abbildung 4C). Die resultierenden Zählungen werden in den Ergebnissen .csv Datei am angegebenen Speicherort gespeichert. - Wenn die nächste Befehlszeile angezeigt wird, ist die Verarbeitung abgeschlossen (siehe Abbildung 4C). Die Konturlinien und die Ergebnistabelle sind jetzt an dem Speicherort verfügbar, der in Schritt 3.4.8 als Ausgabeverzeichnis angegeben wurde (d. h. Pfad/zu/Ausgabe).
- Überprüfen Sie die Konturen visuell, um die Zählqualität zu überprüfen. Die Ergebnisse können nun verwendet werden, um Daten zu normalisieren, wie in Abschnitt 4 beschrieben.
HINWEIS: Wenn bei Konturen Kerne fehlen (z. B. scheinen Kerne zu schwach zu sein) oder Gruppen von Kernen in einem einzigen Element zusammengefasst sind (z. B. überbelichtetes Bild, zu viel Hintergrund, Artefakte), sollte sich der Benutzer auf Tabelle 1 beziehen und muss möglicherweise zu Abschnitt 1 zurückkehren, um die Bildaufnahme zu optimieren, bevor er mit Abschnitt 4 fortfährt.
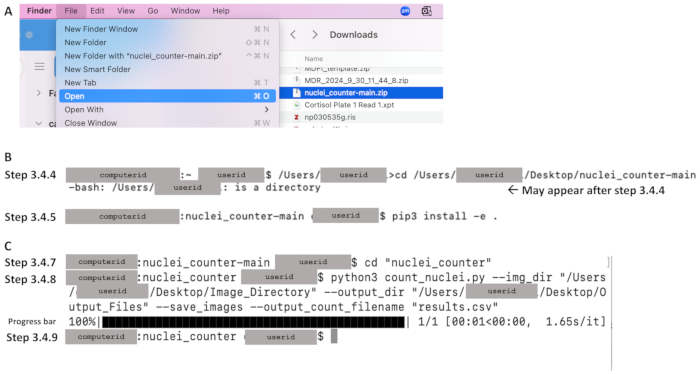
Abbildung 4: Beispiele für wichtige Teile des Workflows für Abschnitt 3 unter Mac OS. Nach dem Herunterladen des Codes (siehe Abbildung 3A) extrahieren Sie (A), indem Sie auf die Zip-Datei (blau dargestellt) klicken und das gewünschte Ziel festlegen. (B) Nach dem Öffnen des Terminals Beispiele für Befehle für die Schritte 3.4.4 (Ändern des Verzeichnisses in das Code-Repository) und 3.4.5 (Installieren von pip). (C) Beispiele für Befehle für die Schritte 3.4.7 und 3.4.8, ein Fortschrittsbalken nach der Ausführung von Schritt 3.4.8 und die Befehlszeile, die den Abschluss in Schritt 3.4.9 anzeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Verwenden von Zählungen zur Normalisierung experimenteller Daten
- Öffnen Sie die results.csv Datei und verknüpfen Sie jedes Bild mit der mit dem automatisierten Programm ermittelten Zellkernquantifizierung.
- Entscheiden Sie sich für die Normalisierungsmethode: 1) Bild-für-Bild-Basis (z. B. bei Mehrfarben-Fluoreszenzmikroskopie), 2) Normalisierung der Daten als Verhältnis der durchschnittlichen Kernzahl pro Probe zu einer Referenzprobe (oder -bedingung), 3) Normalisierung zur Kernzahl pro Probe (z. B. Extrapolation der Gesamtkernzahl auf die Probenfläche). Empfehlungen für die Normalisierungsmethode finden Sie im Abschnitt Diskussion.
- Wenn die Normalisierung auf Bild-für-Bild-Basis durchgeführt wird, dividieren Sie einfach die experimentellen Daten für jedes Bild durch die Anzahl der Kerne für dieses Bild.
- Um Daten als Verhältnis zu normalisieren, führen Sie die unten beschriebenen Schritte aus.
- Berechnen Sie für alle Proben, bei denen mehrere Bilder für ein einzelnes Replikat aufgenommen wurden (z. B. 10-15 Bilder, die von einem einzelnen Well in einer 6-Well-Platte aufgenommen wurden), die durchschnittliche Anzahl von Kernen pro Bild. Diese Kernzahlen werden als die Zellzahl betrachtet, wobei einkernige Zellen oder eine Anzahl von Zellen in mehrkernigen Zellen (z. B. mit Myotuben) miteinander verschmolzen werden.
- Wählen Sie entweder eine Referenzstichprobe oder eine Referenzbedingung (z. B. durchschnittliche Anzahl über alle Stichproben aus der Kontrollbedingung) aus und notieren Sie sich die Anzahl.
- Dividieren Sie die Anzahl für alle Stichproben durch diese Referenzanzahl, was zu einem Normalisierungswert führt. Wenn eine Referenzstichprobe verwendet wurde, verwenden Sie den Normalisierungswert für die Referenzstichprobe als 1.
HINWEIS: Wenn eine Referenzbedingung verwendet wurde, beträgt der durchschnittliche Normalisierungswert über die Bedingung jetzt 1. Proben mit weniger Kernen als die Referenz (d. h. weniger dicht) haben Normalisierungswerte von weniger als 1, und Proben mit mehr Kernen als die Referenz (d. h. dichter) haben Normalisierungswerte von mehr als 1. - Dividieren Sie die experimentellen Daten für jede Vertiefung durch den entsprechenden berechneten Normalisierungswert.
- Um die Daten auf die Kernzahl pro Probe zu normalisieren, führen Sie die unten beschriebenen Schritte aus.
- Berechnen Sie für alle Proben, bei denen mehrere Bilder für ein einzelnes Replikat aufgenommen wurden (z. B. 10-15 Bilder, die von einem einzelnen Well in einer 6-Well-Platte aufgenommen wurden), die durchschnittliche Anzahl von Kernen pro Bild. Diese Kernzahlen werden als die Zellzahl unter der Annahme von einkernigen Zellen oder als die Anzahl der Zellen betrachtet, die in mehrkernigen Zellen (z. B. mit Myotuben) miteinander verschmolzen sind.
- Bestimmen Sie die Oberfläche des Objektträgers (Deckglas), der Vertiefung oder der Schale, von der die Daten abgeleitet wurden. Diese Informationen finden Sie im Abschnitt "Spezifikationen" auf den Produktwebsites.
- Bestimmen Sie den Bereich jedes aufgenommenen Bildes. Diese Informationen sind oft als Metadaten in der Bildgebungs- oder Analysesoftware verfügbar.
- Stellen Sie sicher, dass die Probenoberfläche und der Bildbereich in denselben Einheiten ausgedrückt werden (z. B.mm 2). Wenn nicht, konvertieren Sie in die gewünschte Einheit.
- Berechnen Sie die Gesamtanzahl für jede Stichprobe mit der folgenden Gleichung:
TC = AC • (SA/IA)
wobei TC die Gesamtzählung, AC die Durchschnittszahl, SA die Oberfläche und IA die Bildfläche ist. - Teilen Sie die experimentellen Daten durch den entsprechenden TC für die Probe.
- Normalisieren Sie experimentelle Daten und verwenden Sie sie gegebenenfalls für statistische Analysen.
Ergebnisse
Jeder Batch-Bildlauf erzeugt: 1) einen Satz von Bilddateien mit angewendeten Konturen, die die Umrisse der identifizierten Kerne zeigen (Abbildung 5), und 2) eine .csv Datei (Tabelle), in der die Namen der Bilddateien und die zugehörigen Zählungen verknüpft sind. Durch das Betrachten der Konturen kann der Benutzer die Zählqualität visuell beurteilen. Insbesondere sollten Bilder, die gemäß Abschnitt 1 erhalten wurden, alle (oder fast alle) Kerne von einer durchgezogenen grünen Linie umgeben sein, die anzeigt, dass der Kern vom Programm gezählt wurde. Diese Konturen wurden hier verwendet, um das Programm zu optimieren.
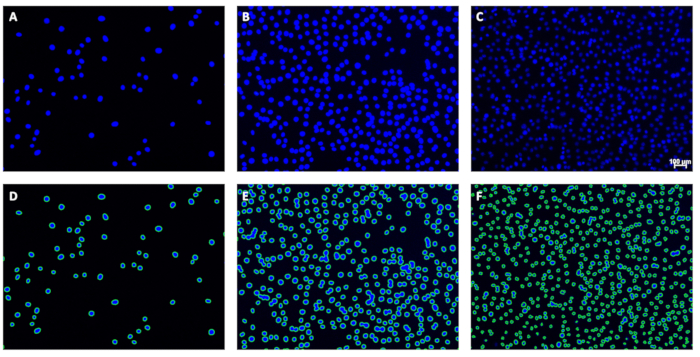
Abbildung 5: Beispiele für Originalbilder und entsprechende Konturen. (A-C) Beispiele für Originalbilder vor der Analyse und (D-F) entsprechende Konturen nach der Analyse direkt darunter. Der Maßstabsbalken (C) zeigt 100 μm an; Maßstabsbalken sollten bei Bildern, die für die Quantifizierung von Kernen verwendet werden, weggelassen werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Validierung anhand erster Ergebnisse
C2C12-Myoblasten mit unterschiedlichen Zelldichten wurden methanolfixiert, mit DAPI in Eindeckmedien gegengefärbt und 120 Bilder bei 10-facher Vergrößerung in .jpg Dateiformat aufgenommen. Das automatisierte Programm wurde ausgeführt, wobei das Image-Verzeichnis auf den Ordner festgelegt wurde, der diese Bilder enthält, und die resultierende .csv Datei wurde gespeichert. Unabhängig davon wurde jedes Bild von zwei geschulten Forschern manuell quantifiziert, die blind für die automatisierten Zählungen und für die Zählungen der anderen waren. Rohbilder, vom automatisierten Programm generierte Konturen und eine Ergebnistabelle mit manuellen und automatisierten Zählungen sind im Open Science Framework (OSF) Repository verfügbar (siehe Link in Abschnitt 1). Intraklassen-Korrelationskoeffizienten (ICCs; Durchschnittsmaße) wurden mit SPSS Statistics berechnet, um die Interrater-Reliabilität zwischen 1) den beiden manuellen Zählungen und 2) dem Durchschnitt der beiden manuellen Zählungen und der mit dem automatisierten Programm erhaltenen Zählungen zu bestimmen. Ähnliche Ansätze wurden verwendet, um andere Programme zur Quantifizierung von Objekten in Bildern zu validieren, die mit Hilfe der Fluoreszenzmikroskopie aufgenommen wurden 16,17,18. Die ICCs wurden auch zwischen durchschnittlichen manuellen und automatisierten Zählungen für jedes Quartil berechnet, um sicherzustellen, dass bei der Bewertung des gesamten Datensatzes keine potenziellen Änderungen der Zuverlässigkeit basierend auf der Zelldichte verdeckt wurden. Die Quartile wurden unter Verwendung der Schnittpunkte des 25., 50. und 75. Perzentils basierend auf der durchschnittlichen manuellen Zählung definiert und reichen von Quartil 1 (Q1; niedrigste Zelldichte) bis Q4 (höchste Zelldichte). ICCs werden auf der Grundlage durchschnittlicher Messungen angegeben und auf der Grundlage der folgenden Definitionen interpretiert: schlecht (ICC <0,50), mäßig (0,50 ≤ICC <0,75), gut (0,75 ≤ICC <0,90) oder ausgezeichnet (ICC ≥0,90)19.
Die Zellkerne pro Bild reichten von 44,5 bis 1160, basierend auf manuellen Zählmitteln. Die Interrater-Reliabilität war zwischen den beiden manuellen Zählungen ausgezeichnet (ICC > 0,999, p < 0,0001; Abbildung 6A) und zwischen durchschnittlichen manuellen und automatisierten Zählungen (ICC = 0,993, p < 0,0001; Abbildung 6B). Die Reliabilität blieb über alle Quartile hinweg ausgezeichnet (Q1: ICC = 0,996, p < 0,0001; Q2: ICC= 0,997, p < 0,0001; Q3: ICC = 0,998, p < 0,0001; Q4: ICC = 0,986, p < 0,0001; Abbildung 6C-F). Der einzelne Datenpunkt, der weit von der Linie mit der besten Anpassung in Abbildung 6B entfernt ist, stellt das in Abbildung 7 gezeigte Bild dar. Das Bild weist mehrere Bereiche mit mehreren Kernen auf, die miteinander gruppiert sind, was verhindert, dass das Computer-Vision-Programm jeden Kern erkennt. Dieses Problem kann durch eine kürzere Belichtungszeit behoben werden. Tabelle 1 beschreibt, wie mit einem solchen Bild umgegangen wird.
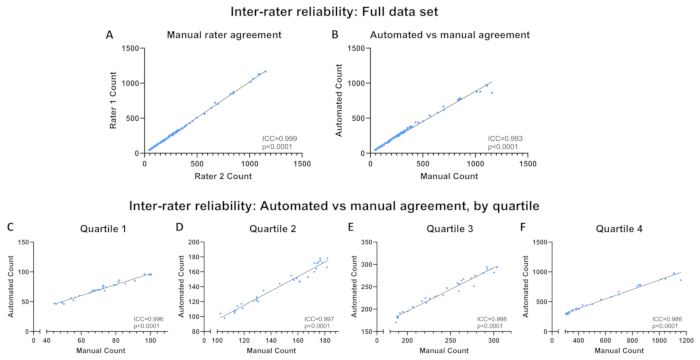
Abbildung 6: Inter-Rater-Reliabilität. Inter-Rater-Reliabilität über den gesamten Datensatz (A) zwischen manuellen Bewerterzählungen und (B) zwischen automatisierten Zählungen und durchschnittlichen manuellen Zählungen. (C-F) Inter-Rater-Zuverlässigkeit zwischen automatisierten Zählungen und durchschnittlichen manuellen Zählungen nach Quartil. Reliabilität, ausgedrückt als Intraklassen-Korrelationskoeffizient (ICC). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 7: Beispiel für ein Bild mit zwei Barrieren für eine genaue Kernquantifizierung. (A) Originalbild und (B) zugehörige Konturen. Pfeil 1 zeigt einen Bereich mit mehreren Kernen, die zusammengeklumpt sind, wodurch die runde Kernform so stark verzerrt wird, dass die Softwareerkennung mehrerer Kerne erschwert wird. Dies tritt an mehreren Stellen in diesem Bild auf. Pfeil 2 zeigt einen verblassten Halo, der hinter mehreren Kernen erscheint; Dieser Halo schien hell genug zu sein, dass die Software ihn als Kern einbeziehen konnte, wodurch die Anzahl der multiplen Kerne, die er umgibt, verloren ging. Dieses Bild stellt den einzelnen Datenpunkt dar, der weit von der am besten angepassten Linie in Abbildung 6B entfernt ist, und unterstreicht die potenziellen Probleme mit diesen Barrieren. Die Maßstabsleiste (A) zeigt 100 μm an; Maßstabsbalken sollten bei Bildern, die für die Quantifizierung von Kernen verwendet werden, weggelassen werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Unser Programm zur Quantifizierung von Kernen hat mehrere Vorteile gegenüber bestehenden Optionen: Es erfordert nur minimale technologische Fähigkeiten, ist für die spezifische Aufgabe der Kernquantifizierung validiert und Open Source; Letzteres überwindet kostenbezogene Barrieren. Letztendlich bietet dieses Programm Zell- und Molekularbiologen eine zusätzliche Möglichkeit, Zellkerne in Bildern, die mit Hilfe der Fluoreszenzmikroskopie aufgenommen wurden, schnell und genau zu quantifizieren. Die derzeit verfügbaren automatisierten Zellzählprogramme werden nicht von allen Anwendern bevorzugt, einige sind unerschwinglich, und die Validierung kann für bestimmte Anwendungen minimal oder gar nicht vorhanden sein. Spezialisierte Geräte, wie z. B. die BioTek Cytation-Linie, werden von technischen Bulletins begleitet, die die In-situ-Zellzählung mit der Kernfärbung20 validieren, zusätzlich zu einer Vielzahl zusätzlicher Anwendungen. Obwohl Validierungsdaten vorhanden sind und die Cytation-Produktlinie weit verbreitet ist, insbesondere wenn sie mit extrazellulären Flussanalysatoren von Seahorse verbunden ist, um Lebendzelldaten einfach und effizient zu normalisieren, kann dieses Produkt für Labore mit begrenzten Gerätemitteln unerschwinglich sein. So lag der Listenpreis für das mit vier Objektiven und vier Filterwürfeln ausgestattete Basismodell für die Fluoreszenzmikroskopie im Kalenderjahr 2024 bei über 100.000 US-Dollar. Für diejenigen, die nicht über die Mittel verfügen, spezielle Geräte zu kaufen, bietet Open-Source-Software kostenlose Alternativen für die Bildanalyse. Zu den Open-Source-Software, die für die Quantifizierung von Zellkernen und Zellen nützlich ist, gehört ImageJ (National Institutes of Health, Bethesda, MD, USA; verfügbar unter 21. Plugins sind jetzt für diejenigen verfügbar, die Makroskripte für die Bildverarbeitungsautomatisierung mit der ImageJ-Plattform schreiben möchten. Die Automatisierung der ImageJ-basierten Zellzählung mittels Phasenkontrast- oder Hellfeldmikroskopie mit Ganzzellbildern wurde bereits beschrieben22,23. CellProfiler ist eine weit verbreitete Anwendung für die Analyse von Zellbildern, die mit Fluoreszenzfarbstoffen oder histologischen Standardfärbungen gefärbt wurden, und ein früher Artikel, der seinen Nutzen in einer Reihe von biologischen Anwendungen beschreibt, wird oft als Validierungzitiert 24. Dieses Manuskript validiert jedoch keinen bestimmten biologischen Befund, so dass eine zusätzliche Validierung und Transparenz für verschiedene verwendete Automatisierungspipelines erforderlich ist, wie z. B. für die Kernzählung25. Darüber hinaus ist CellProfiler zwar ein effizientes Softwarepaket für die Zellanalyse, aber nicht alle Wissenschaftler finden es benutzerfreundlich. Daher haben wir uns zum Ziel gesetzt, ein alternatives automatisiertes Programm zur Quantifizierung von Zellkernen mit Hilfe von Open-Source-Software zu entwickeln und zu validieren. Konkret haben wir vorhandene Computer-Vision-Bibliotheken in Python verwendet, um diesen Workflow zu entwickeln.
Diese Methode beginnt mit Zellen oder Geweben, die der Endbenutzer mit DNA-Färbungen wie DAPI26 gefärbt hat. Es werden allgemeine Richtlinien für das Erfassen und Speichern von Bildern in einer Weise bereitgestellt, die mit Automatisierungssoftware kompatibel ist. Obwohl die Details je nach Mikroskop und Bildgebungssoftware unterschiedlich sein können, ist der allgemeine Arbeitsablauf für Abschnitt 1 derselbe, um quantifizierbare Bilder zu erhalten. Bei der Verwendung von lebenden Proben muss darauf geachtet werden, dass eine geeignete persönliche Schutzausrüstung getragen wird und die Bildgebungsumgebung für diese Proben geeignet ist (z. B. Temperatur, CO2 -Anteil, Zugabe von Puffer usw.).27. Während dieses Protokoll nur die Bildgebung der DNA-Farbstoffe beschreibt, können Benutzer andere Farben desselben Bereichs für Zusammenführungszwecke15 erfassen und die Kernbilder einfach in einen separaten Ordner kopieren, um sie mit dem Quantifizierungsprogramm zu verwenden. Die Bildgebung sollte in einem dunklen Raum mit geeigneter PSA für lebende Proben durchgeführt werden, und bei Bedarf sollte eine angemessene Desinfektion durchgeführt werden (z. B. mit 70 % Ethanol). Verfärbte Proben sollten vor dem Einschalten des Lichts vor Licht geschützt werden. Eine qualitativ hochwertige Bilderfassung ist entscheidend für eine genaue Quantifizierung von Kernen. Selbst bei Bildern mit angemessener Belichtungszeit und starkem Kontrast zwischen hellen Kernen und schwarzem Hintergrund kann das Vorhandensein von übermäßig verklumpten Zellen oder Halos hinter Zellgruppen (siehe Abbildung 7) die Quantifizierungsgenauigkeit verringern. In diesen Fällen müssen die Belichtungszeiten möglicherweise geändert werden, um die Intensität der Signale zu verringern und eine minimale Verschmelzung der Kernsignale und eine Minimierung der Halos zu gewährleisten, während ein Signal erhalten bleibt, das stark genug ist, um die runden Formen der Kerne zu erkennen. Wenn diese Schritte nicht ausreichen, kann die Bildgebung eines angrenzenden Abschnitts der Probe erforderlich sein. Alle Bilder eines einzelnen Experiments müssen mit der gleichen Vergrößerung aufgenommen werden, und die Vergrößerung, die dem verwendeten Objektiv entspricht, muss in der Bildgebungssoftware richtig eingestellt sein. Es ist besonders wichtig, dass das für die Bildgebung verwendete Objektiv mit den Einstellungen der Bildgebungssoftware übereinstimmt, wenn die Kernzahl vom abgebildeten Bereich auf den gesamten Well-Bereich (oder Objektträger) extrapoliert wird. Dieser Schritt sollte bei jedem Umschalten des Objektivs stattfinden, wird aber bei der Einrichtung des Mikroskops und der Software leicht übersehen. Die für Abschnitt 1 erforderliche Zeit hängt von einer Reihe von Faktoren ab (z. B. Benutzererfahrung mit der Fluoreszenzmikroskopie, Anzahl der Bilder und Proben, Mikroskop usw.). Um 10-15 Bilder von gefärbten Zellkernen pro Vertiefung in zwei 6-Well-Platten aufzunehmen, sollte der Benutzer etwa 1-2 Stunden Zeit reservieren.
Sobald die Bilder aufgenommen und in einem speziellen Ordner gespeichert wurden, können sie quantifiziert werden. Die einfachste Methode zur Quantifizierung besteht darin, dieses vorgefertigte automatisierte Programm auszuführen, ohne Python oder irgendwelche Bibliotheken installieren zu müssen (Abschnitt 2). Es ist zu beachten, dass das Programm als ausführbare Datei vorbereitet ist und mit Pyinstaller gepackt wurde. Software, die mit Pyinstaller erstellt wurde, wird häufig von Antivirensoftware gekennzeichnet. Wenn die Antivirensoftware das Öffnen der Datei blockiert, sollte der Benutzer die Datei als sicher markieren, um fortzufahren. Dies ist entscheidend für die erfolgreiche Durchführung von Abschnitt 2. Alternativ kann sich jeder Benutzer dafür entscheiden, das automatisierte Quantifizierungsprogramm als Python-Skript (Abschnitt 3) anstelle einer ausführbaren Datei auszuführen, falls gewünscht. Wenn der Benutzer das Programm mit einem Mac- oder Linux-Betriebssystem ausführen möchte, muss das Programm über Python ausgeführt werden. Diese Methode erfordert keinen Pyinstaller, kann aber technisch anspruchsvoller sein. Das Schritt-für-Schritt-Protokoll zusammen mit Beispielen, die in Abschnitt 3 beschrieben werden, zielt darauf ab, die technischen Hindernisse für die Ausführung dieses Programms als Skript zu beseitigen. Unabhängig davon, ob Abschnitt 2 oder Abschnitt 3 zum Ausführen des Programms verwendet wird, ist es wichtig sicherzustellen, dass der Code ordnungsgemäß in die Eingabeaufforderungen eingegeben wird. Beispielcodezeilen sind im gesamten Protokoll enthalten, und der Benutzer sollte sorgfältig auf Abstände, Anführungszeichen und andere Satzzeichen achten. Der Benutzer sollte auch sicherstellen, dass die Dateipfade korrekt sind. Es ist hilfreich, die Dateipfade für die Bilder und die Ausgabe in einem Word-Dokument zur Verfügung zu haben. Befehle können auch in dieses Word-Dokument eingegeben und bei Bedarf kopiert und eingefügt werden.
Wie in Abschnitt 4 beschrieben, gibt es mehrere Ansätze zur Normalisierung experimenteller Daten unter Verwendung von Zählungen, die aus diesem automatisierten Kernquantifizierungsprogramm erhalten wurden. Bei bildbasierten Experimenten, bei denen experimentelle Daten eine Reihe von Ereignissen in jedem Bild enthalten und ein Anteil gewünscht ist (z. B. Anteil der Annexin-V-positiven Zellen28, der Anteil der Zellen, die ACE2-Rezeptoren exprimieren29, Anzahl der Zellen, die gefärbte Partikel aufnehmen30 usw.), können die experimentellen Daten für jedes Bild einfach durch die resultierende Anzahl dividiert werden (was zu einem Verhältnis von 0 bis 1 führt). Dann kann der durchschnittliche Anteil für replizierte Bilder von derselben Folie, Schale oder Vertiefung berechnet werden. Wenn ein Prozentsatz gewünscht ist, multiplizieren Sie einfach die endgültigen Proportionen mit 100. Schließlich können diese durchschnittlichen Anteile oder Prozentsätze in nachgelagerten statistischen Analysen verwendet werden. Wenn die zu normalisierenden experimentellen Daten aus funktionellen Experimenten in lebenden Zellen (z. B. Sauerstoffverbrauchsraten6, 11, 12) oder aus Zellkulturüberständen (z. B. Laktatdehydrogenaseaktivität im Überstand31 oder Konzentration eines spezifischen sezernierten Zytokins32 usw.) stammen, sollten die Zellkernzahlen zunächst über alle Bilder gemittelt werden, die aus demselben Bohrloch aufgenommen wurden. Dann kann die Kernzahl auf die Oberfläche des Objektträgers (Deckglas), der Vertiefung oder der Schale extrapoliert werden, oder die Anzahl kann als Verhältnis zu einer Referenzprobe oder -bedingung ausgedrückt werden. Die Extrapolation auf die Oberfläche des Objektträgers, der Vertiefung oder der Schale ist nützlich, um experimentelle Daten im Vergleich zur Anzahl der Zellen (bei Verwendung von einkernigen Zellen) auszudrücken, von denen die Daten abgeleitet wurden (z. B. Sauerstoffverbrauch pro Minute pro 103 Zellen). Um diese Methode verwenden zu können, müssen die Oberfläche des Objektträgers (Deckglas), der Vertiefung oder der Schale und der in jedem Bild erfasste Bereich bekannt sein. Wenn die Probe nicht die gesamte verfügbare Oberfläche abdeckt, die experimentellen Daten von einer kleinen Menge Überstand stammen (d. h. ausgedrückt als Aktivität oder Konzentration pro Volumeneinheit) oder das Bild und/oder die Oberfläche nicht bekannt sind, besteht die beste Option darin, die Kernzahl über alle Bilder für eine einzelne Probe zu mitteln und die Kernzahl relativ zu einer Referenzprobe oder -bedingung auszudrücken. Der resultierende Wert wird verwendet, um experimentelle Daten für die Zellkerndichte oder die Zelldichte (bei Verwendung von einkernigen Zellen) zu korrigieren.
Obwohl diese Methode einfach auszuführen ist, gibt es mehrere potenzielle Probleme, über die sich Benutzer im Klaren sein sollten. Diese potenziellen Probleme sind zusammen mit möglichen Ursachen und Schritten zur Problembehandlung zur Behebung der Probleme in Tabelle 1 aufgeführt. Das hier beschriebene Programm hat entscheidende Vorteile gegenüber bestehender Software; Es müssen jedoch einige Einschränkungen anerkannt werden. Der Hauptnachteil besteht darin, dass das Quantifizierungsprogramm nur mit Bildern verwendet werden kann, die mit der Fluoreszenzmikroskopie aufgenommen wurden, nicht mit Hellfeldbildern. Während DAPI-gefärbte Bilder, die mit 10-facher Vergrößerung aufgenommen wurden, zum Erstellen des Programms verwendet wurden, kann das Programm mit anderen DNA-Farbstoffen und -Vergrößerungen verwendet werden, da das Programm so konzipiert ist, dass es die Morphologie der Zellkerne und nicht Farbe oder Größe erkennt. Während wir die Genauigkeit des Programms anhand von Bildern mit einer Vielzahl von Zelldichten validiert haben, kann es sein, dass der Endbenutzer Zelldichten hat, die außerhalb des Bereichs liegen. In diesem Fall gehen wir immer noch von genauen Zählungen aus, vorausgesetzt, es werden qualitativ hochwertige Bilder erhalten. Der Benutzer sollte jedoch die Konturen und den Ausgang sorgfältig überprüfen, um die Genauigkeit zu gewährleisten. Für Zellkulturanwendungen ist die Quantifizierung von Zellkernen in Bildern am besten für Zellen in einer Monoschicht geeignet. Wenn sich kultivierte Zellen überlappen, können Zellkerne maskiert werden, was zu einer Unterschätzung der Zellkernmenge führt. Die zweidimensionale Natur der Quantifizierung von Zellkernen in Bildern schließt die Verwendung dieses Programms zur Bestimmung der Gesamtzahl der Zellkerne aus Zellen in Suspension, dicken Gewebeblöcken oder anderen dreidimensionalen Anwendungen aus. Dieses Programm kann jedoch immer noch Zellkerne quantifizieren, die mit fluoreszierenden DNA-Farbstoffen in zweidimensionalen Bildern aufgenommen wurden, die von dreidimensionalen Modellen wie dicken Gewebeblöcken aufgenommen wurden. Der Nutzen der Quantifizierung von Kernen in Bildern für die gewünschte Anwendung sollte vom Endanwender bestimmt werden.
| Problem | Mögliche Ursachen | Mögliche Lösungen |
| Übermäßiger Hintergrund in Bildern | Belichtungszeit zu hoch | Reduzieren Sie die Belichtungszeit |
| Kerne scheinen in Bildern miteinander zu verschmelzen | Fluoreszierende DNA-Farbstoffkonzentration zu hoch oder zu viel DNA-Farbstoff-Einbindemittel | Reduzieren Sie die Farbstoffkonzentration während der Optimierung; Möglichst wenig Einhängemittel verwenden |
| Kerne erscheinen zu hell oder einige Kerne sind nicht sichtbar | Zu geringe Belichtungszeit | Erhöhen Sie die Belichtungszeit |
| Photobleichen | Verhindern Sie Photobleaching, indem Sie sicherstellen, dass gefärbte Proben im Dunkeln gelagert werden, und indem Sie die Exposition der Proben gegenüber Beleuchtung minimieren (z. B. durch die Verwendung der erforderlichen Mindestbelichtungszeit und durch Ausschalten der Beleuchtung, wenn sie nicht benötigt wird, z. B. nach der Aufnahme eines Bildes oder beim Anfertigen von Notizen) | |
| Halo-Effekte oder nicht-kernspezifische Formen, die erscheinen | Belichtungszeit zu hoch | Reduzieren Sie die Belichtungszeit |
| Flusen von empfindlichen Arbeitswischern oder einigen Tischprotektoren in lebenden Zellen oder an fixierten Zellen | Sorgen Sie für eine fusselfreie Umgebung: Minimieren Sie den Einsatz empfindlicher Wischtücher, wenn Proben exponiert werden (z. B. in der Biosicherheitswerkbank für lebende Zellen, während der Fixierung und Trocknung fixierter Zellen oder während des Färbeprozesses); Stellen Sie sicher, dass Tischprotektoren während des Färbens keine Fusseln auf Objektträgern oder Platten hinterlassen | |
| Andere Anomalien auf der Oberfläche von fixierten Zellen/Geweben | Behandeln Sie Proben mit äußerster Sorgfalt und befolgen Sie die richtigen Vorsichtsmaßnahmen und Best Practices beim Fixieren und Färben | |
| Störungen im Lichtweg | Stellen Sie sicher, dass alle Teile des Mikroskops sauber und frei von Fusseln und Staub sind. Reinigen Sie die Außenseite von Objektträgern/Platten/Schalen mit 70 % Ethanol, um Umgebungsstaub und Flusen vor der Bildgebung zu entfernen | |
| Ausführbare Datei wird nicht geöffnet | Antivirensoftware hat die Datei blockiert | Kennzeichnen der Datei als sicher |
| Fehler in Eingabeaufforderungen | Probleme mit dem Abstand in der Befehlszeile | Vergewissern Sie sich, dass der Anfang des Befehls unmittelbar nach dem Symbol ">" in der Befehlszeile ohne Leerzeichen eingegeben wird, dass nach den offenen Anführungszeichen für Dateipfade keine Leerzeichen vorhanden sind und dass die Abstände und Abstände mit dem bereitgestellten Code und den Beispielen übereinstimmen. |
| Unsachgemäße Groß- und Kleinschreibung in der Befehlszeile | Beim Code wird zwischen Groß- und Kleinschreibung unterschieden. Achten Sie darauf, dass Sie Groß- und Kleinbuchstaben verwenden | |
| Probleme mit dem Dateipfad | Stellen Sie sicher, dass der Dateipfad das Innere des zugeordneten Dateiordners angibt (d. h. der Dateiordner ist geöffnet, wenn der Dateipfad kopiert wird). | |
| Probleme mit dem Programm, die mit den oben genannten Schritten zur Fehlerbehebung nicht behoben werden können | Software- oder Befehlszeilenprobleme, die oben erwähnt wurden. | Reichen Sie ein Problem über GitHub ein. Navigieren Sie zu https://github.com/rbudnar/nuclei_counter. Wählen Sie in der Menüleiste oben "Issues" (zweite Option, rechts neben "Code"). Klicken Sie auf die grüne Schaltfläche auf der rechten Seite mit der Aufschrift "Neue Ausgabe", um sie einzureichen. Es wird eine Aufforderung angezeigt, sich bei GitHub anzumelden, bevor das Problem eingereicht wird. Es wird auch die Möglichkeit geben, bei Bedarf ein kostenloses Konto zu erstellen. |
Tabelle 1: Fehlerbehebung bei der Aufnahme hochwertiger Bilder und der Ausführung des automatisierten Workflows für die Zellkernquantifizierung.
Insgesamt bieten die hier beschriebenen Methoden Schritt-für-Schritt-Details zur Verwendung des automatisierten Kernquantifizierungsprogramms, das wir unter Verwendung von Computer-Vision-Bibliotheken entwickelt und gegen manuelle Zählungen von mehreren Bewertern validiert haben. Dieses Programm wurde entwickelt, um die Nutzbarkeit durch Wissenschaftler mit unterschiedlichen technologischen Fähigkeiten im Bereich der Codierung zu gewährleisten, ist frei verfügbar und wurde speziell für die Aufgabe der Kernquantifizierung validiert. Im Vergleich zur manuellen Zählung spart dieses Programm dem Endbenutzer wertvolle Zeit und liefert genaue und zuverlässige Kernzählungen für die Datennormalisierung.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Arbeit wurde von den National Institutes of Health/National Institute on Aging (R01AG084597; DEL und HYL) und durch Start-up-Fonds der Texas Tech University (DEL). Die Autoren danken den Programmen Undergraduate Research Scholars und TrUE Scholars der Texas Tech University für die finanzielle Unterstützung der Undergraduate-Forscher, die zu dieser Arbeit beigetragen haben (REH, MRD, CJM, AKW). Wir danken auch Dr. Lauren S. Gollahon und Dr. Michael P. Massett für die freundliche Bereitstellung ihrer Laborräume und -geräte.
Materialien
| Name | Company | Catalog Number | Comments |
| Computer with access to results file from method 2 or 3 | - | - | See step 2.6 (for Method 2) or step 3.3.9 or 3.4.9 (for Method 3) |
| Computer with internet access, modern browser | - | - | e.g., Google Chrome |
| Computer with internet access, modern browser, and Windows OS | Varies | Varies | For Mac, Linux, or other OS, use Method 3 |
| Computer with software for image capture | Zeiss | AxioVision | Other software is acceptable; must be compatible with the fluorescence microscope |
| File location for output (results spreadsheet and image contours) | - | - | Can be a new, empty folder |
| Fluorescence microscope | Zeiss | Axiovert 200M | Other fluorescence microscopes are acceptable; must be equipped with appropriate filter cubes, desired objective, and camera |
| Folder containing all images to be quantified | - | - | See step 1.12 |
| Python version 3.10 or higher | Python | - | Available for free download and installation at https://www.python.org/downloads/ |
| Samples to be imaged | - | - | Fixed or live, stained or counterstained with fluorescent DNA dyes |
| Spreadsheet software | Microsoft | Excel | Similar spreadsheet software is also acceptable |
Referenzen
- Chacko, B. K., et al. The bioenergetic health index: A new concept in mitochondrial translational research. Clin Sci. 127 (6), 367-373 (2014).
- Desousa, B. R., et al. Calculation of ATP production rates using the Seahorse XF Analyzer. EMBO Rep. 24 (10), e56380(2023).
- Rayamajhi, M., Zhang, Y., Miao, E. A. detection of pyroptosis by measuring released lactate dehydrogenase activity. Inflammasome. 1040, 85-90 (2013).
- Baskic, D., Popovic, S., Ristic, P., Arsenijevic, N. Analysis of cycloheximide-induced apoptosis in human leukocytes: Fluorescence microscopy using annexin V/propidium iodide versus acridin orange/ethidium bromide. Cell Biol Int. 30 (11), 924-932 (2006).
- Duplanty, A. A., Siggins, R. W., Allerton, T., Simon, L., Molina, P. E. Myoblast mitochondrial respiration is decreased in chronic binge alcohol administered simian immunodeficiency virus-infected antiretroviral-treated rhesus macaques. Physiol Reps. 6 (5), e13625(2018).
- Levitt, D. E., Bourgeois, B. L., Rodríguez-Graciani, K. M., Molina, P. E., Simon, L. Alcohol impairs bioenergetics and differentiation capacity of myoblasts from Simian immunodeficiency virus-infected female Macaques. Int J Mol Sci. 25 (4), 2448(2024).
- Larsen, T. A., Goodsell, D. S., Cascio, D., Grzeskowiak, K., Dickerson, R. E. The structure of DAPI bound to DNA. J Biomol Str Dyn. 7 (3), 477-491 (1989).
- Bazhulina, N. P., et al. Binding of Hoechst 33258 and its derivatives to DNA. J Biomol Str Dyn. 26 (6), 701-718 (2009).
- Njoh, K. L., et al. Spectral analysis of the DNA targeting bisalkylaminoanthraquinone DRAQ5 in intact living cells. Cytomet A. 69A (8), 805-814 (2006).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Levitt, D. E., Chalapati, N., Prendergast, M. J., Simon, L., Molina, P. E. Ethanol-impaired myogenic differentiation is associated with decreased myoblast glycolytic function. Alcohol Clin Exp Res. 44 (11), 2166-2176 (2020).
- Levitt, D. E., et al. Skeletal muscle bioenergetic health and function in people living with HIV: association with glucose tolerance and alcohol use. Am J Physiol Regul, Integ Comp Physiol. 321 (5), R781-R790 (2021).
- Levitt, D. E., Adler, K. A., Simon, L. HEMA 3 staining: A simple alternative for the assessment of myoblast differentiation. Curr Prot Stem Cell Biol. 51 (1), e101(2019).
- Solem, J. E. Programming computer vision with Python. , O'Reilly. Sebastopol, CA. (2012).
- Delery, E. C., Levitt, D. E., Amedee, A. M., Molina, P. E., Simon, L. High fat, high sucrose diet promotes increased expression of ACE2 receptor in the SIV-infected host: implications for SARS-CoV-2 infection. Front Nutrition. 11, 1458106(2024).
- Corrà, M. F., et al. Advantages of an automated method compared with manual methods for the quantification of intraepidermal nerve fiber in skin biopsy. J Neuropathol Exp Neurol. 80 (7), 685-694 (2021).
- Neves, K., et al. The reliability of the isotropic fractionator method for counting total cells and neurons. J Neurosci Meth. 326, 108392(2019).
- Tiong, K. H., et al. QuickCount: a novel automated software for rapid cell detection and quantification. BioTechniques. 65 (6), 322-330 (2018).
- Koo, T. K., Li, M. Y. A guideline of selecting and reporting intraclass correlation coefficients for reliability research. J Chiropractic Med. 15 (2), 155-163 (2016).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. P. Normalization of Agilent Seahorse XF Data by in situ. cell counting using a BioTek Cytation 5. , https://www.agilent.com/cs/library/applications/5991-7908EN.pdf 5991-7908 (2017).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH image to imageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- O'Brien, J., Hayder, H., Peng, C. Automated quantification and analysis of cell counting procedures using ImageJ plugins. J Vis Exp. (117), e54719(2016).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal Biochem. 473, 63-65 (2015).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100(2006).
- Tollemar, V., et al. Quantitative chromogenic immunohistochemical image analysis in cellprofiler software. Cytometry A. 93 (10), 1051-1059 (2018).
- Chazotte, B. Labeling nuclear DNA using DAPI. Cold Spring Harbor. 2011 (1), (2011).
- Cole, R. Live-cell imaging. Cell Adhesion Migrat. 8 (5), 452-459 (2014).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide apoptosis assay for accurate assessment of cell death. J Vis Exp. (50), e2597(2011).
- Hernandez, J. J., Beaty, D. E., Fruhwirth, L. L., Lopes Chaves, A. P., Riordan, N. H. Dodging COVID-19 infection: low expression and localization of ACE2 and TMPRSS2 in multiple donor-derived lines of human umbilical cord-derived mesenchymal stem cells. J Transl Med. 19 (1), 149(2021).
- Bourgeois, B. L., et al. Extracellular vesicle miR-206 improves chronic binge alcohol-mediated decreased myoblast differentiation in SIV-infected female macaques. Am J Physiol Cell Physiol. 327 (6), C1626-C1637 (2024).
- Kumar, P., Nagarajan, A., Uchil, P. D. Analysis of cell viability by the lactate dehydrogenase assay. Cold Spring Harbor. 2018 (6), (2018).
- Hsu, S., et al. Effects of everolimus on macrophage-derived foam cell behavior. Cardiovasc Revasc Med Including Mol Intervent. 15 (5), 269-277 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten