Method Article
Rilevamento rapido dell'antigene fecale dell'infezione da Helicobacter pylori basato sulla tecnologia di rilevamento a sandwich di doppi anticorpi
In questo articolo
Riepilogo
In questo studio, è stata sviluppata una tecnica di rilevamento a sandwich basata su doppi anticorpi per il rilevamento rapido di antigeni fecali infettati da Helicobacter pylori.
Abstract
L'Helicobacter pylori può essere parassitario della mucosa gastrica, che può causare una serie di malattie gastrointestinali dopo l'infezione ed è strettamente correlato alla gastrite, all'ulcera gastrica e al cancro gastrico. L'elevata prevalenza dell'infezione da H. pylori in regioni con scarse infrastrutture mediche e aree igienico-sanitarie inadeguate rimane un notevole problema di salute pubblica. Di conseguenza, lo sviluppo di metodi di screening rapidi, semplici ed economici per la rilevazione di H. pylori in contesti con risorse limitate è di fondamentale importanza.
In questo studio, abbiamo condotto uno screening basato sulla comunità nel villaggio di Shitan, nella provincia del Guangdong, un'area remota e sottosviluppata caratterizzata da una popolazione permanente di circa 300 residenti con un livello di istruzione generalmente basso. Abbiamo impiegato un doppio test anticorpale a sandwich per la rilevazione dell'antigene fecale di H. pylori , un metodo scelto per la sua potenziale applicabilità in contesti con scarse risorse. Sono stati arruolati un totale di 261 partecipanti del villaggio e i loro campioni fecali sono stati analizzati utilizzando questa tecnica. Per la convalida comparativa, gli stessi campioni sono stati sottoposti ad analisi quantitativa della reazione a catena della polimerasi (qPCR). Rispetto ai risultati della qPCR, la sensibilità e la specificità del rilevamento dell'antigene di H. pylori nelle feci erano del 60,24% e dell'80,08%. I risultati hanno dimostrato una forte concordanza tra il metodo di rilevamento dell'antigene fecale e la qPCR (Kappa = 0,630). Questo studio ha chiarito sistematicamente i principi, le procedure, le metodologie e le applicazioni cliniche del rilevamento dell'antigene fecale per l'infezione da H. pylori , con l'obiettivo di esplorare la tecnologia del sandwich di doppi anticorpi a base di lattice e stabilire nuove strategie e linee guida pratiche per la sua diagnosi ausiliaria.
Introduzione
L'Helicobacter pylori (H. pylori, Hp) è unbatterio gram-negativo 1 microaerofilo a forma di spirale. Grazie alla sua capacità di colonizzare e infettare cronicamente lo stomaco umano2, questo patogeno è stato identificato come un importante fattore eziologico nello sviluppo dell'adenocarcinoma gastrico 3,4. Nel 2015, ~4,4 miliardi di persone nel mondo hanno sofferto di infezione da H. pylori 5. La prevalenza dell'infezione da H. pylori mostra notevoli variazioni geografiche, con i paesi in via di sviluppo che registrano tassi significativamente più elevati rispetto alle nazioni sviluppate. In particolare, nei paesi a basso reddito e tra alcune popolazioni vulnerabili, il tasso di infezione può raggiungere il 75%6.
Sebbene i metodi di rilevamento tradizionali come la gastroscopia siano accurati, la loro applicazione nello screening su larga scala e nel follow-up di routine è limitata a causa della loro invasività, del costo elevato e della bassa accettazione da parte deipazienti7. Pertanto, la ricerca di metodi diagnostici non invasivi, semplici e rapidi è diventata una direzione importante della moderna ricerca di medicina clinica. Lo scopo principale di una diagnosi non invasiva di H. pylori è quello di rilevare in modo accurato, sicuro e conveniente se il paziente è infetto da H. pylori senza esame endoscopico. Il test dell'urea del respiro (UBT ), il test dell'antigene fecale (FAT)8 e il test sierologico9 sono tecniche non invasive molto diffuse.
Tra queste, l'UBT è la procedura meno intrusiva e più accurata disponibile10. La sensibilità e la specificità dell'UBT sono superiori al 95%11. Tra i metodi diagnostici non invasivi disponibili, l'UBT ha dimostrato di essere il più accurato e affidabile, come si è visto in uno studio di convalida condotto in Iraq, l'UBT può essere raccomandato come prima scelta grazie alle sue prestazioni più elevate rispetto ad altri metodi12. Tuttavia, l'UBT ha anche i suoi svantaggi, come il costo elevato e la necessità di analisi spettrometriche di massa, che limitano l'applicazione in ambienti remoti o con risorse limitate13.
Lo sviluppo di un metodo diagnostico rapido, non invasivo ed economico per rilevare l'infezione da H. pylori è estremamente necessario, in particolare nelle regioni con risorse limitate. In questo studio, abbiamo sviluppato una tecnologia di rilevamento rapido per l'antigene fecale di H. pylori basata sulla tecnologia di rilevamento a sandwich di doppi anticorpi, in grado di rilevare l'antigene di H. pylori in campioni fecali in modo rapido, efficace e a basso costo. Questa tecnologia offre vantaggi significativi, tra cui convenienza, non invasività, facilità d'uso e adattabilità alle aree remote. Ipotizziamo che il metodo a sandwich di doppi anticorpi per rilevare l'antigene fecale di H. pylori dimostrerà sensibilità e specificità paragonabili alla qPCR, posizionandolo come uno strumento praticabile per la diagnosi clinica non invasiva.
Abbiamo dettagliato in modo completo i principi, le procedure, i punti di forza metodologici e l'applicabilità clinica del rilevamento dell'antigene fecale per l'infezione da H. pylori . Per convalidarne la praticità, abbiamo condotto un'indagine epidemiologica e uno screening nel villaggio di Shitan, nella città di Qingyuan, nella provincia del Guangdong, in Cina, che è un'area remota e sottosviluppata con una popolazione permanente di circa 400 persone e un livello di istruzione limitato. I nostri risultati dimostrano che la tecnologia di rilevamento rapido dell'antigene di H. pylori nelle feci qui progettata può essere completata entro 20 minuti. Si evidenzia che il rilevamento rapido dell'antigene di H. pylori nelle feci è uno strumento potenziale e promettente per una diagnosi rapida e affidabile dell'infezione da H. pylori in aree remote e arretrate.
Protocollo
Questo studio trasversale è stato approvato dal Comitato etico dell'Ospedale del popolo provinciale del Guangdong (numero di approvazione: KY2024-445-01) e i dati personali di tutti i soggetti dello studio sono stati strettamente riservati durante lo studio. Tutti i partecipanti hanno firmato il consenso informato scritto prima degli esperimenti. I residenti del villaggio di Shitan nella città di Qingyuan, nella provincia del Guangdong, nel 2024 sono stati selezionati come soggetti di ricerca e non c'erano restrizioni di età e sesso. Il test del doppio sandwich di anticorpi (un esperimento qualitativo) descritto di seguito è stato eseguito da personale medico e tecnico professionale secondo le istruzioni. L'intera popolazione permanente del villaggio è stata selezionata per questo studio, indipendentemente dal fatto che fosse sana o avesse sintomi o qualsiasi malattia esistente correlata a malattie gastrointestinali.
1. Selezione del paziente
- Escludere i pazienti che hanno utilizzato inibitori della pompa protonica o bismuto, bloccanti del recettore H2 o antibiotici nel 1 mese precedente.
- Chiedi a tutti i partecipanti di compilare il questionario in forma anonima. Il contenuto del questionario comprende cinque domande: Mangi tre pasti regolarmente? Bevi alcolici? Sei a conoscenza di H. pylori? Pensi che sia necessario effettuare un'ispezione di H. pylori ? Comprendi le conseguenze negative dell'infezione da H. pylori ?
2. Procedura per il rilevamento dell'antigene dell'infezione fecale da H. pylori
NOTA: L'algoritmo per il test dell'antigene delle feci di H. pylori è stato progettato per garantire l'accuratezza e l'affidabilità dei risultati, tenendo conto della semplicità e dell'igiene. L'intero processo è suddiviso in cinque fasi: raccolta dei campioni, elaborazione dei campioni, conservazione dei campioni, rilevamento e interpretazione dei risultati. Il diagramma di flusso di lavoro e il diagramma schematico del rilevamento del campione sono mostrati nella Figura 1 e nella Figura 2.
- Raccolta campioni
NOTA: Per la raccolta dei campioni devono essere utilizzati contenitori specifici per l'antigene di H. pylori . I campioni non devono essere mescolati con acqua, urina, disinfettanti e acque reflue.- Al termine della defecazione, rimuovere il coperchio superiore dell'apposito contenitore per estrarre l'asta di campionamento.
- Inserire il sample asta continuamente in cinque diverse posizioni dello sgabello per il campionamento, assicurandosi che l'estremità filettata del sample asta sia completamente inserita nello sgabello.
- Reinserire l'asta di campionamento nella provetta del reagente al termine del campionamento; Il volume del campione è di ~5-50 mg in totale.
- Elaborazione dei campioni
- Agitare la provetta del reagente da un lato all'altro per ~10 s per miscelare accuratamente il campione di feci nel diluente (ingredienti principali: acido etilendiammina tetraacetico, tetrasodio idrato 0,018 g/mL e cloruro di sodio 0,01 g/mL).
- Conservazione dei campioni
- Testare i campioni il prima possibile dopo il campionamento; conservarli a temperatura ambiente per non più di 6 ore e in condizioni refrigerate (non più di 72 ore a 2-8 °C e non più di 6 mesi congelati a -25 °C--15 °C) se necessario per rallentare la degradazione dell'antigene e l'attività microbica.
NOTA: I campioni possono essere congelati e scongelati ripetutamente fino a 3 volte e sia i campioni refrigerati che quelli congelati devono essere riportati a temperatura ambiente prima del test.
- Testare i campioni il prima possibile dopo il campionamento; conservarli a temperatura ambiente per non più di 6 ore e in condizioni refrigerate (non più di 72 ore a 2-8 °C e non più di 6 mesi congelati a -25 °C--15 °C) se necessario per rallentare la degradazione dell'antigene e l'attività microbica.
- Rilevamento del campione
- Aprire il coperchio bianco del tubo del reagente e mantenere il tubo del reagente eretto.
- Premere il coperchio fino al punto più basso in modo che il campione fluisca sulla scheda di prova, passando attraverso l'area di rilevamento (T) e l'area di controllo qualità (C) rivestite con l'anticorpo anti-Hp del topo.
- Interpretazione dei risultati
- Attendere 10-20 minuti in modo che qualsiasi antigene di H. pylori nel campione, se legato all'anticorpo, formi una reazione cromogenica macroscopica nell'area di rilevamento. Questo di solito appare come una linea rossa o viola che contrasta con la linea nell'area del controllo qualità.
- La presenza o l'assenza della reazione cromatica e la profondità del colore sono correlate alla concentrazione dell'antigene di H. pylori nel campione. Confrontare le cartelle colori per interpretare i risultati in modo rapido e accurato (vedere la Figura 3).
3. Estrazione del DNA
- Mettere una quantità adeguata di feci in una provetta, aggiungere la soluzione di PBS e mescolare accuratamente agitando. Centrifugare la miscela a 12.000 × g per 5-10 minuti, quindi raccogliere con cura 200 μl di surnatante per un'ulteriore estrazione.
- In base al numero di campioni, preparare un numero uguale di provette da centrifuga da 1,5 mL. A ciascuna provetta, aggiungere in sequenza 20 μL di proteinasi K (20 mg/mL), 10 μL di biglie magnetiche, 200 μL di surnatante del campione e 200 μL di tampone di lisi A. Mescolare accuratamente capovolgendo le provette, quindi incubare in un bagno di metallo a 55 °C per 10 minuti.
- Al termine della pirolisi, posizionare la provetta da centrifuga sul telaio magnetico, lasciarla riposare e lasciare che l'adsorbimento avvenga per 1 minuto sulle perline. Dopo che il liquido nel tubo è completamente chiarificato, scartare il surnatante e cercare di evitare di disturbare le microsfere magnetiche.
- Rimuovere le provette dal rack magnetico e posizionarle su un rack per provette da centrifuga da 1,5 mL. Utilizzando una pipetta, aggiungere 500 μl di tampone di lavaggio E a ciascuna provetta, mescolare accuratamente per 1 minuto, quindi rimettere le provette nel rack magnetico. Lasciare riposare le provette per 1 minuto, scartare il surnatante dopo la chiarificazione ed evitare di aspirare le microsfere magnetiche.
- Ripetere il processo di lavaggio aggiungendo 500 μL di tampone di lavaggio W2 a ciascuna provetta. Mescolare accuratamente per 1 minuto, posizionare le provette sulla griglia magnetica e lasciarle riposare per 1 minuto. Eliminare il surnatante con cura dopo che la soluzione si è chiarificata, assicurandosi che le microsfere magnetiche rimangano indisturbate.
- Aggiungere 100 μl di eluente, mescolare e incubare a 55 °C per 5 minuti.
- Posizionare le provette sulla griglia magnetica e lasciarle riposare per 2 minuti. Una volta chiarificata la soluzione, trasferire con cura l'acido nucleico eluito in una nuova provetta da centrifuga, evitando l'aspirazione delle microsfere magnetiche.
- Valutare la purezza del DNA estratto misurando l'assorbanza a 260 nm (A260) e 280 nm (A280). Calcola il rapporto A260/A280 , che idealmente dovrebbe essere maggiore di 1,8 per il DNA ad alta purezza.
4. qPCR per la rilevazione di H. pylori e resistenza agli antibiotici
NOTA: Abbiamo eseguito la qPCR per il rilevamento dell'infezione da H. pylori amplificando il gene ureA . Tutti i prodotti per il controllo di qualità negativo sono acqua sterile e purificata. Il prodotto per il controllo di qualità positivo nel kit di rilevamento dell'acido nucleico di H. pylori è costituito da perle standard inattivate di H. pylori (ATCC 43504). A, C, T, ≤ 30 con una tipica curva a forma di S è considerata positiva.
- In base al numero di campioni da testare, estrarre il preparato liofilizzato di H. pylori dal kit e centrifugarlo rapidamente per mantenere la polvere liofilizzata sul fondo del tubo.
- Aprire con cautela il coperchio del preparato liofilizzato; prelevare l'acido nucleico del campione da sottoporre a prova; aggiungere 25 μl di acido nucleico del campione al preparato liofilizzato; e coprire bene il tubo.
- Agitare e mescolare il reagente PCR in modo uniforme per 8-10 s, quindi centrifugarlo rapidamente per 3-5 s.
- Per ciascun campione su una piastra a 32 pozzetti, preparare 25 μl della miscela di reazione PCR (polvere liofilizzata [contenente l'enzima Taq (5 U/μL), desossiribonucleosiside trifosfato (2,5 mmol/L), enzima UNG (2 U/μL), primer ureA forward (5'-ACATTGCGAGCGGGACAG-3') e reverse (5'-CGCCCAATCTCACTTTATCG-3') (40 μmol/L)]) e 25 μl (2,5 μg) del DNA estratto.
- Eseguire la scheda qPCR su piastra a 32 pozzetti sulla macchina qPCR. Programmare il termociclatore: 42 °C e 95 °C (entrambi per un ciclo) per 2 min ciascuno; 95 °C per 10 s e 65 °C per 45 s (due passaggi sono un ciclo) per 10x; 95 °C per 10 s e a 58 °C per 45 s (due fasi sono un ciclo) per 35 cicli.
- Analizza i dati utilizzando un software specifico per qPCR e lascia che lo strumento selezioni automaticamente le soglie di base.
5. Analisi statistica
- Utilizzare il test del chi-quadrato per analizzare i dati sperimentali per valutare la coerenza tra questo metodo e i risultati della qPCR e confrontare il tasso di positività di questa tecnologia di screening con i dati del tasso di positività dell'infezione da H. pylori su scala più ampia (ad esempio, a livello nazionale). Considera le differenze statisticamente significative a P < 0,05.
Risultati
Questionario
Nello studio sono stati arruolati un totale di 261 partecipanti, di cui 144 femmine e 117 maschi, di età compresa tra i 4 e i 99 anni. L'età media dei soggetti era di 48,30 ± 17,61 anni. C'erano 17 minori (0-17 anni), 202 adulti (18-64 anni) e 42 anziani (>64 anni). I risultati del questionario sono riportati nella Tabella 1. La maggioranza (90,8%) dei soggetti ha ritenuto necessario effettuare lo screening di H. pylori .
Risultati dello screening del rilevamento dell'antigene fecale di H. pylori in questo villaggio
Tra i 261 partecipanti testati per l'antigene fecale di H. pylori , 52 (19,92%) sono risultati positivi, mentre 209 sono risultati negativi. Dei 52 casi positivi, 32 erano donne, che rappresentavano il 22,22% di tutti i partecipanti di sesso femminile, e 20 erano uomini, che rappresentavano il 17,09% di tutti i partecipanti di sesso maschile. Stratificati per età, i tassi di positività sono stati i seguenti: i minori (0-17 anni) hanno avuto 2 casi positivi (11,76%), gli adulti (18-64 anni) hanno avuto 41 casi positivi (20,30%) e gli anziani (>64 anni) hanno avuto 9 casi positivi (21,43%).
Confronto della coerenza tra l'antigene fecale di H. pylori e i risultati della qPCR
In questo studio, sono stati selezionati due campioni con diversi risultati del test qPCR per caratterizzare l'affidabilità del protocollo sperimentale (Figura 4). Tra i risultati dell'antigene fecale di H. pylori su 261 soggetti, 52 erano positivi e 209 erano negativi. Tra i risultati della qPCR con le feci di 261 soggetti, 83 erano positivi e 178 erano negativi (Tabella 2). La sensibilità e la specificità del rilevamento dell'antigene fecale di H. pylori sono state rispettivamente del 60,24% e dell'80,08%. Il valore predittivo positivo (PPV) di questo metodo è stato del 96,15% e il valore predittivo negativo (NPV) è stato dell'84,21%. Il test di coerenza Kappa indica che i risultati della diagnosi dei due metodi sono coerenti (Kappa = 0,630, P < 0,05).
Confronto del tasso di positività allo screening tra questo villaggio naturale e l'intero paese
Il tasso di positività è stato del 19,92%, leggermente inferiore al tasso di positività nazionale del 42,8%14 (Tabella 3). Secondo il test del chi-quadrato (P < 0,01), la differenza nei tassi di positività era statisticamente significativa.

Figura 1: Diagramma di flusso del rilevamento del campione. Fare clic qui per visualizzare una versione più grande di questa figura.
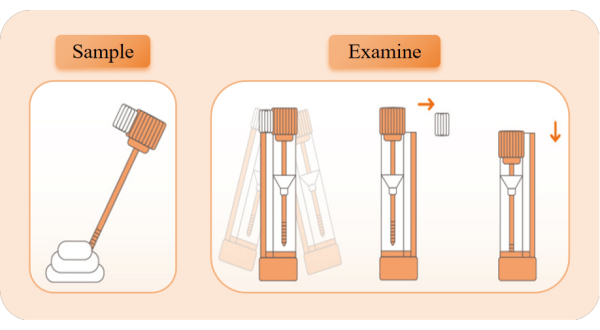
Figura 2: Il diagramma schematico della raccolta e del rilevamento dei campioni. Estrarre il dispositivo di rilevamento, aprire il sacchetto di alluminio ed estrarre il tubo del reagente. Staccare il coperchio del tubo arancione e utilizzare il bastoncino di campionamento per prelevare campioni 5 volte da diverse posizioni delle feci. Reinserire l'asta di campionamento nella provetta del reagente e agitarla a sinistra e a destra per 10 secondi per miscelarla completamente. Rompere il blocco di limite bianco, mantenere la provetta del reagente in posizione verticale e premere il coperchio della provetta nella posizione più bassa per avviare il cronometraggio. Osserva l'aspetto delle bande rosse. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Interpretazione dei risultati dell'antigene. (A) Campionatore. (B,E) Negativo (-): un risultato negativo indica che non è stato rilevato alcun antigene di H. pylori nel campione, suggerendo che il rischio di infezione da H. pylori è basso. Un risultato negativo non può escludere completamente la possibilità di infezione. (C,F) Positivo (+): un risultato positivo significa che l'antigene di H. pylori è stato rilevato nel campione, si sospetta un'infezione da H. pylori . (D) Nessun effetto: risultati non validi potrebbero essere un problema con il processo di raccolta o manipolazione del campione, oppure il reagente si è deteriorato e danneggiato e il campione deve essere raccolto nuovamente o le fasi operative devono essere ripetute. Clicca qui per visualizzare una versione più grande di questa figura.
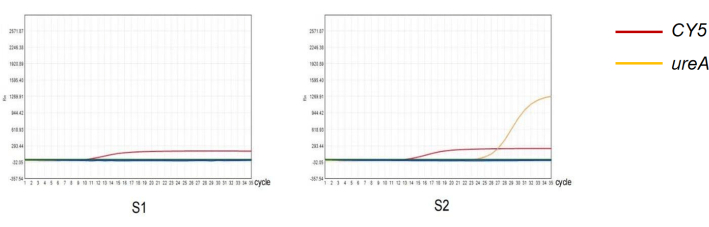
Figura 4: Rilevamento di H. pylori nelle feci con il metodo qPCR. (S1) Risultato negativo dell'amplificazione quantitativa della PCR dell'infezione da H. pylori . (S2) Risultato positivo dell'amplificazione quantitativa della PCR dell'infezione da H. pylori . Clicca qui per visualizzare una versione più grande di questa figura.
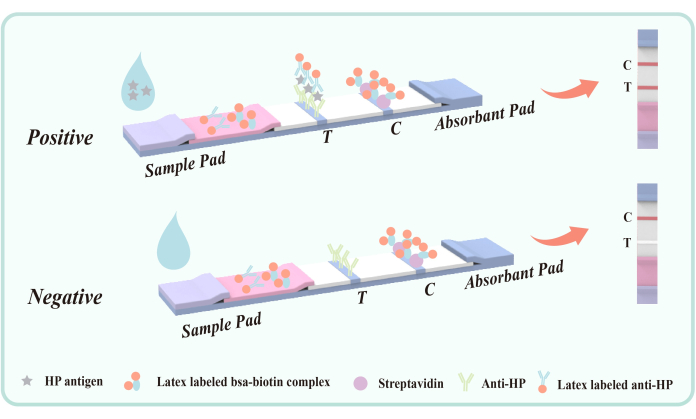
Figura 5: Diagramma schematico del metodo a sandwich di doppi anticorpi per rilevare l'antigene fecale infettato da H. pylori. L'anticorpo anti-Hp di topo (epitopo VacA) è stato fissato in anticipo nell'area di rilevamento (T) sulla membrana. L'area di controllo qualità (C) corregge in anticipo le IgG anti-topo di capra e la streptavidina. Il film di poliestere è stato rivestito con anticorpo anti-Hp di topo marcato con lattice e coniugato lattice-BSA-biotina. Clicca qui per visualizzare una versione più grande di questa figura.
| Se mangiare regolarmente per tre pasti? | Se bere alcolici? | Conosci H. pylori? | Se è necessario effettuare l'ispezione di H. pylori ? | Comprendi le conseguenze negative dell'infezione da H. pylori ? | |
| Sì (tariffa) | 236 (90.4%) | 59 (22.6%) | 117 (44.8%) | 237 (90.8%) | 104 (39.8%) |
| No (tasso) | 25 (9.6%) | 202 (77.4%) | 144 (55.2%) | 24 (9.2%) | 157 (60.2%) |
Tabella 1: I risultati del questionario.
| Antigene di H. pylori | Totale | |||
| Positivo (+) | Negativo (-) | |||
| qPCR | Positivo (+) | 50 | 33 | 83 |
| Negativo (-) | 2 | 176 | 178 | |
| Totale | 52 | 209 | 261 | |
Tabella 2: Risultati diagnostici di due tecniche.
| Positivo | Negativo | Tasso di positività | Totale | |
| Villaggio di Shitan | 52 | 209 | 19.92% | 261 |
| Cina | 314423 | 449404 | 42.80% | 763827 |
Tabella 3: Confronto dei tassi di positività allo screening di H. pylori tra il villaggio di Shitan e la media nazionale.
Discussione
L'H. pylori rappresenta una delle infezioni batteriche più diffuse in tutto il mondo15. I risultati del questionario hanno rivelato che il 55,2% della popolazione non era a conoscenza di H. pylori, mentre il 90,8% riteneva che lo screening di H. pylori fosse necessario. Questi risultati sottolineano l'importanza di implementare programmi di screening di H. pylori in aree economicamente svantaggiate e remote16. Sebbene il test dell'urea del respiro (UBT) sia un metodo diagnostico comune, il suo costo elevato e l'inadeguatezza per i bambini e le donne in gravidanza ne limitano l'applicabilità17,18. Il test degli anticorpi sierici di H. pylori può rilevare gli anticorpi nel siero di un paziente19. Tuttavia, questo test non è adatto per l'analisi post-trattamento poiché gli anticorpi rimangono a lungo dopo che i batteri sono stati eliminati20. Al contrario, il test dell'antigene delle feci offre un approccio non invasivo che elimina la necessità di accesso alla cavità gastrica, richiedendo solo campioni di feci per l'analisi. Questo metodo riduce significativamente il disagio del paziente e i potenziali rischi, rendendolo più conveniente sia per lo screening che per il follow-up. Inoltre, la sua natura non invasiva lo rende più accettabile per le popolazioni delle regioni remote e sottosviluppate.
La prevalenza di pazienti positivi all'H. pylori nella popolazione dello studio è del 19,92%, leggermente inferiore al tasso di positività nazionale del 42,8%14. Secondo il test del chi-quadrato (P < 0,01), c'era una significatività statistica tra di loro. La ragione di questa differenza potrebbe essere il tasso di infezione di H. pylori nelle popolazioni urbane in sette regioni geografiche della Cina che presenta un'evidente differenziazione regionale21. Il tasso di infezione è più alto nella Cina orientale, più alto nella Cina nord-orientale, nella Cina settentrionale e nella Cina nord-occidentale e più basso nella Cina meridionale e nella Cina sud-occidentale21. L'area che abbiamo esaminato appartiene alla Cina meridionale e il tasso di infezione è inferiore al livello nazionale. D'altra parte, l'operazione non standard nel processo di campionamento dei singoli campioni fecali e il lungo tempo per l'invio per il test possono anche essere le ragioni di questa differenza. Sebbene la PCR e i suoi derivati siano ampiamente impiegati per rilevare vari agenti patogeni, tra cui H. pylori, la loro utilità è limitata dalla complessità e dal costo delle apparecchiature per termociclatori e dalla necessità di operatori specializzati22,23. In questo studio, rispetto ai risultati della qPCR, la sensibilità e la specificità del rilevamento dell'antigene di H. pylori nelle feci sono state del 60,24% e dell'80,08%. È stata confrontata la consistenza dell'antigene di H. pylori nelle feci e i risultati della qPCR e i risultati hanno suggerito che erano moderatamente coerenti (Kappa = 0,630, P < 0,05). Le ragioni della sua sensibilità insoddisfacente possono essere influenzate dalla raccolta irregolare del campione, dalla temperatura di conservazione del campione e dai tempi di consegna.
Il metodo a sandwich a doppio anticorpo per la rilevazione dell'antigene fecale dell'infezione da H. pylori è una tecnologia di rilevamento immunologico efficiente e specifica. Il fulcro di questo metodo è quello di sfruttare la capacità di legame specifica degli anticorpi e garantire un'identificazione e una quantificazione accurate degli antigeni bersaglio attraverso un ingegnoso sistema di rilevamento. In particolare, questo approccio prevede due passaggi chiave: la cattura dell'antigene e il rilevamento degli anticorpi. Il diagramma schematico di questa tecnologia è mostrato nella Figura 5.
L'anticorpo anti-Hp di topo rivestito sulla regione di rilevamento della membrana di poliestere (T) ha come bersaglio l'epitopo VacA di H. pylori per ottenere la cattura specifica dell'antigene di H. pylori nelle feci. L'antigene VacA è un'importante esotossina di H. pylori, strettamente associata all'infezione. Gli anticorpi che hanno come bersaglio VacA possono identificare e legarsi efficacemente all'antigene, formando un complesso antigene-anticorpo. Pertanto, questo metodo non è facile da reagire in modo incrociato con altre malattie o malattie gastrointestinali.
Durante il test, il campione liquido entra nel serbatoio di rilevamento e migra verso l'alto attraverso l'azione capillare. Se il campione contiene l'antigene di H. pylori , forma prima un complesso antigene-anticorpo con l'anticorpo anti-H. pylori marcato con il lattice rivestito sulla pellicola di poliestere. Quando il complesso scorre attraverso la zona di rilevamento (T), viene catturato dall'anticorpo anti-H. pylori immobilizzato del topo, risultando in una banda rossa visibile nella zona di rilevamento (T), che indica un risultato positivo. Se nel campione non è presente alcun antigene di H. pylori , nella zona di rilevamento (T) non si forma alcun complesso sandwich di anticorpi doppi e, di conseguenza, non appare alcuna banda rossa, indicando un risultato negativo. Indipendentemente dalla presenza o dall'assenza dell'antigene di H. pylori , il coniugato lattice-BSA-biotina si legherà alla streptavidina immobilizzata sulla membrana durante la cromatografia, producendo una banda rossa nella zona di controllo qualità (C). Questa banda rossa funge sia da indicatore del corretto processo cromatografico che da standard di controllo interno per il reagente.
Il metodo del sandwich a doppio anticorpo realizza la cattura efficiente e la rilevazione qualitativa dell'antigene di H. pylori nelle feci mediante anticorpi e marcatori accuratamente progettati. La sua semplicità, rapidità e alta specificità forniscono un supporto tecnico affidabile per la diagnosi assistenziale dell'infezione da H. pylori . L'applicazione diffusa di questa tecnologia non solo riduce i costi medici e migliora l'efficienza dello screening, ma offre anche ai pazienti un'esperienza di test più confortevole e conveniente. Questo progresso ha un'importanza significativa per la prevenzione e il controllo globale dell'infezione da H. pylori .
Inoltre, l'igiene del test dell'antigene delle feci è una considerazione fondamentale. Questo metodo richiede campioni fecali freschi, che possono essere formati o non formati, senza restrizioni specifiche sui tempi di raccolta. Durante la raccolta dei campioni, è essenziale evitare la contaminazione con acqua, urina, disinfettanti o acque reflue. I campioni fecali devono essere raccolti in contenitori puliti e asciutti, privi di conservanti e detergenti. Se è necessario conservarli, i campioni possono essere conservati a temperatura ambiente per non più di 6 ore o a 2-8 °C per un massimo di 72 ore. I campioni refrigerati devono essere riportati a temperatura ambiente prima del test per garantire risultati accurati. L'elevata efficienza temporale è anche una caratteristica importante del rilevamento dell'antigene dell'infezione da H. pylori nelle feci. Il test a sandwich di doppi anticorpi a base di lattice può fornire risultati in 10-20 minuti rispetto ai metodi tradizionali che richiedono l'attesa dei risultati di laboratorio, il che è importante per il riconoscimento dell'infezione acuta e le decisioni terapeutiche precoci in aree remote e arretrate.
In sintesi, la rilevazione dell'antigene di H. pylori nelle feci offre numerosi vantaggi metodologici, tra cui non invasività, semplicità, igiene, alta efficienza e ampia applicabilità. Queste caratteristiche rendono questa metodica uno strumento prezioso per la pratica clinica e forniscono spunti innovativi per lo sviluppo futuro delle tecnologie diagnostiche. Nelle regioni sottosviluppate con condizioni sanitarie limitate, questo approccio è destinato a svolgere un ruolo fondamentale nella diagnosi e nella gestione dell'infezione da H. pylori .
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Nessuno
Materiali
| Name | Company | Catalog Number | Comments |
| DNA extraction kit | Jiangsu mole biotechnology co., ltd | 20230223 | None |
| H. pylori fecal antigen detection kit | Hangzhou nuohui healthy technology co., ltd | 20213401126 | None |
| H. pylori nucleic acid detection kit | Jiangsu mole biotechnology co., ltd | 20230226 | None |
| Real-time fluorescence quantitative PCR | Shanghai Hongshi medical treatment technology co., ltd | 20183221659 | None |
| Statistical Product and Service Solutions | IBM company | None | None |
Riferimenti
- Ali, A., AlHussaini, K. I. Helicobacter pylori: A contemporary perspective on pathogenesis, diagnosis and treatment strategies. Microorganisms. 12 (1), 222(2024).
- Kusters, J. G., van Vliet, A. H., Kuipers, E. J. Pathogenesis of Helicobacter pylori infection. Clin Microbiol Rev. 19 (3), 449-490 (2006).
- Chen, Y. C., et al. Global prevalence of Helicobacter pylori infection and incidence of gastric cancer between 1980 and 2022. Gastroenterology. 166 (4), 605-619 (2024).
- Malfertheiner, P., et al. Management of Helicobacter pylori infection-the Maastricht V/Florence Consensus Report. Gut. 66 (1), 6-30 (2017).
- Hooi, J. K. Y., et al. Global prevalence of Helicobacter pylori infection: Systematic review and meta-analysis. Gastroenterology. 153 (2), 420-429 (2017).
- Burucoa, C., Axon, A. Epidemiology of Helicobacter pylori infection. Helicobacter. 22 (Suppl 1), (2017).
- Atkinson, N. S., Braden, B. Helicobacter pylori infection: Diagnostic strategies in primary diagnosis and after therapy. Dig Dis Sci. 61 (1), 19-24 (2016).
- Owot, J. C., et al. Diagnostic performance of fecal Helicobacter pylori antigen test in Uganda. BMC Gastroenterol. 22 (1), 518(2022).
- Kornoukhova, L. A., Emanuel, V. L., Denisov, N. L., Nikonov, E. L. Helicobacter pylori infection: place of serological and cultural diagnostics in clinical guidelines. Klin Lab Diagn. 66 (8), 496-501 (2021).
- Boltin, D., et al. Correlation between quantitative 13C-urea breath test and Helicobacter pylori treatment success in a population-based cohort. Gastroenterol Res Pract. 2018, 5439539(2018).
- Gingras, B. A., Maggiore, J. A. Performance of a new molecular assay for the detection of gastrointestinal pathogens. Access Microbiol. 2 (10), acmi000160(2020).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PLoS One. 16 (8), e0256393(2021).
- Ishino, Y., Krupovic, M., Forterre, P. History of CRISPR-Cas from encounter with a mysterious repeated sequence to genome editing technology. J Bacteriol. 200 (7), e00580-e00617 (2018).
- Xie, L., et al. Prevalence of Helicobacter pylori infection in China from 2014-2023: A systematic review and meta-analysis. World J Gastroenterol. 30 (43), 4636-4656 (2024).
- Chmiela, M., Karwowska, Z., Gonciarz, W., Allushi, B., Stączek, P. Host pathogen interactions in Helicobacter pylori-related gastric cancer. World J Gastroenterol. 23 (9), 1521-1540 (2017).
- Dai, B., et al. Rapid and sensitive assay of Helicobacter pylori with one-tube RPA-CRISPR/Cas12 by portable array detector for visible analysis of thermostatic nucleic acid amplification. Front Microbiol. 13, 858247(2022).
- Zubillaga, M., et al. MIN 14C UBT: a combination of gastric basal transit and 14C-urea breath test for the detection of Helicobacter pylori infection in human beings. Nucl Med Biol. 24 (6), 565-569 (1997).
- Bentur, Y., Matsui, D., Koren, G. Safety of 14C-UBT for diagnosis of Helicobacter pylori infection in pregnancy. Can Fam Physician. 55 (5), 479-480 (2009).
- Herbrink, P., van Doorn, L. J. Serological methods for diagnosis of Helicobacter pylori infection and monitoring of eradication therapy. Eur J Clin Microbiol Infect Dis. 19 (3), 164-173 (2000).
- Laheij, R. J., Straatman, H., Jansen, J. B., Verbeek, A. L. Evaluation of commercially available Helicobacter pylori serology kits: a review. J Clin Microbiol. 36 (10), 2803-2809 (1998).
- Wang, L., Li, Z., Tay, C. Y., Marshall, B. J., Gu, B. Multicentre, cross-sectional surveillance of Helicobacter pylori prevalence and antibiotic resistance to clarithromycin and levofloxacin in urban China using the string test coupled with quantitative PCR. Lancet Microbe. 5 (6), e512-e513 (2024).
- Megraud, F., et al. Helicobacter pylori resistance to antibiotics in Europe in 2018 and its relationship to antibiotic consumption in the community. Gut. 70 (10), 1815-1822 (2021).
- Sung, H., et al. Global Cancer Statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 71 (3), 209-249 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon