Method Article
Isolement et identification de cellules souches mésenchymateuses de moelle osseuse porcine et de leurs vésicules extracellulaires dérivées
Dans cet article
Résumé
Cet article élabore une méthode pour isoler et identifier les cellules souches mésenchymateuses de la moelle osseuse porcine (pBM-MSCs) et les vésicules extracellulaires (VE) dérivées de celles-ci, fournissant une base méthodologique pour l’évaluation préclinique de l’efficacité de la transplantation de BM-MSCs et de leurs VE dérivées.
Résumé
Avec le développement de la thérapie par cellules souches dans la recherche translationnelle et la médecine régénérative, les cellules souches mésenchymateuses de la moelle osseuse (BM-MSC), en tant que sorte de cellules souches pluripotentes, sont favorisées pour leur disponibilité instantanée et leur innocuité prouvée. Il a été rapporté que la transplantation de BM-MSCs est d’un grand avantage pour la réparation des tissus blessés dans diverses maladies, qui pourraient être liées à la modulation des réponses immunitaires et inflammatoires via des mécanismes paracrines. Les vésicules extracellulaires (VE), dotées d’une structure membranaire lipidique à double couche, sont considérées comme les principaux médiateurs des effets paracrines des cellules souches. Reconnues pour leur rôle crucial dans la communication cellulaire et la régulation épigénétique, les VE ont déjà été appliquées in vivo pour l’immunothérapie. Cependant, à l’instar de ses cellules maternelles, la plupart des études sur l’efficacité de la transplantation de VE restent encore au niveau des petits animaux, ce qui n’est pas suffisant pour fournir des preuves essentielles pour l’application clinique. Ici, nous utilisons la centrifugation à gradient de densité pour isoler les cellules de moelle osseuse (BMC) de la moelle osseuse porcine dans un premier temps, et obtenons des BM-MSCs porcines (pBM-MSCs) par culture cellulaire par la suite, identifiées par les résultats de l’observation au microscope, du test de différenciation induite et de la cytométrie en flux. De plus, nous isolons avec succès les VE dérivées des pBM-MSCs dans le surnageant cellulaire par ultracentrifugation, prouvée par les techniques de microscopie électronique à transmission (MET), d’analyse de suivi des nanoparticules (NTA) et de western blot. Dans l’ensemble, les pBM-MSC et leurs VE dérivées peuvent être isolées et identifiées efficacement par les protocoles suivants, qui pourraient être largement utilisés dans les études précliniques sur l’efficacité de la transplantation des BM-MSC et de leurs VE dérivées.
Introduction
Au cours des 10 dernières années, la thérapie par cellules souches a promis de grands avantages pour les patients souffrant de diverses maladies et blessures, telles que les traumatismes, les maladies respiratoires et cardiovasculaires. Avec les progrès dans le domaine, les cellules souches mésenchymateuses de la moelle osseuse (BM-MSC) sont progressivement privilégiées par les gens pour leur accessibilité et peu de disputes éthiques1, qui ont été considérées comme l’étalon-or de la recherche clinique malgré d’autres types de cellules2. Les thérapies basées sur les BM-MSC sont également attrayantes pour de plus en plus de chercheurs en raison de leur capacité unique à moduler les réponses immunitaires et inflammatoires et à réparer les tissus lésés via des mécanismes de différenciation ou paracrines3.
Les vésicules extracellulaires (VE), comme l’approuve la Société internationale des vésiculesextracellulaires (ISEV) 4, font référence à l’ensemble des particules avec une structure bicouche lipidique qui sont naturellement libérées des cellules. Avec les découvertes récentes de divers contenus tels que les protéines, les lipides et le matériel génétique (par exemple, les miARN, les ARNm, les molécules d’ADN, ainsi que les longs ARN non codants) dans les VE de différents types cellulaires5, leurs rôles cruciaux dans la communication cellulaire et la régulation épigénétique ont été reconnus6. En tant que nouveau substitut des cellules maternelles, les VE ont été appliqués en immunothérapie et en médecine régénérative avec des études in vivo, qui servent de base à la recherche préclinique en cours et aux essais cliniques de suivi7.
Cependant, à l’heure actuelle, la plupart des études sur l’efficacité de la transplantation de BM-MSC et de leurs VE dérivées restent encore au niveau des petits animaux, ce qui ne suffit pas à fournir les preuves nécessaires à l’application clinique. Par conséquent, il est extrêmement urgent de mener des recherches précliniques sur la transplantation de BM-MSCs et de leurs VE dérivées au niveau des grands animaux tels que les porcs.
Il a été rapporté que les CSM sont présentes en nombre extrêmement faible dans la moelle osseuse, ne représentant que 0,01 % à 0,001 % du total des cellules8. Cependant, l’administration préclinique de BM-MSC nécessite un grand nombre de cellules (≥107 par animal)9 ; la quantité de VE nécessaire est encore plus importante, dont la dose médiane est de 0,25 mg de protéines par kilogramme de poids corporel chez le porc10. Pour atteindre ces grands nombres, il est urgent de disposer d’une méthode sûre et efficace pour isoler et cultiver les CSM à partir de moelle osseuse porcine afin de réaliser leur expansion massive in vitro et d’acquérir leurs VE à forte concentration en protéines par la suite.
Jusqu’à présent, il existe différentes méthodes pour isoler les BM-MSC et leurs VE dérivés. Les méthodes actuelles d’isolement des BM-MSCs comprennent la plantation directe de cellules de moelle osseuse (BMC)11, la centrifugation par gradient de densité, le tri moléculaire des marqueurs moléculaires à la surface cellulaire et le dépistage par cytométrie en flux. Il a été rapporté que le tri des marqueurs moléculaires à la surface cellulaire et le dépistage par cytométrie en flux entraînent une diminution du taux d’adhésion cellulaire, une augmentation de la mortalité sur 24 heures et une inhibition de la prolifération12, tandis que la culture directe de BMC peut entraîner un nombre élevé de cellules hématopoïétiques mixtes. Par conséquent, la centrifugation à gradient de densité est maintenant couramment utilisée pour obtenir des BM-MSCs. Les méthodes actuelles pour isoler les VE des surnageants cellulaires comprennent l’ultracentrifugation, l’ultrafiltration, la précipitation des polymères et l’exclusion stérique13. Par rapport à d’autres méthodes, l’ultracentrifugation présente l’avantage d’être peu coûteuse, facile d’utilisation et compatible avec une préparation de grand volume sans prétraitement compliqué, qui a été la « référence » pour la séparation EV14. Cependant, il existe une grande hétérogénéité dans les réactifs et les techniques entre les différents laboratoires au cours du processus 3,15, ce qui peut induire les lecteurs en erreur. Cet article explique en détail une série d’étapes séquentielles permettant d’isoler en détail les pBM-MSCs et les VE qui en sont dérivées, et les résultats d’identification ultérieurs prouvent que la méthode est réalisable pour obtenir des pBM-MSCs et leurs VE pour une analyse plus approfondie dans la recherche préclinique. Nous espérons que ce travail systématique pourra fournir une base méthodologique aux chercheurs engagés dans l’évaluation préclinique de la transplantation de pBM-MSC et de leurs VE dérivées, afin que des essais cliniques puissent être réalisés le plus tôt possible.
Protocole
Selon le Guide pour le soin et l’utilisation des animaux de laboratoire publié par les National Institutes of Health des États-Unis, toutes les procédures expérimentales ont été approuvées par le Comité institutionnel de soin et d’utilisation des animaux (IACUC) de l’hôpital Fuwai de l’Académie chinoise des sciences médicales.
1. Préparation préopératoire pour les animaux
- Procurez-vous des miniporcs chinois mâles adultes (30 ± 5 kg) à l’âge d’environ 12 mois de l’Institut de zoologie de l’Académie chinoise des sciences et hébergez-les dans les installations du Centre d’expérimentation animale de l’hôpital Fuwai, au moins 2 semaines à l’avance. Effectuer des inspections préopératoires telles que des analyses sanguines de routine pour s’assurer que les animaux sont en bonne santé.
- Nettoyez et rasez la peau de la cuisse du minipig la veille de l’opération. Pour éviter l’aspiration, faites jeûner le minipig pendant 12 h avant l’extraction de la moelle osseuse.
2. Préparation à l’isolement et à la culture des cellules
- Utilisez Percoll (1,130 g/mL) pour isoler les cellules souches mésenchymateuses de la moelle osseuse. Mélangez la solution mère avec 10 fois de PBS concentré dans un rapport de 9:1 pour obtenir un milieu isotonique. Mélangez ensuite le milieu isotonique avec du PBS dans un rapport de 3:2 pour obtenir une solution Percoll à 60 % (1,077 g/mL), qui peut être utilisée comme solution de séparation finale pour isoler les pBM-MSC.
- Préparez le milieu modifié de Dulbecco (IMDM) d’Iscove avec 10 % de sérum fœtal bovin et 1,0 % de pénicilline-streptomycine pour obtenir un milieu complet. Préchauffez le milieu complet et le PBS dans un bain-marie à 37 °C pour la culture cellulaire ultérieure.
3. Anesthésie des animaux
- Administrer une anesthésie générale avec de la kétamine (10 mg/kg) et de la xylazine (2 mg/kg) par voie intramusculaire. Effectuez rapidement l’intubation endotrachéale lorsque le mini-porc a une respiration lente et moins d’activité des membres, et préservez la respiration spontanée pour éviter l’échec de l’intubation.
- Effectuer le maintien de l’anesthésie par inhalation d’isoflurane à 2 %, avec de l’oxygène (1,5 L/min) comme gaz vecteur. Pendant l’anesthésie, surveillez la fréquence cardiaque, la respiration et la saturation en oxygène du minipig en temps réel.
4. Extraction de la moelle osseuse du mini-porc
- Placez le minipig en position latérale. Pour assurer une quantité suffisante de moelle osseuse pour la culture cellulaire ultérieure, localisez le point de ponction de la moelle osseuse au niveau du fémur proximal du miniporc (figure 1). Désinfectez et drapez la peau de la zone de ponction avant l’application de l’opération.
- Serrez la peau autour du point de ponction avec la main non dominante et percez l’aiguille de biopsie de la moelle osseuse verticalement au point avec la main dominante. Lorsque vous sentez l’aiguille en contact avec l’os cortical, tournez doucement la poignée vers la gauche et la droite pour percer l’aiguille. Lorsque l’aiguille pénètre dans la cavité de la moelle osseuse, il y a souvent une sensation de perte de résistance.
- Une fois la ponction en place, retirez l’aiguille centrale. Fixez ensuite une seringue stérile jetable de 50 ml à l’extrémité de l’aiguille extérieure et rincez à l’avance la paroi intérieure de la seringue avec de l’héparine.
- Extrayez lentement 20 ml de moelle osseuse et transférez-les soigneusement dans un tube à centrifuger stérile de 50 ml.
- Une fois la procédure terminée, retirez l’aiguille et retirez le drapé. Désinfectez le site de ponction et appuyez dessus pendant 20 min pour l’hémostase.
- Extubez le mini-porc après avoir rétabli la respiration spontanée. Lorsqu’il est complètement éveillé avec un mouvement libre des membres, remettez le mini-cochon dans la cage pour continuer à se nourrir.
5. Isoler les cellules souches mésenchymateuses de la moelle osseuse
- Ajoutez un volume égal de PBS préchauffé à la moelle osseuse et mélangez-les soigneusement. Ensuite, à l’aide d’une pipette stérile, transférez soigneusement 20 ml de moelle osseuse diluée au-dessus du niveau de solution à gradient de densité de 60 % à un rapport de volume de 1:1 dans un tube à centrifuger stérile de 50 ml.
- Centrifuger le tube à 600 x g (accélération (ACC) = 5, décélération (DEC) = 5) pendant 20 min à température ambiante (RT).
REMARQUE : Quatre phases se forment dans le tube après la centrifugation, y compris la phase sérum, la phase de cellule mononucléaire, la phase moyenne à gradient de densité et la phase de précipitation de haut en bas. La phase cellulaire mononucléée est une fine couche floculante entre le sérum et le milieu à gradient de densité. Les cellules souches mésenchymateuses dérivées de la moelle osseuse sont localisées dans cette phase. - Aspirer la phase de cellule mononucléaire dans un tube à centrifuger stérile de 15 mL et laver deux fois avec du PBS à 800 x g pendant 5 min.
- Remettre en suspension les cellules lavées dans 2 mL de milieu complet et planter les cellules remises en suspension dans une fiole deculture de 2 cellules de 175 cm à une densité de 3-5 x 105/mL.
REMARQUE : Toutes les procédures ci-dessus pour isoler les cellules souches mésenchymateuses sont illustrées à la figure 2.
6. Cultiver des cellules souches mésenchymateuses in vitro
- Incuber le ballon de culture à 37 °C dans une atmosphère saturée et humidifiée à 5 % de CO2. Agitez doucement le ballon de culture toutes les 24 h pour empêcher la croissance adhérente des cellules souches hématopoïétiques précipitées, et observez la croissance, la morphologie et la contamination cellulaires au microscope.
- Remplacez le milieu de culture pour la première fois après 3 jours, puis changez le milieu tous les 2-3 jours. Lorsque les colonies cellulaires atteignent une confluence de 80 à 90 %, sous-cultivez les cellules dans un rapport de 1:2.
7. Différenciation adipogénique, ostéogénique et chondrogénique des pBM-MSC
- Essai de différenciation adipogénique
- Préparez les milieux de différenciation adipogénique A (milieu A) et B (milieu B) pour les BM-MSC selon les instructions du kit. Des informations spécifiques sur le kit se trouvent dans la table des matériaux.
- Ajouter 1 mL de gélatine à 0,1 % dans la plaque à six puits et agiter doucement pour couvrir uniformément le fond de chaque puits. Placez ensuite la plaque à six puits dans un banc propre ou un incubateur de CO2 pendant au moins 30 min.
- Après 30 min, aspirez la gélatine et ajoutez 2 mL de milieu complet général dans chaque puits. Plantez ensuite les pBM-MSC dans la plaque à six puits à une densité cellulaire de 2 x 104 cellules/cm2. Après cela, incubez la plaque à 37 °C dans une humidité saturée de 5 % CO2.
- Lorsque les cellules atteignent 100 % de confluence, retirez soigneusement le milieu complet et ajoutez 2 mL de milieu A dans chaque puits de la plaque. Après 3 jours, retirer le milieu A de la plaque et ajouter 2 ml de milieu B dans chaque puits.
- Après avoir maintenu pendant 1 jour, retirez le milieu B et remplacez-le par le milieu A pour l’induction. Selon la méthode « Medium A pendant 3 jours, Medium B pendant 1 jour », utilisez Medium A et B séquentiellement pour l’induction.
- Observez l’état cellulaire tous les jours pendant les règles. Si les cellules rétrécissent ou meurent pendant le processus d’induction du milieu A, remplacez-le par le milieu B à temps jusqu’à ce que l’état de la cellule se rétablisse.
- Répétez le processus d’induction et d’entretien, et préparez-vous à la coloration lorsque suffisamment de gouttelettes de lipides de taille appropriée sont observées au microscope.
- Retirez le milieu de différenciation adipogénique dans la plaque à six puits et lavez doucement avec 1x PBS. Ajouter 2 ml de solution de paraformaldéhyde à 4 % dans chaque puits et fixer pendant 30 min à RT.
- Retirez le fixateur de paraformaldéhyde et lavez-le avec 1x PBS deux ou trois fois pour vous assurer que le fixateur est bien éliminé. Ajouter 2 ml de colorant Oil Red O dans chaque puits et teindre pendant 30 min à RT.
- Retirez le colorant Oil Red O et lavez-le avec 1x PBS deux ou trois fois. Ajouter 2 mL de 1x PBS dans chaque puits, puis observer l’effet de la différenciation adipogénique au microscope.
- Essai de différenciation ostéogénique
- Préparez le milieu complet pour la différenciation ostéogénique selon les instructions du kit.
- Suivez les étapes 7.1.2-7.1.3 pour planter et cultiver des pBM-MSC.
- Lorsque les cellules atteignent 70 % de confluence, retirer soigneusement le milieu complet général et ajouter 2 ml de milieu pour la différenciation ostéogénique à chaque puits de la plaque.
- Passer à un milieu de différenciation ostéogénique frais tous les 3 jours. Poursuivez l’induction pendant 2 à 4 semaines et préparez-vous à colorer avec le colorant rouge d’alizarine lorsque des nodules calciques évidents apparaissent pendant l’ostéogenèse.
- Retirez le milieu de différenciation ostéogénique dans la plaque à six puits et lavez doucement avec 1x PBS. Ajouter 2 ml de solution de paraformaldéhyde à 4 % dans chaque puits et fixer pendant 30 min à RT.
- Retirez le fixateur de paraformaldéhyde et lavez-le avec 1x PBS deux ou trois fois pour vous assurer que le fixateur est bien nettoyé. Ajouter 2 mL de colorant rouge d’alizarine dans chaque puits et colorer pendant 10 minutes à RT.
- Retirez le colorant rouge d’alizarine et lavez avec 1x PBS deux ou trois fois. Ajoutez 2 ml de 1x PBS dans chaque puits, puis observez l’effet de la différenciation ostéogénique au microscope.
- Essai de différenciation chondrogénique
- Préparez le prémélange pour la différenciation chondrogénique selon les instructions du kit.
- Transvaser 3-4 x 105 pBM-MSC dans un tube à centrifuger stérile de 15 ml. Centrifugeuse à 250 x g pendant 4 min à 20 °C.
- Retirer le surnageant et ajouter 0,5 mL de prémélange pour remettre en suspension la pastille obtenue par centrifugation à l’étape précédente, puis centrifuger à 150 x g pendant 5 min à 20 °C. Répétez cette étape pour laver à nouveau les cellules.
- Préparez le milieu complet pour la différenciation chondrogénique.
- Remettre en suspension la cellule obtenue à l’étape précédente avec 0,5 mL de milieu complet et centrifuger à 150 x g pendant 5 min à 20 °C.
- Dévissez le capuchon du tube de centrifugation pour faciliter l’échange gazeux. Placez-le debout dans un incubateur à 37 °C, 5 % de CO2 et une humidité saturée.
- Lorsque les cellules semblent s’agréger (généralement après 24 à 48 heures, selon la situation réelle), effleurez le bas du tube de centrifugation pour que les boules de cartilage se détachent du fond et restent en suspension dans le milieu.
- Passer à un milieu complet frais pour la différenciation chondrogénique tous les 2-3 jours. Continuez l’induction jusqu’à ce que des boules de cartilage d’un diamètre de 1,5 à 2 mm se forment dans le tube, puis préparez des sections pour la coloration.
- Préparez des coupes de paraffine de boules de cartilage selon les étapes de routine des expériences pathologiques. Ajoutez le colorant bleu Alicia sur les sections déparaffinés et faites teindre à 37 °C pendant 1 h.
- Rincez la lame à l’eau courante pendant 5 min, puis observez l’effet de coloration du bleu Alicia au microscope après séchage.
8. Identification du phénotype cellulaire par cytométrie en flux
- Lorsque les colonies cellulaires atteignent une confluence de 80 à 90 % aux passages 3 à 5, retirez le milieu de culture et lavez les cellules deux fois avec du PBS préchauffé. Ensuite, digérez les cellules avec 3 à 4 ml de trypsine à 0,25 %/EDTA et incuberez-les à 37 °C dans une atmosphère saturée et humidifiée à 5 % de CO2 pendant 2 à 3 minutes jusqu’à ce qu’elles soient détachées du fond du ballon au microscope.
- Récoltez les cellules avec un milieu complet de 10 ml et transférez la suspension cellulaire dans un tube à centrifuger stérile de 15 ml. Centrifuger la suspension cellulaire à 800 x g pendant 5 min à RT. Jeter le surnageant et laver les cellules avec du PBS à 4 °C.
- Remettre les cellules en suspension à 10 mL avec du PBS à 4 °C. La suspension cellulaire est divisée en neuf groupes d’un volume de 1 mL dans chaque microtube de 1,5 mL, appelés respectivement groupe de contrôle négatif, contrôle d’isotype FITC, contrôle d’isotype PE, contrôle d’isotype APC, groupe CD105, CD29, CD90, CD14 et CD45. Assurez-vous que le nombre de cellules dans chaque microtube est compris entre 1 x 105 et 1 x 106.
- Centrifuger la suspension à 800 x g pendant 5 min à 4 °C, et remettre les cellules en suspension dans chaque microtube avec 100 μL de PBS à 4 °C à nouveau. À l’exception du groupe témoin négatif, ajouter 5 μL du contrôle isotypique correspondant (contrôle de l’isotype FITC, PE et APC IgG1 kappa de souris) et des anticorps (anticorps monoclonaux CD105, CD29, CD90, CD14 et CD45) pour la cytométrie en flux dans chaque microtube selon l’ordre de l’étape 8.3. Mélanger doucement et incuber pendant 1,5 h à 4 °C dans l’obscurité.
- Ajouter 1 mL de PBS à 4 °C dans chaque microtube et centrifuger à 300 x g pendant 10 min à 4 °C. Jeter le surnageant et ajouter 200 μL de PBS à 4 °C pour remettre les cellules en suspension.
- Testez au moins 10 000 cellules sur la cytométrie en flux après avoir filtré la suspension cellulaire16 et analysez les données à l’aide du logiciel de cytométrie en flux.
9. Isolement des vésicules extracellulaires (VE) dérivées de cellules souches mésenchymateuses de moelle osseuse porcine
- Lorsque la confluence pBM-MSCs atteint 80 % à 90 %, jetez le surnageant et lavez les cellules avec du PBS 2x. Ajouter ensuite 25 mL d’IMDM sans sérum dans chaque flacon de culture et poursuivre l’incubation à 37 °C dans une atmosphère humidifiée à 5 % de CO2 pendant 48 h.
- Recueillir le surnageant cellulaire (milieu conditionné, CM) dans un tube à centrifuger de 50 mL et centrifuger à 300 x g pendant 10 min à 4 °C pour éliminer les débris cellulaires.
- Prélever à nouveau le surnageant dans un autre tube à centrifuger de 50 ml. Effectuez l’isolement des VE dès que possible après avoir collecté le surnageant. Pour un stockage à long terme, conservez le surnageant au réfrigérateur à -80 °C pour éviter la perte de VE.
- Centrifuger le surnageant à l’étape 9.3 à 2 000 x g pendant 20 min à 4 °C.
- Transférez le surnageant dans un tube stérile utilisé pour la centrifugeuse à grande vitesse et la centrifugeuse à 16 500 x g pendant 30 min à 4 °C.
- Transférez à nouveau le surnageant dans un tube d’ultracentrifugation et centrifugez-le à 120 000 x g pendant au moins 70 min à 4 °C, avec un rotor à angle fixe.
- Jetez complètement le surnageant. Ajouter 1 mL de PBS à 4 °C dans chaque tube d’ultracentrifugeuse et remettre la précipitation en suspension à l’aide d’une micropipette. Mélangez la solution du même groupe dans un tube d’ultracentrifugation, puis ajoutez 4 °C PBS pour porter le volume supérieur aux 3/4 du tube.
- Centrifuger à 120 000 x g pendant 60 min à 4 °C et éliminer le surnageant autant que possible.
- Remettez le précipitant en suspension avec du PBS stérile et conservez les VE dans un réfrigérateur à -80 °C.
REMARQUE : La quantité de PBS pour la remise en suspension peut être déterminée à 100 μL pour les précipitations à partir de deux flacons de culture de175 cm 2 . Toutes les étapes ci-dessus pour isoler les VE sont systématiquement illustrées à la figure 3 .
10. Identification des VE par microscopie électronique à transmission (MET), analyse du suivi des nanoparticules (NTA) et transfert Western
- Placez l’échantillon de VE sur de la glace après l’avoir décongelé dans un bain d’eau à 25 °C, puis diluez-le à la moitié de la concentration précédente avec du PBS pour la détection NTA17.
- Pipetez 10 μL de l’échantillon sur le treillis de cuivre, laissez-le reposer pendant 1 min et absorbez le liquide flottant avec du papier filtre. Ensuite, ajoutez 10 μL d’acide phosphotungstique au treillis de cuivre, laissez-le reposer pendant 1 minute et absorbez l’excès de liquide.
- Après séchage pendant quelques minutes à RT, effectuer une imagerie par microscopie électronique dans des conditions de tension d’accélération de 100 KV18.
- Remettre en suspension l’échantillon EVs de l’étape 9.9 dans le tampon de lyse RIPA (25 mM Tris· HCl (pH 7,6), chlorure de sodium [NaCl] 150 mM, phénoxypolyéthoxyléthanol 1 % de nonyle (NP-40), désoxycholate de sodium à 1 %, dodécylsulfate de sodium (SDS) à 0,1 %, fluorure de phénylméthanesulfonyle (PMSF), inhibiteur de protéase 1x), et détectent l’expression de marqueurs spécifiques des VE tels que Alix, TSG101, CD81 et CD63 par western blot19.
Résultats
Mise en place de cellules souches mésenchymateuses de moelle osseuse porcine
Des cellules souches mésenchymateuses dérivées de la moelle osseuse porcine ont été isolées et cultivées in vitro, et la morphologie des CSM-pBM à différents jours peut être observée sur la figure 4. Dans la culture primaire de pBM-MSC, l’observation microscopique a montré que l’adhérence cellulaire se produisait un jour après la plantation et que les cellules adhérentes étaient généralement de forme ronde. Les pBM-MSC primaires sont généralement restées en phase de repos pendant 3 jours après la plantation, et la prolifération cellulaire a commencé le 4ème jour. La morphologie cellulaire est passée de ronde à fusiforme, multilatérale ou en étoile après la prolifération, et les noyaux sont centraux, avec des nucléoles doubles dans certaines cellules. Les colonies cellulaires se sont formées 7 à 9 jours après le début de la prolifération cellulaire, et 80 à 90 % de la confluence cellulaire a pu être obtenue à 12 à 14 jours. L’observation microscopique a montré que les cellules adhérentes se développaient en colonies dispersées et étaient disposées selon un motif tourbillonnant.
La prolifération cellulaire a été considérablement accélérée après le passage, et une confluence de 80 à 90 % a pu être atteinte en une semaine. La morphologie cellulaire était homogène, en forme de fuseau à partir du deuxième passage, ressemblant à des fibroblastes, avec un rapport longueur/largeur d’environ 2-3:1. Si les cellules étaient différenciées, elles pouvaient apparaître polygonales ou en forme d’étoile. Après le passage, les cellules ne se sont plus développées en colonies dispersées, mais uniformément et radialement dans un arrangement parallèle.
Identification du potentiel de différenciation cellulaire par coloration
Dans l’essai de différenciation adipogénique, la coloration au rouge d’huile O a montré que des gouttelettes lipidiques rondes rouge-orange de différentes tailles apparaissaient autour du noyau (Figure 5A) ; Dans l’essai de différenciation ostéogénique, la coloration au rouge d’alizarine a montré des nodules rouges à la surface de la cellule (Figure 5B), qui ont été causées par la réaction de couleur avec les sels de calcium déposés par les ostéoblastes différenciés des pBM-MSC. Dans l’essai de différenciation chondrogénique, la coloration au bleu d’Alicia a montré que toute la section du tissu était bleue (Figure 5C), ce qui était causé par la coloration du mucopolysaccharide endo-acide dans les boules de cartilage.
Identification du phénotype cellulaire par cytométrie en flux
Des tests des marqueurs de surface cellulaire ont été effectués pour créer un phénotype des pBM-MSC. D’après les résultats de la cytométrie en flux (figure 6), trois marqueurs positifs tels que CD105, CD29 et CD90 ont été exprimés de manière significative à la surface des pBM-MSC, représentant respectivement 96,5 %, 99,8 % et 92 % (figure 6A-C). Cependant, l’expression de CD14 et CD45 était négative (Figure 6D,E). Pendant ce temps, les résultats des contrôles d’isotypes correspondants étaient tous négatifs, ce qui a déjà été superposé dans la figure, excluant la possibilité d’une liaison non spécifique des anticorps.
Identification des VE dérivées des pBM-MSC par NTA, TEM et western blot
Le résultat de la NTA a montré que la taille médiane des particules était de 126,9 nm, ce qui se situait dans la plage des VE. en outre, la concentration initiale de l’échantillon d’EV était de 1,5 x10 10 particules/mL, et la valeur précise attribuée à la taille se trouve dans la figure 7A. Le diagramme de trajectoire des particules est illustré à la figure 7B, illustrant que les particules étaient en mouvement brownien irrégulier. De plus, la vésicule discoïde, en tant que structure classique des VE, pouvait être clairement vue au microscope électronique à des grossissements de 50 000x (Figure 7C). De plus, l’expression de marqueurs spécifiques pour les EV tels que Alix, TSG101, CD81 et CD63 a été détectée dans l’échantillon par western blot (Figure 7D).
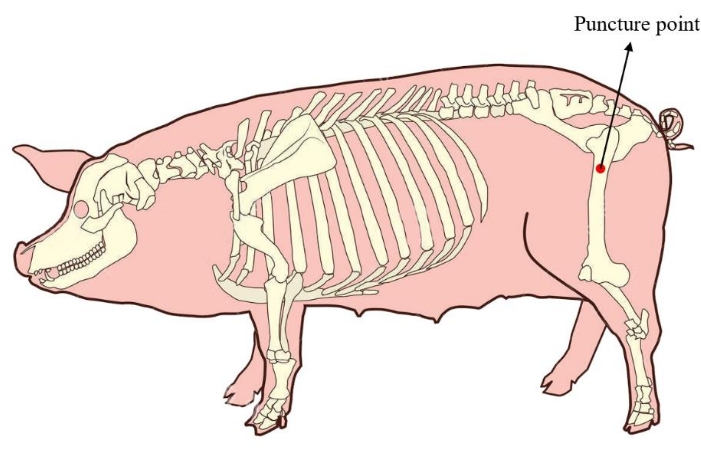
Figure 1 : Point de ponction de la moelle osseuse du mini-porc. La zone rouge montre le point de ponction de l’extraction de la moelle osseuse, situé au niveau du fémur proximal du miniporc. Veuillez cliquer ici pour voir une version agrandie de cette figure.
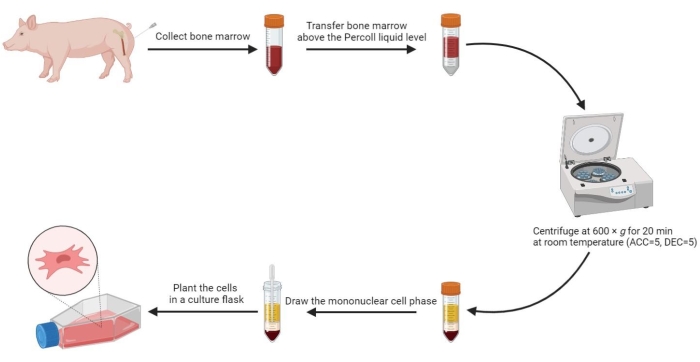
Figure 2 : Isolement de cellules souches mésenchymateuses de la moelle osseuse porcine. Le processus d’isolement des cellules souches mésenchymateuses de la moelle osseuse porcine est illustré dans l’organigramme, et quatre phases liquides sont clairement illustrées après centrifugation par gradient de densité. Veuillez cliquer ici pour voir une version agrandie de cette figure.
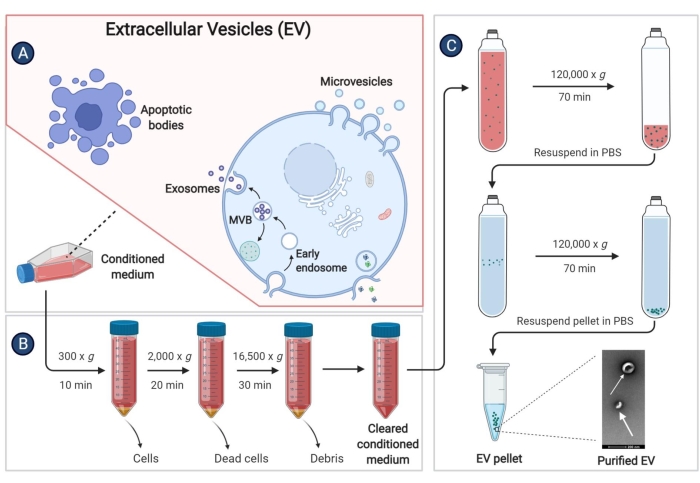
Figure 3 : Isolement des VE dérivés des pBM-MSC. Le schéma montre des étapes spécifiques pour isoler les VE du milieu conditionné par ultracentrifugation. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Caractéristiques morphologiques des pBM-MSC à différents jours. Des caractéristiques morphologiques similaires des pBM-MSC peuvent être observées le 3e, 5e, 7e et 9ejour après la plantation sous le champ microscopique 100x, et des colonies cellulaires se sont formées le 9ejour. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 5. Identification du potentiel de différenciation des pBM-MSCs par coloration. (A) Essai de différenciation adipogénique, (B) ostéogène et (C) chondrogénique des CSM-pBM, respectivement. Le potentiel de différenciation des pBM-MSC peut être identifié par ces résultats de coloration. Veuillez cliquer ici pour voir une version agrandie de cette figure.
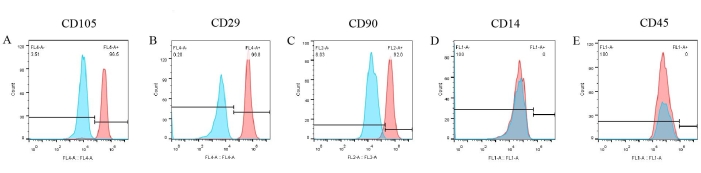
Figure 6 : Résultats d’identification des pBM-MSC par cytométrie en flux. CD105, CD29 et CD90 sont exprimés de manière significative à la surface des pBM-MSC, représentant respectivement 96,5 %, 99,8 % et 92,0 %, tandis que l’expression de CD14 et CD45 est négative. Veuillez cliquer ici pour voir une version agrandie de cette figure.
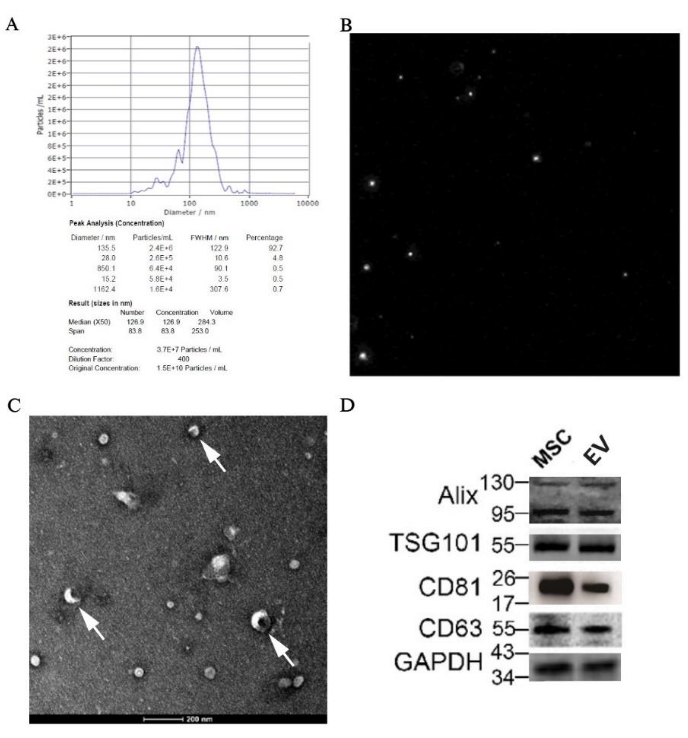
Figure 7 : Résultats d’identification des VE dérivées des pBM-MSCs par morphologie et biologie moléculaire. (A) résultat NTA des VE dérivées des pBM-MSC, avec graphique de distribution granulométrique et (B) diagramme de trajectoire des particules, respectivement ; (C) Image TEM prise à des grossissements de 50 000x, et la flèche blanche montre la structure classique des vésicules discoïdes. (D) Expression de marqueurs spécifiques des VE par western blot. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Le point de ponction traditionnel de la moelle osseuse des miniporcs était positionné à la crête iliaque20. Bien qu’il soit facile à localiser, la quantité d’extraction de moelle osseuse est limitée à21 (seulement environ 5 mL en général), il est donc difficile de répondre à l’exigence d’un grand nombre d’expansions in vitro pour la transplantation in vivo. Dans cette méthode, nous avons repositionné le point de ponction de la moelle osseuse sur le fémur proximal, et au moins 20 ml de moelle osseuse peuvent être extraits de ce site, garantissant une quantité suffisante de pBM-MSCs pour la culture cellulaire ultérieure.
Les deux principales solutions de séparation utilisées pour isoler les BM-MSC par centrifugation à gradient de densité sont Percoll et Ficoll. Percoll est composé de polyvinylpyrrolidone (PVP) siliconé, qui est un nouvel agent centrifuge à gradient de densité non toxique et non irritant. La faible constante de diffusion de Percoll se traduit par un gradient de densité relativement stable ; Par conséquent, une séparation cellulaire satisfaisante peut généralement être obtenue en quelques dizaines de minutes avec de faibles forces centrifuges (200-1000 x g). La méthode d’isolement des CSM-pBM à l’aide de Ficoll a déjà été signalée21. Par rapport au Ficoll, Percoll a été progressivement utilisé en raison de ses avantages d’isoperméabilité facile, de faible viscosité, de non-toxicité et de ne pas provoquer d’agrégation cellulaire, ce qui peut compléter les méthodes existantes d’isolement des pBM-MSC.
Lors de l’isolement et de la culture des pBM-MSC, certaines étapes critiques ne peuvent être ignorées. Tout d’abord, la stratification réussie des différentes phases liquides après centrifugation à gradient de densité est la clé de l’isolement des pBM-MSC purifiés. Les BM-MSC, en tant que type de cellules mononucléées de la moelle osseuse (BM-MNC), ont une densité similaire à celle des lymphocytes et des monocytes, soit environ 1,075 g/mL. La masse volumique d’origine de Percoll est de 1,130 g/mL, et pour obtenir avec succès la couche cellulaire contenant les BM-MSC après centrifugation par gradient de densité, une solution de séparation par gradient de densité isotonique à 60 % (1,077 g/mL) doit être configurée à l’avance selon la relation densité-concentration de Percoll22. De plus, des conditions de centrifugation appropriées contribuent également à une stratification réussie. Compte tenu de la faible constante de diffusion de Percoll, nous avons centrifugé la moelle osseuse extraite à 600 x g pendant 20 min à des niveaux d’accélération/décélération relativement faibles (ACC = 5, DEC = 5), ce qui a permis d’obtenir un bon effet de stratification. Deuxièmement, une densité de plantation appropriée est également essentielle pour la culture cellulaire. Afin d’acquérir un nombre suffisant de CSM (généralement plus de 107 par animal9) pour la transplantation ultérieure, nous utilisons des flacons de culture de 175 cm2 pour la culture cellulaire. Dans une étude précédente20, les BM-MNCs obtenues étaient généralement plantées dans des flacons de culture pour la culture à une densité de 5 x 105/cm2. Il a été rapporté qu’après centrifugation à gradient de densité, on peut obtenir 2-3 x 107 BM-MNCs pour 5 mL de moelle osseuse porcine23. Ainsi, dans ce protocole, nous recommandons de planter des BM-MNC totales isolées de chaque 20 mL de moelle osseuse porcine dans un ballon de culture de175 cm 2 pour une densité appropriée. Troisièmement, les impuretés doivent être évitées lors de l’isolement et de la culture des CSM-pBM. Lors du tirage de la phase de cellule mononucléaire, la pipette ne doit pas être insérée dans la phase Percoll afin de ne pas se mélanger avec le liquide de séparation. De plus, après 24 h de plantation des cellules, le ballon de culture doit être doucement secoué pour réduire l’adhérence des globules rouges.
Au cours du processus d’ultracentrifugation, des niveaux élevés d’agrégats de protéines et de contamination par les lipoprotéines par cette méthode compromettent inévitablement la quantification et l’analyse fonctionnelle des VE14. Afin de réduire autant que possible la contamination au cours du processus, une profondeur de liquide de 5 mm dans le fond doit être conservée à chaque fois lors du transfert du surnageant avant l’étape d’ultracentrifugation. Pendant ce temps, après la première ultracentrifugation, la remise en suspension de la pastille dans du PBS stérile, puis l’ultracentrifugation à nouveau, peuvent réduire efficacement la contamination par les lipoprotéines.
Bien que la centrifugation à gradient de densité et l’ultracentrifugation aient été largement utilisées pour isoler les BM-MSC et leurs VE dérivées, respectivement, ces deux techniques ont également leurs propres limites. D’une part, la technique Percoll est longue et lourde, et l’obtention d’un échantillon de concentré de BM via un dispositif de concentration de cellules de chevet a été signalée comme une méthode alternative pour isoler les CSM24. D’autre part, la méthode d’ultracentrifugation nécessite non seulement des techniciens hautement qualifiés, mais également un équipement coûteux ; par conséquent, l’application combinée de deux techniques ou plus peut constituer une stratégie raisonnable pour une isolation plus efficace des VE25. En outre, l’identification des pBM-MSC et des VE qui en découlent doit également être améliorée. Par exemple, selon les critères internationaux de définition des CSM26, l’expression de certains marqueurs positifs ou négatifs, tels que CD73, CD34 et HLA-DR, est encore absente des résultats d’identification des phénotypes de CSM BM par cytométrie en flux dans cette étude. De plus, bien que des mesures aient été prises pour éviter la contamination lors du processus d’isolement des VE, en raison des limites de notre laboratoire, nous ne sommes pas en mesure d’évaluer la pureté de l’échantillon de VE afin d’améliorer le travail de suivi.
Cette étude combine des méthodes d’isolement séquentiel des pBM-MSCs et de leurs VE dérivées, prouvées par les résultats d’identification ultérieurs systématiques. En particulier, nous avons mis en évidence les opérations clés de cette série d’étapes, en expliquant certaines conditions expérimentales spécifiques qui peuvent résoudre le problème de l’hétérogénéité existant dans différents laboratoires au cours de ce processus dans une certaine mesure. Ce travail méthodique pourrait être largement utilisé dans les études précliniques sur l’efficacité de la transplantation des BM-MSC et de leurs VE dérivées, ce qui pourrait fournir une base expérimentale d’un niveau suffisant pour la recherche clinique.
Déclarations de divulgation
Tous les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Nous remercions Yang Jianzhong et Wang Xuemin pour leurs contributions à l’opération d’extraction de la moelle osseuse. Ce travail a été soutenu par des subventions du Fonds d’innovation CAMS pour les sciences médicales (CIFMS) [numéro de subvention 2016-I2M-1-009], Fondation nationale des sciences naturelles de Chine (n° : 82070307 ; n° : 81874461).
matériels
| Name | Company | Catalog Number | Comments |
| 175 cm2 cell culture flask | Thermo Fisher | 159910 | used for cell culture |
| 0.25% Trypsin/EDTA | Thermo Fisher | 25200056 | used to digest cells |
| Adipogenic Differentiation Kit for Bone Marrow Mesenchymal Stem Cell | OriCell | GUXMX-90031 | used for adipogenic differentiation assay |
| Alix Monoclonal Antibody | Thermo Fisher | MA1-83977 | used to identify extracellular vesicles(Evs) by western blotting |
| APC Mouse IgG1 kappa Isotype Control | Thermo Fisher | 17-4714-42 | used to eliminate the effects of non-specific staining in flow cytometry |
| CD105 (Endoglin) Monoclonal Antibody | Thermo Fisher | 17-1057-42 | used to identify pBM-MSCs by flow cytometry |
| CD14 Monoclonal Antibody | Thermo Fisher | MA1-82074 | used to identify pBM-MSCs by flow cytometry |
| CD29/IGTB1 Monoclonal Antibody | Thermo Fisher | MA1-19458 | used to identify pBM-MSCs by flow cytometry |
| CD45 Monoclonal Antibody | Thermo Fisher | MA5-28383 | used to identify pBM-MSCs by flow cytometry |
| CD63 Monoclonal Antibody | Thermo Fisher | 10628D | used to identify EVs by western blotting |
| CD81 Monoclonal Antibody | Thermo Fisher | MA5-13548 | used to identify EVs by western blotting |
| CD90 Monoclonal Antibody | Thermo Fisher | A15794 | used to identify pBM-MSCs by flow cytometry |
| Chondrogenic Differentiation Kit for Bone Marrow Mesenchymal Stem Cell | OriCell | GUXMX-90041 | used for chondrogenic differentiation assay |
| Fetal Bovine Serum | Gibco | 10099141C | used to prepare complete medium |
| FITC Mouse IgG1 kappa Isotype Control | Thermo Fisher | 11-4714-42 | used to eliminate the effects of non-specific staining in flow cytometry |
| Flow cytometry | BD | Accuri C6 | used for identification of cell phenotype |
| FlowJo software | BD | V10 | used to analyze data from flow cytometry |
| High-speed centrifuge tube (50 mL) | Beckman | 357003 | used for high-speed centrifugation |
| Iscove's Modified Dulbecco's Medium | Gibco | C12440500BT | used for cell culture |
| Low-temperature high-speed floor centrifuge | Avanti | J-26XPI | used for high-speed centrifugation to obtain clean conditioned medium |
| Nonyl phenoxypolyethoxylethanol (NP-40) | Sigma-Aldrich | NP40S | used for the composition of RIPA lysis buffer |
| Osteogenic Differentiation Kit for Bone Marrow Mesenchymal Stem Cell | OriCell | GUXMX-90021 | used for osteogenic differentiation assay |
| PE Mouse IgG1 kappa Isotype Control | Thermo Fisher | 12-4714-42 | used to eliminate the effects of non-specific staining in flow cytometry |
| Percoll | Cytiva | 17089102 | used to isolate porcine bone marrow mesenchymal stem cells(pBM-MSCs) |
| Phenylmethanesulfonyl fluoride (PMSF) | Thermo Scientific | 36978 | used for the composition of RIPA lysis buffer |
| Phosphate Buffered Saline(10x) | Beyotime | ST476 | used to prepare isotonic Percoll solution |
| Phosphate Buffered Saline(1x) | Cytiva | AF29561133 | used to dilute Percoll and wash cells |
| Protease inhibitor (1x) | Thermo Scientific | A32955 | used for the composition of RIPA lysis buffer |
| sodium chloride | Sigma-Aldrich | S9888 | used for the composition of RIPA lysis buffer |
| sodium deoxycholate | Sigma-Aldrich | D6750 | used for the composition of RIPA lysis buffer |
| sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | used for the composition of RIPA lysis buffer |
| Transmission electron microscopy | Hitachi | HT7700 | used for electron microscopy imaging |
| Tris·HCl | Sigma-Aldrich | 93363 | used for the composition of RIPA lysis buffer |
| TSG101 Monoclonal Antibody | Thermo Fisher | MA1-23296 | used to identify EVs by western blotting |
| Ultracentrifuge (Type 50.2 Ti Rotor) | Beckman | optima L-100XP | used for ultracentrifugation to isolate exosomes |
| Ultracentrifuge tube (26.3 mL) | Beckman | 355654 | used for ultracentrifugation |
| ZetaVIEW | Particle Metrix | S/N 17-310 | used for Nanoparticle Tracking Analysis |
Références
- Heslop, J. A., et al. Concise review: workshop review: understanding and assessing the risks of stem cell-based therapies. Stem Cells Translational Medicine. 4 (4), 389-400 (2015).
- Berebichez-Fridman, R., Montero-Olvera, P. R. Sources and clinical applications of mesenchymal stem cells: State-of-the-art review. Sultan Qaboos University Medical Journal. 18 (3), 264-277 (2018).
- Antebi, B., Mohammadipoor, A., Batchinsky, A. I., Cancio, L. C. The promise of mesenchymal stem cell therapy for acute respiratory distress syndrome. The Journal of Trauma and Acute Care Surgery. 84 (1), 183-191 (2018).
- Théry, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750 (2018).
- Valadi, H., et al. Exosome-mediated transfer of mRNAs and microRNAs is a novel mechanism of genetic exchange between cells. Nature Cell Biology. 9 (6), 654-659 (2007).
- Farooqi, A. A., et al. Exosome biogenesis, bioactivities and functions as new delivery systems of natural compounds. Biotechnology Advances. 36 (1), 328-334 (2018).
- Lener, T., et al. Applying extracellular vesicles based therapeutics in clinical trials - an ISEV position paper. Journal of Extracellular Vesicles. 4 (1), 30087 (2015).
- Castro-Malaspina, H., et al. Characterization of human bone marrow fibroblast colony-forming cells (CFU-F) and their progeny. Blood. 56 (2), 289-301 (1980).
- vander Spoel, T. I. G., et al. Human relevance of pre-clinical studies in stem cell therapy: systematic review and meta-analysis of large animal models of ischaemic heart disease. Cardiovascular Research. 91 (4), 649-658 (2011).
- Gupta, D., Zickler, A. M., El Andaloussi, S. Dosing extracellular vesicles. Advanced Drug Delivery Reviews. 178, 113961 (2021).
- Mareschi, K., et al. Multipotent mesenchymal stromal stem cell expansion by plating whole bone marrow at a low cellular density: a more advantageous method for clinical use. Stem Cells International. , 920581 (2012).
- Van Vlasselaer, P., Falla, N., Snoeck, H., Mathieu, E. Characterization and purification of osteogenic cells from murine bone marrow by two-color cell sorting using anti-Sca-1 monoclonal antibody and wheat germ agglutinin. Blood. 84 (3), 753-763 (1994).
- Yang, D., et al. and perspective on exosome isolation - efforts for efficient exosome-based theranostics. Theranostics. 10 (8), 3684-3707 (2020).
- Li, P., Kaslan, M., Lee, S. H., Yao, J., Gao, Z. Progress in exosome isolation techniques. Theranostics. 7 (3), 789-804 (2017).
- Willms, E., Cabañas, C., Mäger, I., Wood, M. J. A., Vader, P. Extracellular vesicle heterogeneity: subpopulations, isolation techniques, and diverse functions in cancer progression. Frontiers In Immunology. 9, 738 (2018).
- Huang, P., et al. Combinatorial treatment of acute myocardial infarction using stem cells and their derived exosomes resulted in improved heart performance. Stem Cell Research & Therapy. 10 (1), 300 (2019).
- Akbar, N., Pinnick, K. E., Paget, D., Choudhury, R. P. Isolation and characterization of human adipocyte-derived extracellular vesicles using filtration and ultracentrifugation. Journal of Visualized Experiments: JoVE. (170), e61979 (2021).
- Jung, M. K., Mun, J. Y. Sample preparation and imaging of exosomes by transmission electron microscopy. Journal of Visualized Experiments: JoVE. (131), e56482 (2018).
- Menck, K., Bleckmann, A., Schulz, M., Ries, L., Binder, C. Isolation and characterization of microvesicles from peripheral blood. Journal of Visualized Experiments: JoVE. (119), e55057 (2017).
- Yang, Y. J., et al. Atorvastatin treatment improves survival and effects of implanted mesenchymal stem cells in post-infarct swine hearts. European Heart Journal. 29 (12), 1578-1590 (2008).
- Lee, W. -. J., et al. Isolation and cellular phenotyping of mesenchymal stem cells derived from synovial fluid and bone marrow of minipigs. Journal of Visualized Experiments: JoVE. (113), e54077 (2016).
- Lindqvist, R., Wilson, I. D. Food Microorganisms: Buoyant Density Centrifugation. Encyclopedia of Separation Science. , 2843-2849 (2000).
- Yao, Y. L., Zhang, H., Gong, D. J., Song, Z. G., Xu, Z. Y. Transfecting hyperpolarization-activated cyclic nucleotide-gated channel 2 gene into porcine bone marrow mesenchymal stem cells. Journal of Clinical Rehabilitative Tissue Engineering Research. 13 (49), 9673-9676 (2009).
- Hegde, V., et al. A prospective comparison of 3 approved systems for autologous bone marrow concentration demonstrated nonequivalency in progenitor cell number and concentration. Journal of Orthopaedic Trauma. 28 (10), 591-598 (2014).
- Gallet, R., et al. Exosomes secreted by cardiosphere-derived cells reduce scarring, attenuate adverse remodelling, and improve function in acute and chronic porcine myocardial infarction. European Heart Journal. 38 (3), 201-211 (2017).
- Dominici, M., et al. Minimal criteria for defining multipotent mesenchymal stromal cells. The International Society for Cellular Therapy position statement. Cytotherapy. 8 (4), 315-317 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon