Method Article
Plazmid DNA Transfeksiyonu ve mRNA Nükleofeksiyon Dağıtımı Kullanılarak İnsan Hücrelerinde CRISPR Epigenom Düzenleme
Bu Makalede
Özet
Protokol, plazmid DNA transfeksiyonu ve mRNA nükleofeksiyonu kullanılarak insan hücre hatlarında Kümelenmiş Düzenli Aralıklı Kısa Palindromik Tekrarlar (CRISPR) tabanlı epigenom düzenleme yöntemlerini açıklar.
Özet
Epigenetik, genlerin ekspresyonunu düzenleyebilen histon proteinlerinin ve DNA'nın kimyasal modifikasyonlarını ifade eder. İnsan epigenomu, hücre farklılaşması ve yaşlanması sırasında dinamik olarak değişir ve birçok hastalık anormal epigenom modellemesi ile ilişkilidir. CRISPR'deki son gelişmeler, hedeflenen genomik lokuslarda epigenetik modifikasyonları düzenlemek için programlanabilir araçların geliştirilmesine yol açmış ve insan hücrelerindeki epigenetik modifikasyonların hassas bir şekilde yeniden yazılmasını sağlamıştır. CRISPR tabanlı epigenom editörleri, sonuçta memeli genomlarında hedeflenen genlerin programlanmış baskılanması veya aktivasyonu ile sonuçlanan epigenetik değiştiricilerle birlikte katalitik olarak ölü Cas9'a güvenir. Geleneksel genom düzenleme yöntemlerinden farklı olarak, epigenom düzenleme, DNA kırılmaları veya insan genom dizisinde değişiklikler gerektirmez ve bu nedenle gen ekspresyonunu kontrol etmek için daha güvenli bir alternatif olarak hizmet eder. Bu protokolde, plazmit DNA transfeksiyonlarını ve CRISPR epigenom editörlerini kodlayan mRNA'ların nükleofeksiyonunu kullanarak insan hücre hatlarında dCas9 aracılı epigenom düzenlemesi gerçekleştirmek için iki farklı yöntemi vurguluyoruz. CRISPR interferansı (CRISPRi) kullanarak genleri geçici olarak baskılamak ve dCas9'un KRAB alanına ve de novo DNA metiltransferaz kompleksine bir füzyonu olan CRISPRoff kullanarak genleri haftalarca dayanıklı bir şekilde susturmak için programlanabilir epigenom düzenlemesini gösteriyoruz. Ayrıca, hedef genlerin başarılı epigenom düzenlemesini ölçmek için kantitatif yöntemler ve deneysel kriterlere bağlı olarak hangi epigenom düzenleme aracının kullanılacağına dair temel hususlar hakkında rehberlik sağlıyoruz.
Giriş
Vücudumuzdaki her hücrenin genomik içeriği neredeyse aynı olsa da, her hücre tipinin transkripsiyonel profili büyük ölçüde farklılık gösterir. DNA ve histon proteinleri üzerindeki epigenetik modifikasyonlar, transkripsiyonel ekspresyonun anahtar düzenleyicileridir. Transkripsiyonel olarak aktif ökromatin, kompakt, transkripsiyonel olarak aktif olmayan heterokromatine kıyasla farklı epigenetik işaretlerle ayırt edilir. Örneğin, heterokromatik bölgeler, histon 3'ün (H3K9me3) lizin 9'u üzerinde trimetilasyon, histon H3'ün (H3K27me3) lizin 27'si üzerinde trimetilasyon ve gen promotörleri1'de guaninlerin (CpG) yanındaki sitozinler üzerinde DNA metilasyonu dahil olmak üzere baskılayıcı histon modifikasyonları ile tanımlanır. Aktif gen ekspresyonunun genomik bölgeleri, tipik olarak histon 3'ün (H3K4me3)1 lizin 4'ü üzerinde histon asetilasyonu ve trimetilasyonu ile tanımlanır.
Kümelenmiş Düzenli Aralıklı Kısa Palindromik Tekrarlar (CRISPR) devrimi, genomik dizilerin programlanmış olarak değiştirilmesini sağlayan çok sayıda araç üretmiştir. CRISPR teknolojisi, programlanabilir hedef dizilerde nükleik asitleri parçalayabilen prokaryotik bir savunma mekanizmasına dayanmaktadır. CRISPR nükleazları 2,3,4, baz düzenleyiciler 5,6 ve asal düzenleyiciler 7,8, DNA kesimi ve bu kırılmaların onarımı yoluyla memeli genomlarının DNA dizisini değiştirebilir. Etkili olmasına rağmen, bu stratejiler hedef dışı bölgelerdeDNA kırılmalarına 9,10 ve büyük ölçekli genomik yapısal mutasyonlara 11,12,13,14,15 neden olabilir. Alternatif CRISPR tabanlı araçlar, altta yatan DNA dizisini değiştirmeden gen aktivasyonunun ve baskılanmasının izlenebilir modülasyonunu sağlar. Bu araçlar, kromatin manzarasını 16,17,18 değiştiren efektör proteinlerle kombinasyon halinde, sgRNA dizisi tarafından dikte edilen hedef bölgelerde DNA bağlanmasına izin veren bir nükleaz eksikliği olan Cas9'u (dCas9) kullanır. Epigenetik yazarlar, okuyucular ve silgiler gibi efektör proteinler doğrudan dCas9'a kaynaştırılabilir veya SunTag gibi dCas9'a kaynaşmış bir peptit iskelesi veya MS2-MCP sistemi 16,17,18 gibi sgRNA üzerindeki bir RNA iskelesi tarafından işe alınabilir. Programlanabilir transkripsiyon kontrol araçlarına örnek olarak CRISPR aktivasyonu (CRISPRa)19,20,21 ve CRISPR girişimi (CRISPRi)22,23 verilebilir. CRISPRa, transkripsiyon mekanizmasını doğrudan işe alarak, hedef transkripsiyonel gen ekspresyonunu artırarakçalışır 19. Buna karşılık, CRISPRi, baskılayıcı bir epigenetik işaret22 olan H3K9me3'ü kurarak transkripsiyonu baskılar.
Epigenom düzenlemedeki ilerlemeler, bu araçların bilimsel alanlarda geniş çapta kullanılmasını sağlamıştır. Farklı efektör alanların ve proteinlerin füzyonları, mevcut epigenom düzenleyicilerinin araç setini genişletti 18,24,25,26,27,28,29,30. Ek olarak, epigenom editörleri, epigenetik modifikasyonların(31,32,33,34,35,36), efektörlerin 37,38,39 ve efektör mutasyonların 28,40,41 rollerini deşifre etmek için kullanılır gen regülasyonunda. Spesifik olarak, CRISPRi ve CRISPRa, hücre sağkalımı42 ve hücre kaderi 43,44,45,46,47 dahil olmak üzere çeşitli biyolojik süreçler için fonksiyonel genomik ekranlarda kullanılır. Ayrıca, epigenom düzenleme, ex vivo hücre mühendisliği ve in vivo tedaviler için terapötik potansiyele sahiptir18.
Burada, insan hücre hatlarında programlanabilir transkripsiyonel baskılama için iki dCas9 tabanlı epigenom düzenleyicisinin uygulanma yöntemlerini açıklıyoruz: CRISPRi22 ve CRISPRoff48. CRISPRi, ZNF10 (KOX1) ve ZIM322,23 gibi bir çinko parmak proteininden baskıcı KRAB alanına dCas9'un bir füzyonudur. CRISPRi belirli bir gen promotörüne hedeflendiğinde, KRAB alanı, hedef geni baskılamak için bir H3K9me3 yazarı olan SETDB1'i işe alır (Şekil 1A). CRISPRi geçici olarak eksprese edildiğinde, hedef lokusta kurulan H3K9me3 korunmaz ve gen ekspresyonu zamanla geri yüklenir32,48. Fonksiyonel genomik uygulamalarda olduğu gibi CRISPRi kullanarak kararlı knockdown'lar yapmak için, sgRNA ile birlikte hücrelerde CRISPRi'nin yapısal ekspresyonu esastır. Son zamanlarda, CRISPRoff kalıtsal epigenom düzenleme48'i programlamak için tasarlandı. CRISPRoff, dCas9'un KRAB alanına ve de novo DNA metiltransferaz kompleksine, DNMT3A ve DNMT3L'ye tek bir protein füzyonudur. İnsan hücrelerinde geçici bir CRISPRoff darbesi, hedeflenen genlerde H3K9me3 ve DNA metilasyonunun birikmesini programlar, bu da DNA metilasyonu ve H3K9me3'ün sürdürülmesiyle hedef genlerin uzun süreli baskılanmasına yol açar (Şekil 1B)48. Ayrıca, epigenom düzenlemeleri tersine çevrilebilir. Örneğin, CRISPRoff tarafından kararlı bir şekilde susturulan bir gen, hedef lokus49'daki DNA metilasyon işaretlerini enzimatik olarak çıkarabilen TET1-dCas9 tarafından yeniden aktive edilebilir.
Bu protokol, epigenom editörlerinin geçici ekspresyonu için iki dağıtım yöntemini detaylandıracaktır: plazmid DNA transfeksiyonu ve mRNA nükleofeksiyonu. Ek olarak, CLTA ve CD55 olmak üzere iki endojen gende epigenom düzenleme etkinliğini değerlendirmek için akış sitometrisinin nasıl kullanılacağını ana hatlarıyla açıklıyoruz. Bu yöntemler, ek düzenleyiciler kullanılarak diğer epigenom düzenleme deneylerine uyarlanabilir ve uygulanabilir veya farklı genleri hedeflemek için kullanılabilir.

Şekil 1: CRISPRi ve CRISPRoff epigenom düzenleme mekanizması ve iş akışı şeması. (A) sgRNA transgeni ve CRISPRi epigenom düzenleyicisinin doğrusal şemaları. CRISPRi ve sgRNA'nın eklenmesi, hedef lokusu susturmak için baskılayıcı H3K9me3 histon işaretinin eklenmesine izin verir. En yüksek susturma seviyesi, CRISPRi ilavesinden sonra erken elde edilir ve gen hedefi genellikle birkaç geçişten sonra yeniden aktive edilir. (B) sgRNA transgeninin ve CRISPRoff epigenom editörünün doğrusal şemaları. İLGILENILEN bir geni hedef alan hücrelere CRISPRoff eklenmesi, hedef geni susturmak için CpG bölgelerinde DNA metilasyonu ile birlikte baskılayıcı H3K9me3'ün eklenmesine yol açar. CRISPRoff ile susturma kalıtsaldır - yüksek düzeyde susturma, transfeksiyon sırasında erken elde edilir ve çoklu hücre bölünmeleri boyunca devam eder. (C) Transfeksiyon yöntemiyle epigenom düzenleme için zaman çizelgesine genel bakış. 0. günde, hücreler transfeksiyon için kaplanır. 1. günde, düzenleyici ve kılavuz plazmit, transfeksiyon yoluyla hücrelere verilebilir. 3. günde, hücreler akış sitometrisi yoluyla BFP ekspresyonu açısından değerlendirilecektir. BFP'nin yüzdesi, her koşul için deneyin nihai susturma etkinliğini belirlemek için normalleştirici bir faktör olarak kullanılır. 6. günden itibaren, hücreler ilgili muhabiri susturmak için analiz edilir, çünkü bu gün en yüksek susturma seviyesine ulaşılacaktır. (D) Epigenom editörü ve sgRNA plazmitlerinin hücrelere damla damla eklendiği transfeksiyon yöntemine genel bakış. (E) Nükleofeksiyon yöntemleri aracılığıyla epigenom düzenleme zaman çizelgesine genel bakış. Bu protokolde mRNA, 0. günde hücrelere nükleofeksiyondur. Hücreler, akış sitometrisi analizi kullanılarak nükleofeksiyondan sonraki 3. günde susturma açısından değerlendirilir. (F) Nükleofeksiyon protokolüne genel bakış. Uygun miktarlarda hücre ve mRNA karıştırılır ve nükleofektör küvetlerine eklenir. sgRNA nükleofeksiyon ile eklenirse, bu karışıma da eklenebilir. Küvetler nükleofektöre konur ve mRNA'yı hücrelere sokmak için uygun darbe kodları kullanılır. Nükleofeksiyon sonrası, hücreler kaplanır ve daha sonraki günlerde analiz için geçirilir. (G) Epigenom düzenleme için plazmit transfeksiyonu ve mRNA nükleofeksiyon stratejileri arasında karşılaştırma. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protokol
NOT: Ek Dosya 1 , temsili verilerimizin sgRNA tasarımı, klonlanması ve hücre hattı oluşturulması hakkında ayrıntılar içerir. Temsili sonuçlar bölümünde ayrıca kontroller için öneriler de ayrıntılı olarak açıklanır.
1. Epigenom düzenleyici eksprese eden plazmitlerin HEK293T hücrelere transfeksiyonu
NOT: Bu protokol, CRISPR kodlayan plazmitlerin HEK293T hücrelere verilmesini açıklar. Hücreleri, endojen olarak GFP ile etiketlenmiş, esansiyel olmayan bir gen olan CLTA'nın promotör bölgesini hedefleyen bir sgRNA'yı (Addgene 217306) eksprese edecek şekilde tasarladık. CLTA-GFP HEK 293T hücreleri, önceki bir çalışmadan50 kökenlidir. Bu örnekte, epigenom editörleri doğrudan bir mavi floresan proteine (BFP) kaynaştırılmıştır, bu da transfeksiyon verimliliğini ölçmemize ve deneysel koşulların doğru bir şekilde değerlendirilmesini sağlamamıza olanak tanır. Yaklaşımın etkinliği, akış sitometrisi kullanılarak tek hücrelerde protein seviyesinde kantitatif olarak ölçülebilen CLTA-GFP'nin susturulması ile gösterilmiştir.
- Hücre kültürü
- %10 FBS ve 1x Penisilin-Streptomisin-Glutamin ile HEK293T hücreleri yüksek glikozlu DMEM'de tutun. Hücreleri her 2-3 günde bir geçirin vekaleten,% 60 -% 70 arasında bir birleşim noktasında kaldıklarından emin olun.
NOT: Optimum transfeksiyon verimliliğini sağlamak için, mikroskop altında günlük olarak izlenebilen hücre birleşmesini %70'in altında tutmak çok önemlidir.
- %10 FBS ve 1x Penisilin-Streptomisin-Glutamin ile HEK293T hücreleri yüksek glikozlu DMEM'de tutun. Hücreleri her 2-3 günde bir geçirin vekaleten,% 60 -% 70 arasında bir birleşim noktasında kaldıklarından emin olun.
- 0. Gün: Transfeksiyon için HEK293T hücrelerinin tohumlanması
- Transfeksiyondan bir gün önce, tripan mavisi canlı / ölü boyama ve 96 oyuklu bir plakada oyuk başına ~ 1.5 x 104 canlı hücre tohumu kullanarak otomatik bir hücre sayacı ile hücreleri sayın. Her kuyucuğun son hacmini 200 μL'de tutun. Hücreler ertesi gün% 60 -% 70 birleşmeye ulaşmalıdır.
NOT: Bu, 24 oyuklu veya 6 oyuklu plakalarda da verimli bir şekilde gerçekleştirilebilir. Transfeksiyon için hücre sayıları ve DNA miktarı için Tablo 1'e bakınız.
- Transfeksiyondan bir gün önce, tripan mavisi canlı / ölü boyama ve 96 oyuklu bir plakada oyuk başına ~ 1.5 x 104 canlı hücre tohumu kullanarak otomatik bir hücre sayacı ile hücreleri sayın. Her kuyucuğun son hacmini 200 μL'de tutun. Hücreler ertesi gün% 60 -% 70 birleşmeye ulaşmalıdır.
- 1. Gün: CRISPRoff ve CRISPRi plazmitlerinin HEK29T hücrelerine transfeksiyonu
- Toplam 3 μL hacimdeki her kuyucuk için 150 ng epigenom düzenleyici plazmid (Addgene 167981) alikot. Bu plazmit seyreltmeleri PCR şerit tüplerinde hazırlanabilir.
NOT: Epigenom editörü ve sgRNA plazmidi birlikte transfekte ediliyorsa, editör:sgRNA plazmitlerinin 3:4 molar oranını denemenizi öneririz. Epigenom düzenlemenin en yüksek verimliliğini sağlamak için oranın optimize edilmesi gerekebilir. - Transfeksiyon reaktifini ve minimum esansiyel ortamı, transfeksiyondan yaklaşık 30-60 dakika önce oda sıcaklığına önceden ısıtın. Oyuk başına 0.6 μL transfeksiyon reaktifi ve 20 μL minimal esansiyel ortamı karıştırın ve oda sıcaklığında 15 dakika inkübe edin.
NOT: Birden fazla transfeksiyon gerçekleştiriliyorsa, transfeksiyon reaktifi ve minimal esansiyel ortamdan oluşan bir ana karışım yapılabilir. - 3 μL plazmit DNA içeren her bir PCR şerit tüpüne 20.6 μL transfeksiyon reaktifi ve minimal esansiyel ortam karışımı ekleyin. Oda sıcaklığında 15 dakika bekletin.
- Karışımı tohumlanmış hücrelerin plakasına yavaşça ekleyin ve karışımı dağıtmak için plakaya hafifçe vurun. Aşağı akış analizlerinde karşılaştırma için uygun bir temel sağlamak için transfekte edilmemiş bir kontrol ekleyin.
NOT: Transfeksiyon karışımını tohumlanmış hücrelere eklerken, transfeksiyon verimliliğini olumsuz yönde etkileyebileceğinden, rahatsızlığı en aza indirmek için bunu damla damla ve nazikçe yapmak önemlidir.
- Toplam 3 μL hacimdeki her kuyucuk için 150 ng epigenom düzenleyici plazmid (Addgene 167981) alikot. Bu plazmit seyreltmeleri PCR şerit tüplerinde hazırlanabilir.
- 2. Gün: Transfeksiyon başarısını kontrol edin
- BFP pozitif olup olmadıklarını belirlemek için hücreleri 10X büyütmede mikroskop altında inceleyin, çünkü bu başarılı transfeksiyonu gösterir. Gözlem işlemi sırasında hücrelerin rahatsız edilmediğinden emin olun.
- 3. Gün: 96 oyuklu plakayı ayırın ve BFP işaretleyicisini kullanarak transfeksiyon verimliliğini değerlendirin
NOT: Başarılı bir transfeksiyon, hücrelerin en az% 30'unu BFP pozitif olarak vermelidir. Bununla birlikte, dCas9, CRISPRi ve CRISPRoff ile, transfeksiyondan iki gün sonra tipik olarak %60'tan fazla BFP pozitif hücre tespit ederiz. Transfeksiyon etkinliği plazmit boyutu ile negatif korelasyon gösterir, bu nedenle farklı epigenom editörleri farklı transfeksiyon verimlilikleri sağlayabilir.- Kullanmadan önce tripsin ve DMEM'i %10 FBS ve 1x Penisilin-Streptomisin-Glutamin ile yaklaşık 30-60 dakika oda sıcaklığına ısıtın.
- Kuyulardan ortam aspire edin; Tabana bağlı hücreleri çıkarmamaya dikkat edin. Kuyucuk başına 50 μL tripsin ekleyin. 37 °C'de 2-3 dakika inkübe edin.
NOT: Özellikle CRISPRoff ile transfeksiyonu takiben bazı yüzen hücreleri gözlemlemek alışılmadık bir durum değildir. Bu hücre ölümünün DNMT3A-3L aşırı ekspresyonunun toksisitesinden kaynaklandığını varsayıyoruz. - Kuyucuk başına 150 μL tam DMEM ekleyerek tripsini inhibe edin. Hücreleri yeniden süspanse etmek ve 5. günde birleşme için hücreleri ayırmak için yukarı ve aşağı pipetleyin (96 oyuklu bir plakanın 200 μL ortamında ~ 20 μL hücre karışımı).
- Transfeksiyon verimliliğini değerlendirmek için akış sitometrisi için 50 μL yeniden süspanse hücre kullanın.
2. Epigenom düzenleyici mRNA'nın K-562 hücrelerine nükleofeksiyonu
NOT: Bu bölüm, CRISPRoff mRNA'yı K-562 hücrelerine nükleofecting işlemini detaylandırır. Basitlik için, K-562 hücrelerini, CD55 endojen geninin promotörünü hedefleyen bir sgRNA'yı yapısal olarak eksprese etmek için önceden tasarladık (Addgene 217306). CRISPRoff mRNA'nın doğrudan hücrelere verilmesi, benzer gen susturma etkinliği elde ederken, plazmid DNA bazlı yaklaşımlara eşlik eden hücresel toksisiteyi azaltma potansiyeline sahiptir. Ek olarak, nükleofeksiyon, K-562'ler gibi verimli bir şekilde transfekte edilmesi zor olan hücre dizilerinde epigenom düzenleyici yapılarını tanıtmak için kullanılabilir.
- Hücre kültürü
- Nükleofeksiyondan önce,% 10 FBS ve 1x Penisilin-Streptomisin-Glutamin içeren RPMI ile bir şişede K-562 hücrelerini koruyun. Hücreleri her gün geçirin ve %60-70 arasında bir birleşim noktasında kalmalarını sağlayın.
- Nükleofeksiyon gününde, CRISPRoff mRNA'yı steril bir RNaz içermeyen mikrosantrifüj tüpünde çözün ve nazikçe girdaplayın ve mRNA'yı buz üzerinde saklayın. Bir şerit nükleocuvetin her bir oyuğunda 2.0 x 10 5 hücre başına2 μg mRNA kullanın. mRNA hacminin toplam nükleofeksiyon hacminin (22 μL) %10'unu geçmediğinden emin olun.
NOT: Nükleofeksiyon, daha fazla hücre ve mRNA'ya sahip daha büyük küvetlerde de gerçekleştirilebilir. mRNA miktarını, ekleyeceğiniz hücre sayısıyla orantılı olarak ölçeklendirmenizi öneririz. Örneğin, 1 x 106 hücre üzerinde büyük bir küvette bir nükleofeksiyon gerçekleştirirken, toplam nükleofeksiyon hacmi 100 μL olan 10 μg mRNA kullanmanızı öneririz. Ek olarak, mRNA'nın dozu, yüksek düzenleme etkinliği sağlamak için optimize edilebilir. Verimli sonuçlar için genellikle her 2.0 x 105 hücre için 2-5 μg düzenleyici mRNA kullanırız. mRNA sentezi sırasında dahil edilmeyen serbest nükleotidler, nicelemeden önce çıkarılmalıdır. - Nükleofeksiyon solüsyonunu üreticinin talimatlarına göre hazırlayın ve nükleofeksiyondan önce 15 dakika oda sıcaklığına ısıtın.
NOT: Nükleofektör çözeltisi farklı olabilir veya farklı hücre tipleri için optimize edilmesi gerekebilir. - Tripan mavisi canlı/ölü boyama ile otomatik bir hücre sayacı kullanarak K-562 hücrelerini hasat edin ve sayın. Bir şerit küvette nükleofeksiyon için, numune başına ~ 2.0 x10 5 hücreyi steril bir mikrosantrifüj tüpüne alikot edin.
- Hücreleri oda sıcaklığında 5 dakika boyunca 500 x g'da santrifüjleyin, ardından süpernatanı atın. Hücreleri 1x oda sıcaklığında PBS ile 500 x g'da 5 dakika yıkayın, ardından süpernatanı atın.
- mRNA hacmini 22 μL'den çıkararak hücreleri yeniden süspanse etmek için nükleofektör çözeltisinin miktarını hesaplayın. Hücreleri uygun miktarda nükleofektör çözeltisinde yeniden süspanse edin.
- 2 μg CRISPRoff mRNA'ya (adım 2.1.2'de belirlenen hacim) hücre çözeltisi ekleyin ve çözeltiyi bir küvete aktarın, kabarcık oluşturmamaya dikkat edin, çünkü bu nükleofeksiyonun verimliliğini tehlikeye atabilir. Hücrelerin altta olduğundan emin olmak için nükleobüte hafifçe vurun.
- Uygun nabız kodu ile 4D-Nükleofektör sistemini kullanarak hücreleri nükleofect edin. FF-120 kodu, K-562 hücreleri için önerilir.
NOT: Darbe kodunun farklı hücre tipleri için optimize edilmesi gerekebilir. Ek optimizasyon gerekiyorsa üreticiye danışın. - Nükleofeksiyondan sonra, her küçük nükleokübte 80 μL RPMI ortamı ekleyin ve hücrelerin 15 dakika boyunca 37 ° C'de oturmasına izin verin.
- Hücre süspansiyonunu, 400 μL önceden ısıtılmış RPMI ortamı (küçük nükleobüvet) içeren 24 oyuklu bir plakanın kuyusuna aktarın.
NOT: Nükleofesyondan sonra, hücreler özellikle kırılgandır ve nükleokümetten kültür kuyucuklarına nazikçe aktarılmalıdır. - Nükleofesyondan sonraki 2. günden sonra: bölüm 3'te tarif edildiği gibi CD55 antikoru ile yüzey işaretleyici boyama protokolünü izleyin ve epigenom düzenleme etkinliğini test etmek için bir akış sitometresinde CD55 APC'nin floresansını ölçün.
NOT: Nükleofeksiyon başarısını test etmek için, GFP veya mCherry mRNA ile bir nükleofeksiyon gerçekleştirmenizi öneririz. Nükleofeksiyondan iki gün sonra, başarılı nükleofeksiyonu doğrulamak için bu hücrelerin akış sitometrisini gerçekleştirin. Genellikle GFP veya mCherry eksprese eden bu hücrelerin %90 ila %100'ünü tespit ederiz. Bir alternatif, birçok hedef geni sağlam bir şekilde susturduğu bilinen CRISPRi mRNA ile bir nükleofeksiyon yapmaktır. 2. günde akış sitometrisi yapılırken, CRISPRi ile muamele edilen numuneler, hücrelerin %90'ından fazlasında hedef gen susturulmasını göstermelidir.
3. Yüzey işaretleyici boyama
NOT: Bu bölüm, K-562 hücrelerinde epigenom düzenlemesinden sonra CD55 protein seviyelerinin ölçülmesini detaylandırır. CRISPRoff aracılı knockdown verimliliğini değerlendirmek için antikor boyama ve akış sitometrisi (aşağıdaki bölüm 4'e bakınız) kullanarak tek hücrelerde CD55 ekspresyonunun azalmasını ölçüyoruz. Ters transkripsiyon kantitatif PCR veya western blotlama dahil olmak üzere ek teknikler, hem transkript hem de protein seviyelerinde knockdown seviyesini doğrulamak için de kullanılabilir.
- Otomatik bir hücre sayacı kullanarak hücreleri sayın ve bir mikrosantrifüj tüpüne yaklaşık 5.0 x 105 ila 1.0 x 106 hücre ekleyin ve hücreleri 5 dakika boyunca 500 x g'da santrifüjleyin.
NOT: Boyama için daha fazla veya daha az hücre kullanılabilir, ancak antikor konsantrasyonunun buna göre ölçeklendirilmesi gerekebilir. - Peleti rahatsız etmemeye dikkat ederek süpernatanı atın ve 500 μL PBS ekleyin ve peleti nazikçe yeniden süspanse edin.
- Hücreleri 5 dakika boyunca 500 x g'da tekrar santrifüjleyin. Süpernatanı atın ve antikoru PBS'de seyreltilmiş istenen konsantrasyonda ekleyin. CD55 boyama için, 49μL PBS'de 1 μL Saflaştırılmış anti-insan CD55 (0.5 mg / mL stok) kullanın.
- Hücreleri nazikçe pipetleyin ve oda sıcaklığında karanlık bir yerde 25 dakika inkübe etmelerine izin verin. İnkübasyondan sonra, hücreleri 5 dakika boyunca 500 x g'da tekrar döndürün ve süpernatanı atın.
- Fazla antikoru yıkamak için peleti 500 μL PBS ile yeniden süspanse edin. Hücreleri 5 dakika boyunca 500 x g'da santrifüjleyin ve PBS süpernatantını atın. 100 μL ila 200 μL taze PBS'de yeniden süspanse edin ve akış sitometrisi analizi için 96 oyuklu bir plakaya ekleyin.
4. Akış sitometrisi
NOT: Bu protokol bir BD FACSymphony A1 Hücre Analizörü kullanmak için yazılmıştır. Ayrıntılar, kullandığınız akış sitometresine bağlı olarak değişebilir. Ayrıntılar için kullandığınız makinenin kullanım kılavuzuna başvurmanızı öneririz.
- Sitometriyi açmadan önce, atık kabının dolu olmadığını ve bol miktarda kılıf sıvısı olduğunu kontrol edin. Kılıf sıvısının yanındaki ana güç anahtarını (yeşil) açın.
- Geri düğmesini kullanarak plaka okuyucuyu açın. Ardından, sağ taraftaki yeşil düğmeyi kullanarak makineyi açın ve makinenin 96 kuyulu plaka okuyucu işlevine ayarlandığından emin olun.
- Bilgisayarı açın ve oturum açın. Yazılımı açın ve kullanıcı adınızı ve şifrenizi kullanarak giriş yapın.
- Denemeyi ayarlayın. Deneme > Yeni Deneme'yi seçerek yeni bir deneme oluşturun. Geçerli adı çift tıklayarak denemeyi yeniden adlandırın.
- Yeni Plaka Deneyi'> tıklayarak bir plaka ekleyin. Kullanılacak kuyu formatını seçin. Çalıştırılacak belirli kuyuları eklemek için, kuyunun plakadaki konumuna tıklayın ve ardından Mavi Şırıngaya tıklayın (kuyu şimdi mavi renkle vurgulanmalıdır). Gerekirse eklemek için aynı anda birden fazla kuyu seçin.
- Plaka ekranının sağındaki Numune'ye sağ tıklayarak her bir kuyucuğa belirli isimler ekleyin. Ardından Deneysel düzen'i seçin. Bu, seçtiğiniz tüm kuyuları listeleyen bir sayfa açacaktır. Kuyuları yeniden adlandırmak için bir kez tıklayın ve istediğiniz adı yazın. İşiniz bittiğinde Tamam'ı tıklayın.
- Genel Çalışma Sayfası'ndaki deneye çizimler ve uygun geçitler ekleyin. Öncelikle ileri saçılma alanı (FSC-A) ve yan saçılma alanı (SSC-A) çizimini yapmanızı öneririz. Ardından, Polygon Tool'a tıklayarak canlı hücrelerin daha sonra iyileştirilmesi için rastgele bir kapı oluşturun.
- Bu kapıya sağ tıklayın ve Detaya Git'e tıklayın. Bu, yalnızca o canlı hücre geçidinden hücreleri görüntüleyen yeni bir çizim oluşturacaktır.
- Bu yeni grafiğin eksenini FSC-A ve ileri saçılma yüksekliği (FSC-H) olarak değiştirin ve tek hücreler için bir geçit oluşturun.
- Hücrelerde sgRNA'yı yapısal olarak eksprese ediyorsa ve lentivirüs ayrıca bir floresan proteini kodluyorsa, bu tek hücre kapısını inceleyin ve sgRNA ekspresyonu için bir vekil olarak bu proteinin ekspresyonu için daha fazla geçit işlemi gerçekleştirin (örnek: PE-CF594-A'ya karşı FSC-A).
- Son kurulum kapısında (tek hücreler veya sgRNA ifade eden hücreler için), sağ tıklayın ve Detaya Git'i seçin. Raportör genin ekspresyonunu test etmek için bir kapı oluşturun (örnek: BB515-A ve FSC-A).
- Transfekte edilmiş hücreler kullanıyorsanız ve BFP ifadesi gibi transfeksiyon verimliliğini test etmek için bir yönteminiz varsa, Raportör İfade Grafiği'ne sağ tıklayın ve Çoğalt'a tıklayın. Eksenleri FSC-A yerine BV421-A olarak değiştirin. BFP pozitif hücreler için rastgele bir kapı oluşturun.
- Sitometre ekranının Parametreler sekmesinde kullanılmayan tüm parametreleri kaldırın.
- Makineyi kurmak ve çalışmaya hazırlamak için, akış sitometresi kontrol panelinde Çalıştır'a tıklayın. Düğme yeşil görünmelidir. Yazılımda, üst paneldeki HTS'ye ve ardından Prime'a gidin. Bitirdikten sonra, makineye iyi bir şekilde yerleştirildiğinden ve kapağın KAPALI olduğundan emin olarak plakayı ekleyin.
- Çalıştırmak için, çalıştırılacak tüm kuyuları seçin ve kontrollerin uygun şekilde ayarlandığından emin olun. Çift tıklayarak iyi bir seçim yapın, numune hacmini 10 μL olarak değiştirin ve Edinme Panosunda Veri Al'a tıklayın. Bu, örneği çalıştırır ancak verileri kaydetmez. Bu örnek çalışırken, lazer voltajlarını gerektiği gibi ayarlayın. Ek olarak, kapıları değiştirin.
NOT: Lazer parametrelerinin ve kapılamanın iyi olduğundan emin olmak için ölçülen tüm renkler için negatif olan ve tüm renkler için pozitif olan bir numune çalıştırmanızı öneririz. - Lazer ayarlarını kontrol ettikten sonra, Plaka Panelindeki Numunelere tıklayın ve çalışma değerlerinin istendiği gibi ayarlandığını doğrulayın.
- Alım Panosunda çalıştırmak ve tüm kuyulardan gelen verileri kaydetmek için Plakayı Çalıştır'a tıklayın. Bitirdikten sonra, plakayı çıkararak makineyi temizleyin.
- 96 oyuklu yeni bir plakaya, üst dört sıraya% 10 çamaşır suyu ve aşağıdaki dört oyuğa H2O ekleyin. HTS > Clean'e gidin. Bitirdikten sonra, programı kapatın ve düğmeleri, yeşil yan düğme, plaka okuyucu > > atık nedeniyle yeşil anahtar açıldıkları sıranın tersine kapatın.
- Hücreli 96 oyuklu plakaya% 20 çamaşır suyu ekleyin ve atın.
5. Veri analizi
NOT: Bu yöntem, akış sitometrisi ile epigenom düzenlemesini ölçmek için geçit stratejilerini ve veri işlemeyi ana hatlarıyla belirtir. Geçit stratejisi, Şekil 2 ve Şekil 3'teki veri analizinden oluşturulan örnek grafiklerle birlikte görsel olarak temsil edilmektedir.
- FlowJo geçit kurulumu
- Tüm FSC dosyalarını yeni bir çalışma sayfasına sürükleyip bırakarak FlowJo'ya yükleyin.
- Kapıları yapmaya başlamak için Tüm Örnekler'e tıklayın. Bu, kapıların tüm numunelere kolayca uygulanabilmesini sağlayacaktır.
- Bu dosyaya çift tıklayarak bir kontrol örneğini (transfekte edilmemiş veya nükleofected gibi) açın.
- Çokgen aracını kullanarak FSC-A ve SSC-A grafiğinde Canlı Hücreler için bir kapı oluşturun. Tüm örneklerin listesinde, artık örnek adının altında bir Canlı Hücreler sekmesi görünmelidir. Bu geçide sağ tıklayın ve bu geçidi tüm örneklere uygulamak için Analizi Gruba Kopyala'yı seçin (Şekil 2A, Şekil 3A).
- Canlı hücrelerin detayına gitmek için Canlı hücreler kapısına çift tıklayın. Eksenleri FSC-A ve FSC-H olarak değiştirin (Şekil 2B, Şekil 3B).
- Yalnızca tek hücreler için geçit vermek üzere bir çokgen çizin. Bu kapıyı bölüm 4'te yapıldığı gibi tüm numunelere uygulayın (Şekil 2B, Şekil 3B).
NOT: Bir floresan raportör ile bir sgRNA yapısını yapısal olarak ifade ediyorsanız, analizin yalnızca sgRNA'yı ifade eden hücrelere odaklanması için bu renk üzerinde daha fazla geçit işlemi gerçekleştirin. Bunun bir örneği, hücreler pLG1 omurgasından bir sgRNA içeriyorsa, mCherry pozitif hücreler (PE-CF594-A'ya karşı FSC-A) için bir kapı oluşturmak olabilir (Şekil 2C, Şekil 3C). - Son Kurulum Kapısına çift tıklayın (tek hücreler veya kılavuz ifade eden hücreler).
- Bir plazmit transfeksiyonu gerçekleştiriyorsa ve epigenom editörü bir BFP füzyonu için kodluyorsa, Gün 2 örnekleri için BFP pozitif hücreler (örnek: BV421-A'ya karşı FSC-A) için bir kapı oluşturun (Şekil 2D).
- Raportör ifadesi için bir geçit oluşturun (örnek: GFP negatif hücreler için geçit için BB515-A ve FSC-A) ve bu geçidi tüm örneklere uygulayın (Şekil 2E, Şekil 3D).
- Transfeksiyon analizi
- Yolluk kurulumunu gerçekleştirdikten sonra üst panelde bulunan Table Editor (Tablo Düzenleyici ) seçeneğine tıklayınız. Analiz için popülasyonları sürükleyin (örnek: BFP pozitif, GFP negatif).
- Tablo düzenleyici panelinde, Tablo Oluştur'u tıklatın. Normalleştirme hesaplamaları yapmak için bu verileri bir elektronik tabloya kopyalayın.
- Bilgi işlem
- Kontrol örneğindeki raportör negatif hücrelerin sayısını (örnek: GFP negatif) diğer tüm örneklerden çıkarın. İhbar eden kişinin arka planda bir miktar susturulması olabilir, bu nedenle bu, verilerden bu arka plan gürültüsünü ortadan kaldırır.
- Tüm değerler, 2. günde ölçülen BFP pozitif hücrelerin (başarıyla transfekte edilmiş hücreler) yüzdesine normalleştirilecektir. Bu normalleştirmeyi yapmak için değeri alın, 2. gündeki BFP pozitif hücrelerin yüzdesine bölün ve 100 ile çarpın. Bu hesaplama, düzenlenen hücrelerin transfeksiyon verimliliği normalleştirilmiş değerini verir. Veriler artık çizilmeye hazırdır. Şekil 2F'de gösterildiği gibi bir epigenom düzenleme zaman seyri boyunca çizgi grafikleri öneriyoruz.
- Nükleofeksiyon analizi
- Yolluk kurulumunu gerçekleştirdikten sonra üst panelde bulunan Table Editor (Tablo Düzenleyici ) seçeneğine tıklayınız. Analiz için popülasyonları sürükleyin (örnekler: APC negatif).
- Tablo düzenleyici panelinde, Tablo Oluştur'u tıklatın. Normalleştirme hesaplamaları yapmak için bu verileri bir elektronik tablo düzenleyicisine kopyalayın.
- Bilgi işlem
- Kontrol örneğindeki raportör negatif hücrelerin sayısını (örnek: APC negatif) diğer tüm örneklerden çıkarın. İhbar eden kişinin arka planda bir miktar susturulması olabilir, bu nedenle bu, verilerden bu arka plan gürültüsünü ortadan kaldırır. Veriler artık çizilmeye hazırdır. Bir epigenom düzenleme zaman kursu boyunca çizgi grafikleri öneririz.
NOT: İstatistikler, iki farklı epigenom editörü arasındaki susturma farklılıklarını ölçmek için kullanılabilir. İstatistiksel analizler yapmak için tüm transfeksiyon ve nükleofeksiyon deneylerini teknik üçlü kopyalar halinde gerçekleştirmenizi öneririz. Zaman içinde susturmanın çizgi grafiklerini gösterirken, teknik kopyaların standart sapmasını çizin. İki epigenom editörü arasındaki susturmayı karşılaştırmak için belirli zaman noktalarında öğrenci t-testleri yapılabilir. Numuneler, eşit olmayan varyansla eşleşmemiş olarak ele alınmalıdır.
- Kontrol örneğindeki raportör negatif hücrelerin sayısını (örnek: APC negatif) diğer tüm örneklerden çıkarın. İhbar eden kişinin arka planda bir miktar susturulması olabilir, bu nedenle bu, verilerden bu arka plan gürültüsünü ortadan kaldırır. Veriler artık çizilmeye hazırdır. Bir epigenom düzenleme zaman kursu boyunca çizgi grafikleri öneririz.
Sonuçlar
Tüm epigenom düzenleme deneyleri için, epigenom düzenleme verimliliğini değerlendirmek için uygun kontroller kritik öneme sahiptir. İnsan genomundaki herhangi bir diziyi hedeflemeyen bir kontrol sgRNA'sı kullanmanızı öneririz. Hedeflemeyen bir kılavuz kontrolünün kullanılması, hedef lokuslardaki değişikliklerin, yalnızca epigenom düzenleyicisinin aşırı ekspresyonu veya spesifik olmayan bağlanmasından ziyade, epigenom düzenleyicisinin o bölgeye yönlendirilmesi tarafından yönlendirildiğine dair güven verecektir. Ek olarak, raportör gen tabanlı deneyler için, raportör ifadesindeki değişikliklerin, dCas9'un hedef lokusa bağlanmasının sterik engellenmesi ve transkripsiyonu geçici olarak engellemesi yerine epigenom düzenleyici füzyonlarından kaynaklandığından emin olmak için yalnızca dCas9 kontrolünün kullanılmasını öneririz (Şekil 2F).
Transfeksiyon deneyleri için, BFP gibi ek bir floresan protein füzyonuna sahip bir epigenom editörü kullanmanızı öneririz. Bu füzyon, mikroskopi ve akış sitometrisi yoluyla epigenom editörü ile başarılı bir şekilde transfekte edilen hücrelerin görselleştirilmesine izin verir. Başarılı bir şekilde transfekte edilmiş hücreler, transfeksiyondan iki gün sonra yüksek BFP seviyelerini eksprese edecektir (Şekil 2D). Başarılı bir şekilde transdüksiyona tabi tutulan hücrelerin miktarı, daha sonraki günlerde epigenom düzenleme etkinliğini normalleştirmek için kullanılır (Şekil 2F).
Hem CRISPRoff hem de CRISPRi, transfeksiyondan sonraki 5. günde pik susturma gösterir (Şekil 2E-F). Farklı epigenom editörleri, CRISPRoff ile kalıtsal susturma ve CRISPRi ile geçici susturma gibi farklı epigenom düzenleme zaman çizelgelerine sahiptir (Şekil 2F). Şekil 2F ayrıca dCas9'un yalnızca epigenom düzenleme deneyleri için önemli bir kontrol olarak kullanımını da göstermektedir. mRNA nükleofeksiyon deneylerinde, CRISPRoff ile başarılı bir şekilde düzenlenen hücreler, nükleofeksiyondan sonraki 3. günde hedef genin güçlü bir şekilde susturulduğunu gösterecektir (Şekil 3E-F).
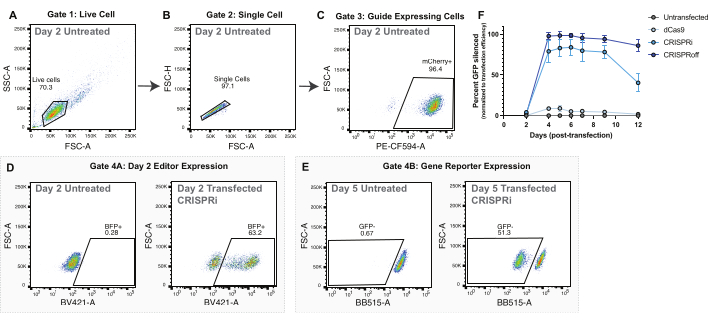
Şekil 2: Plazmit transfeksiyonu ile epigenom düzenleyici iletimi için geçit stratejisi ve temsili veriler. (A-C) Plazmit transfeksiyon deneyleri için geçit stratejisini göstermek için temsili akış grafikleri. Görüntülenen akış grafikleri, transfeksiyondan 2 gün sonra transfekte edilmemiş hücrelere aittir. Grafiklerdeki her nokta bir hücreyi temsil eder. (A) Canlı hücreler için bir kapı ile ileri saçılma alanının (FSC-A) ve yan saçılma alanının (SSC-A) akış grafiği. (B) Tek hücreler için bir geçit ile FSC-A ve ileri saçılma yüksekliğini (FSC-H) gösteren Canlı Hücrelerin akış grafiği. (C) PE-CF594-A (mCherry ifadesi) ve FSC-A'nın grafiğini çizen Tek Hücrelerin akış grafiği. sgRNA ekspresyonu için bir vekil olarak mCherry pozitif hücreler için geçitleme. (D) Transfeksiyondan sonraki 2. günde epigenom düzenleyici ekspresyonu (BFP +) için temsili geçit stratejisi. Ana popülasyon, (C) içinde kapılı kılavuz eksprese eden hücrelerdir. (E) CLA-GFP raportör geninin susturulmasının tahlil edilmesi için temsili geçit stratejisi. Ana popülasyon, panel C'de kapılı kılavuz eksprese eden hücrelerdir. (F) dCas9, CRISPRi ve CRISPRoff'un plazmit iletimini takiben transfeksiyondan sonraki günlerde CLTA-GFP'nin susturulması. Susturulmuş CLTA-GFP yüzdesi, transfeksiyondan 2 gün sonra BFP pozitif hücreler olarak ölçülen transfeksiyon verimliliğine normalize edilir. Puanlar, dört transfeksiyon kopyasının ortalamasıdır. Hata çubukları standart sapmayı temsil eder. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
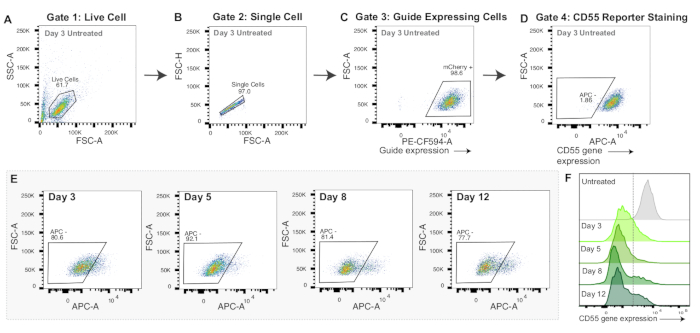
Şekil 3: CRISPRoff mRNA nükleofeksiyonu için geçit stratejisi ve temsili veriler. (M.S.) mRNA nükleofeksiyon deneyleri için geçit stratejisini göstermek için temsili akış grafikleri. Görüntülenen akış grafikleri, nükleofeksiyondan 3 gün sonra APC anti-insan CD55 antikoru ile boyanmış kontrol hücrelerine aittir. Grafiklerdeki her nokta bir hücreyi temsil eder. (A) Canlı hücreler için bir kapı ile ileri saçılma alanı yoğunluğunun (FSC-A) ve yan saçılma alanı yoğunluğunun (SSC-A) akış grafiği. (B) Tek hücreler için bir geçit ile FSC-A ve ileri saçılma yüksekliği yoğunluğunu (FSC-H) gösteren Canlı Hücrelerin akış grafiği. (C) PE-CF594-A (mCherry ifadesi) ve FSC-A'nın grafiğini çizen Tek Hücrelerin akış grafiği. sgRNA ekspresyonu için bir vekil olarak mCherry pozitif hücreler için geçitleme. (D) APC-A'nın FSC-A'ya karşı akış grafiği. CD55 raportör geninin susturulmasını gösterecek olan APC negatif hücreler için çizilen kapı. (E) CRISPRoff mRNA ile nükleofeksiyondan 3, 5, 8 ve 12 gün sonra CD55 susturma (APC - kapısı) akış grafikleri. (F) CRISPRoff mRNA ile nükleofeksiyondan 3, 5, 8 ve 12 gün sonra boyanmış ancak nükleofekte edilmemiş bir kontrole kıyasla CD55 protein ekspresyonunun (APC-A) üst üste bindirilmiş histogramları. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Tabak | Tohumlama Yoğunluğu (kuyu başına hücre) | Plazmid Miktarı |
| 96 kuyulu | 15,000 | 150 ng |
| 24 kuyulu | 90,000 | 500 ng |
| 6 kuyulu | 400,000 | 2 μg |
Tablo 1: Transfeksiyon ölçekleme miktarları. Farklı ölçeklerde HEK293T hücrelere epigenom düzenleyici transfeksiyonu için tohumlama yoğunlukları ve plazmit DNA miktarları.
Ek Tablo 1: epigenom düzenleme deneyleri için sgRNA aralayıcı dizileri. Epigenom editörlerini CLTA ve CD55'e hedeflemek için sgRNA dizileri ve hedeflemeyen bir kontrol kılavuzu için bir dizi. Ek olarak, pLG1 omurgasına klonlama için oligolar listelenmiştir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 1. Bu dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Bu protokol, CRISPR epigenom editörleri için iki farklı geçici dağıtım yöntemini detaylandırır: plazmid DNA transfeksiyonu ve mRNA nükleofeksiyonu. Her iki tekniğin de benzersiz avantajları, dezavantajları ve genel düşünceleri vardır (Şekil 1F).
Plazmid DNA transfeksiyonu, sağlam epigenom düzenleyici ekspresyonuna yol açar ve epigenom düzenleyici yapılarına, akış sitometrisi kullanılarak transfeksiyon etkinliğinin saptanmasına ve nicelleştirilmesine izin veren bir BFP füzyonu ekleriz. Ek olarak, BFP ifadesi, epigenom düzenleyicisini ifade eden hücreleri sıralamak için kullanılabilir veya bu protokolde ayrıntılı olarak açıklandığı gibi, daha sonraki zaman noktalarında susturma verilerini normalleştirmek için nicelleştirilebilir. Bununla birlikte, transfeksiyon verimliliğinin genellikle %100 olmadığını ve bu nedenle, hücreler sıralanmadıkça, editörü alanlar ve almayanlar için popülasyonun heterojen olacağını belirtmek önemlidir. Ayrıca, plazmit DNA iletimi, sitoplazmik çift sarmallı DNA'dan bir bağışıklık tepkisini tetikleyerek bağışıklık yolu aktivasyonuna ve sitotoksisiteye yol açabilir. Son olarak, plazmit DNA transfeksiyonu tüm hücre tiplerine uygun değildir ve plazmitin nükleofektyonu gerekli olabilir.
Plazmid DNA transfeksiyonundan farklı olarak, mRNA nükleofeksiyonu birçok hücre tipi için uygulanabilir ve genellikle yüksek iletim etkinliği ile sonuçlanır. Düzenleyici mRNA'lar, ticari olarak mevcut olan in vitro mRNA sentez kitleri kullanılarak sentezlenebilir ve bunlardan biri daha önce detaylandırılmıştır51. Alternatif olarak, mRNA'lar Aldevron veya Trilink'ten sentezlenebilir. Bununla birlikte, mRNA nükleofeksiyonunun bir uyarısı, hangi hücrelerin epigenom editörlerini aldığını test etmek için yeterli düzenleyici protein üretmemesidir. Yukarıdaki deneyler, 5. günde hedef gen susturulmasının ~% 90'ını tespit ettiğimiz için hemen hemen her hücrenin mRNA'yı aldığını göstermektedir (Şekil 3F). Bununla birlikte, her hücre tipi için nabız kodlarını ve mRNA-hücre oranını optimize etmenizi öneririz.
Diğer bir husus, plazmid DNA transfeksiyonu veya mRNA nükleofeksiyonu ile başlatılan epigenom düzenlemesinin farklı susturma zaman çizelgelerine sahip olmasıdır. Plazmid DNA transfeksiyonu ile, transfeksiyondan sonraki 5. günde CRISPRoff ve CRISPRi ile pik susturma gözlenir (Şekil 2F). Karşılaştırıldığında, mRNA nükleofeksiyonu tarafından sağlanan CRISPRoff ve CRISPRi ile maksimum susturma, nükleofeksiyondan sonraki 3. gün kadar erken bir tarihte görülür (Şekil 3F). Ek olarak, plazmit DNA, mRNA'ya kıyasla hücrelerde kalır ve bu da daha uzun bir editör ekspresyonu süresi ile sonuçlanır. Deneysel hedeflere bağlı olarak, daha uzun veya daha kısa bir susturma zaman çizelgesi ve ifade süresi tercih edilebilir.
Epigenom düzenlemenin evrensel olarak tüm genlere veya hücre tiplerine uygulanamayacağına dikkat etmek de önemlidir. Hedef genlerin genom dizisi veya kromatin durumlarındaki doğal farklılıklar, farklı hücre tiplerinde aynı gen için bile düzenleyici etkinliğini etkileyebilir. Örneğin, açıklamalı CpG adalarına sahip olmayan genlerin, CRISPRoff aracılı DNA metilasyonu48 ile kararlı bir şekilde susturulması zor olabilir. Bu nedenle, bu tür genleri susturmak için farklı epigenom editörlerini test etmek gerekebilir. Ek olarak, daha yüksek epigenom düzenleme verimliliği42 için en iyi kılavuzu belirlemek için en azından ilk üç CRISPRi gRNA'nın test edilmesini de öneririz. Bununla birlikte, sağlam epigenom editörlerinden oluşan sınırlı araç kutumuz göz önüne alındığında, ilgilenilen gene bağlı olarak gen nakavt etmek veya diğer nakavt stratejileri kullanmak daha etkili olabilir.
Bu protokol, mevcut birçok CRISPR tabanlı epigenom düzenleyicisinden ikisi olan CRISPRi ve CRISPRoff'a odaklanmıştır. Son zamanlarda yapılan büyük ölçekli keşif çalışmaları, insan epigenomunu yeniden yazmak için yeni araçlar geliştirmiştir 29,30,52. Epigenom editörlerinin biyomedikal araştırma ve terapötiklerde uygulamaları vardır. Örneğin, son çalışmalar, fare modellerinde ve insan olmayan primatlarda DNA metilasyonu ve H3K9me3 tabanlı epigenom düzenlemesini kullandı ve bu da hastalıkla ilişkili gen53,54,55'in kalıtsal olarak baskılanmasına neden oldu. Epigenom editörleri için gelecekteki dağıtım yöntemlerinin, epigenom düzenlemenin geniş uygulaması için yeni yollar açacağını öngörüyoruz.
Açıklamalar
J.K.N., The Regents of the University of California tarafından dosyalanan CRISPRoff/on teknolojileriyle ilgili patentlerin mucididir.
Teşekkürler
Nuñez laboratuvarı üyelerine, özellikle Rithu Pattali ve Izaiah Ornelas'a, bu el yazmasında açıklanan protokolleri geliştirdikleri ve optimize ettikleri için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
Referanslar
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır