Method Article
Редактирование эпигенома CRISPR в клетках человека с использованием трансфекции плазмидной ДНК и доставки мРНК нуклеофекции
В этой статье
Резюме
Протокол описывает методы редактирования эпигенома на основе кластеризованных регулярно чередующихся коротких палиндромных повторов (CRISPR) в клеточных линиях человека с использованием трансфекции плазмидной ДНК и нуклеофекции мРНК.
Аннотация
Эпигенетика относится к химическим модификациям белков гистонов и ДНК, которые могут регулировать экспрессию генов. Эпигеном человека динамически изменяется во время дифференцировки и старения клеток, и многие заболевания связаны с аберрантным паттерном эпигенома. Последние достижения в области CRISPR привели к разработке программируемых инструментов для редактирования эпигенетических модификаций в целевых геномных локусах, что позволяет точно переписывать эпигенетические модификации в клетках человека. Редакторы эпигенома на основе CRISPR полагаются на каталитически мертвый Cas9 в сочетании с эпигенетическими модификаторами, которые в конечном итоге приводят к запрограммированной репрессии или активации целевых генов в геномах млекопитающих. В отличие от традиционных методов редактирования генома, редактирование эпигенома не требует разрывов ДНК или изменений в последовательности генома человека и, таким образом, служит более безопасной альтернативой контролю экспрессии генов. В этом протоколе мы выделяем два различных метода выполнения dCas9-опосредованного редактирования эпигенома в клеточных линиях человека с использованием трансфекции плазмидной ДНК и нуклеофекции мРНК, кодирующих редакторы эпигенома CRISPR. Мы демонстрируем программируемое редактирование эпигенома для временного подавления генов с помощью интерференции CRISPR (CRISPRi) и для длительного подавления генов в течение многих недель с помощью CRISPRoff, слияния dCas9 с доменом KRAB и комплексом метилтрансферазы de novo ДНК. Мы также даем рекомендации по количественным методам измерения успешного редактирования эпигенома целевых генов и ключевые соображения о том, какой инструмент редактирования эпигенома следует использовать, в зависимости от экспериментальных критериев.
Введение
Хотя геномное содержимое каждой клетки в нашем организме почти идентично, транскрипционный профиль каждого типа клеток сильно отличается. Эпигенетические модификации ДНК и белков гистонов являются ключевыми регуляторами экспрессии транскрипции. Транскрипционно активный эухроматин характеризуется отчетливыми эпигенетическими признаками по сравнению с компактным, транскрипционно неактивным гетерохроматином. Например, гетерохроматические области определяются репрессивными модификациями гистонов, включая триметилирование на лизине 9 гистона 3 (H3K9me3), триметилирование на лизине 27 гистона H3 (H3K27me3) и метилирование ДНК на цитозинах рядом с гуанинами (CpG) в промоторах гена1. Геномные области активной экспрессии генов обычно определяются ацетилированием гистонов и триметилированием лизина 4 гистона 3 (H3K4me3)1.
Революция в области кластеризованных регулярно чередующихся коротких палиндромных повторов (CRISPR) породила множество инструментов, которые позволяют программировать изменение геномных последовательностей. Технология CRISPR основана на прокариотическом защитном механизме, способном расщеплять нуклеиновые кислоты на программируемых целевых последовательностях. Нуклеазы CRISPR 2,3,4, базовые редакторы 5,6 и прайм-редакторы 7,8 могут изменять последовательность ДНК геномов млекопитающих путем разрезания ДНК и восстановления этих разрывов. Несмотря на свою эффективность, эти стратегии могут вызывать разрывы ДНК в нецелевых сайтах 9,10 и крупномасштабные геномные структурные мутации 11,12,13,14,15. Альтернативные инструменты на основе CRISPR обеспечивают управляемую модуляцию активации и репрессии генов без изменения основной последовательности ДНК. Эти инструменты используют дефицит нуклеазы Cas9 (dCas9), обеспечивая связывание ДНК в целевых участках, продиктованных последовательностью sgRNA, в сочетании с эффекторными белками, которые изменяют ландшафт хроматина 16,17,18. Эффекторные белки, такие как эпигенетические писатели, считыватели и ластики, могут быть непосредственно слиты с dCas9 или рекрутированы пептидным скаффолдом, слитым с dCas9, таким как SunTag, или РНК-каркасом на sgRNA, таким как система MS2-MCP 16,17,18. Примерами программируемых инструментов управления транскрипцией являются активация CRISPR (CRISPRa)19,20,21 и интерференция CRISPR (CRISPRi)22,23. CRISPRa функционирует, напрямую рекрутируя механизм транскрипции, увеличивая экспрессию генов-мишенейтранскрипции 19. Напротив, CRISPRi подавляет транскрипцию путем установления H3K9me3, репрессивной эпигенетической метки22.
Достижения в редактировании эпигенома позволили широко использовать эти инструменты в научных областях. Слияния различных эффекторных доменов и белков расширили инструментарий доступных редакторовэпигенома 18,24,25,26,27,28,29,30. Кроме того, редакторы эпигенома используются для расшифровки ролей эпигенетических модификаций 31,32,33,34,35,36, эффекторов 37,38,39 и эффекторных мутаций 28,40,41 в регуляции генов. В частности, CRISPRi и CRISPRa используются в функциональном геномном скрининге различных биологических процессов, включая выживание клеток42 и судьбу клеток 43,44,45,46,47. Кроме того, редактирование эпигенома обладает терапевтическим потенциалом для клеточной инженерии ex vivo и терапии in vivo 18.
Здесь мы описываем методы применения двух редакторов эпигенома на основе dCas9 для программируемой транскрипционной репрессии в клеточных линиях человека: CRISPRi22 и CRISPRoff48. CRISPRi представляет собой слияние dCas9 с репрессивным доменом KRAB из белка цинкового пальца, такого как ZNF10 (KOX1) и ZIM322,23. Когда CRISPRi нацелен на определенный промотор гена, домен KRAB рекрутирует автора H3K9me3, SETDB1, для подавления гена-мишени (рис. 1A). Когда CRISPRi экспрессируется транзиторно, установленный H3K9me3 в целевом локусе не поддерживается, и экспрессия генов восстанавливается с течением времени32,48. Для проведения стабильных нокдаунов с использованием CRISPRi, например, в приложениях функциональной геномики, необходима конститутивная экспрессия CRISPRi в клетках вместе с sgRNA. Недавно CRISPRoff был разработан для программирования редактирования наследуемого эпигенома48. CRISPRoff представляет собой однобелковое слияние dCas9 с доменом KRAB и комплексом метилтрансферазы de novo DNA, DNMT3A и DNMT3L. Переходный импульс CRISPRoff в клетках человека программирует отложение H3K9me3 и метилирование ДНК в генах-мишенях, что приводит к долговременной репрессии генов-мишеней путем поддержания метилирования ДНК и H3K9me3 (рис. 1B)48. Кроме того, редактирование эпигенома может быть отменено. Например, ген, который стабильно подавляется CRISPRoff, может быть реактивирован TET1-dCas9, который может ферментативно удалять метки метилирования ДНК в целевых локусах49.
В этом протоколе будут подробно описаны два метода доставки для транзиторной экспрессии редакторов эпигенома: трансфекция плазмидной ДНК и нуклеофекция мРНК. Кроме того, мы описываем, как использовать проточную цитометрию для оценки эффективности редактирования эпигенома в двух эндогенных генах, CLTA и CD55. Эти методы могут быть адаптированы и применены к другим экспериментам по редактированию эпигенома с использованием дополнительных редакторов или могут быть использованы для нацеливания на различные гены.

Рисунок 1: Схема механизма редактирования эпигенома CRISPRi и CRISPRoff и рабочего процесса. (A) Линейные схемы трансгена sgRNA и редактора эпигенома CRISPRi. Добавление CRISPRi и sgRNA позволяет добавить репрессивную метку гистона H3K9me3 для подавления локуса-мишени. Наивысший уровень сайленсинга достигается на ранних этапах после добавления CRISPRi, и ген-мишень обычно реактивируется после нескольких пассажей. (B) Линейные схемы трансгена sgRNA и редактора эпигенома CRISPRoff. Добавление CRISPRoff к клеткам, нацеленным на интересующий ген, приводит к добавлению репрессивного H3K9me3 вместе с метилированием ДНК в сайтах CpG для подавления гена-мишени. Сайленсинг с помощью CRISPRoff является наследственным - высокий уровень сайленсинга достигается на ранних этапах трансфекции и сохраняется при множественных клеточных делениях. (C) Обзор сроков редактирования эпигенома методом трансфекции. На 0-й день клетки покрываются пластинами для трансфекции. В 1-й день редактор и направляющая плазмида могут быть введены в клетки путем трансфекции. На 3-й день клетки будут оценены на экспрессию BFP с помощью проточной цитометрии. Процент BFP используется в качестве нормирующего фактора для определения окончательной эффективности подавления в эксперименте для каждого состояния. Начиная с 6-го дня, клетки анализируются на предмет подавления замалчивания интересующего репортера в этот день, так как в этот день будет достигнут наивысший уровень молчания. (D) Обзор метода трансфекции, при котором редактор эпигенома и плазмиды sgRNA добавляются в клетки по каплям. (E) Обзор временной шкалы редактирования эпигенома с помощью методов нуклеофекции. В этом протоколе мРНК представляет собой нуклеофекцию в клетки в день 0. Клетки оцениваются на предмет сайленсинга на 3-й день после нуклеофекции с помощью анализа проточной цитометрии. (F) Обзор протокола нуклеофекции. Соответствующее количество клеток и мРНК смешивают и добавляют в нуклеофекторные кюветы. Если sgРНК вводится путем нуклеофекции, ее также можно добавлять в эту смесь. Кюветы помещаются в нуклеофектор, и соответствующие импульсные коды используются для введения мРНК в клетки. После нуклеофекции клетки покрываются и пропускаются для анализа в более поздние дни. (G) Сравнение стратегий трансфекции плазмид и нуклеофекции мРНК для редактирования эпигенома. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
протокол
ПРИМЕЧАНИЕ: Дополнительный файл 1 содержит подробную информацию о дизайне sgРНК, клонировании и создании клеточных линий на основе наших репрезентативных данных. В разделе репрезентативных результатов также подробно описаны предложения по элементам управления.
1. Трансфекция плазмид, экспрессирующих редактор эпигенома, в клетки HEK293T
Примечание: Этот протокол описывает доставку плазмид, кодирующих CRISPR, в HEK293T клетки. Мы сконструировали клетки для экспрессии sgRNA (Addgene 217306), нацеленной на промоторную область CLTA, второстепенного гена, который эндогенно помечен GFP. Клетки CLTA-GFP HEK 293T были получены в результате предыдущего исследования50. В этом примере редакторы эпигенома сливаются непосредственно с синим флуоресцентным белком (BFP), что позволяет количественно оценить эффективность трансфекции и обеспечивает точную оценку условий эксперимента. Эффективность подхода демонстрируется подавлением CLTA-GFP, которое может быть количественно измерено на уровне белка в отдельных клетках с помощью проточной цитометрии.
- Культура клеток
- Поддерживайте HEK293T клеток в DMEM с высоким содержанием глюкозы с помощью 10% FBS и 1x пенициллин-стрептомицина-глутамина. Проходите через клетки каждые 2-3 дня, следя за тем, чтобы они оставались на уровне 60%-70%.
ПРИМЕЧАНИЕ: Для обеспечения оптимальной эффективности трансфекции крайне важно поддерживать конфлюенцию клеток ниже 70%, что можно ежедневно контролировать под микроскопом.
- Поддерживайте HEK293T клеток в DMEM с высоким содержанием глюкозы с помощью 10% FBS и 1x пенициллин-стрептомицина-глутамина. Проходите через клетки каждые 2-3 дня, следя за тем, чтобы они оставались на уровне 60%-70%.
- День 0: Посев HEK293T клеток для трансфекции
- За день до трансфекции подсчитайте клетки с помощью автоматического счетчика клеток с помощью трипанового синего окрашивания живыми/мертвыми и затравите ~1,5 x 104 живых клетки в лунку в 96-луночном планшете. Поддерживайте конечный объем каждой лунки на уровне 200 μл. На следующий день слияние клеток должно достичь 60%-70%.
ПРИМЕЧАНИЕ: Это также может быть эффективно выполнено в 24-луночных или 6-луночных планшетах. В таблице 1 приведены номера клеток и количество ДНК для трансфекции.
- За день до трансфекции подсчитайте клетки с помощью автоматического счетчика клеток с помощью трипанового синего окрашивания живыми/мертвыми и затравите ~1,5 x 104 живых клетки в лунку в 96-луночном планшете. Поддерживайте конечный объем каждой лунки на уровне 200 μл. На следующий день слияние клеток должно достичь 60%-70%.
- День 1: Трансфекция плазмид CRISPRoff и CRISPRi в клетки HEK29T
- Аликвота 150 нг эпигенома редактора плазмиды (Addgene 167981) на каждую лунку общим объемом 3 мкл. Эти плазмидные разведения можно готовить в ПЦР-стрип-пробирках.
ПРИМЕЧАНИЕ: Если редактор эпигенома и плазмида sgRNA подвергаются совместной трансфекции, то мы рекомендуем попробовать молярное соотношение плазмид editor:sgRNA 3:4. Соотношение, возможно, потребуется оптимизировать для достижения максимальной эффективности редактирования эпигенома. - Разогрейте реагент для трансфекции и минимальную необходимую среду до комнатной температуры примерно за 30-60 минут до трансфекции. Смешать 0,6 мкл трансфекционного реагента и 20 мкл минимальной эфирной среды на лунку и инкубировать при комнатной температуре в течение 15 мин.
ПРИМЕЧАНИЕ: При выполнении нескольких трансфекций можно получить мастер-смесь реагента для трансфекции и минимального количества необходимой среды. - Добавьте 20,6 мкл трансфекционного реагента и минимальную смесь незаменимой среды в каждую полосковую пробирку для ПЦР, содержащую 3 мкл плазмидной ДНК. Дать настояться при комнатной температуре в течение 15 минут.
- Медленно добавьте смесь в пластину с засеянными клетками и осторожно постучите по пластине, чтобы диспергировать смесь. Включите нетрансфицированный контроль, чтобы обеспечить надлежащую исходную линию для сравнения в последующих анализах.
ПРИМЕЧАНИЕ: При добавлении трансфекционной смеси в засеянные клетки важно делать это по каплям и осторожно, чтобы свести к минимуму нарушения, так как это может отрицательно повлиять на эффективность трансфекции.
- Аликвота 150 нг эпигенома редактора плазмиды (Addgene 167981) на каждую лунку общим объемом 3 мкл. Эти плазмидные разведения можно готовить в ПЦР-стрип-пробирках.
- День 2: Проверка успешности трансфекции
- Исследуйте клетки под микроскопом при 10-кратном увеличении, чтобы определить, являются ли они BFP-положительными, так как это указывает на успешную трансфекцию. Убедитесь, что клетки не нарушаются во время процесса наблюдения.
- День 3: Разделите 96-луночный планшет и оцените эффективность трансфекции с помощью маркера BFP
Примечание: Успешная трансфекция должна дать не менее 30% клеток как BFP-положительных. Тем не менее, с помощью dCas9, CRISPRi и CRISPRoff мы обычно обнаруживаем более 60% BFP-положительных клеток через два дня после трансфекции. Эффективность трансфекции отрицательно коррелирует с размером плазмиды, поэтому разные редакторы эпигенома могут давать разную эффективность трансфекции.- Предварительно подогрейте трипсин и DMEM с 10% FBS и 1x Пенициллин-Стрептомицин-Глютамин до комнатной температуры в течение примерно 30-60 минут перед использованием.
- Аспирация среды из скважин; Будьте осторожны, чтобы не удалить ячейки, прикрепленные к дну. Добавьте 50 мкл трипсина на лунку. Выдерживать 2-3 минуты при температуре 37 °C.
Примечание: Нет ничего необычного в том, чтобы наблюдать некоторые плавающие клетки, особенно после трансфекции с помощью CRISPRoff. Мы предполагаем, что эта гибель клеток связана с токсичностью сверхэкспрессии DNMT3A-3L. - Ингибируйте трипсин, добавляя 150 мкл полного DMEM на лунку. Пипетку вверх и вниз для ресуспендирования клеток и расщепления клеток для слияния на 5-й день (~20 мкл клеточной смеси в 200 мкл среды 96-луночного планшета).
- Используйте 50 мкл ресуспендированных клеток для проточной цитометрии для оценки эффективности трансфекции.
2. Нуклеофекция мРНК редактора эпигенома в клетки К-562
ПРИМЕЧАНИЕ: В этом разделе подробно описан процесс нуклеофектинга мРНК CRISPRoff в клетки K-562. Для простоты мы предварительно сконструировали клетки K-562 для конститутивной экспрессии sgRNA, которая нацелена на промотор эндогенного гена CD55 (Addgene 217306). Доставка мРНК CRISPRoff непосредственно в клетки может снизить клеточную токсичность, которая сопровождает подходы на основе плазмидной ДНК, при этом сохраняя при этом аналогичную эффективность подавления экспрессии генов. Кроме того, нуклеофекция может быть использована для введения конструкций редактора эпигенома в клеточные линии, которые трудно эффективно трансфицировать, такие как K-562s.
- Культура клеток
- Перед нуклеофекцией поддерживайте клетки K-562 в колбе с RPMI с 10% FBS и 1x пенициллин-стрептомицин-глутамин. Проходите клетки каждый день, следя за тем, чтобы они оставались на уровне 60%-70%.
- В день нуклеофекции разморозьте и аккуратно занесите мРНК CRISPRoff в стерильную микроцентрифужную пробирку, не содержащую РНКазы, и храните мРНК на льду. Используйте 2 мкг мРНК на 2,0 x 105 клеток в каждой лунке полосковой нуклеокуветы. Следите за тем, чтобы объем мРНК не превышал 10% от общего объема нуклеофекции (22 μл).
ПРИМЕЧАНИЕ: Нуклеофекция также может быть выполнена в более крупных кюветах с большим количеством клеток и мРНК. Мы рекомендуем масштабировать количество мРНК пропорционально количеству клеток, которые вы будете добавлять. Например, при выполнении нуклеофекции в большой кювете на клетках размером 1 x 106 мы предлагаем использовать 10 мкг мРНК с общим объемом нуклеофекции 100 мкл. Кроме того, дозировка мРНК может быть оптимизирована для обеспечения высокой эффективности редактирования. Для получения эффективных результатов мы часто используем 2-5 мкг мРНК редактора на каждые 2,0 x 105 клеток. Свободные нуклеотиды, которые не были включены в синтез мРНК, должны быть удалены перед количественным определением. - Приготовьте раствор нуклеофекции в соответствии с инструкциями производителя и подогрейте до комнатной температуры в течение 15 минут до нуклеофекции.
ПРИМЕЧАНИЕ: Раствор нуклеофектора может отличаться или нуждаться в оптимизации для разных типов клеток. - Собирайте и подсчитывайте клетки K-562 с помощью автоматического счетчика клеток с окрашиванием в трипановый синий цвет живых/мертвых. Для нуклеофекции в стрип-кювете аликвота ~2,0 x 105 клеток на образец в стерильную микроцентрифужную пробирку.
- Центрифугируйте клетки при давлении 500 х г в течение 5 минут при комнатной температуре, затем выбросьте надосадочную жидкость. Промойте ячейки 1x с PBS комнатной температуры при 500 х г в течение 5 минут, затем выбросьте надосадочную жидкость.
- Рассчитайте количество раствора нуклеофектора для ресуспензии клеток, вычитая объем мРНК из 22 мкл. Ресуспендируйте клетки в соответствующем количестве раствора нуклеофэктора.
- Добавьте клеточный раствор к 2 мкг мРНК CRISPRoff (объем, определенный на шаге 2.1.2) и перенесите раствор в кювету, стараясь не образовывать пузырьков, так как это может поставить под угрозу эффективность нуклеофекции. Аккуратно постучите по нуклеокубете, чтобы убедиться, что клетки находятся внизу.
- Нуклеофект клеток осуществляется с помощью системы 4D-Nucleofector с соответствующим импульсным кодом. Код FF-120 рекомендуется для элементов K-562.
ПРИМЕЧАНИЕ: Код импульса может потребоваться оптимизировать для различных типов ячеек. Проконсультируйтесь с производителем, если нужна дополнительная оптимизация. - После нуклеофекции добавьте 80 мкл среды RPMI в каждую маленькую лунку для нуклеокуветы и дайте клеткам постоять при температуре 37 °C в течение 15 минут.
- Перенесите клеточную суспензию в лунку 24-луночного планшета, содержащего 400 мкл предварительно подогретой среды RPMI (малой нуклеокуветы).
ПРИМЕЧАНИЕ: После нуклеофекции клетки становятся особенно хрупкими и должны быть бережно перенесены из нуклеокуветы в культуральные лунки. - После 2-го дня после нуклеофэкции: следовать протоколу окрашивания поверхностных маркеров антителом к CD55, как описано в разделе 3, и измерить флуоресценцию CD55 APC на проточном цитометре для определения эффективности редактирования эпигенома.
ПРИМЕЧАНИЕ: Чтобы определить успешность нуклеофекции, мы рекомендуем проводить нуклеофекцию с помощью GFP или мРНК mCherry. Через два дня после нуклеофекции проведите проточную цитометрию этих клеток, чтобы подтвердить успешную нуклеофекцию. Обычно мы обнаруживаем от 90% до 100% этих клеток, экспрессирующих GFP или mCherry. В качестве альтернативы можно провести нуклеофекцию с помощью мРНК CRISPRi, которая, как известно, надежно подавляет многие гены-мишени. При выполнении проточной цитометрии на 2-й день обработанные CRISPRi образцы должны показать сайленсинг генов-мишеней в более чем 90% клеток.
3. Поверхностное окрашивание маркером
ПРИМЕЧАНИЕ: В этом разделе подробно описаны количественные уровни белка CD55 после редактирования эпигенома в клетках K-562. Мы количественно оцениваем снижение экспрессии CD55 в одиночных клетках с помощью окрашивания антителами и проточной цитометрии (см. раздел 4 ниже) для оценки эффективности нокдауна, опосредованного CRISPRoff. Дополнительные методы, включая количественную ПЦР с обратной транскрипцией или вестерн-блоттинг, также могут быть использованы для подтверждения уровня нокдауна как на уровне транскрипта, так и на уровне белка.
- Подсчитайте ячейки с помощью автоматического счетчика клеток и добавьте примерно 5,0 x 105 до 1,0 x 106 клеток в микроцентрифужную пробирку и центрифугируйте ячейки при давлении 500 x g в течение 5 минут.
Примечание: Для окрашивания можно использовать большее или меньшее количество клеток, однако может потребоваться соответствующее масштабирование концентрации антител. - Выбросьте надосадочную жидкость, стараясь не потревожить гранулу, добавьте 500 μл PBS и осторожно ресуспендируйте гранулу.
- Снова центрифугируйте клетки при 500 x g в течение 5 минут. Выбросьте надосадочную жидкость и добавьте антитело в нужной концентрации, разведенное в PBS. Для окрашивания CD55 используйте 1 мкл очищенного античеловеческого CD55 (0,5 мг/мл запаса) в 49 мкл PBS.
- Аккуратно пипетируйте клетки пипеткой и дайте им инкубироваться в течение 25 минут в темном месте при комнатной температуре. После инкубации снова вращайте клетки при давлении 500 x g в течение 5 минут и выбросьте надосадочную жидкость.
- Повторно суспендируйте гранулу 500 μл PBS, чтобы смыть избыток антител. Центрифугируйте клетки при давлении 500 x g в течение 5 минут и выбросьте надосадочную жидкость PBS. Ресуспендируйте в 100 мкл до 200 мкл свежего PBS и добавьте в 96-луночный планшет для анализа проточной цитометрии.
4. Проточная цитометрия
ПРИМЕЧАНИЕ: Этот протокол написан для использования анализатора клеток BD FACSymphony A1. Особенности могут варьироваться в зависимости от используемого вами проточного цитометра. Мы рекомендуем обратиться к руководству пользователя машины, которую вы используете, для уточнения деталей.
- Перед включением цитометрии убедитесь, что контейнер для отходов не заполнен и в нем много жидкости оболочки. Включите главный выключатель питания (зеленого цвета), который находится рядом с жидкостью оболочки.
- Включите считыватель пластин с помощью заднего переключателя. Затем включите машину с помощью зеленой кнопки с правой стороны и убедитесь, что машина настроена на функцию считывателя 96-луночных планшетов.
- Включите компьютер и авторизуйтесь. Откройте программное обеспечение и войдите в систему, используя свое имя пользователя и пароль.
- Настройте эксперимент. Создайте новый эксперимент, выбрав Эксперимент > Новый эксперимент. Переименуйте эксперимент, дважды щелкнув текущее имя.
- Добавьте тарелку, нажав кнопку Экспериментировать > Новой тарелкой. Выберите формат лунки, который будет использоваться. Чтобы добавить определенные лунки для запуска, нажмите на расположение лунки на пластине, а затем нажмите на синий шприц (теперь лунка должна быть выделена синим цветом). При необходимости выберите несколько скважин одновременно для добавления.
- Добавьте конкретные названия к каждой скважине, щелкнув правой кнопкой мыши « Образец» в правой части дисплея планшета. Затем выберите Экспериментальный макет. Откроется страница со списком всех выбранных вами скважин. Чтобы переименовать колодцы, нажмите один раз и введите нужное имя. Нажмите OK , когда закончите.
- Добавьте графики и соответствующие вентили в эксперимент на Глобальном рабочем листе. Мы рекомендуем сначала построить график прямой области рассеяния (FSC-A) в зависимости от области бокового рассеяния (SSC-A). Затем создайте произвольный вентиль для живых ячеек, чтобы позже уточнить, нажав на инструмент «Многоугольник».
- Щелкните правой кнопкой мыши на этом вороте и выберите Drill Down. Это создаст новый график, отображающий только ячейки из этого элемента активной ячейки.
- Измените ось этого нового графика на FSC-A в зависимости от высоты прямого рассеяния (FSC-H) и создайте вентиль для одиночных ячеек.
- Если sgRNA конститутивно экспрессируется в клетках, а лентивирус также кодирует флуоресцентный белок, то просверлите эти одиночные клеточные ворота и выполните дальнейшее гейтирование для экспрессии этого белка в качестве прокси для экспрессии sgRNA (пример: PE-CF594-A против FSC-A).
- На окончательном затворе настройки (для одиночных клеток или для клеток, экспрессирующих sgRNA) щелкните правой кнопкой мыши и выберите Drill Down. Создание затвора для анализа экспрессии репортерного гена (пример: BB515-A против FSC-A).
- Если вы используете трансфицированные клетки и у вас есть метод оценки эффективности трансфекции, такой как экспрессия BFP, щелкните правой кнопкой мыши на графике выражения репортера и выберите Дублировать. Измените оси на BV421-A против FSC-A. Создание произвольного вентита для BFP-положительных ячеек.
- Удалите все параметры, которые не используются, на вкладке «Параметры» на дисплее цитометра.
- Чтобы настроить машину и подготовить ее к работе, нажмите кнопку «Запустить » на панели управления проточным цитометром. Кнопка должна появиться зеленым цветом. В программном обеспечении перейдите в HTS на верхней панели, а затем заправьте. Когда закончите, добавьте тарелку, убедившись, что она правильно установлена в машине и крышка СНЯТА.
- Для запуска выберите все скважины для запуска и убедитесь, что элементы управления настроены правильно. Выберите колодец двойным щелчком мыши, измените объем образца на 10 мкл и нажмите «Получить данные » на панели сбора данных. При этом образец будет выполнен, но данные не будут записываться. Во время работы этого образца отрегулируйте напряжение лазера по мере необходимости. Кроме того, поменяйте ворота.
ПРИМЕЧАНИЕ: Мы рекомендуем использовать образец, который отрицательный для всех измеряемых цветов и положительный для всех цветов, чтобы убедиться в хороших параметрах лазера и стробировании. - После проверки настроек лазера нажмите на «Образцы» на панели пластин и убедитесь, что текущие значения установлены по желанию.
- Нажмите кнопку Run plate, чтобы запустить на панели сбора данных и записать данные со всех скважин. После этого очистите машину, сняв пластину.
- На новую 96-луночную пластину добавьте 10% отбеливателя в четыре верхних ряда и H2O в четыре лунки ниже. Перейдите на сайт HTS > Clean. Закончив, закройте программу и выключите кнопки в порядке, обратном тому, в каком они были включены на зеленой боковой кнопке > пластинчатом считывателе > зеленом переключателе по отработанному ходу.
- Добавьте 20% отбеливателя в 96-луночный планшет с ячейками и выбросьте.
5. Анализ данных
ПРИМЕЧАНИЕ: Этот метод описывает стратегии гейтирования и обработку данных для количественной оценки редактирования эпигенома с помощью проточной цитометрии. Стратегия стробирования представлена визуально вместе с примерами графиков, сгенерированных на основе анализа данных на рисунках 2 и 3.
- Настройка стробирования FlowJo
- Загрузите все файлы FSC в FlowJo, перетащив их на новый лист.
- Нажмите на «Все образцы », чтобы начать изготовление гейтов. Это гарантирует, что затворы можно будет легко применять ко всем образцам.
- Откройте контрольный образец (например, нетрансфицированный или ненуклеофектированный), дважды щелкнув по этому файлу.
- Создайте вентиль для активных ячеек на графике FSC-A против SSC-A с помощью инструмента полигон. В списке всех образцов теперь должна появиться вкладка «Живые клетки» под именем образца. Щелкните правой кнопкой мыши на этом затворе и выберите «Копировать анализ в группу », чтобы применить этот элемент ко всем образцам (Рисунок 2A, Рисунок 3A).
- Дважды щелкните по затвору живых ячеек , чтобы перейти к активным ячейкам. Измените оси на FSC-A и FSC-H (рис. 2B, рис. 3B).
- Нарисуйте многоугольник к воротам только для отдельных ячеек. Примените этот затвор ко всем образцам, как сделано в разделе 4 (рис. 2B, рис. 3B).
ПРИМЕЧАНИЕ: При конститутивной экспрессии конструкции sgRNA с помощью флуоресцентного репортера выполните дальнейшее стробирование на этом цвете, чтобы анализ был сосредоточен только на клетках, экспрессирующих sgRNA. Примером этого может быть создание ворот для mCherry положительных клеток (PE-CF594-A против FSC-A), если клетки содержат sgRNA из основного скелета pLG1 (Рисунок 2C, Рисунок 3C). - Дважды щелкните по Последнему затвору настройки (одиночные ячейки или ячейки, экспрессирующие направляющие).
- Если выполнение трансфекции плазмиды и редактор эпигенома кодирует слияние BFP, то создайте ворота для BFP-положительных клеток (пример: BV421-A против FSC-A) для образцов второго дня (рис. 2D).
- Создайте вентиль для экспрессии репортера (пример: BB515-A против FSC-A для гейта для GFP-отрицательных клеток) и примените этот вентиль ко всем образцам (Рисунок 2E, Рисунок 3D).
- Трансфекционный анализ
- После выполнения настройки стробирования нажмите на «Редактор таблиц » в верхней панели. Перетащите популяции для анализа (примеры: положительный BFP, отрицательный GFP).
- На панели редактора таблиц нажмите кнопку Создать таблицу. Скопируйте эти данные в электронную таблицу для выполнения расчетов нормализации.
- Обработка данных
- Вычтите количество репортерных отрицательных клеток (например, отрицательных GFP) в контрольной выборке из всех остальных образцов. Может произойти некоторое замалчивание работы репортера в фоновом режиме, поэтому этот фоновый шум удаляется из данных.
- Все значения будут нормализованы в соответствии с процентом BFP-положительных клеток (успешно трансфицированных клеток), измеренным на 2-й день. Чтобы сделать эту нормализацию, возьмите значение, разделите на процент положительных клеток BFP на 2-й день и умножьте на 100. Этот расчет дает нормированное значение эффективности трансфекции отредактированных клеток. Теперь данные готовы к построению на графике. Мы рекомендуем использовать линейные графики по всему временному ходу редактирования эпигенома, как показано на рисунке 2F.
- Нуклеофекционный анализ
- После выполнения настройки стробирования нажмите на «Редактор таблиц » в верхней панели. Перетащите популяции для анализа (примеры: отрицательные APC).
- На панели редактора таблиц нажмите кнопку Создать таблицу. Скопируйте эти данные в редактор электронных таблиц для выполнения вычислений нормализации.
- Обработка данных
- Вычтите количество репортерных отрицательных клеток (например, отрицательных APC) в контрольной пробе из всех остальных образцов. Может произойти некоторое замалчивание работы репортера в фоновом режиме, поэтому этот фоновый шум удаляется из данных. Теперь данные готовы к построению на графике. Мы рекомендуем использовать линейные графики по всему временному ходу редактирования эпигенома.
Примечание: Статистические данные могут быть использованы для количественной оценки различий в молчании между двумя разными редакторами эпигенома. Мы рекомендуем проводить все эксперименты по трансфекции и нуклеофекции в технических тройках для выполнения статистического анализа. При изображении линейных графиков глушения во времени стройте график стандартного отклонения технических реплик. Студенческие t-тесты в определенные моменты времени могут быть выполнены для сравнения сайленсинга между двумя редакторами эпигенома. Выборки следует рассматривать как непарные с неравной дисперсией.
- Вычтите количество репортерных отрицательных клеток (например, отрицательных APC) в контрольной пробе из всех остальных образцов. Может произойти некоторое замалчивание работы репортера в фоновом режиме, поэтому этот фоновый шум удаляется из данных. Теперь данные готовы к построению на графике. Мы рекомендуем использовать линейные графики по всему временному ходу редактирования эпигенома.
Результаты
Для всех экспериментов по редактированию эпигенома надлежащий контроль имеет решающее значение для оценки эффективности редактирования эпигенома. Мы рекомендуем использовать контрольную sgRNA, которая не нацелена на какую-либо последовательность в геноме человека. Использование нецелевого направляющего управления даст уверенность в том, что изменения в целевых локусах обусловлены направлением редактора эпигенома на этот сайт, а не только гиперэкспрессией редактора эпигенома или неспецифическим связыванием. Кроме того, для экспериментов на основе репортерных генов мы предлагаем использовать контроль, основанный только на dCas9, чтобы гарантировать, что изменения в экспрессии репортеров обусловлены слиянием редакторов эпигенома, а не стерическим препятствованием связыванию dCas9 с локусом-мишенью и временным препятствованием транскрипции (рис. 2F).
Для экспериментов по трансфекции мы рекомендуем использовать редактор эпигенома с дополнительным слиянием флуоресцентных белков, такой как BFP. Это слияние позволяет визуализировать клетки, успешно трансфицированные с помощью редактора эпигенома, с помощью микроскопии и проточной цитометрии. Успешно трансфицированные клетки будут экспрессировать высокие уровни BFP через два дня после трансфекции (Рисунок 2D). Количественная оценка успешно трансдуцированных клеток используется для нормализации эффективности редактирования эпигенома в последующие дни (рис. 2F).
Как CRISPRoff, так и CRISPRi демонстрируют пик молчания на 5-й день после трансфекции (рисунок 2E-F). Разные редакторы эпигенома имеют различные временные рамки редактирования эпигенома, такие как наследственное сайленсинг с помощью CRISPRoff и временное сайленсинг с помощью CRISPRi (рис. 2F). На рисунке 2F также показано использование dCas9 только в качестве важного элемента управления для экспериментов по редактированию эпигенома. В экспериментах с нуклеофекцией мРНК клетки, успешно отредактированные с помощью CRISPRoff, продемонстрируют сильное подавление экспрессии гена-мишени на 3-й день после нуклеофекции (рис. 3E-F).
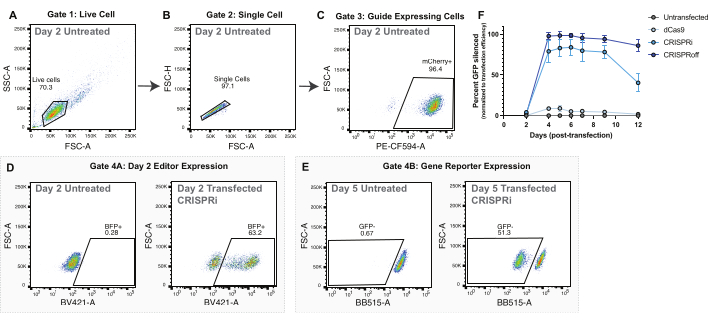
Рисунок 2: Стратегия гейтирования и репрезентативные данные для доставки редактора эпигенома путем трансфекции плазмид. (А-С) Репрезентативные графики потока для отображения стратегии стробирования для экспериментов по трансфекции плазмид. Отображаемые графики потока представляют собой нетрансфицированные клетки через 2 дня после трансфекции. Каждая точка на графиках представляет собой одну клетку. (A) График течения прямой области рассеяния (FSC-A) и боковой области рассеяния (SSC-A) с затвором для живых клеток. (B) График потока активных ячеек, отображающий FSC-A и высоту прямого рассеяния (FSC-H) с затвором для одиночных ячеек. (C) График потока одиночных ячеек с графиками PE-CF594-A (экспрессия mCherry) и FSC-A. Гейтинг для mCherry положительных клеток в качестве прокси для экспрессии sgRNA. (D) Репрезентативная стратегия гейтирования для экспрессии редактора эпигенома (BFP+) на 2-й день после трансфекции. Родительская популяция представляет собой направляющие экспрессирующие клетки, забитые в (C). (E) Репрезентативная стратегия гейтирования для анализа сайленсинга репортерного гена CLTA-GFP. Родительская популяция представляет собой направляющие экспрессирующие клетки, закрытые на панели C. (F) Подавление CLTA-GFP в течение нескольких дней после трансфекции после доставки плазмид dCas9, CRISPRi и CRISPRoff. Процент подавленного CLTA-GFP нормализуется до эффективности трансфекции, измеренной как BFP-положительные клетки через 2 дня после трансфекции. Точки — это средние значения четырех трансфекционных повторений. Полосы погрешностей представляют собой стандартное отклонение. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
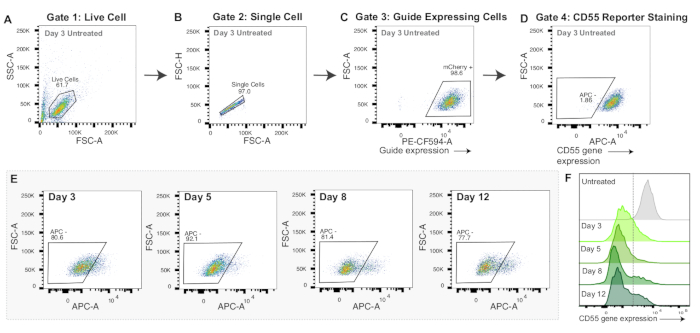
Рисунок 3: Стратегия гейтирования и репрезентативные данные для нуклеофекции мРНК CRISPRoff. (А-Г) Репрезентативные графики потоков для отображения стратегии гейтирования в экспериментах по нуклеофекции мРНК. На дисплее представлены графики потоков контрольных клеток через 3 дня после нуклеофекции, окрашенных античеловеческим антителом к CD55 APC. Каждая точка на графиках представляет собой одну клетку. (A) График течения интенсивности области прямого рассеяния (FSC-A) и интенсивности области бокового рассеяния (SSC-A) с затвором для живых клеток. (B) График потока активных ячеек, отображающий FSC-A и интенсивность высоты прямого рассеяния (FSC-H) с затвором для одиночных ячеек. (C) График потока одиночных ячеек с графиками PE-CF594-A (экспрессия mCherry) и FSC-A. Гейтинг для mCherry положительных клеток в качестве прокси для экспрессии sgRNA. (D) График потока APC-A в сравнении с FSC-A. Ворота нарисованы для APC-отрицательных клеток, что указывает на подавление репортерного гена CD55. (E) Графики потока сайленсинга CD55 (APC - gate) в течение 3, 5, 8 и 12 дней после нуклеофекции мРНК CRISPRoff. (F) Наложенные гистограммы экспрессии белка CD55 (APC-A) в течение 3, 5, 8 и 12 дней после нуклеофекции с помощью мРНК CRISPRoff по сравнению с окрашенной, но ненуклеофектированной контрольной группой. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
| Тарелка | Плотность засева (ячеек на лунку) | Количество плазмид |
| 96 лунок | 15,000 | 150 нг |
| 24 лунки | 90,000 | 500 нг |
| 6-луночный | 400,000 | 2 μг |
Таблица 1: Объемы масштабирования трансфекции. Плотность посева и количество плазмидной ДНК для трансфекции редактора эпигенома в HEK293T клетки в различных масштабах.
Дополнительная таблица 1: последовательности спейсеров sgRNA для экспериментов по редактированию эпигенома. последовательности sgRNA для нацеливания редакторов эпигенома на CLTA и CD55 вместе с последовательностью для нецелевого контрольного руководства. Кроме того, перечислены олигогены для клонирования в остовик pLG1. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительное досье 1. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
В этом протоколе подробно описаны два различных метода транзиторной доставки для редакторов эпигенома CRISPR: трансфекция плазмидной ДНК и нуклеофекция мРНК. Оба метода имеют уникальные преимущества, недостатки и общие соображения (рис. 1F).
Трансфекция плазмидной ДНК приводит к надежной экспрессии редактора эпигенома, и мы включаем слияние BFP в конструкции редактора эпигенома, что позволяет обнаруживать и количественно оценивать эффективность трансфекции с помощью проточной цитометрии. Кроме того, экспрессия BFP может быть использована для сортировки клеток, экспрессирующих редактор эпигенома, или может быть количественно определена для нормализации данных сайленсинга в более поздние моменты времени, как подробно описано в этом протоколе. Однако важно отметить, что эффективность трансфекции обычно не составляет 100%, и, таким образом, популяция будет неоднородной для тех, кто получил редактор, и тех, кто его не получил, если клетки не будут отсортированы. Кроме того, доставка плазмидной ДНК может вызвать иммунный ответ со стороны цитоплазматической двухцепочечной ДНК, что приводит к активации иммунного пути и цитотоксичности. Наконец, трансфекция плазмидной ДНК подходит не для всех типов клеток, и может потребоваться нуклеофекция плазмиды.
В отличие от трансфекции плазмидной ДНК, нуклеофекция мРНК применима для многих типов клеток и часто приводит к высокой эффективности доставки. Редакторные мРНК могут быть синтезированы с использованием коммерчески доступных наборов для синтеза мРНК in vitro , один из которых был подробно описан ранее51. В качестве альтернативы мРНК могут быть синтезированы из Альдеврона или Трилинка. Тем не менее, одним из предостережений нуклеофекции мРНК является то, что она не производит достаточное количество белка-редактора, чтобы определить, какие клетки получили редакторов эпигенома. Приведенные выше эксперименты показывают, что почти каждая клетка получила мРНК, поскольку мы обнаруживаем ~90% сайленсинга гена-мишени на 5-й день (рис. 3F). Тем не менее, мы рекомендуем оптимизировать импульсные коды и соотношение мРНК к клеткам для каждого типа клеток.
Еще одно соображение заключается в том, что редактирование эпигенома, инициированное трансфекцией плазмидной ДНК или нуклеофекцией мРНК, имеет разные временные шкалы сайленсинга. При трансфекции плазмидной ДНК пиковое подавление сайленсинга с помощью CRISPRoff и CRISPRi наблюдается на 5-е сутки после трансфекции (рис. 2F). Для сравнения, максимальное подавление давления с помощью CRISPRoff и CRISPRi, доставляемых мРНК-нуклеофекцией, наблюдается уже на 3-й день после нуклеофекции (рис. 3F). Кроме того, плазмидная ДНК сохраняется в клетках по сравнению с мРНК, что приводит к более длительной экспрессии редактора. В зависимости от целей эксперимента может быть предпочтительным более длительный или более короткий временной шкала сайленсинга и продолжительность экспрессии.
Также важно отметить, что редактирование эпигенома не может быть универсально применено ко всем генам или типам клеток. Внутренние различия в последовательности генома или состояниях хроматина генов-мишеней могут влиять на эффективность редактора, даже для одного и того же гена в разных типах клеток. Например, гены, в которых отсутствуют аннотированные CpG-островки, может быть трудно стабильно заглушить с помощью CRISPRoff-опосредованного метилирования ДНК48. Поэтому, возможно, потребуется протестировать различные редакторы эпигенома, чтобы заставить такие гены замолчать. Кроме того, мы также рекомендуем протестировать по крайней мере три основные гРНК CRISPRi, чтобы определить наилучшее руководство дляповышения эффективности редактирования эпигенома. Тем не менее, учитывая наш ограниченный набор надежных редакторов эпигенома, может быть более эффективным проведение нокаута гена или использование других стратегий нокдауна в зависимости от интересующего гена.
Этот протокол ориентирован на CRISPRi и CRISPRoff, два из многих доступных редакторов эпигенома на основе CRISPR. Недавние крупномасштабные исследования позволили разработать новые инструменты для переписывания эпигенома человека 29,30,52. Редакторы эпигенома находят применение в биомедицинских исследованиях и терапии. Например, в недавних исследованиях использовалось метилирование ДНК и редактирование эпигенома на основе H3K9me3 на мышиных моделях и приматах, что привело к наследственной репрессии гена, связанного с заболеванием 53,54,55. Мы предполагаем, что будущие методы предоставления услуг для редакторов эпигенома откроют новые возможности для широкого применения редактирования эпигенома.
Раскрытие информации
J.K.N. является изобретателем патентов, связанных с технологиями CRISPRoff/on, поданных регентами Калифорнийского университета.
Благодарности
Мы благодарим сотрудников лаборатории Нуньес, особенно Риту Паттали и Изайю Орнеласа, за разработку и оптимизацию протоколов, описанных в этой рукописи.
Материалы
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
Ссылки
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены