Method Article
プラスミドDNAトランスフェクションとmRNAヌクレオフェクションデリバリーを用いたヒト細胞におけるCRISPRエピゲノム編集
要約
このプロトコールは、プラスミドDNAトランスフェクションおよびmRNAヌクレオフェクションを使用したヒト細胞株におけるClustered Regularly Interspaced Short Palindromic Repeats(CRISPR)ベースのエピゲノム編集の方法を説明しています。
要約
エピジェネティクスとは、遺伝子の発現を調節できるヒストンタンパク質とDNAの化学修飾を指します。ヒトエピゲノムは、細胞の分化や老化の際に動的に変化し、多くの疾患がエピゲノムパターンの異常化と関連しています。近年のCRISPRの進歩により、標的ゲノム遺伝子座のエピジェネティックな修飾を編集するためのプログラム可能なツールが開発され、ヒト細胞のエピジェネティックな修飾の正確な書き換えが可能になりました。CRISPRベースのエピゲノムエディターは、触媒的に死んだCas9とエピジェネティックな修飾因子の組み合わせに依存しており、最終的には哺乳類のゲノムにおける標的遺伝子のプログラムされた抑制または活性化をもたらします。従来のゲノム編集法とは異なり、エピゲノム編集はDNAの切断やヒトゲノム配列の変更を必要としないため、遺伝子発現を制御するためのより安全な代替手段として機能します。このプロトコールでは、プラスミドDNAトランスフェクションとCRISPRエピゲノムエディターをコードするmRNAのヌクレオフェクションを使用して、ヒト細胞株でdCas9を介したエピゲノム編集を行う2つの異なる方法に焦点を当てています。私たちは、CRISPR干渉(CRISPRi)を使用して遺伝子を一過性に抑制するプログラム可能なエピゲノム編集と、dCas9のKRABドメインと de novo DNAメチルトランスフェラーゼ複合体の融合であるCRISPRoffを使用して遺伝子を数週間永続的にサイレンシングするプログラム可能なエピゲノム編集を実証しています。また、標的遺伝子のエピゲノム編集の成功を測定するための定量的手法や、実験基準に応じてどのエピゲノム編集ツールを使用するかについての重要な考慮事項に関するガイダンスも提供しています。
概要
私たちの体内のすべての細胞のゲノム含有量はほぼ同じですが、各細胞タイプの転写プロファイルは大きく異なります。DNAおよびヒストンタンパク質のエピジェネティックな修飾は、転写発現の重要な調節因子です。転写活性ユークロマチンは、コンパクトで転写活性のないヘテロクロマチンと比較して、明確なエピジェネティックマークによって特徴付けられます。例えば、ヘテロクロマティック領域は、ヒストン3のリジン9(H3K9me3)上のトリメチル化、ヒストンH3のリジン27(H3K27me3)上のトリメチル化、および遺伝子プロモーター1のグアニンの隣のシトシン(CpG)上のDNAメチル化を含む、抑制的なヒストン修飾によって定義されます。活性遺伝子発現のゲノム領域は、典型的には、ヒストン3(H3K4me3)1のリジン4上のヒストンアセチル化およびトリメチル化によって定義されます。
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)革命は、ゲノム配列のプログラムによる改変を可能にする豊富なツールを生み出しました。CRISPR技術は、プログラム可能な標的配列で核酸を切断できる原核生物の防御メカニズムに基づいています。CRISPRヌクレアーゼ2,3,4、塩基エディター5,6、およびプライムエディター7,8は、DNA切断およびこれらの切断の修復を通じて、哺乳類ゲノムのDNA配列を変化させることができる。これらの戦略は効果的ではあるが、オフターゲット部位9,10および大規模なゲノム構造変異11,12,13,14,15を引き起こす可能性がある。CRISPRベースの代替ツールにより、基礎となるDNA配列を変更することなく、遺伝子の活性化と抑制の扱いやすい調節が可能になります。これらのツールは、ヌクレアーゼ欠損Cas9(dCas9)を利用し、sgRNA配列によって決定される標的部位でのDNA結合を可能にし、クロマチンランドスケープ16,17,18を変化させるエフェクタータンパク質との組み合わせを可能にします。エフェクタータンパク質、例えば、エピジェネティックなライター、リーダー、およびイレイサーは、直接dCas9に融合させるか、またはdCas9に融合したペプチドスカフォールド(SunTagなど)、またはMS2−MCPシステム16,17,18などのsgRNA上のRNAスカフォールドによってリクルートすることができる。プログラム可能な転写制御ツールの例としては、CRISPR活性化(CRISPRa)19,20,21およびCRISPR干渉(CRISPRi)22,23が含まれる。CRISPRaは、転写機構を直接動員することで機能し、標的の転写遺伝子発現を増加させます19。対照的に、CRISPRiは、抑制的なエピジェネティックなマークであるH3K9me3を確立することにより転写を抑制します22。
エピゲノム編集の進歩により、これらのツールを科学分野を超えて広く利用できるようになりました。異なるエフェクタードメインおよびタンパク質の融合により、利用可能なエピゲノムエディター18,24,25,26,27,28,29,30のツールキットが拡大した。さらに、エピゲノムエディタは、エピジェネティックな修飾31,32,33,34,35,36、エフェクター37,38,39、およびエフェクター突然変異28,40,41の役割を解読するために使用されます遺伝子調節において。具体的には、CRISPRiおよびCRISPRaは、細胞生存42および細胞運命43,44,45,46,47を含むさまざまな生物学的プロセスの機能ゲノミクススクリーニングに使用されている。さらに、エピゲノム編集は、ex vivo細胞工学およびin vivo治療の治療可能性を秘めています18。
ここでは、ヒト細胞株におけるプログラム可能な転写抑制のための2つのdCas9ベースのエピゲノムエディター、CRISPRi 22およびCRISPRoff 48を適用する方法について説明します。CRISPRiは、ZNF10(KOX1)やZIM3などのジンクフィンガータンパク質からdCas9を抑制的KRABドメインに融合させたものです22,23。CRISPRiが特定の遺伝子プロモーターを標的とすると、KRABドメインはH3K9me3ライターであるSETDB1をリクルートして標的遺伝子を抑制します(図1A)。CRISPRiが一過性に発現すると、標的遺伝子座で確立されたH3K9me3は維持されず、遺伝子発現は時間とともに回復する32,48。機能ゲノミクスへの応用など、CRISPRiを用いた安定的なノックダウンを行うためには、sgRNAとともに細胞内でCRISPRiが構成的に発現することが不可欠です。最近、CRISPRoffは遺伝性エピゲノム編集をプログラムするように操作された48。CRISPRoffは、dCas9のKRABドメインおよびde novo DNAメチルトランスフェラーゼ複合体であるDNMT3AおよびDNMT3Lへの単一タンパク質融合体です。ヒト細胞におけるCRISPRoffの一過性パルスは、標的遺伝子におけるH3K9me3の沈着とDNAメチル化をプログラムし、DNAメチル化とH3K9me3の維持による標的遺伝子の長期抑制をもたらす(図1B)48。さらに、エピゲノムの編集を元に戻すことができます。例えば、CRISPRoffによって安定にサイレンシングされた遺伝子は、TET1-dCas9によって再活性化することができ、これにより、標的遺伝子座49のDNAメチル化マークを酵素的に除去することができる。
このプロトコールでは、エピゲノムエディターの一過性発現のための2つの導入方法、プラスミドDNAトランスフェクションとmRNAヌクレオフェクションについて詳しく説明します。さらに、フローサイトメトリーを使用して、 CLTA と CD55の2つの内在性遺伝子におけるエピゲノム編集の有効性を評価する方法について概説します。これらの方法は、追加のエディターを使用して他のエピゲノム編集実験に適合させて適用することも、異なる遺伝子を標的とするために使用することもできます。

図1:CRISPRiおよびCRISPRoffエピゲノム編集メカニズムとワークフローの概略図。(A)sgRNA導入遺伝子およびCRISPRiエピゲノムエディターの線形概略図。CRISPRiとsgRNAの添加により、抑制的なH3K9me3ヒストンマークの付加が可能になり、標的遺伝子座を沈黙させることができます。最高レベルのサイレンシングは、CRISPRi添加後早期に達成され、遺伝子ターゲットは通常、数回の継代後に再活性化されます。(B)sgRNA導入遺伝子およびCRISPRoffエピゲノムエディターの線形概略図。目的の遺伝子を標的とする細胞にCRISPRoffを追加すると、CpG部位でのDNAメチル化とともに抑制的なH3K9me3が追加され、標的遺伝子がサイレンシングされます。CRISPRoffによるサイレンシングは遺伝性であり、トランスフェクションの早い段階で高レベルのサイレンシングが達成され、複数の細胞分裂にわたって持続します。(C)トランスフェクション法によるエピゲノム編集のタイムラインの概要。0日目に、細胞をトランスフェクション用にプレーティングします。1日目に、エディタープラスミドとガイドプラスミドをトランスフェクションにより細胞に導入できます。3日目に、細胞はフローサイトメトリーを介してBFP発現について評価されます。BFPの割合は、各条件に対する実験の最終的なサイレンシング効果を決定するための正規化因子として使用されます。6日目以降は、この日に最高レベルのサイレンシングが達成されるため、細胞は対象のレポーターをサイレンシングするために分析されます。(D)エピゲノムエディターとsgRNAプラスミドを細胞に滴下するトランスフェクション法の概要。(E)ヌクレオフェクション法によるエピゲノム編集のタイムラインの概要。このプロトコルでは、mRNAは0日目に細胞にヌクレオフェクションされます。細胞は、フローサイトメトリー分析を使用して、ヌクレオフェクション後3日目にサイレンシングについて評価されます。(F)ヌクレオフェクションプロトコールの概要。適量の細胞とmRNAを混合し、ヌクレオフェクターキュベットに添加します。sgRNAがヌクレオフェクションによって導入される場合、この混合物にsgRNAを添加することもできます。キュベットをヌクレオフェクターに入れ、適切なパルスコードを使用してmRNAを細胞に導入します。ヌクレオフェクション後、細胞はプレーティングされ、後日分析のために継代されます。(G)エピゲノム編集のためのプラスミドトランスフェクションとmRNAヌクレオフェクション戦略の比較。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
注: 補足ファイル1 には、代表的なデータのsgRNAデザイン、クローニング、および細胞株の生成に関する詳細が含まれています。代表的な結果セクションでは、コントロールの提案についても詳しく説明します。
1. エピゲノムエディター発現プラスミドのHEK293T細胞へのトランスフェクション
注:このプロトコルは、CRISPRをコードするプラスミドのHEK293T細胞への導入について説明しています。私たちは、GFPを内因的にタグ付けした非必須遺伝子である CLTAのプロモーター領域を標的とするsgRNA(Addgene 217306)を発現する細胞を改変しました。CLTA-GFP HEK 293T細胞は、以前の研究50に由来しています。この例では、エピゲノムエディターが青色蛍光タンパク質(BFP)に直接融合しているため、トランスフェクション効率を定量化し、実験条件を正確に評価することができます。このアプローチの有効性は、CLTA-GFPのサイレンシングによって実証されており、フローサイトメトリーを使用して単一細胞のタンパク質レベルで定量的に測定できます。
- 細胞培養
- 10% FBS と 1x ペニシリン-ストレプトマイシン-グルタミンを使用して、HEK293T 細胞を高グルコース DMEM に維持します。細胞を2〜3日ごとに継代し、細胞が60%〜70%のコンフルエントに保たれるようにします。
注:最適なトランスフェクション効率を確保するためには、細胞の密度を70%未満に維持することが重要です。これは、顕微鏡下で毎日モニタリングできます。
- 10% FBS と 1x ペニシリン-ストレプトマイシン-グルタミンを使用して、HEK293T 細胞を高グルコース DMEM に維持します。細胞を2〜3日ごとに継代し、細胞が60%〜70%のコンフルエントに保たれるようにします。
- 0日目:トランスフェクション用のHEK293T細胞の播種
- トランスフェクションの前日に、トリパンブルー生/死染色を使用して自動セルカウンターで細胞をカウントし、96ウェルプレートでウェルあたり~1.5 x 104 個の生細胞をシードします。各ウェルの最終容量を200 μLに保ち、翌日には細胞が60%-70%のコンフルエントに達するはずです。
注:これは、24ウェルまたは6ウェルプレートでも効率的に実行できます。トランスフェクションの細胞数とDNA量については 、表1 を参照してください。
- トランスフェクションの前日に、トリパンブルー生/死染色を使用して自動セルカウンターで細胞をカウントし、96ウェルプレートでウェルあたり~1.5 x 104 個の生細胞をシードします。各ウェルの最終容量を200 μLに保ち、翌日には細胞が60%-70%のコンフルエントに達するはずです。
- 1日目:CRISPRoffおよびCRISPRiプラスミドのHEK29T細胞へのトランスフェクション
- エピゲノムエディタープラスミド(Addgene 167981)を各ウェル150 ng分、総容量3 μLで分注します。これらのプラスミド希釈液は、PCRストリップチューブで調製できます。
注:エピゲノムエディターとsgRNAプラスミドを同時トランスフェクトする場合は、エディター:sgRNAプラスミドのモル比を3:4にすることをお勧めします。エピゲノム編集の効率を最大限に高めるためには、この比率を最適化する必要があるかもしれません。 - トランスフェクション試薬を温め、トランスフェクションの約30〜60分前に最小限の必須中温から室温で温めます。0.6 μLのトランスフェクション試薬と20 μLの最小限の必須培地をウェルごとに混合し、室温で15分間インキュベートします。
注:複数のトランスフェクションを行う場合は、トランスフェクション試薬と最小限の必須培地のマスターミックスを作成できます。 - 20.6 μLのトランスフェクション試薬と最小限の必須培地混合物を、3 μLのプラスミドDNAを含む各PCRストリップチューブに加えます。室温で15分間放置します。
- 播種した細胞のプレートに混合物をゆっくりと加え、プレートを軽くたたいて混合物を分散させます。トランスフェクションされていないコントロールを含めて、ダウンストリーム解析での比較のための適切なベースラインを提供します。
注:トランスフェクション混合物を播種細胞に添加する場合、トランスフェクション効率に悪影響を及ぼす可能性があるため、外乱を最小限に抑えるために滴下および穏やかに添加することが不可欠です。
- エピゲノムエディタープラスミド(Addgene 167981)を各ウェル150 ng分、総容量3 μLで分注します。これらのプラスミド希釈液は、PCRストリップチューブで調製できます。
- 2日目:トランスフェクションの成功を確認する
- 10倍の倍率で顕微鏡で細胞を検査し、トランスフェクションが成功したことを示すBFP陽性であるかどうかを判断します。観察プロセス中に細胞が乱されていないことを確認してください。
- 3日目:96ウェルプレートを分割し、BFPマーカーを使用してトランスフェクション効率を評価
注:トランスフェクションが成功すると、細胞の少なくとも30%がBFP陽性となるはずです。しかし、dCas9、CRISPRi、およびCRISPRoffでは、トランスフェクションの2日後に通常60%以上のBFP陽性細胞が検出されます。トランスフェクション効率はプラスミドサイズと負の相関があるため、エピゲノムエディターが異なればトランスフェクション効率も異なる可能性があります。- トリプシンとDMEMを10%FBSと1xペニシリン-ストレプトマイシン-グルタミンで室温で約30〜60分間温めてから使用してください。.
- 井戸から培地を吸引します。底部に付着したセルを取り外さないように注意してください。ウェルあたり50μLのトリプシンを添加します。37°Cで2〜3分間インキュベートします。
注:特にCRISPRoffによるトランスフェクション後に、一部の浮遊細胞が観察されることは珍しくありません。この細胞死は、DNMT3A-3Lの過剰発現の毒性によるものであると仮定しています。 - トリプシンを阻害するには、ウェルあたり150 μLの完全DMEMを添加します。ピペットを上下させて細胞を再懸濁し、5日目に細胞を分裂させてコンフルエンシーにします(96ウェルプレートの培地200μLに~20μLの細胞混合物を入れます)。
- 50 μLの再懸濁細胞をフローサイトメトリーに使用して、トランスフェクション効率を評価します。
2. エピゲノムエディターmRNAのK-562細胞への核吸収
注:このセクションでは、CRISPRoff mRNAをK-562細胞にヌクレオフェクトするプロセスについて詳しく説明します。簡単にするために、K-562細胞を事前に操作して、 CD55 内在性遺伝子(Addgene 217306)のプロモーターを標的とするsgRNAを構成的に発現させました。CRISPRoff mRNAを直接細胞に送達することで、プラスミドDNAベースのアプローチに伴う細胞毒性を低下させると同時に、同様の遺伝子サイレンシング効果を達成できる可能性があります。さらに、ヌクレオフェクションを使用して、K-562などの効率的なトランスフェクションが困難な細胞株にエピゲノムエディターコンストラクトを導入することができます。
- 細胞培養
- ヌクレオフェクションの前に、10% FBSおよび1xペニシリン-ストレプトマイシン-グルタミンを含むRPMIを含むフラスコでK-562細胞を維持します。細胞を毎日継代し、細胞が60%〜70%のコンフルエントに留まるようにします。
- ヌクレオフェクションの日に、無菌のRNaseフリー微量遠心チューブでCRISPRoff mRNAを解凍し、穏やかにボルテックスし、mRNAを氷上に保存します。ストリップ核の2.0 x 105 細胞あたり2 μgのmRNAをストリップ核核の各ウェルで使用します。mRNAの量が全ヌクレオフェクション容量(22 μL)の10%を超えないようにしてください。
注:ヌクレオフェクションは、より多くの細胞とmRNAを含むより大きなキュベットでも実行できます。mRNAの量は、追加する細胞の数に比例してスケーリングすることをお勧めします。例えば、大型キュベットで1 x 106 細胞のヌクレオフェクションを行う場合、10 μgのmRNAを使用し、総ヌクレオフェクション量を100 μLにすることをお勧めします。さらに、mRNAの投与量を最適化して、高い編集効率を確保することができます。効率的な結果を得るために、2.0 x 105 細胞ごとに2〜5 μgのエディターmRNAを使用することがよくあります。mRNA合成中に取り込まれなかった遊離ヌクレオチドは、定量前に除去する必要があります。 - 製造元の指示に従ってヌクレオフェクション溶液を調製し、ヌクレオフェクションの前に15分間室温まで温めます。
注:ヌクレオフェクター溶液は異なる場合や、異なる細胞タイプに対して最適化する必要がある場合があります。 - トリパンブルーの生/死染色を施した自動セルカウンターを使用して、K-562細胞を回収し、カウントします。ストリップキュベットでのヌクレオフェクションには、サンプルあたり~2.0 x 105 細胞を滅菌マイクロ遠心チューブに分注します。
- 細胞を500 x g で室温で5分間遠心分離し、上清を捨てます。細胞を室温PBSで500 x g で5分間1回洗浄し、上清を捨てます。
- 22 μLからmRNAの量を差し引いて、細胞を再懸濁するヌクレオフェクター溶液の量を計算します。適切な量のヌクレオフェクター溶液に細胞を再懸濁します。
- 2 μgのCRISPRoff mRNA(ステップ2.1.2で容量を決定)に細胞溶液を添加し、ヌクレオフェクションの効率を損なう可能性があるため、気泡を形成しないように注意しながら溶液をキュベットに移します。核酸を軽くたたいて、細胞が底にあることを確認します。
- 適切なパルスコードを持つ4D-Nucleofectorシステムを使用して、細胞を核分離します。K-562セルにはFF-120コードをお勧めします。
注:パルスコードは、細胞タイプごとに最適化する必要がある場合があります。追加の最適化が必要な場合は、製造元にご相談ください。 - ヌクレオフェクション後、80 μLのRPMI培地を各小さなヌクレオクベットウェルに加え、細胞を37°Cで15分間放置します。
- 細胞懸濁液を、400 μLの予熱済みRPMI培地(small nucleocuvette)を含む24ウェルプレートのウェルに移します。
注:ヌクレオフェクション後、細胞は特に壊れやすいため、ヌクレオクベットから培養ウェルに穏やかに移す必要があります。 - ヌクレオフェクション後2日目以降:セクション3で説明されているように、CD55抗体による表面マーカー染色プロトコルに従い、フローサイトメーターでCD55 APCの蛍光を測定して、エピゲノム編集の有効性をアッセイします。
注:ヌクレオフェクションの成功を測定するには、GFPまたはmCherry mRNAでヌクレオフェクションを行うことをお勧めします。ヌクレオフェクションの2日後に、これらの細胞のフローサイトメトリーを行い、ヌクレオフェクションが成功したことを確認します。通常、これらの細胞の90%から100%がGFPまたはmCherryを発現していることを検出します。別の方法として、多くの標的遺伝子を強力にサイレンシングすることが知られているCRISPRi mRNAを用いてヌクレオフェクションを行うことがあります。2日目にフローサイトメトリーを実施すると、CRISPRiで処理したサンプルは、細胞の90%以上で標的遺伝子のサイレンシングを示すはずです。
3. 表面マーカー染色
注:このセクションでは、K-562細胞におけるエピゲノム編集後のCD55タンパク質のレベルを定量化することについて詳しく説明します。抗体染色とフローサイトメトリー(以下のセクション4を参照)を使用して、単一細胞におけるCD55発現の減少を定量化し、CRISPRoffを介したノックダウン効率を評価します。逆転写定量PCRやウェスタンブロッティングなどの追加の技術も、転写産物レベルとタンパク質レベルの両方でノックダウンのレベルを確認するために利用できます。
- 自動セルカウンターを使用して細胞をカウントし、約5.0 x 105 〜1.0 x 106 個の細胞をマイクロ遠心チューブに加え、細胞を500 x g で5分間遠心分離します。
注:染色に使用できる細胞の数は多かれ少なかれありますが、それに応じて抗体の濃度をスケーリングする必要がある場合があります。 - ペレットを乱さないようにしながら上清を捨て、PBSを500μL加えてペレットを穏やかに再懸濁します。
- 細胞を再び500 x g で5分間遠心分離します。上清を捨て、PBSで希釈した所望の濃度で抗体を添加します。CD55の染色には、49μLのPBS中に精製抗ヒトCD55(0.5 mg/mLストック)1 μLを使用します。
- 細胞を静かにピペットで動かし、室温の暗い場所で25分間インキュベートします。インキュベーション後、細胞を再び500 x g で5分間回転させ、上清を捨てます。
- ペレットを500μLのPBSで再懸濁し、余分な抗体を洗い流します。細胞を500 x g で5分間遠心分離し、PBS上清を廃棄します。100 μLから200 μLの新鮮なPBSに再懸濁し、フローサイトメトリー分析用の96ウェルプレートに添加します。
4. フローサイトメトリー
注:このプロトコルは、BD FACSymphony A1 Cell Analyzerを使用するために書かれています。詳細は、使用しているフローサイトメーターによって異なる場合があります。詳細については、使用しているマシンのユーザーマニュアルを参照することをお勧めします。
- サイトメトリーをオンにする前に、廃液容器がいっぱいになっていないこと、およびシース液が十分にあることを確認してください。シース液の近くにある主電源スイッチ(緑)をオンにします。
- 背面スイッチを使用してプレートリーダーの電源を入れます。次に、右側の緑色のボタンを使用してマシンの電源を入れ、マシンが96ウェルプレートリーダー機能に設定されていることを確認します。
- コンピュータの電源を入れてログインします。ソフトウェアを開き、ユーザー名とパスワードを使用してログインします。
- テストを設定します。[ 実験] > [新しい実験] を選択して、新しい実験を作成します。現在の名前をダブルクリックして、実験の名前を変更します。
- プレートを追加するには、[Experiment] > [New Plate] をクリックします。使用するウェル形式を選択します。実行する特定のウェルを追加するには、プレート内のウェルの位置をクリックし、 次に青いシリンジ をクリックします(ウェルが青色で強調表示されます)。必要に応じて、複数のウェルを同時に選択して追加します。
- プレートディスプレイの右側にある Specimen を右クリックして、各ウェルに特定の名前を追加します。次に、 試験的なレイアウト を選択します。これにより、選択したすべてのウェルを一覧表示するページが開きます。ウェルの名前を変更するには、一度クリックして目的の名前を入力します。完了したら 、[OK ] をクリックします。
- プロットと適切なゲートをグローバルワークシートの実験に追加します。最初に、前方散乱領域(FSC-A)と側面散乱光領域(SSC-A)をプロットすることをお勧めします。次に、ライブセルの任意のゲートを作成し、後で ポリゴンツールをクリックして調整します。
- このゲートを右クリックし、[ ドリルダウン]をクリックします。これにより、その生細胞ゲートからの細胞のみを表示する新しいプロットが作成されます。
- この新しいプロットの軸を FSC-A vs 前方散乱高さ (FSC-H) に変更し、単一セルのゲートを作成します。
- 細胞内でsgRNAを構成的に発現し、レンチウイルスが蛍光タンパク質にもコードしている場合は、この単一細胞ゲートをドリルダウンし、sgRNA発現のプロキシとしてそのタンパク質の発現をさらにゲーティングします(例:PE-CF594-AとFSC-A)。
- 最終セットアップゲート(シングルセルまたはsgRNA発現細胞用)で、右クリックして Drill Downを選択します。レポーター遺伝子の発現をアッセイするためのゲートを作成します(例:BB515-AとFSC-A)。
- トランスフェクションした細胞を使用し、BFP発現などのトランスフェクション効率をアッセイする方法がある場合は、 Reporter Expression Plot を右クリックして Duplicateをクリックします。軸をBV421-AとFSC-Aに変更します。BFP陽性セルの任意のゲートを作成します。
- サイトメーターディスプレイの[パラメーター]タブで使用されていないパラメーターをすべて削除します。
- マシンをセットアップして実行する準備をするには、フローサイトメーターのコントロールパネルで [実行 ]をクリックします。ボタンは緑色で表示されます。ソフトウェアで、トップパネルのHTSに移動し、次にPrimeに移動します。終了したら、プレートを追加し、プレートが機械にしっかりと配置され、蓋がオフになっていることを確認します。
- 実行するには、実行するすべてのウェルを選択し、コントロールが適切に設定されていることを確認します。ダブルクリックしてウェルを選択し、サンプル量を10 μLに変更して、Acquisition Dashboardの Acquisition Data をクリックします。これにより、サンプルは実行されますが、データは記録されません。このサンプルが動作しているときは、必要に応じてレーザー電圧を調整します。さらに、ゲートを変更します。
注:レーザーパラメータとゲーティングが良好であることを確認するために、測定するすべての色に対して陰性のサンプルとすべての色に対して正のサンプルを実行することをお勧めします。 - レーザー設定を確認した後、 プレートパネルの[Specimens ]をクリックし、ランニング値が希望どおりに設定されていることを確認します。
- [Run plate] をクリックして Acquisition Dashboard で実行し、すべてのウェルからのデータを記録します。終了したら、プレートを取り外して機械を清掃します。
- 新しい96ウェルプレートに、上の4列に10%の漂白剤を加え、下の4つのウェルにH2Oを加えます。 HTS > Cleanに移動します。終了したら、プログラムを閉じ、緑色のサイドボタン>プレートリーダー>緑色のスイッチをオンにしたのと逆の順序でボタンをオフにします。
- 細胞を含む96ウェルプレートに20%漂白剤を加えて廃棄します。
5. データ分析
注:この方法では、フローサイトメトリーによるエピゲノム編集を定量化するためのゲーティング戦略とデータ処理の概要を示しています。ゲーティング戦略は、 図 2 と 図 3 のデータ分析から生成されたプロットの例とともに視覚的に表されています。
- FlowJoゲーティングセットアップ
- すべてのFSCファイルをFlowJoにドラッグアンドドロップして、新しいワークシートにロードします。
- 「All Samples」をクリックして、ゲートの作成を開始します。これにより、ゲートをすべてのサンプルに簡単に適用できるようになります。
- コントロールサンプル(トランスフェクションされていないサンプルやヌクレオフェクトされていないサンプルなど)を開くには、そのファイルをダブルクリックします。
- ポリゴンツールを使用して、FSC-A対SSC-Aプロットで生細胞のゲートを作成します。すべてのサンプルのリストで、サンプル名の下に[Live Cells]タブが表示されます。このゲートを右クリックして[ Copy Analysis to Group ]を選択し、このゲートをすべてのサンプルに適用します(図2A、 図3A)。
- [Live cells] ゲートをダブルクリックして、ライブセルをドリルダウンします。軸をFSC-AとFSC-Hに変更します(図2B、図3B)。
- 1 つのセルのみをゲートするポリゴンを描画します。セクション4(図2B、 図3B)で行ったように、このゲートをすべてのサンプルに適用します。
注:蛍光レポーターを用いてsgRNAコンストラクトを恒常的に発現させる場合は、この色でさらにゲーティングを行い、分析がsgRNAを発現している細胞のみに焦点を当てるようにします。この例として、細胞にpLG1骨格からのsgRNAが含まれている場合、mCherry陽性細胞(PE-CF594-AとFSC-A)のゲートを作成します(図2C、 図3C)。 - Last Setup Gate(単一細胞またはガイド発現細胞)をダブルクリックします。
- プラスミドトランスフェクションを実施し、エピゲノムエディターがBFP融合をコードする場合は、2日目のサンプル(図2D)に対してBFP陽性細胞(BV421-AとFSC-Aの比較)のゲートを作成します。
- レポーター発現用のゲートを作成し(例:BB515-AとFSC-A対GFPネガティブセルのゲート用)、このゲートをすべてのサンプルに適用します(図2E、 図3D)。
- トランスフェクション解析
- ゲート設定を行った後、上部パネルの 「テーブルエディター 」をクリックします。分析のために母集団をドラッグします(例:BFP陽性、GFP陰性)。
- テーブルエディタパネルで、「 Create Table」をクリックします。これらのデータをスプレッドシートにコピーして、正規化計算を実行します。
- データ処理
- コントロールサンプルのレポーターネガティブセル(例:GFP陰性)の数を他のすべてのサンプルから減算します。レポーターのバックグラウンドサイレンシングがある可能性があるため、これによりデータからこのバックグラウンドノイズが除去されます。
- すべての値は、2 日目に測定された BFP 陽性細胞 (トランスフェクションに成功した細胞) の割合に正規化されます。この正規化を行うには、値を取り、2日目のBFP陽性細胞の割合で除算し、100を掛けます。この計算により、編集された細胞のトランスフェクション効率の正規化値が得られます。これで、データをプロットする準備が整いました。 図2Fに示すように、エピゲノム編集の時間経過にわたる折れ線グラフを推奨します。
- ヌクレオフェクション解析
- ゲート設定を行った後、上部パネルの 「テーブルエディター 」をクリックします。分析のために母集団をドラッグします(例:APCネガティブ)。
- テーブルエディタパネルで、「 Create Table」をクリックします。これらのデータをスプレッドシート エディターにコピーして、正規化計算を実行します。
- データ処理
- コントロールサンプルのレポーターネガティブセル(例:APC陰性)の数を他のすべてのサンプルから減算します。レポーターのバックグラウンドサイレンシングがある可能性があるため、これによりデータからこのバックグラウンドノイズが除去されます。これで、データをプロットする準備が整いました。エピゲノム編集の時間経過にわたる折れ線グラフをお勧めします。
注:統計は、2つの異なるエピゲノムエディター間のサイレンシングの違いを定量化するために使用できます。統計解析を行うために、すべてのトランスフェクションおよびヌクレオフェクション実験をテクニカルトリプリケートで実施することをお勧めします。時間の経過に伴う無音化の折れ線グラフを描く場合は、テクニカルレプリケートの標準偏差をプロットします。特定の時点でのスチューデントt検定を実施して、2つのエピゲノムエディター間のサイレンシングを比較できます。サンプルは、不等分散で不対応として扱う必要があります。
- コントロールサンプルのレポーターネガティブセル(例:APC陰性)の数を他のすべてのサンプルから減算します。レポーターのバックグラウンドサイレンシングがある可能性があるため、これによりデータからこのバックグラウンドノイズが除去されます。これで、データをプロットする準備が整いました。エピゲノム編集の時間経過にわたる折れ線グラフをお勧めします。
結果
すべてのエピゲノム編集実験において、エピゲノム編集の効率を評価するためには、適切な制御が重要です。ヒトゲノムのどの配列も標的としないコントロールsgRNAの使用をお勧めします。ノンターゲティングガイドコントロールを使用すると、標的遺伝子座の変化が、エピゲノムエディターの過剰発現や非特異的結合だけでなく、その部位に誘導されるエピゲノムエディターによってもたらされることを確信できます。さらに、レポーター遺伝子ベースの実験では、レポーター発現の変化が、dCas9が標的遺伝子座に結合して一時的に転写を妨げる立体障害ではなく、エピゲノムエディターの融合によるものであることを確認するために、dCas9のみのコントロールを使用することをお勧めします(図2F)。
トランスフェクション実験では、エピゲノムエディターとBFPなどの蛍光タンパク質融合遺伝子を追加で使用することをお勧めします。この融合により、エピゲノムエディターでうまくトランスフェクションされた細胞を顕微鏡法とフローサイトメトリーで可視化することができます。トランスフェクションに成功した細胞は、トランスフェクションの2日後に高レベルのBFPを発現します(図2D)。形質導入に成功した細胞の定量は、後日、エピゲノム編集の有効性を正常化するために使用されます(図2F)。
CRISPRoffとCRISPRiはどちらも、トランスフェクション後5日目にピークサイレンシングを示します(図2E-F)。CRISPRoffによる遺伝性サイレンシングやCRISPRiによる一過性サイレンシングなど、エピゲノム編集のタイムラインは異なれば異なります(図2F)。図2Fは、エピゲノム編集実験の重要なコントロールとしてのみdCas9の使用を示しています。mRNAヌクレオフェクション実験では、CRISPRoffで編集に成功した細胞は、ヌクレオフェクション後3日目までに標的遺伝子の強力なサイレンシングを示します(図3E-F)。
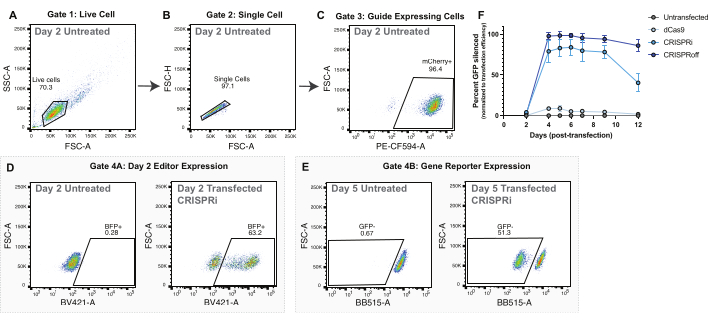
図2:プラスミドトランスフェクションによるエピゲノムエディター導入のゲーティング戦略と代表的なデータ。 (A-C)プラスミドトランスフェクション実験のゲーティング戦略を表示するための代表的なフロープロット。表示されているフロープロットは、トランスフェクション後2日目のトランスフェクションされていない細胞のものです。プロットの各ポイントは 1 つのセルを表します。(A)生細胞のゲート付き前方散乱領域(FSC-A)と側方散乱領域(SSC-A)のフロープロット。(B)FSC-Aと前方散乱高さ(FSC-H)をシングルセルのゲートで表示するライブセルのフロープロット。(C)PE-CF594-A(mCherry発現)とFSC-Aをグラフ化したシングルセルのフロープロット。sgRNA発現のプロキシとしてのmCherry陽性細胞のゲーティング。(D)トランスフェクション後2日目のエピゲノムエディター発現(BFP+)の代表的なゲーティング戦略。親集団は、(C)でゲートされた誘導発現細胞である。(E)CLTA-GFPレポーター遺伝子のサイレンシングをアッセイするための代表的なゲーティング戦略。親集団は、パネルC.(F)dCas9、CRISPRi、およびCRISPRoffのプラスミド送達後数日間にわたるCLTA-GFPのサイレンシングでゲーティングされたガイド発現細胞である。CLTA-GFPのサイレンシング率は、トランスフェクションの2日後にBFP陽性細胞として測定されたトランスフェクション効率に正規化されます。ポイントは、4回のトランスフェクションの繰り返しの平均です。エラーバーは標準偏差を表します。 この図の拡大版を表示するには、ここをクリックしてください。
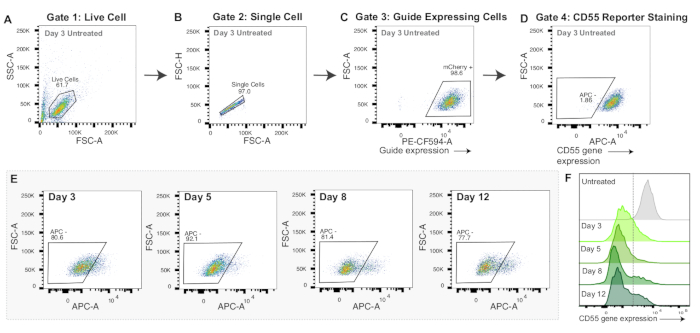
図3:CRISPRoff mRNAヌクレオフェクションのゲーティング戦略と代表的なデータ。(A-D) mRNAヌクレオフェクション実験のゲーティング戦略を表示するための代表的なフロープロット。表示されているフロープロットは、ヌクレオフェクションの3日後にAPC抗ヒトCD55抗体で染色した対照細胞のものです。プロットの各ポイントは 1 つのセルを表します。(A)生細胞のゲート付き前方散乱光強度(FSC-A)と側方散乱光強度(SSC-A)のフロープロット。(B)FSC-Aと前方散乱高さ強度(FSC-H)をシングルセルのゲートで表示する生細胞のフロープロット。(C)PE-CF594-A(mCherry発現)とFSC-Aをグラフ化したシングルセルのフロープロット。sgRNA発現のプロキシとしてのmCherry陽性細胞のゲーティング。(D)APC-AとFSC-Aのフロープロット。APC陰性細胞に対して描かれたゲートは、CD55レポーター遺伝子のサイレンシングを示します。(E)CRISPRoff mRNAによるヌクレオフェクション後3、5、8、および12日間のCD55サイレンシング(APC - ゲート)のフロープロット。(F)ヌクレオフェクション後3、5日、8日、および12日にわたるCD55タンパク質発現(APC-A)のCRISPRoff mRNAによるヒストグラムを、染色したがヌクレオフェクトしていないコントロールと比較したヒストグラムを重ね合わせたもの。 この図の拡大版を表示するには、ここをクリックしてください。
| 皿 | 播種密度(ウェルあたりの細胞数) | プラスミド量 |
| 96ウェル | 15,000 | 150 ng |
| 24ウェル | 90,000 | 500 ng |
| 6ウェル | 400,000 | 2 μg |
表1:トランスフェクションスケーリング量。 エピゲノムエディターをさまざまなスケールでHEK293T細胞にトランスフェクションするためのシード密度とプラスミドDNA量。
補足表1:エピゲノム編集実験のためのsgRNAスペーサー配列。 エピゲノムエディターをCLTAおよびCD55にターゲティングするためのsgRNA配列と、ノンターゲティングコントロールガイドの配列。さらに、pLG1バックボーンにクローニングするためのオリゴがリストされています。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル1。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
このプロトコールでは、CRISPRエピゲノムエディター向けの2つの異なるトランジェントデリバリー方法、プラスミドDNAトランスフェクションとmRNAヌクレオフェクションについて詳しく説明します。どちらの手法にも、独自の長所、短所、および一般的な考慮事項があります(図 1F)。
プラスミドDNAトランスフェクションは、強力なエピゲノムエディターの発現につながり、エピゲノムエディターコンストラクト内にBFP融合を組み込んでおり、フローサイトメトリーを用いたトランスフェクション効果の検出と定量を可能にします。さらに、BFP発現は、エピゲノムエディタを発現する細胞をソートするために使用することも、このプロトコルで詳述されているように、後の時点でサイレンシングデータを正規化するために定量化することもできます。ただし、トランスフェクション効率は通常100%ではないため、細胞を選別しない限り、エディターを受け取った人と受けなかった人で集団が不均一になることに注意することが重要です。さらに、プラスミドDNAの送達は、細胞質の二本鎖DNAからの免疫応答を引き起こし、免疫経路の活性化と細胞毒性を引き起こす可能性があります。最後に、プラスミドDNAトランスフェクションはすべての細胞タイプに適しているわけではなく、プラスミドのヌクレオフェクションが必要になる場合があります。
プラスミドDNAトランスフェクションとは異なり、mRNAヌクレオフェクションは多くの細胞タイプに適用でき、多くの場合、高い導入効果が得られます。エディターmRNAは、市販の in vitro mRNA合成キットを使用して合成することができ、そのうちの1つは以前に詳細に説明されています51。あるいは、AldevronまたはTrilinkからmRNAを合成することもできます。しかし、mRNAヌクレオフェクションの1つの注意点は、どの細胞がエピゲノムエディターを受け取ったかを分析するのに十分なエディタータンパク質を産生しないことです。上記の実験では、5日目に標的遺伝子のサイレンシングの~90%を検出したため、ほぼすべての細胞がmRNAを投与されたことが示唆されています(図3F)。ただし、パルスコードとmRNAと細胞の比率は、細胞タイプごとに最適化することをお勧めします。
また、プラスミドDNAトランスフェクションまたはmRNAヌクレオフェクションによって開始されるエピゲノム編集では、サイレンシングのタイムラインが異なることも考慮されます。プラスミドDNAトランスフェクションでは、トランスフェクション後5日目にCRISPRoffおよびCRISPRiによるピークサイレンシングが観察されます(図2F)。これに対し、mRNAヌクレオフェクションによって送達されるCRISPRoffおよびCRISPRiによる最大のサイレンシングは、ヌクレオフェクション後3日目という早い時期に見られます(図3F)。さらに、プラスミドDNAはmRNAと比較して細胞内に残留するため、エディター発現の持続時間が長くなります。実験の目標によっては、サイレンシングのタイムラインと発現期間を長くしたり短くしたりすることが望ましい場合があります。
また、エピゲノム編集は、すべての遺伝子や細胞種に普遍的に適用されるわけではないことに注意することも重要です。標的遺伝子のゲノム配列やクロマチン状態に固有の違いは、異なる細胞タイプの同じ遺伝子であっても、エディターの有効性に影響を与える可能性があります。例えば、アノテーションされたCpGアイランドを欠く遺伝子は、CRISPRoffを介したDNAメチル化48によって安定的にサイレンシングすることが困難であり得る。したがって、そのような遺伝子をサイレンシングするために、異なるエピゲノムエディターをテストする必要があるかもしれません。さらに、エピゲノム編集効率を高めるための最良のガイドを特定するために、少なくとも上位3つのCRISPRi gRNAをテストすることもお勧めします42。それにもかかわらず、堅牢なエピゲノムエディターの限られたツールボックスを考えると、目的の遺伝子に応じて遺伝子ノックアウトを行うか、他のノックダウン戦略を使用する方が効果的かもしれません。
このプロトコルは、CRISPRベースのエピゲノムエディターのうちの2つであるCRISPRiとCRISPRoffに焦点を当てています。近年の大規模な発見研究により、ヒトエピゲノム29,30,52を書き換えるための新しいツールが開発されている。エピゲノムエディターは、生物医学研究や治療学に応用できます。例えば、最近の研究では、マウスモデルや非ヒト霊長類におけるDNAメチル化とH3K9me3に基づくエピゲノム編集が用いられ、その結果、疾患関連遺伝子53,54,55の遺伝性抑制がもたらされました。私たちは、エピゲノムエディターの将来のデリバリーモダリティが、エピゲノム編集の広範な応用に新たな道を開くと想定しています。
開示事項
J.K.N.は、カリフォルニア大学のリージェンツが出願したCRISPRoff/on技術に関連する特許の発明者です。
謝辞
Nuñez研究室のメンバー、特にRithu Pattali氏とIzaiah Ornelas氏には、この原稿に記載されているプロトコルの開発と最適化について感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
参考文献
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved