Method Article
Edición del epigenoma CRISPR en células humanas mediante transfección de ADN plasmídico y administración de nucleofección de ARNm
En este artículo
Resumen
El protocolo describe los métodos para la edición del epigenoma basada en repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) en líneas celulares humanas mediante la transfección de ADN plasmídico y la nucleofección de ARNm.
Resumen
La epigenética se refiere a las modificaciones químicas de las histonas, las proteínas y el ADN que pueden regular la expresión de los genes. El epigenoma humano se altera dinámicamente durante la diferenciación celular y el envejecimiento, y muchas enfermedades se asocian con patrones epigenómicos aberrantes. Los avances recientes en CRISPR han llevado al desarrollo de herramientas programables para editar modificaciones epigenéticas en loci genómicos específicos, lo que permite una reescritura precisa de las modificaciones epigenéticas en células humanas. Los editores de epigenomas basados en CRISPR se basan en Cas9 catalíticamente muerto junto con modificadores epigenéticos que, en última instancia, dan lugar a la represión programada o la activación de genes específicos en los genomas de los mamíferos. A diferencia de los métodos tradicionales de edición del genoma, la edición del epigenoma no requiere rupturas de ADN ni cambios en la secuencia del genoma humano y, por lo tanto, sirve como una alternativa más segura para controlar la expresión génica. En este protocolo, destacamos dos métodos diferentes para realizar la edición del epigenoma mediado por dCas9 en líneas celulares humanas utilizando transfecciones de ADN plasmídico y nucleofección de ARNm que codifican editores de epigenoma CRISPR. Demostramos la edición programable del epigenoma para reprimir genes de forma transitoria utilizando la interferencia de CRISPR (CRISPRi) y para silenciar genes de forma duradera durante muchas semanas utilizando CRISPRoff, una fusión de dCas9 con el dominio KRAB y el complejo de ADN metiltransferasa de novo . También proporcionamos orientación sobre métodos cuantitativos para medir la edición exitosa del epigenoma de los genes objetivo y consideraciones clave sobre qué herramienta de edición del epigenoma utilizar, según los criterios experimentales.
Introducción
Aunque el contenido genómico de cada célula de nuestro cuerpo es casi idéntico, el perfil transcripcional de cada tipo de célula difiere enormemente. Las modificaciones epigenéticas en el ADN y las proteínas histonas son reguladores clave de la expresión transcripcional. La eucromatina transcripcionalmente activa se caracteriza por marcas epigenéticas distintas en comparación con la heterocromatina compacta y transcripcionalmente inactiva. Por ejemplo, las regiones heterocromáticas se definen por modificaciones represivas de histonas, incluida la trimetilación en la lisina 9 de la histona 3 (H3K9me3), la trimetilación en la lisina 27 de la histona H3 (H3K27me3) y la metilación del ADN en las citosinas junto a las guaninas (CpG) en los promotores del gen1. Las regiones genómicas de la expresión génica activa se definen típicamente por la acetilación y trimetilación de histonas en la lisina 4 de la histona 3 (H3K4me3)1.
La revolución de las repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas (CRISPR) ha generado una gran cantidad de herramientas que permiten la alteración programada de las secuencias genómicas. La tecnología CRISPR se basa en un mecanismo de defensa procariota capaz de escindir ácidos nucleicos en secuencias diana programables. Las nucleasas CRISPR 2,3,4, los editores de bases 5,6 y los editores primarios 7,8 pueden cambiar la secuencia de ADN de los genomas de los mamíferos mediante el corte del ADN y la reparación de estas roturas. Aunque efectivas, estas estrategias pueden causar roturas de ADN en sitios fuera del objetivo 9,10 y mutaciones estructurales genómicas a gran escala 11,12,13,14,15. Las herramientas alternativas basadas en CRISPR permiten una modulación manejable de la activación y represión de genes sin cambiar la secuencia de ADN subyacente. Estas herramientas aprovechan una deficiencia de nucleasa Cas9 (dCas9), lo que permite la unión del ADN en los sitios objetivo dictados por la secuencia de sgRNA, en combinación con proteínas efectoras que alteran el paisaje de la cromatina 16,17,18. Las proteínas efectoras, como los escritores epigenéticos, los lectores y los borradores, pueden fusionarse directamente con dCas9 o reclutarse mediante un andamio peptídico fusionado con dCas9, como SunTag, o un andamio de ARN en el sgRNA, como el sistema MS2-MCP 16,17,18. Algunos ejemplos de herramientas de control de transcripción programables son la activación de CRISPR (CRISPRa)19,20,21 y la interferencia de CRISPR (CRISPRi)22,23. CRISPRa funciona reclutando directamente la maquinaria de transcripción, aumentando la expresión génica transcripcional diana19. Por el contrario, CRISPRi reprime la transcripción mediante el establecimiento de H3K9me3, una marca epigenética represiva22.
Los avances en la edición del epigenoma han permitido un amplio uso de estas herramientas en todos los campos científicos. Las fusiones de diferentes dominios efectores y proteínas han ampliado el conjunto de herramientas de editores de epigenomas disponibles 18,24,25,26,27,28,29,30. Además, los editores del epigenoma se utilizan para descifrar las funciones de las modificaciones epigenéticas 31,32,33,34,35,36, los efectores 37,38,39 y las mutaciones efectoras 28,40,41 en la regulación génica. En concreto, CRISPRi y CRISPRa se utilizan en cribados de genómica funcional para una variedad de procesos biológicos, incluyendo la supervivencia celular42 y el destino celular 43,44,45,46,47. Además, la edición del epigenoma tiene potencial terapéutico para la ingeniería celular ex vivo y las terapias in vivo 18.
Aquí, describimos los métodos para aplicar dos editores de epigenoma basados en dCas9 para la represión transcripcional programable en líneas celulares humanas: CRISPRi22 y CRISPRoff48. CRISPRi es una fusión de dCas9 con el dominio represivo KRAB a partir de una proteína de dedos de zinc como ZNF10 (KOX1) y ZIM322,23. Cuando CRISPRi se dirige a un promotor genético específico, el dominio KRAB recluta a un escritor H3K9me3, SETDB1, para reprimir el gen objetivo (Figura 1A). Cuando CRISPRi se expresa de forma transitoria, el H3K9me3 establecido en el locus diana no se mantiene y la expresión génica se restablece con el tiempo32,48. Para llevar a cabo knockdowns estables utilizando CRISPRi, como en las aplicaciones de genómica funcional, es esencial la expresión constitutiva de CRISPRi en las células junto con el sgRNA. Recientemente, CRISPRoff fue diseñado para programar la edición del epigenoma hereditario48. CRISPRoff es una fusión de una sola proteína de dCas9 con el dominio KRAB y el complejo de ADN metiltransferasa de novo, DNMT3A y DNMT3L. Un pulso transitorio de CRISPRoff en células humanas programa la deposición de H3K9me3 y la metilación del ADN en los genes objetivo, lo que conduce a la represión a largo plazo de los genes objetivo mediante el mantenimiento de la metilación del ADN y H3K9me3 (Figura 1B)48. Además, las ediciones del epigenoma se pueden revertir. Por ejemplo, un gen que es silenciado de manera estable por CRISPRoff puede ser reactivado por TET1-dCas9, que puede eliminar enzimáticamente las marcas de metilación del ADN en los lociobjetivo 49.
Este protocolo detallará dos métodos de administración para la expresión transitoria de los editores del epigenoma: la transfección de ADN plasmídico y la nucleofección de ARNm. Además, describimos cómo utilizar la citometría de flujo para evaluar la eficacia de la edición del epigenoma en dos genes endógenos, CLTA y CD55. Estos métodos se pueden adaptar y aplicar a otros experimentos de edición del epigenoma utilizando editores adicionales o se pueden usar para apuntar a diferentes genes.

Figura 1: Esquema del mecanismo de edición del epigenoma de CRISPRi y CRISPRoff y el flujo de trabajo. (A) Esquemas lineales del transgén sgRNA y editor del epigenoma CRISPRi. La adición de CRISPRi y sgRNA permite la adición de la marca de histonas represiva H3K9me3 para silenciar el locus objetivo. El nivel más alto de silenciamiento se logra temprano después de la adición de CRISPRi, y el gen objetivo generalmente se reactiva después de unos pocos pasos. (B) Esquemas lineales del transgén sgRNA y editor del epigenoma CRISPRoff. La adición de CRISPRoff a las células que se dirigen a un gen de interés conduce a la adición de H3K9me3 represivo junto con la metilación del ADN en los sitios CpG para silenciar el gen objetivo. El silenciamiento por CRISPRoff es hereditario: el alto nivel de silenciamiento se logra temprano durante la transfección y persiste a lo largo de múltiples divisiones celulares. (C) Descripción general de la línea de tiempo para la edición del epigenoma a través del método de transfección. En el día 0, las células se siembran para la transfección. El día 1, el editor y el plásmido guía se pueden introducir en las células a través de la transfección. El día 3, se evaluará la expresión de BFP de las células mediante citometría de flujo. El porcentaje de BFP se utiliza como factor normalizador para determinar la eficacia final de silenciamiento del experimento para cada condición. A partir del día 6, se analizan las células para silenciar al reportero de interés, ya que el nivel más alto de silenciamiento se alcanzará en este día. (D) Descripción general del método de transfección en el que el editor del epigenoma y los plásmidos de sgRNA se agregan a las células de manera gota a gota. (E) Visión general de la línea de tiempo para la edición del epigenoma a través de los métodos de nucleofección. En este protocolo, el ARNm es la nucleofección en las células en el día 0. Se evalúa el silenciamiento de las células en el día 3 después de la nucleofección mediante análisis de citometría de flujo. (F) Descripción general del protocolo de nucleofección. Se mezclan cantidades adecuadas de células y ARNm y se añaden a las cubetas nucleofectoras. Si el sgRNA se introduce por nucleofección, también se puede añadir a esta mezcla. Las cubetas se colocan en el nucleofector y se utilizan los códigos de pulso apropiados para introducir el ARNm en las células. Después de la nucleofección, las células se colocan en placas y se pasan para su análisis en días posteriores. (G) Comparación entre la transfección de plásmidos y las estrategias de nucleofección de ARNm para la edición del epigenoma. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
NOTA: El Archivo Suplementario 1 contiene detalles sobre el diseño del sgRNA, la clonación y la generación de líneas celulares de nuestros datos representativos. En la sección de resultados representativos también se detallan las sugerencias de controles.
1. Transfección de plásmidos que expresan el editor del epigenoma en células HEK293T
NOTA: Este protocolo describe la entrega de plásmidos que codifican CRISPR en HEK293T células. Hemos diseñado células para que expresen un sgRNA (Addgene 217306) dirigido a la región promotora de CLTA, un gen no esencial, que está marcado endógenamente con GFP. Las células CLTA-GFP HEK 293T se originaron a partir de un estudio previo50. En este ejemplo, los editores del epigenoma se fusionan directamente con una proteína fluorescente azul (BFP), lo que nos permite cuantificar la eficiencia de la transfección y garantiza una evaluación precisa de las condiciones experimentales. La eficacia del enfoque se demuestra mediante el silenciamiento de CLTA-GFP, que se puede medir cuantitativamente a nivel de proteína en células individuales mediante citometría de flujo.
- Cultivo celular
- Mantenga las células HEK293T en DMEM de alta glucosa con 10% de FBS y 1x penicilina-estreptomicina-glutamina. Pase las células cada 2-3 días, asegurándose de que permanezcan en una confluencia del 60%-70%.
NOTA: Para garantizar una eficiencia óptima de la transfección, es fundamental mantener la confluencia celular por debajo del 70%, lo que se puede monitorear diariamente bajo un microscopio.
- Mantenga las células HEK293T en DMEM de alta glucosa con 10% de FBS y 1x penicilina-estreptomicina-glutamina. Pase las células cada 2-3 días, asegurándose de que permanezcan en una confluencia del 60%-70%.
- Día 0: Siembra HEK293T células para la transfección
- El día antes de la transfección, cuente las células con un contador de células automatizado utilizando tinción viva/muerta con azul de tripán y siembre ~1,5 x 104 células vivas por pocillo en una placa de 96 pocillos. Mantenga el volumen final de cada pocillo en 200 μL. Las células deben alcanzar una confluencia del 60%-70% al día siguiente.
NOTA: Esto también se puede realizar en placas de 24 o 6 pocillos de manera eficiente. Consulte la Tabla 1 para conocer el número de células y la cantidad de ADN para la transfección.
- El día antes de la transfección, cuente las células con un contador de células automatizado utilizando tinción viva/muerta con azul de tripán y siembre ~1,5 x 104 células vivas por pocillo en una placa de 96 pocillos. Mantenga el volumen final de cada pocillo en 200 μL. Las células deben alcanzar una confluencia del 60%-70% al día siguiente.
- Día 1: Transfección de plásmidos CRISPRoff y CRISPRi en células HEK29T
- Alícuota 150 ng del plásmido editor del epigenoma (Addgene 167981) para cada pocillo en un volumen total de 3 μL. Estas diluciones de plásmidos se pueden preparar en tubos de tiras de PCR.
NOTA: Si el editor del epigenoma y el plásmido de sgRNA se transfectan conjuntamente, recomendamos probar una proporción molar de 3:4 de los plásmidos editor:sgRNA. Es posible que sea necesario optimizar la relación para obtener la mayor eficiencia de la edición del epigenoma. - Precaliente el reactivo de transfección y el medio esencial mínimo a temperatura ambiente unos 30-60 minutos antes de la transfección. Mezclar 0,6 μL de reactivo de transfección y 20 μL de medio esencial mínimo por pocillo e incubar a temperatura ambiente durante 15 min.
NOTA: Se puede hacer una mezcla maestra de reactivo de transfección y medio esencial mínimo si se realizan varias transfecciones. - Añada 20,6 μL del reactivo de transfección y una mezcla mínima de medio esencial a cada tubo de tira de PCR que contenga 3 μL de ADN plasmídico. Deje reposar a temperatura ambiente durante 15 min.
- Agregue la mezcla lentamente a la placa de células sembradas y golpee suavemente la placa para dispersar la mezcla. Incluya un control no transfectado para proporcionar una línea de base adecuada para la comparación en los análisis posteriores.
NOTA: Al agregar la mezcla de transfección a las células sembradas, es esencial hacerlo gota a gota y suavemente para minimizar la perturbación, ya que esto puede afectar negativamente la eficiencia de la transfección.
- Alícuota 150 ng del plásmido editor del epigenoma (Addgene 167981) para cada pocillo en un volumen total de 3 μL. Estas diluciones de plásmidos se pueden preparar en tubos de tiras de PCR.
- Día 2: Comprobar el éxito de la transfección
- Examine las células bajo un microscopio con un aumento de 10X para determinar si son BFP positivas, ya que esto indica una transfección exitosa. Asegúrese de que las células no sean perturbadas durante el proceso de observación.
- Día 3: División de la placa de 96 pocillos y evaluación de la eficiencia de la transfección utilizando el marcador BFP
NOTA: Una transfección exitosa debe producir al menos el 30% de las células como BFP positivas. Sin embargo, con dCas9, CRISPRi y CRISPRoff, normalmente detectamos más del 60% de las células positivas para BFP dos días después de la transfección. La eficiencia de la transfección se correlaciona negativamente con el tamaño del plásmido, por lo que diferentes editores del epigenoma pueden producir diferentes eficiencias de transfección.- Precaliente la tripsina y el DMEM con un 10% de FBS y 1x penicilina-estreptomicina-glutamina a temperatura ambiente durante unos 30-60 minutos antes de su uso.
- Aspirar los medios de los pozos; Tenga cuidado de no quitar las celdas adheridas a la parte inferior. Añadir 50 μL de tripsina por pocillo. Incubar durante 2-3 min a 37 °C.
NOTA: No es inusual observar algunas células flotantes, especialmente después de la transfección con CRISPRoff. Nuestra hipótesis es que esta muerte celular se debe a la toxicidad de la sobreexpresión de DNMT3A-3L. - Inhiba la tripsina añadiendo 150 μL de DMEM completo por pocillo. Pipetear hacia arriba y hacia abajo para resuspender las células y dividir las células para la confluencia en el día 5 (~20 μL de mezcla de células en 200 μL de medio de una placa de 96 pocillos).
- Utilice 50 μl de células resuspendidas para la citometría de flujo para evaluar la eficiencia de la transfección.
2. Nucleofección del ARNm editor del epigenoma en células K-562
NOTA: Esta sección detalla el proceso de nucleofectación del ARNm de CRISPRoff en células K-562. Para simplificar, hemos prediseñado las células K-562 para que expresen constitutivamente un sgRNA que se dirija al promotor del gen endógeno CD55 (Addgene 217306). La administración de ARNm de CRISPRoff directamente a las células tiene el potencial de disminuir la toxicidad celular que acompaña a los enfoques basados en el ADN plasmídico y, al mismo tiempo, lograr una eficacia de silenciamiento génico similar. Además, la nucleofección se puede utilizar para introducir construcciones de editor de epigenoma en líneas celulares que son difíciles de transfectar de manera eficiente, como las K-562.
- Cultivo celular
- Antes de la nucleologización, mantenga las células K-562 en un matraz con RPMI con 10% de FBS y 1x penicilina-estreptomicina-glutamina. Pase las celdas todos los días, asegurándose de que permanezcan en una confluencia del 60%-70%.
- El día de la nucleofección, descongele y agite suavemente el ARNm de CRISPRoff en un tubo de microcentrífuga estéril sin ARNasa y almacene el ARNm en hielo. Utilice 2 μg de ARNm por cada 2,0 x 105 células en cada pocillo de una nucleocubeta en tira. Asegúrese de que el volumen de ARNm no supere el 10% del volumen total de nucleofección (22 μL).
NOTA: La nucleofección también se puede realizar en cubetas más grandes con más células y ARNm. Sugerimos escalar la cantidad de ARNm proporcionalmente a la cantidad de células que agregará. Por ejemplo, cuando se realiza una nucleofección en una cubeta grande en 1 x 106 células, sugerimos utilizar 10 μg de ARNm, con un volumen total de nucleofección de 100 μL. Además, la dosis del ARNm se puede optimizar para garantizar una alta eficacia de edición. A menudo usamos 2-5 μg de ARNm editor por cada 2.0 x 105 células para obtener resultados eficientes. Los nucleótidos libres que no se incorporaron durante la síntesis de ARNm deben eliminarse antes de la cuantificación. - Prepare la solución de nucleofección de acuerdo con las instrucciones del fabricante y caliéntela a temperatura ambiente durante 15 minutos antes de la nucleofectuación.
NOTA: La solución del nucleofactor puede ser diferente o necesitar ser optimizada para diferentes tipos de células. - Coseche y cuente las células K-562 utilizando un contador de células automatizado con tinción viva/muerta con azul de tripán. Para la nucleofección en una cubeta de tiras, alícuota ~ 2.0 x 105 células por muestra en un tubo de microcentrífuga estéril.
- Centrifugar las células a 500 x g durante 5 min a temperatura ambiente, luego desechar el sobrenadante. Lave las celdas 1 vez con PBS a temperatura ambiente a 500 x g durante 5 minutos, luego deseche el sobrenadante.
- Calcule la cantidad de solución nucleofectora para resuspender las células restando el volumen de ARNm de 22 μL. Vuelva a suspender las células en la cantidad adecuada de solución nucleofectora.
- Añada la solución celular a 2 μg de ARNm de CRISPRoff (volumen determinado en el paso 2.1.2) y transfiera la solución a una cubeta, teniendo cuidado de no formar burbujas, ya que esto puede comprometer la eficiencia de la nucleofección. Golpee suavemente el nucleocuvette para asegurarse de que las células estén en la parte inferior.
- Nucleofecte las células utilizando el sistema 4D-Nucleofector con el código de pulso adecuado. El código FF-120 se recomienda para las células K-562.
NOTA: Es posible que sea necesario optimizar el código de pulso para diferentes tipos de celdas. Consulte con el fabricante si se necesita una optimización adicional. - Después de la nucleofección, agregue 80 μL de medio RPMI a cada pocillo pequeño de nucleocuvette y deje que las células se asienten a 37 °C durante 15 minutos.
- Transfiera la suspensión celular a un pocillo de una placa de 24 pocillos que contenga 400 μL de medio RPMI precalentado (nucleocuvette pequeño).
NOTA: Después de la nucleologización, las células son particularmente frágiles y deben transferirse del nucleocuvette a los pocillos de cultivo con cuidado. - Después del día 2 después de la nucleofección: siga el protocolo de tinción de marcadores de superficie con el anticuerpo CD55 como se describe en la sección 3 y mida la fluorescencia de CD55 APC en un citómetro de flujo para analizar la eficacia de la edición del epigenoma.
NOTA: Para ensayar el éxito de la nucleofección, recomendamos realizar una nucleofección con GFP o mCherry mRNA. Dos días después de la nucleologización, realice una citometría de flujo de estas células para confirmar el éxito de la nucleofectición. Por lo general, detectamos entre el 90% y el 100% de estas células que expresan GFP o mCherry. Una alternativa es realizar una nucleofección con ARNm de CRISPRi, que se sabe que silencia de forma robusta muchos genes diana. Al realizar la citometría de flujo en el día 2, las muestras tratadas con CRISPRi deben mostrar silenciamiento del gen objetivo en más del 90% de las células.
3. Tinción de marcadores de superficie
NOTA: Esta sección detalla la cuantificación de los niveles de la proteína CD55 después de la edición del epigenoma en células K-562. Cuantificamos la disminución de la expresión de CD55 en células individuales mediante tinción de anticuerpos y citometría de flujo (ver sección 4 a continuación) para evaluar la eficiencia de la eliminación mediada por CRISPRoff. También se pueden utilizar técnicas adicionales, como la PCR cuantitativa con transcripción inversa o el western blot, para confirmar el nivel de knockdown tanto a nivel de transcripción como de proteínas.
- Cuente las células con un contador de células automatizado y añada aproximadamente 5,0 x 105 a 1,0 x 106 células en un tubo de microcentrífuga y centrifuga las células a 500 x g durante 5 min.
NOTA: Se pueden usar más o menos células para la tinción, sin embargo, es posible que sea necesario escalar la concentración de anticuerpos en consecuencia. - Deseche el sobrenadante asegurándose de no alterar el pellet y agregue 500 μL de PBS y vuelva a suspender el pellet suavemente.
- Vuelva a centrifugar las células a 500 x g durante 5 min. Deseche el sobrenadante y agregue el anticuerpo a la concentración deseada diluido en PBS. Para la tinción de CD55, use 1 μL de CD55 antihumano purificado (0,5 mg/mL de caldo) en 49 μL de PBS.
- Pipetea suavemente las células y déjalas incubar durante 25 minutos en un lugar oscuro a temperatura ambiente. Después de la incubación, vuelva a centrifugar las células a 500 x g durante 5 min y deseche el sobrenadante.
- Vuelva a suspender el pellet con 500 μL de PBS para eliminar el exceso de anticuerpos. Centrifugar las células a 500 x g durante 5 min y desechar el sobrenadante PBS. Vuelva a suspender de 100 μL a 200 μL de PBS fresco y agréguelo a una placa de 96 pocillos para el análisis de citometría de flujo.
4. Citometría de flujo
NOTA: Este protocolo está escrito para el uso de un analizador de células BD FACSymphony A1. Los detalles pueden variar según el citómetro de flujo que esté utilizando. Le sugerimos que consulte el manual del usuario de la máquina que está utilizando para obtener información específica.
- Antes de encender la citometría, compruebe que el contenedor de residuos no esté lleno y que haya suficiente líquido de la vaina. Encienda el interruptor de alimentación principal (verde) que se encuentra cerca del líquido de la funda.
- Encienda el lector de placas con el interruptor de retroceso. Luego, encienda la máquina usando el botón verde en el lado derecho y asegúrese de que la máquina esté configurada con la función de lector de placas de 96 pocillos.
- Encienda la computadora e inicie sesión. Abra el software e inicie sesión con su nombre de usuario y contraseña.
- Configura el experimento. Para crear un nuevo experimento, seleccione Experimento > Nuevo experimento. Cambie el nombre del experimento haciendo doble clic en el nombre actual.
- Agregue una placa haciendo clic en Experimentar > nueva placa. Seleccione el formato de pocillo que se va a utilizar. Para agregar pozos específicos para ejecutar, haga clic en la ubicación del pozo en la placa y luego haga clic en la jeringa azul (el pozo ahora debería estar resaltado en azul). Seleccione varios pozos al mismo tiempo para agregarlos, si es necesario.
- Agregue nombres específicos a cada pocillo haciendo clic con el botón derecho en Espécimen a la derecha de la pantalla de la placa. A continuación, seleccione Diseño experimental. Esto abrirá una página con una lista de todos los pozos que seleccionó. Para cambiar el nombre de los pozos, haga clic una vez y escriba el nombre deseado. Haga clic en Aceptar cuando haya terminado.
- Agregue gráficos y puertas apropiadas al experimento en la hoja de cálculo global. Recomendamos trazar primero el área de dispersión directa (FSC-A) frente al área de dispersión lateral (SSC-A). A continuación, crea una puerta arbitraria para las células vivas para refinarlas más tarde haciendo clic en la herramienta Polígono.
- Haga clic con el botón derecho en esta puerta y haga clic en Desglosar. Esto creará un nuevo gráfico que solo mostrará las celdas de esa puerta de celda viva.
- Cambie el eje de este nuevo gráfico a FSC-A frente a la altura de dispersión directa (FSC-H) y cree una puerta para celdas individuales.
- Si expresa constitutivamente el sgRNA en las células, y el lentivirus también codifica para una proteína fluorescente, entonces profundice en esta puerta de una sola célula y realice una compuerta adicional para la expresión de esa proteína como un proxy para la expresión de sgRNA (ejemplo: PE-CF594-A vs FSC-A).
- En la puerta de configuración final (ya sea para células individuales o para células que expresan sgRNA), haga clic con el botón derecho y seleccione Desglose. Cree una puerta para analizar la expresión del gen reportero (ejemplo: BB515-A frente a FSC-A).
- Si utiliza células transfectadas y tiene un método para evaluar la eficiencia de la transfección, como la expresión BFP, haga clic con el botón derecho en el gráfico de expresión reportera y haga clic en Duplicar. Cambie los ejes a BV421-A vs FSC-A. Cree una puerta arbitraria para las celdas BFP positivas.
- Elimine los parámetros que no estén en uso en la pestaña Parámetros de la pantalla del citómetro.
- Para configurar la máquina y prepararla para funcionar, haga clic en Ejecutar en el panel de control del citómetro de flujo. El botón debería aparecer en verde. En el software, vaya a HTS en el panel superior y luego a Prime. Una vez terminado, agregue la placa, asegurándose de que esté bien colocada en la máquina y que la tapa esté OFF.
- Para ejecutar, seleccione todos los pozos que desea ejecutar y asegúrese de que los controles estén configurados correctamente. Seleccione bien haciendo doble clic, cambie el volumen de la muestra a 10 μL y haga clic en Adquirir datos en el Panel de adquisición. Esto ejecutará la muestra, pero no registrará los datos. A medida que se ejecuta esta muestra, ajuste los voltajes del láser según sea necesario. Además, cambie las puertas.
NOTA: Recomendamos ejecutar una muestra que sea negativa para todos los colores que se miden y una que sea positiva para todos los colores para asegurarse de que los parámetros del láser y la compuerta sean buenos. - Después de verificar la configuración del láser, haga clic en las muestras en el panel de placas y verifique que los valores de ejecución estén configurados como se desea.
- Haga clic en Ejecutar placa para ejecutar en el Panel de adquisición y registrar los datos de todos los pozos. Una vez terminado, limpie la máquina retirando la placa.
- A una nueva placa de 96 pocillos, agregue 10% de lejía en las cuatro filas superiores y H2O en los cuatro pocillos inferiores. Vaya a HTS > Clean. Una vez finalizado, cierre el programa y apague los botones en el orden inverso al que se encendieron botón lateral verde > lector de placas > interruptor verde por residuos.
- Agregue un 20% de lejía a la placa de 96 pocillos con celdas y deséchela.
5. Análisis de datos
NOTA: Este método describe las estrategias de compuerta y el procesamiento de datos para cuantificar la edición del epigenoma mediante citometría de flujo. La estrategia de compuerta se representa visualmente junto con gráficos de ejemplo generados a partir del análisis de datos en la Figura 2 y la Figura 3.
- Configuración de compuertas FlowJo
- Cargue todos los archivos FSC en FlowJo arrastrándolos y soltándolos en una nueva hoja de trabajo.
- Haga clic en Todas las muestras para comenzar a hacer puertas. Esto asegurará que las compuertas se puedan aplicar fácilmente a todas las muestras.
- Abra una muestra de control (como sin transfectar o sin nucleofectar) haciendo doble clic en ese archivo.
- Cree una puerta para celdas vivas en un gráfico FSC-A vs SSC-A utilizando la herramienta polígono. En la lista de todas las muestras, ahora debería aparecer una pestaña Células vivas debajo del nombre de la muestra. Haga clic con el botón derecho en esta puerta y seleccione Copiar análisis a grupo para aplicar esta puerta a todas las muestras (Figura 2A, Figura 3A).
- Haga doble clic en la puerta de celdas vivas para explorar en profundidad las celdas vivas. Cambie los ejes a FSC-A vs FSC-H (Figura 2B, Figura 3B).
- Dibuje un polígono para la puerta solo para celdas individuales. Aplique esta puerta a todas las muestras como se hace en la sección 4 (Figura 2B, Figura 3B).
NOTA: Si expresa constitutivamente una construcción de sgRNA con un reportero fluorescente, realice una compuerta adicional en este color para que el análisis solo se centre en las células que expresan el sgRNA. Un ejemplo de esto sería la creación de una puerta para células mCherry positivas (PE-CF594-A frente a FSC-A) si las células contienen un sgRNA de la columna vertebral pLG1 (Figura 2C, Figura 3C). - Haga doble clic en la última puerta de configuración (celdas individuales o celdas que expresan guía).
- Si se realiza una transfección de plásmidos y el editor del epigenoma codifica para una fusión de BFP, cree una puerta para las células BFP positivas (ejemplo: BV421-A frente a FSC-A) para las muestras del día 2 (Figura 2D).
- Cree una puerta para la expresión reportera (ejemplo: BB515-A vs FSC-A para puerta para celdas GFP negativas) y aplique esta puerta a todas las muestras (Figura 2E, Figura 3D).
- Análisis de transfección
- Después de realizar la configuración de la compuerta, haga clic en Editor de tablas en el panel superior. Poblaciones de arrastre para el análisis (ejemplos: BFP positivo, GFP negativo).
- En el panel del editor de tablas, haga clic en Crear tabla. Copie estos datos en una hoja de cálculo para realizar cálculos de normalización.
- Procesamiento de datos
- Reste el número de celdas reporteras negativas (ejemplo: GFP negativas) en la muestra de control de todas las demás muestras. Es posible que haya algún silenciamiento de fondo del reportero, por lo que esto elimina este ruido de fondo de los datos.
- Todos los valores se normalizarán al porcentaje de células BFP positivas (células transfectadas con éxito) medido el día 2. Para realizar esta normalización, tome el valor, divídalo por el porcentaje de células positivas de BFP en el día 2 y multiplíquelo por 100. Este cálculo arroja el valor normalizado de la eficiencia de transfección de las células editadas. Los datos ya están listos para ser trazados. Recomendamos gráficos de líneas a lo largo de un curso de tiempo de edición del epigenoma, como se muestra en la Figura 2F.
- Análisis de nucleofección
- Después de realizar la configuración de la compuerta, haga clic en Editor de tablas en el panel superior. Arrastre poblaciones para el análisis (ejemplos: APC negativo).
- En el panel del editor de tablas, haga clic en Crear tabla. Copie estos datos en un editor de hojas de cálculo para realizar cálculos de normalización.
- Procesamiento de datos
- Reste el número de células reporteras negativas (ejemplo: APC negativo) en la muestra de control de todas las demás muestras. Es posible que haya algún silenciamiento de fondo del reportero, por lo que esto elimina este ruido de fondo de los datos. Los datos ya están listos para ser trazados. Recomendamos gráficos de líneas a lo largo de un curso de tiempo de edición del epigenoma.
NOTA: La estadística se puede utilizar para cuantificar las diferencias en el silenciamiento entre dos editores de epigenoma diferentes. Recomendamos realizar todos los experimentos de transfección y nucleofección por triplicados técnicos para realizar análisis estadísticos. Al representar gráficos lineales de silenciamiento a lo largo del tiempo, represente la desviación estándar de las réplicas técnicas. Se podrían realizar pruebas t de Student en puntos de tiempo específicos para comparar el silenciamiento entre dos editores de epigenoma. Las muestras deben tratarse como no apareadas con varianza desigual.
- Reste el número de células reporteras negativas (ejemplo: APC negativo) en la muestra de control de todas las demás muestras. Es posible que haya algún silenciamiento de fondo del reportero, por lo que esto elimina este ruido de fondo de los datos. Los datos ya están listos para ser trazados. Recomendamos gráficos de líneas a lo largo de un curso de tiempo de edición del epigenoma.
Resultados
Para todos los experimentos de edición del epigenoma, los controles adecuados son fundamentales para evaluar la eficiencia de la edición del epigenoma. Recomendamos utilizar un sgRNA de control, que no se dirija a ninguna secuencia del genoma humano. El uso de un control guía no dirigido dará la confianza de que los cambios en los loci objetivo son impulsados por el editor del epigenoma que se dirige a ese sitio en lugar de solo por la sobreexpresión del editor del epigenoma o la unión no específica. Además, para los experimentos basados en genes reporteros, sugerimos usar un control de solo dCas9 para garantizar que los cambios en la expresión del editor del epigenoma se deban a fusiones del editor del epigenoma en lugar de un obstáculo estérico de la unión de dCas9 al locus objetivo e impida temporalmente la transcripción (Figura 2F).
Para los experimentos de transfección, recomendamos utilizar un editor de epigenoma con una fusión de proteínas fluorescentes adicional, como BFP. Esta fusión permite la visualización de células transfectadas con éxito con el editor del epigenoma a través de microscopía y citometría de flujo. Las células transfectadas con éxito expresarán altos niveles de BFP dos días después de la transfección (Figura 2D). La cuantificación de las células transducidas con éxito se utiliza para normalizar la eficacia de la edición del epigenoma en días posteriores (Figura 2F).
Tanto CRISPRoff como CRISPRi muestran un silenciamiento máximo en el día 5 después de la transfección (Figura 2E-F). Los diferentes editores de epigenomas tienen distintas líneas de tiempo de edición del epigenoma, como el silenciamiento hereditario con CRISPRoff y el silenciamiento transitorio con CRISPRi (Figura 2F). La Figura 2F también muestra el uso de dCas9 solo como un control importante para los experimentos de edición del epigenoma. En los experimentos de nucleofección de ARNm, las células editadas con éxito con CRISPRoff mostrarán un fuerte silenciamiento del gen objetivo el día 3 después de la nucleofección (Figura 3E-F).
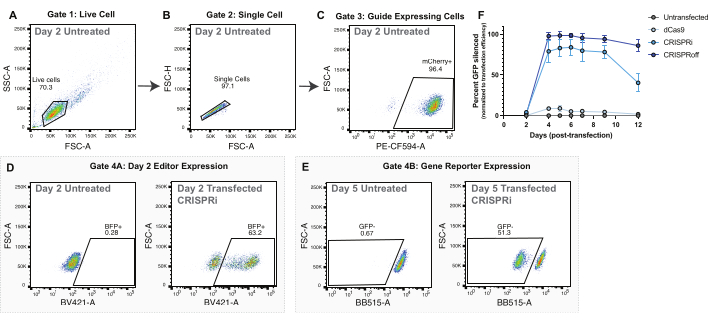
Figura 2: Estrategia de compuerta y datos representativos para la entrega del editor del epigenoma mediante transfección de plásmidos. (A-C) Diagramas de flujo representativos para mostrar la estrategia de compuerta para experimentos de transfección de plásmidos. Los diagramas de flujo mostrados son de células no transfectadas 2 días después de la transfección. Cada punto de las gráficas representa una celda. (A) Diagrama de flujo del área de dispersión directa (FSC-A) y el área de dispersión lateral (SSC-A) con una puerta para celdas vivas. (B) Diagrama de flujo de celdas vivas que muestra FSC-A y altura de dispersión directa (FSC-H) con una puerta para celdas individuales. (C) Diagrama de flujo de celdas individuales graficando PE-CF594-A (expresión de mCherry) y FSC-A. Activación para células mCherry positivas como proxy de la expresión de sgRNA. (D) Estrategia de activación representativa para la expresión del editor del epigenoma (BFP+) en el día 2 después de la transfección. La población parental son células que expresan guía y que están controladas en (C). (E) Estrategia representativa de compuerta para ensayar el silenciamiento del gen reportero CLTA-GFP. La población parental son células que expresan guía y que se encuentran en el panel C. (F) Silenciamiento de CLTA-GFP durante días después de la transfección después de la entrega de plásmidos de dCas9, CRISPRi y CRISPRoff. El porcentaje de CLTA-GFP silenciado se normaliza a la eficiencia de la transfección, medida como células positivas para BFP 2 días después de la transfección. Los puntos son promedios de cuatro réplicas de transfección. Las barras de error representan la desviación estándar. Haga clic aquí para ver una versión más grande de esta figura.
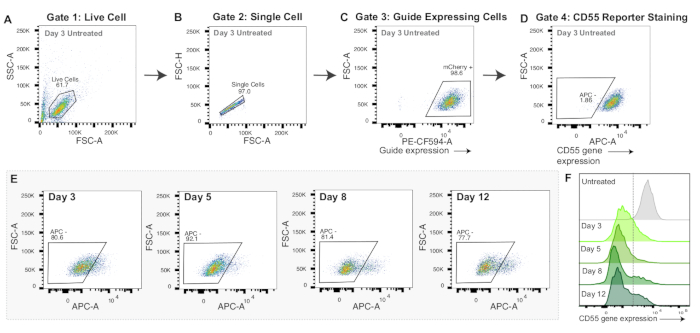
Figura 3: Estrategia de compuerta y datos representativos de la nucleofección de ARNm de CRISPRoff. (A-D) Diagramas de flujo representativos para mostrar la estrategia de compuerta para experimentos de nucleofección de ARNm. Los diagramas de flujo mostrados son de células de control 3 días después de la nucleofección teñidas con el anticuerpo APC anti-humano CD55. Cada punto de las gráficas representa una celda. (A) Diagrama de flujo de la intensidad del área de dispersión directa (FSC-A) y la intensidad del área de dispersión lateral (SSC-A) con una puerta para células vivas. (B) Diagrama de flujo de celdas vivas que muestra FSC-A e intensidad de altura de dispersión directa (FSC-H) con una puerta para celdas individuales. (C) Diagrama de flujo de celdas individuales graficando PE-CF594-A (expresión de mCherry) y FSC-A. Activación para células mCherry positivas como proxy de la expresión de sgRNA. (D) Diagrama de flujo de APC-A frente a FSC-A. Puerta dibujada para células APC negativas, lo que indicaría silenciamiento del gen reportero CD55. (E) Diagramas de flujo de silenciamiento de CD55 (APC - gate) durante 3, 5, 8 y 12 días después de la nucleofección con ARNm de CRISPRoff. (F) Histogramas superpuestos de la expresión de la proteína CD55 (APC-A) durante 3, 5, 8 y 12 días después de la nucleofección con ARNm de CRISPRoff en comparación con un control teñido pero no nucleufectado. Haga clic aquí para ver una versión más grande de esta figura.
| Plato | Densidad de siembra (celdas por pozo) | Cantidad de plásmido |
| 96 pozos | 15,000 | 150 ng |
| 24 pocillos | 90,000 | 500 ng |
| 6 pozos | 400,000 | 2 μg |
Tabla 1: Cantidades de incrustaciones de transfección. Densidades de siembra y cantidades de ADN plasmídico para la transfección del editor del epigenoma en células HEK293T a diferentes escalas.
Tabla suplementaria 1: Secuencias espaciadoras de sgRNA para experimentos de edición de epigenomas. secuencias de sgRNA para dirigir los editores del epigenoma a CLTA y CD55 junto con una secuencia para una guía de control no dirigida. Además, se enumeran los oligonucleótidos para clonar en la red troncal pLG1. Haga clic aquí para descargar este archivo.
Legajo Complementario 1. Haga clic aquí para descargar este archivo.
Discusión
Este protocolo detalla dos métodos diferentes de administración de transitorios para los editores del epigenoma CRISPR: la transfección de ADN plasmídico y la nucleofección de ARNm. Ambas técnicas tienen ventajas, desventajas y consideraciones generales únicas (Figura 1F).
La transfección de ADN plasmídico conduce a una expresión robusta del editor del epigenoma, e incluimos una fusión de BFP dentro de las construcciones del editor del epigenoma que permite la detección y cuantificación de la eficacia de la transfección mediante citometría de flujo. Además, la expresión de BFP se puede usar para ordenar las células que expresan el editor del epigenoma o se puede cuantificar para normalizar los datos de silenciamiento en puntos de tiempo posteriores, como se detalla en este protocolo. Sin embargo, es importante tener en cuenta que la eficiencia de la transfección no suele ser del 100% y, por lo tanto, la población será heterogénea para los que recibieron el editor y los que no, a menos que se ordenen las celdas. Además, la entrega de ADN plasmídico puede desencadenar una respuesta inmunitaria del ADN bicatenario citoplasmático, lo que conduce a la activación de la vía inmunitaria y a la citotoxicidad. Por último, la transfección del ADN plasmídico no es susceptible a todos los tipos de células, y puede ser necesaria la nucleofección del plásmido.
A diferencia de la transfección de ADN plasmídico, la nucleofección de ARNm es aplicable para muchos tipos de células y, a menudo, da lugar a una alta eficacia de administración. El editor: Los ARNm se pueden sintetizar utilizando kits de síntesis de ARNm in vitro disponibles comercialmente, uno de los cuales se ha detallado anteriormente51. Alternativamente, los ARNm se pueden sintetizar a partir de Aldevron o Trilink. Sin embargo, una advertencia de la nucleofección de ARNm es que no produce suficiente proteína editora para determinar qué células recibieron los editores del epigenoma. Los experimentos anteriores sugieren que casi todas las células recibieron el ARNm, ya que detectamos ~ 90% de silenciamiento del gen objetivo en el día 5 (Figura 3F). Sin embargo, recomendamos optimizar los códigos de pulso y la relación ARNm-célula para cada tipo de célula.
Otra consideración es que la edición del epigenoma iniciada por la transfección de ADN plasmídico o la nucleofección de ARNm tiene diferentes líneas de tiempo de silenciamiento. Con la transfección de ADN plasmídico, se observa un silenciamiento máximo con CRISPRoff y CRISPRi en el día 5 después de la transfección (Figura 2F). En comparación, el silenciamiento máximo con CRISPRoff y CRISPRi proporcionado por nucleofección de ARNm se observa ya en el día 3 después de la nucleofección (Figura 3F). Además, el ADN plasmídico persiste en las células en comparación con el ARNm, lo que resulta en una mayor duración de la expresión del editor. Dependiendo de los objetivos experimentales, puede ser preferible una línea de tiempo de silenciamiento más larga o más corta y una duración de la expresión.
También es importante tener en cuenta que la edición del epigenoma no se puede aplicar universalmente a todos los genes o tipos de células. Las diferencias inherentes en la secuencia del genoma o los estados de la cromatina de los genes objetivo pueden afectar la eficacia del editor, incluso para el mismo gen en diferentes tipos de células. Por ejemplo, los genes que carecen de islas CpG anotadas pueden ser difíciles de silenciar de manera estable mediante la metilación del ADN mediada porCRISPRoff 48. Por lo tanto, puede ser necesario probar diferentes editores de epigenomas para silenciar dichos genes. Además, también recomendamos probar al menos los tres principales ARNg de CRISPRi para identificar la mejor guía para una mayor eficiencia de edición del epigenoma42. No obstante, dada nuestra limitada caja de herramientas de editores de epigenomas robustos, puede ser más efectivo llevar a cabo la eliminación de genes o utilizar otras estrategias de eliminación según el gen de interés.
Este protocolo se centra en CRISPRi y CRISPRoff, dos de los muchos editores de epigenomas basados en CRISPR disponibles. Recientes estudios de descubrimiento a gran escala han desarrollado nuevas herramientas para reescribir el epigenoma humano 29,30,52. Los editores de epigenomas tienen aplicaciones en la investigación biomédica y terapéutica. Por ejemplo, estudios recientes utilizaron la metilación del ADN y la edición del epigenoma basada en H3K9me3 en modelos de ratón y primates no humanos, lo que resultó en la represión hereditaria del gen asociado a la enfermedad 53,54,55. Prevemos que las futuras modalidades de entrega para los editores de epigenomas abrirán nuevas vías para la amplia aplicación de la edición del epigenoma.
Divulgaciones
J.K.N. es un inventor de patentes relacionadas con las tecnologías CRISPRoff/on, presentadas por los Regentes de la Universidad de California.
Agradecimientos
Agradecemos a los miembros del laboratorio de Nuñez, especialmente a Rithu Pattali e Izaiah Ornelas, por desarrollar y optimizar los protocolos descritos en este manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
Referencias
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados