Method Article
Plasmid DNA Transfection 및 mRNA Nucleofection Delivery를 사용한 인간 세포의 CRISPR 에피게놈 편집
요약
이 프로토콜은 플라스미드 DNA 형질주입 및 mRNA 핵 박션을 사용하여 인간 세포주에서 CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats) 기반 후성유전체 편집 방법을 설명합니다.
초록
후성유전학(Epigenetics)은 유전자의 발현을 조절할 수 있는 히스톤 단백질과 DNA의 화학적 변형을 말합니다. 인간 후성유전체(epigenome)는 세포 분화와 노화 과정에서 동적으로 변화하며, 많은 질병이 비정상적인 후성유전체 패터닝(epigenome patterning)과 관련이 있습니다. 최근 CRISPR의 발전으로 표적 게놈 자리에서 후성유전학적 변형을 편집할 수 있는 프로그래밍 가능한 도구가 개발되어 인간 세포의 후성유전학적 변형을 정밀하게 다시 작성할 수 있게 되었습니다. CRISPR 기반 후성유전체 편집자는 궁극적으로 포유류 게놈에서 표적 유전자의 프로그래밍된 억제 또는 활성화를 초래하는 후성유전학적 변형자와 결합된 촉매적으로 죽은 Cas9에 의존합니다. 기존의 게놈 편집 방법과 달리 후성유전체 편집은 인간 게놈 염기서열의 DNA 절단이나 변화를 필요로 하지 않으므로 유전자 발현을 제어하는 보다 안전한 대안으로 작용합니다. 이 프로토콜에서는 CRISPR 후성유전체 편집기를 인코딩하는 mRNA의 플라스미드 DNA 형질주입 및 핵절제를 사용하여 인간 세포주에서 dCas9 매개 후성유전체 편집을 수행하는 두 가지 방법을 강조합니다. 당사는 CRISPR 간섭(CRISPRi)을 사용하여 유전자를 일시적으로 억제하고 dCas9를 KRAB 도메인 및 de novo DNA 메틸전이효소 복합체에 융합하는 CRISPRoff를 사용하여 몇 주 동안 유전자를 지속적으로 침묵시키는 프로그래밍 가능한 후성유전체 편집을 시연합니다. 또한 표적 유전자의 성공적인 후성유전체 편집을 측정하기 위한 정량적 방법과 실험 기준에 따라 사용할 후성유전체 편집 도구에 대한 주요 고려 사항에 대한 지침을 제공합니다.
서문
우리 몸에 있는 모든 세포의 게놈 함량은 거의 동일하지만 각 세포 유형의 전사 프로필은 크게 다릅니다. DNA와 히스톤 단백질에 대한 후성유전학적 변형은 전사 발현의 핵심 조절자입니다. 전사 활성 유크로마틴(Euchromatin)은 조밀하고 전사 비활성 헤테로크로마틴(heterochromatin)에 비해 뚜렷한 후성유전학적 표시(epigenetic marks)로 특징지어집니다. 예를 들어, 이색성 영역은 히스톤 3 (H3K9me3)의 라이신 9에 대한 트리메틸화, 히스톤 H3 (H3K27me3)의 라이신 27에 대한 트리메틸화, 유전자 프로모터1에서 구아닌(CpG) 옆의 시토신에 대한 DNA 메틸화를 포함한 억압적 히스톤 변형에 의해 정의됩니다. 활성 유전자 발현의 게놈 영역은 일반적으로 히스톤 3 (H3K4me3) 1의 라이신 4에 대한 히스톤 아세틸화 및 트리메틸화에 의해 정의됩니다.
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats) 혁명은 게놈 염기서열의 프로그래밍된 변경을 가능하게 하는 풍부한 도구를 생성했습니다. CRISPR 기술은 프로그래밍 가능한 표적 염기서열에서 핵산을 절단할 수 있는 원핵 방어 메커니즘을 기반으로 합니다. CRISPR nuclease 2,3,4, base editors 5,6 및 prime editors 7,8은 DNA 절단 및 이러한 절단의 복구를 통해 포유류 게놈의 DNA 염기서열을 변경할 수 있습니다. 이러한 전략은 효과적이기는 하지만, off-target site에서 DNA 파괴 9,10 및 대규모 게놈 구조 돌연변이 11,12,13,14,15를 유발할 수 있습니다. 대체 CRISPR 기반 도구를 사용하면 기본 DNA 염기서열을 변경하지 않고도 유전자 활성화 및 억제를 다루기 쉽게 조절할 수 있습니다. 이러한 도구는 뉴클레아제 결핍 Cas9(dCas9)를 활용하여 염색질 풍경을 변경하는 효과기 단백질과 함께 sgRNA 서열에 의해 지시된 표적 부위에서 DNA 결합을 허용합니다 16,17,18. 후성유전학적 작성자(epigenetic writer), 판독기(reader) 및 지우개(eraser)와 같은 효과기 단백질은 dCas9에 직접 융합되거나 SunTag와 같은 dCas9에 융합된 펩타이드 골격 또는 MS2-MCP 시스템과 같은 sgRNA의 RNA 골격에 의해 모집될 수 있습니다 16,17,18. 프로그래밍 가능한 전사 제어 도구의 예로는 CRISPR 활성화(CRISPRa)19,20,21 및 CRISPR 간섭(CRISPRi)22,23이 있습니다. CRISPRa는 전사 기전을 직접 모집하여 표적 전사 유전자 발현을 증가시킴으로써 작용합니다19. 대조적으로, CRISPRi는 억압적 후성유전학적 표지인 H3K9me3를 확립하여 전사를 억제합니다22.
후성유전체 편집의 발전으로 과학 분야 전반에 걸쳐 이러한 도구를 광범위하게 사용할 수 있게 되었습니다. 서로 다른 effector 도메인과 단백질의 융합은 사용 가능한 후성유전체 편집기 툴킷을 확장했습니다 18,24,25,26,27,28,29,30. 또한 후성유전학적 변형 31,32,33,34,35,36, 효과기37,38,39 및 효과기 돌연변이 28,40,41의 역할을 해독하기 위해 후성유전체 편집기를 사용합니다 유전자 조절에서. 구체적으로, CRISPRi 및 CRISPRa는 세포 생존 42 및 세포 운명 43,44,45,46,47을 포함한 다양한 생물학적 과정을 위한 기능적 유전체학 스크리닝에 사용됩니다. 또한, 후성유전체 편집은 체외세포 공학 및 체내 치료에 대한 치료 잠재력을 가지고 있다18.
여기에서는 인간 세포주에서 프로그래밍 가능한 전사 억제를 위해 두 가지 dCas9 기반 후성유전체 편집기인 CRISPRi22 및 CRISPRoff48을 적용하는 방법에 대해 설명합니다. CRISPRi는 ZNF10 (KOX1) 및 ZIM322,23과 같은 아연 손가락 단백질에서 억제 KRAB 도메인으로 dCas9를 융합한 것입니다. CRISPRi가 특정 유전자 프로모터를 표적으로 삼을 때, KRAB 도메인은 H3K9me3 전달자인 SETDB1을 모집하여 표적 유전자를 억제합니다(그림 1A). CRISPRi가 일시적으로 발현되면 타겟 유전자 자리에서 확립된 H3K9me3가 유지되지 않고 시간이 지남에 따라 유전자 발현이 복원됩니다32,48. 기능적 유전체학 응용 분야와 같이 CRISPRi를 사용하여 안정적인 knockdown을 수행하기 위해서는 sgRNA와 함께 세포에서 CRISPRi의 구성적 발현이 필수적입니다. 최근 CRISPRoff는 유전 가능한 후성유전체 편집을 프로그래밍하도록 설계되었습니다48. CRISPRoff는 dCas9와 KRAB 도메인 및 de novo DNA 메틸전이효소 복합체, DNMT3A 및 DNMT3L의 단일 단백질 융합체입니다. 인간 세포에서 CRISPRoff의 일시적인 펄스는 표적 유전자에서 H3K9me3 및 DNA 메틸화의 증착을 프로그래밍하며, 이는 DNA 메틸화 및 H3K9me3의 유지에 의해 표적 유전자의 장기 억제로 이어집니다(그림 1B)48. 또한 후성유전체 편집을 되돌릴 수 있습니다. 예를 들어, CRISPRoff에 의해 안정적으로 침묵된 유전자는 TET1-dCas9에 의해 재활성화될 수 있으며, 이는 표적 유전자좌49에서 DNA 메틸화 마크를 효소로 제거할 수 있습니다.
이 프로토콜은 후성유전체 편집기의 일시적 발현을 위한 두 가지 전달 방법, 즉 플라스미드 DNA transfection과 mRNA nucleofection에 대해 자세히 설명합니다. 또한 유세포 분석을 사용하여 두 개의 내인성 유전자인 CLTA 및 CD55에서 후성유전체 편집 효능을 평가하는 방법을 간략하게 설명합니다. 이러한 방법은 추가 편집기를 사용하여 다른 후성유전체 편집 실험에 적용 및 적용하거나 다른 유전자를 표적으로 하는 데 사용할 수 있습니다.

그림 1: CRISPRi 및 CRISPRoff 후성유전체 편집 메커니즘 및 워크플로우의 개략도. (A) sgRNA 전이유전자 및 CRISPRi 후성유전체 편집기의 선형 회로도. CRISPRi와 sgRNA를 추가하면 억제적 H3K9me3 히스톤 마크를 추가하여 표적 위치를 침묵시킬 수 있습니다. CRISPRi 첨가 후 가장 높은 수준의 침묵(silencing)이 조기에 이루어지며, 유전자 표적은 일반적으로 몇 번의 계대 후에 재활성화됩니다. (B) sgRNA 전이유전자 및 CRISPRoff 후성유전체 편집기의 선형 회로도. 관심 유전자를 표적으로 하는 세포에 CRISPRoff를 추가하면 CpG 부위에서 DNA 메틸화와 함께 억제성 H3K9me3가 추가되어 표적 유전자를 침묵시킵니다. CRISPRoff에 의한 침묵(silencing)은 유전 가능하며, 높은 수준의 침묵(silencing)은 transfection 중 초기에 달성되며 여러 세포 분열에 걸쳐 지속됩니다. (C) transfection 방법을 통한 후성유전체 편집을 위한 타임라인 개요. 0일차에는 transfection을 위해 세포를 도말합니다. 1일차에는 editor와 guide plasmid를 transfection을 통해 세포에 도입할 수 있습니다. 3일차에는 유세포분석을 통해 세포의 BFP 발현을 평가합니다. BFP의 비율은 각 조건에 대한 실험의 최종 침묵 효능을 결정하기 위한 정규화 계수로 사용됩니다. 6일차부터는 이 날에 가장 높은 수준의 침묵이 달성될 것이므로 관심 보고자를 침묵시키기 위해 세포를 분석합니다. (D) 후성유전체 편집기 및 sgRNA 플라스미드를 적하 방식으로 세포에 첨가하는 형질주입 방법 개요. (E) 핵 절제 방법을 통한 후성유전체 편집을 위한 타임라인 개요. 이 프로토콜에서 mRNA는 0일차에 세포로 핵 절제입니다. 세포는 유세포 분석을 사용하여 핵 절제 후 3일째에 침묵을 평가합니다. (F) 핵 절제 프로토콜의 개요. 적절한 양의 세포와 mRNA를 혼합하여 핵펙터 큐벳에 첨가합니다. sgRNA가 nucleofection에 의해 도입되는 경우, 이 혼합물에 첨가될 수도 있습니다. 큐벳을 핵세포에 넣고 적절한 펄스 코드를 사용하여 mRNA를 세포에 도입합니다. 뉴클레오펙션(nucleofection) 후, 세포는 나중에 분석을 위해 도말화되고 계대분석됩니다. (G) 후성유전체 편집을 위한 플라스미드 형질주입과 mRNA 핵융합 전략의 비교. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
참고: 보충 파일 1 에는 대표 데이터의 sgRNA 설계, 클로닝 및 세포주 생성에 대한 세부 정보가 포함되어 있습니다. 대표 결과 섹션에는 컨트롤에 대한 제안 사항도 자세히 설명되어 있습니다.
1. epigenome editor-expression plasmids를 HEK293T 세포로 transfection
참고: 이 프로토콜은 CRISPR 인코딩 플라스미드를 HEK293T 세포로 전달하는 방법을 설명합니다. 우리는 GFP로 내인성 표지된 비필수 유전자인 CLTA의 프로모터 영역을 표적으로 하는 sgRNA(Addgene 217306)를 발현하도록 세포를 설계했습니다. CLTA-GFP HEK 293T 세포는 이전 연구50에서 유래했습니다. 이 예시에서 후성유전체 편집기는 청색 형광 단백질(BFP)에 직접 융합되어 있어 transfection efficiency를 정량화하고 실험 조건의 정확한 평가를 보장할 수 있습니다. 이 접근법의 효능은 유세포 분석을 사용하여 단일 세포의 단백질 수준에서 정량적으로 측정할 수 있는 CLTA-GFP의 침묵에 의해 입증되었습니다.
- 세포 배양
- 10% FBS와 1x 페니실린-스트렙토마이신-글루타민으로 HEK293T 세포를 고포도당 DMEM으로 유지합니다. 2-3일마다 세포를 통과시켜 60%-70%의 포화도를 유지하도록 합니다.
참고: 최적의 transfection 효율을 보장하려면 세포 밀도를 70% 미만으로 유지하는 것이 중요하며, 이는 현미경으로 매일 모니터링할 수 있습니다.
- 10% FBS와 1x 페니실린-스트렙토마이신-글루타민으로 HEK293T 세포를 고포도당 DMEM으로 유지합니다. 2-3일마다 세포를 통과시켜 60%-70%의 포화도를 유지하도록 합니다.
- Day 0: transfection을 위한 HEK293T 세포 파스팅
- transfection 전날, trypan blue live/dead staining을 사용하여 자동 세포 카운터로 세포를 계수하고 96웰 플레이트에서 웰당 ~1.5 x 104 개의 살아있는 세포를 시드합니다. 각 웰의 최종 부피를 200μL로 유지합니다. 세포는 다음 날 60%-70% 포화도에 도달해야 합니다.
참고: 이 작업은 24웰 또는 6웰 플레이트에서도 효율적으로 수행할 수 있습니다. transfection을 위한 세포 수와 DNA 양에 대해서는 표 1 을 참조하십시오.
- transfection 전날, trypan blue live/dead staining을 사용하여 자동 세포 카운터로 세포를 계수하고 96웰 플레이트에서 웰당 ~1.5 x 104 개의 살아있는 세포를 시드합니다. 각 웰의 최종 부피를 200μL로 유지합니다. 세포는 다음 날 60%-70% 포화도에 도달해야 합니다.
- 1일차: CRISPRoff 및 CRISPRi 플라스미드를 HEK29T 세포로 transfection
- 각 웰에 대해 3μL 총 부피의 후성유전체 편집기 플라스미드(Addgene 167981) 150ng 부분 표본. 이러한 플라스미드 희석액은 PCR 스트립 튜브에서 제조할 수 있습니다.
참고: epigenome editor와 sgRNA plasmid가 동시 transfection되는 경우 editor:sgRNA plasmids의 3:4 몰 비율을 시도하는 것이 좋습니다. 후성유전체 편집의 최고 효율성을 산출하기 위해 비율을 최적화해야 할 수 있습니다. - transfection 시약과 최소 필수 배지를 transfection 약 30-60분 전에 실온으로 예열합니다. 웰당 0.6μL의 transfection 시약과 20μL의 최소 필수 배지를 혼합하고 실온에서 15분 동안 배양합니다.
참고: 여러 transfection을 수행하는 경우 transfection 시약과 최소한의 필수 배지를 혼합할 수 있습니다. - 20.6 μL의 transfection 시약과 최소 필수 배지 혼합물을 3 μL의 plasmid DNA를 함유한 각 PCR 스트립 튜브에 추가합니다. 실온에서 15분 동안 그대로 두십시오.
- 씨앗이 뿌려진 세포의 플레이트에 혼합물을 천천히 추가하고 플레이트를 가볍게 두드려 혼합물을 분산시킵니다. 다운스트림 분석에서 비교를 위한 적절한 기준선을 제공하기 위해 transfection되지 않은 대조군을 포함합니다.
참고: 시딩된 세포에 transfection 혼합물을 첨가할 때는 transfection 효율에 부정적인 영향을 미칠 수 있으므로 방해를 최소화하기 위해 적반하고 부드럽게 첨가하는 것이 필수적입니다.
- 각 웰에 대해 3μL 총 부피의 후성유전체 편집기 플라스미드(Addgene 167981) 150ng 부분 표본. 이러한 플라스미드 희석액은 PCR 스트립 튜브에서 제조할 수 있습니다.
- 2일차: transfection 성공 확인
- 10X 배율로 현미경으로 세포를 검사하여 성공적인 transfection을 나타내는 BFP 양성인지 확인합니다. 관찰 과정에서 세포가 방해받지 않는지 확인하십시오.
- 3일차: 96웰 플레이트를 분할하고 BFP 마커를 사용하여 transfection 효율 평가
참고: transfection이 성공적으로 진행되려면 세포의 30% 이상이 BFP 양성이어야 합니다. 그러나 dCas9, CRISPRi 및 CRISPRoff를 사용하면 일반적으로 transfection 2일 후에 60% 이상의 BFP 양성 세포를 검출할 수 있습니다. Transfection 효율은 플라스미드 크기와 음의 상관관계가 있으므로 다른 epigenome editor는 다른 transfection 효율을 산출할 수 있습니다.- 10% FBS와 1x 페니실린-스트렙토마이신-글루타민이 함유된 트립신과 DMEM을 사용하기 전에 약 30-60분 동안 실온으로 예열합니다.
- 우물에서 매체를 흡입하십시오. 바닥에 붙어있는 세포를 제거하지 않도록 주의하세요. 웰당 50μL의 트립신을 추가합니다. 37 °C에서 2-3분 동안 배양합니다.
참고: 특히 CRISPRoff를 사용한 transfection 후 일부 부유 세포를 관찰하는 것은 드문 일이 아닙니다. 우리는 이러한 세포 사멸이 DNMT3A-3L 과발현의 독성에 기인한다는 가설을 세웠습니다. - 웰당 150μL의 완전한 DMEM을 첨가하여 트립신을 억제합니다. 5일차에 세포를 재현탁하고 세포를 분할하기 위해 위아래로 피펫팅합니다(96웰 플레이트의 배지 200μL에 ~20μL의 세포 혼합물).
- transfection 효율을 평가하기 위해 유세포 분석을 위해 50 μL의 재현 유산소 세포를 사용합니다.
2. 후성유전체 편집기 mRNA를 K-562 세포로 핵 추출
참고: 이 섹션에서는 CRISPRoff mRNA를 K-562 세포로 핵 절단하는 과정에 대해 자세히 설명합니다. 단순화를 위해 K-562 세포를 사전 엔지니어링하여 CD55 내인성 유전자(Addgene 217306)의 프로모터를 표적으로 하는 sgRNA를 구조적으로 발현하도록 했습니다. CRISPRoff mRNA를 세포에 직접 전달하면 플라스미드 DNA 기반 접근법에 수반되는 세포 독성을 줄이면서 유사한 유전자 침묵 효능을 달성할 수 있습니다. 또한 nucleofection은 K-562와 같이 효율적으로 transfection하기 어려운 세포주에 후성유전체 편집기 구조를 도입하는 데 사용할 수 있습니다.
- 세포 배양
- 핵 절제에 앞서, 10% FBS와 1x 페니실린-스트렙토마이신-글루타민이 함유된 RPMI가 있는 플라스크에 K-562 세포를 유지합니다. 매일 세포를 통과시켜 60%-70%의 포화도를 유지하도록 합니다.
- 핵 절제 당일, 멸균 RNase가 없는 미세원심분리 튜브에서 CRISPRoff mRNA를 해동하고 부드럽게 와류로 일그러뜨린 후 mRNA를 얼음에 보관합니다. 스트립 핵융합체의 각 웰에 있는 2.0 x 105 개 세포당 2μg의 mRNA를 사용합니다. mRNA의 부피가 총 핵 절단 부피(22μL)의 10%를 초과하지 않는지 확인합니다.
참고: 핵 절제는 더 많은 세포와 mRNA가 있는 더 큰 큐벳에서도 수행할 수 있습니다. 추가할 세포의 수에 비례하여 mRNA의 양을 조정하는 것이 좋습니다. 예를 들어, 1 x 106 세포의 대형 큐벳에서 핵 절제를 수행하는 경우 총 핵 추출 부피가 100μL인 10μg의 mRNA를 사용하는 것이 좋습니다. 또한 mRNA의 투여량을 최적화하여 높은 편집 효능을 보장할 수 있습니다. 효율적인 결과를 위해 2.0 x 105 세포마다 2-5 μg의 편집기 mRNA를 사용하는 경우가 많습니다. mRNA 합성 중에 통합되지 않은 유리 뉴클레오티드는 정량 전에 제거해야 합니다. - 제조업체의 지침에 따라 핵 제거 용액을 준비하고 핵 제거 전에 15분 동안 실온으로 예열합니다.
참고: nucleofector 용액은 다르거나 다른 세포 유형에 맞게 최적화해야 할 수 있습니다. - 트리판 블루 생염색(trypan blue live/dead staining)이 있는 자동 세포 계수기를 사용하여 K-562 세포를 수확하고 계수합니다. 스트립 큐벳에서 핵 절제의 경우, 샘플당 ~2.0 x 105 세포를 멸균 마이크로 원심분리 튜브에 부분 표본으로 주입합니다.
- 실온에서 500 x g 의 세포를 5분 동안 원심분리한 다음 상층액을 버립니다. 실온 PBS로 500 x g 에서 5분 동안 셀을 1x 세척한 다음 상등액을 버립니다.
- 22 μL에서 mRNA 부피를 빼서 세포를 재현탁시킬 nucleofector 용액의 양을 계산합니다. 적절한 양의 nucleofector 용액에 세포를 재현탁합니다.
- 2μg의 CRISPRoff mRNA(2.1.2단계에서 측정한 부피)에 세포 용액을 첨가하고 용액을 큐벳으로 옮기되, 핵 절제의 효율성을 손상시킬 수 있으므로 기포가 형성되지 않도록 주의합니다. 뉴클레오큐벳을 부드럽게 두드려 세포가 바닥에 있는지 확인합니다.
- 적절한 펄스 코드로 4D-Nucleofector 시스템을 사용하여 세포를 핵 절단합니다. FF-120 코드는 K-562 셀에 권장됩니다.
참고: 펄스 코드는 다양한 세포 유형에 맞게 최적화해야 할 수 있습니다. 추가 최적화가 필요한 경우 제조업체에 문의하십시오. - 핵 절제 후 각 작은 nucleocuvette 웰에 80μL의 RPMI 배지를 추가하고 세포를 37°C에서 15분 동안 그대로 둡니다.
- 세포 현탁액을 400μL의 예열된 RPMI 배지(small nucleocuvette)가 들어 있는 24웰 플레이트의 well로 옮깁니다.
참고: nucleofection 후 세포는 특히 취약하므로 nucleocuvette에서 배양 웰로 부드럽게 전달되어야 합니다. - 2일차 후 뉴클레오펙션(nucleofection) 후: 섹션 3에 설명된 대로 CD55 항체를 사용한 표면 마커 염색 프로토콜에 따라 유세포분석기에서 CD55 APC의 형광을 측정하여 후성유전체 편집 효능을 분석합니다.
참고: 핵 절제 성공을 분석하려면 GFP 또는 mCherry mRNA를 사용하여 핵 절제를 수행하는 것이 좋습니다. nucleofection 2일 후 이 세포의 유세포 분석을 수행하여 성공적인 nucleofection을 확인합니다. 우리는 일반적으로 GFP 또는 mCherry를 발현하는 이러한 세포의 90%에서 100%를 검출합니다. 대안은 많은 표적 유전자를 강력하게 침묵시키는 것으로 알려진 CRISPRi mRNA로 핵 절제를 수행하는 것입니다. 2일차에 유세포분석을 수행할 때 CRISPRi 처리 샘플은 세포의 90% 이상에서 표적 유전자 침묵을 보여야 합니다.
3. 표면 마커 염색
참고: 이 섹션에서는 K-562 세포에서 후성유전체 편집 후 CD55 단백질의 수준을 정량화하는 방법에 대해 자세히 설명합니다. CRISPRoff 매개 녹다운 효율을 평가하기 위해 항체 염색 및 유세포 분석법(아래 섹션 4 참조)을 사용하여 단일 세포에서 CD55 발현 감소를 정량화합니다. 역전사 정량적 PCR 또는 웨스턴 블로팅(western blotting)을 포함한 추가 기술을 사용하여 전사체 및 단백질 수준 모두에서 녹다운(knockdown) 수준을 확인할 수도 있습니다.
- 자동 세포 계수기를 사용하여 세포 수를 계산하고 약 5.0 x 105 - 1.0 x 106 세포를 마이크로 원심분리기 튜브와 원심분리 세포에 500 x g 에서 5분 동안 추가합니다.
참고: 염색에 더 많거나 적은 세포를 사용할 수 있지만 그에 따라 항체 농도를 조정해야 할 수도 있습니다. - 펠릿을 방해하지 않도록 하면서 상등액을 버리고 PBS 500μL를 첨가하고 펠릿을 부드럽게 재현탁합니다.
- 500 x g 에서 5분 동안 세포를 다시 원심분리합니다. 상등액을 버리고 PBS에 희석된 원하는 농도의 항체를 첨가합니다. CD55 염색의 경우 PBS 49μL에 정제된 항인간 CD55(0.5mg/mL 스톡) 1μL를 사용합니다.
- 세포를 부드럽게 피펫팅하고 실온의 어두운 곳에서 25분 동안 배양합니다. 배양 후 세포를 500 x g 에서 5분 동안 다시 회전시키고 상층액을 버립니다.
- 펠릿을 500μL의 PBS로 재현탁하여 과도한 항체를 씻어냅니다. 500 x g 에서 5분 동안 세포를 원심분리하고 PBS 상등액을 버립니다. 100 μL - 200 μL의 새로운 PBS에 재현탁하고 유세포 분석을 위해 96웰 플레이트에 추가합니다.
4. 유세포 분석
참고: 이 프로토콜은 BD FACSymphony A1 세포 분석기를 사용하도록 작성되었습니다. 세부 사항은 사용 중인 유세포 분석기에 따라 달라질 수 있습니다. 자세한 내용은 사용 중인 기계의 사용 설명서를 참조하는 것이 좋습니다.
- 세포 분석을 켜기 전에 폐기물 용기가 가득 차지 않았는지, 피복액이 충분한지 확인하십시오. 피복 유체 근처에 있는 주 전원 스위치(녹색)를 켭니다.
- 후면 스위치를 사용하여 플레이트 리더를 켭니다. 그런 다음 오른쪽에 있는 녹색 버튼을 사용하여 기계를 켜고 기계가 96웰 플레이트 리더 기능으로 설정되어 있는지 확인합니다.
- 컴퓨터를 켜고 로그인합니다. 소프트웨어를 열고 사용자 이름과 비밀번호를 사용하여 로그인하십시오.
- 실험을 설정합니다. 실험 > 새 실험을 선택하여 새 실험을 만듭니다. 현재 이름을 두 번 클릭하여 실험의 이름을 바꿉니다.
- Experiment > New Plate를 클릭하여 플레이트를 추가합니다. 사용할 웰 형식을 선택합니다. 실행할 특정 웰을 추가하려면 플레이트에서 웰의 위치를 클릭한 다음 파란색 주사기를 클릭합니다(이제 웰이 파란색으로 강조 표시되어야 함). 필요한 경우 추가할 웰을 동시에 여러 웰을 선택합니다.
- 플레이트 디스플레이 오른쪽에 있는 Specimen 을 마우스 오른쪽 버튼으로 클릭하여 각 웰에 특정 이름을 추가합니다. 그런 다음 , 실험적 레이아웃을 선택합니다. 그러면 선택한 모든 웰이 나열된 페이지가 열립니다. 웰의 이름을 바꾸려면 한 번 클릭하고 원하는 이름을 입력합니다. 완료되면 확인을 클릭합니다.
- Global Worksheet에서 실험에 플롯과 적절한 게이트를 추가합니다. 먼저 전방 산란 영역(FSC-A) 대 측면 산란 영역(SSC-A)을 플로팅하는 것이 좋습니다. 그런 다음 Polygon Tool을 클릭하여 나중에 구체화할 수 있도록 라이브 셀에 대한 임의의 게이트를 만듭니다.
- 이 게이트를 마우스 오른쪽 버튼으로 클릭하고 Drill Down을 클릭합니다. 이렇게 하면 해당 라이브 셀 게이트의 셀만 표시하는 새 플롯이 생성됩니다.
- 이 새 플롯의 축을 FSC-A 대 전방 산란 높이(FSC-H)로 변경하고 단일 셀에 대한 게이트를 만듭니다.
- 세포에서 sgRNA를 구성적으로 발현하고 렌티바이러스도 형광 단백질을 암호화하는 경우, 이 단일 세포 게이트를 드릴다운하고 sgRNA 발현의 대용물로서 해당 단백질의 발현에 대한 추가 게이팅을 수행합니다(예: PE-CF594-A 대 FSC-A).
- 최종 setup gate(단일 세포 또는 sgRNA 발현 세포)에서 마우스 오른쪽 버튼을 클릭하고 Drill Down을 선택합니다. 리포터 유전자의 발현을 분석하기 위한 게이트를 생성합니다(예: BB515-A 대 FSC-A).
- transfection된 세포를 사용하고 BFP 발현과 같은 transfection 효율을 분석하는 방법이 있는 경우 Reporter Expression Plot 을 마우스 오른쪽 버튼으로 클릭하고 Duplicate를 클릭합니다. 축을 BV421-A 대 FSC-A로 변경합니다. BFP 양성 세포에 대한 임의의 게이트를 생성합니다.
- 세포 분석기 디스플레이의 Parameters 탭에서 사용하지 않는 매개변수를 제거합니다.
- 기계를 설정하고 실행을 준비하려면 유세포 분석기 제어판에서 실행을 클릭합니다. 버튼이 녹색으로 나타나야 합니다. 소프트웨어에서 상단 패널의 HTS로 이동한 다음 Prime으로 이동합니다. 완료되면 플레이트를 추가하고 기계에 잘 배치되어 있고 뚜껑이 꺼져 있는지 확인합니다.
- 실행하려면 실행할 모든 웰을 선택하고 컨트롤이 적절하게 설정되었는지 확인합니다. 두 번 클릭하여 well을 선택하고 샘플 부피를 10μL로 변경한 다음 Acquisition Dashboard에서 Acquire Data 를 클릭합니다. 이렇게 하면 샘플이 실행되지만 데이터는 기록되지 않습니다. 이 샘플이 실행 중일 때 필요에 따라 레이저 전압을 조정합니다. 또한 게이트를 변경하십시오.
참고: 측정되는 모든 색상에 대해 음수인 샘플과 모든 색상에 대해 양수인 샘플을 실행하여 레이저 매개변수와 게이팅이 양호한지 확인하는 것이 좋습니다. - 레이저 설정을 확인한 후 Plate Panel(플레이트 패널)에서 Specimens(표본 )를 클릭하고 실행 값이 원하는 대로 설정되었는지 확인합니다.
- Run plate를 클릭하여 Acquisition Dashboard에서 실행하고 모든 웰의 데이터를 기록합니다. 완료되면 플레이트를 제거하여 기계를 청소하십시오.
- 새로운 96웰 플레이트에 상단 4개 행에 10% 표백제를 추가하고 아래 4개의 웰에 H2O를 추가합니다. HTS > Clean으로 이동합니다. 완료되면 프로그램을 닫고 녹색 측면 버튼 > 플레이트 리더 > 녹색 스위치를 낭비로 켜진 역순으로 버튼을 끕니다.
- 세포가 있는 96웰 플레이트에 20% 표백제를 넣고 버립니다.
5. 데이터 분석
참고: 이 방법은 유세포 분석에 의한 후성유전체 편집을 정량화하기 위한 게이팅 전략 및 데이터 처리를 간략하게 설명합니다. 게이팅 전략은 그림 2 및 그림 3의 데이터 분석에서 생성된 예시 플롯과 함께 시각적으로 표시됩니다.
- FlowJo 게이팅 설정
- 모든 FSC 파일을 새 워크시트로 끌어다 놓아 FlowJo에 로드합니다.
- All Samples를 클릭하여 게이트 제작을 시작합니다. 이렇게 하면 게이트를 모든 샘플에 쉽게 적용할 수 있습니다.
- 해당 파일을 두 번 클릭하여 대조군 샘플(예: transfected 또는 un-nucleofected)을 엽니다.
- 폴리곤 도구를 사용하여 FSC-A 대 SSC-A 플롯에서 라이브 셀에 대한 게이트를 만듭니다. 이제 모든 샘플 목록에서 Live Cells 탭이 샘플 이름 아래에 나타나야 합니다. 이 게이트를 마우스 오른쪽 버튼으로 클릭하고 Copy Analysis to Group 을 선택하여 이 게이트를 모든 샘플에 적용합니다(그림 2A, 그림 3A).
- Live cells 게이트를 두 번 클릭하여 라이브 셀을 드릴다운합니다. 축을 FSC-A 대 FSC-H로 변경합니다(그림 2B, 그림 3B).
- 단일 셀에 대해서만 게이트를 표시하는 다각형을 그립니다. 섹션 4(그림 2B, 그림 3B)에서 수행한 것처럼 이 게이트를 모든 샘플에 적용합니다.
참고: 형광 리포터로 sgRNA 구조체를 구성적으로 발현하는 경우, 분석이 sgRNA를 발현하는 세포에만 초점을 맞추도록 이 색상에 대한 추가 게이팅을 수행합니다. 예를 들어, 세포에 pLG1 골격의 sgRNA가 포함된 경우 mCherry 양성 세포(PE-CF594-A 대 FSC-A)에 대한 게이트를 생성할 수 있습니다(그림 2C, 그림 3C). - Last Setup Gate(마지막 설정 게이트)를 두 번 클릭합니다(단일 셀 또는 가이드 표현 셀).
- 플라스미드 형질주입을 수행하고 후성유전체 편집기가 BFP 융합을 위해 인코딩하는 경우, Day 2 샘플에 대한 BFP 양성 세포(예: BV421-A 대 FSC-A)에 대한 게이트를 생성합니다(그림 2D).
- 리포터 발현을 위한 게이트를 생성하고(예: BB515-A 대 FSC-A를 사용하여 GFP 음성 세포를 게이트) 이 게이트를 모든 샘플에 적용합니다(그림 2E, 그림 3D).
- Transfection 분석
- 게이팅 설정을 수행한 후 상단 패널에서 테이블 편집기를 클릭합니다. 분석을 위해 모집단을 드래그합니다(예: BFP 양성, GFP 음수).
- 테이블 편집기 패널에서 Create Table을 클릭합니다. 이러한 데이터를 스프레드시트에 복사하여 정규화 계산을 수행합니다.
- 데이터 처리
- 다른 모든 샘플에서 대조군 샘플의 리포터 음성 세포(예: GFP 음성)의 수를 뺍니다. 리포터의 일부 배경 침묵이 있을 수 있으므로 데이터에서 이러한 배경 소음이 제거됩니다.
- 모든 값은 2일차에 측정된 BFP 양성 세포(성공적으로 transfection된 세포)의 백분율로 정규화됩니다. 이 정규화를 수행하려면 값을 취하고 2일차에 BFP 양성 세포의 백분율로 나누고 100을 곱합니다. 이 계산은 편집된 세포의 transfection efficiency normalized 값을 산출합니다. 이제 데이터를 그릴 준비가 되었습니다. 그림 2F에 표시된 것처럼 후성유전체 편집 시간 경과에 걸쳐 선 그래프를 사용하는 것이 좋습니다.
- 핵 절제 분석
- 게이팅 설정을 수행한 후 상단 패널에서 테이블 편집기를 클릭합니다. 분석을 위해 모집단을 드래그합니다(예: APC 네거티브).
- 테이블 편집기 패널에서 Create Table을 클릭합니다. 이러한 데이터를 스프레드시트 편집기에 복사하여 정규화 계산을 수행합니다.
- 데이터 처리
- 다른 모든 샘플에서 대조군 샘플의 Reporter 음성 셀(예: APC 음성)의 수를 뺍니다. 리포터의 일부 배경 침묵이 있을 수 있으므로 데이터에서 이러한 배경 소음이 제거됩니다. 이제 데이터를 그릴 준비가 되었습니다. 후성유전체 편집 시간 과정 전반에 걸쳐 선 그래프를 사용하는 것이 좋습니다.
참고: 통계는 두 개의 서로 다른 후성유전체 편집기 간의 침묵 차이를 정량화하는 데 사용할 수 있습니다. 통계적 분석을 수행하기 위해 모든 transfection 및 nucleofection 실험을 기술적 삼중회에서 수행하는 것이 좋습니다. 시간 경과에 따른 침묵의 선 그래프를 나타낼 때 기술 반복실험의 표준 편차를 플롯합니다. 두 후성유전체 편집기 간의 침묵을 비교하기 위해 특정 시점에서 학생 t-테스트를 수행할 수 있습니다. 표본은 불균등 분산과 짝을 이루지 않은 것으로 처리해야 합니다.
- 다른 모든 샘플에서 대조군 샘플의 Reporter 음성 셀(예: APC 음성)의 수를 뺍니다. 리포터의 일부 배경 침묵이 있을 수 있으므로 데이터에서 이러한 배경 소음이 제거됩니다. 이제 데이터를 그릴 준비가 되었습니다. 후성유전체 편집 시간 과정 전반에 걸쳐 선 그래프를 사용하는 것이 좋습니다.
결과
모든 후성유전체 편집 실험에서 적절한 대조군은 후성유전체 편집 효율성을 평가하는 데 중요합니다. 인간 게놈의 염기서열을 표적으로 하지 않는 대조군 sgRNA를 사용하는 것이 좋습니다. 비표적 가이드 대조군을 사용하면 표적 유전자 자리의 변화가 후성유전체 편집기, 과발현 또는 비특이적 결합이 아니라 해당 부위로 유도되는 후성유전체 편집기에 의해 주도된다는 확신을 가질 수 있습니다. 또한 리포터 유전자 기반 실험의 경우 dCas9 단독 대조군을 사용하여 리포터 발현의 변화가 dCas9이 타겟 자리에 결합하는 입체 방해가 아닌 후성유전체 편집기 융합으로 인한 것인지 확인하는 것이 좋습니다(그림 2F).
transfection 실험의 경우 BFP와 같은 추가 형광 단백질 융합과 함께 epigenome editor를 사용하는 것이 좋습니다. 이 융합을 통해 현미경 및 유세포 분석을 통해 epigenome editor로 성공적으로 transfection된 세포를 시각화할 수 있습니다. 성공적으로 transfection된 세포는 transfection 2일 후에 높은 수준의 BFP를 발현합니다(그림 2D). 성공적으로 형질도입된 세포의 정량화는 나중에 후성유전체 편집 효능을 정상화하는 데 사용됩니다(그림 2F).
CRISPRoff와 CRISPRi 모두 transfection 후 5일째에 최대 침묵을 보여줍니다(그림 2E-F). 서로 다른 후성유전체 편집기는 CRISPRoff를 사용한 유전적 침묵 및 CRISPRi를 사용한 일시적인 침묵과 같은 후성유전체 편집의 고유한 타임라인을 가지고 있습니다(그림 2F). 그림 2F는 또한 dCas9를 후성유전체 편집 실험을 위한 중요한 대조군으로만 사용하는 것을 보여줍니다. mRNA 핵 절제 실험에서 CRISPRoff로 성공적으로 편집된 세포는 핵 절제 후 3일차까지 표적 유전자의 강력한 침묵을 보여줍니다(그림 3E-F).
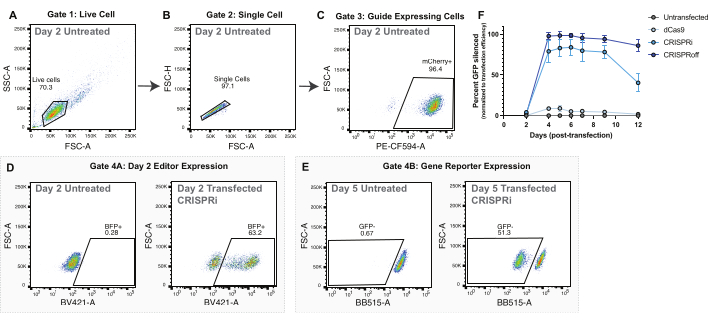
그림 2: 플라스미드 형질주입(transfection)에 의한 후성유전체 편집기 전달을 위한 게이팅(Gating) 전략 및 대표 데이터. (에이씨) plasmid transfection 실험에 대한 게이팅 전략을 표시하기 위한 대표적인 flow plot. 표시된 플로우 플롯은 transfection 후 2일이 지난 후 transfection되지 않은 세포에 대한 것입니다. 플롯의 각 점은 하나의 셀을 나타냅니다. (A) 살아있는 세포에 대한 게이트가 있는 전방 산란 영역(FSC-A) 및 측면 산란 영역(SSC-A)의 흐름도. (B) 단일 셀용 게이트가 있는 FSC-A 및 전방 산란 높이(FSC-H)를 표시하는 라이브 셀의 흐름도. (C) PE-CF594-A(mCherry 발현) 및 FSC-A를 그래프로 표시한 단일 셀의 흐름도. sgRNA 발현을 위한 프록시로서의 mCherry 양성 세포에 대한 게이팅. (D) transfection 후 2일차에 후성유전체 편집기 발현(BFP+)에 대한 대표적인 게이팅 전략. 모 집단은 (C)에서 게이트된 세포를 가이드 발현하고 있습니다. (E) CLTA-GFP 리포터 유전자의 침묵을 분석하기 위한 대표적인 게이팅 전략. Parent Population은 panel C에서 gated 세포를 가이드-발현하고 있습니다. (F) dCas9, CRISPRi 및 CRISPRoff의 플라스미드 전달 후 transfection 후 며칠 동안 CLTA-GFP의 침묵. 침묵된 CLTA-GFP 비율은 transfection 2일 후에 BFP 양성 세포로 측정된 transfection 효율로 정규화됩니다. 점은 4번의 transfection 반복의 평균입니다. 오차 막대는 표준 편차를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
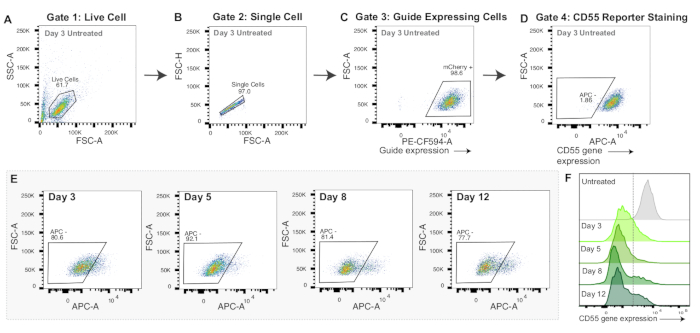
그림 3: CRISPRoff mRNA nucleofection에 대한 게이팅 전략 및 대표 데이터. (A-D) mRNA 핵 절제 실험을 위한 게이팅 전략을 표시하기 위한 대표적인 유동 플롯. 표시된 플로우 플롯은 APC anti-human CD55 항체로 염색된 핵 절제 후 3일 후의 대조 세포입니다. 플롯의 각 점은 하나의 셀을 나타냅니다. (A) 살아있는 세포에 대한 게이트가 있는 전방 산란 영역 강도(FSC-A) 및 측면 산란 영역 강도(SSC-A)의 흐름도. (B) 단일 셀용 게이트가 있는 FSC-A 및 전방 산란 높이 강도(FSC-H)를 표시하는 Live Cells의 흐름도. (C) PE-CF594-A(mCherry 발현) 및 FSC-A를 그래프로 표시한 단일 셀의 흐름도. sgRNA 발현을 위한 프록시로서의 mCherry 양성 세포에 대한 게이팅. (D) APC-A와 FSC-A의 흐름도. APC 음성 세포에 대해 그려진 게이트는 CD55 리포터 유전자 침묵을 나타냅니다. (E) CRISPRoff mRNA를 사용한 핵 절제 후 3, 5, 8, 12일 동안 CD55 침묵(APC-게이트)의 흐름도. (F) CRISPRoff mRNA를 사용한 핵 절단 후 3, 5, 8, 12일 동안 CD55 단백질 발현(APC-A)의 중첩된 히스토그램을 염색되었지만 핵이 없는 대조군과 비교했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 접시 | 파종 밀도 (웰 당 세포) | 플라스미드 양 |
| 96웰 | 15,000 | 150 그램 |
| 24웰 | 90,000 | 500 그램 |
| 6웰 | 400,000 | 2 μg |
표 1: Transfection 스케일링 양. epigenome editor transfection을 위한 seeding density와 plasmid DNA 양은 서로 다른 규모의 HEK293T 세포로 transfection합니다.
보충 표 1: 후성유전체 편집 실험을 위한 sgRNA 스페이서 염기서열. CLTA 및 CD55에 대한 후성유전체 편집기를 표적으로 하기 위한 sgRNA 염기서열과 비표적화 control guide를 위한 염기서열. 또한, pLG1 골격으로의 복제를 위한 올리고가 나열되어 있습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 1. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
이 프로토콜은 CRISPR 후성유전체 편집기에 대한 두 가지 다른 일시적 전달 방법, 즉 플라스미드 DNA transfection과 mRNA nucleofection에 대해 자세히 설명합니다. 두 기술 모두 고유한 장점, 단점 및 일반적인 고려 사항이 있습니다(그림 1F).
Plasmid DNA transfection은 강력한 epigenome editor 발현을 유도하며, 유세포 분석을 사용하여 transfection 효능을 검출하고 정량화할 수 있는 epigenome editor 구조체 내에 BFP 융합을 포함합니다. 또한 BFP 발현은 후성유전체 편집기를 발현하는 세포를 분류하는 데 사용할 수 있으며, 이 프로토콜에 자세히 설명된 바와 같이 나중에 침묵 데이터를 정규화하기 위해 정량화할 수 있습니다. 그러나, transfection 효율은 일반적으로 100%가 아니므로, 세포를 분류하지 않는 한, editor를 받은 개체와 받지 않은 개체군이 이질적일 것이라는 점에 유의하는 것이 중요합니다. 또한 플라스미드 DNA 전달은 세포질 이중 가닥 DNA에서 면역 반응을 유발하여 면역 경로 활성화 및 세포 독성을 유발할 수 있습니다. 마지막으로, 플라스미드 DNA 형질주입은 모든 세포 유형에 적용할 수 있는 것은 아니며, 플라스미드의 핵 삽입이 필요할 수 있습니다.
플라스미드 DNA transfection과 달리 mRNA nucleofection은 많은 세포 유형에 적용할 수 있으며 종종 높은 전달 효능을 제공합니다. 편집기 mRNA는 상업적으로 이용 가능한 시험관 내 mRNA 합성 키트를 사용하여 합성할 수 있으며, 그 중 하나는 이전에 자세히 설명되었습니다51. 대안적으로, mRNA는 Aldevron 또는 Trilink로부터 합성될 수 있다. 그러나 mRNA nucleofection의 한 가지 주의 사항은 어떤 세포가 후성유전체 편집기를 받았는지 분석하기에 충분한 편집 단백질을 생성하지 않는다는 것입니다. 위의 실험은 5일째에 표적 유전자의 ~90%가 침묵하는 것을 감지함에 따라 거의 모든 세포가 mRNA를 받았음을 시사합니다(그림 3F). 그러나 각 세포 유형에 대해 펄스 코드와 mRNA 대 세포 비율을 최적화하는 것이 좋습니다.
또 다른 고려 사항은 플라스미드 DNA 형질주입 또는 mRNA 핵절제에 의해 시작된 후성유전체 편집이 서로 다른 침묵 타임라인을 갖는다는 것입니다. plasmid DNA transfection을 사용하면 transfection 후 5일째에 CRISPRoff 및 CRISPRi를 사용한 피크 침묵이 관찰됩니다(그림 2F). 이에 비해 mRNA 핵 절제에 의해 전달되는 CRISPRoff 및 CRISPRi의 최대 침묵은 핵 절제 후 3일째 되는 날부터 볼 수 있습니다(그림 3F). 또한 plasmid DNA는 mRNA에 비해 세포에 남아 있어 editor 발현 기간이 더 길어집니다. 실험 목표에 따라 더 길거나 더 짧은 침묵 타임라인과 발현 시간이 선호될 수 있습니다.
또한 후성유전체 편집이 모든 유전자 또는 세포 유형에 보편적으로 적용될 수 없다는 점에 유의하는 것이 중요합니다. 표적 유전자의 게놈 염기서열 또는 염색질 상태의 내재적 차이는 다른 세포 유형에서 동일한 유전자에 대해서도 편집기 효능에 영향을 미칠 수 있습니다. 예를 들어, 주석이 달린 CpG 섬이 없는 유전자는 CRISPRoff 매개 DNA 메틸화에 의해 안정적으로 침묵시키기 어려울 수 있습니다48. 그러므로, 그러한 유전자를 침묵시키기 위해 다른 후성유전체 편집기를 검사하는 것이 필요할 수 있습니다. 또한, 보다 높은 후성유전체 편집 효율을 위한 최상의 가이드를 식별하기 위해 상위 3개 이상의 CRISPRi gRNA를 테스트하는 것이 좋습니다42. 그럼에도 불구하고, 강력한 후성유전체 편집기의 제한된 도구 상자를 감안할 때, 관심 유전자에 따라 유전자 knockout을 수행하거나 다른 knockdown 전략을 사용하는 것이 더 효과적일 수 있습니다.
이 프로토콜은 사용 가능한 많은 CRISPR 기반 후성유전체 편집기 중 두 가지인 CRISPRi 및 CRISPRoff에 중점을 둡니다. 최근의 대규모 발견 연구는 인간 후성유전체를 다시 쓰기 위한 새로운 도구를 개발했습니다 29,30,52. Epigenome 편집자는 생물의학 연구 및 치료학에 응용됩니다. 예를 들어, 최근 연구에서는 마우스 모델 및 비인간 영장류에서 DNA 메틸화 및 H3K9me3 기반 후성유전체 편집을 사용하여 질병 관련 유전자 53,54,55의 유전적 억제를 초래했습니다. 우리는 후성유전체 편집자를 위한 미래의 전달 방식이 후성유전체 편집의 광범위한 적용을 위한 새로운 길을 열 것으로 예상합니다.
공개
J.K.N.은 캘리포니아 대학교 리전트(The Regents of the University of California)가 출원한 CRISPRoff/on 기술과 관련된 특허의 발명자입니다.
감사의 말
Nuñez 연구실 구성원들, 특히 Rithu Pattali와 Izaiah Ornelas에게 이 원고에 설명된 프로토콜을 개발하고 최적화해 주신 것에 대해 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
참고문헌
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유