Method Article
Édition de l’épigénome CRISPR dans des cellules humaines à l’aide de la transfection de l’ADN plasmidique et de la nucléofection de l’ARNm
Dans cet article
Résumé
Le protocole décrit les méthodes d’édition de l’épigénome basée sur CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) dans des lignées cellulaires humaines à l’aide de la transfection de l’ADN plasmidique et de la nucléofection de l’ARNm.
Résumé
L’épigénétique fait référence aux modifications chimiques des protéines d’histones et de l’ADN qui peuvent réguler l’expression des gènes. L’épigénome humain est altéré dynamiquement au cours de la différenciation cellulaire et du vieillissement, et de nombreuses maladies sont associées à un motif épigénomique aberrant. Les progrès récents de CRISPR ont conduit au développement d’outils programmables pour modifier les modifications épigénétiques au niveau de loci génomiques ciblés, permettant une réécriture précise des modifications épigénétiques dans les cellules humaines. Les éditeurs d’épigénome basés sur CRISPR s’appuient sur Cas9 catalytiquement mort couplé à des modificateurs épigénétiques qui aboutissent finalement à la répression programmée ou à l’activation de gènes ciblés dans les génomes de mammifères. Contrairement aux méthodes traditionnelles d’édition du génome, l’édition de l’épigénome ne nécessite pas de cassures de l’ADN ou de modifications de la séquence du génome humain et constitue donc une alternative plus sûre pour contrôler l’expression des gènes. Dans ce protocole, nous mettons en évidence deux méthodes différentes pour effectuer l’édition de l’épigénome médiée par dCas9 dans des lignées cellulaires humaines en utilisant des transfections d’ADN plasmidique et la nucléofection d’ARNm codant pour des éditeurs d’épigénome CRISPR. Nous démontrons l’édition programmable de l’épigénome pour réprimer transitoirement les gènes à l’aide de l’interférence CRISPR (CRISPRi) et pour réduire au silence les gènes de manière durable pendant de nombreuses semaines à l’aide de CRISPRoff, une fusion de dCas9 au domaine KRAB et du complexe ADN méthyltransférase de novo . Nous fournissons également des conseils sur les méthodes quantitatives pour mesurer le succès de l’édition de l’épigénome des gènes cibles et des considérations clés sur l’outil d’édition de l’épigénome à utiliser, en fonction de critères expérimentaux.
Introduction
Bien que le contenu génomique de chaque cellule de notre corps soit presque identique, le profil transcriptionnel de chaque type de cellule diffère considérablement. Les modifications épigénétiques de l’ADN et des protéines d’histones sont des régulateurs clés de l’expression transcriptionnelle. L’euchromatine transcriptionnellement active est caractérisée par des marques épigénétiques distinctes par rapport à l’hétérochromatine compacte et transcriptionnellement inactive. Par exemple, les régions hétérochromatiques sont définies par des modifications répressives des histones, y compris la triméthylation sur la lysine 9 de l’histone 3 (H3K9me3), la triméthylation sur la lysine 27 de l’histone H3 (H3K27me3) et la méthylation de l’ADN sur les cytosines à côté des guanines (CpG) sur les promoteurs du gène1. Les régions génomiques d’expression génique active sont définies généralement par l’acétylation et la triméthylation des histones sur la lysine 4 de l’histone 3 (H3K4me3)1.
La révolution CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) a généré une multitude d’outils qui permettent la modification programmée des séquences génomiques. La technologie CRISPR est basée sur un mécanisme de défense procaryote capable de cliver les acides nucléiques à des séquences cibles programmables. Les nucléases CRISPR 2,3,4, les éditeurs de base 5,6 et les éditeurs premiers 7,8 peuvent modifier la séquence d’ADN des génomes de mammifères par la découpe de l’ADN et la réparation de ces cassures. Bien qu’efficaces, ces stratégies peuvent provoquer des cassures de l’ADN sur des sites hors cible 9,10 et des mutations structurelles génomiques à grande échelle 11,12,13,14,15. D’autres outils basés sur CRISPR permettent une modulation traitable de l’activation et de la répression des gènes sans modifier la séquence d’ADN sous-jacente. Ces outils exploitent un Cas9 déficient en nucléases (dCas9), permettant la liaison de l’ADN sur des sites cibles dictés par la séquence de l’ARNsg, en combinaison avec des protéines effectrices qui modifient le paysage chromatinique 16,17,18. Les protéines effectrices, telles que les rédacteurs épigénétiques, les lecteurs et les effaceurs, peuvent être directement fusionnées à dCas9 ou recrutées par un échafaudage peptidique fusionné à dCas9, tel que SunTag, ou un échafaudage d’ARN sur l’ARNsg, tel que le système MS2-MCP 16,17,18. Parmi les exemples d’outils de contrôle de transcription programmables, citons l’activation CRISPR (CRISPRa)19,20,21 et l’interférence CRISPR (CRISPRi)22,23. CRISPRa fonctionne en recrutant directement la machinerie de transcription, augmentant ainsi l’expression des gènes transcriptionnels cibles19. En revanche, CRISPRi réprime la transcription en établissant H3K9me3, une marque épigénétique répressive22.
Les progrès de l’édition de l’épigénome ont permis une large utilisation de ces outils dans tous les domaines scientifiques. Les fusions de différents domaines effecteurs et protéines ont élargi la boîte à outils des éditeurs d’épigénome disponibles 18,24,25,26,27,28,29,30. De plus, les éditeurs d’épigénome sont utilisés pour déchiffrer les rôles des modifications épigénétiques 31,32,33,34,35,36, des effecteurs 37,38,39 et des mutations effectrices 28,40,41 dans la régulation des gènes. Plus précisément, CRISPRi et CRISPRa sont utilisés dans les cribles génomiques fonctionnels pour une variété de processus biologiques, y compris la survie cellulaire42 et le destin cellulaire 43,44,45,46,47. De plus, l’édition de l’épigénome présente un potentiel thérapeutique pour l’ingénierie cellulaire ex vivo et les thérapies in vivo 18.
Ici, nous décrivons des méthodes d’application de deux éditeurs d’épigénome basés sur dCas9 pour la répression transcriptionnelle programmable dans des lignées cellulaires humaines : CRISPRi22 et CRISPRoff48. CRISPRi est une fusion de dCas9 avec le domaine répressif KRAB à partir d’une protéine à doigt de zinc telle que ZNF10 (KOX1) et ZIM3 22,23. Lorsque CRISPRi est ciblé sur un promoteur de gène spécifique, le domaine KRAB recrute un écrivain H3K9me3, SETDB1, pour réprimer le gène cible (Figure 1A). Lorsque CRISPRi est exprimé de manière transitoire, le H3K9me3 établi au locus cible n’est pas maintenu et l’expression génique est restaurée au fil du temps32,48. Pour effectuer des knockdowns stables à l’aide de CRISPRi, comme dans les applications de génomique fonctionnelle, l’expression constitutive de CRISPRi dans les cellules avec l’ARNsg est essentielle. Récemment, CRISPRoff a été conçu pour programmer l’édition héréditaire de l’épigénome48. CRISPRoff est une fusion protéique unique de dCas9 vers le domaine KRAB et le complexe ADN méthyltransférase de novo, DNMT3A et DNMT3L. Une impulsion transitoire de CRISPRoff dans les cellules humaines programme le dépôt de H3K9me3 et la méthylation de l’ADN au niveau des gènes ciblés, ce qui conduit à une répression à long terme des gènes cibles par le maintien de la méthylation de l’ADN et de H3K9me3 (Figure 1B)48. De plus, les modifications de l’épigénome peuvent être inversées. Par exemple, un gène qui est silencieux de manière stable par CRISPRoff peut être réactivé par TET1-dCas9 qui peut supprimer enzymatiquement les marques de méthylation de l’ADN au locicible 49.
Ce protocole détaillera deux méthodes d’administration pour l’expression transitoire des éditeurs d’épigénome : la transfection de l’ADN plasmidique et la nucléofection de l’ARNm. De plus, nous expliquons comment utiliser la cytométrie en flux pour évaluer l’efficacité de l’édition de l’épigénome au niveau de deux gènes endogènes, CLTA et CD55. Ces méthodes peuvent être adaptées et appliquées à d’autres expériences d’édition d’épigénome à l’aide d’éditeurs supplémentaires ou peuvent être utilisées pour cibler différents gènes.

Figure 1 : Schéma du mécanisme et du flux de travail d’édition de l’épigénome CRISPRi et CRISPRoff. (A) Schémas linéaires du transgène sgRNA et de l’éditeur d’épigénome CRISPRi. L’ajout de CRISPRi et d’ARNsg permet l’ajout de la marque d’histone répressive H3K9me3 pour faire taire le locus cible. Le niveau le plus élevé de silençage est atteint tôt après l’ajout de CRISPRi, et la cible du gène est généralement réactivée après quelques passages. (B) Schémas linéaires du transgène sgRNA et de l’éditeur d’épigénome CRISPRoff. L’ajout de CRISPRoff à des cellules ciblant un gène d’intérêt conduit à l’ajout de H3K9me3 répressif ainsi qu’à la méthylation de l’ADN aux sites CpG pour faire taire le gène cible. Le silençage par CRISPRoff est héréditaire - le niveau élevé de silençage est atteint tôt pendant la transfection et persiste sur plusieurs divisions cellulaires. (C) Vue d’ensemble de la chronologie de l’édition de l’épigénome par la méthode de transfection. Le jour 0, les cellules sont plaquées pour la transfection. Le jour 1, l’éditeur et le plasmide guide peuvent être introduits dans les cellules par transfection. Le jour 3, l’expression de BFP dans les cellules sera évaluée par cytométrie en flux. Le pourcentage de BFP est utilisé comme facteur de normalisation pour déterminer l’efficacité finale de l’expérience pour chaque condition. À partir du jour 6, les cellules sont analysées pour faire taire le rapporteur d’intérêt, car le niveau de silence le plus élevé sera atteint ce jour-là. (D) Vue d’ensemble de la méthode de transfection dans laquelle l’éditeur d’épigénome et les plasmides d’ARNsg sont ajoutés aux cellules de manière goutte à goutte. (E) Vue d’ensemble de la chronologie de l’édition de l’épigénome via les méthodes de nucléofection. Dans ce protocole, l’ARNm est nucléofection dans les cellules le jour 0. Les cellules sont évaluées pour le silençage le 3e jour post-nucléofection à l’aide d’une analyse par cytométrie en flux. (F) Vue d’ensemble du protocole de nucléofection. Des quantités appropriées de cellules et d’ARNm sont mélangées et ajoutées dans les cuvettes nucléofectrices. Si l’ARNsg est introduit par nucléofection, il peut également être ajouté à ce mélange. Les cuvettes sont placées dans le nucléofecteur, et des codes d’impulsions appropriés sont utilisés pour introduire l’ARNm dans les cellules. Après la nucléofection, les cellules sont plaquées et passées pour une analyse ultérieure. (G) Comparaison entre les stratégies de transfection de plasmide et de nucléofection d’ARNm pour l’édition de l’épigénome. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
REMARQUE : Le fichier supplémentaire 1 contient des détails sur la conception de l’ARNsg, le clonage et la génération de lignées cellulaires de nos données représentatives. La section des résultats représentatifs détaille également les suggestions de contrôles.
1. Transfection de plasmides exprimant l’éditeur d’épigénome dans des cellules HEK293T
REMARQUE : Ce protocole décrit l’administration de plasmides codant pour CRISPR dans HEK293T cellules. Nous avons modifié des cellules pour exprimer un ARNsg (Addgene 217306) ciblant la région promotrice de CLTA, un gène non essentiel, qui est marqué de manière endogène avec la GFP. Les cellules CLTA-GFP HEK 293T proviennent d’une étude précédente50. Dans cet exemple, les éditeurs d’épigénome sont fusionnés directement à une protéine fluorescente bleue (BFP), ce qui nous permet de quantifier l’efficacité de la transfection et d’assurer une évaluation précise des conditions expérimentales. L’efficacité de l’approche est démontrée par le silençage de CLTA-GFP, qui peut être mesuré quantitativement au niveau protéique dans des cellules uniques à l’aide de la cytométrie en flux.
- Culture cellulaire
- Maintenez HEK293T cellules dans un DMEM à haute teneur en glucose avec 10 % de FBS et 1x pénicilline-streptomycine-glutamine. Passez les cellules tous les 2-3 jours, en veillant à ce qu’elles restent à une confluence de 60 % à 70 %.
REMARQUE : Pour garantir une efficacité de transfection optimale, il est essentiel de maintenir la confluence cellulaire en dessous de 70 %, qui peut être surveillée quotidiennement au microscope.
- Maintenez HEK293T cellules dans un DMEM à haute teneur en glucose avec 10 % de FBS et 1x pénicilline-streptomycine-glutamine. Passez les cellules tous les 2-3 jours, en veillant à ce qu’elles restent à une confluence de 60 % à 70 %.
- Jour 0 : Ensemencement HEK293T cellules pour la transfection
- La veille de la transfection, compter les cellules à l’aide d’un compteur de cellules automatisé à l’aide d’une coloration au bleu trypan vivant/mort et de graines ~1,5 x 104 cellules vivantes par puits dans une plaque de 96 puits. Maintenez le volume final de chaque puits à 200 μL. Les cellules devraient atteindre une confluence de 60 % à 70 % le lendemain.
REMARQUE : Cela peut également être effectué efficacement dans des plaques à 24 ou 6 puits. Voir le tableau 1 pour le nombre de cellules et la quantité d’ADN pour la transfection.
- La veille de la transfection, compter les cellules à l’aide d’un compteur de cellules automatisé à l’aide d’une coloration au bleu trypan vivant/mort et de graines ~1,5 x 104 cellules vivantes par puits dans une plaque de 96 puits. Maintenez le volume final de chaque puits à 200 μL. Les cellules devraient atteindre une confluence de 60 % à 70 % le lendemain.
- Jour 1 : Transfection des plasmides CRISPRoff et CRISPRi dans les cellules HEK29T
- Aliquote 150 ng du plasmide de l’éditeur d’épigénome (Addgene 167981) pour chaque puits dans un volume total de 3 μL. Ces dilutions plasmidiques peuvent être préparées dans des tubes de bandelettes PCR.
REMARQUE : Si l’éditeur d’épigénome et le plasmide sgRNA sont co-transfectés, nous vous recommandons d’essayer un rapport molaire de 3:4 des plasmides editor :sgRNA. Il peut être nécessaire d’optimiser le rapport pour obtenir la plus grande efficacité de l’édition de l’épigénome. - Préchauffez le réactif de transfection et le milieu essentiel minimal à température ambiante environ 30 à 60 minutes avant la transfection. Mélanger 0,6 μL de réactif de transfection et 20 μL de milieu essentiel minimal par puits et incuber à température ambiante pendant 15 min.
REMARQUE : Un mélange principal de réactif de transfection et de milieu essentiel minimal peut être réalisé si vous effectuez plusieurs transfections. - Ajouter 20,6 μL du réactif de transfection et un mélange minimal de milieu essentiel dans chaque tube de bandelette PCR contenant 3 μL d’ADN plasmidique. Laisser reposer à température ambiante pendant 15 min.
- Ajoutez lentement le mélange à la plaque de cellules ensemencées et tapotez doucement la plaque pour disperser le mélange. Inclure un témoin non transfecté afin de fournir une base de référence appropriée pour la comparaison dans les analyses en aval.
REMARQUE : Lors de l’ajout du mélange de transfection dans les cellules ensemencées, il est essentiel de le faire goutte à goutte et doucement pour minimiser les perturbations, car cela peut nuire à l’efficacité de la transfection.
- Aliquote 150 ng du plasmide de l’éditeur d’épigénome (Addgene 167981) pour chaque puits dans un volume total de 3 μL. Ces dilutions plasmidiques peuvent être préparées dans des tubes de bandelettes PCR.
- Jour 2 : Vérifier le succès de la transfection
- Examinez les cellules au microscope à un grossissement de 10X pour déterminer si elles sont positives au BFP, car cela indique une transfection réussie. Assurez-vous que les cellules ne sont pas perturbées pendant le processus d’observation.
- Jour 3 : Fendre la plaque à 96 puits et évaluer l’efficacité de la transfection à l’aide du marqueur BFP
REMARQUE : Une transfection réussie devrait donner au moins 30 % de cellules BFP positives. Cependant, avec dCas9, CRISPRi et CRISPRoff, nous détectons généralement plus de 60 % de cellules BFP positives deux jours après la transfection. L’efficacité de la transfection est négativement corrélée à la taille du plasmide, de sorte que différents éditeurs d’épigénome peuvent produire des efficacités de transfection différentes.- Préchauffer la trypsine et le DMEM avec 10 % de FBS et 1 fois pénicilline-streptomycine-glutamine à température ambiante pendant environ 30 à 60 minutes avant l’utilisation.
- Aspirer le milieu des puits ; Attention à ne pas retirer les cellules attachées au fond. Ajouter 50 μL de trypsine par puits. Incuber pendant 2-3 min à 37 °C.
REMARQUE : Il n’est pas rare d’observer des cellules flottantes, en particulier après une transfection avec CRISPRoff. Nous émettons l’hypothèse que cette mort cellulaire est due à la toxicité de la surexpression de DNMT3A-3L. - Inhiber la trypsine en ajoutant 150 μL de DMEM complet par puits. Pipette de haut en bas pour remettre les cellules en suspension et diviser les cellules pour la confluence le jour 5 (~20 μL de mélange cellulaire dans 200 μL de milieu d’une plaque de 96 puits).
- Utilisez 50 μL de cellules remises en suspension pour la cytométrie en flux afin d’évaluer l’efficacité de la transfection.
2. Nucléofection de l’ARNm de l’éditeur d’épigénome dans les cellules K-562
REMARQUE : Cette section détaille le processus de nucléofectation de l’ARNm CRISPRoff dans les cellules K-562. Pour simplifier, nous avons pré-conçu les cellules K-562 pour exprimer constitutivement un ARNsg qui cible le promoteur du gène endogène CD55 (Addgene 217306). L’administration de l’ARNm CRISPRoff directement aux cellules a le potentiel de réduire la toxicité cellulaire qui accompagne les approches basées sur l’ADN plasmidique tout en obtenant une efficacité similaire de silençage génique. De plus, la nucléofection peut être utilisée pour introduire des constructions d’éditeur d’épigénome dans des lignées cellulaires difficiles à transfecter efficacement, telles que les K-562.
- Culture cellulaire
- Avant la nucléofection, maintenir les cellules K-562 dans un flacon avec RPMI avec 10 % de FBS et 1x pénicilline-streptomycine-glutamine. Passez les cellules tous les jours, en veillant à ce qu’elles restent à une confluence de 60 % à 70 %.
- Le jour de la nucléofection, décongelez et vortex doucement l’ARNm CRISPRoff dans un tube de microcentrifugation stérile sans RNase et stockez l’ARNm sur de la glace. Utiliser 2 μg d’ARNm par 2,0 x 105 cellules dans chaque puits d’une nucléocuvette en bandelettes. S’assurer que le volume d’ARNm ne dépasse pas 10 % du volume total de nucléofection (22 μL).
REMARQUE : La nucléofection peut également être réalisée dans des cuvettes plus grandes avec plus de cellules et d’ARNm. Nous vous suggérons d’augmenter la quantité d’ARNm proportionnellement au nombre de cellules que vous allez ajouter. Par exemple, lors de la réalisation d’une nucléofection dans une grande cuvette sur 1 x 106 cellules, nous suggérons d’utiliser 10 μg d’ARNm, avec un volume total de nucléofection de 100 μL. De plus, le dosage de l’ARNm peut être optimisé pour garantir une efficacité d’édition élevée. Nous utilisons souvent 2 à 5 μg d’ARNm éditeur pour chaque 2,0 x 105 cellules pour des résultats efficaces. Les nucléotides libres qui n’ont pas été incorporés lors de la synthèse de l’ARNm doivent être éliminés avant la quantification. - Préparez la solution de nucléofection selon les instructions du fabricant et réchauffez-la à température ambiante pendant 15 minutes avant la nucléofection.
REMARQUE : La solution nucléofectrice peut être différente ou doit être optimisée pour différents types de cellules. - Récoltez et comptez les cellules K-562 à l’aide d’un compteur de cellules automatisé avec coloration au bleu trypan vivant/mort. Pour la nucléofection dans une cuvette à bandelettes, aliquote ~2,0 x 105 cellules par échantillon dans un tube de microcentrifugation stérile.
- Centrifugez les cellules à 500 x g pendant 5 min à température ambiante, puis jetez le surnageant. Lavez les cellules 1x avec du PBS à température ambiante à 500 x g pendant 5 min, puis jetez le surnageant.
- Calculez la quantité de solution de nucléofecteur dans laquelle remettre les cellules en suspension en soustrayant le volume d’ARNm de 22 μL. Remettre les cellules en suspension dans la quantité appropriée de solution de nucléofecteur.
- Ajouter la solution cellulaire à 2 μg d’ARNm CRISPRoff (volume déterminé à l’étape 2.1.2) et transférer la solution dans une cuvette en prenant soin de ne pas former de bulles, car cela pourrait compromettre l’efficacité de la nucléofection. Tapotez doucement la nucléocuvette pour vous assurer que les cellules sont au fond.
- Nucleofect les cellules à l’aide du système 4D-Nucleofector avec le code d’impulsion approprié. Le code FF-120 est recommandé pour les cellules K-562.
REMARQUE : Le code d’impulsion peut avoir besoin d’être optimisé pour différents types de cellules. Consultez le fabricant si une optimisation supplémentaire est nécessaire. - Après la nucléification, ajouter 80 μL de milieu RPMI dans chaque petit puits de nucléocuvette et laisser les cellules reposer à 37 °C pendant 15 min.
- Transférez la suspension cellulaire dans un puits d’une plaque à 24 puits contenant 400 μL de milieu RPMI préchauffé (petite nucléocuvette).
REMARQUE : Après la nucléofection, les cellules sont particulièrement fragiles et doivent être transférées doucement de la nucléocuvette aux puits de culture. - Après le jour 2 post-nucléofection : suivre le protocole de coloration par marqueur de surface avec l’anticorps CD55 tel que décrit dans la section 3 et mesurer la fluorescence de l’APC CD55 sur un cytomètre en flux pour tester l’efficacité de l’édition de l’épigénome.
REMARQUE : Pour tester le succès de la nucléofection, nous vous recommandons d’effectuer une nucléofection avec GFP ou mCherry mRNA. Deux jours après la nucléofection, effectuez une cytométrie en flux de ces cellules pour confirmer la réussite de la nucléofection. Nous détectons généralement 90 % à 100 % de ces cellules exprimant la GFP ou la mCherry. Une alternative consiste à effectuer une nucléofection avec l’ARNm CRISPRi, qui est connu pour réduire au silence de nombreux gènes cibles. Lors de la cytométrie en flux le jour 2, les échantillons traités par CRISPRi devraient montrer un silençage génique cible dans plus de 90 % des cellules.
3. Coloration au marqueur de surface
REMARQUE : Cette section détaille la quantification des niveaux de protéine CD55 après l’édition de l’épigénome dans les cellules K-562. Nous quantifions la diminution de l’expression de CD55 dans les cellules uniques à l’aide de la coloration des anticorps et de la cytométrie en flux (voir section 4 ci-dessous) pour évaluer l’efficacité de l’inactivation médiée par CRISPRoff. D’autres techniques, notamment la transcription inverse, la PCR quantitative ou le western blot, peuvent également être utilisées pour confirmer le niveau d’inactivation à la fois au niveau de la transcription et de la protéine.
- Comptez les cellules à l’aide d’un compteur de cellules automatisé et ajoutez environ 5,0 x 105 à 1,0 x 106 cellules dans un tube de microcentrifugation et des cellules de centrifugation à 500 x g pendant 5 min.
REMARQUE : Plus ou moins de cellules peuvent être utilisées pour la coloration, mais la concentration d’anticorps peut devoir être mise à l’échelle en conséquence. - Jetez le surnageant en veillant à ne pas déranger la pastille et ajoutez 500 μL de PBS et remettez la pastille en suspension doucement.
- Centrifugez à nouveau les cellules à 500 x g pendant 5 min. Jetez le surnageant et ajoutez l’anticorps à la concentration souhaitée dilué dans du PBS. Pour la coloration au CD55, utilisez 1 μL de CD55 anti-humain purifié (0,5 mg/mL stock) dans 49 μL de PBS.
- Pipetez doucement les cellules et laissez-les incuber pendant 25 minutes dans un endroit sombre à température ambiante. Après l’incubation, faites tourner à nouveau les cellules à 500 x g pendant 5 min et jetez le surnageant.
- Remettez la pastille en suspension avec 500 μL de PBS pour éliminer l’excès d’anticorps. Centrifugez les cellules à 500 x g pendant 5 min et jetez le surnageant PBS. Remettre en suspension dans 100 à 200 μL de PBS frais et l’ajouter à une plaque à 96 puits pour l’analyse par cytométrie en flux.
4. Cytométrie en flux
REMARQUE : Ce protocole est écrit pour l’utilisation d’un analyseur de cellules BD FACSymphony A1. Les spécificités peuvent varier en fonction du cytomètre en flux que vous utilisez. Nous vous suggérons de vous référer au manuel d’utilisation de la machine que vous utilisez pour plus de détails.
- Avant d’allumer la cytométrie, vérifiez que le conteneur à déchets n’est pas plein et qu’il y a beaucoup de liquide de gaine. Allumez l’interrupteur d’alimentation principal (vert) qui se trouve près du liquide de gaine.
- Allumez le lecteur de plaques à l’aide de l’interrupteur arrière. Ensuite, allumez la machine à l’aide du bouton vert sur le côté droit et assurez-vous que la machine est réglée sur la fonction de lecteur de plaques à 96 puits.
- Allumez l’ordinateur et connectez-vous. Ouvrez le logiciel et connectez-vous à l’aide de votre nom d’utilisateur et de votre mot de passe.
- Configurez l’expérience. Créez une nouvelle expérience en sélectionnant Expérience > Nouvelle expérience. Renommez l’expérience en double-cliquant sur le nom actuel.
- Ajoutez une plaque en cliquant sur Tester > Nouvelle plaque. Sélectionnez le format de puits à utiliser. Pour ajouter des puits spécifiques à exécuter, cliquez sur l’emplacement du puits dans la plaque, puis cliquez sur la seringue bleue (le puits doit maintenant être mis en surbrillance en bleu). Sélectionnez plusieurs puits en même temps à ajouter, si nécessaire.
- Ajoutez des noms spécifiques à chaque puits en cliquant avec le bouton droit de la souris sur Spécimen à droite de l’affichage de la plaque. Sélectionnez ensuite Mise en page expérimentale. Cela ouvrira une page répertoriant tous les puits que vous avez sélectionnés. Pour renommer les puits, cliquez une fois et saisissez le nom souhaité. Cliquez sur OK lorsque vous avez terminé.
- Ajoutez des graphiques et des portes appropriées à l’expérience sur la feuille de calcul globale. Nous vous recommandons de tracer d’abord la zone de diffusion vers l’avant (FSC-A) par rapport à la zone de diffusion latérale (SSC-A). Créez ensuite une porte arbitraire pour les cellules vivantes à affiner ultérieurement en cliquant sur l’outil Polygone.
- Faites un clic droit sur cette porte et cliquez sur Drill Down. Cela créera un nouveau graphique affichant uniquement les cellules de cette porte de cellule active.
- Changez l’axe de ce nouveau graphique en FSC-A en fonction de la hauteur de diffusion vers l’avant (FSC-H) et créez une porte pour les cellules individuelles.
- Si l’expression constitutive de l’ARNsg dans les cellules et que le lentivirus code également pour une protéine fluorescente, alors creusez cette porte cellulaire unique et effectuez d’autres contrôles pour l’expression de cette protéine en tant qu’indicateur de l’expression de l’ARNsg (exemple : PE-CF594-A vs FSC-A).
- Sur la dernière porte de configuration (pour les cellules uniques ou pour les cellules exprimant l’ARNsg), cliquez avec le bouton droit de la souris et sélectionnez Drill Down. Créez une porte pour tester l’expression du gène rapporteur (exemple : BB515-A vs FSC-A).
- Si vous utilisez des cellules transfectées et que vous disposez d’une méthode pour mesurer l’efficacité de la transfection telle que l’expression BFP, cliquez avec le bouton droit de la souris sur le graphique d’expression du rapporteur et cliquez sur Dupliquer. Changez les axes en BV421-A vs FSC-A. Créez une porte arbitraire pour les cellules BFP positives.
- Supprimez tous les paramètres qui ne sont pas utilisés dans l’onglet Paramètres de l’affichage du cytomètre.
- Pour configurer l’appareil et le préparer à fonctionner, cliquez sur Exécuter sur le panneau de commande du cytomètre en flux. Le bouton doit apparaître en vert. Dans le logiciel, allez dans HTS dans le panneau supérieur puis Prime. Une fois terminé, ajoutez la plaque en vous assurant qu’elle est bien positionnée dans la machine et que le couvercle est OFF.
- Pour l’exécuter, sélectionnez tous les puits à exécuter et assurez-vous que les commandes sont correctement définies. Sélectionnez bien en double-cliquant, modifiez le volume de l’échantillon à 10 μL et cliquez sur Acquérir des données dans le tableau de bord d’acquisition. Cela exécutera l’échantillon, mais n’enregistrera pas les données. Pendant que cet échantillon est en cours d’exécution, ajustez les tensions laser si nécessaire. De plus, changez les portes.
REMARQUE : Nous vous recommandons d’analyser un échantillon négatif pour toutes les couleurs mesurées et un échantillon positif pour toutes les couleurs afin de vous assurer que les paramètres laser et le déclenchement sont bons. - Après avoir vérifié les paramètres du laser, cliquez sur les échantillons dans le panneau de plaque et vérifiez que les valeurs de fonctionnement sont réglées comme vous le souhaitez.
- Cliquez sur Exécuter la plaque pour l’exécuter dans le tableau de bord d’acquisition et enregistrer les données de tous les puits. Une fois terminé, nettoyez la machine en retirant la plaque.
- Pour une nouvelle plaque à 96 puits, ajoutez 10 % d’eau de Javel dans les quatre rangées supérieures et de H2O dans les quatre puits inférieurs. Allez dans HTS > Clean. Une fois terminé, fermez le programme et éteignez les boutons dans l’ordre inverse de celui dans lequel ils ont été allumés bouton latéral vert > lecteur de plaques >interrupteur vert par déchets.
- Ajoutez 20 % d’eau de Javel dans la plaque à 96 puits avec les cellules et jetez-la.
5. Analyse des données
REMARQUE : Cette méthode décrit les stratégies de contrôle et le traitement des données pour quantifier l’édition de l’épigénome par cytométrie en flux. La stratégie de contrôle est représentée visuellement à côté d’exemples de graphiques générés à partir de l’analyse des données dans les figures 2 et 3.
- Configuration de la porte FlowJo
- Chargez tous les fichiers FSC sur FlowJo en les glissant et en les déposant dans une nouvelle feuille de calcul.
- Cliquez sur Tous les échantillons pour commencer à créer des portes. Cela garantira que les portes peuvent facilement être appliquées à tous les échantillons.
- Ouvrez un échantillon de contrôle (tel que non transfecté ou non nucléofecté) en double-cliquant sur ce fichier.
- Créez une porte pour les cellules vivantes dans un tracé FSC-A ou SSC-A à l’aide de l’outil polygone. Dans la liste de tous les échantillons, un onglet Cellules actives doit maintenant apparaître sous le nom de l’échantillon. Cliquez avec le bouton droit de la souris sur ce contrôle et sélectionnez Copier l’analyse dans le groupe pour appliquer ce contrôle à tous les échantillons (Figure 2A, Figure 3A).
- Double-cliquez sur la porte des cellules actives pour explorer les cellules actives. Remplacez les axes par FSC-A ou FSC-H (Figure 2B, Figure 3B).
- Dessinez un polygone à contrôler pour des cellules uniques uniquement. Appliquez cette porte à tous les échantillons comme indiqué à la section 4 (figure 2B, figure 3B).
REMARQUE : Si vous exprimez constitutivement une construction d’ARNsg avec un rapporteur fluorescent, effectuez un gating supplémentaire sur cette couleur afin que l’analyse se concentre uniquement sur les cellules qui expriment l’ARNsg. Par exemple, la création d’une porte pour les cellules mCherry positives (PE-CF594-A vs FSC-A) si les cellules contiennent un ARNsg provenant du squelette pLG1 (Figure 2C, Figure 3C). - Double-cliquez sur la dernière porte de configuration (cellules simples ou cellules d’expression guide).
- Si vous effectuez une transfection plasmidique et que l’éditeur d’épigénome code pour une fusion BFP, créez une porte pour les cellules BFP positives (exemple : BV421-A vs FSC-A) pour les échantillons du jour 2 (Figure 2D).
- Créez une porte pour l’expression rapporteuse (exemple : BB515-A ou FSC-A pour la porte pour les cellules négatives GFP) et appliquez cette porte à tous les échantillons (Figure 2E, Figure 3D).
- Analyse de transfection
- Après avoir effectué la configuration du gate, cliquez sur Éditeur de table dans le panneau supérieur. Faites glisser les populations pour l’analyse (exemples : BFP positif, GFP négatif).
- Dans le panneau de l’éditeur de tableaux, cliquez sur Créer un tableau. Copiez ces données dans une feuille de calcul pour effectuer des calculs de normalisation.
- Traitement des données
- Soustrayez le nombre de cellules rapporteures négatives (par exemple : GFP négative) dans l’échantillon de contrôle de tous les autres échantillons. Il peut y avoir un certain silence de fond du rapporteur, ce qui supprime ce bruit de fond des données.
- Toutes les valeurs seront normalisées en fonction du pourcentage de cellules BFP positives (cellules transfectées avec succès) mesuré le jour 2. Pour effectuer cette normalisation, prenez la valeur, divisez par le pourcentage de cellules BFP positives au jour 2 et multipliez par 100. Ce calcul donne la valeur normalisée de l’efficacité de transfection des cellules modifiées. Les données sont maintenant prêtes à être tracées. Nous recommandons des graphiques linéaires sur l’ensemble du temps de l’édition de l’épigénome, comme le montre la figure 2F.
- Analyse de nucléofection
- Après avoir effectué la configuration du gate, cliquez sur Éditeur de table dans le panneau supérieur. Faites glisser les populations pour l’analyse (exemples : APC négatif).
- Dans le panneau de l’éditeur de tableaux, cliquez sur Créer un tableau. Copiez ces données dans un tableur pour effectuer des calculs de normalisation.
- Traitement des données
- Soustrayez le nombre de cellules rapporteures négatives (par exemple : APC négatives) dans l’échantillon de contrôle de tous les autres échantillons. Il peut y avoir un certain silence de fond du rapporteur, ce qui supprime ce bruit de fond des données. Les données sont maintenant prêtes à être tracées. Nous recommandons des graphiques linéaires tout au long d’une édition de l’épigénome.
REMARQUE : Les statistiques peuvent être utilisées pour quantifier les différences de silençage entre deux éditeurs d’épigénome différents. Nous recommandons d’effectuer toutes les expériences de transfection et de nucléofection en triplets techniques pour effectuer des analyses statistiques. Lorsque vous illustrez des graphiques linéaires de réduction au silence au fil du temps, tracez l’écart-type des répétitions techniques. Des tests t d’étudiants à des moments précis pourraient être effectués pour comparer le silençage entre deux éditeurs d’épigénome. Les échantillons doivent être traités comme non appariés avec une variance inégale.
- Soustrayez le nombre de cellules rapporteures négatives (par exemple : APC négatives) dans l’échantillon de contrôle de tous les autres échantillons. Il peut y avoir un certain silence de fond du rapporteur, ce qui supprime ce bruit de fond des données. Les données sont maintenant prêtes à être tracées. Nous recommandons des graphiques linéaires tout au long d’une édition de l’épigénome.
Résultats
Pour toutes les expériences d’édition de l’épigénome, des contrôles appropriés sont essentiels pour évaluer l’efficacité de l’édition de l’épigénome. Nous recommandons d’utiliser un ARNsg de contrôle, qui ne cible aucune séquence du génome humain. L’utilisation d’un contrôle guide non ciblé donnera l’assurance que les changements au niveau des loci cibles sont entraînés par l’éditeur d’épigénome dirigé vers ce site plutôt que par une surexpression de l’éditeur d’épigénome ou une liaison non spécifique. De plus, pour les expériences basées sur le gène rapporteur, nous suggérons d’utiliser un contrôle dCas9 uniquement pour s’assurer que les changements dans l’expression du rapporteur sont dus à des fusions d’éditeurs d’épigénome plutôt qu’à une entrave stérique de la liaison du dCas9 au locus cible et à un obstacle temporaire à la transcription (Figure 2F).
Pour les expériences de transfection, nous recommandons d’utiliser un éditeur d’épigénome avec une fusion de protéines fluorescentes supplémentaire, telle que BFP. Cette fusion permet de visualiser les cellules transfectées avec succès avec l’éditeur d’épigénome par microscopie et cytométrie en flux. Les cellules transfectées avec succès exprimeront des niveaux élevés de BFP deux jours après la transfection (Figure 2D). La quantification des cellules transduites avec succès est utilisée pour normaliser l’efficacité de l’édition de l’épigénome dans les jours ultérieurs (Figure 2F).
CRISPRoff et CRISPRi montrent tous deux un pic de silençage au jour 5 après la transfection (Figure 2E-F). Différents éditeurs d’épigénome ont des chronologies distinctes d’édition de l’épigénome, telles que le silençage héréditaire avec CRISPRoff et le silençage transitoire avec CRISPRi (Figure 2F). La figure 2F montre également l’utilisation de dCas9 uniquement comme contrôle important pour les expériences d’édition de l’épigénome. Dans les expériences de nucléofection de l’ARNm, les cellules modifiées avec succès avec CRISPRoff montreront un fort silençage du gène cible au jour 3 après la nucléofection (Figure 3E-F).
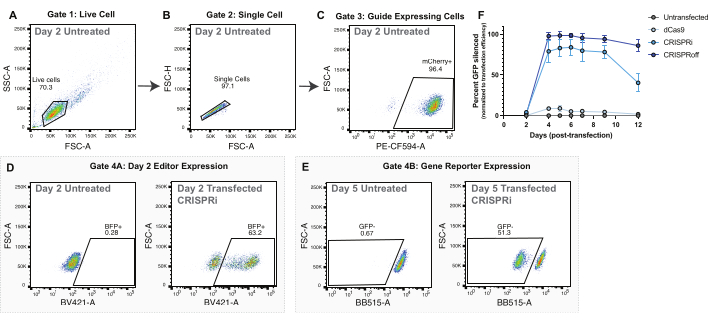
Figure 2 : Stratégie de déclenchement et données représentatives pour l’administration de l’éditeur d’épigénome par transfection de plasmide. (A-C) Tracés d’écoulement représentatifs pour afficher la stratégie de déclenchement pour les expériences de transfection de plasmides. Les graphiques d’écoulement affichés sont ceux de cellules non transfectées 2 jours après la transfection. Chaque point des graphiques représente une cellule. (A) Tracé de l’aire de diffusion vers l’avant (FSC-A) et de l’aire de diffusion latérale (SSC-A) avec une porte pour les cellules vivantes. (B) Tracé d’écoulement de cellules vivantes affichant la FSC-A et la hauteur de diffusion vers l’avant (FSC-H) avec une porte pour les cellules individuelles. (C) Tracé d’écoulement de cellules uniques représentant PE-CF594-A (expression de mCherry) et FSC-A. Gating pour les cellules mCherry positives en tant qu’indicateur de l’expression de l’ARNsg. (D) Stratégie de contrôle représentative de l’expression de l’éditeur d’épigénome (BFP+) au jour 2 post-transfection. La population parente est constituée de cellules guides exprimant des cellules contrôlées en (C). (E) Stratégie de contrôle représentative pour le dosage de l’inactivation du gène rapporteur CLTA-GFP. La population parentale est constituée de cellules guides exprimant des cellules contrôlées dans le panel C. (F) Silençage de CLTA-GFP pendant les jours suivant la transfection après l’administration plasmidique de dCas9, CRISPRi et CRISPRoff. Le pourcentage de CLTA-GFP silencieux est normalisé en fonction de l’efficacité de la transfection mesurée par des cellules BFP positives 2 jours après la transfection. Les points sont des moyennes de quatre répétitions de transfection. Les barres d’erreur représentent l’écart type. Veuillez cliquer ici pour voir une version agrandie de cette figure.
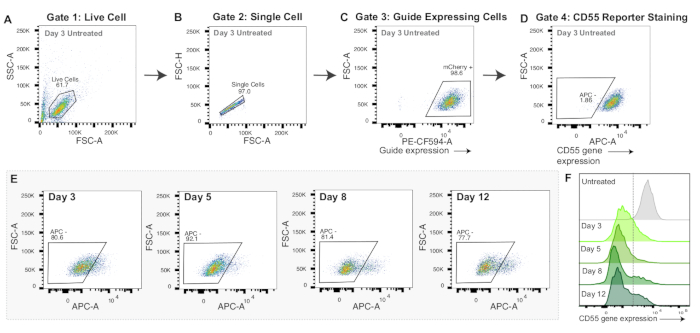
Figure 3 : Stratégie de déclenchement et données représentatives pour la nucléofection de l’ARNm CRISPRoff. (A-D) Graphiques de flux représentatifs pour afficher la stratégie de déclenchement pour les expériences de nucléofection de l’ARNm. Les diagrammes d’écoulement affichés sont ceux de cellules témoins 3 jours après la nucléofection colorées avec l’anticorps anti-humain CD55 APC. Chaque point des graphiques représente une cellule. (A) Tracé de l’intensité de la zone de diffusion vers l’avant (FSC-A) et de l’intensité de la zone de diffusion latérale (SSC-A) avec une porte pour les cellules vivantes. (B) Tracé d’écoulement de cellules vivantes affichant la FSC-A et l’intensité de la hauteur de diffusion vers l’avant (FSC-H) avec une porte pour les cellules individuelles. (C) Tracé d’écoulement de cellules uniques représentant PE-CF594-A (expression de mCherry) et FSC-A. Gating pour les cellules mCherry positives en tant qu’indicateur de l’expression de l’ARNsg. (D) Schéma d’écoulement de l’APC-A par rapport au FSC-A. Porte dessinée pour les cellules APC négatives, ce qui indiquerait le silençage du gène rapporteur CD55. (E) Tracés d’écoulement de l’inactivation de CD55 (APC - gate) sur 3, 5, 8 et 12 jours après la nucléofection avec l’ARNm CRISPRoff. (F) Couches d’histogrammes de l’expression de la protéine CD55 (APC-A) sur 3, 5, 8 et 12 jours après la nucléofection avec l’ARNm CRISPRoff par rapport à un témoin coloré mais non nucléofecté. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Assiette | Densité d’ensemencement (cellules par puits) | Quantité de plasmide |
| 96 puits | 15,000 | 150 ng |
| 24 puits | 90,000 | 500 ng |
| 6 puits | 400,000 | 2 μg |
Tableau 1 : Quantités d’échelle de transfection. Les densités d’ensemencement et les quantités d’ADN plasmidique pour la transfection de l’éditeur d’épigénome dans HEK293T cellules à différentes échelles.
Tableau supplémentaire 1 : Séquences d’espacement des ARNsg pour les expériences d’édition de l’épigénome. séquences d’ARNsg pour cibler les éditeurs d’épigénome de CLTA et CD55 ainsi qu’une séquence pour un guide de contrôle non ciblant. De plus, les oligos pour le clonage dans le squelette pLG1 sont répertoriés. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 1. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Ce protocole détaille deux méthodes différentes d’administration transitoire pour les éditeurs d’épigénome CRISPR : la transfection de l’ADN plasmidique et la nucléofection de l’ARNm. Les deux techniques présentent des avantages, des inconvénients et des considérations générales uniques (Figure 1F).
La transfection de l’ADN plasmidique conduit à une expression robuste de l’éditeur d’épigénome, et nous incluons une fusion BFP dans les constructions de l’éditeur d’épigénome qui permet la détection et la quantification de l’efficacité de la transfection à l’aide de la cytométrie en flux. De plus, l’expression de BFP peut être utilisée pour trier les cellules qui expriment l’éditeur d’épigénome ou peut être quantifiée pour normaliser les données de silence à des points temporels ultérieurs, comme détaillé dans ce protocole. Cependant, il est important de noter que l’efficacité de transfection n’est généralement pas de 100 %, et donc, la population sera hétérogène pour ceux qui ont reçu l’éditeur et ceux qui ne l’ont pas reçu, à moins que les cellules ne soient triées. De plus, l’administration d’ADN plasmidique peut déclencher une réponse immunitaire à partir de l’ADN cytoplasmique double brin, conduisant à l’activation de la voie immunitaire et à la cytotoxicité. Enfin, la transfection de l’ADN plasmidique ne convient pas à tous les types de cellules, et la nucléofection du plasmide peut être nécessaire.
Contrairement à la transfection de l’ADN plasmidique, la nucléofection de l’ARNm est applicable à de nombreux types de cellules et entraîne souvent une efficacité d’administration élevée. Les ARNm de l’éditeur peuvent être synthétisés à l’aide de kits de synthèse d’ARNm in vitro disponibles dans le commerce, dont l’un a été détaillé précédemment51. Alternativement, les ARNm peuvent être synthétisés à partir d’Aldevron ou de Trilink. Cependant, une mise en garde de la nucléofection de l’ARNm est qu’elle ne produit pas suffisamment de protéines d’édition pour déterminer quelles cellules ont reçu les éditeurs d’épigénome. Les expériences ci-dessus suggèrent que presque toutes les cellules ont reçu l’ARNm alors que nous détectons ~90 % du silençage du gène cible au jour 5 (Figure 3F). Cependant, nous recommandons d’optimiser les codes d’impulsions et le rapport ARNm/cellule pour chaque type de cellule.
Une autre considération est que l’édition de l’épigénome initiée par la transfection de l’ADN plasmidique ou la nucléofection de l’ARNm a des chronologies de silençage différentes. Avec la transfection de l’ADN plasmidique, un silençage maximal avec CRISPRoff et CRISPRi est observé le 5e jour après la transfection (Figure 2F). En comparaison, le silençage maximal avec CRISPRoff et CRISPRi délivré par la nucléofection de l’ARNm est observé dès le 3e jour après la nucléofection (Figure 3F). De plus, l’ADN plasmidique persiste dans les cellules par rapport à l’ARNm, ce qui entraîne une durée d’expression plus longue de l’éditeur. Selon les objectifs expérimentaux, une chronologie de réduction au silence et une durée d’expression plus longues ou plus courtes peuvent être préférées.
Il est également important de noter que l’édition de l’épigénome ne peut pas être universellement appliquée à tous les gènes ou types de cellules. Des différences inhérentes dans la séquence du génome ou les états de la chromatine des gènes cibles peuvent avoir un impact sur l’efficacité de l’éditeur, même pour le même gène dans différents types de cellules. Par exemple, les gènes qui n’ont pas d’îlots CpG annotés peuvent être difficiles à faire taire de manière stable par la méthylation de l’ADN médiée par CRISPRoff48. Par conséquent, il peut être nécessaire de tester différents éditeurs d’épigénome pour faire taire ces gènes. De plus, nous recommandons également de tester au moins les trois principaux ARNg CRISPRi afin d’identifier le meilleur guide pour une efficacité d’édition de l’épigénome plus élevée42. Néanmoins, compte tenu de notre boîte à outils limitée d’éditeurs d’épigénome robustes, il peut être plus efficace de procéder à l’inactivation de gènes ou d’utiliser d’autres stratégies d’inactivation en fonction du gène d’intérêt.
Ce protocole est axé sur CRISPRi et CRISPRoff, deux des nombreux éditeurs d’épigénome basés sur CRISPR disponibles. De récentes études de découverte à grande échelle ont mis au point de nouveaux outils pour réécrire l’épigénome humain 29,30,52. Les éditeurs d’épigénome ont des applications dans la recherche biomédicale et thérapeutique. Par exemple, des études récentes ont utilisé la méthylation de l’ADN et l’édition de l’épigénome basée sur H3K9me3 dans des modèles de souris et de primates non humains, ce qui a entraîné une répression héréditaire du gène 53,54,55 associé à la maladie. Nous prévoyons que les futures modalités de livraison pour les éditeurs d’épigénome ouvriront de nouvelles voies pour l’application large de l’édition d’épigénome.
Déclarations de divulgation
J.K.N. est un inventeur de brevets liés aux technologies CRISPRoff/on, déposés par les Régents de l’Université de Californie.
Remerciements
Nous remercions les membres du laboratoire Nuñez, en particulier Rithu Pattali et Izaiah Ornelas, pour le développement et l’optimisation des protocoles décrits dans ce manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
Références
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon