Method Article
使用质粒 DNA 转染和 mRNA 核转染递送在人类细胞中进行 CRISPR 表观基因组编辑
摘要
该方案描述了使用质粒 DNA 转染和 mRNA 核转染在人类细胞系中基于成簇规则间隔短回文重复序列 (CRISPR) 的表观基因组编辑的方法。
摘要
表观遗传学是指组蛋白和 DNA 的化学修饰,可以调节基因的表达。人类表观基因组在细胞分化和衰老过程中发生动态改变,许多疾病与异常的表观基因组模式有关。CRISPR 的最新进展导致了可编程工具的开发,用于编辑目标基因组位点的表观遗传修饰,从而能够精确重写人类细胞中的表观遗传修饰。基于 CRISPR 的表观基因组编辑器依赖于催化死亡的 Cas9 与表观遗传修饰物相结合,最终导致哺乳动物基因组中靶基因的程序性抑制或激活。与传统的基因组编辑方法不同,表观基因组编辑不需要 DNA 断裂或人类基因组序列的改变,因此是控制基因表达的更安全的替代方案。在该协议中,我们重点介绍了两种不同的方法,使用质粒 DNA 转染和编码 CRISPR 表观基因组编辑器的 mRNA 的核转染在人类细胞系中进行 dCas9 介导的表观基因组编辑。我们展示了可编程的表观基因组编辑,以使用 CRISPR 干扰 (CRISPRi) 瞬时抑制基因,并使用 CRISPRoff(dCas9 与 KRAB 结构域和 从头 DNA 甲基转移酶复合物的融合)将基因持久沉默数周。我们还提供了有关测量靶基因表观基因组编辑成功的定量方法的指导,以及根据实验标准使用哪种表观基因组编辑工具的关键考虑因素。
引言
尽管我们体内每个细胞的基因组内容几乎相同,但每种细胞类型的转录谱差异很大。DNA 和组蛋白的表观遗传修饰是转录表达的关键调节因子。与紧凑的、转录失活的异染色质相比,转录活性的常染色质具有明显的表观遗传标记。例如,异色性区域由抑制性组蛋白修饰定义,包括组蛋白 3 赖氨酸 9 (H3K9me3) 上的三甲基化、组蛋白 H3 赖氨酸 27 上的三甲基化 (H3K27me3) 以及基因启动子1 处鸟嘌呤旁胞嘧啶 (CpG) 上的 DNA 甲基化。活性基因表达的基因组区域通常由组蛋白 3 赖氨酸 4 上的组蛋白乙酰化和三甲基化 (H3K4me3) 来定义1。
成簇规则间隔短回文重复序列 (CRISPR) 革命产生了大量工具,可以对基因组序列进行程序化改变。CRISPR 技术基于能够在可编程靶序列处切割核酸的原核防御机制。CRISPR 核酸酶 2,3,4、碱基编辑器 5、6 和引物编辑器 7,8 可以通过 DNA 切割和修复这些断裂来改变哺乳动物基因组的 DNA 序列。尽管有效,但这些策略会导致脱靶位点 9,10 的 DNA 断裂和大规模基因组结构突变11、12、13、14、15。基于 CRISPR 的替代工具能够在不改变潜在 DNA 序列的情况下实现基因激活和抑制的轻松调节。这些工具利用核酸酶缺陷型 Cas9 (dCas9),允许在 sgRNA 序列决定的靶位点结合 DNA,并与改变染色质景观的效应蛋白相结合 16,17,18。效应蛋白(如表观遗传写入蛋白、读取蛋白和擦除蛋白)可以直接与 dCas9 融合,或通过与 dCas9 融合的肽支架(如 SunTag)或 sgRNA 上的 RNA 支架(如 MS2-MCP 系统)募集 16,17,18。可编程转录控制工具的示例包括 CRISPR 激活 (CRISPRa)19,20,21 和 CRISPR 干扰 (CRISPRi)22,23。CRISPRa 通过直接募集转录机制发挥作用,增加靶标转录基因表达19。相比之下,CRISPRi 通过建立抑制性表观遗传标记 H3K9me3 来抑制转录22。
表观基因组编辑的进步使这些工具在科学领域得到广泛使用。不同效应结构域和蛋白质的融合扩展了可用的表观基因组编辑器18、24、25、26、27、28、29、30 的工具包。此外,表观基因组编辑器用于破译表观遗传修饰 31,32,33,34,35,36、效应子 37,38,39 和效应子突变 28,40,41 的作用在基因调控中。具体来说,CRISPRi 和 CRISPRa 用于各种生物过程的功能基因组学筛选,包括细胞存活42 和细胞命运43、44、45、46、47。此外,表观基因组编辑具有离体细胞工程和体内疗法的治疗潜力18。
在这里,我们描述了在人类细胞系中应用两种基于 dCas9 的表观基因组编辑器进行可编程转录抑制的方法:CRISPRi22 和 CRISPRoff48。CRISPRi 是 dCas9 与锌指蛋白(如 ZNF10 (KOX1) 和 ZIM322,23)的抑制性 KRAB 结构域的融合。当 CRISPRi 靶向特定基因启动子时,KRAB 结构域会募集 H3K9me3 编写器 SETDB1 来抑制靶基因(图 1A)。当 CRISPRi 瞬时表达时,在靶基因座上建立的 H3K9me3 得不到维持,基因表达会随着时间的推移而恢复 32,48。为了使用 CRISPRi 进行稳定的敲低,例如在功能基因组学应用中,CRISPRi 在细胞中与 sgRNA 的组成型表达是必不可少的。最近,CRISPRoff 被设计为对可遗传的表观基因组编辑进行编程48。CRISPRoff 是 dCas9 与 KRAB 结构域和从头 DNA 甲基转移酶复合物 DNMT3A 和 DNMT3L 的单一蛋白融合。人类细胞中 CRISPRoff 的瞬时脉冲会触发 H3K9me3 的沉积和 DNA 甲基化在目标基因上,从而通过维持 DNA 甲基化和 H3K9me3 导致目标基因的长期抑制(图 1B)48。此外,表观基因组编辑可以逆转。例如,被 CRISPRoff 稳定沉默的基因可以被 TET1-dCas9 重新激活,TET1-dCas9 可以酶促去除靶基因座49 处的 DNA 甲基化标记。
该协议将详细介绍表观基因组编辑器瞬时表达的两种递送方法:质粒 DNA 转染和 mRNA 核转染。此外,我们概述了如何使用流式细胞术评估两个内源性基因 CLTA 和 CD55 的表观基因组编辑功效。这些方法可以适应并应用于使用其他编辑器的表观基因组编辑实验,或者可用于靶向不同的基因。

图 1:CRISPRi 和 CRISPRoff 表观基因组编辑机制和工作流程示意图。(A) sgRNA 转基因和 CRISPRi 表观基因组编辑器的线性示意图。添加 CRISPRi 和 sgRNA 允许添加抑制性 H3K9me3 组蛋白标记以沉默靶基因座。添加 CRISPRi 后早期达到最高水平的沉默,并且基因靶标通常在几次传代后重新激活。(B) sgRNA 转基因和 CRISPRoff 表观基因组编辑器的线性示意图。向靶向目标基因的细胞中添加 CRISPRoff 会导致在 CpG 位点添加抑制性 H3K9me3 以及 DNA 甲基化,从而沉默靶基因。CRISPRoff 的沉默是可遗传的 - 高水平的沉默在转染的早期实现,并在多次细胞分裂中持续存在。(C) 通过转染方法进行表观基因组编辑的时间表概述。在第 0 天,对细胞进行铺板以进行转染。第 1 天,可以通过转染将编辑器质粒和向导质粒引入细胞中。第 3 天,将通过流式细胞术评估细胞的 BFP 表达。BFP 的百分比用作归一化因子,以确定实验对每种条件的最终沉默效果。从第 6 天开始,分析细胞以沉默感兴趣的报告基因,因为在这一天将达到最高水平的沉默。(D) 转染方法概述,其中表观基因组编辑器和 sgRNA 质粒以逐滴方式添加到细胞中。(E) 通过核转染方法进行表观基因组编辑的时间表概述。在该方案中,mRNA 在第 0 天核转染到细胞中。使用流式细胞术分析在成核后第 3 天评估细胞的沉默。(F) 核转染方案概述。将适量的细胞和 mRNA 混合并加入 nucleofector 比色皿中。如果 sgRNA 是通过核转染引入的,也可以将其添加到该混合物中。将比色皿放入 nucleofector 中,并使用适当的脉冲编码将 mRNA 引入细胞。核转染后,将细胞铺板并传代以备后几天分析。(G) 用于表观基因组编辑的质粒转染和 mRNA 核转染策略的比较。 请单击此处查看此图的较大版本。
研究方案
注: 补充文件 1 包含有关我们代表性数据的 sgRNA 设计、克隆和细胞系生成的详细信息。代表性结果部分还详细介绍了控件的建议。
1. 将表达表观基因组编辑器的质粒转染到HEK293T细胞中
注意:该协议描述了将 CRISPR 编码质粒递送到 HEK293T 细胞中。我们已经对细胞进行了工程改造,以表达靶向 CLTA 启动子区域的 sgRNA (Addgene 217306), CLTA 是一种非必需基因,内源性地标记有 GFP。CLTA-GFP HEK 293T 细胞起源于之前的研究50。在这个例子中,表观基因组编辑器直接融合到蓝色荧光蛋白 (BFP) 上,这使我们能够量化转染效率并确保对实验条件的准确评估。该方法的有效性通过沉默 CLTA-GFP 来证明,CLTA-GFP 可以使用流式细胞术在单细胞的蛋白质水平上定量测量。
- 细胞培养
- 用 10% FBS 和 1x 青霉素 - 链霉素 - 谷氨酰胺将HEK293T细胞维持在高葡萄糖 DMEM 中。每 2-3 天传代一次细胞,确保它们保持在 60%-70% 的汇合度。
注:为确保最佳转染效率,将细胞汇合度保持在 70% 以下至关重要,可以每天在显微镜下监测。
- 用 10% FBS 和 1x 青霉素 - 链霉素 - 谷氨酰胺将HEK293T细胞维持在高葡萄糖 DMEM 中。每 2-3 天传代一次细胞,确保它们保持在 60%-70% 的汇合度。
- 第 0 天:接种 HEK293T 细胞进行转染
- 转染前一天,使用台盼蓝活/死染色,用自动细胞计数仪对细胞进行计数,并在 96 孔板中每孔接种 ~1.5 x 104 个活细胞。将每个孔的最终体积保持在 200 μL。第二天细胞应达到 60%-70% 汇合度。
注:这也可以在 24 孔或 6 孔板中高效进行。有关转染的细胞数量和 DNA 量,请参见 表 1 。
- 转染前一天,使用台盼蓝活/死染色,用自动细胞计数仪对细胞进行计数,并在 96 孔板中每孔接种 ~1.5 x 104 个活细胞。将每个孔的最终体积保持在 200 μL。第二天细胞应达到 60%-70% 汇合度。
- 第 1 天:将 CRISPRoff 和 CRISPRi 质粒转染到 HEK29T 细胞中
- 每个孔分装 150 ng 表观基因组编辑器质粒 (Addgene 167981),总体积为 3 μL。这些质粒稀释液可以在 PCR 联管中制备。
注:如果共转染表观基因组编辑器和 sgRNA 质粒,那么我们建议尝试 3:4 摩尔比的编辑器:sgRNA 质粒。该比率可能需要优化,以产生最高的表观基因组编辑效率。 - 转染前约 30-60 分钟,将转染试剂和最低必需培养基预热至室温。每孔混合 0.6 μL 转染试剂和 20 μL 最低必需培养基,并在室温下孵育 15 分钟。
注:如果进行多次转染,则可以制备转染试剂和最低必需培养基的预混液。 - 向每个含有 3 μL 质粒 DNA 的 PCR 试管中加入 20.6 μL 转染试剂和最低必需培养基混合物。在室温下静置 15 分钟。
- 将混合物缓慢加入到接种细胞板中,轻轻敲击板以分散混合物。包括未转染的对照,为下游分析中的比较提供适当的基线。
注:将转染混合物添加到接种细胞中时,必须逐滴轻柔地添加,以尽量减少干扰,因为这会对转染效率产生不利影响。
- 每个孔分装 150 ng 表观基因组编辑器质粒 (Addgene 167981),总体积为 3 μL。这些质粒稀释液可以在 PCR 联管中制备。
- 第 2 天:检查转染成功
- 在显微镜下以 10 倍放大倍率检查细胞,以确定它们是否为 BFP 阳性,因为这表明转染成功。确保在观察过程中细胞不受干扰。
- 第 3 天:分液 96 孔板并使用 BFP 标记物评估转染效率
注:成功的转染应产生至少 30% 的 BFP 阳性细胞。然而,对于 dCas9、CRISPRi 和 CRISPRoff,我们通常在转染后 2 天检测到超过 60% 的 BFP 阳性细胞。转染效率与质粒大小呈负相关,因此不同的表观基因组编辑器可能会产生不同的转染效率。- 使用前,将含有 10% FBS 和 1x 青霉素-链霉素-谷氨酰胺的胰蛋白酶和 DMEM 预热至室温约 30-60 分钟。
- 从孔中吸出培养基;小心不要去除附着在底部的细胞。每孔加入 50 μL 胰蛋白酶。在 37 °C 下孵育 2-3 分钟。
注:观察到一些漂浮细胞并不罕见,尤其是在使用 CRISPRoff 转染后。我们假设这种细胞死亡是由于 DNMT3A-3L 过表达的毒性。 - 通过每孔添加 150 μL 完全 DMEM 来抑制胰蛋白酶。上下移液以重悬细胞并在第 5 天分流细胞以汇合(~20 μL 细胞混合物在 96 孔板的 200 μL 培养基中)。
- 使用 50 μL 重悬细胞进行流式细胞分析,以评估转染效率。
2. 表观基因组编辑器 mRNA 核转染到 K-562 细胞中
注:本节详细介绍了将 CRISPRoff mRNA 成核到 K-562 细胞中的过程。为简单起见,我们对 K-562 细胞进行了预工程改造,以组成型表达靶向 CD55 内源基因启动子 (Addgene 217306) 的 sgRNA。将 CRISPRoff mRNA 直接递送到细胞有可能降低基于质粒 DNA 的方法伴随的细胞毒性,同时仍能实现类似的基因沉默功效。此外,核转染可用于在难以有效转染的细胞系(如 K-562)中引入表观基因组编辑器构建体。
- 细胞培养
- 在核转染之前,将 K-562 细胞维持在装有 10% FBS 和 1x 青霉素-链霉素-谷氨酰胺的 RPMI 的培养瓶中。每天传代细胞,确保它们保持在 60%-70% 的汇合度。
- 在核转染当天,在无菌无 RNase 微量离心管中解冻并轻轻涡旋 CRISPRoff mRNA,并将 mRNA 储存在冰上。在试纸核比色皿的每个孔中,每 2.0 x 105 个细胞使用 2 μg mRNA。确保 mRNA 的体积不超过总核转染体积 (22 μL) 的 10%。
注:核转染也可以在具有更多细胞和 mRNA 的较大比色皿中进行。我们建议根据您将添加的细胞数量按比例缩放 mRNA 的量。例如,当在大比色皿中对 1 x 106 个细胞进行核转染时,我们建议使用 10 μg mRNA,总核转染体积为 100 μL。此外,可以优化 mRNA 的剂量以确保高编辑效果。我们通常每 2.0 x 105 个细胞使用 2-5 μg 编辑 mRNA,以获得有效的结果。定量前应去除 mRNA 合成过程中未掺入的游离核苷酸。 - 根据制造商的说明准备核转染溶液,并在核转染前加热至室温 15 分钟。
注:核转染基溶液可能不同或需要针对不同的细胞类型进行优化。 - 使用台盼蓝活/死染色的自动细胞计数仪收获和计数 K-562 细胞。对于在条式比色皿中进行成核转染,将每个样品 ~2.0 x 105 个细胞分装到无菌微量离心管中。
- 在室温下以 500 x g 离心细胞 5 分钟,然后弃去上清液。用室温 PBS 以 500 x g 洗涤细胞 1 次 5 分钟,然后弃去上清液。
- 从 22 μL 中减去 mRNA 体积,计算重悬细胞的 nucleofector 溶液量。将细胞重悬于适量的 nucleofector 溶液中。
- 将细胞溶液加入 2 μg CRISPRoff mRNA(体积在步骤 2.1.2 中确定)中,并将溶液转移到比色皿中,注意不要形成气泡,因为这可能会损害核转染的效率。轻轻敲击比色皿以确保细胞位于底部。
- 使用具有适当脉冲代码的 4D-Nucleofector 系统对细胞进行成核。建议将 FF-120 代码用于 K-562 细胞。
注意:脉冲代码可能需要针对不同的细胞类型进行优化。如果需要其他优化,请咨询制造商。 - 核转染后,向每个小核比色皿孔中加入 80 μL RPMI 培养基,让细胞在 37 °C 下静置 15 分钟。
- 将细胞悬液转移到含有 400 μL 预热 RPMI 培养基(小核比色皿)的 24 孔板的孔中。
注:核转染后,细胞特别脆弱,应轻轻地从核比色皿转移到培养孔中。 - 核转染后第 2 天后:按照第 3 节所述使用 CD55 抗体进行表面标志物染色方案,并在流式细胞仪上测量 CD55 APC 的荧光,以测定表观基因组编辑功效。
注:为了检测核转染成功,我们建议使用 GFP 或 mCherry mRNA 进行核转染。核转染后 2 天,对这些细胞进行流式细胞术,以确认核转染成功。我们通常检测到 90% 至 100% 的这些细胞表达 GFP 或 mCherry。另一种方法是使用 CRISPRi mRNA 进行核转染,众所周知,CRISPRi mRNA 可以稳健地沉默许多靶基因。在第 2 天进行流式细胞术时,CRISPRi 处理的样品应在超过 90% 的细胞中显示靶基因沉默。
3. 表面标记物染色
注:本节详细介绍了 K-562 细胞中表观基因组编辑后 CD55 蛋白的定量水平。我们使用抗体染色和流式细胞术(参见下面的第 4 节)量化单细胞中 CD55 表达的降低,以评估 CRISPRoff 介导的敲低效率。其他技术,包括逆转录定量 PCR 或蛋白质印迹,也可用于确认转录本和蛋白质水平的敲低水平。
- 使用自动细胞计数器对细胞进行计数,将约 5.0 x 105 至 1.0 x 106 个细胞加入微量离心管中,并以 500 x g 离心细胞 5 分钟。
注:可以使用更多或更少的细胞进行染色,但抗体浓度可能需要相应地调整。 - 丢弃上清液,同时确保不干扰沉淀,加入 500 μL PBS 并轻轻重悬沉淀。
- 再次以 500 x g 离心细胞 5 分钟。弃去上清液,加入用 PBS 稀释的所需浓度的抗体。对于 CD55 染色,在 49 μL PBS 中使用 1 μL 纯化的抗人 CD55(0.5 mg/mL 储备液)。
- 轻轻吸取细胞,让它们在室温下在黑暗处孵育 25 分钟。孵育后,再次以 500 x g 离心细胞 5 分钟,弃去上清液。
- 用 500 μL PBS 重悬沉淀,以洗去多余的抗体。将细胞以 500 x g 离心 5 分钟,并弃去 PBS 上清液。重悬于 100 μL 至 200 μL 新鲜 PBS 中,并添加到 96 孔板中进行流式细胞术分析。
4. 流式细胞术
注意:该方案是为使用 BD FACSymphony A1 细胞分析仪而编写的。具体细节可能因您使用的流式细胞仪而异。我们建议您参考您正在使用的机器的用户手册以了解具体信息。
- 在打开细胞仪之前,请检查废液容器是否未装满,以及是否有充足的鞘液。打开靠近鞘液的主电源开关(绿色)。
- 使用返回开关打开读板器。然后,使用右侧的绿色按钮打开机器,并确保机器设置为 96 孔读板器功能。
- 打开计算机并登录。打开软件并使用您的用户名和密码登录。
- 设置实验。通过选择 Experiment > New Experiment 创建新实验。双击当前名称来重命名实验。
- 通过单击 Experiment > New Plate 添加板。选择要使用的孔格式。要添加要运行的特定孔,请单击孔在板中的位置,然后单击 蓝色注射器 (孔现在应以蓝色突出显示)。如果需要,可同时选择多个孔进行添加。
- 通过右键单击板显示屏右侧的 Specimen 为每个孔添加特定名称。然后选择 Experimental layout(实验性布局)。这将打开一个页面,列出您选择的所有孔。要重命名井,请单击一次并键入所需的名称。点击 OK 完成后。
- 将绘图和适当的门添加到 Global Worksheet 上的实验中。我们建议首先绘制前向散射区域 (FSC-A) 与侧向散射区域 (SSC-A) 的关系图。然后为活细胞创建一个任意门,以便稍后通过单击 Polygon Tool 进行优化。
- 右键单击此门,然后单击 Drill Down。这将创建一个新图,仅显示来自该活细胞门的细胞。
- 将此新图的轴更改为 FSC-A 与前向散射高度 (FSC-H) 并为单个单元创建一个门。
- 如果在细胞中组成性表达 sgRNA,并且慢病毒也编码荧光蛋白,则深入研究该单细胞门并进一步设门以表达该蛋白质作为 sgRNA 表达的代理(例如:PE-CF594-A 与 FSC-A)。
- 在最终设置门上(对于单个细胞或表达 sgRNA 的细胞),右键单击并选择 Drill Down。创建一个用于检测报告基因表达的门(例如:BB515-A 与 FSC-A)。
- 如果使用转染细胞,并且有检测转染效率的方法,例如 BFP 表达,请右键单击 Reporter Expression Plot ,然后单击 Duplicate。将轴更改为 BV421-A 与 FSC-A。为 BFP 阳性细胞创建任意门。
- 删除细胞仪显示的 Parameters (参数) 选项卡中未使用的任何参数。
- 要设置机器并准备运行,请单击流式细胞仪控制面板上的 Run(运行 )。该按钮应显示为绿色。在软件中,转到顶部面板中的 HTS,然后转到 Prime。完成后,添加印版,确保它在机器中的位置正确且盖子已关闭。
- 要运行,请选择所有要运行的孔,并确保正确设置对照。双击选择孔,将样品体积更改为 10 μL,然后单击 Acquisition Dashboard 中的 Acquire Data 。这将运行样本,但不会记录数据。当该样品正在运行时,根据需要调整激光电压。此外,更改门。
注意:我们建议运行一个对所有被测颜色均为负值的样品,而对所有颜色都呈阳性的样品,以确保激光参数和选通良好。 - 检查激光设置后,单击 “板”面板中的“样品 ”,并验证运行值是否按所需设置。
- 单击 Run plate 以在 Acquisition Dashboard 中运行并记录来自所有孔的数据。完成后,通过取下板来清洁机器。
- 对于新的 96 孔板,将 10% 漂白剂添加到顶部四行中,将 H2O 添加到下面的四个孔中。转到 HTS > Clean。完成后,关闭程序并按照它们在 Plate Reader >绿色开关上打开绿色侧按钮> Waste 开关的相反顺序关闭按钮。
- 向含有细胞的 96 孔板中加入 20% 漂白剂并丢弃。
5. 数据分析
注:该方法概述了门控策略和数据处理,以通过流式细胞术量化表观基因组编辑。门控策略与 图 2 和 图 3 中数据分析生成的示例图一起直观地表示。
- FlowJo 门控设置
- 将所有 FSC 文件拖放到新工作表中,将它们加载到 FlowJo 上。
- 单击 All Samples 开始制作门。这将确保门可以轻松应用于所有样品。
- 双击对照样品(例如未转染或未核染)打开该文件。
- 使用多边形工具在 FSC-A 与 SSC-A 图中为活细胞创建门。在所有样品的列表中,样品名称下方现在应显示 Live Cells 选项卡。右键单击此门,然后选择 Copy Analysis to Group 将此门应用于所有样品(图 2A、 图 3A)。
- 双击 Live cells 门以向下钻取活细胞。将轴更改为 FSC-A 与 FSC-H(图 2B、 图 3B)。
- 绘制一个多边形以仅针对单个单元格进行门控。按照第 4 节(图 2B、图 3B)中所做的,将此门应用于所有样品(图 2B、 图 3B)。
注:如果用荧光报告基因组成型表达 sgRNA 构建体,请对该颜色进行进一步设门,以便分析仅关注表达 sgRNA 的细胞。这方面的一个例子是为 mCherry 阳性细胞(PE-CF594-A 与 FSC-A)创建一个门,如果细胞含有来自 pLG1 骨架的 sgRNA(图 2C、 图 3C)。 - 双击 Last Setup Gate (单个细胞或表达向导的细胞)。
- 如果进行质粒转染并且表观基因组编辑器编码 BFP 融合,则为第 2 天样品的 BFP 阳性细胞(例如:BV421-A 与 FSC-A)创建一个门(图 2D)。
- 为报告基因表达创建一个门(例如:BB515-A 与 FSC-A 对 GFP 阴性细胞的门控)并将该门应用于所有样品(图 2E、 图 3D)。
- 转染分析
- 执行门控设置后,单击上面板中的 Table Editor 。拖动总体进行分析(例如:BFP 阳性、GFP 阴性)。
- 在表编辑器面板中,单击 Create Table。将这些数据复制到电子表格中以执行标准化计算。
- 数据处理
- 从所有其他样品中减去对照样品中报告基因阴性细胞(例如:GFP 阴性)的数量。报告者可能会有一些背景静音,因此这会从数据中消除这种背景噪声。
- 所有值都将标准化为第 2 天测量的 BFP 阳性细胞(成功转染细胞)的百分比。要进行此标准化,请取该值,除以第 2 天 BFP 阳性细胞的百分比,然后乘以 100。该计算得出编辑细胞的转染效率标准化值。现在,数据已准备好进行绘制。我们建议在表观基因组编辑时间过程中使用折线图,如图 2F 所示。
- 核转染分析
- 执行门控设置后,单击上面板中的 Table Editor 。拖动总体进行分析(示例:APC 阴性)。
- 在表编辑器面板中,单击 Create Table。将这些数据复制到电子表格编辑器中以执行标准化计算。
- 数据处理
- 从所有其他样品中减去对照样品中报告基因阴性细胞(例如:APC 阴性)的数量。报告者可能会有一些背景静音,因此这会从数据中消除这种背景噪声。现在,数据已准备好进行绘制。我们建议在表观基因组编辑时间过程中使用折线图。
注意:统计数据可用于量化两种不同表观基因组编辑器之间的沉默差异。我们建议将所有转染和成核转染实验一式三份进行,以进行统计分析。在描述一段时间内沉默的折线图时,绘制技术仿行的标准差。可以进行特定时间点的学生 t 检验来比较两个表观基因组编辑器之间的沉默。样本应被视为方差不相等的未配对样本。
- 从所有其他样品中减去对照样品中报告基因阴性细胞(例如:APC 阴性)的数量。报告者可能会有一些背景静音,因此这会从数据中消除这种背景噪声。现在,数据已准备好进行绘制。我们建议在表观基因组编辑时间过程中使用折线图。
结果
对于所有表观基因组编辑实验,适当的对照对于评估表观基因组编辑效率至关重要。我们建议使用对照 sgRNA,它不靶向人类基因组中的任何序列。使用非靶向指南对照将确信靶基因座的变化是由定向到该位点的表观基因组编辑器驱动的,而不仅仅是来自表观基因组编辑器的过表达或非特异性结合。此外,对于基于报告基因的实验,我们建议使用仅 dCas9 的对照,以确保报告基因表达的变化是由于表观基因组编辑器融合,而不是 dCas9 与靶基因座结合的空间位阻并暂时阻碍转录(图 2F)。
对于转染实验,我们建议使用带有额外荧光蛋白融合的表观基因组编辑器,例如 BFP。这种融合允许通过显微镜和流式细胞术对用表观基因组编辑器成功转染的细胞进行可视化。成功转染的细胞将在转染后两天表达高水平的 BFP(图 2D)。成功转导的细胞的定量用于在以后的日子里使表观基因组编辑功效正常化(图 2F)。
CRISPRoff 和 CRISPRi 在转染后第 5 天均显示峰值沉默(图 2E-F)。不同的表观基因组编辑器具有不同的表观基因组编辑时间表,例如使用 CRISPRoff 的可遗传沉默和使用 CRISPRi 的瞬时沉默(图 2F)。图 2F 还显示了 dCas9 仅作为表观基因组编辑实验的重要对照的使用情况。在 mRNA 核转染实验中,用 CRISPRoff 成功编辑的细胞将在成核转染后第 3 天显示靶基因的强烈沉默(图 3E-F)。
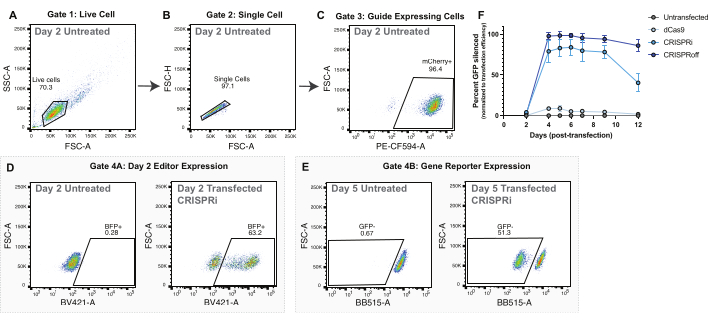
图 2:通过质粒转染进行表观基因组编辑器递送的门控策略和代表性数据。 (A-C)显示质粒转染实验的设门策略的代表性流图。显示的流图是转染后 2 天未转染细胞的流图。图中的每个点都表示一个单元格。(A) 前向散射区 (FSC-A) 和侧向散射区 (SSC-A) 的流图,带有活细胞门。(B) 显示 FSC-A 和前向散射高度 (FSC-H) 的活细胞流图,带有单个细胞的门。(C) 单细胞绘制 PE-CF594-A(mCherry 表达)和 FSC-A 的流程图。mCherry 阳性细胞的门控作为 sgRNA 表达的代理。(D) 转染后第 2 天表观基因组编辑器表达 (BFP+) 的代表性设门策略。亲本群体是在 (C) 中设门的向导表达细胞。(E) 检测 CLTA-GFP 报告基因沉默的代表性门控策略。亲本群体是在图 C 中设门的向导表达细胞。(F) 转染质粒递送 dCas9、CRISPRi 和 CRISPRoff 后几天内 CLTA-GFP 的沉默。将沉默的 CLTA-GFP 百分比标准化为转染效率,以转染后 2 天测量的 BFP 阳性细胞。分数是 4 次转染重复的平均值。误差线表示标准差。 请单击此处查看此图的较大版本。
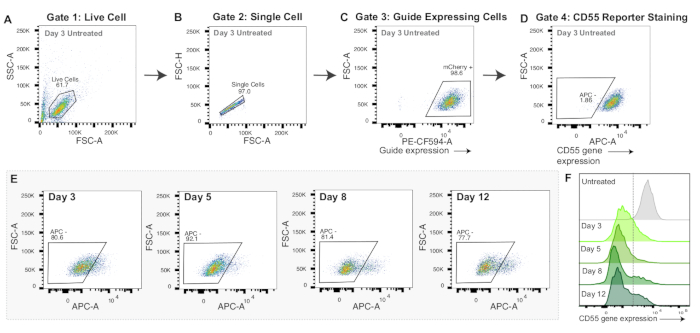
图 3:CRISPRoff mRNA 核转染的门控策略和代表性数据。(A-D) 显示 mRNA 成核转染实验的设门策略的代表性流图。显示的流式图是用 APC 抗人 CD55 抗体染色成核转染后 3 天的对照细胞。图中的每个点都表示一个单元格。(A) 前向散射区强度 (FSC-A) 和侧向散射区强度 (SSC-A) 的流程图,带有活细胞门。(B) 显示 FSC-A 和前向散射高度强度 (FSC-H) 的活细胞流图,带有单个细胞的门。(C) 单细胞绘制 PE-CF594-A(mCherry 表达)和 FSC-A 的流程图。mCherry 阳性细胞的门控作为 sgRNA 表达的代理。(D) APC-A 与 FSC-A 的流程图。为 APC 阴性细胞绘制的门,这表明 CD55 报告基因沉默。(E) 用 CRISPRoff mRNA 成核转染后 3、5、8 和 12 天 CD55 沉默(APC - 门)的流程图。(F) 与染色但未核转染的对照相比,用 CRISPRoff mRNA 成核转染后 3、5、8 和 12 天内 CD55 蛋白表达 (APC-A) 的叠加直方图。 请单击此处查看此图的较大版本。
| 盘子 | 接种密度(每孔细胞数) | 质粒量 |
| 96 孔 | 15,000 | 150 纳克 |
| 24 孔 | 90,000 | 500 纳克 |
| 6 孔 | 400,000 | 2 微克 |
表 1:转染缩放量。 用于表观基因组编辑器转染到不同规模的 HEK293T 细胞中的接种密度和质粒 DNA 量。
补充表 1:用于表观基因组编辑实验的 sgRNA 间隔序列。 用于将表观基因组编辑器靶向 CLTA 和 CD55 的 sgRNA 序列以及用于非靶向对照指南的序列。此外,还列出了用于克隆到 pLG1 骨架中的寡核苷酸。 请点击此处下载此文件。
补充文件 1.请点击此处下载此文件。
讨论
该方案详细介绍了 CRISPR 表观基因组编辑器的两种不同的瞬时递送方法:质粒 DNA 转染和 mRNA 核转染。这两种技术都有独特的优点、缺点和一般注意事项(图 1F)。
质粒 DNA 转染导致稳健的表观基因组编辑器表达,我们在表观基因组编辑器构建体中加入了 BFP 融合,允许使用流式细胞术检测和定量转染功效。此外,BFP 表达可用于分选表达表观基因组编辑器的细胞,或者可以定量以在以后的时间点标准化沉默数据,如本方案中所述。然而,需要注意的是,转染效率通常不是 100%,因此,除非对细胞进行分选,否则接受编辑的细胞群和未接受编辑的细胞群将是异质性的。此外,质粒 DNA 递送可触发细胞质双链 DNA 的免疫反应,导致免疫途径激活和细胞毒性。最后,质粒 DNA 转染并不适合所有细胞类型,可能需要对质粒进行核转染。
与质粒 DNA 转染不同,mRNA 核转染适用于多种细胞类型,并且通常具有很高的递送效率。编辑器 mRNA 可以使用市售 的体外 mRNA 合成试剂盒合成,其中一种已在前面51 中详细介绍。或者,可以从 Aldevron 或 Trilink 合成 mRNA。然而,mRNA 核转染的一个警告是它不能产生足够的编辑器蛋白来检测哪些细胞接受了表观基因组编辑器。上述实验表明,几乎每个细胞都收到了 mRNA,因为我们在第 5 天检测到 ~90% 的靶基因沉默(图 3F)。但是,我们建议优化每种细胞类型的脉冲编码和 mRNA 与细胞比率。
另一个考虑因素是,由质粒 DNA 转染或 mRNA 核转染引发的表观基因组编辑具有不同的沉默时间线。通过质粒 DNA 转染,在转染后第 5 天观察到 CRISPRoff 和 CRISPRi 的峰沉默(图 2F)。相比之下,早在成核转染后第 3 天就可以看到 mRNA 核转染递送的 CRISPRoff 和 CRISPRi 的最大沉默(图 3F)。此外,与 mRNA 相比,质粒 DNA 在细胞中持续存在,导致编辑器表达的持续时间更长。根据实验目标,可能更喜欢更长或更短的沉默时间和表达持续时间。
同样重要的是要注意,表观基因组编辑不能普遍应用于所有基因或细胞类型。靶基因基因组序列或染色质状态的固有差异可能会影响编辑效率,即使对于不同细胞类型中的相同基因也是如此。例如,缺乏注释 CpG 岛的基因可能难以被 CRISPRoff 介导的 DNA 甲基化稳定沉默48。因此,可能需要测试不同的表观基因组编辑器以使此类基因沉默。此外,我们还建议至少测试前三个 CRISPRi gRNA,以确定提高表观基因组编辑效率的最佳指南42。尽管如此,鉴于我们强大的表观基因组编辑器工具箱有限,根据目标基因进行基因敲除或使用其他敲低策略可能更有效。
该协议专注于 CRISPRi 和 CRISPRoff,这是许多可用的基于 CRISPR 的表观基因组编辑器中的两个。最近的大规模发现研究开发了重写人类表观基因组的新工具 29,30,52。表观基因组编辑器在生物医学研究和治疗学中也有应用。例如,最近的研究在小鼠模型和非人灵长类动物中使用了 DNA 甲基化和基于 H3K9me3 的表观基因组编辑,导致疾病相关基因的可遗传抑制 53,54,55。我们设想表观基因组编辑者的未来递送方式将为表观基因组编辑的广泛应用开辟新的途径。
披露声明
J.K.N. 是 The Regents of the University of California 提交的与 CRISPRoff/on 技术相关的专利的发明者。
致谢
我们感谢 Nuñez 实验室的成员,尤其是 Rithu Pattali 和 Izaiah Ornelas,他们开发和优化了本手稿中描述的协议。
材料
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
参考文献
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。