Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Флуоресцентная визуализация в течение жизни NAD(P)H для метаболического анализа кишечника мышей и паразитов во время инфекции нематодами
В этой статье
Резюме
В настоящем протоколе описана флуоресцентная визуализация NAD(P)H эксплантированного кишечника мышей, инфицированного природным паразитом Heligmosomoides polygyrus, что позволяет исследовать метаболические процессы как в тканях хозяина, так и в тканях паразита в пространственном разрешении.
Аннотация
Паразиты, как правило, оказывают негативное влияние на здоровье своего хозяина. Они представляют собой огромное бремя для здоровья, поскольку в глобальном масштабе влияют на здоровье инфицированного человека или животного в долгосрочной перспективе и, таким образом, влияют на сельскохозяйственные и социально-экономические результаты. Тем не менее, были описаны иммунорегуляторные эффекты, вызванные паразитами, с потенциальной терапевтической значимостью для аутоиммунных заболеваний. В то время как метаболизм как у хозяина, так и у паразитов способствует их защите и является основой выживания нематод в кишечнике, он остается в значительной степени малоизученным из-за отсутствия адекватных технологий. Мы разработали и применили флуоресцентную визуализацию NAD(P)H в течение жизни эксплантированной мышиной кишечной ткани во время заражения природной нематодой Heligmosomoides polygyrus для изучения метаболических процессов как у хозяина, так и у паразитов в пространственном разрешении. Использование времени жизни флуоресценции коферментов никотинамидадениндинуклеотида (NADH) и никотинамидадениндинуклеотидфосфата (NADPH), далее NAD(P)H, которые сохраняются у разных видов, зависит от их связывающего статуса и места связывания на ферментах, катализирующих метаболические процессы. Сосредоточив внимание на наиболее широко экспрессируемых NAD(P)H-зависимых ферментах, были выделены метаболические пути, связанные с анаэробным гликолизом, окислительным фосфорилированием/аэробным гликолизом и окислительным взрывом на основе NOX, в качестве основного защитного механизма, а также охарактеризованы метаболические перекрестные помехи между хозяином и паразитом во время инфекции.
Введение
Паразитарные инфекции ложатся огромной нагрузкой на здоровье человека 1,2. Корреляция между ростом аутоиммунных заболеваний и снижением паразитарных инфекций наблюдается в промышленно развитых странах. Известно, что паразиты могут оказывать благотворное воздействие, подавляя чрезмерные иммунные реакции хозяина. H. polygyrus является природным паразитом, обнаруженным в кишечнике грызунов, и известно, что этот паразит индуцирует иммунорегуляторные механизмы, которые снижают антипаразитарный иммунный ответ хозяина посредством, среди прочего, индукции регуляторных Т-клеток (Treg) у инфицированного хозяина 3,4,5,6,7,8,9,10,11 . Эти регуляторные механизмы особенно интересны при дегенеративных аутоиммунных заболеваниях.
Анализ метаболических взаимодействий между хозяином и кишечными нематодами остается широко игнорируемым, хотя метаболизм играет важную роль как в организме хозяина, так и в паразитах для защиты, выживания и функционирования. Мы предлагаем адаптировать и применять флуоресцентную визуализацию NADH и NADPH в течение жизни при двухфотонном возбуждении, технологию, уже широко используемую в различных физиологических и патофизиологических ситуациях в клетках и тканях млекопитающих12, для исследования корреляционного метаболизма хозяина и нематоды в живых тканях.
NADH и NADPH, называемые NAD(P)H, являются вездесущими молекулами, которые сохраняются во всех клеточных формах жизни и играют роль коферментов в различных метаболических путях. Например, они участвуют в производстве энергии, восстановительном биосинтезе и производстве активных форм кислорода (АФК), опосредованных NADPH-оксидазой, которые в основном связаны с защитой клеток и клеточной коммуникацией 13,14,15,16,17,20. Оба кофермента излучают флуоресценцию на длине волны ~450 нм при двухфотонном возбуждении на длине волны 750 нм, что позволяет проводить безмаркерную метаболическую визуализацию в клетках и тканях19,21. Возбуждение как NADH, так и NADPH только одной длиной волны возможно благодаря их схожим и довольно широким спектрам двухфотонного возбуждения21.
Время жизни коэнзима NAD(P)H во флуоресценции напрямую зависит от фермента, с которым он связывается 18,21,22,23. Благодаря своей химической структуре, позволяющей осуществлять внутримолекулярный перенос энергии, возбужденная молекула NADH или NADPH теряет энергию в результате внутренних процессов преобразования, со скоростью, зависящей от ее связывающих свойств, ферментам (катализатору), прежде чем она расслабится и испустит флуоресцентный фотон. Это время жизни дает представление о сайте связывания NAD(P)H на ферменте и, таким образом, о преимущественной биохимической реакции, протекающей 19,21,22,23,24,25. Время жизни флуоресценции свободных молекул NADH и NADPH составляет ~450 пс, в то время как время их жизни флуоресценции при связывании с ферментом значительно больше (~2000 пс) и зависит от места их связывания на соответствующем ферменте21.
Существует более 370 ферментов, участвующих в процессах, связанных с NAD(P)H; однако только наиболее распространенные из них смогут внести свой вклад в результирующее время жизни флуоресценции NAD(P)H в диапазоне возбуждения микроскопа. Используя данные RNASeq из клеток млекопитающих, мы идентифицировали наиболее распространенные NAD(P)H-зависимые ферменты и сгенерировали ссылку на время жизни флуоресценции для интерпретации данных, полученных в образцах тканей и клеток18. Таким образом, в данной работе проводилось различие, например, между преимущественной активностью лактатдегидрогеназы (ЛДГ), которая связана с анаэробными гликолитическими метаболическими путями, и активностью изоцитратдегидрогеназы (ИДГ) и пируватдегидрогеназы (ПДГ), которые в основном участвуют в аэробных метаболических путях гликолиза/окислительного фосфорилирования16,20. Кроме того, связывание NADPH с NADPH-оксидазами, которые являются ферментами, в основном ответственными за окислительный взрыв, может быть легко устранено благодаря характерному расположению этих ферментов в клетке (мембранно-связанному) и из-за особенно длительного времени жизни флуоресценции NADPH (3,650 пс)18,24,29,30,32. Данные RNASeq от H. polygyrus показывают, что эталон, полученный для клеток млекопитающих, также применим в адаптированной форме к этой нематоде27.
Таким образом, в данной работе путем проведения флуоресцентной визуализации (FLIM) NAD(P)H в свежеэксплантированных образцах двенадцатиперстной кишки мышей, инфицированных H. polygyrus, была получена информация о соотношении между свободным и связанным с ферментом NAD(P)H, которая отражала общую метаболическую активность во всех тканях, а также преимущественно активный фермент в каждом пикселе изображения (т.е. фермент, с которым NAD(P)H преимущественно связывается в этом конкретном месте). Успех этих экспериментов зависит от точной подготовки образцов эксплантированной кишки, надежной визуализации времени жизни флуоресценции NAD(P)H в реальном времени с субклеточным разрешением и стандартизированной оценки данных, как описано в этом протоколе.
протокол
Все эксперименты проводились в соответствии с Национальным руководством по защите животных и одобрены Немецким комитетом по этике животных по защите животных (G0176/16 и G0207/19). Протокол описывает данные флуоресцентной визуализации NAD(P)H в течение жизни и оценку данных, которые позволяют оценить общую метаболическую активность и специфические метаболические пути как в кишечнике хозяина, так и у паразитов при заражении естественной мышиной кишечной нематодой, H. polygyrus. С этой целью самки мышей C57BL/6 в возрасте 10-12 недель были инфицированы 200 личинками 3 стадии (L3). В разные моменты инфекции инфицированных мышей умерщвляли, а двенадцатиперстную кишку вырезали и подготавливали к визуализации, как описано ранее33. Двенадцатиперстная кишка неинфицированных мышей соответствующего возраста и пола была аналогичным образом подготовлена и визуализирована для целей контроля. Для поддержания свойств тканей, необходимых для дальнейшей визуализации и анализа, образцы должны быть обработаны сразу после эксплантации, а следующие шаги (шаги 1.1-1.7) должны быть выполнены быстро (рис. 1В).
1. Подготовка образцов
- Отрежьте куски длиной ~1 см от первоначально иссеченной двенадцатиперстной кишки.
- Склейте выпрямленную тканевую трубку в середину небольшой чашки Петри быстроотверждаемым тканевым клеем (диаметр 47 мм, высота обода 10 мм, см. Таблицу материалов) (Рисунок 1А2).
- Нанесите тонкий слой дополнительного клея на дно чашки Петри на большую площадь вокруг ткани с помощью микрокисточки (рисунок 1А2).
- Тупыми ножницами сделайте разрез по всей длине физически неподвижной кишки в районе дна чашки Петри (рисунок 1А3).
- Разверните кишечник тупым пинцетом так, чтобы алюминальная сторона полностью соприкасалась с клеем. Таким образом, кишечник фиксируется клеем просветной стороной вверх (рис. 1А4).
- В случае инфицированной мыши подсчитайте червей под стереомикроскопом (увеличение 10x), чтобы убедиться, что заражение прошло успешно (рис. 1A5).
- Запечатайте кишечник агарозой, чтобы защитить его от пересыхания. Здесь 0,5%-0,9% агарозы использовалась для защиты чувствительной кишечной ткани от ожога из-за ее низкой температуры плавления около 38 °C. Наберите 1 мл агарозы в пипетку и осторожно сбрызните кишечник так, чтобы кончик пипетки и ткань были полностью закрыты тонким слоем толщиной около 0,5 мм (рис. 1A6).
- Заполните чашки Петри образцами, запечатанными в агарозу с PBS (10%) при комнатной температуре, а затем закройте их крышкой. Либо визуализируйте образцы напрямую, либо поместите их на лед в теплоизоляционный бокс, чтобы встать в очередь на измерение. Поместите первый подготовленный образец под объектив микроскопа на нагревательную пластину, установленную на 37 °C (Рисунок 1A8).
2. Визуализация
Микроскопическая система, используемая для проведения NAD(P)H-FLIM в инфицированных и здоровых образцах ткани двенадцатиперстной кишки, состоит из устройств, перечисленных и описанных на рисунке 2 и в таблице материалов. Используйте ImSpector 208 в качестве управляющего программного обеспечения для всех используемых модулей.
- Чтобы изначально найти интересующую область (ROI), поместите образец в чашку Петри под объективом и двигайтесь вдоль плоскости x и y вручную, чтобы найти подходящий ROI путем визуального осмотра в режиме широкопольной флуоресцентной микроскопии.
- Переключите систему формирования изображений на двухфотонное возбуждение с помощью детектирования ФЭУ или детектирования однофотонного подсчета с корреляцией по времени (TCSPC) путем переключения режимов в программном обеспечении. Обратите особое внимание на то, чтобы окружающая среда была темной и свободной от вибрации, закройте микроскоп светонепроницаемыми шторами и, по возможности, используйте пневматический подвесной оптический стол.
- Погрузите линзу объектива в чашку Петри, содержащую образец, с помощью управляющего программного обеспечения, щелкнув значок системы линз и повернув колесико мыши, чтобы изменить положение Z.
- Настройте лазер на 765 нм и установите максимальную мощность лазера на 10 %, что соответствует 30 мВт под объективом, введя желаемую длину волны на панели длин волн программного обеспечения. При необходимости отрегулируйте мощность лазера. Измерьте коэффициент усиления ниже 40% (от ~30 мВт до 100 мВт), чтобы избежать фотоповреждения тканей и нежелательных метаболических сдвигов.
- В программном обеспечении установите размер шага z-ступени на 2 мкм, что соответствует осевому разрешению микроскопа при 765 нм в тканях.
- Установите частоту сканирования линии на 400 Гц на панели гальванического зеркала, в результате чего время задержки пикселей составит 4,95 мкс.
- Установите усреднение изображений по двум-четырем изображениям, выбрав усредненное число в раскрывающемся меню в программном обеспечении (т. е. отсканируйте ROI от двух до четырех раз, чтобы получить более плавные изображения). Таким образом, общее время задержки пикселей увеличивается до 9,9-19,8 мкс за счет более высокого отношения сигнал/шум (SNR) в процессе анализа.
- Установите поле зрения изображения (FOV) на 505 x 505 пикселей (500 x 500 μm²), выбрав параметры на панели FOV в программном обеспечении.
- Для каждого измерения заранее откалибруйте время модуля, чтобы обеспечить оптимальную работу электроники TCSPC, нажав кнопку «Инициализировать» в окне калибровки в меню оборудования TCSPC.
- Отрегулируйте быстрый фотодиод внешне, чтобы обнаружить последовательность импульсов лазера Ti:Sa с помощью быстрого осциллографа.
ПРИМЕЧАНИЕ: Сигнал фотодиода используется для запуска детектирования hPMT и подсчета фотонов TCSPC по отношению к импульсу возбуждения. Детектирование повторяется каждые 12,5 нс (т.е. время между двумя последовательными лазерными импульсами), что соответствует частоте повторения лазера Ti:Sa (80 МГц). - Определите диапазон глубины измерения, сначала установив усреднение на 1 и усиление мощности лазера на ~10%, затем запустите видеорежим (нажмите на кнопку видео ), найдите глубину, с которой сигнал все еще может быть получен, снова щелкнув значок системы объективов , и используя колесико мыши для перехода к образцу с постепенным увеличением усиления.
- Для получения изображений образцов тканей NAD(P)H-FLIM, измеренных в режиме FLIM, установите систему на 765 нм при максимальной номинальной мощности 100 мВт. Детектирование флуоресцентного сигнала NAD(P)H (466/60 нм) с помощью гибридной ФЭУ с коэффициентом усиления 97%, который задан в программном обеспечении. Модуль TCSPC подсчитывает фотоны по 227 временным окнам (ячейкам), каждое из которых имеет длительность 55 пс.
- Щелкните правой кнопкой мыши по отображаемому изображению во время измерения в программном обеспечении и выберите отображение > T-PROFILE , чтобы визуализировать кривую затухания, полученную системой.
- Обратите внимание на количество столбцов перед лазерным импульсом на кривой затухания для последующего анализа.
- Получите данные, нажав на кнопку «Начать измерение».
ПРИМЕЧАНИЕ: Данные должны соответствовать определенному формату. Каждый срез, записанный на любой глубине ткани, разделен расстоянием 2 мкм и имеет площадь 500 мкм x 500 мкм, а срезы получены в виде стопки срезов размером 500 мкм x 500 мкм с временными точками 227 x 55 пс (рис. 3A). Каждый воксель содержит гистограмму времени прихода фотонов с пространственным разрешением (в плоскости x и y). Это представляет собой затухание флуоресценции (рис. 3B). Обычно измеряемый объем должен иметь форму гиперстека с размерами ~500 мкм x 500 мкм x 100-300 мкм и 227 ячейками, где каждый срез в z-плоскости содержит зависящие от времени данные об интенсивности, как описано выше. Для примерного набора данных измерений это дает (505 x 505 x 227) x 100 пикселей (16 бит) и соответствует примерно 4 ГБ.
3. Анализ данных
ПРИМЕЧАНИЕ: Для векторного анализа изображений NAD(P)H-FLIM программа для вычисления времен жизни представляет собой специально написанный код на языке Python33.
- Используйте Anaconda с дистрибутивом Python 3.7 в интегрированной среде разработки Spyder (см. Таблицу материалов). В коде используются стандартные библиотеки Python.
- Загрузите код в среду IDE и выполните его. Откроется диалоговое окно с путями к файлу.
- Выберите папку с исходными данными для анализа. Код запрограммирован на выбор трех параметров перед анализом через диалоговое окно пользовательского ввода Tkinter с флажками, выпадающим меню и текстовыми полями.
- Выберите смещение в диалоговом окне ввода. Определите смещение как количество первых срезов в стеке времени, которые не анализируются.
ПРИМЕЧАНИЕ: Параметр сокращает временные точки перед импульсом возбуждения. При калибровке системы и пускового диода это значение должно составлять примерно пять срезов или количество мертвых ячеек до возбуждения лазера, как описано в шаге 2.14. - Выберите представление векторных графиков в раскрывающемся меню диалогового окна ввода. Здесь выберите внешний вид точек данных (облачный или топографический), а также координаты полукруглой оси времени (энзимы или время [ps]) в качестве опций из выпадающего меню.
- Нажмите на OK.
- Убедитесь, что код вычисляет два типа информации из z-стеков NAD(P)H-FLIM.
- Во-первых, измерьте пространственную информацию каждого объемного среза по коллапсу стеков TCSPC, где 227 гистограмм подсчета фотонов с временным разрешением проецируются на один срез, называемый проекционным изображением интенсивности (рисунок 3G), и из анализа экспоненциальных кривых распада на основе Фурье (как описано Leben et al.18), и из нормализованной действительной и мнимой части для каждого пикселя в каждом объемном срезе (рисунок 3C).
- Из вещественных и мнимых изображений получите векторные графики (рисунок 3D) и изображение среднего времени жизни флуоресценции (t) (цветовая кодировка, пространственно разрешенные средние константы распада для каждого воксела) (рисунок 3E).
Примечание: Как описано во введении, время жизни флуоресценции NAD(P)H при связывании с ферментами определяется сайтом связывания кофермента с соответствующим ферментом. - Определите вклад соответствующего фермента в метаболическую активность путем создания вектора между фазовым вектором каждого пикселя и фазовым вектором несвязанного NAD(P)H и проецирования его на полукруг на векторном графике. Полукруг представляет все возможные моноэкспоненциальные распады времен жизни флуоресценции в чистых соединениях.
- Используя ранее сгенерированный эталон времени жизни флуоресценции NAD(P)H, связанного с наиболее распространенными NAD(P)H-зависимыми ферментами18,33 (дополнительный рисунок 1), и рассчитав вероятность активации этих ферментов.
Примечание: Цветовой код наиболее доминирующего фермента (т.е. фермента, для которого рассчитана наибольшая вероятность активации) присваивается каждому пикселю, таким образом, создается карта ферментов (рис. 3F).
- Исходя из соотношения свободного (несвязанного) и связанного с ферментом NAD(P)H, в частности, из векторного соотношения на векторном графике между свободным NAD(P)H при 450 пс и фермент-связанным состоянием, рассчитайте общую метаболическую активность в процентном соотношении от 0% *только несвязанный NAD(P)H) до 100% (только связанный с ферментом NAD(P)H). Путем присвоения значения метаболической активности (0%-100%) каждому пикселю создается карта активности (с цветовой кодировкой) (рис. 3F).
- Наложите полученные карты на изображения интенсивности, чтобы получить дополнительную морфологическую информацию. Здесь используйте макрос ImageJ (см. Таблицу материалов) для наложения оттенка и насыщенности карты фермента или активности на яркость изображений интенсивности.
- Убедитесь, что макрос выполняет итерацию по всему стеку. Для каждого среза (глубины) в стеке он должен разделить изображение интенсивности и выбранную карту соответственно на HUE, SATURATION и BRIGHTNESS (тип изображения > > стеке HSB).
- Разделите основные каналы для обоих стеков HSB (стеки изображений > > стек в образы).
- Закройте ОТТЕНОК и НАСЫЩЕННОСТЬ стека интенсивности, а также ЯРКОСТЬ стека карты (close(slicename_HUE), close(slicename_BRIGHTNESS), ...).
- Объедините оставшиеся каналы, чтобы сформировать новый стек HSB, состоящий из значений HUE и SATURATION из среза интересующей карты и BRIGHTNESS из изображения интенсивности (Image > stacks > images to stack).
- Измените тип изображения на RGB для лучшего визуального оформления (Тип изображения > > цвет RGB).
4. Сегментация тканей
Примечание: Используйте предварительно обученную сеть на основе U-NET (ILASTIK, см. Таблицу материалов) для сегментации кишечного хозяина и ткани нематоды соответственно, и, кроме того, собственного эпителия и собственной пластинки в организме хозяина, а также областей с высоким флуоресцентным сигналом NAD(P)H и низким флуоресцентным сигнальным участком NAD(P)H в нематоде.
- Откройте ILASTIK и выберите новый проект и классификацию пикселей.
- Загрузите ранее рассчитанную проекцию интенсивности из шага 3.7.1, нажав на кнопку Добавить новую > Добавить отдельные изображения. Загрузите в контейнер случайные срезы из нескольких измерений.
- Нажмите на Выбор функций > выбрать функции. Добавьте значение сигма 50 (безразмерный вес) и отметьте галочкой все активные.
ПРИМЕЧАНИЕ: В то время как значение сигма определяет вес объектов, объекты определяют классы и способность сети распознавать ребра, формы, текстуру или цвет. - Нажмите на кнопку «Тренировка» и назовите метки в соответствии с тканью, которую необходимо сегментировать (например, «Эпителий», «Фон» и т. д.), щелкнув по ним. Выберите метку и раскрасьте все пиксели изображения, соответствующие этой метке.
- Повторите то же самое для других надписей.
- Повторите эти действия примерно для половины изображений в загруженном наборе данных.
- Нажмите « Обновление в реальном времени» и позвольте модели спрогнозировать метки для оставшихся изображений без меток.
- Исправьте примерно половину только предсказанных и непомеченных изображений, переименовав ошибочно оцененные сегментации, и повторите оперативное обновление.
- Повторяйте этот шаг до тех пор, пока сеть не научится правильно оценивать интересующую вас ткань; Убедитесь, что достоверность сети, отображаемая слева рядом с именами меток, находится в диапазоне от 95% до 98%.
- Загрузите новый набор данных, как описано в шаге 3.11, на этот раз со всеми срезами из одного измерения, и нажмите « Экспорт прогнозов».
- В выпадающем меню «Источник» выберите «Простая сегментация»; в поле "Формат" выберите вывод в формате tiff; и в разделе «Выбрать настройки экспорта изображения» выберите путь сохранения для вывода. Нажмите на экспорт.
- При этом создаются бинарные маски для интересующего вас типа ткани. Поскольку они состоят из срезов, в которых сегментированная ткань, представляющая интерес, имеет значение пикселя 1, а остальная — 0 (рисунок 3H), просто умножьте двоичные маски на сгенерированные данные, как указано в шаге 1.1 и шагах 1.4-1.7. Это приводит к маскировке данных (рис. 3 I, J, M). Используйте ImageJ для этого шага (Процесс > Калькулятор изображений > умножения).
- Для каждого объемного среза рассчитайте отношение сигнал/шум (SNR) по замаскированным изображениям интенсивности. Для этой цели используйте макрос ImageJ. Создайте программу таким образом, чтобы она рассчитывала отношение сигнал/шум (SNR) из сегментированных и замаскированных изображений.
- Используйте сегментированный фон из шага 3.21 в качестве фона (BG) и сегментированную ткань интереса в качестве сигнала (SIG), перебирая каждый пиксель каждого среза со значением больше 0 с двумя вложенными циклами for. Вычислите отношение сигнал/шум по следующей формуле33:
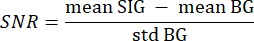
где mean SIG — среднее значение гистограммы сигнала, mean BG — среднее значение фоновой гистограммы, а std BG — стандартное отклонение фоновой гистограммы. - Отбросьте срезы объема со значением SNR ниже 5 из дальнейшего анализа.
- Используйте сегментированный фон из шага 3.21 в качестве фона (BG) и сегментированную ткань интереса в качестве сигнала (SIG), перебирая каждый пиксель каждого среза со значением больше 0 с двумя вложенными циклами for. Вычислите отношение сигнал/шум по следующей формуле33:
- С помощью скрипта Python18,33 можно создать график частоты ферментов на основе карт ферментов для каждого типа тканей путем суммирования и усреднения ферментов по срезам, а затем нормализовать их до объема ткани в 100 мкм³ (рисунок 3K, N).
- Запрограммируйте скрипт таким образом, чтобы он загружал замаскированную карту ферментов.
- Переберите каждый пиксель, вычислите сумму пикселей одного и того же фермента, а затем разделите на анализируемый объем (сумму всех пикселей).
- Сгруппируйте рассчитанную распространенность в их метаболических состояниях и усредните, разделив на количество ферментов в каждой группе. Напишите код, который приписывает метаболические состояния, сгруппированные вокруг ЛДГ, анаэробным гликолизоподобным путям, активность ферментов, сгруппированных вокруг PDH/IDH/GAPDH, аэробному гликолизу/oxPhos-подобным путям, а активность NADPH-оксидазы (активность NOX) окислительному взрыву/окислительному стрессу, используемому для защиты (рис. 3L, O).
- Используйте библиотеку matplotlib из Python для визуализации данных с помощью блочных графиков.
- Рассчитайте активность ткани по замаскированным картам активности (рис. 3F) как среднее значение по каждому объемному срезу для всех пикселей со значением больше 0 с помощью макроса ImageJ, выполнив команду Analyze > Measure.
- Визуализируйте все сгенерированные изображения рядом с помощью ImageJ/FIJI.
Результаты
Используя текущую процедуру NAD(P)H-FLIM 28,29,33 в сочетании с описанным методом фазорного анализа, метаболическую активность и метаболические пути в здоровой и инфицированной двенадцатиперстной кишке измеряли на 6-й, 10-й, 12-й и 14-й день после инфицирования ...
Обсуждение
Критические шаги в рамках протокола происходят во время подготовки и при определении ROI. Волокна частично переваренной пищи представляют собой проблему для визуализации, в основном из-за эндогенной люминесценции волокон, перекрывающихся флуоресценцией NAD(P)H, а также ...
Раскрытие информации
Авторы заявляют об отсутствии конкурирующих финансовых интересов.
Благодарности
Мы благодарим Роберта Гюнтера за отличную техническую поддержку. Финансовая поддержка от Немецкого научно-исследовательского совета (DFG) в рамках грантов SPP2332 HA2542/12-1 (S.H.), NI1167/7-1 (R.A.N.), HA5354/11-1 (A.E.H.) и RA2544/1-1 (S.R.), в рамках гранта SFB1444, P14 (R.A.N., A.E.H.), в рамках гранта HA5354/8-2 (A.E.H.) и в рамках гранта GRK2046 B4 и B5 (S.H., С.Р.) и HA2542/8-1 (S.H.) получили широкое признание. В.Л. получил докторскую степень в Берлинской высшей школе прикладных наук в области медицинской физики/физической инженерии в Берлине.
Материалы
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
Ссылки
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены