JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
선충 감염 중 쥐 장 및 기생충의 대사 분석을 위한 NAD(P)H 형광 수명 이미징
요약
본 프로토콜은 자연 기생충 Heligmosomoides polygyrus에 감염된 이식된 쥐 장의 NAD(P)H 형광 수명 이미징을 설명하며, 이를 통해 숙주 및 기생충 조직 모두에서 공간적으로 해결된 방식으로 대사 과정을 조사할 수 있습니다.
초록
기생충은 일반적으로 숙주의 건강에 부정적인 영향을 미칩니다. 이는 전 세계적으로 감염된 사람이나 동물의 건강에 장기적으로 영향을 미치고 농업 및 사회 경제적 결과에 영향을 미치기 때문에 엄청난 건강 부담을 나타냅니다. 그러나 기생충에 의한 면역 조절 효과가 설명되었으며, 자가면역 질환에 대한 잠재적인 치료 관련성이 있습니다. 숙주와 기생충의 신진대사는 숙주와 기생충 모두의 방어에 기여하고 장에서 선충이 생존할 수 있는 기초가 되지만, 적절한 기술의 부족으로 인해 대체로 연구가 부족한 상태로 남아 있습니다. 우리는 숙주와 기생충 모두의 대사 과정을 공간적으로 해결된 방식으로 연구하기 위해 자연 선충 Heligmosomoides polygyrus 에 감염되는 동안 이식된 쥐 장 조직에 NAD(P)H 형광 수명 이미징을 개발 및 적용했습니다. 종의 맞은편에 보존되는 코엔자임 니코틴아미드 아데닌 디뉴클레오티드(NADH) 및 니코틴아미드 아데닌 디뉴클레오티드 인산염(NADPH)의 형광 수명의 착취는 신진대사 과정을 촉매하는 효소의 결합 상태와 결합 부위에 따라 다릅니다. 가장 풍부하게 발현되는 NAD(P)H 의존성 효소에 초점을 맞추고, 주요 방어 기전인 혐기성 해당작용, 산화적 인산화/호기성 해당작용 및 NOX 기반 산화 폭발과 관련된 대사 경로를 구별하고, 감염 중 숙주와 기생충 사이의 대사 누화를 특성화했습니다.
서문
기생충 감염은 인간의 건강에 큰 부담을 줍니다 1,2. 자가면역 질환의 증가와 기생충 감염의 감소 사이의 상관관계는 산업 국가에서 관찰되었습니다. 기생충은 과도한 숙주 면역 반응을 약화시켜 유익한 효과를 가질 수 있는 것으로 알려져 있습니다. H. polygyrus는 설치류의 장에서 발견되는 천연 기생충이며, 이 기생충은 감염된 숙주에서 조절 T 세포(Treg)의 유도를 통해 숙주의 항기생충 면역 반응을 감소시키는 면역 조절 메커니즘을 유도하는 것으로 알려져 있습니다 3,4,5,6,7,8,9,10,11 . 이러한 조절 메커니즘은 퇴행성 자가면역 질환에서 특히 흥미롭습니다.
숙주와 장 선충 사이의 대사 누화에 대한 분석은 널리 무시되고 있지만, 신진대사는 숙주와 기생충 모두에서 방어, 생존 및 기능을 위해 중요한 역할을 합니다. 우리는 포유류 세포와 조직의 다양한 생리학적 및 병태생리학적 상황에서 이미 널리 사용되는 기술인 이광자 여기(two-photon excitation)에 NADH 및 NADPH 형광 수명 이미징을 적용하고 적용할 것을 제안합니다12 이는 생체 조직의 숙주 및 선충 대사를 상관적으로 조사하기 위함입니다.
NADH(P)H라고 하는 NADH 및 NADPH는 모든 세포 기반 생명체에 보존되어 다양한 대사 경로에서 조효소 역할을 하는 유비쿼터스 분자입니다. 예를 들어, 그들은 에너지 생산, 환원성 생합성 및 NADPH 산화효소 매개 활성산소종(ROS) 생산에 관여하며, 이는 주로 세포 방어 및 세포 통신과 관련이 있습니다 13,14,15,16,17,20. 두 코엔자임은 750nm에서 이광자 여기 시 ~450nm에서 형광을 방출하므로 세포와 조직에서 마커가 없는 대사 이미징을 가능하게 합니다19,21. NADH와 NADPH를 하나의 파장만으로 여기하는 것은 유사하고 다소 넓은 이광자 여기 스펙트럼21로 인해 가능합니다.
코엔자임 NAD(P)H의 형광 수명은 18,21,22,23과 결합하는 효소에 직접적으로 의존합니다. 분자 내 에너지 전달을 허용하는 화학 구조로 인해 여기된 NADH 또는 NADPH 분자는 이완되어 형광 광자를 방출하기 전에 결합 특성에 따라 내부 변환 과정을 통해 효소(촉매)로 에너지를 잃습니다. 이 수명은 효소의 NAD (P) H 결합 부위에 대한 통찰력을 제공하여 19 , 21 , 22 , 23 , 24 , 25 번 일어나는 우선 생화학 반응에 대한 통찰력을 제공합니다. 유리 NADH 및 NADPH 분자의 형광 수명은 ~450ps인 반면, 효소에 결합할 때 형광 수명은 훨씬 더 길고(~2,000ps) 각 효소의 결합 부위에 따라 다릅니다21.
NAD(P)H 연결 과정에 관여하는 효소는 370개 이상입니다. 그러나 가장 풍부한 것만이 현미경의 여기 범위 내에서 결과적인 NAD(P)H 형광 수명에 기여할 수 있습니다. 포유류 세포의 RNASeq 데이터를 사용하여 가장 풍부한 NAD(P)H 의존성 효소를 확인하고 조직 및 세포 샘플에서 생성된 데이터를 해석하기 위해 형광 수명 참조를 생성했습니다18. 따라서, 이 연구는 예를 들어 혐기성 해당작용 대사 경로와 관련된 젖산 탈수소효소(LDH)의 선호 활성과 호기성 해당작용/산화적 인산화 대사 경로에 주로 관여하는 이소시트레이트 탈수소효소(IDH) 및 피루베이트 탈수소효소(PDH) 활성을 구별했습니다16,20. 또한, 산화 파열을 주로 담당하는 효소인 NADPH 산화효소에 대한 NADPH 결합은 세포 내 이러한 효소의 특징적인 위치(멤브레인 결합)와 특히 긴 NADPH 형광 수명(3,650ps)으로 인해 쉽게 분해될 수 있습니다18,24,29,30,32. H. polygyrus의 RNASeq 데이터는 포유류 세포에 대해 생성된 참조가 이 선충에도 적응된 형태로 적용된다는 것을 보여줍니다27.
따라서, 본 연구에서는 H. polygyrus에 감염된 마우스의 갓 이식된 십이지장 샘플에서 NAD(P)H 형광 수명 이미징(FLIM)을 수행하여, 모든 조직에서의 일반적인 대사 활성뿐만 아니라 이미지의 각 픽셀에서 주로 활성화된 효소(즉, NAD(P)H가 특정 위치에서 우선적으로 결합하는 효소). 이러한 실험의 성공 여부는 이식된 장의 정확한 시료 준비, 세포 내 해상도에서 NAD(P)H 형광 수명의 신뢰할 수 있는 실시간 이미징 및 이 프로토콜에서 논의된 바와 같이 표준화된 데이터 평가에 달려 있습니다.
프로토콜
모든 실험은 국가 동물 보호 지침에 따라 수행되었으며 동물 보호를 위한 독일 동물 윤리 위원회(G0176/16 및 G0207/19)의 승인을 받았습니다. 이 프로토콜은 NAD(P)H 형광 수명 이미징 데이터 수집 및 데이터 평가를 설명하며, 이를 통해 천연 쥐 장 선충인 H. polygyrus에 감염되었을 때 숙주 장과 기생충 모두의 일반적인 대사 활동과 특정 대사 경로를 평가할 수 있습니다. 이를 위해 생후 10-12주 된 암컷 C57BL/6 마우스에 200마리의 3기 유충(L3)을 감염시켰습니다. 감염의 다른 시점에서, 감염된 마우스를 희생시키고, 십이지장을 절제하여 앞서 설명한 바와 같이 이미징을 위해 준비하였다33. 감염되지 않은 연령 및 성별이 일치하는 마우스의 십이지장은 대조 목적으로 유사하게 준비되고 이미지화되었습니다. 추가 이미징 및 분석에 필요한 조직 특성을 유지하기 위해 샘플을 이식 직후 처리해야 하며 다음 단계(1.1-1.7단계)를 신속하게 수행해야 합니다(그림 1B).
1. 시료 준비
- 처음에 절제된 십이지장에서 ~1cm 길이의 조각을 자릅니다.
- 작은 페트리 접시 중간에 곧게 펴진 조직 튜브를 속경화 조직 접착제(직경 47mm, 테두리 높이 10mm, 재료 표 참조)로 붙입니다(그림 1A2).
- 마이크로 브러시로 조직 주변의 넓은 영역에 페트리 접시 바닥에 추가 접착제를 얇게 바르십시오(그림 1A, 2).
- 뭉툭한 가위를 사용하여 페트리 접시 바닥 부근에서 물리적으로 고정된 장의 전체 길이를 따라 절개합니다(그림 1A3).
- 뭉툭한 핀셋으로 장을 펴서 abluminal 면이 접착제와 완전히 접촉하도록 합니다. 따라서 창자는 내강 면이 위를 향하도록 접착제에 의해 고정됩니다(그림 1A4).
- 감염된 쥐의 경우 실체현미경(배율 10배)으로 벌레를 세어 감염이 성공했는지 확인합니다(그림 1A5).
- 장이 건조해지지 않도록 아가로스로 장을 밀봉합니다. 여기서, 0.5%-0.9% 아가로스를 사용하여 약 38°C의 저온 융점으로 인해 민감한 장 조직이 연소되는 것을 방지했습니다. 아가로스 1mL를 피펫에 넣고 피펫 팁과 조직 사이의 부드러운 접촉으로 장에 조심스럽게 이슬비를 뿌려 약 0.5mm 두께의 얇은 층이 조직을 완전히 둘러싸도록 합니다(그림 1A6).
- 실온에서 PBS(10%)와 함께 아가로스에 밀봉된 샘플로 페트리 접시를 채우고 뚜껑으로 닫습니다. 샘플을 직접 이미지화하거나 단열 상자에 넣어 얼음 위에 올려 놓고 측정을 위해 대기합니다. 첫 번째 준비된 샘플을 37°C로 설정된 가열판의 현미경 대물렌즈 아래에 놓습니다(그림 1A,8).
2. 이미징
참고: 감염된 건강한 십이지장 조직 샘플에서 NAD(P)H-FLIM을 수행하는 데 사용되는 현미경 시스템은 그림 2 및 재료 표에 나열 및 설명된 장치로 구성됩니다. ImSpector 208을 사용되는 모든 모듈의 제어 소프트웨어로 사용하십시오.
- 초기에 관심 영역(ROI)을 찾으려면 대물렌즈 아래의 페트리 접시에 샘플을 놓고 x-평면과 y-평면을 따라 수동으로 이동하여 광시야 형광 현미경 모드를 사용한 육안 검사로 적절한 ROI를 찾습니다.
- 소프트웨어에서 모드를 전환하여 PMT 감지 또는 TCSPC(Time-Correlated Single-Photon Counting) 감지를 사용하여 이미징 시스템을 이광자 여기(Two-Photon Excitation)로 전환합니다. 환경이 어둡고 진동이 없는지 확인하고, 현미경을 차광 커튼으로 둘러싸고, 가능한 경우 공압식 매달린 광학 테이블을 사용하도록 특히 주의하십시오.
- s가 들어 있는 페트리 접시에 대물 렌즈를 담그십시오.amp렌즈 시스템 아이콘을 클릭하고 마우스 휠을 돌려 z 위치를 변경하여 le.
- 소프트웨어의 파장 패널에 원하는 파장을 입력하여 레이저를 765nm로 조정하고 최대 레이저 출력을 10%로 설정합니다(대물렌즈 아래에서 30mW에 해당). 필요한 경우 레이저 출력을 조정합니다. 조직 광손상과 원치 않는 대사 변화를 방지하기 위해 40%(~30mW에서 100mW) 이하로 측정합니다.
- 소프트웨어에서 z-stage의 단계 크기를 조직 내 765nm에서 현미경의 축 분해능에 해당하는 2μm로 설정합니다.
- 갈보 미러 패널에서 라인 스캐닝 주파수를 400Hz로 설정하면 픽셀 체류 시간이 4.95μs가 됩니다.
- 소프트웨어의 드롭다운 메뉴에서 평균 수치를 선택하여 2-4개의 이미지에 대한 이미지 평균을 설정합니다(즉, 더 부드러운 이미지를 얻기 위해 ROI를 2-4번 스캔). 이러한 방식으로 총 픽셀 체류 시간이 9.9-19.8μs로 증가하여 분석 프로세스에서 더 높은 신호 대 잡음비(SNR)의 이점을 얻을 수 있습니다.
- 소프트웨어의 FOV 패널에서 매개변수를 선택하여 이미지 시야(FOV)를 505픽셀 x 505픽셀(500 x 500μm²)로 설정합니다.
- 각 측정에 대해 TCSPC의 하드웨어 메뉴에 있는 보정 창에서 초기화 를 클릭하여 TCSPC 전자 장치의 최적 기능을 보장하기 위해 모듈 시간을 미리 보정합니다.
- 고속 오실로스코프를 사용하여 Ti:Sa 레이저 펄스 트레인을 감지하도록 고속 포토다이오드를 외부에서 조정합니다.
참고: 포토다이오드의 신호는 여기 펄스와 관련하여 TCSPC에 의해 hPMT 감지 및 광자 계수를 트리거하는 데 사용됩니다. 검출은 Ti:Sa 레이저(80MHz)의 반복률에 따라 12.5ns(즉, 두 개의 연속된 레이저 펄스 사이의 시간)마다 반복됩니다. - 먼저 평균을 1로 설정하고 레이저 출력의 게인을 ~10%로 설정한 다음 비디오 모드를 시작하고( 비디오 버튼 클릭), 렌즈 시스템 아이콘을 다시 클릭하여 신호를 계속 획득할 수 있는 깊이를 검색하고, 마우스 휠을 사용하여 게인을 점진적으로 증가시키면서 샘플로 진행하여 측정 깊이의 범위를 정의합니다.
- 조직의 NAD(P)H-FLIM 이미지의 경우ampFLIM 모드에서 측정된 les, 최대 765mW 공칭 전력에서 100nm로 시스템을 설정합니다. 소프트웨어에서 설정한 97%의 게인에서 하이브리드 PMT를 사용하여 NAD(P)H(466/60nm)의 형광 신호를 감지합니다. TCSPC 모듈은 각각 55ps의 227개의 시간 창(bin)에 걸쳐 광자를 계산합니다.
- 소프트웨어에서 측정하는 동안 표시된 이미지를 마우스 오른쪽 버튼으로 클릭하고 디스플레이 > T-PROFILE 을 선택하여 시스템이 획득한 감쇠 곡선을 시각화합니다.
- 나중에 분석할 수 있도록 감쇠 곡선에서 레이저 펄스 이전의 빈 수를 기록해 두십시오.
- start measurement를 클릭하여 데이터를 수집합니다.
참고: 데이터는 특정 형식과 일치해야 합니다. 임의의 조직 깊이에서 기록된 각 절편은 2μm의 거리만큼 분리되고 500μm x 500μm의 면적을 가지며 절편은 227 x 55ps(그림 3A) 시점에서 500μm x 500μm 절편의 스택으로 획득됩니다. 각 복셀에는 공간적으로 분해된(x-평면 및 y-평면에서) 광자 도착 시간 히스토그램이 포함되어 있습니다. 이것은 형광 붕괴를 나타냅니다(그림 3B). 일반적으로 측정된 부피는 ~500μm x 500μm x 100-300μm 크기의 하이퍼 스택 및 227 빈의 형태여야 하며, 여기서 z 평면의 모든 슬라이스에는 위에서 설명한 대로 시간 종속 강도 데이터가 포함됩니다. 예시적인 측정 데이터 세트의 경우, 이는 (505 x 505 x 227) x 100 픽셀(16비트)이며 약 4GB에 해당합니다.
3. 데이터 분석
참고: NAD(P)H-FLIM 이미지의 위상 분석을 위해 수명을 계산하는 프로그램은 Python33에서 사용자 정의 작성된 코드입니다.
- Spyder IDE에서 Python 3.7 배포판과 함께 Anaconda를 사용합니다( 자료표 참조). 이 코드는 Python의 표준 라이브러리를 사용합니다.
- IDE에 코드를 로드하고 실행합니다. 파일 경로 대화 상자가 열립니다.
- 분석할 원시 데이터가 있는 폴더를 선택합니다. 이 코드는 확인란, 드롭다운 메뉴 및 텍스트 필드가 있는 Tkinter 사용자 입력 대화 상자를 통해 분석하기 전에 세 가지 매개변수를 선택하도록 프로그래밍되어 있습니다.
- 입력 대화 상자에서 오프셋을 선택합니다. 오프셋을 분석되지 않은 시간 스택의 첫 번째 조각 수로 결정합니다.
참고: 매개변수는 여기 펄스 이전의 시점을 자릅니다. 시스템 및 트리거 다이오드를 보정할 때 이 값은 2.14단계에서 설명한 대로 약 5개 슬라이스 또는 레이저 여기 전 데드 빈 수여야 합니다. - 입력 대화 상자의 드롭다운 메뉴에서 페이저 플롯의 표현을 선택합니다. 여기에서 데이터 포인트의 모양(구름 모양 또는 지형)과 반원 시간 축의 축 눈금(효소 또는 시간[ps])을 드롭다운 메뉴에서 옵션으로 선택합니다.
- 확인을 클릭합니다.
- 코드가 NAD(P)H-FLIM z-스택에서 두 가지 유형의 정보를 계산하는지 확인합니다.
- 먼저, 227개의 시간 분해 광자 계수 히스토그램이 강도 투영 이미지(그림 3G)라고 하는 단일 슬라이스에 투영되는 TCSPC 스택의 축소와 지수 붕괴 곡선의 푸리에 기반 분석(Leben et al.18에 의해 설명됨) 및 각 부피 슬라이스의 각 픽셀에 대한 정규화된 실수 및 허수 부분(그림 3C)에서 각 볼륨 슬라이스의 공간 정보를 측정합니다.
- 실제 이미지와 가상 이미지에서 페이저 플롯(그림 3D)과 평균 형광 수명(t) 이미지(각 복셀에 대해 색상으로 구분되고 공간적으로 분해된 평균 감쇠 상수)를 얻습니다(그림 3E).
참고: 소개에서 설명한 바와 같이 효소에 결합할 때 NAD(P)H의 형광 수명은 각 효소에 대한 코엔자임의 결합 부위에 의해 결정됩니다. - 각 픽셀의 위상 벡터와 결합되지 않은 NAD(P)H의 위상 벡터 사이의 벡터를 생성하고 이를 위상 플롯의 반원에 투영하여 대사 활성에 대한 각 효소의 기여도를 결정합니다. 반원은 순수한 화합물에서 형광 수명의 가능한 모든 단일 지수 감소를 나타냅니다.
- 가장 풍부한 NAD(P)H 의존성 효소18,33(보충 그림 1)에 결합된 NAD(P)H의 형광 수명에 대한 이전에 생성된 참조를 사용하여 이러한 효소의 활성화 확률을 계산합니다.
참고: 가장 우세한 효소(즉, 가장 높은 활성화 확률이 계산되는 효소)의 색상 코드가 각 픽셀에 기인하여 효소 맵을 생성합니다(그림 3F).
- 효소 결합 NAD(P)H에 대한 자유(결합되지 않음)의 비율, 특히 450ps의 자유 NAD(P)H와 효소 결합 상태 사이의 위상 플롯의 벡터 비율에서 일반 대사 활성을 0% *결합되지 않은 NAD(P)H만)와 100%(효소 결합 NAD(P)H만) 사이의 백분율로 계산합니다. 각 픽셀에 대사 활성(0%-100%) 값을 부여하여 (색상으로 구분된) 활동 맵이 생성됩니다(그림 3F).
- 결과 맵을 강도 이미지와 오버레이하여 추가적인 형태학적 정보를 얻을 수 있습니다. 여기에서 ImageJ 매크로( Table of Materials(재료 표 참조)를 사용하여 효소 또는 활성 맵의 색조 및 채도를 강도 이미지의 밝기와 오버레이합니다.
- 매크로가 전체 스택에 대해 반복되는지 확인합니다. 스택의 모든 슬라이스(깊이)에 대해 강도 이미지와 선택한 맵을 각각 HUE, SATURATION 및 BRIGHTNESS(HSB 스택> 이미지 > 유형)로 분할해야 합니다.
- 두 HSB 스택의 기본 채널을 분리합니다(이미지 > 스택 > 스택 투 이미지).
- 강도 스택의 HUE 및 SATURATION과 맵 스택의 BRIGHTNESS(close(slicename_HUE), close(slicename_BRIGHTNESS 등)를 닫습니다.
- 나머지 채널을 재결합하여 관심 맵 슬라이스의 HUE 및 SATURATION과 강도 이미지의 BRIGHTNESS(이미지 >는 스택할 > 이미지를 스택함)로 구성된 새로운 HSB 스택을 형성합니다.
- 더 나은 시각적 모양을 위해 이미지 유형을 RGB로 변경합니다(이미지 > 유형 > RGB 색상).
4. 조직 세분화
참고: 사전 훈련된 U-Net 기반 네트워크(ILASTIK, 재료 표 참조)를 사용하여 각각 장 숙주와 선충 조직의 상피 및 층판 및 선충의 높은 NAD(P)H 형광 신호 영역과 낮은 NAD(P)H 형광 신호 영역을 분할합니다.
- ILASTIK을 열고 새 프로젝트와 픽셀 분류를 선택합니다.
- 3.7.1단계에서 이전에 계산된 강도 투영을 Add new > Add separate images를 클릭하여 로드합니다. 여러 측정값의 무작위 슬라이스를 컨테이너로 불러옵니다.
- Feature selection(피처 선택)을 클릭하여 피처> 선택합니다. 시그마 값으로 50(단위 없는 가중치)을 추가하고 활성화할 모든 기능을 선택합니다.
참고: 시그마 값은 기능의 가중치를 결정하지만 기능은 가장자리, 모양, 질감 또는 색상을 인식하는 네트워크의 기능과 클래스를 결정합니다. - Training( 트레이닝)을 클릭하고, 분류할 조직(예: 상피(Epithelium), 배경(Background) 등)에 따라 라벨을 클릭하여 이름을 지정합니다. 레이블을 선택하고 해당 레이블에 해당하는 이미지 내의 모든 픽셀에 색을 지정합니다.
- 다른 레이블에 대해 반복합니다.
- 로드된 데이터 세트에 있는 이미지의 약 절반에 대해 반복합니다.
- Live Update를 클릭하고 모델이 레이블이 지정되지 않은 나머지 이미지의 레이블을 예측하도록 합니다.
- 잘못 추정된 세분화에 레이블을 다시 지정하여 단독으로 예측되고 레이블이 지정되지 않은 이미지의 약 절반을 수정하고 Live Update를 반복합니다.
- 신경망이 원하는 관심 조직을 올바르게 추정하는 방법을 학습할 때까지 이 단계를 반복합니다. 레이블 이름 가까이의 왼쪽에 표시된 네트워크의 신뢰도가 95%에서 98% 사이인지 확인합니다.
- 3.11단계에서 설명한 대로 이번에는 한 측정의 모든 조각이 포함된 새 데이터 세트를 로드하고 Prediction Export를 클릭합니다.
- "Source 드롭다운" 메뉴에서 Simple Segmentation을 선택합니다. "형식"에서 출력을 tiff 형식으로 선택합니다. "Choose Export Image Settings(이미지 내보내기 설정 선택)"에서 출력에 대한 저장 경로를 선택합니다. 내보내기를 클릭합니다.
- 이렇게 하면 관심 있는 조직 유형에 대한 이진 마스크가 생성됩니다. 관심 있는 분절된 조직의 픽셀 값이 1이고 나머지는 픽셀 값이 0인 슬라이스로 구성되므로(그림 3H), 1.1단계 및 1.4-1.7단계에 나열된 대로 생성된 데이터와 이진 마스크를 곱하기만 하면 됩니다. 이로 인해 데이터가 마스킹됩니다(그림 3 I, J, M). 이 단계(곱하기> 이미지 계산기 처리)에는 ImageJ> 사용합니다.
- 각 볼륨 슬라이스에 대해 마스킹된 강도 영상에서 신호 대 잡음비(SNR)를 계산합니다. 이를 위해 ImageJ 매크로를 사용합니다. 분할된 영상과 마스크된 영상에서 신호 대 잡음비(SNR)를 계산하도록 프로그램을 만듭니다.
- 3.21단계의 분할된 배경을 배경(BG)으로 사용하고 두 개의 중첩된 for 루프를 사용하여 0보다 큰 값을 가진 모든 슬라이스의 모든 픽셀을 반복하여 관심 있는 세그먼트 조직(SIG)을 사용합니다. 다음 공식33을 사용하여 SNR을 계산합니다.
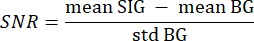
여기서 mean SIG는 신호 히스토그램의 평균값을, mean BG는 백그라운드 히스토그램의 평균값을, std BG는 백그라운드 히스토그램의 표준편차를 나타냅니다. - SNR 값이 5보다 낮은 볼륨 슬라이스는 추가 분석에서 폐기합니다.
- 3.21단계의 분할된 배경을 배경(BG)으로 사용하고 두 개의 중첩된 for 루프를 사용하여 0보다 큰 값을 가진 모든 슬라이스의 모든 픽셀을 반복하여 관심 있는 세그먼트 조직(SIG)을 사용합니다. 다음 공식33을 사용하여 SNR을 계산합니다.
- Python 스크립트18,33을 사용하여 효소를 슬라이스 방식으로 합산하고 평균화한 다음 100μm³ 조직 부피로 정규화하여 각 조직 유형에 대한 효소 맵에서 효소 빈도 그래프를 만듭니다(그림 3K, N).
- masked enzyme map을 로드하도록 스크립트를 코딩합니다.
- 모든 픽셀을 반복하고, 동일한 효소의 픽셀 합을 계산한 다음, 분석된 부피(모든 픽셀의 합계)로 나눕니다.
- 계산된 신진대사 상태의 풍부도를 그룹화하고 각 그룹의 효소 수로 나누어 평균을 구합니다. LDH 주위로 그룹화된 대사 상태를 혐기성 해당작용과 유사한 경로로, PDH/IDH/GAPDH를 중심으로 그룹화된 효소 활성을 호기성 해당작용/옥스포스와 유사한 경로로, NADPH 산화효소 활성(NOX 활성)을 방어에 사용되는 산화 버스트/산화 스트레스에 부여하는 코드를 작성합니다(그림 3L, O).
- Python의 matplotlib 라이브러리를 사용하여 상자 그림으로 데이터를 시각화합니다.
- Analyze > Measure를 실행하여 ImageJ 매크로를 사용하여 값이 0보다 큰 모든 픽셀에 대한 각 볼륨 슬라이스의 평균 값으로 마스킹된 활동 맵(그림 3F)의 조직 활성을 계산합니다.
- 생성된 모든 이미지를 ImageJ/FIJI와 나란히 시각화합니다.
결과
설명된 페이저 분석 방법과 결합된 현재의 NAD(P)H-FLIM 절차 28,29,33을 사용하여 건강한 십이지장과 감염된 십이지장의 대사 활성 및 대사 경로를 쥐 장 선충 H. polygyrus 감염 후 6일, 10일, 12일 및 14일에 측정했습니다.
NAD(P)H-FLIM에 의해 밝혀진 절제된 십이지장에서 장 조직 ...
토론
프로토콜 내의 중요한 단계는 준비 과정과 ROI를 찾을 때 발생합니다. 부분적으로 소화된 식품의 섬유는 주로 NAD(P)H 형광과 겹치는 섬유의 내인성 발광뿐만 아니라 고조파 생성 신호로 인해 이미징에 대한 어려움을 나타냅니다. 대변이 없는 ROI를 찾는 것이 매우 중요합니다. 우리는 배설물이 있는 영역을 측정하지 않는 것을 목표로 했습니다. 세탁은 연약한 융모의 무결성...
공개
저자는 경쟁하는 재정적 이해관계가 없다고 선언합니다.
감사의 말
로버트 귄터(Robert Günther)의 탁월한 기술 지원에 감사드립니다. 그랜트 SPP2332 HA2542/12-1 (S.H.), NI1167/7-1 (R.A.N.), HA5354/11-1 (A.E.H.), RA2544/1-1 (S.R.), 그랜트 SFB1444, P14 (R.A.N., A.E.H.), 그랜트 HA5354/8-2 (A.E.H.), 그리고 그랜트 GRK2046 B4 및 B5 (S.H., S.R.) 및 HA2542/8-1(SH)은 크게 인정받고 있습니다. W.L.은 베를린 응용과학대학(School of Applied Sciences, Berliner Hochschule für Technik)에서 의학 물리학/물리 공학 박사 학위 취득 학위를 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
참고문헌
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유