A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
NAD(P)H הדמיית אורך חיים פלואורסצנטית לניתוח מטבולי של מעי העכברים וטפילים במהלך זיהום בנמטודות
In This Article
Summary
הפרוטוקול הנוכחי מתאר את הדמיית אורך החיים הקרינה של NAD(P)H של מעי עכברי מושתל הנגוע בטפיל הטבעי Heligmosomoides polygyrus, המאפשר לחקור תהליכים מטבוליים הן ברקמות המארח והן ברקמות הטפיל באופן מרחבי.
Abstract
לטפילים יש בדרך כלל השפעה שלילית על בריאות המארח שלהם. הם מהווים נטל בריאותי עצום, מכיוון שהם משפיעים באופן גלובלי על בריאותם של בני אדם או בעלי חיים נגועים בטווח הארוך, ובכך משפיעים על התוצאות החקלאיות והסוציו-אקונומיות. עם זאת, תוארו השפעות ויסות חיסוני המונעות על ידי טפילים, עם רלוונטיות טיפולית פוטנציאלית למחלות אוטואימוניות. בעוד חילוף החומרים הן בפונדקאי והן בטפילים תורם להגנתם ומהווה את הבסיס להישרדות נמטודות במעי, הוא נותר במידה רבה לא נחקר בגלל היעדר טכנולוגיות מתאימות. פיתחנו ויישמנו הדמיית אורך חיים פלואורסצנטית של NAD(P)H על רקמת מעי עכברית מושתלת במהלך זיהום בנמטודה הטבעית Heligmosomoides polygyrus כדי לחקור את התהליכים המטבוליים הן במארח והן בטפילים באופן פתור מרחבי. ניצול אורך החיים הקרינה של הקו-אנזימים ניקוטינמיד אדנין דינוקלאוטיד (NADH) וניקוטינמיד אדנין דינוקלאוטיד פוספט (NADPH), להלן NAD(P)H, שנשמרים בין מינים, תלוי במצב הקישור שלהם ובאתר הקישור באנזימים המזרזים תהליכים מטבוליים. תוך התמקדות באנזימים התלויים ב-NAD(P)H, הובחנו המסלולים המטבוליים הקשורים לגליקוליזה אנאירובית, זרחון חמצוני/גליקוליזה אירובית, והתפרצות חמצונית מבוססת NOX, כמנגנון הגנה עיקרי, ואופיינו את הדיבור המטבולי בין המארח לטפיל במהלך ההדבקה.
Introduction
זיהומים טפיליים מטילים נטל עצום על בריאות האדם 1,2. מתאם בין העלייה במחלות אוטואימוניות לירידה בזיהומים טפיליים נצפה במדינות מתועשות. ידוע כי לטפילים יכולות להיות השפעות מועילות על ידי דיכוי תגובות חיסוניות מוגזמות של המארח. H. polygyrus הוא טפיל טבעי המצוי במעי במכרסמים, וטפיל זה ידוע כגורם למנגנוני ויסות חיסוני המפחיתים את התגובה החיסונית האנטי-טפילית של הפונדקאי באמצעות, בין היתר, השראת תאי T רגולטוריים (Treg) במארח הנגוע 3,4,5,6,7,8,9,10,11 . מנגנוני ויסות אלה מעניינים במיוחד במחלות אוטואימוניות ניווניות.
ניתוח הדיבור המטבולי בין המארח לנמטודות המעי נותר מוזנח באופן נרחב, אם כי חילוף החומרים ממלא תפקיד חשוב הן במארח והן בטפילים להגנה, הישרדות ותפקוד. אנו מציעים להתאים וליישם הדמיית אורך חיים פלואורסצנטית של NADH ו-NADPH על עירור דו-פוטוני, טכנולוגיה שכבר נמצאת בשימוש נרחב במצבים פיזיולוגיים ופתופיזיולוגיים שונים בתאים ורקמות יונקים12, כדי לחקור את חילוף החומרים של המארח והנמטודות ברקמות חיות באופן יחסי.
NADH ו-NADPH, המכונים NAD(P)H, הן מולקולות בכל מקום שנשמרות בכל צורות החיים מבוססות התאים וממלאות את התפקיד של קו-אנזימים במסלולים מטבוליים שונים. לדוגמה, הם מעורבים בייצור אנרגיה, ביוסינתזה רדוקטיבית וייצור מיני חמצן תגובתי בתיווך NADPH אוקסידאז (ROS), הקשורים בעיקר להגנה על התאים ולתקשורת תאית 13,14,15,16,17,20. שני הקו-אנזימים פולטים פלואורסצנטיות ב-~450 ננומטר בעירור דו-פוטוני ב-750 ננומטר, ובכך מאפשרים הדמיה מטבולית ללא סמנים בתאים וברקמות19,21. מרגש גם NADH וגם NADPH עם אורך גל אחד בלבד אפשרי בשל ספקטרום העירור הדו-פוטוני הדומה והרחב למדי שלהם21.
אורך החיים הקרינה של הקו-אנזים NAD(P)H תלוי ישירות באנזים שאליו הוא נקשר 18,21,22,23. בשל המבנה הכימי שלה המאפשר העברת אנרגיה תוך מולקולרית, מולקולת NADH או NADPH הנרגשת מאבדת אנרגיה באמצעות תהליכי המרה פנימיים, בקצב התלוי בתכונות הקישור שלה, לאנזימים (זרז) לפני שהיא נרגעת ופולטת פוטון פלואורסצנטי. אורך חיים זה נותן תובנה לגבי אתר הקישור של NAD(P)H באנזים, וכך, התגובה הביוכימית המועדפת המתרחשת 19,21,22,23,24,25. אורך החיים הקרינה של מולקולות NADH ו-NADPH חופשיות מסתכם ב~450 ps, בעוד שאורך החיים הקרינה שלהן כאשר הן קשורות לאנזים ארוך בהרבה (~2,000 ps) ותלוי באתר הקישור שלהן באנזיםהמתאים 21.
ישנם יותר מ-370 אנזימים המעורבים בתהליכים הקשורים ל-NAD(P)H; עם זאת, רק הנפוצים ביותר יוכלו לתרום לאורך חיי הקרינה של NAD(P)H המתקבל בטווח העירור של המיקרוסקופ. באמצעות נתוני RNASeq מתאי יונקים, זיהינו את האנזימים התלויים ב-NAD(P)H הנפוצים ביותר ויצרנו התייחסות לכל החיים הקרינה כדי לפרש את הנתונים שנוצרו בדגימות רקמה ותאים18. לפיכך, עבודה זו הבחינה למשל בין הפעילות המועדפת של לקטט דהידרוגנאז (LDH), הקשורה למסלולים מטבוליים גליקוליטיים אנאירוביים, לבין פעילות איזוציטראט דהידרוגנאז (IDH) ופירובט דהידרוגנאז (PDH), המעורבים בעיקר במסלולים מטבוליים של גליקוליזה אירובית/זרחון חמצוני16,20. בנוסף, קשירת NADPH ל-NADPH אוקסידאזות, שהם האנזימים האחראים בעיקר להתפרצות חמצונית, ניתנת לפתרון בקלות בשל המיקום האופייני של אנזימים אלה בתא (קשור לממברנה) ובגלל אורך החיים הקרינה הארוך במיוחד של NADPH (3,650 ps)18,24,29,30,32. נתוני RNASeq מ- H. polygyrus מראים כי ההתייחסות שנוצרה לתאי יונקים חלה גם בצורה מותאמת לנמטודה זו27.
לפיכך, בעבודה זו, על ידי ביצוע הדמיית אורך חיים פלואורסצנטית של NAD(P)H (FLIM) בדגימות תריסריון טריות של עכברים הנגועים ב-H. polygyrus, הושג מידע על היחס בין NAD(P)H חופשי לאנזים, שתיאר את הפעילות המטבולית הכללית בכל הרקמות, כמו גם את האנזים הפעיל בעיקר בכל פיקסל של התמונה (כלומר, האנזים שאליו NAD(P)H נקשר באופן מועדף במיקום הספציפי הזה). הצלחת הניסויים הללו מסתמכת על הכנת הדגימה המדויקת של המעי המושתל, הדמיה חיה אמינה של אורך החיים הקרינה של NAD(P)H ברזולוציה תת-תאית, והערכת נתונים סטנדרטית, כפי שנדון בפרוטוקול זה.
Protocol
כל הניסויים בוצעו בהתאם להנחיות הלאומיות להגנת בעלי חיים ואושרו על ידי ועדת האתיקה הגרמנית להגנה על בעלי חיים (G0176/16 ו-G0207/19). הפרוטוקול מתאר איסוף נתוני הדמיה פלואורסצנטית של NAD(P)H והערכת נתונים, המאפשרים להעריך את הפעילות המטבולית הכללית ומסלולים מטבוליים ספציפיים הן במעי המארח והן בטפילים בעת זיהום בנמטודת המעי הטבעית של העכבר, H. polygyrus. למטרה זו, נקבות עכברי C57BL/6 בגילאי 10-12 שבועות נדבקו ב-200 זחלים שלב 3 (L3). בנקודות זמן שונות של ההדבקה, העכברים הנגועים הוקרבו, והתריסריונים נכרתו והוכנו להדמיה כפי שתואר קודם לכן33. התריסריונים של עכברים לא נגועים, תואמי גיל ומין הוכנו באופן דומה וצולמו למטרות בקרה. כדי לשמור על תכונות הרקמה הדרושות להמשך הדמיה וניתוח, יש לעבד את הדגימות מיד לאחר ההשתלה, ולבצע את השלבים הבאים (שלבים 1.1-1.7) במהירות (איור 1B).
1. הכנת מדגם
- חותכים חתיכות באורך ~ 1 ס"מ מהתריסריון שנכרת בתחילה.
- הדביקו את צינור הרקמה המיושר באמצע צלחת פטרי קטנה עם דבק רקמה מהיר (קוטר 47 מ"מ, גובה שפה 10 מ"מ, ראו טבלת חומרים) (איור 1A2).
- מרחו שכבה דקה של דבק נוסף על תחתית צלחת הפטרי על שטח גדול סביב הרקמה בעזרת מיקרו-מברשת (איור 1A2).
- השתמשו במספריים קהים כדי לבצע חתך לכל אורך המעי הקבוע פיזית בקרבת החלק התחתון של צלחת הפטרי (איור 1A3).
- פרש את המעי בפינצטה קהה כך שהצד הבומינלי יבוא במגע מלא עם הדבק. לפיכך, המעי שוכב מקובע על ידי הדבק כשהצד הלומינלי פונה כלפי מעלה (איור 1A4).
- במקרה של עכבר נגוע, ספרו את התולעים תחת סטריאומיקרוסקופ (הגדלה פי 10) כדי לוודא שהזיהום הצליח (איור 1A5).
- אטמו את המעי באגרוז כדי להגן עליו מפני התייבשות. כאן, נעשה שימוש ב-0.5%-0.9% אגרוז כדי להגן על רקמת המעי הרגישה מפני שריפה בשל נקודת ההיתוך שלה בטמפרטורה נמוכה של כ-38 מעלות צלזיוס. משכו 1 מ"ל של אגרוז לתוך פיפטה, וטפטפו בזהירות על המעי במגע עדין בין קצה הפיפטה לרקמה כך ששכבה דקה בעובי של כ-0.5 מ"מ תסגור לחלוטין את הרקמה (איור 1A6).
- מלאו את כלי הפטרי בדגימות אטומות באגרוז עם PBS (10%) בטמפרטורת החדר, ואז סגרו אותן עם המכסה. צלמו את הדגימות ישירות, או הניחו אותן על קרח בקופסה מבודדת תרמית כדי לעמוד בתור למדידה. הנח את הדגימה המוכנה הראשונה מתחת למטרת המיקרוסקופ על צלחת חימום המוגדרת ל-37 מעלות צלזיוס (איור 1A8).
2. הדמיה
הערה: מערכת המיקרוסקופ המשמשת לביצוע NAD(P)H-FLIM בדגימות רקמת תריסריון נגועות ובריאות מורכבת מהמכשירים המפורטים ומתוארים באיור 2 ובטבלת החומרים. השתמש ב-ImSpector 208 כתוכנת הבקרה עבור כל המודולים המשמשים.
- כדי למצוא תחילה את אזור העניין (ROI), הנח את הדגימה בצלחת פטרי מתחת למטרה, ונע לאורך מישור ה-x וה-y ביד כדי למצוא החזר ROI מתאים על ידי בדיקה ויזואלית באמצעות מצב מיקרוסקופיה פלואורסצנטית בשדה רחב.
- העבר את מערכת ההדמיה לעירור דו-פוטוני באמצעות זיהוי PMT או זיהוי ספירת פוטונים בודדים בקורלציה בזמן (TCSPC) על ידי החלפת מצבים בתוכנה. הקפד במיוחד להבטיח שהסביבה חשוכה ונטולת רעידות, לסגור את המיקרוסקופ בווילונות אטומים לאור ולהשתמש בשולחן אופטי תלוי פנאומטי, במידת האפשר.
- טבלו את עדשת האובייקט בצלחת הפטרי המכילה את הדגימה עם תוכנת השליטה על ידי לחיצה על סמל מערכת העדשה וסיבוב גלגל העכבר כדי לשנות את מיקום ה-z.
- כוון את הלייזר ל-765 ננומטר, והגדר את הספק הלייזר המרבי ל-10%, המתאים ל-30 mW מתחת למטרה, על ידי הקלדת אורך הגל הרצוי בלוח אורך הגל של התוכנה. כוונן את עוצמת הלייזר במידת הצורך. מדוד מתחת לרווח של 40% (~30 mW עד 100 mW) כדי למנוע נזק לאור הרקמות ושינויים מטבוליים לא רצויים.
- בתוכנה, הגדר את גודל הצעד של שלב ה-z ל-2 מיקרומטר, המתאים לרזולוציה הצירית של המיקרוסקופ ב-765 ננומטר ברקמה.
- הגדר את תדר סריקת הקו ל-400 הרץ בלוח המראה של galvo, וכתוצאה מכך זמן השתהות פיקסלים של 4.95 מיקרון.
- הגדר את ממוצע התמונה על פני שתיים עד ארבע תמונות על ידי בחירת מספר הממוצע מהתפריט הנפתח בתוכנה (כלומר, סרוק את ההחזר על ההשקעה פעמיים עד ארבע פעמים כדי לקבל תמונות חלקות יותר). באופן זה, זמן השהייה הכולל של הפיקסלים גדל ל-9.9-19.8 מיקרון לטובת יחס אות לרעש (SNR) גבוה יותר בתהליך הניתוח.
- הגדר את שדה הראייה של התמונה (FOV) ל-505 פיקסלים x 505 פיקסלים (500 x 500 מיקרומטר) על ידי בחירת הפרמטרים בחלונית FOV בתוכנה.
- עבור כל מדידה, כייל את זמן המודול מראש כדי להבטיח את התפקוד האופטימלי של האלקטרוניקה TCSPC על ידי לחיצה על אתחול בחלון הכיול בתפריט החומרה של TCSPC.
- כוונן את הפוטו-דיודה המהירה חיצונית כדי לזהות את רכבת פולסי הלייזר Ti:Sa באמצעות אוסצילוסקופ מהיר.
הערה: האות של הפוטו-דיודה משמש להפעלת גילוי hPMT וספירת פוטונים על ידי ה-TCSPC ביחס לפולס העירור. הזיהוי חוזר על עצמו כל 12.5 ns (כלומר, הזמן בין שני פולסי לייזר רצופים), המתאים לקצב החזרה של לייזר Ti:Sa (80 מגה-הרץ). - הגדר את טווח עומק המדידה על ידי הגדרת הממוצע ל-1 והרווח של עוצמת הלייזר ל-~10%, ולאחר מכן התחלת מצב הווידאו (לחץ על כפתור הווידאו ), חיפוש העומק שממנו עדיין ניתן לרכוש את האות על ידי לחיצה נוספת על סמל מערכת העדשות , ושימוש בגלגל העכבר כדי להתקדם לתוך הדגימה תוך הגדלת הרווח בהדרגה.
- עבור תמונות NAD(P)H-FLIM של דגימות הרקמה הנמדדות באופן FLIM, הגדר את המערכת ל- 765 nm בהספק נומינלי מקסימלי של 100 mW. זהה את אות הקרינה של NAD(P)H (466/60 ננומטר) עם ה-PMT ההיברידי ברווח של 97%, המוגדר בתוכנה. מודול TCSPC סופר פוטונים על פני 227 חלונות זמן (פחים), כל אחד של 55 ps.
- לחץ לחיצה ימנית על התמונה המוצגת במהלך המדידה בתוכנה, ובחר תצוגה >-T-PROFILE כדי להמחיש את עקומת הדעיכה שנרכשה על ידי המערכת.
- שימו לב למספר הפחים לפני פולס הלייזר בעקומת הדעיכה לניתוח מאוחר יותר.
- רכוש את הנתונים על ידי לחיצה על התחל מדידה.
הערה: הנתונים חייבים להתאים לתבנית מסוימת. כל פרוסה, שנרשמת בכל עומק רקמה, מופרדת על ידי מרחק של 2 מיקרומטר, ויש לה שטח של 500 מיקרומטר x 500 מיקרומטר, והפרוסות נרכשות כערימה של פרוסות של 500 מיקרומטר x 500 מיקרומטר בנקודות זמן של 227 x 55 ps (איור 3A). כל ווקסל מכיל היסטוגרמה של זמן הגעת פוטונים שנפתרה מרחבית (במישור x ובמישור y). זה מייצג את דעיכת הקרינה (איור 3B). נפח נמדד בדרך כלל חייב להיות בצורת היפר מחסנית עם מידות של ~500 מיקרומטר x 500 מיקרומטר x 100-300 מיקרומטר ו-227 פחים, כאשר כל פרוסה במישור z מכילה את נתוני העוצמה תלויי הזמן כמתואר לעיל. עבור קבוצה מופתית של נתוני מדידה, התוצאה היא (505 x 505 x 227) x 100 פיקסלים (16 סיביות) ומתאימה לכ-4 GB.
3. ניתוח נתונים
הערה: עבור ניתוח הפאזור של תמונות NAD(P)H-FLIM, התוכנית לחישוב אורך החיים היא קוד שנכתב בהתאמה אישית ב-Python33.
- השתמש ב-Anaconda עם הפצת Python 3.7 ב-Spyder IDE (ראה טבלת חומרים). הקוד משתמש בספריות הסטנדרטיות של Pythons.
- טען את הקוד לתוך ה-IDE ובצע. נפתחת תיבת דו-שיח של נתיב קובץ.
- בחר את התיקיה עם הנתונים הגולמיים לניתוח. הקוד מתוכנת לבחור שלושה פרמטרים לפני הניתוח באמצעות תיבת דו-שיח קלט משתמש Tkinter עם תיבות סימון, תפריט נפתח ושדות טקסט.
- בחר היסט בתיבת הדו-שיח של הקלט. קבעו את ההסטה כמספר הפרוסות הראשונות בערימת הזמן שאינן מנותחות.
הערה: הפרמטר חותך את נקודות הזמן לפני דופק העירור. עם כיול המערכת ודיודת ההדק, ערך זה חייב להיות כחמש פרוסות או מספר הפחים המתים לפני עירור לייזר, כמתואר בשלב 2.14. - בחר את הייצוג של עלילות הפאזור בתפריט הנפתח של תיבת הדו-שיח של הקלט. כאן, בחר את המראה של נקודות הנתונים (דמויות ענן או טופוגרפיות), כמו גם את סימני הציר של ציר הזמן של חצי העיגול (אנזימים או זמן [ps]), כאפשרויות מהתפריט הנפתח.
- לחץ על אישור.
- בדוק שהקוד מחשב שני סוגי מידע מערימות ה-z של NAD(P)H-FLIM.
- ראשית, מדדו את המידע המרחבי של כל פרוסת נפח על ידי קריסת ערימות TCSPC, כאשר 227 היסטוגרמות ספירת הפוטונים שנפתרו בזמן מוקרנות על פרוסה אחת הנקראת תמונת הקרנת עוצמה (איור 3G), ומניתוח מבוסס פורייה של עקומות הדעיכה המעריכית (כפי שתואר על ידי Leben et al.18), ומהחלק האמיתי והדמיוני המנורמל עבור כל פיקסל בכל פרוסת נפח (איור 3C).
- מהתמונות האמיתיות והדמיוניות, השיגו את עלילות הפאזור (איור 3D) ואת תמונת אורך החיים הקרינה הממוצעת (t) (קבועי דעיכה ממוצעים מקודדים בצבע, שנפתרו מרחבית עבור כל ווקסל) (איור 3E).
הערה: כפי שמתואר בהקדמה, אורך החיים הקרינה של NAD(P)H, כאשר הוא קשור לאנזימים, נקבע על ידי אתר הקישור של הקו-אנזים לאנזים המתאים. - קבע את תרומתו של האנזים המתאים לפעילות המטבולית על ידי יצירת הווקטור בין וקטור הפאזה של כל פיקסל לווקטור הפאזה של NAD(P)H לא קשור והקרנתו על חצי המעגל בעלילת הפסור. חצי המעגל מייצג את כל הדעיכות החד-מעריכיות האפשריות של תקופות חיים פלואורסצנטיות בתרכובות טהורות .
- שימוש בהתייחסות שנוצרה בעבר של אורך החיים הקרינה של NAD(P)H הקשור לאנזימים התלויים ב-NAD(P)Hהנפוצים ביותר 18,33 (איור משלים 1), ולחשב את ההסתברות להפעלת אנזימים אלה.
הערה: קוד צבע של האנזים הדומיננטי ביותר (כלומר, של האנזים שעבורו מחושבת הסתברות ההפעלה הגבוהה ביותר) מיוחס לכל פיקסל, ובכך מייצר מפת אנזים (איור 3F).
- מהיחס בין NAD(P)H חופשי (לא קשור) ל-NAD(P)H הקשור לאנזים, במיוחד מהיחס הווקטורי בתרשים הפאזור בין NAD(P)H חופשי ב-450 ps למצב הקשור לאנזים, חשב את הפעילות המטבולית הכללית כאחוז בין 0% *רק NAD(P)H לא קשור לאנזים) ל-100% (רק NAD(P)H קשור לאנזים). על ידי ייחוס ערך של פעילות מטבולית (0%-100%) לכל פיקסל, נוצרת מפת פעילות (מקודדת בצבע) (איור 3F).
- שכבו את המפות המתקבלות עם תמונות העוצמה כדי לקבל מידע מורפולוגי נוסף. כאן, השתמש במאקרו ImageJ (ראה טבלת חומרים) כדי לכסות את הגוון והרוויה של האנזים או מפות הפעילות עם הבהירות של תמונות העוצמה.
- ודא שהמאקרו חוזר על עצמו על פני כל המחסנית. עבור כל פרוסה (עומק) בערימה, עליה לפצל את תמונת העוצמה ואת המפה הנבחרת בהתאמה ל-HUE, Saturation ו-BRIGHTNESS (סוג > תמונה > מחסנית HSB).
- הפרד את הערוצים הראשיים לשני אוספי HSB (אוספי > תמונה > אוסף לתמונות).
- סגור את ה-HUE והרוויה של מחסנית העוצמה, כמו גם את ה-BRIGHTNESS של מחסנית המפה (close(slicename_HUE), close(slicename_BRIGHTNESS), ...).
- שלב מחדש את הערוצים הנותרים ליצירת מחסנית HSB חדשה המורכבת מ-HUE ו-Saturation מהפרוסה של המפה המעניינת ומה-BRIGHTNESS מתמונת העוצמה (אוספי > תמונה > תמונות לערימה).
- שנה את סוג התמונה ל- RGB לקבלת מראה חזותי טוב יותר (סוג > תמונה > צבע RGB).
4. פילוח רקמות
הערה: השתמש ברשת מבוססת U-Net מאומנת מראש (ILASTIK, ראה טבלת חומרים) לפילוח מארח המעי ורקמת הנמטודה, בהתאמה, ויתר על כן, האפיתל והלמינה פרופריה במארח ואזורי אות הקרינה הגבוהים של NAD(P)H ואזורי אות הקרינה NAD(P)H נמוכים בנמטודה.
- פתח את ILASTIK ובחר פרויקט חדש וסיווג פיקסלים.
- טען את הקרנת העוצמה שחושבה בעבר משלב 3.7.1 על ידי לחיצה על הוסף > חדש הוסף תמונות נפרדות. טען פרוסות אקראיות ממדידות מרובות לתוך המיכל.
- לחץ על בחירת תכונה > בחר תכונות. הוסף ערך סיגמא של 50 (משקל ללא יחידה) וסמן את כל התכונות כדי שיהיו פעילות.
הערה: בעוד שערך הסיגמא קובע את משקל התכונות, התכונות קובעות את המחלקות ואת היכולת של הרשת לזהות קצוות, צורות, מרקם או צבע. - לחץ על אימון, ותן שמות לתוויות בהתאם לרקמה לפלח (למשל, אפיתל, רקע וכו') על ידי לחיצה עליהן. בחר תווית וצבע את כל הפיקסלים בתוך התמונה המתאימים לתווית זו.
- חזור על הפעולה עבור התוויות האחרות.
- חזור על הפעולה עבור כמחצית מהתמונות במערך הנתונים שנטען.
- לחץ על עדכון חי ותן למודל לחזות את התוויות עבור התמונות הנותרות ללא תווית.
- תקן כמחצית מהתמונות החזויות והלא מסומנות בלבד על-ידי תיוג מחדש של הפילוח שהוערך באופן שגוי, וחזור על העדכון החי.
- חזור על שלב זה עד שהרשת תלמד להעריך נכון את הרקמה הרצויה המעניינת; ודא שהביטחון של הרשת המוצגת בצד שמאל קרוב לשמות התוויות נע בין 95% ל- 98%.
- טען מערך נתונים חדש כמתואר בשלב 3.11, הפעם עם כל הפרוסות ממדידה אחת, ולחץ על ייצוא חיזוי.
- בתפריט הנפתח "מקור", בחר פילוח פשוט; ב"פורמט", בחר את הפלט שיהיה בפורמט tiff; וב"בחר ייצוא הגדרות תמונה", בחר נתיב שמירה עבור הפלט. לחץ על ייצוא.
- זה יוצר מסכות בינאריות לסוג הרקמה המעניין. מכיוון שהם מורכבים מפרוסות שבהן לרקמה המפולחת המעניינת יש ערך פיקסלים של 1 ולשאר יש ערך פיקסלים של 0 (איור 3H), פשוט הכפל את המסכות הבינאריות עם הנתונים שנוצרו כמפורט בשלב 1.1 ובשלבים 1.4-1.7. זה מוביל לנתונים מוסווים (איור 3 I, J, M). השתמש ב-ImageJ עבור שלב זה (עבד > מחשבון תמונה > הכפל).
- עבור כל פרוסת נפח, חשב יחס אות לרעש (SNR) מתמונות העוצמה המוסוות. השתמש במאקרו ImageJ למטרה זו. צור את התוכנית שלך כך שהיא מחשבת את יחס האות לרעש (SNR) מהתמונות המפולחות והמסיכות.
- השתמש ברקע המפולח משלב 3.21 כרקע (BG) וברקמה המפולחת המעניינת כאות (SIG) על ידי איטרציה על כל פיקסל של כל פרוסה עם ערך גדול מ-0 עם שתי לולאות for-מקוננות. חשב את ה- SNR באמצעות הנוסחה הבאה33:
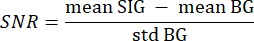
כאשר SIG ממוצע מתייחס לערך הממוצע של היסטוגרמה של האות, ממוצע סוכר מתייחס לערך הממוצע של היסטוגרמה של הרקע, ו-std BG מתייחס לסטיית התקן של היסטוגרמת הרקע. - השלך פרוסות נפח עם ערך SNR נמוך מ-5 מניתוח נוסף.
- השתמש ברקע המפולח משלב 3.21 כרקע (BG) וברקמה המפולחת המעניינת כאות (SIG) על ידי איטרציה על כל פיקסל של כל פרוסה עם ערך גדול מ-0 עם שתי לולאות for-מקוננות. חשב את ה- SNR באמצעות הנוסחה הבאה33:
- עם סקריפט Python18,33, צור גרף תדר אנזים ממפות האנזים עבור כל סוג רקמה על ידי סיכום וממוצע האנזימים בפרוסות ואז נורמליזציה שלהם לנפח רקמה של 100 מיקרומטר מעוקב (איור 3K, N).
- קודד את הסקריפט כך שהוא יטען את מפת האנזים הממוסכה.
- חזור על כל פיקסל, חשב את סכום הפיקסלים של אותו אנזים, ולאחר מכן חלק בנפח המנותח (סך כל הפיקסלים).
- קבץ את השפע המחושב במצבים המטבוליים שלהם, וממוצע על ידי חלוקה במספר האנזימים בכל קבוצה. כתוב קוד המייחס מצבים מטבוליים המקובצים סביב LDH למסלולים דמויי גליקוליזה אנאירובית, פעילות אנזימים מקובצת סביב PDH/IDH/GAPDH למסלולים דמויי גליקוליזה אירובית/oxPhos, ופעילות NADPH oxidase (פעילות NOX) לפרץ חמצוני/מתח חמצוני המשמש להגנה (איור 3L, O).
- השתמש בספריית matplotlib מ-Python כדי לדמיין את הנתונים עם boxplots.
- חשב את פעילות הרקמה ממפות הפעילות עם המסיכה (איור 3F) כערך הממוצע על פני כל פרוסת נפח עבור כל הפיקסלים עם ערך גדול מ-0 באמצעות מאקרו ImageJ על ידי הפעלת Analyze > Measure.
- דמיין את כל התמונות שנוצרו זו לצד זו עם ImageJ/FIJI.
תוצאות
באמצעות הליך NAD(P)H-FLIM הנוכחי 28,29,33 בשילוב עם שיטת ניתוח הפאזור המתוארת, הפעילות המטבולית והמסלולים המטבוליים בתריסריון בריא ונגוע נמדדו ביום 6, יום 10, יום 12 ויום 14 לאחר ההדבקה בנמטודת המעי של העכברים H. polygyrus.
Discussion
השלבים הקריטיים בפרוטוקול מתרחשים במהלך ההכנה ובעת מציאת ההחזר על ההשקעה. סיבים של מזון מעוכל חלקית מהווים אתגר להדמיה, בעיקר בשל הזוהר האנדוגני של הסיבים החופפים לפלואורסצנטיות NAD(P)H, אך גם בשל אות היצירה ההרמוני שלהם. יש חשיבות רבה למצוא החזר ROI נקי מצואה. מטרתנו הייתה ל...
Disclosures
המחברים מצהירים שאין אינטרסים פיננסיים מתחרים.
Acknowledgements
אנו מודים לרוברט גינתר על התמיכה הטכנית המצוינת. תמיכה כספית ממועצת המחקר הגרמנית (DFG) במסגרת מענק SPP2332 HA2542/12-1 (S.H.), NI1167/7-1 (R.A.N.), HA5354/11-1 (A.E.H.), ו-RA2544/1-1 (S.R.), במסגרת מענק SFB1444, P14 (R.A.N., A.E.H.), תחת מענק HA5354/8-2 (A.E.H.), ותחת מענק GRK2046 B4 ו-B5 (S.H., ס.ר.) ו-HA2542/8-1 (S.H.) זוכים להכרה רבה. ו.ל. קיבל מלגת דוקטורט מבית הספר למדעים יישומיים בברלין בפיזיקה רפואית/הנדסה פיזיקלית.
Materials
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
References
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved