Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes de fluorescencia NAD(P)H para el análisis metabólico del intestino murino y los parásitos durante la infección por nematodos
En este artículo
Resumen
El presente protocolo describe la imagen de por vida de fluorescencia de NAD(P)H de un intestino murino explantado infectado con el parásito natural Heligmosomoides polygyrus, lo que permite investigar los procesos metabólicos tanto en los tejidos del huésped como en los del parásito de una manera espacialmente resuelta.
Resumen
Los parásitos generalmente tienen un efecto negativo en la salud de su huésped. Representan una enorme carga para la salud, ya que afectan globalmente a la salud de los seres humanos o animales infestados a largo plazo y, por lo tanto, repercuten en los resultados agrícolas y socioeconómicos. Sin embargo, se han descrito efectos inmunorreguladores impulsados por parásitos, con potencial relevancia terapéutica para enfermedades autoinmunes. Si bien el metabolismo tanto del huésped como de los parásitos contribuye a su defensa y es la base para la supervivencia de los nematodos en el intestino, ha permanecido en gran medida poco estudiado debido a la falta de tecnologías adecuadas. Hemos desarrollado y aplicado imágenes de fluorescencia NAD(P)H a tejido intestinal murino explantado durante la infección con el nematodo natural Heligmosomoides polygyrus para estudiar los procesos metabólicos tanto en el huésped como en los parásitos de una manera espacialmente resuelta. La explotación del tiempo de vida de fluorescencia de las coenzimas nicotinamida adenina dinucleótido (NADH) y nicotinamida adenina dinucleótido fosfato (NADPH), en adelante NAD(P)H, que se conservan en todas las especies, depende de su estado de unión y del sitio de unión a las enzimas que catalizan los procesos metabólicos. Centrándonos en las enzimas dependientes de NAD(P)H más abundantemente expresadas, se distinguieron las vías metabólicas asociadas con la glucólisis anaeróbica, la fosforilación oxidativa/glucólisis aeróbica y el estallido oxidativo basado en NOX, como un mecanismo de defensa importante, y se caracterizó la diafonía metabólica entre el huésped y el parásito durante la infección.
Introducción
Las infecciones parasitarias suponen una enorme carga para la salud humana 1,2. Se ha observado una correlación entre el aumento de las enfermedades autoinmunes y la disminución de las infecciones parasitarias en los países industrializados. Se sabe que los parásitos pueden tener efectos beneficiosos al amortiguar las respuestas inmunitarias excesivas del huésped. H. polygyrus es un parásito natural que se encuentra en el intestino de los roedores, y se sabe que este parásito induce mecanismos inmunorreguladores que reducen la respuesta inmunitaria antiparasitaria del huésped a través, entre otros mecanismos, de la inducción de células T reguladoras (Treg) en el huésped infectado 3,4,5,6,7,8,9,10,11. Esos mecanismos reguladores son especialmente interesantes en las enfermedades autoinmunes degenerativas.
El análisis de la diafonía metabólica entre el huésped y los nematodos intestinales sigue siendo ampliamente descuidado, aunque el metabolismo juega un papel importante tanto en el huésped como en los parásitos para la defensa, la supervivencia y la función. Proponemos adaptar y aplicar imágenes de fluorescencia de NADH y NADPH sobre excitación de dos fotones, una tecnología ya ampliamente utilizada en diferentes situaciones fisiológicas y fisiopatológicas en células y tejidos de mamíferos12, para investigar el metabolismo del huésped y el nematodo en tejidos vivos correlativamente.
El NADH y el NADPH, denominados NAD(P)H, son moléculas ubicuas que se conservan en todas las formas de vida celulares y desempeñan el papel de coenzimas en diversas vías metabólicas. Por ejemplo, están involucrados en la producción de energía, la biosíntesis reductora y la producción de especies reactivas de oxígeno (ROS) mediadas por NADPH oxidasa, que están principalmente relacionadas con la defensa celular y la comunicación celular 13,14,15,16,17,20. Ambas coenzimas emiten fluorescencia a ~450 nm tras la excitación de dos fotones a 750 nm, lo que permite obtener imágenes metabólicas sin marcadores en células y tejidos 19,21. Es posible excitar tanto NADH como NADPH con una sola longitud de onda debido a sus espectros de excitación de dos fotones similares y bastante amplios21.
El tiempo de vida de fluorescencia de la coenzima NAD(P)H depende directamente de la enzima a la que se une 18,21,22,23. Debido a su estructura química que permite la transferencia de energía intramolecular, la molécula de NADH o NADPH excitada pierde energía a través de procesos de conversión interna, a una velocidad que depende de sus propiedades de unión, a las enzimas (catalizador) antes de relajarse y emitir un fotón de fluorescencia. Este tiempo de vida proporciona información sobre el sitio de unión de NAD(P)H en la enzima y, por lo tanto, la reacción bioquímica preferencial que tiene lugar 19,21,22,23,24,25. El tiempo de vida de fluorescencia de las moléculas libres de NADH y NADPH es de ~450 ps, mientras que su tiempo de vida de fluorescencia cuando se unen a una enzima es mucho más largo (~2.000 ps) y depende de su sitio de unión a la enzima21 respectiva.
Hay más de 370 enzimas involucradas en procesos ligados a NAD(P)H; sin embargo, solo los más abundantes podrán contribuir al tiempo de vida de la fluorescencia NAD(P)H resultante dentro del rango de excitación del microscopio. Utilizando datos de RNASeq de células de mamíferos, identificamos las enzimas dependientes de NAD(P)H más abundantes y generamos una referencia de vida útil de fluorescencia para interpretar los datos generados en muestras de tejidos y células18. De este modo, este trabajo distinguió, por ejemplo, entre la actividad preferencial de la lactato deshidrogenasa (LDH), que se asocia con las vías metabólicas glucolíticas anaerobias, y la actividad de la isocitrato deshidrogenasa (IDH) y la piruvato deshidrogenasa (PDH), que están principalmente involucradas en las vías metabólicas de glucólisis aeróbica/fosforilación oxidativa16,20. Además, la unión de NADPH a las NADPH oxidasas, que son las enzimas principales responsables del estallido oxidativo, puede resolverse fácilmente debido a la ubicación característica de estas enzimas en la célula (unidas a la membrana) y debido a la vida útil particularmente larga de la fluorescencia del NADPH (3.650 ps)18,24,29,30,32. Los datos de RNASeq de H. polygyrus muestran que la referencia generada para las células de mamíferos también se aplica en forma adaptada a este nematodo27.
Por lo tanto, en este trabajo, mediante la realización de imágenes de vida útil de fluorescencia de NAD (P) H (FLIM) en muestras de duodeno recién explantadas de ratones infectados con H. polygyrus, se adquirió información sobre la relación entre NAD(P) H libre y unido a enzimas, que representó la actividad metabólica general en todos los tejidos, así como la enzima predominantemente activa en cada píxel de la imagen (es decir, la enzima a la que se une preferentemente el NAD(P)H en esa ubicación específica). El éxito de estos experimentos se basa en la preparación precisa de la muestra del intestino explantado, la obtención de imágenes en vivo fiables del tiempo de vida de la fluorescencia de NAD(P)H a resolución subcelular y la evaluación de datos estandarizada, como se explica en este protocolo.
Protocolo
Todos los experimentos se realizaron de acuerdo con las Directrices Nacionales de Protección Animal y aprobados por el Comité Alemán de Ética Animal para la protección de los animales (G0176/16 y G0207/19). El protocolo describe la adquisición de datos de imágenes de fluorescencia NAD(P)H y la evaluación de datos, lo que permite evaluar la actividad metabólica general y las vías metabólicas específicas tanto en el intestino del huésped como en los parásitos tras la infección con el nematodo intestinal murino natural, H. polygyrus. Para ello, se infectaron ratones hembra C57BL/6 de 10 a 12 semanas de edad con 200 larvas de estadio 3 (L3). En diferentes momentos de la infección, los ratones infectados fueron sacrificados y los duodenos fueron extirpados y preparados para la obtención de imágenes, como se describió anteriormente33. Los duodenos de ratones no infectados, de la misma edad y sexo, se prepararon y tomaron imágenes de manera similar con fines de control. Para mantener las propiedades tisulares necesarias para la obtención de imágenes y análisis posteriores, las muestras deben procesarse inmediatamente después de la explantación, y los siguientes pasos (pasos 1.1-1.7) deben realizarse rápidamente (Figura 1B).
1. Preparación de la muestra
- Corte trozos de ~1 cm de longitud del duodeno inicialmente extirpado.
- Pegue el tubo de tejido enderezado en el centro de una pequeña placa de Petri con pegamento de tejido de curado rápido (47 mm de diámetro, 10 mm de altura del borde, consulte la Tabla de materiales) (Figura 1A2).
- Aplique una capa delgada de pegamento adicional en el fondo de la placa de Petri sobre un área grande alrededor del tejido con un microcepillo (Figura 1A2).
- Utilice unas tijeras romas para hacer una incisión a lo largo de toda la longitud del intestino fijado físicamente en las proximidades del fondo de la placa de Petri (Figura 1A3).
- Despliega el intestino con unas pinzas romas para que el lado abluminal entre en contacto completo con el pegamento. Por lo tanto, el intestino se encuentra fijado por el pegamento con el lado luminal hacia arriba (Figura 1A4).
- En el caso de un ratón infectado, cuente los gusanos bajo un microscopio estereoscópico (aumento 10x) para asegurarse de que la infección fue exitosa (Figura 1A5).
- Sella el intestino con agarosa para protegerlo de la desecación. En este caso, se utilizó agarosa al 0,5%-0,9% para proteger el tejido intestinal sensible de las quemaduras debido a su punto de fusión a baja temperatura de aproximadamente 38 °C. Extraiga 1 ml de agarosa en una pipeta y rocíe cuidadosamente sobre el intestino con un contacto tierno entre la punta de la pipeta y el tejido, de modo que una capa delgada de aproximadamente 0,5 mm de grosor encierre completamente el tejido (Figura 1A6).
- Llene las placas de Petri con las muestras selladas en agarosa con PBS (10%) a temperatura ambiente y luego ciérrelas con la tapa. Tome una imagen de las muestras directamente o colóquelas en hielo en una caja térmicamente aislante para hacer cola para medirlas. Coloque la primera muestra preparada bajo el objetivo del microscopio en una placa calefactora ajustada a 37 °C (Figura 1A8).
2. Imágenes
NOTA: El sistema de microscopio utilizado para realizar NAD(P)H-FLIM en muestras de tejido duodenal infectado y sano consta de los dispositivos enumerados y descritos en la Figura 2 y la Tabla de Materiales. Utilice ImSpector 208 como software de control para todos los módulos utilizados.
- Para encontrar inicialmente la región de interés (ROI), coloque la muestra en la placa de Petri debajo del objetivo y desplácese a lo largo de los planos x e y a mano para encontrar un ROI adecuado mediante inspección visual utilizando el modo de microscopía de fluorescencia de campo amplio.
- Cambie el sistema de imágenes a excitación de dos fotones mediante la detección PMT o la detección de conteo de fotón único correlacionado con el tiempo (TCSPC) cambiando de modo en el software. Tenga especial cuidado de asegurarse de que el entorno esté oscuro y libre de vibraciones, encerrar el microscopio en cortinas a prueba de luz y, si es posible, utilizar una mesa óptica suspendida neumáticamente.
- Sumerja la lente del objetivo en la placa de Petri que contiene la muestra con el software de control haciendo clic en el icono del sistema de lentes y girando la rueda del ratón para modificar la posición z.
- Ajuste el láser a 765 nm y establezca la potencia máxima del láser en 10 %, lo que corresponde a 30 mW debajo del objetivo, escribiendo la longitud de onda deseada en el panel de longitud de onda del software. Ajuste la potencia del láser si es necesario. Mida por debajo de una ganancia del 40% (~30 mW a 100 mW) para evitar el fotodaño tisular y los cambios metabólicos no deseados.
- En el software, establezca el tamaño del paso de la etapa z en 2 μm, que corresponde a la resolución axial del microscopio a 765 nm en tejido.
- Establezca la frecuencia de escaneo de línea en 400 Hz en el panel de espejo galvo, lo que da como resultado un tiempo de permanencia de píxeles de 4,95 μs.
- Establezca el promedio de la imagen en dos a cuatro imágenes eligiendo el número de promedio en el menú desplegable del software (es decir, escanee el ROI de dos a cuatro veces para adquirir imágenes más suaves). De esta manera, el tiempo total de permanencia de los píxeles se incrementa a 9,9-19,8 μs en beneficio de una mayor relación señal-ruido (SNR) en el proceso de análisis.
- Establezca el campo de visión (FOV) de la imagen en 505 píxeles x 505 píxeles (500 x 500 μm²) eligiendo los parámetros en el panel FOV del software.
- Para cada medición, calibrar el tiempo del módulo con anticipación para garantizar el funcionamiento óptimo de la electrónica TCSPC haciendo clic en inicializar en la ventana de calibración en el menú de hardware del TCSPC.
- Ajuste el fotodiodo rápido externamente para detectar el tren de pulsos láser Ti:Sa mediante el uso de un osciloscopio rápido.
NOTA: La señal del fotodiodo se utiliza para activar la detección de hPMT y el conteo de fotones por el TCSPC con respecto al pulso de excitación. La detección se repite cada 12,5 ns (es decir, el tiempo entre dos pulsos láser consecutivos), lo que corresponde a la tasa de repetición del láser Ti:Sa (80 MHz). - Defina el rango de la profundidad de medición ajustando primero el promedio a 1 y la ganancia de la potencia del láser a ~10%, luego iniciando el modo de video (haga clic en el botón de video ), buscando la profundidad desde la cual se puede adquirir la señal haciendo clic nuevamente en el icono del sistema de lentes y usando la rueda del mouse para avanzar en la muestra mientras aumenta la ganancia progresivamente.
- Para las imágenes NAD(P)H-FLIM de las muestras de tejido medidas en el modo FLIM, ajuste el sistema a 765 nm a una potencia nominal máxima de 100 mW. Detecte la señal de fluorescencia de NAD(P)H (466/60 nm) con el PMT híbrido a una ganancia del 97%, que se establece en el software. El módulo TCSPC cuenta fotones en 227 ventanas de tiempo (bins), cada una de 55 ps.
- Haga clic con el botón derecho del ratón en la imagen mostrada durante la medición en el software y elija visualizar > T-PROFILE para visualizar la curva de decaimiento adquirida por el sistema.
- Anote el número de intervalos antes del pulso láser en la curva de decaimiento para un análisis posterior.
- Adquiera los datos haciendo clic en iniciar medición.
NOTA: Los datos deben coincidir con un formato específico. Cada corte, registrado a cualquier profundidad de tejido, está separado por una distancia de 2 μm y tiene un área de 500 μm x 500 μm, y los cortes se adquieren como una pila de cortes de 500 μm x 500 μm a 227 x 55 ps (Figura 3A) puntos de tiempo. Cada vóxel contiene un histograma de tiempo de llegada de fotones resuelto espacialmente (en el plano x y en el plano y). Esto representa el decaimiento de la fluorescencia (Figura 3B). Un volumen medido típicamente debe tener la forma de una hiperpila con dimensiones de ~500 μm x 500 μm x 100-300 μm y 227 bins, donde cada corte en el plano z contiene los datos de intensidad dependientes del tiempo como se describió anteriormente. Para un conjunto ejemplar de datos de medición, esto da como resultado (505 x 505 x 227) x 100 píxeles (16 bits) y corresponde a unos 4 GB.
3. Análisis de datos
NOTA: Para el análisis de fasores de las imágenes NAD(P)H-FLIM, el programa para calcular los tiempos de vida es un código escrito a medida en Python33.
- Utilice Anaconda con una distribución de Python 3.7 en el IDE de Spyder (consulte la Tabla de materiales). El código utiliza las bibliotecas estándar de Python.
- Cargue el código en el IDE y ejecútelo. Se abre un cuadro de diálogo de ruta de archivo.
- Elija la carpeta con los datos sin procesar para analizar. El código está programado para elegir tres parámetros antes del análisis a través de un cuadro de diálogo de entrada de usuario de Tkinter con casillas de verificación, un menú desplegable y campos de texto.
- Elija un desplazamiento en el cuadro de diálogo de entrada. Determine el desplazamiento como el número de los primeros segmentos de la pila de tiempo que no se analizan.
NOTA: El parámetro corta los puntos de tiempo antes del pulso de excitación. Con la calibración del sistema y el diodo de disparo, este valor debe ser de aproximadamente cinco cortes o el número de bins muertos antes de la excitación del láser, como se describe en el paso 2.14. - Elija la representación de los gráficos de fasores dentro del menú desplegable del cuadro de diálogo de entrada. Aquí, elija la apariencia de los puntos de datos (en forma de nube o topográficos), así como las marcas de los ejes del eje de tiempo del semicírculo (enzimas o tiempo [ps]), como opciones del menú desplegable.
- Haga clic en Aceptar.
- Compruebe que el código calcula dos tipos de información a partir de las pilas z de NAD(P)H-FLIM.
- En primer lugar, se mide la información espacial de cada corte de volumen mediante el colapso de las pilas TCSPC, donde los 227 histogramas de conteo de fotones resueltos en el tiempo se proyectan en un solo corte llamado imagen de proyección de intensidad (Figura 3G), y a partir del análisis basado en Fourier de las curvas de decaimiento exponencial (como lo describe Leben et al.18), y de la parte real e imaginaria normalizada para cada píxel en cada corte de volumen (Figura 3C).
- A partir de las imágenes reales e imaginarias, obtenga los diagramas de fasores (Figura 3D) y la imagen de la vida media de la fluorescencia (t) (constantes de decaimiento promedio codificadas por colores, resueltas espacialmente para cada vóxel) (Figura 3E).
NOTA: Como se describe en la introducción, el tiempo de vida de fluorescencia de NAD(P)H, cuando se une a enzimas, está determinado por el sitio de unión de la coenzima a la enzima respectiva. - Determine la contribución de la enzima respectiva a la actividad metabólica generando el vector entre el vector de fase de cada píxel y el vector de fase de NAD(P)H no unido y proyectándolo sobre el semicírculo en el diagrama de fasores. El semicírculo representa todas las posibles desintegraciones monoexponenciales de los tiempos de vida de fluorescencia en compuestos puros.
- Utilizando la referencia previamente generada de los tiempos de vida de fluorescencia de NAD(P)H unido a las enzimas dependientes de NAD(P)H más abundantes18,33 (Figura suplementaria 1), y calcule la probabilidad de la activación de estas enzimas.
NOTA: A cada píxel se le atribuye un código de color de la enzima más dominante (es decir, de la enzima para la que se calcula la mayor probabilidad de activación), produciendo así un mapa de enzimas (Figura 3F).
- A partir de la relación entre el NAD(P)H libre (no unido) y el ligado a la enzima, específicamente a partir de la relación vectorial en el diagrama de fasores entre el NAD(P)H libre a 450 ps y el estado ligado a la enzima, calcule la actividad metabólica general como un porcentaje entre 0% *solo NAD(P)H no unido) y 100% (solo NAD(P)H unido a la enzima). Al atribuir un valor de actividad metabólica (0%-100%) a cada píxel, se genera un mapa de actividad (codificado por colores) (Figura 3F).
- Superponga los mapas resultantes con las imágenes de intensidad para obtener información morfológica adicional. Aquí, use una macro ImageJ (consulte la Tabla de materiales) para superponer el tono y la saturación de los mapas de enzima o actividad con el brillo de las imágenes de intensidad.
- Asegúrese de que la macro itera en toda la pila. Para cada corte (profundidad) de la pila, debe dividir la imagen de intensidad y el mapa de elección respectivamente en TONO, SATURACIÓN y BRILLO (tipo de imagen > > pila HSB).
- Separe los canales principales para ambas pilas HSB (pilas de > de imágenes > pilas de imágenes).
- Cierre el TONO y la SATURACIÓN de la pila de intensidad, así como el BRILLO de la pila del mapa (close(slicename_HUE), close(slicename_BRIGHTNESS), ...).
- Vuelva a combinar los canales restantes para formar una nueva pila HSB que consta de HUE y SATURATION de la porción del mapa de interés y el BRILLO de la imagen de intensidad (Image > apilas > imágenes para apilar).
- Cambie el tipo de imagen a RGB para una mejor apariencia visual (tipo de imagen > > color RGB).
4. Segmentación de tejidos
NOTA: Utilice una red basada en U-Net preentrenada (ILASTIK, consulte la Tabla de materiales) para segmentar el huésped intestinal y el tejido del nematodo, respectivamente, y además, el epitelio y la lámina propia en el huésped y las áreas de señal de fluorescencia de alto NAD(P)H y áreas de señal de fluorescencia de NAD(P)H bajas en el nematodo.
- Abra ILASTIK y elija un nuevo proyecto y clasificación de píxeles.
- Cargue la proyección de intensidad previamente calculada del paso 3.7.1 haciendo clic en Agregar nuevo > Agregar imágenes separadas. Cargue porciones aleatorias de varias medidas en el contenedor.
- Haga clic en Selección de características > seleccionar características. Agregue un valor sigma de 50 (peso sin unidades) y marque todas las funciones para que estén activas.
NOTA: Mientras que el valor sigma determina el peso de las entidades, las entidades determinan las clases y la capacidad de la red para reconocer bordes, formas, texturas o colores. - Haga clic en Entrenamiento y asigne un nombre a las etiquetas de acuerdo con el tejido a segmentar (por ejemplo, Epitelio, Fondo, etc.) haciendo clic en ellas. Seleccione una etiqueta y coloree todos los píxeles de la imagen correspondientes a esa etiqueta.
- Repita para las otras etiquetas.
- Repita el procedimiento para aproximadamente la mitad de las imágenes del conjunto de datos cargado.
- Haga clic en Live Update y deje que el modelo prediga las etiquetas para las imágenes restantes sin etiquetar.
- Corrija aproximadamente la mitad de las imágenes predichas y sin etiquetar volviendo a etiquetar las segmentaciones estimadas falsamente y repita la actualización en vivo.
- Repita este paso hasta que la red haya aprendido a estimar correctamente el tejido de interés deseado; Asegúrese de que la confianza de la red que se muestra a la izquierda cerca de los nombres de las etiquetas oscile entre el 95 % y el 98 %.
- Cargue un nuevo conjunto de datos como se describe en el paso 3.11, esta vez con todos los segmentos de una medición, y haga clic en Exportación de predicción.
- En el menú desplegable "Fuente", elija Segmentación simple; en "Formato", elija la salida para que esté en formato tiff; y en "Elegir configuración de imagen de exportación", seleccione guardar ruta para la salida. Haga clic en exportar.
- Esto crea máscaras binarias para el tipo de tejido de interés. Dado que consisten en cortes donde el tejido segmentado de interés tiene un valor de píxel de 1 y el resto tiene un valor de píxel de 0 (Figura 3H), simplemente multiplique las máscaras binarias con los datos generados como se enumera en el paso 1.1 y los pasos 1.4-1.7. Esto conduce a datos enmascarados (Figura 3 I, J, M). Utilice ImageJ para este paso (Procesar > Calculadora de imágenes > multiplicar).
- Para cada segmento de volumen, calcule una relación señal-ruido (SNR) a partir de las imágenes de intensidad enmascaradas. Utilice una macro ImageJ para ese propósito. Cree el programa de forma que calcule la relación señal-ruido (SNR) a partir de las imágenes segmentadas y enmascaradas.
- Utilice el fondo segmentado del paso 3.21 como fondo (BG) y el tejido de interés segmentado como señal (SIG) iterando sobre cada píxel de cada segmento con un valor mayor que 0 con dos bucles for anidados. Calcule la SNR con la siguiente fórmula33:
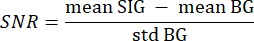
donde mean SIG se refiere al valor medio del histograma de señal, mean BG se refiere al valor medio del histograma de fondo y std BG se refiere a la desviación estándar del histograma de fondo. - Descarte los segmentos de volumen con un valor de SNR inferior a 5 de un análisis posterior.
- Utilice el fondo segmentado del paso 3.21 como fondo (BG) y el tejido de interés segmentado como señal (SIG) iterando sobre cada píxel de cada segmento con un valor mayor que 0 con dos bucles for anidados. Calcule la SNR con la siguiente fórmula33:
- Con el scriptde Python 18,33, cree un gráfico de frecuencia de enzimas a partir de los mapas de enzimas para cada tipo de tejido sumando y promediando las enzimas en cortes y luego normalizándolas a un volumen de tejido de 100 μm³ (Figura 3K, N).
- Codifique el script de modo que cargue el mapa de enzimas enmascarado.
- Iterar sobre cada píxel, calcular la suma de píxeles de la misma enzima y luego dividir por el volumen analizado (el total de todos los píxeles).
- Agrupe las abundancias calculadas en sus estados metabólicos y promedie dividiéndolas por el número de enzimas en cada grupo. Escriba un código que atribuya los estados metabólicos agrupados alrededor de LDH a vías anaerobias similares a la glucólisis, la actividad enzimática agrupada alrededor de PDH/IDH/GAPDH a las vías aerobias de glucólisis/oxPhos, y la actividad de la NADPH oxidasa (actividad NOX) a la explosión oxidativa/estrés oxidativo utilizada para la defensa (Figura 3L, O).
- Utilice la biblioteca matplotlib de Python para visualizar los datos con diagramas de caja.
- Calcule la actividad del tejido a partir de los mapas de actividad enmascarados (Figura 3F) como el valor medio de cada segmento de volumen para todos los píxeles con un valor mayor que 0 utilizando una macro ImageJ mediante la ejecución de Analizar > Medir.
- Visualice todas las imágenes generadas junto con ImageJ/FIJI.
Resultados
Utilizando el procedimiento actual de NAD(P)H-FLIM 28,29,33 combinado con el método de análisis de fasores descrito, se midió la actividad metabólica y las vías metabólicas en duodenos sanos e infectados a los días 6, 10, 12 y 14 después de la infección con el nematodo intestinal murino H. polygyrus.
Viabilidad preservada del tejido intestin...
Discusión
Los pasos críticos dentro del protocolo ocurren durante la preparación y al encontrar el retorno de la inversión. Las fibras de los alimentos parcialmente digeridos representan un desafío para la obtención de imágenes, principalmente debido a la luminiscencia endógena de las fibras que se superponen con la fluorescencia de NAD(P)H, pero también debido a su señal de generación de armónicos. Es de gran importancia encontrar ROIs que estén libres de heces. Nuestro objetivo fue e...
Divulgaciones
Los autores declaran no tener intereses financieros contrapuestos.
Agradecimientos
Agradecemos a Robert Günther por su excelente soporte técnico. Apoyo financiero del Consejo Alemán de Investigación (DFG) en el marco de la subvención SPP2332 HA2542/12-1 (S.H.), NI1167/7-1 (R.A.N.), HA5354/11-1 (A.E.H.) y RA2544/1-1 (S.R.), en el marco de la subvención SFB1444, P14 (R.A.N., A.E.H.), en el marco de la subvención HA5354/8-2 (A.E.H.) y en virtud de la subvención GRK2046 B4 y B5 (S.H., S.R.) y HA2542/8-1 (S.H.) son muy reconocidos. W.L. recibió una beca de doctorado de la Berliner Hochschule für Technik, Escuela de Ciencias Aplicadas, Berlín, en Física Médica/Ingeniería Física.
Materiales
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
Referencias
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados