A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تصوير مدى الحياة الفلوري NAD (P) H للتحليل الأيضي لأمعاء الفئران والطفيليات أثناء عدوى الديدان الخيطية
In This Article
Summary
يصف البروتوكول الحالي التصوير الفلوري NAD (P) H لعمر أمعاء الفئران المزروعة المصابة بالطفيلي الطبيعي Heligmosomoides polygyrus ، والذي يسمح للمرء بالتحقيق في عمليات التمثيل الغذائي في كل من الأنسجة المضيفة والطفيلية بطريقة محلولة مكانيا.
Abstract
الطفيليات بشكل عام لها تأثير سلبي على صحة مضيفها. وهي تمثل عبئا صحيا ضخما، لأنها تؤثر على صحة الإنسان أو الموبوء على الصعيد العالمي، وبالتالي تؤثر على النتائج الزراعية والاجتماعية والاقتصادية. ومع ذلك ، فقد تم وصف التأثيرات التنظيمية المناعية التي يحركها الطفيليات ، مع الأهمية العلاجية المحتملة لأمراض المناعة الذاتية. في حين أن التمثيل الغذائي في كل من المضيف والطفيليات يساهم في دفاعهم وهو أساس بقاء الديدان الخيطية في الأمعاء ، إلا أنه ظل غير مدروس إلى حد كبير بسبب نقص التقنيات الكافية. لقد قمنا بتطوير وتطبيق التصوير الفلوري NAD (P) H على أنسجة الأمعاء المزروعة أثناء الإصابة بالديدان الخيطية الطبيعية Heligmosomoides polygyrus لدراسة عمليات التمثيل الغذائي في كل من المضيف والطفيليات بطريقة محلية محلية. يعتمد استغلال العمر الفلوري للإنزيمات المساعدة نيكوتيناميد الأدينين ثنائي النوكليوتيد (NADH) ونيكوتيناميد الأدينين ثنائي النوكليوتيد الفوسفات (NADPH) ، المشار إليها فيما بعد NAD (P) H ، والتي يتم الحفاظ عليها عبر الأنواع ، على حالة ارتباطها وموقع الارتباط على الإنزيمات التي تحفز عمليات التمثيل الغذائي. مع التركيز على الإنزيمات المعتمدة على NAD (P) H الأكثر تعبيرا عنها بكثرة ، تم تمييز مسارات التمثيل الغذائي المرتبطة بتحلل السكر اللاهوائي ، والفسفرة المؤكسدة / تحلل السكر الهوائي ، والانفجار التأكسدي القائم على أكاسيد النيتروجين ، كآلية دفاع رئيسية ، وتم تمييز الحديث الأيضي بين المضيف والطفيلي أثناء الإصابة.
Introduction
تفرض العدوى الطفيلية عبئا كبيرا على صحة الإنسان1،2. لوحظ وجود علاقة بين ارتفاع أمراض المناعة الذاتية وانخفاض العدوى الطفيلية في البلدان الصناعية. من المعروف أن الطفيليات يمكن أن يكون لها آثار مفيدة عن طريق تثبيط الاستجابات المناعية المفرطة للمضيف. H. polygyrus هو طفيلي طبيعي موجود في الأمعاء في القوارض ، ومن المعروف أن هذا الطفيلي يحفز آليات التنظيم المناعي التي تقلل من الاستجابة المناعية المضادة للطفيليات للمضيف ، من بين آليات أخرى ، تحريض الخلايا التائية التنظيمية (Treg) في المضيف المصاب3،4،5،6،7،8،9،10،11. هذه الآليات التنظيمية ذات أهمية خاصة في أمراض المناعة الذاتية التنكسية.
لا يزال تحليل الحديث المتبادل الأيضي بين المضيف والديدان الخيطية المعوية مهملا على نطاق واسع ، على الرغم من أن التمثيل الغذائي يلعب دورا مهما في كل من المضيف والطفيليات للدفاع والبقاء والوظيفة. نقترح تكييف وتطبيق التصوير الفلوري NADH و NADPH مدى الحياة على إثارة الفوتون ، وهي تقنية مستخدمة بالفعل على نطاق واسع في المواقف الفسيولوجية والفيزيولوجية المرضية المختلفة في خلايا وأنسجة الثدييات12 ، للتحقيق في استقلاب المضيف والديدان الخيطية في الأنسجة الحية بشكل مترابط.
NADH و NADPH ، المشار إليهما باسم NAD (P) H ، هي جزيئات في كل مكان يتم الحفاظ عليها في جميع أشكال الحياة القائمة على الخلايا وتلعب دور الإنزيمات المساعدة في مسارات التمثيل الغذائي المختلفة. على سبيل المثال ، يشاركون في إنتاج الطاقة ، والتخليق الحيوي المختزل ، وإنتاج أنواع الأكسجين التفاعلي بوساطة NADPH أوكسيديز (ROS) ، والتي ترتبط بشكل أساسي بالدفاع الخلوي والاتصال الخلوي13،14،15،16،17،20. ينبعث كلا الإنزيمين المساعدين من التألق عند ~ 450 نانومتر عند إثارة الفوتونين عند 750 نانومتر ، مما يسمح بالتصوير الأيضي الخالي من العلامات في الخلايا والأنسجة19،21. من الممكن إثارة كل من NADH و NADPH بطول موجي واحد فقط بسبب أطياف الإثارة ثنائية الفوتون المتشابهة والواسعةإلى حد ما 21.
يعتمد عمر التألق للإنزيم المساعد NAD (P) H بشكل مباشر على الإنزيم الذي يرتبطبه 18،21،22،23. نظرا لتركيبته الكيميائية التي تسمح بنقل الطاقة داخل الجزيئات ، يفقد جزيء NADH أو NADPH المتحمس الطاقة من خلال عمليات التحويل الداخلية ، بمعدل يعتمد على خصائص الارتباط الخاصة به ، إلى الإنزيمات (المحفز) قبل أن يرتاح وينبعث منه فوتون فلوري. يعطي هذا العمر نظرة ثاقبة لموقع ربط NAD (P) H على الإنزيم ، وبالتالي ، فإن التفاعل الكيميائي الحيوي التفضيلي الذي يحدث19،21،22،23،24،25. يبلغ عمر التألق لجزيئات NADH و NADPH الحرة ~ 450 ps ، في حين أن عمرها المضان عند ربطها بإنزيم يكون أطول بكثير (~ 2,000 ps) ويعتمد على موقع الارتباط على الإنزيمالمعني 21.
هناك أكثر من 370 إنزيما تشارك في العمليات المرتبطة ب NAD (P) H. ومع ذلك ، فإن الأكثر وفرة فقط هي التي ستكون قادرة على المساهمة في عمر مضان NAD (P) H الناتج ضمن نطاق الإثارة للمجهر. باستخدام بيانات RNASeq من خلايا الثدييات ، حددنا الإنزيمات المعتمدة على NAD (P) H الأكثر وفرة وأنشأنا مرجعا لعمر التألق لتفسير البيانات التي تم إنشاؤها في عينات الأنسجة والخلايا18. وبالتالي ، ميز هذا العمل على سبيل المثال بين النشاط التفضيلي لنازعة هيدروجين اللاكتات (LDH) ، والذي يرتبط بمسارات التمثيل الغذائي المحلل للسكر اللاهوائية ، ونشاط نازعة هيدروجين الايزوسترات (IDH) ونازعة هيدروجين البيروفات (PDH) ، والتي تشارك بشكل أساسي في مسارات التمثيل الغذائي لتحلل السكر الهوائي / الفسفرة المؤكسدة16،20. بالإضافة إلى ذلك ، يمكن حل ارتباط NADPH بأكسيدة NADPH ، وهي الإنزيمات المسؤولة بشكل أساسي عن الانفجار التأكسدي ، بسهولة بسبب الموقع المميز لهذه الإنزيمات في الخلية (المرتبطة بالغشاء) وبسبب العمر المضيان الطويل بشكل خاص ل NADPH (3،650 ps) 18،24،29،30،32. تظهر بيانات RNASeq من H. polygyrus أن المرجع الذي تم إنشاؤه لخلايا الثدييات ينطبق أيضا في شكل مكيف على هذه الديدان الخيطية27.
ومن ثم ، في هذا العمل ، من خلال إجراء تصوير العمر المضان NAD (P) H (FLIM) في عينات الاثني عشر المزروعة حديثا للفئران المصابة ب H. polygyrus ، تم الحصول على معلومات عن النسبة بين NAD (P) H الحر والمرتبط بالإنزيم ، والتي تصور النشاط الأيضي العام في جميع الأنسجة ، بالإضافة إلى الإنزيم النشط في الغالب في كل بكسل من الصورة (على سبيل المثال ، الإنزيم الذي يرتبط به NAD (P) H بشكل تفضيلي في هذا الموقع المحدد). يعتمد نجاح هذه التجارب على التحضير الدقيق للعينة من الأمعاء المزروعة ، والتصوير الحي الموثوق به لعمر مضان NAD (P) H بدقة تحت الخلوية ، وتقييم البيانات الموحد ، كما تمت مناقشته في هذا البروتوكول.
Protocol
تم إجراء جميع التجارب وفقا للمبادئ التوجيهية الوطنية لحماية وتمت الموافقة عليها من قبل اللجنة الألمانية لأخلاقيات لحماية (G0176/16 و G0207/19). يصف البروتوكول الحصول على بيانات التصوير مدى الحياة NAD (P) H وتقييم البيانات ، مما يسمح للمرء بتقييم نشاط التمثيل الغذائي العام والمسارات الأيضية المحددة في كل من الأمعاء المضيفة والطفيليات عند الإصابة بالديدان الخيطية المعوية الطبيعية للفئران ، H. polygyrus. لهذا الغرض ، أصيبت إناث الفئران C57BL / 6 التي تتراوح أعمارها بين 10 و 12 أسبوعا ب 200 يرقات من المرحلة 3 (L3). في نقاط زمنية مختلفة من العدوى ، تم التضحية بالفئران المصابة ، وتم استئصال الاثني عشر وإعدادها للتصوير كما هو موضحسابقا 33. تم تحضير الاثني عشر للفئران غير المصابة والمتطابقة مع العمر والجنس وتصويرها بالمثل لأغراض التحكم. للحفاظ على خصائص الأنسجة اللازمة لمزيد من التصوير والتحليل ، يجب معالجة العينات مباشرة بعد الاستخراج ، ويجب تنفيذ الخطوات التالية (الخطوات 1.1-1.7) بسرعة (الشكل 1 ب).
1. تحضير العينة
- قطع قطع بطول ~ 1 سم من الاثني عشر المستأصل في البداية.
- قم بلصق أنبوب المناديل المستقيمة في منتصف طبق بتري صغير بغراء مناديل سريع المعالجة (قطر 47 مم ، ارتفاع حافة 10 مم ، انظر جدول المواد) (الشكل 1 أ2).
- ضع طبقة رقيقة من الغراء الإضافي على قاع طبق بتري على مساحة كبيرة حول الأنسجة بفرشاة دقيقة (الشكل 1 أ2).
- استخدم مقصا غير حاد لعمل شق على طول الأمعاء الثابتة جسديا بالكامل بالقرب من قاع طبق بتري (الشكل 1 أ3).
- افتح الأمعاء بملاقط غير حادة بحيث يتلامس الجانب العظمي تماما مع الغراء. وبالتالي ، يتم تثبيت الأمعاء بواسطة الغراء بحيث يكون الجانب اللمعي متجها لأعلى (الشكل 1 أ4).
- في حالة وجود فأر مصاب ، عد الديدان تحت مجهر مجسم (تكبير 10x) للتأكد من نجاح العدوى (الشكل 1 أ5).
- أغلق الأمعاء بالأغروز لحمايتها من الجفاف. هنا ، تم استخدام 0.5٪ -0.9٪ agarose لحماية الأنسجة المعوية الحساسة من الاحتراق بسبب درجة انصهارها في درجة الحرارة المنخفضة التي تبلغ حوالي 38 درجة مئوية. اسحب 1 مل من الاغاروز في ماصة ، ورشه بعناية على الأمعاء مع ملامسة رقيقة بين طرف الماصة والأنسجة بحيث تحيط طبقة رقيقة بسمك حوالي 0.5 مم بالأنسجة بالكامل (الشكل 1 أ6).
- املأ أطباق بتري بالعينات المختومة في الاغاروز باستخدام PBS (10٪) في درجة حرارة الغرفة ، ثم أغلقها بالغطاء. إما أن تقوم بتصوير العينات مباشرة ، أو وضعها على الجليد في صندوق عازل حراريا للوقوف في قائمة الانتظار للقياس. ضع العينة المحضرة الأولى تحت هدف المجهر على لوحة تسخين مضبوطة على 37 درجة مئوية (الشكل 1 أ8).
2. التصوير
ملاحظة: يتكون نظام المجهر المستخدم لإجراء NAD (P) H-FLIM في عينات أنسجة الاثني عشر المصابة والصحية من الأجهزة المدرجة والموضحة في الشكل 2 وجدول المواد. استخدم ImSpector 208 كبرنامج تحكم لجميع الوحدات النمطية المستخدمة.
- للعثور على منطقة الاهتمام (ROI) في البداية ، ضع العينة في طبق Petri أسفل الهدف ، وتحرك على طول المستوى x و y يدويا للعثور على عائد استثمار مناسب عن طريق الفحص البصري باستخدام وضع الفحص المجهري الفلوري واسع المجال.
- قم بتبديل نظام التصوير إلى إثارة ثنائية الفوتون باستخدام اكتشاف PMT أو اكتشاف عد الفوتون الأحادي المرتبط بالوقت (TCSPC) عن طريق تبديل الأوضاع في البرنامج. احرص بشكل خاص على التأكد من أن البيئة مظلمة وخالية من الاهتزازات ، وإحاطة المجهر بستائر مقاومة للضوء ، واستخدام طاولة بصرية معلقة تعمل بالهواء المضغوط ، إن أمكن.
- اغمر العدسة الموضوعية في طبق Petri الذي يحتوي على العينة باستخدام برنامج التحكم عن طريق النقر على أيقونة نظام العدسة وتدوير عجلة الماوس لتغيير موضع z.
- اضبط الليزر على 765 نانومتر ، واضبط الحد الأقصى لطاقة الليزر على 10٪ ، وهو ما يتوافق مع 30 ميجاوات تحت الهدف ، عن طريق كتابة الطول الموجي المطلوب في لوحة الطول الموجي للبرنامج. اضبط طاقة الليزر إذا لزم الأمر. قم بالقياس أقل من 40٪ (~ 30 ميجاوات إلى 100 ميجاوات) لتجنب التلف الضوئي للأنسجة والتحولات الأيضية غير المرغوب فيها.
- في البرنامج ، اضبط حجم خطوة المرحلة z على 2 ميكرومتر ، وهو ما يتوافق مع الدقة المحورية للمجهر عند 765 نانومتر في الأنسجة.
- اضبط تردد مسح الخط على 400 هرتز في لوحة مرآة galvo ، مما ينتج عنه وقت سكون بكسل يبلغ 4.95 ميكرو ثانية.
- قم بتعيين الصورة بمتوسط أكثر من صورتين إلى أربع صور عن طريق اختيار رقم المتوسط من القائمة المنسدلة في البرنامج (على سبيل المثال ، امسح عائد الاستثمار مرتين إلى أربع مرات للحصول على صور أكثر سلاسة). وبهذه الطريقة، يزداد إجمالي وقت بقاء البكسل إلى 9,9-19,8 ميكرو ثانية لصالح نسبة إشارة إلى ضوضاء أعلى (SNR) في عملية التحليل.
- اضبط مجال رؤية الصورة (FOV) على 505 بكسل × 505 بكسل (500 × 500 ميكرومتر مربع) عن طريق اختيار المعلمات الموجودة في لوحة مجال الرؤية في البرنامج.
- لكل قياس ، قم بمعايرة وقت الوحدة مسبقا لضمان الوظيفة المثلى لإلكترونيات TCSPC بالنقر فوق تهيئة في نافذة المعايرة في قائمة الأجهزة الخاصة ب TCSPC.
- اضبط الصمام الثنائي الضوئي السريع خارجيا للكشف عن قطار نبض الليزر Ti: Sa باستخدام راسم الذبذبات السريع.
ملاحظة: يتم استخدام إشارة الصمام الثنائي الضوئي لتشغيل الكشف عن hPMT وعد الفوتون بواسطة TCSPC فيما يتعلق بنبض الإثارة. يتكرر الكشف كل 12.5 نانوثانية (أي الوقت بين نبضتين ليزريتين متتاليتين) ، بما يتوافق مع معدل تكرار ليزر Ti: Sa (80 ميجاهرتز). - حدد نطاق عمق القياس عن طريق ضبط المتوسط أولا على 1 وكسب طاقة الليزر على ~ 10٪ ، ثم بدء وضع الفيديو (انقر فوق زر الفيديو ) ، والبحث عن العمق الذي لا يزال يمكن الحصول على الإشارة منه عن طريق النقر فوق رمز نظام العدسة مرة أخرى ، واستخدام عجلة الماوس للتقدم في العينة مع زيادة الكسب تدريجيا.
- بالنسبة لصور NAD (P) H-FLIM لعينات الأنسجة المقاسة في وضع FLIM ، اضبط النظام على 765 نانومتر بحد أقصى 100 ميجاوات طاقة اسمية. اكتشف إشارة التألق ل NAD (P) H (466/60 نانومتر) باستخدام PMT الهجين بنسبة 97٪ ، والتي تم تعيينها في البرنامج. تحسب وحدة TCSPC الفوتونات على 227 نافذة زمنية (صناديق) ، كل منها 55 حصا.
- انقر بزر الماوس الأيمن على الصورة المعروضة أثناء القياس في البرنامج ، واختر العرض > T-PROFILE لتصور منحنى الاضمحلال الذي اكتسبه النظام.
- لاحظ عدد الصناديق قبل نبضة الليزر في منحنى الاضمحلال لتحليلها لاحقا.
- احصل على البيانات بالنقر فوق بدء القياس.
ملاحظة: يجب أن تتطابق البيانات مع تنسيق معين. يتم فصل كل شريحة ، مسجلة في أي عمق للأنسجة ، بمسافة 2 ميكرومتر ، وتبلغ مساحتها 500 ميكرومتر × 500 ميكرومتر ، ويتم الحصول على الشرائح على شكل كومة من شرائح 500 ميكرومتر × 500 ميكرومتر عند 227 × 55 نقطة زمنية (الشكل 3 أ). يحتوي كل فوكسل على رسم بياني لوقت وصول الفوتون تم حله مكانيا (في المستوى x والمستوى y). هذا يمثل اضمحلال التألق (الشكل 3 ب). يجب أن يكون الحجم المقاس عادة في شكل مكدس فائق بأبعاد ~ 500 ميكرومتر × 500 ميكرومتر × 100-300 ميكرومتر و 227 صندوقا ، حيث تحتوي كل شريحة في المستوى z على بيانات الشدة المعتمدة على الوقت كما هو موضح أعلاه. بالنسبة لمجموعة نموذجية من بيانات القياس ، ينتج عن ذلك (505 × 505 × 227) × 100 بكسل (16 بت) ويتوافق مع حوالي 4 جيجابايت.
3. تحليل البيانات
ملاحظة: بالنسبة لتحليل الطور لصور NAD (P) H-FLIM ، فإن برنامج حساب العمر الافتراضي هو رمز مكتوب حسب الطلب في Python33.
- استخدم Anaconda مع توزيع Python 3.7 على Spyder IDE (انظر جدول المواد). يستخدم الكود مكتبات Pythons القياسية.
- قم بتحميل التعليمات البرمجية في IDE ، وقم بتنفيذها. يفتح مربع حوار مسار الملف.
- اختر المجلد الذي يحتوي على البيانات الأولية لتحليلها. تمت برمجة الكود لاختيار ثلاث معلمات قبل التحليل من خلال مربع حوار إدخال مستخدم Tkinter مع مربعات الاختيار والقائمة المنسدلة والحقول النصية.
- اختر إزاحة داخل مربع حوار الإدخال. حدد الإزاحة كعدد الشرائح الأولى في المكدس الزمني التي لم يتم تحليلها.
ملاحظة: تقطع المعلمة النقاط الزمنية قبل نبضة الإثارة. مع معايرة النظام والصمام الثنائي الزناد ، يجب أن تكون هذه القيمة حوالي خمس شرائح أو عدد الصناديق الميتة قبل إثارة الليزر ، كما هو موضح في الخطوة 2.14. - اختر تمثيل المخططات الطورية داخل القائمة المنسدلة لمربع حوار الإدخال. هنا ، اختر مظهر نقاط البيانات (الشبيهة بالسحابة أو الطبوغرافية) ، بالإضافة إلى علامات المحور للمحور الزمني نصف الدائرة (الإنزيمات أو الوقت [ps]) ، كخيارات من القائمة المنسدلة.
- انقر فوق موافق.
- تحقق من أن التعليمات البرمجية تحسب نوعين من المعلومات من مكدسات NAD(P)H-FLIM z.
- أولا ، قم بقياس المعلومات المكانية لكل شريحة حجم عن طريق انهيار مداخن TCSPC ، حيث يتم عرض الرسوم البيانية لحساب الفوتون 227 التي تم حلها بمرور الوقت على شريحة واحدة تسمى صورة إسقاط الشدة (الشكل 3G) ، ومن التحليل المستند إلى فورييه لمنحنيات الاضمحلال الأسي (كما هو موضح من قبل Leben et al.18) ، ومن الجزء الحقيقي والخيالي الطبيعي لكل بكسل في كل شريحة حجم (الشكل 3 ج).
- من الصور الحقيقية والخيالية ، احصل على مخططات الطور (الشكل 3D) ومتوسط عمر التألق (t) الصورة (متوسط ثوابت الاضمحلال المرمزة بالألوان والمحلوبة مكانيا لكل فوكسل) (الشكل 3E).
ملاحظة: كما هو موضح في المقدمة ، يتم تحديد العمر الفلوري ل NAD (P) H ، عند ارتباطه بالإنزيمات ، من خلال موقع ارتباط الإنزيم المساعد بالإنزيم المعني. - حدد مساهمة الإنزيم المعني في نشاط التمثيل الغذائي عن طريق توليد المتجه بين متجه الطور لكل بكسل ومتجه الطور ل NAD (P) H غير المرتبط وإسقاطه على نصف الدائرة في مخطط الطور. تمثل نصف الدائرة جميع الاضمحلال الأحادي الأسي المحتمل لعمر التألق في المركبات النقية.
- باستخدام المرجع الذي تم إنشاؤه مسبقا لعمر التألق ل NAD (P) H المرتبط بالإنزيمات المعتمدة على NAD (P) Hالأكثر وفرة 18،33 (الشكل التكميلي 1) ، وحساب احتمالية تنشيط هذه الإنزيمات.
ملاحظة: ينسب رمز اللون للإنزيم الأكثر انتشارا (أي الإنزيم الذي يتم حساب أعلى احتمال تنشيط له) إلى كل بكسل ، وبالتالي إنتاج خريطة إنزيم (الشكل 3F).
- من نسبة NAD (P) H الحرة (غير المرتبطة) إلى الإنزيم ، وتحديدا من النسبة المتجهة في مخطط الطور بين NAD الحر (P) H عند 450 ps والحالة المرتبطة بالإنزيم ، احسب نشاط التمثيل الغذائي العام كنسبة مئوية بين 0٪ * فقط NAD (P) H) غير المرتبط بالإنزيم) و 100٪ (فقط NAD (P) H المرتبط بالإنزيم). من خلال إسناد قيمة نشاط التمثيل الغذائي (0٪ -100٪) إلى كل بكسل ، يتم إنشاء خريطة نشاط (مرمزة بالألوان) (الشكل 3F).
- تراكب الخرائط الناتجة بصور الكثافة للحصول على معلومات مورفولوجية إضافية. هنا ، استخدم ماكرو ImageJ (انظر جدول المواد) لتراكب تدرج اللون وتشبع الإنزيم أو خرائط النشاط مع سطوع صور الشدة.
- تأكد من تكرار الماكرو على المكدس بأكمله. لكل شريحة (عمق) في المكدس، يجب تقسيم صورة الكثافة والخريطة المختارة على التوالي إلى HUE والتشبع والسطوع (نوع > الصورة > مكدس HSB).
- افصل القنوات الرئيسية لكل من مكدسات HSB (تكديس > الصور > التكديس على الصور).
- أغلق HUE والتشبع لمكدس الكثافة، بالإضافة إلى سطوع مكدس الخريطة (close(slicename_HUE)، close(slicename_BRIGHTNESS)، ...).
- أعد تجميع القنوات المتبقية لتشكيل مكدس HSB جديد يتكون من HUE والتشبع من شريحة خريطة الاهتمام والسطوع من صورة الكثافة (تكديس > الصورة > الصور لتكديسها).
- قم بتغيير نوع الصورة إلى RGB للحصول على مظهر مرئي أفضل (نوع > الصورة > لون RGB).
4. تجزئة الأنسجة
ملاحظة: استخدم شبكة قائمة على U-Net مدربة مسبقا (ILASTIK ، انظر جدول المواد) لتجزئة المضيف المعوي وأنسجة الديدان الخيطية ، على التوالي ، وعلاوة على ذلك ، الظهارة والصفيحة المخصوصة في المضيف ومناطق إشارة التألق NAD (P) H العالية ومناطق إشارة مضان NAD (P) H المنخفضة في الديدان الخيطية.
- افتح ILASTIK ، واختر مشروعا جديدا وتصنيفا للبكسل.
- قم بتحميل إسقاط الكثافة المحسوب مسبقا من الخطوة 3.7.1 بالنقر فوق إضافة > جديدة إضافة صور منفصلة. قم بتحميل شرائح عشوائية من قياسات متعددة في الحاوية.
- انقر فوق تحديد الميزة > تحديد الميزات. أضف قيمة سيجما 50 (وزن بدون وحدة)، وحدد جميع الميزات لتكون نشطة.
ملاحظة: بينما تحدد قيمة sigma وزن الميزات، تحدد الميزات الفئات وقدرة الشبكة على التعرف على الحواف أو الأشكال أو النسيج أو اللون. - انقر فوق التدريب ، وقم بتسمية الملصقات وفقا للأنسجة المراد تقسيمها (على سبيل المثال ، الظهارة ، والخلفية ، وما إلى ذلك) بالنقر عليها. حدد تسمية، وقم بتلوين جميع وحدات البكسل داخل الصورة المقابلة لهذا التسمية.
- كرر مع الملصقات الأخرى.
- كرر ما يقرب من نصف الصور في مجموعة البيانات المحملة.
- انقر فوق Live Update ، ودع النموذج يتنبأ بالتسميات للصور المتبقية غير المسماة.
- قم بتصحيح ما يقرب من نصف الصور المتوقعة وغير المسماة فقط عن طريق إعادة تسمية التجزئة المقدرة بشكل خاطئ، وكرر التحديث المباشر.
- كرر هذه الخطوة حتى تتعلم الشبكة تقدير الأنسجة المطلوبة ذات الأهمية بشكل صحيح ؛ تأكد من أن ثقة الشبكة المعروضة على اليسار بالقرب من أسماء الملصقات تتراوح بين 95٪ و 98٪.
- قم بتحميل مجموعة بيانات جديدة كما هو موضح في الخطوة 3.11، هذه المرة مع جميع الشرائح من قياس واحد، وانقر فوق تصدير التنبؤ.
- في القائمة المنسدلة "المصدر"، اختر تجزئة بسيطة. في "التنسيق" ، اختر الإخراج الذي يجب أن يكون بتنسيق tiff ؛ وفي "اختيار إعدادات تصدير الصورة" ، حدد حفظ المسار للإخراج. انقر فوق تصدير.
- هذا يخلق أقنعة ثنائية لنوع الأنسجة ذات الاهتمام. نظرا لأنها تتكون من شرائح حيث يكون للنسيج المجزأ محل الاهتمام قيمة بكسل 1 والباقي له قيمة بكسل تبلغ 0 (الشكل 3H) ، ما عليك سوى ضرب الأقنعة الثنائية بالبيانات التي تم إنشاؤها كما هو موضح في الخطوة 1.1 والخطوات 1.4-1.7. هذا يؤدي إلى بيانات مقنعة (الشكل 3 I ، J ، M). استخدم ImageJ لهذه الخطوة (> ضرب حاسبة الصورة > المعالجة).
- لكل شريحة حجم ، احسب نسبة الإشارة إلى الضوضاء (SNR) من صور الشدة المقنعة. استخدم ماكرو ImageJ لهذا الغرض. قم بإنشاء برنامجك بحيث يحسب نسبة الإشارة إلى الضوضاء (SNR) من الصور المجزأة والمخفية.
- استخدم الخلفية المجزأة من الخطوة 3.21 كخلفية (BG) والأنسجة المجزأة ذات الاهتمام كإشارة (SIG) عن طريق التكرار على كل بكسل من كل شريحة بقيمة أكبر من 0 مع حلقتين متداخلتين ل for. احسب SNR بالصيغة33 التالية:
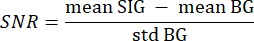
حيث يشير متوسط SIG إلى القيمة المتوسطة للرسم البياني للإشارة ، ويشير متوسط BG إلى القيمة المتوسطة للرسم البياني للخلفية ، ويشير std BG إلى الانحراف المعياري للرسم البياني للخلفية. - تجاهل شرائح وحدة التخزين ذات قيمة SNR أقل من 5 من مزيد من التحليل.
- استخدم الخلفية المجزأة من الخطوة 3.21 كخلفية (BG) والأنسجة المجزأة ذات الاهتمام كإشارة (SIG) عن طريق التكرار على كل بكسل من كل شريحة بقيمة أكبر من 0 مع حلقتين متداخلتين ل for. احسب SNR بالصيغة33 التالية:
- باستخدام برنامج Python النصي18،33 ، قم بإنشاء رسم بياني لتردد الإنزيم من خرائط الإنزيم لكل نوع من أنواع الأنسجة عن طريق جمع الإنزيمات وحسابها حسب الشريحة ثم تطبيعها إلى حجم أنسجة 100 ميكرومتر مكعب (الشكل 3K ، N).
- قم بترميز البرنامج النصي بحيث يقوم بتحميل خريطة الإنزيم المقنعة.
- كرر كل بكسل ، واحسب مجموع وحدات البكسل من نفس الإنزيم ، ثم اقسم على الحجم الذي تم تحليله (إجمالي جميع وحدات البكسل).
- قم بتجميع الوفرة المحسوبة في حالات التمثيل الغذائي ، والمتوسط بالقسمة على عدد الإنزيمات في كل مجموعة. اكتب كودا ينسب حالات التمثيل الغذائي المجمعة حول LDH إلى مسارات تشبه تحلل السكر اللاهوائي ، ونشاط الإنزيم المجمع حول PDH / IDH / GAPDH إلى تحلل السكر الهوائي / المسارات الشبيهة ب oxPhos ، ونشاط NADPH oxidase (نشاط أكاسيد النيتروجين) إلى الانفجار التأكسدي / الإجهاد التأكسدي المستخدم للدفاع (الشكل 3L ، O).
- استخدم مكتبة matplotlib من Python لتصور البيانات باستخدام boxplots.
- احسب نشاط الأنسجة من خرائط النشاط المقنعة (الشكل 3F) كقيمة متوسطة على كل شريحة وحدة تخزين لجميع وحدات البكسل ذات القيمة الأكبر من 0 باستخدام ماكرو ImageJ عن طريق تشغيل Analyze > Measure.
- تصور جميع الصور التي تم إنشاؤها جنبا إلى جنب باستخدام ImageJ / FIJI.
النتائج
باستخدام إجراء NAD (P) H-FLIMالحالي 28،29،33 جنبا إلى جنب مع طريقة تحليل الفاسور الموصوفة ، تم قياس النشاط الأيضي ومسارات التمثيل الغذائي في الاثني عشر السليمة والمصابة في اليوم 6 واليوم 10 واليوم 12 واليوم 14 بعد الإصابة بالديدان ...
Discussion
تحدث الخطوات الحاسمة داخل البروتوكول أثناء الإعداد وعند العثور على عائد الاستثمار. تمثل ألياف الطعام المهضوم جزئيا تحديا للتصوير ، ويرجع ذلك أساسا إلى التلألؤ الداخلي للألياف المتداخلة مع مضان NAD (P) H ، ولكن أيضا بسبب إشارة التوليد التوافقية. من الأهمية بمكان العثور على ...
Disclosures
ويعلن أصحاب البلاغ عدم وجود مصالح مالية متنافسة.
Acknowledgements
نشكر روبرت غونتر على دعمهم الفني الممتاز. الدعم المالي من مجلس البحوث الألماني (DFG) بموجب المنحة SPP2332 HA2542 / 12-1 (S.H.) ، NI1167 / 7-1 (R.A.N.) ، HA5354 / 11-1 (A.E.H.) ، و RA2544 / 1-1 (S.R.) ، بموجب المنحة SFB1444 ، P14 (R.A.N. ، A.E.H.) ، بموجب المنحة HA5354 / 8-2 (A.E.H.) ، وبموجب المنحة GRK2046 B4 و B5 (S.H. ، S.R.) و HA2542 / 8-1 (S.H.) معترف بها إلى حد كبير. حصل WL على زمالة الدكتوراه من Berliner Hochschule für Technik ، كلية العلوم التطبيقية ، برلين في الفيزياء الطبية / الهندسة الفيزيائية.
Materials
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
References
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved