È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging a fluorescenza NAD(P)H per l'analisi metabolica dell'intestino murino e dei parassiti durante l'infezione da nematodi
In questo articolo
Riepilogo
Il presente protocollo descrive l'imaging del tempo di vita della fluorescenza NAD(P)H di un intestino murino espiantato infettato con il parassita naturale Heligmosomoides polygyrus, che consente di studiare i processi metabolici sia nei tessuti dell'ospite che in quelli del parassita in modo spazialmente risolto.
Abstract
I parassiti hanno generalmente un effetto negativo sulla salute del loro ospite. Rappresentano un enorme onere sanitario, in quanto incidono a livello globale sulla salute dell'uomo o dell'animale infestato a lungo termine e, quindi, incidono sui risultati agricoli e socioeconomici. Tuttavia, sono stati descritti effetti immunoregolatori guidati dai parassiti, con potenziale rilevanza terapeutica per le malattie autoimmuni. Sebbene il metabolismo sia nell'ospite che nei parassiti contribuisca alla loro difesa e sia la base per la sopravvivenza dei nematodi nell'intestino, è rimasto in gran parte poco studiato a causa della mancanza di tecnologie adeguate. Abbiamo sviluppato e applicato l'imaging a fluorescenza NAD(P)H al tessuto intestinale murino espiantato durante l'infezione con il nematode naturale Heligmosomoides polygyrus per studiare i processi metabolici sia nell'ospite che nei parassiti in modo spazialmente risolto. Lo sfruttamento del tempo di vita in fluorescenza dei coenzimi nicotinammide adenina dinucleotide (NADH) e nicotinammide adenina dinucleotide fosfato (NADPH), di seguito NAD(P)H, che sono conservati in tutte le specie, dipende dal loro stato di legame e dal sito di legame sugli enzimi che catalizzano i processi metabolici. Concentrandosi sugli enzimi NAD(P)H-dipendenti più abbondantemente espressi, sono state distinte le vie metaboliche associate alla glicolisi anaerobica, alla fosforilazione ossidativa/glicolisi aerobica e al burst ossidativo basato su NOX, come principale meccanismo di difesa, ed è stato caratterizzato il crosstalk metabolico tra l'ospite e il parassita durante l'infezione.
Introduzione
Le infezioni parassitarie impongono un enorme onere alla salute umana 1,2. Nei paesi industrializzati è stata osservata una correlazione tra l'aumento delle malattie autoimmuni e il declino delle infezioni parassitarie. È noto che i parassiti possono avere effetti benefici smorzando le eccessive risposte immunitarie dell'ospite. H. polygyrus è un parassita naturale che si trova nell'intestino nei roditori ed è noto per indurre meccanismi immunoregolatori che riducono la risposta immunitaria antiparassitaria dell'ospite attraverso, tra gli altri meccanismi, l'induzione di cellule T regolatorie (Treg) nell'ospite infetto 3,4,5,6,7,8,9,10,11 . Questi meccanismi regolatori sono particolarmente interessanti nelle malattie autoimmuni degenerative.
L'analisi del crosstalk metabolico tra l'ospite e i nematodi intestinali rimane ampiamente trascurata, sebbene il metabolismo svolga un ruolo importante sia nell'ospite che nei parassiti per la difesa, la sopravvivenza e la funzione. Proponiamo di adattare e applicare l'imaging a fluorescenza NADH e NADPH sull'eccitazione a due fotoni, una tecnologia già ampiamente utilizzata in diverse situazioni fisiologiche e fisiopatologiche in cellule e tessuti di mammifero12, per studiare il metabolismo dell'ospite e del nematode nei tessuti viventi in modo correlato.
NADH e NADPH, indicati come NAD(P)H, sono molecole ubiquitarie che sono conservate in tutte le forme di vita basate sulle cellule e svolgono il ruolo di co-enzimi in varie vie metaboliche. Ad esempio, sono coinvolti nella produzione di energia, nella biosintesi riduttiva e nella produzione di specie reattive dell'ossigeno (ROS) mediate da NADPH ossidasi, che sono principalmente legate alla difesa cellulare e alla comunicazione cellulare 13,14,15,16,17,20. Entrambi i coenzimi emettono fluorescenza a ~450 nm dopo eccitazione a due fotoni a 750 nm, consentendo così l'imaging metabolico privo di marcatori in cellule e tessuti19,21. Eccitare sia il NADH che il NADPH con una sola lunghezza d'onda è possibile a causa dei loro spettri di eccitazione a due fotoni simili e piuttosto ampi21.
Il tempo di vita della fluorescenza del coenzima NAD(P)H dipende direttamente dall'enzima a cui si lega 18,21,22,23. A causa della sua struttura chimica che consente il trasferimento di energia intramolecolare, la molecola eccitata di NADH o NADPH perde energia attraverso processi di conversione interna, ad una velocità che dipende dalle sue proprietà di legame, agli enzimi (catalizzatore) prima di rilassarsi ed emettere un fotone di fluorescenza. Questo tempo di vita fornisce informazioni sul sito di legame del NAD(P)H sull'enzima e, quindi, sulla reazione biochimica preferenziale che avviene 19,21,22,23,24,25. Il tempo di fluorescenza delle molecole libere di NADH e NADPH ammonta a ~450 ps, mentre il loro tempo di fluorescenza quando si legano a un enzima è molto più lungo (~2.000 ps) e dipende dal loro sito di legame sul rispettivo enzima21.
Ci sono più di 370 enzimi coinvolti nei processi legati al NAD(P)H; tuttavia, solo i più abbondanti saranno in grado di contribuire al tempo di vita della fluorescenza del NAD(P)H risultante all'interno dell'intervallo di eccitazione del microscopio. Utilizzando i dati RNASeq provenienti da cellule di mammifero, abbiamo identificato gli enzimi NAD(P)H-dipendenti più abbondanti e abbiamo generato un riferimento del tempo di vita della fluorescenza per interpretare i dati generati in campioni di tessuti e cellule18. In tal modo, questo lavoro ha distinto, ad esempio, tra l'attività preferenziale della lattato deidrogenasi (LDH), che è associata alle vie metaboliche glicolitiche anaerobiche, e l'attività dell'isocitrato deidrogenasi (IDH) e della piruvato deidrogenasi (PDH), che sono principalmente coinvolte nelle vie metaboliche della glicolisi aerobica/fosforilazione ossidativa16,20. Inoltre, il legame del NADPH alle NADPH ossidasi, che sono gli enzimi principalmente responsabili del burst ossidativo, può essere facilmente risolto a causa della posizione caratteristica di questi enzimi nella cellula (legata alla membrana) e a causa della durata di fluorescenza del NADPH particolarmente lunga (3.650 ps)18,24,29,30,32. I dati RNASeq di H. polygyrus mostrano che il riferimento generato per le cellule di mammifero si applica anche in forma adattata a questo nematode27.
Quindi, in questo lavoro, eseguendo l'imaging del tempo di vita della fluorescenza (FLIM) del NAD(P)H in campioni di duodeno appena espiantati di topi infettati da H. polygyrus, sono state acquisite informazioni sul rapporto tra NAD(P)H libero e legato all'enzima, che rappresentava l'attività metabolica generale in tutti i tessuti, così come l'enzima prevalentemente attivo in ogni pixel dell'immagine (ad esempio, l'enzima a cui il NAD(P)H si lega preferenzialmente in quella specifica posizione). Il successo di questi esperimenti si basa sull'accurata preparazione del campione dell'intestino espiantato, sull'affidabile imaging dal vivo del tempo di vita della fluorescenza del NAD(P)H a risoluzione subcellulare e sulla valutazione standardizzata dei dati, come discusso in questo protocollo.
Protocollo
Tutti gli esperimenti sono stati eseguiti in conformità con le linee guida nazionali per la protezione degli animali e approvati dal Comitato tedesco per l'etica degli animali (G0176/16 e G0207/19). Il protocollo descrive l'acquisizione e la valutazione dei dati di imaging a fluorescenza del NAD(P)H, che consentono di valutare l'attività metabolica generale e le vie metaboliche specifiche sia nell'intestino ospite che nei parassiti in seguito all'infezione con il nematode intestinale murino naturale, H. polygyrus. A questo scopo, le femmine di topi C57BL/6 di età compresa tra 10 e 12 settimane sono state infettate con 200 larve di stadio 3 (L3). In diversi momenti dell'infezione, i topi infetti sono stati sacrificati e i duodeni sono stati asportati e preparati per l'imaging come precedentemente descritto33. I duodeni di topi non infetti, di pari età e sesso sono stati preparati e visualizzati in modo simile per scopi di controllo. Per mantenere le proprietà tissutali necessarie per ulteriori immagini e analisi, i campioni devono essere elaborati immediatamente dopo l'espianto e le fasi successive (fasi 1.1-1.7) devono essere eseguite rapidamente (Figura 1B).
1. Preparazione del campione
- Tagliare pezzi di ~1 cm di lunghezza dal duodeno inizialmente asportato.
- Incollare il tubo di tessuto raddrizzato al centro di una piccola piastra di Petri con colla per tessuti a indurimento rapido (diametro 47 mm, altezza del bordo 10 mm, vedere Tabella dei materiali) (Figura 1A2).
- Applicare un sottile strato di colla aggiuntiva sul fondo della capsula di Petri su un'ampia area intorno al tessuto con un micro-pennello (Figura 1A2).
- Utilizzare le forbici smussate per praticare un'incisione lungo l'intera lunghezza dell'intestino fisicamente fissato in prossimità del fondo della capsula di Petri (Figura 1A3).
- Aprire l'intestino con una pinzetta smussata in modo che il lato abluminale entri in completo contatto con la colla. Pertanto, l'intestino giace fissato dalla colla con il lato luminale rivolto verso l'alto (Figura 1A4).
- Nel caso di un topo infetto, contare i vermi al microscopio stereomicroscopio (ingrandimento 10x) per assicurarsi che l'infezione abbia avuto successo (Figura 1A5).
- Sigillare l'intestino con agarosio per proteggerlo dalla disidratazione. Qui, l'agarosio allo 0,5%-0,9% è stato utilizzato per proteggere il tessuto intestinale sensibile dalla combustione a causa del suo punto di fusione a bassa temperatura di circa 38 °C. Aspirare 1 mL di agarosio in una pipetta e versare con cautela sull'intestino con un contatto tenero tra la punta della pipetta e il tessuto in modo che uno strato sottile di circa 0,5 mm di spessore racchiuda completamente il tessuto (Figura 1A6).
- Riempire le piastre di Petri con i campioni sigillati in agarosio con PBS (10%) a temperatura ambiente, quindi chiuderle con il coperchio. Immagini dirette dei campioni o posizionarli sul ghiaccio in una scatola termicamente isolante per metterli in coda per la misurazione. Posizionare il primo campione preparato sotto l'obiettivo del microscopio su una piastra riscaldante impostata a 37 °C (Figura 1A8).
2. Imaging
NOTA: Il sistema di microscopio utilizzato per eseguire NAD(P)H-FLIM in campioni di tessuto duodenale infetto e sano è costituito dai dispositivi elencati e descritti nella Figura 2 e nella Tabella dei materiali. Utilizza ImSpector 208 come software di controllo per tutti i moduli utilizzati.
- Per trovare inizialmente la regione di interesse (ROI), posizionare il campione nella capsula di Petri sotto l'obiettivo e spostarsi manualmente lungo i piani x e y per trovare una ROI adeguata mediante ispezione visiva utilizzando la modalità di microscopia a fluorescenza ad ampio campo.
- Commutare il sistema di imaging sull'eccitazione a due fotoni utilizzando il rilevamento PMT o il rilevamento TCSPC (Time-Related Single-Photon Counting) cambiando le modalità nel software. Prestare particolare attenzione per garantire che l'ambiente sia buio e privo di vibrazioni, per racchiudere il microscopio in tende a prova di luce e, se possibile, per utilizzare un tavolo ottico sospeso pneumatico.
- Immergere la lente dell'obiettivo nella capsula di Petri contenente il campione con il software di controllo facendo clic sull'icona del sistema di lenti e ruotando la rotellina del mouse per modificare la posizione z.
- Sintonizzare il laser su 765 nm e impostare la potenza massima del laser su 10%, che corrisponde a 30 mW sotto l'obiettivo, digitando la lunghezza d'onda desiderata nel pannello della lunghezza d'onda del software. Regolare la potenza del laser, se necessario. Misurare al di sotto di un guadagno del 40% (~30 mW a 100 mW) per evitare fotodanni tissutali e cambiamenti metabolici indesiderati.
- Nel software, impostare la dimensione del passo dello stadio z su 2 μm, che corrisponde alla risoluzione assiale del microscopio a 765 nm nei tessuti.
- Imposta la frequenza di scansione della linea a 400 Hz nel pannello dello specchio galvo, ottenendo un tempo di permanenza dei pixel di 4,95 μs.
- Imposta la media dell'immagine su due o quattro immagini scegliendo il numero di media dal menu a discesa del software (ad esempio, scansiona il ROI da due a quattro volte per acquisire immagini più fluide). In questo modo, il tempo di permanenza totale dei pixel viene aumentato a 9,9-19,8 μs a vantaggio di un rapporto segnale/rumore (SNR) più elevato nel processo di analisi.
- Impostare il campo visivo dell'immagine (FOV) su 505 pixel x 505 pixel (500 x 500 μm²) scegliendo i parametri nel pannello FOV del software.
- Per ogni misurazione, calibrare in anticipo il tempo del modulo per garantire il funzionamento ottimale dell'elettronica TCSPC facendo clic su inizializza nella finestra di calibrazione nel menu hardware del TCSPC.
- Regolare il fotodiodo veloce esternamente per rilevare il treno di impulsi laser Ti:Sa utilizzando un oscilloscopio veloce.
NOTA: Il segnale del fotodiodo viene utilizzato per attivare il rilevamento hPMT e il conteggio dei fotoni da parte del TCSPC rispetto all'impulso di eccitazione. Il rilevamento viene ripetuto ogni 12,5 ns (cioè il tempo tra due impulsi laser consecutivi), corrispondente alla frequenza di ripetizione del laser Ti:Sa (80 MHz). - Definire l'intervallo della profondità di misurazione impostando prima la media su 1 e il guadagno della potenza del laser su ~10%, quindi avviando la modalità video (fare clic sul pulsante video ), cercando la profondità da cui il segnale può ancora essere acquisito facendo nuovamente clic sull'icona del sistema di lenti e utilizzando la rotellina del mouse per avanzare nel campione aumentando progressivamente il guadagno.
- Per le immagini NAD(P)H-FLIM dei campioni di tessuto misurate in modalità FLIM, impostare il sistema su 765 nm a una potenza nominale massima di 100 mW. Rileva il segnale di fluorescenza di NAD(P)H (466/60 nm) con il PMT ibrido con un guadagno del 97%, impostato nel software. Il modulo TCSPC conta i fotoni su 227 finestre temporali (bin), ciascuna di 55 ps.
- Fare clic con il pulsante destro del mouse sull'immagine visualizzata durante la misurazione nel software e scegliere visualizza > T-PROFILE per visualizzare la curva di decadimento acquisita dal sistema.
- Annotare il numero di contenitori prima dell'impulso laser nella curva di decadimento per un'analisi successiva.
- Acquisisci i dati cliccando su avvia misurazione.
NOTA: i dati devono corrispondere a un formato specifico. Ogni fetta, registrata a qualsiasi profondità di tessuto, è separata da una distanza di 2 μm e ha un'area di 500 μm x 500 μm, e le fette vengono acquisite come una pila di fette da 500 μm x 500 μm a 227 x 55 ps (Figura 3A) punti temporali. Ogni voxel contiene un istogramma del tempo di arrivo dei fotoni risolto spazialmente (nel piano x e nel piano y). Questo rappresenta il decadimento della fluorescenza (Figura 3B). Un volume tipicamente misurato deve essere sotto forma di un hyper stack con dimensioni di ~500 μm x 500 μm x 100-300 μm e 227 bin, dove ogni fetta nel piano z contiene i dati di intensità dipendenti dal tempo come descritto sopra. Per un set esemplare di dati di misurazione, il risultato è (505 x 505 x 227) x 100 pixel (16 bit) e corrisponde a circa 4 GB.
3. Analisi dei dati
NOTA: Per l'analisi dei fasori delle immagini NAD(P)H-FLIM, il programma per il calcolo delle durate è un codice scritto su misura in Python33.
- Usa Anaconda con una distribuzione Python 3.7 sull'IDE Spyder (vedi Tabella dei materiali). Il codice utilizza le librerie standard di Python.
- Caricare il codice nell'IDE ed eseguirlo. Si apre una finestra di dialogo del percorso del file.
- Scegli la cartella con i dati grezzi da analizzare. Il codice è programmato per scegliere tre parametri prima dell'analisi attraverso una finestra di dialogo di input dell'utente Tkinter con caselle di controllo, un menu a discesa e campi di testo.
- Scegliere un offset all'interno della finestra di dialogo di input. Determinare l'offset come il numero delle prime sezioni dell'intervallo di tempo che non vengono analizzate.
NOTA: Il parametro taglia i punti temporali prima dell'impulso di eccitazione. Con la calibrazione del sistema e del diodo di attivazione, questo valore deve essere di circa cinque fette o il numero di contenitori morti prima dell'eccitazione laser, come descritto al punto 2.14. - Scegli la rappresentazione dei grafici dei fasori nel menu a discesa della finestra di dialogo di input. Qui, scegli l'aspetto dei punti dati (simili a nuvole o topografici), nonché i segni di spunta dell'asse del tempo a semicerchio (enzimi o tempo [ps]), come opzioni dal menu a discesa.
- Fare clic su OK.
- Verificare che il codice calcoli due tipi di informazioni dagli z-stack NAD(P)H-FLIM.
- In primo luogo, misurare le informazioni spaziali di ogni fetta di volume dal collasso delle pile TCSPC, dove i 227 istogrammi di conteggio dei fotoni risolti nel tempo sono proiettati su una singola fetta chiamata immagine di proiezione dell'intensità (Figura 3G), e dall'analisi basata su Fourier delle curve di decadimento esponenziale (come descritto da Leben et al.18), e dalla parte reale e immaginaria normalizzata per ogni pixel in ogni fetta di volume (Figura 3C).
- Dalle immagini reali e immaginarie, ottenere i grafici dei fasori (Figura 3D) e l'immagine del tempo medio di fluorescenza (t) (costanti di decadimento medie codificate a colori e risolte spazialmente per ciascun voxel) (Figura 3E).
NOTA: Come descritto nell'introduzione, il tempo di vita della fluorescenza del NAD(P)H, quando legato agli enzimi, è determinato dal sito di legame del coenzima al rispettivo enzima. - Determinare il contributo del rispettivo enzima all'attività metabolica generando il vettore tra il vettore di fase di ciascun pixel e il vettore di fase di NAD(P)H non legato e proiettandolo sul semicerchio nel diagramma dei fasori. Il semicerchio rappresenta tutti i possibili decadimenti monoesponenziali dei tempi di vita della fluorescenza nei composti puri.
- Utilizzando il riferimento precedentemente generato dei tempi di vita della fluorescenza di NAD(P)H legato agli enzimi NAD(P)H-dipendenti più abbondanti 18,33 (Figura supplementare 1), e calcolare la probabilità dell'attivazione di questi enzimi.
NOTA: A ciascun pixel viene attribuito un codice colore dell'enzima più dominante (cioè dell'enzima per il quale viene calcolata la più alta probabilità di attivazione), producendo così una mappa enzimatica (Figura 3F).
- Dal rapporto tra il NAD(P)H libero (non legato) e quello legato all'enzima, in particolare dal rapporto vettoriale nel diagramma dei fasori tra il NAD(P)H libero a 450 ps e lo stato legato all'enzima, calcolare l'attività metabolica generale come percentuale compresa tra 0% *solo NAD(P)H non legato) e 100% (solo NAD(P)H legato all'enzima). Attribuendo un valore di attività metabolica (0%-100%) a ciascun pixel, viene generata una mappa dell'attività (codificata a colori) (Figura 3F).
- Sovrapponi le mappe risultanti con le immagini dell'intensità per ottenere ulteriori informazioni morfologiche. In questo caso, utilizzare una macro ImageJ (vedere Tabella dei materiali) per sovrapporre la tonalità e la saturazione dell'enzima o delle mappe di attività con la luminosità delle immagini di intensità.
- Assicurarsi che la macro esegua l'iterazione sull'intero stack. Per ogni fetta (profondità) nella pila, deve dividere l'immagine di intensità e la mappa scelta rispettivamente in HUE, SATURAZIONE e LUMINOSITÀ (tipo di > immagine > pila HSB).
- Separare i canali principali per entrambi gli stack HSB (stack di immagini > > stack di immagini).
- Chiudi la TONALITÀ e la SATURAZIONE della pila di intensità, così come la LUMINOSITÀ della pila di mappe (close(slicename_HUE), close(slicename_BRIGHTNESS), ...).
- Ricombinare i canali rimanenti per formare una nuova pila HSB composta da TONALITÀ e SATURAZIONE dalla fetta della mappa di interesse e LUMINOSITÀ dall'immagine di intensità (Image > stack > immagini da impilare).
- Modificare il tipo di immagine in RGB per un migliore aspetto visivo (Tipo di immagine > > colore RGB).
4. Segmentazione tissutale
NOTA: Utilizzare una rete basata su U-Net pre-addestrata (ILASTIK, vedi Tabella dei materiali) per segmentare rispettivamente l'ospite intestinale e il tessuto del nematode e, inoltre, l'epitelio e la lamina propria nell'ospite e le aree del segnale di fluorescenza ad alto NAD(P)H e le aree del segnale di fluorescenza a basso NAD(P)H nel nematode.
- Apri ILASIK e scegli un nuovo progetto e la classificazione dei pixel.
- Caricare la proiezione dell'intensità calcolata in precedenza dal passaggio 3.7.1 facendo clic su Aggiungi nuovo > Aggiungi immagini separate. Caricare fette casuali da più misurazioni nel contenitore.
- Fare clic su Selezione funzionalità > selezionare le funzionalità. Aggiungi un valore sigma di 50 (peso senza unità) e spunta tutte le funzioni per essere attive.
NOTA: Mentre il valore sigma determina il peso delle feature, le feature determinano le classi e la capacità della rete di riconoscere bordi, forme, texture o colori. - Fare clic su Formazione e denominare le etichette in base al tessuto da segmentare (ad esempio, Epitelio, Sfondo, ecc.) facendo clic su di esse. Selezionate un'etichetta e colorate tutti i pixel all'interno dell'immagine corrispondenti a tale etichetta.
- Ripetere per le altre etichette.
- Ripetere l'operazione per circa la metà delle immagini nel set di dati caricato.
- Fare clic su Aggiornamento in tempo reale e lasciare che il modello preveda le etichette per le immagini rimanenti senza etichetta.
- Correggi circa la metà delle immagini esclusivamente previste e non etichettate rietichettando le segmentazioni erroneamente stimate e ripeti l'aggiornamento in tempo reale.
- Ripetere questo passaggio fino a quando la rete non ha imparato a stimare correttamente il tessuto desiderato di interesse; Assicurarsi che l'affidabilità della rete mostrata a sinistra vicino ai nomi delle etichette sia compresa tra il 95% e il 98%.
- Caricare un nuovo set di dati come descritto nel passaggio 3.11, questa volta con tutte le sezioni di una misurazione, e fare clic su Esportazione previsione.
- Nel menu a discesa "Sorgente", scegli Segmentazione semplice; in "Formato", scegli l'output che deve essere in formato tiff; e in "Scegli impostazioni immagine di esportazione", seleziona il percorso di salvataggio per l'output. Fare clic su esporta.
- In questo modo vengono create maschere binarie per il tipo di tessuto di interesse. Poiché sono costituite da fette in cui il tessuto segmentato di interesse ha un valore di pixel pari a 1 e il resto ha un valore di pixel pari a 0 (Figura 3H), è sufficiente moltiplicare le maschere binarie con i dati generati come elencato nei passaggi 1.1 e 1.4-1.7. Questo porta a dati mascherati (Figura 3 I, J, M). Utilizzare ImageJ per questo passaggio (Elabora > calcolatore di immagini > moltiplica).
- Per ogni fetta di volume, calcolare un rapporto segnale/rumore (SNR) dalle immagini di intensità mascherate. A tale scopo, utilizzare una macro ImageJ. Create il vostro programma in modo che calcoli il rapporto segnale/rumore (SNR) dalle immagini segmentate e mascherate.
- Utilizzare lo sfondo segmentato del passaggio 3.21 come sfondo (BG) e il tessuto segmentato di interesse come segnale (SIG) iterando su ogni pixel di ogni sezione con un valore maggiore di 0 con due cicli for nidificati. Calcola l'SNR con la seguente formula33:
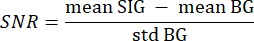
dove SIG medio si riferisce al valore medio dell'istogramma del segnale, BG medio si riferisce al valore medio dell'istogramma di fondo e glicemia std si riferisce alla deviazione standard dell'istogramma di fondo. - Scartare le sezioni di volume con un valore SNR inferiore a 5 da ulteriori analisi.
- Utilizzare lo sfondo segmentato del passaggio 3.21 come sfondo (BG) e il tessuto segmentato di interesse come segnale (SIG) iterando su ogni pixel di ogni sezione con un valore maggiore di 0 con due cicli for nidificati. Calcola l'SNR con la seguente formula33:
- Con lo script Python18,33, creare un grafico enzima-frequenza dalle mappe enzimatiche per ogni tipo di tessuto sommando e facendo la media degli enzimi a fette e quindi normalizzandoli a un volume di tessuto di 100 μm³ (Figura 3K, N).
- Codificare lo script in modo che carichi la mappa enzimatica mascherata.
- Ripeti su ogni pixel, calcola la somma dei pixel dello stesso enzima e poi dividi per il volume analizzato (il totale di tutti i pixel).
- Raggruppa le abbondanze calcolate nei loro stati metabolici e fai la media dividendo per il numero di enzimi in ciascun gruppo. Scrivere codice che attribuisca gli stati metabolici raggruppati intorno a LDH a percorsi simili alla glicolisi anaerobica, l'attività enzimatica raggruppata intorno a PDH/IDH/GAPDH a percorsi aerobici/oxPhos-like e l'attività della NADPH ossidasi (attività NOX) allo stress ossidativo utilizzato per la difesa (Figura 3L, O).
- Usa la libreria matplotlib di Python per visualizzare i dati con i boxplot.
- Calcolare l'attività del tessuto dalle mappe di attività mascherate (Figura 3F) come valore medio su ciascuna fetta di volume per tutti i pixel con un valore maggiore di 0 utilizzando una macro ImageJ eseguendo Analizza > Misura.
- Visualizza tutte le immagini generate fianco a fianco con ImageJ/FIJI.
Risultati
Utilizzando l'attuale procedura NAD(P)H-FLIM 28,29,33 combinata con il metodo di analisi dei fasori descritto, l'attività metabolica e le vie metaboliche nei duodeni sani e infetti sono state misurate al giorno 6, al giorno 10, al giorno 12 e al giorno 14 dopo l'infezione con il nematode intestinale murino H. polygyrus.
Preservata vitalità del tess...
Discussione
I passaggi critici all'interno del protocollo si verificano durante la preparazione e quando si trova il ROI. Le fibre di cibo parzialmente digerito rappresentano una sfida per l'imaging, principalmente a causa della luminescenza endogena delle fibre che si sovrappongono alla fluorescenza NAD(P)H, ma anche a causa del loro segnale di generazione armonica. È di grande importanza trovare ROI privi di feci. Abbiamo cercato di evitare di misurare le aree contenenti feci. Il lavaggio è stat...
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Ringraziamo Robert Günther per l'eccellente supporto tecnico. Sostegno finanziario del Consiglio tedesco della ricerca (DFG) nell'ambito delle sovvenzioni SPP2332 HA2542/12-1 (S.H.), NI1167/7-1 (R.A.N.), HA5354/11-1 (A.E.H.) e RA2544/1-1 (S.R.), nell'ambito della sovvenzione SFB1444, P14 (R.A.N., A.E.H.), della sovvenzione HA5354/8-2 (A.E.H.) e delle sovvenzioni GRK2046 B4 e B5 (S.H., S.R.) e HA2542/8-1 (S.H.) sono molto riconosciuti. W.L. ha ricevuto una borsa di dottorato presso la Berliner Hochschule für Technik, School of Applied Sciences, Berlino, in Fisica Medica/Ingegneria Fisica.
Materiali
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
Riferimenti
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon