このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
線虫感染中のマウス腸および寄生虫の代謝解析のためのNAD(P)H蛍光寿命イメージング
要約
本プロトコルは、天然寄生虫 Heligmosomoides polygyrusに感染した外植マウス腸のNAD(P)H蛍光寿命イメージングを記述し、これにより、宿主組織と寄生虫組織の両方で代謝プロセスを空間的に解決した方法で調査することができます。
要約
寄生虫は一般的に宿主の健康に悪影響を及ぼします。それらは、感染した人間や動物の健康に長期的に世界的に影響を及ぼし、したがって農業および社会経済的結果に影響を与えるため、大きな健康負担を表しています。しかし、寄生虫による免疫調節効果が報告されており、自己免疫疾患に対する治療上の関連性が期待されています。宿主と寄生虫の両方の代謝は、宿主と寄生虫の両方の防御に寄与し、腸内での線虫の生存の基礎ですが、適切な技術が不足しているため、ほとんど研究されていません。私たちは、NAD(P)H蛍光寿命イメージングを開発して、天然の線虫 Heligmosomoides polygyrus に感染したマウス腸組織に移植して適用し、宿主と寄生虫の両方の代謝プロセスを空間的に分解して研究しました。ニコチンアミドアデニンジヌクレオチド(NADH)およびニコチンアミドアデニンジヌクレオチドリン酸(NADPH)(以下、NADPH)は、種を超えて保存されていますが、これらは種を超えて保存されており、それらの結合状態と代謝プロセスを触媒する酵素の結合部位に依存します。最も多く発現するNAD(P)H依存性酵素に着目し、主要な防御機構である嫌気性解糖、酸化的リン酸化/好気的解糖、NOXベースの酸化的バーストに関連する代謝経路を同定し、感染時の宿主と寄生虫との間の代謝クロストークを特徴づけた。
概要
寄生虫感染症は、人間の健康に大きな負担をかけます1,2。自己免疫疾患の増加と寄生虫感染の減少との間には相関関係が先進国で観察されています。寄生虫は、過剰な宿主の免疫応答を抑制することにより、有益な効果をもたらすことが知られています。H. polygyrusは、げっ歯類の腸内に発見される天然寄生虫であり、この寄生虫は、感染した宿主3,4,5,6,7,8,9,10,11において、他のメカニズムの中でも特に、宿主の抗寄生虫免疫応答を低下させる免疫調節メカニズムを誘導することが知られている.これらの制御メカニズムは、変性自己免疫疾患において特に興味深いものです。
宿主と腸内線虫との間の代謝クロストークの解析は、宿主と寄生虫の両方において代謝が防御、生存、および機能に重要な役割を果たしているにもかかわらず、広く無視されたままです。私たちは、哺乳類の細胞や組織のさまざまな生理学的および病態生理学的状況ですでに広く使用されている技術である2光子励起12にNADHおよびNADPH蛍光寿命イメージングを適応させ、適用して、生体組織の宿主と線虫の代謝を相関的に調査することを提案します。
NADHおよびNADPHは、NAD(P)Hと呼ばれ、すべての細胞ベースの生命体に保存され、さまざまな代謝経路で補酵素の役割を果たすユビキタス分子です。例えば、エネルギー生産、還元的生合成、NADPHオキシダーゼ媒介活性酸素種(ROS)産生に関与しており、これらは主に細胞防御と細胞コミュニケーションに関連しています13,14,15,16,17,20。両方の補酵素は、750nmでの2光子励起により~450nmで蛍光を発し、したがって細胞および組織におけるマーカーフリーの代謝イメージングを可能にする19,21。NADHとNADPHの両方を1つの波長のみで励起することは、それらの類似したかなり広い2光子励起スペクトル21のために可能である。
補酵素NAD(P)Hの蛍光寿命は、それが結合する酵素18,21,22,23に直接依存する。その化学構造により分子内エネルギー移動が可能となるため、励起されたNADHまたはNADPH分子は、その結合特性に応じた速度で内部変換プロセスを通じて酵素(触媒)にエネルギーを失い、その後、リラックスして蛍光光子を放出します。この寿命は、酵素上のNAD(P)H結合部位、したがって、19,21,22,23,24,25で起こる優先的な生化学反応についての洞察を与える。遊離NADHおよびNADPH分子の蛍光寿命は~450 psに達するが、酵素に結合したときの蛍光寿命ははるかに長く(~2,000 ps)、それぞれの酵素上の結合部位に依存する21。
NAD(P)H結合プロセスには370以上の酵素が関与しています。ただし、顕微鏡の励起範囲内で得られるNAD(P)H蛍光寿命に寄与できるのは、最も豊富なものだけです。哺乳動物細胞からのRNASeqデータを用いて、我々は最も存在量の多いNAD(P)H依存性酵素を同定し、組織および細胞サンプルで生成されたデータを解釈するための蛍光寿命リファレンスを生成した18。それにより、この研究は、例えば、嫌気性解糖代謝経路に関連する乳酸デヒドロゲナーゼ(LDH)の優先活性と、主に好気性解糖/酸化的リン酸化代謝経路に関与するイソクエン酸デヒドロゲナーゼ(IDH)およびピルビン酸デヒドロゲナーゼ(PDH)活性とを区別した16,20.さらに、酸化バーストの主な原因となる酵素であるNADPHオキシダーゼへのNADPHの結合は、これらの酵素が細胞内に特徴的な位置にある(膜結合している)こと、および特に長いNADPH蛍光寿命(3,650 ps)18,24,29,30,32のために容易に分解することができる。H. polygyrusのRNASeqデータは、哺乳類細胞用に生成された参照が、この線虫27に適応した形でも適用されることを示している。
そこで、本研究では、 H. polygyrusに感染したマウスの新たに摘出した十二指腸試料を用いてNAD(P)H蛍光寿命イメージング(FLIM)を行うことにより、遊離NAD(P)Hと酵素結合NAD(P)Hの比率に関する情報を取得し、全組織における一般的な代謝活性と、画像の各画素における主に活性な酵素(すなわち、 NAD(P)Hがその特定の場所で優先的に結合する酵素)。これらの実験の成功は、このプロトコルで説明されているように、外植された腸の正確なサンプル調製、細胞内分解能でのNAD(P)H蛍光寿命の信頼性の高いライブイメージング、および標準化されたデータ評価にかかっています。
プロトコル
すべての実験は、国家動物保護ガイドラインに従って実施され、動物保護のためのドイツ動物倫理委員会(G0176/16およびG0207/19)によって承認されました。このプロトコルは、NAD(P)H蛍光寿命イメージングデータの取得とデータ評価について説明しており、これにより、天然のマウス腸内線虫である H.polygyrusに感染した際の宿主腸と寄生虫の両方の一般的な代謝活性と特定の代謝経路を評価することができます。この目的のために、10-12週齢の雌C57BL/6マウスを200匹のステージ3幼虫(L3)に感染させた。感染の異なる時点で、感染したマウスを屠殺し、十二指腸を切除し、前述したように画像化の準備をした33。感染していない、年齢と性別が一致したマウスの十二指腸も同様に調製され、制御目的で画像化されました。さらなるイメージングと分析に必要な組織特性を維持するためには、サンプルを外植栽直後に処理し、次のステップ(ステップ1.1-1.7)を迅速に実行する必要があります(図1B)。
1. サンプル調製
- 最初に切除した十二指腸から~1cmの長さの小片を切り取ります。
- 小さなシャーレの中央にまっすぐに伸ばしたティッシュチューブを速硬化ティッシュ接着剤(直径47 mm、リム高さ10 mm、材料表を参照)で接着します(図1A2)。
- マイクロブラシを使用して、ペトリ皿の底に追加の接着剤の薄層を組織の周りの広い領域に塗布します(図1A2)。
- 鈍いはさみを使用して、ペトリ皿の底の近くにある物理的に固定された腸の全長に沿って切開を行います(図1A3)。
- 鈍いピンセットで腸を広げて、アブルミナル側が接着剤と完全に接触するようにします。したがって、腸は、管腔側が上を向くように接着剤で固定されています(図1A4)。
- 感染したマウスの場合は、実体顕微鏡(倍率10倍)で線虫を数え、感染が成功したことを確認します(図1A5)。
- 腸をアガロースで密封して、腸が乾燥しないように保護します。ここでは、0.5%〜0.9%のアガロースを使用して、約38°Cの低温融点により、敏感な腸組織を火傷から保護しました。 1 mLのアガロースをピペットに引き込み、ピペットの先端と組織との間に柔らかく接触させながら腸に慎重に滴下し、厚さ約0.5 mmの薄層が組織を完全に包み込みます(図1A6)。
- PBS(10%)のアガロースに密封したサンプルを室温でペトリ皿に充填し、蓋をして閉じます。サンプルを直接画像化するか、断熱ボックスの氷の上に置いて測定の列に並べます。最初に調製したサンプルを顕微鏡対物レンズの下に置き、37°Cに設定した加熱プレートに置きます(図1A8)。
2. イメージング
注:感染した健康な十二指腸組織サンプルでNAD(P)H-FLIMを実行するために使用される顕微鏡システムは、 図2 および 材料表に記載されているデバイスで構成されています。使用するすべてのモジュールの制御ソフトウェアとしてImSpector208を使用します。
- 最初に関心領域(ROI)を見つけるには、サンプルを対物レンズの下のペトリ皿に置き、x面とy面に沿って手で移動して、広視野蛍光顕微鏡モードを使用した目視検査で適切なROIを見つけます。
- ソフトウェアでモードを切り替えることにより、PMT検出または時間相関単一光子計数(TCSPC)検出のいずれかを使用して、イメージングシステムを2光子励起に切り替えます。環境が暗く、振動がないことを確認し、顕微鏡を遮光カーテンで囲み、可能であれば空気圧吊り下げ式光学テーブルを使用するように、特別な注意を払ってください。
- レンズシステムアイコンをクリックし、マウスホイールを回してz位置を変更することにより、制御ソフトウェアを使用してサンプルを含むシャーレに対物レンズを浸します。
- ソフトウェアの波長パネルに目的の波長を入力して、レーザーを765nmに調整し、最大レーザー出力を対物レンズの下の30mWに対応する10%に設定します。必要に応じてレーザー出力を調整します。40% (~30 mW から 100 mW) のゲイン未満で測定し、組織の光損傷や不要な代謝シフトを回避します。
- ソフトウェアで、zステージのステップサイズを2 μmに設定します。これは、組織中の765 nmでの顕微鏡の軸方向分解能に相当します。
- ガルボミラーパネルでラインスキャン周波数を400Hzに設定すると、ピクセル滞留時間は4.95μsになります。
- ソフトウェアのドロップダウンメニューから平均化番号を選択して、2〜4枚の画像に画像の平均化を設定します(つまり、ROIを2〜4回スキャンして、より滑らかな画像を取得します)。このようにして、総ピクセル滞留時間が9.9〜19.8μsに増加し、分析プロセスでの信号対雑音比(SNR)が向上します。
- ソフトウェアのFOVパネルでパラメータを選択して、画像の視野(FOV)を505ピクセル×505ピクセル(500×500μm²)に設定します。
- 各測定について、TCSPCのハードウェアメニューのキャリブレーションウィンドウで初期化をクリックして、TCSPCエレクトロニクスの最適な機能を確保するために、モジュール時間を事前にキャリブレーションします。
- 高速オシロスコープを使用してTi:Saレーザーパルス列を検出するために、高速フォトダイオードを外部で調整します。
注:フォトダイオードの信号は、励起パルスに対するTCSPCによるhPMT検出とフォトンカウンティングをトリガーするために使用されます。検出は、Ti:Saレーザーの繰り返し周波数(80MHz)に対応して、12.5ns(つまり、2つの連続するレーザーパルス間の時間)ごとに繰り返されます。 - 測定深度の範囲を定義するには、まず平均化を1に、レーザー出力のゲインを~10%に設定し、次にビデオモードを開始( ビデオ ボタンをクリック)し、 レンズシステム アイコンを再度クリックして信号を取得できる深度を検索し、マウスホイールを使用してゲインを徐々に増やしながらサンプルに進みます。
- FLIMモードで測定した組織サンプルのNAD(P)H-FLIM画像では、システムを765 nm、最大公称電力100 mWに設定します。NAD(P)H(466/60 nm)の蛍光シグナルを、ソフトウェアで設定されたゲイン97%のハイブリッドPMTで検出します。TCSPCモジュールは、227のタイムウィンドウ(ビン)でフォトンをカウントし、それぞれが55psです。
- ソフトウェアでの測定中に 表示された画像 を右クリックし、 T-PROFILE>表示 を選択すると、システムで取得した減衰曲線を視覚化できます。
- 後で解析するために、減衰曲線のレーザー パルスの前のビンの数をメモします。
- 「測定開始」をクリックしてデータを集録します。
注: データは特定の形式と一致する必要があります。任意の組織深度で記録された各スライスは、2μmの距離で分離され、500μm x 500μmの面積を持ち、スライスは227 x 55 ps(図3A)の時点で500 μm x 500 μmのスライスのスタックとして取得されます(図3A)。各ボクセルには、空間的に解決された(X平面とY平面の)フォトン到着時間ヒストグラムが含まれています。これは蛍光減衰を表します(図3B)。通常測定されるボリュームは、寸法が ~500 μm x 500 μm x 100-300 μm で、227 ビンのハイパースタックの形式である必要があり、z 平面のすべてのスライスには、上記のように時間依存の強度データが含まれています。測定データの例示的なセットの場合、これは(505 x 505 x 227)x 100ピクセル(16ビット)となり、約4GBに相当します。
3. データ分析
注:NAD(P)H-FLIM画像のフェーザ解析の場合、寿命を計算するプログラムはPython33でカスタム記述されたコードです。
- Spyder IDE で Python 3.7 ディストリビューションの Anaconda を使用します ( 資料の表を参照)。このコードは Python の標準ライブラリを使用しています。
- コードを IDE に読み込み、実行します。ファイルパスダイアログが開きます。
- 分析する生データを含むフォルダーを選択します。このコードは、チェックボックス、ドロップダウンメニュー、およびテキストフィールドを備えたTkinterユーザー入力ダイアログを通じて、分析前に3つのパラメータを選択するようにプログラムされています。
- 入力ダイアログ内でオフセットを選択します。オフセットは、タイムスタック内で解析されない最初のスライスの数として決定します。
注:このパラメータは、励起パルスの前の時間点をカットします。システムとトリガーダイオードのキャリブレーションでは、ステップ2.14で説明したように、この値は約5スライスまたはレーザー励起前のデッドビンの数である必要があります。 - 入力ダイアログのドロップダウンメニュー内でフェーザプロットの表現を選択します。ここでは、データポイントの外観(雲状または地形)と、半円の時間軸の軸目盛(酵素または時間[ps])をドロップダウンメニューからオプションとして選択します。
- [OK]をクリックします。
- コードが NAD(P)H-FLIM z スタックから 2 種類の情報を計算することを確認します。
- まず、TCSPCスタックの崩壊によって各ボリュームスライスの空間情報を測定し、227の時間分解フォトンカウンティングヒストグラムが強度投影画像と呼ばれる単一のスライスに投影されます(図3G)、フーリエベースの指数減衰曲線の解析(Leben et al.18によって記述されているように)、および各ボリュームスライスの各ピクセルの正規化された実数部と虚数部(図3C)から測定します。
- 実数と虚数から、フェーザプロット(図3D)と平均蛍光寿命(t)画像(各ボクセルの色分けされた空間的に分解された平均減衰定数)(図3E)を取得します。
注:序文で説明したように、NAD(P)Hの蛍光寿命は、酵素に結合したとき、補酵素のそれぞれの酵素への結合部位によって決定されます。 - 各ピクセルの位相ベクトルと結合していないNAD(P)Hの位相ベクトルとの間のベクトルを生成し、それをフェーザプロットの半円に投影することにより、代謝活性に対するそれぞれの酵素の寄与を判定します。半円は、純粋な化合物における蛍光寿命の可能性のあるすべての単指数関数的減衰を表します。
- 最も存在量の多いNAD(P)H依存性酵素18,33(補足図1)に結合したNAD(P)Hの蛍光寿命の以前に生成された参照を使用して、これらの酵素の活性化の確率を計算する。
注:最も支配的な酵素(すなわち、最も高い活性化確率が計算される酵素)の色コードが各ピクセルに帰属し、酵素マップが生成されます(図3F)。
- 遊離(非結合)と酵素結合のNAD(P)Hの比から、特に450 psの遊離NAD(P)Hと酵素結合状態との間のフェーザープロットのベクトル比から、一般的な代謝活性を0%*非結合のNAD(P)Hのみ)から100%(酵素結合NAD(P)Hのみ)の間のパーセンテージとして計算します。代謝活性(0%-100%)の値を各ピクセルに帰属させることにより、(色分けされた)活動マップが生成されます(図3F)。
- 結果のマップを強度画像と重ね合わせて、追加の形態学的情報を取得します。ここでは、ImageJマクロ( 「材料の表」を参照)を使用して、酵素または活性マップの色相と彩度を強度画像の明るさと重ね合わせます。
- マクロがスタック全体に対して反復処理されることを確認します。スタック内のすべてのスライス (深度) について、強度イメージと選択したマップをそれぞれ HUE、SATURATION、および BRIGHTNESS に分割する必要があります (HSB スタック>イメージ タイプ>)。
- 両方の HSB スタックのメイン チャネルを分離します (イメージ スタック>スタック>イメージに対して)。
- 強度スタックの色相と彩度、およびマップスタックの明るさを閉じます(close(slicename_HUE)、close(slicename_BRIGHTNESS)、...)。
- 残りのチャネルを再結合して、対象マップのスライスの HUE と SATURATION と強度イメージの BRIGHTNESS で構成される新しい HSB スタックを形成します (Image の > は、スタックするイメージ>スタックします)。
- 画像の種類を RGB に変更して、見栄えを良くします (画像>の種類は RGB カラー>)。
4. 組織セグメンテーション
注:腸宿主と線虫組織をそれぞれセグメント化するために、事前に学習されたU-Netベースのネットワーク(ILASTIK、 材料表を参照)を使用して、宿主の上皮と固有層、線虫の高NAD(P)H蛍光シグナル領域と低NAD(P)H蛍光シグナル領域をセグメント化します。
- ILOSTIKを開き、新しいプロジェクトとピクセル分類を選択します。
- ステップ 3.7.1 で以前に計算した強度投影をロードするには 、[Add new > Add separate images] をクリックします。複数の測定値からランダムなスライスをコンテナにロードします。
- [フィーチャ選択] をクリックして>フィーチャを選択します。シグマ値 50 (単位なしの重み) を追加し、アクティブにするすべてのフィーチャにチェックマークを付けます。
注: シグマ値はフィーチャの重みを決定しますが、フィーチャは、エッジ、形状、テクスチャ、または色を認識するネットワークのクラスと能力を決定します。 - Trainingをクリックし、セグメント化する組織(例:上皮、背景など)に応じてラベルに名前を付けます。ラベルを選択し、そのラベルに対応する画像内のすべてのピクセルに色を付けます。
- 他のラベルについても同じ手順を繰り返します。
- 読み込まれたデータセット内の画像の約半分について繰り返します。
- [Live Update] をクリックし、モデルに残りのラベル付けされていない画像のラベルを予測させます。
- 誤って推定されたセグメンテーションにラベルを付け直して、予測された画像とラベル付けされていない画像の約半分を修正し、 ライブアップデートを繰り返します。
- ネットワークが目的の組織を正しく推定することを学習するまで、この手順を繰り返します。ラベル名の左側に示されているネットワークの信頼度が 95% から 98% の範囲であることを確認してください。
- ステップ 3.11 で説明したように、今度は 1 つの測定値のすべてのスライスを含む新しいデータセットを読み込み、[ Prediction Export] をクリックします。
- [ソース] ドロップダウン メニューで [シンプル セグメンテーション] を選択します。「フォーマット」で、出力をtiff形式にすることを選択します。「エクスポート画像設定の選択」で、出力 の保存パス を選択します。 [エクスポート]をクリックします。
- これにより、対象の組織タイプのバイナリマスクが作成されます。これらは、目的のセグメント化された組織のピクセル値が1で、残りのピクセル値が0であるスライスで構成されているため(図3H)、ステップ1.1およびステップ1.4〜1.7にリストされているように、バイナリマスクに生成されたデータを単純に乗算します。これにより、データがマスクされます(図3 I、J、M)。この手順では ImageJ を使用します (Process > Image calculator > multiply)。
- 各ボリュームスライスについて、マスクされた強度イメージから信号対雑音比(SNR)を計算します。そのためには、ImageJ マクロを使用します。セグメント化された画像とマスクされた画像から信号対雑音比 (SNR) を計算するプログラムを作成します。
- ステップ 3.21 のセグメント化された背景を背景 (BG) として使用し、セグメント化された目的の組織をシグナル (SIG) として使用して、2 つのネストされた for ループを使用して、値が 0 より大きいすべてのスライスのすべてのピクセルを反復処理します。SNR は、次の式33 で計算します。
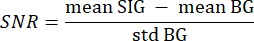
ここで、mean SIG は信号ヒストグラムの平均値、mean BG はバックグラウンド ヒストグラムの平均値、std BG はバックグラウンド ヒストグラムの標準偏差を示します。 - SNR 値が 5 未満のボリューム・スライスは、以降の分析から廃棄します。
- ステップ 3.21 のセグメント化された背景を背景 (BG) として使用し、セグメント化された目的の組織をシグナル (SIG) として使用して、2 つのネストされた for ループを使用して、値が 0 より大きいすべてのスライスのすべてのピクセルを反復処理します。SNR は、次の式33 で計算します。
- Pythonスクリプト18,33を使用して、各組織タイプの酵素マップから、酵素をスライス単位で合計して平均化し、100 μm³の組織体積に正規化することにより、酵素周波数グラフを作成します(図3K、N)。
- マスクされた酵素マップをロードするようにスクリプトをコーディングします。
- すべてのピクセルを反復処理し、同じ酵素のピクセルの合計を計算してから、分析されたボリューム(すべてのピクセルの合計)で除算します。
- 計算された存在量を代謝状態でグループ化し、各グループの酵素数で割って平均化します。LDHの周りにグループ化された代謝状態を嫌気性解糖様経路に、PDH/IDH/GAPDHの周りにグループ化された酵素活性を好気性解糖/oxPhos様経路に、NADPHオキシダーゼ活性(NOx活性)を防御に使用される酸化バースト/酸化ストレスに帰するコードを記述します(図3L、O)。
- Python の matplotlib ライブラリを使用して、箱ひげ図でデータを可視化します。
- マスクされたアクティビティマップ(図3F)から組織の活動量を、ImageJマクロを使用して0より大きい値を持つすべてのピクセルの各ボリュームスライスの平均値として計算し 、Analyze > Measureを実行します。
- 生成されたすべての画像をImageJ/FIJIと並べて視覚化します。
結果
現在のNAD(P)H−FLIM手順28,29,33を記載のフェーザー分析法と組み合わせて使用し、健常で感染した十二指腸における代謝活性および代謝経路を、マウス腸内線虫H. polygyrusの感染後6日目、10日目、12日目、および14日目に測定した。
NAD(P)H-FLIMによって明らかにされた切?...
ディスカッション
プロトコール内の重要なステップは、調製中およびROIを見つけるときに発生します。部分的に消化された食品の繊維は、主にNAD(P)H蛍光と重なる繊維の内因性発光だけでなく、その高調波発生信号によるイメージングの課題となっています。糞便のないROIを見つけることは非常に重要です。糞便が入る場所を測らないようにしました。これは壊れやすい絨毛の完全性?...
開示事項
著者は、競合する金銭的利益を宣言しません。
謝辞
Robert Güntherの優れた技術サポートに感謝します。ドイツ研究評議会(DFG)からの資金援助は、助成金SPP2332 HA2542/12-1(S.H.)、NI1167/7-1(R.A.N.)、HA5354/11-1(A.E.H.)、RA2544/1-1(S.R.)、Grant SFB1444、P14(R.A.N.、A.E.H.)、Grant HA5354/8-2(A.E.H.)、およびGrant GRK2046 B4およびB5(S.H., S.R.)HA2542/8-1(S.H.)が高く評価されています。W.L.は、ベルリンのBerliner Hochschule für Technik, School of Applied Sciencesから医学物理学/物理工学の博士号を取得しました。
資料
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
参考文献
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved