Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Imagerie à vie de fluorescence NAD(P)H pour l’analyse métabolique de l’intestin murin et des parasites lors d’une infection par nématode
Dans cet article
Résumé
Le présent protocole décrit l’imagerie de la durée de vie de la fluorescence NAD(P)H d’un intestin murin explanté infecté par le parasite naturel Heligmosomoides polygyrus, ce qui permet d’étudier les processus métaboliques à la fois dans les tissus de l’hôte et du parasite d’une manière spatialement résolue.
Résumé
Les parasites ont généralement un effet négatif sur la santé de leur hôte. Ils représentent un énorme fardeau pour la santé, car ils affectent globalement la santé de l’homme ou de l’animal infesté à long terme et, par conséquent, ont un impact sur les résultats agricoles et socio-économiques. Cependant, des effets immunorégulateurs induits par les parasites ont été décrits, avec une pertinence thérapeutique potentielle pour les maladies auto-immunes. Bien que le métabolisme de l’hôte et des parasites contribue à leur défense et soit à la base de la survie des nématodes dans l’intestin, il est resté largement sous-étudié en raison d’un manque de technologies adéquates. Nous avons développé et appliqué l’imagerie de la durée de vie de la fluorescence NAD(P)H à des tissus intestinaux murins explantés lors d’une infection par le nématode naturel Heligmosomoides polygyrus afin d’étudier les processus métaboliques chez l’hôte et les parasites de manière spatialement résolue. L’exploitation de la durée de vie en fluorescence des coenzymes nicotinamide adénine dinucléotide (NADH) et nicotinamide adénine dinucléotide phosphate (NADPH), ci-après NAD(P)H, qui sont préservées dans toutes les espèces, dépend de leur statut de liaison et du site de liaison sur les enzymes catalysant les processus métaboliques. En se concentrant sur les enzymes dépendantes du NAD(P)H les plus abondamment exprimées, les voies métaboliques associées à la glycolyse anaérobie, à la phosphorylation oxydative/glycolyse aérobie et à l’explosion oxydative basée sur les NOX, en tant que mécanisme de défense majeur, ont été distinguées, et la diaphonie métabolique entre l’hôte et le parasite pendant l’infection a été caractérisée.
Introduction
Les infections parasitaires imposent un lourd fardeau à la santé humaine 1,2. Une corrélation entre l’augmentation des maladies auto-immunes et le déclin des infections parasitaires a été observée dans les pays industrialisés. On sait que les parasites peuvent avoir des effets bénéfiques en atténuant les réponses immunitaires excessives de l’hôte. H. polygyrus est un parasite naturel présent dans l’intestin chez les rongeurs, et ce parasite est connu pour induire des mécanismes immunorégulateurs qui réduisent la réponse immunitaire antiparasitaire de l’hôte via, entre autres mécanismes, l’induction de lymphocytes T régulateurs (Treg) chez l’hôte infecté 3,4,5,6,7,8,9,10,11 . Ces mécanismes de régulation sont particulièrement intéressants dans les maladies auto-immunes dégénératives.
L’analyse de la diaphonie métabolique entre l’hôte et les nématodes intestinaux reste largement négligée, bien que le métabolisme joue un rôle important à la fois chez l’hôte et les parasites pour la défense, la survie et la fonction. Nous proposons d’adapter et d’appliquer l’imagerie de la durée de vie de la fluorescence du NADH et du NADPH sur l’excitation à deux photons, une technologie déjà largement utilisée dans différentes situations physiologiques et physiopathologiques dans les cellules et les tissus de mammifères12, afin d’étudier corrélativement le métabolisme de l’hôte et du nématode dans les tissus vivants.
Le NADH et le NADPH, appelés NAD(P)H, sont des molécules ubiquitaires qui sont préservées dans toutes les formes de vie cellulaires et jouent le rôle de coenzymes dans diverses voies métaboliques. Par exemple, ils sont impliqués dans la production d’énergie, la biosynthèse réductrice et la production d’espèces réactives de l’oxygène (ROS) médiées par la NADPH oxydase, qui sont principalement liées à la défense cellulaire et à la communication cellulaire 13,14,15,16,17,20. Les deux co-enzymes émettent une fluorescence à ~450 nm lors de l’excitation à deux photons à 750 nm, permettant ainsi une imagerie métabolique sans marqueur dans les cellules et les tissus19,21. Il est possible d’exciter à la fois le NADH et le NADPH avec une seule longueur d’onde en raison de leurs spectres d’excitation à deux photons similaires et assez larges21.
La durée de vie de fluorescence de la coenzyme NAD(P)H dépend directement de l’enzyme à laquelle elle se lie 18,21,22,23. En raison de sa structure chimique permettant le transfert d’énergie intramoléculaire, la molécule de NADH ou NADPH excitée perd de l’énergie par des processus de conversion internes, à un rythme dépendant de ses propriétés de liaison, vers les enzymes (catalyseur) avant de se détendre et d’émettre un photon de fluorescence. Cette durée de vie donne un aperçu du site de liaison du NAD(P)H sur l’enzyme et, par conséquent, de la réaction biochimique préférentielle qui a lieu 19,21,22,23,24,25. La durée de vie de fluorescence des molécules libres de NADH et de NADPH s’élève à ~450 ps, tandis que leur durée de vie de fluorescence lorsqu’elles sont liées à une enzyme est beaucoup plus longue (~2 000 ps) et dépend de leur site de liaison à l’enzymerespective 21.
Il y a plus de 370 enzymes impliquées dans les processus liés au NAD(P)H ; cependant, seuls les plus abondants seront en mesure de contribuer à la durée de vie de la fluorescence NAD(P)H résultante dans la plage d’excitation du microscope. À l’aide des données RNASeq provenant de cellules de mammifères, nous avons identifié les enzymes NAD(P)H dépendantes les plus abondantes et généré une référence de durée de vie en fluorescence pour interpréter les données générées dans les échantillons de tissus et de cellules18. Ainsi, ce travail a distingué par exemple l’activité préférentielle de la lactate déshydrogénase (LDH), qui est associée aux voies métaboliques glycolytiques anaérobies, et l’activité de l’isocitrate déshydrogénase (IDH) et de la pyruvate déshydrogénase (PDH), qui sont principalement impliquées dans les voies métaboliques de glycolyse aérobie/phosphorylation oxydative16,20. De plus, la liaison du NADPH aux NADPH oxydases, qui sont les enzymes principalement responsables de l’explosion oxydative, peut être facilement résolue en raison de l’emplacement caractéristique de ces enzymes dans la cellule (liée à la membrane) et en raison de la durée de vie particulièrement longue de la fluorescence du NADPH (3 650 ps)18,24,29,30,32. Les données RNASeq de H. polygyrus montrent que la référence générée pour les cellules de mammifères s’applique également sous forme adaptée à ce nématode27.
Par conséquent, dans ce travail, en effectuant l’imagerie de la durée de vie de la fluorescence NAD(P)H (FLIM) dans des échantillons de duodénum fraîchement explantés de souris infectées par H. polygyrus, des informations sur le rapport entre le NAD(P)H libre et enzymatique lié à l’enzyme ont été acquises, ce qui a permis de décrire l’activité métabolique générale dans tous les tissus, ainsi que l’enzyme principalement active dans chaque pixel de l’image (c’est-à-dire l’enzyme à laquelle le NAD(P)H se lie préférentiellement à cet endroit spécifique). Le succès de ces expériences repose sur la préparation précise de l’échantillon de l’intestin explanté, l’imagerie en direct fiable de la durée de vie de la fluorescence du NAD(P)H à résolution subcellulaire et l’évaluation standardisée des données, comme discuté dans ce protocole.
Protocole
Toutes les expériences ont été réalisées conformément aux directives nationales de protection des animaux et approuvées par le Comité allemand d’éthique animale pour la protection des animaux (G0176/16 et G0207/19). Le protocole décrit l’acquisition et l’évaluation des données d’imagerie de la durée de vie de la fluorescence NAD(P)H, qui permettent d’évaluer l’activité métabolique générale et les voies métaboliques spécifiques dans l’intestin de l’hôte et les parasites lors de l’infection par le nématode intestinal murin naturel, H. polygyrus. À cette fin, des souris femelles C57BL/6 âgées de 10 à 12 semaines ont été infectées par 200 larves de stade 3 (L3). À différents moments de l’infection, les souris infectées ont été sacrifiées, et les duodénums ont été excisés et préparés pour l’imagerie comme décrit précédemment33. Les duodénums de souris non infectées, appariées selon l’âge et le sexe, ont été préparés et imagés de la même manière à des fins de contrôle. Pour maintenir les propriétés tissulaires nécessaires à l’imagerie et à l’analyse ultérieures, les échantillons doivent être traités immédiatement après l’explantation, et les étapes suivantes (étapes 1.1 à 1.7) doivent être effectuées rapidement (Figure 1B).
1. Préparation de l’échantillon
- Coupez des morceaux de ~1 cm de longueur dans le duodénum initialement excisé.
- Collez le tube de tissu redressé au milieu d’une petite boîte de Pétri avec de la colle à tissu à durcissement rapide (47 mm de diamètre, 10 mm de hauteur de jante, voir le tableau des matériaux) (Figure 1A2).
- Appliquez une fine couche de colle supplémentaire au fond de la boîte de Pétri sur une grande surface autour du tissu à l’aide d’une microbrosse (Figure 1A2).
- À l’aide de ciseaux émoussés, faites une incision sur toute la longueur de l’intestin physiquement fixé à proximité du fond de la boîte de Pétri (Figure 1A3).
- Dépliez l’intestin à l’aide d’une pince à épiler émoussée de manière à ce que le côté abluminal entre en contact complet avec la colle. Ainsi, l’intestin est fixé par la colle avec le côté luminal vers le haut (Figure 1A4).
- Dans le cas d’une souris infectée, comptez les vers au stéréomicroscope (grossissement 10x) pour vous assurer que l’infection a réussi (Figure 1A5).
- Scellez l’intestin avec de l’agarose pour le protéger du dessèchement. Ici, l’agarose à 0,5 % à 0,9 % a été utilisé pour protéger le tissu intestinal sensible contre les brûlures en raison de son point de fusion à basse température d’environ 38 °C. Prélever 1 mL d’agarose dans une pipette et verser délicatement sur l’intestin en laissant un contact tendre entre l’extrémité de la pipette et le tissu de manière à ce qu’une fine couche d’environ 0,5 mm d’épaisseur entoure complètement le tissu (figure 1A6).
- Remplissez les boîtes de Pétri avec les échantillons scellés dans de l’agarose avec du PBS (10 %) à température ambiante, puis fermez-les avec le couvercle. Imagez directement les échantillons ou placez-les sur de la glace dans une boîte isolante thermique pour faire la queue pour les mesures. Placez le premier échantillon préparé sous l’objectif du microscope sur une plaque chauffante réglée à 37 °C (Figure 1A8).
2. Imagerie
REMARQUE : Le système de microscope utilisé pour effectuer la spectrométrie NAD(P)H-FLIM dans des échantillons de tissus duodénaux infectés et sains comprend les dispositifs énumérés et décrits à la figure 2 et dans le tableau des matériaux. Utilisez ImSpector 208 comme logiciel de contrôle pour tous les modules utilisés.
- Pour trouver initialement la région d’intérêt (ROI), placez l’échantillon dans la boîte de Pétri sous l’objectif, et déplacez-vous le long des plans x et y à la main pour trouver un ROI approprié par inspection visuelle à l’aide du mode de microscopie à fluorescence à champ large.
- Basculez le système d’imagerie vers l’excitation à deux photons à l’aide de la détection PMT ou de la détection par comptage de photons uniques corrélés dans le temps (TCSPC) en changeant de mode dans le logiciel. Veillez tout particulièrement à ce que l’environnement soit sombre et exempt de vibrations, à enfermer le microscope dans des rideaux étanches à la lumière et à utiliser une table optique suspendue pneumatique, si possible.
- Immergez la lentille de l’objectif dans la boîte de Pétri contenant l’échantillon avec le logiciel de contrôle en cliquant sur l’icône du système de lentille et en tournant la molette de la souris pour modifier la position z.
- Réglez le laser sur 765 nm et réglez la puissance laser maximale sur 10 %, ce qui correspond à 30 mW sous l’objectif, en tapant la longueur d’onde souhaitée dans le panneau des longueurs d’onde du logiciel. Ajustez la puissance du laser si nécessaire. Mesurez en dessous d’un gain de 40 % (~30 mW à 100 mW) pour éviter les photodommages tissulaires et les changements métaboliques indésirables.
- Dans le logiciel, réglez la taille du pas de la platine z sur 2 μm, ce qui correspond à la résolution axiale du microscope à 765 nm dans les tissus.
- Réglez la fréquence de balayage linéaire sur 400 Hz dans le panneau de miroir galvo, ce qui permet d’obtenir un temps de séjour des pixels de 4,95 μs.
- Réglez la moyenne de l’image sur deux à quatre images en choisissant le nombre de moyenne dans le menu déroulant du logiciel (c’est-à-dire balayez le retour sur investissement deux à quatre fois pour obtenir des images plus fluides). De cette façon, le temps de séjour total des pixels est augmenté à 9,9-19,8 μs au profit d’un rapport signal/bruit (SNR) plus élevé dans le processus d’analyse.
- Réglez le champ de vision de l’image (FOV) sur 505 pixels x 505 pixels (500 x 500 μm²) en choisissant les paramètres dans le panneau FOV du logiciel.
- Pour chaque mesure, calibrez le temps du module à l’avance pour assurer le fonctionnement optimal de l’électronique TCSPC en cliquant sur initialiser dans la fenêtre d’étalonnage du menu matériel du TCSPC.
- Ajustez la photodiode rapide vers l’extérieur pour détecter le train d’impulsions laser Ti :Sa à l’aide d’un oscilloscope rapide.
REMARQUE : Le signal de la photodiode est utilisé pour déclencher la détection hPMT et le comptage des photons par le TCSPC par rapport à l’impulsion d’excitation. La détection est répétée tous les 12,5 ns (c’est-à-dire le temps entre deux impulsions laser consécutives), ce qui correspond au taux de répétition du laser Ti :Sa (80 MHz). - Définissez la plage de la profondeur de mesure en réglant d’abord la moyenne sur 1 et le gain de la puissance laser sur ~10 %, puis en démarrant le mode vidéo (cliquez sur le bouton vidéo ), en recherchant la profondeur à partir de laquelle le signal peut encore être acquis en cliquant à nouveau sur l’icône du système d’objectif , et en utilisant la molette de la souris pour progresser dans l’échantillon tout en augmentant progressivement le gain.
- Pour les images NAD(P)H-FLIM des échantillons de tissus mesurés en mode FLIM, réglez le système sur 765 nm à une puissance nominale maximale de 100 mW. Détectez le signal de fluorescence du NAD(P)H (466/60 nm) avec le PMT hybride à un gain de 97 %, qui est défini dans le logiciel. Le module TCSPC compte les photons sur 227 fenêtres de temps (bacs), chacune de 55 ps.
- Faites un clic droit sur l’image affichée pendant la mesure dans le logiciel, et choisissez afficher > T-PROFILE pour visualiser la courbe de décroissance acquise par le système.
- Notez le nombre de bacs avant l’impulsion laser dans la courbe de décroissance pour une analyse ultérieure.
- Acquérez les données en cliquant sur démarrer la mesure.
REMARQUE : Les données doivent correspondre à un format spécifique. Chaque coupe, enregistrée à n’importe quelle profondeur de tissu, est séparée par une distance de 2 μm et a une aire de 500 μm x 500 μm, et les coupes sont acquises sous la forme d’un empilement de tranches de 500 μm x 500 μm à des points temporels de 227 x 55 ps (Figure 3A). Chaque voxel contient un histogramme de l’heure d’arrivée des photons spatialement résolu (dans le plan x et le plan y). Il s’agit de la décroissance de la fluorescence (Figure 3B). Un volume généralement mesuré doit se présenter sous la forme d’une hyperpile de dimensions de ~500 μm x 500 μm x 100-300 μm et 227 bacs, où chaque tranche dans le plan z contient les données d’intensité dépendantes du temps comme décrit ci-dessus. Pour un ensemble de données de mesure exemplaire, cela donne (505 x 505 x 227) x 100 pixels (16 bits) et correspond à environ 4 Go.
3. Analyse des données
REMARQUE : Pour l’analyse de phaseur des images NAD(P)H-FLIM, le programme de calcul des durées de vie est un code écrit sur mesure en Python33.
- Utilisez Anaconda avec une distribution Python 3.7 sur l’IDE Spyder (voir la Table des matériaux). Le code utilise les bibliothèques standard de Python.
- Chargez le code dans l’IDE et exécutez-le. Une boîte de dialogue de chemin d’accès au fichier s’ouvre.
- Choisissez le dossier contenant les données brutes à analyser. Le code est programmé pour choisir trois paramètres avant l’analyse via une boîte de dialogue de saisie utilisateur Tkinter avec des cases à cocher, un menu déroulant et des champs de texte.
- Choisissez un décalage dans la boîte de dialogue de saisie. Déterminez le décalage comme le nombre de premières tranches de la pile temporelle qui ne sont pas analysées.
REMARQUE : Le paramètre coupe les points de temps avant l’impulsion d’excitation. Avec l’étalonnage du système et de la diode de déclenchement, cette valeur doit être d’environ cinq tranches ou le nombre de bacs morts avant l’excitation laser, comme décrit à l’étape 2.14. - Choisissez la représentation des tracés de phaseur dans le menu déroulant de la boîte de dialogue de saisie. Ici, choisissez l’apparence des points de données (nuageux ou topographiques), ainsi que les graduations de l’axe du demi-cercle de l’axe du temps (enzymes ou temps [ps]), en tant qu’options dans le menu déroulant.
- Cliquez sur OK.
- Vérifiez que le code calcule deux types d’informations à partir des piles z NAD(P)H-FLIM.
- Tout d’abord, mesurez l’information spatiale de chaque tranche de volume par l’effondrement des empilements TCSPC, où les 227 histogrammes de comptage de photons résolus dans le temps sont projetés sur une seule tranche appelée image de projection d’intensité (Figure 3G), et à partir de l’analyse basée sur Fourier des courbes de décroissance exponentielle (telle que décrite par Leben et al.18), et à partir de la partie réelle et imaginaire normalisée pour chaque pixel dans chaque tranche de volume (Figure 3C).
- À partir des images réelles et imaginaires, obtenez les tracés de phaseur (Figure 3D) et l’image de la durée de vie moyenne de la fluorescence (t) (constantes de décroissance moyennes codées en couleur et résolues dans l’espace pour chaque voxel) (Figure 3E).
REMARQUE : Comme décrit dans l’introduction, la durée de vie en fluorescence du NAD(P)H, lorsqu’il est lié à des enzymes, est déterminée par le site de liaison de la coenzyme à l’enzyme respective. - Déterminez la contribution de l’enzyme respective à l’activité métabolique en générant le vecteur entre le vecteur de phase de chaque pixel et le vecteur de phase du NAD(P)H non lié et en le projetant sur le demi-cercle du diagramme de phaseur. Le demi-cercle représente toutes les décroissances mono-exponentielles possibles de la durée de vie de la fluorescence dans les composés purs.
- En utilisant la référence précédemment générée des durées de vie en fluorescence du NAD(P)H lié aux enzymes dépendantes du NAD(P)H les plus abondantes18,33 (figure supplémentaire 1), et calculer la probabilité d’activation de ces enzymes.
REMARQUE : Un code couleur de l’enzyme la plus dominante (c’est-à-dire de l’enzyme pour laquelle la probabilité d’activation la plus élevée est calculée) est attribué à chaque pixel, produisant ainsi une carte enzymatique (Figure 3F).
- À partir du rapport entre le NAD(P)H libre (non lié) et le NAD(P)H lié à l’enzyme, en particulier à partir du rapport vectoriel dans le diagramme de phaseur entre le NAD(P)H libre à 450 ps et l’état enzymatique, calculez l’activité métabolique générale en pourcentage entre 0 % (uniquement du NAD(P)H non lié) et 100 % (uniquement du NAD(P)H lié à l’enzyme). En attribuant une valeur d’activité métabolique (0 %-100 %) à chaque pixel, une carte d’activité (codée en couleur) est générée (Figure 3F).
- Superposez les cartes résultantes avec les images d’intensité pour obtenir des informations morphologiques supplémentaires. Ici, utilisez une macro ImageJ (voir la Table des matériaux) pour superposer la teinte et la saturation de l’enzyme ou des cartes d’activité avec la luminosité des images d’intensité.
- Assurez-vous que la macro effectue une itération sur l’ensemble de la pile. Pour chaque tranche (profondeur) de la pile, il doit diviser l’image d’intensité et la carte de votre choix respectivement en HUE, SATURATION et BRIGHTNESS (type de > d’image > pile TSL).
- Séparez les canaux principaux pour les deux piles HSB (piles de > d’images > pile d’images).
- Fermez la TEINTE et la SATURATION de la pile d’intensité, ainsi que la LUMINOSITÉ de la pile de la carte (close(slicename_HUE), close(slicename_BRIGHTNESS), ...).
- Recombinez les canaux restants pour former une nouvelle pile HSB composée de la teinte et de la SATURATION de la tranche de la carte d’intérêt et de la LUMINOSITÉ de l’image d’intensité (l’image > empile > images à empiler).
- Changez le type d’image en RVB pour une meilleure apparence visuelle (type de > d’image > couleur RVB).
4. Segmentation tissulaire
REMARQUE : Utilisez un réseau basé sur U-Net pré-entraîné (ILASTIK, voir le tableau des matériaux) pour segmenter le tissu intestinal de l’hôte et le tissu du nématode, respectivement, ainsi que l’épithélium et la lamina propria de l’hôte et les zones de signal de fluorescence à haut NAD(P)H et les zones à faible signal de fluorescence NAD(P)H dans le nématode.
- Ouvrez ILASTIK et choisissez un nouveau projet et une nouvelle classification de pixels.
- Chargez la projection d’intensité calculée précédemment à partir de l’étape 3.7.1 en cliquant sur Ajouter une nouvelle > Ajouter des images séparées. Chargez des tranches aléatoires à partir de plusieurs mesures dans le conteneur.
- Cliquez sur Sélection de fonctionnalités > sélectionnez des fonctionnalités. Ajoutez une valeur sigma de 50 (poids sans unité) et cochez toutes les fonctionnalités pour être actives.
REMARQUE : Alors que la valeur sigma détermine le poids des entités, celles-ci déterminent les classes et la capacité du réseau à reconnaître les arêtes, les formes, la texture ou la couleur. - Cliquez sur Entraînement, et nommez les étiquettes en fonction du tissu à segmenter (par exemple, Épithélium, Arrière-plan, etc.) en cliquant dessus. Sélectionnez une étiquette et colorez tous les pixels de l’image correspondant à cette étiquette.
- Répétez l’opération pour les autres étiquettes.
- Répétez l’opération pour environ la moitié des images du jeu de données chargé.
- Cliquez sur Mise à jour en direct et laissez le modèle prédire les étiquettes pour les images non étiquetées restantes.
- Corrigez environ la moitié des images uniquement prédites et non étiquetées en réétiquetant les segmentations faussement estimées, et répétez la mise à jour en direct.
- Répétez cette étape jusqu’à ce que le réseau ait appris à estimer correctement le tissu d’intérêt souhaité ; Assurez-vous que le niveau de confiance dans le réseau indiqué à gauche, près des noms d’étiquette, se situe entre 95 % et 98 %.
- Chargez un nouveau jeu de données comme décrit à l’étape 3.11, cette fois avec toutes les tranches d’une seule mesure, et cliquez sur Exporter les prédictions.
- Dans le menu déroulant « Source », choisissez Segmentation simple ; dans « Format », choisissez la sortie au format tiff ; et dans « Choisir les paramètres d’exportation de l’image », sélectionnez le chemin d’enregistrement pour la sortie. Cliquez sur exporter.
- Cela crée des masques binaires pour le type de tissu d’intérêt. Puisqu’ils sont constitués de tranches où le tissu segmenté d’intérêt a une valeur de pixel de 1 et le reste a une valeur de pixel de 0 (Figure 3H), il suffit de multiplier les masques binaires avec les données générées comme indiqué aux étapes 1.1 et 1.4 à 1.7. Cela conduit à des données masquées (Figure 3 I, J, M). Utilisez ImageJ pour cette étape (Traiter > Calculatrice d’image > multiplier).
- Pour chaque tranche de volume, calculez un rapport signal/bruit (SNR) à partir des images d’intensité masquée. Utilisez une macro ImageJ à cet effet. Créez votre programme de manière à ce qu’il calcule le rapport signal/bruit (SNR) à partir des images segmentées et masquées.
- Utilisez l’arrière-plan segmenté de l’étape 3.21 comme arrière-plan (BG) et le tissu d’intérêt segmenté comme signal (SIG) en itérant sur chaque pixel de chaque tranche d’une valeur supérieure à 0 avec deux boucles for imbriquées. Calculez le SNR à l’aide de la formule suivante33 :
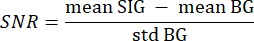
où la moyenne SIG fait référence à la valeur moyenne de l’histogramme du signal, la moyenne BG fait référence à la valeur moyenne de l’histogramme d’arrière-plan et std BG fait référence à l’écart-type de l’histogramme d’arrière-plan. - Éliminez les tranches de volume dont la valeur SNR est inférieure à 5 de l’analyse ultérieure.
- Utilisez l’arrière-plan segmenté de l’étape 3.21 comme arrière-plan (BG) et le tissu d’intérêt segmenté comme signal (SIG) en itérant sur chaque pixel de chaque tranche d’une valeur supérieure à 0 avec deux boucles for imbriquées. Calculez le SNR à l’aide de la formule suivante33 :
- Avec le script Python18,33, créez un graphique de fréquence enzymatique à partir des cartes enzymatiques pour chaque type de tissu en additionnant et en faisant la moyenne des enzymes par tranche, puis en les normalisant à un volume tissulaire de 100 μm³ (Figure 3K, N).
- Codez le script de manière à ce qu’il charge la carte d’enzyme masquée.
- Itérez sur chaque pixel, calculez la somme des pixels d’une même enzyme, puis divisez par le volume analysé (le total de tous les pixels).
- Regroupez les abondances calculées dans leurs états métaboliques et faites la moyenne en divisant par le nombre d’enzymes dans chaque groupe. Écrivez un code qui attribue les états métaboliques regroupés autour de la LDH aux voies de type glycolyse anaérobie, l’activité enzymatique regroupée autour des voies PDH/IDH/GAPDH aux voies de glycolyse aérobie/de type oxPhos, et l’activité de la NADPH oxydase (activité NOX) à l’éclatement oxydatif/stress oxydatif utilisé pour la défense (Figure 3L, O).
- Utilisez la bibliothèque matplotlib de Python pour visualiser les données avec des boîtes à moustaches.
- Calculez l’activité du tissu à partir des cartes d’activité masquées (Figure 3F) en tant que valeur moyenne sur chaque tranche de volume pour tous les pixels d’une valeur supérieure à 0 à l’aide d’une macro ImageJ en exécutant Analyser > mesurer.
- Visualisez toutes les images générées côte à côte avec ImageJ/FIJI.
Résultats
À l’aide de la procédure actuelle NAD(P)H-FLIM 28,29,33 combinée à la méthode d’analyse de phaseur décrite, l’activité métabolique et les voies métaboliques dans les duodénums sains et infectés ont été mesurées au jour 6, au jour 10, au jour 12 et au jour 14 après l’infection par le nématode intestinal murin H. polygyrus.
Viabi...
Discussion
Les étapes critiques du protocole se produisent lors de la préparation et de la recherche du retour sur investissement. Les fibres d’aliments partiellement digérés représentent un défi pour l’imagerie, principalement en raison de la luminescence endogène des fibres chevauchant la fluorescence NAD(P)H, mais aussi en raison de leur signal de génération d’harmoniques. Il est très important de trouver des zones d’investissement exemptes de matières fécales. Nous avons che...
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt financier concurrent.
Remerciements
Nous remercions Robert Günther pour son excellent support technique. Soutien financier du Conseil allemand de la recherche (DFG) au titre des subventions SPP2332 HA2542/12-1 (S.H.), NI1167/7-1 (R.A.N.), HA5354/11-1 (A.E.H.) et RA2544/1-1 (S.R.), au titre de la subvention SFB1444, P14 (R.A.N., A.E.H.), au titre de la subvention HA5354/8-2 (A.E.H.), et au titre des subventions GRK2046 B4 et B5 (S.H., S.R.) et HA2542/8-1 (S.H.) sont grandement reconnus. W.L. a obtenu une bourse de doctorat de la Berliner Hochschule für Technik, École des sciences appliquées, Berlin en physique médicale/génie physique.
matériels
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
Références
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon