Method Article
Etablissement et analyse histologique d’organoïdes œsophagiens modélisant la progression des tissus normaux aux tissus cancéreux
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le présent protocole décrit l’établissement et l’analyse histologique de modèles d’organoïdes œsophagiens qui représentent différents stades de la progression tumorale. Cette méthode permet aux chercheurs d’étudier les changements dans la morphologie cellulaire, l’organisation spatiale et les modèles d’expression des marqueurs moléculaires pendant la transition des tissus normaux aux tissus cancéreux.
Résumé
Les organoïdes sont devenus un outil essentiel pour faire progresser la compréhension de la tumorigenèse et du traitement du cancer. En générant des modèles d’organoïdes humains représentant différents stades tumoraux et en effectuant des analyses histologiques, il est possible d’obtenir une compréhension plus profonde des altérations de la morphologie cellulaire, de l’architecture spatiale et de l’expression des marqueurs moléculaires clés au fur et à mesure que la tumeur progresse. Cette étude présente un protocole complet pour l’établissement et la culture d’organoïdes épidermoïdes œsophagiens. De plus, le protocole décrit des méthodes pour évaluer les modèles d’expression et l’organisation spatiale des molécules critiques dans les organoïdes, en utilisant des techniques telles que la fixation, l’intégration et la coloration. Grâce à ce protocole, des changements significatifs ont été identifiés dans la structure spatiale des cellules épithéliales squameuses de l’œsophage et dans l’expression de divers biomarqueurs tumoraux au cours de la tumorigenèse. Le protocole facilite la construction et l’analyse histologique des organoïdes, permettant aux chercheurs d’étudier l’architecture spatiale et les altérations moléculaires des cellules épithéliales à différentes étapes de la tumorigenèse et de l’intervention thérapeutique.
Introduction
La tumorigenèse est un processus complexe en plusieurs étapes caractérisé par des changements moléculaires et morphologiques progressifs dans les cellules 1,2. Le carcinome épidermoïde de l’œsophage (ESCC), une tumeur maligne prévalente de mauvais pronostic 3,4, illustre cette progression progressive à travers quatre stades distincts : muqueuse normale, néoplasie intra-épithéliale de bas grade (LGIN), néoplasie intra-épithéliale de haut grade (HGIN) et carcinome invasif5. Tout au long de ces étapes, les cellules épithéliales présentent des changements dynamiques dans les modèles d’expression moléculaire et l’organisation spatiale, accompagnés de modifications systématiques de la morphologie des tissus à mesure qu’ils passent d’un état normal à un état malin 6,7. Malgré les progrès réalisés dans la compréhension de la pathogenèse de l’ESCC, l’absence de modèles expérimentaux qui récapitulent fidèlement les aspects spatiaux et temporels de l’évolution tumorale - tout en permettant des analyses histologiques et moléculaires systématiques - a entravé une compréhension mécaniste plus approfondie de la progression de la maladie et du développement thérapeutique.
Bien que les lignées cellulaires cancéreuses immortalisées en 2D aient apporté des contributions significatives à la compréhension de l’oncogenèse, elles sont intrinsèquement limitées dans la reproduction de la complexité biologique et des caractéristiques pathologiques des tumeurs natives8. Les modèles animaux, bien qu’ils fournissent un contexte in vivo, prédisent souvent mal les réponses humaines en raison de différences spécifiques à l’espèce9. En revanche, les organoïdes sont apparus comme une plate-forme préclinique transformatrice qui préserve fidèlement l’hétérogénéité cellulaire, l’architecture et la fonctionnalité des tissus humains 10,11,12,13. En tant que modèles précliniques, les organoïdes capturent mieux les caractéristiques des tumeurs primaires, ce qui permet une étude détaillée des événements moléculaires clés et des changements cellulaires au cours de la progression tumorale14. Par exemple, Chen et al. ont utilisé des organoïdes œsophagiens dérivés de patients à différents stades de l’ESCC pour élucider les interactions épithéliales-fibroblastes, validant ainsi l’axe de signalisation ANXA1-FPR2 en tant que moteur essentiel de la pathogenèse6 de l’ESCC. De même, Ko et al. ont utilisé des organoïdes œsophagiens génétiquement modifiés pour identifier les principaux déterminants génétiques à l’origine de l’initiation de l’ESCC et de l’évasion immunitaire, démontrant ainsi comment les modèles d’organoïdes peuvent récapituler efficacement les caractéristiques de la maladie et révéler de nouvelles cibles thérapeutiques15.
La méthodologie actuelle résout des problèmes importants dans la modélisation du cancer de l’œsophage en établissant un protocole reproductible pour générer des organoïdes ESCC à plusieurs étages qui reflètent la progression histologique de l’épithélium normal au carcinome invasif. Ce système intègre des conditions de culture optimisées à l’aide d’un milieu conditionné par L-WRN pour maintenir la souche épithéliale, combinées à des protocoles standardisés pour le traitement histologique et l’analyse par immunofluorescence multiplex (mIF), fournissant une plate-forme idéale pour analyser longitudinalement les changements spatiaux et moléculaires au cours de la tumorigenèse. Par rapport à d’autres techniques telles que les cultures 2D, cette plateforme organoïde préserve de manière unique l’architecture tissulaire, permettant la visualisation de marqueurs moléculaires organisés spatialement, y compris la protéine de point de contrôle immunitaire-L1 (CD274), qui médie l’évasion immunitaire tumorale en inhibant les réponses des lymphocytes T 16,17,18, et le marqueur de prolifération Ki-67. Le protocole permet le passage des organoïdes à partir de tissus œsophagiens et précancéreux normaux, aidant les chercheurs à construire un modèle organoïde continu du tissu normal à la tumeur19. En permettant une analyse détaillée des changements spatiaux et moléculaires au cours de la tumorigenèse, ce protocole offre aux chercheurs un outil puissant pour comprendre les mécanismes sous-jacents au développement et à la progression du cancer, ce qui pourrait conduire à de meilleures stratégies thérapeutiques.
Cette méthodologie est particulièrement adaptée aux chercheurs qui étudient la carcinogenèse épithéliale, les interactions entre le microenvironnement tumoral ou les réponses thérapeutiques dans l’ESCC et les tumeurs malignes squameuses associées. Sa conception modulaire permet de s’adapter à l’étude d’autres marqueurs moléculaires ou voies de signalisation, à condition d’intégrer des étapes de validation appropriées. En offrant une plateforme standardisée mais flexible, ce protocole vise à faire progresser la recherche préclinique en biologie tumorale et à accélérer la traduction des connaissances mécanistes en thérapies ciblées.
Protocole
Cette étude a été approuvée par le Conseil d’examen institutionnel de l’Hôpital du cancer de l’Académie chinoise des sciences médicales (approbations n° 20/069-2265, 22/221-3423 et 23/305-4047). Des échantillons de tissu œsophagien ont été prélevés sur des patients subissant une intervention chirurgicale ou un dépistage précoce du carcinome épidermoïde de l’œsophage (ESCC) à l’hôpital du cancer de l’Académie chinoise des sciences médicales entre 2021 et 2024, dans le but d’établir des organoïdes œsophagiens humains. Aucun des patients inclus dans cette étude n’avait reçu de chimiothérapie ou de radiothérapie avant le prélèvement de l’échantillon. Le consentement éclairé de tous les participants a été obtenu, et les renseignements cliniques pertinents ont été extraits des dossiers médicaux. Une liste complète des réactifs et de l’équipement utilisés dans cette étude est fournie dans la table des matériaux.
1. Préparation des organoïdes épithéliales de l’œsophage
- Préparation du milieu conditionné L-WRN
- Préparation de la lignée cellulaire L-WRN
- Décongeler la lignée cellulaire L-WRN (stockage à -80 °C) dans un bain-marie à 37 °C pendant 1 à 2 min. Mélanger avec 5 mL de milieu de culture basal préchauffé (DMEM complété par 10 % de FBS).
- Centrifuger le mélange à 200 x g pendant 5 min à température ambiante (RT).
- Jetez le surnageant. Remettre les cellules en suspension avec 2 à 3 ml de milieu de sélection frais préchauffé (DMEM contenant 10 % de FBS, 0,5 mg/mL d’hygromycine B et 0,5 mg/mL de G-418).
REMARQUE : Maintenez le milieu à 4 °C (stockage maximum 1 mois). - Transférez les cellules dans une boîte de culture de 10 cm contenant le milieu de sélection. Cultivez les cellules à 37 °C dans un incubateur humidifié à 5 % de CO2 .
- Passage de la lignée cellulaire L-WRN
- Dissocier les cellules confluentes à l’aide de 0,025 % de trypsine-EDTA (1 à 2 min à 37 °C) et les séparer dans un rapport de 1:2.
- Collecte du fluide conditionné L-WRN
- Remplacer le milieu de culture par un milieu de culture basal à 80 % de confluence cellulaire. Transférez le surnageant dans un tube à centrifuger.
- Filtrer à travers une membrane de 0,22 μm pour éliminer les débris cellulaires. Recueillir le filtrat sous la forme d’un milieu conditionné au L-WRN.
REMARQUE : Conservez le milieu conditionné au L-WRN à 4 °C (1 semaine) ou à -80 °C (6 mois).
- Préparation de la lignée cellulaire L-WRN
- Préparation du milieu de culture d’organoïdes œsophagiens humains (H-EOCM)
- Préparation du H-EOCM
- Vortex : le milieu conditionné par L-WRN (volume de 1,5 mL) après 4 h d’équilibre à 4 °C.
- Combinez le milieu Advanced DMEM/F12 avec ces additifs pour créer H-EOCM : milieu conditionné à 3 % par L-WRN, 1× anti-anti, 1× l-glutamine, 1× supplément N2, 1× supplément de B27, 0,15 mM d’HEPES, 40 ng/mL d’EGF, 10 μM Y-27632 et 50 μM A83-01.
REMARQUE : Maintenir l’entreposage congelé du H-EOCM à -20 °C pendant six mois.
- Préparation du H-EOCM
- Préparation pré-expérimentale
- Dégivrez H-EOCM en le maintenant à 4 °C pendant 4 h.
2. Établissement de l’organoïde œsophagien humain
- Préparation des matériaux
- Décongeler la matrice de la membrane basale et le H-EOCM à 4 °C. Stérilisez les ciseaux chirurgicaux et les pinces à épiler pour la prochaine expérience.
- Pré-refroidissez les pointes de pipette à 4 °C. Préchauffez la plaque à 24 puits à 37 °C.
- Traitement des tissus et isolement cellulaire
REMARQUE : Le tissu tumoral de l’ESCC, les lésions dysplasiques (à ≤2 cm du bord de la tumeur) et le tissu œsophagien normal apparié (à ≥5 cm du bord de la tumeur) ont été prélevés chez les mêmes personnes atteintes d’ESCC qui ont subi une résection chirurgicale. De plus, des échantillons œsophagiens en plusieurs étapes ont été obtenus dans le cadre d’un programme de détection précoce et de dépistage de l’ESCC, tel que décrit dans nos études précédentes 6,7.- Nettoyez l’échantillon avec un tampon de lavage à température ambiante (PBS contenant 1×Anti-Anti et 0,15 mM HEPES) dans des tubes à centrifuger de 5 mL à trois reprises.
REMARQUE : Lavez les échantillons plus de trois fois pour réduire le risque de contamination. - Coupez le tissu en fragments de 1 mm3 à l’aide de ciseaux stériles. Transférer les fragments dans des tubes à centrifuger de 1,5 mL.
- Mettez l’échantillon en suspension avec 1 mL de tampon de digestion et agitez-le à 37 °C, 50-100 tr/min, pendant 10-20 min pour digérer le tissu.
- Centrifuger le mélange à 400 x g pendant 5 min à 4 °C. Jeter le surnageant.
- Remettre le précipité en suspension dans 500 μL de trypsine-EDTA à 0,025 %. Incuber à 37 °C pendant 10 min. Ajouter 1 ml de DMEM complété par 10 % de FBS pour arrêter l’activité enzymatique.
- Passez la suspension à travers un filtre stérile de 70 μm. Prélever le filtrat dans un tube à centrifuger de 1,5 mL.
- Centrifuger le filtrat pendant 5 min à 400 x g à 4 °C. Jeter le surnageant. Remettez les cellules en suspension avec 100 μL de H-EOCM.
- Nettoyez l’échantillon avec un tampon de lavage à température ambiante (PBS contenant 1×Anti-Anti et 0,15 mM HEPES) dans des tubes à centrifuger de 5 mL à trois reprises.
- Ensemencement organoïde
- Déterminez la densité des cellules, puis préparez un nouveau tube à centrifuger de 1,5 ml avec 5 000 à 15 000 cellules en suspension.
- Centrifugeuse pendant 5 min à 400 x g à 4 °C. Aspirez le surnageant avec précaution. Remettez doucement les cellules en suspension dans 50 à 100 μL de matrice de membrane basale, de manière uniforme.
REMARQUE : Stocker la matrice de la membrane basale à 4 °C pour éviter la solidification. - Ajouter 50 μL de matrice de membrane basale avec les cellules mélangées au centre de chaque plaque de 24 puits. Polymériser la matrice de la membrane basale par incubation à 37 °C (durée 30 min).
- Ajouter 500 μL de H-EOCM préchauffé (37 °C) pour couvrir la matrice de la membrane basale. Cultivez les organoïdes dans un incubateur humidifié à 5 % de CO2 à 37 °C.
- Remplacement moyen
- Aspirez le milieu épuisé et remplissez-le de 500 μL de H-EOCM frais (réchauffé à 37 °C).
REMARQUE : Remplacez H-EOCM à des intervalles de 3 jours.
- Aspirez le milieu épuisé et remplissez-le de 500 μL de H-EOCM frais (réchauffé à 37 °C).
3. Passage des organoïdes
- Préparation des matériaux
- Décongeler la matrice de la membrane basale et le H-EOCM à 4 °C. Pré-refroidissez les pointes de pipette à 4 °C. Préchauffez la plaque à 24 puits à 37 °C.
- Digestion des organoïdes
- Retirez délicatement le H-EOCM medium. Ajouter 500 μL de tampon de passage pré-refroidi (Advanced DMEM/F12 contenant 1× Anti-Anti et 0,15 mM HEPES, pré-refroidi à 4 °C) dans le puits pour faire fondre la matrice de la membrane basale.
- Combinez la matrice de la membrane basale avec le tampon de passage dans un tube à centrifuger de 1,5 ml. Lavez le puits avec 500 μL de tampon de passage pré-refroidi et récupérez le tampon pour récupérer les organoïdes restants.
- Centrifuger le tampon pendant 5 min à 400 x g à 4 °C. Aspirez doucement le surnageant. Ajouter 500 μL de trypsine recombinante dans le tube et incuber pour la digestion enzymatique (37 °C, 15 min). Resuspendre à intervalles de 5 minutes.
- Centrifuger l’échantillon digéré (400 x g, 5 min, 4 °C). Jetez le surnageant. Répétez les étapes 3.2.6 et 3.2.7. Remettre les cellules en suspension dans 100 μL de H-EOCM.
- Ensemencement organoïde
- Pour l’ensemencement d’organoïdes, répéter l’étape 2.3.
4. Congélation et récupération des organoïdes
- Congélation d’organoïdes
- Pour la préparation des matériaux, répétez l’étape 2.1.
- Pour la digestion des organoïdes, répétez l’étape 3.2.
- Congélation d’organoïdes
- Déterminez la densité des cellules, puis préparez un nouveau tube à centrifuger de 1,5 ml avec 10 000 cellules en suspension.
- Centrifuger les échantillons à 400 x g pendant 5 min à RT. Aspirer le surnageant soigneusement.
- Remettre les cellules en suspension avec 500-1000 μL de milieu de cryoconservation. Transfert dans un nouveau cryotube. Congelez le cryotube à -80 °C pendant 24 h. Archivage dans un système de stockage d’azote liquide pour une conservation prolongée.
- Décongélation de l’organoïde congelé
- Pour la préparation des matériaux, répétez l’étape 2.1.
- Décongélation de l’organoïde congelé
- Décongelez rapidement le cryotube dans un bain-marie à 37 °C pendant 2-3 min. Transférez la matière congelée dans 9 mL de culture basale préchauffée (37 °C) et remettez les cellules en suspension uniformément.
- Centrifuger la suspension cellulaire pendant 5 min à 400 x g (à RT). Aspirez le surnageant avec précaution.
- Ensemencement organoïde
- Pour ensemencer les organoïdes, répétez l’étape 2.3.
5. Analyse histologique des organoïdes
- Préparation de sections enrobées de paraffine
- Préparation des matériaux
- Stériliser 50 mL de matrice d’enrobage (solution aqueuse : gélose à 2 % + gélatine à 2,5 %) dans une fiole de 250 mL à l’autoclave . Aliquote 5 mL dans des tubes à centrifuger de 15 mL. Conserver à température ambiante.
- Fixation organoïde
- Répétez les étapes 3.2.1 et 3.2.2. Remettre le précipité en suspension avec 1 mL de tampon de passage dans un tube frais de 1,5 mL.
- Centrifugeuse à 400 x g à 4 °C pendant 5 min. Retirez le surnageant. Remettre les organoïdes en suspension dans 500 μL de paraformaldéhyde (PFA) à 4 %. Incuber à température ambiante pendant 1 h ou à 4 °C pendant au moins 6 h.
- Enrobage organoïde
- Centrifuger le mélange (400 x g, 5 min, RT). Retirez le surnageant. Ajouter 1 mL de tampon de passage dans le tube et remettre en suspension la précipitation.
- Centrifuger le mélange (400 x g, 5 min, RT). Retirez le surnageant.
- Placez un tube (15 ml) avec le milieu d’enrobage (5 ml) dans le bain-marie (récipient de 100 ml, 150 ml). Passez au micro-ondes à puissance maximale jusqu’à ce que l’eau commence à bouillir.
REMARQUE : Dévissez le capuchon du tube de 15 ml avant de le cuire au micro-ondes. - Remettre les organoïdes en suspension avec 50 μL de gel d’enrobage dans un tube à centrifuger de 1,5 mL. Refroidissez le tube à 4 °C jusqu’à ce que le gel durcisse complètement.
- Transférez le gel solidifié dans de l’éthanol à 70 %. Conserver à 4 °C.
- Préparation des blocs de paraffine
- Déshydrater le gel par des séries d’éthanol (30 % → 50 % → 70 % → 80 % → 95 % → 100 %), 30 min par concentration.
- Mettez le gel solidifié dans du xylène clair pendant 30 min. Plongez le gel solidifié dans de la paraffine et enfoncez-le à l’aide d’une station de paraffine chauffée.
- Coupez des tranches de 4 m d’épaisseur à l’aide d’un microtome. Montage sur lames et séchage à 65 °C pendant 1 h. Conserver dans des conditions sèches.
- Préparation des matériaux
- Coloration immunohistochimique (IHC) de coupes d’organoïdes
- Déparaffinage et hydratation
- Chauffer les tranches dans un agent nettoyant à 65 °C pendant 40 min. Immergez la lame dans un agent nettoyant frais à température ambiante pendant 20 min.
- Traitez les lames à travers des plateaux en verre contenant des séries d’éthanol : immergez-les dans de l’éthanol à 100 % pendant 10 minutes et répétez l’opération une fois, puis transférez-les à de l’éthanol à 95 % pendant 5 minutes, puis à 85 % d’éthanol pendant 5 minutes, puis à 75 % d’éthanol pendant 5 minutes, et enfin placez dans 4 % de PFA pendant 10 minutes.
- Ajoutez de l’eau stérilisée dans la chambre thermostable et maintenez 5 min sur l’agitateur à 80 tr/min. Répétez cette étape deux fois.
- Récupération d’antigènes
- Ajouter la solution de récupération de l’antigène Tris-EDTA (pH 9,0) dans la chambre thermostable pour immerger les lames. Passez au micro-ondes la chambre thermostable contenant les lames à l’intensité maximale (700 W, 3 min) jusqu’à ébullition. Maintenez le micro-ondes à faible puissance (70 W, 15 min) et laissez refroidir à température ambiante.
- Laver à l’eau distillée pendant 2 min sur un shaker à 80 tr/min. Répétez l’opération deux fois. Encerclez l’emplacement des organoïdes avec un stylo histologique.
- Blocage de la peroxydase
- Déposer la solution bloquant la peroxydase pour couvrir la zone de l’échantillon. Incuber dans une chambre humide à température ambiante pendant 20 min.
- Transférez les lames dans une chambre thermostable. Laver au PBST (PBS + 0,1 % Tween 20) pendant 2 min à 80 tr/min. Répétez l’opération deux fois.
- Incubation primaire de l’anticorps
- Essuyez doucement les gouttelettes PBST en excès sur les lames.
REMARQUE : Évitez de toucher les organoïdes sur les lames. - Déposer l’anticorps primaire dilué pour couvrir la zone de l’organoïde. Incuber dans une chambre d’humidité pendant 2 h à température ambiante, ou 8-14 h à 4 °C. Répétez cette étape.
- Essuyez doucement les gouttelettes PBST en excès sur les lames.
- Incubation secondaire d’anticorps
- Répétez l’étape 5.2.4.1. Déposer l’anticorps secondaire HRP dilué pour couvrir la zone organoïde. Incuber dans une chambre humide à température ambiante pendant 20 min. Répétez cette étape.
- Coloration DAB (3,3′-Diaminobenzidine)
- Répétez l’étape 5.2.4.1. Déposez 1× DAB pour couvrir la zone organoïde. Incuber dans une chambre humide à température ambiante jusqu’à ce que la couleur devienne brune.
- Plongez les lames dans de l’eau distillée pendant 5 à 10 secondes pour terminer le changement de couleur. Répétez l’étape.
- Coloration à l’hématoxyline
- Colorer à l’hématoxyline pendant 5 à 8 min. Différencier dans de l’alcool acide à 1 % (HCl à 1 % dans de l’alcool à 70 %) pendant 5 à 10 s.
- Immergez les lames dans de l’eau distillée pendant 5 à 10 s. Répétez cette étape.
- Déshydratation
- Traitez les lames à travers des plateaux en verre dans l’ordre séquentiel : immergez-les dans de l’éthanol à 75 % pendant 5 min, transférez-les à de l’éthanol à 85 % pendant 5 min, passez à de l’éthanol à 95 % pendant 5 min, puis immergez-les deux fois distinctes dans de l’éthanol à 100 % pendant 10 min chacune, et enfin placez-les dans du xylène pendant deux périodes successives de 20 minutes.
- Montage sur glissière
- Ajoutez du xylène à la gomme neutre jusqu’à ce qu’elle devienne transparente. Sécher légèrement à l’air. Ensuite, déposez de la gomme avec du xylène pour couvrir la zone organoïde.
- Appliquez une lamelle. Numérisez et analysez la diapositive.
- Déparaffinage et hydratation
- Coloration par immunofluorescence multiplex (mIF)
- Pour le déparaffinage et l’hydratation, répétez l’étape 5.2.1.
- Pour le blocage de la peroxydase, répétez l’étape 5.2.3.
- Pour le prélèvement de l’antigène, répétez l’étape 5.2.3.
- Blocage du sérum de mouton.
- Répétez l’étape 5.2.4.1. Ajoutez une solution bloquante de sérum de mouton pour couvrir la zone organoïde. Incuber dans une chambre d’humidité à température ambiante pendant 30 min.
- Laver au PBST pendant 2 min. Répéter deux fois.
- Pour l’incubation primaire de l’anticorps, répéter l’étape 5.2.4.
- Pour l’incubation secondaire des anticorps, répéter l’étape 5.2.4.1.
- Déposez une solution d’anticorps secondaire spécifique pour couvrir la zone des organoïdes. Incuber dans une chambre humide à température ambiante pendant 20 min. Répétez l’étape 5.2.3.2.
REMARQUE : Protégez les lames de la lumière de cette étape jusqu’à la fin de l’expérience.
- Déposez une solution d’anticorps secondaire spécifique pour couvrir la zone des organoïdes. Incuber dans une chambre humide à température ambiante pendant 20 min. Répétez l’étape 5.2.3.2.
- Coloration par colorant fluorescent
- Répétez l’étape 5.3.4.1. Déposez une solution de colorant fluorescent pour couvrir la zone de l’organoïde.
- Incuber dans une chambre d’humidité à température ambiante pendant 10-20 min. Répétez l’étape 5.2.3.2.
- Pour une coloration multiple, répétez les étapes 5.3.3 à 5.3.7.
- Coloration DAPI
- Répétez l’étape 5.2.4.1. Déposez la solution DAPI pour couvrir la zone de l’organoïde. Répétez l’étape 5.2.3.2. Ensuite, répétez l’étape 5.3.4.2.
- Plongez les lames dans de l’eau stérilisée pendant 2 min.
- Montage sur glissière
- Déposez le support de montage anti-décoloration pour couvrir la zone de l’organoïde. Appliquez une lamelle.
- Acquérez des images numériques à l’aide d’un scanner de diapositives et analysez-les.
Résultats
Ce protocole décrit le prélèvement d’organoïdes et l’analyse histologique à différents stades de la tumorigenèse de l’ESCC (Figure 1). En prélevant la muqueuse œsophagienne normale, la néoplasie intra-épithéliale de bas grade (LGIN), la néoplasie intra-épithéliale de haut grade (HGIN) et les tissus tumoraux de patients ESCC, des organoïdes représentant différents stades de la tumorigenèse peuvent être construits. De plus, l’enrobage de paraffine et la section de ces organoïdes ont été effectués, suivis d’une coloration par immunofluorescence.
Pour étudier l’expression des molécules immunosuppressives et les caractéristiques morphologiques des clones cellulaires au cours de la tumorigenèse, des tissus épithéliales œsophagiens ont été prélevés à différents stades du développement tumoral et ont effectué une coloration par immunofluorescence. Une fois la coloration terminée, quatre pseudo-couleurs ont été définies pour les marqueurs avant de numériser les diapositives. -L1, Ki-67 et KRT6A ont reçu des pseudo-couleurs de vert, rouge et gris, respectivement, tandis que les noyaux cellulaires ont été marqués avec DAPI. Les résultats expérimentaux ont montré que la morphologie des organoïdes de la muqueuse œsophagienne a changé au cours de la tumorigenèse, devenant principalement plus désorganisée. La coloration par immunofluorescence a révélé qu’à mesure que la tumorigenèse progressait, la prolifération des cellules épithéliales changeait, accompagnée d’une expression élevée de molécules immunosuppressives telles que-L1 (Figure 2).
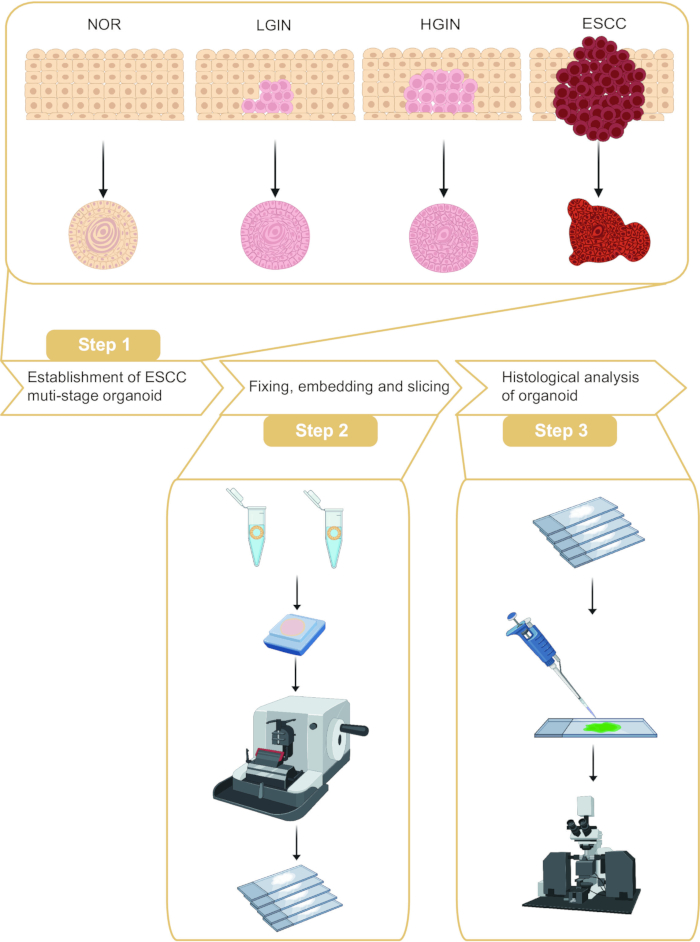
Figure 1 : Déroulement de l’établissement et de l’analyse histologique des organoïdes du carcinome épidermoïde épidermoïde de l’œsophage en plusieurs étapes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
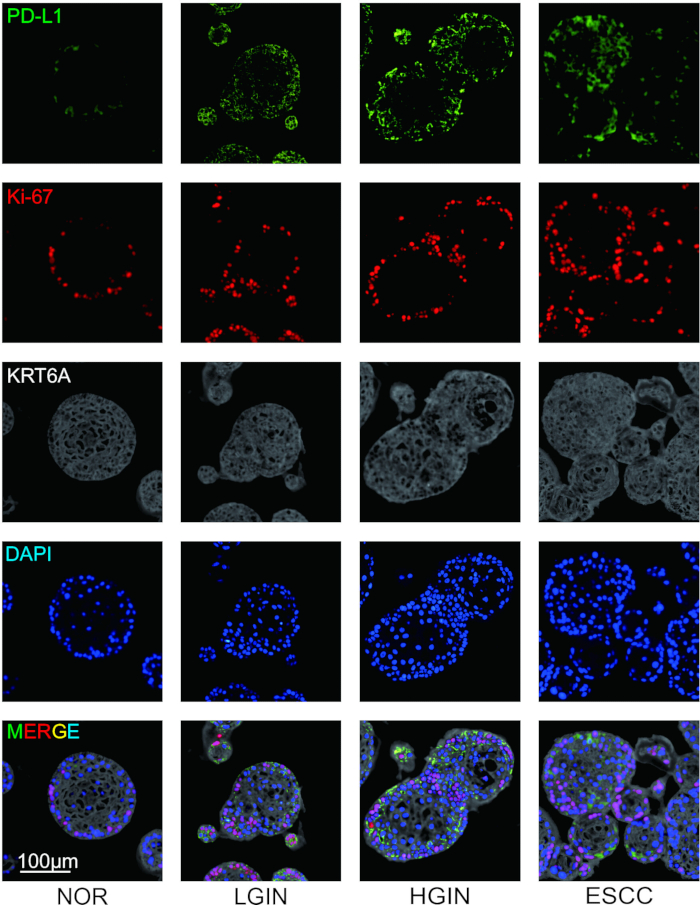
Figure 2 : Coloration par immunofluorescence multiplex (mIF) de l’organoïde ESCC à plusieurs étages. Images représentatives montrant l’expression de-L1 (vert), Ki-67 (rouge) et KRT6A (gris). Barres d’échelle = 100 μm. NOR, muqueuse normale ; LGIN, néoplasie intra-épithéliale de bas grade ; LGIN, néoplasie intra-épithéliale de haut grade. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
L’établissement et l’analyse histologique des organoïdes représentent une avancée significative dans la modélisation de la progression tumorale. Le protocole offre des avantages notables par rapport aux méthodes existantes pour étudier la tumorigenèse20. Contrairement aux systèmes de culture cellulaire 2D traditionnels, les organoïdes conservent une architecture tridimensionnelle complexe et une hétérogénéité cellulaire qui reflètent mieux les conditions in vivo. Par rapport aux modèles animaux, les organoïdes dérivés de tissus humains représentent plus précisément les caractéristiques de la maladie humaine 9,21,22. L’incorporation d’une coloration par immunofluorescence multiplex permet la visualisation simultanée de plusieurs marqueurs, fournissant des informations sur les relations spatiales entre les molécules clés et l’architecture tissulaire au cours de l’évolution tumorale. En utilisant ce protocole avancé, cette étude a révélé des changements dynamiques significatifs dans le carcinome épidermoïde de l’œsophage, démontrant spécifiquement que les niveaux de prolifération des cellules tumorales se sont déplacés progressivement au cours du développement tumoral, accompagnés de changements correspondants dans l’expression de la molécule immunosuppressive-L1.
Ce protocole fournit une méthodologie complète pour étudier les changements morphologiques et moléculaires au cours de la tumorigenèse, avec plusieurs étapes critiques nécessitant une attention particulière. Le temps de digestion pendant le traitement des tissus doit être surveillé de près, car une digestion excessive peut entraîner une mort cellulaire excessive, tandis qu’une digestion insuffisante entraîne une mauvaise isolation cellulaire. De plus, le contrôle de la température lors de la manipulation de la matrice de la membrane basale est crucial - la matrice doit être maintenue à 4 °C pour éviter une polymérisation prématurée tout en maintenant la viabilité cellulaire.
Cependant, certaines limites de la technique doivent être reconnues. L’absence de composants immunitaires et de cellules stromales dans le système de culture organoïde de base peut ne pas récapituler complètement les interactions tumeur-microenvironnement23,24. Cette limitation peut être partiellement résolue par des systèmes de co-culture, bien que de telles modifications nécessitent une optimisation minutieuse. Enfin, la décongélation des organoïdes est également un enjeu important. Selon l’expérience précédente, le taux de réussite de la décongélation des organoïdes congelés n’est pas élevé. Par conséquent, il est conseillé aux utilisateurs de congeler les organoïdes avec prudence.
Les implications cliniques de ce protocole sont substantielles, en particulier pour permettre la modélisation de la progression en plusieurs étapes de la NOR au carcinome invasif6 en passant par la LGIN et la HGIN. Les organoïdes dérivés de patients générés à l’aide de cette méthodologie constituent des outils précieux pour la recherche et la médecine personnalisée, permettant l’identification de marqueurs moléculaires associés à la progression de la maladie25,26.
Ce protocole peut être adapté pour diverses applications dans la recherche sur le cancer et le développement de médicaments. La méthodologie peut être étendue à l’étude d’autres cancers épithéliaux et modifiée pour intégrer des techniques d’analyse supplémentaires telles que le séquençage sur cellule unique27,28. De plus, les adaptations futures pourraient incorporer des fibroblastes et des cellules immunitaires associés au cancer dérivés de patients pour modéliser les interactions tumeur-stroma-immunitaire ou intégrer l’édition CRISPR pour étudier les moteurs génétiques, élargissant ainsi son utilité dans la recherche translationnelle et la prédiction de la réponse thérapeutique29,30.
Ce protocole présente les méthodes de construction et de congélation des organoïdes à partir des différents stades de la tumorigenèse de l’ESCC. Plus important encore, les techniques d’enrobage, de sectionnement, d’IHC et de coloration mIF pour les organoïdes sont décrites dans cet article. Nous pensons que ces méthodes d’enrobage, de tranchage et de coloration des organoïdes peuvent être appliquées aux méthodes de coloration des organoïdes de plusieurs sources d’organes, y compris l’œsophage. Cette approche peut également aider les chercheurs à observer la relation entre l’expression moléculaire pertinente et la structure spatiale.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas de conflits d’intérêts financiers ou concurrents concurrents.
Remerciements
Les auteurs remercient tous les patients et les médecins qui ont participé à la recherche à l’hôpital du cancer, à l’Académie chinoise des sciences médicales (CAMS) et au Peking Union Medical College (PUMC). Cette étude est financée par la Fondation nationale des sciences naturelles de Chine (82203156 à S.Z.), le Programme national de recherche et de développement clés de la Chine (2023YFC3503200 à S.Z.) et le Fonds d’innovation pour les sciences médicales de l’Académie chinoise des sciences médicales (2023-I2M-QJ-002 à S.Z.). La figure 1 est créée avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 μm filter | Merck | Cat#SLGPR33RB | |
| 24-well plate | Corning | Cat#3524 | |
| 4% Paraformaldehyde | Beyotime | Cat# P0099 | |
| 70 μm sterile strainer | Falcon | Cat#352350 | |
| A83-01 | Tocris Bioscience | Cat# 2939 | |
| Advanced DMEM/F12 | Gibco | Cat# 12634028 | |
| Agar | Solarbio | Cat# A8190 | |
| Anti-Anti (Antibiotic-Antimycotic) | Gibco | Cat# 15240062 | |
| B-27 supplement | Gibco | Cat# 17504044 | |
| CO2 incubator | Thermo | Cat#371GPCN | |
| Collagenase IV | Gibco | Cat# 17104019 | |
| Cryostor | STEMCELL | Cat# 07930 | |
| DMEM | Corning | Cat# 10-013-CV | |
| EGF | Gibco | Cat# PHG0313 | |
| Fetal bovine serum | Cell Technologies | Cat# 30070 | |
| G-418 | Sigma | Cat# A1720 | |
| Gelatin | Solarbio | Cat# G8061 | |
| GlutaMAX | Gibco | Cat# 35050061 | |
| Growth factor-reduced Matrigel | Corning | Cat# 354230 | |
| HE staining kit | Beijing Yili Fine Chemicals Co., Ltd | NA | |
| HEPES | Gibco | Cat# 15630080 | |
| Histological pen | Zsbio | Cat#ZLI-9305 | |
| Hygromycin B | Sigma | Cat# 400050 | |
| Immunohistochemical staining kit | ZSGB-BIO | PV-8000 | |
| L-WRN | ATCC | CRL-3276; RRID:CVCL_DA06 | |
| N-2 supplement | Gibco | Cat# 17502048 | |
| Neutral gum | Zsbio | Cat#ZLI-9555 | |
| Opal 5-Color Manual IHC Kit | PANOVUE | Cat# 10144100100 | |
| PBS | MeilunBio | Cat#MA0015 | |
| Rabbit Monoclone anti-PD-L1 | CST | Cat# 13684; RRID:AB_2687655 | |
| Rabbit Polyclonal anti-Ki67 | Abcam | Cat# ab16667; RRID:AB_302459 | |
| Rabbit Polyclonal anti-KRT6A | Proteintech | Cat# 10590-1-AP; RRID: AB_2134306 | |
| Sheep serum | Zsbio | Cat#ZLI-9056 | |
| TrypLE Express | Gibco | Cat# 12604021 | |
| TrypLE-EDTA | Gibco | Cat#15400-054 | |
| Whole slide image scanner | Hamamatsu | Cat#C13210 | |
| Y-27632 | Selleck Chemicals | Cat# S1049 |
Références
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon