Method Article
細胞代謝研究のためのマルチモーダル光学イメージングプラットフォーム
* これらの著者は同等に貢献しました
要約
ラベルフリーの光学イメージングモダリティを組み合わせたマルチモーダルプラットフォームを利用して、細胞のダイナミクスと代謝を視覚化および定量化するためのプロトコルを開発しました。多光子蛍光、第2高調波発生、および誘導ラマン散乱顕微鏡によるイメージングを通じて、細胞および分子環境の全体像を生成することができます。
要約
光学イメージング技術は、生物試料から形態学的情報と機能情報の両方を高い空間分解能で取得する能力において、生物医学研究において重要です。これらの光学プロセスは、細胞、組織、または臓器内の光子と分子との間の散乱、吸収、放出、高調波発生など、さまざまな光分子相互作用を利用します。従来のバイオメディカルイメージングは、歴史的に単一のモダリティの適用に重点を置いてきましたが、最近の研究では、これらの多様な技術が補完的な洞察を提供し、それらの組み合わせた出力により、老化プロセスや疾患発生の分子変化、細胞生物学の基礎をより包括的に理解できることが示されています。
過去数十年で、ラベルフリーの光学イメージング法が進歩し、細胞および細胞内環境の詳細な探索が可能になりました。例えば、多光子蛍光(MPF)は、標的タンパク質のイメージングを容易にするだけでなく、自己蛍光補酵素を介して代謝活性を定量化し、高い浸透深度と空間分解能を実現します。第2高調波発生(SHG)は、細胞外マトリックス中のコラーゲンなどの構造をイメージングするために使用され、誘導ラマン散乱(SRS)は、化学結合と分子組成を細胞内分解能でin situマッピングします。
MPF、SHG、SRSモダリティを組み合わせたマルチモーダルイメージングプラットフォームを開発しました。これらのモダリティを単一のプラットフォームに統合することで、細胞、組織、臓器、さらには体内の同じ局在から多面的な情報を取得できるようになり、細胞代謝、細胞外マトリックス構造、分子組成の間の複雑な関係をより詳細に探索することが容易になります。このマルチモーダルシステムは、細胞内分解能、深部組織への浸透、 in situ 生細胞/組織イメージング、ラベルフリー検出、位置調整、デバイスの切り替え、または解析後のアライメントを必要とせずに、瞬時の同時レジストレーションを提供します。ここでは、このマルチモーダルプラットフォームを使用したラベルフリーイメージングのプロトコルを提示し、老化や疾患を研究するための細胞代謝、細胞や組織の分子不均一性の特性評価への応用を実証します。
概要
光学生物医学イメージングは、生物学的構造と機能の理解を深める上で極めて重要な役割を果たしてきました。画像は、励起光を変調し、光と組織の相互作用からの信号を検出することによって生成されます。1590年頃にハンス・ヤンセンとザカリアス・ヤンセンによって開発された最初の複合顕微鏡は、チューブ内の2つの凸レンズを使用し、最大30倍1の倍率を提供しました。現代の光学顕微鏡は、何世紀にもわたる進歩により、現在では1〜3 nm 2,3の微細な解像度を達成できるようになりました。高度なイメージングシステムは、高解像度を提供するだけでなく、組織のより深い浸透、効率の向上、サンプルの損傷の最小化を実現し、生細胞や組織のイメージングに特に適しています。ラベルフリーイメージングは、細胞内プロセスを中断したり、サンプルの完全性を損なったりすることなく情報をキャプチャできるため、特に有利です。
多光子蛍光(MPF)顕微鏡、特に2光子蛍光顕微鏡は、ラベルフリーイメージングに広く使用されています。線形の単一光子吸収と放出に依存する従来の蛍光顕微鏡法とは異なり、MPF励起は複数の光子の同時吸収を伴い、その結合エネルギーは単一の蛍光色素分子を励起します4,5。これらの光子は、通常は赤外線スペクトルで、単一光子励起に必要なエネルギーの半分以下しか持っていません。この非線形プロセスでは、波長が長く、焦点で局所的な励起が行われるため、散乱が少なくなり、組織への浸透が深くなり、光毒性が減少します。
細胞の代謝情報は、還元型ニコチンアミドアデニンジヌクレオチド(NADH)やフラビンアデニンジヌクレオチド(FAD)などの内因性代謝基質からの自家蛍光シグナルの検出により、ラベルフリーのMPF顕微鏡法で捕捉できます。これらの補酵素は、異なる励起スペクトルと発光スペクトルを示し、酸化還元比(NADH/FAD)として知られる蛍光強度比は、細胞の酸化状態を反映しています。ブリットン・チャンスが1979年に初めて酸化還元比の概念を導入して以来、NAD(P)H/FAD、NAD(P)H/(FAD + NAD(P)H)、FAD/(FAD + NAD(P)H)、FAD/(FAD + NAD(P)H)などの追加の比率が提案されてきた6,7,8,9。MPFイメージングを介してこれらの光学酸化還元比を定量化することで、代謝ダイナミクスに関する貴重な洞察が得られます。例えば、MPFイメージングは、代謝の変化に基づいてがん細胞と正常細胞を区別することができ、がん診断の可能性を示しています10,11,12。ただし、MPFベースの自家蛍光検出には限界があります。ケラチンなどの他の内因性蛍光色素は、蛍光強度に寄与し、スペクトルクロストークおよび不正確なシグナル解釈につながる可能性がある13。さらに、酸化還元比は、細胞全体の酸化還元変化のみを反映しており、異なる供給源(例えば、細胞質またはミトコンドリア)からのNADHまたはNADHとNAD(P)Hを区別しない、なぜなら、両者は450nmで同様のスペクトルピークを示し、その結果、強度シグナルが混合される14。
第2高調波発生(SHG)は、1980年代に生物医学分野で初めて実証された非線形光学プロセスであり、細胞構造のラベルフリーイメージングに広く利用されてきた15,16。MPFと同様に、SHGは、超高速パルスレーザーから同じエネルギーの2つの光子を同時に吸収します。これらの光子は再結合されて、入射光の2倍の周波数の新しい光子を放出し、その結果、第2高調波信号が検出されます。この非線形光学的相互作用は、第2高調波信号を生成するための偏光を誘発する非ゼロの2次感受性を示す非中心対称材料でのみ発生する17,18。これにより、SHGは、外因性蛍光色素15,17,19,20を必要とせずに、コラーゲン、ミオシン、チューブリンなどの糸状タンパク質および繊維状構造のイメージングに特に効果的である。線維症とコラーゲンの存在量、硬さ、整列、および構造の異常は、炎症や癌などの多くの状態に蔓延しており、SHGは特定の疾患状態21,22,23に対する効率的かつ非侵襲的な検出のための有望なツールとなっています。乳がん、卵巣がん、皮膚がんの研究を含む腫瘍学研究におけるSHGイメージングの広範な応用は、基礎研究と潜在的な臨床応用の両方におけるその重要な役割を強調しています24,25,26,27。
異なる分子は異なる振動エネルギーレベルを示し、入射光によって励起されるとさまざまな程度の非弾性散乱を誘発します-1928年にC.V.ラマンによって最初に特徴付けられた現象28。それ以来、ラマン効果は、外因性標識なしで分子組成物や組織組成物を検出するための光学顕微鏡法で広く利用されています。誘導ラマン散乱法(SRS)とコヒーレント反ストークスラマン散乱法(CARS)は、分子振動をコヒーレントに励起し、光の非線形相互作用を利用して、従来の自然ラマン分光法と比較してより強力な信号を生成します。SRS現象は1962年に最初に報告されました29。2008年には、このメカニズムが3次元多光子イメージングに統合され、分子振動遷移によるPumpビームとStokesビームの強度変化に基づいて化学物質を選択的に検出できるようになりました30。この方法により、非共振のバックグラウンド干渉が最小限に抑えられ、CARSを超えるクリーンな強度の信号が生成されます。SRSイメージングは、マルチプレックスイメージングとハイパースペクトルイメージングの提供に優れており、複数の化学結合の同時検出を可能にし、かなりの浸透深さを持つ試料中の分子組成を高解像度で視覚化できます。SRSイメージングは比較的新しい技術ですが、in vivoおよびin vitroの臨床診断と代謝研究の両方で有効であることが証明されています30,31,32,33,34,35,36。例えば、SRSは、脂質対タンパク質比を定量化することにより、脳腫瘍浸潤組織を皮質および白質と区別することができ、ラベルフリーで非侵襲的な方法で腫瘍縁の描写を可能にする37,38。さらに、老化関連疾患や癌関連疾患の特徴としばしば考えられている代謝変化は、重水(D2O)で処理されたサンプル中の炭素-重水素結合を検出することによって達成されるSRSを用いて定量的に評価することができ、タンパク質合成、脂肪生成、およびその他の高分子代謝過程の定量的測定を可能にする31,33,34,35,36.高い時間的および空間的分解能で代謝物を追跡する能力により、SRSは疾患の調査と診断のための有望なツールとして位置付けられ、より広範な臨床応用の可能性を秘めています。
マルチモーダルイメージングは、生物医学研究における強力なアプローチとして登場し、2つ以上のイメージングモダリティを統合して、同じ標本内の複雑な生物学的システムをより包括的に理解することができます。2018年には、2光子蛍光(2PF)、3光子蛍光(3PF)、SHG、および第3高調波発生(THG)を統合した、ラベルフリーの自家蛍光マルチハーモニック(SLAM)顕微鏡法が導入されました39。このアプローチにより、腫瘍微小環境内の細胞相互作用、動的プロセス、および個々のコンポーネントの同時可視化が容易になります。SLAM顕微鏡は、サンプルの摂動を最小限に抑え、レーザー出力要件を削減し、深部組織のプロファイリングを可能にし、より安全な生体内モニタリング方法を提供します40。内因性蛍光分光法、拡散反射分光法、およびラマン分光法を組み合わせた別のマルチモーダルモダリティが、外科的処置中のin situ癌検出のために開発された41。さらに、CARS、SHG、および2光子蛍光(TPF)を統合した最近設計されたマルチモーダル非線形内視鏡システムは、サブミクロンおよび細胞内空間分解能42で生体サンプルをイメージングする能力を実証しました。2PFとSRS顕微鏡の組み合わせは、組織、細胞、およびオルガネラ42,43,44,45の高解像度、生体内イメージングにも同様に利用されている。これらの新しいマルチモーダルイメージング技術は、個々のモダリティの長所を活用し、解像度、浸透深度、画像取得効率の向上につながり、臨床および外科用途にかなりの可能性を示しています。
このマルチモダリティアプローチは、個々の技術に関連する制限を軽減しながら、より広範な測定を提供するため、シングルモダリティイメージングよりもますます好まれています。前述したように、MPFは代謝変化を反映するために内因性蛍光を測定し、SHGは生体サンプル中のコラーゲンなどの非中心対称構造をイメージングでき、SRSは、振動モードに基づいて特徴的なラマンシグナルを生成する化学結合の密度が高いため、主にタンパク質と脂質を検出します。それらのコヒーレントな特性と非線形光学特性の共有原理を考えると、これらのイメージングモダリティは、超短パルスレーザーを使用して単一の顕微鏡セットアップに統合でき、局所領域でのさまざまなバイオマーカーの取得を可能にし、生物学的プロセスのより完全なビューを提供することができる44,45.このホワイトペーパーでは、生物医学研究アプリケーション向けにMPF、SHG、およびSRSを統合したマルチモーダルイメージングプラットフォームを実装するためのプロトコルについて概説します。
プロトコル
1. ラベルフリーのマルチモーダルイメージング実験
注:このプロトコルは、ラベルフリーマルチモーダルイメージングのセットアップと取得手順に焦点を当てています。
- マルチモーダル顕微鏡のセットアップ(図1A)とシステムキャリブレーション
- レーザーをウォームアップし、約15〜20分待ちます。
- コントロールユニットとモニターの電源を次の順序でオンにします:コントロールボックス|タッチパネルコントローラー |メインレーザーリモコン用ACアダプターサブレーザーリモコン用ACアダプター。
- Siフォトダイオード検出器とロックインアンプの電源を入れます。
- ポンプレーザービームとストークスビームを設定します。780 nmから990 nm、5 - 6 psのパルス幅、80 MHzの繰り返し周波数で調整可能なポンプビームでレーザーシステムをセットアップします。ストークスレーザービームの固定波長は1,031nmで、パルスは6ps、繰り返し周波数は80MHzです。ポンプビームとストークスビームの両方が低出力(少なくとも20 mW)で、アライメントプレートに表示されることを確認します。
- 光路上(レーザーボックスの直後)に1枚のアライメントプレートを配置して、ポンプとストークスビームの空間的な重なりを確認し、アライメントプレートの中心で両方のスポットが重なるまでミラーを調整します。
注:ポンプビームとストークスビームはほぼ重なり合っていますが、2つのレーザービームの空間的重なりを微調整することで、最適なSRS信号を得ることができます。 - 空間的なオーバーラップの後、最初に OPO制御 ソフトウェアをクリックして、PSD(Position Sensitive Detector)で微調整します。 六角レンチ を使用して光学ミラー1(OM1)を慎重に調整し、PSDディスプレイでXY位置が変化するのを確認して、中心からの偏差が最小になるようにします。次に、より正確な調整を行うには、位置インジケーターがPSDディスプレイの中央に来るまで、光学ミラー2(OM2)の 六角レンチ を使用します。
注意: OM1とOM2は、レーザーアライメントを調整するためのOPOヘッド内の2つのミラーです。 - 最後に、2 本のコンデンサーセンタリングネジを回してコンデンサーを中央に配置し、アイリスダイアフラム画像を視野の中心に移動します。
注:視野虹彩ダイアフラムは、対物レンズに入る光線の直径を制限し、したがって外来光を排除し、画像のコントラストを向上させることができます。
- ラベルフリーのマルチモーダルイメージング取得ステップ
- 高開口数(1.4 NA)のオイルコンデンサーにオイルを塗布し、顕微鏡スライドをオイルコンデンサーに取り付けます。最後に、25倍水対物レンズの顕微鏡スライドに大きな水滴を置きます。顕微鏡がしっかりと取り付けられ、動かないことを確認してから、zステージを調整して、生物学的サンプルの明視野画像が25倍の水対物レンズの下に見えるまで焦点を調整します。
- MPF、SHG、SRSの正しい順序でイメージングプロセスを開始し、MPFの画質と正確な分析に影響を与える光退色を回避します。MPF と SHG をすばやく切り替えるには、ポンプ ビームから固定ストークス ビームに切り替えます。
注:ポンプレーザーを800 nm(NADHおよびFAD自家蛍光)から791.3 nm(CH3 ラマンシフト)に調整するには、1〜2分かかる場合があります。 - 各モダリティの画像解像度(512 x 512 ピクセル)と適切な滞留時間を選択します。MPF と SHG には 8 μs/ピクセルを使用し、平均フレームは 3 を超えます。SRS モダリティには、平均フレーム 2 で 40 μs/ピクセルを使用します。
注:平均フレームとは、保存する前に平均化する画像の数を意味します。 - MPFで自家蛍光を取得するには、ストークスレーザービームをオフにし、ポンプレーザーを800 nmに調整してNADHとフラビンを励起します。FVOPTフィルターキューブを、NADHとフラビンにそれぞれ460 nm±10 nmと515 ± 10 nmで光路に取り付けます。したがって、サンプルの電力は約15mWです。
- SHGを用いてコラーゲン繊維のシグナルを取得します。ポンプのレーザービームをオフにし、ストークスのレーザービームのみを使用し、電力を500mWに設定します。515nmフィルターに関連するチャネルのみを取得します。
- SRSを用いてタンパク質や脂質の空間分布を取得します。両方のレーザービームをオンのままにし、各分子の特定の振動モードに一致するようにレーザービームの周波数を調整します。
注:通常、791.3 nmはCH3に使用され、797 nmはCH2に使用され、787 nmは不飽和脂質に使用され、794.6は飽和脂質に使用されます。したがって、サンプルの電力は約40mWです。 - SRS ハイパースペクトル画像データセットを取得するには、OPO 制御ソフトウェアを開いてスイープを選択し、波長範囲を 780 nm から 806.5 nm に設定し、スタック番号 を 60 以上選択します。次に、ハイパースペクトル イメージ スタックをキャプチャします。
注:結果のデータセットには60枚の画像が含まれており、それぞれが特定のラマンシフト(2,700 cm-1 から3,150 cm-1)での空間分布を表しています。ハイパースペクトル取得にかかる時間は、画像の視野 (FOV) とスキャン時間によって異なりますが、512 x 512 画像の場合、約 5 分から 8 分の間になります。より大きな画像の場合、2時間後に発生する可能性のある光損傷を防ぐために、レーザーの下の時間を最小限に抑えるように注意する必要があります(サンプルの厚さとFOVサイズによって異なります)。 - 同じ関心領域 (ROI) のすべての画像を同じフォルダーに保存します。画像形式はオリンパス.oirファイルです。
2. 画像解析
- 画像処理ソフトウェアを使用して、保存されたすべてのRAW画像を開き、色を割り当て、画像表示のスケールバーを追加します。また、NADHおよび飽和脂肪酸(SFA、2,880 cm-1)画像のバイナリマスクを生成して、光学酸化還元比および脂質不飽和分析にも使用します。
注:マスクの強度が0と1のみであることを確認してください。 - 自作のPythonスクリプトを使用して、ダウンストリーム解析を行います(コードは https://github.com/lingyanshi2020/HSI_Analysis で入手できます)。
注:このスクリプトは、コントロールサンプルとがんサンプルの両方から複数の関心領域(ROI)を処理するように設計されています。スクリプトのメイン関数に必要な入力は、control フォルダと cancer image フォルダのファイル パスの 2 つだけです。- フォルダを反復処理し、同じベース名(「roi_1_flavin.tif」、「roi_1_nadh.tif」、「roi1_mask.tif」など)の画像を一致させます。
- 一致したイメージのセットごとに、スクリプトでレシオメトリック分析を実行し、これらの計算に基づいて新しいレシオメトリック イメージを生成します。
光学酸化還元比は、フラビン/(NADH +フラビン)として計算されます。
脂質の不飽和度は、USFAs / (SFAs + USFAs)として計算されます。ここで、USFAは不飽和脂肪酸を表します。 - その後、画像のすべてのレシオメトリック値は、簡単に操作できるように pandas DataFrame に保存されます。一元配置ANOVAを実行し、matplotlibをstyple "ggplot"またはseabornと一緒に使用して、定量化のための棒ひげ図または箱ひげ図を行います。
- 2,930 cm-1 (CH3) と 2,850 cm-1 (CH2) の画像を使用して、減算された (CH3 - CH2) 画像を生成します。次いで、RGB色空間のCH2 画像CH3 〜CH2 画像をH&E染色を模倣するカスタムLUTとブレンドして、デジタル組織像46を生成する。
- ハイパースペクトル SRS 画像を取得した後、自作の Python スクリプトを使用して k-means クラスタリングを行い、スペクトルの類似性に基づいて画像をセグメント化します (コードは https://github.com/lingyanshi2020/HSI_Analysis で入手できます)。
- .tiff画像スタックをtifffileパッケージで読み込みます。SRS ハイパースペクトル イメージ スタック (x、y、スペクトル) を 2D 配列 (ピクセル、スペクトル) に形状変更します。
- scikit-learn パッケージを使用して、定義された数のクラスターを使用して、k-means アルゴリズムをこの 2D 配列にデプロイします。
- 結果のクラスター ラベルの形状を元の画像サイズに戻し、セグメンテーション マップを作成します。
- 各クラスターに一意の色を割り当て、組織内のさまざまな生化学的組成を表す偽色画像を生成します。
- matplotlib を使用して、各クラスターのスペクトルを平均値 (実線) と標準偏差 (影の領域) でプロットします
- MATLABを使用してペナルティ付きリファレンスマッチングSRS(PRM-SRS)プロトコルのプロトコルに従って、SRSハイパースペクトル画像データセット47を使用した空間ラベルフリー脂質サブタイプ検出を作成する。
- 画像を整理し、300 dpi .tif形式で保存します。
結果
図2の画像は、FADおよびNADHの自家蛍光を取得するためのプロトコル、ならびにタンパク質、総脂質、不飽和脂質、および飽和脂質の4つのSRSチャネルを追って得られた結果を表しています。ここでは、RGBカラーブレンディングを通じて、プロトコルに記載されている疑似組織学的画像も生成します。MPFチャンネルとSRSチャンネルをアクイジションすると、後でレシオメトリック解析に利用される画像ファイルが得られます。この分析の一例を図3のヒト肺組織で見ることができます。プロトコールでの画像取得に続いて、PythonまたはImageJを使用した画像解析方法論は、異なるチャネルの比率を活用して定量的な代謝情報を提供します。図3の脂質不飽和度および光学酸化還元比の画像に示されているように、レシオメトリック分析は、相対的な代謝活性および分子組成の分布のカラーマップを提供します。これらの測定を活用して、特定の組織、病状、または異なる生物学的因子の代謝経路と脂質含有量の変化に関する観察を行いました。図4に見られるように、これは、2D顕微鏡画像での分布に加えて、酸化ストレスと脂質不飽和の平均値を比較することにより、健康な組織と腫瘍組織の定量的な比較を提供できます。
MPF画像とSRS画像のレシオメトリック解析では、式 (1) と (2)に示すように、光学レドックスと不飽和の比率を一般的に対象としています。
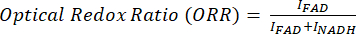 (1)
(1)
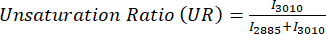 (2)
(2)
ここで、ICはチャネルCからのピクセルの強度です。したがって、ピクセルあたりの比率を測定し、これらの代謝マーカーと分子マーカーの空間分布を取得します。
レシオメトリック解析に加えて、 図4 は、マルチモーダルプラットフォームの別の潜在的なアプリケーションであるハイパースペクトル画像解析も示しています。このプロトコルに記載されているように、チューナブルポンプビームを利用して複数のレーザー波長にわたってスイープを行うことにより、SRSハイパースペクトル画像を取得できます。これにより、顕微鏡画像内の各ピクセルのラマンシフトスペクトルのCH領域を再構成し、ラマン分光法によって提供される化学情報と光学顕微鏡によって提供される空間情報の両方を結合することができます。画像解析のワークフローでは、これらのハイパースペクトル画像を解析するために実装された2つの手法、すなわち生体分子検出のためのPRM-SRSとk-meansクラスタリングに焦点を当てています。 図4E は、マウス肝腫瘍の画像に対するPRM-SRSの適用を示しています。PRM-SRSアルゴリズムは、ラマンスペクトルのシフトに対するペナルティによって調整された、個々のピクセルのスペクトル間の相関を提供します。次に、アルゴリズムは各脂質の確率分布画像を生成します。
ピクセルスペクトルのK平均クラスタリングは、特定のスペクトル表現型のグループ化を視覚化するために実装する別の手法であり、強度とスペクトル形状を通じて、結合濃度と分子組成による分離をそれぞれ可能にします。 図5 は、k-meansクラスタリングによる画像分析をプロトコルにどのように適用できるかの例を示しています。k-mean クラスターは、SRS チャネルや偽ヘマトキシリンおよびエオシン組織学的染色 (H&E) からは容易に識別できない特徴からさらにクラスタリングを提供することに加えて、疑似組織学の図の特徴と構造的に整列すると予想されます。PRM-SRSおよびk-meansクラスタリング解析は、外因性プローブを必要とせずに、特定の分析種および分子結合に関する化学的および半標的化可能な情報を提供することにより、このラベルフリーイメージングプラットフォームを補完します。
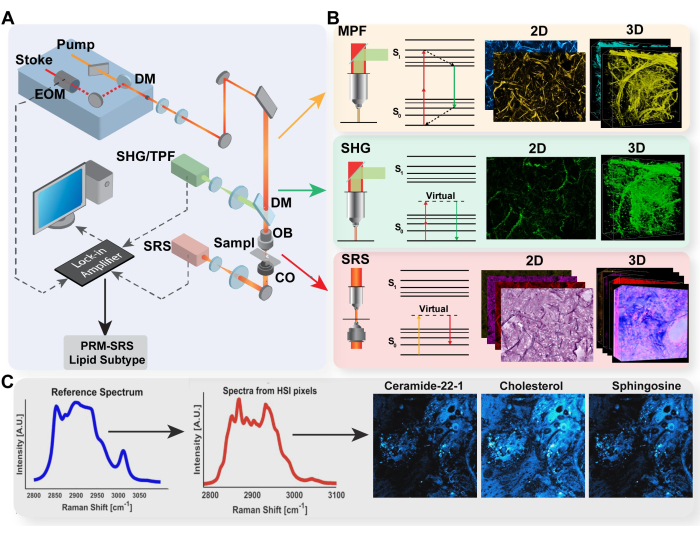
図1:マルチモーダルイメージングプラットフォームとPRM-SRS生体分子検出の図。 (A)MPF/TPF、SHG、およびSRSイメージングシステムのハードウェアセットアップの図。(B)TPF、SHG、およびSRSの光路とヤブロンスキー図、および各モダリティの代表的な取得画像。(C)自発的ラマン分光法とSRSハイパースペクトルイメージング間のスペクトル参照マッチングのためのPRM-SRSワークフロー図。略語:MPF =多光子蛍光;TPF = 2光子蛍光;SHG = 第2高調波発生;SRS = 誘導ラマン散乱;PRM = ペナルティ付き参照マッチング。 この図の拡大版を表示するには、ここをクリックしてください。
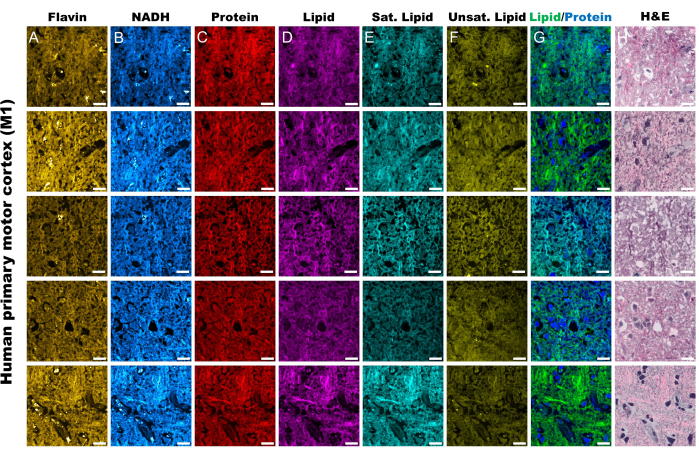
図2:ヒト一次運動皮質組織(M1)からのMPFおよびSRSチャネル。 (A,B)Flavin/FADおよびNADH用のMPF自家蛍光チャンネル。(C-F)特定のラマンシフトピークで捕捉されたSRSチャネル。タンパク質の CH3 不斉伸張ピーク (2,930 cm-1)、脂質の CH2 不斉伸張ピーク (2,845 cm-1)、飽和脂肪酸の 2,885 cm-1 ピーク、不飽和脂肪酸の 3,010 cm-1 ピークです。(G)SRSタンパク質(青)と脂質(緑)のチャネルを結合して、それぞれの空間分布を概説します。(H)H&E染色をミラーリングするSRS疑似組織学的画像(SRH)。スケールバー = 20 μm。略語:MPF =多光子蛍光;FAD =フラビンアデニンジヌクレオチド;NADH =還元型ニコチンアミドアデニンジヌクレオチド;SRS = 誘導ラマン散乱;SRH = SRS組織型。 この図の拡大版を表示するには、ここをクリックしてください。
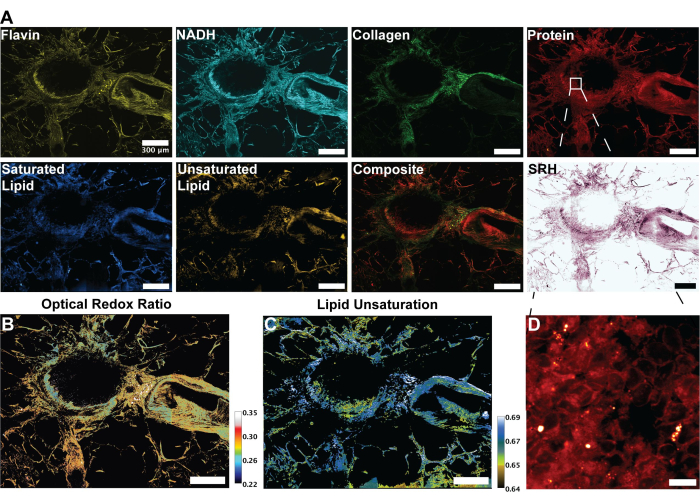
図3:MPF、SHG、およびSRS画像とヒト肺組織の解析。 (A)Flavin/FADおよびNADHのMPF自家蛍光、コラーゲン繊維のSHGシグナル、タンパク質のSRSシグナル(2,930 cm-1)、脂質(2,845 cm-1)、飽和脂質(2,885 cm-1)、不飽和脂質(3,010 cm-1)のピーク、および複数のモダリティの合成合成画像。(B)光学酸化還元比のレシオメトリック画像。(C)脂質不飽和度のレシオメトリック画像。(D)気管支内層の上皮細胞の拡大画像。スケールバー = 200 μm。略語:MPF =多光子蛍光;SHG = 第2高調波発生;SRS = 誘導ラマン散乱;FAD =フラビンアデニンジヌクレオチド;NADH = ニコチンアミドアデニンジヌクレオチドの減少。 この図の拡大版を表示するには、ここをクリックしてください。
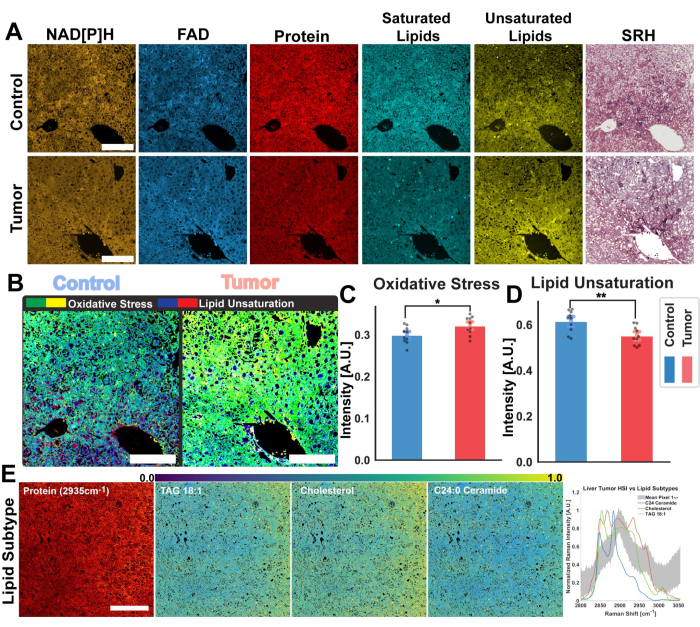
図4:マウス肝腫瘍のレシオメトリックおよびハイパースペクトル分析。 (A)NAD[P]HおよびFADのMPF自家蛍光画像、総タンパク質、飽和脂質、不飽和脂質のSRSチャネル、および健康な対照マウス肝臓(上)および腫瘍組織(下)の疑似組織学的画像。(B)光学酸化還元比で測定される酸化ストレス(黄緑色)、および対照組織(左)と腫瘍組織(右)から計算された脂質不飽和度(青赤)。スケールバー = 100 μm. (C,D) 対照(青)と腫瘍(赤)の肝臓サンプル間の酸化ストレスと脂質不飽和度のレシオメトリック差を示す棒グラフ。有意性統計量: p ≤ 0.05 の有意性の場合 *、p ≤ 0.01 の有意性の場合 **。(E)ハイパースペクトル画像からのPRM-SRS脂質サブタイプ解析結果。左から右へ:構造参照用のSRSタンパク質チャネル、スケールバー=100μm、脂質サブタイプ(TAG 18:1、コレステロール、C24:0セラミド)の確率分布画像。CH領域全体のSRS HSIからの脂質サブタイプ参照スペクトルと平均ピクセルスペクトルのグラフ。略語:MPF =多光子蛍光;SRS = 誘導ラマン散乱;NAD[P]H = 還元型ニコチンアミドアデニンジヌクレオチドリン酸;FAD =フラビンアデニンジヌクレオチド;HSI = ハイパースペクトル画像。 この図の拡大版を表示するには、ここをクリックしてください。
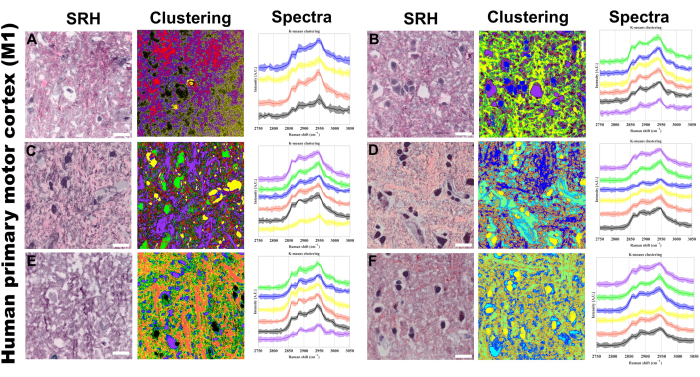
図5:ヒト一次運動野(M1)上のSRSハイパースペクトルk-meansクラスタリング。 (A-F) 6つの代表的な領域:左:H&E様組織可視化を提供する刺激ラマン組織学(SRH)画像;中央:対応するk-meansクラスタリング結果;右:平均強度(実線)と標準偏差(影付きの領域)を持つクラスター固有のラマンスペクトルプロファイル。4から6の範囲の重心の数は平均します。教師なしクラスタリングが使用されていたため、クラスター化された画像の異なる色は、k-means アルゴリズムによって識別された個別の化学組成を表しています。各クラスター (一意の色で表される) は、CH ストレッチ領域内の同様のスペクトル プロファイルを持つ領域に対応します。スケールバー = 20 μm。略語:SRH = SRS組織学。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
このマルチモーダルシステムは、幅広い生物学的起源や病理学的状態にわたるサンプルの分子環境を包括的に可視化するための強力なイメージングプラットフォームです。異なるラベルフリーモダリティを活用する利点は、補完的な情報を取得し、単一のラベルフリーイメージング技術では困難または不可能であった可能性のある特定の分析物を標的にする能力にあります。具体的には、この論文で言及されている3つの非線形イメージング技術(SRS、MPF、SHG)により、高分子組成、光学酸化還元比によるエネルギーダイナミクス、および細胞外マトリックスの組成と形態6,48,49を含む構造情報の定量化が可能になります。さらに、各イメージングモダリティを別々に使用する場合と比較して、1つの組み合わせた顕微鏡システムによるイメージングにより、即時の画像登録が可能になり、サンプルの保存時間が短縮されます。我々は、このアプローチでライブセルイメージングを実施しましたが、これはおそらく共焦点蛍光顕微鏡と比較して光曝露が低いためであり、上記のプロトコルで規定されているように、選択した時点を使用して各モダリティを順次取得します。マルチモーダルプラットフォームは、個々の利点だけでなく、さまざまなバイオマーカーのラベルフリー測定を通じて、生物学的状態に関する複数の視点からも恩恵を受けています。
MPFによる自己蛍光タンパク質のラベルフリーイメージングは、主に、FADのFAD+への酸化とNAD+のNADH50への還元によって促進される酸化還元反応の尺度である光学酸化還元比の定量化に向けられてきました。これは、ATP生成6の2つの主要な経路である酸化的リン酸化と解糖との間の相対活性の測定を提供するため、細胞および組織内の代謝の重要なマーカーです。具体的には、NADH濃度の低下とFAD+の増加は、ミトコンドリアの酸化的リン酸化の増加のマーカーです。一方、細胞質内の解糖系の増加については、その逆が当てはまります。エネルギー生産において一方の代謝経路を他方よりも好む傾向は、癌に関連するいくつかの病理学的変化および経路の活性化と関連しており、早期発見マーカーとしてのORRの潜在的な使用が強調されている6,51。
同様に、SHG線維性コラーゲンシグナルの取得は、非線形ラベルフリーイメージングモダリティを活用して、健康の生物学的マーカーを定量化および視覚化します。SHGは、その非中心対称構造により、I-III型コラーゲンの分布を効果的に追跡できます。SHGによって測定された線維性コラーゲンタンパク質は、癌や線維症を含むいくつかの疾患の重要な診断マーカーとして認識されています25,26,27,52,53。疾患のマーカーとしての検出に加えて、SHGを介したコラーゲンの獲得は、細胞外マトリックスの組成および組織54,55の構造境界における繊維状コラーゲンの役割により、細胞および組織の構造の強力な指標として役立つことができる。SHGコラーゲンシグナルを通じて、細胞膜の外因性マーカーを必要とせずに、別々の細胞または異なる組織機能単位の情報に基づいた観察を行うことができます。全体として、コラーゲンフィブリルのSHG定量は、他のラベルフリーイメージング技術と統合すると、診断能力と分析の容易さの両方に明確な利点をもたらします。
SRS顕微鏡法を利用することで、特に脂質や脂肪酸の高分子組成のいくつかのマーカーを捕捉することができます。このプロトコルでは、同じサンプル内の脂質不飽和のレシオメトリック分析のために、不飽和脂肪酸と飽和脂肪酸のSRSシグナルを取得する方法について説明します。25倍の水対物レンズを備えた正立レーザー走査型顕微鏡を適用して、近赤外スループットを実現しました。レーザービームはサンプルを通過し、高開口オイルコンデンサー(1.4 NA)によって収集されました。高外径ショートパスフィルター(950nm)を使用してストークスビームを遮断し、励起されたラマン損失信号を検出するためにポンプビームをSiフォトダイオード検出器にのみ到達させました。フォトダイオードからの出力電流を終端し、フィルタリングしました。その後、20MHzのロックインアンプがゼロ位相シフトでXの電流を復調し、ソフトウェアモジュールが復調された信号を使用してレーザースキャン中に画像を生成しました。
研究によると、脂質不飽和の変化は細胞膜とオルガネラ膜に大きな影響を及ぼし、脂質代謝の調節不全は、癌や神経変性疾患56,57,58,59を含むいくつかの疾患の強力なマーカーになる可能性があります。脂質調節不全のより包括的な概要として、SRSハイパースペクトルイメージングを使用してサブタイプ組成を分析します。SRSハイパースペクトル画像(HSI)は、SRS顕微鏡法によって取得された高解像度の空間情報と、プロトコルに記載されているシーケンシャルチューニングによるラマン分光法の化学的に重要な信号とを組み合わせます。次に、これらのピクセルスペクトルを、k-meansによるクラスタリングとPRM-SRSによる生体分子検出の2つの方法で分析します。
ピクセルスペクトルのK平均クラスタリングは、SRS信号強度と分子結合濃度60,61との間の線形関係により、分子濃度および組成によって2D画像内の領域を分離する結果となる。この方法論を通じて、プロトコルを適用して、ピクセルスペクトルと共有スペクトル重心によって規定された特定の分子表現型を持つ領域を確認します。特定のピクセルにおける明確な分子発現を徹底的に調べるために、PRM-SRSを実装して、自発ラマン分光法で捕捉された特定の分子の参照スペクトルとSRS HSI取得からのピクセルスペクトルとの間のスペクトルマッチングを実行します。そうすることで、外因性のプローブや標識を利用せずに、異なる分子の発現確率を判断できます。この分析は、組織および生物学的モデル全体で特定の脂質サブタイプを追跡する能力、および脂質組成変化の病理学的変化、したがって脂質異常症を特定する能力を実証した47。さらに、シリカ繊維を通るピコ秒パルス伝搬のスペクトル分割によるスペクトル分解能の向上における最近の進歩は、ラベルフリーの生化学的可視化のためのモダリティとしてのSRS HSIの適用をさらに奨励している62。プロトコルで強調されているこれらの方法を通じて、ラベルフリーのSRSモダリティを利用して包括的なレシオメトリックおよびスペクトル分析を実行するためのプラットフォームを確立します。
これらの手法の相対的な複雑さと新規性に加えて、考慮すべきアプローチにはいくつかの重要な制限があります。一般的に利用されているフェムト秒レーザーとは対照的に、ピコ秒パルスレーザーでMPF画像を取得するには、より高い蛍光色素励起電力が必要であり、光退色のリスクがあります。そのことを念頭に置いて、上記のプロトコルを最適化し、試料に入射するレーザー出力を減らすことにより、プラットフォームからの潜在的な光損傷を軽減しました。さらに、以前の研究では、NADHおよびFADのMPF自家蛍光イメージングからの光学酸化還元比を評価する能力が実証されているが、蛍光寿命イメージング顕微鏡(FLIM)は、エネルギー代謝の評価においてより高い精度を示している63,64,65。これは、NADHとFADのタンパク質結合および非結合濃度を、それらの異なる蛍光寿命に基づいて区別する能力によるものであり、MPFの能力と比較して、これによりFLIMは、酸化的リン酸化および解糖16,63,64,65,66,67を含む異なるエネルギー経路からの代謝活動の包括的な研究に使用することを可能にしました。68。このプロトコルはFLIMを除外していますが、近い将来、このモダリティがこのイメージングプラットフォームに統合され、現在実施しているエネルギー代謝分析が強化されることを期待しています。それにもかかわらず、ここで確立されたプロトコルを使用して、生物学的構造の代謝を複数の視点から測定できるマルチモーダルイメージングアプローチのワークフローとハードウェアのセットアップを提示します。
開示事項
著者は、宣言する利益相反を持っていません。
謝辞
グロリア・プライフーバー博士と彼女のHuBMAPチームメンバーに、ヒト肺組織スライスを提供していただいたことに感謝します。人間の脳組織を提供してくださったKun Zhang博士に感謝します。また、マウスの肝臓サンプルを提供してくださったGen-Sheng Feng博士にも感謝します。NIHU54DK134301、NIH R01GM149976、NIH U01AI167892、NIH R01HL170107、NIH 5R01NS111039、NIH R21NS125395、NIH U54CA132378、UCSD Startup funds、Sloan Research Fellow Award、CZI DAF2023-328667 Award からの支援に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 460 nm Filter Cube | Olympus | OCT-ET 460/50M32 | |
| Bandpass Filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| DC power supply | TopWard | 6302D | |
| Dichroic Mount | Thorlabs | KM100CL | |
| FIJI ImageJ | ImageJ | ||
| High NA oil condenser | Olympus | 6-U130 | |
| High O.D. shortpass filter | Thorlabs | 950 nm | |
| Inverted Laser Scanning Microscope | Olympus | FV1200MPE | |
| Lock-in Amplifier | Zurich Instruments | ||
| picoEmerald Laser System | Applied Physics & Electronics, Inc. | Synchronized pulsed pump beam (tunable 720–990 nm wavelength, 5–6 ps pulse width, and 80 MHz repetition rate) and Stokes (wavelength at 1032 nm, 6 ps pulse width, and 80 MHz repetition rate) | |
| SI Photodiode Detector | Home Built | ||
| Touch Panel Controller | Olympus | ||
| VF-300 Compresstome | Precisionary |
参考文献
- Wollman, A. J. M., Nudd, R., Hedlund, E. G., Leake, M. C. From Animaculum to single molecules: 300 years of the light microscope. Open Biol. 5 (4), 150019(2015).
- Vicidomini, G., Bianchini, P., Diaspro, A. STED super-resolved microscopy. Nat Methods. 15 (3), 173-182 (2018).
- Weber, M., et al. MINSTED fluorescence localization and nanoscopy. Nat Photonics. 15 (5), 361-366 (2021).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. , (1990).
- Hell, S. W., et al. Three-photon excitation in fluorescence microscopy. J Biomed Optics. 1 (1), 71-74 (1996).
- Alhallak, K., Rebello, L. G., Muldoon, T. J., Quinn, K. P., Rajaram, N. Optical redox ratio identifies metastatic potential-dependent changes in breast cancer cell metabolism. Biomed Opt Express. 7 (11), 4364-4374 (2016).
- Pena, A. -M., et al. Multiphoton FLIM imaging of NADH and FAD to analyze cellular metabolic activity of reconstructed human skin in response to UVA light. Multiphoton Microscopy in the Biomedical Sciences XIX. 10882, 23-33 (2019).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomed Opt Express. 11 (10), 5674-5688 (2020).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. J Biol Chem. 254 (11), 4764-4771 (1979).
- Ostrander, J. H., et al. Optical redox ratio differentiates breast cancer cell lines based on estrogen receptor status. Cancer Res. 70 (11), (2010).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proc Natl Acad Sci USA. 104 (49), 19494-19499 (2007).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Res. 74 (11), 3067-3075 (2014).
- Malak, M., James, J., Grantham, J., Ericson, M. B. Contribution of autofluorescence from intracellular proteins in multiphoton fluorescence lifetime imaging. Sci Rep. 12, 16584(2022).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Roth, S., Freund, I. Second harmonic generation in collagen. J Chem Phys. 70 (4), 1637-1643 (1979).
- Freund, I., Deutsch, M. Second-harmonic microscopy of biological tissue. Opt Lett. 11 (2), 94(1986).
- Boyd, R. W., Gaeta, A. L., Giese, E. Nonlinear optics. Springer Handbook of Atomic, Molecular, and Optical Physics. Drake, G. W. F. , Springer. 1097-1110 (2023).
- Aghigh, A., et al. Second harmonic generation microscopy: a powerful tool for bio-imaging. Biophys Rev. 15 (1), 43-70 (2023).
- Mohler, W., Millard, A. C., Campagnola, P. J. Second harmonic generation imaging of endogenous structural proteins. Methods. 29 (1), 97-109 (2003).
- Cox, G. Biological applications of second harmonic imaging. Biophys Rev. 3 (3), 131(2011).
- Brown, E., et al. Dynamic imaging of collagen and its modulation in tumors in vivo using second-harmonic generation. Nat Med. 9 (6), 796-800 (2003).
- Keikhosravi, A., Bredfeldt, J. S., Sagar, A. K., Eliceiri, K. W. Chapter 28 - Second-harmonic generation imaging of cancer. Methods Cell Bio. 123, 531-546 (2014).
- Hui Mingalone, C. K., et al. Bioluminescence and second harmonic generation imaging reveal dynamic changes in the inflammatory and collagen landscape in early osteoarthritis. Lab Invest. 98 (5), 656-669 (2018).
- Tsafas, V., et al. Polarization-dependent second-harmonic generation for collagen-based differentiation of breast cancer samples. J Biophotonics. 13 (10), e202000180(2020).
- Nadiarnykh, O., LaComb, R. B., Brewer, M. A., Campagnola, P. J. Alterations of the extracellular matrix in ovarian cancer studied by Second Harmonic Generation imaging microscopy. BMC Cancer. 10, 94(2010).
- Xiong, S. Y., Yang, J. G., Zhuang, J. Nonlinear spectral imaging of human normal skin, basal cell carcinoma and squamous cell carcinoma based on two-photon excited fluorescence and second-harmonic generation. Laser Phys. 21 (10), 1844-1849 (2011).
- Tilbury, K., Campagnola, P. J. Applications of second-harmonic generation imaging microscopy in ovarian and breast cancer. Perspect Medicin Chem. 7, 21-32 (2015).
- Raman, C. V., Krishnan, K. S. A New type of secondary radiation. Nature. 121 (3048), 501-502 (1928).
- Eckhardt, G., et al. Stimulated Raman scattering from organic liquids. Phys Rev Lett. 9 (11), 455-457 (1962).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Li, Y., et al. Microglial lipid droplet accumulation in tauopathy brain is regulated by neuronal AMPK. Cell Metab. 36 (6), 1351-1370.e8 (2024).
- Lu, F. -K., et al. Label-free neurosurgical pathology with stimulated Raman imaging. Cancer Res. 76 (12), 3451-3462 (2016).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nat Commun. 9 (1), 2995(2018).
- Zhang, L., et al. Spectral tracing of deuterium for imaging glucose metabolism. Nat Biomed Eng. 3 (5), 402-413 (2019).
- Li, Y., et al. Bioorthogonal stimulated Raman scattering imaging uncovers lipid metabolic dynamics in Drosophila brain during aging. GEN Biotechnol. 2 (3), 247-261 (2023).
- Li, X., et al. Quantitative imaging of lipid synthesis and lipolysis dynamics in Caenorhabditis elegans by stimulated Raman scattering microscopy. Anal Chem. 91 (3), 2279-2287 (2019).
- Ji, M., et al. Detection of human brain tumor infiltration with quantitative stimulated Raman scattering microscopy. Sci Transl Med. 7 (309), 309ra163(2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Sci Transl Med. 5 (201ra119), 201ra119(2013).
- You, S., et al. Intravital imaging by simultaneous label-free autofluorescence-multiharmonic microscopy. Nat Commun. 9, 2125(2018).
- You, S., et al. Label-free deep profiling of the tumor microenvironment. Cancer Res. 81 (9), 2534-2544 (2021).
- Jermyn, M., et al. Highly accurate detection of cancer in situ with intraoperative, label-free, multimodal optical spectroscopy. Cancer Res. 77 (14), 3942-3950 (2017).
- Pshenay-Severin, E., et al. Multimodal nonlinear endomicroscopic imaging probe using a double-core double-clad fiber and focus-combining micro-optical concept. Light Sci Appl. 10 (1), 207(2021).
- Wu, W., Li, X., Qu, J. Y., He, S. In vivo imaging of biological tissues with combined two-photon fluorescence and stimulated Raman scattering microscopy. J Vis Exp. (178), e63411(2021).
- Li, X., Jiang, M., Lam, J. W. Y., Tang, B. Z., Qu, J. Y. Mitochondrial imaging with combined fluorescence and stimulated Raman scattering microscopy using a probe of the aggregation-induced emission characteristic. J Am Chem Soc. 139 (47), 17022-17030 (2017).
- Li, Z., et al. Multimodal imaging of metabolic activities for distinguishing subtypes of breast cancer. Biomed Opt Express. 14 (11), 5764-5780 (2023).
- Fung, A. A., et al. Label-free optical biopsy reveals biomolecular and morphological features of diabetic kidney tissue in 2D and 3D. , (2024).
- Zhang, W., et al. Multi-molecular hyperspectral PRM-SRS microscopy. Nat Commun. 15 (1), 1599(2024).
- Xu, F. X., et al. Discrimination of lipid composition and cellular localization in human liver tissues by stimulated Raman scattering microscopy. J Biomed Opt. 29 (1), 016008(2024).
- Zoumi, A., Yeh, A., Tromberg, B. J. Imaging cells and extracellular matrix in vivo by using second-harmonic generation and two-photon excited fluorescence. Proc Natl Acad Sci USA. 99 (17), 11014-11019 (2002).
- Liaudanskaya, V., et al. Mitochondria dysregulation contributes to secondary neurodegeneration progression post-contusion injury in human 3D in vitro triculture brain tissue model. Cell Death Dis. 14 (8), 1-15 (2023).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, sub-types and early treatment response in breast cancer. Cancer Res. 73 (20), 6164-6174 (2013).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. Am J Pathol. 178 (3), 1221-1232 (2011).
- Sun, W., et al. Nonlinear optical microscopy: use of second harmonic generation and two-photon microscopy for automated quantitative liver fibrosis studies. J Biomed Optics. 13 (6), 064010(2008).
- Nejim, Z., Navarro, L., Morin, C., Badel, P. Quantitative analysis of second harmonic generated images of collagen fibers: a review. Res Biomed Eng. 39 (1), 273-295 (2023).
- Burke, K., Brown, E. The use of second harmonic generation to image the extracellular matrix during tumor progression. Intravital. 3 (3), e984509(2015).
- Balasubramanian, P., Prabhakaran, M. P., Sireesha, M., Ramakrishna, S. Collagen in human tissues: Structure, function, and biomedical implications from a tissue engineering perspective. Polymer Composites - Polyolefin Fractionation - Polymeric Peptidomimetics - Collagens. Abe, A., Kausch, H. H., Möller, M., Pasch, H. 251, 173-206 (2012).
- Dadsena, S., et al. Lipid unsaturation promotes BAX and BAK pore activity during apoptosis. Nat Commun. 15, 4700(2024).
- Barelli, H., Antonny, B. Lipid unsaturation and organelle dynamics. Curr Opin Cell Biol. 41, 25-32 (2016).
- Harayama, T., Antonny, B. Beyond fluidity: The role of lipid unsaturation in membrane function. Cold Spring Harb Perspect Biol. 15 (7), a041409(2023).
- Yoon, H., Shaw, J. L., Haigis, M. C., Greka, A. Lipid metabolism in sickness and in health: Emerging regulators of lipotoxicity. Mol Cell. 81 (18), 3708-3730 (2021).
- Villazon, J., Shi, L. Utilizing K-means clustering on hyperspectral DO-SRS images of the mouse hippocampus. CLEO 2024, Technical Digest Series. , Optica Publishing Group. (2024).
- Fu, D., et al. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy. J Am Chem Soc. 134 (8), 3623-3626 (2012).
- Periasamy, A., Diaspro, A. Special section guest editorial: Multiphoton microscopy. J Biomed Optics. 8 (3), 327(2003).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci USA. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nat Commun. 5, 3936(2014).
- Xiong, H., et al. Background-free imaging of chemical bonds by a simple and robust frequency-modulated stimulated Raman scattering microscopy. Opt Express. 28 (10), 15663-15677 (2020).
- Georgakoudi, I., Quinn, K. P. Label-free optical metabolic imaging in cells and tissues. Annu Rev Biomed Eng. 25, 413-443 (2023).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon FLIM microscopy. Sci Rep. 12 (1), 11938(2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved