Method Article
Plate-forme d’imagerie optique multimodale pour l’étude du métabolisme cellulaire
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
À l’aide d’une plate-forme multimodale combinant des modalités d’imagerie optique sans marquage, nous avons développé un protocole pour visualiser et quantifier la dynamique cellulaire et le métabolisme. Grâce à l’imagerie par fluorescence multiphotonique, à la génération de secondes harmoniques et à la microscopie à diffusion Raman stimulée, nous pouvons générer une vue d’ensemble holistique de l’environnement cellulaire et moléculaire.
Résumé
Les technologies d’imagerie optique sont essentielles dans les études biomédicales pour leur capacité à obtenir des informations morphologiques et fonctionnelles à partir d’échantillons biologiques à haute résolution spatiale. Ces processus optiques exploitent diverses interactions lumière-molécule, telles que la diffusion, l’absorption, l’émission et la génération d’harmoniques, entre les photons et les molécules à l’intérieur des cellules, des tissus ou des organes. Alors que l’imagerie biomédicale conventionnelle s’est historiquement concentrée sur l’application d’une seule modalité, des recherches récentes ont montré que ces diverses techniques fournissent des informations complémentaires, et que leurs résultats combinés offrent une compréhension plus complète des changements moléculaires dans les processus de vieillissement et le développement des maladies, ainsi que des principes fondamentaux de la biologie cellulaire.
Au cours des dernières décennies, les méthodes d’imagerie optique sans marquage ont progressé, permettant une exploration détaillée des environnements cellulaires et subcellulaires. Par exemple, la fluorescence multiphotonique (MPF) facilite non seulement l’imagerie ciblée des protéines, mais quantifie également l’activité métabolique grâce à des coenzymes autofluorescentes, ce qui permet d’obtenir une profondeur de pénétration et une résolution spatiale élevées. La génération de secondes harmoniques (SHG) est utilisée pour imager des structures telles que le collagène dans la matrice extracellulaire, tandis que la diffusion Raman stimulée (SRS) cartographie les liaisons chimiques et la composition moléculaire in situ avec une résolution subcellulaire.
Nous avons développé une plate-forme d’imagerie multimodale qui combine les modalités MPF, SHG et SRS. L’intégration de ces modalités dans une seule plateforme permet l’acquisition d’informations à multiples facettes à partir de la même localisation au sein des cellules, des tissus, des organes ou même des corps, facilitant ainsi une exploration plus détaillée des relations complexes entre le métabolisme cellulaire, la structure de la matrice extracellulaire et la composition moléculaire. Ce système multimodal offre une résolution subcellulaire, une pénétration profonde des tissus, une imagerie in situ des cellules vivantes/tissus, ainsi qu’une détection sans marquage et un co-enregistrement instantané sans qu’il soit nécessaire d’ajuster la position, de changer d’appareil ou d’aligner après analyse. Ici, nous présentons un protocole d’imagerie sans marquage avec cette plateforme multimodale et démontrons son application dans la caractérisation du métabolisme cellulaire et de l’hétérogénéité moléculaire dans les cellules et les tissus pour l’étude du vieillissement et des maladies.
Introduction
L’imagerie biomédicale optique a joué un rôle essentiel dans l’avancement de notre compréhension de la structure et de la fonction biologiques. Les images sont générées en modulant la lumière d’excitation et en détectant les signaux des interactions lumière-tissu. Le premier microscope composé, mis au point par Hans et Zacharias Janssen vers 1590, utilisait deux lentilles convexes dans un tube, offrant un grossissement jusqu’à 30x1. Les microscopes optiques modernes, après des siècles de progrès, peuvent maintenant atteindre des résolutions aussi fines que 1-3 nm 2,3. En plus d’offrir une haute résolution, les systèmes d’imagerie avancés offrent désormais une pénétration plus profonde des tissus, une plus grande efficacité et des dommages minimaux aux échantillons, ce qui les rend particulièrement adaptés à l’imagerie des cellules vivantes et des tissus. L’imagerie sans marquage est particulièrement avantageuse car elle permet de capturer des informations sans perturber les processus intracellulaires ni compromettre l’intégrité de l’échantillon.
La microscopie à fluorescence multiphotonique (MPF), en particulier la microscopie à fluorescence à deux photons, a été largement utilisée pour l’imagerie sans marquage. Contrairement à la microscopie à fluorescence conventionnelle, qui repose sur l’absorption et l’émission linéaires de photons uniques, l’excitation MPF implique l’absorption simultanée de plusieurs photons, dont l’énergie combinée excite une seule molécule de fluorophore 4,5. Ces photons, généralement dans le spectre infrarouge, possèdent la moitié ou moins de l’énergie requise pour l’excitation des photons uniques. Les longueurs d’onde plus longues et l’excitation localisée au point focal dans ce processus non linéaire entraînent une diffusion plus faible, une pénétration tissulaire plus profonde et une phototoxicité réduite.
L’information métabolique cellulaire peut être capturée par la microscopie MPF sans marquage grâce à la détection de signaux d’autofluorescence à partir de substrats métaboliques endogènes, tels que le nicotinamide adénine dinucléotide réduit (NADH) et la flavine adénine dinucléotide (FAD). Ces coenzymes présentent des spectres d’excitation et d’émission distincts, et leur rapport d’intensité de fluorescence, connu sous le nom de rapport redox (NADH/FAD), reflète l’état oxydatif de la cellule. Depuis que Britton Chance a introduit pour la première fois le concept du rapport redox en 1979, des rapports supplémentaires, y compris NAD(P)H/FAD, NAD(P)H/(FAD + NAD(P)H) et FAD/(FAD + NAD(P)H), ont été proposés 6,7,8,9. La quantification de ces rapports redox optiques via l’imagerie MPF fournit des informations précieuses sur la dynamique métabolique. Par exemple, l’imagerie MPF peut distinguer les cellules cancéreuses des cellules normales en fonction de leur métabolisme altéré, démontrant ainsi son potentiel pour le diagnostic du cancer 10,11,12. Cependant, la détection d’autofluorescence basée sur MPF présente des limites. D’autres fluorophores intrinsèques, tels que la kératine, peuvent contribuer à l’intensité de la fluorescence, entraînant une diaphonie spectrale et une interprétation inexacte du signal13. De plus, le rapport redox ne reflète que les changements globaux d’oxydoréduction cellulaire et ne fait pas de distinction entre le NADH provenant de différentes sources (p. ex., cytoplasmique ou mitochondriale) ou entre le NADH et le NAD(P)H, car les deux présentent des pics spectraux similaires à 450 nm, ce qui donne des signaux d’intensité mélangés14.
La génération de secondes harmoniques (SHG), un processus optique non linéaire démontré pour la première fois dans le domaine biomédical dans les années 1980, a été largement utilisé pour l’imagerie sans marquage des structures cellulaires15,16. Semblable à MPF, le SHG implique l’absorption simultanée de deux photons de la même énergie à partir d’un laser pulsé ultrarapide. Ces photons sont recombinés pour émettre un nouveau photon deux fois la fréquence de la lumière incidente, ce qui permet de détecter le signal de seconde harmonique. Cette interaction optique non linéaire se produit exclusivement dans les matériaux non centrosymétriques qui présentent une susceptibilité de second ordre non nulle à induire une polarisation pour générer le signal de seconde harmonique17,18. Cela rend SHG particulièrement efficace pour l’imagerie des protéines filamenteuses et des structures fibrillaires, telles que le collagène, la myosine et la tubuline, sans nécessiter de colorants de fluorescence exogènes 15,17,19,20. L’anomalie de l’abondance, de la rigidité, de l’alignement et de la structure de la fibrose et du collagène est répandue dans de nombreuses affections telles que l’inflammation et le cancer, ce qui fait du SHG un outil prometteur pour une détection efficace et non invasive de certaines maladies 21,22,23. L’application généralisée de l’imagerie SHG dans la recherche oncologique, y compris les études sur les cancers du sein, de l’ovaire et de la peau, a mis en évidence son rôle crucial dans la recherche fondamentale et les applications cliniques potentielles 24,25,26,27.
Différentes molécules présentent des niveaux d’énergie vibratoire distincts, qui induisent divers degrés de diffusion inélastique lorsqu’elles sont excitées par la lumière incidente - un phénomène caractérisé pour la première fois par C. V. Raman en 192828. L’effet Raman a depuis été largement utilisé en microscopie optique pour la détection de compositions moléculaires et tissulaires sans marquage exogène. La diffusion Raman stimulée (SRS) et la diffusion Raman anti-Stokes cohérente (CARS) excitent toutes deux des vibrations moléculaires de manière cohérente et exploitent l’interaction non linéaire de la lumière pour produire un signal plus fort par rapport à la spectroscopie Raman spontanée conventionnelle. Le phénomène de la SRS a été signalé pour la première fois en 196229. En 2008, ce mécanisme a été intégré à l’imagerie multiphotonique tridimensionnelle, permettant la détection sélective de produits chimiques en fonction des changements d’intensité dans les faisceaux de la pompe et de Stokes dus aux transitions vibrationnelles moléculaires30. Cette méthode minimise les interférences de fond non résonnantes, générant un signal d’intensité propre qui dépasse celui des CARS. L’imagerie SRS excelle dans la fourniture d’une imagerie multiplexée et hyperspectrale, permettant la détection simultanée de plusieurs liaisons chimiques et permettant une visualisation à haute résolution de la composition moléculaire dans des échantillons avec une profondeur de pénétration considérable. Bien qu’il s’agisse d’une technique relativement nouvelle, l’imagerie SRS s’est avérée efficace à la fois dans le diagnostic clinique et la recherche métabolique, in vivo et in vitro30,31,32,33,34,35,36. Par exemple, la SRS peut différencier les tissus infiltrés dans les tumeurs cérébrales du cortex et de la substance blanche en quantifiant le rapport lipides/protéines, ce qui permet de délimiter les marges tumorales de manière non invasive et sans marquage 37,38. De plus, les altérations métaboliques, souvent considérées comme des caractéristiques des maladies liées au vieillissement et au cancer, peuvent être évaluées quantitativement à l’aide de la SRS obtenue en détectant les liaisons carbone-deutérium dans des échantillons traités à l’eau lourde (D2O), permettant une mesure quantitative de la synthèse des protéines, de la lipogenèse et d’autres processus métaboliques macromoléculaires 31,33,34,35,36. La capacité de suivre les métabolites avec une résolution temporelle et spatiale élevée positionne la SRS comme un outil prometteur pour l’investigation et le diagnostic des maladies, avec un potentiel d’applications cliniques plus larges.
L’imagerie multimodale est devenue une approche puissante dans la recherche biomédicale, intégrant deux ou plusieurs modalités d’imagerie pour obtenir une compréhension plus complète des systèmes biologiques complexes au sein d’un même échantillon. En 2018, une technique de microscopie à autofluorescence-multiharmonique (SLAM) sans marquage a été introduite, intégrant la fluorescence à deux photons (2PF), la fluorescence à trois photons (3PF), le SHG et la génération de troisième harmoniques (THG)39. Cette approche facilite la visualisation simultanée des interactions cellulaires, des processus dynamiques et des composants individuels au sein du microenvironnement tumoral. La microscopie SLAM offre une perturbation minimale et des exigences de puissance laser réduites pour l’échantillon, ce qui permet le profilage des tissus profonds et fournit une méthode plus sûre pour la surveillance intravitale40. Une autre modalité multimodale, combinant la spectroscopie de fluorescence intrinsèque, la spectroscopie de réflectance diffuse et la spectroscopie Raman, a été développée pour la détection in situ du cancer pendant les procédures chirurgicales41. De plus, un système d’endoscopie non linéaire multimodal récemment conçu, qui intègre CARS, SHG et la fluorescence à deux photons (TPF), a démontré sa capacité à imager des échantillons biologiques à une résolution spatiale submicronique et subcellulairede 42. La microscopie combinée 2PF et SRS a également été utilisée pour l’imagerie in vivo à haute résolution des tissus, des cellules et des organites 42,43,44,45. Ces techniques d’imagerie multimodale émergentes exploitent les forces de différentes modalités, ce qui permet d’améliorer la résolution, la profondeur de pénétration et l’efficacité de l’acquisition d’images, montrant ainsi un potentiel considérable pour les applications cliniques et chirurgicales.
Cette approche multimodale est de plus en plus privilégiée par rapport à l’imagerie monomodale, car elle offre une gamme plus large de mesures tout en atténuant les limites associées aux techniques individuelles. Comme nous l’avons vu précédemment, le MPF mesure la fluorescence endogène pour refléter les changements métaboliques, le SHG peut imager des structures non centrosymétriques telles que le collagène dans des échantillons biologiques, et le SRS détecte principalement les protéines et les lipides en raison de la forte densité de liaisons chimiques qui génèrent des signaux Raman distinctifs en fonction de leurs modes vibrationnels. Compte tenu de leurs propriétés cohérentes et du principe commun des propriétés optiques non linéaires, ces modalités d’imagerie peuvent être intégrées dans une seule configuration de microscope à l’aide de lasers pulsés ultracourts, permettant l’acquisition de divers biomarqueurs dans des régions localisées pour fournir une vue plus complète des processus biologiques44,45. Cet article décrit un protocole pour la mise en œuvre d’une plate-forme d’imagerie multimodale qui intègre MPF, SHG et SRS pour des applications de recherche biomédicale.
Protocole
1. Expériences d’imagerie multimodale sans marquage
REMARQUE : Ce protocole se concentre sur la procédure de configuration et d’acquisition de l’imagerie multimodale sans étiquette.
- Configuration du microscope multimodal (Figure 1A) et étalonnage du système
- Réchauffez le laser et attendez environ 15-20 min.
- Allumez les unités de commande et les moniteurs dans l’ordre suivant : Boîtier de commande | Contrôleur à écran tactile | Adaptateur secteur pour télécommande laser principale Adaptateur secteur pour télécommande sous-laser.
- Allumez le détecteur de photodiodes Si et l’amplificateur de verrouillage.
- Configurez les faisceaux laser de la pompe et le faisceau Stokes. Configurez le système laser avec un faisceau de pompe réglable de 780 nm à 990 nm, une largeur d’impulsion de 5 à 6 ps et un taux de répétition de 80 MHz. Le faisceau laser Stokes a une longueur d’onde fixe de 1 031 nm avec une impulsion de 6 ps et un taux de répétition de 80 MHz . Assurez-vous que les faisceaux de la pompe et de Stokes sont à faible puissance (au moins 20 mW) pour être visibles sur la plaque d’alignement.
- Placez une plaque d’alignement dans le chemin optique (juste après le boîtier laser) pour vérifier le chevauchement spatial des pompes et des faisceaux Stokes et ajustez le miroir jusqu’à ce que les deux points se chevauchent au centre de la plaque d’alignement.
REMARQUE : Bien que les pompes et les faisceaux Stokes soient grossièrement chevauchés, le réglage précis du chevauchement spatial de deux faisceaux laser peut obtenir le signal SRS optimal. - Après le chevauchement spatial, affinez le PSD (Position Sensitive Detector) en cliquant d’abord sur le logiciel de contrôle OPO . Utilisez la clé hexagonale pour régler soigneusement le miroir optique 1 (OM1) et surveillez l’écran PSD pour le changement de position X-Y afin de viser l’écart minimal par rapport au centre. Ensuite, pour effectuer des réglages plus précis, utilisez la clé hexagonale sur le miroir optique 2 (OM2) jusqu’à ce que l’indicateur de position soit centré sur l’écran PSD.
REMARQUE : OM1 et OM2 sont deux miroirs dans la tête OPO pour ajuster l’alignement du laser. - Enfin, centrez le condenseur en tournant les deux vis de centrage du condenseur pour déplacer l’image du diaphragme de l’iris vers le centre du champ de vision.
REMARQUE : Le diaphragme à iris de champ peut restreindre le diamètre du faisceau de lumière entrant dans l’objectif et ainsi exclure la lumière étrangère, améliorant ainsi le contraste de l’image.
- Étapes d’acquisition d’imagerie multimodale sans étiquette
- Appliquez de l’huile sur le condenseur d’huile à grande ouverture numérique (1,4 NA) et montez la lame du microscope sur le condenseur huilé. Enfin, placez une grosse goutte d’eau sur la lame du microscope pour l’objectif à eau 25x. Assurez-vous que le microscope est solidement monté et inamovible, puis ajustez la platine z pour régler la mise au point jusqu’à ce que l’image en fond clair de l’échantillon biologique puisse être vue sous l’objectif d’eau 25x.
- Commencez le processus d’imagerie dans le bon ordre : MPF, SHG et SRS pour éviter le photoblanchiment, ce qui aura un impact sur la qualité de l’image et l’analyse précise du MPF. Pour basculer rapidement entre MPF et SHG, passez du faisceau de pompe au faisceau de Stokes fixe.
REMARQUE : Le réglage du laser de la pompe de 800 nm (autofluorescence NADH et FAD) à 791,3 nm (décalage Raman CH3 ) peut prendre 1 à 2 min. - Sélectionnez la résolution de l’image (512 x 512 pixels) et le temps d’attente approprié pour chaque modalité. Utilisez 8 μs/pixel pour le MPF et le SHG avec une image moyenne supérieure à 3. Utilisez 40 μs/pixel avec une image moyenne de 2 pour la modalité SRS.
REMARQUE : L’image moyenne correspond au nombre d’images dont la moyenne est calculée avant l’enregistrement. - Pour acquérir l’autofluorescence avec MPF, désactivez le faisceau laser Stokes et réglez le laser de la pompe à 800 nm pour exciter le NADH et la flavine. Installez le cube de filtre FVOPT dans le trajet lumineux avec 460 ± 10 nm et 515 ± 10 nm pour le NADH et la flavine, respectivement. La puissance sur l’échantillon est donc d’environ 15 mW.
- Acquérez le signal de la fibre de collagène à l’aide de SHG. Éteignez le faisceau laser de la pompe, n’utilisez que le faisceau laser stokes et réglez la puissance sur 500 mW. N’acquérez que le canal lié au filtre 515 nm.
- Obtenir la distribution spatiale des protéines et des lipides à l’aide de la SRS. Gardez les deux faisceaux laser allumés et ajustez la fréquence du faisceau laser pour qu’elle corresponde au mode vibratoire spécifique de chaque molécule.
REMARQUE : En règle générale, 791,3 nm est utilisé pour le CH3, 797 nm pour le CH2, 787 nm pour les lipides insaturés et 794,6 pour les lipides saturés. La puissance sur l’échantillon est donc d’environ 40 mW. - Pour acquérir les ensembles de données d’images hyperspectrales SRS, ouvrez le logiciel de contrôle OPO pour sélectionner le balayage, réglez la plage de longueurs d’onde de 780 nm à 806,5 nm et choisissez un nombre de piles d’au moins 60. Ensuite, capturez la pile d’images hyperspectrales.
REMARQUE : L’ensemble de données résultant contient 60 images, chacune représentant la distribution spatiale à un décalage Raman spécifique (de 2 700 cm-1 à 3 150 cm-1). Le temps d’acquisition hyperspectrale dépend du champ de vision (FOV) de l’image et du temps de balayage, mais sera d’environ 5 min à 8 min pour une image de 512 x 512. Pour les images plus grandes, il faut veiller à minimiser la durée sous le laser afin d’éviter tout photodommage, qui peut survenir après 2 h (en fonction de l’épaisseur de l’échantillon et de la taille du champ de vision). - Enregistrez toutes les images des mêmes régions d’intérêt (ROI) dans le même dossier. Le format de l’image est Olympus .oir file.
2. Analyse d’images
- Utilisez un logiciel de traitement d’image pour ouvrir toutes les images brutes enregistrées afin d’attribuer la couleur et d’ajouter la barre d’échelle pour l’affichage de l’image. Utilisez-le également pour générer le masque binaire d’images de NADH et d’acides gras saturés (AGS, 2 880 cm-1) pour l’analyse optique du rapport redox et de l’insaturation lipidique.
REMARQUE : Assurez-vous que l’intensité du masque n’a que 0 et 1. - Utilisez le script python maison pour effectuer toute analyse en aval (le code est disponible à l’adresse https://github.com/lingyanshi2020/HSI_Analysis).
REMARQUE : Ce script est conçu pour traiter plusieurs régions d’intérêt (ROI) à partir d’échantillons de contrôle et d’échantillons de cancer. La fonction principale du script ne nécessite que deux entrées : les chemins d’accès aux fichiers des dossiers de contrôle et d’image du cancer.- Parcourez les dossiers en faisant correspondre les images portant le même nom de base (par exemple, « roi_1_flavin.tif », « roi_1_nadh.tif », « roi1_mask.tif »).
- Pour chaque ensemble d’images correspondantes, laissez le script effectuer une analyse ratiométrique et générer de nouvelles images ratiométriques en fonction de ces calculs.
Le rapport redox optique est calculé comme suit : flavine / (NADH + flavine).
L’insaturation lipidique est calculée comme suit : AGUS / (AGS + AGUS) où les AGUS représentent les acides gras insaturés. - Toutes les valeurs ratiométriques des images sont ensuite stockées dans un DataFrame pandas pour une manipulation facile. Effectuez une ANOVA unidirectionnelle et utilisez matplotlib avec styple « ggplot » ou seaborn pour faire le barplot ou le boxplot pour la quantification.
- Utilisez les images de 2 930 cm-1 (3 cm) et de 2 850 cm-1 (22) pour générer une image soustraite (3 - 2 en2). Ensuite, fusionnez l’image CH2 CH3 - l’image CH2 dans l’espace colorimétrique RVB avec une LUT personnalisée qui imite la coloration H&E pour générer l’image histologique numérique46.
- Après l’acquisition d’images SRS hyperspectrales, utilisez le script python fait maison pour effectuer le clustering k-means afin de segmenter les images en fonction des similitudes spectrales (le code est disponible à l’adresse https://github.com/lingyanshi2020/HSI_Analysis).
- Chargez les piles d’images .tiff avec le paquet tifffile. Remodelez la pile d’images hyperspectrales SRS (x, y, spectres) en un réseau 2D (pixels, spectres).
- Utilisez le package scikit-learn pour déployer l’algorithme k-means sur ce tableau 2D, avec le nombre défini de clusters.
- Remodelez les étiquettes de cluster résultantes aux dimensions de l’image d’origine, en créant une carte de segmentation.
- Attribuez des couleurs uniques à chaque grappe, générant une image en fausses couleurs qui représente différentes compositions biochimiques au sein du tissu.
- Utilisez matplotlib pour tracer les spectres de chaque cluster avec la valeur moyenne (ligne continue) et l’écart-type (zone d’ombre)
- Suivez le protocole du protocole PRM-SRS (Penalized Reference Matching SRS) à l’aide de MATLAB pour créer la détection spatiale de sous-types lipidiques sans marquage avec des ensembles de données d’images hyperspectrales SRS47.
- Organisez les images et enregistrez-les dans un format .tif de 300 dpi.
Résultats
Les images de la figure 2 sont représentatives des résultats obtenus en suivant le protocole d’acquisition de l’autofluorescence du FAD et du NADH ainsi que les quatre canaux SRS pour les protéines, les lipides totaux, les lipides insaturés et les lipides saturés. Ici, nous générons également une image pseudo-histologique comme décrit dans le protocole grâce à la fusion des couleurs RVB. L’acquisition des canaux MPF et SRS fournit les fichiers d’image qui seront ensuite utilisés dans l’analyse ratiométrique. Un exemple de cette analyse peut être vu dans la figure 3 sur le tissu pulmonaire humain. Suite à l’acquisition d’images dans le protocole, notre méthodologie d’analyse d’images utilisant Python ou ImageJ exploite le rapport des canaux distincts pour fournir des informations métaboliques quantitatives. Comme le montrent les images de l’insaturation lipidique et du rapport redox optique de la figure 3, l’analyse ratiométrique fournit une carte en couleur de la distribution de l’activité métabolique relative et de la composition moléculaire. Nous avons exploité ces mesures pour faire des observations concernant les changements dans les voies métaboliques et la teneur en lipides de tissus spécifiques, de pathologies ou de facteurs biologiques distincts. Comme le montre la figure 4, cela peut fournir une comparaison quantitative des tissus sains et tumoraux en comparant les valeurs moyennes du stress oxydatif et de l’insaturation lipidique en plus de sa distribution dans une image de microscopie 2D.
Pour l’analyse ratiométrique des images MPF et SRS, nous ciblons généralement l’oxydoréduction optique et le rapport d’insaturation, comme le montrent les équations (1) et (2).
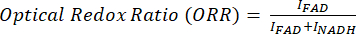 (1)
(1)
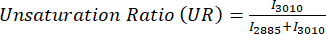 (2)
(2)
Où IC est l’intensité des pixels du canal C. Ainsi, nous mesurons le rapport par pixel et acquérons la distribution spatiale de ces marqueurs métaboliques et moléculaires.
En plus de l’analyse ratiométrique, la figure 4 démontre également une autre voie d’application potentielle pour notre plateforme multimodale : l’analyse d’images hyperspectrales. Comme décrit dans ce protocole, nous pouvons acquérir une image hyperspectrale SRS en effectuant un balayage sur plusieurs longueurs d’onde laser en utilisant un faisceau de pompe accordable. Cela nous permet de reconstruire la région CH du spectre de décalage Raman pour chaque pixel de l’image de microscopie, en joignant à la fois les informations chimiques fournies par la spectroscopie Raman et les informations spatiales fournies par la microscopie optique. Dans le flux de travail pour l’analyse d’images, nous mettons en évidence deux techniques mises en œuvre pour l’analyse de ces images hyperspectrales : PRM-SRS pour la détection de biomolécules et le clustering k-means. La figure 4E montre l’application de la PRM-SRS sur une image d’une tumeur hépatique de souris. L’algorithme PRM-SRS fournit la corrélation entre les spectres de chaque pixel individuel, ajustée par une pénalité pour les décalages dans le spectre Raman ; L’algorithme génère ensuite des images de distribution de probabilité pour chacun des lipides.
Le regroupement des valeurs K des spectres de pixels est une autre technique que nous mettons en œuvre pour visualiser le regroupement de phénotypes spectraux spécifiques, permettant la séparation par concentration de liaisons et composition moléculaire par l’intensité et la forme spectrale respectivement. La figure 5 fournit un exemple de la façon dont l’analyse d’images via le clustering k-means peut être appliquée dans notre protocole. Nous nous attendons à ce que les grappes de la moyenne k s’alignent structurellement avec les caractéristiques des figures pseudo-histologiques, en plus de fournir un regroupement supplémentaire à partir de caractéristiques qui ne sont pas aussi facilement discernables à partir des canaux SRS ou de la coloration histologique de la pseudo-hématoxyline et de l’éosine (H&E). L’analyse PRM-SRS et l’analyse par clustering k-means complètent cette plateforme d’imagerie sans marquage en fournissant des informations chimiques et semi-ciblables concernant des analytes spécifiques et des liaisons moléculaires sans avoir besoin de sonder exogène.
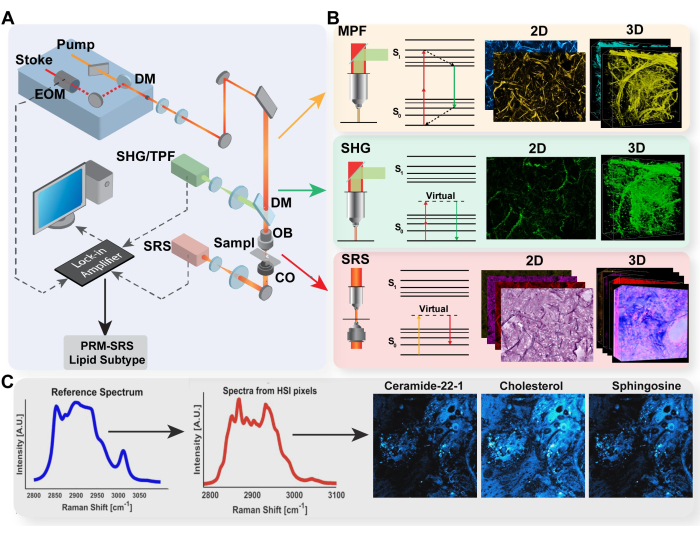
Figure 1 : Schéma de la plateforme d’imagerie multimodale et de la détection de biomolécules PRM-SRS. (A) Schéma de configuration matérielle pour le système d’imagerie MPF/TPF, SHG et SRS. (B) Trajet lumineux et diagramme de Jablonski pour TPF, SHG et SRS avec des images acquises représentatives pour chaque modalité. (C) Diagramme de flux de travail PRM-SRS pour l’appariement des références spectrales entre la spectroscopie Raman spontanée et l’imagerie hyperspectrale SRS. Abréviations : MPF = fluorescence multiphotonique ; TPF = fluorescence à deux photons ; SHG = génération de deuxième harmonique ; SRS = diffusion Raman stimulée ; PRM = correspondance de référence pénalisée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
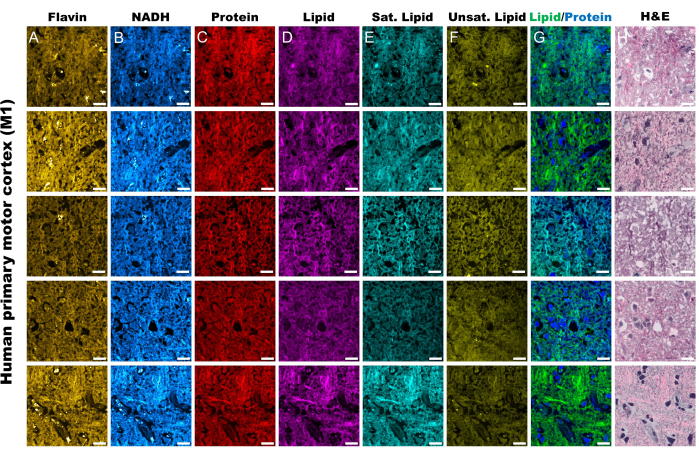
Figure 2 : Canaux MPF et SRS du tissu du cortex moteur primaire humain (M1). (A,B) Canaux d’autofluorescence MPF pour Flavine/FAD et NADH. (C-F) Canaux SRS capturés à des pics de décalage Raman spécifiques ; le pic d’étirement asymétrique CH3 (2 930 cm-1) pour les protéines, le pic d’étirement asymétrique CH2 (2 845 cm-1) pour les lipides, le pic de 2 885 cm-1 pour les acides gras saturés et le pic de 3 010 cm-1 pour les acides gras insaturés. (G) Fusion des canaux protéiques SRS (bleu) et lipidiques (vert) pour décrire leur distribution spatiale respective. (H) Images pseudo-histologiques (SRH) SRS reflétant la coloration H&E. Barres d’échelle = 20 μm. Abréviations : MPF = fluorescence multiphotonique ; FAD = flavine adénine dinucléotide ; NADH = nicotinamide adénine dinucléotide réduit ; SRS = diffusion Raman stimulée ; SRH = histologie SRS. Veuillez cliquer ici pour voir une version agrandie de cette figure.
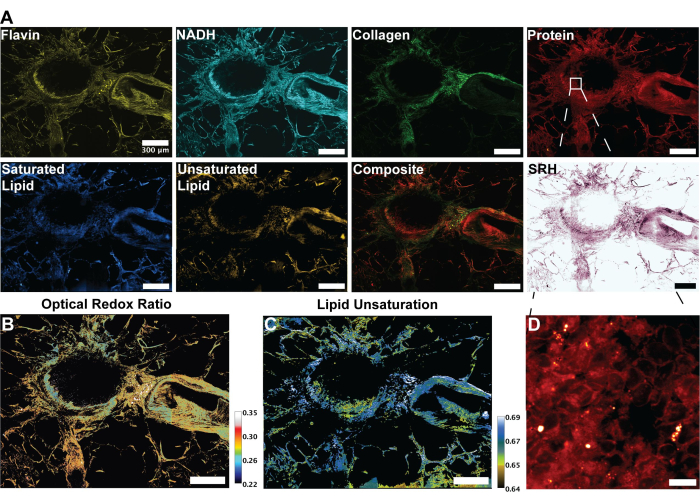
Figure 3 : Images MPF, SHG et SRS et analyse du tissu pulmonaire humain. (A) Autofluorescence MPF pour les pics de flavine/FAD et de NADH, signal SHG pour les fibres de collagène, signal SRS pour les protéines (2 930 cm-1), les lipides (2 845 cm-1), les lipides saturés (2 885 cm-1) et les lipides insaturés (3 010 cm-1), et une image composite fusionnée des multiples modalités. (B) Image ratiométrique du rapport redox optique. (C) Image ratiométrique de l’insaturation lipidique. (D) Image agrandie des cellules épithéliales de la couche interne des bronches. Barres d’échelle = 200 μm. Abréviations : MPF = fluorescence multiphotonique ; SHG = génération de deuxième harmonique ; SRS = diffusion Raman stimulée ; FAD = flavine adénine dinucléotide ; NADH = nicotinamide adénine dinucléotide réduit. Veuillez cliquer ici pour voir une version agrandie de cette figure.
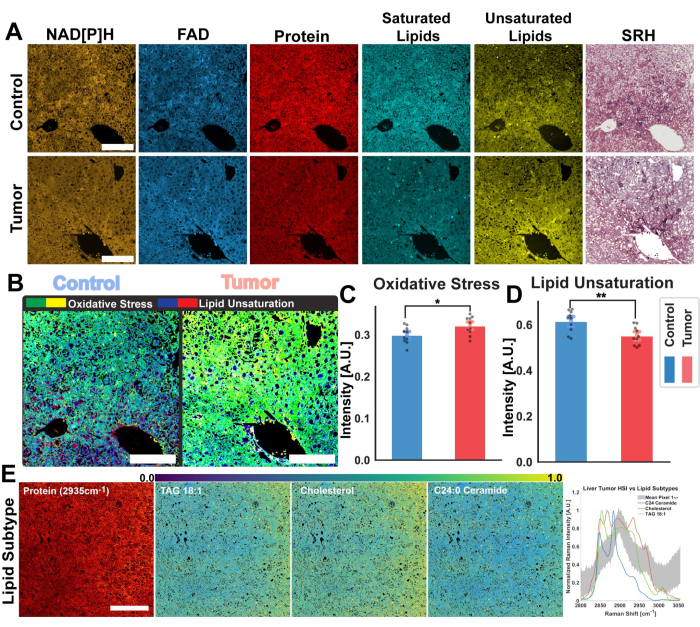
Figure 4 : Analyse ratiométrique et hyperspectrale des tumeurs hépatiques de souris. (A) Images d’autofluorescence MPF pour le NAD[P]H et le FAD, canaux SRS pour les protéines totales, les lipides saturés et insaturés, et images pseudo-histologiques pour le foie de souris témoin saine (en haut) et les tissus tumoraux (en bas). (B) Le stress oxydatif (jaune-vert), mesuré par le rapport redox optique, et l’insaturation lipidique (bleu-rouge) calculée à partir des tissus témoins (gauche) et tumoraux (droite). Barres d’échelle = 100 μm. (C,D) Graphiques à barres montrant les différences ratiométriques de stress oxydatif et d’insaturation lipidique entre les échantillons de foie témoin (bleu) et tumoral (rouge). Statistiques de signification : * pour une signification de p ≤ 0,05, ** pour une signification de p ≤ 0,01. (E) Résultats de l’analyse des sous-types lipidiques PRM-SRS à partir d’images hyperspectrales. De gauche à droite : canal protéique SRS pour référence structurelle, barre d’échelle = 100 μm, image de distribution de probabilité des sous-types lipidiques (TAG 18:1, cholestérol et céramide C24:0). Graphique des spectres de référence des sous-types lipidiques et des spectres de pixels moyens de l’IHM de la SRS dans la région CH. Abréviations : MPF = fluorescence multiphotonique ; SRS = diffusion Raman stimulée ; NAD[P]H = nicotinamide adénine dinucléotide phosphate réduit ; FAD = flavine adénine dinucléotide ; HSI = images hyperspectrales. Veuillez cliquer ici pour voir une version agrandie de cette figure.
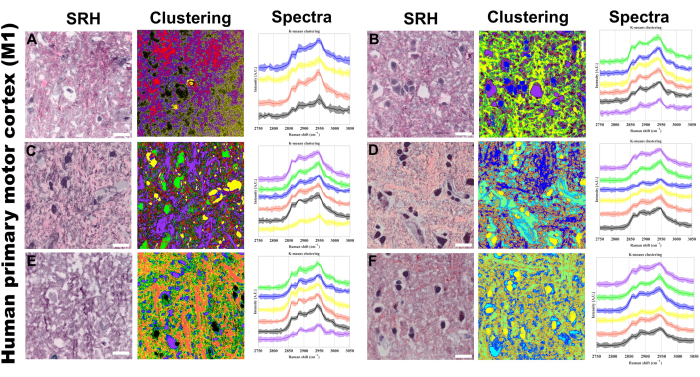
Figure 5 : Regroupement hyperspectral des k-moyennes SRS sur le cortex moteur primaire humain (M1). (A-F) Six régions représentatives montrant : à gauche : images de l’histologie Raman stimulée (SRH) fournissant une visualisation tissulaire de type H&E ; au milieu : résultats correspondants du regroupement des k-moyennes ; À droite : profils spectraux Raman spécifiques aux clusters avec des intensités moyennes (lignes continues) et des écarts-types (zones ombrées). Le nombre de centroïdes allant de 4 à 6 signifie. Le clustering non supervisé a été utilisé, de sorte que différentes couleurs dans les images groupées représentent des compositions chimiques distinctes identifiées par l’algorithme k-means. Chaque cluster (représenté par une couleur unique) correspond à des régions ayant des profils spectraux similaires dans la région d’étirement CH. Barre d’échelle = 20 μm. Abréviations : SRH = Histologie SRS. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Ce système multimodal est une puissante plate-forme d’imagerie permettant de capturer une visualisation holistique de l’environnement moléculaire d’échantillons à travers un large éventail d’origines biologiques et de conditions pathologiques. L’avantage d’exploiter différentes modalités sans marquage réside dans la capacité d’acquérir des informations complémentaires et de cibler des analytes spécifiques qui pourraient autrement être difficiles, voire impossibles, avec une seule technique d’imagerie sans marquage. Plus précisément, les trois techniques d’imagerie non linéaire (SRS, MPF, SHG) mentionnées dans cet article permettent de quantifier la composition des macromolécules, la dynamique de l’énergie via le rapport redox optique et les informations structurales, y compris la composition et la morphologie de la matrice extracellulaire 6,48,49. De plus, par rapport à l’utilisation de chaque modalité d’imagerie séparément, l’imagerie avec un système de microscope combiné permet un enregistrement immédiat des images et des temps de stockage d’échantillons plus courts. Nous avons même réalisé une imagerie de cellules vivantes avec cette approche, peut-être en raison de la photoexposition plus faible par rapport à la microscopie à fluorescence confocale, en utilisant des points temporels sélectionnés pour acquérir chaque modalité séquentiellement comme stipulé dans le protocole ci-dessus. Outre leurs avantages individuels, la plate-forme multimodale bénéficie également des multiples perspectives sur l’état biologique grâce à la mesure sans marquage de différents biomarqueurs.
L’imagerie sans marquage des protéines autofluorescentes via MPF a principalement été orientée vers la quantification du rapport redox optique, une mesure des réactions redox facilitées par l’oxydation de FAD en FAD+ et la réduction de NAD+ en NADH50. Il s’agit d’un marqueur important du métabolisme au sein des cellules et des tissus, car il permet de mesurer l’activité relative entre la phosphorylation oxydative et la glycolyse, les deux principales voies de génération d’ATP6. Plus précisément, les diminutions de la concentration de NADH et les augmentations de FAD+ sont un marqueur de l’augmentation de la phosphorylation oxydative dans les mitochondries ; Pendant ce temps, l’inverse est vrai pour une glycolyse accrue dans le cytoplasme. La tendance à préférer une voie métabolique à l’autre dans la production d’énergie a été associée à plusieurs changements pathologiques et activations de voies liées au cancer, mettant en évidence l’utilisation potentielle de l’ORR comme marqueur de détection précoce 6,51.
De même, l’acquisition du signal de collagène fibreux SHG exploite une modalité d’imagerie non linéaire sans marquage pour quantifier et visualiser un marqueur biologique de la santé. SHG peut suivre efficacement la distribution du collagène de type I-III en raison de sa structure non centrosymétrique. Les protéines de collagène fibreuses mesurées par SHG ont été reconnues comme des marqueurs diagnostiques importants pour plusieurs maladies, notamment le cancer et la fibrose 25,26,27,52,53. En plus de sa détection en tant que marqueur de la maladie, notre acquisition de collagène via SHG peut servir d’indicateur fort de la structure des cellules et des tissus en raison du rôle du collagène fibreux dans la composition de la matrice extracellulaire et les limites structurelles à travers les tissus54,55. Grâce au signal de collagène SHG, nous pouvons ensuite effectuer des observations éclairées de cellules distinctes ou d’unités fonctionnelles tissulaires distinctes sans avoir besoin d’un marqueur exogène pour les membranes cellulaires. Dans l’ensemble, la quantification SHG des fibrilles de collagène présente un avantage évident à la fois pour la capacité de diagnostic et la facilité d’analyse lorsqu’elle est intégrée à d’autres techniques d’imagerie sans marquage.
En utilisant la microscopie SRS, nous pouvons capturer plusieurs marqueurs de la composition des macromolécules, en particulier pour les lipides et les acides gras. Dans ce protocole, nous décrivons comment acquérir le signal SRS pour les acides gras insaturés et saturés pour l’analyse ratiométrique de l’insaturation lipidique au sein du même échantillon. Un microscope à balayage laser vertical avec un objectif à eau 25x a été utilisé pour un débit proche infrarouge. Les faisceaux laser ont traversé l’échantillon et ont été collectés par un condenseur d’huile à grande ouverture numérique (1,4 NA). Un filtre passe-court à haute portée (950 nm) a été utilisé pour bloquer le faisceau de Stokes tout en permettant uniquement au faisceau de la pompe d’atteindre un détecteur de photodiode Si pour détecter le signal de perte Raman stimulé. Le courant de sortie de la photodiode a été terminé et filtré. Par la suite, un amplificateur à verrouillage à 20 MHz a démodulé le courant en X avec un déphasage nul, et un module logiciel a utilisé le signal démodulé pour générer l’image pendant le balayage laser.
Des études ont montré que les changements dans l’insaturation des lipides ont un effet profond sur la membrane cellulaire et des organites et qu’une dérégulation du métabolisme des lipides peut être un marqueur fort de plusieurs maladies, y compris le cancer et les maladies neurodégénératives 56,57,58,59. Pour une vue d’ensemble plus complète de la dérégulation lipidique, nous analysons la composition des sous-types à l’aide de l’imagerie hyperspectrale SRS. Les images hyperspectrales (HSI) SRS combinent les informations spatiales à haute résolution acquises par la microscopie SRS avec le signal chimiquement significatif de la spectroscopie Raman par le biais d’un réglage séquentiel tel que décrit dans le protocole. Ces spectres de pixels sont ensuite analysés selon deux méthodes, le clustering via les k-moyennes et la détection de biomolécules avec PRM-SRS.
Le regroupement des valeurs K des spectres de pixels entraîne la séparation des régions d’une image 2D par concentration moléculaire et composition en raison de la relation linéaire entre l’intensité du signal SRS et la concentration de liaison moléculaire60,61. Grâce à cette méthodologie, nous appliquons le protocole pour déterminer les régions qui possèdent des phénotypes moléculaires spécifiques tels que prescrits par les spectres de pixels et le centroïde spectral partagé. Pour un examen approfondi de l’expression moléculaire distincte dans des pixels spécifiques, nous implémentons PRM-SRS pour effectuer une correspondance spectrale entre un spectre de référence pour une molécule spécifique capturée par spectroscopie Raman spontanée et les spectres de pixels de l’acquisition HSI SRS. Ce faisant, nous pouvons déterminer la probabilité d’expression de molécules distinctes sans utiliser de sondes ou de marqueurs exogènes. Cette analyse a démontré la capacité de suivre certains sous-types de lipides à travers les tissus et les modèles biologiques, et la capacité d’identifier les changements pathologiques dans les changements de composition lipidique et donc la dyslipidémie47. De plus, les progrès récents dans l’amélioration de la résolution spectrale par la division spectrale de la propagation d’impulsions picosecondes à travers les fibres de silice incitent davantage à l’application de l’ISS SRS comme modalité de visualisation biochimique sans marquage62. Grâce à ces méthodes décrites dans le protocole, nous établissons une plate-forme pour effectuer une analyse ratiométrique et spectrale complète en utilisant une modalité SRS sans étiquette.
En plus de la complexité relative et de la nouveauté de ces techniques, il y a quelques limites importantes dans notre approche à prendre en compte. L’acquisition d’images MPF avec un laser pulsé picoseconde, par opposition aux lasers femtosecondes couramment utilisés, nécessite une puissance d’excitation fluorophore plus élevée et risque le photoblanchiment. Dans cet esprit, nous avons optimisé le protocole ci-dessus pour atténuer les photodommages potentiels de notre plateforme en réduisant la puissance laser incidente sur l’échantillon. De plus, alors que des études antérieures ont démontré la capacité d’évaluer le rapport redox optique à partir de l’imagerie par autofluorescence MPF du NADH et du FAD, la microscopie imageuse à durée de vie de fluorescence (FLIM) a démontré une plus grande précision dans l’évaluation du métabolisme énergétique 63,64,65. Cela est dû à la capacité de différencier les concentrations liées et non liées aux protéines de NADH et de FAD en fonction de leurs durées de vie de fluorescence distinctes, par rapport aux capacités de MPF, cela a permis à FLIM d’être utilisé pour des études complètes de l’activité métabolique à partir de voies énergétiques distinctes, y compris la phosphorylation oxydative et la glycolyse 16,63,64,65,66,67, N° 68. Bien que ce protocole exclue FLIM, nous prévoyons que la modalité sera intégrée à cette plateforme d’imagerie dans un avenir proche, améliorant ainsi l’analyse du métabolisme énergétique que nous effectuons actuellement. Néanmoins, avec le protocole établi ici, nous présentons un flux de travail et une configuration matérielle pour une approche d’imagerie multimodale qui peut mesurer le métabolisme de la structure biologique sous plusieurs perspectives.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Nous remercions la Dre Gloria Pryhuber et les membres de son équipe HuBMAP d’avoir fourni des coupes de tissu pulmonaire humain. Nous remercions le Dr Kun Zhang de nous avoir fourni des tissus cérébraux humains. Nous remercions également le Dr Gen-Sheng Feng d’avoir fourni des échantillons de foie de souris. Nous remercions NIHU54DK134301, NIH R01GM149976, NIH U01AI167892, NIH R01HL170107, NIH 5R01NS111039, NIH R21NS125395, NIH U54CA132378, UCSD Startup funds, Sloan Research Fellow Award et CZI DAF2023-328667 Award.
matériels
| Name | Company | Catalog Number | Comments |
| 460 nm Filter Cube | Olympus | OCT-ET 460/50M32 | |
| Bandpass Filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| DC power supply | TopWard | 6302D | |
| Dichroic Mount | Thorlabs | KM100CL | |
| FIJI ImageJ | ImageJ | ||
| High NA oil condenser | Olympus | 6-U130 | |
| High O.D. shortpass filter | Thorlabs | 950 nm | |
| Inverted Laser Scanning Microscope | Olympus | FV1200MPE | |
| Lock-in Amplifier | Zurich Instruments | ||
| picoEmerald Laser System | Applied Physics & Electronics, Inc. | Synchronized pulsed pump beam (tunable 720–990 nm wavelength, 5–6 ps pulse width, and 80 MHz repetition rate) and Stokes (wavelength at 1032 nm, 6 ps pulse width, and 80 MHz repetition rate) | |
| SI Photodiode Detector | Home Built | ||
| Touch Panel Controller | Olympus | ||
| VF-300 Compresstome | Precisionary |
Références
- Wollman, A. J. M., Nudd, R., Hedlund, E. G., Leake, M. C. From Animaculum to single molecules: 300 years of the light microscope. Open Biol. 5 (4), 150019(2015).
- Vicidomini, G., Bianchini, P., Diaspro, A. STED super-resolved microscopy. Nat Methods. 15 (3), 173-182 (2018).
- Weber, M., et al. MINSTED fluorescence localization and nanoscopy. Nat Photonics. 15 (5), 361-366 (2021).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. , (1990).
- Hell, S. W., et al. Three-photon excitation in fluorescence microscopy. J Biomed Optics. 1 (1), 71-74 (1996).
- Alhallak, K., Rebello, L. G., Muldoon, T. J., Quinn, K. P., Rajaram, N. Optical redox ratio identifies metastatic potential-dependent changes in breast cancer cell metabolism. Biomed Opt Express. 7 (11), 4364-4374 (2016).
- Pena, A. -M., et al. Multiphoton FLIM imaging of NADH and FAD to analyze cellular metabolic activity of reconstructed human skin in response to UVA light. Multiphoton Microscopy in the Biomedical Sciences XIX. 10882, 23-33 (2019).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomed Opt Express. 11 (10), 5674-5688 (2020).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. J Biol Chem. 254 (11), 4764-4771 (1979).
- Ostrander, J. H., et al. Optical redox ratio differentiates breast cancer cell lines based on estrogen receptor status. Cancer Res. 70 (11), (2010).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proc Natl Acad Sci USA. 104 (49), 19494-19499 (2007).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Res. 74 (11), 3067-3075 (2014).
- Malak, M., James, J., Grantham, J., Ericson, M. B. Contribution of autofluorescence from intracellular proteins in multiphoton fluorescence lifetime imaging. Sci Rep. 12, 16584(2022).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Roth, S., Freund, I. Second harmonic generation in collagen. J Chem Phys. 70 (4), 1637-1643 (1979).
- Freund, I., Deutsch, M. Second-harmonic microscopy of biological tissue. Opt Lett. 11 (2), 94(1986).
- Boyd, R. W., Gaeta, A. L., Giese, E. Nonlinear optics. Springer Handbook of Atomic, Molecular, and Optical Physics. Drake, G. W. F. , Springer. 1097-1110 (2023).
- Aghigh, A., et al. Second harmonic generation microscopy: a powerful tool for bio-imaging. Biophys Rev. 15 (1), 43-70 (2023).
- Mohler, W., Millard, A. C., Campagnola, P. J. Second harmonic generation imaging of endogenous structural proteins. Methods. 29 (1), 97-109 (2003).
- Cox, G. Biological applications of second harmonic imaging. Biophys Rev. 3 (3), 131(2011).
- Brown, E., et al. Dynamic imaging of collagen and its modulation in tumors in vivo using second-harmonic generation. Nat Med. 9 (6), 796-800 (2003).
- Keikhosravi, A., Bredfeldt, J. S., Sagar, A. K., Eliceiri, K. W. Chapter 28 - Second-harmonic generation imaging of cancer. Methods Cell Bio. 123, 531-546 (2014).
- Hui Mingalone, C. K., et al. Bioluminescence and second harmonic generation imaging reveal dynamic changes in the inflammatory and collagen landscape in early osteoarthritis. Lab Invest. 98 (5), 656-669 (2018).
- Tsafas, V., et al. Polarization-dependent second-harmonic generation for collagen-based differentiation of breast cancer samples. J Biophotonics. 13 (10), e202000180(2020).
- Nadiarnykh, O., LaComb, R. B., Brewer, M. A., Campagnola, P. J. Alterations of the extracellular matrix in ovarian cancer studied by Second Harmonic Generation imaging microscopy. BMC Cancer. 10, 94(2010).
- Xiong, S. Y., Yang, J. G., Zhuang, J. Nonlinear spectral imaging of human normal skin, basal cell carcinoma and squamous cell carcinoma based on two-photon excited fluorescence and second-harmonic generation. Laser Phys. 21 (10), 1844-1849 (2011).
- Tilbury, K., Campagnola, P. J. Applications of second-harmonic generation imaging microscopy in ovarian and breast cancer. Perspect Medicin Chem. 7, 21-32 (2015).
- Raman, C. V., Krishnan, K. S. A New type of secondary radiation. Nature. 121 (3048), 501-502 (1928).
- Eckhardt, G., et al. Stimulated Raman scattering from organic liquids. Phys Rev Lett. 9 (11), 455-457 (1962).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Li, Y., et al. Microglial lipid droplet accumulation in tauopathy brain is regulated by neuronal AMPK. Cell Metab. 36 (6), 1351-1370.e8 (2024).
- Lu, F. -K., et al. Label-free neurosurgical pathology with stimulated Raman imaging. Cancer Res. 76 (12), 3451-3462 (2016).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nat Commun. 9 (1), 2995(2018).
- Zhang, L., et al. Spectral tracing of deuterium for imaging glucose metabolism. Nat Biomed Eng. 3 (5), 402-413 (2019).
- Li, Y., et al. Bioorthogonal stimulated Raman scattering imaging uncovers lipid metabolic dynamics in Drosophila brain during aging. GEN Biotechnol. 2 (3), 247-261 (2023).
- Li, X., et al. Quantitative imaging of lipid synthesis and lipolysis dynamics in Caenorhabditis elegans by stimulated Raman scattering microscopy. Anal Chem. 91 (3), 2279-2287 (2019).
- Ji, M., et al. Detection of human brain tumor infiltration with quantitative stimulated Raman scattering microscopy. Sci Transl Med. 7 (309), 309ra163(2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Sci Transl Med. 5 (201ra119), 201ra119(2013).
- You, S., et al. Intravital imaging by simultaneous label-free autofluorescence-multiharmonic microscopy. Nat Commun. 9, 2125(2018).
- You, S., et al. Label-free deep profiling of the tumor microenvironment. Cancer Res. 81 (9), 2534-2544 (2021).
- Jermyn, M., et al. Highly accurate detection of cancer in situ with intraoperative, label-free, multimodal optical spectroscopy. Cancer Res. 77 (14), 3942-3950 (2017).
- Pshenay-Severin, E., et al. Multimodal nonlinear endomicroscopic imaging probe using a double-core double-clad fiber and focus-combining micro-optical concept. Light Sci Appl. 10 (1), 207(2021).
- Wu, W., Li, X., Qu, J. Y., He, S. In vivo imaging of biological tissues with combined two-photon fluorescence and stimulated Raman scattering microscopy. J Vis Exp. (178), e63411(2021).
- Li, X., Jiang, M., Lam, J. W. Y., Tang, B. Z., Qu, J. Y. Mitochondrial imaging with combined fluorescence and stimulated Raman scattering microscopy using a probe of the aggregation-induced emission characteristic. J Am Chem Soc. 139 (47), 17022-17030 (2017).
- Li, Z., et al. Multimodal imaging of metabolic activities for distinguishing subtypes of breast cancer. Biomed Opt Express. 14 (11), 5764-5780 (2023).
- Fung, A. A., et al. Label-free optical biopsy reveals biomolecular and morphological features of diabetic kidney tissue in 2D and 3D. , (2024).
- Zhang, W., et al. Multi-molecular hyperspectral PRM-SRS microscopy. Nat Commun. 15 (1), 1599(2024).
- Xu, F. X., et al. Discrimination of lipid composition and cellular localization in human liver tissues by stimulated Raman scattering microscopy. J Biomed Opt. 29 (1), 016008(2024).
- Zoumi, A., Yeh, A., Tromberg, B. J. Imaging cells and extracellular matrix in vivo by using second-harmonic generation and two-photon excited fluorescence. Proc Natl Acad Sci USA. 99 (17), 11014-11019 (2002).
- Liaudanskaya, V., et al. Mitochondria dysregulation contributes to secondary neurodegeneration progression post-contusion injury in human 3D in vitro triculture brain tissue model. Cell Death Dis. 14 (8), 1-15 (2023).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, sub-types and early treatment response in breast cancer. Cancer Res. 73 (20), 6164-6174 (2013).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. Am J Pathol. 178 (3), 1221-1232 (2011).
- Sun, W., et al. Nonlinear optical microscopy: use of second harmonic generation and two-photon microscopy for automated quantitative liver fibrosis studies. J Biomed Optics. 13 (6), 064010(2008).
- Nejim, Z., Navarro, L., Morin, C., Badel, P. Quantitative analysis of second harmonic generated images of collagen fibers: a review. Res Biomed Eng. 39 (1), 273-295 (2023).
- Burke, K., Brown, E. The use of second harmonic generation to image the extracellular matrix during tumor progression. Intravital. 3 (3), e984509(2015).
- Balasubramanian, P., Prabhakaran, M. P., Sireesha, M., Ramakrishna, S. Collagen in human tissues: Structure, function, and biomedical implications from a tissue engineering perspective. Polymer Composites - Polyolefin Fractionation - Polymeric Peptidomimetics - Collagens. Abe, A., Kausch, H. H., Möller, M., Pasch, H. 251, 173-206 (2012).
- Dadsena, S., et al. Lipid unsaturation promotes BAX and BAK pore activity during apoptosis. Nat Commun. 15, 4700(2024).
- Barelli, H., Antonny, B. Lipid unsaturation and organelle dynamics. Curr Opin Cell Biol. 41, 25-32 (2016).
- Harayama, T., Antonny, B. Beyond fluidity: The role of lipid unsaturation in membrane function. Cold Spring Harb Perspect Biol. 15 (7), a041409(2023).
- Yoon, H., Shaw, J. L., Haigis, M. C., Greka, A. Lipid metabolism in sickness and in health: Emerging regulators of lipotoxicity. Mol Cell. 81 (18), 3708-3730 (2021).
- Villazon, J., Shi, L. Utilizing K-means clustering on hyperspectral DO-SRS images of the mouse hippocampus. CLEO 2024, Technical Digest Series. , Optica Publishing Group. (2024).
- Fu, D., et al. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy. J Am Chem Soc. 134 (8), 3623-3626 (2012).
- Periasamy, A., Diaspro, A. Special section guest editorial: Multiphoton microscopy. J Biomed Optics. 8 (3), 327(2003).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci USA. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nat Commun. 5, 3936(2014).
- Xiong, H., et al. Background-free imaging of chemical bonds by a simple and robust frequency-modulated stimulated Raman scattering microscopy. Opt Express. 28 (10), 15663-15677 (2020).
- Georgakoudi, I., Quinn, K. P. Label-free optical metabolic imaging in cells and tissues. Annu Rev Biomed Eng. 25, 413-443 (2023).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon FLIM microscopy. Sci Rep. 12 (1), 11938(2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon