Method Article
Plataforma de imagen óptica multimodal para el estudio del metabolismo celular
* Estos autores han contribuido por igual
En este artículo
Resumen
Utilizando una plataforma multimodal que combina modalidades de imágenes ópticas sin etiquetas, hemos desarrollado un protocolo para visualizar y cuantificar la dinámica y el metabolismo celular. A través de imágenes a través de fluorescencia multifotónica, generación de segundos armónicos y microscopía de dispersión Raman estimulada, podemos generar una visión holística del entorno celular y molecular.
Resumen
Las tecnologías de imagen óptica son críticas en los estudios biomédicos por su capacidad para obtener información morfológica y funcional de especímenes biológicos a alta resolución espacial. Estos procesos ópticos explotan diversas interacciones luz-molécula, como la dispersión, la absorción, la emisión y la generación de armónicos, entre los fotones y las moléculas dentro de las células, tejidos u órganos. Si bien las imágenes biomédicas convencionales se han centrado históricamente en la aplicación de una sola modalidad, investigaciones recientes han demostrado que estas diversas técnicas proporcionan conocimientos complementarios, y sus resultados combinados ofrecen una comprensión más completa de los cambios moleculares en los procesos de envejecimiento y el desarrollo de enfermedades, así como los fundamentos de la biología celular.
En las últimas décadas, los métodos de imágenes ópticas sin etiquetas han avanzado, lo que permite una exploración detallada de los entornos celulares y subcelulares. Por ejemplo, la fluorescencia multifotónica (MPF) no solo facilita la obtención de imágenes de proteínas específicas, sino que también cuantifica la actividad metabólica a través de coenzimas autofluorescentes, logrando una alta profundidad de penetración y resolución espacial. La segunda generación de armónicos (SHG) se utiliza para obtener imágenes de estructuras como el colágeno en la matriz extracelular, mientras que la dispersión Raman estimulada (SRS) mapea los enlaces químicos y la composición molecular in situ con resolución subcelular.
Hemos desarrollado una plataforma de imágenes multimodal que combina las modalidades MPF, SHG y SRS. La integración de estas modalidades en una sola plataforma permite la adquisición de información multifacética de la misma localización dentro de células, tejidos, órganos o incluso cuerpos, lo que facilita una exploración más detallada de las intrincadas relaciones entre el metabolismo celular, la estructura de la matriz extracelular y la composición molecular. Este sistema multimodal ofrece resolución subcelular, penetración profunda en tejidos, imágenes in situ de células vivas/tejidos, así como detección sin etiquetas y corregistro instantáneo sin necesidad de ajustes de posición, cambio de dispositivo o alineación posterior al análisis. Aquí, presentamos un protocolo para imágenes sin marcadores con esta plataforma multimodal y demostramos su aplicación en la caracterización del metabolismo celular y la heterogeneidad molecular en células y tejidos para el estudio del envejecimiento y las enfermedades.
Introducción
Las imágenes biomédicas ópticas han sido fundamentales para avanzar en nuestra comprensión de la estructura y función biológicas. Las imágenes se generan modulando la luz de excitación y detectando señales de las interacciones luz-tejido. El primer microscopio compuesto, desarrollado por Hans y Zacharias Janssen alrededor de 1590, utilizaba dos lentes convexas en un tubo, proporcionando un aumento de hasta 30x1. Los microscopios ópticos modernos, después de siglos de avances, ahora pueden alcanzar resoluciones tan finas como 1-3 nm 2,3. Además de ofrecer alta resolución, los sistemas de imágenes avanzados ahora proporcionan una penetración más profunda en el tejido, una mayor eficiencia y un daño mínimo en la muestra, lo que los hace especialmente adecuados para la obtención de imágenes de células y tejidos vivos. Las imágenes sin etiquetas son particularmente ventajosas, ya que capturan información sin interrumpir los procesos intracelulares ni comprometer la integridad de la muestra.
La microscopía de fluorescencia multifotónica (MPF), en particular la microscopía de fluorescencia de dos fotones, se ha utilizado ampliamente para la obtención de imágenes sin marcadores. A diferencia de la microscopía de fluorescencia convencional, que se basa en la absorción y emisión lineal de un solo fotón, la excitación MPF implica la absorción simultánea de múltiples fotones, cuya energía combinada excita una sola molécula de fluoróforo 4,5. Estos fotones, normalmente en el espectro infrarrojo, poseen la mitad o menos de la energía necesaria para la excitación de un solo fotón. Las longitudes de onda más largas y la excitación localizada en el punto focal de este proceso no lineal dan como resultado una menor dispersión, una penetración más profunda en el tejido y una menor fototoxicidad.
La información metabólica celular se puede capturar mediante microscopía MPF sin marcadores a través de la detección de señales de autofluorescencia de sustratos metabólicos endógenos, como el dinucleótido de nicotinamida adenina (NADH) reducido y el dinucleótido de flavina adenina (FAD). Estas coenzimas exhiben distintos espectros de excitación y emisión, y su relación de intensidad de fluorescencia, conocida como relación redox (NADH/FAD), refleja el estado oxidativo de la célula. Desde que Britton Chance introdujo por primera vez el concepto de la relación redox en 1979, sehan propuesto relaciones adicionales, incluyendo NAD(P)H/FAD, NAD(P)H/(FAD + NAD(P)H) y FAD/(FAD + NAD(P)H), 6,7,8,9. La cuantificación de estas proporciones redox ópticas a través de imágenes MPF proporciona información valiosa sobre la dinámica metabólica. Por ejemplo, las imágenes MPF pueden distinguir las células cancerosas de las células normales en función de su metabolismo alterado, lo que demuestra su potencial para el diagnóstico del cáncer 10,11,12. Sin embargo, la detección de autofluorescencia basada en MPF tiene limitaciones. Otros fluoróforos intrínsecos, como la queratina, pueden contribuir a la intensidad de la fluorescencia, lo que conduce a una diafonía espectral y a una interpretación inexacta de la señal13. Además, la relación redox solo refleja los cambios generales de oxidación-reducción celular y no distingue entre NADH de diferentes fuentes (por ejemplo, citoplasmáticas o mitocondriales) o entre NADH y NAD(P)H, ya que ambos exhiben picos espectrales similares a 450 nm, lo que da como resultado señales de intensidad mezcladas14.
La Segunda Generación de Armónicos (SHG), un proceso óptico no lineal demostrado por primera vez en el campo biomédico en la década de 1980, ha sido ampliamente utilizado para la obtención de imágenes sin etiquetas de estructuras celulares15,16. Al igual que el MPF, el SHG implica la absorción simultánea de dos fotones de la misma energía de un láser pulsado ultrarrápido. Estos fotones se recombinan para emitir un nuevo fotón dos veces la frecuencia de la luz incidente, lo que resulta en la detección de la señal del segundo armónico. Esta interacción óptica no lineal ocurre exclusivamente en materiales no centrosimétricos que exhiben una susceptibilidad de segundo orden distinta de cero para inducir una polarización para generar la señal del segundo armónico17,18. Esto hace que SHG sea particularmente eficaz para obtener imágenes de las proteínas filamentosas y las estructuras fibrilares, como el colágeno, la miosina y la tubulina, sin necesidad de colorantes de fluorescencia exógenos 15,17,19,20. La anormalidad en la abundancia, rigidez, alineación y estructura de la fibrosis y el colágeno son prevalentes en muchas afecciones como la inflamación y el cáncer, lo que convierte a la SHG en una herramienta prometedora para la detección eficiente y no invasiva de ciertas condiciones patológicas 21,22,23. La aplicación generalizada de las imágenes SHG en la investigación oncológica, incluidos los estudios sobre los cánceres de mama, ovario y piel, ha puesto de manifiesto su papel crucial tanto en la investigación fundamental como en las posibles aplicaciones clínicas 24,25,26,27.
Diferentes moléculas exhiben distintos niveles de energía vibratoria, que inducen diversos grados de dispersión inelástica cuando son excitadas por la luz incidente, un fenómeno caracterizado por primera vez por C. V. Raman en 192828. Desde entonces, el efecto Raman se ha utilizado ampliamente en microscopía óptica para la detección de composiciones moleculares y de tejidos sin marcaje exógeno. Tanto la dispersión Raman estimulada (SRS) como la dispersión Raman coherente anti-Stokes (CARS) excitan las vibraciones moleculares de manera coherente y aprovechan la interacción no lineal de la luz para producir una señal más fuerte en comparación con la espectroscopia Raman espontánea convencional. El fenómeno SRS se reportó por primera vez en 196229. En 2008, este mecanismo se integró en la obtención de imágenes multifotónicas tridimensionales, lo que permitió la detección selectiva de productos químicos a partir de los cambios de intensidad en los haces de Pump y Stokes debidos a las transiciones vibratorias moleculares30. Este método minimiza la interferencia de fondo no resonante, generando una señal de intensidad limpia que supera a la de CARS. Las imágenes SRS se destacan por proporcionar imágenes multiplexadas e hiperespectrales, lo que permite la detección simultánea de múltiples enlaces químicos y permite la visualización de alta resolución de la composición molecular en muestras con una profundidad de penetración considerable. Aunque es una técnica relativamente nueva, la SRS ha demostrado ser eficaz tanto en el diagnóstico clínico como en la investigación metabólica, in vivo e in vitro30,31,32,33,34,35,36. Por ejemplo, la SRS puede diferenciar los tejidos infiltrados por tumores cerebrales de la corteza y la sustancia blanca mediante la cuantificación de la relación lípido-proteína, lo que permite la delineación de los márgenes tumorales de forma no invasiva y sin etiquetas37,38. Además, las alteraciones metabólicas, a menudo consideradas características distintivas de las enfermedades relacionadas con el envejecimiento y asociadas al cáncer, pueden evaluarse cuantitativamente utilizando SRS logrado mediante la detección de enlaces carbono-deuterio en muestras tratadas con agua pesada (D2O), lo que permite la medición cuantitativa de la síntesis de proteínas, la lipogénesis y otros procesos metabólicos macromoleculares 31,33,34,35,36. La capacidad de rastrear metabolitos con alta resolución temporal y espacial posiciona a SRS como una herramienta prometedora para la investigación y el diagnóstico de enfermedades, con potencial para aplicaciones clínicas más amplias.
Las imágenes multimodales se han convertido en un enfoque poderoso en la investigación biomédica, ya que integran dos o más modalidades de imágenes para obtener una comprensión más completa de los sistemas biológicos complejos dentro de la misma muestra. En 2018, se introdujo una técnica de microscopía de microscopía multiarmónica de autofluorescencia sin marcadores (SLAM), que integra fluorescencia de dos fotones (2PF), fluorescencia de tres fotones (3PF), SHG y tercera generación de armónicos (THG)39. Este enfoque facilita la visualización simultánea de las interacciones celulares, los procesos dinámicos y los componentes individuales dentro del microambiente tumoral. La microscopía SLAM ofrece una perturbación mínima y requisitos de potencia láser reducidos para la muestra, lo que permite la creación de perfiles de tejidos profundos y proporciona un método más seguro para el monitoreo intravital40. Otra modalidad multimodal, que combina espectroscopia de fluorescencia intrínseca, espectroscopia de reflectancia difusa y espectroscopia Raman, ha sido desarrollada para la detección de cáncer in situ durante procedimientos quirúrgicos41. Además, un sistema de endoscopia no lineal multimodal recientemente diseñado, que integra CARS, SHG y fluorescencia de dos fotones (TPF), ha demostrado la capacidad de obtener imágenes de muestras biológicas con una resolución espacial submicrónica y subcelular42. La microscopía combinada de 2PF y SRS se ha utilizado de manera similar para obtener imágenes in vivo de alta resolución de tejidos, células y orgánulos 42,43,44,45. Estas técnicas emergentes de adquisición de imágenes multimodales aprovechan las fortalezas de las modalidades individuales, lo que mejora la resolución, la profundidad de penetración y la eficiencia de adquisición de imágenes, lo que muestra un potencial considerable para aplicaciones clínicas y quirúrgicas.
Este enfoque multimodal se ve cada vez más favorecido sobre las imágenes de monomodalidad porque proporciona una gama más amplia de mediciones al tiempo que mitiga las limitaciones asociadas con las técnicas individuales. Como se ha comentado anteriormente, el MPF mide la fluorescencia endógena para reflejar los cambios metabólicos, el SHG puede obtener imágenes de estructuras no centrosimétricas, como el colágeno, en muestras biológicas, y el SRS detecta predominantemente proteínas y lípidos debido a la alta densidad de enlaces químicos que generan señales Raman distintivas basadas en sus modos vibratorios. Dadas sus propiedades coherentes y el principio compartido de propiedades ópticas no lineales, estas modalidades de imagen pueden integrarse en una sola configuración de microscopio utilizando láseres pulsados ultracortos, lo que permite la adquisición de varios biomarcadores en regiones localizadas para proporcionar una visión más completa de los procesos biológicos44,45. Este artículo describe un protocolo para implementar una plataforma de imágenes multimodal que integra MPF, SHG y SRS para aplicaciones de investigación biomédica.
Protocolo
1. Experimentos de imagen multimodal sin etiquetas
NOTA: Este protocolo se centra en el procedimiento de configuración y adquisición de imágenes multimodales sin etiquetas.
- Configuración del microscopio multimodal (Figura 1A) y calibración del sistema
- Caliente el láser y espere aproximadamente 15-20 min.
- Encienda las unidades de control y los monitores en la siguiente secuencia: Caja de control | Controlador de panel táctil | Adaptador de CA para control remoto láser principal Adaptador de CA para control remoto subláser.
- Encienda el detector de fotodiodos de Si y el amplificador de bloqueo.
- Configure los rayos láser de la bomba y el haz de Stokes. Configure el sistema láser con un haz de bomba ajustable de 780 nm a 990 nm, un ancho de pulso de 5 a 6 ps y una tasa de repetición de 80 MHz. El rayo láser de Stokes tiene una longitud de onda fija de 1.031 nm con un pulso de 6 ps y una tasa de repetición de 80 MHz . Asegúrese de que tanto la bomba como las vigas Stokes estén a baja potencia (al menos 20 mW) para que sean visibles en la placa de alineación.
- Coloque una placa de alineación en la trayectoria óptica (justo después de la caja láser) para verificar la superposición espacial de las bombas y los haces de Stokes y ajuste el espejo hasta que ambos puntos se superpongan en el centro de la placa de alineación.
NOTA: Aunque las bombas y los haces de Stokes están aproximadamente superpuestos, el ajuste fino de la superposición espacial de dos rayos láser puede lograr la señal SRS óptima. - Después de la superposición espacial, ajuste con el PSD (detector sensible a la posición) haciendo clic primero en el software de control OPO . Utilice la tecla hexadecimal para ajustar cuidadosamente el espejo óptico 1 (OM1) y observe la pantalla PSD para ver el cambio de posición X-Y para apuntar a la desviación mínima del centro. A continuación, para realizar ajustes más precisos, utilice la tecla hexadecimal del espejo óptico 2 (OM2) hasta que el indicador de posición se centre en la pantalla PSD.
NOTA: OM1 y OM2 son dos espejos en el cabezal OPO para ajustar la alineación del láser. - Finalmente, centre el condensador girando los dos tornillos de centrado del condensador para mover la imagen del diafragma del iris al centro del campo de visión.
NOTA: El diafragma de iris de campo puede restringir el diámetro del haz de luz que ingresa al objetivo y, por lo tanto, excluir la luz extraña, mejorando el contraste de la imagen.
- Pasos de adquisición de imágenes multimodales sin etiquetas
- Aplique aceite al condensador de aceite de apertura numérica alta (1,4 NA) y monte el portaobjetos del microscopio en el condensador aceitado. Finalmente, coloque una gota de agua grande en el portaobjetos del microscopio para el objetivo de agua 25x. Asegúrese de que el microscopio esté montado de forma segura e inamovible y, a continuación, ajuste la etapa z para ajustar el enfoque hasta que la imagen de campo claro de la muestra biológica se pueda ver bajo el objetivo de agua de 25x.
- Comience el proceso de obtención de imágenes en la secuencia correcta: MPF, SHG y SRS para evitar el fotoblanqueo, que afectará a la calidad de la imagen y al análisis preciso del MPF. Para cambiar rápidamente entre MPF y SHG, cambie de la viga de la bomba a la viga fija de Stokes.
NOTA: El ajuste del láser de la bomba de 800 nm (autofluorescencia NADH y FAD) a 791,3 nm (cambio Raman CH3 ) puede llevar de 1 a 2 minutos. - Seleccione la resolución de la imagen (512 x 512 píxeles) y el tiempo de permanencia adecuado para cada modalidad. Utilice 8 μs/píxel para el MPF y el SHG con un fotograma medio superior a 3. Utilice 40 μs/píxel con un fotograma medio de 2 para la modalidad SRS.
NOTA: El fotograma promedio significa el número de imágenes que se deben promediar antes de guardar. - Para adquirir autofluorescencia con MPF, apague el rayo láser de Stokes y ajuste el láser de la bomba a 800 nm para excitar NADH y flavina. Instale el cubo de filtro FVOPT en la ruta de luz con 460 ± 10 nm y 515 ± 10 nm para el NADH y la flavina, respectivamente. Por lo tanto, la potencia de la muestra es de aproximadamente 15 mW.
- Adquiere la señal de la fibra de colágeno usando SHG. Apague el rayo láser de la bomba, use solo avivar el rayo láser y configure la potencia para que sea de 500 mW. Solo adquiera el canal relacionado con el filtro de 515 nm.
- Obtener la distribución espacial de proteínas y lípidos mediante SRS. Mantenga ambos rayos láser encendidos y ajuste la frecuencia del rayo láser para que coincida con el modo vibratorio específico para cada molécula.
NOTA: Por lo general, 791,3 nm se usa para CH3, 797 nm se usa para CH2, 787 nm es para lípidos insaturados y 794,6 es para lípidos saturados. Por lo tanto, la potencia de la muestra es de aproximadamente 40 mW. - Para adquirir los conjuntos de datos de imágenes hiperespectrales SRS, abra el software de control OPO para seleccionar barrido, establezca el rango de longitud de onda de 780 nm a 806,5 nm y elija un número de pila de al menos 60. A continuación, capture la pila de imágenes hiperespectrales.
NOTA: El conjunto de datos resultante contiene 60 imágenes, cada una de las cuales representa la distribución espacial en un desplazamiento Raman específico (de 2.700 cm-1 a 3.150 cm-1). El tiempo de adquisición hiperespectral depende del campo de visión (FOV) de la imagen y del tiempo de exploración, pero será aproximadamente entre 5 min y 8 min para una imagen de 512 x 512. Para imágenes más grandes, se debe tener cuidado de minimizar la duración bajo el láser para evitar cualquier daño térmico, que puede ocurrir después de 2 h (dependiendo del grosor de la muestra y el tamaño del campo de visión). - Guarde todas las imágenes de las mismas regiones de interés (ROI) en la misma carpeta. El formato de la imagen es un archivo .oir de Olympus.
2. Análisis de imágenes
- Utilice el software de procesamiento de imágenes para abrir todas las imágenes RAW guardadas para asignar el color y agregar la barra de escala para la visualización de la imagen. Utilícelo también para generar la máscara binaria de imágenes de NADH y ácidos grasos de saturación (SFA, 2.880 cm-1) para el análisis óptico de relación redox y unsaturación de lípidos.
NOTA: Asegúrese de que la intensidad de la mascarilla solo tenga 0 y 1. - Utilice el script de python casero para realizar cualquier análisis posterior (el código está disponible en https://github.com/lingyanshi2020/HSI_Analysis).
NOTA: Este script está diseñado para procesar múltiples regiones de interés (ROI) de muestras de control y de cáncer. La función principal del script requiere solo dos entradas: las rutas de archivo para las carpetas de imágenes de control y cáncer.- Itere a través de las carpetas, haciendo coincidir las imágenes con el mismo nombre base (por ejemplo, "roi_1_flavin.tif", "roi_1_nadh.tif", "roi1_mask.tif").
- Para cada conjunto de imágenes coincidentes, deje que el script realice un análisis ratiométrico y genere nuevas imágenes ratiométricas basadas en estos cálculos.
La relación redox óptica se calcula como flavina / (NADH + flavina).
La insaturación de lípidos se calcula como: USFAs / (SFAs + USFAs) donde los USFAs representan ácidos grasos insaturados. - Todos los valores radiométricos de las imágenes se almacenan en un DataFrame de pandas para facilitar su manipulación. Realice ANOVA de un factor y use matplotlib con styple "ggplot" o seaborn para hacer el diagrama de barras o diagrama de caja para la cuantificación.
- Utilice las imágenes de 2.930 cm-1 (CH3) y 2.850 cm-1 (CH2) para generar una imagen sustraída (CH3 - CH2). A continuación, mezcle la imagen CH2 CH3 - CH2 en el espacio de color RGB con una LUT personalizada que imite la tinción H&E para generar la imagen de histología digital46.
- Después de la adquisición de imágenes hiperespectrales SRS, emplee el script de python casero para realizar la agrupación de k-medias para segmentar las imágenes en función de las similitudes espectrales (el código está disponible en https://github.com/lingyanshi2020/HSI_Analysis).
- Cargue las pilas de imágenes .tiff con el paquete tifffile. Cambie la forma de la pila de imágenes hiperespectrales SRS (x, y, espectros) en una matriz 2D (píxeles, espectros).
- Utilice el paquete scikit-learn para implementar el algoritmo k-means en esta matriz 2D, con el número definido de clústeres.
- Vuelva a dar forma a las etiquetas de clúster resultantes a las dimensiones de la imagen original, creando un mapa de segmentación.
- Asigne colores únicos a cada grupo, generando una imagen en falso color que representa diferentes composiciones bioquímicas dentro del tejido.
- Utilice matplotlib para trazar los espectros de cada clúster con el valor medio (línea continua) y la desviación estándar (área de sombra)
- Siga el protocolo del protocolo SRS de coincidencia de referencias penalizadas (PRM-SRS) con MATLAB para crear la detección de subtipos de lípidos sin etiquetas espaciales con conjuntos de datos de imágenes hiperespectrales SRS47.
- Organice las imágenes y guárdelas en un formato de .tif de 300 ppp.
Resultados
Las imágenes de la Figura 2 son representativas de los resultados obtenidos siguiendo el protocolo de adquisición de autofluorescencia de FAD y NADH, así como los cuatro canales SRS para proteínas, lípidos totales, lípidos insaturados y lípidos saturados. Aquí también generamos una imagen pseudohistológica como se describe en el protocolo a través de la mezcla de colores RGB. La adquisición de los canales MPF y SRS proporciona los archivos de imagen que se utilizarán posteriormente en el análisis radiométrico. Un ejemplo de este análisis se puede ver en la Figura 3 sobre tejido pulmonar humano. Después de la adquisición de imágenes en el protocolo, nuestra metodología de análisis de imágenes que utiliza Python o ImageJ aprovecha la proporción de distintos canales para proporcionar información metabólica cuantitativa. Como se muestra en las imágenes de insaturación de lípidos y relación redox óptica de la Figura 3, el análisis radiométrico proporciona un mapa de color de la distribución de la actividad metabólica relativa y la composición molecular. Hemos aprovechado estas mediciones para hacer observaciones sobre los cambios en las rutas metabólicas y el contenido de lípidos de tejidos específicos, patologías o factores biológicos distintos. Como se observa en la Figura 4, esto puede proporcionar una comparación cuantitativa del tejido sano y tumoral al comparar los valores promedio de estrés oxidativo y unsaturación de lípidos, además de su distribución en una imagen de microscopía 2D.
Para el análisis radiométrico de imágenes MPF y SRS, comúnmente nos enfocamos en la relación óptica redox y de insaturación, como se muestra en las ecuaciones (1) y (2).
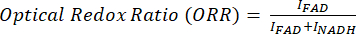 (1)
(1)
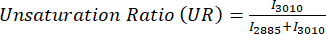 (2)
(2)
Donde IC es la intensidad de los píxeles del canal C. Así, medimos la relación por píxel y adquirimos la distribución espacial de estos marcadores metabólicos y moleculares.
Además del análisis radiométrico, la Figura 4 también muestra otra posible vía de aplicación para nuestra plataforma multimodal: el análisis de imágenes hiperespectrales. Como se describe en este protocolo, podemos adquirir una imagen hiperespectral SRS realizando un barrido a través de múltiples longitudes de onda láser utilizando un haz de bomba sintonizable. Esto nos permite reconstruir la región CH del espectro de desplazamiento Raman para cada píxel dentro de la imagen de microscopía, uniendo tanto la información química proporcionada por la espectroscopía Raman como la información espacial proporcionada por la microscopía óptica. En el flujo de trabajo para el análisis de imágenes, destacamos dos técnicas implementadas para el análisis de estas imágenes hiperespectrales: PRM-SRS para la detección de biomoléculas y k-means clustering. La figura 4E muestra la aplicación de PRM-SRS en una imagen de un tumor hepático de ratón. El algoritmo PRM-SRS proporciona la correlación entre los espectros para cada píxel individual, ajustado por una penalización para los cambios en el espectro Raman; A continuación, el algoritmo genera imágenes de distribución de probabilidad para cada uno de los lípidos.
El agrupamiento de K-means de los espectros de píxeles es otra técnica que implementamos para visualizar el agrupamiento de fenotipos espectrales específicos, lo que permite la separación por concentración de enlaces y composición molecular a través de la intensidad y la forma espectral respectivamente. La Figura 5 proporciona un ejemplo de cómo se puede aplicar el análisis de imágenes a través de la agrupación en clústeres k-means en nuestro protocolo. Esperamos que los grupos de k-medias se alineen estructuralmente con las características de las figuras de pseudohistología, además de proporcionar un mayor agrupamiento de las características que no se distinguen tan fácilmente de los canales SRS o de la tinción histológica (H&E) de pseudohematoxilina y eosina. El análisis de agrupamiento PRM-SRS y k-means complementa esta plataforma de imágenes sin etiquetas al proporcionar información química y semi-apuntable sobre analitos específicos y enlaces moleculares sin la necesidad de sondeo exógeno.
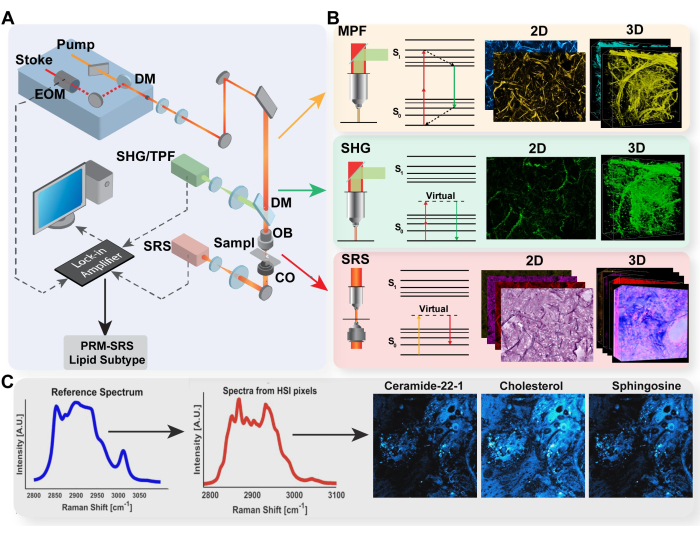
Figura 1: Diagrama de la plataforma de imagen multimodal y la detección de biomoléculas PRM-SRS. (A) Diagrama de la configuración del hardware para el sistema de imágenes MPF/TPF, SHG y SRS. (B) Trayectoria de luz y diagrama de Jablonski para TPF, SHG y SRS con imágenes adquiridas representativas para cada modalidad. (C) Diagrama de flujo de trabajo PRM-SRS para la coincidencia de referencias espectrales entre la espectroscopia Raman espontánea y las imágenes hiperespectrales SRS. Abreviaturas: MPF = fluorescencia multifotónica; TPF = fluorescencia de dos fotones; SHG = segunda generación de armónicos; SRS = dispersión Raman estimulada; PRM = coincidencia de referencias penalizada. Haga clic aquí para ver una versión más grande de esta figura.
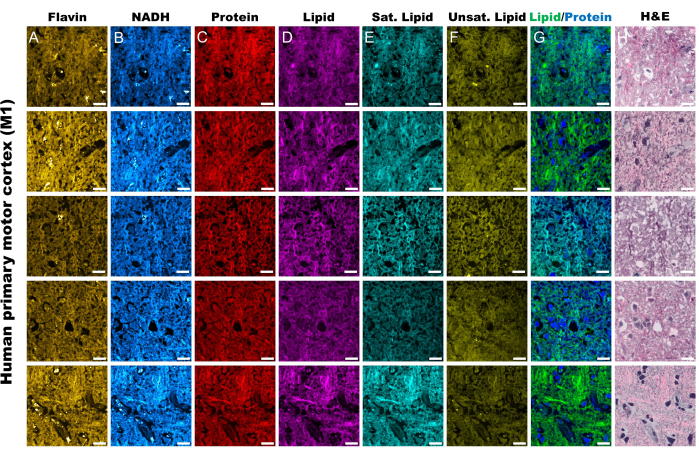
Figura 2: Canales MPF y SRS del tejido de la corteza motora primaria humana (M1). (A,B) Canales de autofluorescencia MPF para Flavin/FAD y NADH. (C-F) Canales SRS capturados en picos de desplazamiento Raman específicos; el pico de estiramiento asimétrico CH3 (2.930 cm-1) para las proteínas, el pico de estiramiento asimétrico CH2 (2.845 cm-1) para los lípidos, el pico de 2.885 cm-1 para los ácidos grasos saturados y el pico de 3.010 cm-1 para los ácidos grasos insaturados. (G) Fusionó los canales de proteínas SRS (azul) y lípidos (verde) para delinear su respectiva distribución espacial. (H) Imágenes pseudohistológicas (SRS) de SRS que reflejan la tinción de H&E. Barras de escala = 20 μm. Abreviaturas: MPF = fluorescencia multifotónica; FAD = flavina adenina dinucleótido; NADH = nicotinamida adenina dinucleótido reducido; SRS = dispersión Raman estimulada; SRH = histología SRS. Haga clic aquí para ver una versión más grande de esta figura.
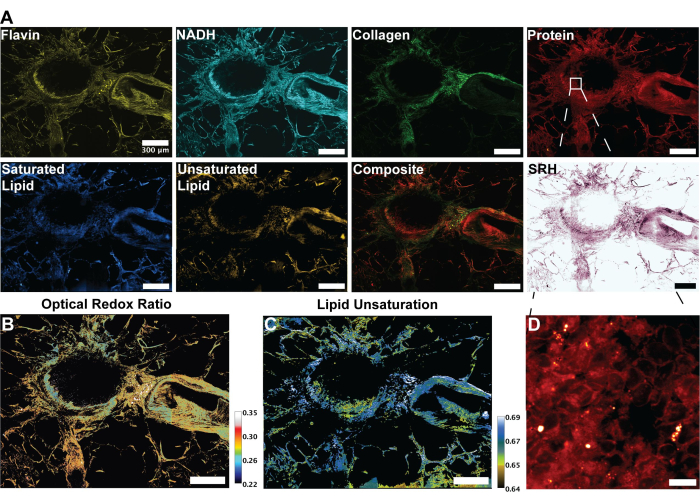
Figura 3: Imágenes y análisis de MPF, SHG y SRS de tejido pulmonar humano. (A) Picos de autofluorescencia MPF para Flavin/FAD y NADH, señal SHG para fibras de colágeno, señal SRS para proteínas (2.930 cm-1), lípidos (2.845 cm-1), lípidos saturados (2.885 cm-1) y lípidos insaturados (3.010 cm-1), y una imagen compuesta fusionada de las múltiples modalidades. (B) Imagen radiométrica de la relación redox óptica. (C) Imagen radiométrica de la insaturación de lípidos. (D) Imagen ampliada de las células epiteliales en la capa interna de los bronquios. Barras de escala = 200 μm. Abreviaturas: MPF = fluorescencia multifotónica; SHG = segunda generación de armónicos; SRS = dispersión Raman estimulada; FAD = flavina adenina dinucleótido; NADH = nicotinamida adenina dinucleótido reducido. Haga clic aquí para ver una versión más grande de esta figura.
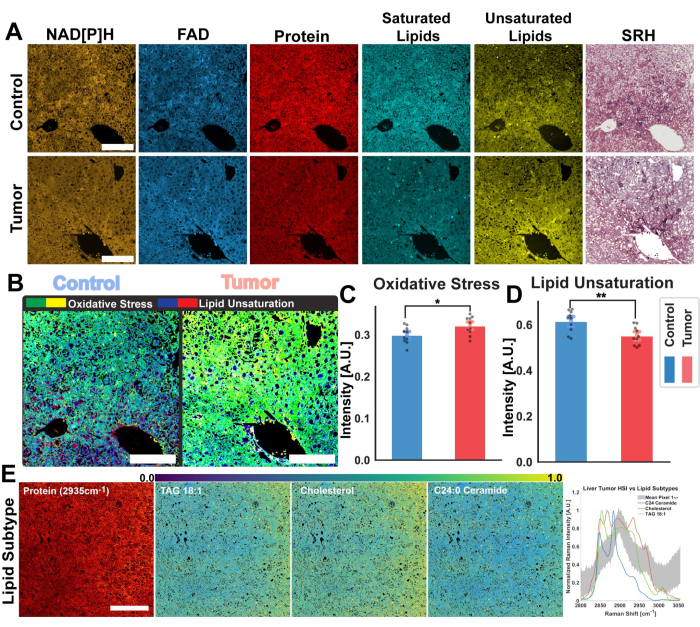
Figura 4: Análisis ratiométrico e hiperespectral de tumores hepáticos de ratón. (A) Imágenes de autofluorescencia MPF para NAD[P]H y FAD, canales SRS para proteínas totales, lípidos saturados e insaturados, e imágenes pseudohistológicas para hígado de ratón de control sano (arriba) y tejido tumoral (abajo). (B) Estrés oxidativo (amarillo-verde), medido por la relación óptica redox, y la insaturación de lípidos (azul-rojo) calculada a partir de los tejidos control (izquierda) y tumoral (derecha). Barras de escala = 100 μm. (C,D) Gráficos de barras que demuestran las diferencias radiométricas en el estrés oxidativo y la insaturación de lípidos entre muestras de hígado de control (azul) y tumorales (rojo). Estadísticos de significación: * para una significación de p ≤ 0,05, ** para una significancia de p ≤ 0,01. (E) El análisis del subtipo de lípidos PRM-SRS resulta de imágenes hiperespectrales. De izquierda a derecha: canal de proteína SRS para referencia estructural, barra de escala = 100 μm, imagen de distribución de probabilidad de subtipos de lípidos (TAG 18:1, colesterol y ceramida C24:0). Gráfico de espectros de referencia de subtipos de lípidos y espectros de píxeles medios de SRS HSI en toda la región CH. Abreviaturas: MPF = fluorescencia multifotónica; SRS = dispersión Raman estimulada; NAD[P]H = nicotinamida adenina dinucleótido fosfato reducido; FAD = flavina adenina dinucleótido; HSI = imágenes hiperespectrales. Haga clic aquí para ver una versión más grande de esta figura.
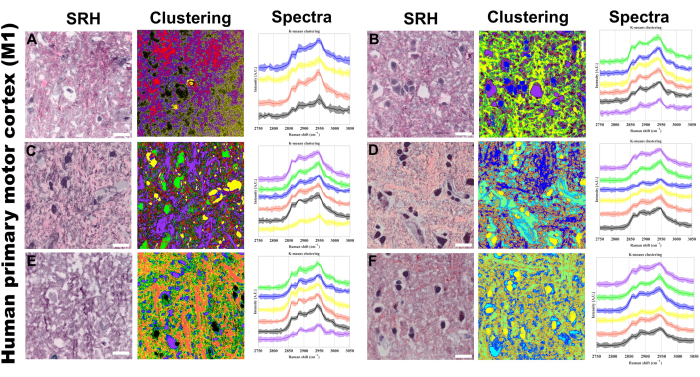
Figura 5: Agrupamiento hiperespectral de k-medias SRS en la corteza motora primaria humana (M1). (A-F) Seis regiones representativas que muestran: izquierda: imágenes de histología Raman estimulada (SRH) que proporcionan visualización de tejidos similares a H&E; medio: resultados correspondientes de agrupamiento de k-medias; Derecha: perfiles espectrales Raman específicos de clúster con intensidades medias (líneas continuas) y desviaciones estándar (áreas sombreadas). El número de centroides que oscila entre 4 y 6 medias. Se utilizó el agrupamiento no supervisado, por lo que los diferentes colores de las imágenes agrupadas representan composiciones químicas distintas identificadas por el algoritmo k-means. Cada clúster (representado por un color único) corresponde a regiones con perfiles espectrales similares en la región de estiramiento CH. Barra de escala = 20 μm. Abreviaturas: SRH = histología SRS. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este sistema multimodal es una potente plataforma de imágenes para capturar una visualización holística del entorno molecular de las muestras en una amplia gama de orígenes biológicos y condiciones patológicas. La ventaja de aprovechar las diferentes modalidades sin marcadores radica en la capacidad de adquirir información complementaria y dirigirse a analitos específicos que, de otro modo, podrían ser difíciles o imposibles en una sola técnica de imagen sin marcadores. Específicamente, las tres técnicas de imagen no lineal (SRS, MPF, SHG) mencionadas en este trabajo permiten la cuantificación de la composición de macromoléculas, la dinámica de energía a través de la relación redox óptica y la información estructural, incluida la composición y morfología de la matriz extracelular 6,48,49. Además, en comparación con la utilización de cada modalidad de adquisición de imágenes por separado, la adquisición de imágenes con un sistema de microscopio combinado permite el registro inmediato de imágenes y tiempos de almacenamiento de muestras más cortos. Incluso hemos realizado imágenes de células vivas con este enfoque, posiblemente debido a la menor fotoexposición en comparación con la microscopía de fluorescencia confocal, utilizando puntos de tiempo seleccionados para adquirir cada modalidad secuencialmente según lo estipulado en el protocolo anterior. Además de sus ventajas individuales, la plataforma multimodal también se beneficia de las múltiples perspectivas sobre la condición biológica a través de la medición sin etiquetas de diferentes biomarcadores.
La obtención de imágenes sin marcadores de proteínas autofluorescentes a través de MPF se ha dirigido principalmente a la cuantificación de la relación redox óptica, una medida de las reacciones redox facilitadas por la oxidación de FAD en FAD+ y la reducción de NAD+ en NADH50. Este es un marcador importante del metabolismo dentro de las células y los tejidos, ya que proporciona una medida de la actividad relativa entre la fosforilación oxidativa y la glucólisis, las dos vías principales para la generación de ATP6. En concreto, las disminuciones en la concentración de NADH y los aumentos en FAD+ son un marcador de aumento de la fosforilación oxidativa en las mitocondrias; Mientras tanto, lo contrario es cierto para el aumento de la glucólisis en el citoplasma. La tendencia a preferir una vía metabólica sobre la otra en la producción de energía se ha relacionado con varios cambios patológicos y activaciones de vías relacionadas con el cáncer, destacando el uso potencial de la TRO como marcador de detección precoz 6,51.
De manera similar, la adquisición de la señal de colágeno fibroso SHG aprovecha una modalidad de imagen no lineal sin etiquetas para cuantificar y visualizar un marcador biológico para la salud. SHG puede rastrear eficazmente la distribución del colágeno tipo I-III debido a su estructura no centrosimétrica. Las proteínas fibrosas de colágeno medidas por SHG han sido reconocidas como marcadores diagnósticos importantes para varias enfermedades, incluyendo el cáncer y la fibrosis 25,26,27,52,53. Además de su detección como marcador de enfermedad, nuestra adquisición de colágeno a través de SHG puede servir como un fuerte indicador de la estructura en células y tejidos debido al papel del colágeno fibroso en la composición de la matriz extracelular y los límites estructurales a través de los tejidos54,55. A través de la señal de colágeno SHG, podemos hacer observaciones informadas de células separadas o unidades funcionales de tejidos distintas sin la necesidad de un marcador exógeno para las membranas celulares. En general, la cuantificación SHG de fibrillas de colágeno presenta un claro beneficio tanto para la capacidad diagnóstica como para la facilidad de análisis cuando se integra con otras técnicas de imagen sin marcaje.
Utilizando la microscopía SRS, podemos capturar varios marcadores para la composición de macromoléculas, particularmente para lípidos y ácidos grasos. En este protocolo, describimos cómo adquirir la señal SRS para ácidos grasos insaturados y saturados para el análisis radiométrico de la insaturación de lípidos dentro de la misma muestra. Se aplicó un microscopio de escaneo láser vertical con un objetivo de agua de 25x para el rendimiento del infrarrojo cercano. Los rayos láser pasaron a través de la muestra y fueron recogidos por un condensador de aceite de alta apertura numérica (1,4 NA). Se utilizó un filtro de paso corto de alto diámetro exterior (950 nm) para bloquear el haz de Stokes, mientras que solo permitía que el haz de la bomba llegara a un detector de fotodiodos de Si para detectar la señal de pérdida Raman estimulada. La corriente de salida del fotodiodo se terminó y filtró. Posteriormente, un amplificador de bloqueo a 20 MHz demoduló la corriente en X con un desplazamiento de fase cero, y un módulo de software utilizó la señal demodulada para generar la imagen durante el escaneo láser.
Los estudios han demostrado que los cambios en la insaturación de lípidos tienen un efecto profundo en la membrana celular y de los orgánulos, y una desregulación en el metabolismo de los lípidos puede ser un fuerte marcador de varias enfermedades, incluyendo el cáncer y las enfermedades neurodegenerativas 56,57,58,59. Para obtener una visión más completa de la desregulación lipídica, analizamos la composición de los subtipos mediante imágenes hiperespectrales SRS. Las imágenes hiperespectrales SRS (HSI) combinan la información espacial de alta resolución adquirida por la microscopía SRS con la señal químicamente significativa de la espectroscopia Raman a través del ajuste secuencial como se describe en el protocolo. A continuación, estos espectros de píxeles se analizan en dos métodos, la agrupación mediante k-means y la detección de biomoléculas con PRM-SRS.
La agrupación de K-means de los espectros de píxeles da como resultado la separación de regiones dentro de una imagen 2D por concentración y composición molecular debido a la relación lineal entre la intensidad de la señal SRS y la concentración de enlace molecular60,61. A través de esta metodología, aplicamos el protocolo para determinar regiones que poseen fenotipos moleculares específicos según lo prescrito por los espectros de píxeles y el centroide espectral compartido. Para un examen exhaustivo de la expresión molecular distintiva en píxeles específicos, implementamos PRM-SRS para realizar la coincidencia espectral entre un espectro de referencia para una molécula específica capturada con espectroscopia Raman espontánea y los espectros de píxeles de la adquisición de SRS HSI. Al hacerlo, podemos determinar la probabilidad de expresión de moléculas distintas sin utilizar sondas o etiquetas exógenas. Este análisis ha demostrado la capacidad de rastrear ciertos subtipos de lípidos a través de tejidos y modelos biológicos, y la capacidad de identificar cambios patológicos en la composición lipídica y, por lo tanto, dislipidemia47. Además, los avances recientes en la mejora de la resolución espectral a través de la división espectral de la propagación de pulsos de picosegundos a través de fibras de sílice incentivan aún más la aplicación de SRS HSI como modalidad para la visualización bioquímica sin etiquetas62. A través de estos métodos destacados en el protocolo, establecemos una plataforma para realizar análisis radiométricos y espectrales exhaustivos utilizando una modalidad SRS sin etiquetas.
Además de la relativa complejidad y novedad de estas técnicas, existen algunas limitaciones importantes en nuestro enfoque a tener en cuenta. La adquisición de imágenes MPF con un láser pulsado de picosegundos, a diferencia de los láseres de femtosegundos comúnmente utilizados, requiere una mayor potencia de excitación de fluoróforos y corre el riesgo de fotoblanqueo. Con eso en mente, hemos optimizado el protocolo anterior para mitigar el posible fotodaño de nuestra plataforma al reducir la potencia del láser incidente en la muestra. Además, mientras que estudios previos han demostrado la capacidad de evaluar la relación óptica redox a partir de imágenes de autofluorescencia MPF de NADH y FAD, la microscopía de imágenes de fluorescencia (FLIM) ha demostrado una mayor precisión en la evaluación del metabolismo energético 63,64,65. Esto se debe a la capacidad de diferenciar las concentraciones de NADH y FAD unidas y no unidas a proteínas en función de sus distintos tiempos de vida de fluorescencia, en comparación con las capacidades de MPF, esto ha permitido que FLIM se utilice para estudios exhaustivos de la actividad metabólica de distintas vías de energía, incluida la fosforilación oxidativa y la glucólisis 16,63,64,65,66,67, Artículo 68. Si bien este protocolo excluye FLIM, esperamos que la modalidad se integre en esta plataforma de imágenes en un futuro próximo, mejorando el análisis del metabolismo energético que realizamos actualmente. Sin embargo, con el protocolo establecido aquí, presentamos un flujo de trabajo y una configuración de hardware para un enfoque de imágenes multimodal que puede medir el metabolismo de la estructura biológica desde múltiples perspectivas.
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
Agradecemos a la Dra. Gloria Pryhuber y a los miembros de su equipo de HuBMAP por proporcionar cortes de tejido pulmonar humano. Agradecemos al Dr. Kun Zhang por proporcionar tejidos cerebrales humanos. También agradecemos al Dr. Gen-Sheng Feng por proporcionar muestras de hígado de ratón. Agradecemos el apoyo de NIHU54DK134301, NIH R01GM149976, NIH U01AI167892, NIH R01HL170107, NIH 5R01NS111039, NIH R21NS125395, NIH U54CA132378, UCSD Startup funds, Sloan Research Fellow Award y CZI DAF2023-328667 Award.
Materiales
| Name | Company | Catalog Number | Comments |
| 460 nm Filter Cube | Olympus | OCT-ET 460/50M32 | |
| Bandpass Filter | KR Electronics | KR2724 | 8 MHz |
| BNC 50 Ohm Terminator | Mini Circuits | STRM-50 | |
| DC power supply | TopWard | 6302D | |
| Dichroic Mount | Thorlabs | KM100CL | |
| FIJI ImageJ | ImageJ | ||
| High NA oil condenser | Olympus | 6-U130 | |
| High O.D. shortpass filter | Thorlabs | 950 nm | |
| Inverted Laser Scanning Microscope | Olympus | FV1200MPE | |
| Lock-in Amplifier | Zurich Instruments | ||
| picoEmerald Laser System | Applied Physics & Electronics, Inc. | Synchronized pulsed pump beam (tunable 720–990 nm wavelength, 5–6 ps pulse width, and 80 MHz repetition rate) and Stokes (wavelength at 1032 nm, 6 ps pulse width, and 80 MHz repetition rate) | |
| SI Photodiode Detector | Home Built | ||
| Touch Panel Controller | Olympus | ||
| VF-300 Compresstome | Precisionary |
Referencias
- Wollman, A. J. M., Nudd, R., Hedlund, E. G., Leake, M. C. From Animaculum to single molecules: 300 years of the light microscope. Open Biol. 5 (4), 150019(2015).
- Vicidomini, G., Bianchini, P., Diaspro, A. STED super-resolved microscopy. Nat Methods. 15 (3), 173-182 (2018).
- Weber, M., et al. MINSTED fluorescence localization and nanoscopy. Nat Photonics. 15 (5), 361-366 (2021).
- Denk, W., Strickler, J. H., Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science. , (1990).
- Hell, S. W., et al. Three-photon excitation in fluorescence microscopy. J Biomed Optics. 1 (1), 71-74 (1996).
- Alhallak, K., Rebello, L. G., Muldoon, T. J., Quinn, K. P., Rajaram, N. Optical redox ratio identifies metastatic potential-dependent changes in breast cancer cell metabolism. Biomed Opt Express. 7 (11), 4364-4374 (2016).
- Pena, A. -M., et al. Multiphoton FLIM imaging of NADH and FAD to analyze cellular metabolic activity of reconstructed human skin in response to UVA light. Multiphoton Microscopy in the Biomedical Sciences XIX. 10882, 23-33 (2019).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomed Opt Express. 11 (10), 5674-5688 (2020).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. J Biol Chem. 254 (11), 4764-4771 (1979).
- Ostrander, J. H., et al. Optical redox ratio differentiates breast cancer cell lines based on estrogen receptor status. Cancer Res. 70 (11), (2010).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proc Natl Acad Sci USA. 104 (49), 19494-19499 (2007).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Res. 74 (11), 3067-3075 (2014).
- Malak, M., James, J., Grantham, J., Ericson, M. B. Contribution of autofluorescence from intracellular proteins in multiphoton fluorescence lifetime imaging. Sci Rep. 12, 16584(2022).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Roth, S., Freund, I. Second harmonic generation in collagen. J Chem Phys. 70 (4), 1637-1643 (1979).
- Freund, I., Deutsch, M. Second-harmonic microscopy of biological tissue. Opt Lett. 11 (2), 94(1986).
- Boyd, R. W., Gaeta, A. L., Giese, E. Nonlinear optics. Springer Handbook of Atomic, Molecular, and Optical Physics. Drake, G. W. F. , Springer. 1097-1110 (2023).
- Aghigh, A., et al. Second harmonic generation microscopy: a powerful tool for bio-imaging. Biophys Rev. 15 (1), 43-70 (2023).
- Mohler, W., Millard, A. C., Campagnola, P. J. Second harmonic generation imaging of endogenous structural proteins. Methods. 29 (1), 97-109 (2003).
- Cox, G. Biological applications of second harmonic imaging. Biophys Rev. 3 (3), 131(2011).
- Brown, E., et al. Dynamic imaging of collagen and its modulation in tumors in vivo using second-harmonic generation. Nat Med. 9 (6), 796-800 (2003).
- Keikhosravi, A., Bredfeldt, J. S., Sagar, A. K., Eliceiri, K. W. Chapter 28 - Second-harmonic generation imaging of cancer. Methods Cell Bio. 123, 531-546 (2014).
- Hui Mingalone, C. K., et al. Bioluminescence and second harmonic generation imaging reveal dynamic changes in the inflammatory and collagen landscape in early osteoarthritis. Lab Invest. 98 (5), 656-669 (2018).
- Tsafas, V., et al. Polarization-dependent second-harmonic generation for collagen-based differentiation of breast cancer samples. J Biophotonics. 13 (10), e202000180(2020).
- Nadiarnykh, O., LaComb, R. B., Brewer, M. A., Campagnola, P. J. Alterations of the extracellular matrix in ovarian cancer studied by Second Harmonic Generation imaging microscopy. BMC Cancer. 10, 94(2010).
- Xiong, S. Y., Yang, J. G., Zhuang, J. Nonlinear spectral imaging of human normal skin, basal cell carcinoma and squamous cell carcinoma based on two-photon excited fluorescence and second-harmonic generation. Laser Phys. 21 (10), 1844-1849 (2011).
- Tilbury, K., Campagnola, P. J. Applications of second-harmonic generation imaging microscopy in ovarian and breast cancer. Perspect Medicin Chem. 7, 21-32 (2015).
- Raman, C. V., Krishnan, K. S. A New type of secondary radiation. Nature. 121 (3048), 501-502 (1928).
- Eckhardt, G., et al. Stimulated Raman scattering from organic liquids. Phys Rev Lett. 9 (11), 455-457 (1962).
- Freudiger, C. W., et al. Label-free biomedical imaging with high sensitivity by stimulated Raman scattering microscopy. Science. 322 (5909), 1857-1861 (2008).
- Li, Y., et al. Microglial lipid droplet accumulation in tauopathy brain is regulated by neuronal AMPK. Cell Metab. 36 (6), 1351-1370.e8 (2024).
- Lu, F. -K., et al. Label-free neurosurgical pathology with stimulated Raman imaging. Cancer Res. 76 (12), 3451-3462 (2016).
- Shi, L., et al. Optical imaging of metabolic dynamics in animals. Nat Commun. 9 (1), 2995(2018).
- Zhang, L., et al. Spectral tracing of deuterium for imaging glucose metabolism. Nat Biomed Eng. 3 (5), 402-413 (2019).
- Li, Y., et al. Bioorthogonal stimulated Raman scattering imaging uncovers lipid metabolic dynamics in Drosophila brain during aging. GEN Biotechnol. 2 (3), 247-261 (2023).
- Li, X., et al. Quantitative imaging of lipid synthesis and lipolysis dynamics in Caenorhabditis elegans by stimulated Raman scattering microscopy. Anal Chem. 91 (3), 2279-2287 (2019).
- Ji, M., et al. Detection of human brain tumor infiltration with quantitative stimulated Raman scattering microscopy. Sci Transl Med. 7 (309), 309ra163(2015).
- Ji, M., et al. Rapid, label-free detection of brain tumors with stimulated Raman scattering microscopy. Sci Transl Med. 5 (201ra119), 201ra119(2013).
- You, S., et al. Intravital imaging by simultaneous label-free autofluorescence-multiharmonic microscopy. Nat Commun. 9, 2125(2018).
- You, S., et al. Label-free deep profiling of the tumor microenvironment. Cancer Res. 81 (9), 2534-2544 (2021).
- Jermyn, M., et al. Highly accurate detection of cancer in situ with intraoperative, label-free, multimodal optical spectroscopy. Cancer Res. 77 (14), 3942-3950 (2017).
- Pshenay-Severin, E., et al. Multimodal nonlinear endomicroscopic imaging probe using a double-core double-clad fiber and focus-combining micro-optical concept. Light Sci Appl. 10 (1), 207(2021).
- Wu, W., Li, X., Qu, J. Y., He, S. In vivo imaging of biological tissues with combined two-photon fluorescence and stimulated Raman scattering microscopy. J Vis Exp. (178), e63411(2021).
- Li, X., Jiang, M., Lam, J. W. Y., Tang, B. Z., Qu, J. Y. Mitochondrial imaging with combined fluorescence and stimulated Raman scattering microscopy using a probe of the aggregation-induced emission characteristic. J Am Chem Soc. 139 (47), 17022-17030 (2017).
- Li, Z., et al. Multimodal imaging of metabolic activities for distinguishing subtypes of breast cancer. Biomed Opt Express. 14 (11), 5764-5780 (2023).
- Fung, A. A., et al. Label-free optical biopsy reveals biomolecular and morphological features of diabetic kidney tissue in 2D and 3D. , (2024).
- Zhang, W., et al. Multi-molecular hyperspectral PRM-SRS microscopy. Nat Commun. 15 (1), 1599(2024).
- Xu, F. X., et al. Discrimination of lipid composition and cellular localization in human liver tissues by stimulated Raman scattering microscopy. J Biomed Opt. 29 (1), 016008(2024).
- Zoumi, A., Yeh, A., Tromberg, B. J. Imaging cells and extracellular matrix in vivo by using second-harmonic generation and two-photon excited fluorescence. Proc Natl Acad Sci USA. 99 (17), 11014-11019 (2002).
- Liaudanskaya, V., et al. Mitochondria dysregulation contributes to secondary neurodegeneration progression post-contusion injury in human 3D in vitro triculture brain tissue model. Cell Death Dis. 14 (8), 1-15 (2023).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, sub-types and early treatment response in breast cancer. Cancer Res. 73 (20), 6164-6174 (2013).
- Conklin, M. W., et al. Aligned collagen is a prognostic signature for survival in human breast carcinoma. Am J Pathol. 178 (3), 1221-1232 (2011).
- Sun, W., et al. Nonlinear optical microscopy: use of second harmonic generation and two-photon microscopy for automated quantitative liver fibrosis studies. J Biomed Optics. 13 (6), 064010(2008).
- Nejim, Z., Navarro, L., Morin, C., Badel, P. Quantitative analysis of second harmonic generated images of collagen fibers: a review. Res Biomed Eng. 39 (1), 273-295 (2023).
- Burke, K., Brown, E. The use of second harmonic generation to image the extracellular matrix during tumor progression. Intravital. 3 (3), e984509(2015).
- Balasubramanian, P., Prabhakaran, M. P., Sireesha, M., Ramakrishna, S. Collagen in human tissues: Structure, function, and biomedical implications from a tissue engineering perspective. Polymer Composites - Polyolefin Fractionation - Polymeric Peptidomimetics - Collagens. Abe, A., Kausch, H. H., Möller, M., Pasch, H. 251, 173-206 (2012).
- Dadsena, S., et al. Lipid unsaturation promotes BAX and BAK pore activity during apoptosis. Nat Commun. 15, 4700(2024).
- Barelli, H., Antonny, B. Lipid unsaturation and organelle dynamics. Curr Opin Cell Biol. 41, 25-32 (2016).
- Harayama, T., Antonny, B. Beyond fluidity: The role of lipid unsaturation in membrane function. Cold Spring Harb Perspect Biol. 15 (7), a041409(2023).
- Yoon, H., Shaw, J. L., Haigis, M. C., Greka, A. Lipid metabolism in sickness and in health: Emerging regulators of lipotoxicity. Mol Cell. 81 (18), 3708-3730 (2021).
- Villazon, J., Shi, L. Utilizing K-means clustering on hyperspectral DO-SRS images of the mouse hippocampus. CLEO 2024, Technical Digest Series. , Optica Publishing Group. (2024).
- Fu, D., et al. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy. J Am Chem Soc. 134 (8), 3623-3626 (2012).
- Periasamy, A., Diaspro, A. Special section guest editorial: Multiphoton microscopy. J Biomed Optics. 8 (3), 327(2003).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci USA. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nat Commun. 5, 3936(2014).
- Xiong, H., et al. Background-free imaging of chemical bonds by a simple and robust frequency-modulated stimulated Raman scattering microscopy. Opt Express. 28 (10), 15663-15677 (2020).
- Georgakoudi, I., Quinn, K. P. Label-free optical metabolic imaging in cells and tissues. Annu Rev Biomed Eng. 25, 413-443 (2023).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon FLIM microscopy. Sci Rep. 12 (1), 11938(2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados