Method Article
Generación y caracterización de astrocitos derivados de células madre pluripotentes inducidas humanamente que carecen de ribonucleoproteína mensajera X frágil
En este artículo
Resumen
Aquí describimos un protocolo que facilita la diferenciación de células madre pluripotentes inducidas humanas en astrocitos funcionales específicos del prosencéfalo. Esto permite investigar el papel de las células gliales en la patogénesis de los trastornos del neurodesarrollo, como el síndrome del cromosoma X frágil, y modelar otros trastornos cerebrales.
Resumen
El síndrome del cromosoma X frágil (SXF), una de las principales causas hereditarias del trastorno del espectro autista y la discapacidad intelectual, se ha estudiado ampliamente utilizando modelos de roedores. Más recientemente, los sistemas modelo derivados de células madre humanas también se han utilizado para obtener información mecanicista sobre la fisiopatología del SXF. Sin embargo, estos estudios se han centrado casi exclusivamente en las neuronas. Además, a pesar de la creciente evidencia de un papel clave de la glía en la función neuronal en la salud y la enfermedad, se sabe poco sobre cómo los astrocitos humanos se ven afectados por el SXF.
Por lo tanto, en este estudio, desarrollamos con éxito un protocolo que captura los hitos espacio-temporales clave del desarrollo del cerebro y también se alinea con el proceso de gliogénesis. En conjunto, esto ofrece un marco útil para estudiar los trastornos del neurodesarrollo. En primer lugar, modelamos las células madre pluripotentes inducidas humanas en el linaje neuroectodérmico con doble supresor de madres contra la inhibición decapentopléjica (SMAD) y moléculas pequeñas. Posteriormente, utilizamos factores de crecimiento específicos y citocinas para generar células progenitoras astrocíticas (APC) derivadas de pacientes de control (CTRL) y FXS. El tratamiento de las APC con factor neurotrófico ciliar, una citocina diferenciadora, reguló y condujo a las células progenitoras hacia la maduración astrocítica, produciendo astrocitos que expresan proteínas ácidas fibrilares gliales específicas del prosencéfalo.
Encontramos que estos astrocitos son funcionales, como lo demuestran sus respuestas de calcio a la aplicación de ATP, y exhiben un metabolismo glucolítico y mitocondrial desregulado en el FXS. Tomados en conjunto, estos hallazgos proporcionan una plataforma experimental útil de origen humano para la investigación de las consecuencias autónomas y no celulares de las alteraciones en la función astrocítica causadas por trastornos del neurodesarrollo.
Introducción
El síndrome del cromosoma X frágil (SXF), una forma hereditaria común de discapacidad intelectual y trastorno del espectro autista (TEA), es causada por la falta de ribonucleoproteína mensajera del cromosoma X frágil (FMRP) producida por el gen de la ribonucleoproteína 1 mensajera del cromosoma X frágil (FMR1) (OMIM: #300624, https://www.omim.org/entry/300624). FMRP desempeña un papel en la regulación de la traducción de ARNm, la formación y el transporte de gránulos de ARNm y la regulación de la expresión génica mediada por microARN1. Por lo tanto, la pérdida de FMRP afecta no solo el desarrollo del cerebro, sino también la función cerebral adulta. Tanto los niveles de transcripción de ARNm de FMR1 como la inmunotinción para FMRP en el cerebro han mostrado una alta expresión neuronal, junto con una expresión significativa en las células gliales2. Sin embargo, la gran mayoría de los estudios anteriores en modelos animales de SXF se centraron principalmente en las neuronas y las aberraciones en su función. En consecuencia, se sabe poco sobre el papel de la glía en FXS3. Tradicionalmente consideradas como células de "soporte pasivo"4, cada vez hay más pruebas de que los astrocitos son fundamentales para mediar una amplia gama de funciones neuronales 5,6, incluyendo la promoción de la sinaptogénesis7, el refinamiento de los circuitos neuronales en desarrollo8 y el reciclaje de neurotransmisores9. Paralelamente, cada vez hay más evidencias del papel de los astrocitos en la patogénesis de la enfermedad y muchas afecciones neurológicas se han asociado con la disfunción astrocítica10.
Si bien gran parte de los trabajos anteriores en los que se utilizaron modelos animales de SXF se centraron en identificar y validar varios objetivos moleculares en las neuronas para el tratamiento del SXF, estos hallazgos preclínicos no siempre han dado lugar a resultados clínicos exitosos. Además, los reveses en los ensayos clínicos recientes también subrayan la necesidad de sistemas modelo basados en humanos. Los modelos de trastornos neurológicos basados en células cerebrales derivadas de células madre humanas ofrecen una poderosa estrategia para cerrar esta brecha entre los conocimientos mecanicistas de los estudios en animales y el éxito limitado con los resultados clínicos para los pacientes. Sin embargo, solo un puñado de estos estudios se han centrado en los astrocitos y también en los astrocitos de origen espinal. Esto, a su vez, es relevante a la luz de los estudios que muestran que la estructura y función de los astrocitos varía entre las regiones cerebrales11,12. Por lo tanto, una mejor comprensión de los cambios inducidos por enfermedades en los astrocitos humanos también debe tener en cuenta estas diferencias específicas de la región cerebral en los astrocitos. Sin embargo, los modelos de trastornos del neurodesarrollo que utilizan astrocitos derivados de células madre humanas que son específicos del prosencéfalo siguen siendo comparativamente poco explorados13. Por lo tanto, para comenzar a abordar estas brechas, describimos protocolos para generar astrocitos específicos del prosencéfalo a partir de células madre pluripotentes inducidas (iPSC) derivadas de pacientes portadoras de mutaciones en el SXF; Además, demostramos que los astrocitos son funcionales y muestran un metabolismo alterado.
Protocolo
Todos los experimentos con células madre pluripotentes inducidas humanas (hiPSCs) (Tabla 1) se realizaron después de obtener las aprobaciones regulatorias institucionales apropiadas. La Figura 1A representa el protocolo completo de diferenciación de las hiPSCs a los astrocitos maduros específicos del prosencéfalo.
1. Mantenimiento y expansión de las hiPSC
- Un día antes de enchapar las iPSC, cubra un plato de 6 pocillos con Matrigel diluido 1:60 (una matriz extracelular [ECM]) en Advanced Dulbecco's Modified Eagle Medium/Ham's F-12 (DMEM/F12) y almacene a 2-8 °C.
NOTA: Evite almacenar placas recubiertas de Matrigel durante >5 días a 2-8 °C, ya que esto puede causar la degradación de proteínas en el ECM con el tiempo. Para evitar que el plato se seque, cubra un mínimo de 1 ml de Matrigel diluido 1:60 por pocillo de un plato de 6 pocillos y extiéndalo uniformemente por todo el plato. Si se usa el mismo día, mantenga la placa en una incubadora humidificada a 37 °C y 5% de CO2 durante 1 h. - El día del cultivo de las hiPSCs, retire el material de recubrimiento y añada 1 mL de medio Essential 8 completo (medio basal E8 con suplemento Essential 8) junto con el inhibidor de ROCK 1x concentración final y mantenga la placa en una incubadora a 37 °C y 5% de CO2 antes de añadir las hiPSCs.
- Vuelva a suspender las hiPSC en medio E8 completo junto con el inhibidor de ROCK a 1x concentración final para una mejor adherencia de las colonias.
NOTA: No prepare grandes cantidades de medio de cultivo. Prepare solo durante 3-4 días como máximo y almacene a 2-8 °C. - Al día siguiente, reponga el medio E8 completo sin el inhibidor de ROCK hasta que las células estén confluentes en un 80% (aproximadamente 4-5 días).
- Cuando alcancen el 80% de confluencia, separar las colonias enzimáticamente.
- Para el paso de las iPSC, retire el medio gastado de la placa y agregue 1 mL por pocillo de mezcla precalentada (37 °C) de colagenasa (2 mg/mL) y dispasa (1 mg/mL) en una proporción de 1:1 e incube a 37 °C durante más de 20-30 min para permitir que las colonias de iPSC se levanten.
NOTA: No deje los platos a 37 °C durante más de 20-30 min; Más allá de este período, las colonias se desintegrarán y las células morirán.
- Para el paso de las iPSC, retire el medio gastado de la placa y agregue 1 mL por pocillo de mezcla precalentada (37 °C) de colagenasa (2 mg/mL) y dispasa (1 mg/mL) en una proporción de 1:1 e incube a 37 °C durante más de 20-30 min para permitir que las colonias de iPSC se levanten.
- Cuando las colonias comiencen a levantarse, saque las placas de la incubadora y neutralice la actividad enzimática agregando 2 mL de solución salina tamponada con fosfato (DPBS) de Dulbecco.
- Raspe las colonias con DPBS y recoja la suspensión en un tubo cónico de 15 mL con una pipeta serológica de 10 mL de diámetro ancho.
- Triture suavemente la suspensión 2-3 veces para romper las colonias con una pipeta serológica de 10 ml y deje que las colonias se asienten.
NOTA: No convierta las colonias en celdas individuales; Esto puede hacer que las células no se adhieran a la placa y causar más muerte celular. Además, no las dejes como grandes colonias; Esto dará lugar a colonias más diferenciadas más adelante. - Una vez que las colonias se asienten (aproximadamente después de 2 minutos), aspire la mezcla de enzimas DPBS dejando aproximadamente 1 mL en el tubo.
- Agregue 2 ml de DPBS al tubo, mezcle las colonias dando golpecitos y deje que se asienten. Repita esto 2 veces para eliminar toda la enzima residual de las colonias. Retire la mayor cantidad posible de sobrenadante después del2º lavado, vuelva a suspender las colonias en 1 ml de medio E8 completo y coloque en un plato recién preparado como se menciona en los pasos 1.2-1.3.
- Alternativamente, criopreservar una proporción de colonias utilizando un 10% de dimetilsulfóxido (DMSO) como solución crioprotectora para su futura expansión y uso.
- Prepare el medio de criopreservación recién hecho mezclando un 90% de medio E8 completo y un 10% de DMSO como solución crioprotectora y colóquelo a 2-8 °C hasta su uso.
- Para criopreservar, siga los pasos 1.5-1.10.
- Durante el paso 1.10, después del2º lavado, deje que las colonias se asienten y elimine el sobrenadante. A las colonias, agregue 1 mL de medio de criopreservación en frío recién preparado y transfiéralo a crioviales.
NOTA: Después de agregar el medio de criopreservación a las colonias, transfiera rápidamente el contenido a los crioviales. El retraso puede resultar en una tasa de reactivación más baja, ya que el DMSO es un crioprotector que puede dañar las células. - Mueva inmediatamente los crioviales a una caja criogénica y manténgalos en un congelador a -80 °C durante la noche.
- Al día siguiente, cambie todos los crioviales a un tanque de nitrógeno líquido (tanque LN2 ) para su uso futuro.
NOTA: Las hiPSC deben ser cariotipadas rutinariamente usando bandas G (Figura Suplementaria S1) para cualquier anormalidad, caracterizadas usando inmunocitoquímica para la pluripotencia (Figura 1B) y analizadas para micoplasma.- Para caracterizar las hiPSCs mediante inmunocitoquímica, coloque las colonias en cubreobjetos de vidrio de 13 mm recubiertos de Matrigel y esterilizados en autoclave a escala 1:60. Una vez que tengan un 40% de confluente, lavar las células con PBS-T (PBS-0,1% Tween 20), fijar con paraformaldehído al 4% durante 10 min, permeabilizar con Triton X-100 al 0,3% en PBS durante 10 min, y bloquear con albúmina sérica bovina (BSA) al 3% en PBS durante 1 h para evitar la unión inespecífica.
- Después del bloqueo, incubar los cultivos con anticuerpos primarios durante 1 h y luego lavar 3 veces con PBS-T durante 5 min cada uno, seguido de los anticuerpos secundarios correspondientes (Tabla de Materiales) en la oscuridad durante 1 h.
- Monte los cubreobjetos en portaobjetos de vidrio con medio de montaje y adquiera imágenes mediante escaneo láser confocal a 405 nm, 488 nm, 561 nm y 633 nm. Captura imágenes a 512 x 512 píxeles; establezca el tamaño del paso Z en 0,5 μm con 1 unidad aireada de diámetro de agujero de alfiler.
2. Generación y caracterización de células progenitoras astrocíticas (APCs)
- Levante enzimáticamente las hiPSC como se menciona en los pasos 1.5-1.10 y colóquelas en una placa de cultivo en suspensión no adherente (100 mm) con medio químicamente definido14 que contenga 50% de medio de Dulbecco modificado de Iscove (IMDM), 50% de mezcla de nutrientes F-12 de Ham (F12), 5 mg/mL de BSA, 1% de concentrado de lípidos químicamente definidos (CD-Lípido), 450 μM de monotioglicerol, 7 μg/mL de insulina, 15 μg/mL de transferrina, Penicilina-estreptomicina al 1% suplementada con mitógenos N-acetilcisteína (1 mM), LDN 193189 (0,1 μM) y SB431542 (10 μM) durante 7 días.
NOTA: Los inhibidores de moléculas pequeñas SB431542 y LDN-193189 (LDN) son inhibidores de la proteína morfogenética ósea y de las vías de señalización del factor de crecimiento beta transformante.
A partir de este momento, el medio se repuso una vez cada 2 días o siguiendo el protocolo de lunes/miércoles/viernes. - Coloque la suspensión celular en un agitador orbital a 40 rpm durante 7 días en condiciones normóxicas para ayudar al desarrollo de corticosferas (Figura 1A).
- El día 8, transfiera las corticosferas a un medio de proliferación celular que contenga DMEM/F12 avanzado con un 1% de antibiótico-antimicótico, un suplemento de N2 al 1%, un sustituto de glutamina al 1%, un suplemento de B27 al 0,1% y 2,5 ng/ml de factor de crecimiento básico de fibroblastos (bFGF) durante 7 días.
- Inducir las esferas para la especificación glial sometiéndolas a un medio de enriquecimiento glial que contenga DMEM/F12 avanzado con 1% de antibiótico-antimicótico, 1% de N2, 1% de sustituto de glutamina, suplemento de 0,1% de B27, 20 ng/mL de factor de crecimiento epidérmico (EGF), bFGF-H (20 ng/mL de bFGF-5 mg/mL de heparina) durante 2 semanas para obtener gliosferas tempranas (Figura 1A).
- Para la maduración de las gliosferas tempranas, reemplace bFGF-H con 20 ng/mL de factor inhibidor de la leucemia (LIF) y mantenga las esferas durante 4 semanas.
- Después de 4 semanas en el medio de maduración, mantener las esferas en medio de enriquecimiento glial durante períodos prolongados. Para evitar la agregación y la pérdida de viabilidad, cada 2 semanas, corte mecánicamente las gliosferas con una cuchilla industrial estéril y reemplace todo el medio con DNasa I para eliminar los fragmentos de ADN generados por el corte.
- Disocie las gliosferas en monocapas de APC utilizando un kit de disociación de papaína y una placa en una placa adherente tratada con cultivo celular con dilución 1:80 de recubrimiento Matrigel.
- Propagar las APCs en un medio de enriquecimiento glial hasta que el 80% confluente y hacerlas pasar enzimáticamente utilizando el medio de desprendimiento de células enzimáticas (ver la Tabla de Materiales).
- Al paso:
- Retire los medios gastados y recójalos en un tubo cónico. A las células, agregue el medio de desprendimiento de células enzimáticas y espere 1-2 minutos. Una vez que las células comiencen a desprenderse, agregue los medios gastados para neutralizar la actividad enzimática.
- Recoja la suspensión de la célula y la centrífuga a 800 x ɡ durante 2 minutos.
- Aspire el sobrenadante, vuelva a suspender las células en un medio de enriquecimiento glial y coloque aproximadamente 1 x 106 células/pocillo en una placa de 6 pocillos recubierta de Matrigel a escala 1:80.
- Para la criopreservación:
- Resuspender las células en una mezcla fría de 90% de medio de proliferación de corticosfera (sin bFGF) y 10% de crioprotector. Transfiera las células resuspendidas a crioviales.
- Mueva inmediatamente los crioviales a una caja criogénica y manténgalos en un congelador a -80 °C durante la noche.
- Al día siguiente, cambie todos los crioviales a un tanque de nitrógeno líquido (tanque LN2 ) para su uso futuro.
NOTA: Las APC fueron criopreservadas usando DMSO al 10% hasta el pasaje número 6.
- Caracterice las APC disociadas mediante inmunotinción (como se menciona en los pasos 1.15.1-1.15.3) con vimentina y marcadores de factor nuclear IA (NFIA) (Figura 1C, D) (consulte la Tabla de materiales para diluciones).
- Para confirmar la especificidad regional del prosencéfalo de las APC derivadas de hiPSC, pruebe las células en busca de un marcador del prosencéfalo, como la caja de horquilla humana G1 (hFOXG1) (positivo) y un marcador del cerebro posterior, como el Homeobox B4 humano (hHOXB4) (negativo) mediante qPCR en tiempo real (Figura 1E,F).
NOTA: Las secuencias de cebadores utilizadas para estos experimentos se han enumerado en la Tabla Suplementaria S1.
3. Generación y caracterización de una población homogénea de astrocitos específicos del prosencéfalo
- Diferenciación de astrocitos de APCs
- Utilice el medio de diferenciación astrocítico (ADM) durante 14 días para diferenciar las APC en astrocitos. El medio de diferenciación astrocítico contiene Neurobasal, 1% antibiótico-antimicótico, 1% sustituto de glutamina, 1% de suplemento de N2, 0,2% de suplemento de B27, 1% de medio de aminoácidos no esenciales (NEAA) y 10 ng/mL de factor neurotrófico ciliar (CNTF).
- Confirmar la identidad astrocítica mediante inmunotinción (como se menciona en los pasos 1.15.1-1.15.3) con marcadores de expresión de proteína ácida fibrilar glial (GFAP) y S100β (Figura 2A,B).
- Síntesis de proteínas de novo en APCs y astrocitos
- Mantenga las APC y los astrocitos del prosencéfalo derivados de hiPSC en cubreobjetos de vidrio estériles de 13 mm esterilizados en autoclave e inmunotiña como se menciona en los pasos 1.15.1-1.15.3.
- Para la síntesis de proteínas de novo , utilice el método de marcado de aminoácidos fluorescente no canónico (FUNCAT), explicado brevemente en los pasos 3.2.3-3.2.6 (Figura 3A).
- Para asegurar la deficiencia de metionina, retire el medio de crecimiento de los cultivos y reemplácelo con un medio libre de metionina y cisteína entremezclado con L-azidohomoalanina (AHA) de 1 mM durante 30 min a 37 °C y 5% de CO2.
- Lavar los cultivos con PBS-T, fijar con paraformaldehído al 4% durante 10 min, permeabilizar con Triton X-100 al 0,3% en PBS durante 10 min y bloquear con 3% de BSA en PBS durante 1 h para evitar la unión inespecífica.
- Incubar los cultivos en la oscuridad durante 1 h a temperatura ambiente con la mezcla de reacción química de células Click y Alkyne Alexa Fluor 647, seguido de anticuerpos primarios durante 1 h y los anticuerpos secundarios correspondientes (Tabla de Materiales) durante 1 h (Figura 3B).
- Monte los cubreobjetos en portaobjetos de vidrio con medio de montaje y utilícelos para un análisis posterior de la imagen.
- Adquiera imágenes mediante escaneo láser confocal a 405 nm, 488 nm, 561 nm y 633 nm.
- Captura imágenes a 512 x 512 píxeles; establezca el tamaño del paso Z en 0,5 μm con 1 unidad aireada de diámetro de agujero de alfiler.
- Mantenga los parámetros de microscopía e imagen en un ajuste constante en todos los tipos de células. Captura imágenes de cada réplica biológica en la misma sesión.
- Realice mediciones de intensidad utilizando cualquier software de análisis de imágenes estándar (por ejemplo, Fiji o Imaris). En Imaris, utilice el módulo SURFACES para obtener mediciones volumétricas de cuerpos celulares positivos para la señal fluorescente de marcado de aminoácidos no canónicos (FUNCAT) con Vimentin (APCs) y GFAP (astrocitos) limitando el rango de vóxeles (250-350) para eliminar los desechos.
NOTA: Mantenga los parámetros en todas las réplicas biológicas para ambos tipos de células.
- Ondas oscilatorias de calcio inducidas por adenosina 5′-trifosfato (ATP)
- Placa de astrocitos en una placa con fondo de vidrio (35 mm) a 5 × 103 células/placa para medir la respuesta celular al ATP.
- Dejar que las células se adhieran al fondo de vidrio durante 24 h, lavarlas 3 veces con HBSS (20 mM HEPES, 137 mM NaCl, 5 mM KCl, 10 mM Glucosa, 1 mM MgCl2, pH = 7,3) sin calcio, e incubar en medio de cultivo con 5 μM de colorante métrico Fura-2AM y 0,02% Pluronics F127 durante 1 h a temperatura ambiente.
NOTA: Fura-2AM es un producto químico sensible a la luz y a la temperatura; almacenar a -20 °C. - Después de la incubación, lave 2 veces con medio de cultivo y reemplácelo con 2 mM de Ca2+ que contenga HBSS y obtenga imágenes de las células a una velocidad de 2 FPS utilizando un objetivo de aceite de 60x (1,35 NA) en un microscopio invertido con compensación de deriva de foco.
- Registre las respuestas de calcio inducidas por ATP mediante la aplicación de ATP en baño a la concentración final de 5 mM a 25th s (Figura 4A).
- Dibuje regiones de interés alrededor de cada celda utilizando Fiji/ImageJ y calcule la relación F340/F380 en todos los puntos de tiempo (Figura 4B).
- Ensayos metabólicos celulares
NOTA: La tasa de consumo de oxígeno (OCR) y la tasa de acidificación extracelular (ECAR) de las células vivas se midieron según el protocolo del fabricante (consulte la Tabla de materiales).- Un día antes del ensayo, dejar que los astrocitos se adhieran a la microplaca (1,5 ×10 4 densidad de siembra) en medio CNTF e incubar a 37 °C con 5% de CO2.
- Para el ensayo de estrés por glucólisis:
- Sustituir el medio de cultivo celular por un medio base suplementado con 2 mM de glutamina (pH ajustado a 7,4) e incubar en una cámara sin CO2 a 37 °C durante 1 h.
NOTA: el pH debe ajustarse en un baño de agua a 37 °C. - Inserte la placa del cartucho en el instrumento para calibrar el sensor. Después de la calibración, reemplace la placa del cartucho con la placa de cultivo celular a una concentración final de los siguientes componentes del kit: 10 mM de glucosa, 1 μM de oligomicina y 50 mM de 2-desoxiglucosa (2-DG) (Figura 5A).
- Al final de la prueba, lisar las células y estimar el contenido total de proteínas.
- Sustituir el medio de cultivo celular por un medio base suplementado con 2 mM de glutamina (pH ajustado a 7,4) e incubar en una cámara sin CO2 a 37 °C durante 1 h.
- Para el ensayo de estrés mitocondrial:
- Sustituya el medio CNTF por un medio base suplementado con 2 mM de glutamina, 1 mM de piruvato y 10 mM de glucosa (pH ajustado a 7,4) en un baño de agua a 37 °C.
- Incubar la placa en una cámara sin CO2 a 37 °C durante 1 h. Mientras tanto, inserte la placa del cartucho en el instrumento para calibrar el sensor.
- Después de la calibración, reemplace la placa del cartucho con la placa de cultivo celular con una concentración final de los siguientes componentes del kit: 1,5 μM de oligomicina, 1 μM de cianuro de carbonilo-4 (trifluorometoxi) fenilhidrazona (FCCP) y 0,5 μM de rotenona/antimicina A (Figura 5C).
- Al final de la prueba, lisar las células y estimar el contenido total de proteínas.
Resultados
Las colonias de células madre pluripotentes inducidas humanas (hiPSC) se mantuvieron utilizando medio definido disponible comercialmente e inmunoteñidas para marcadores de pluripotencia, Oct4 y Nanog (Figura 1B). Las APC FXS y CTRL mostraron proporciones comparables altamente enriquecidas de células inmunopositivas para vimentina y NFIA (Figura 1C,D). Descubrimos que las APC derivadas de hiPSC sanas y FXS mostraron una regulación positiva sustancial de FOXG1 en comparación con HOXB4 (Figura 1F), lo que es consistente con una identidad predominante del prosencéfalo. Observamos una reducción significativa en el número de astrocitos que expresan GFAP en los grupos FXS en comparación con los astrocitos CTRL sin afectar el número de astrocitos que expresan S100β (Figura 2B). El análisis de Western blot mostró que los astrocitos generados a partir de ambas líneas de FXS carecían de expresión de FMRP (Figura 2C, D). La cuantificación de las proteínas recién sintetizadas no reveló ninguna diferencia significativa entre las líneas CTRL frente a las líneas FXS en APC o astrocitos (Figura 3C). Sin embargo, encontramos que la síntesis de proteínas fue consistentemente mayor en las APC que en sus respectivos astrocitos para cada línea (Figura 3C). Estos resultados sugieren una reducción específica de la etapa en la síntesis de proteínas de novo de APC a astrocitos en las líneas FXS y CTRL.
El control, así como los astrocitos del FXS, mostraron transitorios de calcio inducidos por ATP (Figura 4A). Sin embargo, un análisis más detallado reveló varias diferencias clave entre los transitorios de calcio individuales registrados en los astrocitos CTRL, FXS1 y FXS2 (Figura 4B). Clasificamos a los astrocitos como respondedores y no respondedores al ATP en función de la presencia o ausencia de respuesta al calcio, respectivamente (Figura 4C). El número de respondedores en FXS1 y FXS2 fue significativamente menor que el CTRL. En concreto, observamos una reducción significativa en la amplitud máxima del primer transitorio de calcio evocado tras la aplicación de ATP en ambas líneas de astrocitos FXS (Figura 4D). Además, la duración total del primer transitorio de calcio fue significativamente más corta en las líneas FXS1 y FXS2 que en CTRL (Figura 4E). Sin embargo, el número de eventos de calcio (cuantificado como el número total de eventos por 250 s; Figura 4F), fue comparable entre líneas. En conjunto, estos hallazgos revelan alteraciones en las respuestas de calcio provocadas por el ATP en los astrocitos FXS derivados de hiPSC. Los resultados de ECAR sugieren una mayor tasa de glucólisis, capacidad glucolítica y reserva glucolítica en astrocitos FXS derivados de hiPSC (Figura 5A,B). No encontramos diferencias significativas en la respiración basal en los astrocitos CTRL y FXS. La producción celular de ATP no mostró diferencias significativas entre los astrocitos CTRL y FXS. La estimulación de FCCP provocó una respiración máxima significativamente más baja en los astrocitos FXS que en los astrocitos CTRL (Figura 5C, D).
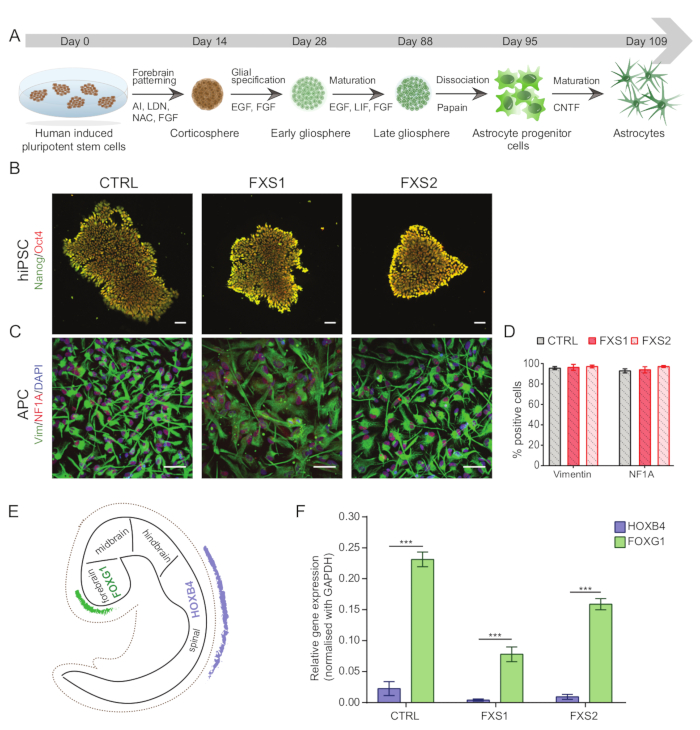
Figura 1: Derivación de células progenitoras astrocíticas específicas del prosencéfalo a partir de hiPSCs. (A) Flujo de trabajo ilustrativo para la generación de astrocitos a partir de hiPSCs. (B) Imágenes representativas de hiPSCs que muestran una expresión comparable de Oct4/Nanog en líneas CTRL y FXS. (C) La población homogénea de APC derivadas de hiPSC expresó un número similar de células positivas para vimentina y NFIA. (D) No hay diferencias significativas en la expresión de Vimentina- y NFIA entre CTRL y FXS APCs. El análisis estadístico se realizó mediante ANOVA de dos factores seguido de la comparación por pares de Tukey. (E) Esquema que ilustra la expresión regional específica de FOXG1 y HOXB4 durante el desarrollo in vivo. (F) Representación gráfica de la expresión de FOXG1 en comparación con HOXB4 (usando qRT-PCR) en todos los genotipos, lo que sugiere propensión hacia el linaje del prosencéfalo. Análisis estadístico por ANOVA de dos factores seguido de la prueba de comparación múltiple de Sidak. Para todos los experimentos, N = 3 réplicas biológicas. p < 0,001. Barra de escala = 50 μm. Las barras de error representan SEM. Abreviaturas: hiPSCs = células madre pluripotentes inducidas humanas; CTRL = Control; FXS = Síndrome del X frágil; APCs = células progenitoras de astrocitos. Haga clic aquí para ver una versión más grande de esta figura.
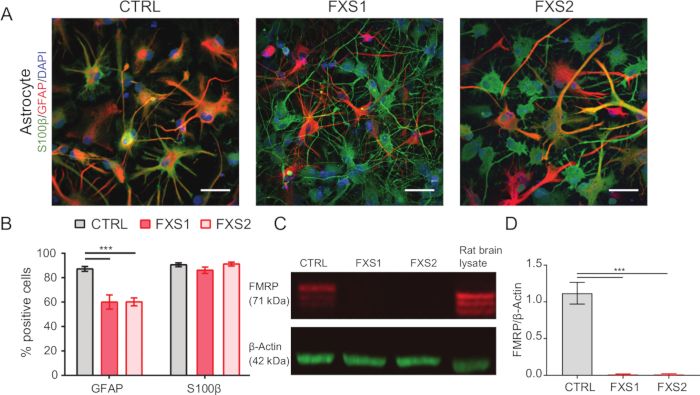
Figura 2: Reducción del número de astrocitos FXS derivados de hiPSC positivos para proteínas ácidas fibrilares gliales. (A) Imágenes representativas de astrocitos derivados de hiPSC que exhiben expresión de S100β y GFAP. (B) El porcentaje de astrocitos positivos para GFAP fue significativamente menor en los astrocitos derivados de FXS en comparación con CTRL, aunque ambos exhibieron una expresión similar de S100β. La significación estadística se determinó mediante ANOVA de dos factores seguido de una comparación por pares de Tukey. (C) Inmunoblot que representa la expresión de FMRP en astrocitos a partir de CTRL y FXS. (D) Gráficos que muestran la ausencia de FMRP en astrocitos FXS derivados de hiPSC. Análisis estadístico realizado por ANOVA de un solo factor con comparación por pares de Tukey. Para todos los experimentos anteriores, N = 3 réplicas biológicas. Barra de escala = 50 μm. ***p < 0,001. Las barras de error representan SEM. Abreviaturas: hiPSCs = células madre pluripotentes inducidas humanas; CTRL = Control; FXS = Síndrome del X frágil; GFAP = proteína ácida fibrilar glial; FMRP = ribonucleoproteína mensajera del cromosoma X frágil. Haga clic aquí para ver una versión más grande de esta figura.
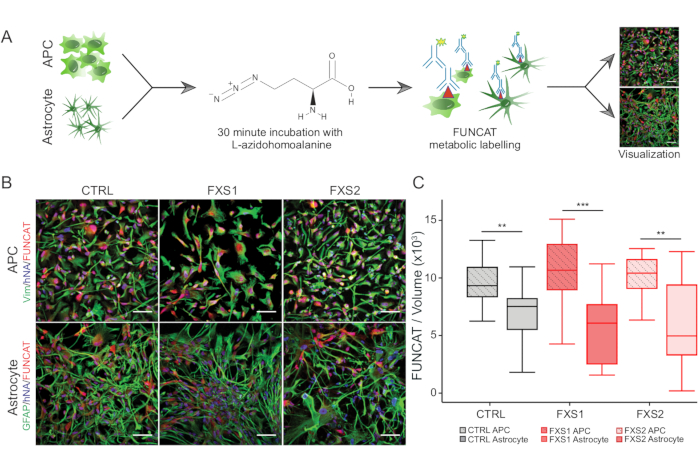
Figura 3: Reducción de la síntesis de proteínas debido a la diferenciación de APC a astrocitos. (A) Flujo de trabajo esquemático que representa el etiquetado y la visualización de la síntesis de proteínas en APC y astrocitos derivados de hiPSC utilizando el marcaje metabólico FUNCAT. (B) Imágenes representativas de (arriba) APC derivadas de hiPSC positivas para vimentina y astrocitos positivos para GFAP (abajo) con marcaje FUNCAT/anticuerpos de núcleos humanos (hNA) en células derivadas de CTRL y FXS. (C) El volumen de FUNCAT/APCs derivadas de hiPSC fue significativamente mayor que el de los astrocitos derivados correspondientes. Para todos los experimentos, N = 3 réplicas biológicas. Barra de escala = 50 μm. **p < 0,01, ***p < 0,001. Los bigotes representan 1,5 × IQR. Abreviaturas: hiPSCs = células madre pluripotentes inducidas humanas; CTRL = Control; FXS = Síndrome del X frágil; GFAP = proteína ácida fibrilar glial; FUNCAT = marcaje fluorescente de aminoácidos no canónicos. Haga clic aquí para ver una versión más grande de esta figura.
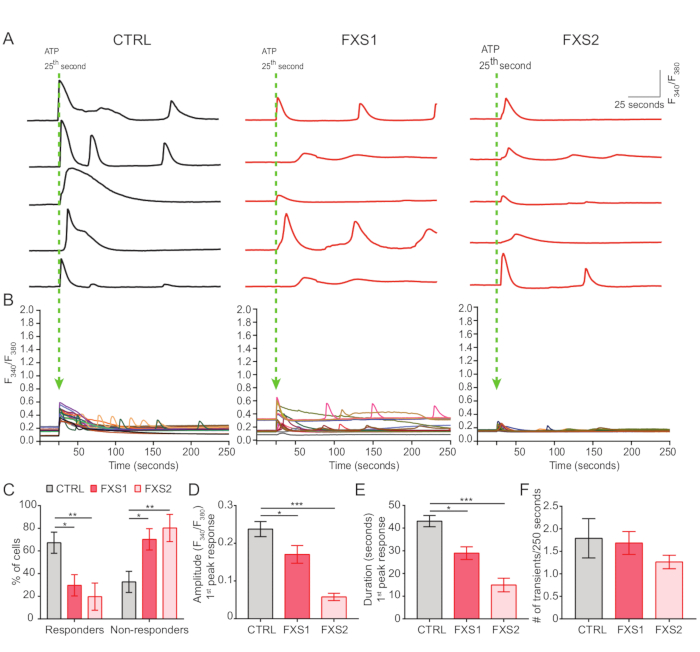
Figura 4: Señalización deficiente de Ca2+ evocada por ATP en astrocitos FXS derivados de hiPSC. (A) Trazas representativas de transitorios de Ca2+ registrados a partir de astrocitos individuales tras la aplicación externa de ATP a 25th . (B) Relaciones promediadas F340/F380 que representan transitorios de Ca2+ durante 250 s después de la aplicación de ATP. (C) Datos agrupados que muestran un mayor porcentaje de no respondedores a ATP en astrocitos FXS derivados de hiPSC. Significación estadística determinada por ANOVA de dos factores con comparación pareada de Tukey. (D-F) Cuantificación de la respuesta del primer pico (amplitud y duración) y frecuencia de eventos. (D) Muestra una reducción significativa en la amplitud. Significación estadística determinada por ANOVA de un solo factor con comparación por pares de Tukey y (E) duración en astrocitos FXS derivados de hiPSC. Significación estadística determinada por la prueba de Kruskal-Wallis con la prueba de comparación múltiple de Dunn; conjunto de datos representados como rangos medios con SEM. (F) Representación gráfica de la frecuencia transitoria de Ca2+ en astrocitos FXS derivados de hiPSC. Análisis estadístico por ANOVA de un solo factor con comparación por pares de Tukey. Para todos los experimentos, N = 3 réplicas biológicas; n = 19 celdas para cada línea celular. *p < 0,05, **p < 0,01, ***p < 0,001. Las barras de error representan SEM. # = Número. Abreviaturas: hiPSCs = células madre pluripotentes inducidas humanas; CTRL = Control; FXS = Síndrome del X frágil; GFAP = proteína ácida fibrilar glial. Haga clic aquí para ver una versión más grande de esta figura.
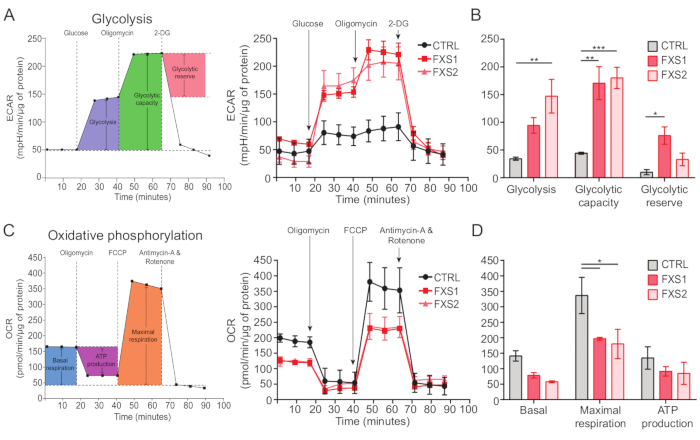
Figura 5: Déficits bioenergéticos en astrocitos FXS derivados de hiPSC. (A) Gráfico lineal que representa la tasa de acidificación extracelular de los astrocitos derivados de hiPSC (CTRL y FXS) después de la adición de 10 mM de glucosa, 1 μM de oligomicina y 50 mM de 2-DG secuencialmente y trazado en un gráfico de líneas. (B) La glucólisis, la capacidad glucolítica y la cuantificación de la reserva glucolítica mostraron un aumento en los astrocitos FXS derivados de hiPSC. Análisis estadístico realizado por ANOVA de dos factores con comparación por pares de Tukey. (C) Gráfico lineal que representa la medición de la tasa de consumo de oxígeno mediante la adición secuencial de 1,5 μM de oligomicina, 1 μM de FCCP y 0,5 μM de antimicina A y rotenona. (D) La respiración basal, la respiración máxima y la producción de ATP se cuantificaron a partir del gráfico lineal y los astrocitos FXS derivados de hiPSC mostraron una disminución significativa en la respiración máxima en comparación con los astrocitos CTRL. Análisis estadístico realizado por ANOVA de dos factores con comparación por pares de Tukey. Para todos los experimentos, N = 2 réplicas biológicas. *p < 0,05, **p < 0,01, ***p < 0,001. Las barras de error representan SEM. Abreviaturas: hiPSCs = células madre pluripotentes inducidas humanas; CTRL = Control; FXS = Síndrome del X frágil; ECAR = tasa de acidificación extracelular; 2-DG = 2-desoxiglucosa; OCR = tasa de consumo de oxígeno; FCCP = cianuro de carbonilo-4 (trifluorometoxi) fenilhidrazona. Haga clic aquí para ver una versión más grande de esta figura.
| ID en el manuscrito | ID en origen | Edad | Sexo | Nombre de línea celular reprogramada | Método de reprogramación | Tipo de celda de inicio | Cariotipo de banda G |
| (años) | |||||||
| CTRL | ND30625 | 76 | M | CS25iCTR-18nxx | Vectores episomales | Fibroblasto | Normal |
| FXS1 | GM07072 | 22 | M | CS072iFXS-n4 | Vectores episomales | Fibroblasto | Normal |
| FXS2 | GM05848 | 4 | M | CS848iFXS-n5 | Vectores episomales | Fibroblasto | Normal |
Tabla 1: Líneas celulares utilizadas en este estudiopara generar astrocitos específicos del prosencéfalo.
Figura suplementaria S1: Informe de análisis cromosómico. Cariotipo de bandas GTG normales de (A) CTRL 46, XY, (B) FXS1 46, XY y (C) FXS2 46, XY. Haga clic aquí para descargar esta figura.
Tabla suplementaria S1: Primers utilizados en este estudio para la caracterización de células. Haga clic aquí para descargar esta tabla.
Discusión
Aquí, describimos un método para generar astrocitos humanos derivados de iPSC que sirven como plataforma de ensayo para caracterizar los cambios funcionales inducidos por el FXS. Estos astrocitos son funcionalmente viables en cultivo y exhiben varias propiedades, como lo demuestran una serie de mediciones realizadas en el presente estudio. Un paso crítico en este protocolo es la conversión inicial de iPSCs en corticosferas utilizando el método de levantamiento enzimático. En esta etapa, es crucial optimizar el tiempo de incubación para la colagenasa tipo IV y la dispasa. Si esto no se optimiza, puede hacer que las iPSC se desintegren o formen células individuales, lo que evitará la formación de corticosferas. Estas corticosferas se modelan hacia la especificidad del prosencéfalo utilizando la inhibición dual de SMAD15 y se diferencian aún más en células progenitoras de astrocitos (APC). Para lograr una alta población de APC, el corte mecánico de las gliosferas es crucial. La qPCR en tiempo real se puede utilizar para confirmar la especificidad del prosencéfalo; observamos un aumento significativo del cambio de pliegue en FOXG1, un marcador para el prosencéfalo, en comparación con HOXB4, un marcador para la médula espinal.
A continuación, las APC se diferencian en astrocitos utilizando CNTF, un activador de la vía de señalización JAK-STAT16. Los resultados de este estudio indican que los astrocitos terminalmente diferenciados derivados de las iPSC del FXS tienen niveles más bajos de GFAP, lo que sugiere una madurez alterada. Aunque nuestros análisis no encontraron ninguna diferencia en la síntesis de proteínas de novo en los astrocitos y APC del FXS en comparación con los controles, encontramos una reducción en la síntesis de proteínas de novo en la transición de las APC a los astrocitos tanto en el SXF como en las líneas de control. La disminución significativa en la traducción entre APCs y astrocitos es consistente con una traducción más simplificada y curada que se ha demostrado para las neuronas frente a las células precursoras neurales17. Trabajos anteriores que han examinado la síntesis de proteínas aberrantes han utilizado tejidos cerebrales completos, neuronas y fibroblastos derivados de pacientes, no astrocitos o APC18. Los únicos reportes publicados sobre la síntesis de proteínas a partir de líneas derivadas de pacientes no se encuentran en astrocitos, sino en células linfoblastoides19 y fibroblastos 20,21. A la luz de estas variaciones entre los estudios, los estudios futuros se beneficiarían del uso de mediciones de FUNCAT en astrocitos derivados de líneas adicionales derivadas de pacientes con FXS.
Se evaluó la funcionalidad de los astrocitos midiendo la dinámica del calcio intracelular22 y la bioenergética. Los astrocitos FXS mostraron una reducción de la amplitud y duración de los picos transitorios de calcio inducidos por ATP, con menos células que respondieron al ATP en comparación con los astrocitos de control. En conjunto, estas alteraciones sugieren que el SXF altera la homeostasis del calcio en los astrocitos humanos. Estos resultados son consistentes con un estudio previo23,24 que reportó una alteración de la actividad del receptor IP3 en fibroblastos FXS. Además, observamos un aumento de la glucólisis, la capacidad glucolítica y la reserva glucolítica en los astrocitos FXS, junto con una reducción de las tasas de consumo de oxígeno mitocondrial.
El presente estudio describe la generación y caracterización de un nuevo modelo in vitro para astrocitos FXS mediante el aprovechamiento de la potencia de las iPSC derivadas de pacientes. En particular, es solo a través de un estudio anterior utilizando astrocitos humanos co-cultivados con neuronas humanas que descubrimos el papel crítico de los astrocitos en la determinación del fenotipo electrofisiológicode las neuronas. Esos estudios, sin embargo, se centraron casi por completo en los cambios inducidos por la enfermedad en la actividad neuronal, y no se exploró nada en los astrocitos. Aquí, este protocolo ofrece un marco novedoso para cambiar nuestro enfoque a las alteraciones funcionales en los astrocitos. Esto, a su vez, arrojará luz sobre las interacciones astrocito-neurona previamente inexploradas, como la señalización del glutamato y las respuestas eléctricas. Nuestros resultados en esta etapa enfatizan la necesidad de priorizar los astrocitos en futuras investigaciones.
Es fundamental reconocer las limitaciones de este protocolo. En primer lugar, implica el corte mecánico, que también conduce a la fragmentación del ADN. Esto puede hacer que las gliosferas cortadas se agrupen, lo que finalmente conduce a la muerte celular. En segundo lugar, la densidad de siembra de APCs durante la etapa de diferenciación terminal a astrocitos es esencial para prevenir la inhibición célula entre células mediada por contacto. Se recomienda optimizar esto a través de las líneas celulares para evitar el desprendimiento de la superficie del recipiente de cultivo.
Aquí, nuestros hallazgos son los primeros análisis de este tipo en astrocitos humanos, ya que todos los estudios anteriores se basaron en otros tipos de células, como las líneas celulares linfoblastoides y los fibroblastos humanos25. El metabolismo energético disfuncional es un actor conocido en la etiología de los trastornos del espectro autista, como lo demuestra el aumento de los niveles de lactato plasmático humano, la tasa de consumo de oxígeno significativamente menor en los granulocitos26 y la reducción de la expresión de los genes de fosforilación oxidativa mitocondrial en el cíngulo anterior, la corteza motora, el tálamo y el cerebelo de los niños con TEA27. Se observan cambios similares en el metabolismo energético en trastornos neurodegenerativos como la enfermedad de Parkinson28 y la enfermedad de Alzheimer29, lo que sugiere que el aumento de la glucólisis puede compensar la reducción de la función funcional30. Se necesitan estudios futuros para explorar si hay mecanismos similares en el metabolismo energético disfuncional observado en los astrocitos derivados de pacientes con SXF.
En la actualidad, la generación de astrocitos específicos del prosencéfalo es un logro significativo, pero los avances futuros podrían permitir la creación de glía específica de la región, como los astrocitos corticales/hipocampales. Esto proporcionaría una visión más profunda de los distintos roles que desempeñan estos astrocitos en las funciones cerebrales regionales, la plasticidad sináptica y las interacciones neuronal-gliales. La capacidad de generar estos astrocitos específicos de la región podría avanzar en modelos de neurodesarrollo y enfermedades neurodegenerativas. En última instancia, esto podría conducir a estrategias terapéuticas más precisas dirigidas a la disfunción de los astrocitos en regiones específicas del cerebro.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos al Prof. Sumantra Chattarji por los fondos internos. Agradecemos al Prof. Gaiti Hasan por el acceso a la configuración de imágenes de calcio, al Centro Central de Imágenes y Flujo-Centro Nacional de Ciencias Biológicas, a Padmanabh Singh y Prangya Hota por la revisión y las sugerencias, y al equipo de Labmate Asia por su ayuda en la realización de los ensayos Seahorse XF.
Materiales
| Name | Company | Catalog Number | Comments |
| 1-Thioglycerol | Sigma-Aldrich | M6145 | |
| Accutase solution | Sigma-Aldrich | A6964 | Enzyme cell detachment medium |
| Adenosine 5′-triphosphate magnesium salt | Sigma-Aldrich | A9187 | |
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634010 | |
| Antibiotic-Antimycotic (100x) | ThermoFisher Scientific | 15240062 | |
| B-27 Supplement (50x), serum-free | ThermoFisher Scientific | 17504044 | |
| Bovine Serum Albumin | Sigma-Aldrich | A9418 | |
| Chemically Defined Lipid Concentrate | ThermoFisher Scientific | 11905031 | |
| Collagenase, Type IV, powder | ThermoFisher Scientific | 17104019 | |
| Deoxyribonuclease I | Worthington Biochemical Corporation | LK003170 | |
| Dimethyl sulfoxide | Sigma-Aldrich | D2650 | |
| Dispase II, powder | ThermoFisher Scientific | 17105041 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| Essential 8 Medium | ThermoFisher Scientific | A1517001 | |
| FXS1, FXS2 | Coriell Institute of Medical Research | GM07072, GM05848 | FXS patient cells |
| GlutaMAX Supplement | ThermoFisher Scientific | 35050061 | glutamine substitute |
| Ham's F-12 Nutrient Mix | ThermoFisher Scientific | 11765054 | |
| Healthy control cells | Cedars-Sinai Medical Center | ND30625 | healthy control cells |
| Heparin sodium salt from porcine intestinal mucosa | Sigma-Aldrich | H3149 | |
| IMDM | ThermoFisher Scientific | 12440053 | |
| Insulin, human | Roche | 11376497001 | |
| LDN 193189 | Stratech Scientific | S2618-SEL | |
| Leukemia Inhibitory Factor human | Sigma-Aldrich | L5283 | |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | Corning | 354230 | |
| MEM Non-Essential Amino Acids Solution (100x) | ThermoFisher Scientific | 11140050 | |
| Mouse FGF-basic (FGF-2/bFGF) Recombinant Protein | Peprotech | 450-33 | |
| Mr. Frosty freezing container | ThermoFisher Scientific | 5100-0001 | cryobox |
| N-2 Supplement (100x) | ThermoFisher Scientific | 17502048 | |
| N-Acetyl-L-cysteine | Sigma-Aldrich | A9165 | |
| Neurobasal Medium | ThermoFisher Scientific | 21103049 | |
| Nunc Biobanking and Cell Culture Cryogenic Tubes | ThermoFisher Scientific | 377267 | |
| Nunc Cell-Culture Treated 6 well dish | ThermoFisher Scientific | 140675 | |
| Papain Dissociation System | Worthington Biochemical Corporation | LK003150 | |
| Penicillin-Streptomycin | ThermoFisher Scientific | 15140122 | |
| Recombinant Human CNTF Protein, CF | R&D Systems | 257-NT-010 | |
| Recombinant Human EGF Protein, CF | R&D Systems | 236-EG-01M | |
| RevitaCell Supplement (100x) | ThermoFisher Scientific | A2644501 | |
| SB431542 | Tocris | 1614 | |
| Seahorse XFe24 Analyzer | Agilent Technologies | ||
| Seahorse XF Cell Mito Stress Test Kit | Agilent Technologies | 103015-100 | |
| Seahorse XF Glycolysis Stress Test Kit | Agilent Technologies | 103020-100 | |
| Tissue Culture Dishes-100 cm | Biostar Lifetech | TCD000100 | |
| Transferrin | Roche | 10652202001 | |
| VWR Razor Blades | VWR International | 55411-050 | |
| Antibodies | |||
| Primary antibody | Company | Catalog number | Dilution |
| Oct4 (C-10) | Santa Cruz Biotechnology | sc-5279 | Dilution: 1:250 Secondary antibody: Goat anti-Mouse IgG, Alexa Fluor 568 |
| Nanog | R & D Systems | AF1997 | Dilution: 1:100 Secondary antibody: Donkey anti-Goat IgG, Alexa Fluor 488 |
| Vimentin | Abcam | Ab5733 | Dilution: 1:500 Secondary antibody: Goat anti-Chicken IgY, Alexa Fluor 488 |
| NFIA | Abcam | Ab41851 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 568 |
| GFAP-cy3 | Sigma | C9205 | Dilution: 1:500 Secondary antibody: NA |
| GFAP | DAKO | Z0334 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 568 |
| S100β | DAKO | IR504 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 488 |
| Anti-Nuclei Antibody, clone 235-1 | Merck Millipore | MAB1281 | Dilution: 1:1000 Secondary antibody: Goat anti-Mouse IgG1, Alexa Fluor 555 |
| Secondary antibodies | Dilution | ||
| Goat anti-Mouse IgG, Alexa Fluor 568 | Thermo Fisher Scientific | A11004 | 1:1000 |
| Donkey anti-Goat IgG, Alexa Fluor 488 | Thermo Fisher Scientific | A11055 | 1:1000 |
| Goat anti-Chicken IgY, Alexa Fluor 488 | Thermo Fisher Scientific | A11039 | 1:1000 |
| Goat anti-Rabbit IgG, Alexa Fluor 568 | Thermo Fisher Scientific | A11011 | 1:1000 |
| Goat anti-Rabbit IgG, Alexa Fluor 488 | Thermo Fisher Scientific | A11034 | 1:1000 |
| Goat anti-Mouse IgG1, Alexa Fluor 555 | Thermo Fisher Scientific | A21127 | 1:1000 |
Referencias
- Santoro, M. R., Bray, S. M., Warren, S. T. Molecular of fragile X syndrome: A twenty-year perspective. Annu Rev Pathol. 7, 219-245 (2012).
- Wang, H., et al. Developmentally-programmed FMRP expression in oligodendrocytes: A potential role of FMRP in regulating translation in oligodendroglia progenitors. Hum Mol Genet. 13 (1), 79-89 (2004).
- Pacey, L. K. K., Doering, L. C. Developmental expression of FMRP in the astrocyte lineage: Implications for fragile X syndrome. Glia. 55 (15), 1601-1609 (2007).
- Zhang, Y., et al. Purification and characterization of progenitor and mature human astrocytes reveals transcriptional and functional differences with mouse. Neuron. 89 (1), 37-53 (2016).
- Allen, N. J., Barres, B. A. Neuroscience: Glia - more than just brain glue. Nature. 457 (7230), 675-677 (2009).
- Khakh, B. S., McCarthy, K. D. Astrocyte calcium signaling: From observations to functions and the challenges therein. Cold Spring Harb Perspect Biol. 7 (4), a020404(2015).
- Allen, N. J., et al. Astrocyte glypicans 4 and 6 promote formation of excitatory synapses via GluA1 AMPA receptors. Nature. 486 (7403), 410-414 (2012).
- Chung, K., et al. Structural and molecular interrogation of intact biological systems. Nature. 497 (7449), 332-337 (2013).
- Rothstein, J. D., et al. Knockout of glutamate transporters reveals a major role for astroglial transport in excitotoxicity and clearance of glutamate. Neuron. 16 (3), 675-686 (1996).
- Almad, A., Maragakis, N. J. A stocked toolbox for understanding the role of astrocytes in disease. Nat Rev Neurol. 14 (6), 351-362 (2018).
- Zhang, Y., Barres, B. A. Astrocyte heterogeneity: An underappreciated topic in neurobiology. Curr Opin Neurobiol. 20 (5), 588-594 (2010).
- Clarke, L. E., Barres, B. A. Emerging roles of astrocytes in neural circuit development. Nat Rev Neurosci. 14 (5), 311-321 (2013).
- Bradley, R. A., et al. Regionally specified human pluripotent stem cell-derived astrocytes exhibit different molecular signatures and functional properties. Development. 146 (13), dev170910(2019).
- Das Sharma, S., et al. Astrocytes mediate cell non-autonomous correction of aberrant firing in human FXS neurons. Cell Rep. 42 (4), 112344(2023).
- Chambers, S. M., et al. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Hu, X., et al. The JAK/STAT signaling pathway: from bench to clinic. Sig Transduct Target Ther. 6, 402(2021).
- Baser, A., et al. Onset of differentiation is post-transcriptionally controlled in adult neural stem cells. Nature. 566 (7742), 100-104 (2019).
- Pal, R., Bhattacharya, A. Modelling protein synthesis as a biomarker in fragile x syndrome patient-derived cells. Brain Sci. 9 (3), 1-12 (2019).
- Gross, C., Bassell, G. J. Excess protein synthesis in FXS patient lymphoblastoid cells can be rescued with a p110β-selective inhibitor. J Mol Med. 18 (3), 336-345 (2012).
- Kumari, D., et al. Identification of fragile X syndrome specific molecular markers in human fibroblasts: A useful model to test the efficacy of therapeutic drugs. Hum Mutat. 35 (12), 1485-1494 (2014).
- Jacquemont, S., et al. Protein synthesis levels are increased in a subset of individuals with fragile X syndrome. Hum Mol Genet. 27 (12), 2039-2051 (2018).
- Bowser, D. N., Khakh, B. S. ATP excites interneurons and astrocytes to increase synaptic inhibition in neuronal networks. J Neurosci. 24 (39), 8606-8620 (2004).
- Schmunk, G., Boubion, B. J., Smith, I. F., Parker, I., Gargus, J. J. Shared functional defect in IP3R-mediated calcium signaling in diverse monogenic autism syndromes. Transl Psychiatry. 5 (9), e643-e710 (2015).
- Peteri, U. K., et al. Generation of the human pluripotent stem-cell-derived astrocyte model with forebrain identity. Brain Sci. 11 (2), 209(2021).
- Alvarez-Mora, M. I., et al. Impaired mitochondrial function and dynamics in the pathogenesis of FXTAS. Mol Neurobiol. 54 (9), 6896-6902 (2017).
- Giulivi, C., et al. Mitochondrial dysfunction in autism. JAMA. 304 (21), 2389-2396 (2010).
- Anitha, A., et al. Brain region-specific altered expression and association of mitochondria-related genes in autism. Mol Autism. 3 (1), 12(2012).
- Teves, J. M. Y., et al. Parkinson's disease skin fibroblasts display signature alterations in growth, redox homeostasis, mitochondrial function, and autophagy. Front Neurosci. 11, 737(2018).
- Yao, J., et al. Mitochondrial bioenergetic deficit precedes Alzheimer's pathology in female mouse model of Alzheimer's disease. Proc Natl Acad Sci USA. 106 (34), 14670-14675 (2009).
- Sonntag, K. C., et al. Late-onset Alzheimer's disease is associated with inherent changes in bioenergetics profiles. Sci Rep. 7 (1), 14038(2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados