Method Article
Generierung und Charakterisierung von humanen induzierten pluripotenten Stammzell-abgeleiteten Astrozyten, denen das fragile X-Boten-Ribonukleoprotein fehlt
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein Protokoll, das die Differenzierung von humanen induzierten pluripotenten Stammzellen in funktionelle Vorderhirn-spezifische Astrozyten erleichtert. Dies ermöglicht die Untersuchung der Rolle von Gliazellen bei der Pathogenese von neurologischen Entwicklungsstörungen, wie dem Fragile-X-Syndrom, und die Modellierung anderer Hirnerkrankungen.
Zusammenfassung
Das Fragile-X-Syndrom (FXS), eine der Hauptursachen für Autismus-Spektrum-Störungen und geistige Behinderungen, wurde ausgiebig anhand von Nagetiermodellen untersucht. In jüngerer Zeit wurden auch von menschlichen Stammzellen abgeleitete Modellsysteme verwendet, um mechanistische Einblicke in die Pathophysiologie des FXS zu gewinnen. Diese Studien haben sich jedoch fast ausschließlich auf Neuronen konzentriert. Trotz zunehmender Beweise für eine Schlüsselrolle von Gliazellen bei der neuronalen Funktion bei Gesundheit und Krankheit ist wenig darüber bekannt, wie menschliche Astrozyten von FXS beeinflusst werden.
Aus diesem Grund haben wir in dieser Studie erfolgreich ein Protokoll entwickelt, das wichtige raumzeitliche Meilensteine der Gehirnentwicklung erfasst und auch mit dem Prozess der Gliogenese übereinstimmt. Zusammen bietet dies einen nützlichen Rahmen für die Untersuchung von neurologischen Entwicklungsstörungen. Zunächst haben wir die humanen induzierten pluripotenten Stammzellen in die neuroektodermale Linie mit dualem Suppressor of Mothers against Decapentaplegic (SMAD) Hemmung und kleinen Molekülen eingeordnet. Anschließend verwendeten wir spezifische Wachstumsfaktoren und Zytokine, um von Patienten stammende astrozytäre Vorläuferzellen (APCs) und FXS-Patienten zu erzeugen. Die Behandlung von APCs mit dem ziliären neurotrophen Faktor, einem differenzierenden Zytokin, regulierte und trieb die Vorläuferzellen zur astrozytären Reifung, was zu Vorderhirn-spezifischen Gliafibrillären, sauren Protein-exprimierenden Astrozyten führte.
Wir fanden heraus, dass diese Astrozyten funktionsfähig sind, was durch ihre Kalziumreaktionen auf die ATP-Anwendung belegt wird, und dass sie einen dysregulierten glykolytischen und mitochondrialen Stoffwechsel bei FXS aufweisen. Zusammengenommen bieten diese Ergebnisse eine nützliche experimentelle Plattform menschlichen Ursprungs für die Untersuchung zellautonomer und nicht-zellautonomer Folgen von Veränderungen der astrozytären Funktion, die durch neurologische Entwicklungsstörungen verursacht werden.
Einleitung
Das Fragile-X-Syndrom (FXS), eine häufige erbliche Form der geistigen Behinderung und der Autismus-Spektrum-Störung (ASS), wird durch den Mangel an fragilem X-Boten-Ribonukleoprotein (FMRP) verursacht, das vom fragilen X-Boten-Ribonukleoprotein 1 (FMR1)-Gen produziert wird (OMIM: #300624, https://www.omim.org/entry/300624). FMRP spielt eine Rolle bei der Regulation der mRNA-Translation, der mRNA-Granulierung und des mRNA-Transports sowie der microRNA-vermittelten Regulation der Genexpression1. Der Verlust von FMRP wirkt sich also nicht nur auf die Gehirnentwicklung, sondern auch auf die Gehirnfunktion von Erwachsenen aus. Sowohl die mRNA-Transkriptspiegel von FMR1 als auch die Immunfärbung für FMRP im Gehirn haben eine hohe neuronale Expression gezeigt, neben einer signifikanten Expression auch in Gliazellen2. Die überwiegende Mehrheit früherer Studien an Tiermodellen von FXS konzentrierte sich jedoch hauptsächlich auf Neuronen und Aberrationen in ihrer Funktion. Folglich ist wenig über die Rolle der Gliazellen bei FXS3 bekannt. Traditionellals "passive Unterstützungszellen" angesehen 4, gibt es immer mehr Hinweise darauf, dass Astrozyten bei der Vermittlung einer Vielzahl von neuronalen Funktionen entscheidend sind 5,6, einschließlich der Förderung der Synaptogenese7, der Verfeinerung der Entwicklung neuronaler Schaltkreise8 und des Neurotransmitterrecyclings9. Parallel dazu gibt es immer mehr Beweise für die Rolle von Astrozyten bei der Pathogenese von Krankheiten, und viele neurologische Erkrankungen wurden mit astrozytärer Dysfunktion in Verbindung gebracht10.
Während sich ein Großteil der früheren Arbeiten mit Tiermodellen von FXS auf die Identifizierung und Validierung verschiedener molekularer Ziele in Neuronen zur Behandlung von FXS konzentrierte, haben diese präklinischen Befunde nicht immer zu erfolgreichen klinischen Ergebnissen geführt. Darüber hinaus unterstreichen auch Rückschläge in den jüngsten klinischen Studien die Notwendigkeit von humanbasierten Modellsystemen. Modelle neurologischer Erkrankungen, die auf aus menschlichen Stammzellen gewonnenen Gehirnzellen basieren, bieten eine leistungsstarke Strategie, um diese Lücke zwischen mechanistischen Erkenntnissen aus Tierversuchen und begrenztem Erfolg mit klinischen Ergebnissen für Patienten zu schließen. Allerdings hat sich nur eine Handvoll dieser Studien auf Astrozyten konzentriert, und zwar hauptsächlich auf Astrozyten, die spinalen Ursprungs sind. Dies wiederum ist relevant im Lichte von Studien, die zeigen, dass die Struktur und Funktion von Astrozyten zwischen den Hirnregionen variiert11,12. Um krankheitsinduzierte Veränderungen in menschlichen Astrozyten besser zu verstehen, müssen daher auch diese hirnregionenspezifischen Unterschiede in Astrozyten berücksichtigt werden. Modelle für neurologische Entwicklungsstörungen mit aus menschlichen Stammzellen gewonnenen Astrozyten, die spezifisch für das Vorderhirn sind, sind jedoch noch vergleichsweise wenig erforscht13. Um diese Lücken zu schließen, beschreiben wir daher Protokolle zur Generierung von Vorderhirn-spezifischen Astrozyten aus patienteneigenen induzierten pluripotenten Stammzellen (iPSCs), die FXS-Mutationen tragen; Darüber hinaus zeigen wir, dass Astrozyten funktionsfähig sind und einen veränderten Stoffwechsel aufweisen.
Protokoll
Alle Experimente mit humanen induzierten pluripotenten Stammzellen (hiPSCs) (Tabelle 1) wurden nach Erhalt der entsprechenden institutionellen behördlichen Genehmigungen durchgeführt. Abbildung 1A zeigt das vollständige Differenzierungsprotokoll von hiPSCs zu reifen Vorderhirn-spezifischen Astrozyten.
1. Erhaltung und Ausbau von hiPSCs
- Einen Tag vor dem Plattieren der iPSCs eine 6-Well-Schale mit 1:60 verdünntem Matrigel (einer extrazellulären Matrix [ECM]) in Advanced Dulbecco's Modified Eagle Medium/Ham's F-12 (DMEM/F12) beschichten und bei 2-8 °C lagern.
HINWEIS: Vermeiden Sie es, mit Matrigel beschichtete Gerichte >5 Tage bei 2-8 °C zu lagern, da dies im Laufe der Zeit zu einem Proteinabbau in der EZM führen kann. Um ein Austrocknen der Schüssel zu vermeiden, beschichten Sie mindestens 1 ml 1:60 verdünntes Matrigel pro Mulde einer 6-Well-Schale und verteilen Sie sie gleichmäßig auf der Schüssel. Wenn Sie die Platte am selben Tag verwenden, bewahren Sie sie 1 h lang in einem befeuchteten Inkubator bei 37 °C und 5 % CO2 auf. - Am Tag der Kultivierung der hiPSCs entfernen Sie das Beschichtungsmaterial und fügen Sie 1 ml vollständiges Essential 8 Medium (E8 Basalmedium mit Essential 8 Supplement) zusammen mit dem ROCK Inhibitorat 1x Endkonzentration hinzu und bewahren Sie die Schale in einem 37 °C und 5 % CO2 -Inkubator auf, bevor Sie die hiPSCs hinzufügen.
- Resuspendieren Sie die hiPSCs in vollständigem E8-Medium zusammen mit dem ROCK-Inhibitor in 1x Endkonzentration, um eine bessere Adhärenz der Kolonien zu gewährleisten.
HINWEIS: Bereiten Sie keine großen Mengen an Nährmedium vor. Nur maximal 3-4 Tage zubereiten und bei 2-8 °C lagern. - Am nächsten Tag füllen Sie das gesamte E8-Medium ohne den ROCK-Inhibitor auf, bis die Zellen zu 80 % konfluent sind (ca. 4-5 Tage).
- Wenn sie eine Konfluenz von 80 % erreichen, lösen Sie die Kolonien enzymatisch ab.
- Um die iPS-Zellen zu passieren, nehmen Sie das verbrauchte Medium aus der Schale und fügen Sie 1 ml pro Vertiefung einer vorgewärmten (37 °C) Mischung aus Kollagenase (2 mg/ml) und Dispase (1 mg/ml) in einem Verhältnis von 1:1 hinzu und inkubieren Sie bei 37 °C für mehr als 20-30 Minuten, damit sich die iPS-Kolonien anheben können.
HINWEIS: Lassen Sie das Geschirr nicht länger als 20-30 Minuten bei 37 °C stehen; Nach dieser Zeit werden sich die Kolonien auflösen und die Zellen sterben ab.
- Um die iPS-Zellen zu passieren, nehmen Sie das verbrauchte Medium aus der Schale und fügen Sie 1 ml pro Vertiefung einer vorgewärmten (37 °C) Mischung aus Kollagenase (2 mg/ml) und Dispase (1 mg/ml) in einem Verhältnis von 1:1 hinzu und inkubieren Sie bei 37 °C für mehr als 20-30 Minuten, damit sich die iPS-Kolonien anheben können.
- Wenn sich die Bienenvölker zu heben beginnen, nehmen Sie die Schalen aus dem Inkubator und neutralisieren Sie die Enzymaktivität, indem Sie 2 ml Dulbeccos phosphatgepufferte Kochsalzlösung (DPBS) hinzufügen.
- Kratzen Sie die Kolonien mit DPBS ab und sammeln Sie die Suspension in einem konischen 15-ml-Röhrchen mit einer serologischen 10-ml-Pipette mit breiter Bohrung.
- Zerreiben Sie die Suspension vorsichtig 2-3x, um die Kolonien mit einer serologischen 10-ml-Pipette aufzubrechen, und lassen Sie die Völker sich setzen.
HINWEIS: Machen Sie die Kolonien nicht zu einzelnen Zellen; Dies kann dazu führen, dass die Zellen nicht an der Schale haften und mehr Zelltod verursachen. Lasst sie ferner nicht als große Völker zurück; Dies wird später zu differenzierteren Kolonien führen. - Sobald sich die Kolonien beruhigt haben (ca. nach 2 Minuten), aspirieren Sie das DPBS-Enzymgemisch und lassen Sie etwa 1 ml im Röhrchen.
- Geben Sie 2 ml DPBS in das Röhrchen, mischen Sie die Kolonien durch Klopfen und lassen Sie sie absetzen. Wiederholen Sie dies 2x, um alle Enzymreste aus den Kolonien zu entfernen. Entfernen Sie nach der 2. Wäsche so viel Überstand wie möglich, resuspendieren Sie die Kolonien in 1 ml vollständigem E8-Medium und geben Sie es in eine frisch zubereitete Schüssel, wie in den Schritten 1.2-1.3 beschrieben.
- Alternativ können Sie einen Teil der Kolonien mit 10 % Dimethylsulfoxid (DMSO) als Kryoprotektivum für die zukünftige Expansion und Verwendung kryokonservieren.
- Bereiten Sie das Kryokonservierungsmedium frisch vor, indem Sie 90 % vollständiges E8-Medium und 10 % DMSO als Kryoprotektivlösung mischen und bis zur Verwendung bei 2-8 °C erhitzen.
- Um Kryokonservierung durchzuführen, befolgen Sie die Schritte 1.5-1.10.
- Lassen Sie die Völker in Schritt 1.10 nach dem 2. Waschen absetzen und entfernen Sie den Überstand. Zu den Kolonien 1 ml frisch zubereitetes kaltes Kryokonservierungsmedium geben und in Kryoröhrchen umfüllen.
HINWEIS: Nachdem Sie das Kryokonservierungsmedium zu den Kolonien hinzugefügt haben, füllen Sie den Inhalt schnell in Kryoröhrchen um. Eine Verzögerung kann zu einer geringeren Wiederbelebungsrate führen, da DMSO ein Kryoprotektivum ist, das Zellen schädigen kann. - Stellen Sie die Kryofläschchen sofort in eine Kryobox und bewahren Sie sie über Nacht in einem -80 °C Gefrierschrank auf.
- Lagern Sie am nächsten Tag alle Kryoröhrchen für die zukünftige Verwendung in einen Flüssigstickstofftank (LN2-Tank ).
HINWEIS: hiPS-Zellen sollten routinemäßig mittels G-Banding (Ergänzende Abbildung S1) auf Anomalien karyotypisiert, mittels Immunzytochemie auf Pluripotenz charakterisiert (Abbildung 1B) und auf Mykoplasmen getestet werden.- Um hiPSCs mittels Immunzytochemie zu charakterisieren, plattieren Sie die Kolonien auf 1:60 Matrigel-beschichteten, autoklavierten 13-mm-Glasdeckgläsern. Sobald sie zu 40 % konfluent sind, waschen Sie die Zellen mit PBS-T (PBS-0,1 % Tween 20), fixieren Sie sie 10 Minuten lang mit 4 % Paraformaldehyd, permeabilisieren Sie 10 Minuten lang mit 0,3 % Triton X-100 in PBS und blockieren Sie 1 h lang mit 3 % Rinderserumalbumin (BSA) in PBS, um eine unspezifische Bindung zu verhindern.
- Nach dem Blocken die Kulturen mit Primärantikörpern für 1 h inkubieren und anschließend 3x mit PBS-T für jeweils 5 min waschen, anschließend entsprechende Sekundärantikörper (Table of Materials) im Dunkeln für 1 h waschen.
- Montieren Sie die Deckgläser mit Eindeckmedium auf Objektträger und nehmen Sie Bilder durch konfokales Laserscanning bei 405 nm, 488 nm, 561 nm und 633 nm auf. Nehmen Sie Bilder mit 512 x 512 Pixeln auf; Stellen Sie die Z-Schrittweite auf 0,5 μm mit 1 luftigen Einheit Lochblendendurchmesser ein.
2. Erzeugung und Charakterisierung astrozytärer Vorläuferzellen (APCs)
- Heben Sie die hiPSCs enzymatisch an, wie in den Schritten 1.5-1.10 erwähnt, und geben Sie sie auf eine nicht adhärente Suspensionskulturschale (100 mm) mit chemisch definiertem Medium14, das 50 % Iscove's Modified Dulbecco's Medium (IMDM), 50 % Ham's F-12 Nutrient Mix (F12), 5 mg/mL BSA, 1 % chemisch definiertes Lipidkonzentrat (CD-Lipid), 450 μM Monothioglycerin, 7 μg/mL Insulin, 15 μg/mL Transferrin enthält. 1% Penicillin-Streptomycin, ergänzt mit den Mitogenen N-Acetylcystein (1 mM), LDN 193189 (0,1 μM) und SB431542 (10 μM) für 7 Tage.
HINWEIS: Die niedermolekularen Inhibitoren SB431542 und LDN-193189 (LDN) sind Inhibitoren des knochenmorphogenetischen Proteins und der transformierenden Wachstumsfaktor-beta-Signalwege.
Von diesem Zeitpunkt an wurde das Medium einmal in 2 Tagen oder nach dem Montag/Mittwoch/Freitag-Protokoll aufgefüllt. - Legen Sie die Zellsuspension 7 Tage lang unter normoxischen Bedingungen bei 40 U/min auf einen Orbitalschüttler, um die Entwicklung von Kortikosphären zu unterstützen (Abbildung 1A).
- Übertragen Sie die Kortikosphären an Tag 8 7 Tage lang in ein Zellproliferationsmedium, das Advanced DMEM/F12 mit 1 % Antibiotikum-Antimykotikum, 1 % N2-Supplement, 1 % Glutaminersatz, 0,1 % B27-Supplement und 2,5 ng/ml basischem Fibroblasten-Wachstumsfaktor (bFGF) enthält.
- Induzieren Sie die Kugeln für die Gliaspezifikation, indem Sie sie 2 Wochen lang einem Gliaanreicherungsmedium aussetzen, das Advanced DMEM/F12 mit 1 % Antibiotikum-Antimykotikum, 1 % N2, 1 % Glutaminersatz, 0,1 % B27-Supplement, 20 ng/ml epidermalem Wachstumsfaktor (EGF), bFGF-H (20 ng/ml bFGF-5 mg/ml Heparin) enthält, um frühe Gliosphären zu erhalten (Abbildung 1A).
- Für die Reifung der frühen Gliosphären ersetzen Sie bFGF-H durch 20 ng/ml Leukämie-Hemmfaktor (LIF) und halten Sie die Kugeln 4 Wochen lang.
- Nach 4 Wochen im Reifemedium halten Sie die Kugeln über einen längeren Zeitraum im Gliaanreicherungsmedium. Um eine Aggregation und einen Verlust der Lebensfähigkeit zu verhindern, werden die Gliosphären alle 2 Wochen mit einer sterilen Industrieklinge mechanisch zerkleinert und das gesamte Medium durch DNase I ersetzt, um DNA-Fragmente zu entfernen, die beim Zerkleinern entstehen.
- Dissoziieren Sie die Gliosphären mit einem Papain-Dissoziationskit in Monoschichten von APCs und plattieren Sie sie mit einer 1:80-Verdünnung der Matrigel-Beschichtung auf eine mit Zellkulturen behandelte Adhärentschale.
- Vermehren Sie die APCs in glialem Anreicherungsmedium, bis sie zu 80 % konfluiert sind, und passagen Sie sie enzymatisch mit dem Enzymzellablösemedium (siehe Materialtabelle).
- Zur Passage:
- Entfernen Sie das verbrauchte Medium und sammeln Sie es in einem konischen Rohr. Geben Sie das Enzym Zellablösungsmedium in die Zellen und warten Sie 1-2 min. Sobald sich die Zellen abzulösen beginnen, fügen Sie das verbrauchte Medium hinzu, um die Enzymaktivität zu neutralisieren.
- Sammeln Sie die Zellsuspension und zentrifugieren Sie sie 2 Minuten lang bei 800 x ɡ .
- Aspirieren Sie den Überstand, resuspendieren Sie die Zellen in glialem Anreicherungsmedium und geben Sie ca. 1 x 106 Zellen/Vertiefung in eine 1:80 Matrigel-beschichtete 6-Well-Schale.
- Für die Kryokonservierung:
- Resuspendieren Sie die Zellen in einer kalten Mischung aus 90 % Kortikosphären-Proliferationsmedium (ohne bFGF) und 10 % Kryoprotektivum. Übertragen Sie die resuspendierten Zellen in Kryofläschchen.
- Stellen Sie die Kryofläschchen sofort in eine Kryobox und bewahren Sie sie über Nacht im -80 °C Gefrierschrank auf.
- Lagern Sie am nächsten Tag alle Kryoröhrchen für die zukünftige Verwendung in einen Flüssigstickstofftank (LN2-Tank ).
HINWEIS: APCs wurden mit 10 % DMSO bis zur Passage Nummer 6 kryokonserviert.
- Charakterisieren Sie die dissoziierten APCs durch Immunfärbung (wie in den Schritten 1.15.1-1.15.3 beschrieben) mit Vimentin- und Nuclear Factor IA (NFIA)-Markern (Abbildung 1C,D) (siehe Materialtabelle für Verdünnungen).
- Um die regionale Spezifität von hiPSC-abgeleiteten APCs im Vorderhirn zu bestätigen, testen Sie die Zellen mittels Echtzeit-qPCR auf einen Vorderhirnmarker wie die humane Forkhead-Box G1 (hFOXG1) (positiv) und einen Hinterhirnmarker wie die humane Homeobox B4 (hHOXB4) (negativ) (Abbildung 1E,F).
HINWEIS: Die für diese Experimente verwendeten Primersequenzen sind in der ergänzenden Tabelle S1 aufgeführt.
3. Generierung und Charakterisierung einer homogenen Population von Vorderhirn-spezifischen Astrozyten
- Unterscheidung von Astrozyten von APCs
- Verwenden Sie 14 Tage lang das astrozytäre Differenzierungsmedium (ADM), um APCs in Astrozyten zu differenzieren. Das astrozytäre Differenzierungsmedium enthält Neurobasal, 1 % Antibiotikum-Antimykotikum, 1 % Glutaminersatz, 1 % N2-Supplement, 0,2 % B27-Supplement, 1 % nicht-essentielles Aminosäuremedium (NEAA) und 10 ng/ml ziliärer neurotropher Faktor (CNTF).
- Bestätigung der astrozytären Identität durch Immunfärbung (wie in den Schritten 1.15.1-1.15.3 erwähnt) mit saurem Gliafibrillärprotein (GFAP) und S100β-Expressionsmarkern (Abbildung 2A,B).
- De-novo-Proteinsynthese in APCs und Astrozyten
- Bewahren Sie hiPSC-abgeleitete APCs und Astrozyten des Vorderhirns auf sterilen 13-mm-Glasdeckgläsern auf und färben Sie sie immunfärblich, wie in den Schritten 1.15.1-1.15.3 beschrieben.
- Für die De-novo-Proteinsynthese verwenden Sie die FUNCAT-Methode (Fluoreszierendes nicht-kanonisches Aminosäure-Tagging), die in den Schritten 3.2.3 bis 3.2.6 kurz erläutert wird (Abbildung 3A).
- Um einen Mangel an Methionin zu gewährleisten, entfernen Sie das Wachstumsmedium aus den Kulturen und ersetzen Sie es durch methionin- und cysteinfreies Medium, gemischt mit 1 mM L-Azidohomoalanin (AHA) für 30 min bei 37 °C und 5 % CO2.
- Waschen Sie die Kulturen mit PBS-T, fixieren Sie sie 10 min lang mit 4 % Paraformaldehyd, permeabilisieren Sie sie 10 min lang mit 0,3 % Triton X-100 in PBS und blockieren Sie sie 1 h lang mit 3 % BSA in PBS, um eine unspezifische Bindung zu verhindern.
- Inkubieren Sie die Kulturen 1 h lang im Dunkeln bei Raumtemperatur mit Click-Zellchemie-Reaktionsmix und Alkin-Alexa-Fluor 647, gefolgt von Primärantikörpern für 1 h und entsprechenden Sekundärantikörpern (Materialtabelle) für 1 h (Abbildung 3B).
- Montieren Sie die Deckgläser mit Montagemedium auf Objektträger und verwenden Sie diese für die weitere Bildanalyse.
- Erfassen Sie Bilder durch konfokales Laserscanning bei 405 nm, 488 nm, 561 nm und 633 nm.
- Nehmen Sie Bilder mit 512 x 512 Pixeln auf; Stellen Sie die Z-Schrittweite auf 0,5 μm mit 1 luftigen Einheit Lochblendendurchmesser ein.
- Halten Sie die Mikroskopie- und Bildgebungsparameter für alle Zelltypen auf einer konstanten Einstellung. Erfassen Sie Bilder für jedes biologische Replikat in derselben Sitzung.
- Führen Sie Intensitätsmessungen mit einer beliebigen Standard-Bildanalysesoftware (z. B. Fiji oder Imaris) durch. Verwenden Sie in Imaris das SURFACES-Modul, um volumetrische Messungen von Zellkörpern zu erhalten, die positiv für das FUNCAT-Signal (Fluoresscent non-canonical Amino Acid Tagging) mit Vimentin (APCs) und GFAP (Astrozyten) sind, wodurch der Voxelbereich (250-350) begrenzt wird, um Ablagerungen zu entfernen.
HINWEIS: Behalten Sie die Parameter für alle biologischen Replikate für beide Zelltypen bei.
- Adenosin-5'-Triphosphat (ATP)-induzierte oszillatorische Calciumwellen
- Teller Astrozyten auf einer Glasbodenschale (35 mm) bei 5 × 103 Zellen/Schale, um die zelluläre Reaktion auf ATP zu messen.
- Lassen Sie die Zellen 24 h am Glasboden haften, waschen Sie sie 3x mit HBSS (20 mM HEPES, 137 mM NaCl, 5 mM KCl, 10 mM Glucose, 1 mM MgCl2, pH = 7,3) ohne Calcium und inkubieren Sie in Kulturmedium mit dem metrischen Farbstoff Fura-2AM mit einem Verhältnis von 5 μM und 0,02 % Pluronics F127 für 1 h bei Raumtemperatur.
HINWEIS: Fura-2AM ist eine licht- und temperaturempfindliche Chemikalie; Bei -20 °C lagern. - Nach der Inkubation 2x mit Kulturmedium waschen und durch 2 mM Ca2+ ersetzen, das HBSS enthält, und die Zellen mit einer Geschwindigkeit von 2 FPS unter Verwendung eines 60-fachen Ölobjektivs (1,35 NA) in einem inversen Mikroskop mit Fokusdrift-Kompensation abbilden.
- Aufzeichnung von ATP-induzierten Calciumreaktionen durch Badapplikation von ATP in der Endkonzentration von 5 mM bei 25μs (Abbildung 4A).
- Zeichnen Sie mit Fiji/ImageJ interessante Bereiche um jede Zelle und berechnen Sie das Verhältnis F340/F380 über alle Zeitpunkte (Abbildung 4B).
- Assays für den Zellstoffwechsel
HINWEIS: Die Sauerstoffverbrauchsrate (OCR) und die extrazelluläre Ansäuerungsrate (ECAR) lebender Zellen wurden gemäß dem Protokoll des Herstellers gemessen (siehe Materialtabelle).- Lassen Sie die Astrozyten einen Tag vor dem Assay an der Mikroplatte (1,5 × 104 Aussaatdichte) in CNTF-Medium haften und inkubieren Sie bei 37 °C mit 5 % CO2.
- Für den Glykolyse-Stress-Assay:
- Ersetzen Sie das Zellkulturmedium durch ein Basismedium, das mit 2 mM Glutamin (pH-Wert auf 7,4 eingestellt) ergänzt wurde, und inkubieren Sie in einer Nicht-CO2 -Kammer bei 37 °C für 1 h.
HINWEIS: Der pH-Wert muss in einem 37 °C warmen Wasserbad eingestellt werden. - Setzen Sie die Patronenplatte in das Gerät ein, um den Sensor zu kalibrieren. Ersetzen Sie nach der Kalibrierung die Kartuschenplatte durch die Zellkulturplatte mit einer Endkonzentration der folgenden Kit-Komponenten: 10 mM Glukose, 1 μM Oligomycin und 50 mM 2-Desoxyglukose (2-DG) (Abbildung 5A).
- Am Ende des Tests lysieren Sie die Zellen und schätzen Sie den Gesamtproteingehalt.
- Ersetzen Sie das Zellkulturmedium durch ein Basismedium, das mit 2 mM Glutamin (pH-Wert auf 7,4 eingestellt) ergänzt wurde, und inkubieren Sie in einer Nicht-CO2 -Kammer bei 37 °C für 1 h.
- Für den Mitochondrien-Stress-Assay:
- Ersetzen Sie das CNTF-Medium durch ein Basismedium, das mit 2 mM Glutamin, 1 mM Pyruvat und 10 mM Glukose (pH-Wert auf 7,4 eingestellt) in einem Wasserbad bei 37 °C ergänzt wird.
- Die Platte wird in einer Nicht-CO2 -Kammer bei 37 °C 1 h lang inkubiert. Setzen Sie in der Zwischenzeit die Patronenplatte in das Gerät ein, um den Sensor zu kalibrieren.
- Ersetzen Sie nach der Kalibrierung die Kartuschenplatte durch die Zellkulturplatte mit einer Endkonzentration der folgenden Kit-Komponenten: 1,5 μM Oligomycin, 1 μM Carbonylcyanid-4 (Trifluormethoxy)phenylhydrazon (FCCP) und 0,5 μM Rotenon/Antimycin A (Abbildung 5C).
- Am Ende des Tests lysieren Sie die Zellen und schätzen Sie den Gesamtproteingehalt.
Ergebnisse
Die Kolonien humaner induzierter pluripotenter Stammzellen (hiPSC) wurden unter Verwendung eines kommerziell erhältlichen definierten Mediums gehalten und mit den Pluripotenzmarkern Oct4 und Nanog immungefärbt (Abbildung 1B). FXS- und CTRL-APCs zeigten vergleichbare hochangereicherte Anteile von Zellen, die immunpositiv für Vimentin und NFIA waren (Abbildung 1C,D). Wir fanden heraus, dass APCs, die von gesunden und FXS-hiPSCs abgeleitet wurden, eine signifikante Hochregulation von FOXG1 im Vergleich zu HOXB4 zeigten (Abbildung 1F), was mit einer vorherrschenden Vorderhirnidentität übereinstimmt. Wir beobachteten eine signifikante Reduktion der Anzahl von GFAP-exprimierenden Astrozyten in FXS-Gruppen im Vergleich zu CTRL-Astrozyten, ohne die Anzahl der S100β-exprimierenden Astrozyten zu beeinflussen (Abbildung 2B). Die Western-Blot-Analyse zeigte, dass die Astrozyten, die aus beiden FXS-Linien erzeugt wurden, keine Expression von FMRP aufwiesen (Abbildung 2C,D). Die Quantifizierung neu synthetisierter Proteine ergab keinen signifikanten Unterschied zwischen CTRL- und FXS-Linien in APCs oder Astrozyten (Abbildung 3C). Wir fanden jedoch, dass die Proteinsynthese in APCs für jede Linie konsistent höher war als in ihren jeweiligen Astrozyten (Abbildung 3C). Diese Ergebnisse deuten auf eine stadienspezifische Reduktion der de novo Proteinsynthese von APCs zu Astrozyten sowohl in der FXS- als auch in der CTRL-Linie hin.
Sowohl die Kontroll- als auch die FXS-Astrozyten wiesen ATP-induzierte Kalziumtransienten auf (Abbildung 4A). Eine detailliertere Analyse ergab jedoch mehrere wichtige Unterschiede zwischen den einzelnen Kalziumtransienten, die von CTRL-, FXS1- und FXS2-Astrozyten aufgezeichnet wurden (Abbildung 4B). Wir klassifizierten die Astrozyten als Responder und Nicht-Responder auf ATP, basierend auf dem Vorhandensein bzw. Fehlen einer Kalziumreaktion (Abbildung 4C). Die Anzahl der Responder in FXS1 und FXS2 war signifikant niedriger als bei CTRL. Insbesondere beobachteten wir eine signifikante Verringerung der Peakamplitude des ersten Calciumtransienten, die nach ATP-Applikation in beiden FXS-Astrozytenlinien hervorgerufen wurde (Abbildung 4D). Darüber hinaus war die Gesamtdauer des ersten Calciumtransienten in den FXS1- und FXS2-Linien signifikant kürzer als in der CTRL (Abbildung 4E). Die Anzahl der Kalziumereignisse (quantifiziert als Gesamtzahl der Ereignisse pro 250 s; Abbildung 4F) war zwischen den Linien vergleichbar. Zusammengenommen zeigen diese Ergebnisse Veränderungen in den Kalziumreaktionen, die durch ATP in hiPSC-abgeleiteten FXS-Astrozyten ausgelöst werden. Die ECAR-Ergebnisse deuten auf eine höhere Glykolyserate, glykolytische Kapazität und glykolytische Reserve in hiPSC-abgeleiteten FXS-Astrozyten hin (Abbildung 5A,B). Wir fanden keinen signifikanten Unterschied in der basalen Atmung bei CTRL- und FXS-Astrozyten. Die zelluläre ATP-Produktion zeigte keinen signifikanten Unterschied zwischen CTRL- und FXS-Astrozyten. Die FCCP-Stimulation führte bei FXS-Astrozyten zu einer signifikant geringeren maximalen Atmung als bei CTRL-Astrozyten (Abbildung 5C,D).
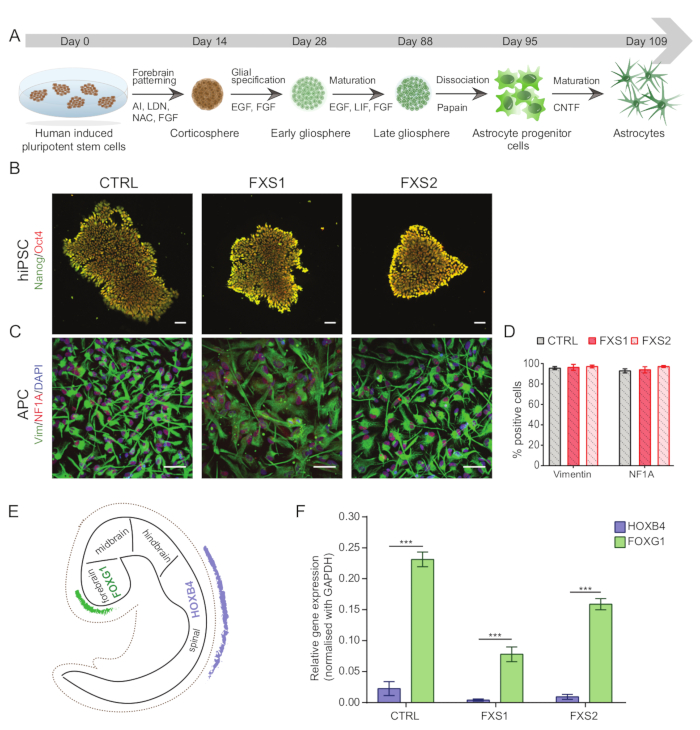
Abbildung 1: Ableitung von Vorderhirn-spezifischen astrozytären Vorläuferzellen aus hiPSCs. (A) Illustrativer Arbeitsablauf zur Generierung von Astrozyten aus hiPSCs. (B) Repräsentative Bilder von hiPSCs, die eine vergleichbare Expression von Oct4/Nanog in CTRL- und FXS-Linien zeigen. (C) Eine homogene Population von hiPSC-abgeleiteten APCs exprimierte eine ähnliche Anzahl von Vimentin- und NFIA-positiven Zellen. (D) Kein signifikanter Unterschied in der Expression von Vimentin- und NFIA zwischen CTRL- und FXS-APCs. Die statistische Analyse erfolgte unter Verwendung einer zweifaktorigen ANOVA, gefolgt von Tukeys paarweisem Vergleich. (E) Schematische Darstellung der regionenspezifischen Expression von FOXG1 und HOXB4 während der Entwicklung in vivo. (F) Grafische Darstellung der FOXG1-Expression im Vergleich zu HOXB4 (mittels qRT-PCR) bei allen Genotypen, was auf eine Neigung zur Abstammung des Vorderhirns hindeutet. Statistische Analyse durch Zwei-Faktoren-ANOVA, gefolgt von Sidaks Mehrfachvergleichstest. Für alle Experimente ist N = 3 biologische Replikate. p < 0,001. Maßstabsleiste = 50 μm. Fehlerbalken stellen REM dar. Abkürzungen: hiPSCs = humane induzierte pluripotente Stammzellen; STRG = Steuerung; FXS = Fragile-X-Syndrom; APCs = Astrozyten-Vorläuferzellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
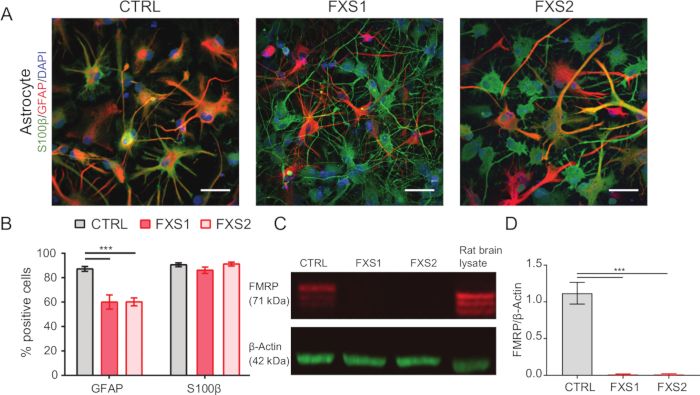
Abbildung 2: Reduzierte Anzahl von Gliafibrillen-positiven sauren Protein-positiven hiPSC-abgeleiteten FXS-Astrozyten. (A) Repräsentative Bilder von hiPSC-abgeleiteten Astrozyten mit S100β- und GFAP-Expression. (B) Der Prozentsatz der GFAP-positiven Astrozyten war in FXS-abgeleiteten Astrozyten im Vergleich zu CTRL signifikant niedriger, obwohl beide eine ähnliche S100β-Expression aufwiesen. Statistische Signifikanz, bestimmt durch zweifaktorige ANOVA, gefolgt von Tukeys paarweisem Vergleich. (C) Immunoplot, der die Expression von FMRP in Astrozyten aus CTRL und FXS darstellt. (D) Diagramme, die das Fehlen von FMRP in hiPSC-abgeleiteten FXS-Astrozyten zeigen. Statistische Analyse durch einfaktorige ANOVA mit dem paarweisen Vergleich von Tukey. Für alle oben genannten Experimente gilt: N = 3 biologische Replikate. Maßstabsleiste = 50 μm. ***p < 0,001. Fehlerbalken stellen REM dar. Abkürzungen: hiPSCs = humane induzierte pluripotente Stammzellen; STRG = Steuerung; FXS = Fragile-X-Syndrom; GFAP = saures Protein der Gliafaser; FMRP = Fragiles X Boten-Ribonukleoprotein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
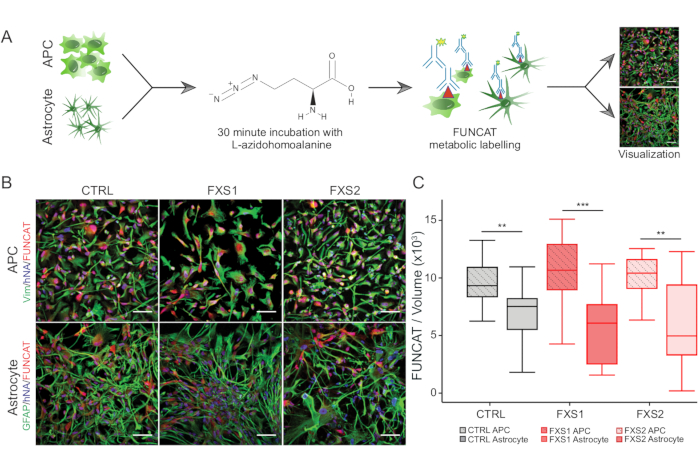
Abbildung 3: Reduzierte Proteinsynthese aufgrund der Differenzierung von APCs zu Astrozyten. (A) Schematischer Arbeitsablauf zur Darstellung der Markierung und Visualisierung der Proteinsynthese in hiPSC-abgeleiteten APCs und Astrozyten unter Verwendung der metabolischen Markierung von FUNCAT. (B) Repräsentative Bilder von (oben) Vimentin-positiven hiPSC-abgeleiteten APCs und (unten) GFAP-positiven Astrozyten mit FUNCAT/humanen Zellkern-Antikörpern (hNA)-Markierung in CTRL- und FXS-abgeleiteten Zellen. (C) FUNCAT/Volumen von hiPSC-abgeleiteten APCs war signifikant höher als bei entsprechenden abgeleiteten Astrozyten. Für alle Experimente ist N = 3 biologische Replikate. Maßstabsleiste = 50 μm. **p < 0,01, ***p < 0,001. Whisker repräsentieren 1,5 × IQR. Abkürzungen: hiPSCs = humane induzierte pluripotente Stammzellen; STRG = Steuerung; FXS = Fragile-X-Syndrom; GFAP = saures Protein der Gliafaser; FUNCAT = fluoreszierendes nicht-kanonisches Aminosäure-Tagging. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
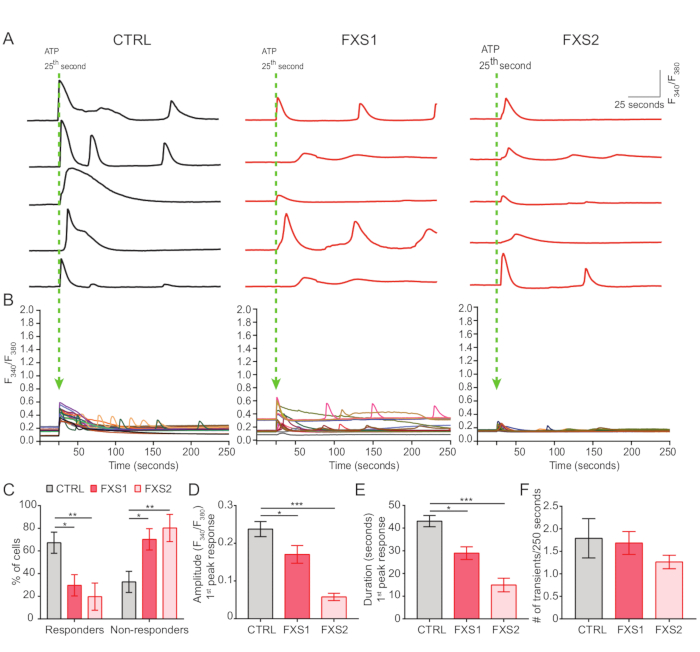
Abbildung 4: Defizienter Ca2+-Signalweg, der durch ATP in hiPSC-abgeleiteten FXS-Astrozyten hervorgerufen wird. (A) Repräsentative Spuren von Ca2+-Transienten, die von einzelnen Astrozyten bei externer Gabe von ATP bei 25μm aufgezeichnet wurden. (B) Gemittelte F340/F380-Verhältnisse, die Ca2+-Transienten über 250 s nach ATP-Applikation darstellen. (C) Gruppierte Daten, die einen höheren Prozentsatz von Non-Respondern auf ATP in hiPSC-abgeleiteten FXS-Astrozyten zeigen. Statistische Signifikanz, bestimmt durch zweifaktorige ANOVA mit dem paarweisen Vergleich nach Tukey. (D-F) Quantifizierung der ersten Peakantwort (Amplitude und Dauer) und der Häufigkeit von Ereignissen. (D) Zeigt eine signifikante Verringerung der Amplitude. Statistische Signifikanz, bestimmt durch Einzelfaktor-ANOVA mit Tukeys paarweisem Vergleich und (E) Dauer in hiPSC-abgeleiteten FXS-Astrozyten. Statistische Signifikanz, bestimmt durch Kruskal-Wallis-Test mit dem Mehrfachvergleichstest nach Dunn; Der Datensatz wird als Mittelwert mit REM dargestellt. (F) Grafische Darstellung der transienten Häufigkeit von Ca2+ in hiPSC-abgeleiteten FXS-Astrozyten. Statistische Analyse durch einfaktorige ANOVA mit dem paarweisen Vergleich von Tukey. Für alle Experimente N = 3 biologische Replikate; n = 19 Zellen für jede Zelllinie. *p < 0,05, **p < 0,01, ***p < 0,001. Fehlerbalken stellen SEM dar. # = Zahl. Abkürzungen: hiPSCs = humane induzierte pluripotente Stammzellen; STRG = Steuerung; FXS = Fragile-X-Syndrom; GFAP = saures gliales fibrilläres Protein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
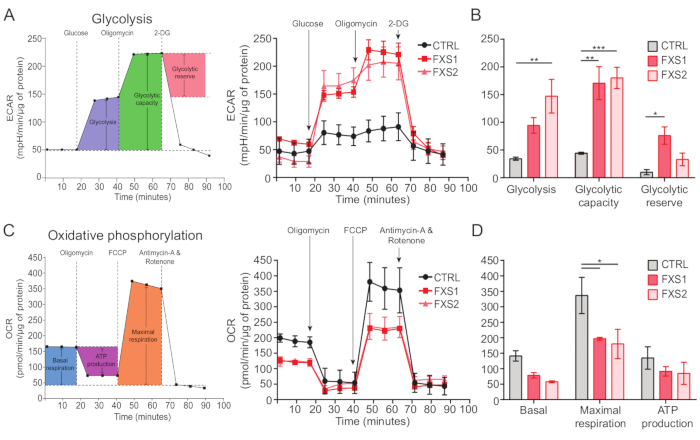
Abbildung 5: Bioenergetische Defizite in hiPSC-abgeleiteten FXS-Astrozyten. (A) Liniendiagramm, das die extrazelluläre Versauerungsrate von hiPSC-abgeleiteten Astrozyten (CTRL und FXS) nach Zugabe von 10 mM Glucose, 1 μM Oligomycin und 50 mM 2-DG nacheinander darstellt und auf einem Liniendiagramm dargestellt wird. (B) Die Quantifizierung der Glykolyse, der glykolytischen Kapazität und der glykolytischen Reserve zeigte einen Anstieg der hiPSC-abgeleiteten FXS-Astrozyten. Statistische Analyse durch Zwei-Faktor-ANOVA mit dem paarweisen Vergleich von Tukey. (C) Liniendiagramm, das die Messung der Sauerstoffverbrauchsrate durch sequenzielle Zugabe von 1,5 μM Oligomycin, 1 μM FCCP und 0,5 μM Antimycin A und Rotenon darstellt. (D) Die basale Atmung, die maximale Atmung und die ATP-Produktion wurden anhand des Liniendiagramms quantifiziert und hiPSC-abgeleitete FXS-Astrozyten zeigten eine signifikante Abnahme der maximalen Atmung im Vergleich zu CTRL-Astrozyten. Statistische Analyse durch Zwei-Faktor-ANOVA mit dem paarweisen Vergleich von Tukey. Für alle Experimente ist N = 2 biologische Replikate. *p < 0,05, **p < 0,01, ***p < 0,001. Fehlerbalken stellen REM dar. Abkürzungen: hiPSCs = humane induzierte pluripotente Stammzellen; STRG = Steuerung; FXS = Fragile-X-Syndrom; ECAR = extrazelluläre Versauerungsrate; 2-DG = 2-Desoxyglukose; OCR = Sauerstoffverbrauchsrate; FCCP = Carbonylcyanid-4 (Trifluormethoxy)phenylhydrazon. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| ID im Manuskript | ID an der Quelle | Alter | Sex | Name der neu programmierten Zelllinie | Methode der Neuprogrammierung | Zellentyp "Start" | G-Band-Karyotyp |
| (Jahre) | |||||||
| STRG | ND30625 | 76 | M | CS25iCTR-18nxx | Episomale Vektoren | Fibroblasten | Normal |
| FXS1 | GM07072 | 22 | M | CS072iFXS-n4 | Episomale Vektoren | Fibroblasten | Normal |
| FXS2 | GM05848 | 4 | M | CS848iFXS-n5 | Episomale Vektoren | Fibroblasten | Normal |
Tabelle 1: Zelllinien, die in dieser Studie zurErzeugung von Vorderhirn-spezifischen Astrozyten verwendet wurden.
Ergänzende Abbildung S1: Bericht zur Chromosomenanalyse. Normaler GTG-Banding-Karyotyp von (A) CTRL 46, XY, (B) FXS1 46, XY und (C) FXS2 46, XY. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Ergänzende Tabelle S1: Primer, die in dieser Studie zur Charakterisierung von Zellen verwendet wurden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Hier beschreiben wir eine Methode zur Generierung humaner iPSC-abgeleiteter Astrozyten, die als Assay-Plattform zur Charakterisierung funktioneller Veränderungen dienen, die durch FXS induziert werden. Diese Astrozyten sind in Kultur funktionell lebensfähig und weisen verschiedene Eigenschaften auf, wie eine Reihe von Messungen in der vorliegenden Studie zeigt. Ein kritischer Schritt in diesem Protokoll ist die anfängliche Umwandlung von iPSCs in Kortikosphären mit der enzymatischen Lifting-Methode. In diesem Stadium ist die Optimierung der Inkubationszeit für Kollagenase Typ IV und Dispase von entscheidender Bedeutung. Wenn dies nicht optimiert wird, kann es dazu führen, dass sich die iPS-Zellen auflösen oder einzelne Zellen bilden, was die Bildung von Kortikosphären verhindert. Diese Kortikosphären sind unter Verwendung der dualen SMAD-Hemmung15 in Richtung Vorderhirnspezifität gemustert und werden weiter zu Astrozyten-Vorläuferzellen (APCs) differenziert. Um eine hohe Population von APCs zu erreichen, ist das mechanische Zerkleinern der Gliosphären entscheidend. Die real-time qPCR kann verwendet werden, um die Spezifität des Vorderhirns zu bestätigen; Wir beobachteten eine signifikante Zunahme der Faltenveränderung bei FOXG1, einem Marker für das Vorderhirn, im Vergleich zu HOXB4, einem Marker für das Rückenmark.
APCs werden dann mit Hilfe von CNTF, einem Aktivator des JAK-STAT-Signalwegs, zu Astrozyten differenziert16. Die Ergebnisse dieser Studie deuten darauf hin, dass terminale differenzierte Astrozyten, die aus FXS-iPSCs stammen, niedrigere GFAP-Spiegel aufweisen, was auf eine beeinträchtigte Reife hindeutet. Obwohl unsere Analysen keinen Unterschied in der de novo Proteinsynthese in FXS-Astrozyten und APCs im Vergleich zu Kontrollen fanden, fanden wir eine Verringerung der de novo Proteinsynthese beim Übergang von APCs zu Astrozyten sowohl in der FXS- als auch in der Kontrolllinie. Die signifikante Verringerung der Translation zwischen APCs und Astrozyten steht im Einklang mit einer stromlinienförmigeren und kuratierteren Translation, die für Neuronen im Vergleich zu neuralen Vorläuferzellen gezeigt wurde17. Frühere Arbeiten, die die aberrante Proteinsynthese untersucht haben, haben ganze Hirngewebe, Neuronen und von Patienten stammende Fibroblasten verwendet, keine Astrozyten oder APCs18. Die einzigen veröffentlichten Berichte über die Proteinsynthese aus patienteneigenen Linien stammen nicht in Astrozyten, sondern aus lymphoblastoiden Zellen19 und Fibroblasten20,21. Angesichts dieser Unterschiede zwischen den Studien würden zukünftige Studien von der Verwendung von FUNCAT-Messungen in Astrozyten profitieren, die aus zusätzlichen FXS-Patientenlinien stammen.
Wir untersuchten die Funktionalität von Astrozyten durch Messung der intrazellulären Kalziumdynamik22 und der Bioenergetik. FXS-Astrozyten zeigten eine reduzierte maximale Amplitude und Dauer von ATP-induzierten Kalziumtransienten, wobei weniger Zellen im Vergleich zu Kontroll-Astrozyten auf ATP reagierten. Zusammengenommen deuten diese Veränderungen darauf hin, dass FXS die Kalziumhomöostase in menschlichen Astrozyten stört. Diese Ergebnisse stehen im Einklang mit einer früheren Studie23,24, in der über eine gestörte IP3-Rezeptoraktivität in FXS-Fibroblasten berichtet wurde. Darüber hinaus beobachteten wir eine erhöhte Glykolyse, glykolytische Kapazität und glykolytische Reserve in FXS-Astrozyten, zusammen mit einem reduzierten mitochondrialen Sauerstoffverbrauch.
Die vorliegende Studie beschreibt die Generierung und Charakterisierung eines neuen in vitro Modells für FXS-Astrozyten unter Nutzung der Leistungsfähigkeit von patienteneigenen iPSCs. Bemerkenswert ist, dass wir erst durch eine frühere Studie mit menschlichen Astrozyten, die mit menschlichen Neuronen kokultiviert wurden, die entscheidende Rolle von Astrozyten bei der Bestimmung des elektrophysiologischen Phänotyps der Neuronen aufdeckten14. Diese Studien konzentrierten sich jedoch fast ausschließlich auf krankheitsinduzierte Veränderungen der neuronalen Aktivität, und bei Astrozyten wurde nichts erforscht. Hier bietet dieses Protokoll einen neuartigen Rahmen, um unseren Fokus auf funktionelle Veränderungen in Astrozyten zu verlagern. Dies wiederum wird Licht auf bisher unerforschte Astrozyten-Neuronen-Wechselwirkungen werfen, wie z. B. die Glutamat-Signalübertragung und elektrische Reaktionen. Unsere Ergebnisse in diesem Stadium unterstreichen die Notwendigkeit, Astrozyten in der zukünftigen Forschung zu priorisieren.
Es ist wichtig, die Grenzen dieses Protokolls zu erkennen. Zunächst handelt es sich um mechanisches Zerkleinern, was ebenfalls zu einer DNA-Fragmentierung führt. Dies kann dazu führen, dass die zerstückelten Gliosphären verklumpen, was schließlich zum Zelltod führt. Zweitens ist die Aussaatdichte von APCs während des terminalen Differenzierungsschritts zu Astrozyten essentiell, um eine kontaktvermittelte Zell-Zell-Hemmung zu verhindern. Es wird empfohlen, dies über Zelllinien hinweg zu optimieren, um eine Ablösung von der Oberfläche des Kulturgefäßes zu verhindern.
Hier sind unsere Ergebnisse die erste derartige Analyse in menschlichen Astrozyten, da alle bisherigen Studien auf anderen Zelltypen wie lymphoblastoiden Zelllinien und menschlichen Fibroblasten basierten25. Ein dysfunktionaler Energiestoffwechsel ist ein bekannter Akteur in der Ätiologie von Autismus-Spektrum-Störungen, wie die erhöhten Spiegel an humanem Plasmalaktat, die signifikant niedrigere Sauerstoffverbrauchsrate in den Granulozyten26 und die verminderte Expression von mitochondrialen oxidativen Phosphorylierungsgenen im anterioren cingulierten, motorischen Kortex, Thalamus und Kleinhirn von Kindern mit ASDzeigen 27. Ähnliche Veränderungen im Energiestoffwechsel werden bei neurodegenerativen Erkrankungen wie der Parkinson-Krankheit28 und der Alzheimer-Krankheit29 beobachtet, was darauf hindeutet, dass eine erhöhte Glykolyse die verminderte funktionelle Funktion kompensieren kann30. Zukünftige Studien sind erforderlich, um zu untersuchen, ob ähnliche Mechanismen bei einem dysfunktionalen Energiestoffwechsel im Spiel sind, der bei FXS-Patienten beobachtet wurde.
Gegenwärtig ist die Generierung von Vorderhirn-spezifischen Astrozyten eine bedeutende Errungenschaft, aber zukünftige Fortschritte könnten die Schaffung regionenspezifischer Gliazellen ermöglichen, wie z. B. kortikale/hippokampale Astrozyten. Dies würde tiefere Einblicke in die unterschiedlichen Rollen dieser Astrozyten bei regionalen Gehirnfunktionen, synaptischer Plastizität und neuronal-glialen Interaktionen ermöglichen. Die Fähigkeit, solche regionenspezifischen Astrozyten zu generieren, könnte Modelle der Neuroentwicklung und neurodegenerativer Erkrankungen voranbringen. Letztendlich könnte dies zu präziseren therapeutischen Strategien führen, die auf die Dysfunktion von Astrozyten in bestimmten Hirnregionen abzielen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken Prof. Sumantra Chattarji für die internen Fonds. Wir danken Prof. Gaiti Hasan für den Zugang zum Calcium-Imaging-Setup, der Central Imaging and Flow Facility - National Centre for Biological Sciences, Padmanabh Singh und Prangya Hota für das Korrekturlesen und die Vorschläge und dem Team von Labmate Asia für ihre Unterstützung bei der Durchführung von Seahorse XF-Assays.
Materialien
| Name | Company | Catalog Number | Comments |
| 1-Thioglycerol | Sigma-Aldrich | M6145 | |
| Accutase solution | Sigma-Aldrich | A6964 | Enzyme cell detachment medium |
| Adenosine 5′-triphosphate magnesium salt | Sigma-Aldrich | A9187 | |
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634010 | |
| Antibiotic-Antimycotic (100x) | ThermoFisher Scientific | 15240062 | |
| B-27 Supplement (50x), serum-free | ThermoFisher Scientific | 17504044 | |
| Bovine Serum Albumin | Sigma-Aldrich | A9418 | |
| Chemically Defined Lipid Concentrate | ThermoFisher Scientific | 11905031 | |
| Collagenase, Type IV, powder | ThermoFisher Scientific | 17104019 | |
| Deoxyribonuclease I | Worthington Biochemical Corporation | LK003170 | |
| Dimethyl sulfoxide | Sigma-Aldrich | D2650 | |
| Dispase II, powder | ThermoFisher Scientific | 17105041 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| Essential 8 Medium | ThermoFisher Scientific | A1517001 | |
| FXS1, FXS2 | Coriell Institute of Medical Research | GM07072, GM05848 | FXS patient cells |
| GlutaMAX Supplement | ThermoFisher Scientific | 35050061 | glutamine substitute |
| Ham's F-12 Nutrient Mix | ThermoFisher Scientific | 11765054 | |
| Healthy control cells | Cedars-Sinai Medical Center | ND30625 | healthy control cells |
| Heparin sodium salt from porcine intestinal mucosa | Sigma-Aldrich | H3149 | |
| IMDM | ThermoFisher Scientific | 12440053 | |
| Insulin, human | Roche | 11376497001 | |
| LDN 193189 | Stratech Scientific | S2618-SEL | |
| Leukemia Inhibitory Factor human | Sigma-Aldrich | L5283 | |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | Corning | 354230 | |
| MEM Non-Essential Amino Acids Solution (100x) | ThermoFisher Scientific | 11140050 | |
| Mouse FGF-basic (FGF-2/bFGF) Recombinant Protein | Peprotech | 450-33 | |
| Mr. Frosty freezing container | ThermoFisher Scientific | 5100-0001 | cryobox |
| N-2 Supplement (100x) | ThermoFisher Scientific | 17502048 | |
| N-Acetyl-L-cysteine | Sigma-Aldrich | A9165 | |
| Neurobasal Medium | ThermoFisher Scientific | 21103049 | |
| Nunc Biobanking and Cell Culture Cryogenic Tubes | ThermoFisher Scientific | 377267 | |
| Nunc Cell-Culture Treated 6 well dish | ThermoFisher Scientific | 140675 | |
| Papain Dissociation System | Worthington Biochemical Corporation | LK003150 | |
| Penicillin-Streptomycin | ThermoFisher Scientific | 15140122 | |
| Recombinant Human CNTF Protein, CF | R&D Systems | 257-NT-010 | |
| Recombinant Human EGF Protein, CF | R&D Systems | 236-EG-01M | |
| RevitaCell Supplement (100x) | ThermoFisher Scientific | A2644501 | |
| SB431542 | Tocris | 1614 | |
| Seahorse XFe24 Analyzer | Agilent Technologies | ||
| Seahorse XF Cell Mito Stress Test Kit | Agilent Technologies | 103015-100 | |
| Seahorse XF Glycolysis Stress Test Kit | Agilent Technologies | 103020-100 | |
| Tissue Culture Dishes-100 cm | Biostar Lifetech | TCD000100 | |
| Transferrin | Roche | 10652202001 | |
| VWR Razor Blades | VWR International | 55411-050 | |
| Antibodies | |||
| Primary antibody | Company | Catalog number | Dilution |
| Oct4 (C-10) | Santa Cruz Biotechnology | sc-5279 | Dilution: 1:250 Secondary antibody: Goat anti-Mouse IgG, Alexa Fluor 568 |
| Nanog | R & D Systems | AF1997 | Dilution: 1:100 Secondary antibody: Donkey anti-Goat IgG, Alexa Fluor 488 |
| Vimentin | Abcam | Ab5733 | Dilution: 1:500 Secondary antibody: Goat anti-Chicken IgY, Alexa Fluor 488 |
| NFIA | Abcam | Ab41851 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 568 |
| GFAP-cy3 | Sigma | C9205 | Dilution: 1:500 Secondary antibody: NA |
| GFAP | DAKO | Z0334 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 568 |
| S100β | DAKO | IR504 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 488 |
| Anti-Nuclei Antibody, clone 235-1 | Merck Millipore | MAB1281 | Dilution: 1:1000 Secondary antibody: Goat anti-Mouse IgG1, Alexa Fluor 555 |
| Secondary antibodies | Dilution | ||
| Goat anti-Mouse IgG, Alexa Fluor 568 | Thermo Fisher Scientific | A11004 | 1:1000 |
| Donkey anti-Goat IgG, Alexa Fluor 488 | Thermo Fisher Scientific | A11055 | 1:1000 |
| Goat anti-Chicken IgY, Alexa Fluor 488 | Thermo Fisher Scientific | A11039 | 1:1000 |
| Goat anti-Rabbit IgG, Alexa Fluor 568 | Thermo Fisher Scientific | A11011 | 1:1000 |
| Goat anti-Rabbit IgG, Alexa Fluor 488 | Thermo Fisher Scientific | A11034 | 1:1000 |
| Goat anti-Mouse IgG1, Alexa Fluor 555 | Thermo Fisher Scientific | A21127 | 1:1000 |
Referenzen
- Santoro, M. R., Bray, S. M., Warren, S. T. Molecular of fragile X syndrome: A twenty-year perspective. Annu Rev Pathol. 7, 219-245 (2012).
- Wang, H., et al. Developmentally-programmed FMRP expression in oligodendrocytes: A potential role of FMRP in regulating translation in oligodendroglia progenitors. Hum Mol Genet. 13 (1), 79-89 (2004).
- Pacey, L. K. K., Doering, L. C. Developmental expression of FMRP in the astrocyte lineage: Implications for fragile X syndrome. Glia. 55 (15), 1601-1609 (2007).
- Zhang, Y., et al. Purification and characterization of progenitor and mature human astrocytes reveals transcriptional and functional differences with mouse. Neuron. 89 (1), 37-53 (2016).
- Allen, N. J., Barres, B. A. Neuroscience: Glia - more than just brain glue. Nature. 457 (7230), 675-677 (2009).
- Khakh, B. S., McCarthy, K. D. Astrocyte calcium signaling: From observations to functions and the challenges therein. Cold Spring Harb Perspect Biol. 7 (4), a020404(2015).
- Allen, N. J., et al. Astrocyte glypicans 4 and 6 promote formation of excitatory synapses via GluA1 AMPA receptors. Nature. 486 (7403), 410-414 (2012).
- Chung, K., et al. Structural and molecular interrogation of intact biological systems. Nature. 497 (7449), 332-337 (2013).
- Rothstein, J. D., et al. Knockout of glutamate transporters reveals a major role for astroglial transport in excitotoxicity and clearance of glutamate. Neuron. 16 (3), 675-686 (1996).
- Almad, A., Maragakis, N. J. A stocked toolbox for understanding the role of astrocytes in disease. Nat Rev Neurol. 14 (6), 351-362 (2018).
- Zhang, Y., Barres, B. A. Astrocyte heterogeneity: An underappreciated topic in neurobiology. Curr Opin Neurobiol. 20 (5), 588-594 (2010).
- Clarke, L. E., Barres, B. A. Emerging roles of astrocytes in neural circuit development. Nat Rev Neurosci. 14 (5), 311-321 (2013).
- Bradley, R. A., et al. Regionally specified human pluripotent stem cell-derived astrocytes exhibit different molecular signatures and functional properties. Development. 146 (13), dev170910(2019).
- Das Sharma, S., et al. Astrocytes mediate cell non-autonomous correction of aberrant firing in human FXS neurons. Cell Rep. 42 (4), 112344(2023).
- Chambers, S. M., et al. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Hu, X., et al. The JAK/STAT signaling pathway: from bench to clinic. Sig Transduct Target Ther. 6, 402(2021).
- Baser, A., et al. Onset of differentiation is post-transcriptionally controlled in adult neural stem cells. Nature. 566 (7742), 100-104 (2019).
- Pal, R., Bhattacharya, A. Modelling protein synthesis as a biomarker in fragile x syndrome patient-derived cells. Brain Sci. 9 (3), 1-12 (2019).
- Gross, C., Bassell, G. J. Excess protein synthesis in FXS patient lymphoblastoid cells can be rescued with a p110β-selective inhibitor. J Mol Med. 18 (3), 336-345 (2012).
- Kumari, D., et al. Identification of fragile X syndrome specific molecular markers in human fibroblasts: A useful model to test the efficacy of therapeutic drugs. Hum Mutat. 35 (12), 1485-1494 (2014).
- Jacquemont, S., et al. Protein synthesis levels are increased in a subset of individuals with fragile X syndrome. Hum Mol Genet. 27 (12), 2039-2051 (2018).
- Bowser, D. N., Khakh, B. S. ATP excites interneurons and astrocytes to increase synaptic inhibition in neuronal networks. J Neurosci. 24 (39), 8606-8620 (2004).
- Schmunk, G., Boubion, B. J., Smith, I. F., Parker, I., Gargus, J. J. Shared functional defect in IP3R-mediated calcium signaling in diverse monogenic autism syndromes. Transl Psychiatry. 5 (9), e643-e710 (2015).
- Peteri, U. K., et al. Generation of the human pluripotent stem-cell-derived astrocyte model with forebrain identity. Brain Sci. 11 (2), 209(2021).
- Alvarez-Mora, M. I., et al. Impaired mitochondrial function and dynamics in the pathogenesis of FXTAS. Mol Neurobiol. 54 (9), 6896-6902 (2017).
- Giulivi, C., et al. Mitochondrial dysfunction in autism. JAMA. 304 (21), 2389-2396 (2010).
- Anitha, A., et al. Brain region-specific altered expression and association of mitochondria-related genes in autism. Mol Autism. 3 (1), 12(2012).
- Teves, J. M. Y., et al. Parkinson's disease skin fibroblasts display signature alterations in growth, redox homeostasis, mitochondrial function, and autophagy. Front Neurosci. 11, 737(2018).
- Yao, J., et al. Mitochondrial bioenergetic deficit precedes Alzheimer's pathology in female mouse model of Alzheimer's disease. Proc Natl Acad Sci USA. 106 (34), 14670-14675 (2009).
- Sonntag, K. C., et al. Late-onset Alzheimer's disease is associated with inherent changes in bioenergetics profiles. Sci Rep. 7 (1), 14038(2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten