Method Article
Imaging longitudinale intravitale della dinamica vascolare nel midollo osseo calvariale
In questo articolo
Riepilogo
La microscopia intravitale consente lo studio di processi biologici dinamici come la rigenerazione dei tessuti e lo sviluppo del tumore. Il midollo osseo calvariale, un tessuto altamente dinamico, offre informazioni sull'emopoiesi e sulla funzione vascolare. L'utilizzo di un impianto di fissazione della testa biocompatibile stampato in 3D consente l'imaging longitudinale ripetitivo, migliorando la nostra comprensione delle dinamiche dei tessuti e del microambiente tumorale.
Abstract
L'imaging al microscopio a fluorescenza longitudinale intravitale è emerso come una tecnica cruciale per lo studio dei processi biologici dinamici, in particolare nel contesto della rigenerazione tissutale, dello sviluppo tumorale e delle risposte terapeutiche. In particolare, il midollo osseo calvario è un tessuto altamente dinamico, in cui il destino ematopoietico è interconnesso con il microambiente circostante, con vasi specializzati che rispondono all'emopoiesi normale e patologica. L'imaging tradizionale dei tessuti fissati offre informazioni statiche, spesso limitando una comprensione completa di questi processi.
L'integrazione di animali transgenici che esprimono marcatori specifici per le cellule, traccianti di cellule vive, i progressi nelle apparecchiature di imaging e l'uso di camere specializzate hanno elevato la microscopia intravitale a uno strumento fondamentale per ottenere informazioni sui fenomeni biologici dinamici. Un'applicazione dell'imaging intravitale è l'indagine sul comportamento dei vasi tumorali e sugli effetti terapeutici. Un impianto di fissazione della testa in titanio stampato in 3D di nuova concezione può essere collegato stabilmente al cranio del topo ed è adatto per l'imaging longitudinale durante più sessioni.
Il protocollo proposto consente l'esame spaziale e temporale delle dinamiche vascolari nel midollo osseo calvariano, compresa la visualizzazione e la quantificazione dell'eterogeneità vascolare, l'interazione con le cellule stromali ed ematopoietiche e la misurazione dei parametri funzionali vascolari. Inoltre, la tecnica consente la visualizzazione di letti vascolari stabiliti e il monitoraggio degli effetti terapeutici, la mobilizzazione delle cellule staminali e la localizzazione dei composti chemioterapici nel tempo utilizzando la microscopia a due fotoni. Nel complesso, questo protocollo di imaging longitudinale intravitale fornisce una piattaforma completa per studiare sia il comportamento dei vasi tumorali che la dinamica delle cellule ematopoietiche, offrendo preziose informazioni sugli intricati processi che regolano questi fenomeni biologici.
Introduzione
L'imaging al microscopio intravitale del midollo osseo calvariale (BM) funge da tecnica potente e indispensabile per studiare i processi dinamici dell'emopoiesi, la regolazione del microambiente tissutale e la dinamica vascolare all'interno della nicchia del midollo osseo. Lo scopo principale di questo approccio metodologico è quello di consentire la visualizzazione e l'analisi in tempo reale dei comportamenti cellulari, delle interazioni e dell'organizzazione spaziale all'interno del microambiente BM in vivo. Osservando direttamente la calvaria del midollo osseo utilizzando tecniche di imaging avanzate abbinate alla marcatura fluorescente, i ricercatori possono chiarire la complessa interazione tra le cellule staminali ematopoietiche (HSC), le cellule stromali e il sistema vascolare circostante, fornendo così informazioni cruciali sulla regolazione dell'emopoiesi e delle risposte immunitarie.
Lo sviluppo e l'utilizzo di cellule staminali di imaging al microscopio intravitale dai limiti dei tradizionali metodi di imaging istologico ed ex vivo, che spesso non riescono a catturare la natura dinamica dei comportamenti cellulari e delle interazioni di un tessuto. A differenza delle tecniche di imaging statico, la microscopia intravitale consente ai ricercatori di osservare le dinamiche cellulari in tempo reale, consentendo lo studio di processi longitudinali come la migrazione, la proliferazione e la differenziazione cellulare all'interno della loro nicchia nativa, senza sacrificare gli animali da esperimento. Inoltre, la microscopia intravitale offre il vantaggio unico di studiare i comportamenti funzionali in vivo, come la funzionalità vascolare (ad esempio, perfusione, permeabilità, ipossia), preservando così la rilevanza fisiologica ed evitando artefatti associati alla fissazione e al processamento dei tessuti. Studi pionieristici nel campo hanno dimostrato gli enormi vantaggi di questo approccio 1,2 e i loro risultati sono stati corroborati e ampliati da approcci più recentemente perfezionati 3,4,5 che hanno utilizzato la microscopia intravitale per tracciare la localizzazione, la migrazione e le interazioni endogene delle HSC con la vascolarizzazione all'interno della nicchia del midollo osseo. Inoltre, la microscopia intravitale è stata determinante per chiarire i meccanismi alla base dei disturbi ematopoietici, come la leucemia e le sindromi da insufficienza midollare, offrendo nuove informazioni sulla motilità delle cellule leucemiche 6,7, sulle implicazioni vascolari associate alla malattia8 e sulla risposta ai farmaci9.
Ci sono diversi vantaggi dell'imaging al microscopio intravitale dei siti ossei alternativi della copertura del midollo osseo calvariale. In primo luogo, il midollo osseo contenuto all'interno dell'osso cranico fornisce una facile accessibilità per l'imaging intravitale rispetto alle ossa più profonde come il femore o la tibia. Questa accessibilità facilita l'osservazione diretta del microambiente tissutale, compreso l'osso stesso, tramite la visualizzazione di generazione di seconda armonica (SHG)10, senza la necessità di procedure chirurgiche invasive. In secondo luogo, il cranio è relativamente sottile e traslucido, consentendo una migliore visualizzazione delle dinamiche cellulari all'interno della nicchia del midollo osseo. Questa trasparenza facilita l'imaging ad alta risoluzione con microscopia a due fotoni, microscopia confocale e microscopia a foglio luminoso, senza la necessità di tecniche di assottigliamento o pulizia ossea. L'osso calvario offre una piattaforma stabile, piatta e rigida per esperimenti di imaging intravitale, riducendo al minimo gli artefatti di movimento dei tessuti e garantendo condizioni di imaging coerenti per periodi di osservazione prolungati. Questa stabilità è particolarmente vantaggiosa per gli studi longitudinali che monitorano i comportamenti e le risposte cellulari nel tempo. La riproducibilità è un altro grande vantaggio, data la struttura relativamente piccola e spazialmente definita dell'osso calvario negli animali da esperimento. Questa uniformità facilita il confronto tra diversi gruppi sperimentali e consente una solida analisi statistica dei dati di imaging.
Qui descriviamo un metodo per visualizzare il midollo osseo calvario del topo tramite microscopia intravitale a due fotoni introducendo un impianto di fissazione della testa11 di nuova concezione, stampato in 3D utilizzando una lega di titanio biocompatibile di grado 23 (Ti6Al4V), che presenta una copertura rigida dedicata e facilmente posizionabile realizzata con la stessa lega di titanio, che consente una chiusura sicura della ferita per evitare infezioni o danni al sito chirurgico. L'impianto fissa la testa del topo in modo saldo e stabile al tavolino del microscopio tramite un supporto per impianto, riducendo al minimo gli artefatti respiratori e consentendo l'imaging longitudinale delle stesse aree nel tempo. Vengono forniti alcuni esempi di immagini multicolore raffiguranti cellule e strutture del microambiente BM (SHG+ superficie ossea; nestina-GFP+ cellule mesenchimali; cdh5-DSRED+, cdh5-GFP+ o pdgfb-GFP+) e il compartimento ematopoietico maligno (cellule AML tdTOMATO+), nonché agenti di contrasto fluorescenti che raffigurano il lume dei vasi (destrano-TRITC). I parametri vascolari misurati nell'arco di ore o giorni, tra cui la lunghezza, la rettilineità e il diametro dei vasi, nonché la permeabilità in diverse regioni vascolari, possono fornire informazioni importanti sul comportamento e sulla salute dei tessuti.
Protocollo
Tutti gli esperimenti sugli animali sono stati eseguiti nell'ambito dell'accordo etico APAFIS#27215-2020041513522374 v6, approvato dal "Ministère de l'enseignement supérieur, de la recherche et de l'innovation" francese.
1. Progettazione di un impianto di fissazione della testa in titanio biocompatibile per la stampa 3D
NOTA: Abbiamo progettato un impianto di fissazione della testa biocompatibile utilizzando un software CAD (Computer-Aided Design) di modellazione parametrica con funzionalità integrate di analisi degli elementi finiti (FEA) (vedere la Tabella dei materiali). Gli input primari per il processo di progettazione includono un modello anatomico ad alta risoluzione del cranio del topo, un modello dell'obiettivo del microscopio e un modello del sistema di fissaggio, che insieme informano le dimensioni e la configurazione dell'impianto. L'output finale è un file mesh indipendente dalla piattaforma, tipicamente in formato STL o STEP, compatibile con la maggior parte dei software di stampa 3D. Questo formato di file garantisce un trasferimento senza soluzione di continuità alla stampante 3D per una fabbricazione accurata dell'impianto.
- Preparazione del modello 3D del mouse
- Posizionare il cranio del topo, preparato o in vivo, nello scanner micro-CT.
- Imposta i parametri di scansione per una risoluzione ottimale dei dettagli anatomici fini: apri il Pannello di controllo e nella sezione Condizioni di scansione , scegli un Campo visivo (FOV). Utilizzare LiveView per definire il volume di scansione.
NOTA: Attraverso la risoluzione del rivelatore, il FOV determinerà la dimensione del voxel in base alla risoluzione del rivelatore. Nelle macchine micro-CT più recenti, un FOV di 5 mm corrisponde tipicamente a una dimensione del voxel di 5 μm, un FOV di 10 mm a una dimensione del voxel di 10 μm, un FOV di 25 mm a una dimensione del voxel di 21 μm. Il FOV limitato a questa risoluzione può richiedere più scansioni per coprire l'intero cranio. Le scansioni multiple richiedono la sovrapposizione tra le scansioni. - Unisci più scansioni ad alta risoluzione nel software di imaging per creare un unico modello completo del cranio.
- Importa file DICOM micro-CT in software di segmentazione (ad es. 3D Slicer, Amira, Avizo o Mimics) per isolare il cranio.
- Esegui la segmentazione basata su soglia per separare il tessuto osseo dalle strutture circostanti ed effettua regolazioni manuali per affinare regioni complesse o rumorose.
- In 3D Slicer, importa prima i file DICOM e carica il set di dati e osserva le sezioni CT nelle viste assiale, sagittale e coronale .
- Apri il segmento Edittor, fai clic su Aggiungi per creare una nuova segmentazione | Aggiungi per creare un nuovo segmento, che deve essere sottoposto a soglia.
- Nel pannello Editor segmenti , fai clic sull'effetto Soglia . Regolare i dispositivi di scorrimento della soglia inferiore e superiore (o digitare valori numerici) in modo che la regione da segmentare sia evidenziata in rosso nelle viste sezione. Un segmento 3D con soglia è pronto.
- Per esportare il teschio segmentato come file STL, passare al modulo di segmentazione. Assicurati che il segmento con soglia sia selezionato. Nella sezione Esporta/Importa modelli e mappe etichetta , impostare Tipo di esportazione su Modelli. e Formato file: STL. Fare clic sul pulsante Esporta .
NOTA: I file STL utilizzano un formato a maglie triangolari, che rappresenta la superficie del cranio con migliaia di triangoli, catturando la forma anatomica nei minimi dettagli.- Se necessario, eseguire la cucitura in MeshLab come segue:
- Importare le mesh facendo clic su File | Importa Mesh e selezionane una come scansione di riferimento.
- Allineare altre scansioni utilizzando lo strumento Allinea (Filtri | Iscrizione | Allinea) contrassegnando i punti corrispondenti e utilizzando Processo.
- Unisci le scansioni allineate facendo clic su Filtri | Strato di rete | Appiattisci i livelli visibili con l'opzione Unisci solo i livelli visibili .
- Se necessario, eseguire la cucitura in MeshLab come segue:
- Pulire e riparare la mesh, eliminando vertici e triangoli duplicati come segue, per garantire la rimozione dei triangoli indotti dal rumore e il riempimento di eventuali fori non intenzionali, per ottenere una superficie liscia e continua:
- Clicca su Filtri | Pulizia e riparazione | Rimuovi i vertici duplicati.
- Clicca su Filtri | Pulizia e riparazione | Rimuovi le facce duplicate.
- Clicca su Filtri | Pulizia e riparazione | Rimuovere i pezzi isolati (per numero di faccia). Specificare una soglia per il numero minimo di facce da mantenere (ad esempio, impostare questa soglia su un valore basso per rimuovere piccoli cluster o disturbo).
- Clicca su Filtri | Remeshing, semplificazione e ricostruzione | Chiudere i fori.
- Esegui la semplificazione della mesh a ~10.000-30.000 triangoli poiché i file STL iniziali da micro-CT contengono un numero di triangoli molto elevato che non è compatibile con il software CAD. Nelle aree ad alto dettaglio, mantieni i triangoli più piccoli sulla parte superiore del cranio per garantire un adattamento accurato dell'impianto. Nelle regioni più grossolane, semplificate le regioni meno critiche con triangoli più grandi per ridurre le dimensioni del file e ottimizzare l'efficienza dell'elaborazione.
NOTA: La semplificazione della mesh è fatta per aree e dettagliata in 1.1.8.1-1.1.8.3.- Utilizzare lo strumento di selezione Seleziona componenti connessi in una regione per selezionare le aree da semplificare.
- Usa la decimazione di compressione del bordo quadratico sulle aree selezionate con i parametri. Clicca su Filtri | Remeshing, semplificazione e ricostruzione | Semplificazione: Decimazione del collasso del bordo della quadrica. Impostare Soglia di qualità su 0,8, Mantieni il contorno della mesh su SÌ, Mantieni topologia su SÌ, Numero di facce target: impostare un numero di destinazione corrispondente alla risoluzione necessaria.
- Ripetere se necessario fino a quando il numero totale di triangoli non viene ridotto al livello desiderato. Quindi, salvate il modello semplificato in un file STL.
- Preparazione del modello del cranio nel modello CAD
- Importa il modello semplificato del cranio in formato STL nel software CAD come nuova parte o modello. Assicurarsi che il file importato venga convertito correttamente in un corpo solido o di superficie, se necessario, a seconda delle capacità del software. Salva il modello nel formato di file CAD nativo per consentire ulteriori modifiche e garantire la compatibilità con le fasi di progettazione successive.
- Posizionate i punti di riferimento su Bregma e Lambda e create l'asse Bregma-Lambda in questo modello.
- Create il piano mediano compreso l'asse Bregma-Lambda.
- nel piano mediano, creare un asse tangente alla calvaria, quindi salvare il cranio preparato (Figura supplementare S1).
- Create un nuovo assieme e inserite il cranio preparato in modo che Bregma coincida con l'origine, il piano mediano con il piano destro e l'asse calvario sia orizzontale.
- L'impianto di fissazione della testa sarà progettato direttamente all'interno dell'assemblaggio per garantire un perfetto adattamento con il modello di cranio preparato. Per raggiungere questo obiettivo, creare l'impianto come una nuova parte all'interno dell'assieme. Per iniziare, passare a Inserisci | Componente | Nuovo pezzo nel software CAD. Quando richiesto, selezionare la superficie calvaria o il piano superiore dell'assieme come riferimento per posizionare e ancorare la nuova parte.
- Crea una serie di piani equidistanti (spaziatura di 2 mm) attraverso il cranio. Su ogni piano, creare uno schizzo e su ogni schizzo, disegnare una curva proiettata con offset di 0,2 mm sotto forma di una spline simmetrica con nove punti, seguendo la sezione della superficie superiore del cranio 0,2 mm sopra il cranio (Figura supplementare S2).
- Crea una superficie spazzata utilizzando queste scanalature - la superficie limite del cranio - assicurandoti che questa superficie sia liscia senza alcun difetto, da utilizzare per la costruzione delle caratteristiche dell'impianto.
- Definizione della superficie osservabile sul cranio
NOTA: Sulla base del modello 3D, viene identificata la regione più piatta dell'osso della calvaria ideale per l'osservazione 3D. Questa regione è stata scelta con cura per garantire un accesso ottimale all'obiettivo del microscopio a due fotoni, considerando l'integrità biologica e strutturale del cranio. La definizione può utilizzare coordinate stereotassiche in cui il punto di Bregma funge da origine del sistema di coordinate.- Create uno schizzo nel piano calvariale e create una spline a forma di pera da AP +6,5 a -2, 6 mm di larghezza a AP 0,0. Questo sarà il contorno della superficie osservabile (Figura supplementare S3).
NOTA: L'obiettivo del microscopio è posizionato virtualmente rispetto alla superficie osservabile precedentemente definita sul cranio. Questa fase consiste nel determinare i vincoli spaziali imposti dalle dimensioni e dalla forma dell'obiettivo, assicurando che il design dell'impianto soddisfi i requisiti del microscopio. - Inserire verticalmente il modello dell'obiettivo del microscopio nell'assieme. Vincolare il suo punto di messa a fuoco su un piano parallelo al piano calvario, l'osso calvario, e definire il volume limite dell'obiettivo del microscopio.
- Create un volume eseguendo lo sweep dell'obiettivo seguendo il contorno della superficie osservabile.
- Crea una finestra di osservazione attorno alla superficie osservabile definita, garantendo un accesso senza ostacoli per l'obiettivo del microscopio.
NOTA: La finestra di osservazione è una struttura ad anello con funzioni secondarie come la ritenzione del liquido per immersione e la struttura solida per la corretta chiusura del coperchio di protezione. - Creare uno schizzo nel piano calvariale e copiare il contorno della superficie osservabile.
- Creare un contorno parallelo di 0,5 mm di spessore. Estrudere questo doppio contorno verso l'alto di 0,9 mm e verso il basso fino alla superficie della superficie Skull Limit con un angolo di sformo di 4°.
- Create uno schizzo nel piano calvariale e create una spline a forma di pera da AP +6,5 a -2, 6 mm di larghezza a AP 0,0. Questo sarà il contorno della superficie osservabile (Figura supplementare S3).
- Definizione delle strutture di cementazione, coda e coda di rondine.
- Create uno schizzo nel piano calvariale e disegnate una forma a C spessa 0,5 mm che si collega alla finestra di osservazione ed estrudete 2 mm di altezza e verso il basso fino alla superficie della superficie limite del cranio con un angolo di sformo di 4° (Figura S4 supplementare).
- Creare uno schizzo nel piano calvariale e riempire questo spazio con rettangoli di 0,4 mm di spessore posizionati radialmente con uno spazio di 1 mm tra di loro.
- Estrudete in modo simile alla finestra di osservazione.
- Creare la Coda dell'impianto affidandosi al cranio per la fissazione rispettando il volume disponibile.
- Create un piano trasversale tangente alla struttura di cementazione e un altro 12,5 mm all'indietro.
- Disegna rettangoli su questi piani. Sweepare un corpo piramidale tra di loro, impostando gli angoli a circa 20° e 37° dall'orizzontale.
- Aggiungere un foro filettato M1.6 nella coda vicino alla struttura di cementazione.
- Create uno schizzo all'estremità della coda e disegnate il profilo a coda di rondine.
- Estrudere questo profilo di 8 mm.
- Verifica della non interferenza con l'obiettivo.
- Esaminare l'intero design dell'impianto, compresa la finestra di osservazione e la coda di rondine, per assicurarsi che non vi siano interferenze meccaniche con l'obiettivo del microscopio. Verificare la presenza di interferenze con il volume creato nel passaggio 1.3.3.
NOTA: Questa fase di verifica è fondamentale per garantire che l'obiettivo possa essere posizionato e spostato come richiesto senza ostacoli.
- Esaminare l'intero design dell'impianto, compresa la finestra di osservazione e la coda di rondine, per assicurarsi che non vi siano interferenze meccaniche con l'obiettivo del microscopio. Verificare la presenza di interferenze con il volume creato nel passaggio 1.3.3.
- Rimuovere il materiale non necessario dalla coda dell'impianto per ridurre al minimo il peso e ridurre il potenziale disagio per l'animale.
- Create uno schizzo nel piano mediano e disegnate ritagli triangolari su di esso per formare una struttura a travatura reticolare quando viene estratto per estrusione dal corpo della coda.
- Ripetere l'operazione sul piano perpendicolare.
- Verifica lo spostamento massimo mediante simulazione con il metodo degli elementi finiti (FEM) per analizzare le proprietà meccaniche dell'impianto in varie condizioni.
NOTA: Questa simulazione aiuta a prevedere lo spostamento massimo e le sollecitazioni all'interno dell'impianto durante l'uso, garantendone l'affidabilità.- Abilitare il componente aggiuntivo di simulazione del software CAD.
- Nella barra degli strumenti Simulazione, creare un nuovo studio statico. Vai alla scheda Simulazione (se visibile) o vai a Simulazione | Studiare. Nella finestra di dialogo Studio , scegliere Statico come tipo di studio. Fare clic su OK per creare il nuovo studio (Figura supplementare S5).
NOTA: Questo tipo di studio è adatto per analizzare la deformazione e le sollecitazioni sotto carichi statici, come previsto nell'uso dell'impianto. - Scegliere un materiale per l'impianto che corrisponda alla composizione effettiva dell'impianto (Ti6Al4V).
- Definisci vincoli fissi per simulare i punti di attacco dell'impianto nel punto in cui entrerebbe in contatto con la fissazione a coda di rondine. Definite la superficie superiore della coda di rondine come fissa.
- Definire il carico verticale sulla superficie inferiore dell'impianto. Impostare una forza verticale di 1 N.
- Generare una mesh. Affinare la mesh nelle aree in cui è prevista un'elevata sollecitazione o deformazione (Figura S6 supplementare). A tale scopo, fare clic con il pulsante destro del mouse sull'elemento mesh dell'albero dello studio e selezionare Crea mesh... Nella finestra di dialogo Mesh , regolare la densità della mesh con il cursore o attivando la casella di controllo della mesh per impostare le dimensioni massime e minime degli elementi in modo indipendente. Fare clic su OK per generare la mesh.
NOTA: Il software genera automaticamente una mesh iniziale per lo studio, ma per ottenere risultati più accurati, la mesh può essere perfezionata per selezionare una mesh più fine, in particolare nelle regioni critiche per la stabilità dell'impianto (come la finestra di osservazione). Una mesh più fine in queste regioni migliora l'accuratezza della simulazione catturando le concentrazioni di stress locali. - Avvia l'analisi statica per calcolare le distribuzioni di spostamento, sollecitazione e deformazione nell'impianto.
- Analizza i risultati. Esaminare il grafico dello spostamento per identificare le aree di massima deformazione. Questo grafico mostra quanto l'impianto si deformerebbe sotto i carichi applicati, evidenziando eventuali aree a rischio di movimento eccessivo. Assicurarsi che la deformazione all'interno della finestra di osservazione non superi i 3 micron.
- Infine, progetta una copertura protettiva per proteggere la finestra di osservazione, quando non è in uso. Questo coperchio protegge l'osso esposto da contaminanti ambientali e danni fisici.
2. Trattamenti sui topi, anestesia e impianto chirurgico dell'impianto della testa
NOTA: In questo caso, maschi o femmine, C57BL/6 di età compresa tra 7 e 12 settimane o topi transgenici, come raffigurato. Per indurre la colonizzazione leucemica del midollo osseo, le cellule leucemiche generate, come descritto da Horton et al.12, vengono somministrate per via endovenosa 2-3 settimane prima dell'imaging. Per garantire la salute della ferita devono essere utilizzate tecniche sterili.
- Sotto il cofano, assemblare gli strumenti chirurgici sterili (forbici smussate e pinzette), il rifinitore e i materiali di consumo, tra cui gel per gli occhi, soluzione fisiologica, disinfettante, cemento dentale, tamponi assorbenti e fazzoletti per la pulizia. Accendere lo sterilizzatore di perline.
- Riempire la camera di induzione dell'anestetico con isoflurano al 4% e ossigeno atmosferico e posizionare il topo nella camera. Quando l'animale è completamente anestetizzato (verificare la presenza di una perdita del riflesso di raddrizzamento e di un modello di respirazione più profondo e lento), commutare il flusso di isoflurano al cono nasale della maschera anestetica e abbassare la concentrazione di isoflurano al 2%.
- Posizionare il mouse su un termoforo a 37 °C e monitorare visivamente la frequenza respiratoria. Facoltativo: per i topi con un fenotipo patologico, aggiungere una coperta di sopravvivenza per preservare la temperatura corretta durante l'anestesia.
- Somministrare antidolorifici per via sottocutanea (Buprecare, 0,01 mg/kg) 30 minuti prima dell'intervento.
- Radere la testa del mouse con un rasoio elettrico.
- Aggiungere una goccia di gel oftalmico sugli occhi del topo per prevenire la secchezza durante l'anestesia, assicurandosi che il gel non goccioli sul cuoio capelluto.
- Pulire il mouse per rimuovere eventuali peli residui dall'area chirurgica. Tamponare la parte superiore del cuoio capelluto con disinfettante su un fazzoletto di cotone. Assicurati di rimuovere tutti i peli per evitare artefatti di imaging e il rischio di infezione della ferita.
- Utilizzando pinze e forbici sterili (dopo 30 s nello sterilizzatore), praticare una piccola incisione nella parte centrale del cuoio capelluto per esporre la cicatrice ossea centrale. Seguirlo per definire la lunghezza e la larghezza corrette dell'area di imaging. Rimuovere con cura il pezzo di cuoio capelluto, sollevare la pelle tra le orecchie con una pinza e praticare una piccola incisione nella parte posteriore della testa. Tenendo la pelle sollevata, passare le forbici sotto la pelle e tagliare con cura al di fuori dell'area di imaging.
- Rimuovere il tessuto connettivo tra il cranio e il cuoio capelluto. Pulire l'osso esposto con PBS sterile su un batuffolo di cotone. Procedere rapidamente all'attacco dell'impianto della testa al cranio del topo per evitare l'esposizione prolungata dell'osso del cranio (<10 min).
- Preparare una pasta con abbastanza cemento dentale in una capsula di Petri seguendo le istruzioni del produttore e applicarla rapidamente sul fondo dell'impianto di fissazione della testa.
- Senza far entrare il cemento dentale all'interno dell'area di imaging, posizionare il supporto per la testa sul cranio esposto del topo, quindi attendere che si solidifichi.
- Una volta indurito (normalmente entro 3-5 minuti), aggiungere alcune gocce di PBS a 37 °C nella camera di imaging per mantenere il cranio idratato.
3. Imaging con un microscopio a due fotoni
- Prima di iniziare l'intervento chirurgico, accendere il laser e lasciarlo riscaldare per stabilizzarsi. Accendere il microscopio e avviare il software di acquisizione. Selezionare l'obiettivo appropriato (immersione in ACQUA 25x/0,95 per questo esperimento).
- Utilizzare un supporto per microscopio verticale a tavolino fisso che abbia spazio sufficiente per posizionare una breadboard veterinaria con un cuscinetto termico, il tutto da spostare integralmente con il tavolino xy. Utilizzare una solida breadboard ottica in alluminio come piattaforma per fissare l'impianto della testa e la maschera stereotassica. Collegare i tubi flessibili e i relativi connettori associati all'iniezione e all'estrazione dell'isoflurano alla maschera nella gabbia del microscopio.
- Configura le impostazioni di acquisizione per una scansione veloce: formato 512 x 512 pixel; velocità 600 Hz; e fattore di zoom 1.
- Sintonizza la linea laser a 880 nm per essere in grado di rilevare SHG, GFP e tdTOMATO con la stessa lunghezza d'onda di eccitazione.
- Spegnere la luce nella stanza e chiudere correttamente la scatola attorno al supporto del microscopio prima di attivare i rivelatori NDD e definire le porte di rilevamento spettrale: PMT1, per rilevare SHG [388 - 431 nm]; HyD2, per GFP [485 - 548 nm] e HyD3 per tdTOMATO [551 - 645 nm]. Lasciate l'offset a 0.
- Creare un nuovo set di dati del progetto e rinominarlo in base all'esperimento specifico.
- Una volta che l'impianto di fissazione della testa è attaccato al cranio del topo (confermato dall'indurimento del cemento), accendere l'isoflurano verso la maschera stereotassica del microscopio.
- Avvicinare rapidamente il mouse al microscopio. Inserire con cautela i denti del topo nella maschera per consentire la penetrazione dell'isoflurano attraverso il naso del topo sollevando il naso con una mano.
- Tenendo il mouse con una mano, infilare delicatamente la coda di rondine dell'impianto di fissaggio della testa nel supporto di fissaggio con l'altra mano e fissarla con mezzo giro della manopola a vite.
- Introdurre una sonda rettale preincorporata con un gel a base d'acqua per il monitoraggio della temperatura e aggiungere gocce oftalmiche agli occhi del topo.
- Riempire la camera di imaging della testa con una grande quantità di gel a base d'acqua o PBS e abbassare l'obiettivo ad immersione in acqua per immergerlo completamente per ottenere un'eccitazione e una rilevazione ottimali dei segnali generati dall'eccitazione a due fotoni.
- Sposta il tavolino x-y e l'unità z per concentrarti sull'osso centrale nella cicatrice dell'impalcatura.
NOTA: È conveniente utilizzare gli oculari del microscopio e una lampada ad alogenuri metallici come sorgente luminosa, filtrata da un filtro di emissione di eccitazione tri-banda. La mancanza di sezionamento ottico rende impossibile ottenere un'immagine nitida; Ma con la pratica, l'area centrale del cranio è facilmente riconoscibile. - Dopo aver identificato il tessuto, contrassegnato da una superficie ossea e da una vena centrale, spegnere la lampada ad alogenuri metallici e chiudere le finestre della gabbia del microscopio per proteggere i rivelatori HyD e consentire il passaggio del laser a infrarossi (IR).
- Impostare il guadagno PMT/SHG su 850 V (il suo intervallo di funzionamento lineare) e impostare i guadagni HyD al 100%. Aumentare la potenza del laser IR fino ad ottenere un'immagine con una gamma dinamica di 200 livelli di grigio (con rivelatori dinamici a 8 bit) sul più basso dei tre canali. Ridurre i guadagni degli altri rilevatori se le differenze di emissione sono troppo importanti o se c'è saturazione utilizzando la modalità LUT (Look Up Table) "sotto il basso".
NOTA: Nel nostro caso, una potenza laser del 30% era sufficiente per ottenere il contrasto in tutti i canali. - Sul software di acquisizione, cercare una regione costituita da tasche BM (GFP+, tdTOMATO+) racchiuse all'interno delle superfici ossee (SHG).
- Per trovare diversi ROI, attiva LAS NAVIGATOR e crea una panoramica dell'intera regione utilizzando la modalità a spirale. Interrompere l'acquisizione quando la superficie acquisita è sufficientemente grande. Salva la panoramica, uniscila e rinominala: questa sarà l'immagine di riferimento in caso di imaging longitudinale.
NOTA: Fare attenzione a non generare una spirale troppo grande in quanto ciò potrebbe causare il contatto del supporto del dispositivo con l'obiettivo. - Registra alcune posizioni ROI facendo clic sull'icona dell'immagine singola e rinomina ogni posizione nell'elenco delle attività. Acquisisci una schermata per sovrapporre la posizione relativa delle varie ROI alla panoramica precedentemente salvata.
NOTA: Questa cattura dello schermo sarà essenziale per sostituire le ROI nell'imaging longitudinale, poiché il software di acquisizione non consente di richiamare questo gruppo di posizioni nella versione utilizzata per questo esperimento. - Per acquisire un volume z-stack, selezionare la modalità z-stack, definire la dimensione del passo a 3 μm e deselezionare l'opzione la stessa dimensione dello stack per tutte le regioni . Seleziona la prima posizione, definisci le posizioni superiore e inferiore , controlla la dimensione del passo z e fai clic su ridefinisci pila. Viene visualizzata un'icona a forma di cubo accanto al nome della posizione nell'elenco delle posizioni. Ripeti questo passaggio per tutte le posizioni dei diversi ROI.
- Premere acquisisci e salvare le immagini (tre canali z-stack con ~50 piani) nell'apposita cartella. Questo passaggio segna Tctrl dell'esperimento; Verificare che i parametri di acquisizione siano corretti (ottenere la corretta gamma dinamica nei tre canali, evitare il più possibile la saturazione del segnale e controllare il tempo di acquisizione per un ciclo completo).
- Per misurare una caratteristica dinamica (permeabilità vascolare in questo caso), acquisire un timelapse.
- In questa configurazione, i tre rivelatori consentono l'imaging simultaneo di tre canali; se per l'esperimento è necessario un quarto canale (in questo caso il monitoraggio destrano), attivare la modalità sequenziale e aggiungere una seconda sequenza di acquisizione. Sintonizza la linea laser IR a 820 nm per poter eccitare il fluoroforo Dextran Cy5. Modificare l'intervallo di rilevamento Hyd2 a [650 - 744 nm] e disattivare gli altri rilevatori. Seleziona tra la modalità di acquisizione degli stack .
NOTA: Per impostazione predefinita, la seconda sequenza di acquisizione ha gli stessi parametri della sequenza 1. - Prima di iniziare il timelapse, avviare l'acquisizione di tutte le posizioni per assicurarsi che il tempo di registrazione non superi i 3 min (tenendo conto dell'aggiunta della modalità sequenziale), il tempo minimo necessario per rilevare la perdita di destrano.
- Prima dell'iniezione di destrano, modificare la modalità di acquisizione in xyzt. Nel modulo t, regolare l'intervallo di tempo a 3 min e la durata a 1 ora.
- In questa configurazione, i tre rivelatori consentono l'imaging simultaneo di tre canali; se per l'esperimento è necessario un quarto canale (in questo caso il monitoraggio destrano), attivare la modalità sequenziale e aggiungere una seconda sequenza di acquisizione. Sintonizza la linea laser IR a 820 nm per poter eccitare il fluoroforo Dextran Cy5. Modificare l'intervallo di rilevamento Hyd2 a [650 - 744 nm] e disattivare gli altri rilevatori. Seleziona tra la modalità di acquisizione degli stack .
- Iniettare 100 μL di destrano 70 KDa-TRITC (o 500 KDa-Cy5) a 3 mg/topo per via endovenosa e iniziare l'acquisizione.
- Dopo l'acquisizione, salvare il set di dati nella cartella appropriata.
NOTA: Durante questi numerosi passaggi, monitorare frequentemente la frequenza respiratoria e la temperatura del topo e regolare il flusso di isoflurano se necessario. Idealmente, la temperatura, l'ECG e la frequenza respiratoria potrebbero essere registrati e inclusi come metadati dell'esperimento.
4. Recupero del mouse
- Al termine della sessione di imaging, disattivare il rivelatore HyDs all'interno del software di acquisizione e accendere la scatola di riscaldamento del mouse (37 °C). Aprire la gabbia del microscopio, sollevare l'obiettivo e rimuovere la sonda rettale dal mouse. Quindi, sfilare delicatamente la coda di rondine dell'impianto di fissazione della testa fuori dal supporto di fissazione e spostare il mouse dalla maschera stereotassica del microscopio alla maschera chirurgica sul cuscinetto termico.
- Spegnere l'iniezione di isoflurano al microscopio e spegnere il cuscinetto termico.
- Rimuovere delicatamente il gel a base d'acqua o il PBS dal cranio del topo con un tampone sterile.
- Aggiungere gel intrasito per mantenere l'umidità del cranio tra le sessioni di imaging, come descritto in precedenza13.
- Chiudere l'area di imaging dell'impianto cranico con l'apposito coperchio e fissarla accuratamente con la vite.
- Metti il mouse nella scatola di riscaldamento a 37 °C e attendi che si svegli. Porta il topo alla struttura per animali e ospitalo in una gabbia pulita con idrogel e arricchimento. Monitorare gli animali ogni giorno per individuare eventuali segni di dolore o infezione, nel qual caso somministrare Buprecare ogni giorno. Per preservare l'umidità calvariare, cambiare il gel intrasite 2 volte a settimana.
NOTA: Scegli una gabbia alta con rifugi di cartone e materiale per il nido e assicurati di mettere i topi in gabbie individuali.
5. Acquisizioni longitudinali
NOTA: Il mouse può essere nuovamente ripreso nei giorni successivi. Tuttavia, assicurati di non ripetere più di tre sessioni di imaging a settimana per evitare effetti indesiderati dell'anestesia ripetuta, come secchezza oculare o affaticamento eccessivo, nonché distress respiratorio e ipotermia.
- Per ricreare l'immagine di un mouse con un supporto per la testa già installato, seguire il passaggio 2.2 e posizionare una piccola goccia di gel oftalmico sugli occhi del topo per prevenire la secchezza durante la procedura.
- Seguire la sezione 3 fino al passaggio 3.16 per ottenere l'immagine panoramica.
NOTA: L'utilizzo dell'impianto in titanio della testa senza dover modificare l'altezza o l'angolo della posizione del supporto rende molto facile trovare l'area di interesse complessiva. - Se necessario, riallineare le immagini precedenti e nuove utilizzando il modulo Apri immagine e allinea.
- Aprire l'immagine di cattura dello schermo dal passaggio 3.17 per contrassegnare le posizioni nella loro posizione originale.
NOTA: L'uso del contrasto SHG è molto utile soprattutto quando ci si aspetta un rimodellamento dei vasi sanguigni e delle cellule associate che esprimono GFP. - Acquisisci z-stack e timelapse nello stesso modo descritto nei passaggi 3.18-3.22.
6. Quantificazione dei parametri vascolari
- Parametri vascolari
- Per aprire le immagini generate con il software (file .lif) in IMARIS, convertitele prima in formato ".ims" con il convertitore di file. Apri il file ".ims" con IMARIS, controlla i metadati (scale X, Y, Z) e seleziona il modulo Filamento . Seleziona salta la creazione automatica, modifica manualmente.
- Per una migliore visualizzazione durante la generazione del filamento, vai su Impostazioni (simbolo della foglia), seleziona Linea nello stile e lascia il numero di pixel al valore predefinito (1). Seleziona le caselle Mostra dendriti, inclusi Punto iniziale, Punti di diramazione e Punti terminali. Deselezionare Mostra dorso.
- In Disegna (simbolo del pennello), selezionare Metodo AutoPath; 5 μm come diametro e la sorgente del canale associata al recipiente (in questo caso TRITC-Destrano). Infine, spuntare il centro automatico e il diametro automaticor.
- Inizia a disegnare i filamenti usando Maiusc + clic destro del mouse per scegliere un punto iniziale di ramo e Maiusc + clic sinistro per scegliere un punto finale di un ramo.
NOTA: Il punto iniziale è in azzurro, i punti di ramificazione sono in rosso o blu scuro e i punti terminali sono in verde. - Per impostare il diametro corretto del filamento durante il disegno, utilizzare il pulsante di scorrimento del mouse.
- Per unire due filamenti con punti terminali distinti, creare un nuovo punto iniziale tra di loro e collegarlo a ciascun punto finale.
NOTA: Pertanto, i punti terminali verdi vengono visualizzati nei punti di diramazione. - Una volta creato l'albero vascolare, torna alle impostazioni e assicurati che i numeri dei punti iniziali, di ramificazione e terminali siano corretti. Selezionare il cono per visualizzare i diametri e verificarne l'aspetto.
- Se il diametro di un filamento (o una connessione) non sembra corretto, vai su Modifica (simbolo della matita), seleziona il filamento da rimuovere con un clic destro e fai clic su Elimina. Ridisegna il filamento regolando il diametro sul filamento con il pulsante di scorrimento del mouse.
- Per estrarre tutti i dati statistici generati e salvarli, fare clic sull'icona Statistica | esporta tutte le statistiche su file-(icona di più floppy disk nella parte inferiore dello schermo).
NOTA: Per evitare filamenti potenzialmente aberranti che non corrispondono ai recipienti, nell'analisi statistica sono stati inclusi solo filamenti di lunghezza superiore a 20 μm.
- Quantificazione della perdita
NOTA: Per aprire le immagini generate con il software (file .lif) in IMARIS, convertirle prima in formato ".ims" con il convertitore di file.- Per unire le immagini scattate prima e dopo l'iniezione di destrano nella stessa serie temporale, vai alla modalità Surpass, fai clic sia sulla prima immagine (Tctrl , prima dell'iniezione di destrano) che sull'immagine timelapse (tempo 1, 60 min) per aprirle e combinarle facendo clic su Modifica e aggiungi punti temporali.
NOTA: Per correggere eventuali deviazioni 3D, eseguire i passaggi 6.2.2-6.2.7 utilizzando la funzione di ponte tra Fiji e IMARIS. Installa i seguenti plugin nelle Fiji: "IMARIS_Bridge96.jar" e "IMARISBridgeUtils.jar". La registrazione della deriva 3D viene eseguita utilizzando "StackReg Pluging" (già disponibile nelle Fiji). Se non è necessario, andare al passaggio 6.2.8 - Per eseguire la correzione della deriva 3D, apri Fiji per collegare le immagini da IMARIS facendo clic su Plugin | IMARIS | Immagine da IMARIS.
- Nelle Figi, seleziona Plugin | Iscrizione | Corretta la deriva 3D.
- Nelle nuove finestre visualizzate, scegli il canale appropriato per la registrazione.
NOTA: In questo caso si trattava di Nestin-GFP, ma poteva essere qualsiasi canale che evidenziasse oggetti immobili e un elevato rapporto segnale/sfondo. - Per migliorare il rilevamento di piccole deviazioni, selezionare Calcolo multiscala temporale, Correzione deviazione pixel secondari e Migliora immagini dai bordi e lasciare le altre impostazioni predefinite.
- Per collegare l'immagine corretta a IMARIS, selezionarla e fare clic su Plugin | IMARIS | Immagine per IMARIS.
- Salvarlo nella cartella appropriata facendo clic su File | Salva con nome.
- Per visualizzare tutti i canali acquisiti, clicca su Modifica | Mostra la regolazione del display. Selezionare il canale del destrano e il tempo 1 (fotogramma 2 nella parte inferiore dello schermo).
- Creare una nuova superficie che definisca il lume vascolare al punto temporale 1 selezionando la casella di controllo Aggiungi nuova superficie (oggetto blu)
- In Surface 1, vai su Proprietà e deseleziona tutti i parametri in Impostazioni algoritmo. Fare clic sul passaggio successivo (freccia blu)
- Selezionare il canale vascolare (destrano in questo caso) sotto il canale sorgente e spuntare Sottrazione uniforme e sfondo. Lasciare tutti gli altri parametri ai valori predefiniti. Fare clic sul passaggio successivo (freccia blu).
- Regolare il valore di Soglia in base all'aspetto della superficie in termini di spessore e copertura di tutte le aree positive. Fare clic sul passaggio successivo (freccia blu).
- Escludi le superfici non specifiche (per lo più piccoli punti) utilizzando il filtro per impostazione predefinita Numero di Voxel. Fare clic sul passaggio finale (freccia verde).
- Se necessario, continuare a regolare manualmente la superficie facendo clic su modifica (icona a forma di matita) in Impostazione parametri. Per selezionare un oggetto da rimuovere, fare clic con il pulsante sinistro del mouse su di esso ed eliminarlo selezionando Elimina. Se l'oggetto da eliminare viene fascicolato con un altro, scollegare le due parti separate della superficie facendo clic su Maiusc + clic a sinistra tra le due e selezionando Taglia superficie. Ripetere questo passaggio tutte le volte che è necessario.
- Infine, combina tutti i segmenti utilizzando la modalità di selezione (icona del bersaglio a destra) e premi Ctrl + Scorri per recuperare tutti gli oggetti. Quando tutti sono selezionati, fare clic con il pulsante sinistro del mouse e fare clic su Unisci.
- Per generare la superficie di riferimento, copiatela negli altri punti temporali. Selezionare la superficie globale e duplicarla facendo clic su Duplica e rinominarla come Intravascolare.
- Selezionare la superficie Intravascolare e fare clic su Duplica in tutti i punti temporali.
- Dividere il segnale del destrano per distinguere le aree intra (IN) ed extravascolari (OUT). Per creare un canale IN per tutti i punti temporali, fare clic sulla superficie Intravascular | Modifica (pulsante matita) | Maschera Tutto.
- In Selezione canale, selezionare il canale appropriato e selezionare Duplica canale prima di applicare la maschera. In Impostazioni maschera, selezionare solo Costante interno/esterno con la superficie esterna dei voxel impostata su 0. Infine, spunta anche Applica a tutti i punti temporali.
- In Regolazione visualizzazione, per impostazione predefinita viene creato un nuovo canale con il nome mascherato. Rinominalo IN.
- Per creare il canale OUT per tutti i punti temporali, ripetere i passaggi 6.2.18-6.2.20, ma selezionare Imposta voxel all'interno della superficie su 0. Rinominare questo nuovo canale OUT.
- Per estrarre i dati statistici, fare clic sull'icona Statistica | Esporta tutte le statistiche su file (icona di più dischi floppy nella parte inferiore dello schermo)
- Per analizzare la perdita, dividere il canale della somma dell'intensità all'esterno per il canale della somma dell'intensità all'interno per ogni punto temporale.
- Per unire le immagini scattate prima e dopo l'iniezione di destrano nella stessa serie temporale, vai alla modalità Surpass, fai clic sia sulla prima immagine (Tctrl , prima dell'iniezione di destrano) che sull'immagine timelapse (tempo 1, 60 min) per aprirle e combinarle facendo clic su Modifica e aggiungi punti temporali.
Risultati
Nella Figura 1 e nella Figura 2, viene mostrato il modello CAD di un impianto di fissazione della testa in titanio posizionato su un cranio di topo scansionato, progettato per seguire la struttura anatomica del cranio e fornire un dispositivo leggero e biocompatibile in grado di aderire saldamente allo stadio del microscopio garantendo stabilità a livello cellulare. Seguendo questo protocollo passo-passo, l'impianto è fissato stabilmente al cranio del topo e può essere fissato saldamente al supporto del microscopio tramite la sua coda di rondine, consentendo un'area di imaging piatta per la ritenzione di liquidi e l'osservazione intravitale nel tempo. Può essere chiuso con un coperchio per ridurre al minimo eventuali danni o infezioni della ferita, consentendo l'imaging ripetuto della stessa area di tessuto per settimane. Una volta sveglio, il topo che indossa un impianto per la testa può camminare liberamente, nutrirsi e avere una routine regolare.
La Figura 3 mostra una vista a scansione a piastrelle del sistema vascolare del midollo osseo calvariale costituito da capillari eterogenei, tra cui arteriole, capillari di transizione e sinusoidi. I vasi sono incorporati in un complesso microambiente tissutale a stretto contatto con la superficie ossea e le cellule mesenchimali perivascolari. Durante lo sviluppo della leucemia, singole cellule leucemiche isolate possono essere rilevate all'interno del microambiente midollare in prossimità dei vasi sanguigni e il loro attecchimento aumenta nel tempo, riempiendo la calvaria nelle fasi avanzate della malattia.
La Figura 4 mostra come le immagini ottenute con questo protocollo possano fornire dati quantitativi, che possono essere analizzati con metodi statistici. Mostriamo come segmentare i vasi con lo strumento a filamento IMARIS e misuriamo la lunghezza e il diametro dei frammenti vascolari, nonché la loro rettilineità. È inoltre possibile valutare la correlazione di questi parametri.
La Figura 5 mostra l'acquisizione di imaging longitudinale di due diverse posizioni del midollo osseo calvario durante la progressione della LMA ai giorni 4, 7 e 10, con il giorno 10 associato a un attecchimento del ~50% del midollo osseo con cellule leucemiche, come misurato tramite citometria a flusso (non mostrata). Possiamo osservare un importante rimodellamento delle dimensioni dei vasi preesistenti, nonché la formazione di nuovi vasi in aree specifiche associate alla perdita ossea locale.
Infine, nella Figura 6, mostriamo come la permeabilità vascolare possa essere misurata come parametro dinamico con l'imaging time lapse che mostra la capacità di diverse barriere vascolari di trattenere un colorante fluorescente nel tempo.

Figura 1: Progettazione e produzione di un portatestina biocompatibile a base di titanio. (A) Parti dell'impianto in situ: 1 anello di osservazione, 2 elemento di cementazione, 3 ancoraggi stabilizzanti, 4 coda, 5 coda di rondine, 6 fori filettati, 7 Bregma. (B) Collegamento dell'impianto della testa al supporto: 8 corpi di fissaggio, 9 morsetti, 10 leva eccentrica, 11 struttura, 12 obiettivo del microscopio. (C) Deformazione dell'impianto rispetto al carico mediante simulazione FEM in cui lo spostamento massimo è di 0,23 μm contro una forza di 0,04 N. (D) Coperchio di protezione e relativa vite. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Preparazione del topo per l'imaging intravitale. (A) Vista dell'impianto della testa e dell'area di imaging esposta chirurgicamente prima dell'imaging. (B) Impianto della testa saldamente attaccato al cranio del topo. (C) Topo sveglio nella gabbia di recupero con il coperchio chiuso sull'impianto della testa. Clicca qui per visualizzare una versione più grande di questa figura.
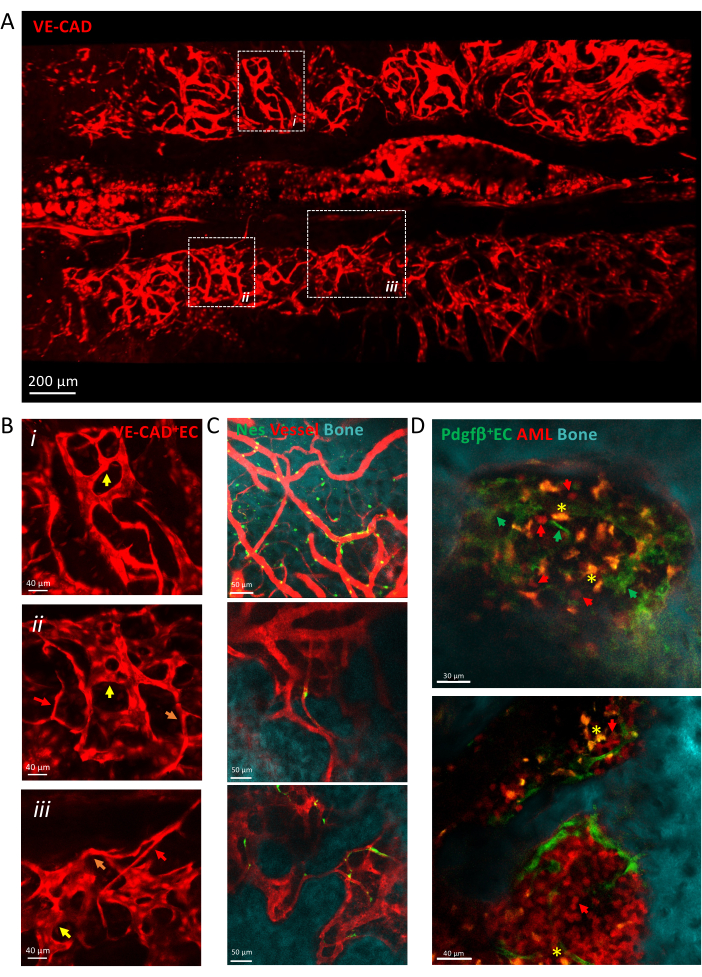
Figura 3: Imaging intravitale della vascolarizzazione della calvaria. (A) Proiezione z della vista a scansione a piastrelle della vascolarizzazione della calvaria etichettata da cdh5-DSRED. (B) Ingrandisci le aree raffigurate per descrivere diversi tipi di vasi raffigurati da frecce, arteriole da frecce rosse, capillari di transizione da frecce arancioni, sinusoidi da frecce gialle. (i) e (ii) proiezioni z di tessuto X μm; (iii) singola fetta. (C) Singole fette di diversi campi visivi di vasi BM, che mostrano la superficie ossea (SHG), le cellule perivascolari (nes-GFP+) e il lume vascolare (destrano-TRITC). (D) Nicchia vascolare associata alla progressione della LMA. Sezioni rappresentative dei punti temporali iniziali (in alto) e tardivi (in basso) dello sviluppo dell'AML. La leucemia MLL-AF9 è marcata con tdTOMATO (frecce rosse), mentre i vasi sono marcati con pdgfb-GFP (frecce verdi), la superficie ossea con SHG e i macrofagi in giallo (autofluorescenza, asterisco giallo). Barre di scala = 200 μm (A), 40 μm (B,D-pannello inferiore), 50 μm (C). Abbreviazioni: BM = midollo osseo; GFP = proteina fluorescente verde; LMA = leucemia mieloide acuta; EC= Cellule endoteliali. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Quantificazione dei parametri vascolari. (A) Misurazione dei parametri vascolari tramite lo strumento del filamento IMARIS in una proiezione z rappresentativa di vasi del midollo osseo marcati con TRITC-destrano. Vengono mostrate le rappresentazioni lineari e a cono. (B) Quantificazione dei parametri della nave nell'immagine mostrata in A. (C) Correlazione tra parametri vascolari che mostrano una correlazione opposta tra rettilineità e lunghezza del vaso (negativo, Spearman r = -3523; p < 0,0001; R2 = 0,2102) vs diametro (positivo; Lanciere r = 0,4110; p < 0,0001; R2 = 0,1299). Barre di scala = 100 μm. Clicca qui per visualizzare una versione più grande di questa figura.
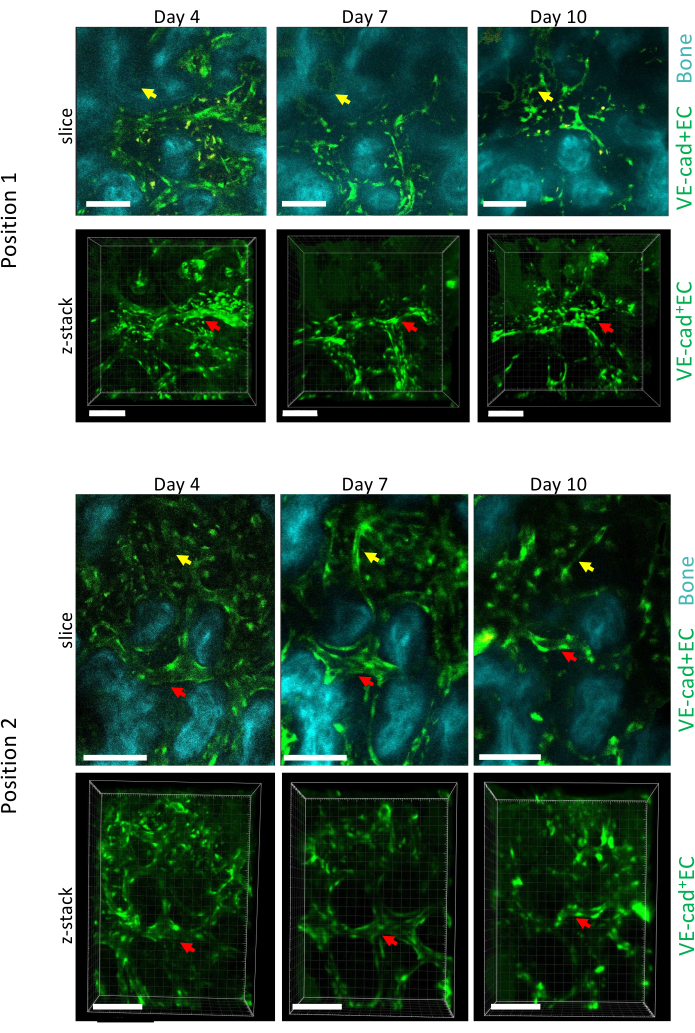
Figura 5: Imaging longitudinale di due diverse posizioni del midollo osseo della calvaria sullo sviluppo di LMA. Le cellule endoteliali che rivestono i vasi sono marcate con cdh5-GFP, la superficie ossea con SHG e i macrofagi in giallo (autofluorescenza). Vengono mostrati il rimodellamento di vasi preesistenti (frecce rosse) e la formazione di nuovi vasi (frecce gialle). Barre di scala = 100 μm. Clicca qui per visualizzare una versione più grande di questa figura.
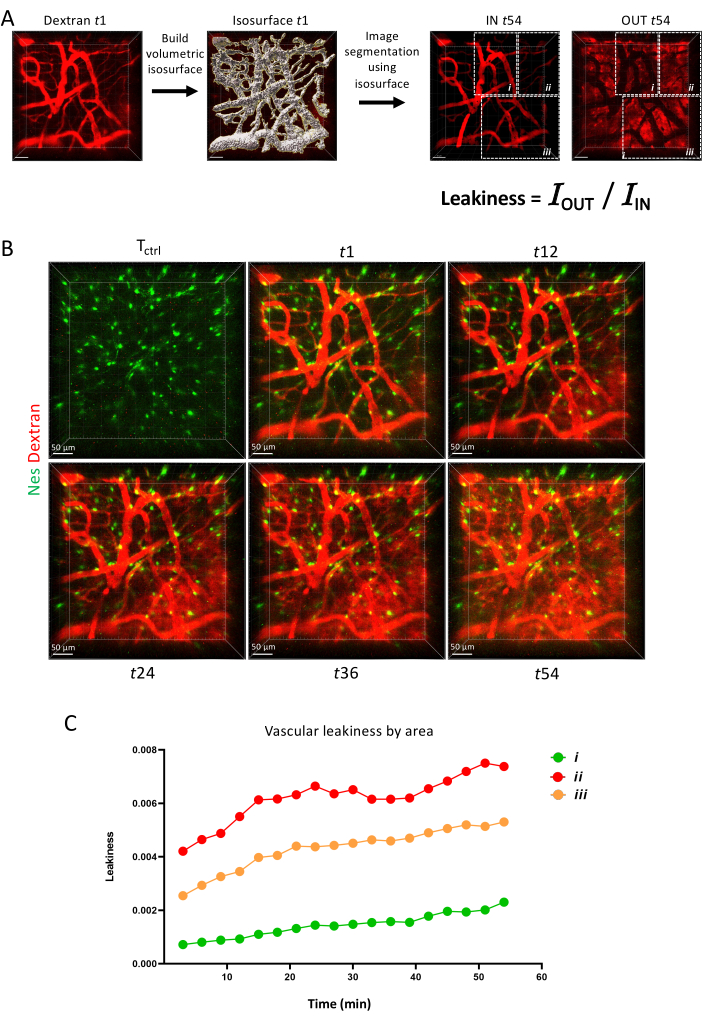
Figura 6: Permeabilità vascolare. (A) Schemi della misurazione della permeabilità vascolare tramite lo strumento di superficie IMARIS. (B) Proiezione Z della stessa area ripresa longitudinalmente nell'arco di 1 ora. (C) Quantificazione della permeabilità vascolare all'interno delle aree come illustrato in A. Clicca qui per visualizzare una versione più grande di questa figura.
Figura supplementare S1: Cranio preparato. Nel piano mediano, creare un asse tangente alla calvaria, quindi salvare il cranio preparato. Clicca qui per scaricare questo file.
Figura supplementare S2: Piani distanziati di 2 mm. Come creare una serie di piani equidistanti (spaziatura di 2 mm) attraverso il cranio. Clicca qui per scaricare questo file.
Figura supplementare S3: Contorno della superficie osservabile. Create uno schizzo nel piano calvariale e create una spline a forma di pera da AP +6,5 a -2, 6 mm di larghezza a AP 0,0. Clicca qui per scaricare questo file.
Figura supplementare S4: Osservazione vindow. Crea uno schizzo nel piano calvariale e disegna una forma a C spessa 0,5 mm che si collega alla finestra di osservazione . Clicca qui per scaricare questo file.
Figura supplementare S5: Fare clic su un nuovo studio. Vai alla scheda Simulazione (se visibile) o vai a Simulazione | Studiare. Nella finestra di dialogo Studio , scegliere Statico come tipo di studio. Fare clic su OK per creare il nuovo studio. Clicca qui per scaricare questo file.
Figura supplementare S6: Fare clic con il pulsante destro del mouse per creare una mesh. Rifinisci la mesh nelle aree in cui sono previste sollecitazioni o deformazioni elevate. Clicca qui per scaricare questo file.
Discussione
Il principale punto di forza della microscopia intravitale è la sua capacità di catturare processi cellulari dinamici in tempo reale all'interno del loro ambiente nativo, superando i limiti dei tradizionali metodi di imaging istologico ed ex vivo. Osservando direttamente il midollo osseo calvariale utilizzando le tecniche di imaging avanzate accoppiate con la marcatura fluorescente come descritto in questo protocollo, i ricercatori possono studiare non solo i parametri funzionali vascolari 8,14, ma anche i processi longitudinali come l'attecchimento e la migrazione delle cellule leucemiche 6,15, la proliferazione e l'attività metabolica16,17 preservando la rilevanza fisiologica ed evitando artefatti associati alla fissazione e alla lavorazione dei tessuti.
I vantaggi dell'imaging intravitale del calvario BM rispetto ai siti ossei alternativi includono la facile accessibilità, la trasparenza dell'osso del cranio che consente una migliore visualizzazione senza danni ossei, la stabilità dell'osso calvario che riduce al minimo gli artefatti di movimento dei tessuti e la riproducibilità tra gli animali da esperimento che facilita il confronto tra diversi gruppi sperimentali. La possibilità di recuperare gli animali ed eseguire l'imaging longitudinale consente inoltre di ridurre il numero di animali utilizzati in uno studio. Vale la pena ricordare che, poiché sono state osservate differenze specifiche del ceppo nei modelli di crescita dei vasi cranici e nella conseguente osteogenesi18, è importante tenerne conto quando si progetta l'impianto di imaging specifico per il ceppo di topo desiderato per consentire una perfetta compatibilità anatomica e l'accesso al sito di imaging desiderato.
Un altro punto importante da menzionare è la potenziale differenza nelle proprietà vascolari ed ematopoietiche all'interno del midollo osseo calvariale rispetto ad altre ossa, una questione poco esplorata fino a pochi anni fa. Nuovi studi suggeriscono funzioni localizzate per diverse ossa, con il midollo osseo calvario che differisce dalle altre ossa in termini di emopoiesi, struttura ossea e vascolare e funzione 19,20,21,22,23, nonché risposta a patologie neurologiche24. Queste differenze devono essere ulteriormente esplorate e prese in considerazione prima di generalizzare i risultati specifici della calvaria.
La progettazione e la costruzione di un impianto di imaging è un passo fondamentale per questa configurazione sperimentale, a partire dalla scelta del materiale. Gli impianti biocompatibili svolgono un ruolo fondamentale nella ricerca biomedica, facilitando un'ampia gamma di applicazioni che vanno dall'ingegneria tissutale e la medicina rigenerativa ai sistemi di somministrazione di farmaci e ai dispositivi di monitoraggio in vivo . La scelta del materiale per un impianto di testa adattato per l'imaging intravitale è fondamentale. Idealmente, il materiale dovrebbe mostrare un'eccellente biocompatibilità, proprietà meccaniche adatte a garantire una flessibilità minima e stabilità complessiva e, infine, la capacità di integrarsi all'interno del cranio senza indurre infiammazione. Il titanio è ideale per la sua buona tollerabilità all'interno del corpo animale, senza induzione di reazioni avverse a contatto con i tessuti biologici, nonché per la sua resistenza alla corrosione, garantendo stabilità a lungo termine anche se esposto a fluidi corporei o soluzioni sperimentali. Inoltre, la sua resistenza meccanica lo rende resistente alla deformazione e alla frattura. Nonostante la sua resistenza, il titanio ha una densità relativamente bassa, il che si traduce in dispositivi di imaging intravitali leggeri che riducono al minimo il carico su animali da esperimento e ricercatori. Infine, la sua versatilità nella fabbricazione consente la personalizzazione dei dispositivi di imaging intravitale per soddisfare specifici requisiti sperimentali, come dimensioni, forma e funzionalità.
In questo caso, abbiamo progettato un impianto di fissazione della testa biocompatibile utilizzando un software CAD di modellazione parametrica con funzionalità di analisi degli elementi finiti (FEA) integrate, in particolare SolidWorks. Questo approccio consente regolazioni precise e iterative dei requisiti strutturali e spaziali dell'impianto, garantendo sia la compatibilità anatomica che la resilienza meccanica. Le alternative gratuite, come FreeCAD, offrono funzionalità di modellazione e simulazione simili. Gli input primari per il processo di progettazione includono un modello anatomico ad alta risoluzione del cranio del topo, un modello dell'obiettivo del microscopio e un modello del sistema di fissaggio, che insieme informano le dimensioni e la configurazione dell'impianto. L'output finale è un file mesh indipendente dalla piattaforma, tipicamente in formato STL o STEP, compatibile con la maggior parte dei software di stampa 3D.
La fase iniziale prevede l'acquisizione di caratteristiche anatomiche dettagliate della testa del topo utilizzando tecniche di imaging ad alta risoluzione, come la scansione micro-CT in vivo . Questo approccio fornisce il massimo dettaglio anatomico e precisione, catturando le microstrutture del cranio. Anche la scansione 3D di un teschio nudo preparato potrebbe essere un'opzione. Questo metodo, che utilizza laser o scanner a luce strutturata, è comunemente impiegato su un cranio preparato e fornisce contorni superficiali accurati, anche se con meno dettagli interni rispetto alla micro-CT. In caso contrario, i modelli anatomici possono essere scaricati da pubblicazioni e database open source25 o DigiMorph {https://www.digimorph.org/specimens/Mus_musculus/}. Sebbene convenienti, questi modelli potrebbero non avere dettagli specifici per il campione, quindi sono spesso necessari aggiustamenti per l'animale specifico nello studio, come il ridimensionamento alla distanza effettiva di Bregma-Lambda. I dati acquisiti vengono quindi utilizzati per creare un modello 3D preciso del cranio del topo, che funge da modello di base per la progettazione dell'impianto.
Per fissare l'impianto sul cranio, è stata progettata una struttura cementante per coprire la superficie cranica rimanente non occupata dalla finestra di osservazione. Questa struttura deve fornire punti di attacco robusti evitando caratteristiche anatomiche critiche. La struttura cementante presenta molteplici aperture per garantire una polimerizzazione ottimale del cemento sotto l'impianto attraverso la diffusione. Inoltre, le pareti della struttura cementativa hanno un piccolo angolo di sformo, che consente al cemento di ancorarsi saldamente contro queste pareti angolate. La coda dell'impianto, che si estende dal corpo principale al sistema di fissaggio, è progettata. Questo componente è fondamentale per l'allineamento e la stabilizzazione dell'impianto durante l'osservazione e il suo design deve considerare lo spazio disponibile e i vincoli anatomici della testa del topo. Infine, un meccanismo a coda di rondine è integrato nel design dell'impianto per facilitare l'attacco e il distacco dell'impianto dal sistema di fissaggio. Questa caratteristica migliora la praticità e l'usabilità dell'impianto durante le osservazioni ripetute. Il meccanismo a coda di rondine fornisce una fissazione ripetibile per un facile recupero dei tessuti osservati per sessioni di imaging ripetute.
I lettori che stanno valutando l'adozione dell'imaging al microscopio intravitale del midollo osseo calvariale dovrebbero valutare attentamente i loro obiettivi di ricerca e i requisiti sperimentali per determinare se questo metodo è appropriato per i loro studi. Sebbene la microscopia intravitale offra informazioni senza precedenti sull'emopoiesi, sulla regolazione del microambiente tissutale e sulle dinamiche vascolari in vivo, presenta anche alcune sfide e limitazioni tecniche. I ricercatori dovrebbero essere preparati ad affrontare queste sfide attraverso un'attenta progettazione sperimentale, l'ottimizzazione dei parametri di imaging e l'utilizzo di controlli appropriati. Inoltre, i ricercatori dovrebbero considerare la disponibilità di apparecchiature di imaging specializzate, competenze nelle tecniche di etichettatura fluorescente e risorse computazionali per l'analisi delle immagini. Nel complesso, l'imaging al microscopio intravitale del midollo osseo calvariale ha un enorme potenziale per far progredire la nostra comprensione dell'emopoiesi e della biologia vascolare, offrendo una finestra unica sui processi dinamici che si verificano all'interno del microambiente del midollo osseo.
Divulgazioni
Jozsua Fodor è il fondatore della società YMETRY (numero di registro 888312352). Gli altri autori non hanno conflitti di interesse.
Riconoscimenti
Gli autori ringraziano tutto il personale dell'IMAG'IC e delle strutture per animali dell'Institut Cochin per il loro supporto negli esperimenti di microscopia e nell'alloggiamento dei topi. I topi Tg(Nes-EGFP)33Enik e Tg(Pdgfb-icre/ERT2)1Frut sono stati un gentile dono del Dr. Bonnet (The Francis Crick Institute, Londra). Tg(Cdh5-cre/ERT2)1Rha e B6. I topi Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze/J sono stati un gentile dono del dottor Rafii (Weill Cornell Medicine, New York). Il lavoro descritto è stato sostenuto dal CNRS, dall'INSERM e dall'Université de Paris Cité, e da sovvenzioni di ATIP-AVENIR, Fondation ARC pour la recherche sur le cancer (R19084KS - RSE20008KSA), Ville de Paris "Emergence" (R20192KK - RPH20192KKA), Laurette Fugain (R23197KK), Cancéropôle IDF (RPH23177KKA), INCA PLBIO (RPH21162KKA), Fondation de France (RAF23152KKA), Ligue contre le cancer (282273/807251), Institut du cancer Paris Carpem, Associazione europea di ematologia (RAK23130KKA) e Consiglio europeo della ricerca ERC-STG (EEA24092KKA). La struttura principale di IMAG'IC è supportata dalla National Infrastructure France BioImaging (sovvenzione ANR-10-INBS-04). Il laboratorio Passaro è affiliato all'"Institut Hors Murs des Sciences Cardiovasculaires" e all'"Istituto per la Leucemia Paris Saint-Louis".
Materiali
| Name | Company | Catalog Number | Comments |
| Amira | Thermo ScientificTM | ||
| Anesthesia | Isoflurane 2% to 3% | ||
| Anesthesic mask (animal detection) | Minerve | ||
| Anesthesic unit | Minerve | ||
| B6.Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze | Jackson laboratories | MGI:3809523 | |
| CalvariaVue | Ymetry | https://ymetry.com/site/head-fixation-implants/44-mouse-head-fixation-implant-for-calvaria.html | |
| Dental cement | Kemdent | SUN527 | |
| Dextran 500 kDa-Cy5 | Tebu-bio | DX500-S5-1 | 3 mg/mouse |
| Dextran 70 kDa-TRITC | Sigma | T1162 | 3 mg/mouse |
| Disinfectant | MP-Labo | Dermidine-60ml | |
| Electric razor | Aescular | Isis | |
| Eye gel | Ocry-gel | 10g | |
| Fiji | https://imagej.net/software/fiji/downloads | v 17 May 30 | |
| Fiji plugings to bridge with Imaris | https://imagej.net/software/fiji/downloads | Imaris_Bridge96.jar ; ImarisBridgeUtils.jar | |
| Heating box | Datesand | Thermacage | |
| Heating pad for surgery | Minerve | ||
| Imaging heating pad & rectal probe | F. Haer | ||
| Imaris v9.6.0 | Oxford instruments | ||
| Intrasite gel | Chinoxia | 2390766 | |
| LAS AF Software | Leica | LAS X 3.5.7.23225 | |
| Medication | Buprecare, 0.01 mg/kg | ||
| Objective HCX IRAPO L 25x/0.95 WATER | Leica | 506374 | |
| Saline buffer (PBS 1x) | Sigma | P4417 | Sterilize by autoclave |
| SP8 DIVE FALCON Multiphoton Microscope | Leica | ||
| Stereotoxic mask | Minerve | 1201261 | |
| Sterilizator beads | Sigma | Z742555 | |
| Surgery tools | Moria | 4877A; 2183 | |
| Survival blanket | SECURIMED | 11006 | |
| Swabs / Tissues | Sterilize by autoclave | ||
| Syringe 1 mL 26 G | BD Plastipak | 305501 | |
| Temperature controller | F. Haer | 40-90-5D-02 | |
| Tg(Nes-EGFP)33Enik mice | Jackson laboratories | MGI:5523870 | |
| Tg(Pdgfb-icre/ERT2)1Frut mice | Jackson laboratories | MGI:3793852 | |
| Tg(Cdh5-cre/ERT2)1Rha mice | Jackson laboratories | MGI:3848982 | |
| Ultrasound gel | Parker laboratories | Aquasonic 100 |
Riferimenti
- Lo Celso, C., et al. Live-animal tracking of individual haematopoietic stem/progenitor cells in their niche. Nature. 457 (7225), 92-96 (2009).
- Pittet, M. J., Weissleder, R. Intravital imaging. Cell. 147 (5), 983-991 (2011).
- Itkin, T., et al. Distinct bone marrow blood vessels differentially regulate haematopoiesis. Nature. 532 (7599), 323-328 (2016).
- Christodoulou, C., et al. Live-animal imaging of native haematopoietic stem and progenitor cells. Nature. 578 (7794), 278-283 (2020).
- Upadhaya, S., et al. Intravital imaging reveals motility of adult hematopoietic stem cells in the bone marrow niche. Cell Stem Cell. 27 (2), 336-345.e4 (2020).
- Hawkins, E. D., et al. T-cell acute leukaemia exhibits dynamic interactions with bone marrow microenvironments. Nature. 538 (7626), 518-522 (2016).
- Duarte, D., et al. Defining the in vivo characteristics of acute myeloid leukemia cells behavior by intravital imaging. Immunol Cell Biol. 97 (2), 229-235 (2019).
- Passaro, D., et al. Increased vascular permeability in the bone marrow microenvironment contributes to disease progression and drug response in acute myeloid leukemia. Cancer Cell. 32 (3), 324-341.e6 (2017).
- Jia, Y., et al. FLT3 inhibitors upregulate CXCR4 and E-selectin ligands via ERK suppression in AML cells and CXCR4/E-selectin inhibition enhances anti-leukemia efficacy of FLT3-targeted therapy in AML. Leukemia. 37 (6), 1379-1383 (2023).
- Pendleton, E. G., Tehrani, K. F., Barrow, R. P., Mortensen, L. J. Second harmonic generation characterization of collagen in whole bone. Biomed Opt Express. 11 (8), 4379-4396 (2020).
- Ventalon, C., Bourdieu, L., Leger, J. F., Fodor, J. Dispositif de liaison d'un animal de laboratoire à au moins un système d'expérimentation, et procédé de fixation d'un tel dispositif. France patent. , (2021).
- Horton, S. J., et al. Acute myeloid leukemia induced by MLL-ENL is cured by oncogene ablation despite acquisition of complex genetic abnormalities. Blood. 113 (20), 4922-4929 (2009).
- Scott, M. K., Akinduro, O., Lo Celso, C. In vivo 4-dimensional tracking of hematopoietic stem and progenitor cells in adult mouse calvarial bone marrow. J Vis Exp. (91), e51683 (2014).
- Jung, Y., et al. Intravital imaging of mouse bone marrow: Hemodynamics and vascular permeability. Methods Mol Biol. 1763, 11-22 (2018).
- Le, V. H., et al. In vivo longitudinal visualization of bone marrow engraftment process in mouse calvaria using two-photon microscopy. Sci Rep. 7, 44097 (2017).
- Spencer, J. A., et al. Direct measurement of local oxygen concentration in the bone marrow of live animals. Nature. 508 (7495), 269-273 (2014).
- Yang, M., Mahanty, A., Jin, C., Wong, A. N. N., Yoo, J. S. Label-free metabolic imaging for sensitive and robust monitoring of anti-CD47 immunotherapy response in triple-negative breast cancer. J Immunother Cancer. 10 (9), e005199 (2022).
- Li, W., et al. Tracking strain-specific morphogenesis and angiogenesis of murine calvaria with large-scale optoacoustic and Ultrasound Microscopy. J Bone Miner Res. 37 (5), 1032-1043 (2022).
- Lassailly, F., Foster, K., Lopez-Onieva, L., Currie, E., Bonnet, D. Multimodal imaging reveals structural and functional heterogeneity in different bone marrow compartments: functional implications on hematopoietic stem cells. Blood. 122 (10), 1730-1740 (2013).
- Rindone, A. N., et al. Quantitative 3D imaging of the cranial microvascular environment at single-cell resolution. Nat Commun. 12 (1), 6219 (2021).
- Mills, W. A., Coburn, M. A., Eyo, U. B. The emergence of the calvarial hematopoietic niche in health and disease. Immunol Rev. 311 (1), 26-38 (2022).
- Bixel, M. G., et al. Angiogenesis is uncoupled from osteogenesis during calvarial bone regeneration. Nat Commun. 15 (1), 4575 (2024).
- Koh, B. I., et al. Adult skull bone marrow is an expanding and resilient haematopoietic reservoir. Nature. 636 (8041), 172-181 (2024).
- Kolabas, Z. I., et al. Distinct molecular profiles of skull bone marrow in health and neurological disorders. Cell. 186 (17), 3706-3725.e9 (2023).
- Rosenhain, S., et al. A preclinical micro-computed tomography database including 3D whole body organ segmentations. Sci Data. 5, 180294 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon