Method Article
Imagerie longitudinale intravitale de la dynamique vasculaire dans la moelle osseuse calvaire
Dans cet article
Résumé
La microscopie intravitale permet d’étudier des processus biologiques dynamiques tels que la régénération tissulaire et le développement tumorale. La moelle osseuse calvaire, un tissu très dynamique, offre des informations sur l’hématopoïèse et la fonction vasculaire. L’utilisation d’un implant de fixation de la tête biocompatible imprimé en 3D permet une imagerie longitudinale répétitive, améliorant ainsi notre compréhension de la dynamique des tissus et du microenvironnement tumoral.
Résumé
L’imagerie par microscopie à fluorescence longitudinale intravitale est apparue comme une technique cruciale pour l’étude des processus biologiques dynamiques, notamment dans le contexte de la régénération tissulaire, du développement tumoral et des réponses thérapeutiques. En particulier, la moelle osseuse calvariale est un tissu très dynamique, où le destin hématopoïétique est interconnecté avec le microenvironnement environnant, avec des vaisseaux spécialisés répondant à l’hématopoïèse normale et pathologique. L’imagerie traditionnelle des tissus fixés offre des informations statiques, ce qui limite souvent une compréhension complète de ces processus.
L’intégration d’animaux transgéniques exprimant des marqueurs spécifiques aux cellules, des traceurs de cellules vivantes, les progrès de l’équipement d’imagerie et l’utilisation de chambres spécialisées ont élevé la microscopie intravitale au rang d’outil essentiel pour mieux comprendre les phénomènes biologiques dynamiques. L’une des applications de l’imagerie intravitale est l’étude du comportement des vaisseaux tumoraux et des effets thérapeutiques. Un nouvel implant de fixation de la tête en titane imprimé en 3D peut être connecté de manière stable au crâne de la souris et convient à l’imagerie longitudinale pendant plusieurs séances.
Le protocole proposé permet l’examen spatial et temporel de la dynamique vasculaire dans la moelle osseuse calvaire, y compris la visualisation et la quantification de l’hétérogénéité vasculaire, l’interaction avec les cellules stromales et hématopoïétiques, et la mesure des paramètres fonctionnels vasculaires. De plus, la technique permet de visualiser les lits vasculaires établis et de surveiller les effets thérapeutiques, la mobilisation des cellules souches et la localisation des composés chimiothérapeutiques au fil du temps à l’aide de la microscopie à deux photons. Dans l’ensemble, ce protocole d’imagerie longitudinale intravitale fournit une plate-forme complète pour étudier à la fois le comportement des vaisseaux tumoraux et la dynamique des cellules hématopoïétiques, offrant des informations précieuses sur les processus complexes régissant ces phénomènes biologiques.
Introduction
L’imagerie par microscopie intravitale de la moelle osseuse calvaire (BM) est une technique puissante et indispensable pour étudier les processus dynamiques de l’hématopoïèse, la régulation du microenvironnement tissulaire et la dynamique vasculaire au sein de la niche BM. L’objectif principal de cette approche méthodologique est de permettre la visualisation et l’analyse en temps réel des comportements, des interactions et de l’organisation spatiale cellulaires au sein du microenvironnement BM in vivo. En observant directement la calvaria BM à l’aide de techniques d’imagerie avancées couplées à un marquage fluorescent, les chercheurs peuvent élucider l’interaction complexe entre les cellules souches hématopoïétiques (CSH), les cellules stromales et le système vasculaire environnant, fournissant ainsi des informations cruciales sur la régulation de l’hématopoïèse et des réponses immunitaires.
Le développement et l’utilisation des cellules souches d’imagerie par microscopie intravitale à partir des limites des méthodes d’imagerie histologique et ex vivo traditionnelles, qui ne parviennent souvent pas à capturer la nature dynamique des comportements cellulaires et des interactions d’un tissu. Contrairement aux techniques d’imagerie statique, la microscopie intravitale permet aux chercheurs d’observer la dynamique cellulaire en temps réel, ce qui permet d’étudier les processus longitudinaux tels que la migration, la prolifération et la différenciation cellulaires au sein de leur niche d’origine, sans sacrifier les animaux de laboratoire. De plus, la microscopie intravitale offre l’avantage unique d’étudier les comportements fonctionnels in vivo, tels que la fonctionnalité vasculaire (par exemple, la perfusion, la perméabilité, l’hypoxie), préservant ainsi la pertinence physiologique et évitant les artefacts associés à la fixation et au traitement des tissus. Des études pionnières dans le domaine ont démontré les énormes avantages de cette approche 1,2, et leurs résultats ont été corroborés et élargis par des approches plus récemment affinées 3,4,5 qui ont utilisé la microscopie intravitale pour suivre la localisation, la migration et les interactions des CSH endogènes avec le système vasculaire dans la niche de la BM. De plus, la microscopie intravitale a joué un rôle déterminant dans l’élucidation des mécanismes sous-jacents aux troubles hématopoïétiques, tels que la leucémie et les syndromes d’échec de la BM, offrant de nouvelles perspectives sur la motilité des cellules leucémiques 6,7, les implications vasculaires associées à la maladie 8 et la réponse aux médicaments9.
L’imagerie par microscopie intravitale de la couverture osseuse calvaire présente plusieurs avantages. Tout d’abord, le BM contenu dans l’os du crâne offre un accès facile pour l’imagerie intravitale par rapport aux os plus profonds tels que le fémur ou le tibia. Cette accessibilité facilite l’observation directe du microenvironnement tissulaire, y compris l’os lui-même, via la visualisation de la génération de secondes harmoniques (SHG)10, sans avoir besoin d’interventions chirurgicales invasives. Deuxièmement, le crâne est relativement mince et translucide, ce qui permet une meilleure visualisation de la dynamique cellulaire dans la niche BM. Cette transparence facilite l’imagerie haute résolution avec la microscopie à deux photons, la microscopie confocale et la microscopie à feuillet de lumière, sans avoir besoin de techniques d’amincissement ou de nettoyage des os. L’os calvaire offre une plate-forme stable, plate et rigide pour les expériences d’imagerie intravitale, minimisant les artefacts de mouvement des tissus et assurant des conditions d’imagerie constantes sur des périodes d’observation prolongées. Cette stabilité est particulièrement avantageuse pour les études longitudinales suivant les comportements et les réponses cellulaires au fil du temps. La reproductibilité est un autre grand avantage, étant donné la structure relativement petite et spatialement définie de l’os calvaire chez les animaux de laboratoire. Cette uniformité facilite la comparaison entre différents groupes expérimentaux et permet une analyse statistique robuste des données d’imagerie.
Nous décrivons ici une méthode d’imagerie de la BM calvaire de souris via la microscopie intravitale à deux photons en introduisant un nouvel implant de fixation de la tête11, imprimé en 3D à l’aide d’un alliage de titane biocompatible de grade 23 (Ti6Al4V), qui comporte une couverture rigide dédiée et facilement positionnée dans le même alliage de titane, permettant une fermeture sûre de la plaie pour éviter les infections ou les dommages au site de la chirurgie. L’implant fixe fermement et de manière stable la tête de souris à la platine du microscope via un porte-implant, minimisant ainsi les artefacts respiratoires et permettant une imagerie longitudinale des mêmes zones au fil du temps. Quelques exemples d’images multicolores représentant des cellules et des structures du microenvironnement BM (surface osseuse SHG+ ; cellules mésenchymateuses nestine-GFP+ ; cellules endothéliales cdh5-DSRED+, cdh5-GFP+ ou pdgfb-GFP+) et le compartiment hématopoïétique malin (cellules AML tdTOMATO+), ainsi que des agents de contraste fluorescents représentant la lumière des vaisseaux (dextran-TRITC). Les paramètres vasculaires mesurés sur des heures ou des jours, notamment la longueur, la rectitude et le diamètre des vaisseaux, ainsi que la perméabilité dans différentes régions vasculaires, peuvent fournir des informations importantes sur le comportement et la santé des tissus.
Protocole
Toutes les expériences sur les animaux ont été réalisées dans le cadre de l’accord éthique APAFIS#27215-2020041513522374 v6, approuvé par le Ministère de l’enseignement supérieur, de la recherche et de l’innovation.
1. Conception d’un implant de fixation de tête en titane biocompatible pour l’impression 3D
REMARQUE : Nous avons conçu un implant de fixation de la tête biocompatible à l’aide d’un logiciel de conception assistée par ordinateur (CAO) de modélisation paramétrique avec des capacités intégrées d’analyse par éléments finis (FEA) (voir le tableau des matériaux). Les principales entrées du processus de conception comprennent un modèle anatomique haute résolution du crâne de la souris, un modèle de l’objectif du microscope et un modèle du système de fixation, qui informent ensemble les dimensions et la configuration de l’implant. Le résultat final est un fichier de maillage indépendant de la plate-forme, généralement au format STL ou STEP, compatible avec la plupart des logiciels d’impression 3D. Ce format de fichier assure un transfert transparent vers l’imprimante 3D pour une fabrication précise de l’implant.
- Préparation du modèle 3D de la souris
- Positionnez le crâne de la souris, préparé ou in vivo, dans le scanner micro-CT.
- Définissez les paramètres de balayage pour une résolution optimale des détails anatomiques fins : ouvrez le panneau de configuration et dans la section Conditions de balayage , choisissez un champ de vision (FOV). Utilisez LiveView pour définir le volume d’analyse.
REMARQUE : Grâce à la résolution du détecteur, le FOV déterminera la taille du voxel en fonction de la résolution du détecteur. Dans les appareils micro-CT plus récents, un champ de vision de 5 mm correspond généralement à une taille de voxel de 5 μm, un champ de vision de 10 mm à une taille de voxel de 10 μm, un champ de vision de 25 mm à une taille de voxel de 21 μm. Le champ de vision limité à cette résolution peut nécessiter plusieurs balayages pour couvrir l’ensemble du crâne. Plusieurs numérisations nécessitent un chevauchement entre les numérisations. - Assemblez plusieurs numérisations haute résolution dans un logiciel d’imagerie pour créer un modèle unique et complet du crâne.
- Importez des fichiers DICOM micro-CT dans un logiciel de segmentation (par exemple, 3D Slicer, Amira, Avizo ou Mimics) pour isoler le crâne.
- Effectuez une segmentation basée sur des seuils pour séparer le tissu osseux des structures environnantes et effectuez des ajustements manuels pour affiner les régions complexes ou bruyantes.
- Dans 3D Slicer, importez d’abord des fichiers DICOM, chargez l’ensemble de données et observez les coupes CT dans les vues axiale, sagittale et coronale .
- Ouvrez le Segment Editor, cliquez sur Ajouter pour créer une nouvelle segmentation | Ajouter pour créer un nouveau segment, qui doit être seuillé.
- Dans le panneau Éditeur de segments , cliquez sur l’effet Seuil . Ajustez les curseurs Seuil inférieur et supérieur (ou saisissez des valeurs numériques) de sorte que la région à segmenter soit mise en surbrillance en rouge dans les vues de tranche. Un segment 3D seuillé est prêt.
- Pour exporter le crâne segmenté sous forme de fichier STL, passez au module de segmentation. Assurez-vous que le segment seuillé est sélectionné. Dans la section Exporter/Importer des modèles et des cartes d’étiquettes , définissez le type d’exportation sur Modèles. et Format de fichier : STL. Cliquez sur le bouton Exporter .
REMARQUE : Les fichiers STL utilisent un format de maillage triangulaire, représentant la surface du crâne avec des milliers de triangles, capturant la forme anatomique dans les moindres détails.- Si nécessaire, effectuez l’assemblage dans MeshLab comme suit :
- Importez des maillages en cliquant sur Fichier | Importez le maillage et sélectionnez-en un comme balayage de référence.
- Alignez d’autres numérisations à l’aide de l’outil Aligner (Filtres | Inscription | Aligner) en marquant les points correspondants et en utilisant Traiter.
- Fusionnez les numérisations alignées en cliquant sur Filtres | Couche de maille | Aplatissez les calques visibles à l’aide de l’option Fusionner uniquement les calques visibles .
- Si nécessaire, effectuez l’assemblage dans MeshLab comme suit :
- Nettoyez et réparez le maillage, en éliminant les sommets et les triangles dupliqués comme suit, pour assurer l’élimination des triangles induits par le bruit et le remplissage de tous les trous involontaires, pour obtenir une surface lisse et continue :
- Cliquez sur Filtres | Nettoyage et réparation | Supprimez les sommets dupliqués.
- Cliquez sur Filtres | Nettoyage et réparation | Supprimez les faces dupliquées.
- Cliquez sur Filtres | Nettoyage et réparation | Retirez les morceaux isolés (par numéro de face). Spécifiez un seuil pour le nombre minimum de faces à conserver (par exemple, définissez ce seuil à un niveau bas pour supprimer les petits groupes ou le bruit).
- Cliquez sur Filtres | Remaillage, simplification et reconstruction | Fermez les trous.
- Effectuez la simplification du maillage à ~10 000-30 000 triangles, car les fichiers STL initiaux de micro-CT contiennent un nombre très élevé de triangles qui ne sont pas compatibles avec le logiciel de CAO. Dans les zones très détaillées, conservez des triangles plus petits sur le dessus du crâne pour assurer un ajustement précis de l’implant. Dans les régions plus grossières, simplifiez les régions moins critiques avec des triangles plus grands pour réduire la taille du fichier et optimiser l’efficacité du traitement.
REMARQUE : La simplification du maillage est effectuée par zones et détaillée aux paragraphes 1.1.8.1-1.1.8.3.- Utilisez l’outil Sélectionner les composants connectés dans une région pour sélectionner les zones à simplifier.
- Utilisez la décimation de l’effondrement des arêtes quadratiques sur les zones sélectionnées avec des paramètres. Cliquez sur Filtres | Remaillage, simplification et reconstruction | Simplification : décimation de l’effondrement des bords quadriques. Définir le seuil de qualité à 0,8, Préserver la limite du maillage à OUI, Préserver la topologie à OUI, Nombre cible de faces : définissez un nombre cible correspondant à la résolution nécessaire.
- Itérez si nécessaire jusqu’à ce que le nombre total de triangles soit réduit au niveau souhaité. Ensuite, enregistrez le modèle simplifié dans un fichier STL.
- Préparation du modèle de crâne dans le modèle CAO
- Importez le modèle de crâne simplifié au format STL dans le logiciel de CAO en tant que nouvelle pièce ou modèle. Assurez-vous que le fichier importé est correctement converti en corps solide ou en surface si nécessaire, en fonction des capacités du logiciel. Enregistrez le modèle dans le format de fichier CAO natif pour permettre d’autres modifications et assurer la compatibilité avec les étapes de conception ultérieures.
- Placez des points de référence sur Bregma et Lambda et créez l’axe Bregma-Lambda dans ce modèle.
- Créez le plan médian incluant l’axe Bregma-Lambda.
- dans le plan médian, créez un axe tangent à la calvaire, puis enregistrez le crâne préparé (figure supplémentaire S1).
- Créez un nouvel assemblage et insérez le crâne préparé de manière à ce que Bregma coïncide avec l’origine, le plan médian avec le plan droit et l’axe calvaire soit horizontal.
- L’implant de fixation de la tête sera conçu directement dans l’ensemble pour assurer un ajustement parfait avec le modèle de crâne préparé. Pour y parvenir, créez l’implant en tant que nouvelle pièce au sein de l’assemblage. Commencez par accéder à Insertion | Composant | Nouvelle pièce dans le logiciel de CAO. Lorsque vous y êtes invité, sélectionnez la surface calvaria ou le plan supérieur de l’assemblage comme référence pour positionner et ancrer la nouvelle pièce.
- Créez un ensemble de plans équidistants (espacement de 2 mm) sur le crâne. Sur chaque plan, créez un croquis et, sur chaque croquis, dessinez une courbe projetée décalée de 0,2 mm sous la forme d’une cannelure symétrique à neuf points, en suivant la section de la surface supérieure du crâne à 0,2 mm au-dessus du crâne (figure supplémentaire S2).
- Créez une surface balayée à l’aide de ces cannelures - la surface limite du crâne - en veillant à ce que cette surface soit lisse sans aucun défaut, à utiliser pour la construction des caractéristiques de l’implant.
- Définition de la surface observable sur le crâne
REMARQUE : Sur la base du modèle 3D, la région la plus plate de l’os de la calvariose idéale pour l’observation 3D est identifiée. Cette région est soigneusement choisie pour assurer un accès optimal à l’objectif du microscope à deux photons tout en tenant compte de l’intégrité biologique et structurelle du crâne. La définition peut utiliser des coordonnées stéréotaxiques où le point Bregma sert d’origine au système de coordonnées.- Créez une esquisse dans le plan calvaire et créez une cannelure en forme de poire de AP +6,5 à -2, largeur de 6 mm à AP 0,0. Il s’agira du contour de surface observable (figure supplémentaire S3).
REMARQUE : L’objectif du microscope est virtuellement positionné par rapport à la surface observable précédemment définie sur le crâne. Cette étape consiste à déterminer les contraintes spatiales imposées par la taille et la forme de l’objectif, en s’assurant que la conception de l’implant répond aux exigences du microscope. - Insérez verticalement le modèle d’objectif de microscope dans l’ensemble. Contraindre son point focal sur un plan parallèle au plan calvarial de l’os calvaire et définir le volume limite de l’objectif du microscope.
- Créez un volume en balayant l’objectif en suivant le contour de surface observable.
- Créez une fenêtre d’observation autour de la surface observable définie, assurant un accès libre à l’objectif du microscope.
REMARQUE : La fenêtre d’observation est une structure en forme d’anneau ayant des fonctions secondaires telles que la rétention de liquide d’immersion et une structure solide pour la fermeture correcte du couvercle de protection. - Créez une esquisse dans le plan calvaire et copiez le contour de surface observable.
- Créez un contour parallèle de 0,5 mm d’épaisseur. Extrudez ce double contour vers le haut sur 0,9 mm et vers le bas jusqu’à la surface Skull Limit Surface avec un angle de dépouille de 4°.
- Créez une esquisse dans le plan calvaire et créez une cannelure en forme de poire de AP +6,5 à -2, largeur de 6 mm à AP 0,0. Il s’agira du contour de surface observable (figure supplémentaire S3).
- Définition des structures de cimentation, de queue et de queue d’aronde.
- Créez un croquis dans le plan calvaire et dessinez une forme en C de 0,5 mm d’épaisseur reliée à la fenêtre d’observation et extrudez 2 mm de haut et vers le bas jusqu’à la surface limite du crâne avec un angle de dépouille de 4° (figure supplémentaire S4).
- Créez une esquisse dans le plan calvaire et remplissez cet espace avec des rectangles de 0,4 mm d’épaisseur placés radialement avec un espace de 1 mm entre eux.
- Extrudez de la même manière que la fenêtre d’observation.
- Créez la queue de l’implant en vous appuyant sur le crâne pour la fixation en respectant le volume disponible.
- Créez un plan transversal tangentiellement à la structure de cimentation et un autre de 12,5 mm en arrière.
- Esquissez des rectangles sur ces plans. Balayez un corps pyramidal entre les deux, en réglant les angles d’environ 20° et 37° par rapport à l’horizontale.
- Ajoutez un trou fileté M1.6 dans la queue près de la structure de cimentation.
- Créez une esquisse à l’extrémité de la queue et dessinez le profil en queue d’aronde.
- Extrudez ce profil de 8 mm.
- Vérification de la non-interférence avec l’objectif.
- Examinez l’ensemble de la conception de l’implant, y compris la fenêtre d’observation et la queue d’aronde, pour vous assurer qu’il n’y a pas d’interférence mécanique avec l’objectif du microscope. Vérifiez s’il n’y a pas d’interférences avec le volume créé à l’étape 1.3.3.
REMARQUE : Cette étape de vérification est essentielle pour s’assurer que l’objectif peut être positionné et déplacé au besoin sans obstruction.
- Examinez l’ensemble de la conception de l’implant, y compris la fenêtre d’observation et la queue d’aronde, pour vous assurer qu’il n’y a pas d’interférence mécanique avec l’objectif du microscope. Vérifiez s’il n’y a pas d’interférences avec le volume créé à l’étape 1.3.3.
- Retirez le matériau inutile de la queue de l’implant pour minimiser le poids et réduire l’inconfort potentiel pour l’animal.
- Créez une esquisse dans le plan médian et dessinez des découpes triangulaires dessus pour former une structure de treillis lorsqu’elle est extraite par extrusion du corps de queue.
- Répétez l’opération dans le plan perpendiculaire.
- Vérifiez le déplacement maximal par simulation par la méthode des éléments finis (FEM) pour analyser les propriétés mécaniques de l’implant dans diverses conditions.
REMARQUE : Cette simulation aide à prédire le déplacement maximal et les contraintes à l’intérieur de l’implant pendant l’utilisation, garantissant ainsi sa fiabilité.- Activez le complément de simulation de logiciel de CAO.
- Dans la barre d’outils Simulation, créez une étude statique. Accédez à l’onglet Simulation (s’il est visible) ou accédez à Simulation | Étudier. Dans la boîte de dialogue Etude , choisissez Statique comme type d’étude. Cliquez sur OK pour créer la nouvelle étude (figure supplémentaire S5).
REMARQUE : Ce type d’étude convient à l’analyse de la déformation et des contraintes sous des charges statiques, comme prévu dans l’utilisation de l’implant. - Choisissez un matériau pour l’implant qui correspond à la composition réelle de l’implant (Ti6Al4V).
- Définissez des contraintes fixes pour simuler les points d’attache de l’implant où il entrerait en contact avec la fixation en queue d’aronde. Définissez la surface supérieure de la queue d’aronde comme fixe.
- Définissez la charge verticale sur la surface inférieure de l’implant. Réglez 1 N force verticale.
- Générez un maillage. Affinez le maillage dans les zones où une contrainte ou une déformation élevée est attendue (figure supplémentaire S6). Pour ce faire, faites un clic droit sur l’élément de maillage de l’arbre d’étude et sélectionnez Créer un maillage... Dans la boîte de dialogue Maillage , ajustez la densité du maillage à l’aide du curseur ou en activant la zone Contrôle du maillage pour définir indépendamment les tailles maximale et minimale des éléments. Cliquez sur OK pour générer le maillage.
REMARQUE : Le logiciel génère automatiquement un maillage initial pour l’étude, mais pour des résultats plus précis, le maillage peut être affiné pour sélectionner un maillage plus fin, en particulier dans les régions critiques pour la stabilité de l’implant (comme la fenêtre d’observation). Un maillage plus fin dans ces régions améliore la précision de la simulation en capturant les concentrations de contraintes locales. - Démarrez l’analyse statique pour calculer le déplacement, les contraintes et les distributions de déformation sur l’implant.
- Analysez les résultats. Examinez le graphique de déplacement pour identifier les zones de déformation maximale. Ce graphique montre à quel point l’implant se déformerait sous les charges appliquées, en mettant en évidence les zones à risque de mouvement excessif. Assurez-vous que la déformation à l’intérieur de la fenêtre d’observation ne dépasse pas 3 microns.
- Enfin, concevez une housse de protection pour protéger la fenêtre d’observation, lorsqu’elle n’est pas utilisée. Ce couvercle protège l’os exposé des contaminants environnementaux et des dommages physiques.
2. Traitements de souris, anesthésie et implantation chirurgicale de l’implant de tête
REMARQUE : Ici, des souris C57BL/6 ou transgéniques mâles ou femelles de 7 à 12 semaines peuvent être utilisées, comme illustré. Pour induire la colonisation leucémique de la BM, les cellules leucémiques générées, telles que décrites par Horton et coll.12, sont administrées par voie intraveineuse 2 à 3 semaines avant l’imagerie. Pour garantir la santé de la plaie, des techniques stériles doivent être utilisées.
- Sous le capot, assemblez les outils chirurgicaux stériles (ciseaux émoussés et pince à épiler), la tondeuse et les consommables, y compris le gel pour les yeux, le sérum physiologique, le désinfectant, le ciment dentaire, les écouvillons absorbants et les chiffons de nettoyage. Allumez le stérilisateur à billes.
- Remplissez la chambre d’induction de l’anesthésique avec 4 % d’isoflurane et d’oxygène atmosphérique et placez la souris dans la chambre. Lorsque l’animal est complètement anesthésié (vérifiez s’il n’y a pas une perte du réflexe de redressement et une respiration plus profonde et plus lente), basculez le flux d’isoflurane vers le cône nasal du masque anesthésique et abaissez la concentration d’isoflurane à 2 %.
- Placez la souris sur un coussin chauffant à 37 °C et surveillez visuellement la fréquence respiratoire. Facultatif : Pour les souris présentant un phénotype pathologique, ajoutez une couverture de survie pour préserver la bonne température tout au long de l’anesthésie.
- Administrer des analgésiques par voie sous-cutanée (Buprecare, 0,01 mg/kg) 30 min avant l’intervention.
- Rasez la tête de la souris avec un rasoir électrique.
- Ajoutez une goutte de gel ophtalmique sur les yeux de la souris pour prévenir la sécheresse sous anesthésie, en vous assurant que le gel ne coule pas sur le cuir chevelu.
- Nettoyez la souris pour éliminer tous les poils résiduels de la zone chirurgicale. Tamponnez le haut du cuir chevelu avec un désinfectant sur un mouchoir en coton. Assurez-vous d’enlever tous les cheveux pour éviter les artefacts d’imagerie ainsi que le risque d’infection de la plaie.
- À l’aide d’une pince et de ciseaux stériles (après 30 secondes dans le stérilisateur), faites une petite incision dans la partie centrale du cuir chevelu pour exposer la cicatrice osseuse centrale. Suivez-le pour définir la longueur et la largeur correctes de la zone d’imagerie. Retirez soigneusement le morceau du cuir chevelu, soulevez la peau entre les oreilles à l’aide d’une pince et faites une petite incision à l’arrière de la tête. En tenant la peau vers le haut, passez les ciseaux sous la peau et coupez soigneusement à l’extérieur de la zone d’imagerie.
- Retirez le tissu conjonctif entre le crâne et le cuir chevelu. Essuyez l’os exposé avec du PBS stérile sur un coton-tige. Procéder rapidement à la fixation de l’implant crânien sur le crâne de la souris pour éviter une exposition prolongée de l’os du crâne (<10 min).
- Faites une pâte avec suffisamment de ciment dentaire dans une boîte de Pétri en suivant les instructions du fabricant et appliquez-la rapidement au bas de l’implant de fixation de la tête.
- Sans mettre de ciment dentaire à l’intérieur de la zone d’imagerie, placez le support de tête sur le crâne exposé de la souris, puis attendez qu’il prenne.
- Une fois réglé (normalement dans les 3 à 5 minutes), ajoutez quelques gouttes de PBS à 37 °C dans la chambre d’imagerie pour maintenir le crâne hydraté.
3. Imagerie à l’aide d’un microscope à deux photons
- Avant de commencer l’opération, allumez le laser et laissez-le se réchauffer pour se stabiliser. Allumez le microscope et démarrez le logiciel d’acquisition. Sélectionnez l’objectif approprié (immersion 25x/0,95 WATER pour cette expérience).
- Utilisez un support de microscope vertical à platine fixe qui a suffisamment d’espace pour placer une planche à pain vétérinaire avec un coussin chauffant, le tout devant être déplacé intégralement avec la platine xy. Utilisez une plaque d’essai optique en aluminium solide comme plate-forme pour fixer l’implant de tête et le masque stéréotaxique. Relier les tuyaux souples et leurs connecteurs associés à l’injection et à l’extraction de l’isoflurane au masque dans la cage du microscope.
- Configurez les paramètres d’acquisition pour numériser rapidement : format 512 x 512 pixels ; vitesse 600 Hz ; et le facteur de zoom 1.
- Réglez la ligne laser à 880 nm pour pouvoir détecter le SHG, le GFP et le tdTOMATO avec la même longueur d’onde d’excitation.
- Éteignez la lumière dans la pièce et fermez correctement le boîtier autour du support du microscope avant d’activer les détecteurs NDD et de définir les portes de détection spectrale : PMT1, pour détecter SHG [388 - 431 nm] ; HyD2, pour GFP [485 - 548 nm] et HyD3 pour tdTOMATO [551 - 645 nm]. Laissez le décalage à 0.
- Créez un nouveau jeu de données de projet et renommez-le en fonction de l’expérience spécifique.
- Une fois l’implant de fixation de la tête fixé sur le crâne de la souris (confirmé par le durcissement du ciment), allumez l’isoflurane vers le masque stéréotaxique du microscope.
- Amenez rapidement la souris au microscope. Insérez soigneusement les dents de la souris dans le masque pour permettre la pénétration de l’isoflurane par le nez de la souris en soulevant son nez d’une main.
- Tout en tenant la souris d’une main, glissez doucement la queue d’aronde de l’implant de fixation de la tête dans le support de fixation avec l’autre main et fixez-la avec un demi-tour du bouton à vis.
- Introduisez une sonde rectale pré-incrustée d’un gel à base d’eau pour la surveillance de la température et ajoutez des gouttes ophtalmiques dans les yeux de la souris.
- Remplissez la chambre d’imagerie de la tête avec une grande quantité de gel à base d’eau ou PBS et abaissez l’objectif d’immersion dans l’eau pour l’immerger complètement afin d’obtenir une excitation et une détection optimales des signaux générés par l’excitation à deux photons.
- Déplacez la platine x-y et le lecteur z pour vous concentrer sur l’os central dans la cicatrice de l’échafaudage.
REMARQUE : Il est pratique d’utiliser les oculaires du microscope et une lampe de type halogénure métallique comme source lumineuse, filtrée par un filtre d’émission d’excitation tri-bande. L’absence de sectionnement optique rend impossible l’obtention d’une image claire ; Mais avec de la pratique, la zone centrale du crâne est facilement reconnaissable. - Après avoir identifié le tissu, marqué par une surface osseuse et une veine centrale, éteignez la lampe aux halogénures métalliques et fermez les fenêtres de la cage du microscope pour protéger les détecteurs HyD et laisser passer le laser infrarouge (IR).
- Réglez le gain PMT/SHG sur 850 V (sa plage de fonctionnement linéaire) et réglez les gains HyD à 100 %. Augmentez la puissance du laser IR jusqu’à ce qu’une image avec une plage dynamique de 200 niveaux de gris (avec des détecteurs de 8 bits dynamiques) soit obtenue sur le plus bas des trois canaux. Réduisez les gains des autres détecteurs si les différences d’émission sont trop importantes ou s’il y a saturation en utilisant le mode de table de conversion (LUT) « sous le bas ».
REMARQUE : Dans notre cas, une puissance laser de 30 % a suffi pour obtenir un contraste dans tous les canaux. - Sur le logiciel d’acquisition, recherchez une région constituée de poches de BM (GFP+, tdTOMATO+) enfermées dans les surfaces osseuses (SHG).
- Pour trouver différents ROI, activez LAS NAVIGATOR et créez une vue d’ensemble de toute la région à l’aide du mode spirale. Arrêtez l’acquisition lorsque la surface acquise est suffisamment grande. Enregistrez la vue d’ensemble, fusionnez-la et renommez-la : ce sera l’image de référence en cas d’imagerie longitudinale.
REMARQUE : Veillez à ne pas générer une spirale trop grande car cela pourrait amener le support de l’appareil à entrer en contact avec l’objectif. - Enregistrez certaines positions de retour sur investissement en cliquant sur l’icône de l’image unique et renommez chaque position dans la liste des tâches. Faites une capture d’écran pour que la position relative des différents ROI soit superposée à la vue d’ensemble précédemment enregistrée.
REMARQUE : Cette capture d’écran sera essentielle pour remplacer les ROI en imagerie longitudinale, car le logiciel d’acquisition ne permet pas de rappeler ce groupe de positions dans la version utilisée pour cette expérience. - Pour acquérir un volume z-stack, sélectionnez le mode z-stack, définissez la taille du pas à 3 μm et désélectionnez l’option Même taille de pile pour toutes les régions . Sélectionnez la première position, définissez les positions supérieure et inférieure , vérifiez la taille du pas z et cliquez sur redéfinir la pile. Une icône de cube apparaît à côté du nom de la position dans la liste des positions. Répétez cette étape pour toutes les positions des différents ROI.
- Appuyez sur Acquérir et enregistrez les images (trois canaux, z-stacks avec ~50 plans) dans le dossier approprié. Cette étape marque le Tctrl de l’expérience ; Vérifiez que les paramètres d’acquisition sont corrects (obtenir la plage dynamique correcte dans les trois canaux, éviter autant que possible la saturation du signal et vérifier le temps d’acquisition pour un cycle complet).
- Pour mesurer une caractéristique dynamique (perméabilité vasculaire dans ce cas), acquérez un timelapse.
- Dans cette configuration, les trois détecteurs permettent l’imagerie simultanée de trois canaux ; si un quatrième canal est nécessaire pour l’expérience (surveillance Dextran dans ce cas), activez le mode séquentiel et ajoutez une deuxième séquence d’acquisition. Réglez la ligne laser IR à 820 nm pour pouvoir exciter le fluorophore Dextran Cy5. Réglez la plage de détection Hyd2 sur [650 - 744 nm] et désactivez les autres détecteurs. Choisissez entre le mode d’acquisition des piles .
REMARQUE : Par défaut, la deuxième séquence d’acquisition a les mêmes paramètres que la séquence 1. - Avant de lancer le timelapse, lancez l’acquisition de toutes les positions pour vous assurer que le temps d’enregistrement ne dépasse pas 3 min (en tenant compte de l’ajout du mode séquentiel), le temps minimum nécessaire pour détecter la fuite de dextran.
- Avant l’injection de Dextran, changez le mode d’acquisition en xyzt. Dans le module t, réglez l’intervalle de temps à 3 min et la durée à 1 h.
- Dans cette configuration, les trois détecteurs permettent l’imagerie simultanée de trois canaux ; si un quatrième canal est nécessaire pour l’expérience (surveillance Dextran dans ce cas), activez le mode séquentiel et ajoutez une deuxième séquence d’acquisition. Réglez la ligne laser IR à 820 nm pour pouvoir exciter le fluorophore Dextran Cy5. Réglez la plage de détection Hyd2 sur [650 - 744 nm] et désactivez les autres détecteurs. Choisissez entre le mode d’acquisition des piles .
- Injecter 100 μL de Dextran 70 KDa-TRITC (ou 500 KDa-Cy5) à 3 mg/souris par voie intraveineuse et commencer l’acquisition.
- Après l’acquisition, enregistrez l’ensemble de données dans le dossier approprié.
REMARQUE : Au cours de ces nombreuses étapes, surveillez fréquemment la fréquence de respiration et la température de la souris et ajustez le débit d’isoflurane si nécessaire. Idéalement, la température, l’ECG et la fréquence respiratoire pourraient être enregistrés et inclus dans les métadonnées de l’expérience.
4. Récupération de la souris
- Une fois la session d’imagerie terminée, désactivez le détecteur HyDs dans le logiciel d’acquisition et allumez le boîtier de chauffage de la souris (37 °C). Ouvrez la cage du microscope, soulevez l’objectif et retirez la sonde rectale de la souris. Ensuite, glissez doucement la queue d’aronde de l’implant de fixation de la tête hors du support de fixation et déplacez la souris du masque stéréotaxique du microscope au masque chirurgical sur le coussin chauffant.
- Coupez l’injection d’isoflurane au microscope et éteignez le coussin chauffant.
- Retirez délicatement le gel à base d’eau ou PBS du crâne de la souris à l’aide d’un écouvillon stérile.
- Ajouter du gel intrasite pour maintenir l’humidité du crâne entre les séances d’imagerie, comme décrit précédemment13.
- Fermez la zone d’imagerie de l’implant crânien avec le couvercle spécifique et fixez-la soigneusement avec la vis.
- Placez la souris dans le boîtier chauffant à 37 °C et attendez qu’elle se réveille. Amenez la souris à l’animalerie et logez-la dans une cage propre avec de l’hydrogel et de l’enrichissement. Surveillez les animaux chaque jour pour détecter tout signe de douleur ou d’infection, auquel cas administrez Buprecare quotidiennement. Pour préserver l’humidité calvaire, changez le gel intrasite 2 fois par semaine.
REMARQUE : Choisissez une cage haute avec des abris en carton et du matériel de nidification et assurez-vous de mettre les souris dans des cages individuelles.
5. Acquisitions longitudinales
REMARQUE : La souris peut être à nouveau imagée au cours des jours suivants. Cependant, assurez-vous de ne pas répéter plus de trois séances d’imagerie par semaine pour éviter les effets indésirables des anesthésies répétées, tels que la sécheresse oculaire ou la fatigue excessive, ainsi que la détresse respiratoire et l’hypothermie.
- Pour réimager une souris avec un support de tête déjà installé, suivez l’étape 2.2 et placez une petite goutte de gel ophtalmique sur les yeux de la souris pour éviter la sécheresse pendant la procédure.
- Suivez la section 3 jusqu’à l’étape 3.16 pour obtenir l’image de synthèse.
REMARQUE : L’utilisation de l’implant de tête en titane sans avoir à modifier la hauteur ou l’angle de la position du support permet de trouver très facilement la zone d’intérêt globale. - Si nécessaire, réalignez les images précédentes et nouvelles à l’aide du module d’ouverture d’image et d’alignement.
- Ouvrez l’image de capture d’écran de l’étape 3.17 pour marquer les positions à leur place d’origine.
REMARQUE : L’utilisation du contraste SHG est très utile, en particulier lorsque l’on s’attend à un remodelage des vaisseaux sanguins et des cellules associées exprimant la GFP. - Acquérez des piles z et des timelapses de la même manière que celle décrite aux étapes 3.18 à 3.22.
6. Quantification des paramètres vasculaires
- Paramètres vasculaires
- Pour ouvrir les images générées avec le logiciel (fichiers .lif) dans IMARIS, convertissez-les d’abord au format « .ims » avec le convertisseur de fichiers. Ouvrez le fichier « .ims » avec IMARIS, vérifiez les métadonnées (échelles X, Y, Z) et sélectionnez le module Filament . Sélectionnez Ignorer la création automatique, modifier manuellement.
- Pour une meilleure visualisation lors de la génération du filament, accédez à Paramètres (symbole de feuille), sélectionnez Ligne dans Style et laissez le nombre de pixels à la valeur par défaut (1). Cochez les cases Afficher les dendrites, y compris le point de départ, les points de branche et les points finaux. Décochez Afficher la colonne vertébrale.
- Sous draw (symbole de pinceau), sélectionnez Method AutoPath ; 5 μm de diamètre et la source de canal associée au récipient (TRITC-Dextran dans ce cas). Enfin, cochez centre automatique et diamètre automatiquer.
- Commencez à dessiner les filaments en utilisant Maj + clic droit de la souris pour choisir un point de départ de branche et Maj + clic gauche pour choisir un point final d’une branche.
REMARQUE : Le point de départ est en bleu clair, les points de ramification sont en rouge ou en bleu foncé et les points terminaux sont en vert. - Pour régler le bon diamètre du filament pendant le dessin, utilisez le bouton de défilement de la souris.
- Pour joindre deux filaments avec des points terminaux distincts, créez un nouveau point de départ entre eux et connectez-le à chaque point d’extrémité.
REMARQUE : Ainsi, les points terminaux verts apparaissent dans les points de ramification. - Une fois l’arbre vasculaire créé, revenez aux paramètres et assurez-vous que les nombres de points de départ, de ramification et de fin sont corrects. Sélectionnez cône pour visualiser les diamètres et vérifier leur apparence.
- Si le diamètre d’un filament (ou d’une connexion) semble incorrect, accédez à Modifier (symbole du crayon), sélectionnez le filament à retirer avec un clic droit, puis cliquez sur Supprimer. Dessinez à nouveau le filament en ajustant le diamètre du filament à l’aide du bouton de défilement de la souris.
- Pour extraire toutes les données statistiques générées et les sauvegarder, cliquez sur l’icône Statistiques | exporter toutes les statistiques dans un fichier-(icône de plusieurs disquettes en bas de l’écran).
REMARQUE : Pour éviter tout filament potentiellement aberrant ne correspondant pas aux vaisseaux, seuls les filaments de plus de 20 μm ont été inclus dans l’analyse statistique.
- Quantification de l’étanchéité
REMARQUE : Pour ouvrir les images générées avec le logiciel (fichier .lif) dans IMARIS, convertissez-les d’abord au format « .ims » avec le convertisseur de fichiers.- Pour fusionner des images prises avant et après l’injection de dextran dans la même série temporelle, allez en mode Surpass, cliquez à la fois sur la première image (Tctrl , avant l’injection de dextran) et sur l’image timelapse (durée 1, 60 min) pour les ouvrir, et combinez-les en cliquant sur Modifier et ajouter des points temporels.
REMARQUE : Pour corriger une éventuelle dérive 3D, suivez les étapes 6.2.2 à 6.2.7 en utilisant la fonction de pont entre Fiji et IMARIS. Installez les plugins suivants aux Fidji : « IMARIS_Bridge96.jar » et « IMARISBridgeUtils.jar ». L’enregistrement de la dérive 3D est effectué à l’aide de « StackReg Pluging » (déjà disponible aux Fidji). Si ce n’est pas nécessaire, passez à l’étape 6.2.8 - Pour faire une correction de dérive 3D, ouvrez Fiji pour relier les images d’IMARIS en cliquant sur Plugins | IMARIS | Image de IMARIS.
- Aux Fidji, sélectionnez Plugins | Inscription | Dérive 3D correcte.
- Dans les nouvelles fenêtres qui s’affichent, choisissez le canal approprié pour l’enregistrement.
REMARQUE : Il s’agissait de Nestin-GFP dans ce cas, mais il pouvait s’agir de n’importe quel canal mettant en évidence des objets immobiles et un rapport signal/arrière-plan élevé. - Pour améliorer la détection des petites dérives, sélectionnez Calcul à plusieurs échelles de temps, Correction de la dérive de sous-pixel et Images d’amélioration des bords, et laissez les autres paramètres par défaut.
- Pour relier l’image corrigée à IMARIS, sélectionnez-la et cliquez sur Plugins | IMARIS | Image à IMARIS.
- Enregistrez-le dans le dossier approprié en cliquant sur Fichier | Enregistrer sous.
- Pour visualiser toutes les chaînes acquises, cliquez sur Modifier | Afficher le réglage de l’affichage. Sélectionnez le canal dextran et la durée 1 (image 2 en bas de l’écran).
- Créez une nouvelle surface définissant la lumière vasculaire au point temporel 1 en cochant la case Ajouter une nouvelle surface (objet bleu)
- Dans Surface 1, allez dans Propriétés et décochez tous les paramètres dans Paramètres de l’algorithme. Cliquez sur l’étape suivante (flèche bleue)
- Sélectionnez le canal vasculaire (dextran dans ce cas) sous le canal source et cochez Lisse et soustraction d’arrière-plan. Laissez tous les autres paramètres aux valeurs par défaut. Cliquez sur l’étape suivante (flèche bleue).
- Ajustez la valeur du seuil en fonction de l’aspect de la surface en termes d’épaisseur et de couverture de toutes les zones positives. Cliquez sur l’étape suivante (flèche bleue).
- Excluez les surfaces non spécifiques (principalement les petits points) à l’aide du filtre par défaut Nombre de voxels. Cliquez sur l’étape finale (flèche verte).
- Si nécessaire, poursuivez le réglage manuel de la surface en cliquant sur Modifier (icône en forme de crayon) dans Réglage des paramètres. Pour sélectionner un objet à supprimer, cliquez dessus avec le bouton gauche de la souris et supprimez-le en sélectionnant Supprimer. Si l’objet à supprimer est assemblé à un autre, déconnectez les deux parties séparées de la surface en cliquant sur Maj + clic gauche entre les deux et en sélectionnant Couper la surface. Répétez cette étape autant de fois que nécessaire.
- Enfin, combinez tous les segments en utilisant le mode de sélection (icône cible à droite) et appuyez sur Ctrl + défilement pour récupérer tous les objets. Lorsque tous sont sélectionnés, cliquez avec le bouton gauche de la souris et cliquez sur unifier.
- Pour générer la surface de référence, copiez cette surface dans les autres points temporels. Sélectionnez la surface globale et dupliquez-la en cliquant sur Dupliquer et renommez-la en Intravasculaire.
- Sélectionnez la surface Intravasculaire et cliquez sur Dupliquer pour tous les points temporels.
- Divisez le signal dextran pour distinguer les zones intra (IN) et extravasculaires (OUT). Pour créer un canal IN pour tous les points temporels, cliquez sur la surface Intravasculaire | Modifier (bouton crayon) | Masquez tout.
- Dans la sélection des canaux, sélectionnez le canal approprié et cochez la case Dupliquer le canal avant d’appliquer le masque. Dans les paramètres du masque, cochez uniquement Constant à l’intérieur/à l’extérieur avec les voxels définis sur la surface extérieure à 0. Enfin, cochez également Appliquer à tous les points temporels.
- Dans le réglage de l’affichage, un nouveau canal est créé par défaut avec le nom masqué. Renommez-le EN.
- Pour créer le canal OUT pour tous les points temporels, répétez les étapes 6.2.18 à 6.2.20, mais sélectionnez Définir les voxels à l’intérieur de la surface sur 0. Renommez ce nouveau canal OUT.
- Pour extraire des données statistiques, cliquez sur l’icône Statistique | Exporter toutes les statistiques dans un fichier (icône de plusieurs disquettes en bas de l’écran)
- Pour analyser les fuites, divisez le canal de somme d’intensité à l’extérieur par le canal de somme d’intensité à l’intérieur pour chaque point temporel.
- Pour fusionner des images prises avant et après l’injection de dextran dans la même série temporelle, allez en mode Surpass, cliquez à la fois sur la première image (Tctrl , avant l’injection de dextran) et sur l’image timelapse (durée 1, 60 min) pour les ouvrir, et combinez-les en cliquant sur Modifier et ajouter des points temporels.
Résultats
Dans les figures 1 et 2, le modèle CAO d’un implant de fixation de la tête en titane positionné sur un crâne de souris numérisé, qui est conçu pour suivre la structure anatomique du crâne et fournir un dispositif léger et biocompatible capable de tenir fermement à la platine du microscope assurant la stabilité au niveau cellulaire. En suivant ce protocole étape par étape, l’implant est fixé de manière stable au crâne de la souris et peut être solidement fixé au support de microscope par sa queue d’aronde, permettant une zone d’imagerie plate pour la rétention de liquide et l’observation intravitale au fil du temps. Il peut être fermé avec un couvercle pour minimiser tout dommage ou infection de la plaie, permettant une imagerie répétée de la même zone tissulaire pendant des semaines. Une fois réveillée, la souris portant un implant de tête peut se promener librement, se nourrir et avoir une routine régulière.
La figure 3 montre une vue par balayage tuile du système vasculaire calvaire BM composé de capillaires hétérogènes, y compris les artérioles, les capillaires de transition et les sinusoïdes. Les vaisseaux sont intégrés dans un microenvironnement tissulaire complexe en contact étroit avec la surface de l’os et les cellules mésenchymateuses périvasculaires. Au cours du développement de la leucémie, des cellules leucémiques isolées peuvent être détectées dans le microenvironnement de la BM à proximité des vaisseaux, et leur greffe augmente avec le temps, remplissant la calvaria aux stades avancés de la maladie.
La figure 4 montre comment les images obtenues avec ce protocole peuvent fournir des données quantitatives, qui peuvent être analysées avec des méthodes statistiques. Nous montrons comment segmenter les vaisseaux à l’aide de l’outil à filament IMARIS et mesurer la longueur et le diamètre des fragments vasculaires, ainsi que leur rectitude. La corrélation de ces paramètres peut également être évaluée.
La figure 5 montre l’acquisition d’une imagerie longitudinale de deux positions différentes de la BM calvaire au cours de la progression de la LMA aux jours 4, 7 et 10, le jour 10 étant associé à une greffe de ~50 % de la BM avec des cellules leucémiques, mesurée par cytométrie en flux (non illustrée). On peut observer un remodelage important de la taille des vaisseaux préexistants, ainsi que la formation de nouveaux vaisseaux dans des zones spécifiques associées à une perte osseuse locale.
Enfin, dans la figure 6, nous montrons comment la perméabilité vasculaire peut être mesurée en tant que paramètre dynamique avec l’imagerie en accéléré montrant la capacité de différentes barrières vasculaires à retenir un colorant fluorescent au fil du temps.

Figure 1 : Conception et production d’un support de tête biocompatible à base de titane. (A) Parties de l’implant in situ : 1 anneau d’observation, 2 éléments de cimentation, 3 ancrages stabilisateurs, 4 queues, 5 queues d’aronde, 6 trous filetés, 7 Bregma. (B) Connexion de l’implant de tête au support : 8 corps de fixation, 9 pinces, 10 leviers excentriques, 11 structure, 12 objectifs de microscope. (C) Déformation de l’implant contre charge par simulation FEM où le déplacement maximal est de 0,23 μm contre une force de 0,04 N. (D) Couvercle de protection et sa vis. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Préparation de la souris pour l’imagerie intravitale. (A) Vue de l’implant crânien et de la zone d’imagerie exposée chirurgicalement avant l’imagerie. (B) Implant de tête fermement fixé au crâne de la souris. (C) Souris réveillée dans la cage de récupération avec le couvercle fermé sur l’implant de tête. Veuillez cliquer ici pour voir une version agrandie de cette figure.
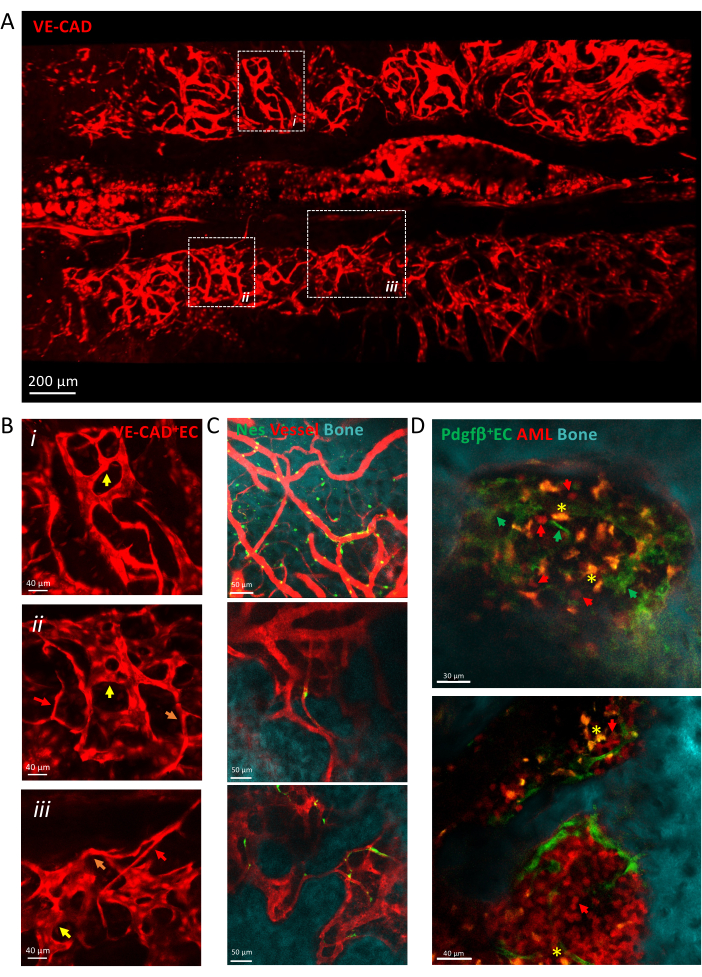
Figure 3 : Imagerie intravitale du système vasculaire de la calvarie. (A) Projection z de la vue du balayage de la tuile du système vasculaire de la calvaria marquée par cdh5-DSRED. (B) Zoomez sur les zones représentées pour décrire différents types de vaisseaux représentés par des flèches, les artérioles par des flèches rouges, les capillaires de transition par des flèches orange, les sinusoïdes par des flèches jaunes. (i) et (ii) projections z de tissu X μm ; (iii) tranche unique. (C) Coupes uniques de plusieurs champs de vision de vaisseaux BM, montrant la surface osseuse (SHG), les cellules périvasculaires (nes-GFP+) et la lumière vasculaire (dextran-TRITC). (D) Niche vasculaire associée à la progression de la LAM. Tranches représentatives des premiers (en haut) et des derniers (en bas) du développement de la lutte contre le blanchiment d’argent. La leucémie MLL-AF9 est marquée avec tdTOMATO (flèches rouges), tandis que les vaisseaux sont marqués avec pdgfb-GFP (flèches vertes), la surface osseuse avec SHG et les macrophages en jaune (autofluorescence, astérisque jaune). Barres d’échelle = 200 μm (A), 40 μm (B, D-panneau inférieur), 50 μm (C). Abréviations : BM = moelle osseuse ; GFP = protéine fluorescente verte ; LMA = leucémie myéloïde aiguë ; EC= Cellules endothéliales. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Quantification des paramètres vasculaires. (A) Mesure des paramètres vasculaires à l’aide de l’outil de filament IMARIS dans une projection z représentative des vaisseaux de la moelle osseuse marqués avec TRITC-dextran. Des représentations linéaires et coniques sont présentées. (B) Quantification des paramètres du récipient dans l’image illustrée en A. (C) Corrélation entre les paramètres vasculaires montrant une corrélation opposée entre la rectitude et la longueur des vaisseaux (négative, Spearman r = -3523 ; p < 0,0001 ; R2 = 0,2102) en fonction du diamètre (positif ; Spearman r = 0,4110 ; p < 0,0001 ; R2 = 0,1299). Barres d’échelle = 100 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
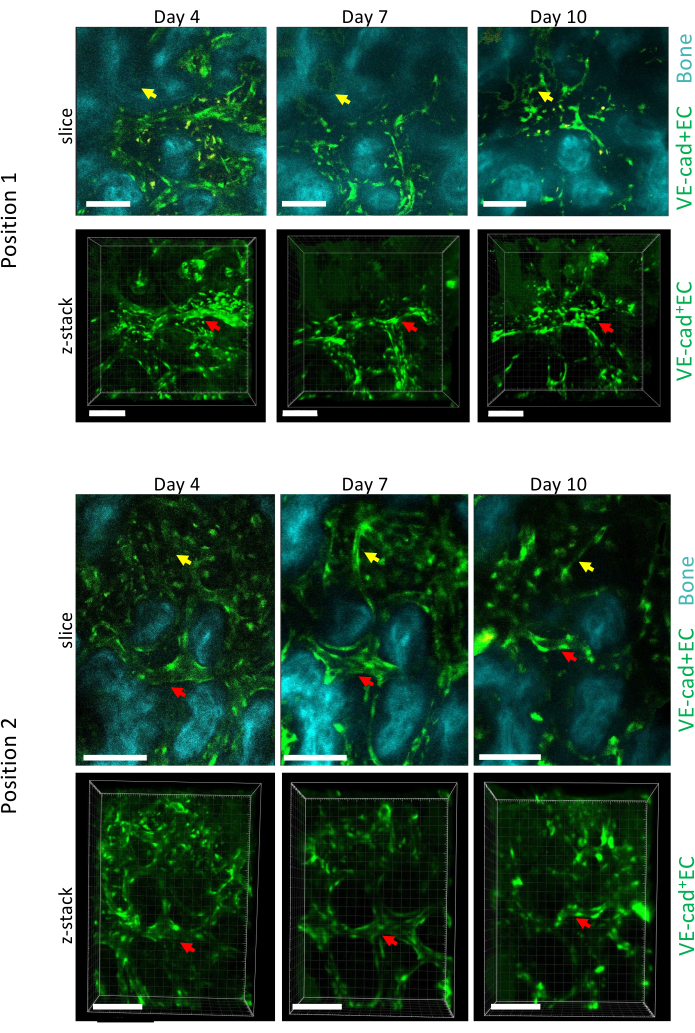
Figure 5 : Imagerie longitudinale de deux positions différentes de la calvaria BM au cours du développement de la LAM. Les cellules endothéliales qui tapissent les vaisseaux sont marquées avec du cdh5-GFP, la surface osseuse avec SHG et les macrophages en jaune (autofluorescence). Le remodelage des vaisseaux préexistants (flèches rouges) et la formation de nouveaux vaisseaux (flèches jaunes) sont illustrés. Barres d’échelle = 100 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
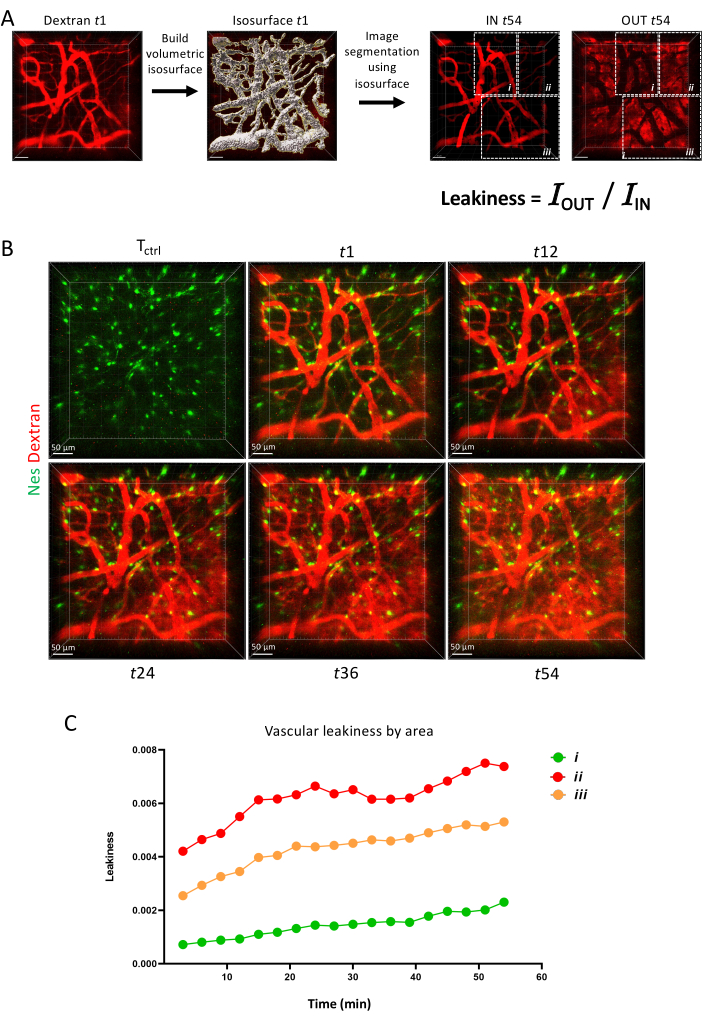
Figure 6 : Perméabilité vasculaire. (A) Schémas de mesure de la perméabilité vasculaire à l’aide de l’outil de surface IMARIS. (B) Projection Z de la même zone imagée longitudinalement sur 1 h. (C) Quantification de la perméabilité vasculaire à l’intérieur des aires comme illustré en A. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire S1 : Crâne préparé. Dans le plan médian, créez un axe tangent à la calvariose, puis enregistrez le crâne préparé. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S2 : Plans espacés de 2 mm. Comment créer un ensemble de plans équidistants (espacement de 2 mm) sur le crâne. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S3 : Contour de surface observable. Créez une esquisse dans le plan calvaire et créez une cannelure en forme de poire de AP +6,5 à -2, largeur de 6 mm à AP 0,0. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S4 : Observation vindow. Créez un croquis dans le plan calvaire et dessinez une forme en C de 0,5 mm d’épaisseur en vous connectant à la fenêtre d’observation . Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S5 : Cliquez sur Nouvelle étude. Accédez à l’onglet Simulation (s’il est visible) ou accédez à Simulation | Étudier. Dans la boîte de dialogue Etude , choisissez Statique comme type d’étude. Cliquez sur OK pour créer l’étude. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S6 : Clic droit pour créer un maillage. Affinez le maillage dans les zones où une contrainte ou une déformation élevée est attendue. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
La principale force de la microscopie intravitale est sa capacité à capturer les processus cellulaires dynamiques en temps réel dans leur environnement natif, surmontant ainsi les limites des méthodes traditionnelles d’imagerie histologique et ex vivo. En observant directement la BM calvaire à l’aide des techniques d’imagerie avancées couplées au marquage fluorescent comme décrit dans ce protocole, les chercheurs peuvent étudier non seulement les paramètres fonctionnels vasculaires 8,14, mais aussi les processus longitudinaux tels que la greffe et la migration des cellules leucémiques 6,15, la prolifération et l’activité métabolique16,17 préserver la pertinence physiologique et éviter les artefacts associés à la fixation et au traitement des tissus.
Les avantages de l’imagerie intravitale BM calvaria par rapport aux autres sites osseux comprennent l’accessibilité facile, la transparence de l’os du crâne permettant une meilleure visualisation sans lésions osseuses, la stabilité de l’os du calvarium minimisant les artefacts de mouvement des tissus et la reproductibilité entre les animaux de laboratoire facilitant la comparaison entre différents groupes expérimentaux. La possibilité de récupérer les animaux et de réaliser des images longitudinales permet également de réduire le nombre d’animaux utilisés dans une étude. Il convient de mentionner que, étant donné que des différences spécifiques à la souche dans les schémas de croissance des vaisseaux du crâne et l’ostéogenèse qui en résulte ont été observées18, il est important d’en tenir compte lors de la conception de l’implant d’imagerie spécifique à la souche de souris souhaitée afin de permettre une compatibilité anatomique parfaite et l’accès au site d’imagerie souhaité.
Un autre point important à mentionner est les différences potentielles de propriétés vasculaires et hématopoïétiques au sein de la BM calvaire par rapport à d’autres os, une question peu explorée jusqu’à ces dernières années. De nouvelles études suggèrent des fonctions localisées pour différents os, la BM calvaire différant des autres os en termes d’hématopoïèse, de structure et de fonction osseuses et vasculaires 19,20,21,22,23, ainsi que de réponse aux pathologies neurologiques24. Ces différences doivent être explorées plus avant d’être prises en compte avant de généraliser des résultats spécifiques à la calvariose.
La conception et la construction d’un implant d’imagerie est une étape clé pour ce dispositif expérimental, à commencer par le choix du matériau. Les implants biocompatibles jouent un rôle central dans la recherche biomédicale, facilitant un large éventail d’applications allant de l’ingénierie tissulaire et de la médecine régénérative aux systèmes d’administration de médicaments et aux dispositifs de surveillance in vivo . Le choix du matériau d’un implant crânien adapté à l’imagerie intravitale est critique. Idéalement, le matériau doit présenter une excellente biocompatibilité, des propriétés mécaniques permettant d’assurer une flexibilité minimale et une stabilité globale, et enfin, la capacité de s’intégrer dans le crâne sans induire d’inflammation. Le titane est idéal pour sa bonne tolérance dans le corps animal, sans induction de réactions indésirables au contact des tissus biologiques, ainsi que pour sa résistance à la corrosion, assurant une stabilité à long terme même en cas d’exposition à des fluides corporels ou à des solutions expérimentales. De plus, sa résistance mécanique le rend résistant à la déformation et à la rupture. Malgré sa résistance, le titane a une densité relativement faible, ce qui permet d’obtenir des appareils d’imagerie intravitaux légers qui minimisent le fardeau des animaux de laboratoire et des chercheurs. Enfin, sa polyvalence dans la fabrication permet de personnaliser les dispositifs d’imagerie intravitale pour répondre à des exigences expérimentales spécifiques, telles que la taille, la forme et la fonctionnalité.
Ici, nous avons conçu un implant de fixation de la tête biocompatible à l’aide d’un logiciel de CAO de modélisation paramétrique avec des capacités intégrées d’analyse par éléments finis (FEA), en particulier SolidWorks. Cette approche permet des ajustements précis et itératifs aux exigences structurelles et spatiales de l’implant, garantissant à la fois la compatibilité anatomique et la résilience mécanique. Les alternatives gratuites, telles que FreeCAD, offrent des fonctionnalités de modélisation et de simulation similaires. Les principales entrées du processus de conception comprennent un modèle anatomique haute résolution du crâne de la souris, un modèle de l’objectif du microscope et un modèle du système de fixation, qui informent ensemble les dimensions et la configuration de l’implant. Le résultat final est un fichier de maillage indépendant de la plate-forme, généralement au format STL ou STEP, compatible avec la plupart des logiciels d’impression 3D.
La première étape consiste à capturer les caractéristiques anatomiques détaillées de la tête de la souris à l’aide de techniques d’imagerie à haute résolution, telles que le balayage micro-CT in vivo . Cette approche fournit les détails anatomiques et la plus grande précision, en capturant les microstructures du crâne. Le scan 3D d’un crâne nu préparé pourrait également être une option. Cette méthode, utilisant des scanners laser ou à lumière structurée, est couramment utilisée sur un crâne préparé et fournit des contours de surface précis, bien qu’avec moins de détails internes par rapport à la micro-tomodensitométrie. Sinon, les modèles d’anatomie peuvent être téléchargés à partir de publications open source et de bases de données25 ou DigiMorph {https://www.digimorph.org/specimens/Mus_musculus/}. Bien que pratiques, ces modèles peuvent manquer de détails spécifiques au spécimen, de sorte que des ajustements sont souvent nécessaires pour l’animal spécifique de l’étude, tels que la mise à l’échelle à la distance réelle Bregma-Lambda. Les données acquises sont ensuite utilisées pour créer un modèle 3D précis du crâne de la souris, qui sert de modèle de base pour la conception de l’implant.
Pour fixer l’implant sur le crâne, une structure de cimentation est conçue pour couvrir la surface restante du crâne non occupée par la fenêtre d’observation. Cette structure doit fournir des points d’attache robustes tout en évitant les caractéristiques anatomiques critiques. La structure de cimentation comporte de multiples ouvertures pour assurer une polymérisation optimale du ciment sous l’implant par diffusion. De plus, les murs de la structure de cimentation ont un petit angle de tirage, ce qui permet au ciment de s’ancrer solidement contre ces murs inclinés. La queue de l’implant, qui s’étend du corps principal au système de fixation, est conçue. Ce composant est crucial pour l’alignement et la stabilisation de l’implant pendant l’observation, et sa conception doit tenir compte de l’espace disponible et des contraintes anatomiques de la tête de la souris. Enfin, un mécanisme à queue d’aronde est intégré dans la conception de l’implant pour faciliter la fixation et le détachement de l’implant du système de fixation. Cette caractéristique améliore l’aspect pratique et la facilité d’utilisation de l’implant lors d’observations répétées. Le mécanisme à queue d’aronde permet une fixation reproductible pour une récupération facile des tissus observés pour des séances d’imagerie répétées.
Les lecteurs qui envisagent l’adoption de l’imagerie par microscopie intravitale de la BM calvaire doivent évaluer soigneusement leurs objectifs de recherche et leurs exigences expérimentales afin de déterminer si cette méthode est appropriée pour leurs études. Bien que la microscopie intravitale offre des informations inégalées sur l’hématopoïèse, la régulation du microenvironnement tissulaire et la dynamique vasculaire in vivo, elle présente également certains défis et limites techniques. Les chercheurs doivent être prêts à relever ces défis grâce à une conception expérimentale minutieuse, à l’optimisation des paramètres d’imagerie et à l’utilisation de contrôles appropriés. De plus, les chercheurs doivent tenir compte de la disponibilité d’équipements d’imagerie spécialisés, d’une expertise dans les techniques de marquage fluorescent et de ressources informatiques pour l’analyse d’images. Dans l’ensemble, l’imagerie par microscopie intravitale de la BM calvaire présente un énorme potentiel pour faire progresser notre compréhension de l’hématopoïèse et de la biologie vasculaire, offrant une fenêtre unique sur les processus dynamiques qui se produisent dans le microenvironnement de la BM.
Déclarations de divulgation
Jozsua Fodor est le fondateur de la société YMETRY (numéro d’enregistrement 888312352). Les autres auteurs n’ont aucun conflit d’intérêts.
Remerciements
Les auteurs tiennent à remercier tout le personnel de l’IMAG’IC et des animaleries de l’Institut Cochin pour leur soutien dans les expériences de microscopie et l’hébergement de souris. Les souris Tg(Nes-EGFP)33Enik et Tg(Pdgfb-icre/ERT2)1Frut ont été un aimable cadeau du Dr Bonnet (The Francis Crick Institute, Londres). Tg(Cdh5-cre/ERT2)1Rha et B6. Les souris Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze/J étaient un aimable cadeau du Dr Rafii (Weill Cornell Medicine, New York). Les travaux décrits ont été soutenus par le CNRS, l’INSERM et Université de Paris Cité, et des subventions de l’ATIP-AVENIR, de la Fondation ARC pour la recherche sur le cancer (R19084KS - RSE20008KSA), de la Ville de Paris « Emergence » (R20192KK - RPH20192KKA), de Laurette Fugain (R23197KK), du Cancéropôle IDF (RPH23177KKA), de l’INCA PLBIO (RPH21162KKA), de la Fondation de France (RAF23152KKA), de la Ligue contre le cancer (282273/807251), de l’Institut du cancer Paris Carpem, Association européenne d’hématologie (RAK23130KKA) et Conseil européen de la recherche ERC-STG (EEA24092KKA). La plateforme IMAG’IC est soutenue par l’Infrastructure Nationale France BioImaging (subvention ANR-10-INBS-04). Le laboratoire Passaro est affilié à l’Institut Hors Murs des Sciences Cardiovasculaires et à l’Institut de Leucémie Paris Saint-Louis.
matériels
| Name | Company | Catalog Number | Comments |
| Amira | Thermo ScientificTM | ||
| Anesthesia | Isoflurane 2% to 3% | ||
| Anesthesic mask (animal detection) | Minerve | ||
| Anesthesic unit | Minerve | ||
| B6.Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze | Jackson laboratories | MGI:3809523 | |
| CalvariaVue | Ymetry | https://ymetry.com/site/head-fixation-implants/44-mouse-head-fixation-implant-for-calvaria.html | |
| Dental cement | Kemdent | SUN527 | |
| Dextran 500 kDa-Cy5 | Tebu-bio | DX500-S5-1 | 3 mg/mouse |
| Dextran 70 kDa-TRITC | Sigma | T1162 | 3 mg/mouse |
| Disinfectant | MP-Labo | Dermidine-60ml | |
| Electric razor | Aescular | Isis | |
| Eye gel | Ocry-gel | 10g | |
| Fiji | https://imagej.net/software/fiji/downloads | v 17 May 30 | |
| Fiji plugings to bridge with Imaris | https://imagej.net/software/fiji/downloads | Imaris_Bridge96.jar ; ImarisBridgeUtils.jar | |
| Heating box | Datesand | Thermacage | |
| Heating pad for surgery | Minerve | ||
| Imaging heating pad & rectal probe | F. Haer | ||
| Imaris v9.6.0 | Oxford instruments | ||
| Intrasite gel | Chinoxia | 2390766 | |
| LAS AF Software | Leica | LAS X 3.5.7.23225 | |
| Medication | Buprecare, 0.01 mg/kg | ||
| Objective HCX IRAPO L 25x/0.95 WATER | Leica | 506374 | |
| Saline buffer (PBS 1x) | Sigma | P4417 | Sterilize by autoclave |
| SP8 DIVE FALCON Multiphoton Microscope | Leica | ||
| Stereotoxic mask | Minerve | 1201261 | |
| Sterilizator beads | Sigma | Z742555 | |
| Surgery tools | Moria | 4877A; 2183 | |
| Survival blanket | SECURIMED | 11006 | |
| Swabs / Tissues | Sterilize by autoclave | ||
| Syringe 1 mL 26 G | BD Plastipak | 305501 | |
| Temperature controller | F. Haer | 40-90-5D-02 | |
| Tg(Nes-EGFP)33Enik mice | Jackson laboratories | MGI:5523870 | |
| Tg(Pdgfb-icre/ERT2)1Frut mice | Jackson laboratories | MGI:3793852 | |
| Tg(Cdh5-cre/ERT2)1Rha mice | Jackson laboratories | MGI:3848982 | |
| Ultrasound gel | Parker laboratories | Aquasonic 100 |
Références
- Lo Celso, C., et al. Live-animal tracking of individual haematopoietic stem/progenitor cells in their niche. Nature. 457 (7225), 92-96 (2009).
- Pittet, M. J., Weissleder, R. Intravital imaging. Cell. 147 (5), 983-991 (2011).
- Itkin, T., et al. Distinct bone marrow blood vessels differentially regulate haematopoiesis. Nature. 532 (7599), 323-328 (2016).
- Christodoulou, C., et al. Live-animal imaging of native haematopoietic stem and progenitor cells. Nature. 578 (7794), 278-283 (2020).
- Upadhaya, S., et al. Intravital imaging reveals motility of adult hematopoietic stem cells in the bone marrow niche. Cell Stem Cell. 27 (2), 336-345.e4 (2020).
- Hawkins, E. D., et al. T-cell acute leukaemia exhibits dynamic interactions with bone marrow microenvironments. Nature. 538 (7626), 518-522 (2016).
- Duarte, D., et al. Defining the in vivo characteristics of acute myeloid leukemia cells behavior by intravital imaging. Immunol Cell Biol. 97 (2), 229-235 (2019).
- Passaro, D., et al. Increased vascular permeability in the bone marrow microenvironment contributes to disease progression and drug response in acute myeloid leukemia. Cancer Cell. 32 (3), 324-341.e6 (2017).
- Jia, Y., et al. FLT3 inhibitors upregulate CXCR4 and E-selectin ligands via ERK suppression in AML cells and CXCR4/E-selectin inhibition enhances anti-leukemia efficacy of FLT3-targeted therapy in AML. Leukemia. 37 (6), 1379-1383 (2023).
- Pendleton, E. G., Tehrani, K. F., Barrow, R. P., Mortensen, L. J. Second harmonic generation characterization of collagen in whole bone. Biomed Opt Express. 11 (8), 4379-4396 (2020).
- Ventalon, C., Bourdieu, L., Leger, J. F., Fodor, J. Dispositif de liaison d'un animal de laboratoire à au moins un système d'expérimentation, et procédé de fixation d'un tel dispositif. France patent. , (2021).
- Horton, S. J., et al. Acute myeloid leukemia induced by MLL-ENL is cured by oncogene ablation despite acquisition of complex genetic abnormalities. Blood. 113 (20), 4922-4929 (2009).
- Scott, M. K., Akinduro, O., Lo Celso, C. In vivo 4-dimensional tracking of hematopoietic stem and progenitor cells in adult mouse calvarial bone marrow. J Vis Exp. (91), e51683 (2014).
- Jung, Y., et al. Intravital imaging of mouse bone marrow: Hemodynamics and vascular permeability. Methods Mol Biol. 1763, 11-22 (2018).
- Le, V. H., et al. In vivo longitudinal visualization of bone marrow engraftment process in mouse calvaria using two-photon microscopy. Sci Rep. 7, 44097 (2017).
- Spencer, J. A., et al. Direct measurement of local oxygen concentration in the bone marrow of live animals. Nature. 508 (7495), 269-273 (2014).
- Yang, M., Mahanty, A., Jin, C., Wong, A. N. N., Yoo, J. S. Label-free metabolic imaging for sensitive and robust monitoring of anti-CD47 immunotherapy response in triple-negative breast cancer. J Immunother Cancer. 10 (9), e005199 (2022).
- Li, W., et al. Tracking strain-specific morphogenesis and angiogenesis of murine calvaria with large-scale optoacoustic and Ultrasound Microscopy. J Bone Miner Res. 37 (5), 1032-1043 (2022).
- Lassailly, F., Foster, K., Lopez-Onieva, L., Currie, E., Bonnet, D. Multimodal imaging reveals structural and functional heterogeneity in different bone marrow compartments: functional implications on hematopoietic stem cells. Blood. 122 (10), 1730-1740 (2013).
- Rindone, A. N., et al. Quantitative 3D imaging of the cranial microvascular environment at single-cell resolution. Nat Commun. 12 (1), 6219 (2021).
- Mills, W. A., Coburn, M. A., Eyo, U. B. The emergence of the calvarial hematopoietic niche in health and disease. Immunol Rev. 311 (1), 26-38 (2022).
- Bixel, M. G., et al. Angiogenesis is uncoupled from osteogenesis during calvarial bone regeneration. Nat Commun. 15 (1), 4575 (2024).
- Koh, B. I., et al. Adult skull bone marrow is an expanding and resilient haematopoietic reservoir. Nature. 636 (8041), 172-181 (2024).
- Kolabas, Z. I., et al. Distinct molecular profiles of skull bone marrow in health and neurological disorders. Cell. 186 (17), 3706-3725.e9 (2023).
- Rosenhain, S., et al. A preclinical micro-computed tomography database including 3D whole body organ segmentations. Sci Data. 5, 180294 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon