Method Article
Intravitale longitudinale Bildgebung der Gefäßdynamik im Schädeldachknochenmark
In diesem Artikel
Zusammenfassung
Die Intravitalmikroskopie ermöglicht die Untersuchung dynamischer biologischer Prozesse wie Geweberegeneration und Tumorentwicklung. Das Schädeldachknochenmark, ein hochdynamisches Gewebe, bietet Einblicke in die Blutbildung und Gefäßfunktion. Die Verwendung eines biokompatiblen 3D-gedruckten Kopffixationsimplantats ermöglicht eine repetitive Längsbildgebung und verbessert so unser Verständnis der Gewebedynamik und der Mikroumgebung des Tumors.
Zusammenfassung
Die intravitale longitudinale Fluoreszenzmikroskopie hat sich zu einer entscheidenden Technik für die Untersuchung dynamischer biologischer Prozesse entwickelt, insbesondere im Zusammenhang mit der Geweberegeneration, der Tumorentwicklung und dem therapeutischen Ansprechen. Insbesondere das Schädeldachknochenmark ist ein hochdynamisches Gewebe, in dem das hämatopoetische Schicksal mit der umgebenden Mikroumgebung verbunden ist, mit spezialisierten Gefäßen, die auf die normale und pathologische Hämatopoese reagieren. Die traditionelle Bildgebung von fixiertem Gewebe bietet statische Informationen, die ein umfassendes Verständnis dieser Prozesse oft einschränken.
Die Integration von transgenen Tieren, die zellspezifische Marker exprimieren, Lebendzell-Tracer, Fortschritte bei bildgebenden Geräten und die Verwendung spezieller Kammern haben die Intravitalmikroskopie zu einem zentralen Werkzeug gemacht, um Einblicke in dynamische biologische Phänomene zu gewinnen. Eine Anwendung der intravitalen Bildgebung ist die Untersuchung des Verhaltens von Tumorgefäßen und der therapeutischen Wirkungen. Ein neu entwickeltes, 3D-gedrucktes Titan-Kopffixationsimplantat kann stabil mit dem Schädel der Maus verbunden werden und eignet sich für die Längsschnittbildgebung während mehrerer Sitzungen.
Das vorgeschlagene Protokoll ermöglicht die räumliche und zeitliche Untersuchung der vaskulären Dynamik im Schädeldachknochenmark, einschließlich der Visualisierung und Quantifizierung der vaskulären Heterogenität, der Interaktion mit stromalen und hämatopoetischen Zellen und der Messung vaskulärer funktioneller Parameter. Darüber hinaus ermöglicht die Technik die Visualisierung etablierter Gefäßbetten und die Überwachung therapeutischer Effekte, die Mobilisierung von Stammzellen und die Lokalisierung chemotherapeutischer Verbindungen im Zeitverlauf mittels Zwei-Photonen-Mikroskopie. Insgesamt bietet dieses intravitale longitudinale Bildgebungsprotokoll eine umfassende Plattform für die Untersuchung sowohl des Tumorgefäßverhaltens als auch der hämatopoetischen Zelldynamik und bietet wertvolle Einblicke in die komplizierten Prozesse, die diese biologischen Phänomene steuern.
Einleitung
Die intravitale mikroskopische Bildgebung des Schädeldachknochenmarks (BM) dient als leistungsfähige und unverzichtbare Technik zur Untersuchung der dynamischen Prozesse der Hämatopoese, der Regulation der Gewebemikroumgebung und der Gefäßdynamik innerhalb der BM-Nische. Der Hauptzweck dieses methodischen Ansatzes besteht darin, die Echtzeit-Visualisierung und -Analyse von zellulärem Verhalten, Interaktionen und räumlicher Organisation innerhalb der BM-Mikroumgebung in vivo zu ermöglichen. Durch die direkte Beobachtung der BM-Kalvaria mit Hilfe fortschrittlicher bildgebender Verfahren in Verbindung mit Fluoreszenzmarkierung können Forscher das komplexe Zusammenspiel zwischen hämatopoetischen Stammzellen (HSCs), Stromazellen und dem umgebenden Gefäßsystem aufklären und so entscheidende Einblicke in die Regulation der Hämatopoese und der Immunantwort gewinnen.
Die Entwicklung und Nutzung von intravitalen Mikroskopie-bildgebenden Stammzellen aus den Grenzen traditioneller histologischer und ex vivo-Bildgebungsmethoden, die oft nicht in der Lage sind, die dynamische Natur zellulärer Verhaltensweisen und Interaktionen mit einem Gewebe zu erfassen. Im Gegensatz zu statischen bildgebenden Verfahren ermöglicht die Intravitalmikroskopie den Forschern, die zelluläre Dynamik in Echtzeit zu beobachten und so Längsschnittprozesse wie Zellmigration, Proliferation und Differenzierung innerhalb ihrer ursprünglichen Nische zu untersuchen, ohne Versuchstiere zu opfern. Darüber hinaus bietet die Intravitalmikroskopie den einzigartigen Vorteil, funktionelle Verhaltensweisen in vivo zu untersuchen, wie z. B. die Gefäßfunktionalität (z. B. Perfusion, Permeabilität, Hypoxie), wodurch die physiologische Relevanz erhalten bleibt und Artefakte vermieden werden, die mit der Gewebefixierung und -verarbeitung verbunden sind. Bahnbrechende Studien auf diesem Gebiet haben die enormen Vorteile dieses Ansatzes gezeigt 1,2, und ihre Ergebnisse wurden durch neuere verfeinerte Ansätze 3,4,5 bestätigt und erweitert, die die Intravitalmikroskopie nutzten, um die endogene HSC-Lokalisation, Migration und Wechselwirkungen mit dem Gefäßsystem innerhalb der BM-Nische zu verfolgen. Darüber hinaus hat die Intravitalmikroskopie maßgeblich zur Aufklärung der Mechanismen beigetragen, die hämatopoetischen Erkrankungen wie Leukämie und BM-Versagenssyndromen zugrunde liegen, und bietet neue Einblicke in die Beweglichkeit leukämischer Zellen 6,7, die krankheitsassoziierten vaskulären Implikationen8 und das Ansprechen auf Medikamente9.
Es gibt mehrere Vorteile der intravitalen mikroskopischen Bildgebung des Schädeldach-BM, die alternative Knochenstellen abdeckt. Erstens bietet das im Schädelknochen enthaltene BM im Vergleich zu tieferen Knochen wie dem Femur oder der Tibia einen einfachen Zugang für die intravitale Bildgebung. Diese Zugänglichkeit erleichtert die direkte Beobachtung der Mikroumgebung des Gewebes, einschließlich des Knochens selbst, über die Visualisierung der zweiten harmonischen Erzeugung (SHG)10, ohne dass invasive chirurgische Eingriffe erforderlich sind. Zweitens ist der Schädel relativ dünn und durchscheinend, was eine bessere Visualisierung der zellulären Dynamik innerhalb der BM-Nische ermöglicht. Diese Transparenz ermöglicht eine hochauflösende Bildgebung mit Zwei-Photonen-Mikroskopie, konfokaler Mikroskopie und Lichtblattmikroskopie, ohne dass Knochenverdünnungs- oder -reinigungstechniken erforderlich sind. Der Schädeldachknochen bietet eine stabile, flache und starre Plattform für intravitale Bildgebungsexperimente, minimiert Gewebebewegungsartefakte und gewährleistet konsistente Bildgebungsbedingungen über längere Beobachtungszeiträume. Diese Stabilität ist besonders vorteilhaft für Längsschnittstudien, die zelluläre Verhaltensweisen und Reaktionen über die Zeit verfolgen. Ein weiterer großer Vorteil ist die Reproduzierbarkeit, da die Struktur des Schädeldachknochens bei Versuchstieren relativ klein und räumlich definiert ist. Diese Einheitlichkeit erleichtert den Vergleich zwischen verschiedenen Versuchsgruppen und ermöglicht eine robuste statistische Analyse von Bilddaten.
Hier beschreiben wir eine Methode zur Abbildung des Schädeldach-BM der Maus mittels intravitaler Zwei-Photonen-Mikroskopie durch die Einführung eines neu entwickelten Kopffixationsimplantats11, das unter Verwendung einer biokompatiblen Titanlegierung Grad 23 (Ti6Al4V) 3D-gedruckt wurde und über eine spezielle und leicht zu positionierende Hardcover aus derselben Titanlegierung verfügt, die einen sicheren Verschluss der Wunde ermöglicht, um Infektionen oder Schäden an der Operationsstelle zu vermeiden. Das Implantat fixiert den Mauskopf über einen Implantathalter fest und stabil auf dem Mikroskoptisch, wodurch Atemartefakte minimiert werden und eine Längsdarstellung der gleichen Bereiche über die Zeit ermöglicht wird. Es werden einige Beispiele für mehrfarbige Bilder gegeben, die Zellen und Strukturen aus der BM-Mikroumgebung darstellen (SHG+ Knochenoberfläche; Nestin-GFP+ mesenchymale Zellen; cdh5-DSRED+, cdh5-GFP+ oder pdgfb-GFP+ Endothelzellen) und das maligne hämatopoetische Kompartiment (tdTOMATO+ AML-Zellen) sowie fluoreszierende Kontrastmittel, die das Lumen der Gefäße darstellen (Dextran-TRITC). Über Stunden oder Tage gemessene vaskuläre Parameter, darunter Gefäßlänge, Geradheit und Durchmesser sowie die Permeabilität in verschiedenen Gefäßregionen, können wichtige Informationen über das Verhalten und die Gesundheit des Gewebes liefern.
Protokoll
Alle Tierversuche wurden im Rahmen der ethischen Vereinbarung APAFIS#27215-2020041513522374 v6 durchgeführt, die vom französischen "Ministère de l'enseignement supérieur, de la recherche et de l'innovation" genehmigt wurde.
1. Design eines biokompatiblen Titan-Kopffixationsimplantats für den 3D-Druck
HINWEIS: Wir haben ein biokompatibles Kopffixationsimplantat unter Verwendung einer parametrischen Modellierungssoftware für computergestütztes Design (CAD) mit integrierten Funktionen für die Finite-Elemente-Analyse (FEA) entwickelt (siehe Materialtabelle). Zu den wichtigsten Eingaben für den Designprozess gehören ein hochauflösendes anatomisches Modell des Schädels der Maus, ein Modell des Mikroskopobjektivs und ein Modell des Fixationssystems, die zusammen die Abmessungen und die Konfiguration des Implantats bestimmen. Die endgültige Ausgabe ist eine plattformunabhängige Mesh-Datei, in der Regel im STL- oder STEP-Format, die mit den meisten 3D-Druckprogrammen kompatibel ist. Dieses Dateiformat gewährleistet eine nahtlose Übertragung auf den 3D-Drucker für eine präzise Implantatherstellung.
- Vorbereitung des 3D-Modells der Maus
- Positionieren Sie den Schädel der Maus, präpariert oder in vivo, im Mikro-CT-Scanner.
- Stellen Sie die Scanparameter für eine optimale Auflösung feiner anatomischer Details ein: Öffnen Sie die Systemsteuerung und wählen Sie im Abschnitt Scanbedingungen ein Sichtfeld (FOV) aus. Verwenden Sie LiveView , um das Scan-Volumen zu definieren.
HINWEIS: Durch die Detektorauflösung bestimmt das FOV die Voxelgröße basierend auf der Detektorauflösung. In neueren Mikro-CT-Geräten entspricht ein Sichtfeld von 5 mm in der Regel einer Voxelgröße von 5 μm, einem Sichtfeld von 10 mm bis einer Voxelgröße von 10 μm und einem Sichtfeld von 25 mm bis einer Voxelgröße von 21 μm. Das begrenzte Sichtfeld bei dieser Auflösung kann mehrere Scans erfordern, um den gesamten Schädel abzudecken. Mehrere Scans erfordern eine Überlappung zwischen den Scans. - Fügen Sie mehrere hochauflösende Scans in einer Bildgebungssoftware zusammen, um ein einziges, umfassendes Modell des Schädels zu erstellen.
- Importieren Sie Micro-CT-DICOM-Dateien in eine Segmentierungssoftware (z. B. 3D Slicer, Amira, Avizo oder Mimics), um den Schädel zu isolieren.
- Führen Sie eine schwellenwertbasierte Segmentierung durch, um Knochengewebe von umgebenden Strukturen zu trennen, und nehmen Sie manuelle Anpassungen vor, um komplexe oder verrauschte Bereiche zu verfeinern.
- Importieren Sie in 3D Slicer zuerst DICOM-Dateien, laden Sie den Datensatz und beobachten Sie die CT-Schichten in den axialen, sagittalen und koronalen Schnittansichten.
- Öffnen Sie das Segment-Editor, klicken Sie auf Hinzufügen , um eine neue Segmentierung zu erstellen | Hinzufügen , um ein neues Segment zu erstellen, das mit einem Schwellenwert versehen werden soll.
- Klicken Sie im Bereich Segmenteditor auf den Effekt Schwellenwert . Passen Sie die Schieberegler für den unteren und oberen Schwellenwert an (oder geben Sie numerische Werte ein), sodass der zu segmentierende Bereich in den Segmentansichten rot hervorgehoben wird. Ein 3D-Segment mit Schwellenwerten ist fertig.
- Um den segmentierten Schädel als STL-Datei zu exportieren, wechseln Sie zum Segmentierungsmodul. Stellen Sie sicher, dass das Segment mit Schwellenwert ausgewählt ist. Legen Sie im Abschnitt Modelle und Labelmaps exportieren/importieren den Exporttyp auf Modelle fest. und Dateiformat: STL. Klicken Sie auf die Schaltfläche Exportieren.
HINWEIS: STL-Dateien verwenden ein dreieckiges Netzformat, das die Oberfläche des Schädels mit Tausenden von Dreiecken darstellt und die anatomische Form bis ins kleinste Detail erfasst.- Führen Sie bei Bedarf das Stitching in MeshLab wie folgt durch:
- Importieren Sie Netze, indem Sie auf Datei | Importieren Sie ein Mesh und wählen Sie eines als Referenzscan aus.
- Richten Sie andere Scans mit dem Ausrichten-Werkzeug aus (Filter | Anmeldung | Ausrichten), indem Sie entsprechende Punkte markieren und die Option Verarbeiten verwenden.
- Führen Sie die ausgerichteten Scans zusammen, indem Sie auf Filter | Mesh-Schicht | Reduzieren Sie sichtbare Ebenen mit der Option Nur sichtbare Ebenen zusammenführen .
- Führen Sie bei Bedarf das Stitching in MeshLab wie folgt durch:
- Reinigen und reparieren Sie das Netz, indem Sie doppelte Scheitelpunkte und Dreiecke wie folgt entfernen, um sicherzustellen, dass rauschbedingte Dreiecke entfernt und unbeabsichtigte Löcher gefüllt werden, um eine glatte und kontinuierliche Oberfläche zu erhalten:
- Klicken Sie auf Filter | Reinigung und Reparatur | Entfernen Sie duplizierte Scheitelpunkte.
- Klicken Sie auf Filter | Reinigung und Reparatur | Entfernen Sie duplizierte Flächen.
- Klicken Sie auf Filter | Reinigung und Reparatur | Entfernen Sie isolierte Teile (nach Flächennummer). Geben Sie einen Schwellenwert für die Mindestanzahl von Flächen an, die beibehalten werden sollen (legen Sie diesen Schwellenwert z. B. niedrig fest, um kleine Cluster oder Rauschen zu entfernen).
- Klicken Sie auf Filter | Neuvernetzung, Vereinfachung und Rekonstruktion | Löcher schließen.
- Führen Sie eine Netzvereinfachung auf ~10.000-30.000 Dreiecke durch, da die anfänglichen STL-Dateien von micro-CT sehr hohe Dreieckszahlen enthalten, die nicht mit der CAD-Software kompatibel sind. Behalten Sie in Bereichen mit hohen Details kleinere Dreiecke auf der Oberseite des Schädels bei, um eine genaue Passform des Implantats zu gewährleisten. Vereinfachen Sie in gröberen Bereichen weniger kritische Bereiche mit größeren Dreiecken, um die Dateigröße zu reduzieren und die Verarbeitungseffizienz zu optimieren.
HINWEIS: Die Vereinfachung des Netzes erfolgt nach Bereichen und wird in den Abschnitten 1.1.8.1-1.1.8.3 detailliert beschrieben.- Verwenden Sie Verbundene Komponenten in einem Bereichsauswahlwerkzeug auswählen, um Bereiche zur Vereinfachung auszuwählen.
- Verwenden Sie die Dezimierung des quadratischen Kantenkollapses für die ausgewählten Bereiche mit Parametern. Klicken Sie auf Filter | Neuvernetzung, Vereinfachung und Rekonstruktion | Vereinfachung: Dezimierung des quadratischen Kantenkollapses. Setzen Sie den Qualitätsschwellenwert auf 0,8, die Begrenzung des Netzes beibehalten auf JA, die Topologie auf JA beibehalten, die Zielanzahl der Flächen: Legen Sie eine Zielzahl fest, die der erforderlichen Auflösung entspricht.
- Iterieren Sie nach Bedarf, bis die Gesamtzahl der Dreiecke auf das gewünschte Niveau reduziert ist. Speichern Sie dann das vereinfachte Modell in einer STL-Datei.
- Aufbereitung des Schädelmodells im CAD-Modell
- Importieren Sie das vereinfachte Schädelmodell im STL-Format als neues Teil oder Modell in die CAD-Software. Stellen Sie sicher, dass die importierte Datei je nach den Fähigkeiten der Software ordnungsgemäß in einen Volumen- oder Flächenkörper konvertiert wird. Speichern Sie das Modell im nativen CAD-Dateiformat, um weitere Änderungen zu ermöglichen und die Kompatibilität mit nachfolgenden Konstruktionsschritten sicherzustellen.
- Platzieren Sie Referenzpunkte auf Bregma und Lambda und erstellen Sie die Bregma-Lambda-Achse in diesem Modell.
- Erstellen Sie die Medianebene einschließlich der Bregma-Lambda-Achse.
- Erstellen Sie in der Medianebene eine Achse, die tangential zum Schädel verläuft, und speichern Sie dann den präparierten Schädel (Ergänzende Abbildung S1).
- Erstellen Sie eine neue Baugruppe und setzen Sie den vorbereiteten Schädel so ein, dass Bregma mit dem Ursprung, der Medianebene mit der rechten Ebene, übereinstimmt und die Schädeldachachse horizontal ist.
- Das Kopffixationsimplantat wird direkt in der Baugruppe entworfen, um eine perfekte Passform mit dem präparierten Schädelmodell zu gewährleisten. Um dies zu erreichen, legen Sie das Implantat als neues Teil innerhalb der Baugruppe an. Navigieren Sie zunächst zu Einfügen | Komponente | Neues Teil in der CAD-Software. Wenn Sie dazu aufgefordert werden, wählen Sie die Schädelfläche oder die obere Ebene der Baugruppe als Referenz aus, um das neue Teil zu positionieren und zu verankern.
- Erstellen Sie einen Satz gleichmäßig verteilter Ebenen (2 mm Abstand) über den Schädel. Erstellen Sie auf jeder Ebene eine Skizze, und zeichnen Sie auf jeder Skizze eine um 0,2 mm versetzte projizierte Kurve in Form eines symmetrischen Splines mit neun Punkten, wobei Sie dem Schnitt der oberen Oberfläche des Schädels 0,2 mm über dem Schädel folgen (Ergänzende Abbildung S2).
- Erstellen Sie mit diesen Splines eine gesweepte Oberfläche - die Skull Limit Surface -, die sicherstellt, dass diese Oberfläche glatt und ohne Defekte ist, die für die Konstruktion der Implantatmerkmale verwendet werden kann.
- Definition der beobachtbaren Oberfläche auf dem Schädel
HINWEIS: Basierend auf dem 3D-Modell wird die flachste Region des Schädeldachknochens identifiziert, die sich ideal für die 3D-Betrachtung eignet. Dieser Bereich wird sorgfältig ausgewählt, um einen optimalen Zugang für das Objektiv des Zwei-Photonen-Mikroskops zu gewährleisten und gleichzeitig die biologische und strukturelle Integrität des Schädels zu berücksichtigen. Bei der Definition können stereotaktische Koordinaten verwendet werden, wobei der Bregma-Punkt als Ursprung des Koordinatensystems dient.- Erstellen Sie eine Skizze in der Schädelbahn, und erstellen Sie einen birnenförmigen Spline von AP +6,5 bis -2, 6 mm Breite bei AP 0,0. Dabei handelt es sich um die beobachtbare Oberflächenkontur (Ergänzende Abbildung S3).
HINWEIS: Das Mikroskopobjektiv wird virtuell relativ zu der zuvor definierten beobachtbaren Oberfläche auf dem Schädel positioniert. In diesem Schritt werden die räumlichen Einschränkungen bestimmt, die durch die Größe und Form des Objektivs auferlegt werden, um sicherzustellen, dass das Implantatdesign den Anforderungen des Mikroskops entspricht. - Setzen Sie das Mikroskopobjektivmodell vertikal in die Baugruppe ein. Beschränken Sie den Fokuspunkt auf eine Ebene, die parallel zur Schädeldachebene des Schädeldachknochens verläuft, und definieren Sie das Begrenzungsvolumen des Mikroskopobjektivs.
- Erstellen Sie ein Volumen, indem Sie das Ziel entlang der beobachtbaren Oberflächenkontur ziehen.
- Erstellen Sie ein Beobachtungsfenster um die definierte beobachtbare Oberfläche, um einen ungehinderten Zugang zum Mikroskopobjektiv zu gewährleisten.
HINWEIS: Das Beobachtungsfenster ist eine ringförmige Struktur mit sekundären Funktionen wie dem Zurückhalten von Tauchflüssigkeit und der festen Struktur für das korrekte Schließen der Schutzabdeckung. - Erstellen Sie eine Skizze in der Schädelebene, und kopieren Sie die beobachtbare Oberflächenkontur.
- Erstellen Sie eine parallele Kontur mit einer Dicke von 0,5 mm. Extrudieren Sie diese Doppelkontur 0,9 mm nach oben und nach unten zur Oberfläche Skull Limit Surface mit einem Entformungswinkel von 4°.
- Erstellen Sie eine Skizze in der Schädelbahn, und erstellen Sie einen birnenförmigen Spline von AP +6,5 bis -2, 6 mm Breite bei AP 0,0. Dabei handelt es sich um die beobachtbare Oberflächenkontur (Ergänzende Abbildung S3).
- Definieren der Zement-, Schwanz- und Schwalbenschwanzstrukturen.
- Erstellen Sie eine Skizze in der Schädelebene und zeichnen Sie eine 0,5 mm dicke C-Form, die mit dem Beobachtungsfenster verbunden ist, und extrudieren Sie sie 2 mm hoch und nach unten zur Oberfläche Schädelgrenzfläche mit einem Entformungswinkel von 4° (Ergänzende Abbildung S4).
- Erstellen Sie eine Skizze in der Schädelbahn, und füllen Sie diesen Raum mit 0,4 mm dicken Rechtecken, die radial mit einem Abstand von 1 mm dazwischen angeordnet sind.
- Extrudieren Sie auf ähnliche Weise wie das Beobachtungsfenster.
- Erstellen Sie den Schwanz des Implantats, indem Sie sich auf den Schädel zur Fixierung verlassen, indem Sie das verfügbare Volumen berücksichtigen.
- Es entsteht eine Querebene, die tangential zur Zementstruktur verläuft und weitere 12,5 mm nach hinten.
- Skizzieren Sie Rechtecke auf diesen Ebenen. Ziehen Sie einen pyramidenförmigen Körper dazwischen und stellen Sie die Winkel etwa 20° und 37° von der Horizontalen ein.
- Fügen Sie ein M1,6-Gewindeloch in den Schwanz in der Nähe der Zementstruktur ein.
- Erstellen Sie eine Skizze am Ende des Schwanzes, und zeichnen Sie das Schwalbenschwanzprofil.
- Extrudieren Sie dieses Profil um 8 mm.
- Überprüfung der Nichteinmischung in das Ziel.
- Untersuchen Sie das gesamte Implantatdesign, einschließlich des Beobachtungsfensters und des Schwalbenschwanzes, um sicherzustellen, dass keine mechanischen Interferenzen mit dem Mikroskopobjektiv auftreten. Überprüfen Sie, ob das in Schritt 1.3.3 erstellte Volume gestört ist.
HINWEIS: Dieser Überprüfungsschritt ist entscheidend, um sicherzustellen, dass das Objektiv wie gewünscht ohne Behinderung positioniert und bewegt werden kann.
- Untersuchen Sie das gesamte Implantatdesign, einschließlich des Beobachtungsfensters und des Schwalbenschwanzes, um sicherzustellen, dass keine mechanischen Interferenzen mit dem Mikroskopobjektiv auftreten. Überprüfen Sie, ob das in Schritt 1.3.3 erstellte Volume gestört ist.
- Entfernen Sie unnötiges Material vom Schwanz des Implantats, um das Gewicht zu minimieren und mögliche Beschwerden für das Tier zu reduzieren.
- Erstellen Sie eine Skizze in der Mittelebene, und zeichnen Sie dreieckige Ausschnitte darauf, um eine Fachwerkstruktur zu bilden, wenn Sie sie durch Extrusion aus dem Leitwerk extrahieren.
- Wiederholen Sie den Vorgang in der senkrechten Ebene.
- Überprüfen Sie die maximale Verschiebung durch die Finite-Elemente-Methode (FEM)-Simulation, um die mechanischen Eigenschaften des Implantats unter verschiedenen Bedingungen zu analysieren.
HINWEIS: Diese Simulation hilft bei der Vorhersage der maximalen Verschiebung und der Spannungen innerhalb des Implantats während des Gebrauchs und gewährleistet so seine Zuverlässigkeit.- Aktivieren Sie das CAD-Software-Simulations-Add-In.
- Erstellen Sie in der Symbolleiste Simulation eine neue statische Studie. Navigieren Sie zur Registerkarte Simulation (falls sichtbar) oder gehen Sie zu Simulation | Studie. Wählen Sie im Dialogfeld Studie die Option Statisch als Studientyp aus. Klicken Sie auf OK , um die neue Studie zu erstellen (Ergänzende Abbildung S5).
HINWEIS: Dieser Studientyp eignet sich für die Analyse von Verformungen und Spannungen unter statischen Belastungen, wie sie bei der Verwendung des Implantats zu erwarten sind. - Wählen Sie ein Material für das Implantat, das der tatsächlichen Implantatzusammensetzung (Ti6Al4V) entspricht.
- Definieren Sie feste Randbedingungen, um die Befestigungspunkte des Implantats zu simulieren, an denen es die Schwalbenschwanzfixierung berühren würde. Definieren Sie die obere Fläche des Schwalbenschwanzes als fest.
- Definieren Sie die vertikale Belastung der Unterseite des Implantats. Stellen Sie 1 N Vertikalkraft ein.
- Generieren Sie ein Netz. Verfeinern Sie das Netz in Bereichen, in denen hohe Spannungen oder Verformungen zu erwarten sind (Ergänzende Abbildung S6). Klicken Sie dazu mit der rechten Maustaste auf das Netzelement des Studienbaums und wählen Sie Netz erstellen... Passen Sie im Dialogfeld Netz die Netzdichte entweder mit dem Schieberegler an oder indem Sie das Feld Netzsteuerung aktivieren, um die maximale und minimale Elementgröße unabhängig voneinander festzulegen. Klicken Sie auf OK , um das Netz zu generieren.
HINWEIS: Die Software generiert automatisch ein erstes Netz für die Studie, aber für genauere Ergebnisse kann das Netz verfeinert werden, um ein feineres Netz auszuwählen, insbesondere in Bereichen, die für die Implantatstabilität entscheidend sind (wie z. B. das Beobachtungsfenster). Eine feinere Vernetzung in diesen Bereichen verbessert die Genauigkeit der Simulation, indem lokale Spannungskonzentrationen erfasst werden. - Starten Sie die statische Analyse, um die Verschiebungs-, Spannungs- und Dehnungsverteilungen über das Implantat zu berechnen.
- Analysieren Sie die Ergebnisse. Untersuchen Sie das Verschiebungsdiagramm, um Bereiche mit maximaler Verformung zu identifizieren. Dieses Diagramm zeigt, wie stark sich das Implantat unter den aufgebrachten Lasten verformen würde, und hebt alle Bereiche hervor, in denen das Risiko einer übermäßigen Bewegung besteht. Stellen Sie sicher, dass die Verformung im Inneren des Beobachtungsfensters 3 Mikrometer nicht überschreitet.
- Entwerfen Sie schließlich eine Schutzabdeckung, um das Beobachtungsfenster zu schützen, wenn es nicht verwendet wird. Dieser Deckel schützt den freiliegenden Knochen vor Umweltschadstoffen und physischen Schäden.
2. Behandlungen bei Mäusen, Anästhesie und chirurgische Implantation des Kopfimplantats
HINWEIS: Hier können entweder männlich oder weiblich, 7 bis 12 Wochen alte C57BL/6 oder transgene Mäuse verwendet werden, wie abgebildet. Um eine Leukämiebesiedlung des BM zu induzieren, werden die erzeugten leukämischen Zellen, wie von Horton et al.12 beschrieben, 2-3 Wochen vor der Bildgebung intravenös verabreicht. Um die Gesundheit der Wunde zu gewährleisten, müssen sterile Techniken angewendet werden.
- Unter der Haube montieren Sie sterile Operationswerkzeuge (stumpfe Schere und Pinzette), Trimmer und die Verbrauchsmaterialien, einschließlich Augengel, Kochsalzlösung, Desinfektionsmittel, Zahnzement, saugfähige Tupfer und Reinigungstuch. Schalten Sie den Bead-Sterilisator ein.
- Füllen Sie die Narkose-Induktionskammer mit 4% Isofluran und Luftsauerstoff und setzen Sie die Maus in die Kammer. Wenn das Tier vollständig betäubt ist (auf Verlust des Aufrichtreflexes und tieferes und langsameres Atemmuster prüfen), schalten Sie den Isofluranfluss auf den Nasenkegel der Anästhesiemaske um und senken Sie die Isoflurankonzentration auf 2%.
- Legen Sie die Maus auf ein 37 °C heißes Heizkissen und überwachen Sie die Atemfrequenz visuell. Optional: Bei Mäusen mit einem pathologischen Phänotyp fügen Sie eine Überlebensdecke hinzu, um die richtige Temperatur während der gesamten Narkose zu erhalten.
- Verabreichen Sie 30 min vor der Operation subkutan Schmerzmittel (Buprecare, 0,01 mg/kg).
- Rasieren Sie den Mauskopf mit einem Elektrorasierer.
- Geben Sie einen Tropfen Augengel auf die Augen der Maus, um Trockenheit während der Narkose zu verhindern, und stellen Sie sicher, dass das Gel nicht auf die Kopfhaut tropft.
- Reinigen Sie die Maus, um alle Resthaare aus dem Operationsbereich zu entfernen. Tupfen Sie die Oberseite der Kopfhaut mit Desinfektionsmittel auf ein Baumwolltuch ab. Achten Sie darauf, alle Haare zu entfernen, um bildgebende Artefakte sowie das Risiko einer Wundinfektion zu vermeiden.
- Machen Sie mit einer sterilen Pinzette und Schere (nach 30 s im Sterilisator) einen kleinen Schnitt im zentralen Teil der Kopfhaut, um die zentrale Knochennarbe freizulegen. Befolgen Sie diese Angaben, um den Bildbereich für die richtige Länge und Breite zu definieren. Entfernen Sie vorsichtig das Stück der Kopfhaut, heben Sie die Haut zwischen den Ohren mit einer Pinzette an und machen Sie einen kleinen Schnitt am Hinterkopf. Halten Sie die Haut hoch, führen Sie die Schere unter die Haut und schneiden Sie vorsichtig außerhalb des Bildgebungsbereichs.
- Entfernen Sie das Bindegewebe zwischen dem Schädel und der Kopfhaut. Wischen Sie den freiliegenden Knochen mit sterilem PBS auf einem Wattestäbchen ab. Fahren Sie schnell mit der Befestigung des Kopfimplantats am Schädel der Maus fort, um eine längere Freilegung des Schädelknochens (<10 min) zu vermeiden.
- Stellen Sie eine Paste mit ausreichend Zahnzement in einer Petrischale gemäß den Anweisungen des Herstellers her und tragen Sie sie schnell auf die Unterseite des Kopffixationsimplantats auf.
- Platzieren Sie den Kopfhalter auf dem freiliegenden Schädel der Maus und warten Sie, bis er fest ist, ohne dass Zahnzement in den Bildgebungsbereich gelangt.
- Nach dem Abbinden (normalerweise innerhalb von 3-5 Minuten) geben Sie einige Tropfen 37 °C PBS in die Bildgebungskammer, um den Schädel mit Feuchtigkeit zu versorgen.
3. Bildgebung mit einem Zwei-Photonen-Mikroskop
- Schalten Sie vor Beginn der Operation den Laser ein und lassen Sie ihn sich aufwärmen, um sich zu stabilisieren. Schalten Sie das Mikroskop ein und starten Sie die Erfassungssoftware. Wählen Sie das passende Objektiv (25x/0,95 WATER Immersion für dieses Experiment).
- Verwenden Sie ein aufrechtes Mikroskopstativ mit festem Tisch, das genügend Platz bietet, um ein veterinärmedizinisches Steckbrett mit einem Heizkissen zu platzieren, das alle integral mit dem xy-Tisch bewegt werden kann. Verwenden Sie ein optisches Steckbrett aus massivem Aluminium als Plattform, um das Kopfimplantat und die stereotaktische Maske zu befestigen. Verbinden Sie flexible Schläuche und deren Anschlüsse, die für die Isofluran-Injektion und -Extraktion verbunden sind, mit der Maske im Mikroskopkäfig.
- Konfigurieren Sie die Aufnahmeeinstellungen so, dass schnell gescannt wird: Format 512 x 512 Pixel; Geschwindigkeit 600 Hz; und Zoomfaktor 1.
- Stimmen Sie die Laserlinie auf 880 nm ab, um SHG, GFP und tdTOMATO mit der gleichen Anregungswellenlänge detektieren zu können.
- Schalten Sie das Licht im Raum aus und schließen Sie den Kasten um das Mikroskopstativ ordnungsgemäß, bevor Sie die NDD-Detektoren aktivieren, und definieren Sie die spektralen Detektionsgates: PMT1, um SHG [388 - 431 nm] zu detektieren; HyD2 für GFP [485 - 548 nm] und HyD3 für tdTOMATO [551 - 645 nm]. Belassen Sie den Offset bei 0.
- Erstellen Sie ein neues Projektdataset, und benennen Sie es entsprechend dem jeweiligen Experiment um.
- Sobald das Kopffixationsimplantat am Schädel der Maus befestigt ist (bestätigt durch die Zementhärtung), schalten Sie das Isofluran in Richtung der stereotaktischen Maske des Mikroskops ein.
- Bringen Sie die Maus schnell ans Mikroskop. Führen Sie die Mauszähne vorsichtig in die Maske ein, um das Eindringen von Isofluran durch die Mausnase zu ermöglichen, indem Sie die Nase mit einer Hand anheben.
- Während Sie die Maus mit einer Hand halten, schieben Sie mit der anderen Hand den Schwalbenschwanz des Kopffixationsimplantats vorsichtig in den Fixierhalter und sichern Sie ihn mit einer halben Umdrehung des Schraubknopfes.
- Führen Sie eine rektale Sonde ein, die mit einem Gel auf Wasserbasis zur Temperaturüberwachung voreingebettet ist, und geben Sie ophthalmische Tropfen auf die Augen der Maus.
- Füllen Sie die Bildgebungskammer des Kopfes mit einer großen Menge Gel oder PBS auf Wasserbasis und senken Sie das Wasserimmersionsobjektiv ab, um es vollständig einzutauchen, um eine optimale Anregung und Detektion der durch die Zwei-Photonen-Anregung erzeugten Signale zu erzielen.
- Verschieben Sie den x-y-Tisch und den z-Antrieb, um sich auf den zentralen Knochen zu konzentrieren, in die Gerüstnarbe.
HINWEIS: Es ist zweckmäßig, die Okulare des Mikroskops und eine Halogen-Metalldampflampe als Lichtquelle zu verwenden, die durch einen Tri-Band-Anregungsemissionsfilter gefiltert wird. Das Fehlen von optischen Schnitten macht es unmöglich, ein klares Bild zu erhalten; Aber mit etwas Übung ist der zentrale Bereich des Schädels leicht zu erkennen. - Schalten Sie nach der Identifizierung des Gewebes, das durch eine Knochenoberfläche und eine zentrale Vene gekennzeichnet ist, die Halogen-Metalldampflampe aus und schließen Sie die Fenster des Mikroskopkäfigs, um die HyD-Detektoren zu schützen und den Infrarot-Laser (IR) passieren zu lassen.
- Stellen Sie die PMT/SHG-Verstärkung auf 850 V (linearen Betriebsbereich) und die HyD-Verstärkung auf 100 % ein. Erhöhen Sie die IR-Laserleistung, bis auf dem untersten der drei Kanäle ein Bild mit einem Dynamikumfang von 200 Graustufen (mit dynamischen 8-Bit-Detektoren) erhalten wird. Reduzieren Sie die Verstärkungen der anderen Detektoren, wenn die Emissionsunterschiede zu groß sind oder wenn eine Sättigung vorliegt, indem Sie den LUT-Modus (Under Lower) verwenden.
HINWEIS: In unserem Fall reichte eine Laserleistung von 30% aus, um Kontrast in allen Kanälen zu erzielen. - Suchen Sie in der Akquisitionssoftware nach einer Region, die aus BM-Taschen (GFP+, tdTOMATO+) besteht, die von den Knochenoberflächen (SHG) umschlossen sind.
- Um verschiedene ROIs zu finden, aktivieren Sie LAS NAVIGATOR und erstellen Sie im Spiralmodus einen Überblick über die gesamte Region. Stoppen Sie die Erfassung, wenn die erfasste Oberfläche groß genug ist. Speichern Sie die Übersicht, fügen Sie sie zusammen und benennen Sie sie um: Dies wird das Referenzbild im Falle der Längsschnittbildgebung sein.
HINWEIS: Achten Sie darauf, keine zu große Spirale zu erzeugen, da dies dazu führen kann, dass der Gerätehalter mit dem Objektiv in Berührung kommt. - Erfassen Sie einige ROI-Positionen, indem Sie auf das Einzelbildsymbol klicken und jede Position in der Aufgabenliste umbenennen. Machen Sie einen Bildschirm, um die relative Position der verschiedenen ROIs über die zuvor gespeicherte Übersicht zu legen.
HINWEIS: Diese Bildschirmaufnahme ist wichtig, um die ROIs in der Längsschnittbildgebung zu ersetzen, da die Erfassungssoftware es nicht zulässt, diese Gruppe von Positionen in der für dieses Experiment verwendeten Version abzurufen. - Um ein Z-Stack-Volume zu erfassen, wählen Sie den Z-Stack-Modus, definieren Sie die Schrittweite bei 3 μm und deaktivieren Sie die Option Gleiche Stack-Größe für alle Regionen . Wählen Sie die erste Position aus, definieren Sie die obere und untere Position, überprüfen Sie die Z-Schrittgröße und klicken Sie auf Stapel neu definieren. Ein Würfelsymbol wird neben dem Namen der Position in der Positionsliste angezeigt. Wiederholen Sie diesen Schritt für alle Positionen der verschiedenen ROIs.
- Drücken Sie auf "Erfassen " und speichern Sie die Bilder (drei Kanäle, Z-Stapel mit ~50 Plänen) im entsprechenden Ordner. Dieser Schritt markiert dieT-Strg des Experiments; Überprüfen Sie, ob die Erfassungsparameter korrekt sind (erhalten Sie den korrekten Dynamikbereich in den drei Kanälen, vermeiden Sie die Signalsättigung so weit wie möglich und überprüfen Sie die Erfassungszeit für einen vollständigen Zyklus).
- Um ein dynamisches Merkmal (in diesem Fall die Gefäßpermeabilität) zu messen, erfassen Sie einen Zeitraffer.
- In diesem Aufbau ermöglichen die drei Detektoren die gleichzeitige Abbildung von drei Kanälen; Wenn für das Experiment ein vierter Kanal benötigt wird (in diesem Fall Dextran-Monitoring), aktivieren Sie den sequentiellen Modus und fügen Sie eine zweite Erfassungssequenz hinzu. Stimmen Sie die IR-Laserlinie auf 820 nm ab, um das Dextran Cy5-Fluorophor anregen zu können. Ändern Sie den Hyd2-Nachweisbereich auf [650 - 744 nm] und deaktivieren Sie die anderen Detektoren. Wählen Sie zwischen den Stack-Erfassungsmodi .
HINWEIS: Standardmäßig hat die zweite Erfassungssequenz die gleichen Parameter wie Sequenz 1. - Starten Sie vor dem Start des Zeitraffers die Erfassung aller Positionen, um sicherzustellen, dass die Aufnahmezeit 3 Minuten (unter Berücksichtigung der Hinzufügung des sequenziellen Modus) nicht überschreitet, die Mindestzeit, die erforderlich ist, um das Dextran-Leck zu erkennen.
- Ändern Sie vor der Dextran-Injektion den Erfassungsmodus auf xyzt. Stellen Sie im t-Modul das Zeitintervall auf 3 min und die Dauer auf 1 h ein.
- In diesem Aufbau ermöglichen die drei Detektoren die gleichzeitige Abbildung von drei Kanälen; Wenn für das Experiment ein vierter Kanal benötigt wird (in diesem Fall Dextran-Monitoring), aktivieren Sie den sequentiellen Modus und fügen Sie eine zweite Erfassungssequenz hinzu. Stimmen Sie die IR-Laserlinie auf 820 nm ab, um das Dextran Cy5-Fluorophor anregen zu können. Ändern Sie den Hyd2-Nachweisbereich auf [650 - 744 nm] und deaktivieren Sie die anderen Detektoren. Wählen Sie zwischen den Stack-Erfassungsmodi .
- Injizieren Sie 100 μl Dextran 70 KDa-TRITC (oder 500 KDa-Cy5) in einer Dosis von 3 mg/Maus intravenös und beginnen Sie mit der Erfassung.
- Speichern Sie den Datensatz nach der Erfassung im entsprechenden Ordner.
HINWEIS: Überwachen Sie während dieser zahlreichen Schritte häufig die Atemfrequenz und -temperatur der Maus und passen Sie den Isofluranfluss bei Bedarf an. Im Idealfall könnten Temperatur, EKG und Atemfrequenz aufgezeichnet und als Metadaten des Experiments eingebunden werden.
4. Wiederherstellung der Maus
- Sobald die Bildgebungssitzung abgeschlossen ist, deaktivieren Sie den HyDs-Detektor in der Erfassungssoftware und schalten Sie die Maus-Heizbox (37 °C) ein. Öffnen Sie den Mikroskopkäfig, heben Sie das Objektiv an und entfernen Sie die Rektalsonde aus der Maus. Schieben Sie anschließend den Schwalbenschwanz des Kopffixationsimplantats vorsichtig aus dem Fixationshalter und bewegen Sie die Maus von der stereotaktischen Maske des Mikroskops zur OP-Maske auf dem Wärmekissen.
- Schalten Sie die Isofluran-Injektion unter dem Mikroskop aus und schalten Sie das Wärmekissen aus.
- Entfernen Sie das wasserbasierte Gel oder PBS vorsichtig mit einem sterilen Tupfer vom Schädel der Maus.
- Fügen Sie Intrasite-Gel hinzu, um die Schädelfeuchtigkeit zwischen den Bildgebungssitzungen aufrechtzuerhalten, wie zuvor beschrieben13.
- Verschließen Sie den bildgebenden Bereich des Kopfimplantats mit der speziellen Abdeckung und befestigen Sie diese vorsichtig mit der Schraube.
- Lege die Maus bei 37 °C in die Heizbox und warte, bis sie aufwacht. Bringen Sie die Maus in die Tierhaltung und bringen Sie sie in einem sauberen Käfig mit Hydrogel und Anreicherung unter. Überwachen Sie die Tiere jeden Tag, um Anzeichen von Schmerzen oder Infektionen zu erkennen, und verabreichen Sie in diesem Fall täglich Buprecare. Um die Feuchtigkeit des Schädeldachs zu erhalten, wechseln Sie das Intrasite-Gel 2x pro Woche.
HINWEIS: Wählen Sie einen hohen Käfig mit Pappunterständen und Nistmaterial und achten Sie darauf, dass die Mäuse in einzelnen Käfigen untergebracht werden.
5. Längsschnittliche Akquisitionen
HINWEIS: Die Maus kann in den folgenden Tagen erneut abgebildet werden. Achten Sie jedoch darauf, nicht mehr als drei Bildgebungssitzungen pro Woche zu wiederholen, um unerwünschte Auswirkungen einer wiederholten Anästhesie wie Augentrockenheit oder übermäßige Müdigkeit sowie Atemnot und Unterkühlung zu vermeiden.
- Um eine Maus mit bereits installiertem Kopfhalter neu abzubilden, befolgen Sie Schritt 2.2 und geben Sie einen kleinen Tropfen Augengel auf die Augen der Maus, um Trockenheit während des Eingriffs zu vermeiden.
- Folgen Sie Abschnitt 3 bis Schritt 3.16, um das Übersichtsbild zu erhalten.
HINWEIS: Die Verwendung des Kopf-Titan-Implantats, ohne die Höhe oder den Winkel der Halterposition ändern zu müssen, macht es sehr einfach, den gesamten Interessenbereich zu finden. - Richten Sie bei Bedarf das vorherige und das neue Bild mit dem Modul "Bild öffnen und ausrichten" neu aus.
- Öffnen Sie das Screenshot-Bild aus Schritt 3.17, um Positionen an ihrer ursprünglichen Stelle zu markieren.
HINWEIS: Die Verwendung von SHG-Kontrast ist sehr nützlich, insbesondere wenn ein Umbau der Blutgefäße und der zugehörigen Zellen, die GFP exprimieren, erwartet wird. - Erfassen Sie Z-Stacks und Zeitraffer auf die gleiche Weise, wie in den Schritten 3.18 bis 3.22 beschrieben.
6. Quantifizierung der vaskulären Parameter
- Vaskuläre Parameter
- Um die mit der Software erzeugten Bilder (.lif-Dateien) in IMARIS zu öffnen, konvertieren Sie sie zuerst mit dem Dateikonverter in das Format ".ims". Öffnen Sie die Datei ".ims" mit IMARIS, überprüfen Sie die Metadaten (X-, Y-, Z-Skalen) und wählen Sie das Filament-Modul aus. Wählen Sie Automatische Erstellung überspringen, manuell bearbeiten.
- Für eine bessere Visualisierung während der Filamentgenerierung navigieren Sie zu Einstellungen (Blattsymbol), wählen Sie Linie im Stil und belassen Sie die Anzahl der Pixel auf dem Standardwert (1). Aktivieren Sie die Kontrollkästchen Dendriten anzeigen, einschließlich Anfangspunkt, Verzweigungspunkte und Endpunkte. Deaktivieren Sie "Wirbelsäule anzeigen".
- Wählen Sie unter Zeichnen (Pinselsymbol) die Option Methode AutoPfad mit 5 μm Durchmesser und der dem Gefäß zugeordneten Kanalquelle (in diesem Fall TRITC-Dextran). Zum Schluss kreuzen Sie die automatische Zentrierung und den automatischen Durchmesserr an.
- Beginnen Sie mit dem Zeichnen der Filamente, indem Sie mit Umschalt + rechter Maustaste einen Startpunkt eines Astes auswählen und Umschalt + Linksklick , um einen Endpunkt eines Astes auszuwählen.
HINWEIS: Der Startpunkt ist hellblau, Verzweigungspunkte sind rot oder dunkelblau und Endpunkte sind grün. - Um den richtigen Durchmesser des Filaments während des Zeichnens einzustellen, verwenden Sie die Maus-Scroll-Taste.
- Um zwei Filamente mit unterschiedlichen Endpunkten zu verbinden, erstellen Sie einen neuen Startpunkt zwischen ihnen und verbinden Sie ihn an jedem Endpunkt.
HINWEIS: Daher erscheinen grüne Endpunkte in Verzweigungspunkten. - Sobald der Gefäßbaum erstellt wurde, kehren Sie zu den Einstellungen zurück und stellen Sie sicher, dass die Anzahl der Start-, Verzweigungs- und Endpunkte korrekt ist. Wählen Sie Kegel aus, um die Durchmesser zu visualisieren und ihre Darstellung zu überprüfen.
- Wenn ein Filamentdurchmesser (oder eine Verbindung) falsch erscheint, navigieren Sie zu Bearbeiten (Bleistiftsymbol), wählen Sie das zu entfernende Filament mit einem Rechtsklick aus und klicken Sie auf Löschen. Zeichnen Sie das Filament neu, indem Sie den Durchmesser des Filaments mit der Maus-Scroll-Taste anpassen.
- Um alle generierten Statistikdaten zu extrahieren und zu speichern, klicken Sie auf das Statistik-Symbol | alle Statistiken in eine Datei exportieren-(Symbol für mehrere Disketten am unteren Bildschirmrand).
HINWEIS: Um potentiell abweichende Filamente zu vermeiden, die nicht den Gefäßen entsprechen, wurden nur Filamente mit einer Länge von mehr als 20 μm in die statistische Analyse einbezogen.
- Quantifizierung der Leckage
HINWEIS: Um die mit der Software erzeugten Bilder (.lif-Datei) in IMARIS zu öffnen, konvertieren Sie sie zuerst mit dem Dateikonverter in das Format ".ims".- Um Bilder, die vor und nach der Dextraninjektion in derselben Zeitserie aufgenommen wurden, zusammenzuführen, gehen Sie in den Surpass-Modus, klicken Sie sowohl auf das erste Bild (TStrg , vor der Dextraninjektion) als auch auf das Zeitrafferbild (Zeit 1, 60 min), um sie zu öffnen, und kombinieren Sie sie, indem Sie auf Bearbeiten klicken und Zeitpunkte hinzufügen.
HINWEIS: Um eine eventuelle 3D-Drift zu korrigieren, führen Sie die Schritte 6.2.2-6.2.7 mit der Brückenfunktion zwischen Fiji und IMARIS aus. Installieren Sie die folgenden Plugins in Fidschi: "IMARIS_Bridge96.jar" und "IMARISBridgeUtils.jar". Die 3D-Driftregistrierung erfolgt mit Hilfe von "StackReg Pluging" (in Fidschi bereits verfügbar). Wenn dies nicht erforderlich ist, fahren Sie mit Schritt 6.2.8 fort - Um eine 3D-Drift-Korrektur durchzuführen, öffnen Sie Fidschi, um Bilder von IMARIS zu überbrücken, indem Sie auf Plugins | IMARIS | Bild von IMARIS.
- Wählen Sie in Fidschi Plugins | Anmeldung | Korrigieren Sie die 3D-Drift.
- Wählen Sie in den neuen Fenstern, die erscheinen, den entsprechenden Kanal für die Registrierung aus.
HINWEIS: In diesem Fall handelte es sich um Nestin-GFP, aber es könnte sich um einen beliebigen Kanal handeln, der unbewegliche Objekte und ein hohes Signal-Hintergrund-Verhältnis hervorhebt. - Um die Erkennung kleiner Drifts zu verbessern, wählen Sie Multi-Time-Scale-Berechnung, Subpixel-Drift-Korrektur und Kantenverbesserung für Bilder aus und behalten Sie die anderen Einstellungen als Standard bei.
- Um das korrigierte Bild mit IMARIS zu überbrücken, wählen Sie es aus und klicken Sie auf Plugins | IMARIS | Bild zu IMARIS.
- Speichern Sie es im entsprechenden Ordner, indem Sie auf Datei | Speichern unter.
- Um alle erfassten Kanäle zu visualisieren, klicken Sie auf Bearbeiten | Anzeigeanpassung anzeigen. Wählen Sie den Dextran-Kanal und die Zeit 1 (Bild 2 am unteren Bildschirmrand).
- Erstellen Sie eine neue Oberfläche, die das Gefäßlumen zum Zeitpunkt 1 definiert, indem Sie das Kontrollkästchen Neue Oberfläche hinzufügen aktivieren (blaues Objekt)
- Wechseln Sie in Surface 1 zu Eigenschaften , und deaktivieren Sie alle Parameter in den Algorithmuseinstellungen. Klicken Sie auf Nächster Schritt (blauer Pfeil)
- Wählen Sie den Gefäßkanal (in diesem Fall Dextran) unter Quellkanal aus und aktivieren Sie Glatt und Hintergrundsubtraktion. Belassen Sie alle anderen Parameter auf den Standardwerten. Klicken Sie auf Nächster Schritt (blauer Pfeil).
- Passen Sie den Schwellenwert entsprechend der Oberflächendarstellung in Bezug auf Dicke und Abdeckung aller positiven Bereiche an. Klicken Sie auf Nächster Schritt (blauer Pfeil).
- Unspezifische Flächen (meist kleine Punkte) mit dem Filter standardmäßig ausschließen Anzahl der Voxel. Klicken Sie auf den letzten Schritt (grüner Pfeil).
- Fahren Sie bei Bedarf mit der manuellen Anpassung der Oberfläche fort, indem Sie in den Einstellungsparametern auf Bearbeiten (Stiftsymbol) klicken. Um ein Objekt auszuwählen, das entfernt werden soll, klicken Sie mit der linken Maustaste darauf und löschen Sie es, indem Sie Löschen auswählen. Wenn das zu löschende Objekt mit einem anderen Objekt sortiert ist, trennen Sie die beiden getrennten Teile der Fläche, indem Sie Umschalt + Linksklick zwischen den beiden drücken und Schnittfläche auswählen. Wiederholen Sie diesen Schritt so oft wie nötig.
- Kombinieren Sie abschließend alle Segmente im Auswahlmodus (Zielsymbol rechts) und drücken Sie Strg + Scrollen , um alle Objekte wiederherzustellen. Wenn alle ausgewählt sind, klicken Sie mit der linken Maustaste und klicken Sie auf Vereinheitlichen.
- Um die Referenzoberfläche zu generieren, kopieren Sie diese Oberfläche in die anderen Zeitpunkte. Wählen Sie die globale Oberfläche aus, und duplizieren Sie sie, indem Sie auf Duplizieren klicken, und benennen Sie sie in Intravaskulär um.
- Wählen Sie die Oberfläche Intravaskulär aus, und klicken Sie auf Auf alle Zeitpunkte duplizieren.
- Teilen Sie das Dextransignal auf, um intra (IN) und extravaskuläre (OUT) Bereiche zu unterscheiden. Um einen IN-Kanal für alle Zeitpunkte zu erstellen, klicken Sie auf die Oberfläche Intravaskulär | Bearbeiten (Bleistift-Schaltfläche) | Alles maskieren.
- Wählen Sie in der Kanalauswahl den entsprechenden Kanal aus und aktivieren Sie Kanal duplizieren, bevor Sie die Maske anwenden. Aktivieren Sie in den Maskeneinstellungen nur Konstante innen/außen, wobei die Voxel außerhalb der Oberfläche auf 0 gesetzt werden. Setzen Sie abschließend ein Häkchen bei Auf alle Zeitpunkte anwenden.
- In der Anzeigeanpassung wird standardmäßig ein neuer Kanal erstellt, bei dem der Name maskiert ist. Benennen Sie es in IN um.
- Um einen Kanalausgang für alle Zeitpunkte zu erstellen, wiederholen Sie die Schritte 6.2.18 bis 6.2.20, wählen Sie jedoch Voxel innerhalb der Oberfläche auf 0 setzen. Benennen Sie diesen neuen Kanal in OUT um.
- Um statistische Daten zu extrahieren, klicken Sie auf das Symbol Statistik | Alle Statistiken in Datei exportieren (Symbol für mehrere Disketten am unteren Bildschirmrand)
- Um die Leckage zu analysieren, dividieren Sie für jeden Zeitpunkt den Intensitätssummenkanal nach außen durch den Intensitätssummenkanal nach innen .
- Um Bilder, die vor und nach der Dextraninjektion in derselben Zeitserie aufgenommen wurden, zusammenzuführen, gehen Sie in den Surpass-Modus, klicken Sie sowohl auf das erste Bild (TStrg , vor der Dextraninjektion) als auch auf das Zeitrafferbild (Zeit 1, 60 min), um sie zu öffnen, und kombinieren Sie sie, indem Sie auf Bearbeiten klicken und Zeitpunkte hinzufügen.
Ergebnisse
In Abbildung 1 und Abbildung 2 ist das CAD-Modell eines Titan-Kopffixationsimplantats dargestellt, das auf einem gescannten Mausschädel positioniert ist und der anatomischen Struktur des Schädels folgt und ein leichtes und biokompatibles Gerät bietet, das fest am Mikroskoptisch befestigt werden kann und die Stabilität auf zellulärer Ebene gewährleistet. Durch die Befolgung dieses Schritt-für-Schritt-Protokolls wird das Implantat stabil mit dem Schädel der Maus verbunden und kann durch seinen Schwalbenschwanz fest am Mikroskophalter befestigt werden, was einen flachen Bildgebungsbereich für die Flüssigkeitsretention und intravitale Beobachtung über die Zeit ermöglicht. Es kann mit einer Abdeckung verschlossen werden, um Schäden oder Infektionen der Wunde zu minimieren, und ermöglicht so eine wiederholte Bildgebung desselben Gewebebereichs über Wochen. Sobald sie wach ist, kann die Maus, die ein Kopfimplantat trägt, frei herumlaufen, füttern und eine regelmäßige Routine haben.
Abbildung 3 zeigt eine Kachelansicht des Schädeldach-BM-Gefäßsystems, das aus heterogenen Kapillaren besteht, einschließlich Arteriolen, Übergangskapillaren und Sinusoiden. Die Gefäße sind in eine komplexe Gewebemikroumgebung eingebettet und stehen in engem Kontakt mit der Knochenoberfläche und den perivaskulären mesenchymalen Zellen. Während der Entwicklung einer Leukämie können einzelne isolierte leukämische Zellen in der BM-Mikroumgebung in unmittelbarer Nähe der Gefäße nachgewiesen werden, und ihre Transplantation nimmt mit der Zeit zu, wodurch sich die Schädeldecke in späten Stadien der Erkrankung füllt.
Abbildung 4 zeigt, wie Bilder, die mit diesem Protokoll aufgenommen wurden, quantitative Daten liefern können, die mit statistischen Methoden analysiert werden können. Wir zeigen, wie man Gefäße mit dem IMARIS Filamentwerkzeug segmentiert und die Länge und den Durchmesser von Gefäßfragmenten sowie deren Geradheit misst. Auch die Korrelation dieser Parameter kann ausgewertet werden.
Abbildung 5 zeigt die longitudinale bildgebende Erfassung von zwei verschiedenen Positionen des Schädeldach-BM während des AML-Fortschreitens an den Tagen 4, 7 und 10, wobei Tag 10 mit einer ~50%igen Transplantation des BM mit leukämischen Zellen assoziiert war, gemessen mittels Durchflusszytometrie (nicht gezeigt). Wir können eine wichtige Umgestaltung der Größe der bereits bestehenden Gefäße sowie die Bildung neuer Gefäße in bestimmten Bereichen beobachten, die mit lokalem Knochenverlust verbunden sind.
In Abbildung 6 zeigen wir schließlich, wie die Gefäßpermeabilität als dynamischer Parameter gemessen werden kann, wobei die Zeitrafferbildgebung die Fähigkeit verschiedener Gefäßbarrieren zeigt, einen Fluoreszenzfarbstoff über die Zeit zu erhalten.

Abbildung 1: Design und Produktion eines biokompatiblen Kopfhalters auf Titanbasis. (A) Teile des Implantats in situ: 1 Beobachtungsring, 2 Zementierungsmerkmale, 3 Stabilisierungsanker, 4 Schwanz, 5 Schwalbenschwanz, 6 Gewindebohrungen, 7 Bregma. (B) Verbindung des Kopfimplantats mit dem Halter: 8 Fixierkörper, 9 Klemme, 10 Exzenterhebel, 11 Struktur, 12 Mikroskopobjektiv. (C) Verformung des Implantats gegen Last durch FEM-Simulation, bei der die maximale Verschiebung 0,23 μm bei 0,04 N Kraft beträgt. (D) Schutz der Abdeckung und ihrer Schraube. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Vorbereitung der Maus für die intravitale Bildgebung. (A) Blick auf das Kopfimplantat und den vor der Bildgebung chirurgisch freigelegten Bildgebungsbereich. (B) Kopfimplantat, das fest mit dem Schädel der Maus verbunden ist. (C) Die Maus ist im Aufwachkäfig mit geschlossener Abdeckung auf dem Kopfimplantat wach. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
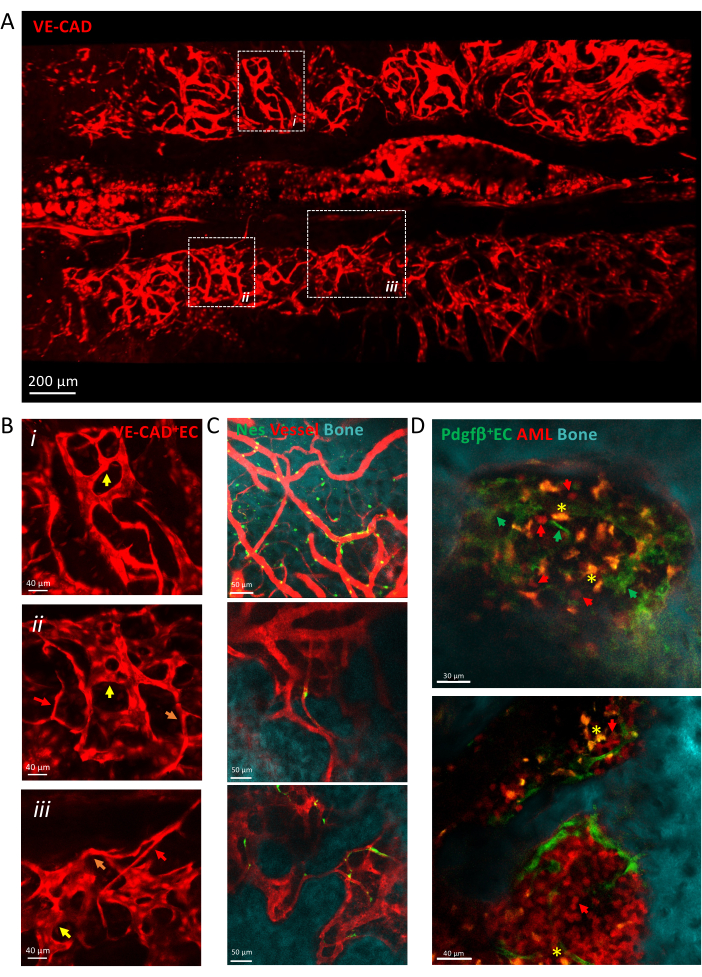
Abbildung 3: Intravitale Bildgebung des Gefäßsystems der Calvaria. (A) Z-Projektion der Kachelansicht des mit cdh5-DSRED markierten Gefäßsystems der Calvaria. (B) Vergrößern Sie die dargestellten Bereiche, um verschiedene Arten von Gefäßen zu beschreiben, die durch Pfeile, Arteriolen durch rote Pfeile, Übergangskapillaren durch orangefarbene Pfeile und Sinusoide durch gelbe Pfeile dargestellt werden. i) und ii) z-Projektionen von X μm Gewebe; iii) einzelne Scheibe. (C) Einzelne Ausschnitte aus mehreren Gesichtsfeldern von BM-Gefäßen, die die Knochenoberfläche (SHG), die perivaskulären Zellen (nes-GFP+) und das Gefäßlumen (dextran-TRITC) zeigen. (D) Vaskuläre Nische, die mit dem Fortschreiten der AML assoziiert ist. Repräsentative Ausschnitte der frühen (oben) und späten (unten) Zeitpunkte der AML-Entwicklung. MLL-AF9-Leukämie ist mit tdTOMATO (rote Pfeile) markiert, während Gefäße mit pdgfb-GFP (grüne Pfeile), Knochenoberfläche mit SHG und Makrophagen mit gelb (Autofluoreszenz, gelbes Sternchen) markiert sind. Maßstabsleisten = 200 μm (A), 40 μm (B,D-untere Platte), 50 μm (C). Abkürzungen: BM = Knochenmark; GFP = grün fluoreszierendes Protein; AML = akute myeloische Leukämie; EC= Endothelzellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Quantifizierung der vaskulären Parameter. (A) Messung der vaskulären Parameter mittels IMARIS-Filamentwerkzeug in einer repräsentativen z-Projektion von Knochenmarkgefäßen, die mit TRITC-Dextran markiert sind. Linien- und Kegeldarstellungen werden angezeigt. (B) Quantifizierung der Schiffsparameter in dem in A gezeigten Bild. (C) Korrelation zwischen vaskulären Parametern, die eine gegensätzliche Korrelation zwischen Geradheit und Länge des Gefäßes zeigt (negativ, Spearman r = -3523; p < 0,0001; R2 = 0,2102) vs. Durchmesser (positiv; Spearman r = 0,4110; p < 0,0001; R2 = 0,1299). Maßstabsbalken = 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
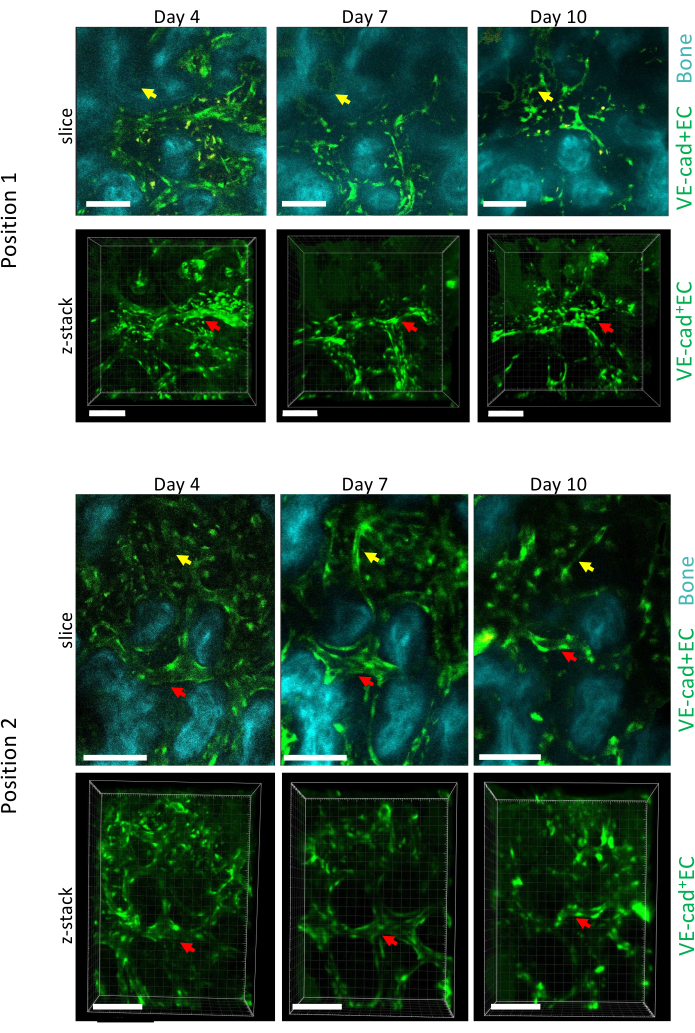
Abbildung 5: Längsschnittdarstellung von zwei verschiedenen Positionen des BM der Calvaria über der AML-Entwicklung. Endothelzellen, die die Gefäße auskleiden, werden mit cdh5-GFP markiert, die Knochenoberfläche mit SHG und Makrophagen gelb (Autofluoreszenz). Dargestellt sind der Umbau bereits bestehender Gefäße (rote Pfeile) und die Bildung neuer Gefäße (gelbe Pfeile). Maßstabsbalken = 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
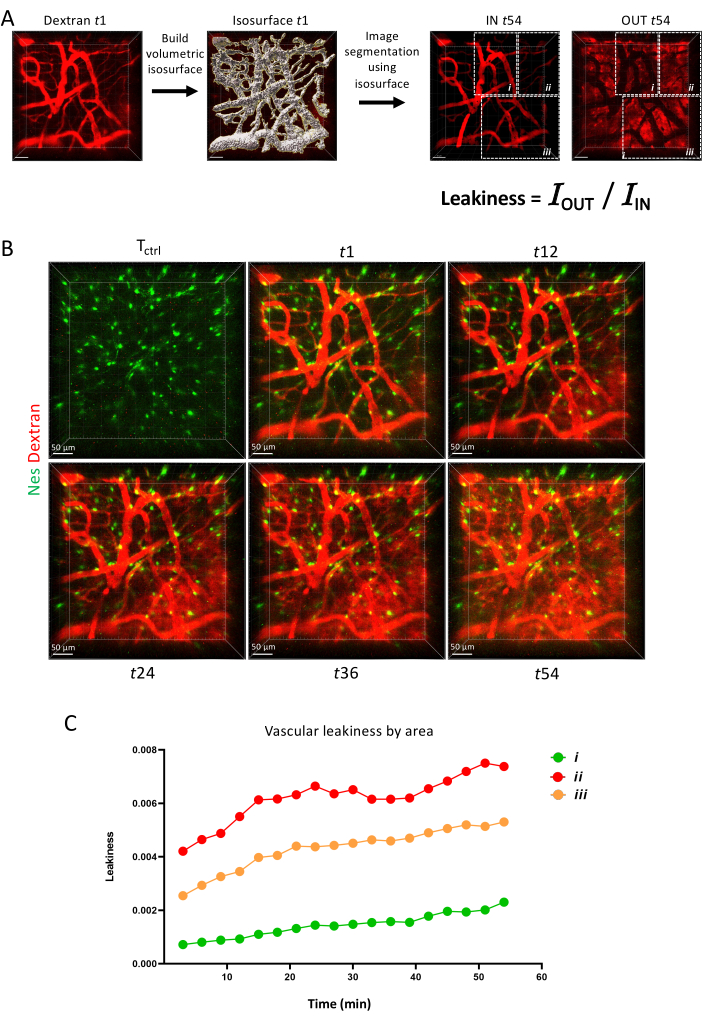
Abbildung 6: Gefäßpermeabilität. (A) Schematische Darstellung der Messung der Gefäßpermeabilität mit dem IMARIS-Oberflächenwerkzeug. (B) Z-Projektion des gleichen Bereichs, der über 1 h in Längsrichtung abgebildet wurde. (C) Quantifizierung der Gefäßpermeabilität innerhalb der in A dargestellten Bereiche. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung S1: Präparierter Schädel. Erstellen Sie in der Medianebene eine Achse, die tangential zum Schädeldach verläuft, und speichern Sie dann den vorbereiteten Schädel. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S2: Pläne im Abstand von 2 mm. Wie man einen Satz von gleichmäßig verteilten Ebenen (2 mm Abstand) über den Schädel erstellt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S3: Beobachtbare Oberflächenkontur. Erstellen Sie eine Skizze in der Schädelbahn, und erstellen Sie einen birnenförmigen Spline von AP +6,5 bis -2, 6 mm Breite bei AP 0,0. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S4: Beobachtung vindow. Erstellen Sie eine Skizze in der Schädelebene, und zeichnen Sie eine 0,5 mm dicke C-Form, die mit dem Beobachtungsfenster verbunden ist. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S5: Klicken Sie auf Neue Studie. Navigieren Sie zur Registerkarte Simulation (falls sichtbar) oder gehen Sie zu Simulation | Studie. Wählen Sie im Dialogfeld Studie die Option Statisch als Studientyp aus. Klicken Sie auf OK , um die neue Studie zu erstellen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S6: Rechtsklick, um ein Netz zu erstellen. Verfeinern Sie das Netz in Bereichen, in denen hohe Spannungen oder Verformungen zu erwarten sind. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die Hauptstärke der Intravitalmikroskopie liegt in ihrer Fähigkeit, dynamische zelluläre Prozesse in Echtzeit in ihrer natürlichen Umgebung zu erfassen und dabei die Grenzen traditioneller histologischer und ex vivo-Bildgebungsverfahren zu überwinden. Durch die direkte Beobachtung des Schädeldach-BM unter Verwendung der fortschrittlichen bildgebenden Verfahren in Verbindung mit der Fluoreszenzmarkierung, wie in diesem Protokoll beschrieben, können die Forscher nicht nur vaskuläre Funktionsparameter 8,14 untersuchen, sondern auch longitudinale Prozesse wie leukämische Zelltransplantation und -migration 6,15, Proliferation und metabolische Aktivität16,17 Erhaltung der physiologischen Relevanz und Vermeidung von Artefakten, die mit der Gewebefixierung und -verarbeitung verbunden sind.
Zu den Vorteilen der intravitalen Bildgebung der BM-Kalvaria gegenüber alternativen Knochenstellen gehören die einfache Zugänglichkeit, die Transparenz des Schädelknochens, die eine bessere Visualisierung ohne Knochenschäden ermöglicht, die Stabilität des Schädeldachknochens, die Bewegungsartefakte des Gewebes minimiert, und die Reproduzierbarkeit bei Versuchstieren, die den Vergleich zwischen verschiedenen Versuchsgruppen erleichtert. Die Möglichkeit, die Tiere zu bergen und eine longitudinale Bildgebung durchzuführen, ermöglicht es auch, die Anzahl der in einer Studie verwendeten Tiere zu reduzieren. Es ist erwähnenswert, dass es wichtig ist, dies bei der Gestaltung des spezifischen bildgebenden Implantats für den gewünschten Mausstamm zu berücksichtigen, da stammspezifische Unterschiede in den Wachstumsmustern der Schädelgefäße und der daraus resultierenden Osteogenese beobachtet wurden18, um eine perfekte anatomische Kompatibilität und einen perfekten Zugang zur gewünschten Bildgebungsstelle zu ermöglichen.
Ein weiterer wichtiger Punkt sind die möglichen Unterschiede in den vaskulären und hämatopoetischen Eigenschaften des Schädeldach-BM im Vergleich zu anderen Knochen, eine Frage, die bis vor wenigen Jahren wenig erforscht wurde. Neuartige Studien deuten auf lokalisierte Funktionen für verschiedene Knochen hin, wobei sich der BM des Schädeldachs von anderen Knochen in Bezug auf Hämatopoese, Knochen- und Gefäßstruktur und -funktionunterscheidet 19,20,21,22,23 sowie das Ansprechen auf neurologische Pathologien24. Diese Unterschiede müssen weiter erforscht und berücksichtigt werden, bevor spezifische Calvaria-Befunde verallgemeinert werden können.
Das Design und die Konstruktion eines bildgebenden Implantats ist ein wichtiger Schritt für diesen Versuchsaufbau, beginnend mit der Wahl des Materials. Biokompatible Implantate spielen eine zentrale Rolle in der biomedizinischen Forschung und ermöglichen eine breite Palette von Anwendungen, die von Tissue Engineering und regenerativer Medizin bis hin zu Arzneimittelverabreichungssystemen und In-vivo-Überwachungsgeräten reichen. Die Wahl des Materials für ein Kopfimplantat, das für die intravitale Bildgebung geeignet ist, ist von entscheidender Bedeutung. Idealerweise sollte das Material eine ausgezeichnete Biokompatibilität aufweisen, mechanische Eigenschaften, die geeignet sind, ein Minimum an Flexibilität und Gesamtstabilität zu gewährleisten, und schließlich die Fähigkeit, sich in den Schädel zu integrieren, ohne Entzündungen auszulösen. Titan ist ideal für seine gute Verträglichkeit im tierischen Körper, ohne Induktion von Nebenwirkungen bei Kontakt mit biologischem Gewebe, sowie für seine Korrosionsbeständigkeit, die eine Langzeitstabilität gewährleistet, auch wenn es Körperflüssigkeiten oder experimentellen Lösungen ausgesetzt ist. Darüber hinaus ist es aufgrund seiner mechanischen Festigkeit widerstandsfähig gegen Verformungen und Brüche. Trotz seiner Festigkeit hat Titan eine relativ geringe Dichte, was zu leichten Intravital-Bildgebungsgeräten führt, die die Belastung für Versuchstiere und Forscher minimieren. Schließlich ermöglicht seine Vielseitigkeit in der Herstellung die Anpassung von Intravital-Bildgebungsgeräten an spezifische experimentelle Anforderungen wie Größe, Form und Funktionalität.
Hier haben wir ein biokompatibles Kopffixationsimplantat unter Verwendung einer CAD-Software für die parametrische Modellierung mit integrierten Funktionen für die Finite-Elemente-Analyse (FEA), insbesondere SolidWorks, entwickelt. Dieser Ansatz ermöglicht eine präzise, iterative Anpassung an die strukturellen und räumlichen Anforderungen des Implantats und stellt sowohl die anatomische Kompatibilität als auch die mechanische Belastbarkeit sicher. Kostenlose Alternativen, wie FreeCAD, bieten ähnliche Modellierungs- und Simulationsfunktionen. Zu den wichtigsten Eingaben für den Designprozess gehören ein hochauflösendes anatomisches Modell des Schädels der Maus, ein Modell des Mikroskopobjektivs und ein Modell des Fixationssystems, die zusammen die Abmessungen und die Konfiguration des Implantats bestimmen. Die endgültige Ausgabe ist eine plattformunabhängige Mesh-Datei, in der Regel im STL- oder STEP-Format, die mit den meisten 3D-Druckprogrammen kompatibel ist.
Der erste Schritt besteht darin, detaillierte anatomische Merkmale des Mauskopfes mit hochauflösenden bildgebenden Verfahren wie In-vivo-Mikro-CT-Scans zu erfassen. Dieser Ansatz bietet höchste anatomische Detailgenauigkeit und erfasst die Mikrostrukturen des Schädels. Das 3D-Scannen eines präparierten, nackten Schädels könnte ebenfalls eine Option sein. Diese Methode, bei der Laser- oder Streifenlichtscanner verwendet werden, wird häufig an einem präparierten Schädel angewendet und liefert genaue Oberflächenkonturen, wenn auch mit weniger internen Details im Vergleich zur Mikro-CT. Andernfalls können Anatomiemodelle aus Open-Source-Publikationen und Datenbanken25 oder DigiMorph {https://www.digimorph.org/specimens/Mus_musculus/} heruntergeladen werden. Diese Modelle sind zwar praktisch, aber es fehlen möglicherweise probenspezifische Details, so dass oft Anpassungen für das jeweilige Tier in der Studie erforderlich sind, wie z. B. die Skalierung auf den tatsächlichen Bregma-Lambda-Abstand. Die erfassten Daten werden dann verwendet, um ein präzises 3D-Modell des Schädels der Maus zu erstellen, das als Grundlage für das Implantatdesign dient.
Um das Implantat auf dem Schädel zu befestigen, wird eine Zementierungsstruktur entwickelt, die die verbleibende Schädeloberfläche abdeckt, die nicht vom Beobachtungsfenster eingenommen wird. Diese Struktur muss robuste Befestigungspunkte bieten und gleichzeitig kritische anatomische Merkmale vermeiden. Die Zementierungsstruktur hat mehrere Öffnungen, um eine optimale Polymerisation des Zements unter dem Implantat durch Diffusion zu gewährleisten. Zusätzlich haben die Wände der Zementierkonstruktion einen kleinen Entformungswinkel, der es dem Zement ermöglicht, sich sicher an diesen abgewinkelten Wänden zu verankern. Der Schwanz des Implantats, der sich vom Hauptkörper bis zum Fixationssystem erstreckt, ist gestaltet. Diese Komponente ist entscheidend für die Ausrichtung und Stabilisierung des Implantats während der Beobachtung, und ihr Design muss den verfügbaren Platz und die anatomischen Einschränkungen des Mauskopfes berücksichtigen. Schließlich ist ein Schwalbenschwanzmechanismus in das Implantatdesign integriert, der ein einfaches Anbringen und Lösen des Implantats vom Fixationssystem ermöglicht. Diese Eigenschaft verbessert die Praktikabilität und Benutzerfreundlichkeit des Implantats bei wiederholten Beobachtungen. Der Schwalbenschwanzmechanismus ermöglicht eine wiederholbare Fixierung für eine einfache Entnahme des beobachteten Gewebes für wiederholte Bildgebungssitzungen.
Leser, die die Einführung einer intravitalen mikroskopischen Bildgebung des Schädeldach-BM in Betracht ziehen, sollten ihre Forschungsziele und experimentellen Anforderungen sorgfältig abwägen, um festzustellen, ob diese Methode für ihre Studien geeignet ist. Die Intravitalmikroskopie bietet zwar beispiellose Einblicke in die Hämatopoese, die Regulation der Mikroumgebung des Gewebes und die Gefäßdynamik in vivo, birgt aber auch gewisse technische Herausforderungen und Einschränkungen. Forscher sollten darauf vorbereitet sein, diese Herausforderungen durch sorgfältiges experimentelles Design, Optimierung der Bildgebungsparameter und Verwendung geeigneter Kontrollen zu bewältigen. Darüber hinaus sollten Forscher die Verfügbarkeit von spezialisierten Bildgebungsgeräten, Fachwissen über Fluoreszenzmarkierungstechniken und Rechenressourcen für die Bildanalyse berücksichtigen. Insgesamt birgt die intravitale mikroskopische Bildgebung des Schädeldach-BM ein enormes Potenzial für unser Verständnis der Hämatopoese und der Gefäßbiologie und bietet einen einzigartigen Einblick in die dynamischen Prozesse, die in der BM-Mikroumgebung ablaufen.
Offenlegungen
Jozsua Fodor ist der Gründer der Firma YMETRY (Register-Nr. 888312352). Die anderen Autoren haben keine Interessenkonflikte.
Danksagungen
Die Autoren danken allen Mitarbeitern des IMAG'IC und der Tierschutzeinrichtungen am Institut Cochin für ihre Unterstützung bei den Mikroskopie-Experimenten und der Maushaltung. Tg(Nes-EGFP)33Enik und Tg(Pdgfb-icre/ERT2)1Frut Mäuse waren ein freundliches Geschenk von Dr. Bonnet (The Francis Crick Institute, London). Tg(Cdh5-cre/ERT2)1Rha und B6. Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze/J Mäuse waren ein freundliches Geschenk von Dr. Rafii (Weill Cornell Medicine, New York). Die beschriebene Arbeit wurde unterstützt vom CNRS, INSERM und der Université de Paris Cité sowie von ATIP-AVENIR, Fondation ARC pour la recherche sur le cancer (R19084KS - RSE20008KSA), Ville de Paris "Emergence" (R20192KK - RPH20192KKA), Laurette Fugain (R23197KK), Cancéropôle IDF (RPH23177KKA), INCA PLBIO (RPH21162KKA), Fondation de France (RAF23152KKA), Ligue contre le cancer (282273/807251), Institut du cancer Paris Carpem, European Hematology Association (RAK23130KKA) und Europäischer Forschungsrat ERC-STG (EEA24092KKA). Die IMAG'IC Core Facility wird von der National Infrastructure France BioImaging unterstützt (Grant ANR-10-INBS-04). Das Labor Passaro ist dem "Institut Hors Murs des Sciences Cardiovasculaires" und dem "Leukämie-Institut Paris Saint-Louis" angegliedert.
Materialien
| Name | Company | Catalog Number | Comments |
| Amira | Thermo ScientificTM | ||
| Anesthesia | Isoflurane 2% to 3% | ||
| Anesthesic mask (animal detection) | Minerve | ||
| Anesthesic unit | Minerve | ||
| B6.Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze | Jackson laboratories | MGI:3809523 | |
| CalvariaVue | Ymetry | https://ymetry.com/site/head-fixation-implants/44-mouse-head-fixation-implant-for-calvaria.html | |
| Dental cement | Kemdent | SUN527 | |
| Dextran 500 kDa-Cy5 | Tebu-bio | DX500-S5-1 | 3 mg/mouse |
| Dextran 70 kDa-TRITC | Sigma | T1162 | 3 mg/mouse |
| Disinfectant | MP-Labo | Dermidine-60ml | |
| Electric razor | Aescular | Isis | |
| Eye gel | Ocry-gel | 10g | |
| Fiji | https://imagej.net/software/fiji/downloads | v 17 May 30 | |
| Fiji plugings to bridge with Imaris | https://imagej.net/software/fiji/downloads | Imaris_Bridge96.jar ; ImarisBridgeUtils.jar | |
| Heating box | Datesand | Thermacage | |
| Heating pad for surgery | Minerve | ||
| Imaging heating pad & rectal probe | F. Haer | ||
| Imaris v9.6.0 | Oxford instruments | ||
| Intrasite gel | Chinoxia | 2390766 | |
| LAS AF Software | Leica | LAS X 3.5.7.23225 | |
| Medication | Buprecare, 0.01 mg/kg | ||
| Objective HCX IRAPO L 25x/0.95 WATER | Leica | 506374 | |
| Saline buffer (PBS 1x) | Sigma | P4417 | Sterilize by autoclave |
| SP8 DIVE FALCON Multiphoton Microscope | Leica | ||
| Stereotoxic mask | Minerve | 1201261 | |
| Sterilizator beads | Sigma | Z742555 | |
| Surgery tools | Moria | 4877A; 2183 | |
| Survival blanket | SECURIMED | 11006 | |
| Swabs / Tissues | Sterilize by autoclave | ||
| Syringe 1 mL 26 G | BD Plastipak | 305501 | |
| Temperature controller | F. Haer | 40-90-5D-02 | |
| Tg(Nes-EGFP)33Enik mice | Jackson laboratories | MGI:5523870 | |
| Tg(Pdgfb-icre/ERT2)1Frut mice | Jackson laboratories | MGI:3793852 | |
| Tg(Cdh5-cre/ERT2)1Rha mice | Jackson laboratories | MGI:3848982 | |
| Ultrasound gel | Parker laboratories | Aquasonic 100 |
Referenzen
- Lo Celso, C., et al. Live-animal tracking of individual haematopoietic stem/progenitor cells in their niche. Nature. 457 (7225), 92-96 (2009).
- Pittet, M. J., Weissleder, R. Intravital imaging. Cell. 147 (5), 983-991 (2011).
- Itkin, T., et al. Distinct bone marrow blood vessels differentially regulate haematopoiesis. Nature. 532 (7599), 323-328 (2016).
- Christodoulou, C., et al. Live-animal imaging of native haematopoietic stem and progenitor cells. Nature. 578 (7794), 278-283 (2020).
- Upadhaya, S., et al. Intravital imaging reveals motility of adult hematopoietic stem cells in the bone marrow niche. Cell Stem Cell. 27 (2), 336-345.e4 (2020).
- Hawkins, E. D., et al. T-cell acute leukaemia exhibits dynamic interactions with bone marrow microenvironments. Nature. 538 (7626), 518-522 (2016).
- Duarte, D., et al. Defining the in vivo characteristics of acute myeloid leukemia cells behavior by intravital imaging. Immunol Cell Biol. 97 (2), 229-235 (2019).
- Passaro, D., et al. Increased vascular permeability in the bone marrow microenvironment contributes to disease progression and drug response in acute myeloid leukemia. Cancer Cell. 32 (3), 324-341.e6 (2017).
- Jia, Y., et al. FLT3 inhibitors upregulate CXCR4 and E-selectin ligands via ERK suppression in AML cells and CXCR4/E-selectin inhibition enhances anti-leukemia efficacy of FLT3-targeted therapy in AML. Leukemia. 37 (6), 1379-1383 (2023).
- Pendleton, E. G., Tehrani, K. F., Barrow, R. P., Mortensen, L. J. Second harmonic generation characterization of collagen in whole bone. Biomed Opt Express. 11 (8), 4379-4396 (2020).
- Ventalon, C., Bourdieu, L., Leger, J. F., Fodor, J. Dispositif de liaison d'un animal de laboratoire à au moins un système d'expérimentation, et procédé de fixation d'un tel dispositif. France patent. , (2021).
- Horton, S. J., et al. Acute myeloid leukemia induced by MLL-ENL is cured by oncogene ablation despite acquisition of complex genetic abnormalities. Blood. 113 (20), 4922-4929 (2009).
- Scott, M. K., Akinduro, O., Lo Celso, C. In vivo 4-dimensional tracking of hematopoietic stem and progenitor cells in adult mouse calvarial bone marrow. J Vis Exp. (91), e51683 (2014).
- Jung, Y., et al. Intravital imaging of mouse bone marrow: Hemodynamics and vascular permeability. Methods Mol Biol. 1763, 11-22 (2018).
- Le, V. H., et al. In vivo longitudinal visualization of bone marrow engraftment process in mouse calvaria using two-photon microscopy. Sci Rep. 7, 44097 (2017).
- Spencer, J. A., et al. Direct measurement of local oxygen concentration in the bone marrow of live animals. Nature. 508 (7495), 269-273 (2014).
- Yang, M., Mahanty, A., Jin, C., Wong, A. N. N., Yoo, J. S. Label-free metabolic imaging for sensitive and robust monitoring of anti-CD47 immunotherapy response in triple-negative breast cancer. J Immunother Cancer. 10 (9), e005199 (2022).
- Li, W., et al. Tracking strain-specific morphogenesis and angiogenesis of murine calvaria with large-scale optoacoustic and Ultrasound Microscopy. J Bone Miner Res. 37 (5), 1032-1043 (2022).
- Lassailly, F., Foster, K., Lopez-Onieva, L., Currie, E., Bonnet, D. Multimodal imaging reveals structural and functional heterogeneity in different bone marrow compartments: functional implications on hematopoietic stem cells. Blood. 122 (10), 1730-1740 (2013).
- Rindone, A. N., et al. Quantitative 3D imaging of the cranial microvascular environment at single-cell resolution. Nat Commun. 12 (1), 6219 (2021).
- Mills, W. A., Coburn, M. A., Eyo, U. B. The emergence of the calvarial hematopoietic niche in health and disease. Immunol Rev. 311 (1), 26-38 (2022).
- Bixel, M. G., et al. Angiogenesis is uncoupled from osteogenesis during calvarial bone regeneration. Nat Commun. 15 (1), 4575 (2024).
- Koh, B. I., et al. Adult skull bone marrow is an expanding and resilient haematopoietic reservoir. Nature. 636 (8041), 172-181 (2024).
- Kolabas, Z. I., et al. Distinct molecular profiles of skull bone marrow in health and neurological disorders. Cell. 186 (17), 3706-3725.e9 (2023).
- Rosenhain, S., et al. A preclinical micro-computed tomography database including 3D whole body organ segmentations. Sci Data. 5, 180294 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten