É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Cell-free Dot Blot como uma plataforma de imunoensaio prática e adaptável para a detecção da resposta de anticorpos em soros humanos e animais
Neste Artigo
Resumo
Descrevemos uma plataforma de imunoensaio recentemente desenvolvida com base nos princípios da biologia sintética livre de células e na técnica de dot-blot para detecção personalizável da resposta de anticorpos em soros humanos e animais.
Resumo
A série de surtos patogênicos globais nas últimas duas décadas destacou a importância das estratégias de sorovigilância. As plataformas de imunoensaio que servem para detectar anticorpos específicos da doença no soro dos pacientes estão no centro da sorovigilância. Exemplos comuns incluem ensaios de imunoabsorção enzimática e ensaios de fluxo lateral; No entanto, embora esses sejam métodos padrão-ouro, eles exigem consumíveis específicos para patógenos e equipamentos especializados, o que limita seu uso fora de laboratórios com bons recursos.
Recentemente, desenvolvemos uma nova plataforma de imunoensaio chamada Cell-Free Dot-Blot (CFDB) e a validamos usando soros humanos e animais contra SARS-CoV-2. Ao contrário dos imunoensaios convencionais, as amostras de soro de pacientes com CFDB são imobilizadas em uma fase sólida (membrana de nitrocelulose), enquanto o antígeno alvo é suspenso na fase móvel do ensaio. Para melhorar o acesso aos recursos de sorovigilância, os antígenos CFDB são produzidos sob demanda e com infraestrutura de baixa carga usando a expressão de proteínas in vitro . Aqui, o antígeno é fundido com uma marca peptídica que pode ser detectada usando uma única proteína repórter universal para qualquer ensaio de CFDB. O resultado é que o CFDB não requer acesso a um leitor de placas de vários poços ou componentes de ensaios moleculares comerciais purificados. Com essas considerações de design, o CFDB aborda as deficiências das plataformas de imunoensaio existentes, fornecendo acessibilidade a laboratórios não centralizados, adaptabilidade para patógenos emergentes e acessibilidade para comunidades de baixa renda.
No artigo atual, forneceremos um protocolo passo a passo para preparar e realizar um imunoensaio CFDB. Usando nosso trabalho recente sobre SARS-CoV-2 CFDB como exemplo, abordaremos o projeto de DNA de antígeno para produção livre de células sob demanda, seguido pela preparação da proteína repórter CFDB, imobilização de amostras de soro na fase sólida e, finalmente, etapas de ligação e detecção de antígeno do ensaio. Prevemos que, seguindo essas instruções, os pesquisadores serão capazes de adaptar o ensaio CFDB para detectar respostas imunes em soros humanos e animais a qualquer patógeno.
Introdução
A pandemia de COVID-19 revelou a necessidade crítica de ferramentas de diagnóstico acessíveis e escaláveis, principalmente para ambientes com poucos recursos1. Os imunoensaios convencionais, como os ensaios imunoenzimáticos (ELISAs), têm se mostrado essenciais para a detecção de respostas imunes 2,3. No entanto, seu alto custo, dependência de reagentes complexos e dependência de equipamentos especializados limitam sua acessibilidade, especialmente durante crises globais de saúde. Em resposta a esses desafios, desenvolvemos o Cell-Free Dot Blot (CFDB), uma plataforma de imunoensaio adaptável e de baixo custo projetada para a detecção de anticorpos anti-SARS-CoV-2 em soros humanos e animais.
O CFDB aproveita a biologia sintética livre de células para a produção rápida e sob demanda de antígenos virais usando modelos lineares de DNA 4,5. Isso elimina a necessidade de processos tradicionais de clonagem, expressão e purificação baseados em células, acelerando significativamente a produção de antígenos e reduzindo custos. O método CFDB simplifica a detecção de anticorpos usando um formato dot blot, onde os soros são detectados diretamente nas membranas de nitrocelulose. Este sistema evita a necessidade de placas caras de vários poços e equipamentos de laboratório especializados, permitindo um fluxo de trabalho simples de "imersão" para as etapas de incubação e lavagem. A plataforma também utiliza um sistema SpyCatcher-SpyTag, onde uma quimera de peroxidase SpyCatcher2-Apex2 atua como um reagente de detecção secundária universal 5,6. Isso é produzido usando a expressão padrão baseada em Escherichia coli, que elimina a dependência de conjugados de anticorpos comerciais caros. Como resultado, o sistema CFDB pode realizar ensaios sorológicos com desempenho comparável aos ELISAs a um custo significativamente menor - cerca de US$ 3 por ensaio de 96 amostras em comparação com mais de US$ 300 para um kit ELISA comercial5.
Para demonstrar a eficácia do CFDB, testamos sua capacidade de detectar anticorpos em soros humanos e animais pré-caracterizados. Nossos resultados se correlacionaram estreitamente com o ELISA na identificação de amostras positivas e negativas para COVID-19. Além do diagnóstico humano, avaliamos a utilidade do CFDB em modelos animais, testando soros de hamsters infectados com SARS-CoV-2 e aqueles vacinados com proteína recombinante do nucleocapsídeo. Esses testes confirmaram o potencial do CFDB para uso em diagnósticos humanos e veterinários, tornando-o uma ferramenta versátil para monitorar as respostas imunológicas entre as espécies. Uma das principais vantagens do CFDB é sua flexibilidade. Simplesmente modificando o modelo de DNA que codifica o antígeno de interesse, a plataforma pode ser rapidamente adaptada para detectar anticorpos contra diferentes patógenos, tornando-a valiosa para a preparação para futuras pandemias. Seu baixo custo, fluxo de trabalho simples e requisitos mínimos de infraestrutura o tornam particularmente adequado para laboratórios descentralizados e ambientes de poucos recursos, onde o acesso a diagnósticos comerciais é limitado.
Neste trabalho, forneceremos instruções passo a passo para preparar e conduzir um ensaio CFDB. Primeiro, cobrimos o design e a síntese de modelos lineares de DNA para a produção de antígenos livres de células, que são os principais reagentes de detecção do ensaio. Em seguida, descrevemos as etapas para a preparação do reagente de detecção secundária do ensaio, SpyCatcher2-Apex2. Depois disso, fornecemos instruções para a produção livre de células e verificação de qualidade dos próprios antígenos. Finalmente, descrevemos em detalhes o processo para a realização de um ensaio CFDB em amostras de soro humano ou animal.
Protocolo
Todos os experimentos com hamsters foram realizados no Laboratório Nacional de Microbiologia (NML) da Agência de Saúde Pública do Canadá, aprovados pelo Centro Canadense de Ciências para Saúde Humana e Animal e seguindo as diretrizes do Conselho Canadense de Cuidados com Animais. Todas as amostras de soro/plasma humano foram obtidas comercialmente para testes internos ou fornecidas por colaboradores clínicos ao NML para testes independentes no NML.
1. Conceção e preparação de modelos de expressão linear de antigénio (LETs)
- Projete os LETs de expressão livre de células para o antígeno alvo com base nas instruções de Norouzi et al.7 e contendo um SpyTag N/C-terminal, conforme descrito em Norouzi et al.5.
NOTA: Aqui fornecemos detalhes para a preparação da proteína do nucleocapsídeo SARS-CoV-2 (NP, aminoácidos 2-419).- Certifique-se de que o LET para o SARS-CoV-2 NP (Figura 1 e Arquivo Suplementar 1) contém locais de 5' e 3' Ter com uma sequência de buffer de 50 pares de bases antes do promotor T7 e após o códon de parada.
- Marque a proteína NP no terminal N com His 6-SpyTag-TEV.
- Obtenha a sequência de proteínas do banco de dados UniProt (código de acesso P0DTC9) e otimize-a para expressão baseada em E. coli usando a ferramenta de otimização de códonsIDT 8. Encomende esta sequência para síntese comercial como um fragmento de DNA de fita simples ressuspenso em água a 10 ng / μL.
NOTA: O local de clivagem da protease TEV e a etiqueta His6 não são características essenciais no design do LET.
- A PCR amplifica o fragmento de DNA com o Ter FW universal (GGCTCCGAATAAGTATGTTGTAACTAAAAGTGCGGCC
ACGATGCGTCCGGCGTAGAGGATCG) e Ter RV (CCGAGGCAATAAGTATGTTGTAACTAAAAGTGCTCAG
CTTCCTTTCGGGCTTTGTTAGCAGCC) usando o kit de DNA polimerase de alta fidelidade (consulte a Tabela de Materiais)- Configure uma reação de 100 μL da seguinte forma: 75 μL de água livre de nuclease, 20 μL de tampão 5x DNA polimerase, 2 μL de 10 mM dNTPs (concentração final de 200 μM), 0,5 μL de primer 100 μM Ter-FW, 0,5 μL de primer Ter-RV 100 μM, 1 μL de LET de fita simples 10 ng/μL, 1 μL(2U) de DNA polimerase.
- Use as seguintes configurações de PCR: desnaturação inicial a 98 °C por 30 s; 35 ciclos: 98 °C durante 6 s, 60 °C durante 15 s, 72 °C durante 90 s; manter a 4 °C.
- Purifique o produto de PCR usando um kit de purificação de PCR comercial, verifique sua qualidade executando uma amostra em um gel de agarose a 1% e meça sua concentração em um espectrofotômetro UV-Vis.
NOTA: Os produtos brutos de PCR também podem ser usados diretamente para expressão livre de células. No entanto, a purificação permite um procedimento mais padronizado.

Figura 1: Modelo de expressão linear para SARS-CoV-2-NP. Um esquema representando as características do modelo de DNA linear His-SpyTag-SARS-CoV-2 NP. Os principais elementos do molde de DNA são rotulados. A sequência de codificação da proteína de interesse, aqui a NP, é colocada sob o controle transcricional de um promotor T7 para expressão eficiente. No terminal N, a proteína NP é anexada com um SpyTag para detecção específica usando o reagente de detecção SpyCatcher2-Apex2. Os locais de protease x6His-tag e TEV, embora incluídos como parte do projeto geral do LET, são dispensáveis para fins de CFDB. Nos terminais do molde de DNA linear, os sítios Ter "a montante" e "a jusante", cada um precedido pelas respectivas sequências tampão de 50 pares de bases, são incluídos para proteção mediada por Tus contra a degradação do DNA exonucleolítico no lisado livre de células. Abreviaturas: NP = proteína do nucleocapsídeo; LET = modelo de expressão linear; TEV = vírus da corrosão do tabaco; CFDB = dot blot livre de células. Clique aqui para ver uma versão maior desta figura.
2. Purificação da proteína repórter SpyCatcher2-Apex2
- Use o plasmídeo pET24b-SpyCatcher2-Apex2, originalmente construído em Norouzi et al.5 (sequência completa no Arquivo Suplementar 2 e mapa de plasmídeo na Figura Suplementar S1) para preparar a proteína repórter SpyCatcher2-Apex-2.
- Prepare placas de ágar e caldo de lisogenia (LB) contendo 50 μg / mL de canamicina. Transforme as células BL21 (DE3) de E. coli com o plasmídeo pET24b-SpyCatcher2-Apex2. Inocular uma única colónia numa cultura inicial de 15 ml de LB e crescer durante a noite a 37 °C com agitação a 250 RPM.
- No dia seguinte, adicione 10 mL da cultura inicial em 500 mL de meio LB fresco contendo 50 μg / mL de canamicina. Incubar a 37 °C com agitação a 250 RPM até que a cultura atinja uma densidade óptica a 600 nm de 0,6-0,8 (~3 h).
- Induza a expressão de SpyCatcher2-Apex2 suplementando a cultura com 0,5 mM de isopropil-β-D-1-tiogalactopiranosídeo e 1 mM de cloridrato de ácido 5-aminolevulínico. Reduzir a temperatura de crescimento para 30 °C e deixar incubar a cultura durante mais 4 h.
- Colha as bactérias por centrifugação a 8.000 × g por 15 min. Proceder à lise celular ou, em alternativa, conservar o pellet a -80 °C até à utilização.
- Ressuspenda o pellet em 20 mL de tampão de lise contendo 50 mM de Tris-HCl (pH 7,8), 300 mM de NaCl, 1 mg/mL de lisozima, comprimido inibidor de protease sem EDTA e 1 mM de ditiotreitol (DTT). Lisar as células por sonicação a 50 % de amplitude com intervalos de 5 s ON e 10 s OFF para um tempo total de ON de 3 min.
- Clarificar o lisado por centrifugação a 20.000 × g durante 1 h a 4 °C. Passe o sobrenadante por um filtro de seringa de 0,2 μm.
- Adicionar cloreto de hemina a uma concentração final de 250 μM ao lisado clarificado e incubar durante a noite a 4 °C. Prossiga para a purificação de proteínas.
NOTA: A incubação com cloreto de hemina maximiza a incorporação de heme na peroxidase Apex-2 para uma atividade enzimática ideal. - Adicione 2,5 ml de resina de Ni ao lisado clarificado e incube a 4 °C com agitação suave durante 45 min. Aplique a mistura em uma coluna de fluxo por gravidade e lave a resina com 50 mL de tampão Tris (50 mM Tris-HCl (pH 7,8), 300 mM NaCl e 1 mM DTT).
- Eluir a proteína SpyCatcher2-Apex2 em 25 mL de tampão Tris contendo 400 mM de imidazol. Concentre e troque o eluído em tampão Tris usando uma unidade de filtro centrífugo, visando um volume final de 0,5-1,0 mL.
- Use o coeficiente de extinção molar de SpyCatcher2-Apex2 (27.390 M-1 cm-1) para determinar a concentração de proteína em um espectrofotômetro UV-Vis. Adicione glicerol a uma concentração final de 40% e armazene as alíquotas a -20 °C.
NOTA: Espera-se que este protocolo produza aproximadamente 40 mg de SpyCatcher2-Apex2 altamente puro e ativo, suficiente para 400 execuções de CFDB em um tamanho de borrão padrão de 96 amostras. - Para detecção de proteínas SpyTagged usando western blot9 convencional e para experimentos CFDB, sempre bloqueie as membranas em leite em pó desnatado a 5% em 1x solução salina tamponada com Tris contendo 0,05% de Tween-20 (TBST). Use a proteína SpyCatcher2-Apex2 em uma concentração final de 10 μg/mL em solução de bloqueio.
NOTA: O repórter SpyCatcher2-Apex2 requer uma solução de quimioluminescência aprimorada (ECL) para o desenvolvimento do sinal. A solução de ECL pode ser obtida comercialmente ou preparada internamente de acordo com as instruções de Mruk et al.10. A solução final de ECL aqui consiste em 0,4 mM de ácido p-cumárico, 2,5 mM de luminol e 0,015% de H2O2 em 100 mM de Tris-HCl (pH 8,6).
3. Produção sem células e verificação da qualidade dos antigénios
- Prepare e monte um lisado de expressão livre de células E . coli BL21 e componentes de reação seguindo as instruções em Levine et al.11 e Norouzi et al.5. Suplementar a mistura de reação final com 5 μM de proteína Tus e 1,2 μM de RNA polimerase T7 da seguinte forma para uma reação representativa de 100 μL: 14,6 μL de solução A, 14 μL de solução B, 33,3 μL de lisado BL21 de E. coli , 2,5 μL de proteína Tus 200 mM, 1,2 μL de RNA polimerase T7 100 mM, 1 μL de molde de DNA linear de 1,5 μM, 33,4 μL de água livre de nuclease.
NOTA: Consulte o Arquivo Suplementar 3 para obter instruções importantes sobre a preparação de um lisado livre de células E . coli BL21 e a Tabela Suplementar S1 para obter uma receita detalhada das Soluções A e B. O sistema de expressão livre de células de E. coli também pode ser preparado usando protocolos alternativos ou obtido comercialmente, desde que a reação seja suplementada com Tus e T7 RNA Polimerase. - Realize um teste inicial de expressão em escala de 5 μL adicionando 10% (v / v) de produto de PCR bruto à reação livre de células em um tubo de PCR. Incubar sem agitar a 30 °C durante 15 h.
- Verificar a qualidade da expressão do antigénio NP carregando 1 μL da reacção sem células num gel de poliacrilamida de dodecil sulfato de sódio a 12% (SDS-PAGE) e, em seguida, transferi-lo para uma membrana de nitrocelulose para western blot, conforme descrito no passo 2.12. Use a proteína repórter SpyCatcher2-Apex2 para marcação de manchas por meio do SpyTag e, opcionalmente, um anticorpo comercial específico (aqui um anti-SARS-CoV-2-NP) contra o antígeno alvo.
- Monte uma reação livre de células em escala de 1 mL com produto LET purificado de 15 nM para ser usado para o ensaio CFDB. Incubar a mistura de extração num tubo cónico de 15 ml, agitando a 80 RPM durante 15 h a 30 °C. Verificar a qualidade da expressão com western blot e armazenar alíquotas de 50 μL a -20 °C.
4. Amostras de soro
- Obtenha amostras de soro (ou plasma) humano/animal de fontes comerciais ou colaboradores clínicos ou internamente usando procedimentos padrão e seguindo as diretrizes do comitê institucional de ética em pesquisa humana ou do comitê de cuidados e uso de animais. Garantir que as medidas apropriadas de pré-tratamento da amostra foram tomadas para mitigar os riscos de contaminação.
NOTA: As medidas de pré-tratamento podem incluir tratamento com solvente-detergente, inativação por calor e teste de marcadores de vírus transmitidos pelo sangue. Um protocolo útil para isolamento de soro e plasma de sangue total está disponível12. Tal como acontece com outras plataformas de imunoensaio, não se espera que as amostras de soro coletadas precocemente (<3 semanas) após a infecção inicial ou o início da doença contenham níveis adequados de resposta de anticorpos e seus resultados devem ser tratados com cautela. - Para verificação da qualidade dos reagentes SARS-CoV-2 NP CFDB, use o painel de referência internacional do Instituto Nacional de Padrões e Controle Biológico (NIBSC) da Organização Mundial da Saúde (OMS) para imunoglobulina anti-SARS-CoV-2, que contém x1 amostras pré-COVID-19 e x4 SARS-CoV-2-positivas com níveis variados de imunoglobulinas anti-NP13. Como alternativa, use amostras de soro saudáveis e positivas pré-caracterizadas de outras fontes.
- Para experimentos CFDB, obter ou preparar uma amostra de controle negativo agrupando várias (>3) amostras de soro saudáveis para aumentar a precisão e facilitar a análise e interpretação dos resultados.
5. Procedimento de Cell-free Dot Blot (CFDB)
- Baixe e imprima o arquivo de imagem da grade mestra (Figura Suplementar S2). A grade mestra é um padrão de 6 x 6 cm contendo 12 x 12 círculos de 2 mm de diâmetro cada e fornece uma capacidade de localização semelhante a uma placa de 96 poços.
- Coloque a grade firmemente entre duas camadas de filme de vedação de placa PCR adesiva e corte no tamanho certo ao longo das bordas externas da grade. Use um punção de biópsia de 2 mm para esvaziar cada círculo marcado.
NOTA: Esta grade mestra pode ser reutilizada várias vezes após a limpeza com etanol 70%. - Corte um pedaço de membrana de nitrocelulose de 6.5 x 6.5 cm e posicione-o sob a grade mestre em uma superfície limpa, prendendo a configuração usando fita adesiva conforme mostrado na Figura 2 e na Figura Suplementar S3. Use uma caneta marcadora para marcar as posições do círculo mais externo na membrana de nitrocelulose a ser usada como guia para cortar a membrana após a detecção da amostra.
- Dilua as amostras de soro 1/10 em 1x solução salina tamponada com fosfato (pH 7,4). Use uma micropipeta para dispensar volumes triplicados de 0,4 μL de cada amostra na membrana de nitrocelulose em posições de grade predeterminadas. Levará aproximadamente 15 s por ponto para dispensar.
NOTA: Certifique-se de incluir amostras de controle negativas e positivas em cada ensaio, pois isso será necessário para a análise dos resultados. - Aguarde 10 minutos em temperatura ambiente para que as amostras manchadas se liguem e sequem. Usando uma pinça, recupere cuidadosamente a membrana de nitrocelulose e corte ao longo dos círculos externos marcados.
- Bloquear a membrana em 10 mL de solução bloqueadora (5% de leite em pó desnatado em TBST) em uma placa de Petri de 10 cm por 30 min em temperatura ambiente, agitando suavemente a 100 RPM.
- Descongelar e adicionar uma alíquota de 50 μL da mistura de expressão do antigénio sem células a 5 ml de solução de bloqueio numa placa de Petri de 10 cm. Transferir a membrana directamente para esta solução contendo antigénio e incubar à temperatura ambiente durante 1 h, agitando a 100 RPM.
NOTA: Esta será a etapa de detecção primária em que o antígeno SpyTagged se ligará a posições pontuais contendo anticorpos, se presentes. - Enxágue a membrana, lave por 5 min e enxágue novamente em TBST antes de prosseguir para a etapa de detecção secundária.
- Incube a membrana em 10 mL de tampão de bloqueio contendo 10 μg / mL de proteína SpyCatcher2-Apex2 purificada, por 1 h agitando a 100 RPM.
- Enxágue a membrana e lave 2 x 5 min em TBST, com um enxágue final em TBS.
- Cole e prenda um pedaço de parafilme em uma área de trabalho limpa perto do instrumento de imagem de borrão. Remova o excesso de líquido batendo na membrana com uma pinça e coloque a membrana em cima do parafilme.
- Adicione imediatamente 3 mL (100 μL / cm2) de solução de ECL no topo da membrana e incube em temperatura ambiente por exatamente 90 s.
- Seque imediatamente a membrana e transfira para um instrumento de imagem compatível com quimioluminescência para visualização dos resultados.
NOTA: O tempo ideal de imagem pode variar em diferentes instrumentos. Recomenda-se usar as configurações padrão de tempo de aquisição automática do instrumento e, se ausente, usar amostras de soro pré-caracterizadas para otimizar o tempo de aquisição da imagem. - Use a função de análise de imagem do instrumento para obter intensidades pontuais, inclusive para três posições em branco (fundo) na membrana de nitrocelulose; Mantenha um volume de medição constante por ponto. Exporte os dados para uma planilha, lembrando-se de rotular corretamente cada posição pontual.
- Calcule a média e o desvio padrão (DP) das intensidades pontuais triplicadas. Em seguida, subtraia o fundo da membrana de nitrocelulose de todas as amostras.
- Use a seguinte equação para obter um valor de corte para a interpretação dos resultados -/+:
(Média dos controles negativos) + (3 x DP dos controles negativos). Considere as amostras positivas se sua intensidade média cair acima do valor de corte e negativas se sua intensidade média cair abaixo do valor de corte.
NOTA: Um esquema do procedimento CFDB é fornecido na Figura 3.
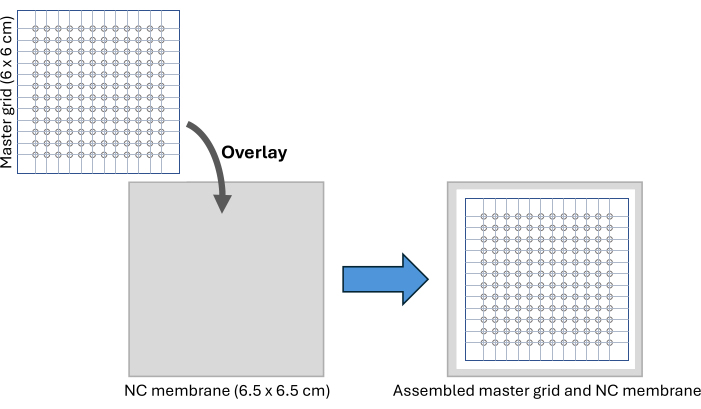
Figura 2: Montagem CFDB. Um esquema da grade mestre CFDB e da configuração da montagem da membrana NC. A grade mestra é sobreposta à membrana NC para fornecer um padrão regular e endereçável para detecção e imobilização de amostras de soro. Abreviaturas: CFDB = dot blot livre de células; NC = nitrocelulose. Clique aqui para ver uma versão maior desta figura.

Figura 3: Uma representação esquemática do fluxo de trabalho do CFDB. Em um ensaio CFDB, uma pequena quantidade (<0,4 μL) de amostras de soro diluídas 10x é dispensada manualmente em uma membrana de nitrocelulose pré-cortada (painel esquerdo) em locais discretos e endereçáveis (painel do meio). Depositar uma amostra de soro por ponto em pontos triplicados e imobilizar o conteúdo proteico, incluindo o reservatório total de anticorpos do soro, no substrato NC sólido (pontos bege no painel central). Neste exemplo, os anticorpos anti-NP contidos nas amostras de soro podem ser primeiro ligados pelo reagente de detecção primária CFDB SpyTag-NP e, finalmente, detectados pelo reagente de detecção secundária CFDB SpyCatcher2-Apex2 (bolha ampliada no painel direito). Essa figura foi retirada de Norouzi et al.5. Abreviaturas: CFDB = dot blot livre de células; NP = proteína do nucleocapsídeo; LET = modelo de expressão linear. Clique aqui para ver uma versão maior desta figura.
Resultados
Amplificação por PCR do modelo de expressão linear para antígeno alvo
Para amplificar o SARS-CoV-2 NP LET, foram utilizados primers universais Ter direto e reverso conforme descrito na seção 1.2 do protocolo e 1 μL do produto foi verificado em um gel de agarose (Figura 4) antes de prosseguir para a purificação do produto de PCR.
Discussão
A COVID-19 destacou a importância de diagnósticos acessíveis e robustos para controlar surtos de infecção e otimizar estratégias globais de saúde. A testagem sorológica que detecta anticorpos protetores mostrou-se essencial para rastrear padrões de transmissibilidade de novas variantes, identificar pontos quentes, orientar o desenvolvimento de vacinas, fazer a triagem de casos suspeitos e proteger populações vulneráveis14. A pandemia também expôs des...
Divulgações
M.N. e K.P. são co-inventores do método de dot blot sem células. Foi depositado um pedido provisório de patente relacionado a este trabalho (PCT/CA2024/050097, depositado em janeiro de 2024).
Agradecimentos
SS e RZ são apoiados por financiamento da Agência de Projetos de Pesquisa Avançada de Defesa (DARPA), Contrato nº N66001-23-2-4042. As visões, opiniões e/ou descobertas expressas são de responsabilidade dos autores e não devem ser interpretadas como representando as visões ou políticas oficiais do Departamento de Defesa ou do governo dos EUA. Este trabalho foi apoiado por fundos para K.P. do programa de subsídios da Fundação CIHR (201610FDN-375469), Programa de Cátedra de Pesquisa do CIHR Canadá (950-231075 e 950-233107), Iniciativa de Medicina por Design da Universidade de Toronto, que recebe financiamento do Fundo de Excelência em Pesquisa do Canadá e fundos para K.P., do Programa de Segurança e Proteção do Canadá de Pesquisa e Desenvolvimento de Defesa do Canadá (contrato 39903-200137). A Figura 1 e a Figura Suplementar S1 foram criadas usando o SnapGene Viewer.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 kb DNA ladder | NEB | N3232 | Used as a size marker for agarose gels |
| 20 Amino acids | Sigma-Aldrich | LAA21-1KT | A component of the cell-free reaction Solution B |
| 2 mm biopsy punch | Integra Miltex | 33-31-P/25 | For preparation of CFDB master grid |
| 5-aminolevulinic acid hydrochloride | Sigma-Aldrich | A3785 | Heme precursor for induction of SpyCatcher2-Apex2 |
| Agarose powder | BioShop | AGA002 | For electrophoretic analysis of DNA |
| Anti-SARS-CoV2-Nucleocapsid antibody | Sinobiological | 40588-T62 | For WB detection of cell-free produced NP antigen |
| Bio-Rad ChemiDoc XRS+ | Bio-Rad | N/A | Gel imager instrument |
| Centrifugal concentrators | Cytiva | 28-9323-60 | For concentration and buffer exchange of proteins |
| Centrifuge | Eppendorf | EP022628257 | For harvesting bacterial culture |
| Coenzyme A sodium salt hydrate (CoA) | Sigma-Aldrich | C3144 | A component of the cell-free reaction Solution A |
| Color pre-stained protein standard (broad range) | NEB | P7719 | Used as a size marker for SDS-PAGE gels |
| Covid-19 serum panel | RayBiotech | CoV-PosSet | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| D-(−)-3-Phosphoglyceric acid disodium salt (3-PGA) | Sigma-Aldrich | P8877 | A component of the cell-free reaction Solution B |
| Dithiothreitol (DTT) | Sigma-Aldrich | 10197777001 | Component of buffers |
| E. coli 5-alpha | NEB | C2987 | for plasmid preparation |
| E. coli BL21 | NEB | C2530 | For preparation of cell-free lysates |
| E. coli BL21 (DE3) | NEB | C2527 | For expression of SpyCatcher2-Apex2 |
| EDTA-free protease inhibitor tablet | Sigma-Aldrich | 11836153001 | A component of E. coli lysis buffer |
| Eppendorf New Brunswick Innova 43/43R Incubator Shaker | Eppendorf | EPM1320 | Incubator for growing E. coli cells for protein expression and cell-free lysate preparation |
| Folinic acid | Sigma-Aldrich | 47612 | A component of the cell-free reaction Solution A |
| Glycerol | Sigma-Aldrich | G9012 | Component of SpyCatcher2-Apex2 storage buffer |
| Hemin chloride | Sigma-Aldrich | H9039 | For Heme supplementation of SpyCatcher2-Apex2 |
| Hydrogen peroxide 30% | Sigma-Aldrich | H1009 | For making ECL reagent |
| Image Lab Software | Bio-Rad | 1709690 | Software for ChemiDoc gel imaging instrument |
| Isopropyl-b-D-1-thiogalactopyranoside | Bioshop | IPT001 | For induction of SpyCatcher2-Apex2 expressikon |
| Kanamycin Sulfate | Sigma-Aldrich | 60615 | For preparation of SpyCatcher2-Apex2 bacterial culture |
| LB agar | BioShop | LBL406 | For E. coli growth |
| LB broth | BioShop | LBL407 | For E. coli growth |
| luminol | Sigma-Aldrich | A4685 | For making ECL reagent |
| Lysozyme | Sigma-Aldrich | L6876 | For lysis of bacterial cells |
| Magnesium Acetate | Sigma-Aldrich | M5661 | A component of the cell-free reaction Solution B |
| NEBExpress Ni resin | NEB | S1428S | For purification of SpyCatcher2-Apex2 |
| Non-fat dry milk | Bioshop | SKI400 | For blocking of WB and CFDB membranes |
| Parafilm | Bemis | 2099-1337410 | For ECL-incubation of CFDB blots |
| p-coumaric acid | Sigma-Aldrich | C9008 | For making ECL reagent |
| pET24b-SpyCatcher2-Apex2 plasmid | Pardee Laboratory | N/A | Used for the expression of SpyCatcher2-Apex2 protein |
| Petri dish | Fisherbrand | FB0875712 | Container for WB and CFDB membrane incubation |
| Phosphate buffered saline 10% | BioShop | PBS405 | Component of buffers |
| Potassium Glutamate | Sigma-Aldrich | G1501 | A component of the cell-free reaction Solution B |
| Potassium Oxalate Monohydrate (Oxalic acid) | Sigma-Aldrich | 223425 | A component of the cell-free reaction Solution A |
| Precast SDS-PAGE gel | BioRad | 4561036EDU | For electrophoretic analysis of protein |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification of LETs |
| QIAquick PCR purification kit | Qiagen | 28106 | For purification of LETs |
| Ribonucleotide Solution Set | NEB | N0450S | A component of the cell-free reaction Solution A |
| Skim milk powder | BioShop | SKI400 | Used as a blocking agent for western blot and CFDB |
| Sodium Chloride | Sigma-Aldrich | S9625 | A component of protein purification buffers |
| Sonicator | Qsonica | Q500 | For lysis of bacterial cells |
| Spermidine | Sigma-Aldrich | S2626 | A component of the cell-free reaction Solution A |
| Syringe filter | Sigma-Aldrich | SLGSR33SS | For sterilisation of buffers and solutions |
| Thermocycler | BioRad | T100 | Instrument for incubating PCR reactions |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8759 | A component of the cell-free reaction Solution A |
| Tris buffered saline | Thermo Scientific | J60764.K2 | Component of WB and CFDB wash buffer |
| Trizma base | Sigma-Aldrich | T1503 | For making tris buffer |
| Tunair SS-5012 Half-Baffle Shake Flask, 2.5 L | Cole-Parmer | RK-01835-39 | Vessel for growing E. coli cells for protein expression and cell-free lysate preparation |
| Tween-20 | BioShop | TWN510 | Component of WB and CFDB wash buffer |
| Tweezer | Almedic | 7728-A10-100 | For handling CFDB NC membrane |
| UV-Vis spectrophotometer | Thermo Scientific | 13400518 | Used for the measurement of nucleic acid and DNA concentration and bacterial culture OD |
| WHO International Reference Panel for anti-SARS-CoV-2 immunoglubulin | NISBC | 20/268 | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| β-Nicotinamide adenine dinucleotide hydrate (NAD) | Sigma-Aldrich | 10127965001 | A component of the cell-free reaction Solution A |
Referências
- Carter, L. J., et al. Assay techniques and test development for COVID-19 diagnosis. ACS Cent Sci. 6, 591-605 (2020).
- Amanat, F., et al. A serological assay to detect SARS-CoV-2 seroconversion in humans. Nat Med. 26 (7), 1033-1036 (2020).
- Stadlbauer, D., et al. SARS-CoV-2 seroconversion in humans: A detailed protocol for a serological assay, antigen production, and test setup. Curr Protoc Microbiol. 57 (1), e100 (2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259.e12 (2016).
- Norouzi, M., et al. Cell-free dot blot: an ultra-low-cost and practical immunoassay platform for detection of anti-SARS-CoV-2 antibodies in human and animal sera. Microbiol Spectr. 11 (2), e0245722 (2023).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Norouzi, M., Panfilov, S., Pardee, K. High-efficiency protection of linear DNA in cell-free extracts from Escherichia coli and Vibrio natriegens. ACS Synth Biol. 10 (7), 1615-1624 (2021).
- . IDT codon optimization tool Available from: https://www.idtdna.com/pages/tools/codon-optimization-tool (2025)
- . General protocol for western blotting Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6376.pdf (2025)
- Mruk, D. D., Cheng, C. Y. Enhanced chemiluminescence (ECL) for routine immunoblotting. Spermatogenesis. 1, 121-122 (2011).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-based cell-free protein synthesis: Protocols for a robust, flexible, and accessible platform technology. J Vis Exp. (144), (2019).
- . Plasma and serum preparation Available from: https://www.thermofisher.com/ca/en/home/references/protocols/cell-and-tissue-analysis/elisa-protocol/elisa-sample-preparation-protocols/plasma-and-serum-preparation.html (2025)
- Mattiuzzo, G., et al. . WHO/BS.2020.2403 Establishment of the WHO International Standard and Reference Panel for anti-SARS-CoV-2 antibody. , (2020).
- Gong, F., Wei, H., Li, Q., Liu, L., Li, B. Evaluation and comparison of serological methods for COVID-19 diagnosis. Front Mol Biosci. 8, 682405 (2021).
- Peeling, R. W., et al. Serology testing in the COVID-19 pandemic response. Lancet Infect Dis. 20 (9), e245-e249 (2020).
- Smith, M. T., Berkheimer, S. D., Werner, C. J., Bundy, B. C. Lyophilized Escherichia coli-based cell-free systems for robust, high-density, long-term storage. Biotechniques. 56 (4), 186-193 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados