Method Article
Cell-free dot blot como plataforma de inmunoensayo práctica y adaptable para la detección de la respuesta de anticuerpos en sueros humanos y animales
En este artículo
Resumen
Describimos una plataforma de inmunoensayo recientemente desarrollada basada en los principios de la biología sintética libre de células y la técnica dot-blot para la detección personalizable de la respuesta de anticuerpos en sueros humanos y animales.
Resumen
La serie de brotes patógenos mundiales de las dos últimas décadas ha puesto de relieve la importancia de las estrategias de serovigilancia. Las plataformas de inmunoensayo que sirven para detectar anticuerpos específicos de la enfermedad en los sueros de los pacientes son el núcleo de la serovigilancia. Algunos ejemplos comunes son los ensayos de inmunoabsorción enzimática y los ensayos de flujo lateral; Sin embargo, si bien estos son métodos estándar de oro, requieren consumibles específicos para patógenos y equipos especializados, lo que limita su uso fuera de laboratorios con buenos recursos.
Recientemente hemos desarrollado una novedosa plataforma de inmunoensayo llamada Cell-Free Dot-Blot (CFDB) y la hemos validado utilizando sueros humanos y animales contra el SARS-CoV-2. A diferencia de los inmunoensayos convencionales, las muestras de suero de pacientes con CFDB se inmovilizan a una fase sólida (membrana de nitrocelulosa), mientras que el antígeno objetivo se suspende en la fase móvil del ensayo. Para mejorar el acceso a las capacidades de serovigilancia, los antígenos CFDB se producen bajo demanda y con una infraestructura de baja carga utilizando la expresión de proteínas in vitro . Aquí, el antígeno se fusiona con una etiqueta peptídica que se puede detectar utilizando una sola proteína reportera universal para cualquier ensayo CFDB. El resultado es que el CFDB no requiere acceso a un lector de placas de pocillos múltiples ni a componentes de ensayos moleculares comerciales purificados. Con estas consideraciones de diseño, CFDB aborda las deficiencias de las plataformas de inmunoensayo existentes al proporcionar accesibilidad a laboratorios no centralizados, adaptabilidad para patógenos emergentes y asequibilidad para las comunidades de bajos ingresos.
En el presente artículo, proporcionaremos un protocolo paso a paso para preparar y realizar un inmunoensayo CFDB. Utilizando nuestro trabajo reciente sobre el SARS-CoV-2 CFDB como ejemplo, cubriremos el diseño de ADN de antígenos para la producción libre de células bajo demanda, seguido de la preparación de la proteína reportera CFDB, la inmovilización de muestras de suero en la fase sólida y, finalmente, los pasos de unión y detección de antígenos del ensayo. Anticipamos que siguiendo estas instrucciones, los investigadores podrán adaptar el ensayo CFDB para detectar respuestas inmunitarias en sueros humanos y animales a cualquier patógeno dado.
Introducción
La pandemia de COVID-19 puso de manifiesto la necesidad crítica de contar con herramientas de diagnóstico asequibles y escalables, en particular para entornos de bajos recursos1. Los inmunoensayos convencionales, como los ensayos de inmunoabsorción enzimática (ELISA), han demostrado ser esenciales para detectar respuestas inmunitarias 2,3. Sin embargo, su alto costo, la dependencia de reactivos complejos y la dependencia de equipos especializados limitan su accesibilidad, especialmente durante las crisis sanitarias mundiales. En respuesta a estos desafíos, desarrollamos el Cell-Free Dot Blot (CFDB), una plataforma de inmunoensayo adaptable y de bajo costo diseñada para la detección de anticuerpos anti-SARS-CoV-2 en sueros humanos y animales.
CFDB aprovecha la biología sintética libre de células para la producción rápida y bajo demanda de antígenos virales utilizando plantillas lineales de ADN 4,5. Esto elimina la necesidad de procesos tradicionales de clonación, expresión y purificación basados en células, lo que acelera significativamente la producción de antígenos y reduce los costos. El método CFDB simplifica la detección de anticuerpos mediante el uso de un formato de transferencia de puntos, en el que los sueros se manchan directamente en las membranas de nitrocelulosa. Este sistema evita la necesidad de costosas placas de pocillos múltiples y equipos de laboratorio especializados, lo que permite un flujo de trabajo de "inmersión" simple para los pasos de incubación y lavado. La plataforma también utiliza un sistema SpyCatcher-SpyTag, donde una quimera peroxidasa SpyCatcher2-Apex2 actúa como reactivo de detección secundaria universal 5,6. Esto se produce utilizando la expresión estándar basada en Escherichia coli, lo que elimina la dependencia de costosos conjugados de anticuerpos comerciales. Como resultado, el sistema CFDB puede realizar ensayos serológicos con un rendimiento comparable al de los ELISA a un costo significativamente menor: alrededor de $3 USD por ensayo de 96 muestras en comparación con más de $300 USD para un kit ELISA comercial5.
Para demostrar la eficacia de CFDB, probamos su capacidad para detectar anticuerpos en sueros humanos y animales precaracterizados. Nuestros resultados se correlacionaron estrechamente con ELISA en la identificación de muestras positivas y negativas de COVID-19. Además del diagnóstico en humanos, evaluamos la utilidad de CFDB en modelos animales, analizando sueros de hámsteres infectados con SARS-CoV-2 y vacunados con proteína nucleocápside recombinante. Estas pruebas confirmaron el potencial de CFDB para su uso tanto en el diagnóstico humano como en el veterinario, lo que lo convierte en una herramienta versátil para monitorear las respuestas inmunitarias en todas las especies. Una de las principales ventajas de CFDB es su flexibilidad. Con solo modificar la plantilla de ADN que codifica el antígeno de interés, la plataforma se puede adaptar rápidamente para detectar anticuerpos contra diferentes patógenos, lo que la hace valiosa para la preparación ante futuras pandemias. Su bajo costo, flujo de trabajo simple y requisitos mínimos de infraestructura lo hacen particularmente adecuado para laboratorios descentralizados y entornos de bajos recursos, donde el acceso a los diagnósticos comerciales es limitado.
En este trabajo, proporcionaremos instrucciones paso a paso para preparar y realizar un ensayo CFDB. En primer lugar, abordamos el diseño y la síntesis de plantillas lineales de ADN para la producción libre de células de antígenos, que son los principales reactivos de detección del ensayo. A continuación, describimos los pasos para la preparación del reactivo de detección secundaria del ensayo SpyCatcher2-Apex2. Después de eso, proporcionamos instrucciones para la producción libre de células y el control de calidad de los antígenos mismos. Finalmente, describimos en detalle el proceso para realizar un ensayo de CFDB en muestras de suero humano o animal.
Protocolo
Todos los experimentos con hámsters se realizaron en el Laboratorio Nacional de Microbiología (NML) de la Agencia de Salud Pública de Canadá, aprobados por el Centro Canadiense de Ciencias para la Salud Humana y Animal, y siguiendo las directrices del Consejo Canadiense de Cuidado Animal. Todas las muestras de suero/plasma humano se obtuvieron comercialmente para pruebas internas o fueron proporcionadas por colaboradores clínicos al NML para pruebas independientes en el NML.
1. Diseño y preparación de plantillas de expresión lineal de antígenos (LETs)
- Diseñar los LETs de expresión libre de células para el antígeno diana basándose en las instrucciones de Norouzi et al.7 y que contengan un SpyTag N/C-terminal como se describe en Norouzi et al.5.
NOTA: Aquí proporcionamos detalles para la preparación de la proteína de la nucleocápside del SARS-CoV-2 (NP, aminoácidos 2-419).- Asegúrese de que el LET para el NP del SARS-CoV-2 (Figura 1 y Archivo complementario 1) contenga sitios Ter 5' y 3' con una secuencia de tampón de 50 pares de bases antes del promotor T7 y después del codón de parada.
- Marque la proteína NP en el extremo N-terminal con su 6-SpyTag-TEV.
- Obtenga la secuencia de proteínas de la base de datos UniProt (código de acceso P0DTC9) y optimícela para la expresión basada en E. coli utilizando la herramienta de optimización de codones IDT8. Solicite esta secuencia para la síntesis comercial como un fragmento de ADN monocatenario resuspendido en agua a 10 ng/μL.
NOTA: El sitio de escisión de la proteasa TEV y la etiqueta His6 no son características esenciales en el diseño LET.
- La PCR amplifica el fragmento de ADN con el Ter FW universal (GGCTCCGAATAAGTGTTGTAACTAAAGTGCGGCC
ACGATGCGTCCGGCGTAGAGGATCG) y Ter RV (CCGAGGCAATAAGTGTTGTAACTAAAGTGCTCAG
CTTCCTTTCGGGCTTTGTTAGCAGCC) utilizando el kit de ADN polimerasa de alta fidelidad (ver la Tabla de Materiales)- Configure una reacción de 100 μL de la siguiente manera: 75 μL de agua libre de nucleasas, 20 μL de tampón de ADN polimerasa 5x, 2 μL de dNTP de 10 mM (concentración final de 200 μM), 0,5 μL de cebador Ter-FW de 100 μM, 0,5 μL de cebador Ter-RV de 100 μM, 1 μL de LET monocatenario de 10 ng/μL, 1 μL (2U) de ADN polimerasa.
- Utilice los siguientes ajustes de PCR: desnaturalización inicial a 98 °C durante 30 s; 35 ciclos: 98 °C durante 6 s, 60 °C durante 15 s, 72 °C durante 90 s; mantener a 4 °C.
- Purifique el producto de PCR utilizando un kit de purificación de PCR comercial, verifique su calidad ejecutando una muestra en un gel de agarosa al 1% y mida su concentración en un espectrofotómetro UV-Vis.
NOTA: Los productos de PCR crudos también se pueden usar directamente para la expresión sin células. Sin embargo, la purificación permite un procedimiento más estandarizado.

Figura 1: Plantilla de expresión lineal para SARS-CoV-2-NP. Un esquema que representa las características de la plantilla de ADN lineal His-SpyTag-SARS-CoV-2 NP. Los elementos clave de la plantilla de ADN están etiquetados. La secuencia codificante de la proteína de interés, en este caso la NP, se coloca bajo el control transcripcional de un promotor T7 para una expresión eficiente. En el extremo N, a la proteína NP se le añade una etiqueta SpyTag para la detección específica utilizando el reactivo de detección SpyCatcher2-Apex2. Los sitios de proteasa x6His-tag y TEV, aunque se incluyen como parte del diseño general de LET, son prescindibles para fines de CFDB. En el extremo de la plantilla lineal de ADN, se incluyen los sitios Ter "aguas arriba" y "aguas abajo", cada uno precedido por secuencias tampón de 50 pares de bases respectivas, para la protección mediada por Tus contra la degradación del ADN exonucleolítico en el lisado libre de células. Abreviaturas: NP = proteína nucleocápside; LET = plantilla de expresión lineal; TEV = virus del grabado del tabaco; CFDB = transferencia de puntos sin celdas. Haga clic aquí para ver una versión más grande de esta figura.
2. Purificación de la proteína reportera SpyCatcher2-Apex2
- Utilice el plásmido pET24b-SpyCatcher2-Apex2, construido originalmente en Norouzi et al.5 (secuencia completa en el Archivo Suplementario 2 y mapa de plásmidos en la Figura Suplementaria S1) para preparar la proteína reportera SpyCatcher2-Apex-2.
- Prepare placas de agar y caldo de lisogénia (LB) que contenga 50 μg/mL de kanamicina. Transforme células de E. coli BL21 (DE3) con el plásmido pET24b-SpyCatcher2-Apex2. Inocular una sola colonia en un cultivo iniciador de LB de 15 mL y cultivar durante la noche a 37 °C con agitación a 250 RPM.
- Al día siguiente, agregue 10 mL del cultivo iniciador en 500 mL de medio LB fresco que contenga 50 μg/mL de kanamicina. Incubar a 37 °C con agitación a 250 RPM hasta que el cultivo alcance una densidad óptica a 600 nm de 0,6-0,8 (~3 h).
- Inducir la expresión de SpyCatcher2-Apex2 suplementando el cultivo con 0,5 mM de isopropil-β-D-1-tiogalactopiranósido y 1 mM de clorhidrato de ácido 5-aminolevulínico. Reduzca la temperatura de crecimiento a 30 °C y deje que el cultivo se incube durante otras 4 h.
- Recolectar las bacterias por centrifugación a 8.000 × g durante 15 min. Proceda a la lisis celular o, alternativamente, almacene el pellet a -80 °C hasta su uso.
- Vuelva a suspender el pellet en 20 mL de tampón de lisis que contenga 50 mM de Tris-HCl (pH 7,8), 300 mM de NaCl, 1 mg/mL de lisozima, comprimido inhibidor de la proteasa sin EDTA y 1 mM de ditiotreitol (DTT). Lisar las células por sonicación al 50 % de amplitud con intervalos de 5 s ON y 10 s OFF durante un tiempo total de ON de 3 min.
- Clarificar el lisado por centrifugación a 20.000 × g durante 1 h a 4 °C. Pase el sobrenadante a través de un filtro de jeringa de 0,2 μm.
- Añadir cloruro de hemina hasta una concentración final de 250 μM al lisado clarificado e incubar durante la noche a 4 °C. Proceda a la purificación de proteínas.
NOTA: La incubación con cloruro de hemina maximiza la incorporación de hemo en la peroxidasa Apex-2 para una actividad enzimática óptima. - Añadir 2,5 mL de resina de Ni al lisado clarificado e incubar a 4 °C agitando suavemente durante 45 min. Aplique la mezcla a una columna de flujo por gravedad y lave la resina con 50 mL de tampón Tris (50 mM de Tris-HCl (pH 7,8), 300 mM de NaCl y 1 mM de DTT).
- Eluir la proteína SpyCatcher2-Apex2 en 25 mL de tampón Tris que contiene 400 mM de imidazol. El concentrado y el tampón intercambian el eluido en tampón Tris utilizando una unidad de filtro centrífugo, con el objetivo de obtener un volumen final de 0,5-1,0 mL.
- Utilice el coeficiente de extinción molar de SpyCatcher2-Apex2 (27.390 M-1 cm-1) para determinar la concentración de proteínas en un espectrofotómetro UV-Vis. Añadir glicerol hasta una concentración final del 40% y almacenar las alícuotas a -20 °C.
NOTA: Se espera que este protocolo produzca aproximadamente 40 mg de SpyCatcher2-Apex2 altamente puro y activo, suficiente para 400 ejecuciones de CFDB en un tamaño de transferencia estándar de 96 muestras. - Para la detección de proteínas SpyTagged utilizando Western blot9 convencional y para experimentos con CFDB, siempre bloquee las membranas en leche en polvo descremada al 5% en solución salina tamponada con 1x Tris que contenga 0,05% de Tween-20 (TBST). Utilice la proteína SpyCatcher2-Apex2 a una concentración final de 10 μg/mL en solución bloqueante.
NOTA: El reportero SpyCatcher2-Apex2 requiere una solución de quimioluminiscencia mejorada (ECL) para el desarrollo de señales. La solución ECL puede obtenerse comercialmente o prepararse internamente de acuerdo con las instrucciones de Mruk et al.10. La solución final de ECL aquí consiste en 0,4 mM de ácido p-cumárico, 2,5 mM de luminol y 0,015% deH2O2 en 100 mM de Tris-HCl (pH 8,6).
3. Producción libre de células y control de calidad de antígenos
- Prepare y ensamble un lisado de expresión libre de células y componentes de reacción de E. coli BL21 siguiendo las instrucciones de Levine et al.11 y Norouzi et al.5. Complemente la mezcla de reacción final con 5 μM de proteína Tus y 1,2 μM de ARN polimerasa T7 de la siguiente manera para una reacción representativa de 100 μL: 14,6 μL de solución A, 14 μL de solución B, 33,3 μL de lisado BL21 de E. coli , 2,5 μL de proteína Tus 200 mM, 1,2 μL de ARN polimerasa T7 100 mM, 1 μL de molde de ADN lineal de 1,5 μM, 33,4 μL de agua libre de nucleasas.
NOTA: Consulte el Archivo Suplementario 3 para obtener instrucciones clave sobre cómo preparar un lisado libre de células de E. coli BL21 y la Tabla Suplementaria S1 para obtener una receta detallada de las Soluciones A y B. El sistema de expresión libre de células de E. coli también se puede preparar utilizando protocolos alternativos u obtener comercialmente, siempre que la reacción se complemente con Tus y ARN polimerasa T7. - Realice una prueba de expresión inicial a escala de 5 μL añadiendo un 10% (v/v) de producto de PCR crudo a la reacción libre de células en un tubo de PCR. Incubar sin agitar a 30 °C durante 15 h.
- Compruebe la calidad de expresión del antígeno NP cargando 1 μL de la reacción libre de células en un gel de poliacrilamida de dodecil sulfato de sodio al 12% (SDS-PAGE) y posteriormente transfiéralo a una membrana de nitrocelulosa para Western blot como se describe en el paso 2.12. Utilice la proteína reportera SpyCatcher2-Apex2 para el etiquetado de la transferencia a través de SpyTag y, opcionalmente, un anticuerpo comercial específico (en este caso, un anti-SARS-CoV-2-NP) contra el antígeno objetivo.
- Ensamble una reacción libre de células a escala de 1 mL con un producto LET purificado de 15 nM para utilizarlo en el ensayo CFDB. Incubar la mezcla de expresión en un tubo cónico de 15 mL agitando a 80 rpm durante 15 h a 30 °C. Compruebe la calidad de la expresión con Western blot y almacene 50 μL de alícuotas a -20 °C.
4. Muestras de suero
- Obtener muestras de suero (o plasma) humano/animal de fuentes comerciales o colaboradores clínicos o internamente utilizando procedimientos estándar y siguiendo las pautas del comité institucional de ética de investigación humana o del comité de cuidado y uso animal. Asegúrese de que se han tomado las medidas adecuadas de pretratamiento de la muestra para mitigar los riesgos de contaminación.
NOTA: Las medidas de pretratamiento pueden incluir tratamiento con solvente-detergente, inactivación por calor y pruebas de marcadores de virus transmitidos por la sangre. Se dispone de un protocolo útil para el aislamiento sérico y plasmático de sangre total12. Al igual que con otras plataformas de inmunoensayo, no se espera que las muestras de suero recolectadas temprano (<3 semanas) después de la infección inicial o el inicio de la enfermedad contengan niveles adecuados de una respuesta de anticuerpos y sus resultados deben tratarse con precaución. - Para la verificación de la calidad de los reactivos NP CFDB del SARS-CoV-2, utilice el panel de referencia internacional de la Organización Mundial de la Salud (OMS) del Instituto Nacional de Normas y Control Biológicos (NIBSC) para la inmunoglobulina anti-SARS-CoV-2, que contiene x1 muestras pre-COVID-19 y x4 muestras positivas para el SARS-CoV-2 con niveles variables de inmunoglobulinas anti-NP13. Alternativamente, use muestras de suero sanas y positivas precaracterizadas de otras fuentes.
- En el caso de los experimentos con CFDB, obtenga o prepare una muestra de control negativa agrupando varias (>3) muestras de suero sano para aumentar la precisión y facilitar el análisis y la interpretación de los resultados.
5. Procedimiento de transferencia de puntos sin células (CFDB)
- Descargue e imprima el archivo de imagen de la cuadrícula maestra (Figura complementaria S2). La rejilla maestra es un patrón de 6 x 6 cm que contiene 12 x 12 círculos de 2 mm de diámetro cada uno y proporciona una capacidad de detección similar a una placa de 96 pocillos.
- Intercala firmemente la rejilla entre dos capas de película adhesiva de sellado de placa PCR y córtela a medida a lo largo de los bordes exteriores de la rejilla. Use un punzón de biopsia de 2 mm para ahuecar cada círculo marcado.
NOTA: Esta rejilla maestra se puede reutilizar varias veces después de limpiarla con etanol al 70%. - Corte un trozo de membrana de nitrocelulosa de 6,5 x 6,5 cm y colóquelo debajo de la rejilla maestra sobre una superficie limpia, asegurando la configuración con cinta adhesiva como se muestra en la Figura 2 y la Figura complementaria S3. Utilice un rotulador para marcar las posiciones de los círculos más exteriores de la membrana de nitrocelulosa que se utilizará como guía para cortar la membrana después de la detección de la muestra.
- Diluir las muestras de suero 1/10 en solución salina tamponada con fosfato 1x (pH 7,4). Utilice una micropipeta para dispensar volúmenes triplicados de 0,4 μL de cada muestra en la membrana de nitrocelulosa en posiciones de rejilla predeterminadas. Se necesitarán aproximadamente 15 s por punto para dispensar.
NOTA: Asegúrese de incluir muestras de control negativas y positivas con cada ensayo, ya que esto será necesario para el análisis de los resultados. - Espere 10 minutos a temperatura ambiente para que las muestras manchadas se unan y sequen. Con unas pinzas, recupere con cuidado la membrana de nitrocelulosa y corte a lo largo de los círculos exteriores marcados.
- Bloquee la membrana en 10 mL de solución bloqueante (5% de leche en polvo descremada en TBST) en una placa de Petri de 10 cm durante 30 min a temperatura ambiente, agitando suavemente a 100 RPM.
- Descongele y añada una alícuota de 50 μL de la mezcla de expresión de antígeno libre de células a 5 mL de solución de bloqueo en una placa de Petri de 10 cm. Transfiera la membrana directamente a esta solución que contiene antígeno e incube a temperatura ambiente durante 1 h, agitando a 100 RPM.
NOTA: Este será el paso de detección principal en el que el antígeno SpyTagged se unirá a las posiciones puntuales que contienen anticuerpos, si están presentes. - Enjuague la membrana, lávela durante 5 minutos y vuelva a enjuagar en TBST antes de continuar con el paso de detección secundario.
- Incubar la membrana en 10 mL de tampón de bloqueo que contenga 10 μg/mL de proteína SpyCatcher2-Apex2 purificada, durante 1 h mientras se agita a 100 RPM.
- Enjuague la membrana y lave 2 x 5 min en TBST, con un enjuague final en TBS.
- Pegue y asegure un trozo de parafilm en un área de trabajo limpia cerca del instrumento de adquisición de imágenes de mancha. Retire el exceso de líquido golpeando la membrana con unas pinzas y coloque la membrana encima del parafilm.
- Añada inmediatamente 3 mL (100 μL/cm2) de solución de ECL sobre la membrana e incube a temperatura ambiente durante exactamente 90 s.
- Seque inmediatamente la membrana y transfiérala a un instrumento de imágenes compatible con la quimioluminiscencia para la visualización de los resultados.
NOTA: El tiempo óptimo de obtención de imágenes puede variar en diferentes instrumentos. Se recomienda utilizar los ajustes de tiempo de adquisición automática predeterminados del instrumento y, si están ausentes, utilizar muestras de suero precaracterizadas para optimizar el tiempo de adquisición de imágenes. - Utilice la función de análisis de imágenes del instrumento para obtener intensidades puntuales, incluso para tres posiciones en blanco (fondo) en la membrana de nitrocelulosa; Mantenga un volumen de medición constante por punto. Exporte los datos a una hoja de cálculo, recordando etiquetar correctamente cada posición de punto.
- Calcule la media y la desviación estándar (DE) de las intensidades puntuales por triplicado. A continuación, reste el fondo de la membrana de nitrocelulosa de todas las muestras.
- Utilice la siguiente ecuación para obtener un valor de corte para la interpretación de los resultados de -/+:
(Media de los controles negativos) + (3 x DE de los controles negativos). Considere las muestras positivas si su intensidad media cae por encima del valor de corte y negativas si su intensidad media cae por debajo del valor de corte.
NOTA: En la Figura 3 se proporciona un esquema del procedimiento CFDB.
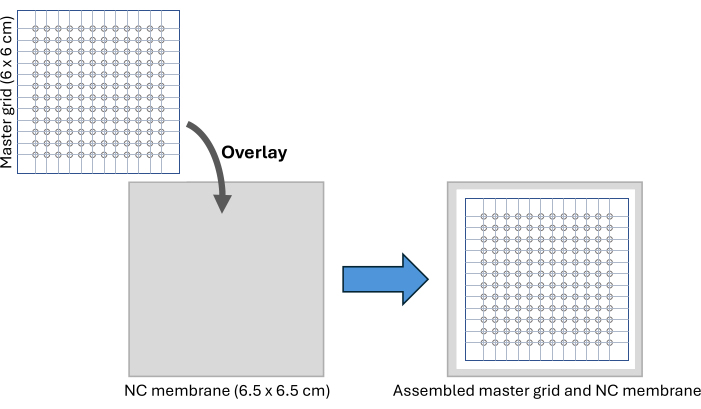
Figura 2: Ensamblaje de CFDB. Un esquema de la rejilla maestra CFDB y la configuración del ensamblaje de membrana NC. La rejilla maestra se superpone a la membrana NC para proporcionar un patrón regular y direccionable para la detección e inmovilización de muestras de suero. Abreviaturas: CFDB = transferencia de puntos sin células; NC = nitrocelulosa. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Una representación esquemática del flujo de trabajo de CFDB. En un ensayo CFDB, una pequeña cantidad (<0,4 μL) de muestras de suero diluido 10x se dispensa manualmente en una membrana de nitrocelulosa precortada (panel izquierdo) en ubicaciones discretas y direccionables (panel central). Depositar una muestra de suero por mancha por triplicado e inmovilizar el contenido de proteína, incluido el reservorio total de anticuerpos del suero, sobre el sustrato NC sólido (manchas beige en el panel central). En este ejemplo, los anticuerpos anti-NP contenidos en las muestras de suero pueden ser unidos primero por el reactivo de detección primaria CFDB SpyTag-NP y finalmente detectados por el reactivo de detección secundaria CFDB SpyCatcher2-Apex2 (burbuja ampliada en el panel derecho). Esta figura fue tomada de Norouzi et al.5. Abreviaturas: CFDB = transferencia de puntos sin células; NP = proteína de la nucleocápside; LET = plantilla de expresión lineal. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Amplificación por PCR de la plantilla de expresión lineal para el antígeno diana
Para amplificar por PCR el LET NP del SARS-CoV-2, se utilizaron cebadores universales Ter directos e inversos como se describe en la sección 1.2 del protocolo y se comprobó 1 μL del producto en un gel de agarosa (Figura 4) antes de proceder a la purificación del producto PCR.

Figura 4: PCR de la plantilla de expresión lineal. Un gel de agarosa al 1% que se muestra en el carril 1: 1 kb DNA Ladder y en el carril 2: la plantilla de ADN lineal amplificada por PCR para el His-SpyTag-SARS-CoV-2 NP (1.630 pb). Abreviaturas: PCR = reacción en cadena de la polimerasa; NP = proteína de la nucleocápside. Haga clic aquí para ver una versión más grande de esta figura.
Purificación de la proteína reportera SpyCatcher2-Apex2
La proteína SpyCatcher2-Apex2 se preparó como se describe en la sección 2 del protocolo. Para asegurar una incorporación adecuada de hemo en la peroxidasa Apex2, se añadieron clorhidrato de ácido 5-aminolevulínico (un precursor del hemo) y cloruro de hemina durante la síntesis de proteínas y después de la lisis celular, respectivamente. Las muestras de todos los pasos de purificación se cargaron en un gel SDS-PAGE para garantizar la calidad del producto final (Figura 5).
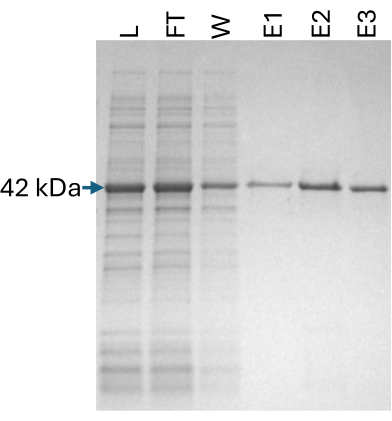
Figura 5: Purificación de SpyCatcher2-Apex2. Un gel SDS-PAGE al 12% que muestra fracciones de los pasos de purificación de la proteína SpyCatcher2-Apex2. L: lisado (2 μL), FT: flujo (2 μL), W: lavado (10 μL), E1-3: elución 1-3 (10 μL) cada uno. La banda esperada a 42 kDa está marcada. Abreviatura: SDS-PAGE = electroforesis en gel de poliacrilamida dodecil sulfato de sodio. Haga clic aquí para ver una versión más grande de esta figura.
Expresión libre de células y verificación de la calidad del antígeno diana
Después de realizar una expresión piloto de la proteína NP a escala de 5 μL y un control de calidad mediante SDS-PAGE, se realizó una expresión a escala de 1 mL como se describe en la sección 3 del protocolo. Una muestra de 1 μL de la mezcla final de expresión libre de células se sometió a Western blot por separado para su detección mediante SpyCatcher2-Apex2 y un anticuerpo comercial anti-NP (Figura 6). Este paso garantiza que el antígeno se exprese con éxito y que pueda unirse específicamente tanto a las inmunoglobulinas anti-NP como a la proteína reportera SpyCatcher2-Apex2.
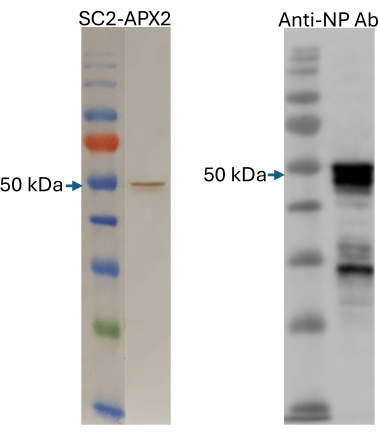
Figura 6: Control de calidad de antígenos producidos sin células. Detección por Western blot de la proteína NP producida libremente mediante SpyCatcher2-Apex2 (panel izquierdo) y un anticuerpo policlonal anti-NP de conejo (panel derecho). Se marca el producto de longitud completa esperado a 50 kDa. El marcador de tamaño en ambos geles es el Color Pre-stained Protein Standard, Broad Range. Esta figura fue adaptada de Norouzi et al.5. Abreviatura: NP = proteína nucleocápside. Haga clic aquí para ver una versión más grande de esta figura.
Verificación de reactivos CFDB utilizando sueros humanos precaracterizados
Para confirmar que los reactivos de CFDB son de alta calidad y que los protocolos de CFDB se implementan con éxito, utilizamos un panel de suero precaracterizado proporcionado por el NISBC-panel internacional de referencia de la OMS para la inmunoglobulina anti-SARS-CoV-2 20/26813. Este panel consta de x1 muestra pre-COVID-19 (negativa) y x4 muestras positivas para SARS-CoV-2 con diferentes niveles de respuesta de anticuerpos (Tabla suplementaria S2). También adquirimos un panel de suero de RayBiotech (CoV-PosSet) que contenía 10 muestras pre-COVID-19 (negativas) y 20 presuntas positivas para SARS-CoV-2. Agrupamos fracciones iguales de los 10 sueros negativos para utilizarlas como control negativo en todos los experimentos realizados en nuestro laboratorio. Esta estrategia de agrupación aumenta la precisión y la solidez de los resultados, ya que las muestras negativas individuales pueden exhibir niveles sutiles de señal de fondo que pueden complicar la interpretación de los resultados.
Se realizó un ensayo de CFDB en el panel de referencia de la OMS precaracterizado y en el CoV-PosSet de RayBiotech de acuerdo con la sección 5 del protocolo, y se obtuvieron imágenes del blot. Se utilizó la herramienta de volumen del software Image Lab para dibujar círculos uniformes en cada posición puntual y exportar intensidades de señal individuales. Después de restar la intensidad de fondo de la membrana NC de todas las muestras, el valor de corte ((Media de los controles negativos) + (3 x SD de los controles negativos)) también se restó de las intensidades medias de la señal. Las muestras se consideran anti-NP positivas si la intensidad de la señal cae por encima del valor de corte. En la Figura 7 se presenta una imagen de la mancha y el gráfico de intensidad de señal correspondiente. Los resultados demuestran que, utilizando una proteína NP del SARS-CoV-2 producida sin células, la técnica CFDB puede identificar correctamente todas las muestras negativas y todas las muestras positivas, excepto dos muestras del conjunto de RayBiotech (RB P5 recolectado 34 días después del inicio de los síntomas (dpso) y RB P10 recolectado 4 dpso). Una inspección más detallada de la hoja de datos del proveedor confirmó que ambas muestras también parecen negativas para IgG anti-S1RBD (Spike Receptor Binding Domain) por ELISA y anti-N IgG/M por ensayo de flujo lateral (Tabla Suplementaria S3), lo que sugiere una falta de niveles de anticuerpos detectables en estas muestras.

Figura 7: Verificación de CFDB utilizando sueros humanos comerciales precaracterizados. (A) Imagen de transferencia que representa un CFDB NP anti-SARS-CoV-2 en el panel de referencia internacional de la OMS y muestras de suero RayBiotech PosSet. Las muestras se detectaron por triplicado en columna y se identificaron mediante etiquetas. (B) Gráfico que representa los valores cuantificados de intensidad de la señal de quimioluminiscencia para el blot en el panel A. Se utilizó el control negativo para establecer el valor de corte, y todas las mediciones son medias de puntos triplicados ± desviaciones estándar (DE). Esta cifra fue tomada de Norouzi et al.5. Abreviaturas: Neg Cont = muestra de control negativa obtenida mediante la combinación de las 10 muestras anteriores a la COVID-19 de RayBiotech; RB N1 a N10 = Muestras presuntamente negativas de RayBiotech 1 a 10; RB P1 a P20 = RayBiotech presuntamente positivo muestras 1 a 20; CFDB = transferencia de puntos sin células; NP = proteína de la nucleocápside; OMS = Organización Mundial de la Salud. Haga clic aquí para ver una versión más grande de esta figura.
Pruebas independientes del ensayo CFDB con sueros de pacientes humanos
Para demostrar la aplicabilidad del ensayo CFDB, proporcionamos materiales e instrucciones de CFDB a un laboratorio colaborador independiente en una ubicación diferente (Laboratorio Nacional de Microbiología (NML), Winnipeg). El objetivo era probar la plataforma de forma independiente utilizando muestras de pacientes humanos recogidas en la clínica por el grupo de estudios de campo del NML. En total, 24 muestras (x12 negativas y x12 positivas) confirmadas por ELISA para anticuerpos NP (Tabla Suplementaria S4) se sometieron a CFDB y los resultados se analizaron como se ha descrito anteriormente. Como se ve en la Figura 8, CFDB identificó correctamente todas las muestras x12 negativas y x12 positivas, lo que confirma la solidez y practicidad del ensayo en una prueba independiente.

Figura 8: Pruebas independientes de CFDB utilizando sueros de pacientes humanos en el NML. (A) Imagen de transferencia que representa un CFDB NP anti-SARS-CoV-2 en 12 muestras de pacientes COVID-19 negativos (N1 a N12) o 12 positivos (P1 a P12). Los sueros se vieron por triplicados a lo largo de la columna, como se etiquetó. (B) Valores cuantificados de intensidad de la señal de quimioluminiscencia del blot en el panel A. Todas las mediciones son medias de puntos triplicados ± desviaciones estándar (SD). Esta cifra fue tomada de Norouzi et al.5. Abreviaturas: CFDB = transferencia de puntos sin células; NML = laboratorio nacional de microbiología; NP = proteína de la nucleocápside. Haga clic aquí para ver una versión más grande de esta figura.
Pruebas independientes del ensayo CFDB con sueros de hámster infectados con SARS-CoV-2
Utilizando los mismos reactivos e instrucciones de CFDB, el equipo de NML realizó un ensayo de CFDB en sueros de hámster infectados con SARS-CoV-2 para probar si este ensayo también se puede utilizar para detectar la respuesta de anticuerpos en muestras animales. En total, se analizaron 36 muestras, x12 preinfección y 24 sueros postinfección. Los sueros presuntamente positivos procedían de animales infectados con dosis infecciosas de cultivo de tejidos del 10al 5-50 % (TCID50) de SARS-CoV-2 que se muestrearon 140 días después de la infección (dpi 140, n = 3); o reinfectados en DPI 140 y muestreados 5 días después (DPI 5, n = 21). Como se muestra en la Figura 9, todos los sueros previos a la infección tuvieron intensidades de señal por debajo del valor de corte y 18 de los 24 sueros posteriores a la infección (todos desde 5 días después de la reinfección) provocaron intensidades de señal positivas discretas. Curiosamente, de las seis muestras presuntamente positivas que el CFDB pasó por alto, tres muestras (todas del dpi inicial 140) también fueron indetectables utilizando un ELISA anti-NP estándar (Tabla suplementaria S5), lo que indica una pérdida o disminución de los niveles de anticuerpos con el tiempo.
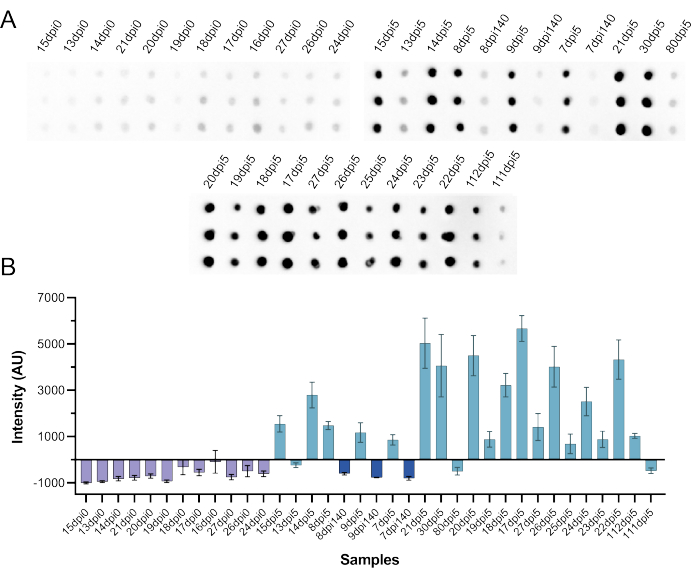
Figura 9: Pruebas independientes de CFDB utilizando sueros de hámster infectados con SARS-CoV-2 en el NML. (A) Imagen de transferencia que presenta un CFDB NP anti-SARS-CoV-2 en sueros de hámster antes de la infección (ppp 0, n = 12), 140 días después de la infección (ppp 140, n = 3) y 5 días después de la reinfección (ppp 5, n = 21) con 105 TCID50 de SARS-CoV-2. Cada muestra se depositó como puntos triplicados en forma de columna y se etiquetó de acuerdo con el esquema del estudio original de NML. (B) Valores cuantificados de intensidad de la señal de quimioluminiscencia a partir de la transferencia en el panel A. Todas las mediciones son medias de puntos triplicados ± desviaciones estándar (DE). Esta cifra fue tomada de Norouzi et al.5. Abreviaturas: CFDB = transferencia de puntos sin células; NML = laboratorio nacional de microbiología; NP = proteína de la nucleocápside; DPI = días después de la infección; TCID50 = 50% de dosis infecciosa de cultivo de tejidos. Haga clic aquí para ver una versión más grande de esta figura.
Archivo complementario 1: Secuencia de nucleótidos para la plantilla de expresión lineal NP His-SpyTag-SARS-CoV-2. Abreviatura: NP = proteína nucleocápside. Haga clic aquí para descargar este archivo.
Archivo suplementario 2: Secuencia completa de nucleótidos del plásmido pET24b SpyCatcher2-Apex2. Haga clic aquí para descargar este archivo.
Archivo complementario 3: Una descripción general de los pasos clave en la preparación de un lisado de expresión de proteínas libres de células de E. coli BL21. Haga clic aquí para descargar este archivo.
Figura suplementaria S1: Mapa de construcción para el plásmido pET24b SpyCatcher2-Apex2. Haga clic aquí para descargar este archivo.
Figura suplementaria S2: Una copia imprimible de la cuadrícula maestra de CFDB. Abreviatura: CFDB = transferencia de puntos sin células. Haga clic aquí para descargar este archivo.
Figura complementaria S3: Una imagen de la membrana CFDB NC y la configuración del ensamblaje de la rejilla maestra. La membrana NC se intercala entre una placa de vidrio en la parte inferior y el patrón de rejilla maestra en la parte superior, asegurando el conjunto con cinta adhesiva. Esta cifra fue tomada de Norouzi et al.5. Abreviaturas: CFDB = transferencia de puntos sin células; NC = nitrocelulosa Haga clic aquí para descargar este archivo.
Tabla suplementaria S1: Recetas completas para los componentes de la solución A y la solución B de la mezcla de reacción libre de celdas. Haga clic aquí para descargar este archivo.
Tabla suplementaria S2: Información de muestra para el Panel de Referencia 20/268 de la OMS (véase también la Figura 7). Abreviaturas: NISBC = Instituto Nacional de Normas y Control Biológicos; OMS = Organización Mundial de la Salud. Haga clic aquí para descargar este archivo.
Tabla complementaria S3: Información de muestra RayBiotech CoV-PosSet (véase también la Figura 7). Haga clic aquí para descargar este archivo.
Tabla suplementaria S4: Resultados de NP ELISA para muestras de suero de pacientes humanos (realizadas en NML). Véase también la figura 8. Abreviaturas: NP = proteína nucleocápside; ELISA = ensayo de inmunoabsorción ligado a enzimas; NML = laboratorio nacional de microbiología. Haga clic aquí para descargar este archivo.
Tabla suplementaria S5: Resultados de NP ELISA para sueros de hámster infectados con SARS-CoV-2 (realizados en NML). Véase también la figura 9. Abreviaturas: NP = proteína nucleocápside; ELISA = ensayo de inmunoabsorción ligado a enzimas; NML = laboratorio nacional de microbiología. Haga clic aquí para descargar este archivo.
Discusión
La COVID-19 ha puesto de manifiesto la importancia de contar con diagnósticos accesibles y sólidos para controlar los brotes de infección y optimizar las estrategias sanitarias mundiales. Las pruebas serológicas que detectan anticuerpos protectores resultaron esenciales para rastrear los patrones de transmisibilidad de las nuevas variantes, identificar los puntos calientes, guiar el desarrollo de vacunas, clasificar los casos sospechosos y proteger a las poblaciones vulnerables14. La pandemia también puso de manifiesto las desigualdades en la accesibilidad de las pruebas, exasperada por los retrasos y los requisitos de instalaciones con equipos sofisticados15. Por lo tanto, la atención se centra ahora en fortalecer nuestra respuesta mundial a futuros brotes, lo que requerirá nuevas innovaciones que reduzcan las barreras de las pruebas y puedan adaptarse rápidamente a amenazas impredecibles.
CFDB es un inmunoensayo en papel que simplifica las pruebas serológicas, realizadas convencionalmente por ELISA o ensayos de flujo lateral, al inmovilizar muestras de pacientes en fase sólida y administrar antígenos producidos sin células en fase móvil para la detección de anticuerpos antivirales5. El ensayo se puede llevar a cabo en entornos de laboratorio básicos con capacidades de Western blot. Mientras tanto, los reactivos para CFDB se pueden producir localmente en entornos de investigación básica e intercambiarse fácilmente para reutilizar el ensayo para nuevos patógenos. El reactivo de detección universal quimérico SpyCatcher2-Apex2 es una característica habilitadora que evita la necesidad de obtener conjugados de anticuerpos secundarios comerciales y se produce de manera eficiente en un sistema de expresión de E. coli . CFDB también proporciona capacidades de alto rendimiento que son paralelas a los ensayos clínicos a una fracción del costo, a solo $ 3 USD por kit de ensayo en comparación con aproximadamente $ 300 USD para un ELISA comercial de 96 pocillos.
La configuración de la CFDB para las pruebas serológicas de COVID-19 se logró mediante el diseño de plantillas de ADN lineal que instruyeron al sistema de transcripción-traducción libre de células para producir el antígeno objetivo, el SARS-CoV-2 NP. A continuación, se detectaron sueros humanos y animales en rejillas de membrana de nitrocelulosa y se realizaron incubaciones con la mezcla de expresión de antígeno libre de células seguida de la proteína reportera SpyCatcher2-Apex2. En este estudio, la CFDB se realizó en sueros de sujetos negativos y positivos para COVID-19, tanto en nuestro laboratorio como en un entorno independiente (NML), y en general, se logró una buena correlación con ELISA para distinguir las muestras positivas de las negativas. Mediante el uso de sueros sanos agrupados como control negativo CFDB y el cálculo de un valor de corte del ensayo basado en esta muestra, se minimizó el riesgo de identificación de falsos negativos/positivos.
La alta sensibilidad del ensayo CFDB se manifiesta por su capacidad para distinguir claramente todas las muestras en el Panel de Referencia de la OMS 20/26813 del NIBSC ampliamente precaracterizado, particularmente la muestra con el título de anticuerpos más bajo (Muestra 20/140 bajo, ver Tabla Suplementaria S2 y Figura 7). Del mismo modo, la alta especificidad de CFDB queda demostrada por la falta de identificación de falsos positivos para ninguno de los 35 sueros sanos analizados en este trabajo. Es importante destacar que la validación clínica en múltiples sitios confirmó que el protocolo CFDB es lo suficientemente sencillo para su uso generalizado y mantiene un alto rendimiento, comparable a los ELISA estándar, cuando es ejecutado por diferentes usuarios. En nuestra experiencia y como lo han demostrado otros16, los antígenos libres de células producidos en los lisados de E. coli son estables durante al menos 3 meses a -20 °C, y las preparaciones de lotes de múltiples antígenos libres de células se comportan de manera indistinguible entre sí.
Se deben tener en cuenta varias consideraciones antes de adoptar ampliamente la plataforma CFDB y orientar el diseño del sistema hacia nuevos patógenos. La plataforma CFDB requiere que el antígeno viral o el fragmento de antígeno se sintetice correctamente mediante un sistema de expresión libre de células para que las partes antigénicas estén presentes y disponibles para la unión de anticuerpos. Por ejemplo, inicialmente teníamos la intención de incluir también el dominio de unión al receptor de pico (RBD) del SARS-CoV-2 en nuestros ensayos de CFDB. Sin embargo, a pesar de una expresión de alto rendimiento y debido a su requerimiento de enlaces disulfuro complejos (a diferencia del antígeno NP que carece de aminoácidos cisteína), el producto RBD se agregó fácilmente y parecía insoluble en el extracto de E. coli , lo que impidió su uso como una sonda CFDB confiable. En tales escenarios donde se requieren modificaciones postraduccionales complejas para un plegamiento adecuado y una función antigénica, se recomienda el uso de sistemas libres de células basados en eucariotas. Al igual que con otros inmunoensayos basados en peroxidasa, también es imperativo que se excluyan las muestras de suero con hemólisis visible debido a la posibilidad de que la actividad de la hemoglobina peroxidasa confunda los resultados. Por último, el CFDB está diseñado para detectar la respuesta total de anticuerpos y no informa sobre la clase específica de inmunoglobulina implicada en las interacciones anticuerpo-antígeno.
En conclusión, el CFDB es una nueva técnica diagnóstica que combina elementos de la biología sintética libre de células y la transferencia de puntos para agilizar las pruebas serológicas. El CFDB ofrece una alternativa rentable a los ELISA convencionales que puede desplegarse fácilmente en los laboratorios de investigación de todo el mundo cuando se necesita capacidad de prueba adicional, como suele ser el caso durante una pandemia. Por lo tanto, el CFDB puede servir como una herramienta de primera línea en la próxima respuesta mundial a la pandemia, que se puede adaptar rápidamente a las amenazas emergentes y fortalecerá las capacidades de prueba de los países de ingresos bajos y medianos.
Divulgaciones
M.N. y K.P. son coinventores del método de transferencia de puntos sin células. Se ha presentado una solicitud provisional de patente relacionada con esta obra (PCT/CA2024/050097, presentada en enero de 2024).
Agradecimientos
S.S. y R.Z. cuentan con el apoyo financiero de la Agencia de Proyectos de Investigación Avanzada de Defensa (DARPA), Contrato No. N66001-23-2-4042. Los puntos de vista, opiniones y/o hallazgos expresados son los de los autores y no deben interpretarse como representativos de los puntos de vista o políticas oficiales del Departamento de Defensa o del Gobierno de los EE. UU. Este trabajo fue apoyado por fondos para K.P. del programa de subvenciones de la Fundación CIHR (201610FDN-375469), el Programa de Cátedras de Investigación de CIHR Canadá (950-231075 y 950-233107), la Iniciativa de Medicina por Diseño de la Universidad de Toronto, que recibe fondos del Fondo de Excelencia en Investigación de Canadá y fondos para K.P., del Programa Canadiense de Seguridad y Protección de Investigación y Desarrollo de Defensa de Canadá (contrato 39903-200137). La Figura 1 y la Figura Complementaria S1 se crearon utilizando SnapGene Viewer.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 kb DNA ladder | NEB | N3232 | Used as a size marker for agarose gels |
| 20 Amino acids | Sigma-Aldrich | LAA21-1KT | A component of the cell-free reaction Solution B |
| 2 mm biopsy punch | Integra Miltex | 33-31-P/25 | For preparation of CFDB master grid |
| 5-aminolevulinic acid hydrochloride | Sigma-Aldrich | A3785 | Heme precursor for induction of SpyCatcher2-Apex2 |
| Agarose powder | BioShop | AGA002 | For electrophoretic analysis of DNA |
| Anti-SARS-CoV2-Nucleocapsid antibody | Sinobiological | 40588-T62 | For WB detection of cell-free produced NP antigen |
| Bio-Rad ChemiDoc XRS+ | Bio-Rad | N/A | Gel imager instrument |
| Centrifugal concentrators | Cytiva | 28-9323-60 | For concentration and buffer exchange of proteins |
| Centrifuge | Eppendorf | EP022628257 | For harvesting bacterial culture |
| Coenzyme A sodium salt hydrate (CoA) | Sigma-Aldrich | C3144 | A component of the cell-free reaction Solution A |
| Color pre-stained protein standard (broad range) | NEB | P7719 | Used as a size marker for SDS-PAGE gels |
| Covid-19 serum panel | RayBiotech | CoV-PosSet | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| D-(−)-3-Phosphoglyceric acid disodium salt (3-PGA) | Sigma-Aldrich | P8877 | A component of the cell-free reaction Solution B |
| Dithiothreitol (DTT) | Sigma-Aldrich | 10197777001 | Component of buffers |
| E. coli 5-alpha | NEB | C2987 | for plasmid preparation |
| E. coli BL21 | NEB | C2530 | For preparation of cell-free lysates |
| E. coli BL21 (DE3) | NEB | C2527 | For expression of SpyCatcher2-Apex2 |
| EDTA-free protease inhibitor tablet | Sigma-Aldrich | 11836153001 | A component of E. coli lysis buffer |
| Eppendorf New Brunswick Innova 43/43R Incubator Shaker | Eppendorf | EPM1320 | Incubator for growing E. coli cells for protein expression and cell-free lysate preparation |
| Folinic acid | Sigma-Aldrich | 47612 | A component of the cell-free reaction Solution A |
| Glycerol | Sigma-Aldrich | G9012 | Component of SpyCatcher2-Apex2 storage buffer |
| Hemin chloride | Sigma-Aldrich | H9039 | For Heme supplementation of SpyCatcher2-Apex2 |
| Hydrogen peroxide 30% | Sigma-Aldrich | H1009 | For making ECL reagent |
| Image Lab Software | Bio-Rad | 1709690 | Software for ChemiDoc gel imaging instrument |
| Isopropyl-b-D-1-thiogalactopyranoside | Bioshop | IPT001 | For induction of SpyCatcher2-Apex2 expressikon |
| Kanamycin Sulfate | Sigma-Aldrich | 60615 | For preparation of SpyCatcher2-Apex2 bacterial culture |
| LB agar | BioShop | LBL406 | For E. coli growth |
| LB broth | BioShop | LBL407 | For E. coli growth |
| luminol | Sigma-Aldrich | A4685 | For making ECL reagent |
| Lysozyme | Sigma-Aldrich | L6876 | For lysis of bacterial cells |
| Magnesium Acetate | Sigma-Aldrich | M5661 | A component of the cell-free reaction Solution B |
| NEBExpress Ni resin | NEB | S1428S | For purification of SpyCatcher2-Apex2 |
| Non-fat dry milk | Bioshop | SKI400 | For blocking of WB and CFDB membranes |
| Parafilm | Bemis | 2099-1337410 | For ECL-incubation of CFDB blots |
| p-coumaric acid | Sigma-Aldrich | C9008 | For making ECL reagent |
| pET24b-SpyCatcher2-Apex2 plasmid | Pardee Laboratory | N/A | Used for the expression of SpyCatcher2-Apex2 protein |
| Petri dish | Fisherbrand | FB0875712 | Container for WB and CFDB membrane incubation |
| Phosphate buffered saline 10% | BioShop | PBS405 | Component of buffers |
| Potassium Glutamate | Sigma-Aldrich | G1501 | A component of the cell-free reaction Solution B |
| Potassium Oxalate Monohydrate (Oxalic acid) | Sigma-Aldrich | 223425 | A component of the cell-free reaction Solution A |
| Precast SDS-PAGE gel | BioRad | 4561036EDU | For electrophoretic analysis of protein |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification of LETs |
| QIAquick PCR purification kit | Qiagen | 28106 | For purification of LETs |
| Ribonucleotide Solution Set | NEB | N0450S | A component of the cell-free reaction Solution A |
| Skim milk powder | BioShop | SKI400 | Used as a blocking agent for western blot and CFDB |
| Sodium Chloride | Sigma-Aldrich | S9625 | A component of protein purification buffers |
| Sonicator | Qsonica | Q500 | For lysis of bacterial cells |
| Spermidine | Sigma-Aldrich | S2626 | A component of the cell-free reaction Solution A |
| Syringe filter | Sigma-Aldrich | SLGSR33SS | For sterilisation of buffers and solutions |
| Thermocycler | BioRad | T100 | Instrument for incubating PCR reactions |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8759 | A component of the cell-free reaction Solution A |
| Tris buffered saline | Thermo Scientific | J60764.K2 | Component of WB and CFDB wash buffer |
| Trizma base | Sigma-Aldrich | T1503 | For making tris buffer |
| Tunair SS-5012 Half-Baffle Shake Flask, 2.5 L | Cole-Parmer | RK-01835-39 | Vessel for growing E. coli cells for protein expression and cell-free lysate preparation |
| Tween-20 | BioShop | TWN510 | Component of WB and CFDB wash buffer |
| Tweezer | Almedic | 7728-A10-100 | For handling CFDB NC membrane |
| UV-Vis spectrophotometer | Thermo Scientific | 13400518 | Used for the measurement of nucleic acid and DNA concentration and bacterial culture OD |
| WHO International Reference Panel for anti-SARS-CoV-2 immunoglubulin | NISBC | 20/268 | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| β-Nicotinamide adenine dinucleotide hydrate (NAD) | Sigma-Aldrich | 10127965001 | A component of the cell-free reaction Solution A |
Referencias
- Carter, L. J., et al. Assay techniques and test development for COVID-19 diagnosis. ACS Cent Sci. 6, 591-605 (2020).
- Amanat, F., et al. A serological assay to detect SARS-CoV-2 seroconversion in humans. Nat Med. 26 (7), 1033-1036 (2020).
- Stadlbauer, D., et al. SARS-CoV-2 seroconversion in humans: A detailed protocol for a serological assay, antigen production, and test setup. Curr Protoc Microbiol. 57 (1), e100(2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259.e12 (2016).
- Norouzi, M., et al. Cell-free dot blot: an ultra-low-cost and practical immunoassay platform for detection of anti-SARS-CoV-2 antibodies in human and animal sera. Microbiol Spectr. 11 (2), e0245722(2023).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Norouzi, M., Panfilov, S., Pardee, K. High-efficiency protection of linear DNA in cell-free extracts from Escherichia coli and Vibrio natriegens. ACS Synth Biol. 10 (7), 1615-1624 (2021).
- IDT codon optimization tool. , Integrated DNA Technologies. https://www.idtdna.com/pages/tools/codon-optimization-tool (2025).
- General protocol for western blotting. , Bio-Rad. https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6376.pdf (2025).
- Mruk, D. D., Cheng, C. Y. Enhanced chemiluminescence (ECL) for routine immunoblotting. Spermatogenesis. 1, 121-122 (2011).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-based cell-free protein synthesis: Protocols for a robust, flexible, and accessible platform technology. J Vis Exp. (144), (2019).
- Plasma and serum preparation. , ThermoFisher Scientific. https://www.thermofisher.com/ca/en/home/references/protocols/cell-and-tissue-analysis/elisa-protocol/elisa-sample-preparation-protocols/plasma-and-serum-preparation.html (2025).
- Mattiuzzo, G., et al. WHO/BS.2020.2403 Establishment of the WHO International Standard and Reference Panel for anti-SARS-CoV-2 antibody. , (2020).
- Gong, F., Wei, H., Li, Q., Liu, L., Li, B. Evaluation and comparison of serological methods for COVID-19 diagnosis. Front Mol Biosci. 8, 682405(2021).
- Peeling, R. W., et al. Serology testing in the COVID-19 pandemic response. Lancet Infect Dis. 20 (9), e245-e249 (2020).
- Smith, M. T., Berkheimer, S. D., Werner, C. J., Bundy, B. C. Lyophilized Escherichia coli-based cell-free systems for robust, high-density, long-term storage. Biotechniques. 56 (4), 186-193 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados