Method Article
ヒトおよび動物の血清中の抗体応答を検出するための実用的で適応性の高いイムノアッセイプラットフォームとしての無細胞ドットブロット
要約
ここでは、無細胞合成生物学の原理と、ヒトおよび動物の血清における抗体応答のカスタマイズ可能な検出のためのドットブロット技術に基づいて最近開発されたイムノアッセイプラットフォームについて説明します。
要約
過去20年間にわたる一連の世界的な病原性アウトブレイクは、血清監視戦略の重要性を浮き彫りにしています。患者の血清中の疾患特異的抗体を検出するのに役立つイムノアッセイプラットフォームは、血清サーベイランスの中核をなすものです。一般的な例としては、酵素結合免疫吸着アッセイやラテラルフローアッセイなどがあります。しかし、これらはゴールドスタンダードの方法ですが、病原体に特化した消耗品や特殊な機器が必要なため、リソースの豊富な研究所以外での使用は限られています。
私たちは最近、Cell-Free Dot-Blot (CFDB) と呼ばれる新しい免疫測定プラットフォームを開発し、SARS-CoV-2 に対するヒトおよび動物の血清を使用して検証しました。従来のイムノアッセイとは異なり、CFDB患者の血清サンプルは固相(ニトロセルロース膜)に固定化され、標的抗原はアッセイの移動相に懸濁されます。セロサーベイランス機能へのアクセスを改善するために、CFDB抗原はオンデマンドで、 in vitro タンパク質発現を使用して低負荷のインフラストラクチャで産生されます。ここでは、抗原はペプチドタグと融合しており、任意のCFDBアッセイで単一のユニバーサルレポータータンパク質を使用して検出できます。その結果、CFDBはマルチウェルプレートリーダーや精製された市販の分子アッセイコンポーネントにアクセスする必要がありません。これらの設計上の考慮事項により、CFDBは、非中央集権的な研究所へのアクセス性、新興病原体への適応性、および低所得コミュニティ向けの手頃な価格を提供することにより、既存の免疫測定プラットフォームの欠点に対処します。
本稿では、CFDBイムノアッセイを調製および実施するためのステップバイステップのプロトコルを提供します。SARS-CoV-2 CFDBに関する最近の研究を例にとり、オンデマンドの無細胞生産のための抗原DNA設計、CFDBレポータータンパク質の調製、固相への血清サンプルの固定化、そして最後にアッセイの抗原結合と検出ステップについて説明します。これらの指示に従うことで、研究者はCFDBアッセイを適応させて、任意の病原体に対するヒトおよび動物の血清中の免疫応答を検出できると期待しています。
概要
COVID-19のパンデミックにより、特にリソースの少ない環境に対して、手頃な価格でスケーラブルな診断ツールが不可欠であることが明らかになりました1。酵素結合免疫吸着アッセイ(ELISA)のような従来の免疫アッセイは、免疫応答の検出に不可欠であることが証明されています2,3。しかし、その高コスト、複雑な試薬への依存、特殊な機器への依存は、特に世界的な健康危機の際には、その入手を制限します。これらの課題に対応するため、ヒトおよび動物血清中の抗SARS-CoV-2抗体を検出するために設計された低コストで適応性の高い免疫測定プラットフォームであるCell-Free Dot Blot(CFDB)を開発しました。
CFDBは、無細胞合成生物学を活用して、直鎖状DNAテンプレート4,5を用いたウイルス抗原の迅速かつオンデマンドな生産を行っています。これにより、従来の細胞ベースのクローニング、発現、精製プロセスが不要になり、抗原産生が大幅にスピードアップし、コストが削減されます。CFDB法では、血清をニトロセルロースメンブレンに直接スポットするドットブロットフォーマットを使用することで、抗体の検出を簡素化します。このシステムにより、高価なマルチウェルプレートや特殊なラボ機器が不要になり、インキュベーションと洗浄のステップにおけるシンプルな「ディッピング」ワークフローが可能になります。このプラットフォームは、SpyCatcher-SpyTagシステムも利用しており、SpyCatcher2-Apex2ペルオキシダーゼキメラが汎用的な二次検出試薬として作用します5,6。これは、標準的な大腸菌ベースの発現を使用して製造されるため、高価な市販の抗体コンジュゲートへの依存が排除されます。その結果、CFDBシステムは、市販のELISAキット5の300米ドル以上と比較して、96サンプルアッセイあたり約3米ドルという大幅に低いコストで、ELISAに匹敵する性能で血清学的アッセイを実施できます。
CFDBの有効性を実証するために、事前に特徴付けられたヒトおよび動物の血清中の抗体を検出する能力をテストしました。私たちの結果は、COVID-19陽性および陰性のサンプルを特定する際にELISAと密接に関連していました。ヒトの診断に加えて、SARS-CoV-2に感染したハムスターと組換えヌクレオカプシドタンパク質をワクチン接種したハムスターの血清を試験し、動物モデルにおけるCFDBの有用性を評価しました。これらの試験により、CFDBがヒトと獣医の両方の診断に使用できる可能性が確認され、種を超えた免疫応答をモニタリングするための汎用性の高いツールとなりました。CFDBの主な利点の1つは、その柔軟性です。目的の抗原をコードするDNAテンプレートを変更するだけで、このプラットフォームはさまざまな病原体に対する抗体を検出するために迅速に適応させることができ、将来のパンデミックへの備えに役立ちます。その低コスト、シンプルなワークフロー、最小限のインフラストラクチャ要件により、商用診断へのアクセスが制限されている分散型の研究所やリソースの少ない環境に特に適しています。
この作業では、CFDBアッセイの準備と実施について、段階的に説明します。まず、アッセイの主要な検出試薬である抗原を無細胞で生産するための線状DNAテンプレートの設計と合成について説明します。次に、アッセイの二次検出試薬SpyCatcher2-Apex2の調製手順について説明します。その後、無細胞生産の指示と抗原自体の品質チェックを行います。最後に、ヒトまたは動物の血清サンプルでCFDBアッセイを実施するプロセスについて詳しく説明します。
プロトコル
すべてのハムスターの実験は、カナダ公衆衛生庁の国立微生物学研究所(NML)で行われ、カナダ人間動物衛生科学センターによって承認され、カナダ動物ケア評議会のガイドラインに従って行われました。すべてのヒト血清/血漿サンプルは、社内試験のために商業的に取得されたか、または臨床協力者によってNMLに提供され、NMLでの独立した試験が行われました。
1. 抗原線状発現テンプレート(LET)の設計と調製
- Norouzi et al.7 の指示に基づき、Norouzi et al.5 に記載されている N/C 末端 SpyTag を含む標的抗原の無細胞発現 LET を設計します。
注:ここでは、SARS-CoV-2ヌクレオカプシドタンパク質(NP、アミノ酸2-419)の調製について詳しく説明します。- SARS-CoV-2 NP の LET (図 1 および 補足ファイル 1) に、T7 プロモーターの前と終止コドンの後に 50 塩基対のバッファー配列を持つ 5' および 3' Ter サイトが含まれていることを確認します。
- N末端のNPタンパク質にHis 6-SpyTag-TEVをタグします。
- UniProtデータベース(アクセッションコードP0DTC9)からタンパク質配列を取得し、IDTコドン最適化ツール8を使用して大腸菌ベースの発現用にコドン最適化します。この配列を、10 ng/μLの水に再懸濁した一本鎖DNAフラグメントとして商業合成のためにご注文ください。
注:TEVプロテアーゼ切断部位とHis6 タグは、LET設計の重要な機能ではありません。
- PCRは、ユニバーサルTer FW(GGCTCCGAATAAGTATGTTGTAACTAAAGTGCGGCC)でDNA断片を増幅します。
ACGATGCGTCCGGCGTAGAGGATCG) と Ter RV (CCGAGGCAAGTATATGTTGTAACTAAAGTGCTCAG
CTTCCTTTCGGGCTTTTTGTTAGCAGCC)プライマーをハイフィデリティDNAポリメラーゼキット( 材料表を参照)を使用- 100 μL の反応液を次のように設定します: ヌクレアーゼフリー水 75 μL、5x DNA ポリメラーゼバッファー 20 μL、10 mM dNTP 2 μL (最終濃度 200 μM)、0.5 μL 100 μM Ter-FW プライマー、0.5 μL 100 μM Ter-RV プライマー、10 ng/μL 一本鎖 LET 1 μL、1 μL(2U) DNA ポリメラーゼ。
- 以下のPCR設定を使用してください:98°Cで30秒間の初期変性。35サイクル:98°Cで6秒、60°Cで15秒、72°Cで90秒。4°Cで保持します。
- 市販のPCR精製キットを使用してPCR産物を精製し、1%アガロースゲルでサンプルを泳動して品質を確認し、UV-Vis分光光度計でその濃度を測定します。
注:粗PCR産物は、無細胞発現に直接使用することもできます。しかし、精製により、より標準化された手順が可能になります。

図1:SARS-CoV-2-NPの線形発現テンプレート。 His-SpyTag-SARS-CoV-2 NP直鎖状DNAテンプレートの特徴を表す概略図。主要なDNAテンプレートエレメントがラベル付けされています。目的のタンパク質(ここではNP)のコード配列は、効率的な発現のためにT7プロモーターの転写制御下に置かれます。N末端で、NPタンパク質にSpyTagを付加し、SpyCatcher2-Apex2検出試薬を使用して特異的に検出します。x6His-tagおよびTEVプロテアーゼ部位は、一般的なLET設計の一部として含まれていますが、CFDBの目的には不要です。直鎖状DNAテンプレートの末端には、それぞれ50の塩基対バッファー配列が先行する「上流」および「下流」のTerサイトが含まれており、無細胞ライセート中の外核分解性DNA分解に対するTusを介した保護のために含まれています。略語:NP =ヌクレオカプシドタンパク質;LET = 線形式テンプレート;TEV = タバコエッチングウイルス;CFDB = 無細胞ドットブロット。 この図の拡大版を表示するには、ここをクリックしてください。
2. SpyCatcher2-Apex2レポータータンパク質の精製
- Norouzi et al.5 で最初に構築されたpET24b-SpyCatcher2-Apex2プラスミド( Supplemental File 2 の全配列および Supplemental Figure S1のプラスミドマップ)を使用して、SpyCatcher2-Apex-2レポータータンパク質を調製します。
- 寒天プレートと50 μg/mLのカナマイシンを含む溶解ブロス(LB)を調製します。 大腸菌 BL21(DE3)細胞をpET24b-SpyCatcher2-Apex2プラスミドで形質転換します。15 mLのスターターLB培養物に1つのコロニーを接種し、250 RPMで振とうしながら37°Cで一晩増殖します。
- 翌日、50 μg/mL カナマイシンを含む新鮮な LB 培地 500 mL に 10 mL のスターター培養液を加えます。250 RPMで振とうしながら37°Cでインキュベートし、培養物が600 nmで0.6-0.8(~3時間)の光学密度に達するまでインキュベートします。
- 0.5 mM イソプロピル-β-D-1-チオガラクトピラノシドと 1 mM 5-アミノレブリン酸塩酸塩を培養液に添加することにより、SpyCatcher2-Apex2 の発現を誘導します。増殖温度を30°Cに下げ、培養物をさらに4時間インキュベートします。
- 8,000 × g で15分間遠心分離することにより、細菌を回収します。細胞溶解に進むか、または使用するまでペレットを-80°Cで保存します。
- 50 mM Tris-HCl(pH 7.8)、300 mM NaCl、1 mg/mLリゾチーム、EDTAフリープロテアーゼ阻害剤錠剤、および1 mMジチオスレイトール(DTT)を含む20 mLの溶解緩衝液にペレットを再懸濁します。50 秒の振幅で超音波処理を行い、5 秒の ON および 10 秒の OFF 間隔で細胞を溶解し、合計 ON 時間 3 分にします。
- ライセートを20,000 × g 、4°Cで1時間遠心分離して清澄化します。 上清を0.2μmシリンジフィルターに通します。
- 清澄化したライセートに最終濃度250 μMの塩化ヘミンを添加し、4 °Cで一晩インキュベートします。 タンパク質精製に進みます。
注:ヘミンクロリドとのインキュベーションにより、ヘムがApex-2ペルオキシダーゼに最大化され、最適な酵素活性が得られます。 - 清澄化したライセートに2.5 mLのNiレジンを加え、4°Cで45分間穏やかに振とうしながらインキュベートします。混合物を重力フローカラムに塗布し、50 mLのTrisバッファー(50 mM Tris-HCl(pH 7.8)、300 mM NaCl、および1 mM DTT)でレジンを洗浄します。
- SpyCatcher2-Apex2タンパク質を、400 mMイミダゾールを含む25 mLのTrisバッファーで溶出します。濃縮液とバッファーは、遠心フィルターユニットを使用して溶出液をトリスバッファーに交換し、最終容量0.5〜1.0 mLを目指します。
- SpyCatcher2-Apex2 (27,390 M-1 cm-1) のモル吸光係数を使用して、UV-Vis 分光光度計でタンパク質濃度を決定します。グリセロールを最終濃度40%まで添加し、アリコートを-20°Cで保存します。
注:このプロトコルでは、標準的な96サンプルブロットサイズで400 CFDBの実行に十分な、約40 mgの高純度で活性なSpyCatcher2-Apex2が得られることが期待されます。 - 従来のウェスタンブロット9 を使用したSpyTaggedタンパク質の検出、およびCFDB実験では、0.05% Tween-20(TBST)を含む1x Tris緩衝生理食塩水中の5%無脂粉乳中のメンブレンを常にブロックしてください。SpyCatcher2-Apex2タンパク質を最終濃度10 μg/mLのブロッキング溶液で使用してください。
注:SpyCatcher2-Apex2レポーターは、シグナル開発のために強化された化学発光(ECL)ソリューションを必要とします。ECL溶液は、市販して入手することも、Mruk et al.10の指示に従って社内で調製することもできます。ここでの最終的なECL溶液は、0.4 mM p-クマル酸、2.5 mMルミノール、および0.015% H2O2 in 100 mM Tris-HCl(pH 8.6)で構成されています。
3. 無細胞での抗原生産と品質確認
- Levine et al.11およびNorouzi et al.5の指示に従って、大腸菌BL21の無細胞発現ライセートおよび反応成分を調製し、組み立てます。代表的な100 μLの反応には、最終反応混合物に5 μM Tusタンパク質と1.2 μM T7 RNAポリメラーゼを次のように添加します:14.6 μLの溶液A、14 μLの溶液B、33.3 μLの大腸菌BL21ライセート、2.5 μLの200 mM Tusタンパク質、1.2 μLの100 mM T7 RNAポリメラーゼ、 1 μL の 1.5 μM 直鎖状 DNA テンプレート、33.4 μL のヌクレアーゼフリー水。
注:大腸菌BL21無細胞溶解物の調製に関する主要な指示については補足ファイル3を、溶液AおよびBの詳細なレシピについては補足表S1を参照してください。 - PCRチューブ内の無細胞反応に10%(v/v)粗PCR産物を添加することにより、最初の5 μLスケールの発現試験を実施します。30°Cで15時間振とうせずにインキュベートします。
- 12%ドデシル硫酸ナトリウムポリアクリルアミドゲル(SDS-PAGE)に1 μLの無細胞反応をロードし、その後、ステップ2.12で説明したように、ウェスタンブロット用のニトロセルロースメンブレンに移して、NP抗原の発現品質を確認します。SpyTagによるブロット標識にはSpyCatcher2-Apex2レポータータンパク質を使用し、オプションで標的抗原に対する特異的な市販抗体(ここでは抗SARS-CoV-2-NP)を使用します。
- 1 mLスケールの無細胞反応を15 nMの精製LET生成物とアセンブルし、CFDBアッセイに使用します。発現混合物を15 mLのコニカルチューブ内で、80 RPMで30°C、15時間振とうしながらインキュベートします。 ウェスタンブロットを使用して発現品質を確認し、50 μLアリコートを-20°Cで保存します。
4. 血清サンプル
- ヒト/動物の血清(または血漿)サンプルは、標準的な手順を使用し、機関のヒト研究倫理委員会または動物管理および使用委員会のガイドラインに従って、商業供給源または臨床協力者から、または社内で入手します。汚染のリスクを軽減するために、適切なサンプル前処理対策が講じられていることを確認してください。
注:前処理手段には、溶剤洗剤処理、熱不活化、および血液媒介ウイルスマーカーの試験が含まれる場合があります。全血からの血清および血漿の単離のための有用なプロトコルが利用可能である12。他のイムノアッセイプラットフォームと同様に、最初の感染または疾患発症の早い時期(<3週間)に採取された血清サンプルには、適切なレベルの抗体反応が含まれているとは予想されず、その結果は慎重に扱う必要があります。 - SARS-CoV-2 NP CFDB試薬の品質検証には、米国国立生物標準管理研究所(NIBSC)の世界保健機関(WHO)の抗SARS-CoV-2免疫グロブリンに関する国際リファレンスパネル(COVID-19前x1および抗NP免疫グロブリンのレベルが異なるx4のSARS-CoV-2陽性サンプルを含む)を使用してください13。あるいは、他の供給源から得られた、事前に特徴付けられた健康で陽性の血清サンプルを使用します。
- CFDB実験では、複数の(>3)健康な血清サンプルをプールしてネガティブコントロールサンプルを取得または調製し、精度を高め、結果の解析と解釈を容易にします。
5. 無細胞ドットブロット(CFDB)法
- マスターグリッドイメージファイル(補足図S2)をダウンロードして印刷します。マスターグリッドは、直径2mmの円を12個ずつ含む6 x 6 cmのパターンで、96ウェルプレートと同様のスポッティング能力を提供します。
- グリッドを接着性PCRプレートシーリングフィルムの2つの層の間にしっかりと挟み、外側のグリッド境界に沿ってサイズにカットします。2 mmの生検パンチを使用して、マークされた各円をくり抜きます。
注:このマスターグリッドは、70%エタノールで拭いた後、複数回再利用できます。 - 6.5 x 6.5 cmのニトロセルロースメンブレンをカットし、マスターグリッドの下のきれいな表面に配置し、図2および補足図S3に示すように粘着テープでセットアップを固定します。マーカーペンを使用して、サンプルスポッティング後にメンブレンを切断するためのガイドとして使用するニトロセルロースメンブレン上の最も外側の円の位置をマークします。
- 血清サンプルを1倍リン酸緩衝生理食塩水(pH 7.4)で1/10に希釈します。マイクロピペットを使用して、各サンプルの0.4 μLの容量を3倍にして、所定のグリッド位置でニトロセルロースメンブレンに分注します。ディスペンスするには、スポットあたり約15秒かかります。
注:各アッセイには、結果の分析に必要となる陰性および陽性のコントロールサンプルの両方を必ず含めてください。 - スポットされたサンプルが結合して乾燥するまで、周囲温度で10分間待ちます。ピンセットを使用して、ニトロセルロース膜を慎重に取り出し、マークされた外側の円に沿って切断します。
- 10 cmのシャーレに10 mLのブロッキング溶液(TBST中の5%脱脂粉乳)でメンブレンを10 mのシャーレで室温で30分間ブロックし、100 RPMで穏やかに振とうします。
- 10 cmのシャーレで、無細胞抗原発現混合物の50 μLアリコートを解凍し、5 mLのブロッキング溶液に加えます。メンブレンをこの抗原含有溶液に直接移し、室温で1時間インキュベートし、100 RPMで振とうします。
注:これは、SpyTagged抗原が抗体含有スポット位置(存在する場合)に結合する主要な検出ステップになります。 - メンブレンをすすぎ、5分間洗浄した後、TBSTで再度すすぎてから、二次検出ステップに進みます。
- 10 μg/mLの精製SpyCatcher2-Apex2タンパク質を含むブロッキングバッファー10 mLでメンブレンを100 RPMで振とうしながら1時間インキュベートします。
- メンブレンをすすぎ、TBSTで2 x 5分間洗浄し、最後にTBSですすぎます。
- パラフィルムをブロットイメージング装置の近くの清潔な作業領域に貼り付けて固定します。ピンセットでメンブレンを軽くたたいて余分な液体を取り除き、パラフィルムの上にメンブレンを置きます。
- 直ちに3 mL(100 μL/cm2)のECL溶液をメンブレンの上に加え、室温で正確に90秒間インキュベートします。
- すぐにメンブレンを直火乾燥し、化学発光適合イメージング装置に移して結果を視覚化します。
注:最適なイメージング時間は、機器によって異なる場合があります。装置のデフォルトの自動取得時間設定を使用し、設定されていない場合は、事前に特性化された血清サンプルを使用して画像取得時間を最適化することをお勧めします。 - 装置の画像解析機能を使用して、ニトロセルロースメンブレン上の3つのブランク(バックグラウンド)位置を含むスポット強度を取得します。スポットあたりの測定量を一定に保ちます。データをスプレッドシートにエクスポートし、各スポット位置に正しくラベルを付けることを忘れないでください。
- 3重のスポット強度の平均と標準偏差(SD)を計算します。次に、すべてのサンプルからニトロセルロース膜のバックグラウンドを差し引きます。
- 次の式を使用して、-/+ 結果の解釈のカットオフ値を取得します。
(ネガティブコントロールの平均)+(ネガティブコントロールの3 x SD)。サンプルの平均強度がカットオフ値を上回っている場合はサンプルを正とし、平均強度がカットオフ値を下回っている場合は負と見なします。
注 : CFDB の手順の概略図を 図 3 に示します。
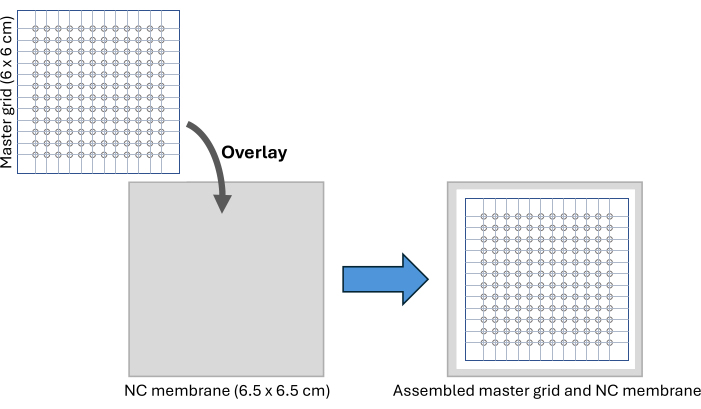
図2:CFDBアセンブリ。 CFDBマスターグリッドとNCメンブレンアセンブリのセットアップの概略図。マスターグリッドはNCメンブレンに重ねられ、血清サンプルのスポッティングと固定化のための規則的でアドレス指定可能なパターンを提供します。略語:CFDB =無細胞ドットブロット;NC =ニトロセルロース。 この図の拡大版を表示するには、ここをクリックしてください。

図3:CFDBワークフローの概略図。 CFDBアッセイでは、少量(<0.4 μL)の10倍希釈血清サンプルを、プレカットされたニトロセルロースメンブレン(左パネル)の個別のアドレス指定可能な場所(中央パネル)に手動で分注します。1スポットにつき1つの血清サンプルを三重のスポットに沈着させ、血清の総抗体リザーバーを含むタンパク質含有量を固体NC基質(中央パネルのベージュのスポット)に固定化します。この例では、血清サンプルに含まれる抗NP抗体は、最初にCFDB一次検出試薬SpyTag-NPによって結合され、最後にCFDB二次検出試薬SpyCatcher2-Apex2(右パネル拡大バブル)によって検出されます。この図は、Norouzi et al.5から取得されました。略語:CFDB =無細胞ドットブロット;NP =ヌクレオカプシドタンパク質;LET = 線形表現テンプレート。 この図の拡大版を表示するには、ここをクリックしてください。
結果
標的抗原の直鎖状発現テンプレートのPCR増幅
SARS-CoV-2 NP LETをPCR増幅するために、プロトコルセクション1.2に記載されているようにユニバーサルTerフォワードプライマーとリバースプライマーを使用し、PCR産物の精製に進む前に、製品の1μLをアガロースゲル(図4)でチェックしました。

図4:線状発現テンプレートのPCR。 Lane 1:1 kb DNA LadderおよびLane 2:His-SpyTag-SARS-CoV-2 NP(1,630 bp)のPCR増幅直鎖状DNAテンプレートを示す1%アガロースゲル。略語:PCR =ポリメラーゼ連鎖反応;NP =ヌクレオカプシドタンパク質。 この図の拡大版を表示するには、ここをクリックしてください。
SpyCatcher2-Apex2レポータータンパク質の精製
SpyCatcher2-Apex2タンパク質は、プロトコールセクション2に記載されているように調製しました。Apex2ペルオキシダーゼへのヘムの適切な取り込みを確実にするために、5-アミノレブリン酸塩酸塩(ヘム前駆体)とヘミンクロリドをそれぞれタンパク質合成時と細胞溶解後に添加しました。すべての精製ステップからのサンプルをSDS-PAGEゲルにロードして、最終製品の品質を確認しました(図5)。
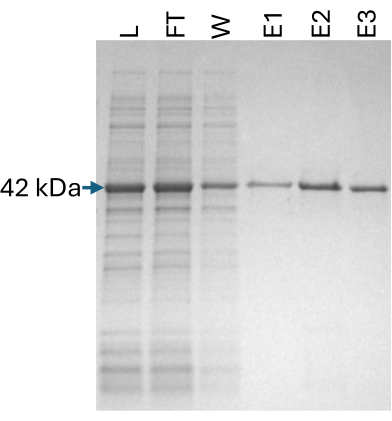
図5:SpyCatcher2-Apex2の精製。 SpyCatcher2-Apex2タンパク質の精製ステップからの画分を示す12%SDS-PAGEゲル。L:ライセート(2μL)、FT:フロースルー(2μL)、W:洗浄(10μL)、E1-3:溶出1-3(10μL)各42 kDaの予想バンドがマークされています。略語:SDS-PAGE = ナトリウムドデシル硫酸ポリアクリルアミドゲル電気泳動。 この図の拡大版を表示するには、ここをクリックしてください。
標的抗原の無細胞発現と品質検証
NPタンパク質の5 μLスケールのパイロット発現とSDS-PAGEを使用した品質チェックを行った後、プロトコルセクション3に記載されているように1 mLスケールの発現を行いました。最終的な無細胞発現混合物の1 μLサンプルを、SpyCatcher2-Apex2と抗NP市販抗体で検出するために、別々にウェスタンブロットにかけました(図6)。このステップにより、抗原がうまく発現し、抗NP免疫グロブリンとSpyCatcher2-Apex2レポータータンパク質の両方に特異的に結合できることが保証されます。
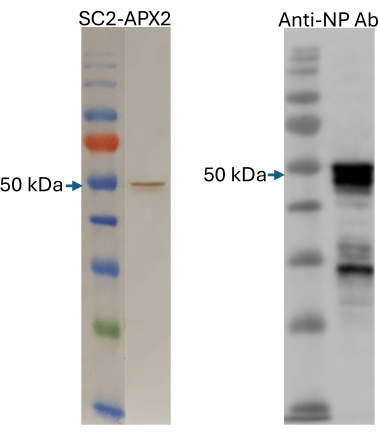
図6:無細胞産生抗原の品質チェック。 SpyCatcher2-Apex2(左パネル)および抗NPウサギポリクローナル抗体(右パネル)を用いた無細胞産生NPタンパク質のウェスタンブロット検出。50 kDaで予想される全長積がマークされています。両方のゲルのサイズマーカーは、Color Pre-stained Protein Standard, Broad Rangeです。この図は、Norouzi et al.5 から採用されました。略語:NP =ヌクレオカプシドタンパク質。 この図の拡大版を表示するには、ここをクリックしてください。
事前に特徴付けられたヒト血清を用いたCFDB試薬の検証
CFDB試薬が高品質であり、CFDBプロトコルが成功裏に実施されていることを確認するために、NISBC-WHOの抗SARS-CoV-2免疫グロブリン国際参照パネルから提供された事前特性評価血清パネルを使用した20/26813。このパネルは、COVID-19 以前の x1 サンプル (陰性) と x4 の SARS-CoV-2 陽性サンプルで構成されており、抗体反応のレベルはさまざまです (補足表 S2)。また、RayBiotech(CoV-PosSet)から、COVID-19以前(陰性)10のサンプルとSARS-CoV-2陽性と推定されるサンプル20のサンプルを含む血清パネルを取得しました。10の陰性血清すべてから等しい画分をプールして、私たちの研究室で行われたすべての実験で陰性コントロールとして使用しました。このプーリング戦略により、単一のネガティブサンプルが微妙なレベルのバックグラウンドシグナルを示す可能性があり、結果の解釈を複雑にする可能性があります。
事前に特徴付けられたWHOリファレンスパネルとRayBiotechのCoV-PosSetに対して、プロトコルセクション5に従ってCFDBアッセイを実施し、ブロットをイメージングしました。Image Lab Softwareのボリュームツールを使用して、各スポット位置に均一な円を描き、個々の信号強度をエクスポートしました。すべてのサンプルからNC膜バックグラウンド強度を差し引いた後、カットオフ値((ネガティブコントロールの平均)+(ネガティブコントロールの3 x SD))も平均シグナル強度から差し引かれました。サンプルは、信号強度がカットオフ値を超える場合、アンチNP陽性と見なされます。ブロットと対応するシグナル強度チャートの画像を 図7に示します。この結果は、無細胞で産生されたSARS-CoV-2 NPタンパク質を使用すると、CFDB技術が、RayBiotechセットの2つのサンプル(症状発症後34日で採取したRB P5(dpso)とRB P10で採取した4 dpso)を除くすべての陰性サンプルとすべての陽性サンプルを正しく同定できることを示しています。サプライヤーのデータシートをさらに調査したところ、これらのサンプルは両方とも、ELISAによる抗S1RBD(Spike Receptor Binding Domain)IgGおよびラテラルフローアッセイによる抗N IgG/Mに対しても陰性であることが確認されました(補足表S3)ことから、これらのサンプルには検出可能な抗体レベルが不足していることが示唆されています。

図7:事前に特徴付けられた市販のヒト血清を用いたCFDBの検証 (A)WHO国際リファレンスパネルおよびRayBiotech PosSet血清サンプル上の抗SARS-CoV-2 NP CFDBを表すブロット画像。サンプルは列方向に三重に発見され、ラベルによって識別されました。(B)パネル Aのブロットの定量化された化学発光シグナル強度値を表すグラフ。ネガティブコントロールはカットオフ値の設定に使用され、すべての測定値は標準偏差(SD)±3重スポットの平均です。この図はNorouziらから取得されました。5.略語:Neg Cont = RayBiotechの10個前のCOVID-19サンプルすべてをプールして得られたネガティブコントロールサンプル。RB N1 から N10 = RayBiotech 推定陰性サンプル 1 から 10;RB P1 から P20 = RayBiotech は陽性サンプルを 1 から 20 と推定しました。CFDB = 無細胞ドットブロット;NP =ヌクレオカプシドタンパク質;WHO=世界保健機関。 この図の拡大版を表示するには、ここをクリックしてください。
ヒト患者血清を用いたCFDBアッセイの独立試験
CFDBアッセイの適用性を実証するために、別の場所にある独立した協力研究所(National Microbiology Laboratory(NML)、ウィニペグ)にCFDBの資料と指示を提供しました。その目的は、NMLのフィールド研究グループによってクリニックで収集されたヒト患者サンプルを使用して、プラットフォームを独立してテストすることでした。合計24検体(NP抗体についてELISAで確認されたx12陰性およびx12陽性(補足表S4))をCFDBに供し、その結果を上記のように解析した。 図8に示すように、CFDBはすべてのx12陰性サンプルとx12陽性サンプルを正しく同定し、独立したテストでアッセイの堅牢性と実用性を確認しました。

図8:NMLでのヒト患者血清を用いたCFDBの独立試験 (A)12人のCOVID-19陰性(N1からN12)または12人の陽性(P1からP12)の患者サンプルに対する抗SARS-CoV-2 NP CFDBを表すブロット画像。セラは、ラベル付けされたように、列ごとに三重に発見されました。(B)パネルAのブロットからの定量化された化学発光シグナル強度値 。 すべての測定値は、標準偏差(SD)±3重のスポットの平均です。この図はNorouziらから取得されました。5.略語:CFDB =無細胞ドットブロット;NML = 国立微生物学研究所;NP =ヌクレオカプシドタンパク質。 この図の拡大版を表示するには、ここをクリックしてください。
SARS-CoV-2感染ハムスター血清を用いたCFDBアッセイの独立試験
その後、NMLチームは、同じCFDB試薬と指示書を使用して、SARS-CoV-2感染したハムスター血清に対してCFDBアッセイを実施し、このアッセイが動物サンプルの抗体応答の検出にも使用できるかどうかを検証しました。合計で36のサンプルがテストされ、x12の感染前と24の感染後血清がテストされました。陽性と推定された血清は、感染後140日(dpi 140、n = 3)にサンプリングされたSARS-CoV-2の105 50%組織培養感染用量(TCID50)に感染した動物からのものでした。またはDPI 140で再感染し、5日後にサンプリングされました(DPI 5、N = 21)。 図9に示すように、感染前のすべての血清のシグナル強度はカットオフ値を下回っており、感染後24人中18人(すべて再感染後5日目)の血清は、個別の陽性シグナル強度を誘発しました。興味深いことに、CFDBが見逃したと推定される6つの陽性サンプルのうち、3つのサンプル(すべて最初のdpi 140からのもの)も標準的な抗NP ELISA(補足表S5)を使用しても検出できず、時間の経過とともに抗体レベルが減少または減少したことを示しています。
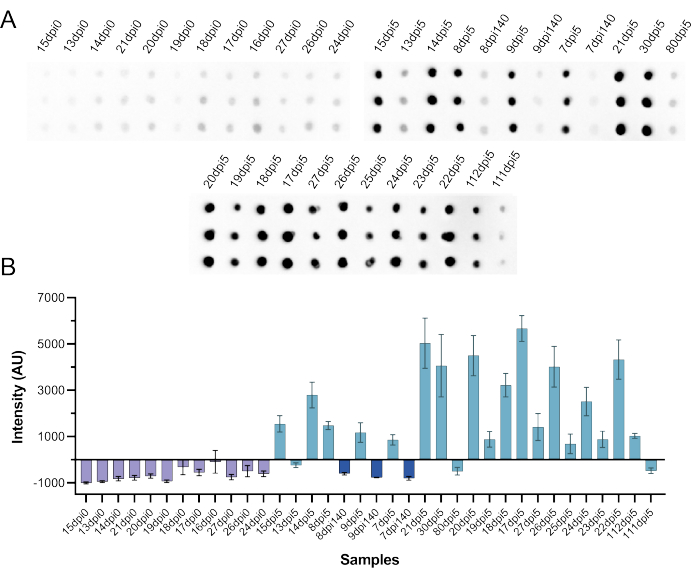
図9:NMLでのSARS-CoV-2感染ハムスター血清を使用したCFDBの独立試験。 (A)ハムスター血清の抗SARS-CoV-2 NP CFDBを示す抗SARS-CoV-2 NP CFDBを示すブロット画像(dpi 0、n = 12)、感染後140日(dpi 140、n = 3)、および再感染の5日後(dpi 5、n = 21)で、SARS-CoV-2の105 TCID50 。各サンプルは、列方向に三重のスポットとして堆積し、元のNML研究のスキームに従って標識しました。(B)パネルAのブロットからの定量化された化学発光シグナル強度値 。 すべての測定値は、標準偏差(SD)±3重のスポットの平均です。この図はNorouziらから取得されました。5.略語:CFDB =無細胞ドットブロット;NML = 国立微生物学研究所;NP =ヌクレオカプシドタンパク質;dpi = 感染後日数;TCID50 = 50% 組織培養感染線量。 この図の拡大版を表示するには、ここをクリックしてください。
補足ファイル1:His-SpyTag-SARS-CoV-2 NP直鎖発現テンプレートのヌクレオチド配列。 略語:NP =ヌクレオカプシドタンパク質。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル2:pET24b SpyCatcher2-Apex2プラスミドの全ヌクレオチド配列。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル3:大腸菌BL21無細胞タンパク質発現ライセートの調製における主要なステップの概要。このファイルをダウンロードするには、ここをクリックしてください。
補足図S1:pET24b SpyCatcher2-Apex2プラスミドのマップを作成します。このファイルをダウンロードするには、ここをクリックしてください。
補足図S2:CFDBマスターグリッドの印刷可能なコピー。略語:CFDB = cell-free dot blot.このファイルをダウンロードするには、ここをクリックしてください。
補足図S3:CFDB NCメンブレンとマスターグリッドアセンブリのセットアップの画像。 底面のガラス板と上面のマスターグリッドパターンの間にNCメンブレンは挟まれており、固定テープで組み立てを固定しています。この図はNorouziらから取得されました。5.略語:CFDB =無細胞ドットブロット;NC = ニトロセルロース このファイルをダウンロードするには、ここをクリックしてください。
補足表S1:無細胞反応混合物の溶液Aおよび溶液B成分の完全なレシピ。このファイルをダウンロードするには、ここをクリックしてください。
補足表S2:NIBSC WHO参照パネル20/268のサンプル情報( 図7も参照)。 略語:NISBC =国立生物標準管理研究所。WHO=世界保健機関。 このファイルをダウンロードするには、ここをクリックしてください。
補足表S3:サンプル情報RayBiotech CoV-PosSet(図7も参照)。このファイルをダウンロードするには、ここをクリックしてください。
補足表S4:ヒト患者血清サンプルのNP ELISA結果(NMLで実施)。図 8 も参照してください。略語:NP =ヌクレオカプシドタンパク質;ELISA = 酵素結合免疫吸着アッセイ;NML = 国立微生物学研究所。このファイルをダウンロードするには、ここをクリックしてください。
補足表S5:SARS-CoV-2感染ハムスター血清のNP ELISA結果(NMLで実施)。 図 9 も参照してください。略語:NP =ヌクレオカプシドタンパク質;ELISA = 酵素結合免疫吸着アッセイ;NML = 国立微生物学研究所。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
COVID-19は、感染の発生を制御し、世界の健康戦略を最適化するためのアクセス可能で堅牢な診断の重要性を浮き彫りにしました。防御抗体を検出する血清学的検査は、新しい変異株の感染パターンを追跡し、ホットスポットを特定し、ワクチン開発を指導し、疑わしい症例をトリアージし、脆弱な集団を保護するために不可欠であることが証明されました14。また、パンデミックは、高度な機器を備えた施設のバックログや要件に悩まされ、アクセシビリティのテストにおける不公平さも露呈した15。したがって、現在、焦点は、将来のアウトブレイクに対する世界的な対応を強化することにあり、そのためには、検査の障壁を減らし、予測不可能な脅威に迅速に適応できる新たなイノベーションが必要になります。
CFDBは、患者サンプルを固相に固定化し、抗ウイルス抗体5を検出するための移動相無細胞産生抗原を送達することにより、従来はELISAまたはラテラルフローアッセイによって行われていた血清学的検査を簡素化する紙ベースのイムノアッセイです。このアッセイは、ウェスタンブロット機能を備えた基本的なラボ環境で実施できます。一方、CFDB用の試薬は、基礎研究の現場で局所的に作成でき、新しい病原体のアッセイを再利用するために容易に交換できます。キメラSpyCatcher2-Apex2ユニバーサル検出試薬は、市販の二次抗体コンジュゲートを調達する必要性を排除し、 大腸菌 発現システムで効率的に生産されることを可能にする機能です。また、CFDBは、市販の96ウェルELISAの約300米ドルに対し、アッセイキットあたりわずか3米ドルというわずかなコストで臨床アッセイを並行して行うハイスループット機能も提供します。
COVID-19血清学的検査のためのCFDBの構成は、無細胞転写翻訳システムに標的抗原であるSARS-CoV-2 NPを生成するように指示する線形DNAテンプレートを設計することによって達成されました。次に、ヒトおよび動物の血清をニトロセルロース膜グリッド上にスポットし、無細胞抗原発現混合物とそれに続くSpyCatcher2-Apex2レポータータンパク質を用いてインキュベーションを行いました。この研究では、COVID-19陰性および陽性被験者の血清に対して、当研究室内および独立した環境(NML)の両方でCFDBを実施し、全体として、陽性サンプルと陰性サンプルを区別するためにELISAとの良好な相関が達成されました。プールされた健康な血清をCFDB陰性コントロールとして使用し、このサンプルに基づいてアッセイカットオフ値を計算することにより、偽陰性/偽陽性の同定のリスクが最小限に抑えられました。
CFDBアッセイの高感度は、広範に事前に特徴付けられたNIBSCのWHOリファレンスパネル20/26813のすべてのサンプル、特に抗体価が最も低いサンプル(サンプル20/140が低い、 補足表S2 および 図7を参照)を明確に区別する能力によって明らかにされています。同様に、CFDBの高い特異性は、この研究でテストされた35の健康な血清のいずれについても偽陽性の同定がないことによって実証されています。重要なことは、マルチサイト臨床検証により、CFDBプロトコルが広く使用するために十分に容易であり、異なるユーザーが実行した場合に標準のELISAに匹敵する高いパフォーマンスを維持することが確認されたことです。私たちの経験では、また他の研究者が示しているように16、 大腸菌 溶解物で産生される無細胞抗原は、-20°Cで少なくとも3ヶ月間安定であり、複数の無細胞抗原バッチ調製物は互いに区別がつかないほど機能します。
CFDBプラットフォームを広く採用し、システム設計を新しい病原体に軸足を移す前に、いくつかの考慮事項を考慮する必要があります。CFDBプラットフォームでは、ウイルス抗原または抗原フラグメントが無細胞発現システムによって適切に合成され、抗原部分が存在し、抗体結合に利用可能であることが必要です。例えば、当初はSARS-CoV-2 Spike受容体結合ドメイン(RBD)もCFDBアッセイに含める予定でした。しかし、高収率の発現にもかかわらず、また(システインアミノ酸を欠くNP抗原とは異なり)複雑なジスルフィド結合が必要であるため、RBD産物は容易に凝集し、 大腸菌 抽出物に不溶性に見えたため、信頼性の高いCFDBプローブとしての使用が妨げられました。このようなシナリオでは、適切なフォールディングと抗原機能のために複雑な翻訳後修飾が必要なため、真核生物ベースの無細胞システムの使用が推奨されます。他のペルオキシダーゼベースのイムノアッセイと同様に、ヘモグロビンペルオキシダーゼ活性が結果を交絡させる可能性があるため、目に見える溶血を伴う血清サンプルを除外することも不可欠です。最後に、CFDBは抗体の総応答を検出するように設計されており、抗体-抗原相互作用に関与する特定の免疫グロブリンクラスについては報告しません。
結論として、CFDBは、無細胞合成生物学とドットブロッティングの要素を融合して血清学的検査を効率化する新しい診断技術です。CFDBは、従来のELISAに代わる費用対効果の高い方法を提供し、パンデミック時によく見られるように、追加の検査能力が必要な場合に、世界中の研究所に容易に導入することができます。したがって、CFDBは、新たな脅威に迅速に適応し、低・中所得国の検査能力を強化する、次の世界的なパンデミック対応の最前線のツールとして機能することができる。
開示事項
M.N.とK.P.は、無細胞ドットブロット法の共同発明者です。 この研究に関連する仮特許出願が提出されました(PCT/CA2024/050097、2024年1月出願)。
謝辞
S.S.とR.Z.は、国防高等研究計画局(DARPA)からの資金提供を受けています(契約番号N66001-23-2-4042)。表明された見解、意見、および/または調査結果は著者のものであり、国防総省または米国政府の公式見解または政策を表すものと解釈されるべきではありません。この研究は、CIHR Foundation助成プログラム(201610FDN-375469)、CIHR Canada Research Chair Program(950-231075および950-233107)、Canada First Research Excellence Fundから資金提供を受け、カナダ国防研究開発省のCanada Safety and Security Program(契約39903-200137)からK.P.に資金提供を受ける、CIHR Foundation助成プログラム(201610FDN-375469)、Canada First Research Excellence FundからK.P.への資金提供を受けたK.P.への資金提供によって支援されました。 図1 と 補足図S1 は、SnapGene Viewerを使用して作成されました。
資料
| Name | Company | Catalog Number | Comments |
| 1 kb DNA ladder | NEB | N3232 | Used as a size marker for agarose gels |
| 20 Amino acids | Sigma-Aldrich | LAA21-1KT | A component of the cell-free reaction Solution B |
| 2 mm biopsy punch | Integra Miltex | 33-31-P/25 | For preparation of CFDB master grid |
| 5-aminolevulinic acid hydrochloride | Sigma-Aldrich | A3785 | Heme precursor for induction of SpyCatcher2-Apex2 |
| Agarose powder | BioShop | AGA002 | For electrophoretic analysis of DNA |
| Anti-SARS-CoV2-Nucleocapsid antibody | Sinobiological | 40588-T62 | For WB detection of cell-free produced NP antigen |
| Bio-Rad ChemiDoc XRS+ | Bio-Rad | N/A | Gel imager instrument |
| Centrifugal concentrators | Cytiva | 28-9323-60 | For concentration and buffer exchange of proteins |
| Centrifuge | Eppendorf | EP022628257 | For harvesting bacterial culture |
| Coenzyme A sodium salt hydrate (CoA) | Sigma-Aldrich | C3144 | A component of the cell-free reaction Solution A |
| Color pre-stained protein standard (broad range) | NEB | P7719 | Used as a size marker for SDS-PAGE gels |
| Covid-19 serum panel | RayBiotech | CoV-PosSet | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| D-(−)-3-Phosphoglyceric acid disodium salt (3-PGA) | Sigma-Aldrich | P8877 | A component of the cell-free reaction Solution B |
| Dithiothreitol (DTT) | Sigma-Aldrich | 10197777001 | Component of buffers |
| E. coli 5-alpha | NEB | C2987 | for plasmid preparation |
| E. coli BL21 | NEB | C2530 | For preparation of cell-free lysates |
| E. coli BL21 (DE3) | NEB | C2527 | For expression of SpyCatcher2-Apex2 |
| EDTA-free protease inhibitor tablet | Sigma-Aldrich | 11836153001 | A component of E. coli lysis buffer |
| Eppendorf New Brunswick Innova 43/43R Incubator Shaker | Eppendorf | EPM1320 | Incubator for growing E. coli cells for protein expression and cell-free lysate preparation |
| Folinic acid | Sigma-Aldrich | 47612 | A component of the cell-free reaction Solution A |
| Glycerol | Sigma-Aldrich | G9012 | Component of SpyCatcher2-Apex2 storage buffer |
| Hemin chloride | Sigma-Aldrich | H9039 | For Heme supplementation of SpyCatcher2-Apex2 |
| Hydrogen peroxide 30% | Sigma-Aldrich | H1009 | For making ECL reagent |
| Image Lab Software | Bio-Rad | 1709690 | Software for ChemiDoc gel imaging instrument |
| Isopropyl-b-D-1-thiogalactopyranoside | Bioshop | IPT001 | For induction of SpyCatcher2-Apex2 expressikon |
| Kanamycin Sulfate | Sigma-Aldrich | 60615 | For preparation of SpyCatcher2-Apex2 bacterial culture |
| LB agar | BioShop | LBL406 | For E. coli growth |
| LB broth | BioShop | LBL407 | For E. coli growth |
| luminol | Sigma-Aldrich | A4685 | For making ECL reagent |
| Lysozyme | Sigma-Aldrich | L6876 | For lysis of bacterial cells |
| Magnesium Acetate | Sigma-Aldrich | M5661 | A component of the cell-free reaction Solution B |
| NEBExpress Ni resin | NEB | S1428S | For purification of SpyCatcher2-Apex2 |
| Non-fat dry milk | Bioshop | SKI400 | For blocking of WB and CFDB membranes |
| Parafilm | Bemis | 2099-1337410 | For ECL-incubation of CFDB blots |
| p-coumaric acid | Sigma-Aldrich | C9008 | For making ECL reagent |
| pET24b-SpyCatcher2-Apex2 plasmid | Pardee Laboratory | N/A | Used for the expression of SpyCatcher2-Apex2 protein |
| Petri dish | Fisherbrand | FB0875712 | Container for WB and CFDB membrane incubation |
| Phosphate buffered saline 10% | BioShop | PBS405 | Component of buffers |
| Potassium Glutamate | Sigma-Aldrich | G1501 | A component of the cell-free reaction Solution B |
| Potassium Oxalate Monohydrate (Oxalic acid) | Sigma-Aldrich | 223425 | A component of the cell-free reaction Solution A |
| Precast SDS-PAGE gel | BioRad | 4561036EDU | For electrophoretic analysis of protein |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification of LETs |
| QIAquick PCR purification kit | Qiagen | 28106 | For purification of LETs |
| Ribonucleotide Solution Set | NEB | N0450S | A component of the cell-free reaction Solution A |
| Skim milk powder | BioShop | SKI400 | Used as a blocking agent for western blot and CFDB |
| Sodium Chloride | Sigma-Aldrich | S9625 | A component of protein purification buffers |
| Sonicator | Qsonica | Q500 | For lysis of bacterial cells |
| Spermidine | Sigma-Aldrich | S2626 | A component of the cell-free reaction Solution A |
| Syringe filter | Sigma-Aldrich | SLGSR33SS | For sterilisation of buffers and solutions |
| Thermocycler | BioRad | T100 | Instrument for incubating PCR reactions |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8759 | A component of the cell-free reaction Solution A |
| Tris buffered saline | Thermo Scientific | J60764.K2 | Component of WB and CFDB wash buffer |
| Trizma base | Sigma-Aldrich | T1503 | For making tris buffer |
| Tunair SS-5012 Half-Baffle Shake Flask, 2.5 L | Cole-Parmer | RK-01835-39 | Vessel for growing E. coli cells for protein expression and cell-free lysate preparation |
| Tween-20 | BioShop | TWN510 | Component of WB and CFDB wash buffer |
| Tweezer | Almedic | 7728-A10-100 | For handling CFDB NC membrane |
| UV-Vis spectrophotometer | Thermo Scientific | 13400518 | Used for the measurement of nucleic acid and DNA concentration and bacterial culture OD |
| WHO International Reference Panel for anti-SARS-CoV-2 immunoglubulin | NISBC | 20/268 | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| β-Nicotinamide adenine dinucleotide hydrate (NAD) | Sigma-Aldrich | 10127965001 | A component of the cell-free reaction Solution A |
参考文献
- Carter, L. J., et al. Assay techniques and test development for COVID-19 diagnosis. ACS Cent Sci. 6, 591-605 (2020).
- Amanat, F., et al. A serological assay to detect SARS-CoV-2 seroconversion in humans. Nat Med. 26 (7), 1033-1036 (2020).
- Stadlbauer, D., et al. SARS-CoV-2 seroconversion in humans: A detailed protocol for a serological assay, antigen production, and test setup. Curr Protoc Microbiol. 57 (1), e100(2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259.e12 (2016).
- Norouzi, M., et al. Cell-free dot blot: an ultra-low-cost and practical immunoassay platform for detection of anti-SARS-CoV-2 antibodies in human and animal sera. Microbiol Spectr. 11 (2), e0245722(2023).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Norouzi, M., Panfilov, S., Pardee, K. High-efficiency protection of linear DNA in cell-free extracts from Escherichia coli and Vibrio natriegens. ACS Synth Biol. 10 (7), 1615-1624 (2021).
- IDT codon optimization tool. , Integrated DNA Technologies. https://www.idtdna.com/pages/tools/codon-optimization-tool (2025).
- General protocol for western blotting. , Bio-Rad. https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6376.pdf (2025).
- Mruk, D. D., Cheng, C. Y. Enhanced chemiluminescence (ECL) for routine immunoblotting. Spermatogenesis. 1, 121-122 (2011).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-based cell-free protein synthesis: Protocols for a robust, flexible, and accessible platform technology. J Vis Exp. (144), (2019).
- Plasma and serum preparation. , ThermoFisher Scientific. https://www.thermofisher.com/ca/en/home/references/protocols/cell-and-tissue-analysis/elisa-protocol/elisa-sample-preparation-protocols/plasma-and-serum-preparation.html (2025).
- Mattiuzzo, G., et al. WHO/BS.2020.2403 Establishment of the WHO International Standard and Reference Panel for anti-SARS-CoV-2 antibody. , (2020).
- Gong, F., Wei, H., Li, Q., Liu, L., Li, B. Evaluation and comparison of serological methods for COVID-19 diagnosis. Front Mol Biosci. 8, 682405(2021).
- Peeling, R. W., et al. Serology testing in the COVID-19 pandemic response. Lancet Infect Dis. 20 (9), e245-e249 (2020).
- Smith, M. T., Berkheimer, S. D., Werner, C. J., Bundy, B. C. Lyophilized Escherichia coli-based cell-free systems for robust, high-density, long-term storage. Biotechniques. 56 (4), 186-193 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved