Method Article
כתם נקודות נטול תאים כפלטפורמת בדיקה חיסונית מעשית וניתנת להתאמה לזיהוי תגובת נוגדנים בסרום של בני אדם ובעלי חיים
In This Article
Summary
אנו מתארים פלטפורמת בדיקה חיסונית שפותחה לאחרונה המבוססת על עקרונות הביולוגיה הסינתטית נטולת התאים וטכניקת כתמי הנקודות לזיהוי מותאם אישית של תגובת נוגדנים בבני אדם ובעלי חיים.
Abstract
שרשרת ההתפרצויות הפתוגניות העולמיות בשני העשורים האחרונים הדגישה את החשיבות של אסטרטגיות מעקב סרולוגי. פלטפורמות בדיקה חיסונית המשמשות לאיתור נוגדנים ספציפיים למחלה בסרום של חולים נמצאות בליבת המעקב הסרולוגי. דוגמאות נפוצות כוללות מבחני אימונוסורבנט הקשורים לאנזים ומבחני זרימה רוחבית; עם זאת, בעוד שמדובר בשיטות סטנדרטיות זהב, הן דורשות חומרים מתכלים ספציפיים לפתוגן וציוד מיוחד, מה שמגביל את השימוש בהם מחוץ למעבדות בעלות משאבים טובים.
לאחרונה פיתחנו פלטפורמת בדיקה חיסונית חדשה בשם Cell-Free Dot-Blot (CFDB) ואימתנו אותה באמצעות נסיוב אנושי ובעלי חיים כנגד SARS-CoV-2. בניגוד לבדיקות אימונולוגיות קונבנציונליות, דגימות סרום של חולי CFDB משותקות לשלב מוצק (קרום ניטרוצלולוזה), בעוד אנטיגן המטרה מושעה בשלב הנייד של הבדיקה. כדי לשפר את הגישה ליכולות מעקב סרולוגי, אנטיגנים של CFDB מיוצרים לפי דרישה ועם תשתית עומס נמוכה באמצעות ביטוי חלבון במבחנה . כאן, האנטיגן מתמזג עם תג פפטיד שניתן לזהות באמצעות חלבון מדווח אוניברסלי יחיד עבור כל בדיקת CFDB. התוצאה היא שה-CFDB אינו דורש גישה לקורא צלחות מרובה בארות או לרכיבי בדיקה מולקולרית מסחריים מטוהרים. עם שיקולי תכנון אלה, CFDB מטפל בחסרונות של פלטפורמות הבדיקה החיסוניות הקיימות על ידי מתן נגישות למעבדות לא ריכוזיות, יכולת הסתגלות לפתוגנים מתעוררים ומחיר סביר לקהילות בעלות הכנסה נמוכה.
במאמר הנוכחי נספק פרוטוקול שלב אחר שלב להכנה וביצוע של בדיקת חיסון CFDB. באמצעות עבודתנו האחרונה על SARS-CoV-2 CFDB כדוגמה, נסקור תכנון DNA של אנטיגן לייצור נטול תאים לפי דרישה, ואחריו הכנת חלבון מדווח CFDB, קיבוע דגימות סרום בשלב המוצק, ולבסוף, שלבי קשירת אנטיגן וזיהוי של הבדיקה. אנו צופים כי על ידי ביצוע הוראות אלה, חוקרים יוכלו להתאים את בדיקת ה-CFDB כדי לזהות תגובות חיסוניות בנסיוב אנושי ובעלי חיים לכל פתוגן נתון.
Introduction
מגיפת COVID-19 חשפה את הצורך הקריטי בכלי אבחון זולים וניתנים להרחבה, במיוחד עבור סביבות דלות משאבים1. בדיקות אימונולוגיות קונבנציונליות כמו מבחני אימונוסורבנט מקושרים לאנזים (ELISAs) הוכחו כחיוניים לאיתור תגובות חיסוניות 2,3. עם זאת, עלותם הגבוהה, ההסתמכות על ריאגנטים מורכבים והתלות בציוד מיוחד מגבילים את נגישותם, במיוחד במהלך משברי בריאות עולמיים. בתגובה לאתגרים אלה, פיתחנו את כתם הנקודות ללא תאים (CFDB), פלטפורמת בדיקה חיסונית בעלות נמוכה וניתנת להתאמה המיועדת לזיהוי נוגדנים נגד SARS-CoV-2 בבני אדם ובבעלי חיים.
CFDB ממנף ביולוגיה סינתטית נטולת תאים לייצור מהיר לפי דרישה של אנטיגנים נגיפיים באמצעות תבניות DNA ליניאריות 4,5. זה מבטל את הצורך בתהליכי שיבוט, ביטוי וטיהור מסורתיים מבוססי תאים, ומאיץ משמעותית את ייצור האנטיגן תוך הפחתת עלויות. שיטת CFDB מפשטת את זיהוי הנוגדנים על ידי שימוש בפורמט כתם נקודות, שבו הסרום מזוהה ישירות על ממברנות הניטרוצלולוזה. מערכת זו מייתרת את הצורך בצלחות מרובות בארות יקרות וציוד מעבדה מיוחד, מה שמאפשר זרימת עבודה פשוטה של "טבילה" לשלבי הדגירה והשטיפה. הפלטפורמה משתמשת גם במערכת SpyCatcher-SpyTag, שבה כימרה של SpyCatcher2-Apex2 peroxidase פועלת כמגיב זיהוי משני אוניברסלי 5,6. זה מיוצר באמצעות ביטוי סטנדרטי מבוסס Escherichia coli, המבטל את התלות במצומדים מסחריים יקרים של נוגדנים. כתוצאה מכך, מערכת CFDB יכולה לבצע בדיקות סרולוגיות עם ביצועים דומים ל-ELISA בעלות נמוכה משמעותית - כ-3 דולר לבדיקת דגימה של 96 בהשוואה ליותר מ-300 דולר לערכת ELISA מסחרית5.
כדי להדגים את יעילותו של CFDB, בדקנו את יכולתו לזהות נוגדנים בסדרות אנושיות ובעלי חיים מוגדרות מראש. התוצאות שלנו היו בקורלציה הדוקה עם ELISA בזיהוי דגימות חיוביות ושליליות ל-COVID-19. בנוסף לאבחון אנושי, הערכנו את התועלת של CFDB במודלים של בעלי חיים, בדקנו סרום מאוגרים נגועים ב-SARS-CoV-2 וכאלה שחוסנו בחלבון נוקלאוקפסיד רקומביננטי. בדיקות אלו אישרו את הפוטנציאל של CFDB לשימוש באבחון אנושי ווטרינרי כאחד, מה שהופך אותו לכלי רב-תכליתי לניטור תגובות חיסוניות בין מינים. אחד היתרונות המרכזיים של CFDB הוא הגמישות שלו. פשוט על ידי שינוי תבנית ה-DNA המקודדת את האנטיגן המעניין, ניתן להתאים את הפלטפורמה במהירות לאיתור נוגדנים נגד פתוגנים שונים, מה שהופך אותה לבעלת ערך למוכנות למגיפה עתידית. העלות הנמוכה, זרימת העבודה הפשוטה ודרישות התשתית המינימליות שלו הופכים אותו למתאים במיוחד עבור מעבדות מבוזרות וסביבות דלות משאבים, שבהן הגישה לאבחון מסחרי מוגבלת.
בעבודה זו נספק הוראות שלב אחר שלב להכנה וביצוע בדיקת CFDB. ראשית, אנו מכסים את התכנון והסינתזה של תבניות DNA ליניאריות לייצור אנטיגנים ללא תאים, שהם ריאגנטים לזיהוי העיקריים של הבדיקה. לאחר מכן אנו מתארים שלבים להכנת מגיב הזיהוי המשני של הבדיקה SpyCatcher2-Apex2. לאחר מכן, אנו מספקים הוראות לייצור נטול תאים ובדיקת איכות של האנטיגנים עצמם. לבסוף, אנו מתארים בפירוט את התהליך לביצוע בדיקת CFDB על דגימות סרום של בני אדם או בעלי חיים.
Protocol
כל הניסויים באוגרים בוצעו במעבדה הלאומית למיקרוביולוגיה (NML) בסוכנות לבריאות הציבור של קנדה, באישור המרכז הקנדי למדע לבריאות האדם ובעלי החיים, ובהתאם להנחיות המועצה הקנדית לטיפול בבעלי חיים. כל דגימות הסרום/פלזמה האנושיות הושגו באופן מסחרי לבדיקה פנימית או סופקו על ידי משתפי פעולה קליניים ל-NML לבדיקה עצמאית ב-NML.
1. תכנון והכנה של תבניות ביטוי ליניאריות אנטיגן (LETs)
- תכנן את הביטוי נטול התאים LETs עבור אנטיגן המטרה על סמך ההוראות ב-Norouzi et al.7 ומכיל מסוף N/C SpyTag כמתואר ב-Norouzi et al.5.
הערה: כאן אנו מספקים פרטים להכנת חלבון נוקלאוקפסיד SARS-CoV-2 (NP, חומצות אמינו 2-419).- ודא שה-LET עבור SARS-CoV-2 NP (איור 1 וקובץ משלים 1) מכיל אתרי 5' ו-3' Ter עם רצף מאגר של 50 זוגות בסיסים לפני מקדם T7 ואחרי קודון העצירה.
- סמן את חלבון ה-NP בקצה ה-N עם 6-SpyTag-TEV שלו.
- השג את רצף החלבון ממסד הנתונים של UniProt (קוד גישה P0DTC9) ובצע אופטימיזציה של קודון לביטוי מבוסס E. coli באמצעות כלי אופטימיזציה של קודון IDT8. הזמינו רצף זה לסינתזה מסחרית כמקטע DNA חד-גדילי התלוי במים ב-10 ננוגרם/מיקרוליטר.
הערה: אתר מחשוף הפרוטאז TEV ותג His6 אינם מאפיינים חיוניים בעיצוב ה-LET.
- PCR מגביר את שבר ה-DNA עם ה-Ter FW האוניברסלי (GGCTCCGAATAAGTATGTTGTAACTAAAGTGCGGCC
ACGATGCGTCCGGCGTAGAGGATCG) ו-Ter RV (CCGAGGCAATAAGTATGTTGTAACTAAAGTGCTCAG
CTTCCTTTCGGGCTTTGTTAGCAGCC) באמצעות ערכת DNA פולימראז בנאמנות גבוהה (ראה טבלת החומרים)- הגדר תגובה של 100 מיקרוליטר באופן הבא: 75 מיקרוליטר של מים נטולי נוקלאז, 20 מיקרוליטר של מאגר DNA פולימראז פי 5, 2 מיקרוליטר של 10 מ"מ dNTPs (ריכוז סופי של 200 מיקרומטר), 0.5 מיקרוליטר של פריימר 100 מיקרומטר Ter-FW, 0.5 מיקרוליטר של פריימר 100 מיקרומטר Ter-RV, 1 מיקרוליטר של 10 ננוגרם/מיקרוליטר LET חד-גדילי, 1 מיקרוליטר (2U) של DNA פולימראז.
- השתמש בהגדרות ה-PCR הבאות: דנטורציה ראשונית ב-98 מעלות צלזיוס למשך 30 שניות; 35 מחזורים: 98 מעלות צלזיוס למשך 6 שניות, 60 מעלות צלזיוס למשך 15 שניות, 72 מעלות צלזיוס למשך 90 שניות; יש להחזיק בטמפרטורה של 4 מעלות צלזיוס.
- לטהר את מוצר ה-PCR באמצעות ערכת טיהור PCR מסחרית, לבדוק את איכותו על ידי הפעלת דגימה על ג'ל אגרוז 1%, ולמדוד את ריכוזו בספקטרופוטומטר UV-Vis.
הערה: ניתן להשתמש במוצרי PCR גולמיים גם ישירות לביטוי נטול תאים. עם זאת, הטיהור מאפשר הליך סטנדרטי יותר.

איור 1: תבנית ביטוי ליניארית עבור SARS-CoV-2-NP. סכמטי המייצג תכונות של תבנית ה-DNA הליניארית His-SpyTag-SARS-CoV-2 NP. מרכיבי תבנית DNA מרכזיים מסומנים. רצף הקידוד של החלבון המבוקש, כאן ה-NP, ממוקם תחת בקרת השעתוק של מקדם T7 לביטוי יעיל. בקצה ה-N, חלבון ה-NP מצורף ל-SpyTag לזיהוי ספציפי באמצעות מגיב הזיהוי SpyCatcher2-Apex2. אתרי הפרוטאז x6His-tag ו-TEV, למרות שהם כלולים כחלק מעיצוב ה-LET הכללי, ניתנים לוויתור למטרות CFDB. בקצה תבנית ה-DNA הליניארית, אתרי Ter "במעלה הזרם" ו"במורד הזרם", שלכל אחד מהם קודמים 50 רצפי חיץ זוגות בסיסים בהתאמה, כלולים להגנה בתיווך Tus מפני פירוק DNA אקסונוקלאוליטי בליזאט נטול התאים. קיצורים: NP = חלבון נוקלאוקפסיד; LET = תבנית ביטוי ליניארית; TEV = וירוס תחריט טבק; CFDB = כתם נקודות נטול תאים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
2. טיהור חלבון המדווח SpyCatcher2-Apex2
- השתמש בפלסמיד pET24b-SpyCatcher2-Apex2, שנבנה במקור ב-Norouzi et al.5 (רצף מלא בקובץ משלים 2 ומפת פלסמיד באיור משלים S1) כדי להכין את חלבון המדווח SpyCatcher2-Apex-2.
- הכינו צלחות אגר ומרק ליזוגני (LB) המכיל 50 מיקרוגרם/מ"ל קנמיצין. הפוך תאי E. coli BL21 (DE3) עם הפלסמיד pET24b-SpyCatcher2-Apex2. יש לחסן מושבה בודדת לתרבית LB התחלתית של 15 מ"ל ולגדל בן לילה בטמפרטורה של 37 מעלות צלזיוס עם ניעור ב-250 סל"ד.
- למחרת, הוסף 10 מ"ל של תרבית המתנע ל-500 מ"ל של מדיה LB טרייה המכילה 50 מיקרוגרם/מ"ל קנמיצין. דגירה ב-37 מעלות צלזיוס עם טלטול ב-250 סל"ד עד שהתרבית מגיעה לצפיפות אופטית ב-600 ננומטר של 0.6-0.8 (~3 שעות).
- לגרום לביטוי של SpyCatcher2-Apex2 על ידי השלמת התרבית עם 0.5 מ"מ isopropyl-β-D-1-thiogalactopyranoside ו-1 mM 5-aminolevulinic acid hydrochloride. הפחיתו את טמפרטורת הגידול ל -30 מעלות צלזיוס ואפשרו לתרבות לדגור למשך 4 שעות נוספות.
- קצרו את החיידקים על ידי צנטריפוגה בחום של 8,000 × גרם למשך 15 דקות. המשך לליזה של תאים או לחילופין אחסן את הגלולה ב-80 מעלות צלזיוס עד לשימוש.
- השהה מחדש את הגלולה ב-20 מ"ל של מאגר ליזה המכיל 50 מ"מ Tris-HCl (pH 7.8), 300 מ"מ NaCl, 1 מ"ג/מ"ל ליזוזים, טבלית מעכב פרוטאז ללא EDTA ו-1 מ"מ דיתיותרייטול (DTT). לייז את התאים על ידי סוניקציה במשרעת של 50% עם מרווחים של 5 שניות ON ו-10 שניות OFF למשך זמן ON כולל של 3 דקות.
- הבהירו את הליזאט על ידי צנטריפוגה ב -20,000 × גרם למשך שעה ב -4 מעלות צלזיוס. העבירו את הסופרנטנט דרך מסנן מזרק של 0.2 מיקרומטר.
- הוסף המין כלוריד לריכוז סופי של 250 מיקרומטר לליזט המובהר ודגר למשך הלילה ב-4 מעלות צלזיוס. המשך לטיהור חלבון.
הערה: דגירה עם המין כלוריד ממקסמת את שילוב הם בפרוקסידאז Apex-2 לפעילות אנזימים מיטבית. - הוסף 2.5 מ"ל של שרף Ni לליזט המובהר ודגירה בטמפרטורה של 4 מעלות צלזיוס עם ניעור עדין למשך 45 דקות. מרחו את התערובת על עמוד זרימת כוח הכבידה ושטפו את השרף עם 50 מ"ל של מאגר טריס (50 מ"מ Tris-HCl (pH 7.8), 300 מ"מ NaCl ו-1 מ"מ DTT).
- הסר את החלבון SpyCatcher2-Apex2 ב-25 מ"ל של מאגר Tris המכיל 400 מ"מ אימידזול. תרכיז וחוצץ מחליפים את הפליטה למאגר Tris באמצעות יחידת מסנן צנטריפוגלית, במטרה לנפח סופי של 0.5-1.0 מ"ל.
- השתמש במקדם ההכחדה המולרית של SpyCatcher2-Apex2 (27,390 M-1 cm-1) כדי לקבוע את ריכוז החלבון בספקטרופוטומטר UV-Vis. מוסיפים גליצרול לריכוז סופי של 40% ומאחסנים את המינונים בטמפרטורה של -20 מעלות צלזיוס.
הערה: פרוטוקול זה צפוי להניב כ-40 מ"ג של SpyCatcher2-Apex2 טהור ופעיל במיוחד, מספיק ל-400 ריצות CFDB בגודל כתם סטנדרטי של 96 דגימות. - לזיהוי חלבוני SpyTagged באמצעות כתם מערבי קונבנציונאלי9 ולניסויי CFDB, חסום תמיד ממברנות בחלב יבש ללא שומן 5% בתמיסת מלח 1x Tris-buffered המכילה 0.05% Tween-20 (TBST). השתמש בחלבון SpyCatcher2-Apex2 בריכוז סופי של 10 מיקרוגרם/מ"ל בתמיסת חסימה.
הערה: מדווח SpyCatcher2-Apex2 דורש פתרון כימילומינסנציה משופר (ECL) לפיתוח אותות. ניתן להשיג את תמיסת ה-ECL באופן מסחרי או להכין אותה בבית על פי ההוראות ב-Mruk et al.10. תמיסת ה-ECL הסופית כאן מורכבת מ-0.4 מ"מ חומצה p-קומרית, 2.5 מ"מ לומינול ו-0.015% H2O2 ב-100 מ"מ Tris-HCl (pH 8.6).
3. ייצור ללא תאים ובדיקת איכות של אנטיגנים
- הכן והרכיב רכיבי ליזאט ותגובה נטולי תאים של E. coli BL21 על ידי ביצוע ההוראות ב- Levine et al.11 ו- Norouzi et al.5. השלם את תערובת התגובה הסופית עם 5 μM חלבון Tus ו-1.2 μM T7 RNA פולימראז כדלקמן לתגובה מייצגת של 100 מיקרוליטר: 14.6 מיקרוליטר של תמיסה A, 14 מיקרוליטר של תמיסה B, 33.3 מיקרוליטר של E. coli BL21 ליזאט, 2.5 מיקרוליטר של 200 מ"מ חלבון Tus, 1.2 מיקרוליטר של 100 מ"מ T7 RNA פולימראז, 1 מיקרוליטר של תבנית DNA ליניארית של 1.5 מיקרומטר, 33.4 מיקרוליטר של מים נטולי נוקלאז.
הערה: ראה קובץ משלים 3 להוראות מפתח להכנת ליזט נטול תאים E. coli BL21 וטבלה משלימה S1 למתכון מפורט של פתרונות A ו-B. ניתן להכין את מערכת הביטוי ללא תאי E. coli גם באמצעות פרוטוקולים חלופיים או להשיג באופן מסחרי, כל עוד התגובה מתווספת ל-Tus ו-T7 RNA Polymerase. - בצע בדיקת ביטוי ראשונית בקנה מידה של 5 מיקרוליטר על ידי הוספת תוצר PCR גולמי של 10% (v/v) לתגובה נטולת התאים בשפופרת PCR. דגירה ללא ניעור בטמפרטורה של 30 מעלות צלזיוס למשך 15 שעות.
- בדוק את איכות הביטוי של אנטיגן NP על ידי העמסת 1 מיקרוליטר של התגובה נטולת התאים על ג'ל פוליאקרילאמיד 12% נתרן דודציל סולפט (SDS-PAGE) ולאחר מכן העבר אותו לקרום ניטרוצלולוזה לכתם מערבי כמתואר בשלב 2.12. השתמש בחלבון המדווח SpyCatcher2-Apex2 לתיוג כתמים באמצעות SpyTag, ואופציונלי, נוגדן מסחרי ספציפי (כאן אנטי-SARS-CoV-2-NP) כנגד אנטיגן המטרה.
- הרכיבו תגובה נטולת תאים בקנה מידה של 1 מ"ל עם מוצר LET מטוהר של 15 ננומטר שישמש לבדיקת CFDB. דגרו את תערובת הביטוי בצינור חרוטי של 15 מ"ל עם ניעור ב-80 סל"ד למשך 15 שעות ב-30 מעלות צלזיוס. בדוק את איכות הביטוי באמצעות כתם מערבי ואחסן 50 מיקרוליטר ב-20 מעלות צלזיוס.
4. דגימות סרום
- השג דגימות סרום (או פלזמה) של בני אדם/בעלי חיים ממקורות מסחריים או משתפי פעולה קליניים או בתוך החברה תוך שימוש בנהלים סטנדרטיים ובהתאם להנחיות של ועדת אתיקה מוסדית למחקר בבני אדם או ועדת טיפול ושימוש בבעלי חיים. ודא שננקטו אמצעים מתאימים לטיפול מקדים בדגימה כדי להפחית את הסיכונים לזיהום.
הערה: אמצעי טיפול מקדים עשויים לכלול טיפול בחומר ניקוי ממס, השבתת חום ובדיקת סמני וירוסים הנישאים בדם. פרוטוקול שימושי לבידוד סרום ופלזמה מדם מלא זמין12. בדומה לפלטפורמות אחרות של בדיקות חיסוניות, דגימות סרום שנאספו מוקדם (<3 שבועות) לאחר הזיהום הראשוני או הופעת המחלה אינן צפויות להכיל רמות נאותות של תגובת נוגדנים ויש להתייחס לתוצאותיהן בזהירות. - לאימות איכות של ריאגנטים של SARS-CoV-2 NP CFDB, השתמש בפאנל הייחוס הבינלאומי של המכון הלאומי לתקנים ובקרה ביולוגיים (NIBSC) של ארגון הבריאות העולמי (WHO) לאימונוגלובולין נגד SARS-CoV-2, המכיל דגימות חיוביות ל-x1 לפני COVID-19 ו-x4 SARS-CoV-2 עם רמות שונות של אימונוגלובולינים נגד NP13. לחלופין, השתמש בדגימות סרום בריאות וחיוביות ממקורות אחרים.
- עבור ניסויי CFDB, השג או הכן דגימת בקרה שלילית על ידי איגום מספר (>3) דגימות סרום בריאות כדי להגביר את הדיוק ולהקל על הניתוח והפרשנות של התוצאות.
5. הליך כתם נקודות ללא תאים (CFDB).
- הורד והדפס את קובץ תמונת הרשת הראשית (איור משלים S2). רשת המאסטר היא תבנית בגודל 6X6 ס"מ המכילה 12X12 עיגולים בקוטר 2 מ"מ כל אחד ומספקת יכולת איתור הדומה לצלחת של 96 בארות.
- סנדוויץ' את הרשת בחוזקה בין שתי שכבות של סרט איטום לוחית PCR דביק וחתוך לגודל לאורך גבולות הרשת החיצונית. השתמש באגרוף ביופסיה של 2 מ"מ כדי לחלול כל עיגול מסומן.
הערה: ניתן לעשות שימוש חוזר ברשת ראשית זו מספר פעמים לאחר ניגוב עם 70% אתנול. - חותכים חתיכה של קרום ניטרוצלולוזה בגודל 6.5 על 6.5 ס"מ ומקמו אותה מתחת לרשת הראשית על משטח נקי, ואבטחו את ההתקנה באמצעות סרט דבק כפי שמוצג באיור 2 ובאיור משלים S3. השתמש בעט טוש כדי לסמן את מיקומי המעגל החיצוניים ביותר על קרום הניטרוצלולוזה שישמש כמדריך לחיתוך הממברנה לאחר איתור הדגימה.
- יש לדלל את דגימות הסרום 1/10 בתמיסת מלח 1x עם חוצץ פוספט (pH 7.4). השתמש במיקרופיפטה כדי להוציא נפחים משולשים של 0.4 מיקרוליטר מכל דגימה על קרום הניטרוצלולוזה במיקומי רשת שנקבעו מראש. זה ייקח בערך 15 שניות למקום לחלק.
הערה: הקפד לכלול דגימות בקרה שליליות וחיוביות בכל בדיקה, מכיוון שהדבר יידרש לניתוח התוצאות. - אפשר 10 דקות בטמפרטורת הסביבה עד שהדגימות המנוקדות נקשרות ומתייבשות. בעזרת פינצטה שולפים בזהירות את קרום הניטרוצלולוזה וחותכים לאורך העיגולים החיצוניים המסומנים.
- חסום את הממברנה ב-10 מ"ל של תמיסת חסימה (5% חלב יבש ללא שומן ב-TBST) בצלחת פטרי בגודל 10 ס"מ למשך 30 דקות בטמפרטורת החדר, תוך ניעור עדין ב-100 סל"ד.
- הפשירו והוסיפו 50 מיקרוליטר של תערובת ביטוי האנטיגן נטולת התאים ל-5 מ"ל של תמיסת חסימה בצלחת פטרי בגודל 10 ס"מ. העבירו את הממברנה ישירות לתמיסה המכילה אנטיגן זו ודגרו בטמפרטורת החדר למשך שעה, תוך ניעור ב -100 סל"ד.
הערה: זה יהיה שלב הזיהוי העיקרי שבו האנטיגן SpyTagged ייקשר למיקומים נקודתיים המכילים נוגדנים, אם קיימים. - שטפו את הממברנה, שטפו במשך 5 דקות ושטפו שוב ב-TBST לפני שתמשיך לשלב הזיהוי המשני.
- דגרו את הממברנה ב-10 מ"ל של מאגר חוסם המכיל 10 מיקרוגרם/מ"ל של חלבון SpyCatcher2-Apex2 מטוהר, למשך שעה אחת תוך ניעור ב-100 סל"ד.
- שוטפים את הממברנה ושוטפים 2 x 5 דקות ב- TBST, עם שטיפה אחרונה ב- TBS.
- הדביקו ואבטחו פיסת פרפילם על אזור עבודה נקי קרוב למכשיר הדמיית הכתמים. הסר עודפי נוזלים על ידי הקשה על הממברנה באמצעות פינצטה והנח את הממברנה על גבי הפרפילם.
- מוסיפים מיד 3 מ"ל (100 מיקרוליטר/ס"מ2) של תמיסת ECL על גבי הממברנה ודוגרים בטמפרטורת החדר למשך 90 שניות בדיוק.
- יבש מיד את הממברנה והעביר למכשיר הדמיה תואם כימילומינסנציה להדמיה של התוצאות.
הערה: זמן ההדמיה האופטימלי עשוי להשתנות במכשירים שונים. מומלץ להשתמש בהגדרות ברירת המחדל של זמן הרכישה האוטומטית של המכשיר, ואם נעדר, השתמש בדגימות סרום מאופיינות מראש כדי לייעל את זמן רכישת התמונה. - השתמש בפונקציית ניתוח התמונה של המכשיר כדי להשיג עוצמות נקודתיות, כולל עבור שלושה מיקומים ריקים (רקע) על קרום הניטרוצלולוזה; שמור על נפח מדידה קבוע לכל נקודה. ייצא את הנתונים לגיליון אלקטרוני, וזכור לתייג נכון כל מיקום נקודתי.
- חשב את הממוצע וסטיית התקן (SD) של עוצמות נקודתיות משולשות. לאחר מכן, הפחיתו את רקע קרום הניטרוצלולוזה מכל הדגימות.
- השתמש במשוואה הבאה כדי לקבל ערך חיתוך לפירוש של תוצאות -/+:
(ממוצע של בקרות שליליות) + (3 x SD של הבקרות השליליות). קחו בחשבון דגימות חיוביות אם העוצמה הממוצעת שלהן יורדת מעל ערך החיתוך ושליליות אם העוצמה הממוצעת שלהן יורדת מתחת לערך החיתוך.
הערה: סכימה של נוהל CFDB מסופקת באיור 3.
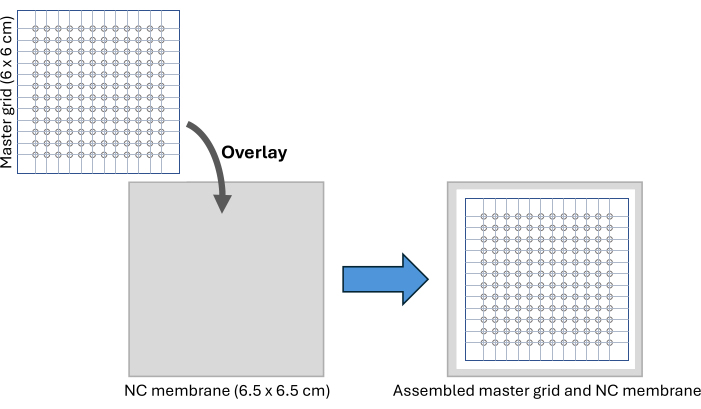
איור 2: מכלול CFDB. סכמטי של רשת האב CFDB ומערך מכלול הממברנה NC. הרשת הראשית מונחת על קרום ה-NC כדי לספק דפוס קבוע וניתן להתייחסות לאיתור וקיבוע של דגימות סרום. קיצורים: CFDB = כתם נקודות נטול תאים; NC = ניטרוצלולוז. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 3: ייצוג סכמטי של זרימת העבודה CFDB. בבדיקת CFDB, כמות קטנה (<0.4 מיקרוליטר) של דגימות סרום מדוללות פי 10 מופצת ידנית על קרום ניטרוצלולוזה חתוך מראש (פאנל שמאלי) במיקומים נפרדים וניתנים להתייחסות (פאנל אמצעי). הנחת דגימת סרום אחת לכל נקודה בכתמים משולשים וקיבוע תכולת החלבון, כולל מאגר הנוגדנים הכולל של הסרה, על מצע ה-NC המוצק (כתמים בז' בפאנל האמצעי). בדוגמה זו, נוגדנים נגד NP הכלולים בדגימות הסרום יכולים להיות קשורים תחילה על ידי מגיב הזיהוי הראשי של CFDB SpyTag-NP ולבסוף מזוהים על ידי מגיב הזיהוי המשני CFDB SpyCatcher2-Apex2 (בועה מוגדלת בפאנל הימני). נתון זה נלקח מ-Norouzi et al.5. קיצורים: CFDB = כתם נקודות נטול תאים; NP = חלבון נוקלאוקפסיד; LET = תבנית ביטוי ליניארית. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
תוצאות
הגברת PCR של תבנית הביטוי הליניארי עבור אנטיגן יעד
כדי להגביר את ה-PCR של SARS-CoV-2 NP LET, נעשה שימוש בפריימרים אוניברסליים של Ter קדימה ואחורה כמתואר בסעיף פרוטוקול 1.2 ו-1 מיקרוליטר של המוצר נבדק על ג'ל אגרוז (איור 4) לפני שהמשיכו לטיהור מוצר ה-PCR.

איור 4: PCR של תבנית ביטוי ליניארי. ג'ל אגרוז של 1% המופיע ב-Lane 1: 1 kb DNA Ladder ו-Lane 2: תבנית ה-DNA הליניארית המוגברת ב-PCR עבור His-SpyTag-SARS-CoV-2 NP (1,630 bp). קיצורים: PCR = תגובת שרשרת פולימראז; NP = חלבון נוקלאוקפסיד. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
טיהור חלבון המדווח SpyCatcher2-Apex2
החלבון SpyCatcher2-Apex2 הוכן כמתואר בסעיף פרוטוקול 2. כדי להבטיח שילוב הם הולם בפרוקסידאז Apex2, נוספו 5-aminolevulinic acid hydrochloride (מבשר heme) והמין כלוריד במהלך סינתזת החלבון ולאחר ליזיס התאים, בהתאמה. דגימות מכל שלבי הטיהור הועמסו על ג'ל SDS-PAGE כדי להבטיח את איכות המוצר הסופי (איור 5).
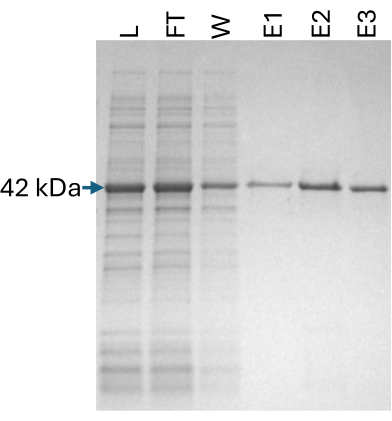
איור 5: טיהור SpyCatcher2-Apex2. ג'ל 12% SDS-PAGE המציג שברים משלבי הטיהור של חלבון SpyCatcher2-Apex2. L: ליזאט (2 מיקרוליטר), FT: זרימה (2 מיקרוליטר), W: שטיפה (10 מיקרוליטר), E1-3: פליטה 1-3 (10 מיקרוליטר) כל אחד. הרצועה הצפויה ב-42 kDa מסומנת. קיצור: SDS-PAGE = נתרן דודציל סולפט אלקטרופורזה של ג'ל פוליאקרילאמיד. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
ביטוי ללא תאים ואימות איכות של אנטיגן היעד
לאחר ביצוע ביטוי פיילוט בקנה מידה של 5 מיקרוליטר של חלבון NP ובדיקת איכות באמצעות SDS-PAGE, נערך ביטוי בקנה מידה של 1 מ"ל כמתואר בסעיף פרוטוקול 3. דגימה של 1 מיקרוליטר מתערובת הביטוי הסופית נטולת התאים נחשפה לכתם מערבי בנפרד לזיהוי על ידי SpyCatcher2-Apex2 ונוגדנים מסחריים נגד NP (איור 6). שלב זה מבטיח שהאנטיגן מתבטא בהצלחה ושהוא יכול להיקשר באופן ספציפי הן לאימונוגלובולינים נגד NP והן לחלבון המדווח SpyCatcher2-Apex2.
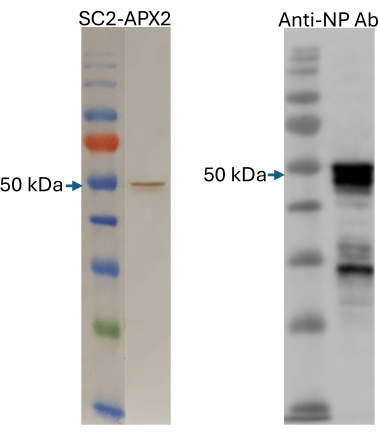
איור 6: בדיקת איכות של אנטיגנים המיוצרים ללא תאים. זיהוי כתמים מערביים של חלבון NP המיוצר ללא תאים באמצעות SpyCatcher2-Apex2 (פאנל שמאלי) ונוגדנים רב-שבטיים של ארנב אנטי-NP (פאנל ימני). המוצר הצפוי באורך מלא ב-50 kDa מסומן. סמן הגודל בשני הג'לים הוא תקן חלבון צבוע מראש, טווח רחב. איור זה אומץ מ-Norouzi et al.5. קיצור: NP = חלבון נוקלאוקפסיד. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
אימות ריאגנטים CFDB באמצעות סרום אנושי מאופיין מראש
כדי לאשר כי ריאגנטים של CFDB הם באיכות גבוהה וכי פרוטוקולי CFDB מיושמים בהצלחה, השתמשנו בפאנל סרום מאופיין מראש שסופק על ידי NISBC-פאנל הייחוס הבינלאומי של ארגון הבריאות העולמי לאימונוגלובולין נגד SARS-CoV-2 20/26813. פאנל זה מורכב מ-x1 דגימות טרום-COVID-19 (שליליות) ו-x4 SARS-CoV-2 חיוביות עם רמות שונות של תגובת נוגדנים (טבלה משלימה S2). רכשנו גם פאנל סרום מ-RayBiotech (CoV-PosSet) המכיל 10 דגימות טרום-COVID-19 (שליליות) ו-20 דגימות חיוביות ל-SARS-CoV-2. איגדנו שברים שווים מכל 10 הסרומים השליליים כדי להשתמש בהם כבקרה שלילית בכל הניסויים שבוצעו במעבדה שלנו. אסטרטגיית איגום זו מגבירה את הדיוק והחוסן של התוצאות מכיוון שדגימות שליליות בודדות עשויות להציג רמות עדינות של אות רקע שעלולות לסבך את פירוש התוצאות.
בדיקת CFDB בוצעה על פאנל הייחוס של ארגון הבריאות העולמי המאופיין מראש ועל CoV-PosSet של RayBiotech בהתאם לסעיף פרוטוקול 5 והכתם צולם. כלי עוצמת הקול בתוכנת Image Lab שימש לציור עיגולים אחידים בכל מיקום נקודה ולייצוא עוצמות אות בודדות. לאחר הפחתת עוצמת הרקע של ממברנת NC מכל הדגימות, ערך החיתוך ((ממוצע בקרות שליליות) + (3 x SD של הבקרות השליליות)) הופחת גם מעוצמת האות הממוצעת. דגימות נחשבות חיוביות נגד NP אם עוצמת האות שלהן יורדת מעל ערך החיתוך. תמונה של הכתם ותרשים עוצמת האות המתאים מוצגת באיור 7. התוצאות מדגימות כי באמצעות חלבון SARS-CoV-2 NP המיוצר ללא תאים, טכניקת CFDB יכולה לזהות נכון את כל הדגימות השליליות ואת כל הדגימות החיוביות, למעט שתי דגימות ממערך RayBiotech (RB P5 שנאסף 34 ימים לאחר הופעת התסמינים (dpso) ו-RB P10 שנאסף 4 dpso). בדיקה נוספת של גיליון הנתונים של הספק אישרה כי שתי הדגימות הללו נראות שליליות גם ל-anti-S1RBD (Spike Receptor Binding Domain) IgG על ידי ELISA ואנטי-N IgG/M על ידי בדיקת זרימה רוחבית (טבלה משלימה S3), מה שמרמז על היעדר רמות נוגדנים ניתנות לזיהוי בדגימות אלה.

איור 7: אימות CFDB באמצעות סרה אנושית מסחרית מאופיינת מראש. (A) תמונת כתם המייצגת CFDB NP נגד SARS-CoV-2 בפאנל הייחוס הבינלאומי של ארגון הבריאות העולמי ובדגימות סרום RayBiotech PosSet. הדגימות נצפו בשלוש עמודות וזוהו על ידי תוויות. (B) גרף שמייצג את ערכי עוצמת האות של הכימילומינסנציה הכמותית עבור הכתם בפאנל A. הבקרה השלילית שימשה לקביעת ערך החיתוך, וכל המדידות הן אמצעים של כתמים משולשים ± סטיות תקן (SD). נתון זה נלקח מנורוזי ואחרים.5. קיצורים: Neg Cont = דגימת בקרה שלילית שהתקבלה על ידי איגום כל 10 דגימות טרום COVID-19 מ-RayBiotech; RB N1 עד N10 = דגימות שליליות משוערות של RayBiotech 1 עד 10; RB P1 עד P20 = RayBiotech הניחה דגימות חיוביות 1 עד 20; CFDB = כתם נקודות נטול תאים; NP = חלבון נוקלאוקפסיד; ארגון הבריאות העולמי = ארגון הבריאות העולמי. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
בדיקה עצמאית של בדיקת CFDB באמצעות נסיוב מטופלים אנושיים
כדי להדגים את הישימות של בדיקת CFDB, סיפקנו חומרים והוראות CFDB למעבדה משתפת פעולה עצמאית במיקום אחר (המעבדה הלאומית למיקרוביולוגיה (NML), וויניפג). המטרה הייתה לבחון באופן עצמאי את הפלטפורמה באמצעות דגימות מטופלים אנושיים שנאספו במרפאה על ידי קבוצת מחקרי השדה ב-NML. בסך הכל, 24 דגימות (x12 שליליות ו-x12 חיוביות שאושרו על ידי ELISA לנוגדני NP (טבלה משלימה S4) נחשפו ל-CFDB והתוצאות נותחו כמתואר לעיל. כפי שניתן לראות באיור 8, CFDB זיהה נכון את כל הדגימות השליליות x12 והחיוביות x12, מה שמאשר את החוסן והמעשיות של הבדיקה בבדיקה עצמאית.

איור 8: בדיקה עצמאית של CFDB באמצעות נסיוב מטופלים אנושיים ב-NML. (A) תמונת כתם המייצגת CFDB NP נגד SARS-CoV-2 ב-12 דגימות חולים שליליות ל-COVID-19 (N1 עד N12) או 12 חיוביות (P1 עד P12). סרות נצפו בשלושה עמודים, כפי שמסומן. (B) ערכי עוצמת אות כימילומינסנציה מכומתים מהכתם בפאנל A. כל המדידות הן אמצעים של כתמים משולשים ± סטיות תקן (SD). נתון זה נלקח מנורוזי ואחרים.5. קיצורים: CFDB = כתם נקודות נטול תאים; NML = מעבדה לאומית למיקרוביולוגיה; NP = חלבון נוקלאוקפסיד. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
בדיקה עצמאית של בדיקת CFDB באמצעות נסיוב אוגרים נגוע ב-SARS-CoV-2
באמצעות אותם ריאגנטים והוראות CFDB, צוות ה-NML ביצע בדיקת CFDB על נסיוב אוגרים נגוע ב-SARS-CoV-2 כדי לבדוק אם ניתן להשתמש בבדיקה זו גם לאיתור תגובת נוגדנים בדגימות של בעלי חיים. בסך הכל נבדקו 36 דגימות, x12 לפני הדבקה ו-24 לאחר הדבקה. הנסיוב החיובי המשוער היה מבעלי חיים שנדבקו ב-105 50% מנות זיהומיות של תרבית רקמה (TCID50) של SARS-CoV-2 שנדגמו 140 יום לאחר ההדבקה (dpi 140, n = 3); או נדבק מחדש ב-DPI 140 ונדגם 5 ימים לאחר מכן (DPI 5, N = 21). כפי שמוצג באיור 9, לכל הסרומים שלפני ההדבקה היו עוצמות אות מתחת לערך החתך ו-18 מתוך 24 סריות לאחר ההדבקה (כולן מ-5 ימים לאחר ההדבקה החוזרת) עוררו עוצמות אות חיוביות נפרדות. מעניין לציין שמתוך שש הדגימות החיוביות המשוערות שהוחמצו על ידי CFDB, שלוש דגימות (כולן מ-dpi 140 הראשוני) לא היו ניתנות לזיהוי באמצעות אנטי-NP ELISA סטנדרטי (טבלה משלימה S5), מה שמצביע על אובדן או ירידה ברמות הנוגדנים לאורך זמן.
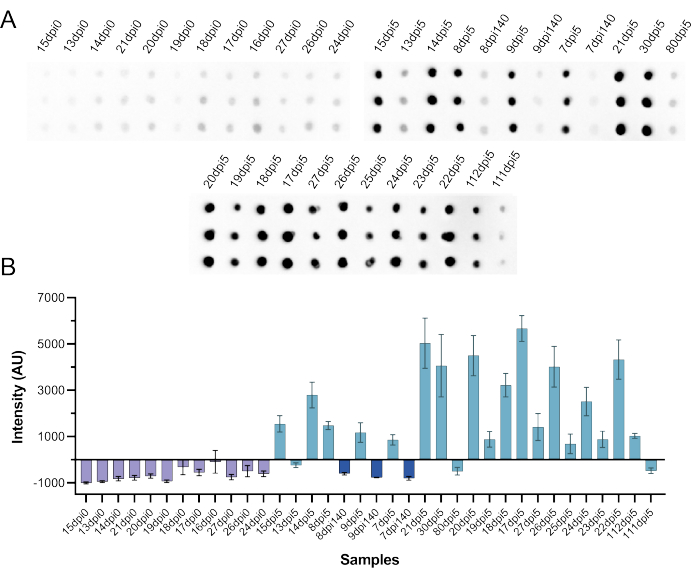
איור 9: בדיקה עצמאית של CFDB באמצעות נסיוב אוגרים נגוע ב-SARS-CoV-2 ב-NML. (A) תמונת כתם שמציגה CFDB נגד SARS-CoV-2 NP על הדבקה מוקדמת בנסיוב האוגר (dpi 0, n = 12), 140 ימים לאחר ההדבקה (dpi 140, n = 3), ו-5 ימים לאחר הדבקה חוזרת (dpi 5, n = 21) עם 105 TCID50 של SARS-CoV-2. כל דגימה הופקדה ככתמים משולשים לפי עמודות ותויגה בהתאם לתוכנית במחקר ה-NML המקורי. (B) ערכי עוצמת אות כימילומינסנציה מכומתים מהכתם בפאנל A. כל המדידות הן אמצעים של כתמים משולשים ± סטיות תקן (SD). נתון זה נלקח מנורוזי ואחרים.5. קיצורים: CFDB = כתם נקודות נטול תאים; NML = מעבדה לאומית למיקרוביולוגיה; NP = חלבון נוקלאוקפסיד; dpi = ימים לאחר ההדבקה; TCID50 = 50% מינון זיהומי בתרבית רקמה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
קובץ משלים 1: רצף נוקלאוטידים עבור תבנית הביטוי הליניארי His-SpyTag-SARS-CoV-2 NP. קיצור: NP = חלבון נוקלאוקפסיד. אנא לחץ כאן להורדת קובץ זה.
קובץ משלים 2: רצף נוקלאוטידים מלא של הפלסמיד pET24b SpyCatcher2-Apex2. אנא לחץ כאן להורדת קובץ זה.
קובץ משלים 3: סקירה כללית של השלבים העיקריים בהכנת ליזאט ביטוי חלבון נטול תאים E. coli BL21. אנא לחץ כאן להורדת קובץ זה.
איור משלים S1: בניית מפת פלסמיד pET24b SpyCatcher2-Apex2. אנא לחץ כאן להורדת קובץ זה.
איור משלים S2: עותק להדפסה של רשת האב CFDB. קיצור: CFDB = כתם נקודות נטול תאים. אנא לחץ כאן להורדת קובץ זה.
איור משלים S3: תמונה של מערך מכלול הממברנה CFDB NC ורשת האב. קרום ה- NC דחוס בין צלחת זכוכית בתחתית לתבנית רשת המאסטר בחלק העליון, ומאבטח את המכלול באמצעות סרט נייח. נתון זה נלקח מנורוזי ואחרים.5. קיצורים: CFDB = כתם נקודות נטול תאים; NC = ניטרוצלולוזה אנא לחץ כאן להורדת קובץ זה.
טבלה משלימה S1: מתכונים מלאים לרכיבי פתרון A ו-B של תערובת התגובה נטולת התאים. אנא לחץ כאן להורדת קובץ זה.
טבלה משלימה S2: מידע לדוגמה עבור פאנל ההתייחסות של NIBSC WHO 20/268 (ראה גם איור 7). קיצורים: NISBC = המכון הלאומי לתקנים ובקרה ביולוגיים; ארגון הבריאות העולמי = ארגון הבריאות העולמי. אנא לחץ כאן להורדת קובץ זה.
טבלה משלימה S3: מידע לדוגמה RayBiotech CoV-PosSet (ראה גם איור 7). אנא לחץ כאן להורדת קובץ זה.
טבלה משלימה S4: תוצאות NP ELISA עבור דגימות סרום של מטופלים אנושיים (בוצעו ב-NML). ראה גם איור 8. קיצורים: NP = חלבון נוקלאוקפסיד; ELISA = בדיקת אימונוסורבנט מקושרת לאנזים; NML = מעבדה לאומית למיקרוביולוגיה. אנא לחץ כאן להורדת קובץ זה.
טבלה משלימה S5: תוצאות NP ELISA עבור נסיוב אוגר נגוע ב-SARS-CoV-2 (מבוצע ב-NML). ראה גם איור 9. קיצורים: NP = חלבון נוקלאוקפסיד; ELISA = בדיקת אימונוסורבנט מקושרת לאנזים; NML = מעבדה לאומית למיקרוביולוגיה. אנא לחץ כאן להורדת קובץ זה.
Discussion
COVID-19 הדגיש את החשיבות של אבחון נגיש וחזק לשליטה בהתפרצויות זיהום ואופטימיזציה של אסטרטגיות בריאות גלובליות. בדיקות סרולוגיות המגלות נוגדנים מגנים הוכחו כחיוניות למעקב אחר דפוסי הדבקה של וריאנטים חדשים, זיהוי נקודות חמות, הנחיית פיתוח חיסונים, מיון מקרים חשודים והגנה על אוכלוסיות פגיעות14. המגפה חשפה גם אי שוויון בנגישות הבדיקות, שהחמיר על ידי צבר ודרישות למתקנים עם ציוד מתוחכם15. לכן ההתמקדות כעת היא בחיזוק התגובה הגלובלית שלנו להתפרצויות עתידיות, מה שידרוש חידושים חדשים שמפחיתים את מחסומי הבדיקה וניתן להתאים אותם במהירות לאיומים בלתי צפויים.
CFDB הוא מבחן חיסוני מבוסס נייר המפשט בדיקות סרולוגיות, המבוצעות באופן קונבנציונלי על ידי ELISA או מבחני זרימה רוחבית, על ידי קיבוע דגימות חולים בשלב המוצק ואספקת אנטיגנים המיוצרים ללא תאים בשלב נייד לזיהוי נוגדנים אנטי-ויראליים5. הבדיקה יכולה להתבצע בסביבות מעבדה בסיסיות עם יכולות כתמים מערביים. בינתיים, ניתן לייצר ריאגנטים ל-CFDB באופן מקומי במסגרות מחקר בסיסיות ולהחליף אותם בקלות כדי לייעד מחדש את הבדיקה לפתוגנים חדשים. מגיב הזיהוי האוניברסלי הכימרי SpyCatcher2-Apex2 הוא תכונה מאפשרת המייתרת את הצורך ברכישת מצומדי נוגדנים משניים מסחריים ומיוצרת ביעילות במערכת ביטוי E. coli . CFDB מספק גם יכולות תפוקה גבוהות המקבילות לבדיקות קליניות בשבריר מהעלות, ב-3 דולר בלבד לערכת בדיקה בהשוואה לכ-300 דולר עבור ELISA מסחרי של 96 בארות.
הגדרת ה-CFDB לבדיקות סרולוגיות של COVID-19 הושגה על ידי תכנון תבניות DNA ליניאריות שהורו למערכת תרגום השעתוק-תרגום נטולת התאים לייצר את אנטיגן המטרה, SARS-CoV-2 NP. לאחר מכן נצפו סרוולים של בני אדם ובעלי חיים על רשתות ממברנות הניטרוצלולוזה, והדגירה בוצעה עם תערובת ביטוי האנטיגן נטולת התאים ואחריה החלבון המדווח SpyCatcher2-Apex2. במחקר זה, CFDB בוצע על סרום מנבדקים שליליים וחיוביים ל-COVID-19, הן במעבדה שלנו והן בסביבה עצמאית (NML), ובסך הכל, הושג מתאם טוב עם ELISA להבחנה בין דגימות חיוביות לעומת שליליות. על ידי שימוש בסרום בריא מאוחד כבקרה שלילית CFDB וחישוב ערך חיתוך בדיקה על סמך דגימה זו, הסיכון לזיהוי שלילי/חיובי כוזב היה ממוזער.
רגישות גבוהה של בדיקת CFDB באה לידי ביטוי ביכולתה להבחין בבירור בין כל הדגימות בפאנל הייחוס של ארגון הבריאות העולמי 20/26813 של NIBSC, במיוחד המדגם עם טיטר הנוגדנים הנמוך ביותר (מדגם 20/140 נמוך, ראה טבלה משלימה S2 ואיור 7). באופן דומה, הספציפיות הגבוהה של CFDB מודגמת על ידי היעדר זיהוי חיובי כוזב עבור כל אחד מ-35 הנסיובים הבריאים שנבדקו בעבודה זו. חשוב לציין, אימות קליני מרובה אתרים אישר כי פרוטוקול CFDB פשוט מספיק לשימוש נרחב ושומר על ביצועים גבוהים, בהשוואה ל-ELISA סטנדרטיים, כאשר הוא מבוצע על ידי משתמשים שונים. מניסיוננו וכפי שהוכח על ידי אחרים16, האנטיגנים נטולי התאים המיוצרים בליזטים של E. coli יציבים לפחות 3 חודשים ב-20 מעלות צלזיוס, ותכשירי אצווה מרובים של אנטיגן נטול תאים מתפקדים ללא הבחנה זה מזה.
יש לקחת בחשבון מספר שיקולים לפני אימוץ פלטפורמת CFDB באופן נרחב וסיבוב תכנון המערכת לעבר פתוגנים חדשים. פלטפורמת ה-CFDB אכן דורשת שהאנטיגן הנגיפי או מקטע האנטיגן יסונתז כראוי על ידי מערכת ביטוי נטולת תאים, כך שחלקים אנטיגניים יהיו קיימים וזמינים לקשירת נוגדנים. לדוגמה, בתחילה התכוונו לכלול גם את תחום קשירת קולטן הספייק (RBD) של SARS-CoV-2 במבחני ה-CFDB שלנו. עם זאת, למרות ביטוי בעל תפוקה גבוהה ובגלל דרישתו לקישורי דיסולפיד מורכבים (בניגוד לאנטיגן NP שחסר חומצות אמינו ציסטאין), מוצר ה-RBD הצטבר בקלות ונראה בלתי מסיס בתמצית E. coli , מה שמנע את השימוש בו כבדיקת CFDB אמינה. בתרחישים כאלה שבהם נדרשים שינויים מורכבים לאחר התרגום לקיפול תקין ותפקוד אנטיגני, מומלץ להשתמש במערכות נטולות תאים מבוססות איקריוטיות. כמו בבדיקות אימונולוגיות אחרות המבוססות על פרוקסידאז, הכרחי גם שלא לכלול דגימות סרום עם המוליזה גלויה בשל הפוטנציאל לפעילות המוגלובין פרוקסידאז המבלבלת את התוצאות. לבסוף, ה-CFDB נועד לזהות את תגובת הנוגדנים הכוללת ואינו מדווח על קבוצת האימונוגלובולין הספציפית המעורבת באינטראקציות נוגדנים-אנטיגן.
לסיכום, ה-CFDB היא טכניקת אבחון חדשה הממזגת אלמנטים של ביולוגיה סינתטית נטולת תאים וכתמי נקודות כדי לייעל את הבדיקות הסרולוגיות. ה-CFDB מציע אלטרנטיבה חסכונית ל-ELISA קונבנציונליים שניתן לפרוס בקלות במעבדות מחקר ברחבי העולם כאשר יש צורך ביכולת בדיקה נוספת, כפי שקורה לעתים קרובות במהלך מגיפה. ה-CFDB יכול לשמש אפוא ככלי קדמי בתגובה העולמית הבאה למגיפה, שניתן להתאים במהירות לאיומים מתעוררים ויחזק את יכולות הבדיקה של מדינות בעלות הכנסה נמוכה ובינונית.
Disclosures
M.N. ו-K.P. הם ממציאים שותפים של שיטת כתמי הנקודות נטולות התאים. הוגשה בקשת פטנט זמנית הקשורה לעבודה זו (PCT/CA2024/050097, הוגשה בינואר 2024).
Acknowledgements
ס.ס. ור.ז. נתמכים על ידי מימון מהסוכנות לפרויקטי מחקר מתקדמים של ההגנה (DARPA), חוזה מס' N66001-23-2-4042. הדעות, הדעות ו/או הממצאים המובעים הם של המחברים ואין לפרש אותם כמייצגים את העמדות או המדיניות הרשמית של משרד ההגנה או ממשלת ארה"ב. עבודה זו נתמכה על ידי מימון ל-K.P. מתוכנית המענקים של קרן CIHR (201610FDN-375469), תוכנית קתדרת המחקר של CIHR קנדה (950-231075 ו-950-233107), יוזמת הרפואה של אוניברסיטת טורונטו, המקבלת מימון מקרן המצוינות במחקר הראשון של קנדה וכספים ל-K.P., מתוכנית המחקר והפיתוח הביטחוני הקנדית של קנדה (חוזה 39903-200137). איור 1 ואיור משלים S1 נוצרו באמצעות SnapGene Viewer.
Materials
| Name | Company | Catalog Number | Comments |
| 1 kb DNA ladder | NEB | N3232 | Used as a size marker for agarose gels |
| 20 Amino acids | Sigma-Aldrich | LAA21-1KT | A component of the cell-free reaction Solution B |
| 2 mm biopsy punch | Integra Miltex | 33-31-P/25 | For preparation of CFDB master grid |
| 5-aminolevulinic acid hydrochloride | Sigma-Aldrich | A3785 | Heme precursor for induction of SpyCatcher2-Apex2 |
| Agarose powder | BioShop | AGA002 | For electrophoretic analysis of DNA |
| Anti-SARS-CoV2-Nucleocapsid antibody | Sinobiological | 40588-T62 | For WB detection of cell-free produced NP antigen |
| Bio-Rad ChemiDoc XRS+ | Bio-Rad | N/A | Gel imager instrument |
| Centrifugal concentrators | Cytiva | 28-9323-60 | For concentration and buffer exchange of proteins |
| Centrifuge | Eppendorf | EP022628257 | For harvesting bacterial culture |
| Coenzyme A sodium salt hydrate (CoA) | Sigma-Aldrich | C3144 | A component of the cell-free reaction Solution A |
| Color pre-stained protein standard (broad range) | NEB | P7719 | Used as a size marker for SDS-PAGE gels |
| Covid-19 serum panel | RayBiotech | CoV-PosSet | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| D-(−)-3-Phosphoglyceric acid disodium salt (3-PGA) | Sigma-Aldrich | P8877 | A component of the cell-free reaction Solution B |
| Dithiothreitol (DTT) | Sigma-Aldrich | 10197777001 | Component of buffers |
| E. coli 5-alpha | NEB | C2987 | for plasmid preparation |
| E. coli BL21 | NEB | C2530 | For preparation of cell-free lysates |
| E. coli BL21 (DE3) | NEB | C2527 | For expression of SpyCatcher2-Apex2 |
| EDTA-free protease inhibitor tablet | Sigma-Aldrich | 11836153001 | A component of E. coli lysis buffer |
| Eppendorf New Brunswick Innova 43/43R Incubator Shaker | Eppendorf | EPM1320 | Incubator for growing E. coli cells for protein expression and cell-free lysate preparation |
| Folinic acid | Sigma-Aldrich | 47612 | A component of the cell-free reaction Solution A |
| Glycerol | Sigma-Aldrich | G9012 | Component of SpyCatcher2-Apex2 storage buffer |
| Hemin chloride | Sigma-Aldrich | H9039 | For Heme supplementation of SpyCatcher2-Apex2 |
| Hydrogen peroxide 30% | Sigma-Aldrich | H1009 | For making ECL reagent |
| Image Lab Software | Bio-Rad | 1709690 | Software for ChemiDoc gel imaging instrument |
| Isopropyl-b-D-1-thiogalactopyranoside | Bioshop | IPT001 | For induction of SpyCatcher2-Apex2 expressikon |
| Kanamycin Sulfate | Sigma-Aldrich | 60615 | For preparation of SpyCatcher2-Apex2 bacterial culture |
| LB agar | BioShop | LBL406 | For E. coli growth |
| LB broth | BioShop | LBL407 | For E. coli growth |
| luminol | Sigma-Aldrich | A4685 | For making ECL reagent |
| Lysozyme | Sigma-Aldrich | L6876 | For lysis of bacterial cells |
| Magnesium Acetate | Sigma-Aldrich | M5661 | A component of the cell-free reaction Solution B |
| NEBExpress Ni resin | NEB | S1428S | For purification of SpyCatcher2-Apex2 |
| Non-fat dry milk | Bioshop | SKI400 | For blocking of WB and CFDB membranes |
| Parafilm | Bemis | 2099-1337410 | For ECL-incubation of CFDB blots |
| p-coumaric acid | Sigma-Aldrich | C9008 | For making ECL reagent |
| pET24b-SpyCatcher2-Apex2 plasmid | Pardee Laboratory | N/A | Used for the expression of SpyCatcher2-Apex2 protein |
| Petri dish | Fisherbrand | FB0875712 | Container for WB and CFDB membrane incubation |
| Phosphate buffered saline 10% | BioShop | PBS405 | Component of buffers |
| Potassium Glutamate | Sigma-Aldrich | G1501 | A component of the cell-free reaction Solution B |
| Potassium Oxalate Monohydrate (Oxalic acid) | Sigma-Aldrich | 223425 | A component of the cell-free reaction Solution A |
| Precast SDS-PAGE gel | BioRad | 4561036EDU | For electrophoretic analysis of protein |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification of LETs |
| QIAquick PCR purification kit | Qiagen | 28106 | For purification of LETs |
| Ribonucleotide Solution Set | NEB | N0450S | A component of the cell-free reaction Solution A |
| Skim milk powder | BioShop | SKI400 | Used as a blocking agent for western blot and CFDB |
| Sodium Chloride | Sigma-Aldrich | S9625 | A component of protein purification buffers |
| Sonicator | Qsonica | Q500 | For lysis of bacterial cells |
| Spermidine | Sigma-Aldrich | S2626 | A component of the cell-free reaction Solution A |
| Syringe filter | Sigma-Aldrich | SLGSR33SS | For sterilisation of buffers and solutions |
| Thermocycler | BioRad | T100 | Instrument for incubating PCR reactions |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8759 | A component of the cell-free reaction Solution A |
| Tris buffered saline | Thermo Scientific | J60764.K2 | Component of WB and CFDB wash buffer |
| Trizma base | Sigma-Aldrich | T1503 | For making tris buffer |
| Tunair SS-5012 Half-Baffle Shake Flask, 2.5 L | Cole-Parmer | RK-01835-39 | Vessel for growing E. coli cells for protein expression and cell-free lysate preparation |
| Tween-20 | BioShop | TWN510 | Component of WB and CFDB wash buffer |
| Tweezer | Almedic | 7728-A10-100 | For handling CFDB NC membrane |
| UV-Vis spectrophotometer | Thermo Scientific | 13400518 | Used for the measurement of nucleic acid and DNA concentration and bacterial culture OD |
| WHO International Reference Panel for anti-SARS-CoV-2 immunoglubulin | NISBC | 20/268 | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| β-Nicotinamide adenine dinucleotide hydrate (NAD) | Sigma-Aldrich | 10127965001 | A component of the cell-free reaction Solution A |
References
- Carter, L. J., et al. Assay techniques and test development for COVID-19 diagnosis. ACS Cent Sci. 6, 591-605 (2020).
- Amanat, F., et al. A serological assay to detect SARS-CoV-2 seroconversion in humans. Nat Med. 26 (7), 1033-1036 (2020).
- Stadlbauer, D., et al. SARS-CoV-2 seroconversion in humans: A detailed protocol for a serological assay, antigen production, and test setup. Curr Protoc Microbiol. 57 (1), e100(2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259.e12 (2016).
- Norouzi, M., et al. Cell-free dot blot: an ultra-low-cost and practical immunoassay platform for detection of anti-SARS-CoV-2 antibodies in human and animal sera. Microbiol Spectr. 11 (2), e0245722(2023).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Norouzi, M., Panfilov, S., Pardee, K. High-efficiency protection of linear DNA in cell-free extracts from Escherichia coli and Vibrio natriegens. ACS Synth Biol. 10 (7), 1615-1624 (2021).
- IDT codon optimization tool. , Integrated DNA Technologies. https://www.idtdna.com/pages/tools/codon-optimization-tool (2025).
- General protocol for western blotting. , Bio-Rad. https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6376.pdf (2025).
- Mruk, D. D., Cheng, C. Y. Enhanced chemiluminescence (ECL) for routine immunoblotting. Spermatogenesis. 1, 121-122 (2011).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-based cell-free protein synthesis: Protocols for a robust, flexible, and accessible platform technology. J Vis Exp. (144), (2019).
- Plasma and serum preparation. , ThermoFisher Scientific. https://www.thermofisher.com/ca/en/home/references/protocols/cell-and-tissue-analysis/elisa-protocol/elisa-sample-preparation-protocols/plasma-and-serum-preparation.html (2025).
- Mattiuzzo, G., et al. WHO/BS.2020.2403 Establishment of the WHO International Standard and Reference Panel for anti-SARS-CoV-2 antibody. , (2020).
- Gong, F., Wei, H., Li, Q., Liu, L., Li, B. Evaluation and comparison of serological methods for COVID-19 diagnosis. Front Mol Biosci. 8, 682405(2021).
- Peeling, R. W., et al. Serology testing in the COVID-19 pandemic response. Lancet Infect Dis. 20 (9), e245-e249 (2020).
- Smith, M. T., Berkheimer, S. D., Werner, C. J., Bundy, B. C. Lyophilized Escherichia coli-based cell-free systems for robust, high-density, long-term storage. Biotechniques. 56 (4), 186-193 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved