Method Article
Análise abrangente da resposta a medicamentos usando o ensaio FLICK
Neste Artigo
Resumo
Este protocolo descreve como usar o ensaio FLICK para avaliar as respostas a medicamentos, incluindo instruções detalhadas para usar este ensaio para calcular as taxas de crescimento e mortalidade induzidas por drogas e avaliar o mecanismo de morte celular induzida por drogas.
Resumo
Para entender a eficácia do medicamento, uma necessidade crítica é caracterizar a extensão da morte celular induzida por medicamentos. Os esforços para quantificar o nível de morte celular induzida por drogas são desafiados pela existência de mais de uma dúzia de formas molecularmente distintas de morte regulada, cada uma com seu próprio tempo de ativação e características bioquímicas. Além disso, para alguns subtipos de morte necrótica, as características marcantes são observadas apenas transitoriamente e são rapidamente perdidas devido à ruptura celular. Assim, mesmo ao usar uma combinação de ensaios específicos da via de morte, é um desafio quantificar com precisão a quantidade total de morte celular ou as contribuições relativas de cada subtipo de morte. Outra questão é que muitos ensaios específicos de morte ignoram como as drogas afetam a proliferação celular, tornando difícil interpretar se uma população tratada com drogas está se expandindo ou diminuindo. O ensaio FLICK permite quantificar o nível total de morte celular após estimulação de uma maneira específica para a morte, mas também amplamente agnóstica para o(s) tipo(s) de morte ativado(s). Além disso, o ensaio FLICK retém informações sobre o tamanho total da população e a taxa de proliferação celular. Neste manuscrito, descrevemos o uso básico do ensaio FLICK, como solucionar problemas desse ensaio ao usar diferentes tipos de material biológico e como usar o ensaio FLICK para quantificar as contribuições de cada tipo de morte celular para uma resposta a medicamentos observada.
Introdução
Para medicamentos anticâncer, a avaliação pré-clínica da sensibilidade aos medicamentos geralmente envolve testar como os medicamentos afetam a viabilidade das células em cultura1. A viabilidade celular após a exposição ao medicamento é um produto de pelo menos dois efeitos separados: inibição da proliferação celular induzida por drogas e ativação da morte celular2. Infelizmente, embora a morte celular seja uma característica crítica necessária para respostas duráveis a medicamentos, as abordagens padrão não esclarecem o grau em que um medicamento ativa a morte celular3.
Os ensaios comuns de resposta a medicamentos incluem aqueles que contam diretamente as células (por exemplo, Coulter Counter, alguns usos de citometria de fluxo ou microscopia), quantificam a capacidade das células de proliferar (por exemplo, ensaio de formação de colônias) ou quantificam uma atividade metabólica (por exemplo, CellTiter-Glo, ensaios MTT ou MTS baseados em tetrazólio). Uma característica compartilhada desses ensaios é que os dados gerados são proporcionais ao número de células vivas. Como as drogas variam consideravelmente na forma como coordenam a inibição do crescimento e a morte celular, o número de células vivas após a exposição à droga fornece uma visão não confiável do nível de morte celular induzida por drogas3. Além disso, como as células cancerígenas geralmente proliferam rapidamente na cultura de células, o número de células vivas pode ser drasticamente reduzido em relação à população não tratada sem induzir qualquer morte celular4. Assim, uma falha central é que o grau de morte celular não pode ser quantificado sem medir o número de células vivas e mortas.
Quantificar o número de células mortas após a exposição ao medicamento é um desafio para fazer com precisão por vários motivos2. Primeiro, existem mais de uma dúzia de vias de morte celular regulamentadas 5,6,7,8. Embora geralmente existam marcadores bioquímicos para identificar cada tipo de morte celular regulada, esses marcadores variam em sua especificidade, e nenhum ensaio único pode ser usado para quantificar simultaneamente todos os subtipos de morte. Em segundo lugar, o momento da ativação para cada forma de morte celular pode variar drasticamente dependendo do contexto, de modo que um quadro completo não pode emergir a menos que a morte seja quantificada ao longo do tempo 9,10. Muitos ensaios bioquímicos para quantificar a morte celular produzem uma medição de ponto final, portanto, a geração de dados cinéticos pode ser desafiadora e limitada pelo custo. Uma terceira complicação é que a própria célula morta é um estado intermediário transitório entre o estado da célula viva e os detritos de células dissociadas. A estabilidade das células mortas varia dependendo do subtipo de morte, com alguns tipos, como a apoptose, criando cadáveres relativamente estáveis, enquanto outros tipos de morte causam lise rápida. Assim, métodos de quantificação da morte que requerem coleta e contagem de células mortas também produzirão uma compreensão tendenciosa da morte celular. Finalmente, uma quarta limitação é que os ensaios bioquímicos que quantificam o grau de morte celular normalmente não fornecem informações sobre como uma droga altera a proliferação. Assim, o tamanho geral da população - e, mais importante, se a população está se expandindo ou diminuindo - não pode ser interpretado.
Alguns ensaios baseados em microscopia, como STACK e SPARKL, são eficazes na medição de células vivas e mortas ao longo do tempo, e esses ensaios podem produzir informações abrangentes sobre a morte celular induzida por drogas10 , 11 . Esses ensaios, no entanto, requerem instrumentos especializados, como o microscópio Incucyte, criando limitações no rendimento e no acesso a essas abordagens. Além disso, as técnicas baseadas em microscopia exigem que as células mortas permaneçam no plano focal do microscópio durante toda a duração do experimento, comprometendo a capacidade de quantificar as células mortas quando elas perdem a aderência da placa ou ao longo do tempo à medida que as células mortas se decompõem. Da mesma forma, os ensaios baseados em microscopia enfrentam desafios quando aplicados no contexto de culturas em suspensão, pois as células entram e saem de um determinado plano focal.
Para resolver os problemas destacados acima, geramos um ensaio chamado FLICK (Fluorescência baseada em e Inferência dependente de Lise da Cinética de Morte Celular) 12 , 13 . O objetivo do ensaio FLICK é determinar o nível de morte celular induzida por drogas, independentemente de como as células estão morrendo. O método FLICK usa corantes impermeáveis de células, cuja fluorescência depende da ligação ao DNA. Uma característica fundamental do FLICK é o uso desses fluoróforos para rotular o acúmulo de células mortas ao longo do tempo em virtude de seu DNA acessível, seguido por uma lise mecânica à base de detergente para permeabilizar quaisquer células vivas no final do ensaio. Esses dados, combinados com modelagem matemática, permitem a quantificação de populações de células vivas e mortas com resolução temporal contínua e sem exigir a coleta ou manuseio de células mortas. Além disso, o uso de um leitor de placas para avaliar a fluorescência de células mortas permite a avaliação de células mortas sem exigir que as células mortas permaneçam intactas, aliviando assim o viés contra formas necróticas de morte que resultam em ruptura celular. Finalmente, o ensaio FLICK requer manuseio mínimo da placa e pode gerar rapidamente medições cinéticas, permitindo a triagem de medicamentos de alto rendimento. Neste protocolo, nos concentramos no uso do ensaio FLICK, incluindo como usar o FLICK para inferir a taxa de crescimento induzida por drogas, taxa de mortalidade e/ou os mecanismos de morte celular.
Protocolo
1. Otimização do tempo de permeabilização para cada linhagem celular de interesse
NOTA: Os volumes e quantidades descritos são para otimizar uma linha celular. Esses valores devem ser ampliados com base no número de linhagens celulares que devem ser testadas.
- Coloque o número desejado de células em cada poço de uma placa de parede preta com fundo óptico de 96 poços. Adicione 100 μL de meio completo e deixe as células aderirem à placa durante a noite.
NOTA: O número de células plaqueadas deve ser otimizado levando em consideração a taxa de crescimento das células, densidades ideais e comprimento do ensaio. Os números iniciais típicos de células para linhagens de células cancerígenas aderentes medidos ao longo de 72 h são de 1500 a 5000 células por poço. Por exemplo, as células U2OS podem ser plaqueadas a 2000 células por poço em DMEM com 10% de FBS, 2 mM de glutamina e 1% de Pen-Strep e cultivadas durante a noite em condições padrão (5% de CO2, 37 ° C, com umidade). - Em um tubo de centrífuga cônico de 15 mL, prepare 1,5 mL de uma solução a 1,5% de Triton-X em solução salina tamponada com fosfato (PBS). Vortex a solução Triton-X a 1,5% por 5 s na velocidade máxima. Colocar em banho-maria a 37 °C durante, pelo menos, 30 min.
- Inspecione visualmente a solução para garantir que o Triton-X tenha se dissolvido totalmente por vórtice por vários segundos. Certifique-se de que a solução seja homogênea.
- Adicione 10 μL da solução de Triton-X a 1,5% a cada poço das células plaqueadas. Não misture.
NOTA: Nesta fase, não é desejável misturar usando pipetagem repetida devido à formação de bolhas. - Retorne as células ao CO2 a 5%, 37 °C com incubadora de cultura de células de umidade por pelo menos 1 h.
NOTA: Após a aplicação da solução Triton-X, a maioria das linhagens celulares é totalmente lisada em 2-3 h. Esperar até 24 h tem impacto mínimo no sinal de fluorescência ao usar corantes como SYTOX Green. Aumentar a porcentagem de Triton-X pode encurtar o tempo de incubação de linhagens celulares difíceis de lisar. - Observe as morfologias celulares em um microscópio óptico usando uma objetiva de 10x. Inspecione as células uma vez a cada hora até que os corpos celulares não estejam mais visíveis. Registre o tempo necessário para a permeabilização celular.
NOTA: Se um microscópio fluorescente estiver disponível, a lise pode ser visualizada usando um corante de DNA impermeável a células, como o SYTOX Green. O corante pode ser adicionado em uma concentração final de 10x à solução Triton-X, e a permeabilização pode ser confirmada por microscopia de fluorescência.
2. Seleção e calibração da coloração de DNA
NOTA: Um requisito para o ensaio FLICK é o uso de um fluoróforo impermeável de célula que emite um sinal de maneira dependente da ligação ao DNA, não afeta a viabilidade celular e produz um sinal que escala linearmente com o número de células. Este protocolo usa SYTOX Green. Outros corantes com propriedades semelhantes também podem ser adequados para o ensaio FLICK, mas cada um deles deve ser avaliado e calibrado. Veja a Tabela 1 para exemplos.
- Para o corante de DNA selecionado, determine a faixa de concentrações a serem testadas com base na recomendação do fabricante.
- Para cada concentração a ser testada, coloque 40.000 células em 180 μL de meio de cultura celular em triplicado ao longo da coluna mais à esquerda de uma placa de parede preta de fundo óptico de 96 poços. Adicione 90 μL de meio aos poços restantes.
NOTA: Isso será usado para gerar uma titulação de célula linear nas linhas da placa. A concentração final dos primeiros poços será de 20.000 células/poço. As placas ópticas inferiores não são necessárias para leitores de placas que leem fluorescência do topo do poço. O meio de cultura celular deve ser o mesmo meio usado para o crescimento das células que estão sendo testadas. Por exemplo, se estiver usando células U2OS, 40.000 células devem ser ressuspensas em 180 μL de DMEM. - Usando uma pipeta multicanal, crie uma diluição serial de 1:2 transferindo 90 μL da coluna mais à esquerda para a coluna adjacente à direita. Pipete 15x para misturar.
- Repita a etapa 2.3, passando da segunda coluna para a terceira coluna, depois da terceira coluna para a quarta coluna e assim por diante. Termine a titulação na penúltima coluna da placa. Deixar a última coluna sem células para que se possa obter o sinal de fundo dos meios de cultura celular.
- Para a penúltima coluna, remova 90 μL para que todos os poços tenham 90 μL de meio contendo um número variado de células. Deixe as células aderirem por 6 h em uma incubadora de cultura de células a 37 ° C.
- Prepare uma solução 10x de corante de DNA para cada concentração de corante a ser testada, com o corante de DNA diluído em meio de crescimento completo usado para cultivar as células de interesse. Prepare 1,5 mL de uma solução de Triton-X a 1,5% em PBS, conforme descrito na etapa 1.
- Para cada poço da placa de 96 poços, adicione 10 μL do corante de DNA 10x. Adicione 10 μL de solução Triton-X a 1,5% a cada poço para permeabilizar as células. Incube as células nesta solução pelo tempo ideal, que foi determinado na etapa 1.
NOTA: Após a adição de Triton-X, a concentração final de corante de DNA é ligeiramente inferior a 1x, mas isso é irrelevante. Após a incubação, toda a placa deve ser lisada. A titulação celular manual será usada para identificar a concentração ideal de corante de DNA e as configurações ideais para adquirir fluorescência. - Meça as intensidades de fluorescência na placa de titulação da célula. Quantifique a fluorescência em uma variedade de configurações de aquisição, conforme descrito pelo fabricante do corante de DNA. Dependendo do leitor de placas usado, modifique os comprimentos de onda de excitação e emissão e/ou o ganho digital.
- Remova o sinal de fundo de cada medição subtraindo o sinal médio da coluna que não contém células. Esses valores podem ser encontrados na coluna mais à direita dos poços na placa de diluição seriada.
- Determine a linearidade de cada concentração de corante de DNA para cada configuração de aquisição. A linearidade pode ser determinada representando graficamente o número da célula em relação ao sinal de fluorescência e realizando regressão linear. O coeficiente de determinação (r2) indica o grau em que o sinal de fluorescência está linearmente relacionado com o número de células.
- Escolha as configurações de aquisição e a concentração do corante de DNA que tem a melhor combinação de linearidade e faixa dinâmica.
NOTA: Considere todo o intervalo de números de células de 0 a 20.000 células. Certifique-se de que o ganho e a concentração do corante de DNA sejam lineares em números de células baixos, entre 0 e 1000 células. Várias configurações do instrumento ou concentrações de corante de DNA podem fornecer uma forte correlação em toda a faixa. No entanto, é essencial que a sensibilidade de baixo custo seja robusta, de modo que pequenas alterações nas células mortas possam ser medidas com precisão.
3. Revestimento celular, aplicação de medicamentos em poços e medição de fluorescência de células mortas ao longo do tempo em placas tratadas com medicamentos
NOTA: As placas de diluição de medicamentos podem ser projetadas de forma flexível com base nas necessidades experimentais. Geralmente, as placas de diluição de medicamentos incluem uma série de diluição logarítmica ou semilogarítmica de um ou vários medicamentos.
- Considere o layout de revestimento ideal para o experimento. Evite medições dos poços externos de uma placa de 96 poços para diminuir o ruído associado à variação de crescimento, pois os poços externos experimentam diferentes temperaturas, oxigenação e evaporação em comparação com os poços internos da placa.
NOTA: A inclusão de controles em toda a paisagem da placa aumentará a robustez dos dados de crescimento. Considere incluir poços de controle no centro e nas bordas da placa. - Determine o número de placas necessárias para o experimento. Inclua uma placa adicional a ser lisada no início do ensaio para estabelecer o número médio de células por alvéolo no momento da droga (ou seja, a placa de controle T0).
- Prepare uma suspensão celular em meio de crescimento completo.
- Com base no número de placas necessárias para o experimento (calculado na etapa 3.2), determine o volume da suspensão celular necessária. Se semear placas a 90 μL por poço, cerca de 10 mL são necessários por placa. Multiplique o número de placas necessárias por 10 mL.
NOTA: É desejável aumentar este volume para levar em conta o volume morto do reservatório e a perda de volume durante a pipetagem. Por exemplo, o volume calculado pode ser multiplicado por 1,2. - Conte as células e prepare uma diluição celular com base no número desejado de células por poço. Por exemplo, se pretende semear 2000 células em 90 μL de meio por poço de uma placa de 96 poços, para 3 placas totais, calcule o número total de células necessárias para o experimento conforme abaixo:
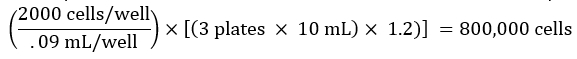
Da mesma forma, se a suspensão celular contada for de 400.000 células/mL, o volume da suspensão celular necessário para o experimento seria:
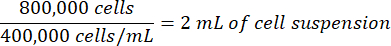
- Corrija o volume do meio de cultura celular usado para o plaqueamento, subtraindo o volume da suspensão de células contada a ser adicionada.
36 mL de mídia - 2 mL de suspensão celular = 34 mL de volume de mídia corrigido
- Com base no número de placas necessárias para o experimento (calculado na etapa 3.2), determine o volume da suspensão celular necessária. Se semear placas a 90 μL por poço, cerca de 10 mL são necessários por placa. Multiplique o número de placas necessárias por 10 mL.
- Misturar a suspensão de células contadas com o volume de meio correcto utilizando uma pipeta serológica. Transferir esta suspensão celular para um reservatório de reagente de fundo em V.
- Usando uma pipeta multicanal, adicione 90 μL da suspensão celular a cada poço das placas de 96 poços. Misturar regularmente a suspensão celular no reservatório do reagente, utilizando pipetagem repetida com uma pipeta serológica, a fim de garantir a manutenção da concentração desejada de células por volume.
- Deixe as células aderirem durante a noite em uma incubadora de cultura de células a 37 ° C (geralmente 12-24 h).
- Prepare uma solução de Triton-X a 1,5% em PBS, conforme descrito na etapa 1. O volume desta solução deve ser suficiente para lisar todas as placas. Cerca de 1,5 mL de Triton-X a 1,5% em PBS é suficiente para lisar uma placa de 96 poços.
NOTA: A solução de Triton-X a 1,5% em PBS é estável por mais de uma semana e pode ser feita com antecedência por conveniência. - Determine a quantidade de meios de cultura de células necessários para fazer uma placa de diluição de medicamentos. Prepare placas de diluição de medicamentos em placas transparentes de 96 poços com fundo em U ou V.
- Para garantir que o volume do medicamento diluído seja suficiente para o experimento, aumente o volume mínimo necessário para levar em conta a perda de volume durante a pipetagem. Por exemplo, se os medicamentos forem adicionados em 10 μL a cada poço de uma placa de 96 poços e o experimento incluir três placas, serão necessários 30 μL de medicamento diluído por poço. Prepare 40-50 μL de mídia drogada por poço para compensar a perda de volume. Adicione 1,5 mL ao volume de mídia necessário para contabilizar a mídia necessária para a placa de controle T0.
- Prepare uma concentração de 10x do corante de DNA selecionado em meios de crescimento completos. Esta concentração baseia-se na concentração seleccionada no passo 2.11. O volume total desta solução é calculado no passo 3.8.
- Usando o corante de DNA + meio de crescimento da etapa 3.9, prepare uma concentração de 10x de cada medicamento a ser testado. Crie apenas a dose mais alta de cada medicamento na placa de diluição do medicamento e dilua os medicamentos em série com uma pipeta multicanal, misturando 15x entre cada poço.
NOTA: Ao fazer a dose mais alta para um determinado medicamento na placa de diluição do medicamento, o volume do medicamento deve ser contabilizado para garantir que a concentração seja precisa. - Usando uma pipeta multicanal, adicione 10 μL de medicamento + solução de corante de DNA da etapa 3.10 às placas que contêm as células.
NOTA: Para placas de diluição de medicamentos nas quais uma diluição em série foi criada, se os medicamentos forem adicionados às células começando com a concentração mais baixa do medicamento e trabalhando até a concentração mais alta, as pontas da pipeta multicanal não precisam ser alteradas. No entanto, as ponteiras da pipeta multicanal devem ser trocadas entre cada placa e sempre que as ponteiras forem utilizadas em um poço contendo um medicamento diferente ou uma concentração mais alta do fármaco. - Leia a fluorescência de todas as placas nas quais os medicamentos foram adicionados. Esta leitura de fluorescência é a leitura T0 (ou seja, células mortas no Tempo = 0 h).
- Retorne as placas para a incubadora após fazer a leitura de fluorescência T0.
- Adicione 10 μL da solução de corante de DNA 10x criada na etapa 3.9 e 10 μL da solução Triton-X a 1,5% na etapa 3.7 à placa de controle T0. Retorne esta placa à incubadora pelo tempo selecionado na etapa 1.8.
- Leia a placa de controle T0 assim que as células estiverem totalmente lisadas, conforme descrito na etapa 1.6.
4. Meça a fluorescência de células mortas ao longo do tempo para placas tratadas com medicamentos
NOTA: Minimize as placas de tempo fora da incubadora. Mudanças prolongadas na temperatura podem afetar a viabilidade celular, e a exposição à luz pode comprometer os fluoróforos de DNA, como o SYTOX Green.
- Adquira leituras de fluorescência para todas as placas tratadas com medicamentos a cada 3-4 h após a droga. As placas não precisam ser lidas durante a noite, a menos que seja necessária uma cinética de morte muito precisa.
NOTA: Os pontos de tempo não precisam ser feitos em intervalos fixos/padrão. Em geral, os pontos de tempo podem ser obtidos exclusivamente durante o horário normal de trabalho, sem causar erros na análise a jusante da cinética da morte. A cinética pode ser inferida desde que algumas medidas sejam feitas no período anterior ao início da morte, durante a fase crescente da morte e durante a fase de saturação/platô. - No ponto de tempo final desejado, adquira uma leitura de fluorescência. Imediatamente depois, adicione 10 μL da solução de Triton-X a 1,5% preparada na etapa 3.7 para lisar as células.
- Retorne as placas para a incubadora e deixe as células permeabilizarem pelo tempo determinado na etapa 1.
- Adquira leituras de fluorescência após a permeabilização. Este valor de fluorescência é proporcional ao número total de células (ou seja, células vivas + mortas) para cada poço.
5. Calcule a cinética da fração letal
NOTA: Os cálculos descritos neste protocolo podem ser analisados em qualquer formato ou software. No entanto, o uso de um ambiente de programação como MATLAB, R ou Python permitirá uma análise mais rápida e flexível.
- Calcule os valores médios de fluorescência da placa de controle T0 usando a média aparada de 50%. Este valor é proporcional ao número total de células antes da droga.
- Usando o ajuste da curva e uma função de crescimento exponencial, calcule a cinética do crescimento populacional para todos os poços do experimento. O tamanho inicial da população para cada poço é o valor médio de fluorescência T0 calculado na etapa 5.1. O tamanho final da população para cada poço é o valor pós-permeabilização calculado na etapa 4.4. A duração do ensaio é de 0 h até ao ponto final do ensaio selecionado no passo 4.4.
- Usando os parâmetros de crescimento determinados na etapa 5.2, calcule o número total de células em cada ponto de tempo medido no ensaio para cada poço. Determine o número de células vivas em cada ponto de tempo medido subtraindo a medição de células mortas do total de células calculado na etapa 5.2.
NOTA: Devido a pequenas quantidades de ruído no ensaio, ocasionalmente, o número de células vivas pode ser um pequeno número negativo. Isso pode ser definido manualmente como 0, pois não pode haver um número negativo de células vivas e provavelmente indica que todas as células desse poço estavam mortas. - Calcule a fração letal (LF) para cada ponto de tempo dividindo o sinal de fluorescência de células mortas pelo sinal total de células para cada ponto de tempo.
- Ajuste uma equação de Morte Exponencial de Lag (LED) aos dados do curso de tempo de fração letal10. Para evitar parâmetros cinéticos arbitrários para doses de um medicamento que não induzem letalidade significativa, ajuste para um modelo linear com inclinação igual a 0. Determine níveis significativos de letalidade com base no ruído no ensaio ou no LF observado para condições não drogadas.
- Registre o tempo de início da morte (DO) da equação do LED. A equação LED tem quatro parâmetros: o LF inicial (LFi), o LF no platô (LFp), a taxa de mortalidade inicial (DR) e o DO. Inferir esses parâmetros a partir dos dados usando uma regressão não linear (ou seja, ajuste de curva).
- Calcule a viabilidade fracionada (FV) no ponto final do ensaio para cada medicamento em cada dose. Subtraia o valor LF do ponto final de 1 ou divida o número de células vivas pelo total de células.
NOTA: O FV pode ser calculado a qualquer momento, não apenas no ponto final do ensaio. Esses dados são usados para avaliar a farmacologia da dose (por exemplo, medicamento IC50 ou EC50).
6. Calcule o valor de GR
- Determine o número médio de células vivas para cada poço no início do ensaio. Calcule esse valor usando a leitura T0 pós-permeabilização calculada na etapa 3.15 e subtraindo a leitura T0 para cada poço, que foi coletada na etapa 3.12. Na equação GR abaixo (etapa 6.3), esse valor é referido como x0.
NOTA: Ocasionalmente, a dose mais alta de um medicamento pode interferir na leitura de fluorescência do corante de DNA. Pode ser desejável calcular o número médio de células vivas no tempo 0 usando os poços de controle. - Para os alvéolos de controle (xctrl), determine o número médio de células vivas para cada poço no ponto final do ensaio. Determine o número médio de células vivas no ponto final do ensaio para cada condição tratada com medicamento [x(c)].
NOTA: Dependendo da variação no crescimento celular na placa, diferentes valores dex ctrl podem ser usados para normalização de cada poço. Por exemplo, se os valores de xctrl variam sistematicamente na placa, pode ser desejável usar o valor xctrl mais próximo em vez do valor médio xctrl da placa. - Para cada poço tratado com medicamento, calcule o valor de inibição da taxa de crescimento normalizado (GR) usando a seguinte equação4:
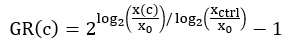
Execute o ajuste da curva usando uma regressão logística de 4 parâmetros. GR está em uma escala de -1 a 1.
7. Calcule o crescimento induzido por drogas e as taxas de mortalidade usando o método GRADE
NOTA: GR representa a taxa líquida de crescimento populacional, não a verdadeira taxa de proliferação celular. O crescimento populacional induzido por drogas e as taxas de mortalidade podem ser calculados usando uma combinação do GR e da fração letal (LF).
- Determinar o tempo de duplicação (τ) da linhagem celular durante a experiência, utilizando o número médio de células vivas da leitura de controlo T0 (x0), o número médio de células vivas da condição de controlo no ponto final do ensaio (xctrl) e a duração do ensaio em horas (t) para resolver a seguinte equação:

NOTA: O cálculo preciso do tempo de duplicação da célula (τ) requer que as células dobrem continuamente ao longo do comprimento do ensaio. Para células que experimentam inibição de contato, as células devem ser semeadas em densidades iniciais que não se tornarão confluentes durante o tempo de ensaio desejado. Por exemplo, o U2OS tem um tempo médio de duplicação de 24 h. A semeadura de 2000 células por poço em um ensaio de 72 h é ideal para interpretar sua taxa de crescimento. - Determine a relação entre as taxas de crescimento/mortalidade e o tamanho da população. Para fazer isso, use um modelo de nascimento/morte e uma simulação inicializada com todas as combinações plausíveis de taxas de crescimento e mortalidade. Esta simulação requer o número médio de células vivas da placa de controle T0 (x0), o comprimento do ensaio (t) e uma faixa definida pelo usuário de taxas de proliferação plausíveis (p) e taxas de mortalidade (d).

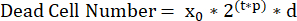
- Para determinar valores plausíveis para as taxas de crescimento, comece com a taxa de crescimento não tratada na população de células dobrando por h (1/T) como a maior taxa de crescimento e 0 como a menor taxa de crescimento. Divida esse intervalo em 500 segmentos igualmente espaçados. Uma operação semelhante pode ser aplicada para a taxa de mortalidade, testando um intervalo de 0 a 1.
NOTA: Todas as combinações de pares de 500 taxas podem ser executadas usando qualquer ambiente de programação, mas podem ser muito intensas computacionalmente se estiver usando algum software. Reduzir isso a todas as combinações de pares de 50 taxas de crescimento e mortalidade pode aliviar esse problema, com uma redução na precisão da inferência. Um modelo de planilha foi fornecido com este protocolo.
- Para determinar valores plausíveis para as taxas de crescimento, comece com a taxa de crescimento não tratada na população de células dobrando por h (1/T) como a maior taxa de crescimento e 0 como a menor taxa de crescimento. Divida esse intervalo em 500 segmentos igualmente espaçados. Uma operação semelhante pode ser aplicada para a taxa de mortalidade, testando um intervalo de 0 a 1.
- Determine os valores de GR e LF (descritos acima) para cada par de taxas de proliferação (p) e morte (d) simuladas.
NOTA: As etapas 7.2-7.3 gerarão uma tabela que contém cada par teórico de taxas de proliferação e mortalidade, com os valores de GR e LF calculados para esse par teórico. Isso funcionará como uma tabela de pesquisa para associar um par de valores de GR e LF observados experimentalmente a um par de taxas de proliferação e mortalidade induzidas por drogas. Testar mais de 500 valores discretos para taxas de proliferação e mortalidade resultará em um valor numericamente mais preciso, mas esse nível de precisão provavelmente estará além da precisão do ensaio. - Calcule a distância entre cada par GR/LF calculado experimentalmente e cada par GR/LF teórico na tabela de pesquisa. Inferir o par teórico com a distância mínima para o par de valores GR/LF observado experimentalmente como sendo as verdadeiras taxas de proliferação e mortalidade induzidas por drogas.
8. Determinação de vias de morte usando inibidores químicos seletivos de vias
NOTA: Os inibidores químicos por si só são insuficientes para determinar definitivamente o mecanismo de morte de um determinado medicamento. Inibidores químicos das vias de morte devem ser usados para determinar quais respostas bioquímicas ou fenotípicas devem ser exploradas em experimentos subsequentes, que provavelmente incluirão avaliação morfológica, marcadores bioquímicos específicos da via e avaliação de dependências genéticas.
- Otimize a dose de cada inibidor de morte celular na linha celular de interesse, testando uma faixa de dose de concentrações de inibidores no contexto de um ativador canônico da(s) via(s) de morte de interesse (Tabela 2). Idealmente, a dose selecionada de inibidor não deve afetar a viabilidade celular por si só (verifique a métrica GR).
NOTA: Nem todas as linhagens celulares podem ativar todas as vias de morte. A validação bioquímica ou fenotípica pode ser necessária ao testar ativadores/inibidores de diferentes vias de morte. - Considere o layout de revestimento ideal para uma tela inibidora. Para minimizar os efeitos do lote, mantenha cada medicamento na mesma placa que o(s) inibidor(es) que está(ão) sendo avaliado(s), com réplicas em placas separadas.
- Semeie o número desejado de células em cada poço de uma placa de 96 poços, fundo óptico e paredes pretas, semelhante à etapa 3.3. Diminua o volume de plaqueamento para 80 μL para contabilizar os volumes do inibidor e do medicamento.
- Prepare 10x concertação dos inibidores a serem testados em meio completo e adicione 10 μL a cada poço. Pré-trate as células com inibidores da via da morte por 2-4 h. Depois, adicione os medicamentos de interesse e adquira a leitura de fluorescência, como nas etapas 3-5.
- Avalie a mudança no tempo de início da morte (OD) e/ou na fração letal máxima para avaliar a eficácia do(s) inibidor(es). O DO é um parâmetro inferido do modelo Lag Exponential Death (LED). Consulte as etapas 5.6 a 5.7.
Resultados
Usando este protocolo, exploramos a sensibilidade das células U2OS ao inibidor de HDAC Belinostat. Esses experimentos foram realizados usando 2 μM de SYTOX Green para marcar células mortas (Figura 1A). As leituras cinéticas foram feitas usando um leitor de placa fluorescente em uma configuração de ganho de 130 (Figura 1B). As células foram lisadas em solução de Triton-X a 1,5% em PBS por 2 h no final do ensaio (Figura 1C-D).
O protocolo FLICK produz insights sobre a taxa de crescimento populacional e a taxa de morte celular induzida por drogas. Esses insights podem ser visualizados separadamente ou usando a estrutura de visualização e análise do medicamento GRADE, vistos em conjunto (Figura 1E). A avaliação da sensibilidade do Belinostat usando o valor de GR revela que 1 μM de Belinostat resulta em um valor de GR de aproximadamente 0 (Figura 2A). Na escala GR, os valores positivos relatam a taxa de expansão populacional e os valores negativos relatam a taxa de encolhimento populacional. Assim, um valor de GR de 0 revela que a população permanece em estase (ou seja, uma resposta citostática a drogas). Dada a interpretação convencional da citostase, pode-se concluir que 1 μM de Belinostat causa inibição total do crescimento sem qualquer morte celular. No entanto, a avaliação da fração letal induzida por drogas revela que o Belinostat 1 μM causou cerca de 50% de letalidade ao longo deste ensaio (Figura 2B). Na maioria dos casos, esses dois insights seriam derivados de experimentos diferentes que não podem ser comparados de maneira igualitária. É importante ressaltar que, usando o ensaio FLICK, ambos os insights são derivados dos mesmos dados experimentais, mas analisados de forma diferente para capturar os efeitos do Belinostat no crescimento populacional versus morte celular.
Esses dois insights também podem ser integrados usando o método analítico GRADE (Figura 2C). O medicamento GRADE justapõe a Fração Letal, que é proporcional à taxa média de mortalidade, contra o valor GR, a taxa líquida de crescimento populacional. A integração dos dados dessa maneira fornece uma visualização de como uma droga coordena a inibição do crescimento e a ativação da morte. Dentro do gráfico GRADE: drogas que inibem o crescimento sem ativar a morte (ou seja, drogas citostáticas, usando a interpretação comum) ocuparão o limite superior, drogas que apenas matam sem alterar as taxas de proliferação celular ocuparão o limite direito, e drogas que primeiro causam inibição do crescimento, seguida pela morte de células paradas no crescimento ocuparão o limite esquerdo (ou seja, drogas bifásicas; Figura 2C). Drogas que resultam em respostas GR / LF que se enquadram dentro desses limites podem ser inferidas para inibir simultaneamente a proliferação até certo ponto, ao mesmo tempo em que ativam a morte celular (ou seja, drogas coincidentes) 3. É importante ressaltar que a posição de cada resposta a medicamentos dentro do espaço GR / LF pode ser usada para calcular a verdadeira taxa de proliferação (p) e a taxa média de mortalidade (d) para cada concentração de um medicamento (Figura 2D-E). Observe que o valor de GR não é a verdadeira taxa de proliferação celular, mas sim a taxa líquida de crescimento populacional (ou seja, o efeito líquido combinando a verdadeira taxa de proliferação e a taxa de mortalidade induzida por drogas). Além disso, a taxa de mortalidade produzida em uma análise GRADE (d, que é a taxa média de mortalidade) é distinta do parâmetro de taxa de mortalidade na análise cinética de LED (DR, a velocidade máxima ou taxa máxima de mortalidade).
O ensaio FLICK pode gerar dados específicos de morte, como a fração letal induzida por drogas; no entanto, esses dados são agnósticos quanto ao mecanismo de morte celular. Para obter informações sobre o mecanismo de morte celular usando FLICK, o método mais simples é determinar como a cinética da fração letal é alterada pela inclusão de inibidores específicos da via de morte. A inibição de um mecanismo de morte relevante deve resultar na diminuição da fração letal induzida por drogas e/ou no atraso do tempo de início da morte. Aqui, mostramos que 50 μM do inibidor da pan-caspase z-VAD resgatam cerca de 50% da letalidade induzida por 1 μM de Belinostat (Figura 2F). É importante ressaltar que, como os medicamentos, os inibidores têm especificidade limitada e podem inibir ou exacerbar acidentalmente outros mecanismos de morte. Assim, antes que uma conclusão definitiva seja feita, esses dados devem ser complementados com outros insights, como morfologia celular, medição de marcadores bioquímicos de ativação e/ou avaliação de dependências genéticas específicas da via14.
Para ilustrar formalmente como a interpretação de uma resposta a medicamentos pode ser alterada por uma avaliação baseada em FLICK e análise baseada em GRADE, exploramos a seguir três medicamentos que apresentam diferentes tipos de coordenação de crescimento / morte. O palbociclibe é um inibidor de Cdk4/6 que resulta em uma resposta citostática (inibição do crescimento sem morte celular). A camptotecina é um inibidor da topoisomerase I que causa uma resposta bifásica (inibição do crescimento, seguida de morte celular em altas doses). Belinostat é um inibidor de HDAC que causa uma resposta coincidente (inibição parcial do crescimento e ativação da morte em cada dose, mas com proporções variadas entre as doses). Usando uma análise convencional desses dados, pode-se observar que todos os três medicamentos diminuem substancialmente a viabilidade relativa das células U2OS (Figura 2G). Embora o Palbociclibe seja visivelmente menos potente, não está claro a partir desses dados que o Palbociclibe não ativa a morte celular. Usando uma análise baseada em GR, que relata como os medicamentos afetam a taxa líquida de crescimento populacional, pode-se interpretar com mais precisão que as populações tratadas com Palbociclibe continuam a se expandir em todas as doses, enquanto altas doses de Camptotecina ou Belinostat resultam em encolhimento populacional e, portanto, a morte celular deve ter sido ativada após altas doses desses medicamentos (Figura 2H). No entanto, uma análise baseada em RG não leva em conta a coordenação variada entre crescimento e morte, o que pode levar a conclusões errôneas sobre o nível de morte celular ativado por um determinado medicamento. Por exemplo, com base nos dados de RG, pode-se concluir que 1 μM de Camptotecina ativa a morte celular em maior extensão do que 1 μM de Belinostat, uma vez que a Camptotecina resulta em um valor de GR de -0,25 e Belinostat resulta em um valor de GR de 0; um valor de -0,25 GR significa que a população está encolhendo 25% tão rápido quanto a população não tratada está se expandindo. Usando uma visualização e análise baseadas em GRADE, pode-se observar que a camptotecina e o belinostat diferem em sua coordenação de crescimento / morte. Assim, na dose de 1 μM, ambos os Belinostat ativam a morte a uma taxa mais alta do que a Camptotecina (0,8% de morte/hora para Belinostat, em comparação com 0,7% de morte/hora para Camptotecina), mas a população tratada com Camptotecina encolhe mais rapidamente devido a uma inibição de crescimento mais pronunciada induzida por este medicamento (Figura 2I).
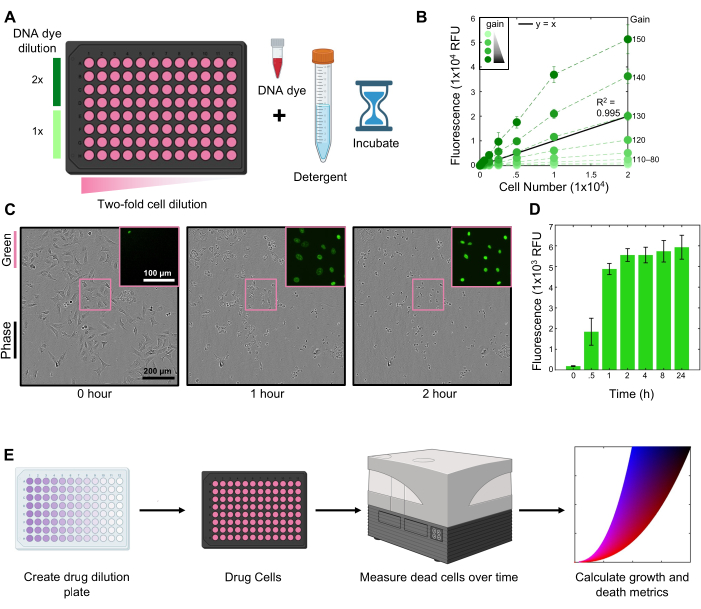
Figura 1: Fluxograma do protocolo FLICK. (A) Crie uma diluição celular para otimizar a concentração do corante de DNA e a linearidade das medições de fluorescência em um leitor de microplacas. (B) Titulação de ganho em todo o número de células para 2 μM de SYTOX Green em células U2OS. Um ganho de 130 fornece o sinal mais linear em todos os números de células. (C) Imagens de fase de células U2OS lisadas com 1,5% de Triton-X ao longo do tempo usando uma objetiva de 10x. A região encaixotada é uma visão ampliada da fluorescência verde SYTOX. As barras de escala são representativas de todas as imagens. (D) SYTOX Sinal verde após a lise nos horários indicados. Os dados são médios +/- DP para 3 réplicas biológicas. (E) Uma visão geral conceitual do ensaio, desde a configuração até a análise. As principais etapas são a criação de uma placa de diluição de medicamentos, a drogação de células em uma placa de vários poços, a aquisição de medições de fluorescência ao longo do tempo e o cálculo de métricas de crescimento e morte. Para os painéis B e D, os dados são ± média DP para n = 3 réplicas biológicas independentes. Clique aqui para ver uma versão maior desta figura.
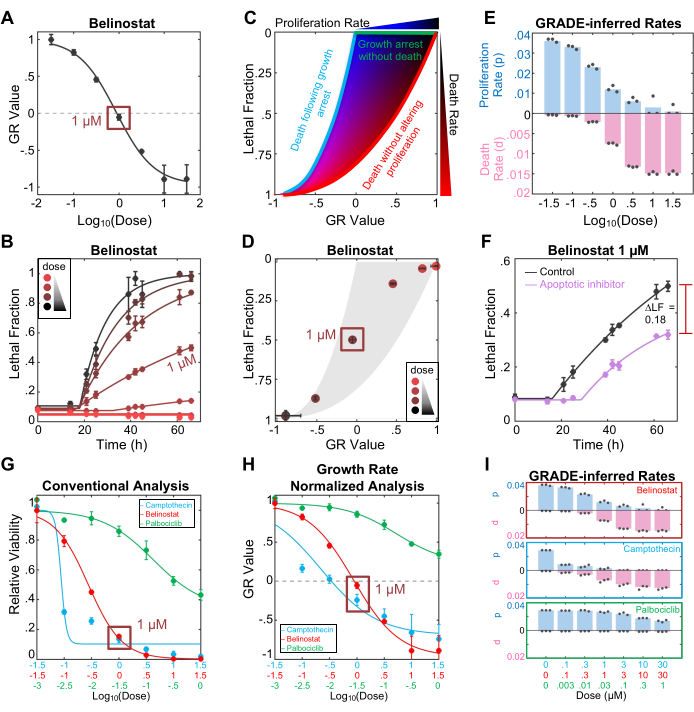
Figura 2: Caracterização das respostas aos medicamentos usando o ensaio FLICK e a análise baseada em GRADE. (A) Métrica GR para o inibidor de HDAC, Belinostat, em uma dose de doses. Os dados mostrados são do endpoint do ensaio, que foi de 66 h. A 1 μM, o Belinostat tem um valor de GR de aproximadamente 0, indicando uma resposta citostática. (B) Cinética da fração letal para Belinostat. A 1 μM, o Belinostat induz quase 50% de letalidade, que não é capturada pelo valor de GR. (C) Diagrama de fase GRADE baseado em uma simulação de todas as taxas possíveis de crescimento e mortalidade. A simulação foi baseada no tempo de duplicação do U2OS neste ensaio (27,12 h). (D) Gráfico GRADE para Belinostat entre doses no ponto final do ensaio. (E) Gráfico de barras das taxas de crescimento inferidas pelo GRADE (p, duplicações da população por hora) e morte (d, fração da população que morre por hora) para doses de Belinostat. As taxas foram determinadas a partir das taxas simuladas em (C) e dos dados de (D). (F) Gráfico de fração letal para 1 μM de Belinostat tratado com (roxo) ou sem (preto) 50 μM do inibidor apoptótico z-VAD. ΔLF = 0,18 foi calculado subtraindo-se o LF máximo médio da condição tratada com inibidor do LF máximo médio do controle. (G) Viabilidade relativa (número de células vivas na condição tratada com o medicamento, dividido pelo número de células vivas na condição não tratada) para três medicamentos após uma exposição de 72 horas. Todos os medicamentos reduzem a viabilidade celular, mas a contribuição da morte celular para a resposta não é conhecida. (H) Métrica GR para três medicamentos em (G). Esses dados demonstram que dois dos três medicamentos resultam em encolhimento populacional, indicado pelo GR < 0. A contribuição da morte celular ainda não está clara, embora a morte celular deva ter sido ativada quando o valor de GR é negativo. (I) Taxas inferidas pelo GRADE para três drogas. O palbociclibe não é letal, mas resulta em parada do crescimento. A camptotecina é bifásica; Doses mais baixas resultam em parada do crescimento, enquanto doses mais altas resultam em parada do crescimento seguida de letalidade. Belinostat é uma droga coincidente; a faixa de dose corta o meio do gráfico GRADE, indicando que todas as doses resultam em uma perturbação do crescimento e ativação da morte celular. Os dados em todos os painéis são a média ± DP para três experimentos de replicação biológica independentes. Clique aqui para ver uma versão maior desta figura.
| Tingir | Cinético | Ponto final |
| SYTOX Verde | Y | Y |
| CellTox Verde | Y | Y |
| NucSpot 500/515, 594/615, 750/780 | Y | Y |
| YOYO-3 | Y | Y |
| Iodeto de propídio | N | Y |
| 7-AAD | N | Y |
Tabela 1: Corantes de DNA e sua aplicação no FLICK. Muitos tipos de corantes podem ser usados para ensaios FLICK somente de ponto final. Menos corantes atendem às condições para serem incubados continuamente durante todo o ensaio. Outros corantes, incluindo corantes que não são verdes, não foram profundamente caracterizados e devem ser cuidadosamente avaliados antes do uso.
| Caminho da Morte | Inibidor | Dose típica |
| Apoptose intrínseca | ZVAD-FMK | 50 μM |
| Apoptose extrínseca | Z-IETD | 30 μM |
| Ferroptose | Ferrostatina-1 | 10 μM |
| Necroptose | Necrostatina-1 | 50 μM |
| Piroxase | VX-765 | 50 μM |
| Parthanatos | Rucaparibe | 1 μM |
| Morte celular dependente autofágica | Hidroxicloroquina (HCQ) | 10 μM |
| Morte Celular Dependente de Lisossomo | E-64D | 10 μM |
| Cuproptose | Tetratiomolibdato (TTM) | 5 μM |
| Oxeiptose | N-acetil-l-cisteína (NAC) | 2 mM |
| Necrose induzida por MPT | Ciclosporina A (CsA) | 10 μM |
Tabela 2: Vias de morte celular e seus inibidores. Os inibidores de morte celular devem ser validados em relação a um ativador canônico para cada via de morte. Idealmente, as doses de inibidores selecionadas não devem afetar a viabilidade celular.
Tabela Suplementar 1: Calculadora GRADE. Planilha para calcular o crescimento induzido por drogas e as taxas de mortalidade a partir de dados de GR e FV baseados em FLICK. Os usuários devem inserir dados de GR e FV, juntamente com a duração do ensaio em horas, o tempo de duplicação da célula em horas e o tamanho da população inicial/final. Clique aqui para baixar este arquivo.
Discussão
O ensaio FLICK é um método robusto para gerar uma avaliação abrangente do efeito de um medicamento no crescimento e morte de uma população de células. Como esse método não conta diretamente as células, as etapas críticas do protocolo são garantir a linearidade do ensaio e a lise completa durante as etapas de permeabilização do tritão. O tempo correto de permeabilização pode ser identificado visualmente, conforme destacado neste protocolo, ou quantitativamente pela leitura da fluorescência da placa ao longo do tempo e identificação de quando o sinal se estabiliza. Em nossa experiência, no entanto, em caso de dúvida, não parece haver nenhum custo em esperar mais: para os corantes de DNA que usamos no ensaio FLICK, a intensidade do sinal permanece estável por vários dias após a lise. Usamos regularmente a coloração de ácido nucleico SYTOX Green e, embora não tenhamos validado cada fluoróforo referenciado na Tabela 1, vários outros protocolos demonstram seu uso efetivo 13,15,16. Avaliar a linearidade de um determinado corante de DNA impermeável à célula em uma variedade de números de células e configurações de instrumentos garantirá dados robustos e quantitativamente confiáveis.
Algumas linhagens celulares são mais difíceis de lisar do que outras, e nunca se deve presumir que o tempo de lise de uma linhagem celular é o mesmo que o de outras13. Para algumas linhagens celulares, em densidades mais altas, a lise celular faz com que as células se levantem do poço como uma folha, o que pode causar erro no leitor de placas. Isso pode ser evitado reduzindo a densidade inicial de semeadura ou pipeta misturando os poços após a lise e antes da leitura. Para melhorar o tempo de lise ou a eficiência da lise, a solução Triton-X pode ser ajustada para uma porcentagem maior ou menor, mas ainda não encontramos um cenário em que isso fosse necessário.
O projeto experimental e o layout da placa de medicamentos devem ser cuidadosamente considerados quando dados robustos de crescimento são desejados. A criação de uma placa de diluição de medicamentos para o ensaio FLICK é a mesma que qualquer outro ensaio de placa de 96 poços. No entanto, incluir muitos controles cobrindo diferentes partes da paisagem de uma placa ajudará a criar insights robustos sobre a cinética de crescimento e pode ajudar a determinar se existe variação sistemática de crescimento. Evitar os poços externos de uma placa produzirá dados de crescimento mais precisos, pois esses poços são mais sensíveis às flutuações de temperatura, oxigênio ou umidade, afetando seu crescimento. Por fim, o uso de um padrão de drogas pseudo-randomizado, como mudar a localização de uma droga em placas replicadas, fornecerá uma avaliação mais precisa do comportamento da droga.
Existem algumas limitações para o uso preciso do ensaio FLICK. Essas limitações estão principalmente relacionadas à inferência cinética, pois os dados do endpoint são valores observados experimentalmente. Tanto a cinética de proliferação de células vivas quanto a cinética de morte dependem de alguma suposição sobre a trajetória de crescimento das células vivas ao longo do tempo na condição tratada com drogas. Neste protocolo, descrevemos essa trajetória de crescimento como exponencial e com uma taxa uniforme ao longo do tempo. Embora seja provável que essas características sejam observadas na ausência de um medicamento, elas podem não ser respeitadas na presença de um medicamento. O método FLICK é preciso, não porque as suposições sobre as trajetórias de crescimento estejam sempre corretas, mas porque o impacto de quaisquer suposições incorretas é minimizado se a população tratada com drogas não proliferar muito (ou seja, menos de 2 ou 3 duplicações populacionais no período do ensaio). Não encontramos drogas que não possam ser perfiladas com precisão no FLICK; no entanto, a teoria sugeriria que o ensaio FLICK deixará de ser preciso para um medicamento no qual a morte ocorre muito lentamente, por longos períodos de tempo que são vários múltiplos do tempo de duplicação da célula (nota: não identificamos nenhum medicamento com essas características3). No entanto, o impacto de quaisquer suposições incorretas para a cinética de crescimento da população total não afetará o tempo de início da morte obtido a partir do ajuste do LED ou os valores máximos finais de LF, pois estes são limitados pelos valores de células mortas medidos empiricamente e os valores totais finais de células que foram determinados experimentalmente no final do ensaio.
Apesar dessas limitações, o ensaio FLICK permite insights difíceis de gerar usando outros métodos de resposta a medicamentos. A maioria dos métodos de resposta a medicamentos gera um sinal proporcional ao número de células vivas, e esses métodos podem ser usados para quantificar como os medicamentos afetam a taxa líquida de crescimento populacional (ou seja, valor GR ou equivalente), mas não podem discriminar com precisão entre os efeitos citotóxicos e citostáticos de um medicamento. Alternativamente, ensaios baseados em microscopia que medem células vivas e mortas certamente podem gerar uma imagem abrangente. No entanto, os ensaios baseados em microscopia podem ter dificuldade em contar as células mortas depois que as células se decompõem em detritos, como acontecerá rapidamente no contexto de muitas formas não apoptóticas de morte celular. Uma característica fundamental do FLICK é que as medições são feitas em um leitor de placas, que agrega a fluorescência total de células mortas em vez de contar diretamente as células mortas. Assim, o sinal de células mortas no FLICK não depende da celularidade intacta das células mortas, que, no contexto da morte celular não apoptótica, é exclusivamente essencial. Além disso, uma característica única do ensaio FLICK é a capacidade de medir as populações de células vivas e mortas usando o mesmo reagente. Assim, o ensaio FLICK produz medições de células vivas e mortas com igual sensibilidade. Esse recurso permite a análise usando o método GRADE e melhora a precisão dos cálculos simultâneos de crescimento e taxa de mortalidade do GRADE. A Tabela Suplementar 1 incluída inclui um modelo usado para visualização do GRADE do medicamento, onde o usuário pode inserir o comprimento do ensaio, a taxa de crescimento calculada a partir da etapa 7.1, o número de células inicial e final ou as leituras fluorescentes das condições de controle e o LF e GR calculados das etapas 5.4 e 6.3. O arquivo contém uma simulação de todas as combinações de pares de 50 taxas de crescimento e mortalidade que serão atualizadas automaticamente com base nos parâmetros definidos pelo usuário. O modelo gera uma visualização baseada em GRADE da resposta ao medicamento e das taxas de crescimento (p) e morte (d) inferidas pelo GRADE.
Trabalhos futuros devem explorar a aplicação do ensaio FLICK em cenários de cultura de células 3D ou na avaliação das respostas a medicamentos de organoides derivados de tumores. A linearidade e a sensibilidade dos dados do FLICK não foram profundamente questionadas nesses contextos, mas, em teoria, o ensaio do FLICK deve ser eficaz com algumas modificações para integrar a fluorescência em amostras tridimensionais. Além disso, adicionar rótulos adicionais específicos de células ajudaria a discriminar entre dois ou mais tipos de células em co-cultura. Esses avanços serão valiosos para explorar as interações das células imunológicas com as células cancerígenas. Finalmente, ensaios como o SPARKL inspiram o uso de repórteres específicos de morte no formato de ensaio FLICK, o que pode melhorar o rendimento, mantendo a precisão dos insights de morte celular11,16.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Agradecemos a todos os membros anteriores e atuais do Lee Lab por suas contribuições para a perspectiva de nosso laboratório na avaliação de respostas a medicamentos. Este trabalho foi apoiado por financiamento dos Institutos Nacionais de Saúde para MJL (R21CA294000 e R35GM152194).
Materiais
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
Referências
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados