Method Article
Análisis exhaustivo de la respuesta a los fármacos mediante el ensayo FLICK
En este artículo
Resumen
Este protocolo describe cómo usar el ensayo FLICK para evaluar las respuestas a los medicamentos, incluidas instrucciones detalladas para usar este ensayo para calcular las tasas de crecimiento y mortalidad inducidas por medicamentos y para evaluar el mecanismo de la muerte celular inducida por medicamentos.
Resumen
Para comprender la eficacia de los fármacos, una necesidad crítica es caracterizar el alcance de la muerte celular inducida por los fármacos. Los esfuerzos para cuantificar el nivel de muerte celular inducida por fármacos se ven desafiados por la existencia de más de una docena de formas molecularmente distintas de muerte regulada, cada una con su propio momento de activación y características bioquímicas distintivas. Además, para algunos subtipos de muerte necrótica, las características distintivas solo se observan transitoriamente y se pierden rápidamente debido a la ruptura celular. Por lo tanto, incluso cuando se utiliza una combinación de ensayos específicos de la vía de muerte, es difícil cuantificar con precisión la cantidad total de muerte celular o las contribuciones relativas de cada subtipo de muerte. Otro problema es que muchos ensayos específicos de muerte ignoran cómo los fármacos afectan a la proliferación celular, lo que dificulta la interpretación de si una población tratada con fármacos se está expandiendo o disminuyendo. El ensayo FLICK permite cuantificar el nivel total de muerte celular después de la estimulación de una manera que es específica para la muerte, pero también en gran medida independiente del tipo o tipos de muerte activados. Además, el ensayo FLICK retiene información sobre el tamaño total de la población y la tasa de proliferación celular. En este manuscrito, describimos el uso básico del ensayo FLICK, cómo solucionar problemas de este ensayo cuando se utilizan diferentes tipos de material biológico y cómo usar el ensayo FLICK para cuantificar las contribuciones de cada tipo de muerte celular a una respuesta farmacológica observada.
Introducción
En el caso de los fármacos contra el cáncer, la evaluación preclínica de la sensibilidad a los fármacos suele implicar la comprobación de cómo afectan los fármacos a la viabilidad de las células encultivo. La viabilidad celular después de la exposición al fármaco es producto de al menos dos efectos separados: la inhibición de la proliferación celular inducida por el fármaco y la activación de la muerte celular2. Desafortunadamente, aunque la muerte celular es una característica crítica que se requiere para respuestas duraderas a los medicamentos, los enfoques estándar no aclaran el grado en que un medicamento activala muerte celular.
Los ensayos comunes de respuesta a fármacos incluyen aquellos que cuentan directamente las células (p. ej., contador de Coulter, algunos usos de la citometría de flujo o la microscopía), cuantifican la capacidad de las células para proliferar (p. ej., ensayo de formación de colonias) o cuantifican una actividad metabólica (p. ej., CellTiter-Glo, ensayos MTT o MTS basados en tetrazolio). Una característica compartida de estos ensayos es que los datos generados son proporcionales al número de células vivas. Debido a que los fármacos varían considerablemente en la forma en que coordinan la inhibición del crecimiento y la muerte celular, el número de células vivas tras la exposición al fármaco proporciona una visión poco fiable del nivel de muerte celular inducida por el fármaco. Además, debido a que las células cancerosas generalmente proliferan rápidamente en cultivos celulares, el número de células vivas puede reducirse drásticamente en relación con la población no tratada sin inducirninguna muerte celular. Por lo tanto, una falla central es que el grado de muerte celular no se puede cuantificar sin medir tanto el número de células vivas como el de células muertas.
Cuantificar el número de células muertas después de la exposición a un fármaco es difícil de hacer con precisión porvarias razones. En primer lugar, existen más de una docena de vías de muerte celular reguladas 5,6,7,8. Aunque generalmente existen marcadores bioquímicos para identificar cada tipo de muerte celular regulada, estos marcadores varían en su especificidad, y no se puede utilizar un solo ensayo para cuantificar simultáneamente todos los subtipos de muerte. En segundo lugar, el momento de la activación de cada forma de muerte celular puede variar drásticamente dependiendo del contexto, por lo que no puede surgir una imagen completa a menos que la muerte se cuantifique a lo largo del tiempo9,10. Muchos ensayos bioquímicos para cuantificar la muerte celular producen una medición de punto final, por lo que la generación de datos cinéticos puede ser un desafío y estar limitada por el costo. Una tercera complicación es que la célula muerta en sí misma es un estado intermedio transitorio entre el estado de la célula viva y los restos de células disociadas. La estabilidad de las células muertas varía según el subtipo de muerte, con algunos tipos, como la apoptosis, creando cadáveres relativamente estables, mientras que otros tipos de muerte causan una lisis rápida. Por lo tanto, los métodos de cuantificación de la muerte que requieren la recolección y el recuento de células muertas también producirán una comprensión sesgada de la muerte celular. Por último, una cuarta limitación es que los ensayos bioquímicos que cuantifican el grado de muerte celular no suelen proporcionar ninguna información sobre cómo un fármaco altera la proliferación. Por lo tanto, no se puede interpretar el tamaño total de la población y, lo que es más importante, si la población está aumentando o disminuyendo.
Algunos ensayos basados en microscopía, como STACK y SPARKL, son eficaces para medir las células vivas y muertas a lo largo del tiempo, y estos ensayos pueden producir información completa sobre la muerte celular inducida por fármacos10,11. Sin embargo, estos ensayos requieren instrumentos especializados, como el microscopio Incucyte, lo que crea limitaciones en el rendimiento y el acceso a estos enfoques. Además, las técnicas basadas en microscopía requieren que las células muertas permanezcan en el plano focal del microscopio durante todo el experimento, lo que compromete la capacidad de cuantificar las células muertas cuando pierden adherencia a la placa o con el tiempo a medida que las células muertas se descomponen. De manera similar, los ensayos basados en microscopía enfrentan desafíos cuando se aplican en el contexto de cultivos en suspensión, ya que las células entran y salen de un plano focal determinado.
Para abordar los problemas señalados anteriormente, hemos generado un ensayo llamado FLICK (Fluorescence-based and Lysis-dependent Inference of Cell Death Kinetics)12,13. El objetivo del ensayo FLICK es determinar el nivel de muerte celular inducida por fármacos, independientemente de cómo estén muriendo las células. El método FLICK utiliza tintes celulares impermeabilizados, cuya fluorescencia depende de la unión del ADN. Una característica clave de FLICK es el uso de estos fluoróforos para etiquetar la acumulación de células muertas a lo largo del tiempo en virtud de su ADN accesible, seguido de una lisis mecánica basada en detergente para permeabilizar cualquier célula viva al final del ensayo. Estos datos, combinados con modelos matemáticos, permiten la cuantificación de poblaciones de células vivas y muertas con una resolución temporal continua y sin necesidad de recoger o manipular células muertas. Además, el uso de un lector de placas para evaluar la fluorescencia de las células muertas permite la evaluación de las células muertas sin necesidad de que las células muertas permanezcan intactas, aliviando así el sesgo contra las formas necróticas de muerte que dan lugar a la ruptura celular. Por último, el ensayo FLICK requiere una manipulación mínima de la placa y puede generar rápidamente mediciones cinéticas, lo que permite el cribado de fármacos de alto rendimiento. En este protocolo, nos centramos en el uso del ensayo FLICK, incluyendo cómo utilizar FLICK para inferir la tasa de crecimiento inducida por fármacos, la tasa de mortalidad y/o los mecanismos de muerte celular.
Protocolo
1. Optimización del tiempo de permeabilización para cada línea celular de interés
NOTA: Los volúmenes y cantidades descritos son para optimizar una línea celular. Estos valores deben ampliarse en función del número de líneas celulares que se van a probar.
- Coloque el número deseado de celdas en cada pocillo de una placa de 96 pocillos, fondo óptico, de paredes negras. Agregue 100 μL de medio completo y deje que las células se adhieran a la placa durante la noche.
NOTA: El número de células en placa debe optimizarse teniendo en cuenta la tasa de crecimiento de las células, las densidades óptimas y la longitud del ensayo. El número de células iniciales típicas para las líneas celulares cancerosas adherentes medido durante 72 h es de 1500 a 5000 células por pocillo. Por ejemplo, las células U2OS pueden sembrarse a 2000 células por pocillo en DMEM con 10% de FBS, 2 mM de glutamina y 1% de Pen-Strep y cultivarse durante la noche en condiciones estándar (5% de CO2, 37 °C, con humedad). - En un tubo centrífugo cónico de 15 mL, prepare 1,5 mL de una solución al 1,5% de Triton-X en solución salina tamponada con fosfato (PBS). Vortex, la solución Triton-X al 1,5% durante 5 s a velocidad máxima. Colocar en un baño de agua a 37 °C durante al menos 30 min.
- Inspeccione visualmente la solución para asegurarse de que Triton-X se haya disuelto completamente por vórtice durante varios segundos. Asegúrese de que la solución sea homogénea.
- Añada 10 μL de la solución Triton-X al 1,5% a cada pocillo de las células plateadas. No mezclar.
NOTA: En esta etapa, no es deseable mezclar mediante pipeteo repetido debido a la formación de burbujas. - Regrese las células al 5% de CO2, 37 °C con humedad en la incubadora de cultivos celulares durante al menos 1 h.
NOTA: Después de la aplicación de la solución Triton-X, la mayoría de las líneas celulares se lisan completamente en 2-3 h. Esperar hasta 24 horas tiene un impacto mínimo en la señal de fluorescencia cuando se utilizan colorantes como SYTOX Green. El aumento del porcentaje de Triton-X puede acortar el tiempo de incubación de líneas celulares difíciles de lisar. - Observe las morfologías celulares en un microscopio óptico utilizando un objetivo 10x. Inspeccione las celdas una vez cada hora hasta que los cuerpos celulares ya no sean visibles. Registre el tiempo necesario para la permeabilización celular.
NOTA: Si se dispone de un microscopio fluorescente, la lisis se puede visualizar utilizando un tinte de ADN impermeable a las células, como SYTOX Green. El colorante se puede agregar a una concentración final de 10 veces a la solución de Triton-X, y la permeabilización se puede confirmar mediante microscopía de fluorescencia.
2. Selección y calibración de la tinción de ADN
NOTA: Un requisito para el ensayo FLICK es el uso de un fluoróforo impermeable a la célula que emita una señal de manera dependiente de la unión al ADN, que no afecte a la viabilidad de la célula y que produzca una señal que escale linealmente con el número de células. Este protocolo utiliza SYTOX Green. Otros colorantes con propiedades similares también pueden ser adecuados para el ensayo FLICK, pero cada uno de ellos debe ser evaluado y calibrado. Consulte la Tabla 1 para ver ejemplos.
- Para el tinte de ADN seleccionado, determine el rango de concentraciones que se probarán según la recomendación del fabricante.
- Para cada concentración que se vaya a probar, coloque 40.000 células en 180 μL de medio de cultivo celular por triplicado a lo largo de la columna más a la izquierda de una placa de pared negra de 96 pocillos, con fondo óptico. Agregue 90 μL de medio a los pocillos restantes.
NOTA: Esto se utilizará para generar una valoración lineal de la celda a través de las filas de la placa. La concentración final de los primeros pozos será de 20.000 células/pocillo. Las placas inferiores ópticas no son necesarias para los lectores de placas que leen la fluorescencia desde la parte superior del pocillo. El medio de cultivo celular debe ser el mismo que se utiliza para cultivar las células que se están analizando. Por ejemplo, si se utilizan células U2OS, se deben resuspender 40.000 células en 180 μL de DMEM. - Con una pipeta multicanal, cree una dilución en serie 1:2 transfiriendo 90 μL de la columna situada más a la izquierda a la columna adyacente de la derecha. Pipetear 15x para mezclar.
- Repita el paso 2.3, pasando de la segunda columna a la tercera, luego de la tercera columna a la cuarta columna, y así sucesivamente. Termine la valoración en la penúltima columna de la placa. Deje la última columna sin celdas para que se pueda obtener la señal de fondo de los medios de cultivo celular.
- Para la penúltima columna, retire 90 μL para que todos los pocillos tengan 90 μL de medio que contenga un número variado de células. Deje que las células se adhieran durante 6 h en una incubadora de cultivo celular a 37 °C.
- Prepare una solución 10x de tinte de ADN para cada concentración de tinte que se va a analizar, con el tinte de ADN diluido en un medio de crecimiento completo utilizado para cultivar las células de interés. Prepare 1,5 ml de una solución de Triton-X al 1,5% en PBS, como se describe en el paso 1.
- A cada pocillo de la placa de 96 pocillos, agregue 10 μL del tinte de ADN 10x. Añadir 10 μL de solución de Triton-X al 1,5% a cada pocillo para permeabilizar las células. Incubar las células en esta solución durante el tiempo óptimo, que se determinó en el paso 1.
NOTA: Después de la adición de Triton-X, la concentración final de tinte de ADN es ligeramente inferior a 1x, pero esto es intrascendente. Después de la incubación, se debe lisar toda la placa. La valoración celular manual se utilizará para identificar la concentración óptima de colorante de ADN y los ajustes óptimos para adquirir fluorescencia. - Mida las intensidades de fluorescencia en la placa de valoración de la célula. Cuantifique la fluorescencia en una variedad de configuraciones de adquisición según lo descrito por el fabricante del tinte de ADN. Dependiendo del lector de placas utilizado, modifique las longitudes de onda de excitación y emisión y/o la ganancia digital.
- Elimine la señal de fondo de cada medición restando la señal promedio de la columna que no contiene celdas. Estos valores se pueden encontrar en la columna de pocillos más a la derecha en la placa de dilución en serie.
- Determine la linealidad de cada concentración de colorante de ADN para cada configuración de adquisición. La linealidad se puede determinar graficando el número de celdas frente a la señal de fluorescencia y realizando una regresión lineal. El coeficiente de determinación (r2) informa el grado en que la señal de fluorescencia está relacionada linealmente con el número de células.
- Elija la configuración de adquisición y la concentración de colorante de ADN que tenga la mejor combinación de linealidad y rango dinámico.
NOTA: Considere todo el rango de números de celda de 0 a 20,000 celdas. Asegúrese de que la ganancia y la concentración de colorante de ADN sean lineales a números de células bajos, entre 0 y 1000 células. Varios ajustes del instrumento o concentraciones de colorante de ADN pueden proporcionar una fuerte correlación en todo el rango. Sin embargo, es esencial que la sensibilidad del extremo inferior sea robusta, de modo que se puedan medir con precisión los pequeños cambios en las células muertas.
3. Colocación de placas celulares, aplicación de fármacos en pocillos y medición de la fluorescencia de células muertas a lo largo del tiempo en placas tratadas con fármacos
NOTA: Las placas de dilución de fármacos pueden diseñarse de forma flexible en función de las necesidades experimentales. Por lo general, las placas de dilución de fármacos incluirán una serie de diluciones logarítmicas o semilogarítmicas de uno o varios fármacos.
- Considere el diseño de recubrimiento óptimo para el experimento. Evite las mediciones de los pozos exteriores de una placa de 96 pocillos para disminuir el ruido asociado con la variación del crecimiento, ya que los pozos exteriores experimentan diferentes temperaturas, oxigenación y evaporación en comparación con los pocillos internos de la placa.
NOTA: Incluir controles en todo el paisaje de la placa aumentará la solidez de los datos de crecimiento. Considere incluir pozos de control en el centro y los bordes de la placa. - Determine el número de placas necesarias para el experimento. Incluya una placa adicional que se lisará al comienzo del ensayo para establecer el número promedio de células por pocillo en el momento del fármaco (es decir, la placa de control T0).
- Prepare una suspensión celular en un medio de crecimiento completo.
- En función del número de placas necesarias para el experimento (calculado en el paso 3.2), determine el volumen de la suspensión celular necesario. Si las placas de siembra se siembran a 90 μL por pocillo, se requieren aproximadamente 10 mL por placa. Multiplique el número de placas necesarias por 10 mL.
NOTA: Es deseable aumentar este volumen para tener en cuenta el volumen muerto del depósito y la pérdida de volumen durante el pipeteo. Por ejemplo, el volumen calculado se puede multiplicar por 1,2. - Cuente las células y prepare una dilución de células en función del número deseado de células por pocillo. Por ejemplo, si el objetivo es sembrar 2000 células en 90 μL de medio por pocillo de una placa de 96 pocillos, para un total de 3 placas, calcule el número total de células necesarias para el experimento de la siguiente manera:
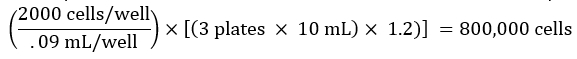
De manera similar, si la suspensión celular contada es de 400.000 células/mL, el volumen de la suspensión celular necesario para el experimento sería:
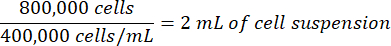
- Corrija el volumen de los medios de cultivo celular utilizados para la siembra restando el volumen de la suspensión celular contada que se va a agregar.
Medios de 36 mL - Suspensión de células de 2 mL = volumen de medio corregido de 34 mL
- En función del número de placas necesarias para el experimento (calculado en el paso 3.2), determine el volumen de la suspensión celular necesario. Si las placas de siembra se siembran a 90 μL por pocillo, se requieren aproximadamente 10 mL por placa. Multiplique el número de placas necesarias por 10 mL.
- Mezcle la suspensión de células contadas con el volumen de medio correcto utilizando una pipeta serológica. Transfiera esta suspensión celular a un depósito de reactivos de fondo en V.
- Con una pipeta multicanal, añada 90 μL de la suspensión celular a cada pocillo de las placas de 96 pocillos. Mezcle la suspensión celular en el depósito de reactivo regularmente utilizando pipeteo repetido con una pipeta serológica para asegurarse de que se mantenga la concentración deseada de células por volumen.
- Deje que las células se adhieran durante la noche en una incubadora de cultivo celular a 37 °C (generalmente 12-24 h).
- Prepare una solución de Triton-X al 1,5 % en PBS, como se describe en el paso 1. El volumen de esta solución debe ser suficiente para lisar todas las placas. Aproximadamente 1,5 mL de Triton-X al 1,5% en PBS es suficiente para lisar una placa de 96 pocillos.
NOTA: La solución Triton-X al 1,5% en PBS es estable durante más de una semana y se puede fabricar con anticipación para mayor comodidad. - Determine la cantidad de medios de cultivo celular necesarios para hacer una placa de dilución de medicamentos. Prepare placas de dilución de medicamentos en placas transparentes de 96 pocillos con fondo en U o V.
- Para asegurarse de que el volumen del fármaco diluido es suficiente para el experimento, aumente el volumen mínimo necesario para tener en cuenta la pérdida de volumen durante el pipeteo. Por ejemplo, si se van a añadir fármacos en 10 μL a cada pocillo de una placa de 96 pocillos y el experimento incluye tres placas, se requieren 30 μL de fármaco diluido por pocillo. Prepare 40-50 μL de medio drogado por pocillo para tener en cuenta la pérdida de volumen. Agregue 1.5 mL al volumen de medios requerido para tener en cuenta los medios necesarios para la placa de control T0.
- Prepare una concentración de 10x del tinte de ADN seleccionado en un medio de crecimiento completo. Esta concentración se basa en la concentración seleccionada en el paso 2.11. El volumen total de esta solución se calcula en el paso 3.8.
- Usando el tinte de ADN + medio de crecimiento del paso 3.9, prepare una concentración 10x de cada medicamento que se va a probar. Cree solo la dosis más alta de cada fármaco en la placa de dilución del fármaco y diluya en serie los fármacos con una pipeta multicanal, mezclando 15 veces entre cada pocillo.
NOTA: Al hacer la dosis más alta para un medicamento determinado en la placa de dilución del medicamento, se debe tener en cuenta el volumen del medicamento para garantizar que la concentración sea precisa. - Con una pipeta multicanal, añada 10 μL de fármaco + solución de colorante de ADN del paso 3.10 a las placas que contienen las células.
NOTA: En el caso de las placas de dilución de fármacos en las que se ha creado una dilución en serie, si se añaden fármacos a las células empezando por la concentración más baja del fármaco y subiendo hasta la concentración más alta, no es necesario cambiar las puntas de la pipeta multicanal. Sin embargo, las puntas de la pipeta multicanal deben cambiarse entre cada placa y siempre que las puntas se hayan utilizado en un pocillo que contenga un fármaco diferente o una concentración más alta del fármaco. - Lea la fluorescencia de todas las placas en las que se agregaron medicamentos. Esta lectura de fluorescencia es la lectura T0 (es decir, células muertas en el tiempo = 0 h).
- Regrese las placas a la incubadora después de tomar la lectura de fluorescencia T0.
- Añada 10 μL de la solución de colorante de ADN 10x creada en el paso 3.9 y 10 μL de la solución Triton-X al 1,5% en el paso 3.7 a la placa de control T0. Regrese esta placa a la incubadora durante la cantidad de tiempo seleccionada en el paso 1.8.
- Lea la placa de control T0 una vez que las células estén completamente lisadas, como se describe en el paso 1.6.
4. Mida la fluorescencia de las células muertas a lo largo del tiempo para placas tratadas con medicamentos
NOTA: Minimice el tiempo que las placas están fuera de la incubadora. Los cambios prolongados de temperatura pueden afectar a la viabilidad celular, y la exposición a la luz puede comprometer los fluoróforos de ADN, como SYTOX Green.
- Adquiera lecturas de fluorescencia para todas las placas tratadas con medicamentos cada 3-4 horas después de la administración del medicamento. Las placas no necesitan leerse durante la noche a menos que se requiera una cinética de muerte muy precisa.
NOTA: No es necesario tomar los puntos de tiempo a intervalos fijos/estándar. En general, los puntos de tiempo se pueden tomar exclusivamente durante las horas normales de trabajo sin causar errores en el análisis posterior de la cinética de muerte. La cinética se puede inferir siempre y cuando se tomen algunas mediciones en el período anterior al inicio de la muerte, durante la fase ascendente de la muerte y durante la fase de saturación/meseta. - En el punto de tiempo final deseado, adquiera una lectura de fluorescencia. Inmediatamente después, añadir 10 μL de la solución de Triton-X al 1,5% preparada en el paso 3.7 para lisar las células.
- Regrese las placas a la incubadora y deje que las células se permeabilicen durante el tiempo determinado en el paso 1.
- Adquirir lecturas de fluorescencia después de la permeabilización. Este valor de fluorescencia es proporcional al número total de células (es decir, células vivas + células muertas) de cada pocillo.
5. Calcular la cinética de la fracción letal
NOTA: Los cálculos descritos en este protocolo se pueden analizar en cualquier formato o software. Sin embargo, el uso de un entorno de programación como MATLAB, R o Python permitirá un análisis más rápido y flexible.
- Calcule los valores medios de fluorescencia de la placa de control T0 utilizando la media recortada al 50%. Este valor es proporcional al número total de células antes de la medicación.
- Usando el ajuste de la curva y una función de crecimiento exponencial, calcule la cinética del crecimiento de la población para todos los pozos del experimento. El tamaño inicial de la población para cada pocillo es el valor medio de fluorescencia T0 calculado en el paso 5.1. El tamaño final de la población para cada pozo es el valor posterior a la permeabilización calculado en el paso 4.4. La duración del ensayo es desde 0 h hasta el punto final del ensayo seleccionado en el paso 4.4.
- Utilizando los parámetros de crecimiento determinados en el paso 5.2, calcule el número total de células en cada punto de tiempo medido en el ensayo para cada pocillo. Determine el número de células vivas en cada punto de tiempo medido restando la medición de células muertas del total de células calculadas en el paso 5.2.
NOTA: Debido a las pequeñas cantidades de ruido en el ensayo, en ocasiones, el número de células vivas puede ser un pequeño número negativo. Esto se puede establecer manualmente en 0 ya que no puede haber un número negativo de células vivas y lo más probable es que indique que todas las células de ese pozo estaban muertas. - Calcule la fracción letal (LF) para cada punto de tiempo dividiendo la señal de fluorescencia de la célula muerta por la señal de célula total para cada punto de tiempo.
- Ajuste una ecuación de muerte exponencial por retraso (LED) a los datos del curso de tiempo de la fracción letal10. Para evitar parámetros cinéticos arbitrarios para dosis de un fármaco que no inducen ninguna letalidad significativa, ajústelo a un modelo lineal con una pendiente igual a 0. Determine los niveles significativos de letalidad en función del ruido en el ensayo o la LF observada para las condiciones no farmacológicas.
- Registre el tiempo de inicio de la muerte (DO) de la ecuación del LED. La ecuación del LED tiene cuatro parámetros: el LF inicial (LFi), el LF en la meseta (LFp), la tasa de mortalidad inicial (DR) y el OD. Deduzca estos parámetros a partir de los datos utilizando una regresión no lineal (es decir, ajuste de curva).
- Calcule la viabilidad fraccional (FV) en el criterio de valoración del ensayo para cada fármaco en cada dosis. Reste el valor LF del punto final de 1 o divida el número de celdas vivas por el total de celdas.
NOTA: El FV se puede calcular en cualquier punto temporal, no solo en el punto final del ensayo. Estos datos se utilizan para evaluar la farmacología de la dosis (p. ej., fármaco IC50 o EC50).
6. Calcular el valor GR
- Determine el número promedio de células vivas para cada pocillo al comienzo del ensayo. Calcule este valor utilizando la lectura de T0 posterior a la permeabilización calculada en el paso 3.15 y restando la lectura de T0 para cada pocillo, que se recopiló en el paso 3.12. En la ecuación GR que se muestra a continuación (paso 6.3), este valor se denomina x0.
NOTA: Ocasionalmente, la dosis más alta de un medicamento puede interferir con la lectura de fluorescencia del tinte de ADN. Puede ser conveniente calcular el número medio de células vivas en el tiempo 0 utilizando los pozos de control. - Para los pocillos de control (xctrl), determine el número promedio de celdas vivas para cada pocillo en el punto final del ensayo. Determine el número promedio de células vivas en el criterio de valoración del ensayo para cada afección tratada con fármaco (x(c)).
NOTA: Dependiendo de la variación en el crecimiento celular a través de la placa, se pueden usar diferentes valores de xctrl para la normalización de cada pocillo. Por ejemplo, si los valores de xctrl varían sistemáticamente a lo largo de la placa, puede ser conveniente utilizar el valor dex ctrl más cercano en lugar del valor medio de xctrl de la placa. - Para cada pozo tratado con fármaco, calcule el valor de inhibición de la tasa de crecimiento (GR) normalizado utilizando la siguiente ecuación4:
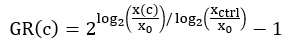
Realice el ajuste de curvas utilizando una regresión logística de 4 parámetros. GR está en una escala de -1 a 1.
7. Calcular las tasas de crecimiento y mortalidad inducidas por fármacos utilizando el método GRADE
NOTA: GR representa la tasa neta de crecimiento de la población, no la verdadera tasa de proliferación celular. Las tasas de crecimiento poblacional y mortalidad inducidas por drogas se pueden calcular utilizando una combinación de la GR y la fracción letal (LF).
- Determine el tiempo de duplicación (τ) de la línea celular durante el experimento utilizando el número promedio de células vivas de la lectura de control T0 (x0), el número promedio de células vivas de la condición de control en el punto final del ensayo (xctrl) y la longitud del ensayo en horas (t) para resolver la siguiente ecuación:

NOTA: El cálculo preciso del tiempo de duplicación de células (τ) requiere que las células se dupliquen continuamente a lo largo del ensayo. En el caso de las células que experimentan inhibición por contacto, las células deben sembrarse a densidades iniciales que no confluyan durante el tiempo de ensayo deseado. Por ejemplo, U2OS tiene un tiempo medio de duplicación de 24 h. La siembra de 2000 células por pocillo en un ensayo de 72 h es óptima para interpretar su tasa de crecimiento. - Determinar la relación entre las tasas de crecimiento/mortalidad y el tamaño de la población. Para ello, utilice un modelo de nacimiento/muerte y una simulación inicializada con todas las combinaciones plausibles de tasas de crecimiento y mortalidad. Esta simulación requiere el número promedio de células vivas de la placa de control T0 (x0), la longitud del ensayo (t) y un rango definido por el usuario de tasas de proliferación plausibles (p) y tasas de mortalidad (d).

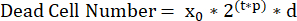
- Para determinar valores plausibles para las tasas de crecimiento, comience con la tasa de crecimiento no tratada en la población celular duplicada por h (1/T) como la tasa de crecimiento más alta y 0 como la tasa de crecimiento más baja. Divida este rango en 500 segmentos igualmente espaciados. Se puede aplicar una operación similar para la tasa de mortalidad, probando un rango de 0 a 1.
NOTA: Todas las combinaciones de pares de 500 velocidades se pueden realizar utilizando cualquier entorno de programación, pero pueden ser demasiado intensas computacionalmente si se utiliza algún software. Reducir esto a todas las combinaciones de pares de 50 tasas de crecimiento y mortalidad puede aliviar este problema, con una reducción en la precisión de la inferencia. Se ha proporcionado una plantilla de hoja de cálculo con este protocolo.
- Para determinar valores plausibles para las tasas de crecimiento, comience con la tasa de crecimiento no tratada en la población celular duplicada por h (1/T) como la tasa de crecimiento más alta y 0 como la tasa de crecimiento más baja. Divida este rango en 500 segmentos igualmente espaciados. Se puede aplicar una operación similar para la tasa de mortalidad, probando un rango de 0 a 1.
- Determine los valores de GR y LF (descritos anteriormente) para cada par de tasas de proliferación (p) y mortalidad (d) simuladas.
NOTA: Los pasos 7.2-7.3 generarán una tabla que contiene cada par teórico de tasas de proliferación y mortalidad, con los valores calculados de GR y LF para ese par teórico. Esto funcionará como una tabla de búsqueda para asociar un par de valores de GR y LF observados experimentalmente con un par de tasas de proliferación y mortalidad inducidas por fármacos. El análisis de más de 500 valores discretos para las tasas de proliferación y mortalidad dará como resultado un valor más preciso numéricamente, pero es probable que este nivel de precisión esté más allá de la precisión del ensayo. - Calcule la distancia por pares entre cada par GR/LF calculado experimentalmente y cada par GR/LF teórico en la tabla de búsqueda. Inferir que el par teórico con la distancia mínima al par de valores GR/LF observados experimentalmente son las verdaderas tasas de proliferación y mortalidad inducidas por fármacos.
8. Determinación de vías de muerte mediante inhibidores químicos selectivos de vías
NOTA: Los inhibidores químicos por sí solos son insuficientes para determinar definitivamente el mecanismo de muerte de un fármaco determinado. Los inhibidores químicos de las vías de muerte deben utilizarse para determinar qué respuestas bioquímicas o fenotípicas deben explorarse en experimentos posteriores, que probablemente incluyan la evaluación morfológica, los marcadores bioquímicos específicos de la vía y la evaluación de las dependencias genéticas.
- Optimizar la dosis de cada inhibidor de la muerte celular en la línea celular de interés mediante la prueba de un rango de dosis de concentraciones de inhibidores en el contexto de un activador canónico de la(s) vía(s) de muerte de interés (Tabla 2). Idealmente, la dosis seleccionada de inhibidor no debería afectar la viabilidad celular por sí sola (consulte la métrica GR).
NOTA: No todas las líneas celulares pueden activar todas las vías de muerte. Es posible que se requiera una validación bioquímica o fenotípica cuando se prueban activadores/inhibidores de diferentes vías de muerte. - Considere el diseño óptimo de la placa para una pantalla inhibidora. Para minimizar los efectos del lote, mantenga cada medicamento en la misma placa que los inhibidores que se están evaluando, con réplicas en placas separadas.
- Siembre el número deseado de células en cada pocillo de una placa de 96 pocillos, con fondo óptico y paredes negras, similar al paso 3.3. Disminuya el volumen de siembra a 80 μL para tener en cuenta los volúmenes del inhibidor y del fármaco.
- Prepare 10 veces la concertación de los inhibidores que se van a probar en medios completos y agregue 10 μL a cada pocillo. Tratamiento previo de las células con inhibidores de la vía de muerte durante 2-4 h. Posteriormente, agregue los medicamentos de interés y adquiera la lectura de fluorescencia, como en los pasos 3-5.
- Evaluar el cambio en el tiempo de inicio de la muerte (OD) y/o la fracción letal máxima para evaluar la efectividad de los inhibidores. El DO es un parámetro inferido del modelo de muerte exponencial de retraso (LED). Consulte los pasos 5.6 y 5.7.
Resultados
Utilizando este protocolo, exploramos la sensibilidad de las células U2OS al inhibidor de HDAC Belinostat. Estos experimentos se realizaron utilizando 2 μM de SYTOX Green para etiquetar las células muertas (Figura 1A). Las lecturas cinéticas se realizaron utilizando un lector de placas fluorescentes a un ajuste de ganancia de 130 (Figura 1B). Las células se lisaron en una solución de Triton-X al 1,5% en PBS durante 2 h al final del ensayo (Figura 1C-D).
El protocolo FLICK proporciona información sobre la tasa de crecimiento de la población y la tasa de muerte celular inducida por fármacos. Estos datos se pueden ver por separado o mediante el marco de visualización y análisis de GRADE del fármaco, en conjunto (Figura 1E). La evaluación de la sensibilidad de Belinostat utilizando el valor GR revela que 1 μM de Belinostat da como resultado un valor de GR de aproximadamente 0 (Figura 2A). En la escala GR, los valores positivos informan de la tasa de expansión de la población, y los valores negativos informan de la tasa de contracción de la población. Por lo tanto, un valor de GR de 0 revela que la población permanece en estasis (es decir, una respuesta citostática al fármaco). Dada la interpretación convencional de la citostasis, se puede concluir que 1 μM de Belinostat provoca una inhibición completa del crecimiento sin ninguna muerte celular. Sin embargo, la evaluación de la fracción letal inducida por el fármaco revela que 1 μM de Belinostat causó aproximadamente un 50% de letalidad en el transcurso de este ensayo (Figura 2B). En la mayoría de los casos, estas dos percepciones se derivarían de diferentes experimentos que no se pueden comparar de una manera comparada. Es importante destacar que, al utilizar el ensayo FLICK, ambos conocimientos se derivan de los mismos datos experimentales, pero se analizan de manera diferente para capturar los efectos de Belinostat en el crecimiento de la población frente a la muerte celular.
Estos dos conocimientos también se pueden integrar utilizando el método analítico GRADE (Figura 2C). El grado de fármaco yuxtapone la fracción letal, que es proporcional a la tasa de mortalidad media, con el valor de GR, la tasa neta de crecimiento de la población. La integración de los datos de esta manera proporciona una visualización de cómo un fármaco coordina la inhibición del crecimiento y la activación de la muerte. Dentro de la gráfica GRADE: los fármacos que inhiben el crecimiento sin activar la muerte (es decir, los fármacos citostáticos, utilizando la interpretación común) ocuparán el límite superior, los fármacos que sólo matan sin alterar las tasas de proliferación celular ocuparán el límite derecho, y los fármacos que primero causan inhibición del crecimiento, seguida de la muerte de las células detenidas en el crecimiento ocuparán el límite izquierdo (es decir, fármacos bifásicos; Figura 2C). Se puede inferir que los fármacos que dan lugar a respuestas GR/LF que caen dentro de estos límites inhiben simultáneamente la proliferación hasta cierto punto y al mismo tiempo activan la muerte celular (es decir, fármacos coincidentes)3. Es importante destacar que la posición de la respuesta de cada fármaco dentro del espacio GR/LF puede utilizarse para calcular la tasa de proliferación real (p) y la tasa media de mortalidad (d) para cada concentración de un fármaco (Figura 2D-E). Tenga en cuenta que el valor de GR no es la tasa de proliferación celular real, sino más bien la tasa neta de crecimiento de la población (es decir, el efecto neto que combina la tasa de proliferación real y la tasa de mortalidad inducida por fármacos). Además, la tasa de mortalidad producida en un análisis GRADE (d, que es la tasa de mortalidad promedio) es distinta del parámetro de tasa de mortalidad en el análisis cinético de LED (DR, la velocidad máxima o la tasa de mortalidad máxima).
El ensayo FLICK puede generar datos específicos de la muerte, como la fracción letal inducida por el fármaco; Sin embargo, estos datos son independientes del mecanismo de muerte celular. Para obtener información sobre el mecanismo de la muerte celular utilizando FLICK, el método más simple es determinar cómo se altera la cinética de la fracción letal mediante la inclusión de inhibidores específicos de la vía de muerte. La inhibición de un mecanismo de muerte relevante debería resultar en una disminución de la fracción letal inducida por el fármaco y/o retrasar el momento de inicio de la muerte. Aquí, mostramos que 50 μM del inhibidor de pancaspasa z-VAD rescatan aproximadamente el 50% de la letalidad inducida por 1 μM de Belinostat (Figura 2F). Es importante destacar que, al igual que los fármacos, los inhibidores tienen una especificidad limitada y pueden inhibir o exacerbar accidentalmente otros mecanismos de muerte. Por lo tanto, antes de llegar a una conclusión definitiva, estos datos deben complementarse con otros conocimientos, como la morfología celular, la medición de los marcadores bioquímicos de activación y/o la evaluación de las dependencias genéticas específicas de la vía14.
Para ilustrar formalmente cómo la interpretación de la respuesta a un fármaco puede verse alterada por una evaluación basada en FLICK y un análisis basado en GRADE, a continuación exploramos tres fármacos que presentan diferentes tipos de coordinación de crecimiento/muerte. Palbociclib es un inhibidor de Cdk4/6 que produce una respuesta citostática (inhibición del crecimiento sin muerte celular). La camptotecina es un inhibidor de la topoisomerasa I que provoca una respuesta bifásica (inhibición del crecimiento, seguida de muerte celular a dosis altas). Belinostat es un inhibidor de HDAC que provoca una respuesta coincidente (inhibición parcial del crecimiento y activación de la muerte en cada dosis, pero con proporciones variadas entre dosis). Utilizando un análisis convencional de estos datos, se puede observar que los tres fármacos disminuyen sustancialmente la viabilidad relativa de las células U2OS (Figura 2G). Aunque el palbociclib es notablemente menos potente, no está claro a partir de estos datos que el palbociclib no active la muerte celular. Utilizando un análisis basado en GR, que informa sobre cómo los fármacos afectan a la tasa de crecimiento neto de la población, se puede interpretar con mayor precisión que las poblaciones tratadas con palbociclib continúan expandiéndose con todas las dosis, mientras que las dosis altas de Camptotecina o Belinostat dan lugar a una reducción de la población y, por lo tanto, la muerte celular debe haberse activado después de dosis altas de estos fármacos (Figura 2H). Sin embargo, un análisis basado en GR no tiene en cuenta la coordinación variada entre el crecimiento y la muerte, lo que puede llevar a conclusiones erróneas sobre el nivel de muerte celular activado por un fármaco determinado. Por ejemplo, sobre la base de los datos de GR, se podría concluir que 1 μM de Camptotecina activa la muerte celular en mayor medida que 1 μM de Belinostat, dado que Camptotecina da como resultado un valor de GR de -0,25 y Belinostat da como resultado un valor de GR de 0; un valor de -0,25 GR significa que la población se está reduciendo un 25% tan rápido como se está expandiendo la población no tratada. Utilizando una visualización y análisis basados en GRADE, se puede observar que Camptothecin y Belinostat difieren en su coordinación de crecimiento/muerte. Por lo tanto, a la dosis de 1 μM, ambos Belinostat activan la muerte a una tasa mayor que la Camptotecina (0,8% de muerte/hora para Belinostat, en comparación con 0,7% de muerte/hora para Camptotecina), pero la población tratada con Camptotecina se reduce más rápidamente debido a una inhibición del crecimiento más pronunciada inducida por este fármaco (Figura 2I).
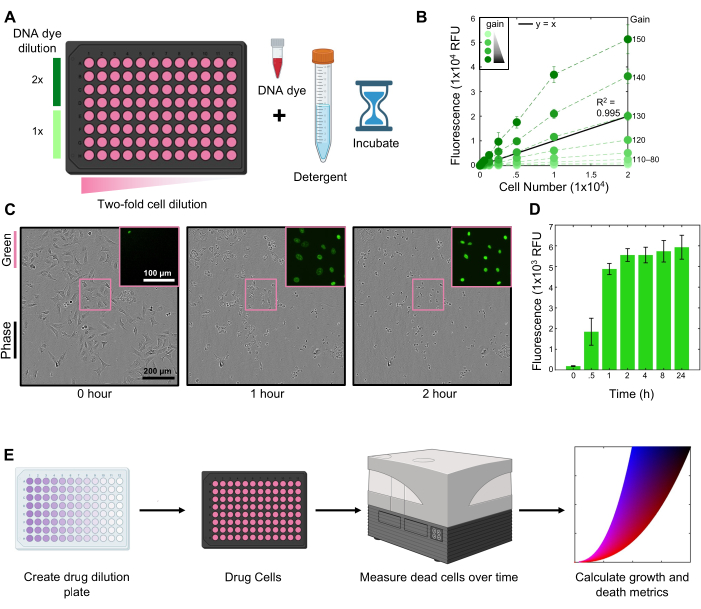
Figura 1: Diagrama de flujo del protocolo FLICK. (A) Cree una dilución celular para optimizar la concentración de colorante de ADN y la linealidad de las mediciones de fluorescencia en un lector de microplacas. (B) Titulación de ganancia a través del número de células para 2 μM SYTOX Green en células U2OS. Una ganancia de 130 proporciona la señal más lineal en todos los números de celda. (C) Imágenes de fase de células U2OS lisadas con Triton-X al 1,5% a lo largo del tiempo utilizando un objetivo 10x. La región en caja es una vista ampliada de la fluorescencia verde de SYTOX. Las barras de escala son representativas de todas las imágenes. (D) Señal verde SYTOX después de la lisis en los momentos indicados. Los datos son media +/- DE para 3 réplicas biológicas. (E) Una visión conceptual del ensayo, desde la configuración hasta el análisis. Los pasos clave son la creación de una placa de dilución del fármaco, la farmacología de las células en una placa de pocillos múltiples, la adquisición de mediciones de fluorescencia a lo largo del tiempo y el cálculo de las métricas de crecimiento y muerte. Para los paneles B y D, los datos son la media ± SD para n = 3 réplicas biológicas independientes. Haga clic aquí para ver una versión más grande de esta figura.
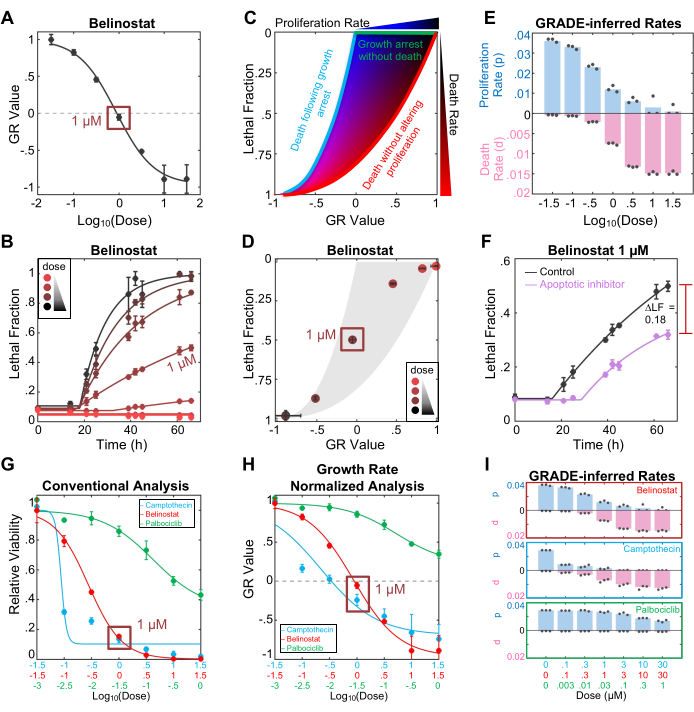
Figura 2: Caracterización de las respuestas a los fármacos mediante el ensayo FLICK y el análisis basado en GRADE. (A) Métrica GR para el inhibidor de HDAC, Belinostat, sobre una dosis de dosis. Los datos mostrados son del punto final del ensayo, que fue de 66 h. A 1 μM, Belinostat tiene un valor GR de aproximadamente 0, lo que indica una respuesta citostática. (B) Cinética de fracción letal para Belinostat. A 1 μM, Belinostat induce casi un 50% de letalidad, que no es capturada por el valor GR. (C) Diagrama de fases GRADE basado en una simulación de todas las tasas posibles de crecimiento y mortalidad. La simulación se basó en el tiempo de duplicación de U2OS en este ensayo (27,12 h). (D) Gráfico GRADE para Belinostat a través de la dosis en el punto final del ensayo. (E) Diagrama de barras de las tasas de crecimiento inferido por GRADE (p, duplicación de la población por hora) y muerte (d, fracción de la población que muere por hora) para las dosis de Belinostat. Las tasas se determinaron a partir de las tasas simuladas en (C) y los datos de (D). (F) Gráfico de fracción letal para 1 μM de Belinostat tratado con (púrpura) o sin (negro) 50 μM del inhibidor apoptótico z-VAD. ΔLF = 0,18 se calculó restando la LF máx. promedio de la afección tratada con inhibidores de la LF máx. promedio del control. (G) Viabilidad relativa (número de células vivas en la afección tratada con fármaco, dividido por el número de células vivas en la afección no tratada) para tres fármacos después de una exposición de 72 h. Todos los fármacos reducen la viabilidad celular, pero se desconoce la contribución de la muerte celular a la respuesta. (H) Métrica GR para tres medicamentos en (G). Estos datos demuestran que dos de los tres fármacos provocan una disminución de la población, indicada por el GR < 0. La contribución de la muerte celular aún no está clara, aunque la muerte celular debe haberse activado cuando el valor de GR es negativo. (I) Tasas inferidas por GRADE para tres medicamentos. El palbociclib no es letal, pero provoca la detención del crecimiento. La camptotecina es bifásica; Las dosis más bajas dan como resultado la detención del crecimiento, mientras que las dosis más altas resultan en la detención del crecimiento seguida de la letalidad. Belinostat es un fármaco coincidente; el rango de dosis corta a través de la mitad de la gráfica GRADE, lo que indica que todas las dosis dan como resultado una perturbación del crecimiento y la activación de la muerte celular. Los datos de todos los paneles son la media ± SD para tres experimentos de réplicas biológicas independientes. Haga clic aquí para ver una versión más grande de esta figura.
| Teñir | Cinético | Punto final |
| SYTOX Verde | Y | Y |
| CellTox Verde | Y | Y |
| NucSpot 500/515, 594/615, 750/780 | Y | Y |
| YOYO-3 | Y | Y |
| Yoduro de propidio | N | Y |
| 7-AAD | N | Y |
Tabla 1: Colorantes de ADN y su aplicación en FLICK. Se pueden utilizar muchos tipos de colorantes para los ensayos FLICK de solo punto final. Menos colorantes cumplen las condiciones para ser incubados continuamente durante todo el ensayo. Otros tintes, incluidos los tintes que no son verdes, no se han caracterizado profundamente y deben evaluarse cuidadosamente antes de su uso.
| Camino de la muerte | Inhibidor | Dosis típica |
| Apoptosis intrínseca | ZVAD-FMK | 50 μM |
| Apoptosis extrínseca | Z-IETD | 30 μM |
| Ferroptosis | Ferrostatina-1 | 10 μM |
| Necroptosis | Necrostatina-1 | 50 μM |
| Piroptosis | VX-765 | 50 μM |
| Parthanatos | Rucaparib | 1 μM |
| Muerte de células dependientes autofágicas | Hidroxicloroquina (HCQ) | 10 μM |
| Muerte de células lisosomales dependientes | E-64D | 10 μM |
| Cuproptosis | Tetratiomolibdato (TTM) | 5 μM |
| Oxeiptosis | N-acetil-l-cisteína (NAC) | 2 mM |
| Necrosis impulsada por MPT | Ciclosporina A (CsA) | 10 μM |
Tabla 2: Vías de muerte celular y sus inhibidores. Los inhibidores de la muerte celular deben validarse frente a un activador canónico para cada vía de muerte. Lo ideal es que las dosis seleccionadas de inhibidores no afecten a la viabilidad celular.
Tabla complementaria 1: Calculadora GRADE. Hoja de cálculo para calcular las tasas de crecimiento y mortalidad inducidas por fármacos a partir de datos de GR y FV basados en FLICK. Los usuarios deben ingresar los datos de GR y FV, junto con la duración del ensayo en horas, el tiempo de duplicación de celdas en horas y los tamaños de población inicial/final. Haga clic aquí para descargar este archivo.
Discusión
El ensayo FLICK es un método sólido para generar una evaluación completa del efecto de un fármaco en el crecimiento y la muerte de una población celular. Debido a que este método no cuenta directamente las células, los pasos críticos del protocolo son garantizar la linealidad del ensayo y la lisis completa durante los pasos de permeabilización del tritón. El tiempo de permeabilización correcto se puede identificar visualmente, como se destaca en este protocolo, o cuantitativamente mediante la lectura de la fluorescencia de la placa a lo largo del tiempo e identificando cuándo se estabiliza la señal. Sin embargo, en nuestra experiencia, en caso de duda, no parece haber ningún coste por esperar más tiempo: para los tintes de ADN que hemos utilizado en el ensayo FLICK, la intensidad de la señal permanece estable durante varios días después de la lisis. Utilizamos regularmente la tinción de ácido nucleico SYTOX Green, y aunque no hemos validado cada fluoróforo al que se hace referencia en la Tabla 1, varios otros protocolos demuestran su uso efectivo 13,15,16. La evaluación de la linealidad de un determinado colorante de ADN impregnado de células en una gama de números de células y configuraciones de instrumentos garantizará datos sólidos y cuantitativamente fiables.
Algunas líneas celulares son más difíciles de lisar que otras, y nunca se debe asumir que el tiempo de lisis de una línea celular es el mismo que el de otras13. Para algunas líneas celulares, a densidades más altas, la lisis celular hace que las células se levanten del pocillo como una lámina, lo que puede causar un error en el lector de placas. Esto se puede evitar reduciendo la densidad de siembra inicial o mezclando los pocillos con pipeta después de la lisis y antes de la lectura. Para mejorar el tiempo de lisis o la eficiencia de la lisis, la solución Triton-X se puede ajustar a un porcentaje mayor o menor, pero aún no hemos encontrado un escenario en el que esto sea necesario.
El diseño experimental y la disposición de la siembra de fármacos deben considerarse cuidadosamente cuando se desean datos de crecimiento sólidos. La creación de una placa de dilución de fármaco para el ensayo FLICK es la misma que cualquier otro ensayo de placa de 96 pocillos. Sin embargo, incluir muchos controles que cubran diferentes partes del paisaje de una placa ayudará a crear información sólida sobre la cinética del crecimiento y puede ayudar a determinar si existe una variación sistemática del crecimiento. Evitar los pozos exteriores de una placa producirá datos de crecimiento más precisos, ya que estos pozos son más sensibles a las fluctuaciones de temperatura, oxígeno o humedad, afectando su crecimiento. Por último, el uso de un patrón de farmacograma pseudoaleatorio, como el cambio de la ubicación de un fármaco en placas replicadas, proporcionará una evaluación más precisa del comportamiento de los fármacos.
Existen algunas limitaciones para el uso preciso del ensayo FLICK. Estas limitaciones están relacionadas principalmente con la inferencia cinética, ya que los datos de los puntos finales son valores observados experimentalmente. Tanto la cinética de proliferación de células vivas como la cinética de muerte dependen de alguna suposición con respecto a la trayectoria de crecimiento de las células vivas a lo largo del tiempo en la afección tratada con fármaco. En este protocolo, describimos esta trayectoria de crecimiento como exponencial y con una tasa uniforme en el tiempo. Si bien es probable que estas características se observen en ausencia de un medicamento, es posible que no se cumplan en presencia de un medicamento. El método FLICK es preciso, no porque las suposiciones sobre las trayectorias de crecimiento sean siempre correctas, sino porque el impacto de cualquier suposición incorrecta se minimiza si la población tratada con fármacos no prolifera mucho (es decir, menos de 2 o 3 duplicaciones de población en el período de ensayo). No hemos encontrado fármacos que no puedan perfilarse con precisión en FLICK; sin embargo, la teoría sugeriría que el ensayo FLICK dejará de ser preciso para un fármaco en el que la muerte se produce muy lentamente, durante largos períodos de tiempo que son varios múltiplos del tiempo de duplicación de la célula (nota: no hemos identificado ningún fármaco con estas características3). Sin embargo, el impacto de cualquier suposición incorrecta para la cinética de crecimiento de la población total no afectará el tiempo de inicio de la muerte obtenido a partir del ajuste del LED o los valores máximos finales de LF, ya que estos están limitados por los valores de células muertas medidos empíricamente y los valores finales de células totales que se determinaron experimentalmente al final del ensayo.
A pesar de estas limitaciones, el ensayo FLICK permite obtener información que es difícil de generar con otros métodos de respuesta a los fármacos. La mayoría de los métodos de respuesta a fármacos generan una señal proporcional al número de células vivas, y estos métodos pueden utilizarse para cuantificar cómo afectan los fármacos a la tasa neta de crecimiento de la población (es decir, el valor de GR o equivalente), pero no pueden discriminar con precisión entre los efectos citotóxicos y citostáticos de un fármaco. Por otra parte, los ensayos basados en microscopía que miden tanto las células vivas como las muertas pueden generar una imagen completa. Sin embargo, los ensayos basados en microscopía pueden tener dificultades para contar las células muertas después de que las células se descomponen en desechos, como sucederá rápidamente en el contexto de muchas formas no apoptóticas de muerte celular. Una característica clave de FLICK es que las mediciones se realizan en un lector de placas, que agrega la fluorescencia total de las células muertas en lugar de contar directamente las células muertas. Por lo tanto, la señal de células muertas en FLICK no depende de la celularidad intacta de las células muertas, que, en el contexto de la muerte celular no apoptótica, es excepcionalmente esencial. Además, una característica única del ensayo FLICK es la capacidad de medir tanto las poblaciones de células vivas como las muertas utilizando el mismo reactivo. Por lo tanto, el ensayo FLICK produce mediciones de células vivas y muertas con la misma sensibilidad. Esta característica permite el análisis utilizando el método GRADE y mejora la precisión de los cálculos simultáneos de la tasa de crecimiento y mortalidad de GRADE. La Tabla Suplementaria 1 incluida incluye una plantilla utilizada para la visualización del GRADE del fármaco, donde el usuario puede ingresar la longitud del ensayo, la tasa de crecimiento calculada del paso 7.1, los números de células iniciales y finales o las lecturas fluorescentes de las condiciones de control, y la LF y GR calculadas de los pasos 5.4 y 6.3. El archivo contiene una simulación de todas las combinaciones por pares de 50 tasas de crecimiento y mortalidad que se actualizarán automáticamente en función de los parámetros definidos por el usuario. La plantilla genera una visualización basada en GRADE de la respuesta al fármaco y las tasas de crecimiento (p) y mortalidad (d) inferidas por GRADE.
El trabajo futuro debería explorar la aplicación del ensayo FLICK en escenarios de cultivo celular en 3D o en la evaluación de las respuestas a los fármacos de organoides derivados de tumores. La linealidad y la sensibilidad de los datos FLICK no se han cuestionado en profundidad en estos contextos, pero en teoría, el ensayo FLICK debería ser efectivo con algunas modificaciones para integrar la fluorescencia en muestras tridimensionales. Además, la adición de etiquetas adicionales específicas para cada célula ayudaría a discriminar entre dos o más tipos de células en el cocultivo. Estos avances serán valiosos para explorar las interacciones de las células inmunitarias con las células cancerosas. Por último, ensayos como SPARKL inspiran el uso de indicadores específicos de muerte en el formato de ensayo FLICK, lo que puede mejorar el rendimiento al tiempo que mantiene la precisión de los conocimientos sobre la muerte celular11,16.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos a todos los miembros pasados y presentes del Laboratorio Lee por sus contribuciones a la perspectiva de nuestro laboratorio sobre la evaluación de las respuestas a los medicamentos. Este trabajo fue apoyado por fondos de los Institutos Nacionales de Salud para MJL (R21CA294000 y R35GM152194).
Materiales
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
Referencias
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados