Method Article
Umfassende Analyse des Wirkstoffansprechens mit dem FLICK Assay
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt, wie der FLICK-Assay zur Bewertung von Arzneimittelreaktionen verwendet wird, einschließlich detaillierter Anweisungen für die Verwendung dieses Assays zur Berechnung der arzneimittelinduzierten Wachstumsraten und Todesraten und zur Bewertung des Mechanismus des arzneimittelinduzierten Zelltods.
Zusammenfassung
Um die Wirksamkeit von Medikamenten zu verstehen, ist es von entscheidender Bedeutung, das Ausmaß des medikamenteninduzierten Zelltods zu charakterisieren. Die Bemühungen, das Ausmaß des medikamenteninduzierten Zelltods zu quantifizieren, werden durch die Existenz von mehr als einem Dutzend molekular unterschiedlicher Formen des regulierten Todes in Frage gestellt, von denen jede ihren eigenen Aktivierungszeitpunkt und biochemischen Merkmale aufweist. Darüber hinaus werden bei einigen nekrotischen Todessubtypen charakteristische Merkmale nur vorübergehend beobachtet und gehen aufgrund von Zellrupturen schnell verloren. Selbst bei Verwendung einer Kombination von Assays, die für den Todesweg spezifisch sind, ist es daher schwierig, die Gesamtmenge des Zelltods oder die relativen Beiträge der einzelnen Todessubtypen genau zu quantifizieren. Ein weiteres Problem ist, dass viele todesspezifische Assays ignorieren, wie sich Medikamente auf die Zellproliferation auswirken, was es schwierig macht, zu interpretieren, ob eine medikamentös behandelte Population wächst oder schrumpft. Der FLICK-Assay ermöglicht die Quantifizierung des Gesamtgrades des Zelltods nach der Stimulation auf eine Weise, die spezifisch für den Tod, aber auch weitgehend unabhängig von der Art und Weise des aktivierten Todes ist. Darüber hinaus speichert der FLICK-Assay Informationen über die Gesamtpopulationsgröße und die Zellproliferationsrate. In diesem Manuskript beschreiben wir die grundlegende Anwendung des FLICK-Assays, wie dieser Assay bei der Verwendung verschiedener Arten von biologischem Material behoben werden kann und wie der FLICK-Assay verwendet werden kann, um den Beitrag jeder Art von Zelltod zu einer beobachteten Arzneimittelreaktion zu quantifizieren.
Einleitung
Bei Krebsmedikamenten umfasst die präklinische Bewertung der Arzneimittelempfindlichkeit in der Regel die Prüfung, wie sich Arzneimittel auf die Lebensfähigkeit von Zellen in Kulturauswirken 1. Die Lebensfähigkeit von Zellen nach Arzneimittelexposition ist ein Produkt von mindestens zwei getrennten Effekten: der arzneimittelinduzierten Hemmung der Zellproliferation und der Aktivierung des Zelltods2. Obwohl der Zelltod ein kritisches Merkmal ist, das für ein dauerhaftes Ansprechen auf Medikamente erforderlich ist, können die Standardansätze leider nicht klären, inwieweit ein Medikament den Zelltod aktiviert3.
Zu den gängigen Assays für das Ansprechen auf Arzneimittel gehören solche, die Zellen direkt zählen (z. B. Coulter Counter, einige Anwendungen der Durchflusszytometrie oder Mikroskopie), die Fähigkeit von Zellen zur Proliferation quantifizieren (z. B. Assay zur Koloniebildung) oder eine metabolische Aktivität quantifizieren (z. B. CellTiter-Glo, MTT- oder MTS-Assays auf Tetrazoliumbasis). Ein gemeinsames Merkmal dieser Assays ist, dass die generierten Daten proportional zur Anzahl der lebenden Zellen sind. Da sich die Art und Weise, wie Medikamente Wachstumshemmung und Zelltod koordinieren, erheblich unterscheiden, liefert die Anzahl lebender Zellen nach Medikamentenexposition einen unzuverlässigen Einblick in das Ausmaß des medikamenteninduzierten Zelltods3. Da sich Krebszellen in Zellkulturen in der Regel schnell vermehren, kann die Anzahl der lebenden Zellen im Vergleich zur unbehandelten Population drastisch reduziert werden, ohne dass es zu Zelltod kommt4. Ein zentraler Fehler besteht also darin, dass der Grad des Zelltods nicht quantifiziert werden kann, ohne sowohl die Anzahl der lebenden als auch der toten Zellen zu messen.
Die genaue Quantifizierung der Anzahl toter Zellen nach Arzneimittelexposition ist aus mehreren Gründen schwierig zu bewerkstelligen2. Erstens gibt es mehr als ein Dutzend regulierte Zelltodwege 5,6,7,8. Obwohl es im Allgemeinen biochemische Marker gibt, um jede Art von reguliertem Zelltod zu identifizieren, variieren diese Marker in ihrer Spezifität, und kein einzelner Assay kann verwendet werden, um alle Todessubtypen gleichzeitig zu quantifizieren. Zweitens kann der Zeitpunkt der Aktivierung für jede Form des Zelltods je nach Kontext sehr stark variieren, so dass ein vollständiges Bild nur entstehen kann, wenn der Tod im Laufe der Zeit quantifiziert wird 9,10. Viele biochemische Assays zur Quantifizierung des Zelltods erzeugen eine Endpunktmessung, so dass die Generierung kinetischer Daten eine Herausforderung darstellen und durch die Kosten begrenzt sein kann. Eine dritte Komplikation besteht darin, dass die tote Zelle selbst ein vorübergehender Zwischenzustand zwischen dem lebenden Zellzustand und dissoziierten Zelltrümmern ist. Die Stabilität toter Zellen variiert je nach Todessubtyp, wobei einige Typen, wie z. B. die Apoptose, relativ stabile Leichen erzeugen, während andere Arten des Todes eine schnelle Lyse verursachen. Daher werden Methoden zur Quantifizierung des Todes, die das Sammeln und Zählen toter Zellen erfordern, auch zu einem verzerrten Verständnis des Zelltods führen. Eine vierte Einschränkung besteht darin, dass biochemische Assays, die den Grad des Zelltods quantifizieren, in der Regel keinen Einblick in die Veränderung der Proliferation eines Medikaments liefern. Daher kann die Gesamtbevölkerungsgröße - und vor allem die Frage, ob die Bevölkerung wächst oder schrumpft - nicht interpretiert werden.
Einige mikroskopiebasierte Assays, wie z. B. STACK und SPARKL, sind wirksam bei der Messung lebender und toter Zellen im Laufe der Zeit, und diese Assays können umfassende Erkenntnisse über den medikamenteninduzierten Zelltod liefern10,11. Diese Assays erfordern jedoch spezielle Instrumente, wie z. B. das Incucyte-Mikroskop, was zu Einschränkungen des Durchsatzes und des Zugangs zu diesen Ansätzen führt. Darüber hinaus erfordern mikroskopiebasierte Techniken, dass tote Zellen während der gesamten Dauer des Experiments in der Brennebene des Mikroskops verbleiben, was die Fähigkeit zur Quantifizierung toter Zellen beeinträchtigt, wenn sie die Haftung an der Platte verlieren oder im Laufe der Zeit, wenn tote Zellen zerfallen. In ähnlicher Weise stehen mikroskopiebasierte Assays vor Herausforderungen, wenn sie im Zusammenhang mit Suspensionskulturen angewendet werden, da Zellen in eine bestimmte Brennebene hinein und aus ihr heraus driften.
Um die oben genannten Probleme zu lösen, haben wir einen Assay namens FLICK (Fluorescence-based and Lysis-dependent Inference of Cell Death Kinetics)12,13 entwickelt. Ziel des FLICK-Assays ist es, den Grad des medikamenteninduzierten Zelltods zu bestimmen, unabhängig davon, wie die Zellen absterben. Bei der FLICK-Methode werden zellundurchlässige Farbstoffe verwendet, deren Fluoreszenz von der DNA-Bindung abhängt. Ein Hauptmerkmal von FLICK ist die Verwendung dieser Fluorophore, um die Akkumulation toter Zellen im Laufe der Zeit aufgrund ihrer zugänglichen DNA zu markieren, gefolgt von einer auf mechanischen Reinigungsmitteln basierenden Lyse, um alle lebenden Zellen am Ende des Assays zu permeabilisieren. Diese Daten, kombiniert mit mathematischer Modellierung, ermöglichen die Quantifizierung sowohl lebender als auch toter Zellpopulationen mit kontinuierlicher zeitlicher Auflösung und ohne dass tote Zellen gesammelt oder gehandhabt werden müssen. Darüber hinaus ermöglicht die Verwendung eines Plattenreaders zur Bewertung der Fluoreszenz toter Zellen die Bewertung toter Zellen, ohne dass tote Zellen intakt bleiben müssen, wodurch eine Verzerrung gegen nekrotische Formen des Todes, die zu einem Zellbruch führen, gemildert wird. Schließlich erfordert der FLICK-Assay nur eine minimale Plattenhandhabung und kann schnell kinetische Messungen erzeugen, was ein Wirkstoffscreening mit hohem Durchsatz ermöglicht. In diesem Protokoll konzentrieren wir uns auf die Verwendung des FLICK-Assays, einschließlich der Verwendung von FLICK, um die medikamenteninduzierte Wachstumsrate, die Sterberate und/oder die Mechanismen des Zelltods abzuleiten.
Protokoll
1. Optimierung der Permeabilisierungszeit für jede Zelllinie von Interesse
HINWEIS: Die beschriebenen Volumina und Mengen dienen zur Optimierung einer Zelllinie. Diese Werte sollten basierend auf der Anzahl der Zelllinien, die getestet werden sollen, hochskaliert werden.
- Plattieren Sie die gewünschte Anzahl von Zellen in jeder Vertiefung einer 96-Well-Platte mit schwarzem Boden und optischem Boden. Fügen Sie 100 μl vollständiges Medium hinzu und lassen Sie die Zellen über Nacht an der Platte haften.
HINWEIS: Die Anzahl der plattierten Zellen sollte unter Berücksichtigung der Wachstumsrate der Zellen, der optimalen Dichte und der Länge des Assays optimiert werden. Typische Ausgangszellzahlen für adhärente Krebszelllinien, die über 72 h gemessen werden, betragen 1500 - 5000 Zellen pro Well. Zum Beispiel können U2OS-Zellen mit 2000 Zellen pro Vertiefung in DMEM mit 10 % FBS, 2 mM Glutamin und 1 % Pen-Strep plattiert und über Nacht unter Standardbedingungen (5 % CO2, 37 °C, mit Feuchtigkeit) kultiviert werden. - Bereiten Sie in einem konischen 15-ml-Zentrifugenröhrchen 1,5 ml einer 1,5%igen Lösung von Triton-X in phosphatgepufferter Kochsalzlösung (PBS) vor. Wirbeln Sie die 1,5%ige Triton-X-Lösung 5 s lang bei maximaler Geschwindigkeit auf. Mindestens 30 min in ein 37 °C warmes Wasserbad stellen.
- Überprüfen Sie die Lösung visuell, um sicherzustellen, dass sich Triton-X durch Vortexen mehrere Sekunden lang vollständig aufgelöst hat. Stellen Sie sicher, dass die Lösung homogen ist.
- Geben Sie 10 μl der 1,5%igen Triton-X-Lösung in jede Vertiefung der plattierten Zellen. Nicht mischen.
HINWEIS: In diesem Stadium ist es nicht wünschenswert, durch wiederholtes Pipettieren zu mischen, da sich Blasen bilden. - Bringen Sie die Zellen für mindestens 1 h in den 5% CO2, 37 °C mit Feuchtigkeits-Zellkultur-Inkubator zurück.
HINWEIS: Nach der Anwendung der Triton-X-Lösung sind die meisten Zelllinien in 2-3 h vollständig lysiert. Eine Wartezeit von bis zu 24 Stunden hat bei der Verwendung von Farbstoffen wie SYTOX Green nur minimale Auswirkungen auf das Fluoreszenzsignal. Eine Erhöhung des Triton-X-Anteils kann die Inkubationszeit für schwer zu lysierende Zelllinien verkürzen. - Beobachten Sie die Zellmorphologien am Lichtmikroskop mit einem 10-fach-Objektiv. Untersuchen Sie die Zellen einmal pro Stunde, bis die Zellkörper nicht mehr sichtbar sind. Notieren Sie die Zeit, die für die Zellpermeabilisierung erforderlich ist.
HINWEIS: Wenn ein Fluoreszenzmikroskop zur Verfügung steht, kann die Lyse mit einem zellundurchlässigen DNA-Farbstoff wie SYTOX Green sichtbar gemacht werden. Der Farbstoff kann in einer 10-fachen Endkonzentration zur Triton-X-Lösung hinzugefügt werden, und die Permeabilisierung kann durch Fluoreszenzmikroskopie bestätigt werden.
2. Auswahl und Kalibrierung des DNA-Farbstoffs
HINWEIS: Eine Voraussetzung für den FLICK-Assay ist die Verwendung eines zellundurchlässigen Fluorophors, der ein Signal in einer DNA-bindungsabhängigen Weise sendet, die Lebensfähigkeit der Zellen nicht beeinträchtigt und ein Signal erzeugt, das linear mit der Zellzahl skaliert. Dieses Protokoll verwendet SYTOX Green. Auch andere Farbstoffe mit ähnlichen Eigenschaften können für den FLICK-Assay geeignet sein, diese sollten jedoch jeweils bewertet und kalibriert werden. Beispiele finden Sie in Tabelle 1 .
- Bestimmen Sie für den ausgewählten DNA-Farbstoff den zu prüfenden Konzentrationsbereich auf der Grundlage der Empfehlung des Herstellers.
- Für jede zu testende Konzentration werden 40.000 Zellen in 180 μl Zellkulturmedium in dreifacher Ausfertigung entlang der äußersten linken Säule einer 96-Well-Platte mit optischem Boden und schwarzen Wänden plattiert. Geben Sie 90 μl Medium in die verbleibenden Vertiefungen.
HINWEIS: Dies wird verwendet, um eine lineare Zelltitration über die Reihen der Platte zu erzeugen. Die Endkonzentration der ersten Wells wird 20.000 Zellen/Well betragen. Optische Bodenplatten sind für Platten-Reader, die Fluoreszenz von der Oberseite des Wells lesen, nicht erforderlich. Das Zellkulturmedium sollte das gleiche Medium sein, das auch für die Züchtung der zu testenden Zellen verwendet wird. Wenn Sie beispielsweise U2OS-Zellen verwenden, sollten 40.000 Zellen in 180 μl DMEM resuspendiert werden. - Erzeugen Sie mit einer Mehrkanalpipette eine serielle Verdünnung von 1:2, indem Sie 90 μl von der Säule ganz links in die benachbarte Säule rechts übertragen. Zum Mischen 15x pipettieren.
- Wiederholen Sie Schritt 2.3, indem Sie von der zweiten Spalte in die dritte Spalte wechseln, dann von der dritten Spalte in die vierte Spalte und so weiter. Beenden Sie die Titration in der vorletzten Spalte der Platte. Lassen Sie die letzte Säule ohne Zellen, damit das Hintergrundsignal des Zellkulturmediums abgerufen werden kann.
- Entfernen Sie für die vorletzte Säule 90 μl, sodass alle Vertiefungen 90 μl Medium mit einer unterschiedlichen Anzahl von Zellen enthalten. Lassen Sie die Zellen 6 h lang in einem 37 °C Zellkultur-Inkubator haften.
- Bereiten Sie für jede zu testende Farbstoffkonzentration eine 10-fache Lösung des DNA-Farbstoffs vor, wobei der DNA-Farbstoff zu einem vollständigen Wachstumsmedium verdünnt wird, das für die Kultivierung der interessierenden Zellen verwendet wird. Bereiten Sie 1,5 ml einer 1,5%igen Triton-X-Lösung in PBS vor, wie in Schritt 1 beschrieben.
- Geben Sie in jede Vertiefung der 96-Well-Platte 10 μl des 10x DNA-Farbstoffs. Geben Sie 10 μl 1,5%ige Triton-X-Lösung in jede Vertiefung, um die Zellen zu permeabilisieren. Inkubieren Sie die Zellen in dieser Lösung für die optimale Zeit, die in Schritt 1 bestimmt wurde.
HINWEIS: Nach der Zugabe von Triton-X beträgt die Endkonzentration des DNA-Farbstoffs etwas weniger als 1x, aber das ist unbedeutend. Nach der Inkubation sollte die gesamte Platte lysiert werden. Die manuelle Zelltitration wird verwendet, um die optimale DNA-Farbstoffkonzentration und die optimalen Einstellungen für die Erfassung der Fluoreszenz zu ermitteln. - Messen Sie die Fluoreszenzintensität über die Zelltitrationsplatte. Quantifizieren Sie die Fluoreszenz über eine Reihe von Erfassungseinstellungen, wie vom Hersteller des DNA-Farbstoffs beschrieben. Je nach verwendetem Plattenleser können Sie die Anregungs- und Emissionswellenlängen und/oder die digitale Verstärkung ändern.
- Entfernen Sie das Hintergrundsignal aus jeder Messung, indem Sie das durchschnittliche Signal der Spalte subtrahieren, die keine Zellen enthält. Diese Werte finden Sie in der äußersten rechten Spalte der Vertiefungen in der seriellen Verdünnungsplatte.
- Bestimmen Sie die Linearität jeder Konzentration des DNA-Farbstoffs für jede Aufnahmeeinstellung. Die Linearität kann bestimmt werden, indem die Zellzahl gegen das Fluoreszenzsignal grafisch dargestellt und eine lineare Regression durchgeführt wird. Der Bestimmungskoeffizient (r2) gibt an, inwieweit das Fluoreszenzsignal linear mit der Zellzahl zusammenhängt.
- Wählen Sie die Erfassungseinstellungen und die DNA-Farbstoffkonzentration aus, die die beste Kombination aus Linearität und Dynamikbereich bieten.
HINWEIS: Berücksichtigen Sie den gesamten Bereich der Zellzahlen von 0 bis 20.000 Zellen. Stellen Sie sicher, dass die Verstärkung und die DNA-Farbstoffkonzentration bei niedrigen Zellzahlen zwischen 0 und 1000 Zellen linear sind. Verschiedene Geräteeinstellungen oder DNA-Farbstoffkonzentrationen können eine starke Korrelation über den gesamten Bereich liefern. Es ist jedoch wichtig, dass die Empfindlichkeit im unteren Drehzahlbereich robust ist, damit kleine Veränderungen in toten Zellen genau gemessen werden können.
3. Zellplattierung, In-Well-Anwendung von Arzneimitteln und Messung der Fluoreszenz toter Zellen im Laufe der Zeit in medikamentenbehandelten Platten
HINWEIS: Wirkstoffverdünnungsplatten können je nach experimentellem Bedarf flexibel gestaltet werden. Im Allgemeinen umfassen Arzneimittelverdünnungsplatten eine logarithmisch oder halblogithmische Verdünnungsreihe von einem oder mehreren Arzneimitteln.
- Berücksichtigen Sie das optimale Beschichtungslayout für das Experiment. Vermeiden Sie Messungen aus den äußeren Vertiefungen einer 96-Well-Platte, um das mit Wachstumsschwankungen verbundene Rauschen zu verringern, da die äußeren Vertiefungen im Vergleich zu den inneren Vertiefungen der Platte unterschiedliche Temperaturen, Sauerstoffversorgung und Verdunstung aufweisen.
HINWEIS: Durch das Einbeziehen von Steuerelementen in der gesamten Landschaft der Platte wird die Robustheit der Wachstumsdaten erhöht. Erwägen Sie, Kontrollvertiefungen in der Mitte und an den Rändern der Platte einzuschließen. - Bestimmen Sie die Anzahl der für das Experiment erforderlichen Platten. Fügen Sie eine zusätzliche Platte hinzu, die zu Beginn des Assays lysiert wird, um die durchschnittliche Zellzahl pro Well zum Zeitpunkt der Verabreichung zu ermitteln (d. h. die T0-Kontrollplatte).
- Bereiten Sie eine Zellsuspension in vollständigem Wachstumsmedium vor.
- Bestimmen Sie anhand der Anzahl der für den Versuch benötigten Platten (berechnet in Schritt 3.2) das Volumen der erforderlichen Zellsuspension. Bei der Aussaat von Platten mit 90 μl pro Vertiefung sind etwa 10 ml pro Platte erforderlich. Multiplizieren Sie die Anzahl der benötigten Platten mit 10 ml.
HINWEIS: Es ist wünschenswert, dieses Volumen zu erhöhen, um das Totvolumen des Reservoirs und den Volumenverlust während des Pipettierens zu berücksichtigen. Zum Beispiel kann das berechnete Volumen mit 1,2 multipliziert werden. - Zählen Sie die Zellen und bereiten Sie eine Zellverdünnung basierend auf der gewünschten Anzahl von Zellen pro Vertiefung vor. Wenn Sie beispielsweise darauf abzielen, 2000 Zellen in 90 μl Medium pro Vertiefung einer 96-Well-Platte für insgesamt 3 Platten zu säen, berechnen Sie die Gesamtzahl der für das Experiment benötigten Zellen wie folgt:
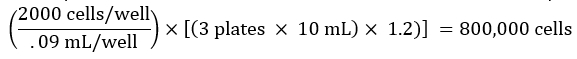
Wenn die gezählte Zellsuspension 400.000 Zellen/ml beträgt, beträgt das Volumen der Zellsuspension, das für das Experiment benötigt wird, wie folgt:
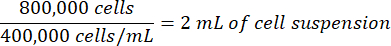
- Korrigieren Sie das Volumen des für die Beschichtung verwendeten Zellkulturmediums, indem Sie das Volumen der hinzuzufügenden Zählzellsuspension subtrahieren.
36 mL Medien - 2 mL Zellsuspension = 34 mL korrigiertes Medienvolumen
- Bestimmen Sie anhand der Anzahl der für den Versuch benötigten Platten (berechnet in Schritt 3.2) das Volumen der erforderlichen Zellsuspension. Bei der Aussaat von Platten mit 90 μl pro Vertiefung sind etwa 10 ml pro Platte erforderlich. Multiplizieren Sie die Anzahl der benötigten Platten mit 10 ml.
- Mischen Sie die gezählte Zellsuspension mit einer serologischen Pipette mit dem richtigen Medienvolumen. Übertragen Sie diese Zellsuspension in ein Reagenzreservoir mit V-Boden.
- Geben Sie mit einer Mehrkanalpipette 90 μl der Zellsuspension in jede Vertiefung der 96-Well-Platten. Mischen Sie die Zellsuspension im Reagenzreservoir regelmäßig durch wiederholtes Pipettieren mit einer serologischen Pipette, um sicherzustellen, dass die gewünschte Konzentration von Zellen pro Volumen beibehalten wird.
- Lassen Sie die Zellen über Nacht in einem 37 °C heißen Zellkultur-Inkubator anhaften (i.d.R. 12-24 h).
- Bereiten Sie eine 1,5%ige Triton-X-Lösung in PBS vor, wie in Schritt 1 beschrieben. Das Volumen dieser Lösung sollte ausreichen, um alle Platten zu lysieren. Etwa 1,5 ml 1,5 % Triton-X in PBS reichen aus, um eine 96-Well-Platte zu lysieren.
HINWEIS: Die 1,5%ige Triton-X-Lösung in PBS ist über eine Woche lang stabil und kann der Einfachheit halber im Voraus hergestellt werden. - Bestimmen Sie die Menge an Zellkulturmedien, die für die Herstellung einer Arzneimittelverdünnungsplatte erforderlich ist. Bereiten Sie Arzneimittelverdünnungsplatten in klaren 96-Well-Platten mit U- oder V-Boden vor.
- Um sicherzustellen, dass das Volumen des verdünnten Arzneimittels für den Versuch ausreicht, erhöhen Sie das erforderliche Mindestvolumen, um den Volumenverlust während des Pipettierens zu berücksichtigen. Wenn beispielsweise Arzneimittel in 10 μl zu jeder Vertiefung einer 96-Well-Platte hinzugefügt werden sollen und das Experiment drei Platten umfasst, sind 30 μl verdünntes Arzneimittel pro Vertiefung erforderlich. Bereiten Sie 40-50 μl betäubtes Medium pro Vertiefung vor, um den Volumenverlust auszugleichen. Fügen Sie 1,5 ml zu dem Medienvolumen hinzu, das erforderlich ist, um das für die T0-Kontrollplatte benötigte Medium zu berücksichtigen.
- Bereiten Sie eine 10-fache Konzentration des ausgewählten DNA-Farbstoffs in vollständigem Wachstumsmedium vor. Diese Konzentration basiert auf der in Schritt 2.11 gewählten Konzentration. Das Gesamtvolumen dieser Lösung wird in Schritt 3.8 berechnet.
- Bereiten Sie unter Verwendung des DNA-Farbstoffs + Wachstumsmediums aus Schritt 3.9 eine 10-fache Konzentration jedes zu testenden Arzneimittels vor. Stellen Sie nur die höchste Dosis jedes Arzneimittels in der Wirkstoffverdünnungsplatte her und verdünnen Sie die Arzneimittel seriell mit einer Mehrkanalpipette, wobei Sie 15x zwischen den einzelnen Vertiefungen mischen.
HINWEIS: Wenn Sie die höchste Dosis für ein bestimmtes Arzneimittel in der Arzneimittelverdünnungsplatte herstellen, sollte das Volumen des Arzneimittels berücksichtigt werden, um sicherzustellen, dass die Konzentration genau ist. - Geben Sie mit einer Mehrkanalpipette 10 μl Arzneimittel + DNA-Farbstofflösung aus Schritt 3.10 auf die Platten mit den Zellen.
HINWEIS: Bei Arzneimittelverdünnungsplatten, bei denen eine serielle Verdünnung erstellt wurde, müssen die Spitzen der Mehrkanalpipette nicht gewechselt werden, wenn Arzneimittel den Zellen zugesetzt werden, beginnend mit der niedrigsten Konzentration des Arzneimittels und bis zur höchsten Konzentration. Die Spitzen der Mehrkanalpipette sollten jedoch zwischen jeder Platte und immer dann gewechselt werden, wenn die Spitzen in einer Vertiefung verwendet wurden, die ein anderes Arzneimittel oder eine höhere Konzentration des Arzneimittels enthielt. - Lesen Sie die Fluoreszenz für alle Platten ab, in denen Arzneimittel zugesetzt wurden. Dieser Fluoreszenzwert ist der T0-Messwert (d. h. tote Zellen zu Zeit = 0 h).
- Bringen Sie die Platten nach der T0-Fluoreszenzmessung wieder in den Inkubator zurück.
- 10 μl der in Schritt 3.9 hergestellten 10x DNA-Farbstofflösung und 10 μl der 1,5%igen Triton-X-Lösung in Schritt 3.7 auf die T0-Kontrollplatte geben. Stellen Sie diese Platte für die in Schritt 1.8 ausgewählte Zeit wieder in den Inkubator.
- Lesen Sie die T0-Kontrollplatte ab, sobald die Zellen vollständig lysiert sind, wie in Schritt 1.6 beschrieben.
4. Messen Sie die Fluoreszenz toter Zellen im Laufe der Zeit für medikamentös behandelte Platten
HINWEIS: Minimieren Sie die Zeit, in der sich die Platten außerhalb des Inkubators befinden. Anhaltende Temperaturänderungen können die Lebensfähigkeit der Zellen beeinträchtigen, und die Lichteinwirkung kann DNA-Fluorophore wie SYTOX Green beeinträchtigen.
- Erfassen Sie alle 3-4 Stunden nach der Verabreichung Fluoreszenzwerte für alle medikamentös behandelten Platten. Die Platten müssen nicht über Nacht abgelesen werden, es sei denn, es ist eine sehr genaue Todeskinetik erforderlich.
HINWEIS: Zeitpunkte müssen nicht in festen/standardmäßigen Intervallen genommen werden. Generell können Zeitpunkte ausschließlich während der normalen Arbeitszeit genommen werden, ohne dass es bei der nachgelagerten Analyse der Todeskinetik zu Fehlern kommt. Die Kinetik kann abgeleitet werden, solange einige Messungen in der Zeit vor dem Einsetzen des Todes, während der aufsteigenden Phase des Todes und während der Sättigungs-/Plateauphase durchgeführt werden. - Erfassen Sie zum gewünschten Endpunkt einen Fluoreszenzmesswert. Unmittelbar danach werden 10 μl der in Schritt 3.7 hergestellten 1,5%igen Triton-X-Lösung zugegeben, um die Zellen zu lysieren.
- Stellen Sie die Platten wieder in den Inkubator und lassen Sie die Zellen für die in Schritt 1 festgelegte Zeit permeabilisieren.
- Erfassen Sie Fluoreszenzmesswerte nach der Permeabilisierung. Dieser Fluoreszenzwert ist proportional zur Gesamtzahl der Zellen (d. h. lebende + tote Zellen) für jede Vertiefung.
5. Berechnen Sie die Kinetik der letalen Fraktion
HINWEIS: Die in diesem Protokoll beschriebenen Berechnungen können in jedem Format und jeder Software analysiert werden. Die Verwendung einer Programmierumgebung wie MATLAB, R oder Python ermöglicht jedoch eine schnellere und flexiblere Analyse.
- Berechnen Sie die durchschnittlichen Fluoreszenzwerte von der T0-Kontrollplatte unter Verwendung des getrimmten Mittelwerts von 50 %. Dieser Wert ist proportional zur Gesamtzahl der Zellen vor der Verabreichung.
- Berechnen Sie mithilfe der Kurvenanpassung und einer Funktion für exponentielles Wachstum die Kinetik des Populationswachstums für alle Bohrlöcher im Experiment. Die anfängliche Populationsgröße für jede Vertiefung entspricht dem in Schritt 5.1 berechneten durchschnittlichen T0-Fluoreszenzwert. Die endgültige Populationsgröße für jede Vertiefung ist der in Schritt 4.4 berechnete Wert nach der Permeabilisierung. Die Dauer des Assays beträgt 0 h bis zu dem in Schritt 4.4 ausgewählten Assay-Endpunkt.
- Berechnen Sie unter Verwendung der in Schritt 5.2 ermittelten Wachstumsparameter die Gesamtzahl der Zellen zu jedem gemessenen Zeitpunkt im Assay für jede Vertiefung. Bestimmen Sie die Anzahl der lebenden Zellen zu jedem gemessenen Zeitpunkt, indem Sie die Messung der toten Zellen von der in Schritt 5.2 berechneten Gesamtzahl der Zellen subtrahieren.
HINWEIS: Aufgrund des geringen Rauschens im Assay kann es gelegentlich vorkommen, dass die Anzahl der lebenden Zellen eine kleine negative Zahl ist. Dies kann manuell auf 0 gesetzt werden, da es keine negative Anzahl lebender Zellen geben kann und höchstwahrscheinlich bedeutet, dass alle Zellen in dieser Vertiefung tot waren. - Berechnen Sie die letale Fraktion (LF) für jeden Zeitpunkt, indem Sie das Fluoreszenzsignal der toten Zellen durch das Gesamtzellsignal für jeden Zeitpunkt dividieren.
- Passen Sie eine LED-Gleichung (Lag Exponential Death) an die Daten des letalen Bruchteils des Zeitverlaufs10 an. Um willkürliche kinetische Parameter für Dosen eines Arzneimittels zu vermeiden, die keine signifikante Letalität induzieren, passen Sie es an ein lineares Modell mit einer Steigung gleich 0 an. Bestimmen Sie signifikante Letalitätsniveaus basierend auf dem Rauschen im Assay oder dem bei nicht medikamentösen Erkrankungen beobachteten LF.
- Erfassen Sie die Todeszeit (DO) aus der LED-Gleichung. Die LED-Gleichung besteht aus vier Parametern: dem anfänglichen LF (LFi), dem LF auf dem Plateau (LFp), der anfänglichen Sterberate (DR) und dem DO. Diese Parameter lassen sich aus den Daten mit einer nichtlinearen Regression (d. h. Kurvenanpassung) ableiten.
- Berechnen Sie die fraktionale Viabilität (FV) am Assay-Endpunkt für jedes Medikament bei jeder Dosis. Subtrahieren Sie den Endpunkt-LF-Wert von 1 oder dividieren Sie die Anzahl der lebenden Zellen durch die Gesamtzahl der Zellen.
HINWEIS: Die FV kann zu jedem Zeitpunkt berechnet werden, nicht nur am Endpunkt des Assays. Diese Daten werden zur Bewertung der Dosispharmakologie (z. B. Arzneimittel IC50 oder EC50) verwendet.
6. Berechnen Sie den WE-Wert
- Bestimmen Sie die durchschnittliche Anzahl lebender Zellen für jede Vertiefung zu Beginn des Assays. Berechnen Sie diesen Wert unter Verwendung des in Schritt 3.15 berechneten T0-Messwerts nach der Permeabilisierung und subtrahieren Sie den T0-Messwert für jede Vertiefung, der in Schritt 3.12 erfasst wurde. In der folgenden GR-Gleichung (Schritt 6.3) wird dieser Wert als x0 bezeichnet.
HINWEIS: Gelegentlich kann die höchste Dosis eines Arzneimittels die Fluoreszenzmessung des DNA-Farbstoffs beeinträchtigen. Es kann wünschenswert sein, die durchschnittliche Anzahl lebender Zellen zum Zeitpunkt 0 unter Verwendung der Kontrollvertiefungen zu berechnen. - Bestimmen Sie für die Kontrollvertiefungen (xStrg) die durchschnittliche Anzahl lebender Zellen für jede Vertiefung am Assay-Endpunkt. Bestimmen Sie die durchschnittliche Anzahl lebender Zellen am Assay-Endpunkt für jede medikamentös behandelte Erkrankung (x(c)).
HINWEIS: Abhängig von der Variation des Zellwachstums auf der Platte können unterschiedliche xctrl-Werte für die Normalisierung jeder Vertiefung verwendet werden. Wenn z. B. diex-Strg-Werte auf der Platte systematisch variieren, kann es wünschenswert sein, den nächstgelegenenx-Strg-Wert anstelle des durchschnittlichen x-Strg-Werts der Platte zu verwenden. - Berechnen Sie für jede medikamentös behandelte Vertiefung den normalisierten Hemmwert (GR) der Wachstumsrate mit der folgenden Gleichung4:
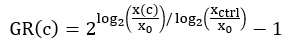
Führen Sie die Kurvenanpassung mit einer logistischen Regression mit 4 Parametern durch. GR liegt auf einer Skala von -1 bis 1.
7. Berechnen Sie drogeninduzierte Wachstums- und Sterberaten mit der GRADE-Methode
HINWEIS: GR stellt die Nettowachstumsrate der Population dar, nicht die tatsächliche Zellproliferationsrate. Das drogeninduzierte Bevölkerungswachstum und die Sterberaten können mit einer Kombination aus GR und letaler Fraktion (LF) berechnet werden.
- Bestimmen Sie die Verdopplungszeit (τ) der Zelllinie während des Versuchs anhand der durchschnittlichen Anzahl lebender Zellen aus dem T0-Kontrollwert (x0), der durchschnittlichen Anzahl lebender Zellen aus der Kontrollbedingung am Assay-Endpunkt (xctrl) und der Länge des Assays in Stunden (t), um die folgende Gleichung zu lösen:

HINWEIS: Eine genaue Berechnung der Zellverdopplungszeit (τ) erfordert, dass sich die Zellen während der gesamten Dauer des Assays kontinuierlich verdoppeln. Bei Zellen, bei denen eine Kontakthemmung auftritt, sollten die Zellen bei einer Ausgangsdichte ausgesät werden, die während der gewünschten Assay-Zeit nicht konfluent wird. Zum Beispiel hat U2OS eine durchschnittliche Verdopplungszeit von 24 h. Die Aussaat von 2000 Zellen pro Well in einem 72-Stunden-Assay ist optimal für die Interpretation ihrer Wachstumsrate. - Bestimmen Sie die Beziehung zwischen Wachstums-/Sterberaten und der Populationsgröße. Verwenden Sie dazu ein Geburten-/Sterbemodell und eine Simulation, die mit allen plausiblen paarweisen Kombinationen von Wachstums- und Sterberaten initialisiert ist. Diese Simulation erfordert die durchschnittliche Anzahl lebender Zellen von der T0-Kontrollplatte (x0), die Länge des Assays (t) und einen benutzerdefinierten Bereich plausibler Proliferationsraten (p) und Todesraten (d).

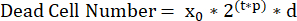
- Um plausible Werte für Wachstumsraten zu ermitteln, beginnen wir mit der unbehandelten Wachstumsrate in Zellpopulationsverdopplungen pro h (1/T) als höchste Wachstumsrate und 0 als niedrigste Wachstumsrate. Teilen Sie diesen Bereich in 500 Segmente mit gleichmäßigem Abstand auf. Eine ähnliche Operation kann für die Sterberate angewendet werden, wobei ein Bereich von 0 bis 1 getestet wird.
HINWEIS: Alle paarweisen Kombinationen von 500 Raten können mit jeder Programmierumgebung ausgeführt werden, können jedoch bei Verwendung einer Software zu rechenintensiv sein. Die Reduzierung auf alle paarweisen Kombinationen von 50 Wachstums- und Sterberaten kann dieses Problem lindern, wobei die Genauigkeit der Inferenz verringert wird. Mit diesem Protokoll wurde eine Tabellenkalkulationsvorlage bereitgestellt.
- Um plausible Werte für Wachstumsraten zu ermitteln, beginnen wir mit der unbehandelten Wachstumsrate in Zellpopulationsverdopplungen pro h (1/T) als höchste Wachstumsrate und 0 als niedrigste Wachstumsrate. Teilen Sie diesen Bereich in 500 Segmente mit gleichmäßigem Abstand auf. Eine ähnliche Operation kann für die Sterberate angewendet werden, wobei ein Bereich von 0 bis 1 getestet wird.
- Bestimmen Sie die GR- und LF-Werte (oben beschrieben) für jedes simulierte Paar aus Proliferation (p) und Todesrate (d).
HINWEIS: In den Schritten 7.2 bis 7.3 wird eine Tabelle generiert, die jedes theoretische Paar von Proliferations- und Sterberaten mit den berechneten GR- und LF-Werten für dieses theoretische Paar enthält. Dies dient als Nachschlagetabelle, um ein experimentell beobachtetes Paar von GR- und LF-Werten mit einem Paar von arzneimittelinduzierten Proliferations- und Todesraten zu verknüpfen. Das Testen von mehr als 500 diskreten Werten für Proliferations- und Todesraten führt zu einem numerisch genaueren Wert, aber dieses Maß an Präzision wird wahrscheinlich über die Präzision des Assays hinausgehen. - Berechnen Sie den paarweisen Abstand zwischen jedem experimentell berechneten GR/LF-Paar und jedem theoretischen GR/LF-Paar in der Nachschlagetabelle. Leiten Sie ab, dass das theoretische Paar mit dem minimalen Abstand zum experimentell beobachteten Paar von GR/LF-Werten die wahren arzneimittelinduzierten Proliferations- und Todesraten ist.
8. Bestimmung der Todeswege mit Hilfe von Signalweg-selektiven chemischen Inhibitoren
HINWEIS: Chemische Inhibitoren allein reichen nicht aus, um den Todesmechanismus eines bestimmten Arzneimittels endgültig zu bestimmen. Chemische Inhibitoren von Todeswegen sollten verwendet werden, um zu bestimmen, welche biochemischen oder phänotypischen Reaktionen in nachfolgenden Experimenten untersucht werden sollten, die wahrscheinlich die morphologische Bewertung, signalwegspezifische biochemische Marker und die Bewertung genetischer Abhängigkeiten umfassen.
- Optimieren Sie die Dosis jedes Zelltodinhibitors in der interessierenden Zelllinie, indem Sie einen Dosisbereich von Inhibitorkonzentrationen im Kontext eines kanonischen Aktivators des interessierenden Todeswegs (der Todeswege) von Interesse testen (Tabelle 2). Im Idealfall sollte die gewählte Dosis des Inhibitors allein die Lebensfähigkeit der Zellen nicht beeinflussen (überprüfen Sie die GR-Metrik).
HINWEIS: Nicht alle Zelllinien können alle Todeswege aktivieren. Eine biochemische oder phänotypische Validierung kann erforderlich sein, wenn Aktivatoren/Inhibitoren verschiedener Todeswege getestet werden. - Berücksichtigen Sie das optimale Beschichtungslayout für ein Inhibitorsieb. Um Batch-Effekte zu minimieren, bewahren Sie jedes Medikament auf derselben Platte wie den/die Inhibitor(en) auf, die untersucht werden, mit Replikaten auf separaten Platten.
- Die gewünschte Anzahl von Zellen in jede Vertiefung einer 96-Well-Platte mit optischem Boden und schwarzen Wänden aussäen, ähnlich wie in Schritt 3.3. Verringern Sie das Plattierungsvolumen auf 80 μl, um das Volumen des Inhibitors und des Arzneimittels zu berücksichtigen.
- Bereiten Sie eine 10-fache Konzertierung der zu testenden Inhibitoren in vollständigen Medien vor und geben Sie 10 μl in jede Vertiefung. Die Zellen werden 2-4 h lang mit Inhibitoren des Todeswegs vorbehandelt. Fügen Sie anschließend die gewünschten Arzneimittel hinzu und führen Sie eine Fluoreszenzmessung durch, wie in den Schritten 3-5.
- Bewerten Sie die Veränderung der Todesbeginnzeit (DO) und/oder der max. letalen Fraktion, um die Wirksamkeit des/der Inhibitor(en) zu beurteilen. Der DO ist ein abgeleiteter Parameter aus dem LED-Modell (Lag Exponential Death). Siehe Schritte 5.6 - 5.7.
Ergebnisse
Mit Hilfe dieses Protokolls untersuchten wir die Empfindlichkeit von U2OS-Zellen gegenüber dem HDAC-Inhibitor Belinostat. Diese Experimente wurden mit 2 μM SYTOX Green durchgeführt, um tote Zellen zu markieren (Abbildung 1A). Die kinetischen Messungen wurden mit einem Fluoreszenzplatten-Reader mit einer Verstärkungseinstellung von 130 durchgeführt (Abbildung 1B). Die Zellen wurden am Ende des Assays 2 Stunden lang in 1,5%iger Triton-X-Lösung in PBS lysiert (Abbildung 1C-D).
Das FLICK-Protokoll liefert Einblicke in die Wachstumsrate der Population und die medikamenteninduzierte Zelltodrate. Diese Erkenntnisse können separat oder mit dem Drug GRADE-Visualisierungs- und Analyse-Framework zusammen betrachtet werden (Abbildung 1E). Die Bewertung der Belinostat-Sensitivität anhand des GR-Wertes zeigt, dass 1 μM Belinostat zu einem GR-Wert von etwa 0 führt (Abbildung 2A). Auf der GR-Skala geben positive Werte die Rate der Bevölkerungsexpansion an, und negative Werte geben die Rate der Bevölkerungsschrumpfung an. Ein GR-Wert von 0 zeigt also, dass die Population in der Stasis verharrt (d. h. in einer zytostatischen Arzneimittelreaktion). Bei der herkömmlichen Interpretation der Zytostase kann man daraus schließen, dass 1 μM Belinostat eine vollständige Wachstumshemmung ohne Zellabtötung bewirkt. Die Bewertung der arzneimittelinduzierten letalen Fraktion zeigt jedoch, dass 1 μM Belinostat im Verlauf dieses Assays eine Letalität von etwa 50 % verursachte (Abbildung 2B). In den meisten Fällen stammen diese beiden Erkenntnisse aus unterschiedlichen Experimenten, die nicht wie Äpfel mit Äpfeln verglichen werden können. Wichtig ist, dass mit dem FLICK-Assay beide Erkenntnisse aus denselben experimentellen Daten abgeleitet, aber unterschiedlich analysiert werden, um die Auswirkungen von Belinostat auf das Populationswachstum im Vergleich zum Zelltod zu erfassen.
Diese beiden Erkenntnisse können auch mit der Analysemethode GRADE integriert werden (Abbildung 2C). Drug GRADE stellt die letale Fraktion, die proportional zur durchschnittlichen Sterberate ist, dem GR-Wert, der Nettowachstumsrate der Bevölkerung, gegenüber. Die Integration der Daten auf diese Weise ermöglicht eine Visualisierung, wie ein Medikament die Wachstumshemmung und die Aktivierung des Todes koordiniert. Innerhalb des GRADE-Diagramms: Medikamente, die das Wachstum hemmen, ohne den Tod auszulösen (d.h. Zytostatika, nach der gängigen Interpretation), werden die obere Grenze einnehmen, Medikamente, die nur töten, ohne die Zellproliferationsraten zu verändern, werden die rechte Grenze einnehmen, und Medikamente, die zuerst eine Wachstumshemmung verursachen, gefolgt vom Tod von wachstumshemmenden Zellen, werden die linke Grenze einnehmen (d.h. biphasische Arzneimittel; Abbildung 2C). Medikamente, die zu GR/LF-Reaktionen führen, die innerhalb dieser Grenzen liegen, können so abgeleitet werden, dass sie gleichzeitig die Proliferation bis zu einem gewissen Grad hemmen und gleichzeitig den Zelltod aktivieren (d. h. koinzidente Medikamente)3. Wichtig ist, dass die Position jeder Wirkstoffreaktion innerhalb des GR/LF-Raums verwendet werden kann, um die wahre Proliferationsrate (p) und die durchschnittliche Sterblichkeitsrate (d) für jede Konzentration eines Medikaments zu berechnen (Abbildung 2D-E). Beachten Sie, dass der GR-Wert nicht die wahre Zellproliferationsrate ist, sondern die Nettowachstumsrate der Population (d. h. der Nettoeffekt, der die tatsächliche Proliferationsrate und die drogeninduzierte Sterberate kombiniert). Außerdem unterscheidet sich die in einer GRADE-Analyse ermittelte Sterberate (d, d, d. die durchschnittliche Sterberate) von dem Sterberatenparameter in der kinetischen LED-Analyse (DR, maximale Geschwindigkeit oder maximale Sterberate).
Der FLICK-Assay kann todesspezifische Daten generieren, wie z. B. die medikamenteninduzierte letale Fraktion; Diese Daten sind jedoch unabhängig vom Mechanismus des Zelltods. Um Einblicke in den Mechanismus des Zelltods mit FLIP zu erhalten, besteht die einfachste Methode darin, zu bestimmen, wie die Kinetik der letalen Fraktion durch die Einbeziehung von Inhibitoren des Todeswegs verändert wird. Die Hemmung eines relevanten Sterbemechanismus sollte zu einer Verringerung der arzneimittelinduzierten Letalfraktion und/oder einer Verzögerung des Zeitpunkts des Beginns des Todes führen. Hier zeigen wir, dass 50 μM des Pan-Caspase-Inhibitors z-VAD etwa 50% der durch 1 μM Belinostat induzierten Letalität retten (Abbildung 2F). Wichtig ist, dass Inhibitoren wie Medikamente eine begrenzte Spezifität aufweisen und versehentlich andere Todesmechanismen hemmen oder verschlimmern können. Bevor eine endgültige Schlussfolgerung gezogen wird, sollten diese Daten daher durch andere Erkenntnisse ergänzt werden, wie z. B. die Zellmorphologie, die Messung biochemischer Aktivierungsmarker und/oder die Bewertung von Signalweg-spezifischen genetischen Abhängigkeiten14.
Um formal zu veranschaulichen, wie die Interpretation einer Arzneimittelreaktion durch eine FLICK-basierte Bewertung und eine GRADE-basierte Analyse verändert werden kann, untersuchten wir als nächstes drei Medikamente, die unterschiedliche Arten der Wachstums-/Todeskoordination aufweisen. Palbociclib ist ein Cdk4/6-Inhibitor, der zu einer zytostatischen Reaktion (Wachstumshemmung ohne Zelltod) führt. Camptothecin ist ein Topoisomerase-I-Hemmer, der eine biphasische Reaktion (Wachstumshemmung, gefolgt vom Zelltod bei hohen Dosen) hervorruft. Belinostat ist ein HDAC-Inhibitor, der eine gleichzeitige Reaktion hervorruft (partielle Wachstumshemmung und Todesaktivierung bei jeder Dosis, jedoch mit unterschiedlichen Anteilen zwischen den Dosen). Bei einer konventionellen Analyse dieser Daten kann beobachtet werden, dass alle drei Medikamente die relative Lebensfähigkeit von U2OS-Zellen erheblich verringern (Abbildung 2G). Obwohl Palbociclib deutlich weniger wirksam ist, ist aus diesen Daten nicht klar, dass Palbociclib den Zelltod nicht aktiviert. Anhand einer GR-basierten Analyse, die berichtet, wie sich Medikamente auf die Nettowachstumsrate der Population auswirken, kann genauer interpretiert werden, dass die mit Palbociclib behandelten Populationen bei allen Dosen weiter wachsen, während hohe Dosen von Camptothecin oder Belinostat zu einer Schrumpfung der Population führen und daher der Zelltod nach hohen Dosen dieser Medikamente aktiviert worden sein muss (Abbildung 2H). Eine GR-basierte Analyse berücksichtigt jedoch nicht die unterschiedliche Koordination zwischen Wachstum und Tod, was zu falschen Schlussfolgerungen über den Grad des Zelltods führen kann, der durch ein bestimmtes Medikament aktiviert wird. Basierend auf den GR-Daten könnte man beispielsweise den Schluss ziehen, dass 1 μM Camptothecin den Zelltod in größerem Maße aktiviert als 1 μM Belinostat, da Camptothecin zu einem GR-Wert von -0,25 und Belinostat zu einem GR-Wert von 0 führt; Ein GR-Wert von -0,25 bedeutet, dass die Population um 25 % so schnell schrumpft, wie die unbehandelte Population wächst. Anhand einer GRADE-basierten Visualisierung und Analyse kann beobachtet werden, dass sich Camptothecin und Belinostat in ihrer Wachstums-/Todeskoordination unterscheiden. Bei der Dosis von 1 μM aktivieren beide Belinostaten den Tod mit einer höheren Rate als Camptothecin (0,8 % Tod/h für Belinostat im Vergleich zu 0,7 % Tod/h für Camptothecin), aber die mit Camptothecin behandelte Population schrumpft aufgrund einer ausgeprägteren Wachstumshemmung, die durch dieses Medikament induziert wird, schneller (Abbildung 2I).
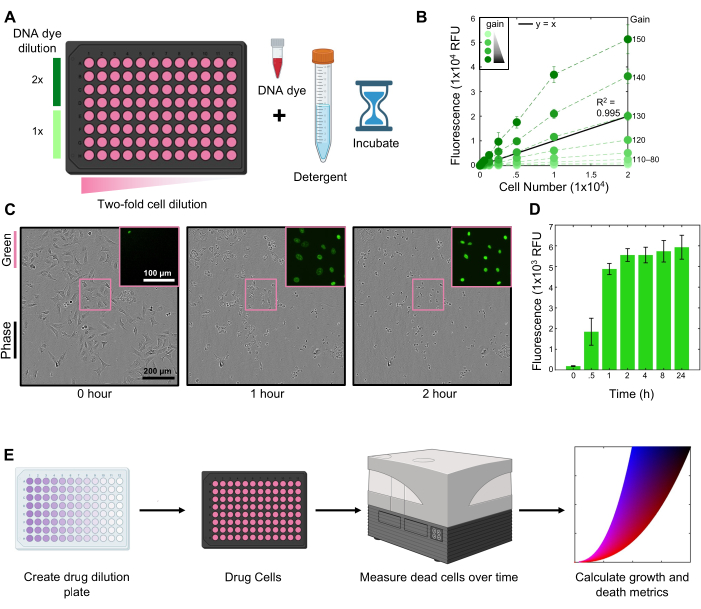
Abbildung 1: Flussdiagramm des FLICK-Protokolls. (A) Erstellen einer Zellverdünnung zur Optimierung der DNA-Farbstoffkonzentration und der Linearität von Fluoreszenzmessungen auf einem Mikroplatten-Reader. (B) Verstärkungstitration über die Zellzahl für 2 μM SYTOX Green in U2OS-Zellen. Eine Verstärkung von 130 liefert das linearste Signal über alle Zellzahlen hinweg. (C) Phasenbilder von U2OS-Zellen, die mit 1,5 % Triton-X über die Zeit unter Verwendung eines 10-fachen Objektivs lysiert wurden. Der eingerahmte Bereich ist eine vergrößerte Ansicht der SYTOX Green-Fluoreszenz. Maßstabsleisten sind repräsentativ für alle Bilder. (D) SYTOX Grünes Signal nach der Lyse zu den angegebenen Zeiten. Die Daten sind Mittelwerte +/- SD für 3 biologische Replikate. (E) Ein konzeptioneller Überblick über den Assay, von der Einrichtung bis zur Analyse. Zu den wichtigsten Schritten gehören die Erstellung einer Wirkstoffverdünnungsplatte, die Verabreichung von Zellen in einer Multi-Well-Platte, die Erfassung von Fluoreszenzmessungen über die Zeit und die Berechnung von Wachstums- und Todesmetriken. Für die Panels B und D sind die Daten Mittelwerte ± SD für n = 3 unabhängige biologische Replikate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
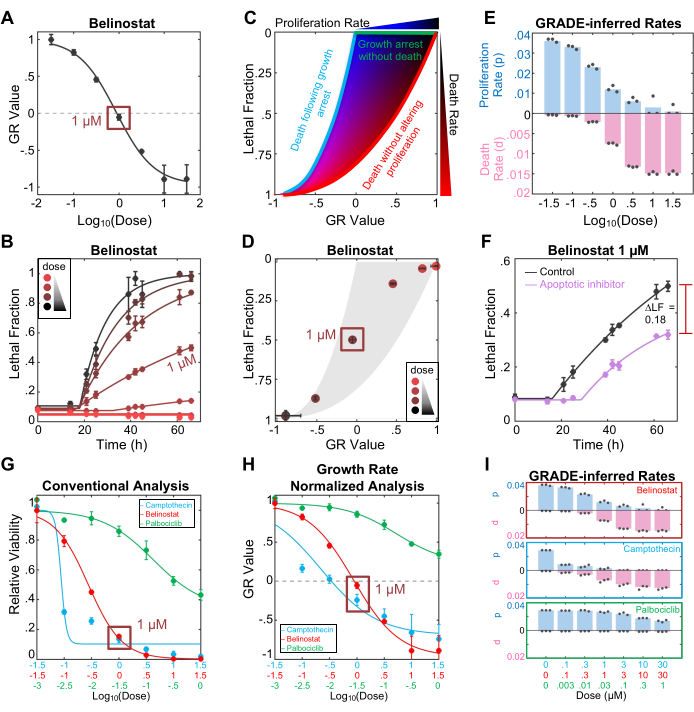
Abbildung 2: Charakterisierung des Arzneimittelansprechens mit dem FLICK-Assay und der GRADE-basierten Analyse. (A) GR-Metrik für den HDAC-Inhibitor Belinostat über eine Dosis von Dosen. Die gezeigten Daten stammen vom Endpunkt des Assays, der 66 Stunden betrug. Bei 1 μM hat Belinostat einen GR-Wert von etwa 0, was auf eine zytostatische Reaktion hinweist. (B) Kinetik der letalen Fraktion für Belinostat. Bei 1 μM induziert Belinostat eine Letalität von fast 50 %, die vom GR-Wert nicht erfasst wird. (C) GRADE-Phasendiagramm basierend auf einer Simulation aller möglichen Wachstums- und Sterberaten. Die Simulation basierte auf der Verdopplungszeit von U2OS in diesem Assay (27,12 h). (D) GRADE-Diagramm für Belinostat über die Dosis am Endpunkt des Assays. (E) Balkendiagramm der von GRADE abgeleiteten Wachstums- (p, Verdopplung der Population pro Stunde) und der Todesraten (d, Anteil der Bevölkerung, der pro Stunde stirbt) für die Dosen von Belinostat. Die Raten wurden aus den simulierten Raten in (C) und den Daten aus (D) bestimmt. (F) Diagramm der letalen Fraktion für 1 μM Belinostat, behandelt mit (violett) oder ohne (schwarz) 50 μM des apoptotischen Inhibitors z-VAD. ΔLF = 0,18 wurde berechnet, indem die durchschnittliche maximale LF von der mit dem Inhibitor behandelten Bedingung von der durchschnittlichen maximalen LF der Kontrolle subtrahiert wurde. (G) Relative Lebensfähigkeit (Anzahl lebender Zellen im arzneimittelbehandelten Zustand, dividiert durch die Anzahl lebender Zellen im unbehandelten Zustand) für drei Arzneimittel nach einer 72-stündigen Exposition. Alle Medikamente verringern die Lebensfähigkeit der Zellen, aber der Beitrag des Zelltods zur Reaktion ist nicht bekannt. (H) GR-Metrik für drei Arzneimittel in (G). Diese Daten zeigen, dass zwei der drei Medikamente zu einer Schrumpfung der Population führen, was durch GR < 0 angezeigt wird. Der Beitrag des Zelltods ist noch unklar, obwohl der Zelltod aktiviert worden sein muss, wenn der GR-Wert negativ ist. (I) Abgeleitete GRADE-Sätze für drei Medikamente. Palbociclib ist nicht tödlich, führt aber zu einem Wachstumsstillstand. Camptothecin ist zweiphasig; Niedrigere Dosen führen zu einem Wachstumsstillstand, während höhere Dosen zu einem Wachstumsstillstand führen, gefolgt von Letalität. Belinostat ist ein zufälliges Medikament; Der Dosisbereich schneidet durch die Mitte des GRADE-Diagramms, was darauf hinweist, dass alle Dosen zu einer Wachstumsstörung und einer Aktivierung des Zelltods führen. Die Daten in allen Panels sind der Mittelwert ± SD für drei unabhängige biologische Replikatexperimente. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Färben | Kinetisch | Endpunkt |
| SYTOX Grün | Y | Y |
| CellTox Grün | Y | Y |
| NucSpot 500/515, 594/615, 750/780 | Y | Y |
| JOJO-3 | Y | Y |
| Propidiumiodid | N | Y |
| 7-AAD | N | Y |
Tabelle 1: DNA-Farbstoffe und ihre Anwendung in FLICK. Viele Arten von Farbstoffen können für FLICK-Assays nur für Endpunkte verwendet werden. Weniger Farbstoffe erfüllen die Bedingungen, um während des gesamten Assays kontinuierlich inkubiert zu werden. Andere Farbstoffe, einschließlich Farbstoffe, die nicht grün sind, wurden nicht gründlich charakterisiert und sollten vor der Verwendung sorgfältig bewertet werden.
| Todesweg | Inhibitor | Typische Dosis |
| Intrinsische Apoptose | ZVAD-FMK | 50 μM |
| Extrinsische Apoptose | Z-IETD | 30 μM |
| Ferroptose | Ferrostatin-1 | 10 μM |
| Nekroptose | Necrostatin-1 | 50 μM |
| Pyroptose | VX-765 | 50 μM |
| Parthanatos | Rucaparib | 1 μM |
| Autophagischer abhängiger Zelltod | Hydroxychloroquin (HCQ) | 10 μM |
| Lysosomal-abhängiger Zelltod | E-64D | 10 μM |
| Cuproptosis | Tetrathiomolybdat (TTM) | 5 μM |
| Oxeiptose | N-Acetyl-L-Cystein (NAC) | ca. 2 mM |
| MPT-getriebene Nekrose | Cyclosporin A (CsA) | 10 μM |
Tabelle 2: Zelltodwege und ihre Inhibitoren. Zelltodinhibitoren sollten gegen einen kanonischen Aktivator für jeden Todesweg validiert werden. Ausgewählte Inhibitordosen sollten die Lebensfähigkeit der Zellen idealerweise nicht beeinflussen.
Ergänzende Tabelle 1: NOTENRECHNER. Tabelle zur Berechnung von drogeninduzierten Wachstums- und Sterberaten aus FLICK-basierten GR- und FV-Daten. Benutzer sollten GR- und FV-Daten zusammen mit der Länge des Assays in Stunden, der Zellverdopplungszeit in Stunden und der Anfangs-/Endpopulationsgröße eingeben. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Der FLICK-Assay ist eine robuste Methode, um eine umfassende Bewertung der Wirkung eines Medikaments auf das Wachstum und den Tod einer Zellpopulation zu erstellen. Da bei dieser Methode die Zellen nicht direkt gezählt werden, bestehen die kritischen Schritte im Protokoll darin, die Linearität des Assays und die vollständige Lyse während der Triton-Permeabilisierungsschritte sicherzustellen. Die korrekte Permeabilisierungszeit kann visuell bestimmt werden, wie in diesem Protokoll hervorgehoben, oder quantitativ durch Lesen der Plattenfluoreszenz im Laufe der Zeit und Identifizieren, wann das Signal ein Plateau erreicht. Unserer Erfahrung nach scheint es aber im Zweifelsfall keinen Preis zu geben, länger zu warten: Bei den DNA-Farbstoffen, die wir im FLICK-Assay verwendet haben, bleibt die Signalintensität nach der Lyse über mehrere Tage stabil. Wir verwenden regelmäßig die Nukleinsäurefärbung SYTOX Green, und obwohl wir nicht jedes in Tabelle 1 genannte Fluorophor validiert haben, zeigen mehrere andere Protokolle ihre effektive Verwendung 13,15,16. Die Beurteilung der Linearität eines gegebenen zellunbelasteten DNA-Farbstoffs über eine Reihe von Zellzahlen und Geräteeinstellungen gewährleistet robuste und quantitativ zuverlässige Daten.
Einige Zelllinien sind schwieriger zu lysieren als andere, und es sollte niemals davon ausgegangen werden, dass die Lysezeit für eine Zelllinie die gleiche ist wie für eine andere13. Zelllinie. Bei einigen Zelllinien führt die Zelllyse bei höheren Dichten dazu, dass sich die Zellen als Blatt von der Vertiefung abheben, was zu einem Fehler beim Plattenleser führen kann. Dies kann vermieden werden, indem die Anfangsdichte der Aussaat reduziert oder die Wells nach der Lyse und vor dem Ablesen mit einer Pipette gemischt werden. Um die Lysezeit oder die Lyseeffizienz zu verbessern, kann die Triton-X-Lösung auf einen höheren oder niedrigeren Prozentsatz eingestellt werden, aber wir haben noch kein Szenario erlebt, in dem dies notwendig war.
Das Versuchsdesign und das Layout der Wirkstoffbeschichtung sollten sorgfältig abgewogen werden, wenn robuste Wachstumsdaten gewünscht werden. Die Erstellung einer Wirkstoffverdünnungsplatte für den FLICK-Assay ist die gleiche wie bei jedem anderen 96-Well-Plattenassay. Die Einbeziehung vieler Kontrollen, die verschiedene Teile der Landschaft einer Platte abdecken, wird jedoch dazu beitragen, robuste Einblicke in die Wachstumskinetik zu gewinnen und festzustellen, ob systematische Wachstumsvariationen existieren. Das Vermeiden der äußeren Vertiefungen einer Platte führt zu genaueren Wachstumsdaten, da diese Vertiefungen empfindlicher auf Temperatur-, Sauerstoff- oder Feuchtigkeitsschwankungen reagieren, was sich auf ihr Wachstum auswirkt. Schließlich ermöglicht die Verwendung eines pseudo-randomisierten Medikamentenmusters, wie z. B. das Wechseln der Position eines Medikaments auf Replikatplatten, eine genauere Bewertung des Arzneimittelverhaltens.
Es gibt einige Einschränkungen für die genaue Anwendung des FLICK-Assays. Diese Einschränkungen hängen hauptsächlich mit der kinetischen Inferenz zusammen, da es sich bei den Endpunktdaten um experimentell beobachtete Werte handelt. Sowohl die Proliferationskinetik lebender Zellen als auch die Todeskinetik hängen von einer Annahme ab, die den Wachstumsverlauf lebender Zellen im Laufe der Zeit in der medikamentös behandelten Bedingung betrifft. In diesem Protokoll beschreiben wir diesen Wachstumspfad als exponentiell und mit einer einheitlichen Rate über die Zeit. Während diese Merkmale wahrscheinlich in Abwesenheit eines Arzneimittels beobachtet werden, können sie in Gegenwart eines Arzneimittels nicht eingehalten werden. Die FLICK-Methode ist nicht deshalb genau, weil die Annahmen über die Wachstumspfade immer korrekt sind, sondern weil die Auswirkungen falscher Annahmen minimiert werden, wenn sich die medikamentös behandelte Population nicht sehr stark vermehrt (d. h. weniger als 2 oder 3 Populationsverdopplungen im Testzeitraum). Wir sind noch nicht auf Medikamente gestoßen, die in FLICK nicht genau profiliert werden können; Die Theorie deutet jedoch darauf hin, dass der FLICK-Assay für ein Medikament, bei dem der Tod sehr langsam eintritt, über lange Zeiträume, die mehrere Vielfache der Zellverdopplungszeit betragen, nicht mehr genau ist (Hinweis: Wir haben keine Medikamente mit diesen Merkmalen identifiziert3). Die Auswirkungen falscher Annahmen für die Wachstumskinetik der Gesamtpopulation haben jedoch keinen Einfluss auf die Zeit des Beginns des Todes, die aus der LED-Anpassung oder den endgültigen maximalen LF-Werten erhalten wird, da diese durch die empirisch gemessenen Werte toter Zellen und die endgültigen Gesamtzellwerte, die am Ende des Assays experimentell bestimmt wurden, begrenzt sind.
Ungeachtet dieser Einschränkungen ermöglicht der FLICK-Assay Erkenntnisse, die mit anderen Methoden des Wirkstoffansprechens nur schwer zu gewinnen sind. Die meisten Arzneimittelreaktionsmethoden erzeugen ein Signal, das proportional zur Anzahl der lebenden Zellen ist, und diese Methoden können verwendet werden, um zu quantifizieren, wie Arzneimittel die Nettowachstumsrate der Population beeinflussen (d. h. den GR-Wert oder ein Äquivalent), können jedoch nicht genau zwischen den zytotoxischen und zytostatischen Wirkungen eines Arzneimittels unterscheiden. Alternativ können mikroskopiebasierte Assays, die sowohl lebende als auch tote Zellen messen, sicherlich ein umfassendes Bild erzeugen. Mikroskopie-basierte Assays können jedoch Schwierigkeiten haben, tote Zellen zu zählen, nachdem sich die Zellen zu Trümmern zersetzt haben, wie dies im Zusammenhang mit vielen nicht-apoptotischen Formen des Zelltods schnell der Fall sein wird. Ein wesentliches Merkmal von FLICK ist, dass die Messungen in einem Plattenlesegerät durchgeführt werden, das die gesamte Fluoreszenz toter Zellen aggregiert, anstatt tote Zellen direkt zu zählen. Somit hängt das Signal toter Zellen in FLICK nicht von der intakten Zellularität toter Zellen ab, die im Zusammenhang mit dem nicht-apoptotischen Zelltod einzigartig essentiell ist. Darüber hinaus ist ein einzigartiges Merkmal des FLICK-Assays die Möglichkeit, sowohl die lebende als auch die tote Zellpopulation mit demselben Reagenz zu messen. So ermöglicht der FLICK-Assay Messungen von lebenden und toten Zellen mit gleicher Empfindlichkeit. Diese Funktion ermöglicht die Analyse mit der GRADE-Methode und verbessert die Genauigkeit der gleichzeitigen Berechnung der Wachstums- und Sterberate von GRADE. Die beigefügte ergänzende Tabelle 1 enthält eine Vorlage, die zur Visualisierung der Wirkstoff-GRADE verwendet wird, in der der Benutzer seine Assay-Länge, die berechnete Wachstumsrate aus Schritt 7.1, die Start- und Endzellzahlen oder die Fluoreszenzmesswerte aus den Kontrollbedingungen und die berechneten LF und GR aus den Schritten 5.4 und 6.3 eingeben kann. Die Datei enthält eine Simulation aller paarweisen Kombinationen von 50 Wachstums- und Sterberaten, die basierend auf den benutzerdefinierten Parametern automatisch aktualisiert wird. Das Template generiert eine GRADE-basierte Visualisierung des Arzneimittelansprechens und der von GRADE abgeleiteten Wachstums- (p) und Todesraten (d).
Zukünftige Arbeiten sollten die Anwendung des FLICK-Assays in 3D-Zellkulturszenarien oder bei der Bewertung des Wirkstoffansprechens von Tumor-abgeleiteten Organoiden untersuchen. Linearität und Sensitivität der FLICK-Daten wurden in diesem Zusammenhang nicht eingehend hinterfragt, aber theoretisch sollte der FLICK-Assay mit einigen Modifikationen wirksam sein, um die Fluoreszenz in 3-dimensionale Proben zu integrieren. Darüber hinaus würde das Hinzufügen zusätzlicher zellspezifischer Markierungen helfen, zwischen zwei oder mehr Zelltypen in Co-Kultur zu unterscheiden. Diese Fortschritte werden für die Erforschung der Wechselwirkungen von Immunzellen mit Krebszellen wertvoll sein. Schließlich inspirieren Assays wie SPARKL zur Verwendung von todesspezifischen Reportern im FLICK-Assay-Format, was den Durchsatz verbessern und gleichzeitig die Genauigkeit der Erkenntnisse über den Zelltod beibehalten kann11,16.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken allen ehemaligen und derzeitigen Mitgliedern des Lee Lab für ihre Beiträge zur Perspektive unseres Labors zur Bewertung des Ansprechens auf Medikamente. Diese Arbeit wurde durch Mittel der National Institutes of Health an MJL (R21CA294000 und R35GM152194) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
Referenzen
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten