Method Article
Analisi completa della risposta ai farmaci utilizzando il test FLICK
In questo articolo
Riepilogo
Questo protocollo descrive come utilizzare il test FLICK per valutare le risposte ai farmaci, comprese le istruzioni dettagliate per l'utilizzo di questo test per calcolare i tassi di crescita e di mortalità indotti dai farmaci e per valutare il meccanismo della morte cellulare indotta dai farmaci.
Abstract
Per comprendere l'efficacia dei farmaci, una necessità fondamentale è quella di caratterizzare l'entità della morte cellulare indotta dai farmaci. Gli sforzi per quantificare il livello di morte cellulare indotta da farmaci sono messi in discussione dall'esistenza di più di una dozzina di forme molecolarmente distinte di morte regolata, ognuna con i propri tempi di attivazione e caratteristiche biochimiche. Inoltre, per alcuni sottotipi di morte necrotica, le caratteristiche distintive sono osservate solo transitoriamente e vengono rapidamente perse a causa della rottura cellulare. Pertanto, anche quando si utilizza una combinazione di saggi specifici per la via di morte, è difficile quantificare con precisione la quantità totale di morte cellulare o i contributi relativi di ciascun sottotipo di morte. Un altro problema è che molti test specifici per la morte ignorano il modo in cui i farmaci influenzano la proliferazione cellulare, rendendo difficile interpretare se una popolazione trattata con farmaci è in espansione o in diminuzione. Il test FLICK consente di quantificare il livello totale di morte cellulare in seguito alla stimolazione in un modo specifico per la morte, ma anche in gran parte agnostico rispetto al tipo o ai tipi di morte attivati. Inoltre, il test FLICK conserva informazioni sulla dimensione totale della popolazione e sul tasso di proliferazione cellulare. In questo manoscritto, descriviamo l'uso di base del test FLICK, come risolvere i problemi di questo test quando si utilizzano diversi tipi di materiale biologico e come utilizzare il test FLICK per quantificare i contributi di ciascun tipo di morte cellulare a una risposta farmacologica osservata.
Introduzione
Per i farmaci antitumorali, la valutazione preclinica della sensibilità ai farmaci comporta generalmente la verifica di come i farmaci influenzano la vitalità delle cellule in coltura1. La vitalità cellulare a seguito dell'esposizione al farmaco è il prodotto di almeno due effetti distinti: l'inibizione della proliferazione cellulare indotta dal farmaco e l'attivazione della morte cellulare2. Purtroppo, sebbene la morte cellulare sia una caratteristica critica necessaria per risposte durature ai farmaci, gli approcci standard non riescono a chiarire il grado in cui un farmaco attiva la morte cellulare3.
I saggi comuni di risposta ai farmaci includono quelli che contano direttamente le cellule (ad esempio, Coulter Counter, alcuni usi della citometria a flusso o della microscopia), quantificano la capacità delle cellule di proliferare (ad esempio, saggio di formazione di colonie) o quantificano un'attività metabolica (ad esempio, CellTiter-Glo, test MTT o MTS basati su tetrazolio). Una caratteristica comune di questi saggi è che i dati generati sono proporzionali al numero di cellule vive. Poiché i farmaci variano considerevolmente nel modo in cui coordinano l'inibizione della crescita e la morte cellulare, il numero di cellule vive dopo l'esposizione al farmaco fornisce una visione inaffidabile del livello di morte cellulare indotta dal farmaco3. Inoltre, poiché le cellule tumorali generalmente proliferano rapidamente in coltura cellulare, il numero di cellule vive può essere drasticamente ridotto rispetto alla popolazione non trattata senza indurre alcuna morte cellulare4. Pertanto, un difetto centrale è che il grado di morte cellulare non può essere quantificato senza misurare sia il numero di cellule vive che quelle morte.
Quantificare il numero di cellule morte in seguito all'esposizione al farmaco è difficile da fare con precisione per diversi motivi2. In primo luogo, esistono più di una dozzina di vie di morte cellulare regolate 5,6,7,8. Sebbene esistano generalmente marcatori biochimici per identificare ogni tipo di morte cellulare regolata, questi marcatori variano nella loro specificità e nessun singolo test può essere utilizzato per quantificare contemporaneamente tutti i sottotipi di morte. In secondo luogo, i tempi di attivazione per ogni forma di morte cellulare possono variare notevolmente a seconda del contesto, quindi un quadro completo non può emergere a meno che la morte non sia quantificata nel tempo 9,10. Molti saggi biochimici per quantificare la morte cellulare producono una misurazione end-point, quindi la generazione di dati cinetici può essere impegnativa e limitata dai costi. Una terza complicazione è che la cellula morta stessa è uno stato intermedio transitorio tra lo stato di cellula viva e i detriti cellulari dissociati. La stabilità delle cellule morte varia a seconda del sottotipo di morte, con alcuni tipi, come l'apoptosi, che creano cadaveri relativamente stabili, mentre altri tipi di morte causano una rapida lisi. Pertanto, i metodi di quantificazione della morte che richiedono la raccolta e il conteggio delle cellule morte produrranno anche una comprensione distorta della morte cellulare. Infine, una quarta limitazione è che i saggi biochimici che quantificano il grado di morte cellulare in genere non riescono a fornire alcuna informazione su come un farmaco altera la proliferazione. Pertanto, la dimensione complessiva della popolazione - e, soprattutto, se la popolazione è in espansione o in riduzione - non può essere interpretata.
Alcuni saggi basati sulla microscopia, come STACK e SPARKL, sono efficaci nel misurare le cellule vive e morte nel tempo e questi saggi possono produrre informazioni complete sulla morte cellulare indotta da farmaci10,11. Questi saggi, tuttavia, richiedono strumenti specializzati, come il microscopio a incuciti, creando limitazioni nella produttività e nell'accesso a questi approcci. Inoltre, le tecniche basate sulla microscopia richiedono che le cellule morte rimangano nel piano focale del microscopio per tutta la durata dell'esperimento, compromettendo la capacità di quantificare le cellule morte quando perdono aderenza dalla piastra o nel tempo man mano che le cellule morte decadono. Allo stesso modo, i saggi basati sulla microscopia affrontano sfide quando vengono applicati nel contesto delle colture in sospensione, poiché le cellule entrano ed escono da un determinato piano focale.
Per risolvere i problemi evidenziati sopra, abbiamo generato un test chiamato FLICK (Fluorescence-based and Lysis-dependent Inference of Cell Death Kinetics)12,13. L'obiettivo del test FLICK è determinare il livello di morte cellulare indotta da farmaci, indipendentemente da come le cellule stanno morendo. Il metodo FLICK utilizza coloranti irregolari cellulari, la cui fluorescenza dipende dal legame con il DNA. Una caratteristica chiave di FLICK è l'uso di questi fluorofori per marcare l'accumulo di cellule morte nel tempo in virtù del loro DNA accessibile, seguito da una lisi meccanica basata su detergenti per permeabilizzare qualsiasi cellula viva alla fine del saggio. Questi dati, combinati con la modellizzazione matematica, consentono la quantificazione di popolazioni di cellule vive e morte con risoluzione temporale continua e senza richiedere la raccolta o la manipolazione di cellule morte. Inoltre, l'uso di un lettore di piastre per valutare la fluorescenza delle cellule morte consente la valutazione delle cellule morte senza richiedere che le cellule morte rimangano intatte, alleviando così una distorsione contro le forme necrotiche di morte che provocano la rottura delle cellule. Infine, il test FLICK richiede una manipolazione minima della piastra e può generare rapidamente misure cinetiche, consentendo lo screening di farmaci ad alto rendimento. In questo protocollo, ci concentriamo sull'uso del test FLICK, incluso il modo in cui utilizzare FLICK per dedurre il tasso di crescita indotto dal farmaco, il tasso di mortalità e/o i meccanismi di morte cellulare.
Protocollo
1. Ottimizzazione del tempo di permeabilizzazione per ogni linea cellulare di interesse
NOTA: I volumi e le quantità descritti sono per l'ottimizzazione di una linea cellulare. Questi valori devono essere ingranditi in base al numero di linee cellulari da testare.
- Piastra il numero desiderato di celle in ciascun pozzetto di una piastra a 96 pozzetti, fondo ottico, pareti nere. Aggiungere 100 μl di terreno completo e lasciare che le cellule aderiscano alla piastra per una notte.
NOTA: Il numero di cellule piastrate deve essere ottimizzato tenendo conto del tasso di crescita delle cellule, delle densità ottimali e della lunghezza del saggio. Il numero tipico di cellule iniziali per le linee cellulari tumorali aderenti misurato nell'arco di 72 ore è di 1500 - 5000 cellule per pozzetto. Ad esempio, le cellule U2OS possono essere piastrate a 2000 cellule per pozzetto in DMEM con il 10% di FBS, 2 mM di glutammina e l'1% di Pen-Strep e coltivate durante la notte in condizioni standard (5% di CO2, 37 °C, con umidità). - In una provetta da centrifuga conica da 15 mL, preparare 1,5 mL di una soluzione all'1,5% di Triton-X in soluzione salina tamponata con fosfato (PBS). Agitare la soluzione Triton-X all'1,5% per 5 s alla massima velocità. Porre a bagnomaria a 37 °C per almeno 30 min.
- Ispezionare visivamente la soluzione per assicurarsi che Triton-X si sia completamente dissolto mediante vortice per alcuni secondi. Assicurarsi che la soluzione sia omogenea.
- Aggiungere 10 μl della soluzione di Triton-X all'1,5% a ciascun pozzetto delle celle piastre. Non mescolare.
NOTA: In questa fase, non è consigliabile miscelare utilizzando pipettaggi ripetuti a causa della formazione di bolle. - Riportare le cellule all'incubatore per colture cellulari al 5%di CO 2, 37 °C con umidità per almeno 1 ora.
NOTA: Dopo l'applicazione della soluzione Triton-X, la maggior parte delle linee cellulari viene completamente lisata in 2-3 ore. L'attesa fino a 24 ore ha un impatto minimo sul segnale di fluorescenza quando si utilizzano coloranti come SYTOX Green. L'aumento della percentuale di Triton-X può ridurre il tempo di incubazione per linee cellulari difficili da lisare. - Osserva le morfologie cellulari su un microscopio ottico utilizzando un obiettivo 10x. Ispezionare le cellule una volta ogni ora fino a quando i corpi cellulari non sono più visibili. Registrare il tempo necessario per la permeabilizzazione cellulare.
NOTA: Se è disponibile un microscopio a fluorescenza, la lisi può essere visualizzata utilizzando un colorante di DNA impermeabile alle cellule, come SYTOX Green. Il colorante può essere aggiunto a una concentrazione finale di 10 volte superiore alla soluzione di Triton-X e la permeabilizzazione può essere confermata dalla microscopia a fluorescenza.
2. Selezione e calibrazione della colorazione del DNA
NOTA: Un requisito per il test FLICK è l'uso di un fluoroforo impenetrante cellulare che emette un segnale in modo dipendente dal legame con il DNA, non influisce sulla vitalità cellulare e produce un segnale che scala linearmente con il numero di cellule. Questo protocollo utilizza SYTOX Green. Anche altri coloranti con proprietà simili possono essere adatti per il test FLICK, ma ciascuno di questi deve essere valutato e calibrato. Vedere la Tabella 1 per esempi.
- Per il colorante a base di DNA selezionato, determinare l'intervallo di concentrazioni da testare in base alle raccomandazioni del produttore.
- Per ogni concentrazione da testare, piastra 40.000 cellule in 180 μL di terreno di coltura cellulare in triplicato lungo la colonna più a sinistra di una piastra a 96 pozzetti, fondo ottico, pareti nere. Aggiungere 90 μl di terreno ai pozzetti rimanenti.
NOTA: Questo verrà utilizzato per generare una titolazione cellulare lineare tra le file della piastra. La concentrazione finale dei primi pozzi sarà di 20.000 cellule/pozzetto. Le piastre ottiche inferiori non sono necessarie per i lettori di piastre che leggono la fluorescenza dalla parte superiore del pozzetto. Il terreno di coltura cellulare deve essere lo stesso utilizzato per la crescita delle cellule da testare. Ad esempio, se si utilizzano celle U2OS, 40.000 cellule devono essere risospese in 180 μL di DMEM. - Utilizzando una pipetta multicanale, creare una diluizione seriale 1:2 trasferendo 90 μl dalla colonna più a sinistra alla colonna adiacente a destra. Pipettare 15 volte per mescolare.
- Ripetere il passaggio 2.3, passando dalla seconda colonna alla terza, quindi dalla terza alla quarta colonna e così via. Terminare la titolazione nella penultima colonna della piastra. Lasciare l'ultima colonna senza celle in modo da poter ottenere il segnale di fondo dei terreni di coltura cellulare.
- Per la penultima colonna, rimuovere 90 μl in modo che tutti i pozzetti abbiano 90 μl di terreno contenente un numero variabile di cellule. Lasciare aderire le cellule per 6 ore in un incubatore per colture cellulari a 37 °C.
- Preparare una soluzione 10x di colorante DNA per ogni concentrazione di colorante da testare, con il colorante DNA diluito in terreno di crescita completo utilizzato per la coltura delle cellule di interesse. Preparare 1,5 mL di una soluzione di Triton-X all'1,5% in PBS, come descritto al punto 1.
- A ciascun pozzetto della piastra a 96 pozzetti, aggiungere 10 μl del colorante DNA 10x. Aggiungere 10 μl di soluzione Triton-X all'1,5% in ciascun pozzetto per permeabilizzare le cellule. Incubare le cellule in questa soluzione per il tempo ottimale, che è stato determinato nella fase 1.
NOTA: Dopo l'aggiunta di Triton-X, la concentrazione finale del colorante DNA è leggermente inferiore a 1x, ma questo è irrilevante. Dopo l'incubazione, l'intera piastra deve essere lisata. La titolazione cellulare manuale verrà utilizzata per identificare la concentrazione ottimale del colorante di DNA e le impostazioni ottimali per l'acquisizione della fluorescenza. - Misurare l'intensità della fluorescenza attraverso la piastra di titolazione cellulare. Quantifica la fluorescenza in una serie di impostazioni di acquisizione, come indicato dal produttore del colorante per DNA. A seconda del lettore di piastre utilizzato, modificare le lunghezze d'onda di eccitazione ed emissione e/o il guadagno digitale.
- Rimuovere il segnale di fondo da ogni misurazione sottraendo il segnale medio della colonna che non contiene celle. Questi valori si trovano nella colonna più a destra dei pozzetti nella piastra di diluizione seriale.
- Determinare la linearità di ciascuna concentrazione di colorante DNA per ogni impostazione di acquisizione. La linearità può essere determinata rappresentando graficamente il numero di cellule rispetto al segnale di fluorescenza ed eseguendo la regressione lineare. Il coefficiente di determinazione (r2) riporta il grado in cui il segnale di fluorescenza è linearmente correlato al numero di cellule.
- Scegli le impostazioni di acquisizione e la concentrazione del colorante del DNA che ha la migliore combinazione di linearità e gamma dinamica.
NOTA: Considerare l'intero intervallo di numeri di celle da 0 a 20.000 celle. Assicurarsi che il guadagno e la concentrazione del colorante del DNA siano lineari a un numero basso di cellule, tra 0 e 1000 cellule. Diverse impostazioni dello strumento o concentrazioni di coloranti di DNA possono fornire una forte correlazione sull'intero intervallo. Tuttavia, è essenziale che la sensibilità di fascia bassa sia robusta, in modo tale che piccoli cambiamenti nelle cellule morte possano essere misurati con precisione.
3. Placcatura cellulare, applicazione di farmaci in pozzetto e misurazione della fluorescenza delle cellule morte nel tempo in piastre trattate con farmaci
NOTA: Le piastre di diluizione dei farmaci possono essere progettate in modo flessibile in base alle esigenze sperimentali. Generalmente, le piastre di diluizione dei farmaci includeranno una serie di diluizione logaritmica o semi-logaritmica di uno o più farmaci.
- Considerare il layout di placcatura ottimale per l'esperimento. Evitare le misurazioni dai pozzetti esterni di una piastra a 96 pozzetti per ridurre il rumore associato alla variazione di crescita, poiché i pozzetti esterni subiscono temperature, ossigenazione ed evaporazione diverse rispetto ai pozzetti interni della piastra.
NOTA: L'inclusione di controlli in tutto il paesaggio della piastra aumenterà la robustezza dei dati di crescita. Prendi in considerazione l'inclusione di pozzetti di controllo al centro e ai bordi della piastra. - Determina il numero di piastre necessarie per l'esperimento. Includere una piastra aggiuntiva da lisare all'inizio del test per stabilire il numero medio di cellule per pozzetto al momento della somministrazione del farmaco (ad esempio, la piastra di controllo T0).
- Preparare una sospensione cellulare in un terreno di coltura completo.
- In base al numero di piastre necessarie per l'esperimento (calcolato al punto 3.2), determinare il volume della sospensione cellulare richiesta. Se si seminano piastre a 90 μl per pozzetto, sono necessari circa 10 mL per piastra. Moltiplicare il numero di piastre necessarie per 10 mL.
NOTA: Si consiglia di aumentare questo volume per tenere conto del volume morto del serbatoio e della perdita di volume durante il pipettaggio. Ad esempio, il volume calcolato può essere moltiplicato per 1,2. - Contare le cellule e preparare una diluizione cellulare in base al numero desiderato di cellule per pozzetto. Ad esempio, se si mira a seminare 2000 cellule in 90 μL di terreno per pozzetto di una piastra a 96 pozzetti, per 3 piastre totali, calcolare il numero totale di cellule necessarie per l'esperimento come di seguito:
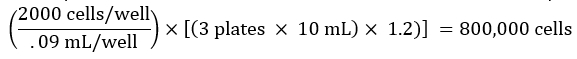
Allo stesso modo, se la sospensione cellulare contata è di 400.000 cellule/mL, il volume della sospensione cellulare necessaria per l'esperimento sarebbe:
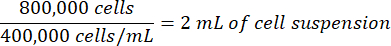
- Correggere il volume dei terreni di coltura cellulare utilizzati per la piastrella sottraendo il volume della sospensione cellulare contata da aggiungere.
36 mL di terreno - 2 mL di sospensione cellulare = 34 mL di volume del terreno corretto
- In base al numero di piastre necessarie per l'esperimento (calcolato al punto 3.2), determinare il volume della sospensione cellulare richiesta. Se si seminano piastre a 90 μl per pozzetto, sono necessari circa 10 mL per piastra. Moltiplicare il numero di piastre necessarie per 10 mL.
- Miscelare la sospensione di cellule contate con il volume di terreno corretto utilizzando una pipetta sierologica. Trasferire questa sospensione cellulare in un serbatoio di reagenti con fondo a V.
- Utilizzando una pipetta multicanale, aggiungere 90 μl della sospensione cellulare a ciascun pozzetto delle piastre a 96 pozzetti. Miscelare regolarmente la sospensione cellulare nel serbatoio del reagente utilizzando pipettaggio ripetuto con una pipetta sierologica per garantire che venga mantenuta la concentrazione desiderata di cellule per volume.
- Lasciare aderire le cellule per una notte in un incubatore per colture cellulari a 37 °C (generalmente 12-24 ore).
- Preparare una soluzione di Triton-X all'1,5% in PBS, come descritto al punto 1. Il volume di questa soluzione dovrebbe essere sufficiente per lisare tutte le piastre. Circa 1,5 mL di Triton-X all'1,5% in PBS sono sufficienti per lisare una piastra a 96 pozzetti.
NOTA: La soluzione di Triton-X all'1,5% in PBS è stabile per oltre una settimana e può essere preparata in anticipo per comodità. - Determinare la quantità di terreno di coltura cellulare necessaria per realizzare una piastra di diluizione del farmaco. Preparare le piastre di diluizione del farmaco in piastre trasparenti a 96 pozzetti con fondo a U o V.
- Per garantire che il volume del farmaco diluito sia sufficiente per l'esperimento, aumentare il volume minimo richiesto per tenere conto della perdita di volume durante il pipettaggio. Ad esempio, se i farmaci devono essere aggiunti in 10 μL a ciascun pozzetto di una piastra da 96 pozzetti e l'esperimento include tre piastre, sono necessari 30 μL di farmaco diluito per pozzetto. Preparare 40-50 μl di terreno drogato per pozzetto per tenere conto della perdita di volume. Aggiungere 1,5 mL al volume di terreno necessario per tenere conto del supporto necessario per la piastra di controllo T0.
- Preparare una concentrazione 10x del colorante di DNA selezionato in un terreno di coltura completo. Questa concentrazione si basa sulla concentrazione selezionata al punto 2.11. Il volume totale di questa soluzione è calcolato al punto 3.8.
- Utilizzando il colorante DNA + terreno di crescita del passaggio 3.9, preparare una concentrazione 10x di ciascun farmaco da testare. Creare solo la dose più alta di ciascun farmaco nella piastra di diluizione del farmaco e diluire in serie i farmaci con una pipetta multicanale, mescolando 15 volte tra ogni pozzetto.
NOTA: Quando si effettua la dose più alta per un determinato farmaco nella piastra di diluizione del farmaco, il volume del farmaco deve essere preso in considerazione per garantire che la concentrazione sia accurata. - Utilizzando una pipetta multicanale, aggiungere 10 μL di farmaco + soluzione colorante di DNA dal passaggio 3.10 alle piastre contenenti le cellule.
NOTA: Per le piastre di diluizione dei farmaci in cui è stata creata una diluizione seriale, se i farmaci vengono aggiunti alle cellule partendo dalla concentrazione più bassa di farmaco e arrivando alla concentrazione più alta, non è necessario sostituire le punte della pipetta multicanale. Tuttavia, i puntali della pipetta multicanale devono essere cambiati tra una piastra e l'altra e ogni volta che i puntali sono stati utilizzati in un pozzetto contenente un farmaco diverso o una concentrazione più elevata del farmaco. - Leggere la fluorescenza di tutte le piastre in cui sono stati aggiunti farmaci. Questa lettura della fluorescenza è la lettura T0 (cioè cellule morte al Tempo = 0 h).
- Rimettere le piastre nell'incubatore dopo aver effettuato la lettura della fluorescenza T0.
- Aggiungere 10 μl della soluzione di colorante DNA 10x creata nella fase 3.9 e 10 μl della soluzione Triton-X all'1,5% nella fase 3.7 alla piastra di controllo T0. Rimetti questa piastra nell'incubatrice per il periodo di tempo selezionato al punto 1.8.
- Leggere la piastra di controllo T0 una volta che le cellule sono completamente lisate, come descritto al punto 1.6.
4. Misurare la fluorescenza delle cellule morte nel tempo per le piastre trattate con farmaci
NOTA: Ridurre al minimo le targhe orarie fuori dall'incubatrice. I cambiamenti prolungati di temperatura possono influire sulla vitalità cellulare e l'esposizione alla luce può compromettere i fluorofori del DNA, come SYTOX Green.
- Acquisire le letture della fluorescenza per tutte le piastre trattate con il farmaco ogni 3-4 ore dopo la somministrazione del farmaco. Le piastre non devono essere lette durante la notte, a meno che non siano necessarie cinetiche di morte molto precise.
NOTA: Non è necessario acquisire punti temporali a intervalli fissi/standard. In generale, i punti temporali possono essere rilevati esclusivamente durante il normale orario di lavoro senza causare errori nell'analisi a valle della cinetica di morte. La cinetica può essere dedotta a condizione che vengano effettuate alcune misurazioni nel periodo precedente l'inizio della morte, durante la fase ascendente della morte e durante la fase di saturazione/plateau. - Al punto temporale finale desiderato, acquisire una lettura della fluorescenza. Subito dopo, aggiungere 10 μl della soluzione di Triton-X all'1,5% preparata al punto 3.7 per lisare le cellule.
- Rimettere le piastre nell'incubatore e lasciare che le cellule permeabilizzino per il tempo determinato nel passaggio 1.
- Acquisire letture di fluorescenza dopo la permeabilizzazione. Questo valore di fluorescenza è proporzionale al numero totale di cellule (cioè cellule vive + morte) per ciascun pozzetto.
5. Calcolare la cinetica della frazione letale
NOTA: I calcoli descritti in questo protocollo possono essere analizzati in qualsiasi formato o software. Tuttavia, l'utilizzo di un ambiente di programmazione come MATLAB, R o Python consentirà un'analisi più rapida e flessibile.
- Calcolare i valori medi di fluorescenza dalla piastra di controllo T0 utilizzando la media trimmata del 50%. Questo valore è proporzionale al numero totale di cellule prima del farmaco.
- Utilizzando l'adattamento della curva e una funzione di crescita esponenziale, calcolare la cinetica di crescita della popolazione per tutti i pozzi dell'esperimento. La dimensione iniziale della popolazione per ciascun pozzetto è il valore medio di fluorescenza T0 calcolato al punto 5.1. La dimensione finale della popolazione per ciascun pozzo è il valore post-permeabilizzazione calcolato nel passaggio 4.4. La durata del saggio è compresa tra 0 h e il punto finale del test selezionato al punto 4.4.
- Utilizzando i parametri di crescita determinati al punto 5.2, calcolare il numero totale di cellule in ogni punto temporale misurato nel saggio per ogni pozzetto. Determinare il numero di cellule vive in ogni punto temporale misurato sottraendo la misurazione delle celle morte dal totale delle celle calcolato al punto 5.2.
NOTA: A causa di piccole quantità di rumore nel test, occasionalmente, il numero di cellule vive può essere un piccolo numero negativo. Questo può essere impostato manualmente su 0 poiché non può esserci un numero negativo di cellule vive e molto probabilmente indica che tutte le cellule in quel pozzo erano morte. - Calcola la frazione letale (LF) per ogni punto temporale dividendo il segnale di fluorescenza delle cellule morte per il segnale totale delle cellule per ogni punto temporale.
- Adattare un'equazione LED (Lag Exponential Death) ai dati del corso della frazione di tempo letale10. Per evitare parametri cinetici arbitrari per dosi di un farmaco che non inducono alcuna letalità significativa, adattarla a un modello lineare con una pendenza pari a 0. Determinare i livelli significativi di letalità in base al rumore nel test o alla LF osservata per condizioni non drogate.
- Registra il tempo di insorgenza della morte (DO) dall'equazione LED. L'equazione LED ha quattro parametri: l'LF iniziale (LFi), l'LF al plateau (LFp), il tasso di mortalità iniziale (DR) e il DO. Deduci questi parametri dai dati utilizzando una regressione non lineare (ad esempio, l'adattamento della curva).
- Calcolare la vitalità frazionaria (FV) all'endpoint del test per ciascun farmaco a ciascuna dose. Sottrarre il valore LF del punto finale da 1 o dividere il numero di celle attive per le celle totali.
NOTA: La FV può essere calcolata in qualsiasi momento, non solo all'endpoint del saggio. Questi dati vengono utilizzati per valutare la farmacologia della dose (ad esempio, il farmaco IC50 o EC50).
6. Calcola il valore GR
- Determinare il numero medio di cellule vive per ciascun pozzetto all'inizio del saggio. Calcolare questo valore utilizzando la lettura T0 post-permeabilizzazione calcolata nel passaggio 3.15 e sottraendo la lettura T0 per ciascun pozzetto, raccolta nel passaggio 3.12. Nell'equazione GR sottostante (passaggio 6.3), questo valore è indicato come x0.
NOTA: Occasionalmente la dose più alta di un farmaco può interferire con la lettura della fluorescenza del colorante del DNA. Può essere opportuno calcolare il numero medio di cellule vive al tempo 0 utilizzando i pozzetti di controllo. - Per i pozzetti di controllo (xctrl), determinare il numero medio di cellule vive per ciascun pozzetto all'endpoint del saggio. Determinare il numero medio di cellule vive all'endpoint del test per ciascuna condizione trattata con il farmaco (x(c)).
NOTA: A seconda della variazione della crescita cellulare attraverso la piastra, è possibile utilizzare diversi valori di xctrl per la normalizzazione di ciascun pozzetto. Ad esempio, se i valori xctrl variano sistematicamente tra la piastra, potrebbe essere desiderabile utilizzare il valore xctrl più vicino piuttosto che il valore xctrl medio della piastra. - Per ciascun pozzetto trattato con farmaco, calcolare il valore normalizzato di inibizione del tasso di crescita (GR) utilizzando la seguente equazione4:
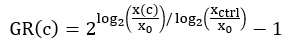
Esegui l'adattamento della curva utilizzando una regressione logistica a 4 parametri. La GR è su una scala da -1 a 1.
7. Calcolare i tassi di crescita e mortalità indotti da farmaci utilizzando il metodo GRADE
NOTA: La GR rappresenta il tasso netto di crescita della popolazione, non il vero tasso di proliferazione cellulare. La crescita della popolazione e i tassi di mortalità indotti dai farmaci possono essere calcolati utilizzando una combinazione del GR e della frazione letale (LF).
- Determinare il tempo di raddoppio (τ) della linea cellulare durante l'esperimento utilizzando il numero medio di cellule vive dalla lettura del controllo T0 (x0), il numero medio di cellule vive dalla condizione di controllo al punto finale del saggio (xctrl) e la durata del test in ore (t) per risolvere la seguente equazione:

NOTA: Il calcolo accurato del tempo di raddoppio delle cellule (τ) richiede che le cellule raddoppino continuamente per tutta la durata del saggio. Per le cellule che presentano l'inibizione del contatto, le cellule devono essere seminate a densità iniziali che non diventeranno confluenti durante il tempo di analisi desiderato. Ad esempio, U2OS ha un tempo medio di raddoppio di 24 ore. La semina di 2000 cellule per pozzetto in un saggio di 72 ore è ottimale per interpretare il loro tasso di crescita. - Determinare la relazione tra i tassi di crescita/mortalità e le dimensioni della popolazione. Per fare ciò, utilizzare un modello di nascita/morte e una simulazione inizializzata con tutte le combinazioni plausibili a coppie di tassi di crescita e di mortalità. Questa simulazione richiede il numero medio di cellule vive dalla piastra di controllo T0 (x0), la lunghezza del test (t) e un intervallo definito dall'utente di tassi di proliferazione plausibili (p) e tassi di mortalità (d).

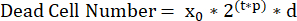
- Per determinare valori plausibili per i tassi di crescita, iniziare con il tasso di crescita non trattato nei raddoppi della popolazione cellulare per h (1/T) come tasso di crescita più alto e 0 come tasso di crescita più basso. Dividi questo intervallo in 500 segmenti equidistanti. Un'operazione simile può essere applicata per il tasso di mortalità, testando un intervallo da 0 a 1.
NOTA: Tutte le combinazioni a coppie di 500 rate possono essere eseguite utilizzando qualsiasi ambiente di programmazione, ma potrebbero essere troppo impegnative dal punto di vista computazionale se si utilizza un software. Ridurre questo a tutte le combinazioni a coppie di 50 tassi di crescita e mortalità può alleviare questo problema, con una riduzione della precisione dell'inferenza. Con questo protocollo è stato fornito un modello di foglio di calcolo.
- Per determinare valori plausibili per i tassi di crescita, iniziare con il tasso di crescita non trattato nei raddoppi della popolazione cellulare per h (1/T) come tasso di crescita più alto e 0 come tasso di crescita più basso. Dividi questo intervallo in 500 segmenti equidistanti. Un'operazione simile può essere applicata per il tasso di mortalità, testando un intervallo da 0 a 1.
- Determinare i valori GR e LF (descritti sopra) per ogni coppia di tassi di proliferazione (p) e di morte (d) simulata.
NOTA: I passaggi 7.2-7.3 genereranno una tabella che contiene ogni coppia teorica di tassi di proliferazione e mortalità, con i valori GR e LF calcolati per quella coppia teorica. Questa funzionerà come una tabella di ricerca per associare una coppia osservata sperimentalmente di valori GR e LF con una coppia di tassi di proliferazione e mortalità indotti da farmaci. L'analisi di più di 500 valori discreti per i tassi di proliferazione e mortalità si tradurrà in un valore numericamente più preciso, ma questo livello di precisione sarà probabilmente superiore alla precisione del test. - Calcola la distanza a coppie tra ogni coppia GR/LF calcolata sperimentalmente e ogni coppia GR/LF teorica nella tabella di ricerca. Dedurre la coppia teorica con la distanza minima dalla coppia di valori GR/LF osservata sperimentalmente per essere i veri tassi di proliferazione e mortalità indotti dal farmaco.
8. Determinazione delle vie di morte mediante inibitori chimici selettivi per via
NOTA: Gli inibitori chimici da soli non sono sufficienti per determinare definitivamente il meccanismo di morte per un dato farmaco. Gli inibitori chimici delle vie di morte dovrebbero essere utilizzati per determinare quali risposte biochimiche o fenotipiche dovrebbero essere esplorate negli esperimenti successivi, che probabilmente includeranno la valutazione morfologica, i marcatori biochimici specifici del percorso e la valutazione delle dipendenze genetiche.
- Ottimizzare la dose di ciascun inibitore della morte cellulare nella linea cellulare di interesse testando un intervallo di dosi di concentrazioni di inibitori nel contesto di un attivatore canonico della via di morte di interesse (Tabella 2). Idealmente, la dose selezionata di inibitore non dovrebbe influenzare la vitalità cellulare da sola (controllare la metrica GR).
NOTA: Non tutte le linee cellulari possono attivare tutti i percorsi di morte. La convalida biochimica o fenotipica può essere necessaria quando si testano attivatori/inibitori di diverse vie di morte. - Considerare il layout di placcatura ottimale per uno schermo a inibitori. Per ridurre al minimo gli effetti dei lotti, mantenere ogni farmaco sulla stessa piastra degli inibitori che vengono valutati, con le repliche su piastre separate.
- Seminare il numero desiderato di cellule in ciascun pozzetto di una piastra a 96 pozzetti, fondo ottico, pareti nere, simile al passaggio 3.3. Ridurre il volume di placcatura a 80 μl per tenere conto dei volumi dell'inibitore e del farmaco.
- Preparare 10 volte la concertazione degli inibitori da testare in terreni completi e aggiungere 10 μl a ciascun pozzetto. Pretrattare le cellule con inibitori della via di morte per 2-4 ore. Successivamente, aggiungi i farmaci di interesse e acquisisci la lettura della fluorescenza, come nei passaggi 3-5.
- Valutare la variazione del tempo di insorgenza della morte (DO) e/o della frazione letale massima per valutare l'efficacia dell'inibitore/i. Il DO è un parametro dedotto dal modello LED (Lag Exponential Death). Vedere i passaggi 5.6 - 5.7.
Risultati
Utilizzando questo protocollo, abbiamo esplorato la sensibilità delle cellule U2OS all'inibitore dell'HDAC Belinostat. Questi esperimenti sono stati eseguiti utilizzando 2 μM di SYTOX Green per marcare le cellule morte (Figura 1A). Le letture cinetiche sono state effettuate utilizzando un lettore di piastre fluorescenti con un'impostazione di guadagno di 130 (Figura 1B). Le cellule sono state lisate in soluzione di Triton-X all'1,5% in PBS per 2 ore alla fine del saggio (Figura 1C-D).
Il protocollo FLICK produce informazioni sul tasso di crescita della popolazione e sul tasso di morte cellulare indotta da farmaci. Queste informazioni possono essere visualizzate separatamente o utilizzando il framework di visualizzazione e analisi GRADE dei farmaci, visti insieme (Figura 1E). La valutazione della sensibilità di Belinostat utilizzando il valore GR rivela che 1 μM di Belinostat produce un valore GR di circa 0 (Figura 2A). Sulla scala GR, i valori positivi riportano il tasso di espansione della popolazione e i valori negativi riportano il tasso di contrazione della popolazione. Pertanto, un valore GR di 0 rivela che la popolazione rimane in stasi (cioè una risposta citostatica ai farmaci). Data l'interpretazione convenzionale della citostasi, si può concludere che 1 μM di Belinostat provoca la piena inibizione della crescita senza alcuna uccisione cellulare. Tuttavia, la valutazione della frazione letale indotta dal farmaco rivela che 1 μM di Belinostat ha causato circa il 50% di letalità nel corso di questo test (Figura 2B). Nella maggior parte dei casi, queste due intuizioni deriverebbero da esperimenti diversi che non possono essere confrontati in modo da mele a mele. È importante sottolineare che, utilizzando il test FLICK, entrambe le intuizioni derivano dagli stessi dati sperimentali, ma analizzate in modo diverso per catturare gli effetti di Belinostat sulla crescita della popolazione rispetto alla morte cellulare.
Queste due intuizioni possono essere integrate anche utilizzando il metodo analitico GRADE (Figura 2C). Il farmaco GRADE contrappone la Frazione Letale, che è proporzionale al tasso medio di mortalità, al valore GR, il tasso netto di crescita della popolazione. L'integrazione dei dati in questo modo fornisce una visualizzazione di come un farmaco coordina l'inibizione della crescita e l'attivazione della morte. All'interno del grafico GRADE: i farmaci che inibiscono la crescita senza attivare la morte (cioè i farmaci citostatici, usando l'interpretazione comune) occuperanno il limite superiore, i farmaci che uccidono solo senza alterare i tassi di proliferazione cellulare occuperanno il limite destro e i farmaci che causano prima l'inibizione della crescita, seguita dalla morte delle cellule arrestate dalla crescita occuperanno il limite sinistro (cioè, farmaci bifasici; Figura 2C). Si può dedurre che i farmaci che provocano risposte GR/LF che rientrano in questi limiti inibiscono simultaneamente la proliferazione in una certa misura e attivano anche la morte cellulare (cioè i farmaci coincidenti)3. È importante sottolineare che la posizione di ciascuna risposta farmacologica all'interno dello spazio GR/LF può essere utilizzata per calcolare il tasso di proliferazione reale (p) e il tasso medio di mortalità (d) per ciascuna concentrazione di un farmaco (Figura 2D-E). Si noti che il valore GR non è il vero tasso di proliferazione cellulare, ma piuttosto il tasso netto di crescita della popolazione (cioè, l'effetto netto che combina il vero tasso di proliferazione e il tasso di mortalità indotta dai farmaci). Inoltre, il tasso di mortalità prodotto in un'analisi GRADE (d, che è il tasso di mortalità medio) è distinto dal parametro del tasso di mortalità nell'analisi cinetica LED (DR, la velocità massima o il tasso di mortalità massimo).
Il test FLICK può generare dati specifici per la morte, come la frazione letale indotta da farmaci; Tuttavia, questi dati sono agnostici rispetto al meccanismo di morte cellulare. Per ottenere informazioni sul meccanismo di morte cellulare utilizzando FLICK, il metodo più semplice consiste nel determinare in che modo la cinetica della frazione letale viene alterata dall'inclusione di inibitori specifici della via di morte. L'inibizione di un meccanismo di morte rilevante dovrebbe comportare una riduzione della frazione letale indotta dal farmaco e/o un ritardo nel tempo di insorgenza della morte. Qui, mostriamo che 50 μM dell'inibitore della pan-caspasi z-VAD recuperano circa il 50% della letalità indotta da 1 μM di Belinostat (Figura 2F). È importante sottolineare che, come i farmaci, gli inibitori hanno una specificità limitata e possono accidentalmente inibire o esacerbare altri meccanismi di morte. Pertanto, prima di giungere a una conclusione definitiva, questi dati dovrebbero essere integrati con altre intuizioni, come la morfologia cellulare, la misurazione dei marcatori biochimici di attivazione e/o la valutazione delle dipendenze genetiche specifiche del percorso14.
Per illustrare formalmente come l'interpretazione di una risposta ai farmaci possa essere alterata da una valutazione basata su FLICK e da un'analisi basata su GRADE, abbiamo successivamente esplorato tre farmaci che presentano diversi tipi di coordinazione crescita/morte. Palbociclib è un inibitore di Cdk4/6 che provoca una risposta citostatica (inibizione della crescita senza morte cellulare). La camptotecina è un inibitore della topoisomerasi I che provoca una risposta bifasica (inibizione della crescita, seguita da morte cellulare ad alte dosi). Belinostat è un inibitore dell'HDAC che provoca una risposta coincidente (parziale inibizione della crescita e attivazione della morte ad ogni dose, ma con proporzioni variabili tra le dosi). Utilizzando un'analisi convenzionale di questi dati, si può osservare che tutti e tre i farmaci riducono sostanzialmente la vitalità relativa delle cellule U2OS (Figura 2G). Sebbene Palbociclib sia notevolmente meno potente, non è chiaro da questi dati che Palbociclib non riesca ad attivare la morte cellulare. Utilizzando un'analisi basata sulla GR, che riporta come i farmaci influenzano il tasso di crescita netto della popolazione, si può interpretare in modo più accurato che le popolazioni trattate con Palbociclib continuano ad espandersi a tutte le dosi, mentre alte dosi di camptotecina o belinostato provocano una riduzione della popolazione e, quindi, la morte cellulare deve essere stata attivata a seguito di alte dosi di questi farmaci (Figura 2H). Tuttavia, un'analisi basata sulla GR non riesce a tenere conto della varia coordinazione tra crescita e morte, il che può portare a conclusioni errate sul livello di morte cellulare attivato da un determinato farmaco. Ad esempio, sulla base dei dati GR, si potrebbe concludere che 1 μM di Camptotecina attiva la morte cellulare in misura maggiore rispetto a 1 μM di Belinostat, dato che la Camptotecina produce un valore GR di -0,25 e Belinostat un valore GR di 0; un valore di -0,25 GR significa che la popolazione si sta riducendo del 25% alla stessa velocità con cui si sta espandendo la popolazione non trattata. Utilizzando una visualizzazione e un'analisi basate su GRADE, si può osservare che Camptothecin e Belinostat differiscono nella loro coordinazione crescita/morte. Pertanto, alla dose di 1 μM, sia Belinostat attiva la morte a un tasso più elevato rispetto alla camptotecina (0,8% di decessi/ora per Belinostat, rispetto allo 0,7% di decessi/ora per la camptotecina), ma la popolazione trattata con camptotecina si riduce più rapidamente a causa di un'inibizione della crescita più pronunciata indotta da questo farmaco (Figura 2I).
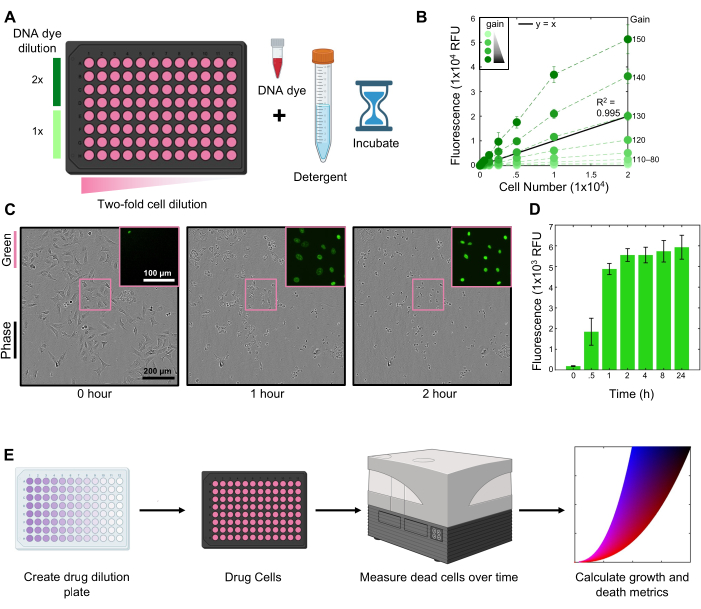
Figura 1: Diagramma di flusso del protocollo FLICK. (A) Creare una diluizione cellulare per ottimizzare la concentrazione del colorante del DNA e la linearità delle misure di fluorescenza su un lettore di micropiastre. (B) Ottenere la titolazione attraverso il numero di cellule per 2 μM di SYTOX Green nelle cellule U2OS. Un guadagno di 130 fornisce il segnale più lineare in tutti i numeri di cella. (C) Immagini di fase di cellule U2OS lisate con Triton-X all'1,5% nel tempo utilizzando un obiettivo 10x. La regione in scatola è una vista ingrandita della fluorescenza di SYTOX Green. Le barre di scala sono rappresentative di tutte le immagini. (D) Segnale verde SYTOX dopo la lisi agli orari indicati. I dati sono medi +/- SD per 3 repliche biologiche. (E) Una panoramica concettuale del saggio, dalla messa a punto all'analisi. Le fasi chiave sono la creazione di una piastra di diluizione del farmaco, la somministrazione di farmaci in una piastra a più pozzetti, l'acquisizione delle misurazioni della fluorescenza nel tempo e il calcolo delle metriche di crescita e morte. Per i pannelli B e D, i dati sono medi ± SD per n = 3 repliche biologiche indipendenti. Clicca qui per visualizzare una versione più grande di questa figura.
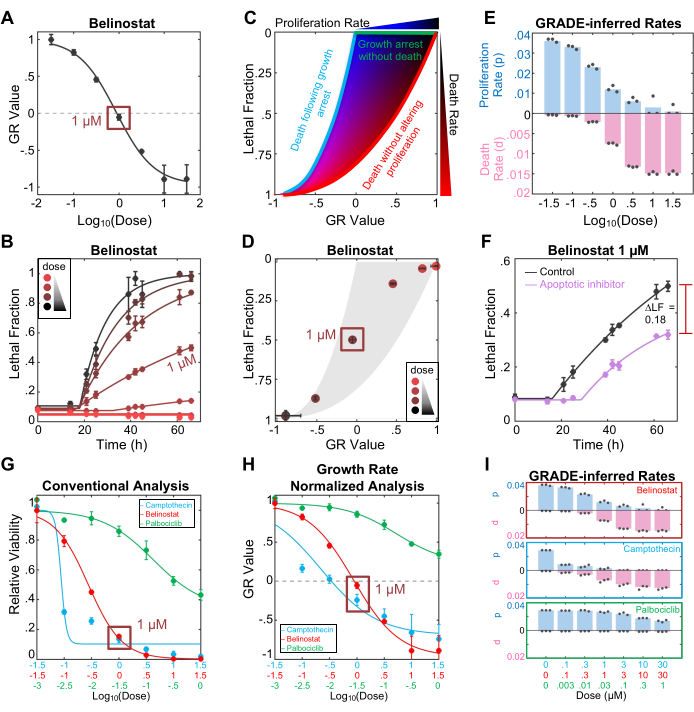
Figura 2: Caratterizzazione delle risposte ai farmaci utilizzando il saggio FLICK e l'analisi basata su GRADE. (A) Metrica GR per l'inibitore dell'HDAC, Belinostat, su una dose di dosi. I dati mostrati provengono dall'endpoint del saggio, che era di 66 ore. A 1 μM, Belinostat ha un valore GR di circa 0, che indica una risposta citostatica. (B) Cinetica della frazione letale per Belinostat. A 1 μM Belinostat induce quasi il 50% di letalità, che non viene catturata dal valore GR. (C) Diagramma di fase GRADE basato su una simulazione di tutti i possibili tassi di crescita e mortalità. La simulazione si è basata sul tempo di raddoppio di U2OS in questo test (27.12 h). (D) Grafico GRADE per Belinostat attraverso la dose all'endpoint del saggio. (E) Grafico a barre dei tassi di crescita (p, raddoppi della popolazione all'ora) e di mortalità (d, frazione della popolazione che muore all'ora) dedotti dal GRADE per le dosi di Belinostat. I tassi sono stati determinati dai tassi simulati in (C) e dai dati di (D). (F) Grafico della frazione letale per 1 μM di Belinostat trattato con (viola) o senza (nero) 50 μM dell'inibitore apoptotico z-VAD. ΔLF = 0,18 è stato calcolato sottraendo la LF massima media dalla condizione trattata con l'inibitore dalla LF massima media del controllo. (G) Vitalità relativa (numero di cellule vive nella condizione trattata con il farmaco, diviso per il numero di cellule vive nella condizione non trattata) per tre farmaci dopo un'esposizione di 72 ore. Tutti i farmaci riducono la vitalità cellulare, ma il contributo della morte cellulare alla risposta non è noto. (H) Metrica GR per tre farmaci in (G). Questi dati dimostrano che due dei tre farmaci provocano una riduzione della popolazione, indicata da GR < 0. Il contributo della morte cellulare non è ancora chiaro, anche se la morte cellulare deve essere stata attivata quando il valore GR è negativo. (I) Tassi dedotti dal GRADE per tre farmaci. Palbociclib non è letale ma provoca l'arresto della crescita. La camptotecina è bifasica; Dosi più basse provocano l'arresto della crescita, mentre dosi più elevate provocano l'arresto della crescita seguito da letalità. Belinostat è un farmaco casuale; l'intervallo di dosaggio attraversa il centro del grafico GRADE, indicando che tutte le dosi provocano una perturbazione della crescita e l'attivazione della morte cellulare. I dati in tutti i pannelli sono la media ± SD per tre esperimenti di replicazione biologica indipendenti. Clicca qui per visualizzare una versione più grande di questa figura.
| Tingere | Cinetico | Punto finale |
| SYTOX Verde | Y | Y |
| CellTox Verde | Y | Y |
| NucSpot 500/515, 594/615, 750/780 | Y | Y |
| YOYO-3 | Y | Y |
| Ioduro di Propidio | N | Y |
| 7 AAD | N | Y |
Tabella 1: Coloranti al DNA e loro applicazione in FLICK. Molti tipi di coloranti possono essere utilizzati per i saggi FLICK solo per l'end point. Un numero inferiore di coloranti soddisfa le condizioni per essere incubato continuamente durante il saggio. Altri coloranti, compresi i coloranti che non sono verdi, non sono stati caratterizzati in profondità e devono essere attentamente valutati prima dell'uso.
| Percorso della morte | Inibitore | Dose tipica |
| Apoptosi intrinseca | ZVAD-FMK | 50 μM |
| Apoptosi estrinseca | Norme Z IETD | 30 μM |
| Ferroptosi | Ferrostatina-1 | 10 μM |
| Necroptosi | Necrostatina-1 | 50 μM |
| Piroptosi | VX-765 | 50 μM |
| Parthanatos | Rucaparib | 1 μM |
| Morte cellulare autofagica dipendente | Idrossiclorochina (HCQ) | 10 μM |
| Morte cellulare lisosomiale dipendente | E-64D | 10 μM |
| Cuproptosi | Tetratiomolibdato (TTM) | 5 μM |
| Ossiiptosi | N-acetil-l-cisteina (NAC) | 2 mM |
| Necrosi guidata da MPT | Ciclosporina A (CsA) | 10 μM |
Tabella 2: Vie di morte cellulare e loro inibitori. Gli inibitori della morte cellulare dovrebbero essere convalidati rispetto a un attivatore canonico per ogni via di morte. Le dosi selezionate di inibitori non dovrebbero idealmente influenzare la vitalità cellulare.
Tabella supplementare 1: Calcolatore del GRADO. Foglio di calcolo per il calcolo dei tassi di crescita e mortalità indotti da farmaci da dati GR e FV basati su FLICK. Gli utenti devono inserire i dati GR e FV, insieme alla durata del test in ore, al tempo di raddoppio delle cellule in ore e alle dimensioni della popolazione iniziale/finale. Clicca qui per scaricare questo file.
Discussione
Il test FLICK è un metodo robusto per generare una valutazione completa dell'effetto di un farmaco sulla crescita e la morte di una popolazione cellulare. Poiché questo metodo non conta direttamente le cellule, le fasi critiche del protocollo consistono nel garantire la linearità del saggio e la lisi completa durante le fasi di permeabilizzazione dei tritoni. Il tempo di permeabilizzazione corretto può essere identificato visivamente, come evidenziato in questo protocollo, o quantitativamente leggendo la fluorescenza della piastra nel tempo e identificando quando il segnale si stabilizza. Nella nostra esperienza, tuttavia, in caso di dubbio, non sembra esserci alcun costo per un'attesa più lunga: per i coloranti di DNA che abbiamo utilizzato nel test FLICK, l'intensità del segnale rimane stabile per diversi giorni dopo la lisi. Utilizziamo regolarmente il colorante per acido nucleico SYTOX Green e, sebbene non abbiamo convalidato tutti i fluorofori citati nella Tabella 1, diversi altri protocolli ne dimostrano l'uso efficace 13,15,16. La valutazione della linearità di un dato colorante di DNA impenetrabile nelle cellule in una gamma di numeri di cellule e impostazioni dello strumento garantirà dati robusti e quantitativamente affidabili.
Alcune linee cellulari sono più difficili da lisare di altre e non si dovrebbe mai presumere che il tempo di lisi per una linea cellulare sia lo stesso di altre13. Per alcune linee cellulari, a densità più elevate, la lisi cellulare provoca il sollevamento delle cellule dal pozzetto come un foglio, il che può causare un errore del lettore di piastre. Questo può essere evitato riducendo la densità di semina iniziale o mescolando con pipette i pozzetti dopo la lisi e prima della lettura. Per migliorare il tempo di lisi o l'efficienza della lisi, la soluzione Triton-X può essere regolata su una percentuale più alta o più bassa, ma non abbiamo ancora trovato uno scenario in cui ciò fosse necessario.
Il disegno sperimentale e il layout della placcatura dei farmaci devono essere attentamente considerati quando si desiderano dati di crescita robusti. La creazione di una piastra di diluizione del farmaco per il test FLICK è la stessa di qualsiasi altro test su piastra a 96 pozzetti. Tuttavia, l'inclusione di molti controlli che coprono diverse parti del paesaggio di una piastra aiuterà a creare solide informazioni sulla cinetica di crescita e può aiutare a determinare se esiste una variazione sistematica della crescita. Evitare i pozzetti esterni di una piastra produrrà dati di crescita più precisi, poiché questi pozzetti sono più sensibili alle fluttuazioni di temperatura, ossigeno o umidità, influenzando la loro crescita. Infine, l'utilizzo di un modello di droga pseudo-randomizzato, come lo scambio della posizione di un farmaco su piastre replicate, fornirà una valutazione più accurata del comportamento del farmaco.
Esistono alcune limitazioni per l'uso accurato del test FLICK. Queste limitazioni sono per lo più legate all'inferenza cinetica, poiché i dati del punto finale sono valori osservati sperimentalmente. Sia la cinetica di proliferazione delle cellule vive che la cinetica di morte dipendono da alcune ipotesi riguardanti la traiettoria di crescita delle cellule vive nel tempo nella condizione trattata con il farmaco. In questo protocollo, descriviamo questa traiettoria di crescita come esponenziale e con un tasso uniforme nel tempo. Sebbene sia probabile che queste caratteristiche vengano osservate in assenza di un farmaco, potrebbero non essere rispettate in presenza di un farmaco. Il metodo FLICK è accurato, non perché le ipotesi sulle traiettorie di crescita siano sempre corrette, ma perché l'impatto di qualsiasi ipotesi errata è ridotto al minimo se la popolazione trattata con il farmaco non prolifera molto (cioè meno di 2 o 3 raddoppi della popolazione nel periodo del test). Non abbiamo riscontrato farmaci che non possono essere accuratamente profilati in FLICK; tuttavia, la teoria suggerirebbe che il test FLICK cesserà di essere accurato per un farmaco in cui la morte avviene molto lentamente, per lunghi periodi di tempo che sono diversi multipli del tempo di raddoppio cellulare (nota: non abbiamo identificato alcun farmaco con queste caratteristiche3). Tuttavia, l'impatto di eventuali ipotesi errate per la cinetica di crescita della popolazione totale non influenzerà il tempo di insorgenza della morte ottenuto dall'adattamento del LED o i valori massimi finali di LF, poiché questi sono vincolati dai valori delle cellule morte misurati empiricamente e dai valori finali delle cellule totali che sono stati determinati sperimentalmente alla fine del test.
Nonostante queste limitazioni, il test FLICK consente di ottenere informazioni difficili da generare utilizzando altri metodi di risposta ai farmaci. La maggior parte dei metodi di risposta ai farmaci genera un segnale proporzionale al numero di cellule vive e questi metodi possono essere utilizzati per quantificare il modo in cui i farmaci influenzano il tasso di crescita netto della popolazione (ad esempio, valore GR o equivalente), ma non possono discriminare con precisione tra gli effetti citotossici e citostatici di un farmaco. In alternativa, i saggi basati sulla microscopia che misurano sia le cellule vive che quelle morte possono certamente generare un quadro completo. Tuttavia, i saggi basati sulla microscopia possono avere difficoltà a contare le cellule morte dopo che le cellule si sono decomposte in detriti, come accadrà rapidamente nel contesto di molte forme non apoptotiche di morte cellulare. Una caratteristica chiave di FLICK è che le misurazioni vengono effettuate in un lettore di piastre, che aggrega la fluorescenza totale delle cellule morte piuttosto che contare direttamente le cellule morte. Pertanto, il segnale delle cellule morte in FLICK non dipende dalla cellularità intatta delle cellule morte, che, nel contesto della morte cellulare non apoptotica, è essenziale in modo univoco. Inoltre, una caratteristica unica del test FLICK è la capacità di misurare sia la popolazione di cellule vive che quelle morte utilizzando lo stesso reagente. Pertanto, il test FLICK produce misurazioni di cellule vive e morte con la stessa sensibilità. Questa funzione consente l'analisi utilizzando il metodo GRADE e migliora l'accuratezza dei calcoli simultanei del tasso di crescita e mortalità di GRADE. La Tabella supplementare 1 inclusa include un modello utilizzato per la visualizzazione del GRADO del farmaco, in cui l'utente può inserire la lunghezza del test, il tasso di crescita calcolato dal passaggio 7.1, il numero di cellule iniziali e finali o le letture fluorescenti dalle condizioni di controllo e la LF e la GR calcolate dai passaggi 5.4 e 6.3. Il file contiene una simulazione di tutte le combinazioni a coppie di 50 tassi di crescita e mortalità che si aggiorneranno automaticamente in base ai parametri definiti dall'utente. Il modello genera una visualizzazione basata su GRADE della risposta al farmaco e dei tassi di crescita (p) e di mortalità (d) dedotti da GRADE.
Il lavoro futuro dovrebbe esplorare l'applicazione del saggio FLICK in scenari di colture cellulari 3D o nella valutazione delle risposte ai farmaci di organoidi derivati dal tumore. La linearità e la sensibilità dei dati FLICK non sono state approfondite in questi contesti, ma in teoria, il test FLICK dovrebbe essere efficace con alcune modifiche per integrare la fluorescenza in campioni tridimensionali. Inoltre, l'aggiunta di ulteriori etichette specifiche per le cellule aiuterebbe a discriminare tra due o più tipi di cellule in co-coltura. Questi progressi saranno preziosi per esplorare le interazioni delle cellule immunitarie con le cellule tumorali. Infine, saggi come SPARKL ispirano l'uso di reporter specifici per la morte nel formato del saggio FLICK, che può migliorare la produttività mantenendo l'accuratezza delle intuizioni sulla morte cellulare11,16.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Ringraziamo tutti i membri passati e presenti del Lee Lab per il loro contributo alla prospettiva del nostro laboratorio sulla valutazione delle risposte ai farmaci. Questo lavoro è stato sostenuto dai finanziamenti del National Institutes of Health a MJL (R21CA294000 e R35GM152194).
Materiali
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
Riferimenti
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon