Method Article
Analyse complète de la réponse médicamenteuse à l’aide du test FLICK
Dans cet article
Résumé
Ce protocole décrit comment utiliser le test FLICK pour évaluer les réponses aux médicaments, y compris des instructions détaillées pour utiliser ce test afin de calculer les taux de croissance et de mortalité induits par les médicaments et d’évaluer le mécanisme de la mort cellulaire induite par les médicaments.
Résumé
Pour comprendre l’efficacité des médicaments, il est essentiel de caractériser l’étendue de la mort cellulaire induite par les médicaments. Les efforts visant à quantifier le niveau de mort cellulaire induite par les médicaments sont mis à l’épreuve par l’existence de plus d’une douzaine de formes moléculairement distinctes de mort régulée, chacune ayant son propre moment d’activation et ses propres caractéristiques biochimiques. De plus, pour certains sous-types de mort nécrotique, les caractéristiques caractéristiques ne sont observées que de manière transitoire et sont rapidement perdues en raison de la rupture cellulaire. Ainsi, même en utilisant une combinaison de tests spécifiques à la voie de mort, il est difficile de quantifier avec précision la quantité totale de mort cellulaire ou les contributions relatives de chaque sous-type de mort. Un autre problème est que de nombreux tests spécifiques à la mort ignorent comment les médicaments affectent la prolifération cellulaire, ce qui rend difficile l’interprétation de l’expansion ou de la diminution d’une population traitée par un médicament. Le test FLICK permet de quantifier le niveau total de mort cellulaire après stimulation d’une manière qui est spécifique à la mort, mais aussi largement agnostique au(x) type(s) de mort activé(s). De plus, le test FLICK conserve des informations sur la taille totale de la population et le taux de prolifération cellulaire. Dans ce manuscrit, nous décrivons l’utilisation de base du test FLICK, comment dépanner ce test lors de l’utilisation de différents types de matériel biologique et comment utiliser le test FLICK pour quantifier les contributions de chaque type de mort cellulaire à une réponse médicamenteuse observée.
Introduction
Pour les médicaments anticancéreux, l’évaluation préclinique de la sensibilité aux médicaments consiste généralement à tester comment les médicaments affectent la viabilité des cellules en culture1. La viabilité cellulaire à la suite d’une exposition à un médicament est le produit d’au moins deux effets distincts : l’inhibition de la prolifération cellulaire induite par le médicament et l’activation de la mort cellulaire2. Malheureusement, bien que la mort cellulaire soit une caractéristique essentielle nécessaire à des réponses médicamenteuses durables, les approches standard ne parviennent pas à clarifier le degré d’activation de la mort cellulaire par un médicament3.
Les tests de réponse aux médicaments courants comprennent ceux qui comptent directement les cellules (par exemple, Coulter Counter, certaines utilisations de la cytométrie en flux ou de la microscopie), quantifient la capacité des cellules à proliférer (par exemple, test de formation de colonies) ou quantifient une activité métabolique (par exemple, tests CellTiter-Glo, MTT à base de tétrazolium ou MTS). Une caractéristique commune de ces tests est que les données générées sont proportionnelles au nombre de cellules vivantes. Étant donné que les médicaments varient considérablement dans la façon dont ils coordonnent l’inhibition de la croissance et la mort cellulaire, le nombre de cellules vivantes après l’exposition au médicament fournit un aperçu peu fiable du niveau de mort cellulaire induite par les médicaments3. De plus, comme les cellules cancéreuses prolifèrent généralement rapidement en culture cellulaire, le nombre de cellules vivantes peut être considérablement réduit par rapport à la population non traitée sans induire de mort cellulaire4. Ainsi, un défaut central est que le degré de mort cellulaire ne peut pas être quantifié sans mesurer à la fois le nombre de cellules vivantes et de cellules mortes.
Il est difficile de quantifier avec précision le nombre de cellules mortes après une exposition à un médicament pour plusieurs raisons: 2. Tout d’abord, il existe plus d’une douzaine de voies de mort cellulaire régulées 5,6,7,8. Bien qu’il existe généralement des marqueurs biochimiques pour identifier chaque type de mort cellulaire régulée, ces marqueurs varient dans leur spécificité, et aucun test unique ne peut être utilisé pour quantifier simultanément tous les sous-types de mort. Deuxièmement, le moment de l’activation pour chaque forme de mort cellulaire peut varier considérablement en fonction du contexte, de sorte qu’une image complète ne peut pas émerger à moins que la mort ne soit quantifiée dans le temps 9,10. De nombreux tests biochimiques permettant de quantifier la mort cellulaire produisent une mesure finale, de sorte que la génération de données cinétiques peut être difficile et limitée par le coût. Une troisième complication est que la cellule morte elle-même est un état intermédiaire transitoire entre l’état de cellule vivante et les débris cellulaires dissociés. La stabilité des cellules mortes varie en fonction du sous-type de mort, certains types, tels que l’apoptose, créant des cadavres relativement stables, tandis que d’autres types de mort provoquent une lyse rapide. Ainsi, les méthodes de quantification de la mort qui nécessitent la collecte et le comptage des cellules mortes produiront également une compréhension biaisée de la mort cellulaire. Enfin, une quatrième limite est que les tests biochimiques qui quantifient le degré de mort cellulaire ne fournissent généralement aucun aperçu de la façon dont un médicament modifie la prolifération. Ainsi, la taille globale de la population - et, surtout, si la population augmente ou diminue - ne peut pas être interprétée.
Certains tests basés sur la microscopie, tels que STACK et SPARKL, sont efficaces pour mesurer les cellules vivantes et mortes au fil du temps, et ces tests peuvent produire des informations complètes sur la mort cellulaire induite par les médicaments10,11. Cependant, ces tests nécessitent des instruments spécialisés, tels que le microscope Incucyte, ce qui limite le débit et l’accès à ces approches. De plus, les techniques basées sur la microscopie exigent que les cellules mortes restent dans le plan focal du microscope pendant toute la durée de l’expérience, ce qui compromet la capacité de quantifier les cellules mortes lorsqu’elles perdent leur adhérence de la plaque ou au fil du temps à mesure que les cellules mortes se décomposent. De même, les tests basés sur la microscopie rencontrent des défis lorsqu’ils sont appliqués dans le contexte de cultures en suspension, car les cellules entrent et sortent d’un plan focal donné.
Pour résoudre les problèmes mis en évidence ci-dessus, nous avons généré un test appelé FLICK (Fluorescence-based and Lysis-dependent Inference of Cell Death Kinetics)12,13. L’objectif du test FLICK est de déterminer le niveau de mort cellulaire induite par le médicament, quelle que soit la façon dont les cellules meurent. La méthode FLICK utilise des colorants imperméables aux cellules, dont la fluorescence dépend de la liaison à l’ADN. L’une des principales caractéristiques du FLICK est l’utilisation de ces fluorophores pour marquer l’accumulation de cellules mortes au fil du temps grâce à leur ADN accessible, suivie d’une lyse mécanique à base de détergent pour perméabiliser toutes les cellules vivantes à la fin du test. Ces données, combinées à la modélisation mathématique, permettent de quantifier les populations de cellules vivantes et mortes avec une résolution temporelle continue et sans nécessiter la collecte ou la manipulation de cellules mortes. De plus, l’utilisation d’un lecteur de plaques pour évaluer la fluorescence des cellules mortes permet d’évaluer les cellules mortes sans exiger que les cellules mortes restent intactes, atténuant ainsi le biais contre les formes nécrotiques de mort qui entraînent une rupture cellulaire. Enfin, le test FLICK nécessite une manipulation minimale des plaques et peut générer rapidement des mesures cinétiques, ce qui permet un criblage de médicaments à haut débit. Dans ce protocole, nous nous concentrons sur l’utilisation du test FLICK, y compris sur la façon d’utiliser FLICK pour déduire le taux de croissance induit par le médicament, le taux de mortalité et/ou les mécanismes de mort cellulaire.
Protocole
1. Optimisation du temps de perméabilisation pour chaque lignée cellulaire d’intérêt
REMARQUE : Les volumes et les quantités décrits sont destinés à optimiser une lignée cellulaire. Ces valeurs doivent être mises à l’échelle en fonction du nombre de lignées cellulaires à tester.
- Plaquez le nombre souhaité de cellules dans chaque puits d’une plaque à fond optique à paroi noire de 96 puits. Ajoutez 100 μL de milieu complet et laissez les cellules adhérer à la plaque pendant la nuit.
REMARQUE : Le nombre de cellules plaquées doit être optimisé en tenant compte du taux de croissance des cellules, des densités optimales et de la durée du test. Le nombre de cellules de départ typique pour les lignées cellulaires cancéreuses adhérentes mesurées sur 72 h est de 1500 à 5000 cellules par puits. Par exemple, les cellules U2OS peuvent être plaquées à 2000 cellules par puits dans du DMEM avec 10 % de FBS, 2 mM de glutamine et 1 % de Pen-Strep et cultivées pendant la nuit dans des conditions standard (5 % de CO2, 37 °C, avec de l’humidité). - Dans un tube à centrifuger conique de 15 ml, préparer 1,5 mL d’une solution à 1,5 % de Triton-X dans une solution saline tamponnée au phosphate (PBS). Vortex la solution Triton-X à 1,5 % pendant 5 s à vitesse maximale. Placer dans un bain-marie à 37 °C pendant au moins 30 min.
- Inspectez visuellement la solution pour vous assurer que Triton-X s’est complètement dissous par vortex pendant plusieurs secondes. Assurez-vous que la solution est homogène.
- Ajouter 10 μL de la solution de Triton-X à 1,5 % dans chaque puits des cellules plaquées. Ne pas mélanger.
REMARQUE : À ce stade, il n’est pas souhaitable de mélanger à l’aide de pipetages répétés en raison de la formation de bulles. - Remettre les cellules dans l’incubateur de culture cellulaire à 5 % de CO2, 37 °C avec humidité pendant au moins 1 h.
REMARQUE : Après l’application de la solution Triton-X, la plupart des lignées cellulaires sont complètement lysées en 2-3 h. Attendre jusqu’à 24 h a un impact minimal sur le signal de fluorescence lors de l’utilisation de colorants tels que SYTOX Green. L’augmentation du pourcentage de Triton-X peut raccourcir le temps d’incubation des lignées cellulaires difficiles à lyser. - Observez les morphologies cellulaires au microscope optique à l’aide d’un objectif 10x. Inspectez les cellules une fois toutes les heures jusqu’à ce que les corps cellulaires ne soient plus visibles. Notez le temps nécessaire à la perméabilisation des cellules.
REMARQUE : Si un microscope fluorescent est disponible, la lyse peut être visualisée à l’aide d’un colorant d’ADN imperméable aux cellules, tel que SYTOX Green. Le colorant peut être ajouté à une concentration finale de 10x à la solution Triton-X, et la perméabilisation peut être confirmée par microscopie à fluorescence.
2. Sélection et étalonnage de la coloration de l’ADN
REMARQUE : L’une des exigences du test FLICK est l’utilisation d’un fluorophore imprégné des cellules qui émet un signal dépendant de la liaison à l’ADN, n’affecte pas la viabilité cellulaire et produit un signal qui s’adapte linéairement au nombre de cellules. Ce protocole utilise SYTOX Green. D’autres colorants ayant des propriétés similaires peuvent également convenir au test FLICK, mais chacun d’entre eux doit être évalué et calibré. Voir le tableau 1 pour des exemples.
- Pour le colorant d’ADN sélectionné, déterminer la plage de concentrations à tester en fonction des recommandations du fabricant.
- Pour chaque concentration à tester, disposez 40 000 cellules dans 180 μL de milieu de culture cellulaire en trois exemplaires le long de la colonne la plus à gauche d’une plaque à fond optique à paroi noire à 96 puits. Ajouter 90 μL de fluide dans les puits restants.
REMARQUE : Cela sera utilisé pour générer un titrage linéaire des cellules sur les rangées de la plaque. La concentration finale des premiers puits sera de 20 000 cellules/puits. Les plaques inférieures optiques ne sont pas nécessaires pour les lecteurs de plaques qui lisent la fluorescence à partir du haut du puits. Le milieu de culture cellulaire doit être le même que celui utilisé pour la croissance des cellules testées. Par exemple, si vous utilisez des cellules U2OS, 40 000 cellules doivent être remises en suspension dans 180 μL de DMEM. - À l’aide d’une pipette multicanaux, créez une dilution en série de 1:2 en transférant 90 μL de la colonne la plus à gauche dans la colonne adjacente à droite. Pipette 15x pour mélanger.
- Répétez l’étape 2.3, en passant de la deuxième colonne à la troisième colonne, puis de la troisième colonne à la quatrième colonne, et ainsi de suite. Terminez le titrage dans l’avant-dernière colonne de la plaque. Laissez la dernière colonne sans cellules afin d’obtenir le signal de fond des milieux de culture cellulaire.
- Pour l’avant-dernière colonne, retirez 90 μL afin que tous les puits aient 90 μL de milieu contenant un nombre variable de cellules. Laissez les cellules adhérer pendant 6 h dans un incubateur de culture cellulaire à 37 °C.
- Préparez une solution 10x de colorant d’ADN pour chaque concentration de colorant à tester, avec le colorant d’ADN dilué dans un milieu de croissance complet utilisé pour la culture des cellules d’intérêt. Préparez 1,5 mL d’une solution de Triton-X à 1,5 % dans du PBS, comme décrit à l’étape 1.
- Dans chaque puits de la plaque à 96 puits, ajoutez 10 μL du colorant ADN 10x. Ajouter 10 μL de solution de Triton-X à 1,5 % dans chaque puits pour perméabiliser les cellules. Incuber les cellules dans cette solution pendant le temps optimal, qui a été déterminé à l’étape 1.
REMARQUE : Après l’ajout de Triton-X, la concentration finale de colorant d’ADN est légèrement inférieure à 1x, mais cela n’a pas d’importance. Après l’incubation, toute la plaque doit être lysée. Le titrage manuel des cellules sera utilisé pour identifier la concentration optimale de colorant d’ADN et les paramètres optimaux pour l’acquisition de la fluorescence. - Mesurez les intensités de fluorescence à travers la plaque de titrage de la cellule. Quantifiez la fluorescence sur une gamme de paramètres d’acquisition tels que décrits par le fabricant du colorant d’ADN. En fonction du lecteur de plaques utilisé, modifiez les longueurs d’onde d’excitation et d’émission et/ou le gain numérique.
- Supprimez le signal d’arrière-plan de chaque mesure en soustrayant le signal moyen de la colonne ne contenant aucune cellule. Ces valeurs se trouvent dans la colonne la plus à droite des puits de la plaque de dilution en série.
- Déterminez la linéarité de chaque concentration de colorant d’ADN pour chaque paramètre d’acquisition. La linéarité peut être déterminée en comparant le nombre de cellules au signal de fluorescence et en effectuant une régression linéaire. Le coefficient de détermination (r2) indique le degré de parenté linéaire entre le signal de fluorescence et le nombre de cellules.
- Choisissez les paramètres d’acquisition et la concentration de colorant d’ADN qui offrent la meilleure combinaison de linéarité et de plage dynamique.
REMARQUE : Considérez toute la plage de numéros de cellules de 0 à 20 000 cellules. Assurez-vous que le gain et la concentration du colorant ADN sont linéaires à un faible nombre de cellules, entre 0 et 1000 cellules. Plusieurs réglages d’instruments ou concentrations de colorants d’ADN peuvent fournir une forte corrélation sur l’ensemble de la plage. Cependant, il est essentiel que la sensibilité à bas terme soit robuste, de sorte que les petits changements dans les cellules mortes puissent être mesurés avec précision.
3. Placage cellulaire, application de médicament dans le puits et mesure de la fluorescence des cellules mortes au fil du temps dans des plaques traitées par médicament
REMARQUE : Les plaques de dilution de médicament peuvent être conçues de manière flexible en fonction des besoins expérimentaux. En général, les plaques de dilution de médicament comprennent une série de dilution logarithmique ou semi-logarithmique d’un ou de plusieurs médicaments.
- Tenez compte de la disposition optimale du placage pour l’expérience. Évitez les mesures à partir des puits extérieurs d’une plaque de 96 puits pour réduire le bruit associé à la variation de croissance, car les puits extérieurs subissent des températures, une oxygénation et une évaporation différentes de celles des puits intérieurs de la plaque.
REMARQUE : L’inclusion de contrôles dans l’ensemble du paysage de la plaque augmentera la robustesse des données de croissance. Envisagez d’inclure des puits de contrôle au centre et sur les bords de la plaque. - Déterminez le nombre de plaques nécessaires pour l’expérience. Inclure une plaque supplémentaire à lyser au début de l’essai pour établir le nombre moyen de cellules par puits au moment de l’administration du médicament (c.-à-d. la plaque de contrôle T0).
- Préparez une suspension cellulaire dans un milieu de croissance complet.
- En fonction du nombre de plaques nécessaires à l’expérience (calculé à l’étape 3.2), déterminez le volume de suspension cellulaire requis. Si l’ensemencement des plaques est de 90 μL par puits, il faut environ 10 mL par plaque. Multipliez le nombre de plaques nécessaires par 10 ml.
REMARQUE : Il est souhaitable d’augmenter ce volume pour tenir compte du volume mort du réservoir et de la perte de volume pendant le pipetage. Par exemple, le volume calculé peut être multiplié par 1,2. - Comptez les cellules et préparez une dilution cellulaire en fonction du nombre souhaité de cellules par puits. Par exemple, si vous visez à ensemencer 2000 cellules dans 90 μL de milieu par puits d’une plaque de 96 puits, pour 3 plaques au total, calculez le nombre total de cellules nécessaires pour l’expérience comme ci-dessous :
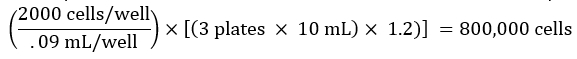
De même, si la suspension cellulaire comptée est de 400 000 cellules/mL, le volume de la suspension cellulaire nécessaire à l’expérience serait de :
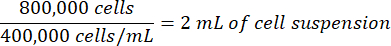
- Corriger le volume de milieu de culture cellulaire utilisé pour le placage en soustrayant le volume de la suspension cellulaire comptée à ajouter.
Média de 36 mL - suspension cellulaire de 2 mL = volume de média corrigé de 34 mL
- En fonction du nombre de plaques nécessaires à l’expérience (calculé à l’étape 3.2), déterminez le volume de suspension cellulaire requis. Si l’ensemencement des plaques est de 90 μL par puits, il faut environ 10 mL par plaque. Multipliez le nombre de plaques nécessaires par 10 ml.
- Mélangez la suspension cellulaire comptée avec le volume de milieu correct à l’aide d’une pipette sérologique. Transférez cette suspension cellulaire dans un réservoir de réactif à fond en V.
- À l’aide d’une pipette multicanaux, ajoutez 90 μL de suspension cellulaire dans chaque puits des plaques de 96 puits. Mélangez régulièrement la suspension cellulaire dans le réservoir de réactif à l’aide d’un pipetage répété avec une pipette sérologique pour vous assurer que la concentration souhaitée de cellules par volume est maintenue.
- Laissez les cellules adhérer toute la nuit dans un incubateur de culture cellulaire à 37 °C (généralement 12-24 h).
- Préparez une solution de Triton-X à 1,5 % dans du PBS, comme décrit à l’étape 1. Le volume de cette solution doit être suffisant pour lyser toutes les plaques. Environ 1,5 mL de Triton-X à 1,5 % dans du PBS est suffisant pour lyser une plaque de 96 puits.
REMARQUE : La solution Triton-X à 1,5 % dans PBS est stable pendant plus d’une semaine et peut être préparée à l’avance pour plus de commodité. - Déterminer la quantité de milieu de culture cellulaire nécessaire à la fabrication d’une plaque de dilution de médicament. Préparez des plaques de dilution de médicament dans des plaques transparentes à 96 puits à fond en U ou en V.
- Pour s’assurer que le volume de médicament dilué est suffisant pour l’expérience, augmentez le volume minimum requis pour tenir compte de la perte de volume pendant le pipetage. Par exemple, si des médicaments doivent être ajoutés dans 10 μL dans chaque puits d’une plaque de 96 puits et que l’expérience comprend trois plaques, 30 μL de médicament dilué sont nécessaires par puits. Préparez 40 à 50 μL de milieu médicamenteux par puits pour tenir compte de la perte de volume. Ajouter 1,5 mL au volume de support requis pour tenir compte du support nécessaire à la plaque de contrôle T0.
- Préparez une concentration 10x du colorant d’ADN sélectionné dans un milieu de croissance complet. Cette concentration est basée sur la concentration sélectionnée à l’étape 2.11. Le volume total de cette solution est calculé à l’étape 3.8.
- À l’aide du colorant d’ADN + milieu de croissance de l’étape 3.9, préparez une concentration 10x de chaque médicament à tester. Créez uniquement la dose la plus élevée de chaque médicament dans la plaque de dilution du médicament et diluez les médicaments en série avec une pipette multicanaux, en mélangeant 15 fois entre chaque puits.
REMARQUE : Lors de la préparation de la dose la plus élevée pour un médicament donné dans la plaque de dilution du médicament, le volume du médicament doit être pris en compte pour s’assurer que la concentration est exacte. - À l’aide d’une pipette multicanaux, ajoutez 10 μL de solution de médicament + colorant ADN de l’étape 3.10 dans les plaques contenant les cellules.
REMARQUE : Pour les plaques de dilution de médicament dans lesquelles une dilution en série a été créée, si des médicaments sont ajoutés aux cellules en commençant par la concentration la plus faible de médicament et en allant jusqu’à la concentration la plus élevée, il n’est pas nécessaire de changer les pointes de la pipette multicanaux. Cependant, les pointes de la pipette multicanaux doivent être changées entre chaque plaque et chaque fois que les pointes ont été utilisées dans un puits contenant un médicament différent ou une concentration plus élevée du médicament. - Lire la fluorescence de toutes les plaques dans lesquelles des médicaments ont été ajoutés. Cette lecture de fluorescence est la lecture T0 (c’est-à-dire les cellules mortes au temps = 0 h).
- Remettez les plaques dans l’incubateur après avoir pris la lecture de fluorescence T0.
- Ajouter 10 μL de la solution de colorant d’ADN 10x créée à l’étape 3.9 et 10 μL de la solution Triton-X à 1,5 % à l’étape 3.7 sur la plaque de contrôle T0. Remettez cette plaque dans l’incubateur pendant la durée sélectionnée à l’étape 1.8.
- Lire la plaque de contrôle T0 une fois que les cellules sont complètement lysées, comme décrit à l’étape 1.6.
4. Mesurer la fluorescence des cellules mortes au fil du temps pour les plaques traitées par médicament
REMARQUE : Minimisez le temps pendant lequel les plaques sont hors de l’incubateur. Des changements prolongés de température peuvent affecter la viabilité cellulaire, et l’exposition à la lumière peut compromettre les fluorophores de l’ADN, tels que SYTOX Green.
- Obtenir des lectures de fluorescence pour toutes les plaques traitées par le médicament toutes les 3 à 4 heures après le traitement. Les plaques n’ont pas besoin d’être lues pendant la nuit, à moins qu’une cinétique de mort très précise ne soit requise.
REMARQUE : Il n’est pas nécessaire de prendre des points temporels à intervalles fixes/standard. En général, les points temporels peuvent être pris exclusivement pendant les heures normales de travail sans provoquer d’erreurs dans l’analyse en aval de la cinétique de mort. La cinétique peut être déduite tant que certaines mesures sont prises dans la période précédant le début de la mort, pendant la phase ascendante de la mort et pendant la phase de saturation/plateau. - Au point temporel final souhaité, obtenez une lecture de fluorescence. Immédiatement après, ajoutez 10 μL de la solution Triton-X à 1,5 % préparée à l’étape 3.7 pour lyser les cellules.
- Remettez les plaques dans l’incubateur et laissez les cellules perméabiliser pendant le temps déterminé à l’étape 1.
- Obtenir des lectures de fluorescence après perméabilisation. Cette valeur de fluorescence est proportionnelle au nombre total de cellules (c’est-à-dire cellules vivantes + cellules mortes) pour chaque puits.
5. Calculer la cinétique de la fraction létale
REMARQUE : Les calculs décrits dans ce protocole peuvent être analysés dans n’importe quel format ou logiciel. Cependant, l’utilisation d’un environnement de programmation tel que MATLAB, R ou Python permettra une analyse plus rapide et plus flexible.
- Calculez les valeurs moyennes de fluorescence à partir de la plaque de contrôle T0 en utilisant la moyenne tronquée de 50 %. Cette valeur est proportionnelle au nombre total de cellules avant la prise de médicament.
- À l’aide de l’ajustement de la courbe et d’une fonction de croissance exponentielle, calculez la cinétique de la croissance de la population pour tous les puits de l’expérience. La taille initiale de la population pour chaque puits est la valeur moyenne de fluorescence T0 calculée à l’étape 5.1. La taille finale de la population pour chaque puits est la valeur post-perméabilisation calculée à l’étape 4.4. La durée de l’essai est de 0 h jusqu’au point final de l’essai sélectionné à l’étape 4.4.
- À l’aide des paramètres de croissance déterminés à l’étape 5.2, calculer le nombre total de cellules à chaque point de l’essai pour chaque puits. Déterminez le nombre de cellules vivantes à chaque point temporel mesuré en soustrayant la mesure des cellules mortes du nombre total de cellules calculé à l’étape 5.2.
REMARQUE : En raison de petites quantités de bruit dans le test, il peut arriver que le numéro de cellule vivante soit un petit nombre négatif. Cela peut être réglé manuellement sur 0 car il ne peut pas y avoir un nombre négatif de cellules vivantes et indique très probablement que toutes les cellules de ce puits étaient mortes. - Calculez la fraction létale (LF) pour chaque point temporel en divisant le signal de fluorescence des cellules mortes par le signal total de la cellule pour chaque point temporel.
- Ajustez une équation de mort exponentielle décalée (LED) à la fraction létale données de trajectoiretemporelle 10. Pour éviter des paramètres cinétiques arbitraires pour des doses d’un médicament qui n’induisent pas de létalité significative, ajustez à un modèle linéaire avec une pente égale à 0. Déterminer les niveaux significatifs de létalité en fonction du bruit dans le test ou de la LF observée pour des conditions non médicamenteuses.
- Enregistrez l’heure de début de la mort (DO) à partir de l’équation de la LED. L’équation de la LED comporte quatre paramètres : le LF initial (LFi), le LF au plateau (LFp), le taux de mortalité initial (DR) et le DO. Déduisez ces paramètres des données à l’aide d’une régression non linéaire (c’est-à-dire l’ajustement de la courbe).
- Calculer la viabilité fractionnelle (FV) au point final du test pour chaque médicament à chaque dose. Soustrayez la valeur LF du point final de 1 ou divisez le nombre de cellules vivantes par le nombre total de cellules.
REMARQUE : La FV peut être calculée à n’importe quel point temporel, et pas seulement au point final du test. Ces données sont utilisées pour évaluer la pharmacologie de la dose (p. ex., CI50 ou CE50 du médicament).
6. Calculer la valeur GR
- Déterminer le nombre moyen de cellules vivantes pour chaque puits au début de l’essai. Calculez cette valeur en utilisant la lecture T0 post-perméabilisation calculée à l’étape 3.15 et en soustrayant la lecture T0 pour chaque puits, qui a été recueillie à l’étape 3.12. Dans l’équation GR ci-dessous (étape 6.3), cette valeur est appelée x0.
REMARQUE : Parfois, la dose la plus élevée d’un médicament peut interférer avec la lecture de la fluorescence du colorant ADN. Il peut être souhaitable de calculer le nombre moyen de cellules vivantes au temps 0 à l’aide des puits de contrôle. - Pour les puits témoins (xctrl), déterminer le nombre moyen de cellules vivantes pour chaque puits au point final de l’essai. Déterminer le nombre moyen de cellules vivantes au critère d’évaluation de l’essai pour chaque affection médicamenteuse traitée (x(c)).
REMARQUE : En fonction de la variation de la croissance cellulaire sur la plaque, différentes valeurs xctrl peuvent être utilisées pour la normalisation de chaque puits. Par exemple, si les valeurs xctrl varient systématiquement sur la plaque, il peut être souhaitable d’utiliser la valeur xctrl la plus proche plutôt que la valeur xctrl moyenne de la plaque. - Pour chaque bien traité par le médicament, calculer la valeur normalisée d’inhibition du taux de croissance (RG) à l’aide de l’équation4 suivante :
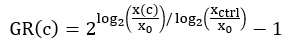
Effectuez un ajustement de courbe à l’aide d’une régression logistique à 4 paramètres. Le GR est sur une échelle de -1 à 1.
7. Calculer les taux de croissance et de mortalité induits par les médicaments à l’aide de la méthode GRADE
REMARQUE : Le RG représente le taux de croissance net de la population, et non le véritable taux de prolifération cellulaire. La croissance démographique et les taux de mortalité induits par les médicaments peuvent être calculés en utilisant une combinaison du RG et de la fraction létale (LF).
- Déterminer le temps de doublement (τ) de la lignée cellulaire au cours de l’expérience en utilisant le nombre moyen de cellules vivantes à partir de la lecture du témoin T0 (x0), le nombre moyen de cellules vivantes à partir de la condition de contrôle au point final de l’essai (xctrl) et la durée de l’essai en heures (t) pour résoudre l’équation suivante :

REMARQUE : Le calcul précis du temps de doublement des cellules (τ) exige que les cellules doublent continuellement pendant toute la durée du test. Pour les cellules qui subissent une inhibition de contact, les cellules doivent être ensemencées à des densités de départ qui ne deviendront pas confluentes pendant le temps de test souhaité. Par exemple, U2OS a un temps de doublement moyen de 24 h. L’ensemencement de 2000 cellules par puits dans un test de 72 heures est optimal pour interpréter leur taux de croissance. - Déterminer la relation entre les taux de croissance/mortalité et la taille de la population. Pour ce faire, utilisez un modèle naissance/mort et une simulation initialisée avec toutes les combinaisons plausibles par paires de taux de croissance et de mortalité. Cette simulation nécessite le nombre moyen de cellules vivantes de la plaque de contrôle T0 (x0), la durée du test (t) et une plage définie par l’utilisateur de taux de prolifération plausibles (p) et de taux de mortalité (d).

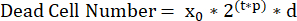
- Pour déterminer les valeurs plausibles des taux de croissance, il faut commencer par le taux de croissance non traité dans les doublages de population cellulaire par h (1/T) comme taux de croissance le plus élevé et 0 comme taux de croissance le plus bas. Divisez cette gamme en 500 segments équidistants. Une opération similaire peut être appliquée pour le taux de mortalité, en testant une plage de 0 à 1.
REMARQUE : Toutes les combinaisons par paires de taux 500 peuvent être effectuées à l’aide de n’importe quel environnement de programmation, mais peuvent être trop intenses en calcul si vous utilisez un logiciel. Réduire cela à toutes les combinaisons par paires de 50 taux de croissance et de mortalité peut atténuer ce problème, avec une réduction de la précision de l’inférence. Un modèle de tableur a été fourni avec ce protocole.
- Pour déterminer les valeurs plausibles des taux de croissance, il faut commencer par le taux de croissance non traité dans les doublages de population cellulaire par h (1/T) comme taux de croissance le plus élevé et 0 comme taux de croissance le plus bas. Divisez cette gamme en 500 segments équidistants. Une opération similaire peut être appliquée pour le taux de mortalité, en testant une plage de 0 à 1.
- Déterminez les valeurs GR et LF (décrites ci-dessus) pour chaque paire de taux de prolifération (p) et de mortalité (d) simulée.
REMARQUE : Les étapes 7.2 et 7.3 généreront un tableau contenant chaque paire théorique de taux de prolifération et de mortalité, avec les valeurs calculées de la RG et de la LF pour cette paire théorique. Cela fonctionnera comme une table de recherche pour associer une paire de valeurs GR et LF observées expérimentalement avec une paire de taux de prolifération et de mortalité induits par la drogue. L’analyse de plus de 500 valeurs discrètes pour les taux de prolifération et de mortalité permettra d’obtenir une valeur numériquement plus précise, mais ce niveau de précision sera probablement au-delà de la précision du test. - Calculez la distance par paire entre chaque paire GR/LF calculée expérimentalement et chaque paire GR/LF théorique dans la table de recherche. Déduire que la paire théorique avec la distance minimale par rapport à la paire de valeurs GR/LF observée expérimentalement est le véritable taux de prolifération et de mortalité induit par la drogue.
8. Détermination des voies de mort à l’aide d’inhibiteurs chimiques sélectifs de voie
REMARQUE : Les inhibiteurs chimiques seuls ne suffisent pas à déterminer définitivement le mécanisme de mort d’un médicament donné. Des inhibiteurs chimiques des voies de mort devraient être utilisés pour déterminer quelles réponses biochimiques ou phénotypiques devraient être explorées dans les expériences ultérieures, qui sont susceptibles d’inclure une évaluation morphologique, des marqueurs biochimiques spécifiques aux voies et une évaluation des dépendances génétiques.
- Optimiser la dose de chaque inhibiteur de la mort cellulaire dans la lignée cellulaire d’intérêt en testant une gamme de doses de concentrations d’inhibiteurs dans le contexte d’un activateur canonique de la ou des voies de mort d’intérêt (tableau 2). Idéalement, la dose choisie d’inhibiteur ne devrait pas affecter la viabilité cellulaire à elle seule (vérifiez la métrique GR).
REMARQUE : Toutes les lignées cellulaires ne peuvent pas activer toutes les voies de mort. Une validation biochimique ou phénotypique peut être nécessaire lors de l’essai d’activateurs/inhibiteurs de différentes voies de mort. - Considérez la disposition optimale du placage pour un tamis inhibiteur. Pour minimiser les effets de lot, conservez chaque médicament sur la même plaque que le ou les inhibiteurs évalués, avec des répétitions sur des plaques distinctes.
- Ensemencez le nombre souhaité de cellules dans chaque puits d’une plaque à 96 puits, à fond optique et à paroi noire, comme à l’étape 3.3. Réduire le volume de placage à 80 μL pour tenir compte des volumes de l’inhibiteur et du médicament.
- Préparez une concertation 10 fois des inhibiteurs à tester dans des milieux complets et ajoutez 10 μL dans chaque puits. Prétraiter les cellules avec des inhibiteurs de la voie de mort pendant 2 à 4 h. Ensuite, ajoutez les médicaments d’intérêt et obtenez une lecture de fluorescence, comme aux étapes 3 à 5.
- Évaluez la variation du temps d’apparition de la mort (OD) et/ou de la fraction létale maximale pour évaluer l’efficacité du ou des inhibiteurs. L’OD est un paramètre déduit du modèle de mort exponentielle de décalage (LED). Voir les étapes 5.6 et 5.7.
Résultats
À l’aide de ce protocole, nous avons exploré la sensibilité des cellules U2OS à l’inhibiteur de HDAC Belinostat. Ces expériences ont été réalisées à l’aide de 2 μM SYTOX Green pour marquer les cellules mortes (Figure 1A). Les lectures cinétiques ont été effectuées à l’aide d’un lecteur de plaques fluorescentes à un réglage de gain de 130 (figure 1B). Les cellules ont été lysées dans une solution de Triton-X à 1,5 % dans du PBS pendant 2 h à la fin de l’essai (Figure 1C-D).
Le protocole FLICK permet de mieux comprendre le taux de croissance de la population et le taux de mortalité cellulaire induit par les médicaments. Ces informations peuvent être visualisées séparément ou à l’aide du cadre de visualisation et d’analyse des médicaments GRADE, visualisées ensemble (Figure 1E). L’évaluation de la sensibilité du Belinostat à l’aide de la valeur GR révèle que 1 μM Belinostat donne une valeur GR d’environ 0 (Figure 2A). Sur l’échelle des RG, les valeurs positives indiquent le taux d’expansion de la population, et les valeurs négatives indiquent le taux de diminution de la population. Ainsi, une valeur GR de 0 révèle que la population reste en stase (c’est-à-dire une réponse cytostatique aux médicaments). Compte tenu de l’interprétation conventionnelle de la cytostase, on peut conclure que 1 μM Belinostat provoque une inhibition complète de la croissance sans aucune destruction cellulaire. Cependant, l’évaluation de la fraction létale induite par le médicament révèle que 1 μM de bélinostat a causé une létalité d’environ 50 % au cours de cet essai (figure 2B). Dans la plupart des cas, ces deux connaissances seraient dérivées d’expériences différentes qui ne peuvent pas être comparées de manière à des pommes et des pommes. Il est important de noter qu’à l’aide du test FLICK, les deux informations sont dérivées des mêmes données expérimentales, mais analysées différemment pour saisir les effets du Belinostat sur la croissance de la population par rapport à la mort cellulaire.
Ces deux insights peuvent également être intégrés à l’aide de la méthode analytique GRADE (Figure 2C). Le médicament GRADE juxtapose la fraction létale, qui est proportionnelle au taux de mortalité moyen, à la valeur GR, le taux de croissance net de la population. L’intégration des données de cette manière permet de visualiser comment un médicament coordonne l’inhibition de la croissance et l’activation de la mort. Dans le graphique GRADE : les médicaments qui inhibent la croissance sans activer la mort (c’est-à-dire les médicaments cytostatiques, selon l’interprétation courante) occuperont la limite supérieure, les médicaments qui ne tuent que sans modifier les taux de prolifération cellulaire occuperont la limite droite, et les médicaments qui provoquent d’abord l’inhibition de la croissance, suivie de la mort des cellules arrêtées par la croissance, occuperont la limite gauche (c’est-à-dire médicaments biphasiques ; Figure 2C). On peut en déduire que les médicaments qui entraînent des réponses GR/LF qui se situent à l’intérieur de ces limites inhibent simultanément la prolifération dans une certaine mesure tout en activant la mort cellulaire (c’est-à-dire les médicaments coïncidents)3. Il est important de noter que la position de chaque réponse médicamenteuse dans l’espace GR/LF peut être utilisée pour calculer le taux de prolifération réel (p) et le taux de mortalité moyen (d) pour chaque concentration d’un médicament (Figure 2D-E). Il convient de noter que la valeur RG n’est pas le taux réel de prolifération cellulaire, mais plutôt le taux de croissance net de la population (c’est-à-dire l’effet net combinant le taux de prolifération réel et le taux de mortalité induit par la drogue). De plus, le taux de mortalité produit dans une analyse GRADE (d, qui est le taux de mortalité moyen) est distinct du paramètre de taux de mortalité dans l’analyse cinétique des LED (DR, la vitesse maximale ou le taux de mortalité maximal).
Le test FLICK peut générer des données spécifiques à la mort, telles que la fraction létale induite par le médicament ; Cependant, ces données sont agnostiques au mécanisme de la mort cellulaire. Pour mieux comprendre le mécanisme de la mort cellulaire à l’aide de FLICK, la méthode la plus simple consiste à déterminer comment la cinétique de la fraction létale est modifiée par l’inclusion d’inhibiteurs spécifiques à la voie de mort. L’inhibition d’un mécanisme de mort pertinent devrait entraîner une diminution de la fraction létale induite par le médicament et/ou un retard dans le moment du début de la mort. Ici, nous montrons que 50 μM de l’inhibiteur de la pan-caspase z-VAD sauve environ 50 % de la létalité induite par 1 μM de bélinostat (Figure 2F). Il est important de noter que, comme les médicaments, les inhibiteurs ont une spécificité limitée et peuvent accidentellement inhiber ou exacerber d’autres mécanismes de mort. Ainsi, avant qu’une conclusion définitive ne soit tirée, ces données doivent être complétées par d’autres informations, telles que la morphologie cellulaire, la mesure des marqueurs biochimiques d’activation et/ou l’évaluation des dépendances génétiques spécifiques à la voie14.
Pour illustrer formellement comment l’interprétation d’une réponse médicamenteuse peut être modifiée par une évaluation basée sur Flick et une analyse basée sur GRADE, nous avons ensuite exploré trois médicaments qui présentent différents types de coordination croissance/mort. Le palbociclib est un inhibiteur de Cdk4/6 qui entraîne une réponse cytostatique (inhibition de la croissance sans mort cellulaire). La camptothécine est un inhibiteur de la topoisomérase I qui provoque une réponse biphasique (inhibition de la croissance, suivie de la mort cellulaire à fortes doses). Le bélinostat est un inhibiteur de l’HDAC qui provoque une réponse coïncidente (inhibition partielle de la croissance et activation de la mort à chaque dose, mais avec des proportions variables selon les doses). En utilisant une analyse conventionnelle de ces données, on peut observer que les trois médicaments diminuent considérablement la viabilité relative des cellules U2OS (Figure 2G). Bien que le palbociclib soit nettement moins puissant, il n’est pas clair à partir de ces données que le palbociclib ne parvient pas à activer la mort cellulaire. À l’aide d’une analyse basée sur la RG, qui indique comment les médicaments affectent le taux de croissance net de la population, on peut interpréter plus précisément que les populations traitées au palbociclib continuent de croître à toutes les doses, tandis que des doses élevées de camptothécine ou de belinostat entraînent une diminution de la population, et donc, la mort cellulaire doit avoir été activée après de fortes doses de ces médicaments (Figure 2H). Cependant, une analyse basée sur la RG ne tient pas compte de la coordination variée entre la croissance et la mort, ce qui peut conduire à des conclusions erronées sur le niveau de mort cellulaire activé par un médicament donné. Par exemple, sur la base des données RG, on pourrait conclure que 1 μM de camptothécine active la mort cellulaire dans une plus grande mesure que 1 μM de Belinostat, étant donné que la camptothécine donne une valeur de RG de -0,25 et Belinostat donne une valeur de RG de 0 ; une valeur de -0,25 GR signifie que la population diminue de 25 % aussi vite que la population non traitée augmente. À l’aide d’une visualisation et d’une analyse basées sur GRADE, on peut observer que la camptothécine et le belinostat diffèrent dans leur coordination croissance/mort. Ainsi, à la dose de 1 μM, les deux belinostat activent la mort à un taux plus élevé que la camptothécine (0,8 % de décès/h pour le bélinostat, contre 0,7 % de décès/h pour la camptothécine), mais la population traitée par la camptothécine diminue plus rapidement en raison d’une inhibition de croissance plus prononcée induite par ce médicament (Figure 2I).
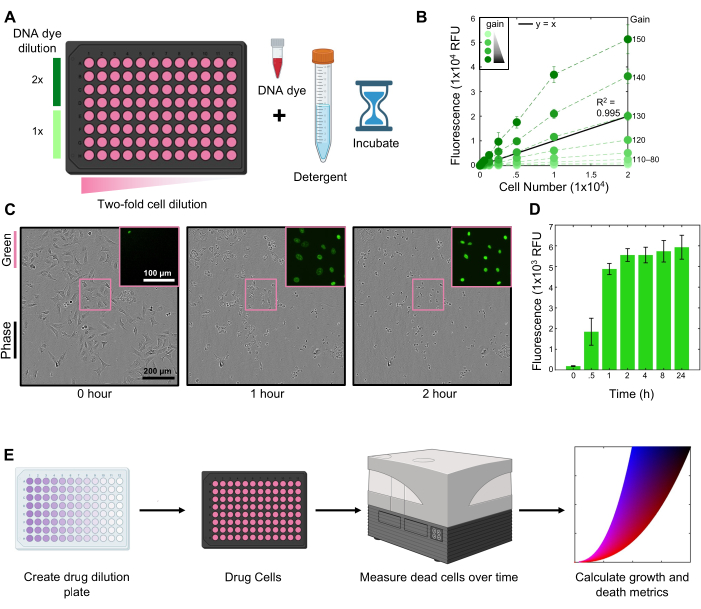
Figure 1 : Organigramme du protocole FLICK. (A) Créer une dilution cellulaire pour optimiser la concentration du colorant d’ADN et la linéarité des mesures de fluorescence sur un lecteur de microplaques. (B) Titrage du gain en fonction du nombre de cellules pour 2 μM SYTOX Green dans les cellules U2OS. Un gain de 130 fournit le signal le plus linéaire sur tous les numéros de cellules. (C) Images de phase de cellules U2OS lysées avec 1,5 % de Triton-X au fil du temps à l’aide d’un objectif 10x. La région encadrée est une vue agrandie de la fluorescence SYTOX Green. Les barres d’échelle sont représentatives de toutes les images. (D) SYTOX Signal vert après la lyse aux moments indiqués. Les données sont la moyenne +/- ET pour 3 réplicats biologiques. (E) Un aperçu conceptuel de l’essai, de la mise en place à l’analyse. Les étapes clés sont la création d’une plaque de dilution de médicament, la mise en médicament des cellules dans une plaque multi-puits, l’acquisition de mesures de fluorescence au fil du temps et le calcul des paramètres de croissance et de mort. Pour les panels B et D, les données sont moyennes ± ET pour n = 3 réplicats biologiques indépendants. Veuillez cliquer ici pour voir une version agrandie de cette figure.
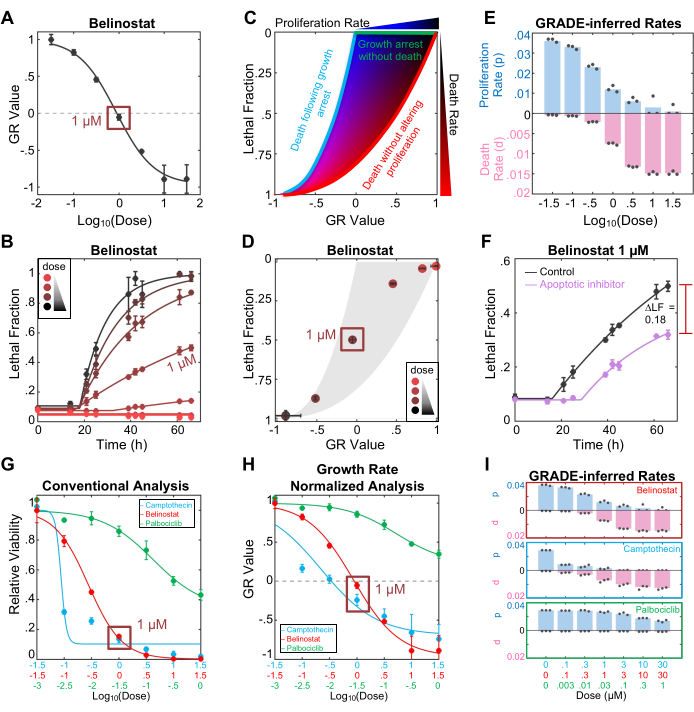
Figure 2 : Caractérisation des réponses médicamenteuses à l’aide du test FLICK et de l’analyse basée sur GRADE. (A) Mesure GR pour l’inhibiteur de l’HDAC, Belinostat, sur une dose de doses. Les données présentées proviennent du critère d’évaluation de l’essai, qui était de 66 h. À 1 μM, Belinostat a une valeur GR d’environ 0, indiquant une réponse cytostatique. (B) Cinétique de fraction létale pour Belinostat. À 1 μM, Belinostat induit une létalité de près de 50 %, qui n’est pas prise en compte par la valeur GR. (C) Diagramme de phase GRADE basé sur une simulation de tous les taux de croissance et de mortalité possibles. La simulation a été basée sur le temps de doublement d’U2OS dans ce test (27,12 h). (D) Graphique GRADE pour Belinostat en fonction de la dose au critère d’évaluation. (E) Diagramme à barres des taux de croissance (p, doublement de la population par heure) et de décès (d, fraction de la population qui meurt par heure) inférés par GRADE pour les doses de Belinostat. Les taux ont été déterminés à partir des taux simulés en (C) et des données en (D). (F) Graphique de la fraction létale pour 1 μM de belinostat traité avec (violet) ou sans (noir) 50 μM de l’inhibiteur apoptotique z-VAD. ΔLF = 0,18 a été calculé en soustrayant la LF maximale moyenne de l’affection traitée par inhibiteur de la LF maximale moyenne du témoin. (G) Viabilité relative (nombre de cellules vivantes dans l’état traité par le médicament, divisé par le nombre de cellules vivantes dans l’état non traité) pour trois médicaments après une exposition de 72 heures. Tous les médicaments réduisent la viabilité cellulaire, mais la contribution de la mort cellulaire à la réponse n’est pas connue. (H) Mesure RG pour trois médicaments en (G). Ces données démontrent que deux des trois médicaments entraînent une diminution de la population, indiquée par le RG < 0. La contribution de la mort cellulaire n’est pas encore claire, bien que la mort cellulaire ait dû être activée lorsque la valeur GR est négative. (I) Taux déduits par GRADE pour trois médicaments. Le palbociclib n’est pas létal mais entraîne un arrêt de la croissance. La camptothécine est biphasique ; Des doses plus faibles entraînent un arrêt de la croissance, tandis que des doses plus élevées entraînent un arrêt de la croissance suivi d’une létalité. Le bélinostat est une coïncidence ; la gamme de doses passe au milieu du graphique GRADE, indiquant que toutes les doses entraînent une perturbation de la croissance et une activation de la mort cellulaire. Les données de tous les panels correspondent à la moyenne ± ET de trois expériences biologiques répliquées indépendantes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Teindre | Cinétique | Point final |
| SYTOX Vert | Y | Y |
| CellTox Vert | Y | Y |
| NucSpot 500/515, 594/615, 750/780 | Y | Y |
| YOYO-3 | Y | Y |
| Iodure de propidium | N | Y |
| 7-AAD | N | Y |
Tableau 1 : Les colorants d’ADN et leur application dans FLICK. De nombreux types de colorants peuvent être utilisés pour les tests FLICK finaux uniquement. Moins de colorants remplissent les conditions pour être incubés en continu tout au long de l’essai. D’autres colorants, y compris les colorants qui ne sont pas verts, n’ont pas été caractérisés en profondeur et doivent être soigneusement évalués avant d’être utilisés.
| Chemin de la mort | Inhibiteur | Dose typique |
| Apoplose intrinsèque | ZVAD-FMK | 50 μM |
| Apoplose extrinsèque | Z-IETD | 30 μM |
| Ferroptose | Ferrostatine-1 | 10 μM |
| Nécroptose | Nécrostatine-1 | 50 μM |
| Pyroptose | VX-765 | 50 μM |
| Parthanatos | Le rucaparib | 1 μM |
| Mort cellulaire autophagique dépendante | Hydroxychloroquine (HCQ) | 10 μM |
| Mort cellulaire dépendante lysosomale | E-64D | 10 μM |
| Cuproptose | Tétrathiomolybdate (TTM) | 5 μM |
| Oxeiptose | N-acétyl-l-cystéine (NAC) | 2 millions d’euros |
| Nécrose induite par MPT | Cyclosporine A (CsA) | 10 μM |
Tableau 2 : Voies de mort cellulaire et leurs inhibiteurs. Les inhibiteurs de la mort cellulaire doivent être validés par rapport à un activateur canonique pour chaque voie de mort. Idéalement, les doses d’inhibiteurs sélectionnées ne devraient pas affecter la viabilité cellulaire.
Tableau supplémentaire 1 : Calculateur GRADE. Tableur pour calculer les taux de croissance et de mortalité induits par les médicaments à partir de données RG et FV basées sur FLICK. Les utilisateurs doivent saisir des données sur les RG et la VF, ainsi que la durée de l’essai en heures, le temps de doublement des cellules en heures et la taille de la population au début et à la fin. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le test FLICK est une méthode robuste pour générer une évaluation complète de l’effet d’un médicament sur la croissance et la mort d’une population cellulaire. Étant donné que cette méthode ne compte pas directement les cellules, les étapes critiques du protocole consistent à assurer la linéarité du test et la lyse complète pendant les étapes de perméabilisation des tritons. Le temps de perméabilisation correct peut être identifié visuellement, comme mis en évidence dans ce protocole, ou quantitativement en lisant la fluorescence de la plaque au fil du temps et en identifiant le moment où le signal se stabilise. D’après notre expérience, cependant, en cas de doute, il ne semble pas y avoir de coût à attendre plus longtemps : pour les colorants d’ADN que nous avons utilisés dans le test FLICK, l’intensité du signal reste stable pendant plusieurs jours après la lyse. Nous utilisons régulièrement le colorant d’acide nucléique SYTOX Green, et bien que nous n’ayons pas validé chaque fluorophore référencé dans le tableau 1, plusieurs autres protocoles démontrent leur utilisation efficace 13,15,16. L’évaluation de la linéarité d’un colorant d’ADN imprégné d’une cellule donnée sur une gamme de nombres de cellules et de paramètres d’instruments garantira des données robustes et quantitativement fiables.
Certaines lignées cellulaires sont plus difficiles à lyser que d’autres, et il ne faut jamais supposer que le temps de lyse d’une lignée cellulaire est le même quecelui de 13 autres. Pour certaines lignées cellulaires, à des densités plus élevées, la lyse cellulaire provoque le décollage des cellules du puits sous forme de feuille, ce qui peut provoquer une erreur de lecture de plaques. Cela peut être évité en réduisant la densité de semis de départ ou en mélangeant les puits à la pipette après la lyse et avant la lecture. Pour améliorer le temps de lyse ou l’efficacité de la lyse, la solution Triton-X peut être ajustée à un pourcentage plus ou moins élevé, mais nous n’avons pas encore rencontré de scénario où cela était nécessaire.
La conception expérimentale et la disposition du placage du médicament doivent être soigneusement prises en compte lorsque des données de croissance robustes sont souhaitées. La création d’une plaque de dilution de médicament pour le test FLICK est la même que pour tout autre test sur plaque de 96 puits. Cependant, l’inclusion de nombreux contrôles couvrant différentes parties du paysage d’une plaque aidera à créer des informations solides sur la cinétique de croissance et peut aider à déterminer s’il existe une variation systématique de la croissance. En évitant les puits extérieurs d’une plaque, vous obtiendrez des données de croissance plus précises, car ces puits sont plus sensibles aux fluctuations de température, d’oxygène ou d’humidité, ce qui affecte leur croissance. Enfin, l’utilisation d’un modèle de médicament pseudo-randomisé, comme le changement d’emplacement d’un médicament sur des plaques répétées, fournira une évaluation plus précise du comportement du médicament.
Il existe certaines limites pour l’utilisation précise du test FLICK. Ces limites sont principalement liées à l’inférence cinétique, car les données du paramètre sont des valeurs observées expérimentalement. La cinétique de prolifération des cellules vivantes et la cinétique de mort dépendent d’une hypothèse concernant la trajectoire de croissance des cellules vivantes au fil du temps dans la condition traitée par médicament. Dans ce protocole, nous décrivons cette trajectoire de croissance comme exponentielle et avec un taux uniforme dans le temps. Bien que ces caractéristiques soient susceptibles d’être observées en l’absence d’un médicament, elles peuvent ne pas être respectées en présence d’un médicament. La méthode FLICK est exacte, non pas parce que les hypothèses concernant les trajectoires de croissance sont toujours correctes, mais plutôt parce que l’impact de toute hypothèse incorrecte est minimisé si la population traitée par le médicament ne prolifère pas beaucoup (c’est-à-dire moins de 2 ou 3 doublements de population au cours de la période d’essai). Nous n’avons pas rencontré de médicaments qui ne peuvent pas être profilés avec précision dans FLICK ; cependant, la théorie suggère que le test FLICK cessera d’être précis pour un médicament dans lequel la mort se produit très lentement, sur de longues périodes de temps qui sont plusieurs fois du temps de doublement cellulaire (remarque : nous n’avons identifié aucun médicament présentant ces caractéristiques3). Cependant, l’impact de toute hypothèse incorrecte sur la cinétique de croissance de la population totale n’affectera pas le temps d’apparition de la mort obtenu à partir de l’ajustement des LED ou les valeurs maximales finales des cellules mortes, car celles-ci sont limitées par les valeurs de cellules mortes mesurées empiriquement et les valeurs totales finales des cellules qui ont été déterminées expérimentalement à la fin de l’essai.
Malgré ces limites, le test FLICK permet d’obtenir des informations qui sont difficiles à générer à l’aide d’autres méthodes de réponse aux médicaments. La plupart des méthodes de réponse aux médicaments génèrent un signal proportionnel au nombre de cellules vivantes, et ces méthodes peuvent être utilisées pour quantifier comment les médicaments affectent le taux de croissance net de la population (c’est-à-dire la valeur GR ou équivalent), mais ne peuvent pas faire la distinction avec précision entre les effets cytotoxiques et cytostatiques d’un médicament. Alternativement, des tests basés sur la microscopie qui mesurent à la fois les cellules vivantes et mortes peuvent certainement générer une image complète. Cependant, les tests basés sur la microscopie peuvent avoir du mal à compter les cellules mortes après leur décomposition en débris, comme cela se produira rapidement dans le contexte de nombreuses formes non apoptotiques de mort cellulaire. L’une des principales caractéristiques de FLICK est que les mesures sont effectuées dans un lecteur de plaques, qui agrège la fluorescence totale des cellules mortes plutôt que de compter directement les cellules mortes. Ainsi, le signal des cellules mortes dans FLICK ne dépend pas de la cellularité intacte des cellules mortes, qui, dans le contexte de la mort cellulaire non apoptotique, est particulièrement essentielle. De plus, une caractéristique unique du test FLICK est la capacité de mesurer à la fois les populations de cellules vivantes et mortes à l’aide du même réactif. Ainsi, le test FLICK produit des mesures de cellules vivantes et mortes avec la même sensibilité. Cette fonctionnalité permet l’analyse à l’aide de la méthode GRADE et améliore la précision des calculs simultanés des taux de croissance et de mortalité de GRADE. Le tableau supplémentaire 1 inclus comprend un modèle utilisé pour la visualisation du GRADE du médicament, où l’utilisateur peut saisir la longueur du dosage, le taux de croissance calculé à l’étape 7.1, les numéros de cellules de début et de fin ou les lectures fluorescentes à partir des conditions de contrôle, ainsi que la LF et la RG calculées à partir des étapes 5.4 et 6.3. Le fichier contient une simulation de toutes les combinaisons par paires de 50 taux de croissance et de mortalité qui seront automatiquement mises à jour en fonction des paramètres définis par l’utilisateur. Le modèle génère une visualisation basée sur GRADE de la réponse au médicament et des taux de croissance (p) et de mortalité (d) déduits par GRADE.
Les travaux futurs devraient explorer l’application du test FLICK dans des scénarios de culture cellulaire 3D ou dans l’évaluation des réponses médicamenteuses d’organoïdes dérivés de tumeurs. La linéarité et la sensibilité des données FLICK n’ont pas été profondément remises en question dans ces contextes, mais en théorie, le test FLICK devrait être efficace avec quelques modifications pour intégrer la fluorescence dans des échantillons tridimensionnels. De plus, l’ajout de marqueurs cellulaires supplémentaires aiderait à distinguer deux ou plusieurs types de cellules en coculture. Ces avancées seront précieuses pour explorer les interactions entre les cellules immunitaires et les cellules cancéreuses. Enfin, des tests comme SPARKL inspirent l’utilisation de rapporteurs spécifiques à la mort dans le format de test FLICK, ce qui peut améliorer le débit tout en maintenant la précision des informations sur la mort cellulaire11,16.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions tous les membres passés et actuels du Lee Lab pour leurs contributions à la perspective de notre laboratoire sur l’évaluation des réponses aux médicaments. Ce travail a été soutenu par un financement des National Institutes of Health à MJL (R21CA294000 et R35GM152194).
matériels
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
Références
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon