Method Article
Imagem e Quantificação da Morfologia Mitocondrial em C. elegans Durante o Envelhecimento
Neste Artigo
Resumo
Este protocolo fornece uma abordagem padronizada para imagens da morfologia mitocondrial em múltiplos tecidos de C. elegans durante o envelhecimento.
Resumo
As mitocôndrias, importantes organelas celulares encontradas na maioria das células eucarióticas, são os principais locais de produção de energia por meio da respiração aeróbica. Além desse papel bem conhecido como a 'usina de energia celular', as mitocôndrias também estão envolvidas em muitos outros processos celulares essenciais, incluindo a regulação do metabolismo celular, proliferação, sinalização imunológica e sinalização hormonal. A deterioração da função mitocondrial durante o envelhecimento ou sob estresse mitocondrial é frequentemente caracterizada por mudanças distintas na morfologia e no volume mitocondrial. O nematóide C. elegans é um modelo ideal para estudar essas alterações devido ao seu corpo transparente e curta vida útil, o que facilita a microscopia ao vivo ao longo de sua vida. No entanto, mesmo dentro do campo de C. elegans , numerosas construções e métodos transgênicos para imagens mitocondriais estão disponíveis, cada um com suas próprias limitações. Aqui, as construções GFP localizadas em matriz de cópia única são apresentadas como um método robusto e confiável para imagens da morfologia mitocondrial em C. elegans. Este estudo se concentra especificamente em fatores experimentalmente controláveis para minimizar erros e reduzir a variabilidade entre réplicas e entre estudos ao realizar imagens mitocondriais durante o processo de envelhecimento. Além disso, o mitoMAPR é recomendado como um método robusto para quantificar as mudanças na morfologia mitocondrial entre os tipos de tecido durante o envelhecimento.
Introdução
As mitocôndrias são organelas celulares envoltas por uma membrana fosfolipídica dupla e desempenham um papel importante na manutenção da bioenergética celular como o local chave para o metabolismo e a produção de energia1. Um grande número de mitocôndrias gera continuamente energia na forma de ATP para atender às demandas celulares2. Além desses papéis importantes, as mitocôndrias também participam de processos celulares complexos, como transdução de sinal, autofagia, imunidade inata, ciclo celular e vias de morte celular 2,3. As mitocôndrias exibem diversas morfologias, variando de pequenas organelas individuais a extensas redes tubulares interconectadas, dependendo dos diferentes requisitos energéticos e da saúde mitocondrial4.
Uma característica fundamental das mitocôndrias é sua natureza dinâmica, por meio da qual elas podem alternar entre essas várias formas por meio de uma sequência contínua e bem coordenada de eventos de fissão e fusão4. O equilíbrio preciso entre esses processos opostos regula a morfologia, número, tamanho e posicionamento mitocondrial dentro do citoplasma5. Além disso, esses processos de fusão e fissão são importantes para manter o controle de qualidade das mitocôndrias5. Por exemplo, a fissão mitocondrial é um componente essencial da remoção de mitocôndrias danificadas por meio de mitofagia e remoção seletiva de mitocôndrias por meio de autofagia2. A dinâmica mitocondrial também desempenha um papel importante na divisão celular, desenvolvimento, resistência a vários estressores e manutenção do metabolismo celular6.
A interrupção da dinâmica mitocondrial está implicada em inúmeras doenças, incluindo distúrbios neurodegenerativos, doenças metabólicas, doenças cardiovasculares e cânceres4. Além disso, o envelhecimento está associado à remodelação dramática das mitocôndrias, em grande parte devido a mudanças nessas dinâmicas de fusão-fissão7. Portanto, visualizar e monitorar as mudanças na morfologia mitocondrial sob várias condições de estresse ou doença e ao longo do processo de envelhecimento oferece informações valiosas para entender a função celular, o mecanismo da doença e as possíveis estratégias terapêuticas.
Como muitas vias moleculares, a dinâmica e a função mitocondriais também são altamente conservadas em eucariotos, incluindo organismos modelo como o nematóide Caenorhabditis elegans. Semelhante às células humanas, a fusão mitocondrial em C. elegans é alcançada pela função das proteínas guanina trifosfatase (GTPase) relacionadas à dinamina, incluindo FZO-1 (ortólogo de mamíferos Mfn1/2) e EAT-3 (ortólogo de mamíferos Opa1) que controlam a fusão da membrana mitocondrial externa (OMM) e da membrana mitocondrial interna (IMM), respectivamente8. A fissão mitocondrial é regulada pela proteína relacionada à dinamina (DRP-1, ortólogo do Drp1 humano), que promove a fissão mitocondrial formando complexos semelhantes a anéis ao redor da membrana externa mitocondrial que contraem e, eventualmente, separam as membranas mitocondriais9. A fusão mitocondrial desempenha um papel fundamental no controle de qualidade mitocondrial, permitindo a mistura de conteúdos mitocondriais, incluindo DNA mitocondrial, proteínas e lipídios, permitindo a complementação de mitocôndrias parcialmente danificadas por conteúdos de mitocôndrias saudáveis9. Por outro lado, a fissão mitocondrial permite que as mitocôndrias se dividam, criando novas mitocôndrias e facilitando sua distribuição não apenas dentro do citoplasma, mas também para as células-filhas durante a divisão celular, garantindo herança e função mitocondrial adequadas9. Este evento também é essencial para segregar segmentos mitocondriais danificados ou disfuncionais, que podem então ser alvo de degradação por meio da mitofagia9.
C. elegans tem sido considerado um dos sistemas de modelo genético mais poderosos devido ao genoma completo e à disponibilidade de uma variedade de ferramentas genéticas, incluindo CRISPR / Cas9 que facilita modificações genéticas10, vários métodos para superexpressão de genes e um método baseado em alimentos bacterianos para interferência de RNA (RNAi) 11 . Além disso, sua anatomia transparente permite imagens microscópicas em organismos vivos12. Finalmente, sua vida útil relativamente curta, baixos custos de manutenção e facilidade de produzir um grande número de animais pareados por idade os tornam um excelente sistema modelo para a biologia do envelhecimento13. Esses benefícios, combinados com a conhecida conservação das principais vias regulatórias mitocondriais, tornam C. elegans um sistema modelo altamente atraente para estudar a dinâmica mitocondrial durante o envelhecimento.
Os marcadores fluorescentes são amplamente utilizados em pesquisas biológicas para visualizar e estudar componentes celulares, incluindo mitocôndrias. Existem corantes permeáveis a células específicos, como MitoTracker e éster etílico de tetrametilrodamina (TMRE), que são comumente usados14. O primeiro é usado para tingir a matriz mitocondrial geral carregada negativamente15, enquanto o último é usado para avaliar o potencial relativo da membrana mitocondrial por meio de seu íon trifenilfosfônio carregado positivamente16. Embora eles se beneficiem de não exigir transgênicos, a cutícula espessa dos vermes que muda de estrutura e permeabilidade durante o envelhecimento e a variabilidade da infiltração de corante em diferentes tecidos tornam as abordagens baseadas em corantes desafiadoras em C. elegans17. Além disso, a avaliação da morfologia mitocondrial é confundida por potenciais efeitos fora do alvo dos corantes, como a agregação do corante17. Em vez disso, métodos genéticos para expressar fluoróforos localizados nas mitocôndrias são comumente usados no modelo de vermes.
Aqui, este estudo se concentra em destacar cepas que expressam uma GFP localizada na matriz mitocondrial (doravante referida como MLS :: GFP) sob promotores específicos do tipo de célula. É importante ressaltar que essas linhagens transgênicas foram feitas usando o método MosSCI para garantir a expressão de cópia única do repórter em um locus genômico conhecido, o que evita problemas com outras cepas disponíveis. Por exemplo, a alta expressão de cópias de algumas proteínas direcionadas às mitocôndrias leva à variabilidade nos níveis de expressão devido à integração de matrizes de plasmídeos extracromossômicos em um locus aleatório com um número de cópia desconhecido18. Além disso, eles já demonstraram induzir danos às mitocôndrias, uma vez que é uma alta carga para as células localizarem adequadamente as proteínas mitocondriais superexpressas19. Portanto, a expressão gênica precisa, estável e baixa e a facilidade de cruzamento com loci conhecidos tornam esses transgênicos MosSCI o método preferido. Usando essas cepas, este estudo padroniza métodos para obter imagens de mitocôndrias no músculo, intestino e hipoderme de C. elegans. Além disso, destaca as importantes aplicações técnicas e métodos de solução de problemas que são importantes para considerar e garantir a análise experimental reprodutível das mitocôndrias durante o envelhecimento em C. elegans.
Protocolo
Detalhes sobre os reagentes e equipamentos usados neste estudo estão listados na Tabela de Materiais.
1. Crescimento e manutenção de C. elegans
- Preparação de placas de meios de crescimento de nematóides (NGM)
- Para a cultura de C. elegans, use placas de ágar 2% padrão com NGM contendo 1 mM de CaCl2, 12,93 μM (5 μg/mL) de colesterol, 25 mM de KPO4 (pH 6,0), 1 mM de MgSO4, 2,5 mM (0,25% p/v) de peptona e 51,3 mM de NaCl. Veja o método detalhado de vazamento de placas NGM descrito em Castro Torres et al.20.
NOTA: Para experimentos de interferência de RNA (RNAi), adicione 1 mL de IPTG 1 M e 1 mL de carbenicilina 100 mg / mL para placas de ágar 1 L NGM. - Depois que as placas NGM estiverem solidificadas, cultive uma cultura de OP50 em caldo de lisogenia (LB) por 24-48 h à temperatura ambiente ou cultive uma cultura de HT115 carregando um plasmídeo vetor vazio pL4440 em LB com antibióticos (ampicilina / carbenicilina 100 μg / mL + tetraciclina 5 μg / mL) com agitação a 37 ° C por 12-16 h.
NOTA: Este plasmídeo está disponível em fontes comerciais (consulte a Tabela de Materiais). - Semear 200 μL de cultura OP50 ou HT115 em placas de 60 mm ou 1 mL em placas de 100 mm.
- Seque as placas até que não estejam mais molhadas e guarde-as em recipientes fechados a 4 °C por até 2 meses.
- Opcional: Adicione 100 μL de 10 mg / mL de 5-fluoro-2'-desoxiuridina (FUDR) diretamente em placas de ágar NGM semeadas com bactérias para esterilizar quimicamente vermes L4 / adultos e prevenir o desenvolvimento de vermes mais jovens.
NOTA: As estirpes desenvolvidas e utilizadas neste estudo são:
(1) RHS191 - uthSi17[myo3p::MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] I
(2) RHS192 - uthSi83[col19p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)]I
(3) RHS193 - uthSi80[vha-6p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] IV
- Para a cultura de C. elegans, use placas de ágar 2% padrão com NGM contendo 1 mM de CaCl2, 12,93 μM (5 μg/mL) de colesterol, 25 mM de KPO4 (pH 6,0), 1 mM de MgSO4, 2,5 mM (0,25% p/v) de peptona e 51,3 mM de NaCl. Veja o método detalhado de vazamento de placas NGM descrito em Castro Torres et al.20.
- Sincronização C. elegans por branqueamento
- Para um ensaio em grande escala que requer um grande número de nematóides, corte uma placa de ágar NGM de 60 mm cheia de animais em pedaços pequenos e corte-os em placas de 100 mm semeadas com bactérias para expansão. Aguarde 2-3 dias para que as placas de 100 mm fiquem cheias de vermes quando cultivadas a 20 ° C após o corte. Para recomendações mais detalhadas para animais em expansão, consulte Castro Torres et al.20.
- Para coletar os vermes para sincronização, despeje 5-10 mL de solução M9 (22 mM de KH2PO4 monobásico, 42,3 mM de Na2HPO4, 85,6 mM de NaCl, 1 mM de MgSO4) em placas de ágar NGM de 100 mm com minhocas e agite a solução M9 suavemente para soltar as minhocas dos gramados bacterianos.
- Retirar os vermes com uma pipeta serológica e transferi-los para tubos cónicos de 15 ml.
NOTA: Pipetas sorológicas de vidro são recomendadas, pois as pipetas sorológicas de plástico tendem a fazer com que os vermes grudem na parede interna das pipetas de plástico. - Centrifugue por 30 s a 1.100 x g à temperatura ambiente para derrubar os vermes e aspirar o sobrenadante usando uma bomba de vácuo.
- Prepare 5 mL de solução clareadora contendo 1,5 mL de hipoclorito de sódio a 6%, 0,75 mL de 5 M NaOH ou KOH e 2,75 mL de dH2O. Despeje os 5 mL de solução clareadora no pellet de minhoca.
CUIDADO: Recomenda-se o uso de luvas e jaleco nesta etapa, pois as soluções de hipoclorito de sódio e hidróxido são corrosivas. - Incube a mistura de minhoca + solução de branqueamento por ~ 4-6 min até que todos os corpos dos animais estejam totalmente dissolvidos e apenas os ovos permaneçam. Agite a mistura vigorosamente para ajudar no processo de dissolução.
NOTA: Verifique os vermes a cada minuto sob um microscópio de dissecação. Encurte ou estenda o tempo de branqueamento com base em quanto os corpos dos vermes adultos são dissolvidos. Deixar os ovos dentro de uma solução de branqueamento por um longo período resultará em danos aos ovos e afetará a viabilidade dos nematóides. - Centrifugue a mistura de ovos/solução de branqueamento por 30 s a 1.100 x g à temperatura ambiente para granular os ovos e, em seguida, aspire cuidadosamente o sobrenadante usando uma bomba de vácuo, certificando-se de não aspirar o pellet de ovo.
NOTA: Recomenda-se realizar lavagens rapidamente para evitar a exposição prolongada dos ovos à solução de branqueamento. - Lave os ovos com 5-10 mL de solução M9 4 vezes.
- Ressuspenda os ovos em um pequeno volume de solução M9 e coloque-os em placas de NGM semeadas com bactérias pipetando até 50 μL. Calcule uma estimativa aproximada do número de ovos pipetando 3-5 μL da mistura de ovos em uma placa e contando o número de ovos neste volume. Adicione M9 para diluir a mistura de ovos em um número contável de ovos neste volume.
NOTA: Recomenda-se o plaqueamento de menos de 100 minhocas por placa, pois as minhocas podem morrer de fome antes do dia 1 da idade adulta, quando há muitos animais banhados. Para recomendações mais detalhadas para o número de animais em placas, consulte Castro Torres et al.20. - Como alternativa, Prenda os nematóides em L1 para uma sincronização de idade mais estreita. Para a parada de L1, adicione o volume apropriado de solução M9 ao pellet de ovo em um tubo cônico de 15 mL a um volume de 10-12 mL. Deixe as minhocas girarem em um rotador por 16-24 h a 20 °C. Centrifugar (como na etapa 1.2.7) e aspirar o sobrenadante. Calcular a concentração de animais L1 e colocá-los em placas NGM, conforme descrito para os ovos no passo 1.2.9.
- Envelheça a população de C. elegans até a idade desejada: Transfira os animais L4 / adultos para placas contendo 5-fluoro-2'-desoxiuridina (FUDR), conforme descrito na etapa 1.1.6, para evitar a progênie e eliminar qualquer prole já produzida. Para métodos alternativos de envelhecimento de vermes sem FUDR, consulte Castro Torres et al.20.
NOTA: Alguns fatores importantes precisam ser considerados antes de escolher o branqueamento como método para sincronizar vermes em seu primeiro estágio larval. Conforme destacado anteriormente, a superexposição ao alvejante pode danificar embriões, reduzir as taxas de eclosão e criar pressão de seleção para cepas resistentes ao alvejante, potencialmente alterando a genética da população. Além disso, se os embriões não forem alimentados por longos períodos de tempo, a depleção de nutrientes pode prejudicar o metabolismo e o desenvolvimento. Portanto, os métodos de postura de ovos ou NemaSync podem ser usados como uma alternativa aos métodos de branqueamento. O método para o procedimento de postura de ovos é descrito passo a passo no referido estudo de Castro Torres et al.20. O NemaSync é um método comercial mais novo e mais caro, onde um sincronizador de worm manual é usado para sincronizar worms sem o uso de produtos químicos21.
2. Imagem de mitocôndrias em C. elegans
- Preparação de lâminas para imageamento de C. elegans
- Adicione 10-20 μL de solução M9 em lâminas de vidro e transfira um número desejado de vermes adultos nas idades específicas para a solução na lâmina. Aproximadamente 25 vermes são recomendados.
NOTA: O uso de métodos tradicionais para paralisar vermes (por exemplo, tetramisol ou azida de sódio) pode causar fragmentação mitocondrial sob certas condições (veja abaixo). Em vez disso, recomenda-se o uso de lâminas HistoBond aqui, que têm uma superfície com uma carga superficial positiva permanente, reduzirá o movimento de vermes sem o uso de drogas que possam introduzir artefatos. É importante colocar uma proporção adequada de minhocas para solução M9, pois volumes baixos podem fazer com que os animais sejam esmagados entre a lâmina e a lamínula, enquanto volumes altos resultarão em animais se movendo durante a aquisição da imagem. As recomendações podem ser encontradas na Tabela 1. - Alternativamente, para o uso de medicamentos que paralisam vermes, consulte os resultados representativos deste manuscrito para uma variedade de concentrações e pontos de tempo que podem ser usados com fragmentação limitada das mitocôndrias.
NOTA: Embora não tenha havido grande fragmentação em animais saudáveis do tipo selvagem, é importante lembrar que certos mutantes ou condições podem sofrer fragmentação mitocondrial com a mesma concentração desses medicamentos, portanto, alguns testes são necessários ao usar esses medicamentos. - Cubra os vermes com solução M9 na lâmina com uma lamínula e use esmalte para selar as laterais e evitar a evaporação da solução M9.
- Opcional: Para imagens musculares, role os vermes empurrando suavemente a lamínula. Isso permite que os músculos da parede do corpo nas laterais dos vermes fiquem melhor alinhados com o plano focal para imagens de alta qualidade.
- Adicione 10-20 μL de solução M9 em lâminas de vidro e transfira um número desejado de vermes adultos nas idades específicas para a solução na lâmina. Aproximadamente 25 vermes são recomendados.
- Imagem de mitocôndrias usando um microscópio composto
- Use um microscópio de campo amplo padrão para mitocôndrias musculares e hipodérmicas (myo-3p::MLS::GFP e col-19p::MLS::GFP).
NOTA: Um gerador de imagens disponível comercialmente equipado com uma objetiva Plan Aprochromat 63x/1.4, filtro GFP (11525314), câmera DFC9000 GT e fonte de luz LED5 foi usado, e software de microscópio compatível foi usado. - Otimize as configurações de imagem para cada microscópio e configuração experimental.
NOTA: No entanto, como ponto de partida, os parâmetros para imagens com o gerador de imagens usado neste manuscrito são os seguintes: As dimensões são 2048 (x) * 2048 (y) pixels em um tamanho de passo de 0,50 μm para mitocôndrias hipodérmicas e "Otimizado para o sistema" calculado automaticamente pelo software para mitocôndrias musculares. O tempo de exposição é de 50 ms para as mitocôndrias musculares e 50 ms para as mitocôndrias hipodérmicas. O LED de 475 nm foi ajustado para 50% da potência e o filtro de densidade neutra foi ajustado para 30% para o músculo e 17% para as mitocôndrias hipodérmicas. A faixa Z começa de onde o sinal GFP é visível até onde termina. Para minimizar o ruído em uma projeção máxima, pode-se diminuir o número de planos z usados para a projeção máxima. - Para imagens de mitocôndrias intestinais (vha-6p::MLS::GFP), use um microscópio confocal.
NOTA: A imagem foi realizada em um microscópio confocal disponível comercialmente equipado com uma objetiva Plan ApoChromat 63x/1,4, laser de luz branca (WLL), divisor de feixe acústico-óptico, detectores HyD S e executado em software de microscópio. Como mencionado anteriormente, deve-se otimizar cada configuração experimental individual, mas como ponto de partida, os parâmetros para imagens com o Stellaris usado neste estudo são os seguintes: O WLL foi ajustado para 85,00% de potência máxima e com uma linha de laser de 485 nm a uma intensidade de 3,00%. O detector HyD S foi ajustado para 490-590 nm com um ganho de 25 na configuração analógica. Fizemos a varredura unidirecional de uma área de 1024 (x) * 1024 (y) ou 82,01 μm (x) * 82,01 μm (y) com 5 (z) pixels em um tamanho de passo de 0,495 μm (a dimensão z é variável dependendo do tamanho dos vermes ou tecidos) com uma velocidade de varredura de 1.000 Hz, zoom de 2,25, média de linha de 2 e orifício de 1 UA (95,5 μm). A faixa Z começa de onde o sinal GFP é visível até onde termina. Para minimizar o ruído em uma projeção máxima, pode-se diminuir o número de planos z usados para a projeção máxima.
- Use um microscópio de campo amplo padrão para mitocôndrias musculares e hipodérmicas (myo-3p::MLS::GFP e col-19p::MLS::GFP).
- Quantificação da morfologia mitocondrial
NOTA: Para obter detalhes, consulte Schindelin et al.22 e consulte a Figura Suplementar 1A-D.- Baixe e instale o software FIJI (Fiji é apenas ImageJ, https://fiji.sc/). Em seguida, abra FIJI.
- Para baixar e instalar a macro MitoMAPR, baixe o código-fonte 1 do https://doi.org/10.7554/eLife.49158.033. Em seguida, copie todo o código mencionado em "A.). Código IJM para MitoMAPR-1.0." Em seguida, abra Fiji, vá para Plugins > New > Macro e cole o código copiado anteriormente na janela de macro. Vá para Arquivo > opção Salvar como e salve a macro para uso futuro.
- Abra uma imagem microscópica 3D com pilhas Z em FIJI arrastando e soltando arquivos na barra de ferramentas Fiji ou acessando Arquivo > Abrir.
- Crie uma projeção máxima da imagem acessando Pilhas de > de imagem > Projeto Z e, em seguida, selecionando o intervalo de fatias com imagens em foco a serem projetadas no máximo e selecionando Intensidade máxima como o tipo de projeção.
- Salve a imagem como um TIFF acessando Arquivo > Salvar como > Tiff.
- Cortar regiões de interesse (ROIs) de imagens completas usando a ferramenta "Retângulo", desenhando um ROI e, em seguida, indo para Imagem > Corte.
- Salve a imagem como um TIFF acessando Arquivo > Salvar como > Tiff.
- Execute a macro MitoMAPR salva anteriormente arrastando e soltando o arquivo de macro na barra de ferramentas Fiji e clique em Executar. Uma nova janela solicitará a seleção da pasta onde o arquivo Tiff foi salvo na versão 2.3.6.
- Uma nova janela solicitará a seleção de uma área; crie um retângulo na imagem usando a ferramenta retângulo conforme mencionado anteriormente na etapa 2.3.4 e pressione Ok.
NOTA: A imagem inteira pode ser selecionada se tiver sido cortada anteriormente, conforme descrito na etapa 2.3.6. Salve a janela "Dados" com todos os valores relacionados à morfologia mitocondrial como um arquivo Excel acessando Arquivo > Salvar como > Salvar.
- Processamento em lote de amostras usando macros FIJI
- Crie uma macro para especificar a região das imagens a serem analisadas. Abra Fiji e vá para Plugins > Nova > Macro. Cole o código a seguir na caixa de texto e salve a macro conforme descrito em 2.3.2 (Figura Suplementar 1E-H).
NOTA: Faça retângulo (100, 100, 200, 200); run (Especificar..., largura = 100, altura = 100, x = 100, y = 100 escalado). - Crie uma macro para salvar todas as imagens abertas como arquivos TIFF. Na janela de macro, vá para Arquivo > Novo e cole o código a seguir na caixa de texto. Salve a macro conforme descrito em 2.3.2.
NOTA: dir = getDirectory("Selecione uma pasta"); ids=newArray(nImagens); para (i=0; i - Crie uma macro para o lote MitoMAPR. Vá para https://doi.org/10.7554/eLife.49158.033 código-fonte 1 e copie todos os códigos mencionados em B.). Código IJM para MitoMAPR-1.0_Batch". Na janela de macro, vá para Arquivo > Novo, cole o código do código-fonte 1 na caixa de texto e salve a macro conforme descrito na etapa 2.3.2.
- Crie uma macro para corte iterativo. Vá para https://doi.org/10.7554/eLife.49158.033 código-fonte 1 e copie todos os códigos mencionados em C.) Código IJM para CropR". Na janela de macro, vá para Arquivo > Novo, cole o código do código-fonte 1 na caixa de texto e salve a macro conforme descrito na etapa 2.3.2.
- Para iniciar o processamento em lote, primeiro salve todas as imagens a serem processadas como TIFF abrindo todas as imagens e executando a macro salva na etapa 2.4.2. Todas as imagens serão salvas como TIFF na pasta especificada. Feche todas as imagens depois de salvas.
- Se as imagens salvas na etapa 2.4.5 forem imagens 3D (imagens contendo pilhas z), converta-as em imagens 2D tornando-as projeções Z. Para projetar as imagens em lote em Z, abra a janela de processamento em lote acessando Processar > Macro de > em lote na janela principal do FIJI.
- Para "Entrada", selecione o local da pasta que contém todas as imagens 3D a serem processadas a partir de 2.4.5. Para "Saída", selecione o local desejado para as imagens salvas após o processamento. Selecione o formato de saída como TIFF. Cole o seguinte código na caixa de texto grande na janela e pressione o botão de processo : run("Z Project...", "projection=[Max Intensity]").
- Cortar ROIs com dimensões idênticas de todas as imagens 2D a serem processadas usando a macro de corte iterativa da etapa 2.4.4 em combinação com a macro de região de especificação da etapa 2.4.3. Execute a macro de corte iterativa da etapa 2.4.4. Uma janela aparecerá solicitando um diretório.
- Selecione a pasta que contém apenas imagens 2D da etapa 2.4.5 ou as imagens 2D projetadas em Z da etapa 2.4.6 e pressione o botão Selecionar . A macro abrirá uma das imagens na pasta selecionada e uma janela chamada "fazer uma seleção" com dois botões aparecerá.
- Execute a macro de seleção especificada na etapa 2.4.3. Modifique os valores de largura e altura nessa caixa de texto de macro conforme necessário para alterar a dimensão de seleção e pressione Executar para observar a nova seleção. Alterar os valores X e Y na macro modificará a posição do canto superior esquerdo da seleção.
- Quando as dimensões da seleção forem satisfatórias, arraste o retângulo para a área desejada e pressione o botão OK na janela "fazer uma seleção". A região cortada será salva na pasta selecionada na etapa 2.4.7.
- A macro irá iterar por todas as imagens restantes na pasta. Para cada imagem, execute a macro da etapa 2.4.8 sem alterar os valores de dimensão para garantir que as mesmas dimensões sejam cortadas para todas as imagens. Para cada imagem, arraste o retângulo de seleção para o local desejado e pressione o botão OK na janela "fazer uma seleção".
- Abra a pasta que contém as imagens cortadas das etapas 2.4.8 a 2.4.9 e mova todas as imagens cortadas para uma nova pasta.
- Analise todas as imagens cortadas executando a macro MitoMAPR em lote da etapa 2.4.3. Uma nova janela solicitará que você selecione um diretório. Selecione a pasta com todas as imagens cortadas da etapa 2.4.10. Depois de concluído, uma janela chamada "Dados" aparecerá com todos os valores relacionados à morfologia mitocondrial. Salve todos os dados como um arquivo do Excel, conforme descrito na etapa 2.3.9.
- Crie uma macro para especificar a região das imagens a serem analisadas. Abra Fiji e vá para Plugins > Nova > Macro. Cole o código a seguir na caixa de texto e salve a macro conforme descrito em 2.3.2 (Figura Suplementar 1E-H).
Resultados
C. elegans é um ótimo modelo para imagens mitocondriais devido ao seu corpo transparente, que permite imagens fáceis de animais vivos sem preparação excessiva de amostras. Além disso, a morfologia mitocondrial em diferentes tecidos pode ser facilmente visualizada usando promotores específicos do tecido para expressar proteínas fluorescentes direcionadas às mitocôndrias. Aqui, myo-3p (para mitocôndrias musculares), vha-6p (para mitocôndrias intestinais) e col-19p (para mitocôndrias hipodérmicas) foram utilizados para impulsionar a expressão de uma GFP direcionada às mitocôndrias (sequência de localização mitocondrial da proteína ATP-1). As mitocôndrias dos músculos da parede corporal de C. elegans exibem morfologia tubular, alinhando-se ao longo das fibras musculares (miofibrilas) (Figura 1A); as mitocôndrias intestinais apresentaram estruturas altamente interconectadas, semelhantes a teias, com alinhamento menos uniforme (Figura 1B); e a hipoderme possui mitocôndrias tubulares que parecem mais arredondadas ou ovais em comparação com o intestino ou músculo (Figura 1C). A morfologia mitocondrial é geralmente consistente em todo o comprimento do verme no músculo e no intestino, mas a hipoderme exibe algumas pequenas diferenças na interconexão das mitocôndrias entre as extremidades proximal e distal do verme. Assim, para obter resultados experimentais reprodutíveis, recomenda-se focar em uma região específica de seus corpos. Aqui, a área entre a faringe e a vulva ~ 100-200 μm abaixo da faringe é consistentemente visualizada.
Para mitocôndrias musculares e hipodérmicas, a imagem sob um microscópio composto fornece resolução suficiente devido ao achatamento das células e aos baixos níveis de luz fora de foco. No entanto, as mitocôndrias intestinais são difíceis de observar com um microscópio composto, pois o grande volume do intestino limita a resolução devido a uma grande quantidade de luz fora de foco de outras seções do intestino (Figura 2). Portanto, recomenda-se obter imagens das mitocôndrias intestinais sob um microscópio confocal para reduzir a luz fora de foco e visualizar a morfologia mitocondrial adequada.
A forma das mitocôndrias é altamente dinâmica e muda com base no ambiente metabólico dos animais23 ou mesmo devido à exposição a rigidez variável do substrato24. Portanto, deixar os animais em lâminas de microscópio por longos períodos de tempo na ausência de uma fonte de alimento e no substrato rígido do vidro pode afetar potencialmente a morfologia mitocondrial. Aqui, este estudo descobriu que as mitocôndrias dos vermes se fragmentam após aproximadamente 30 minutos em uma lâmina em solução M9, com as mitocôndrias hipodérmicas exibindo a maior fragmentação (Figura 3). Portanto, a imagem das mitocôndrias deve ser realizada rapidamente após a preparação da amostra.
Produtos químicos que restringem a mobilidade do verme, incluindo azida de sódio e tetramisol, são comumente usados para imagens ao vivo de C. elegans, já que vermes estacionários são necessários para capturar várias seções z de animais para imagens 3D. Estudos anteriores mostraram que a exposição à azida sódica ou tetramisol pode resultar em fragmentação mitocondrial25. Surpreendentemente, verificou-se que a azida de sódio - mesmo em altas concentrações - teve um impacto limitado na morfologia mitocondrial no músculo ou intestino. No entanto, a hipoderme exibiu fragmentação mitocondrial mais precoce do que os controles M9 (Figura 3). É importante ressaltar que altas concentrações de tetramisol (100 mM) resultaram em fragmentação mitocondrial significativa imediatamente após a exposição em todos os tipos de células. Em comparação, concentrações médias (10 mM) resultaram em fragmentação mais rápida em comparação com um controle M9. Baixas concentrações (1 mM) tiveram um impacto limitado na morfologia mitocondrial. Esses dados sugerem que a utilização de azida sódica pode ser uma opção viável para imagens rápidas de mitocôndrias, enquanto o tetramisol geralmente deve ser evitado.
Como esperado, as mitocôndrias de todos os tecidos de C. elegans exibem fragmentação durante o processo natural de envelhecimento (Figura 4A). A fragmentação pode ser visualizada como mitocôndrias apresentando-se como estruturas mais truncadas e esféricas, que são dramaticamente diferentes das mitocôndrias tubulares lineares exibidas em animais jovens. É importante notar que o intestino aumenta em estruturas autofluorescentes esféricas em uma idade posterior, portanto, deve-se tomar cuidado para não confundir autofluorescência com estruturas mitocondriais reais. Como pode haver variabilidade de mudanças na estrutura mitocondrial entre os vermes, é importante realizar a quantificação da morfologia mitocondrial em um tamanho de amostra significativo, em vez de apenas obter imagens de alguns vermes. Aqui, foi usado o mitoMAPR, que permite a quantificação automatizada da morfologia mitocondrial usando um conjunto diversificado de métricas, incluindo objetos, redes, junções por rede, pontos de junção, comprimento do objeto, pegada mitocondrial, cobertura mitocondrial e área do objeto (Tabela Suplementar 1 e Tabela Suplementar 2). A automação remove o viés subjetivo do usuário. Aqui, relatamos que a métrica de comprimento do objeto é ideal para medir quantitativamente as mudanças na morfologia mitocondrial muscular e intestinal durante o envelhecimento, e métricas de pontos de junção para medir as mudanças na morfologia mitocondrial hipodérmica durante o envelhecimento (Figura 4B).
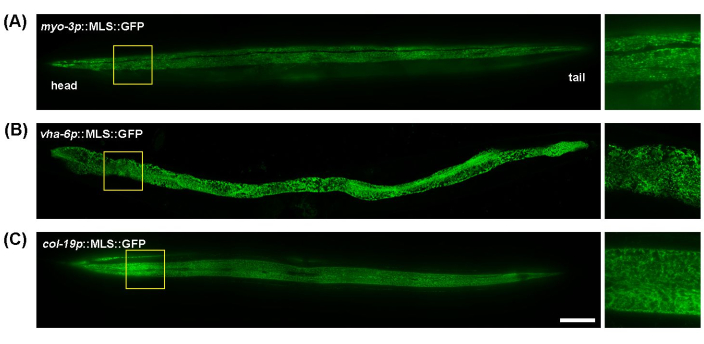
Figura 1: Imagens de mitocôndrias em todo o corpo de C. elegans. A imagem mitocondrial foi realizada durante toda a duração do dia 5 de animais adultos cultivados em EV a partir do estágio L1 em animais transgênicos com expressão de MLS::GFP no músculo (A), intestino (B) e hipoderme (C). Barra de escala: 100 μm. Clique aqui para ver uma versão maior desta figura.

Figura 2: Comparação da imagem mitocondrial entre microscópio composto e microscópio confocal. A imagem mitocondrial foi realizada em animais adultos de dia 1 cultivados em EV a partir do estágio L1 em animais transgênicos com expressão de MLS::GFP no músculo (A), intestino (B) e hipoderme (C). Barra de escala: 5 μm. Clique aqui para ver uma versão maior desta figura.

Figura 3: Avaliação da fragmentação mitocondrial de diferentes tecidos após tratamento com M9, azida sódica e tetramisol. A imagem mitocondrial foi realizada em animais adultos do dia 1 com expressão de MLS::GFP no músculo, intestino e hipoderme. Os animais foram criados em EV a partir do estágio L1. Os animais foram colocados em lâminas contendo M9, azida sódica (1 mM, 10 mM e 100 mM) ou tetramisol (1 mM, 10 mM e 100 mM) e a imagem foi realizada imediatamente após o preparo da lâmina (0 min) ou 15 min ou 30 min após o preparo da lâmina. Imagens representativas são mostradas para n > 5 animais por cepa para 2 réplicas biológicas. Barras de escala: 5 μm. Clique aqui para ver uma versão maior desta figura.

Figura 4: Imagem mitocondrial de C. elegans durante o envelhecimento em diferentes tecidos e quantificação da morfologia mitocondrial usando MitoMAPR. (A) A imagem mitocondrial foi realizada em animais adultos nos dias 1, 5, 9, 13 e 15 da idade adulta com expressão de MLS::GFP no músculo, intestino e hipoderme. Os animais foram criados em EV a partir do estágio L1 e movidos para placas EV contendo FUDR a partir do estágio adulto do dia 1. As imagens são representativas de n ≥ 5 animais por cepa para ≥ 3 repetições biológicas. Barra de escala: 5 μm. (B) A quantificação do comprimento do objeto das mitocôndrias musculares e intestinais de vermes nos dias 1, 5, 9, 13 e 15, e a quantificação dos pontos de junção das mitocôndrias hipodérmicas de vermes nos dias 1, 5, 9, 13 e 15. Todos os pontos de dados individuais combinados com a média ± DP. Os gráficos foram plotados e analisados estatisticamente usando um teste t de Student. ns = não significativo, *p < 0,03; **p < 0,002; p < 0,0002; p < 0,0001. Clique aqui para ver uma versão maior desta figura.
| Dias de vermes | Número de vermes | Volume do tampão (mL) |
| 1 | 25 | 13 |
| 5 | 25 | 14 |
| 9 | 20 | 15 |
| 13 | 20 | 15 |
Tabela 1: Números recomendados de minhocas e volumes de buffer ao preparar lâminas.
Figura suplementar 1: Fluxos de trabalho da aplicação de uma única quantificação de imagem usando MitoMAPR e processamento em lote para várias imagens. (A) Fluxo de trabalho de quantificação da morfologia mitocondrial a partir de uma única imagem usando a macro MitoMAPR. (B) Projeção Z de arquivo de imagem 3D bruta usando Fiji (projeção máxima). (C) Recortar a região de interesse da imagem Z projetada. (D) Imagens do esqueleto adquiridas da macro MitoMAPR. Barras de escala: 5 μm. (E) Fluxo de trabalho de quantificação da morfologia mitocondrial de várias imagens usando processo em lote usando macro MitoMAPR. (F) Captura de tela da macro de corte. (G) Captura de tela de um processo em lote para projeção Z. (H) Captura de tela da janela de confirmação de seleção da macro de corte em lote. Clique aqui para baixar este arquivo.
Tabela Suplementar 1: Análise quantitativa das métricas de morfologia mitocondrial no tecido muscular de C. elegans . As métricas registradas incluem objetos, redes, junções por rede, pontos de junção, comprimento do objeto, pegada mitocondrial, área do objeto e cobertura mitocondrial, medida em dias diferentes. Os dados fornecem uma base para avaliar as mudanças na morfologia mitocondrial com o envelhecimento. Clique aqui para baixar este arquivo.
Tabela suplementar 2: Efeitos de produtos químicos imobilizadores na morfologia mitocondrial em C. elegans. Os dados incluem medições como objetos, redes, junções por rede, pontos de junção, comprimento do objeto, pegada mitocondrial, área do objeto e cobertura mitocondrial em concentrações e pontos de tempo variados após a preparação da amostra com tampão M9 ou diferentes concentrações de tetramisol e azida de sódio. Clique aqui para baixar este arquivo.
Discussão
A imagem fluorescente da morfologia mitocondrial é a maneira mais comum de determinar alterações nas mitocôndrias. Embora as técnicas avançadas de microscopia, como microscopia eletrônica de transmissão (TEM), microscopia de força atômica e microscopia crioeletrônica, ofereçam maior resolução, a microscopia de fluorescência permanece mais acessível e acessível para a maioria dos pesquisadores. Além disso, a microscopia fluorescente pode ser realizada em células vivas e, em organismos modelo claros como C. elegans, a imagem pode ser realizada em animais inteiros26,27. A imagem de C. elegans transgênico é bastante simples, e os marcadores fluorescentes geneticamente codificados permitem imagens mais confiáveis e robustas das mitocôndrias, uma vez que não requerem processamento complexo de amostras ou sofrem coloração variável entre tipos de células ou condições de corantes mitocondriais convencionais como MitoTracker ou TMRE 28,29,30. Os marcadores fluorescentes geneticamente codificados geralmente envolvem a marcação direta de proteínas mitocondriais com fluoróforos ou conjugação de fluoróforos com uma sequência mínima de localização mitocondrial. Essas construções são frequentemente conduzidas por promotores específicos do tecido, permitindo a visualização das mitocôndrias em diferentes tecidos, como músculos, intestino ou tecidos da hipoderme31. Geralmente, essas proteínas marcadas com fluorescência são superexpressas, o que pode ter efeitos fisiológicos potencialmente indesejados se as proteínas mitocondriais de comprimento total forem superexpressas; assim, sequências mínimas de MLS são uma opção melhor32. No entanto, mesmo com a superexpressão de fusões mínimas de proteínas fluorescentes MLS, a alta superexpressão deve ser evitada, pois a importação de uma grande quantidade de proteína para as mitocôndrias pode colapsar o potencial da membrana mitocondrial e impactar a saúde animal33. Embora uma caracterização exaustiva de todas as cepas de C. elegans atualmente disponíveis esteja além do escopo deste manuscrito, uma análise comparativa detalhada de vários repórteres mitocondriais e os prós e contras de cada um pode ser encontrada aqui31.
Para imagens de células vivas de mitocôndrias em C. elegans, a microscopia composta padrão ou de campo amplo pode ser uma opção preferida devido à alta velocidade e facilidade desses métodos em relação à microscopia confocal. Neste estudo, mostra-se que células planas como o músculo e a hipoderme se beneficiam minimamente da microscopia confocal, e a microscopia composta permite a aquisição com resolução suficiente para visualizar a morfologia mitocondrial adequada. Células maiores, como o intestino, tornam a microscopia composta desafiadora devido à luz fora de foco. Portanto, a microscopia confocal é necessária para imagens confiáveis da morfologia mitocondrial intestinal.
Uma consideração importante para imagens 3D em toda a espessura do tecido em animais vivos é evitar o movimento dos vermes durante a aquisição da imagem. Os pesquisadores costumam usar métodos para paralisar vermes, como tetramisol ou azida sódica34. A azida sódica inibe a citocromo c oxidase (complexo IV), uma enzima crítica na cadeia de transporte de elétrons das mitocôndrias, levando à paralisia geral devido à falta de ATP necessário para contrações musculares e outras funções celulares 34,35. O tetramisol age mimetizando a acetilcolina nas junções neuromusculares, causando despolarização e contração muscular persistentes36. No entanto, a exposição a essas drogas pode induzir a fragmentação mitocondrial, causando estresse oxidativo23. Neste estudo, verificou-se que o tetramisol induziu muito rapidamente a fragmentação mitocondrial, mas a azida sódica teve um efeito muito mais limitado.
C. elegans oferece uma maneira muito simples e fácil de estudar o impacto do envelhecimento na morfologia mitocondrial devido à sua curta vida útil e facilidade de envelhecimento dos animais. Aqui, optamos por usar a exposição ao FUDR, que é um método robusto para esterilizar quimicamente os animais, impedindo a replicação do DNA 20,37,38. No entanto, o FUDR pode ter efeitos indesejados em parâmetros específicos de envelhecimento e, para aqueles preocupados com os efeitos fora do alvo do FUDR, outras estratégias para remover a progênie podem ser utilizadas39. Por exemplo, existem mutantes estéreis, incluindo o mutante da linha germinativa sensível à temperatura glp-4, ou mutantes deficientes em esperma, como CF512 40,41,42. Como alternativa, os animais também podem ser envelhecidos naturalmente, escolhendo manualmente os adultos de sua progênie diariamente.
A quantificação das mudanças na morfologia mitocondrial também é uma consideração muito importante, pois pode haver variabilidade significativa na morfologia mitocondrial entre os animais. Portanto, a realização de análises quantitativas e estatísticas em um tamanho de amostra suficiente é essencial para tirar conclusões significativas sobre as mudanças na morfologia mitocondrial. No entanto, a análise e quantificação de imagens podem sofrer muito com vieses subjetivos e desafios de imagens de estruturas altamente complexas, como mitocôndrias interconectadas. Para esse fim, aqui está um resumo de um método automatizado para quantificar a morfologia mitocondrial usando várias métricas desenvolvidas pelo laboratório Mair. O MitoMAPR permite a quantificação objetiva e automatizada da morfologia mitocondrial, medindo vários aspectos da mitocôndria, incluindo a rede mitocondrial, comprimento do objeto, distribuição, cobertura da rede e pegada mitocondrial. O MitoMAPR é uma macro gratuita para o ImageJ e, portanto, está disponível para todos os laboratórios com um computador funcional. Um aspecto importante do uso do MitoMAPR é realizar a quantificação em um grande tamanho de amostra para determinar qual métrica da morfologia mitocondrial é mais robusta para determinar mudanças nas condições experimentais que estão sendo testadas43,44. Aqui, verifica-se que o comprimento do objeto e os pontos de junção são as melhores métricas para determinar as mudanças durante o envelhecimento no músculo, intestino e hipoderme. Uma abordagem alternativa para analisar mudanças na morfologia mitocondrial é a geração de representações 3D de mitocôndrias a partir de imagens z-stack, seguidas de análise 3D16,45. Isso pode ser alcançado usando software disponível comercialmente, como o Image-Pro Plus com os módulos SharpStack Total Deconvolution e 3D Constructor. A quantificação de representações mitocondriais 3D demonstrou fornecer informações mais precisas sobre a forma mitocondrial e as propriedades da rede46. No entanto, esse método é tecnicamente mais complexo e caro em comparação com a abordagem semi-3D, que envolve o recolhimento de várias seções de imagem de uma pilha z de campo amplo ou confocal em uma única projeção 2D e analisá-las com ferramentas como o mitoMAPR. Este estudo resume o uso da expressão de cópia única de GFP direcionada à matriz mitocondrial e detalha algumas armadilhas a serem evitadas durante a imagem.
Limitações e considerações de tempo
Embora a microscopia confocal seja recomendada para tecidos grandes e espessos, como tecido intestinal, as técnicas padrão de microscopia confocal de varredura de linha podem ser muito lentas para realizar imagens rápidas das mitocôndrias. Isso é especialmente verdadeiro considerando nossos dados que mostram que manter vermes em lâminas por longos períodos de tempo pode resultar na fragmentação das mitocôndrias. Embora a tecnologia de microscopia confocal tenha avançado significativamente, os microscópios que podem realizar imagens rápidas, como disco giratório, Airyscan ou o usado neste estudo, podem ter um custo proibitivo para alguns laboratórios. Nesses casos, a microscopia composta pode ser combinada com métodos de liberação computacional para remover a luz fora de foco, como a deconvolução47.
Além disso, conforme descrito, o MitoMAPR é uma poderosa macro automatizada para medir quantitativamente o comprimento das mitocôndrias e a interconexão da rede mitocondrial. No entanto, ele deve ser usado com cuidado, considerando as limitações listadas aqui. Primeiro, o uso de uma GFP direcionada à matriz observa apenas a fragmentação mitocondrial da membrana interna, o que pode não recapitular totalmente a morfologia mitocondrial da membrana externa, pois os eventos de fissão da membrana interna podem ocorrer na ausência de fissão da membrana externa. Assim, para imagens mais refinadas de ambas as membranas, deve-se usar um fluoróforo direcionado à matriz e à membrana externa mitocondrial. Como os marcadores da membrana mitocondrial externa podem sofrer as mesmas consequências que as proteínas localizadas na matriz se altamente superexpressos, recomenda-se o uso de marcadores de cópia única, especialmente aqueles que usam sequências mínimas de localização mitocondrial em vez de proteínas completas, como as usadas aqui31.
Conforme descrito na Figura 3, as mitocôndrias podem se fragmentar rapidamente ao microscópio, e diferentes métodos para preparar lâminas de amostra podem afetar amplamente a morfologia, dependendo do tampão que está sendo usado. Os dados representativos descritos neste manuscrito mostram que as mitocôndrias dos vermes sofrem fragmentação mínima sob M9 e baixas concentrações de azida sódica. O tetramisol, outro produto químico amplamente utilizado para paralisar vermes, fragmenta rapidamente as mitocôndrias, indicando que seu uso deve ser evitado em geral. Embora M9 e azida sódica não tenham mostrado fragmentação significativa das mitocôndrias, é importante notar que diferentes cepas podem responder de maneira diferente dos resultados mostrados aqui. Embora outros estudos tenham mostrado que a azida sódica pode fragmentar as mitocôndrias, é possível que nossas cepas não apresentem fragmentação mitocondrial significativa com a exposição à azida sódica devido ao baixo nível de expressão de nossas construções. A alta expressão de proteínas localizadas nas mitocôndrias pode colapsar o potencial de membrana e, portanto, diferentes cepas usadas em outros estudos podem tornar as mitocôndrias mais suscetíveis à fragmentação das mesmas concentrações de azida sódica usadas neste estudo. Independentemente disso, deve-se tomar cuidado para garantir que os produtos químicos que paralisam os vermes não causem fragmentação mitocondrial em condições específicas a serem testadas antes do uso em todos os estudos, pois mutantes ou condições específicas podem ser mais suscetíveis à fragmentação mitocondrial induzida por drogas. Além disso, mesmo manter os vermes em M9 pode resultar em mudanças em sua morfologia e atividade mitocondrial natural, uma vez que sua atividade de natação no tampão M9 já demonstrou afetar a fissão mitocondrial e a dinâmica de fusão. Além disso, deixar vermes em M9 por longos períodos de tempo pode ativar respostas de hipóxia que afetam significativamente as mitocôndrias, causando interrupções da proteostase mitocondrial48,49.
Finalmente, outra consideração importante é que, embora as mudanças na morfologia mitocondrial sejam frequentemente correlacionadas com mudanças na função mitocondrial, nem sempre há uma correlação direta entre as duas. Assim, recomenda-se uma análise mais completa da função mitocondrial. Por exemplo, a taxa de consumo de oxigênio pode ser medida usando um instrumento Seahorse50, o potencial de membrana mitocondrial pode ser medido usando corantes de potencial de membrana, como JC9 ou TMRE51, o estado oxidativo mitocondrial pode ser medido usando corantes sensíveis a redox, como roGFP52, e a resiliência ao estresse mitocondrial pode ser medida usando sensibilidade a estressores, como rotenona53. Como a imagem das mitocôndrias pode ser bastante rápida, oferecemos os métodos aqui como uma primeira passagem fácil para determinar se as condições experimentais afetam a morfologia mitocondrial. Esses métodos são passíveis até mesmo para triagem em larga escala de medicamentos ou genes, com acompanhamento de análises mitocondriais mais completas usando métricas adicionais recomendadas. Ao todo, revisamos aqui o que se acredita serem os métodos mais simples para obter imagens da morfologia mitocondrial em C. elegans com erros experimentais mínimos.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
JK é apoiado pela USC Provost Fellowship; MA e GG são apoiados por T32AG052374; MV é suportado por 1R25AG076400; e R.H.S. é apoiado por R01AG079806 do Instituto Nacional do Envelhecimento e 2022-A-010-SUP da Fundação Larry L. Hillblom. Algumas cepas foram fornecidas pelo CGC, que é financiado pelo Escritório de Programas de Infraestrutura de Pesquisa do NIH conceder P40 OD010440. Algumas análises genéticas foram realizadas usando o Wormbase, que é financiado por uma bolsa U41 HG002223.
Materiais
| Name | Company | Catalog Number | Comments |
| 5-fluoro-2'-deoxyuridine (FUDR) | Spectrum Chemical | F2026-10GMBL | for proliferation inhibition |
| APEX IPTG | Genesee | 18-242 | for RNAi |
| Bacto Agar | VWR | 90000-764 | for NGM plates |
| Bacto Peptone | VWR | 97064-330 | for NGM plates |
| Calcium chloride dihydrate | VWR | 97061-904 | for NGM plates |
| Carbenicillin | VWR | 76345-522 | for RNAi |
| Cholesterol | VWR | 80057-932 | for NGM plates |
| Confocal microscope | Stellaris 5 | ||
| DMSO | VWR | BDH1115-1LP | solvent for drugs |
| HistoBond microscope slides | VWR | 16005-110 | for slides preparation |
| LB Broth | VWR | 95020-778 | for LB |
| LEICA S7E Dissecting Scope | Leica | 10450840 | Standard dissecting microscope |
| LEICA STELLARIS 5 | Leica | 158101100 | Confocal Microscope |
| LEICA THUNDER | Leica | 11525679 | Compound Microscope |
| Magnesium sulfate heptahydrate | VWR | 97062-132 | for NGM plates, M9 |
| pL4440 empty vector plasmid | addgene | plasmid #1654 | for empty vector plasmid |
| Potassium Chloride | VWR | 97061-566 | for bleach soluton |
| Potassium phosphate dibasic | VWR | EM-PX1570-2 | for NGM plates |
| Potassium phosphate monobasic | VWR | EM-PX1565-5 | for M9 |
| Sodium azide | VWR | 97064-646 | for paralyzing worms |
| Sodium Chloride | VWR | EM-SX0420-5 | for NGM plates, M9 |
| Sodium hypochlorite | VWR | RC7495.7-32 | for bleach solution |
| Sodium phosphate dibasic | VWR | 71003-472 | for M9 |
| Tetracycline hydrochloride | VWR | 97061-638 | for RNAi |
| WOB-L® Dry Vacuum Pumps, Standard-Duty, Welch® | VWR | 80077-612 | for aspiration |
Referências
- Protasoni, M., Zeviani, M. Mitochondrial structure and bioenergetics in normal and disease conditions. Int J Mol Sci. 22 (2), 586 (2021).
- Wang, Y., et al. The role of mitochondrial dynamics in disease. MedComm. 4 (6), e462 (2023).
- Kamer, K. J., Mootha, V. K. The molecular era of the mitochondrial calcium uniporter. Nat Rev Mol Cell Biol. 16 (9), 545-553 (2015).
- Chen, W., Zhao, H., Li, Y. Mitochondrial dynamics in health and disease: mechanisms and potential targets. Signal Transduct Target Ther. 8 (1), 333 (2023).
- Adebayo, M., Singh, S., Singh, A. P., Dasgupta, S. Mitochondrial fusion and fission: The fine-tune balance for cellular homeostasis. FASEB J. 35 (6), e21620 (2021).
- Madan, S., Uttekar, B., Chowdhary, S., Rikhy, R. Mitochondria lead the way: mitochondrial dynamics and function in cellular movements in development and disease. Front Cell Dev Biol. 9, 781933 (2021).
- Sharma, A., Smith, H. J., Yao, P., Mair, W. B. Causal roles of mitochondrial dynamics in longevity and healthy aging. EMBO Rep. 20 (12), e48395 (2019).
- Rolland, S. G., Lu, Y., David, C. N., Conradt, B. The BCL-2-like protein CED-9 of C. elegans promotes FZO-1/Mfn1,2- and EAT-3/Opa1-dependent mitochondrial fusion. J Cell Biol. 186 (4), 525-540 (2009).
- Labrousse, A. M., Zappaterra, M. D., Rube, D. A., van der Bliek, A. M. C. elegans dynamin-related protein DRP-1 controls severing of the mitochondrial outer membrane. Mol Cell. 4 (5), 815-826 (1999).
- Dickinson, D. J., Pani, A. M., Heppert, J. K., Higgins, C. D., Goldstein, B. Streamlined genome engineering with a self-excising drug selection cassette. Genetics. 200 (4), 1035-1049 (2015).
- Bosher, J. M., Labouesse, M. RNA interference: Genetic wand and genetic watchdog. Nat Cell Biol. 2 (2), E31-E36 (2000).
- Yu, C. C. J., et al. Expansion microscopy of C. elegans. eLife. 9, e46249 (2020).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Front Endocrinol. 11, 554994 (2020).
- Dingley, S., et al. Mitochondrial respiratory chain dysfunction variably increases oxidant stress in Caenorhabditis elegans. Mitochondrion. 10 (2), 125-136 (2010).
- Presley, A. D., Fuller, K. M., Arriaga, E. A. MitoTracker Green labeling of mitochondrial proteins and their subsequent analysis by capillary electrophoresis with laser-induced fluorescence detection. J Chromatogr B Anal Technol Biomed Life Sci. 793 (1), 141-150 (2003).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Curr Protoc Cell Biol. Chapter 4, Unit 4.25.1-Unit 4.25.21 (2010).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. Novel imaging tools to study mitochondrial morphology in Caenorhabditis elegans. Life Sci Alliance. 7 (11), e202402918 (2024).
- Benedetti, C., Haynes, C. M., Yang, Y., Harding, H. P., Ron, D. Ubiquitin-like protein 5 positively regulates chaperone gene expression in the mitochondrial unfolded protein response. Genetics. 174 (1), 229-239 (2006).
- Begelman, D. V., et al. An aco-2::gfp knock-in enables the monitoring of mitochondrial morphology throughout C. elegans lifespan. microPublication Biol. 2022, (2022).
- Castro Torres, T., et al. Surveying low-cost methods to measure lifespan and healthspan in Caenorhabditis elegans. J Vis Exp. 183, e64091 (2022).
- Rasmussen, N. R., Reiner, D. J. Nuclear translocation of the tagged endogenous MAPK MPK-1 denotes a subset of activation events in C. elegans development. J Cell Sci. 134 (17), jcs258456 (2021).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Luz, A. L., et al. Mitochondrial morphology and fundamental parameters of the mitochondrial respiratory chain are altered in Caenorhabditis elegans strains deficient in mitochondrial dynamics and homeostasis processes. PLoS One. 10 (6), e0130940 (2015).
- Oorloff, M., et al. Mechanical stress through growth on stiffer substrates impacts animal health and longevity in C. elegans. bioRxiv. , (2024).
- Chu, X., Wu, S., Raju, R. NLRX1 regulation following acute mitochondrial injury. Front Immunol. 10, 2431 (2019).
- Shah, P., Bao, Z., Zaidel-Bar, R. Visualizing and quantifying molecular and cellular processes in Caenorhabditis elegans using light microscopy. Genetics. 221 (4), iyac068 (2022).
- Wang, Y., Wang, P., Li, C. Fluorescence microscopic platforms imaging mitochondrial abnormalities in neurodegenerative diseases. Adv Drug Deliv Rev. 197, 114841 (2023).
- Ding, J., et al. An expanded GCaMP reporter toolkit for functional imaging in Caenorhabditis elegans. G3 (Bethesda, Md). 13 (10), jkad183 (2023).
- Sarasija, S., Norman, K. R. Analysis of mitochondrial structure in the body wall muscle of Caenorhabditis elegans. Bio-Protocol. 8 (7), e2801 (2018).
- Chaweeborisuit, P., Suriyonplengsaeng, C., Suphamungmee, W., Sobhon, P., Meemon, K. Nematicidal effect of plumbagin on Caenorhabditis elegans: A model for testing a nematicidal drug. Z Naturforsch C J Biosci. 71 (5-6), 121-131 (2016).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. . Novel imaging tools to study mitochondrial dynamics in Caenorhabditis elegans. 7, 11 (2024).
- Bolognesi, B., Lehner, B. Reaching the limit. eLife. 7, e39804 (2018).
- Jishi, A., Qi, X. Altered mitochondrial protein homeostasis and proteinopathies. Front Mol Neurosci. 15, 867935 (2022).
- Morton, K. S., Wahl, A. K., Meyer, J. N. The effect of common paralytic agents used for fluorescence imaging on redox tone and ATP levels in Caenorhabditis elegans. PLoS One. 19 (4), e0292415 (2024).
- Noumi, T., Maeda, M., Futai, M. Mode of inhibition of sodium azide on H+-ATPase of Escherichia coli. FEBS Lett. 213 (2), 381-384 (1987).
- Siete, C., Xiong, R., Khalid, A., Hsieh, Y. -. W., Chuang, C. -. F. Immobilization of C. elegans with different concentrations of an anesthetic for time-lapse imaging of dynamic protein trafficking in neurons. microPublication Biology. 2024, (2024).
- Mitchell, D. H., Stiles, J. W., Santelli, J., Sanadi, D. R. Synchronous growth and aging of Caenorhabditis elegans in the presence of fluorodeoxyuridine. J Gerontol. 34 (1), 28-36 (1979).
- Santi, D. V., McHenry, C. S. 5-Fluoro-2'-deoxyuridylate: covalent complex with thymidylate synthetase. Proc Natl Acad Sci U S A. 69 (7), 1855-1857 (1972).
- Wang, H., Zhao, Y., Zhang, Z. Age-dependent effects of floxuridine (FUdR) on senescent pathology and mortality in the nematode Caenorhabditis elegans. Biochem Biophys Res Commun. 509 (3), 694-699 (2019).
- Beanan, M. J., Strome, S. Characterization of a germ-line proliferation mutation in C. elegans. Development (Cambridge, England). 116 (3), 755-766 (1992).
- Austin, J., Kimble, J. glp-1 is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans. Cell. 51 (4), 589-599 (1987).
- Garigan, D., et al. Genetic analysis of tissue aging in Caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Zhang, Y., et al. Neuronal TORC1 modulates longevity via AMPK and cell nonautonomous regulation of mitochondrial dynamics in C. elegans. eLife. 8, e49158 (2019).
- Dutta, N., et al. Investigating impacts of the mycothiazole chemotype as a chemical probe for the study of mitochondrial function and aging. GeroScience. 46 (6), 6009-6028 (2024).
- Tronstad, K., et al. Regulation and quantification of cellular mitochondrial morphology and content. Curr Pharm Des. 20 (35), 5634-5652 (2014).
- Nikolaisen, J., et al. Automated quantification and integrative analysis of 2D and 3D mitochondrial shape and network properties. PLoS One. 9 (7), e101365 (2014).
- Lee, J. S., Wee, T. L. E., Brown, C. M. Calibration of wide-field deconvolution microscopy for quantitative fluorescence imaging. J Biomol Tech. 25 (1), 31-40 (2014).
- Hong, M., Kwon, J. Y., Shim, J., Lee, J. Differential hypoxia response of hsp-16 genes in the nematode. J Mol Biol. 344 (2), 369-381 (2004).
- Yan, J., Sun, C. L., Shin, S., Van Gilst, M., Crowder, C. M. Effect of the mitochondrial unfolded protein response on hypoxic death and mitochondrial protein aggregation. Cell Death Dis. 12 (7), 711 (2021).
- Koopman, M., et al. A screening-based platform for the assessment of cellular respiration in Caenorhabditis elegans. Nat Protoc. 11 (10), 1798-1816 (2016).
- Shpilka, T., et al. UPRmt scales mitochondrial network expansion with protein synthesis via mitochondrial import in Caenorhabditis elegans. Nat Comm. 12 (1), 479 (2021).
- Braeckman, B. P., Smolders, A., Back, P., De Henau, S. In vivo detection of reactive oxygen species and redox status in Caenorhabditis elegans. antioxidants & redox signaling. 25 (10), 577-592 (2016).
- Bar-Ziv, R., et al. Measurements of physiological stress responses in C. elegans. J Vis Exp. 159, e61001 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados