Method Article
衰老过程中 秀丽隐杆线 虫线粒体形态的成像和定量
摘要
该协议提供了一种标准化方法,用于对 秀丽隐杆线虫 衰老过程中多个组织中的线粒体形态进行成像。
摘要
线粒体是大多数真核细胞中发现的重要细胞器,是通过有氧呼吸产生能量的主要场所。除了作为“细胞发电厂”的众所周知的作用外,线粒体还参与许多其他重要的细胞过程,包括细胞代谢、增殖、免疫信号传导和激素信号传导的调节。衰老过程中或线粒体应激下线粒体功能的恶化通常以线粒体形态和体积的明显变化为特征。线虫秀 丽隐杆 线虫是研究这些变化的理想模型,因为它的身体透明且寿命短,这在其整个生命周期中都有利于实时显微镜检查。然而,即使在 秀丽隐杆线虫 领域内,也有许多用于线粒体成像的转基因构建体和方法可用,每种方法都有自己的局限性。在这里,单拷贝、基质定位的 GFP 构建体被介绍为一种稳健可靠的线粒体形态成像方法 。本研究特别关注实验可控因素,以最大限度地减少在衰老过程中进行线粒体成像时重复和研究之间的变异性。此外,mitoMAPR 被推荐作为一种可靠的方法来量化衰老过程中不同组织类型的线粒体形态变化。
引言
线粒体是被双磷脂膜包围的细胞器,作为新陈代谢和能量产生的关键部位,在维持细胞生物能量学方面发挥着重要作用1。大量的线粒体不断以 ATP 的形式产生能量以满足细胞需求2。除了这些重要作用外,线粒体还参与复杂的细胞过程,如信号转导、自噬、先天免疫、细胞周期和细胞死亡途径 2,3。线粒体表现出多种形态,从小的单个细胞器到广泛的互连管状网络,具体取决于不同的能量需求和线粒体健康状况4。
线粒体的一个关键特征是它们的动态性质,因此它们可以通过紧密协调和连续的裂变和融合事件序列在这些不同形式之间交替4。这些相反过程之间的精确平衡调节线粒体形态、数量、大小和在细胞质内的位置5。此外,这些融合和裂变过程对于维持线粒体5 的质量控制非常重要。例如,线粒体裂变是通过线粒体自噬去除受损线粒体和通过自噬选择性去除线粒体的重要组成部分2。线粒体动力学在细胞分裂、发育、对各种压力源的抵抗力和细胞代谢的维持中也起着重要作用6。
线粒体动力学的破坏与许多疾病有关,包括神经退行性疾病、代谢性疾病、心血管疾病和癌症4。此外,衰老与线粒体的剧烈重塑有关,这主要是由于这些聚变-裂变动力学的变化7。因此,在各种压力或疾病条件下以及整个衰老过程中可视化和监测线粒体形态的变化为了解细胞功能、疾病机制和潜在治疗策略提供了有价值的见解。
与许多分子途径一样,线粒体动力学和功能在真核生物中也高度保守,包括线虫 秀丽隐杆线虫等模式生物。与人类细胞类似, 秀丽隐 杆线虫中的线粒体融合是通过动力蛋白相关的鸟嘌呤三磷酸酶 (GTP 酶) 蛋白的功能实现的,包括 FZO-1(哺乳动物 Mfn1/2 的直系同源物)和 EAT-3(哺乳动物 Opa1 的直系同源物),它们分别控制线粒体外膜 (OMM) 和线粒体内膜 (IMM) 的融合8.线粒体裂变受动力蛋白相关蛋白(DRP-1,人 Drp1 的直系同源物)的调节,该蛋白通过在线粒体外膜周围形成环状复合物来促进线粒体裂变,这些复合物会收缩并最终分离线粒体膜9。线粒体融合在线粒体质量控制中起着关键作用,它允许线粒体内容物(包括线粒体 DNA、蛋白质和脂质)混合,从而允许健康线粒体的内容物补充部分受损的线粒体9。另一方面,线粒体裂变允许线粒体分裂,产生新的线粒体,并促进它们不仅在细胞质内分布,而且在细胞分裂过程中分布到子细胞,确保线粒体的正确遗传和功能9。该事件对于分离受损或功能失调的线粒体片段也是必不可少的,然后可以通过线粒体自噬9 靶向降解。
秀丽隐杆 线虫长期以来一直被认为是最强大的遗传模型系统之一,因为它具有完整的基因组, 并且有多种遗传工具可用,包括促进基因修饰的 CRISPR/Cas910、多种基因过表达方法,以及基于细菌食物的 RNA 干扰 (RNAi) 方法 (RNAi)11。此外,它们透明的解剖结构允许在活体生物体中进行显微镜成像12。最后,它们的寿命相对较短、维护成本低以及易于生产大量年龄匹配的动物,使它们成为衰老生物学的优秀模型系统13。这些好处,再加上已知的主要线粒体调节途径的保守性,使 秀丽隐 杆线虫成为研究衰老过程中线粒体动力学的极具吸引力的模型系统。
荧光标记物广泛用于生物学研究,以可视化和研究细胞成分,包括线粒体。常用的细胞渗透性染料如 MitoTracker 和四甲基罗丹明乙酯 (TMRE)等 14.前者用于染色带负电荷的整体线粒体基质15,而后者用于通过其带正电荷的三苯基鏻离子16 评估相对线粒体膜电位。虽然它们受益于不需要转基因,但在衰老过程中蠕虫的厚角质层结构和渗透性发生变化,以及染料在不同组织中浸润的变化,使得基于染料的方法在秀丽隐杆线虫中具有挑战性 17。此外,线粒体形态的评估受到染料的潜在脱靶效应(例如染料聚集)的混淆17。相反,蠕虫模型中通常使用表达线粒体定位荧光团的遗传方法。
在这里,本研究的重点是突出在细胞类型特异性启动子下表达线粒体基质定位 GFP(以下简称 MLS::GFP)的菌株。重要的是,这些转基因品系是使用 MosSCI 方法制备的,以确保报告基因在已知基因组位点中的单拷贝表达,从而避免了其他可用菌株的问题。例如,由于将染色体外质粒阵列整合到拷贝数未知的随机基因座中,一些线粒体靶向蛋白的高拷贝表达导致表达水平的变异。此外,它们先前已被证明会诱导线粒体损伤,因为细胞正确定位过表达的线粒体蛋白是一项高负担19。因此,精确、稳定和低基因表达以及易于杂交到已知基因座使这些 MosSCI 转基因成为首选方法。使用这些菌株,本研究标准化了对秀丽隐杆线虫肌肉、肠道和皮下线粒体进行成像的方法。此外,它强调了重要的技术应用和故障排除方法,这些方法对于考虑和确保秀丽隐杆线虫衰老过程中线粒体的可重复实验分析非常重要。
研究方案
材料 表中列出了本研究中使用的试剂和设备的详细信息。
1. 秀丽隐杆线虫的生长和维持
- 制备线虫生长培养基 (NGM) 板
- 对于 秀丽隐杆线虫的培养,使用标准的 2% 琼脂平板,其中含有 1 mM CaCl2、12.93 μM (5 μg/mL) 胆固醇、25 mM KPO4 (pH 6.0)、1 mM MgSO4、2.5 mM (0.25% w/v) 蛋白胨和 51.3 mM NaCl。请参阅 Castro Torres 等人 20 中描述的倾注 NGM 板的详细方法。
注:对于 RNA 干扰 (RNAi) 实验,对于 1 L NGM 琼脂平板,添加 1 mL 1 M IPTG 和 1 mL 100 mg/mL 羧青霉素。 - NGM 板固化后,在室温下在溶原肉汤 (LB) 中培养 OP50 培养物 24-48 小时,或在 LB 中培养携带 pL4440 空载体质粒的 HT115 培养物与抗生素(氨苄青霉素/羧苄青霉素 100 μg/mL + 四环素 5 μg/mL),在 37 °C 下摇动 12-16 小时。
注:该质粒可从商业来源获得(参见 材料表)。 - 将 200 μL OP50 或 HT115 培养物接种到 60 mm 平板上,或将 1 mL 接种到 100 mm 平板上。
- 将板干燥至不再湿润,并将板存放在 4 °C 的密封容器中长达 2 个月。
- 可选:将 100 μL 10 mg/mL 5-氟-2'-脱氧尿嘧啶 (FUDR) 直接添加到接种有细菌的 NGM 琼脂平板上,以对 L4/成虫进行化学灭菌并防止年轻蠕虫的发育。
注:本研究中开发和使用的菌株是:
(1) RHS191 - uthSi17[myo3p::MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] I
(2) RHS192 - uthSi83[col19p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)]I
(3) RHS193 - uthSi80[vha-6p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] IV
- 对于 秀丽隐杆线虫的培养,使用标准的 2% 琼脂平板,其中含有 1 mM CaCl2、12.93 μM (5 μg/mL) 胆固醇、25 mM KPO4 (pH 6.0)、1 mM MgSO4、2.5 mM (0.25% w/v) 蛋白胨和 51.3 mM NaCl。请参阅 Castro Torres 等人 20 中描述的倾注 NGM 板的详细方法。
- 同步 C. elegans 通过漂白
- 对于需要大量线虫的大规模检测,将装满动物的 60 mm NGM-琼脂平板切成小块,然后将它们分块到 100 mm 平板上,这些平板上接种有细菌以进行扩增。分块后在 20 °C 下生长时,让 100 mm 板充满蠕虫 2-3 天。有关扩张动物的更详细建议,请参阅 Castro Torres 等人20。
- 为了收集蠕虫进行同步,将 5-10 mL 的 M9 溶液(22 mM 的 KH2PO4 二元、42.3 mM 的 Na2HPO4、85.6 mM 的 NaCl、1 mM 的 MgSO4)倒入 100 mm NGM 琼脂平板上带有蠕虫,然后轻轻旋转 M9 溶液以从细菌草坪上松开蠕虫。
- 用血清移液管吸取蠕虫并将其转移到 15 mL 锥形管中。
注意:建议使用玻璃血清移液器,因为塑料血清移液器往往会使蠕虫粘在塑料移液器的内壁上。 - 在室温下以 1,100 x g 离心 30 秒,以沉淀蠕虫并使用真空泵吸出上清液。
- 准备 5 mL 漂白溶液,其中含有 1.5 mL 6% 次氯酸钠、0.75 mL 5 M NaOH 或 KOH 和 2.75 mL dH2O。将 5 mL 漂白溶液倒入蠕虫颗粒中。
注意:在此步骤中,建议戴手套和穿实验服,因为次氯酸钠和氢氧化物溶液具有腐蚀性。 - 将蠕虫 + 漂白溶液混合物孵育 ~4-6 分钟,直到所有动物尸体完全溶解,只剩下卵。用力摇晃混合物以帮助溶解过程。
注意:在解剖显微镜下每分钟检查一次蠕虫。根据成虫体内的溶解量缩短或延长漂白时间。将卵长时间留在漂白溶液中会导致虫卵损坏并影响线虫的活力。 - 在室温下以 1,100 x g 离心鸡蛋/漂白溶液混合物 30 秒以沉淀鸡蛋,然后使用真空泵小心吸出上清液,确保不要吸出鸡蛋沉淀。
注意:建议快速进行洗涤,以防止鸡蛋长时间暴露在漂白溶液中。 - 用 5-10 mL 的 M9 溶液清洗鸡蛋 4 次。
- 将鸡蛋重悬于少量 M9 溶液中,然后通过移液至 50 μL 将它们接种到接种有细菌的 NGM 板上。通过将 3-5 μL 鸡蛋混合物移液到板上并计算该体积中的鸡蛋数量,粗略估计鸡蛋的数量。加入 M9 将鸡蛋混合物稀释至该体积中可计数数量的鸡蛋。
注意:建议每板铺板少于 100 条蠕虫,因为当铺板的动物过多时,蠕虫可能会在成年的第 1 天之前挨饿。有关动物接种数量的更详细建议,请参阅 Castro Torres 等人20。 - 或者,L1 阻滞线虫以实现更紧密的年龄同步。对于 L1 阻滞,将适当体积的 M9 溶液添加到 15 mL 锥形管中的鸡蛋沉淀中,体积为 10-12 mL。让蠕虫在 20 °C 下在旋转器中旋转 16-24 小时。 离心(如步骤 1.2.7 所示)并吸出上清液。计算 L1 动物的浓度并将其接种到 NGM 平板上,如步骤 1.2.9 中的鸡蛋所述。
- 将 秀丽隐杆线虫 种群老化到所需的年龄:如步骤 1.1.6 所述,将 L4/成年动物转移到含有 5-氟-2'-脱氧尿嘧啶 (FUDR) 的平板上,以防止后代并消除任何已经产生的后代。有关在没有 FUDR 的情况下使蠕虫老化的替代方法,请参阅 Castro Torres 等人20。
注意:在选择漂白作为在蠕虫的第一个幼虫阶段同步的方法之前,需要考虑一些重要因素。如前所述,过度暴露于漂白剂会损害胚胎,降低孵化率,并为抗漂白剂菌株产生选择压力,从而可能改变种群的遗传学。此外,如果胚胎长时间不喂食,营养消耗会损害新陈代谢和发育。因此,产蛋或 NemaSync 方法可以用作漂白方法的替代方法。Castro Torres 等人20 的参考研究中逐步描述了产卵程序的方法。NemaSync 是一种更新且更昂贵的商业方法,其中使用手动蜗杆同步器来同步蜗杆,而无需使用化学品21。
2. 秀丽隐杆线虫线粒体成像
- 用于秀丽隐杆线虫成像的载玻片的制备
- 将 10-20 μL M9 溶液添加到载玻片上,并将所需数量的特定年龄的成虫转移到载玻片上的溶液中。建议使用大约 25 条蠕虫。
注:在某些条件下,使用传统方法麻痹蠕虫(例如,四咪唑或叠氮化钠)可能会导致线粒体碎裂(见下文)。相反,这里建议使用 HistoBond 玻片,其表面具有永久的正表面电荷,将减少蠕虫的运动,而无需使用可能引入伪影的药物。将蠕虫与 M9 溶液的适当比例放在一起很重要,因为低体积可能会导致动物在载玻片和盖玻片之间被压碎,而高体积会导致动物在图像采集过程中移动。 表 1 中提供了相关建议。 - 或者,对于使用麻痹蠕虫的药物,请参阅本手稿的代表性结果,了解可用于线粒体有限碎裂的浓度和时间点范围。
注意:尽管在健康的野生型动物中没有重大断裂,但重要的是要记住,某些突变体或病症可能会在这些药物浓度相同的情况下发生线粒体断裂,因此在使用这些药物时需要进行一些测试。 - 用盖玻片覆盖载玻片上 M9 溶液中的蠕虫,并使用指甲油密封侧面并防止 M9 溶液蒸发。
- 可选:对于肌肉成像,通过轻轻轻推盖玻片来滚动蠕虫。这使得蠕虫侧面的体壁肌肉能够更好地与焦平面对齐,以获得更高质量的图像。
- 将 10-20 μL M9 溶液添加到载玻片上,并将所需数量的特定年龄的成虫转移到载玻片上的溶液中。建议使用大约 25 条蠕虫。
- 使用复合显微镜对线粒体进行成像
- 使用标准的宽视场显微镜检测肌肉和皮下线粒体(myo-3p:MLS::GFP 和 col-19p:MLS::GFP)。
注意:使用了配备 63x/1.4 平场全色差物镜、GFP 滤光片 (11525314)、DFC9000 GT 相机和 LED5 光源的市售成像仪,并使用了兼容的显微镜软件。 - 优化每个显微镜和实验设置的成像设置。
注意:然而,作为起点,使用本手稿中使用的成像仪进行成像的参数如下:皮下线粒体的尺寸为 2048 (x) * 2048(y) 像素,步长为 0.50 μm,由软件自动计算肌肉线粒体。肌肉线粒体的暴露时间为 50 毫秒,皮下线粒体的暴露时间为 50 毫秒。475 nm LED 设置为 50% 功率,中性密度滤光片设置为肌肉 30% 和皮下线粒体 17%。Z 范围从 GFP 信号可见的位置开始,到信号结束的位置。为了最小化 max 投影中的噪声,可以减少用于 max 投影的 z 平面的数量。 - 对于肠道线粒体 (vha-6p::MLS::GFP) 的成像,请使用共聚焦显微镜。
注:成像是在配备 63x/1.4 Plan ApoChromat 物镜、白光激光器 (WLL)、声光分束器、HyD S 探测器的市售共聚焦显微镜上进行的,并在显微镜软件上运行。如前所述,应该优化每个单独的实验设置,但作为起点,使用本研究中使用的 Stellaris 进行成像的参数如下:WLL 设置为 85.00% 最大功率,并使用强度为 3.00% 的 485 nm 激光线。HyD S 检测器设置为 490-590 nm,模拟设置增益为 25。我们以 0.495 μm 的步长(尺寸 z 随蠕虫或组织的大小而变化)单向扫描了 1024 (x) * 1024(y) 或 82.01 μm (x) * 82.01 μm (y) 的区域,具有 5 个 (z) 像素(尺寸 z 随蠕虫或组织的大小而变化),扫描速度为 1,000 Hz,缩放 2.25 倍,线平均值为 2,针孔为 1 AU (95.5 μm)。Z 范围从 GFP 信号可见的位置开始到结束的位置。为了最小化 max 投影中的噪声,可以减少用于 max 投影的 z 平面的数量。
- 使用标准的宽视场显微镜检测肌肉和皮下线粒体(myo-3p:MLS::GFP 和 col-19p:MLS::GFP)。
- 线粒体形态的定量
注:有关详细信息,请参阅 Schindelin 等人 22 并参见 补充图 1A-D。- 下载并安装斐济(斐济只是 ImageJ,https://fiji.sc/)软件。然后打开 FIJI。
- 要下载并安装 MitoMAPR 宏,请从 https://doi.org/10.7554/eLife.49158.033 下载源代码 1。然后复制 “A.” 下提到的所有代码。MitoMAPR-1.0 的 IJM 代码。接下来,打开 Fiji,转到 Plugins > New > Macro,然后将之前复制的代码粘贴到宏窗口中。前往 文件 > 另存为 选项并保存宏以备将来使用。
- 通过将文件拖放到 Fiji 工具栏或转到 File > Open,在 FIJI 中使用 Z 轴堆栈打开 3D 显微图像。
- 通过转到 Image > Stacks > Z Project,然后选择具有最大投影的聚焦图像的切片范围,并选择 Max Intensity 作为投影类型,创建图像的最大投影。
- 通过转到 文件 > 另存为 > Tiff 将图像另存为 TIFF。
- 使用 “矩形” 工具从完整图像中裁剪感兴趣区域 (ROI),绘制 ROI,然后转到 Image > Crop。
- 通过转到文件 >另存为 > Tiff 将图像另存为 TIFF。
- 通过将宏文件拖放到斐济工具栏上来运行之前保存的 MitoMAPR 宏,然后单击 运行。将出现一个新窗口,提示选择 2.3.6 中保存 Tiff 文件的文件夹。
- 将出现一个新窗口,提示您选择一个区域;如前面的步骤 2.3.4 中所述,使用矩形工具在图像中创建一个矩形,然后按 Ok。
注意:如果之前已按照步骤 2.3.6 中的说明裁剪了整个图像,则可以选择整个图像。通过转到 File > Save as > Save,将与线粒体形态学相关的所有值保存为 Excel 文件。
- 使用 FIJI 宏对样品进行批处理
- 创建一个宏以指定要分析的图像区域。打开 斐济 并转到 插件 > 新建 > 宏。将以下代码粘贴到文本框中,并按照 2.3.2 中的说明保存宏(补充图 1E-H)。
注意:制作 矩形 (100, 100, 200, 200);run (指定...,宽度 = 100,高度 = 100,x = 100,y =100 缩放)。 - 创建一个宏,将所有打开的图像另存为 TIFF 文件。在宏窗口中,转到 File > New 并将以下代码粘贴到文本框中。按照 2.3.2 中的说明保存宏。
注意:dir = getDirectory(“选择 A 文件夹”);ids=newArray(nImages);for (i=0;i - 为批处理 MitoMAPR 创建宏。转到 https://doi.org/10.7554/eLife.49158.033 源代码 1 并复制 B. 下提到的所有代码。MitoMAPR-1.0_Batch 的 IJM 代码”。在宏窗口中,转到 File > New,将源代码 1 中的代码粘贴到文本框中,然后按照步骤 2.3.2 中的说明保存宏。
- 创建用于迭代裁剪的宏。转到 https://doi.org/10.7554/eLife.49158.033 源代码 1) 并复制 C.) CropR 的 IJM 代码下提到的所有代码。在宏窗口中, 转到 File > New,将源代码 1 中的代码粘贴到文本框中,然后按照步骤 2.3.2 中的说明保存宏。
- 要开始批处理,首先通过打开所有图像并运行步骤 2.4.2 中保存的宏,将所有要处理的图像保存为 TIFF。所有图像都将以 TIFF 格式保存在指定文件夹中。保存后关闭所有图像。
- 如果在步骤 2.4.5 中保存的图像是 3D 图像(包含 z 堆栈的图像),请通过将其设为 Z 投影来将其转换为 2D 图像。要批量 Z 投影图像,请转到 FIJI 主窗口中的 Process > Batch > Macro 打开批处理窗口。
- 对于 “Input(输入)”,选择包含 2.4.5 中所有要处理的 3D 图像的文件夹的位置。对于“输出”,为处理后保存的图像选择所需的位置。选择输出格式作为 TIFF。将以下代码粘贴到窗口的大文本框中,然后按 process 按钮: run(“Z Project...”, “projection=[Max Intensity]”)。
- 通过使用步骤 2.4.4 中的迭代裁剪宏与步骤 2.4.3 中的指定区域宏相结合,从所有 2D 图像中裁剪具有相同尺寸的 ROI。运行步骤 2.4.4 中的迭代 crop 宏。将出现一个窗口,要求提供目录。
- 选择仅包含步骤 2.4.5 中的 2D 图像或步骤 2.4.6 中的 2D Z 投影图像的文件夹,然后按 选择 按钮。宏将打开所选文件夹中的一张图像,并出现一个标有“make a selection”的窗口,其中包含两个按钮。
- 运行步骤 2.4.3 中指定的选择宏。根据需要修改该宏文本框中的宽度和高度值以更改选区尺寸,然后按 Run (运行 ) 观察新的选区。更改宏中的 X 和 Y 值将修改所选内容的左上角位置。
- 一旦选择尺寸令人满意,将矩形拖动到所需区域,然后按“建立选择”窗口上的 OK 按钮。裁剪的区域将保存到步骤 2.4.7 中选择的文件夹中。
- 该宏将遍历文件夹中的所有剩余图像。对于每个图像,从步骤 2.4.8 开始运行宏,而不更改尺寸值,以确保为所有图像裁剪相同的尺寸。对于每个图像,将选择矩形拖动到所需位置,然后按“建立选择”窗口中的 “确定 ”按钮。
- 打开包含步骤 2.4.8-2.4.9 中裁剪图像的文件夹,然后将所有裁剪的图像移动到新文件夹中。
- 通过运行步骤 2.4.3 中的批处理 MitoMAPR 宏来分析所有裁剪的图像。将出现一个新窗口,要求您选择一个目录。选择包含步骤 2.4.10 中所有裁剪图像的文件夹。完成后,将弹出一个名为 “Data” 的窗口,其中包含与线粒体形态相关的所有值。按照步骤 2.3.9 中的说明将所有数据另存为 Excel 文件。
- 创建一个宏以指定要分析的图像区域。打开 斐济 并转到 插件 > 新建 > 宏。将以下代码粘贴到文本框中,并按照 2.3.2 中的说明保存宏(补充图 1E-H)。
结果
秀丽隐杆线 虫是一种很好的线粒体成像模型,因为它的透明体,可以很容易地进行全蠕虫、活体动物成像,而无需过多的样品制备。此外,通过使用组织特异性启动子表达线粒体靶向荧光蛋白,可以很容易地可视化不同组织中的线粒体形态。在这里, myo-3p (用于肌肉线粒体)、 vha-6p (用于肠道线粒体)和 col-19p (用于皮下线粒体) 用于驱动线粒体靶向 GFP (ATP-1 蛋白的线粒体定位序列) 的表达。 秀丽隐杆线 虫体壁肌肉的线粒体呈现管状形态,沿肌纤维(肌原纤维)排列(图 1A);肠道线粒体表现出高度互连的网状结构,排列不太均匀(图 1B);皮下组织具有管状线粒体,与肠道或肌肉相比,线粒体看起来更圆或椭圆形(图 1C)。线粒体形态在蠕虫的肌肉和肠道长度上通常是一致的,但皮下组织确实在蠕虫近端和远端之间线粒体的相互联系方面表现出一些细微的差异。因此,为了获得可重复的实验结果,建议专注于他们身体的特定区域。在这里,咽部和咽部下方 ~100-200 μm 之间的区域始终成像。
对于肌肉和皮下线粒体,由于细胞的平坦性和低水平的离焦光,在复合显微镜下对它们进行成像可提供足够的分辨率。然而,肠道线粒体很难用复合显微镜观察,因为由于来自肠道其他部分的大量离焦光,肠道的体积大限制了分辨率(图 2)。因此,建议在共聚焦显微镜下对肠道线粒体进行成像,以减少离焦光并可视化适当的线粒体形态。
线粒体的形状是高度动态的,并且根据动物的代谢环境而变化23 甚至由于暴露于可变的基质刚度24。因此,在没有食物来源的情况下将动物长时间放在显微镜载玻片上和玻璃的坚硬基材上可能会影响线粒体形态。在这里,这项研究发现,蠕虫的线粒体在 M9 溶液中的载玻片上大约 30 分钟后碎裂,其中皮下线粒体显示最多的碎裂(图 3)。因此,应在样品制备后迅速进行线粒体成像。
限制蠕虫移动的化学物质,包括叠氮化钠和四咪唑,通常用于 秀丽隐杆线虫的实时成像,因为需要固定蠕虫来捕获动物的多个 z 切片以进行 3D 成像。先前的研究表明,暴露于叠氮化钠或四咪唑会导致线粒体碎裂25。令人惊讶的是,发现叠氮化钠 - 即使是高浓度 - 对肌肉或肠道中的线粒体形态的影响有限。然而,皮下组织比 M9 对照更早地显示线粒体碎裂(图 3)。重要的是,高浓度的四咪唑 (100 mM) 在所有细胞类型中暴露后立即导致显著的线粒体断裂。相比之下,与 M9 对照相比,中等浓度 (10 mM) 导致更快的碎裂。低浓度 (1 mM) 对线粒体形态的影响有限。这些数据表明,使用叠氮化钠可能是线粒体快速成像的可行选择,而通常应避免使用四咪唑。
正如预期的那样, 秀丽隐杆线虫 所有组织的线粒体在自然衰老过程中都表现出碎裂(图 4A)。碎片化可以可视化为线粒体表现为更截短和球形的结构,这与年轻动物中显示的线性管状线粒体截然不同。重要的是要注意,肠道在较晚的年龄确实会增加球形自发荧光结构,因此必须注意不要将自发荧光与实际的线粒体结构混淆。由于蠕虫之间线粒体结构的变化可能存在差异,因此在显着的样本量上进行线粒体形态的定量非常重要,而不仅仅是对一些蠕虫进行成像。这里使用了 mitoMAPR,它允许使用一组不同的指标自动量化线粒体形态,包括对象、网络、每个网络的连接点、连接点、对象长度、线粒体足迹、线粒体覆盖和对象面积(补充表 1 和 补充表 2)。自动化消除了用户的主观偏见。在这里,我们报告了物体长度指标是定量测量衰老过程中肌肉和肠道线粒体形态变化的最佳选择,而连接点指标是测量衰老过程中皮下线粒体形态变化的最佳选择(图 4B)。
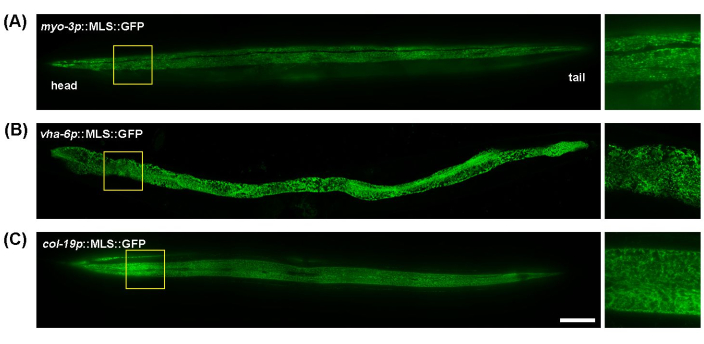
图 1: 秀丽隐杆线虫全身线粒体图像。 在肌肉 (A)、肠道 (B) 和皮下组织 (C) 中表达 MLS::GFP 的转基因动物中,从 L1 阶段开始在 EV 上生长的成年动物在第 5 天的整个长度内进行线粒体成像。比例尺:100 μm。 请单击此处查看此图的较大版本。

图 2:复合显微镜和共聚焦显微镜的线粒体成像比较。 在肌肉 (A)、肠道 (B) 和皮下组织 (C) 中表达 MLS::GFP 的转基因动物中,从 L1 阶段开始在 EV 上生长的第 1 天成年动物中进行线粒体成像。比例尺:5 μm。 请单击此处查看此图的较大版本。

图 3:用 M9、叠氮化钠和四咪唑处理后不同组织的线粒体碎裂评估。 在肌肉、肠道和皮下表达 MLS::GFP 的第 1 天成年动物中进行线粒体成像。动物从 L1 阶段开始在 EV 上生长。将动物置于含有 M9、叠氮化钠(1 mM、10 mM 和 100 mM)或四咪唑(1 mM、10 mM 和 100 mM)的载玻片上,并在载玻片制备后立即进行成像 (0 min) 或载玻片制备后 15 min 或 30 min。显示了每个菌株 n > 5 只动物的代表性图像,用于 2 次生物学重复。比例尺:5 μm。 请单击此处查看此图的较大版本。

图 4:不同组织中 秀丽隐杆线 虫衰老过程中的线粒体成像以及使用 MitoMAPR 对线粒体形态进行定量。 (A) 在成年后的第 1 、 5 、 9 、 13 和 15 天对成年动物进行线粒体成像,在肌肉、肠道和皮下组织表达 MLS::GFP。动物从 L1 阶段开始在 EV 上生长,并从第 1 天成虫阶段转移到含有 FUDR 的 EV 平板上。图像代表每个菌株 n ≥ 5 只动物,≥ 3 次生物学重复。比例尺:5 μm。(B) 第 1 、 5 天、 9 天、第 13 天和第 15 天蠕虫肌肉线粒体和肠道线粒体物体长度的量化,以及第 1 、 5 、 9 、 13 和 15 天蠕虫皮下线粒体连接点的量化。所有单独的数据点都与平均值± SD 相结合。使用学生 t 检验绘制图表并进行统计分析。ns = 不显著,*p < 0.03;**p < 0.002;p < 0.0002;p < 0.0001。 请单击此处查看此图的较大版本。
| 蠕虫的日子 | 蠕虫数 | 缓冲液体积 (mL) |
| 1 | 25 | 13 |
| 5 | 25 | 14 |
| 9 | 20 | 15 |
| 13 | 20 | 15 |
表 1:制备玻片时推荐的蠕虫数量和缓冲液体积。
补充图 1:使用 MitoMAPR 进行单图像定量和多图像批处理的应用工作流程。 (A) 使用 MitoMAPR 宏从单个图像中量化线粒体形态的工作流程。(B) 使用斐济的原始 3D 图像文件的 Z 投影(最大投影)。(C) 从 Z 投影图像中裁剪感兴趣区域。(D) 从 MitoMAPR 宏获取的骨骼图像。比例尺:5 μm。(E) 使用 MitoMAPR 宏使用批处理从多个图像中量化线粒体形态的工作流程。(F) 裁剪宏的屏幕截图。(G) Z 投影的批处理过程的屏幕截图。(H) 批量裁剪宏的选择确认窗口的屏幕截图。 请点击此处下载此文件。
补充表 1: 秀丽隐杆线 虫肌肉组织中线粒体形态指标的定量分析。 记录的指标包括对象、网络、每个网络的交汇点、交汇点、对象长度、线粒体足迹、对象面积和线粒体覆盖率,在不同日期测量。数据为评估线粒体形态随衰老的变化提供了基础。 请点击此处下载此文件。
补充表 2:固定化化学物质对 秀丽隐杆线虫线粒体形态的影响。 数据包括使用 M9 缓冲液或不同浓度的四咪唑和叠氮化钠进行样品制备后,不同浓度和时间点的测量结果,例如对象、网络、每个网络的交界点、交界点、对象长度、线粒体足迹、对象面积以及线粒体覆盖率。 请点击此处下载此文件。
讨论
线粒体形态的荧光成像是确定线粒体变化的最常用方法。虽然透射电子显微镜 (TEM)、原子力显微镜和冷冻电子显微镜等先进显微镜技术提供了更高的分辨率,但大多数研究人员仍然更实惠且更容易获得荧光显微镜。此外,荧光显微镜可以在活细胞中进行,在秀丽隐杆线虫等透明模式生物中,可以在整只动物中进行成像26,27。转基因秀丽隐杆线虫的成像非常简单,基因编码的荧光标记物可以对线粒体进行更可靠和稳健的成像,因为它不需要复杂的样品处理,也不需要使用常规线粒体染料(如 MitoTracker 或 TMRE)在不同细胞类型或条件下进行可变染色 28,29,30.基因编码的荧光标记通常涉及用荧光团直接标记线粒体蛋白或将荧光团与最小的线粒体定位序列偶联。这些构建体通常由组织特异性启动子驱动,能够可视化肌肉、肠道或皮下组织等不同组织中的线粒体31。通常,这些荧光标记的蛋白质过表达,如果全长线粒体蛋白过表达,可能会产生潜在的不良生理效应;因此,最小的 MLS 序列是更好的选择32.然而,即使过表达最少的 MLS-荧光蛋白融合,也应避免高过表达,因为将大量蛋白质输入线粒体会破坏线粒体膜的潜力并影响动物健康33。虽然对所有目前可用的秀丽隐杆线虫菌株的详尽描述超出了本手稿的范围,但可以在此处找到对众多线粒体报告基因的详细比较分析以及每个线粒体报告的优缺点31。
对于 秀丽隐杆线虫中线粒体的活细胞成像,标准化合物或宽场显微镜可能是首选,因为这些方法比共聚焦显微镜更快、更容易。在这项研究中,结果表明,肌肉和皮下组织等扁平细胞从共聚焦显微镜中受益最小,而复合显微镜允许以足够的分辨率进行采集以可视化适当的线粒体形态。由于离焦光,像肠道这样的较大细胞使复合显微镜检查具有挑战性。因此,需要共聚焦显微镜对肠道线粒体形态进行可靠的成像。
在活体动物的整个组织厚度上进行 3D 成像的一个重要考虑因素是防止蠕虫在图像采集过程中移动。研究人员经常使用麻痹蠕虫的方法,例如四咪唑或叠氮化钠34。叠氮化钠抑制细胞色素 c 氧化酶(复合物 IV),这是线粒体电子传递链中的一种关键酶,由于缺乏肌肉收缩和其他细胞功能所需的 ATP,导致全身瘫痪34,35。四咪唑通过在神经肌肉接头处模拟乙酰胆碱发挥作用,导致持续的去极化和肌肉收缩36。然而,暴露于这些药物会通过引起氧化应激来诱导线粒体碎裂23。在这项研究中,发现四咪唑非常迅速地诱导线粒体碎裂,但叠氮化钠的作用要有限得多。
秀丽隐杆线虫提供了一种非常简单易行的方法来研究衰老对线粒体形态的影响,因为它的寿命短且容易使动物衰老。在这里,我们选择使用 FUDR,这是一种通过阻止 DNA 复制对动物进行化学绝育的可靠方法 20,37,38。然而,FUDR 可能对特定的衰老参数产生不良影响,对于那些担心 FUDR 脱靶效应的人来说,可以使用其他策略来去除后代39。例如,有不育突变体,包括温度敏感的种系突变体 glp-4,或精子缺陷突变体,如 CF512 40,41,42。作为替代方案,动物也可以通过每天手动从后代中挑选成年动物来自然衰老。
线粒体形态变化的量化也是一个非常重要的考虑因素,因为动物之间的线粒体形态可能存在显着差异。因此,在足够的样本量中进行定量分析和统计对于对线粒体形态变化得出重要结论至关重要。然而,图像分析和定量可能会受到主观偏见和成像高度复杂结构(如互连线粒体)的挑战的很大影响。为此,这里总结了一种使用 Mair 实验室开发的大量指标量化线粒体形态的自动化方法。MitoMAPR 允许客观、自动地量化线粒体形态,测量线粒体的各个方面,包括线粒体网络、对象长度、分布、网络覆盖率和线粒体足迹。MitoMAPR 是 ImageJ 的免费宏,因此可供所有拥有功能计算机的实验室使用。使用 MitoMAPR 的一个重要方面是对大样本量进行定量,以确定哪种线粒体形态指标最可靠,以确定所测试实验条件的变化43,44。在这里,发现物体长度和连接点是确定肌肉、肠道和皮下组织衰老过程中变化的最佳指标。分析线粒体形态变化的另一种方法是从 z 堆栈图像生成线粒体的 3D 表示,然后进行 3D 分析16,45。这可以使用市售软件来实现,例如带有 SharpStack Total Deconvolution 和 3D Constructor 模块的 Image-Pro Plus。3D 线粒体表示的量化已被证明可以更准确地了解线粒体形状和网络特性46。然而,与半 3D 方法相比,这种方法在技术上更复杂且成本更高,半 3D 方法涉及将来自宽场或共聚焦 z 堆栈的多个图像部分折叠成单个 2D 投影,并使用 mitoMAPR 等工具对其进行分析。本研究总结了线粒体基质靶向 GFP 的单拷贝表达的使用,并详细介绍了成像过程中需要避免的一些陷阱。
限制和时间注意事项
虽然建议对大而厚的组织(如肠组织)进行共聚焦显微镜检查,但标准的线扫描共聚焦显微镜技术可能太慢,无法对线粒体进行快速成像。考虑到我们的数据表明,将蠕虫长时间放在载玻片上会导致线粒体碎裂,这一点尤其正确。尽管共聚焦显微镜技术已经取得了长足的进步,但对于某些实验室来说,可以进行快速成像的显微镜(例如转盘式显微镜、Airyscan 显微镜或本研究中使用的显微镜)可能成本高昂。在这些情况下,化合物显微镜可以与计算清除方法相结合,以去除离焦光,例如反卷积47。
此外,如前所述,MitoMAPR 是一种强大的自动化宏,用于定量测量线粒体的长度和线粒体网络的互连。但是,考虑到此处列出的限制,应谨慎使用它。首先,使用基质靶向 GFP 仅观察到内膜的线粒体碎裂,这可能无法完全概括外膜的线粒体形态,因为在没有外膜裂变的情况下可能会发生内膜裂变事件。因此,为了对两种膜进行更精细的成像,应同时使用基质靶向和线粒体外膜靶向荧光团。由于线粒体外膜标志物如果高度过表达,可能会遭受与基质定位蛋白相同的后果,因此建议使用单拷贝标记物,尤其是那些使用最小线粒体定位序列而不是完整蛋白质的标记物,例如此处使用的31。
如图 3 所示,线粒体可以在显微镜下迅速碎裂,制备样品载玻片的不同方法会在很大程度上影响形态,具体取决于所使用的缓冲液。本手稿中描述的代表性数据表明,蠕虫的线粒体在 M9 和低浓度叠氮化钠下发生最小碎裂。四咪唑是另一种广泛使用的麻痹蠕虫的化学物质,会迅速使线粒体碎裂,表明通常应避免使用。尽管 M9 和叠氮化钠未显示线粒体的显着碎裂,但重要的是要注意不同的菌株可能对线粒体的反应可能与此处显示的结果不同。虽然其他研究表明叠氮化钠可以碎裂线粒体,但由于我们的构建体的表达水平低,我们的菌株在暴露于叠氮化钠时可能不会显示明显的线粒体碎裂。线粒体定位蛋白的高表达会破坏膜电位,因此,其他研究中使用的不同菌株可能使线粒体更容易从本研究中使用的相同浓度的叠氮化钠中碎裂。无论如何,应注意确保麻痹蠕虫的化学物质在特定条件下不会导致线粒体断裂,以便在所有研究中使用之前进行测试,因为特定突变体或条件可能更容易受到药物诱导的线粒体断裂的影响。此外,即使将蠕虫保持在 M9 中也会导致其天然线粒体形态和活动发生变化,因为它们在 M9 缓冲液中的游泳活动先前已被证明会影响线粒体裂变和融合动力学。此外,将蠕虫长时间留在 M9 中会激活缺氧反应,通过引起线粒体蛋白质稳态中断来显着影响线粒体48,49。
最后,另一个重要的考虑因素是,虽然线粒体形态的变化通常与线粒体功能的变化相关,但两者之间并不总是直接相关。因此,建议对线粒体功能进行更彻底的分析。例如,可以使用 Seahorse 仪器50 测量耗氧率,可以使用 JC9 或 TMRE51 等膜电位染料测量线粒体膜电位,可以使用氧化还原敏感染料(如 roGFP52)测量线粒体氧化状态,可以使用鱼藤酮53 等应激源的敏感性来测量线粒体应激恢复力.由于线粒体的成像可能非常迅速,因此我们在此处提供这些方法作为确定实验条件是否影响线粒体形态的简单第一步。这些方法甚至适用于药物或基因的大规模筛选,并使用推荐的其他指标进行更彻底的线粒体分析。总而言之,我们在这里回顾了被认为是对 秀丽隐杆线 虫线粒体形态进行成像的最简单方法,同时具有最小的实验误差。
披露声明
作者没有什么可披露的。
致谢
J.K. 得到了 USC Provost Fellowship 的支持;M.A. 和 G.G. 由 T32AG052374 提供支持;M.V. 由 1R25AG076400 支持;RHS 得到了美国国家老龄化研究所 R01AG079806 和 Larry L. Hillblom 基金会的 2022-A-010-SUP 的支持。一些菌株由 CGC 提供,该 CGC 由 NIH 研究基础设施计划办公室拨款 P40 OD010440资助。使用 Wormbase 进行了一些基因分析,该 Wormbase 由 U41 赠款HG002223资助。
材料
| Name | Company | Catalog Number | Comments |
| 5-fluoro-2'-deoxyuridine (FUDR) | Spectrum Chemical | F2026-10GMBL | for proliferation inhibition |
| APEX IPTG | Genesee | 18-242 | for RNAi |
| Bacto Agar | VWR | 90000-764 | for NGM plates |
| Bacto Peptone | VWR | 97064-330 | for NGM plates |
| Calcium chloride dihydrate | VWR | 97061-904 | for NGM plates |
| Carbenicillin | VWR | 76345-522 | for RNAi |
| Cholesterol | VWR | 80057-932 | for NGM plates |
| Confocal microscope | Stellaris 5 | ||
| DMSO | VWR | BDH1115-1LP | solvent for drugs |
| HistoBond microscope slides | VWR | 16005-110 | for slides preparation |
| LB Broth | VWR | 95020-778 | for LB |
| LEICA S7E Dissecting Scope | Leica | 10450840 | Standard dissecting microscope |
| LEICA STELLARIS 5 | Leica | 158101100 | Confocal Microscope |
| LEICA THUNDER | Leica | 11525679 | Compound Microscope |
| Magnesium sulfate heptahydrate | VWR | 97062-132 | for NGM plates, M9 |
| pL4440 empty vector plasmid | addgene | plasmid #1654 | for empty vector plasmid |
| Potassium Chloride | VWR | 97061-566 | for bleach soluton |
| Potassium phosphate dibasic | VWR | EM-PX1570-2 | for NGM plates |
| Potassium phosphate monobasic | VWR | EM-PX1565-5 | for M9 |
| Sodium azide | VWR | 97064-646 | for paralyzing worms |
| Sodium Chloride | VWR | EM-SX0420-5 | for NGM plates, M9 |
| Sodium hypochlorite | VWR | RC7495.7-32 | for bleach solution |
| Sodium phosphate dibasic | VWR | 71003-472 | for M9 |
| Tetracycline hydrochloride | VWR | 97061-638 | for RNAi |
| WOB-L® Dry Vacuum Pumps, Standard-Duty, Welch® | VWR | 80077-612 | for aspiration |
参考文献
- Protasoni, M., Zeviani, M. Mitochondrial structure and bioenergetics in normal and disease conditions. Int J Mol Sci. 22 (2), 586 (2021).
- Wang, Y., et al. The role of mitochondrial dynamics in disease. MedComm. 4 (6), e462 (2023).
- Kamer, K. J., Mootha, V. K. The molecular era of the mitochondrial calcium uniporter. Nat Rev Mol Cell Biol. 16 (9), 545-553 (2015).
- Chen, W., Zhao, H., Li, Y. Mitochondrial dynamics in health and disease: mechanisms and potential targets. Signal Transduct Target Ther. 8 (1), 333 (2023).
- Adebayo, M., Singh, S., Singh, A. P., Dasgupta, S. Mitochondrial fusion and fission: The fine-tune balance for cellular homeostasis. FASEB J. 35 (6), e21620 (2021).
- Madan, S., Uttekar, B., Chowdhary, S., Rikhy, R. Mitochondria lead the way: mitochondrial dynamics and function in cellular movements in development and disease. Front Cell Dev Biol. 9, 781933 (2021).
- Sharma, A., Smith, H. J., Yao, P., Mair, W. B. Causal roles of mitochondrial dynamics in longevity and healthy aging. EMBO Rep. 20 (12), e48395 (2019).
- Rolland, S. G., Lu, Y., David, C. N., Conradt, B. The BCL-2-like protein CED-9 of C. elegans promotes FZO-1/Mfn1,2- and EAT-3/Opa1-dependent mitochondrial fusion. J Cell Biol. 186 (4), 525-540 (2009).
- Labrousse, A. M., Zappaterra, M. D., Rube, D. A., van der Bliek, A. M. C. elegans dynamin-related protein DRP-1 controls severing of the mitochondrial outer membrane. Mol Cell. 4 (5), 815-826 (1999).
- Dickinson, D. J., Pani, A. M., Heppert, J. K., Higgins, C. D., Goldstein, B. Streamlined genome engineering with a self-excising drug selection cassette. Genetics. 200 (4), 1035-1049 (2015).
- Bosher, J. M., Labouesse, M. RNA interference: Genetic wand and genetic watchdog. Nat Cell Biol. 2 (2), E31-E36 (2000).
- Yu, C. C. J., et al. Expansion microscopy of C. elegans. eLife. 9, e46249 (2020).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Front Endocrinol. 11, 554994 (2020).
- Dingley, S., et al. Mitochondrial respiratory chain dysfunction variably increases oxidant stress in Caenorhabditis elegans. Mitochondrion. 10 (2), 125-136 (2010).
- Presley, A. D., Fuller, K. M., Arriaga, E. A. MitoTracker Green labeling of mitochondrial proteins and their subsequent analysis by capillary electrophoresis with laser-induced fluorescence detection. J Chromatogr B Anal Technol Biomed Life Sci. 793 (1), 141-150 (2003).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Curr Protoc Cell Biol. Chapter 4, Unit 4.25.1-Unit 4.25.21 (2010).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. Novel imaging tools to study mitochondrial morphology in Caenorhabditis elegans. Life Sci Alliance. 7 (11), e202402918 (2024).
- Benedetti, C., Haynes, C. M., Yang, Y., Harding, H. P., Ron, D. Ubiquitin-like protein 5 positively regulates chaperone gene expression in the mitochondrial unfolded protein response. Genetics. 174 (1), 229-239 (2006).
- Begelman, D. V., et al. An aco-2::gfp knock-in enables the monitoring of mitochondrial morphology throughout C. elegans lifespan. microPublication Biol. 2022, (2022).
- Castro Torres, T., et al. Surveying low-cost methods to measure lifespan and healthspan in Caenorhabditis elegans. J Vis Exp. 183, e64091 (2022).
- Rasmussen, N. R., Reiner, D. J. Nuclear translocation of the tagged endogenous MAPK MPK-1 denotes a subset of activation events in C. elegans development. J Cell Sci. 134 (17), jcs258456 (2021).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Luz, A. L., et al. Mitochondrial morphology and fundamental parameters of the mitochondrial respiratory chain are altered in Caenorhabditis elegans strains deficient in mitochondrial dynamics and homeostasis processes. PLoS One. 10 (6), e0130940 (2015).
- Oorloff, M., et al. Mechanical stress through growth on stiffer substrates impacts animal health and longevity in C. elegans. bioRxiv. , (2024).
- Chu, X., Wu, S., Raju, R. NLRX1 regulation following acute mitochondrial injury. Front Immunol. 10, 2431 (2019).
- Shah, P., Bao, Z., Zaidel-Bar, R. Visualizing and quantifying molecular and cellular processes in Caenorhabditis elegans using light microscopy. Genetics. 221 (4), iyac068 (2022).
- Wang, Y., Wang, P., Li, C. Fluorescence microscopic platforms imaging mitochondrial abnormalities in neurodegenerative diseases. Adv Drug Deliv Rev. 197, 114841 (2023).
- Ding, J., et al. An expanded GCaMP reporter toolkit for functional imaging in Caenorhabditis elegans. G3 (Bethesda, Md). 13 (10), jkad183 (2023).
- Sarasija, S., Norman, K. R. Analysis of mitochondrial structure in the body wall muscle of Caenorhabditis elegans. Bio-Protocol. 8 (7), e2801 (2018).
- Chaweeborisuit, P., Suriyonplengsaeng, C., Suphamungmee, W., Sobhon, P., Meemon, K. Nematicidal effect of plumbagin on Caenorhabditis elegans: A model for testing a nematicidal drug. Z Naturforsch C J Biosci. 71 (5-6), 121-131 (2016).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. . Novel imaging tools to study mitochondrial dynamics in Caenorhabditis elegans. 7, 11 (2024).
- Bolognesi, B., Lehner, B. Reaching the limit. eLife. 7, e39804 (2018).
- Jishi, A., Qi, X. Altered mitochondrial protein homeostasis and proteinopathies. Front Mol Neurosci. 15, 867935 (2022).
- Morton, K. S., Wahl, A. K., Meyer, J. N. The effect of common paralytic agents used for fluorescence imaging on redox tone and ATP levels in Caenorhabditis elegans. PLoS One. 19 (4), e0292415 (2024).
- Noumi, T., Maeda, M., Futai, M. Mode of inhibition of sodium azide on H+-ATPase of Escherichia coli. FEBS Lett. 213 (2), 381-384 (1987).
- Siete, C., Xiong, R., Khalid, A., Hsieh, Y. -. W., Chuang, C. -. F. Immobilization of C. elegans with different concentrations of an anesthetic for time-lapse imaging of dynamic protein trafficking in neurons. microPublication Biology. 2024, (2024).
- Mitchell, D. H., Stiles, J. W., Santelli, J., Sanadi, D. R. Synchronous growth and aging of Caenorhabditis elegans in the presence of fluorodeoxyuridine. J Gerontol. 34 (1), 28-36 (1979).
- Santi, D. V., McHenry, C. S. 5-Fluoro-2'-deoxyuridylate: covalent complex with thymidylate synthetase. Proc Natl Acad Sci U S A. 69 (7), 1855-1857 (1972).
- Wang, H., Zhao, Y., Zhang, Z. Age-dependent effects of floxuridine (FUdR) on senescent pathology and mortality in the nematode Caenorhabditis elegans. Biochem Biophys Res Commun. 509 (3), 694-699 (2019).
- Beanan, M. J., Strome, S. Characterization of a germ-line proliferation mutation in C. elegans. Development (Cambridge, England). 116 (3), 755-766 (1992).
- Austin, J., Kimble, J. glp-1 is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans. Cell. 51 (4), 589-599 (1987).
- Garigan, D., et al. Genetic analysis of tissue aging in Caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Zhang, Y., et al. Neuronal TORC1 modulates longevity via AMPK and cell nonautonomous regulation of mitochondrial dynamics in C. elegans. eLife. 8, e49158 (2019).
- Dutta, N., et al. Investigating impacts of the mycothiazole chemotype as a chemical probe for the study of mitochondrial function and aging. GeroScience. 46 (6), 6009-6028 (2024).
- Tronstad, K., et al. Regulation and quantification of cellular mitochondrial morphology and content. Curr Pharm Des. 20 (35), 5634-5652 (2014).
- Nikolaisen, J., et al. Automated quantification and integrative analysis of 2D and 3D mitochondrial shape and network properties. PLoS One. 9 (7), e101365 (2014).
- Lee, J. S., Wee, T. L. E., Brown, C. M. Calibration of wide-field deconvolution microscopy for quantitative fluorescence imaging. J Biomol Tech. 25 (1), 31-40 (2014).
- Hong, M., Kwon, J. Y., Shim, J., Lee, J. Differential hypoxia response of hsp-16 genes in the nematode. J Mol Biol. 344 (2), 369-381 (2004).
- Yan, J., Sun, C. L., Shin, S., Van Gilst, M., Crowder, C. M. Effect of the mitochondrial unfolded protein response on hypoxic death and mitochondrial protein aggregation. Cell Death Dis. 12 (7), 711 (2021).
- Koopman, M., et al. A screening-based platform for the assessment of cellular respiration in Caenorhabditis elegans. Nat Protoc. 11 (10), 1798-1816 (2016).
- Shpilka, T., et al. UPRmt scales mitochondrial network expansion with protein synthesis via mitochondrial import in Caenorhabditis elegans. Nat Comm. 12 (1), 479 (2021).
- Braeckman, B. P., Smolders, A., Back, P., De Henau, S. In vivo detection of reactive oxygen species and redox status in Caenorhabditis elegans. antioxidants & redox signaling. 25 (10), 577-592 (2016).
- Bar-Ziv, R., et al. Measurements of physiological stress responses in C. elegans. J Vis Exp. 159, e61001 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。