Method Article
Imaging e quantificazione della morfologia mitocondriale in C. elegans durante l'invecchiamento
In questo articolo
Riepilogo
Questo protocollo fornisce un approccio standardizzato per l'imaging della morfologia mitocondriale in più tessuti di C. elegans durante l'invecchiamento .
Abstract
I mitocondri, importanti organelli cellulari presenti nella maggior parte delle cellule eucariotiche, sono i principali siti di produzione di energia attraverso la respirazione aerobica. Oltre a questo ruolo ben noto come "centrale elettrica cellulare", i mitocondri sono anche coinvolti in molti altri processi cellulari essenziali, tra cui la regolazione del metabolismo cellulare, la proliferazione, la segnalazione immunitaria e la segnalazione ormonale. Il deterioramento della funzione mitocondriale durante l'invecchiamento o sotto stress mitocondriale è spesso caratterizzato da cambiamenti distinti nella morfologia e nel volume mitocondriale. Il nematode C. elegans è un modello ideale per studiare questi cambiamenti grazie al suo corpo trasparente e alla breve durata, che facilitano la microscopia dal vivo per tutta la sua vita. Tuttavia, anche all'interno del campo di C. elegans , sono disponibili numerosi costrutti transgenici e metodi per l'imaging mitocondriale, ognuno con le proprie limitazioni. Qui, i costrutti GFP a copia singola e localizzati in matrice sono presentati come un metodo robusto e affidabile per l'imaging della morfologia mitocondriale in C. elegans. Questo studio si concentra in particolare sui fattori controllabili sperimentalmente per ridurre al minimo gli errori e ridurre la variabilità tra le repliche e tra gli studi durante l'esecuzione dell'imaging mitocondriale durante il processo di invecchiamento. Inoltre, mitoMAPR è raccomandato come metodo robusto per quantificare i cambiamenti nella morfologia mitocondriale tra i tipi di tessuto durante l'invecchiamento.
Introduzione
I mitocondri sono organelli cellulari racchiusi da una doppia membrana fosfolipidica e svolgono un ruolo importante nel mantenimento della bioenergetica cellulare come sito chiave per il metabolismo e la produzione di energia1. Un gran numero di mitocondri genera continuamente energia sotto forma di ATP per soddisfare le richieste cellulari2. Oltre a questi importanti ruoli, i mitocondri partecipano anche a processi cellulari complessi come la trasduzione del segnale, l'autofagia, l'immunità innata, il ciclo cellulare e le vie di morte cellulare 2,3. I mitocondri presentano morfologie diverse, che vanno da piccoli organelli individuali a estese reti tubolari interconnesse, a seconda delle diverse esigenze energetiche e della salute mitocondriale4.
Una caratteristica chiave dei mitocondri è la loro natura dinamica, per cui possono alternarsi tra queste varie forme attraverso una sequenza strettamente coordinata e continua di eventi di fissione e fusione4. Il preciso equilibrio tra questi processi opposti regola la morfologia, il numero, le dimensioni e il posizionamento mitocondriale all'interno del citoplasma5. Inoltre, questi processi di fusione e fissione sono importanti per mantenere il controllo di qualità dei mitocondri5. Ad esempio, la fissione mitocondriale è una componente essenziale della rimozione dei mitocondri danneggiati attraverso la mitofagia e della rimozione selettiva dei mitocondri attraverso l'autofagia2. La dinamica mitocondriale svolge anche un ruolo importante nella divisione cellulare, nello sviluppo, nella resistenza a vari fattori di stress e nel mantenimento del metabolismo cellulare6.
L'interruzione della dinamica mitocondriale è implicata in numerose malattie, tra cui disturbi neurodegenerativi, malattie metaboliche, malattie cardiovascolari e tumori4. Inoltre, l'invecchiamento è associato a un drammatico rimodellamento dei mitocondri, in gran parte dovuto a cambiamenti in queste dinamiche di fusione-fissione7. Pertanto, la visualizzazione e il monitoraggio dei cambiamenti nella morfologia mitocondriale in varie condizioni di stress o malattia e durante il processo di invecchiamento offre preziose informazioni per comprendere la funzione cellulare, il meccanismo della malattia e le potenziali strategie terapeutiche.
Come molte vie molecolari, anche la dinamica e la funzione mitocondriale sono altamente conservate negli eucarioti, compresi gli organismi modello come il nematode Caenorhabditis elegans. Analogamente alle cellule umane, la fusione mitocondriale in C. elegans è ottenuta dalla funzione delle proteine guanina trifosfatasi (GTPasi) correlate alla dinamina, tra cui FZO-1 (ortologo di Mfn1/2 di mammifero) e EAT-3 (ortologo di Opa1 di mammifero) che controllano la fusione della membrana mitocondriale esterna (OMM) e della membrana mitocondriale interna (IMM), rispettivamente8. La fissione mitocondriale è regolata dalla proteina correlata alla dinamina (DRP-1, ortologo di Drp1 umano), che promuove la fissione mitocondriale formando complessi ad anello attorno alla membrana esterna mitocondriale che restringono e infine separano le membrane mitocondriali9. La fusione mitocondriale svolge un ruolo fondamentale nel controllo della qualità mitocondriale consentendo la miscelazione dei contenuti mitocondriali, tra cui DNA mitocondriale, proteine e lipidi, consentendo la complementazione dei mitocondri parzialmente danneggiati con contenuti provenienti da mitocondri sani9. D'altra parte, la fissione mitocondriale consente ai mitocondri di dividersi, creando nuovi mitocondri e facilitando la loro distribuzione non solo all'interno del citoplasma ma anche alle cellule figlie durante la divisione cellulare, garantendo una corretta eredità e funzione mitocondriale9. Questo evento è anche essenziale per segregare i segmenti mitocondriali danneggiati o disfunzionali, che possono quindi essere presi di mira per la degradazione attraverso la mitofagia9.
C. elegans è stato a lungo considerato uno dei più potenti sistemi modello genetici grazie al genoma completo e alla disponibilità di una varietà di strumenti genetici, tra cui CRISPR/Cas9 che facilita le modifiche genetiche10, numerosi metodi per la sovraespressione dei geni e un metodo batterico basato sul cibo per l'interferenza dell'RNA (RNAi)11. Inoltre, la loro anatomia trasparente consente l'imaging microscopico in organismi viventi12. Infine, la loro durata di vita relativamente breve, i bassi costi di manutenzione e la facilità di produrre un gran numero di animali di pari età li rendono un eccellente sistema modello per la biologia dell'invecchiamento13. Questi benefici, combinati con la nota conservazione delle principali vie di regolazione mitocondriale, rendono C. elegans un sistema modello molto interessante per lo studio delle dinamiche mitocondriali durante l'invecchiamento.
I marcatori fluorescenti sono ampiamente utilizzati nella ricerca biologica per visualizzare e studiare i componenti cellulari, compresi i mitocondri. Esistono coloranti specifici permeabili alle cellule, come il MitoTracker e l'estere etilico tetrametilrodamina (TMRE) che sono comunemente usati14. Il primo viene utilizzato per tingere la matrice mitocondriale15 caricata negativamente, mentre il secondo viene utilizzato per valutare il potenziale relativo della membrana mitocondriale attraverso il suo ione trifenilfosfonio16 caricato positivamente. Sebbene traggano vantaggio dal fatto di non richiedere transgenici, la spessa cuticola dei vermi che cambia in struttura e permeabilità durante l'invecchiamento e la variabilità dell'infiltrazione del colorante tra i diversi tessuti rendono difficili gli approcci basati sui coloranti in C. elegans17. Inoltre, la valutazione della morfologia mitocondriale è confusa da potenziali effetti off-target dei coloranti, come l'aggregazione del colorante17. Invece, i metodi genetici per esprimere i fluorofori localizzati nei mitocondri sono comunemente usati nel modello di verme.
Qui, questo studio si concentra sull'evidenziazione di ceppi che esprimono una GFP localizzata nella matrice mitocondriale (d'ora in poi denominata MLS::GFP) sotto promotori specifici del tipo di cellula. È importante sottolineare che queste linee transgeniche sono state realizzate utilizzando il metodo MosSCI per garantire l'espressione di una singola copia del reporter in un locus genomico noto, evitando problemi con altri ceppi disponibili. Ad esempio, l'espressione ad alta copia di alcune proteine mirate ai mitocondri porta a variabilità nei livelli di espressione a causa dell'integrazione di array di plasmidi extracromosomici in un locus casuale con un numero di copie sconosciuto18. Inoltre, è stato precedentemente dimostrato che inducono danni ai mitocondri poiché è un onere elevato per le cellule localizzare correttamente le proteine mitocondriali sovraespresse19. Pertanto, l'espressione genica precisa, stabile e bassa e la facilità di attraversamento con loci noti rendono questi transgenici MosSCI il metodo preferito. Utilizzando questi ceppi, questo studio standardizza i metodi per visualizzare i mitocondri nel muscolo, nell'intestino e nell'ipoderma di C. elegans. Inoltre, evidenzia le importanti applicazioni tecniche e i metodi di risoluzione dei problemi che sono importanti da considerare e garantire un'analisi sperimentale riproducibile dei mitocondri durante l'invecchiamento in C. elegans.
Protocollo
I dettagli sui reagenti e sulle attrezzature utilizzate in questo studio sono elencati nella Tabella dei materiali.
1. Crescita e mantenimento di C. elegans
- Preparazione delle piastre per terreni di crescita per nematodi (NGM)
- Per la coltura di C. elegans, utilizzare piastre di agar standard al 2% con NGM contenenti 1 mM di CaCl2, 12,93 μM (5 μg/mL) di colesterolo, 25 mM di KPO4 (pH 6,0), 1 mM di MgSO4, 2,5 mM (0,25% p/v) di peptone e 51,3 mM di NaCl. Si veda il metodo dettagliato di colata delle piastre NGM descritto in Castro Torres et al.20.
NOTA: Per gli esperimenti di interferenza dell'RNA (RNAi), aggiungere 1 mL di IPTG da 1 M e 1 mL di Carbenicillina da 100 mg/mL per piastre NGM-agar da 1 L. - Dopo che le piastre NGM si sono solidificate, coltivare una coltura di OP50 in brodo di lisogenia (LB) per 24-48 ore a temperatura ambiente o coltivare una coltura di HT115 contenente un plasmide vettore vuoto pL4440 in LB con antibiotici (ampicillina/carbenicillina 100 μg/mL + tetraciclina 5 μg/mL) con agitazione a 37 °C per 12-16 ore.
NOTA: Questo plasmide è disponibile da fonti commerciali (vedi Tabella dei materiali). - Seminare 200 μL di coltura OP50 o HT115 su piastre da 60 mm o 1 mL su piastre da 100 mm.
- Asciugare le piastre fino a quando non sono più bagnate e conservare le piastre in contenitori sigillati a 4 °C per un massimo di 2 mesi.
- Opzionale: aggiungere 100 μL di 10 mg/mL di 5-fluoro-2'-deossiuridina (FUDR) direttamente su piastre NGM-agar seminate con batteri per sterilizzare chimicamente i vermi L4/adulti e prevenire lo sviluppo di vermi più giovani.
NOTA: I ceppi sviluppati e utilizzati in questo studio sono:
(1) RHS191 - uthSi17[myo3p::MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] I
(2) RHS192 - uthSi83[col19p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)]I
(3) RHS193 - uthSi80[vha-6p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] IV
- Per la coltura di C. elegans, utilizzare piastre di agar standard al 2% con NGM contenenti 1 mM di CaCl2, 12,93 μM (5 μg/mL) di colesterolo, 25 mM di KPO4 (pH 6,0), 1 mM di MgSO4, 2,5 mM (0,25% p/v) di peptone e 51,3 mM di NaCl. Si veda il metodo dettagliato di colata delle piastre NGM descritto in Castro Torres et al.20.
- Sincronizzazione C. elegans per decolorazione
- Per un test su larga scala che richiede un numero elevato di nematodi, tagliare una piastra NGM-agar da 60 mm piena di animali in piccoli pezzi e spezzettarli su piastre da 100 mm seminate con batteri per l'espansione. Attendere 2-3 giorni affinché le piastre da 100 mm siano piene di vermi quando vengono coltivate a 20 °C dopo la spezzettatura. Per raccomandazioni più dettagliate per l'espansione degli animali, fare riferimento a Castro Torres et al.20.
- Per raccogliere i vermi per la sincronizzazione, versare 5-10 mL di soluzione M9 (22 mM di KH2PO4 monobasico, 42,3 mM di Na2HPO4, 85,6 mM di NaCl, 1 mM di MgSO4) su piastre NGM-agar da 100 mm con vermi e agitare delicatamente la soluzione M9 per allentare i vermi dai prati batterici.
- Aspirare i vermi con una pipetta sierologica e trasferirli in provette coniche da 15 mL.
NOTA: Si consigliano pipette sierologiche in vetro poiché le pipette sierologiche in plastica tendono a far aderire i vermi alla parete interna delle pipette in plastica. - Centrifugare per 30 s a 1.100 x g a temperatura ambiente per pellettare le viti senza fine e aspirare il surnatante utilizzando una pompa a vuoto.
- Preparare 5 mL di soluzione sbiancante contenente 1,5 mL di ipoclorito di sodio al 6%, 0,75 mL di 5 M NaOH o KOH e 2,75 mL di dH2O. Versare i 5 mL di soluzione sbiancante nel pellet di verme.
ATTENZIONE: In questa fase si consiglia di indossare guanti e camice da laboratorio poiché le soluzioni di ipoclorito di sodio e idrossido sono corrosive. - Incubare la miscela di verme + soluzione sbiancante per ~4-6 minuti fino a quando tutti i corpi degli animali sono completamente disciolti e rimangono solo le uova. Agitare energicamente la miscela per favorire il processo di dissoluzione.
NOTA: Controllare i vermi ogni minuto al microscopio da dissezione. Accorciare o prolungare il tempo di sbiancamento in base a quanto i corpi dei vermi adulti sono disciolti. Lasciare le uova all'interno di una soluzione sbiancante per un periodo prolungato provocherà danni alle uova e influirà sulla vitalità dei nematodi. - Centrifugare la miscela di uova/soluzione sbiancante per 30 s a 1.100 x g a temperatura ambiente per pellettare le uova e quindi aspirare accuratamente il surnatante utilizzando una pompa a vuoto, facendo attenzione a non aspirare la pellet di uova.
NOTA: Si consiglia di eseguire lavaggi rapidi per evitare l'esposizione prolungata delle uova alla soluzione sbiancante. - Lavare le uova con 5-10 ml di soluzione M9 4 volte.
- Risospendere le uova in un piccolo volume di soluzione M9 e piastrle su piastre NGM seminate con batteri pipettando fino a 50 μl. Calcolare una stima approssimativa del numero di uova pipettando 3-5 μl di miscela di uova su una piastra e contando il numero di uova in questo volume. Aggiungere M9 per diluire il composto di uova fino a ottenere un numero numerabile di uova in questo volume.
NOTA: Si consiglia di placcare meno di 100 vermi per piastra poiché i vermi possono morire di fame prima del giorno 1 dell'età adulta quando ci sono troppi animali placcati. Per raccomandazioni più dettagliate per il numero di esemplari di piastra, fare riferimento a Castro Torres et al.20. - In alternativa, arrestare i nematodi con L1 per una sincronizzazione più stretta dell'età. Per l'arresto di L1, aggiungere il volume appropriato di soluzione M9 al pellet di uova in una provetta conica da 15 mL a un volume di 10-12 mL. Lasciare girare i vermi in un rotatore per 16-24 ore a 20 °C. Centrifugare (come al punto 1.2.7) e aspirare il surnatante. Calcolare la concentrazione di animali L1 e disporli su piastre NGM come descritto per le uova al punto 1.2.9.
- Invecchiare la popolazione di C. elegans all'età desiderata: trasferire gli animali L4/adulti su piastre contenenti 5-fluoro-2'-deossiuridina (FUDR) come descritto al punto 1.1.6 per prevenire la progenie ed eliminare la prole già prodotta. Per metodi alternativi per invecchiare i vermi senza FUDR, fare riferimento a Castro Torres et al.20.
NOTA: Alcuni fattori importanti devono essere considerati prima di scegliere lo sbiancamento come metodo per sincronizzare i vermi al loro primo stadio larvale. Come evidenziato in precedenza, la sovraesposizione alla candeggina può danneggiare gli embrioni, ridurre i tassi di schiusa e creare pressione selettiva per i ceppi resistenti alla candeggina, alterando potenzialmente la genetica della popolazione. Inoltre, se gli embrioni vengono lasciati non nutriti per lunghi periodi di tempo, l'esaurimento dei nutrienti può compromettere il metabolismo e lo sviluppo. Pertanto, i metodi di deposizione delle uova o NemaSync possono essere utilizzati come alternativa ai metodi di sbiancamento. Il metodo per la procedura di deposizione delle uova è descritto gradualmente nello studio citato da Castro Torres et al.20. NemaSync è un metodo commerciale più recente e più costoso, in cui viene utilizzato un sincronizzatore manuale di worm per sincronizzare i worm senza l'uso di sostanze chimiche21.
2. Imaging dei mitocondri in C. elegans
- Preparazione di vetrini per l'imaging di C. elegans
- Aggiungere 10-20 μl di soluzione M9 sui vetrini e trasferire il numero desiderato di vermi adulti all'età specifica nella soluzione sul vetrino. Si consigliano circa 25 vermi.
NOTA: L'uso di metodi tradizionali per paralizzare i vermi (ad esempio, tetramsolo o azide di sodio) può causare la frammentazione mitocondriale in determinate condizioni (vedi sotto). Invece, si consiglia l'uso di vetrini HistoBond, che hanno una superficie con una carica superficiale positiva permanente, ridurrà il movimento dei vermi senza l'uso di farmaci che possono introdurre artefatti. È importante inserire un rapporto appropriato tra vermi e soluzione M9, poiché volumi bassi possono causare lo schiacciamento degli animali tra il vetrino e il vetrino coprioggetti, mentre volumi elevati provocheranno il movimento degli animali durante l'acquisizione dell'immagine. Le raccomandazioni sono riportate nella Tabella 1. - In alternativa, per l'uso di farmaci che paralizzano i vermi, fare riferimento ai risultati rappresentativi di questo manoscritto per una gamma di concentrazioni e punti temporali che possono essere utilizzati con una frammentazione limitata dei mitocondri.
NOTA: Sebbene non vi sia stata una frammentazione importante negli animali sani di tipo selvatico, è importante ricordare che alcuni mutanti o condizioni possono sperimentare la frammentazione mitocondriale con la stessa concentrazione di questi farmaci, quindi sono necessari alcuni test quando si utilizzano questi farmaci. - Coprire i vermi in soluzione M9 sul vetrino con un vetro di copertura e utilizzare lo smalto per unghie per sigillare i lati e prevenire l'evaporazione della soluzione M9.
- Opzionale: per l'imaging muscolare, arrotolare i vermi spingendo delicatamente il vetrino coprioggetto. Ciò consente ai muscoli della parete corporea ai lati dei vermi di essere meglio allineati con il piano focale per immagini di qualità superiore.
- Aggiungere 10-20 μl di soluzione M9 sui vetrini e trasferire il numero desiderato di vermi adulti all'età specifica nella soluzione sul vetrino. Si consigliano circa 25 vermi.
- Imaging dei mitocondri utilizzando un microscopio composto
- Utilizzare un microscopio standard a campo largo per mitocondri muscolari e ipodermici (myo-3p::MLS::GFP e col-19p::MLS::GFP).
NOTA: È stato utilizzato un imager disponibile in commercio dotato di un obiettivo Plan Aprochromat 63x/1.4, filtro GFP (11525314), DFC9000 fotocamera GT e sorgente luminosa LED5 ed è stato utilizzato un software per microscopio compatibile. - Ottimizza le impostazioni di imaging per ogni microscopio e configurazione sperimentale.
NOTA: Tuttavia, come punto di partenza, i parametri per l'imaging con l'imager utilizzato in questo manoscritto sono i seguenti: le dimensioni sono 2048 (x) * 2048 (y) pixel a una dimensione del passo di 0,50 μm per i mitocondri ipodermici e "System Optimized" calcolato automaticamente dal software per i mitocondri muscolari. Il tempo di esposizione è di 50 ms per i mitocondri muscolari e di 50 ms per i mitocondri ipodermici. Il LED a 475 nm è stato impostato al 50% di potenza e il filtro a densità neutra è stato impostato al 30% per i muscoli e al 17% per i mitocondri ipodermici. La gamma Z inizia dal punto in cui il segnale GFP è visibile fino al punto in cui termina. Per ridurre al minimo il rumore in una proiezione massima, è possibile ridurre il numero di piani z utilizzati per la proiezione massima. - Per l'imaging dei mitocondri intestinali (vha-6p::MLS::GFP), utilizzare un microscopio confocale.
NOTA: L'imaging è stato eseguito su un microscopio confocale disponibile in commercio dotato di un obiettivo Plan ApoChromat 63x/1.4, laser a luce bianca (WLL), splitter di fascio acusto-ottico, rivelatori HyD S ed eseguito su software per microscopio. Come accennato in precedenza, si dovrebbe ottimizzare ogni singola configurazione sperimentale, ma come punto di partenza, i parametri per l'imaging con lo Stellaris utilizzato in questo studio sono i seguenti: La WLL è stata impostata all'85,00% di potenza massima e con una linea laser a 485 nm a un'intensità del 3,00%. Il rivelatore HyD S è stato impostato a 490-590 nm con un guadagno di 25 nell'impostazione analogica. Abbiamo scansionato unidirezionalmente un'area di 1024 (x) * 1024 (y) o 82,01 μm (x) * 82,01 μm (y) con 5 (z) pixel a un passo di 0,495 μm (la dimensione z è variabile a seconda delle dimensioni dei vermi o dei tessuti) con una velocità di scansione di 1.000 Hz, 2,25 zoom, media delle linee di 2 e foro stenopeico di 1 UA (95,5 μm). La gamma Z inizia dal punto in cui il segnale GFP è visibile fino al punto in cui termina. Per ridurre al minimo il rumore in una proiezione massima, è possibile ridurre il numero di piani z utilizzati per la proiezione massima.
- Utilizzare un microscopio standard a campo largo per mitocondri muscolari e ipodermici (myo-3p::MLS::GFP e col-19p::MLS::GFP).
- Quantificazione della morfologia mitocondriale
NOTA: Per i dettagli, fare riferimento a Schindelin et al.22 e vedere la Figura supplementare 1A-D.- Scarica e installa il software FIJI (Fiji è solo ImageJ, https://fiji.sc/). Quindi apri FIJI.
- Per scaricare e installare la macro MitoMAPR, scaricare il codice sorgente 1 da https://doi.org/10.7554/eLife.49158.033. Quindi copiare tutto il codice menzionato in "A.). Codice IJM per MitoMAPR-1.0." Quindi, apri Fiji, vai su Plugin > Nuova macro > e incolla il codice precedentemente copiato nella finestra della macro. Vai all'opzione File > Salva con nome e salva la macro per un uso futuro.
- Apri un'immagine microscopica 3D con Z-stack in FIJI trascinando e rilasciando i file sulla barra degli strumenti delle Fiji o andando su File > Apri.
- Crea una proiezione massima dell'immagine andando su Pile di immagini > > Progetto Z, quindi selezionando l'intervallo di sezioni con immagini a fuoco da proiettare al massimo e selezionando Intensità massima come tipo di proiezione.
- Salva l'immagine come TIFF andando su File > Salva come > TIFF.
- Ritaglia le regioni di interesse (ROI) da immagini complete utilizzando lo strumento "Rettangolo", disegnando un ROI e quindi andando su Immagine > Ritaglia.
- Salva l'immagine come TIFF andando su File > Salva come > TIFF.
- Esegui la macro MitoMAPR precedentemente salvata trascinando e rilasciando il file della macro sulla barra degli strumenti delle Fiji, quindi fai clic su Esegui. Una nuova finestra chiederà di selezionare la cartella in cui è stato salvato il file Tiff nella versione 2.3.6.
- Una nuova finestra chiederà di selezionare un'area; creare un rettangolo nell'immagine utilizzando lo strumento rettangolo, come indicato in precedenza nel passaggio 2.3.4, e premere Ok.
NOTA: L'intera immagine può essere selezionata se è stata precedentemente ritagliata come descritto nel passaggio 2.3.6. Salva la finestra "Dati" con tutti i valori relativi alla morfologia mitocondriale come file Excel andando su File > Salva con > Salva.
- Elaborazione batch di campioni utilizzando macro FIJI
- Creare una macro per specificare la regione delle immagini da analizzare. Apri Fiji e vai su Plugin > Nuova > Macro. Incollare il seguente codice nella casella di testo e salvare la macro come descritto in 2.3.2 (Figura supplementare 1E-H).
NOTA: Crea rettangolo (100, 100, 200, 200); corsa (Specificare..., larghezza = 100, altezza = 100, x = 100, y =100 in scala). - Crea una macro per salvare tutte le immagini aperte come file TIFF. All'interno della finestra della macro, vai su File > nuovo e incolla il seguente codice nella casella di testo. Salvare la macro come descritto in 2.3.2.
NOTA: dir = getDirectory(" Seleziona una cartella"); ids=newArray(nImmagini); per (i=0; i - Creare una macro per il batch MitoMAPR. Vai al https://doi.org/10.7554/eLife.49158.033 codice sorgente 1 e copia tutti i codici menzionati al punto B.). Codice IJM per MitoMAPR-1.0_Batch". All'interno della finestra della macro, passare a File > nuovo, incollare il codice dal codice sorgente 1 nella casella di testo e salvare la macro come descritto nel passaggio 2.3.2.
- Creare una macro per il ritaglio iterativo. Vai al https://doi.org/10.7554/eLife.49158.033 codice sorgente 1 e copia tutti i codici menzionati in C.) Codice IJM per CropR". All'interno della finestra della macro, vai su File > nuovo, incolla il codice dal codice sorgente 1 nella casella di testo e salva la macro come descritto nel passaggio 2.3.2.
- Per avviare l'elaborazione batch, salvare innanzitutto tutte le immagini da elaborare come TIFF aprendo tutte le immagini ed eseguendo la macro salvata nel passaggio 2.4.2. Tutte le immagini verranno salvate come TIFF nella cartella specificata. Chiudi tutte le immagini una volta salvate.
- Se le immagini salvate nel passaggio 2.4.5 sono immagini 3D (immagini contenenti z-stack), convertitele in immagini 2D trasformandole in proiezioni Z. Per eseguire il batch Z-project delle immagini, apri la finestra di elaborazione batch andando su Elabora > Batch > Macro nella finestra principale delle FIJI.
- Per "Input", selezionare la posizione della cartella contenente tutte le immagini 3D da elaborare da 2.4.5. Per "Output", selezionare la posizione desiderata per le immagini salvate dopo l'elaborazione. Seleziona il formato di output come TIFF. Incolla il seguente codice nella grande casella di testo della finestra e premi il pulsante di processo : run("Z Project...", "projection=[Max Intensity]").
- Ritaglia le ROI con dimensioni identiche da tutte le immagini 2D da elaborare utilizzando la macro di ritaglio iterativo del passaggio 2.4.4 in combinazione con la macro della regione specificata dal passaggio 2.4.3. Eseguire la macro di ritaglio iterativo dal passaggio 2.4.4. Apparirà una finestra che richiede una directory.
- Selezionare la cartella contenente solo le immagini 2D del passaggio 2.4.5 o le immagini 2D proiettate Z dal passaggio 2.4.6 e premere il pulsante Seleziona . La macro aprirà una delle immagini nella cartella selezionata e apparirà una finestra etichettata "effettua una selezione" con due pulsanti.
- Eseguire la macro di selezione specificata dal passaggio 2.4.3. Modificare i valori di larghezza e altezza nella casella di testo della macro in base alle esigenze per modificare la quota di selezione e premere Esegui per osservare la nuova selezione. La modifica dei valori X e Y nella macro modificherà la posizione nell'angolo superiore sinistro della selezione.
- Una volta che le dimensioni della selezione sono soddisfacenti, trascinare il rettangolo nell'area desiderata e quindi premere il pulsante OK nella finestra "effettua una selezione". La regione ritagliata verrà salvata nella cartella selezionata al punto 2.4.7.
- La macro scorrerà tutte le immagini rimanenti nella cartella. Per ogni immagine, eseguire la macro dal passaggio 2.4.8 senza modificare i valori delle dimensioni per assicurarsi che le stesse dimensioni vengano ritagliate per tutte le immagini. Per ogni immagine, trascinare il rettangolo di selezione nella posizione desiderata e premere il pulsante OK nella finestra "effettua una selezione".
- Aprire la cartella contenente le immagini ritagliate dai passaggi 2.4.8-2.4.9 e spostare tutte le immagini ritagliate in una nuova cartella.
- Analizzare tutte le immagini ritagliate eseguendo la macro batch MitoMAPR dal passaggio 2.4.3. Una nuova finestra ti chiederà di selezionare una directory. Seleziona la cartella con tutte le immagini ritagliate dal passaggio 2.4.10. Una volta completato, apparirà una finestra chiamata "Dati" con tutti i valori relativi alla morfologia mitocondriale. Salvare tutti i dati come file Excel come descritto nel passaggio 2.3.9.
- Creare una macro per specificare la regione delle immagini da analizzare. Apri Fiji e vai su Plugin > Nuova > Macro. Incollare il seguente codice nella casella di testo e salvare la macro come descritto in 2.3.2 (Figura supplementare 1E-H).
Risultati
C. elegans è un ottimo modello per l'imaging mitocondriale grazie al suo corpo trasparente, che consente un facile imaging di vermi interi e animali vivi senza un'eccessiva preparazione del campione. Inoltre, la morfologia mitocondriale in diversi tessuti può essere facilmente visualizzata utilizzando promotori tessuto-specifici per esprimere proteine fluorescenti mirate ai mitocondri. Qui, myo-3p (per i mitocondri muscolari), vha-6p (per i mitocondri intestinali) e col-19p (per i mitocondri ipodermici) sono stati utilizzati per guidare l'espressione di una GFP (sequenza di localizzazione mitocondriale della proteina ATP-1) mirata ai mitocondri. I mitocondri dei muscoli della parete corporea di C. elegans mostrano una morfologia tubulare, allineandosi lungo le fibre muscolari (miofibrille) (Figura 1A); i mitocondri intestinali mostravano strutture altamente interconnesse, simili a ragnatele, con un allineamento meno uniforme (Figura 1B); e l'ipoderma ha mitocondri tubulari che appaiono più arrotondati o di forma ovale rispetto all'intestino o al muscolo (Figura 1C). La morfologia mitocondriale è generalmente coerente per tutta la lunghezza del verme nel muscolo e nell'intestino, ma l'ipoderma mostra alcune differenze minori nell'interconnessione dei mitocondri tra le estremità prossimale e distale del verme. Pertanto, per ottenere risultati sperimentali riproducibili, si consiglia di concentrarsi su una regione specifica del loro corpo. Qui, l'area tra la faringe e la vulva ~100-200 μm sotto la faringe è costantemente visualizzata.
Per i mitocondri muscolari e ipodermici, l'imaging al microscopio composto fornisce una risoluzione sufficiente a causa della piattezza delle cellule e dei bassi livelli di luce sfocata. Tuttavia, i mitocondri intestinali sono difficili da osservare con un microscopio composto, poiché il grande volume dell'intestino limita la risoluzione a causa di una grande quantità di luce sfocata proveniente da altre sezioni dell'intestino (Figura 2). Pertanto, si consiglia di visualizzare i mitocondri intestinali con un microscopio confocale per ridurre la luce sfocata e visualizzare la corretta morfologia mitocondriale.
La forma dei mitocondri è altamente dinamica e cambia in base all'ambiente metabolico degli animali23 o anche a causa dell'esposizione a rigidezze variabili del substrato24. Pertanto, lasciare gli animali sui vetrini del microscopio per lunghi periodi di tempo in assenza di una fonte di cibo e sul substrato rigido di vetro può potenzialmente influire sulla morfologia mitocondriale. Qui, questo studio ha scoperto che i mitocondri dei vermi si frammentano dopo circa 30 minuti su un vetrino in soluzione M9, con i mitocondri ipodermici che mostrano la maggiore frammentazione (Figura 3). Pertanto, l'imaging dei mitocondri deve essere eseguito rapidamente dopo la preparazione del campione.
Le sostanze chimiche che limitano la mobilità dei vermi, tra cui l'azide di sodio e il tetramisolo, sono comunemente utilizzate per l'imaging dal vivo di C. elegans, poiché i vermi stazionari sono necessari per catturare più sezioni z di animali per l'imaging 3D. Studi precedenti hanno dimostrato che l'esposizione all'azide di sodio o al tetramisolo può provocare la frammentazione mitocondriale25. Sorprendentemente, è stato scoperto che l'azide di sodio, anche ad alte concentrazioni, ha un impatto limitato sulla morfologia mitocondriale nel muscolo o nell'intestino. Tuttavia, l'ipoderma mostrava una frammentazione mitocondriale più precoce rispetto ai controlli M9 (Figura 3). È importante sottolineare che alte concentrazioni di tetramisolo (100 mM) hanno provocato una significativa frammentazione mitocondriale subito dopo l'esposizione in tutti i tipi di cellule. In confronto, le concentrazioni medie (10 mM) hanno portato a una frammentazione più rapida rispetto a un controllo M9. Basse concentrazioni (1 mM) hanno avuto un impatto limitato sulla morfologia mitocondriale. Questi dati suggeriscono che l'utilizzo dell'azide di sodio può essere un'opzione praticabile per l'imaging rapido dei mitocondri, mentre il tetramisolo dovrebbe essere generalmente evitato.
Come previsto, i mitocondri di tutti i tessuti di C. elegans mostrano frammentazione durante il naturale processo di invecchiamento (Figura 4A). La frammentazione può essere visualizzata come mitocondri che si presentano come strutture più tronche e sferiche, che sono notevolmente diverse dai mitocondri lineari e tubolari mostrati negli animali giovani. È importante notare che l'intestino aumenta nelle strutture sferiche autofluorescenti in età avanzata, quindi è necessario prestare attenzione a non confondere l'autofluorescenza con le strutture mitocondriali effettive. Poiché può esserci variabilità dei cambiamenti nella struttura mitocondriale tra i vermi, è importante eseguire la quantificazione della morfologia mitocondriale su un campione di dimensioni significative piuttosto che limitarsi a visualizzare alcuni vermi. In questo caso è stato utilizzato mitoMAPR, che consente la quantificazione automatizzata della morfologia mitocondriale utilizzando un insieme diversificato di metriche, tra cui oggetti, reti, giunzioni per rete, punti di giunzione, lunghezza dell'oggetto, impronta mitocondriale, copertura mitocondriale e area dell'oggetto (Tabella supplementare 1 e Tabella supplementare 2). L'automazione elimina i pregiudizi soggettivi dall'utente. Qui, riportiamo che la metrica della lunghezza dell'oggetto è ottimale per misurare quantitativamente i cambiamenti nella morfologia mitocondriale muscolare e intestinale durante l'invecchiamento e le metriche dei punti di giunzione per misurare i cambiamenti nella morfologia mitocondriale ipodermica durante l'invecchiamento (Figura 4B).
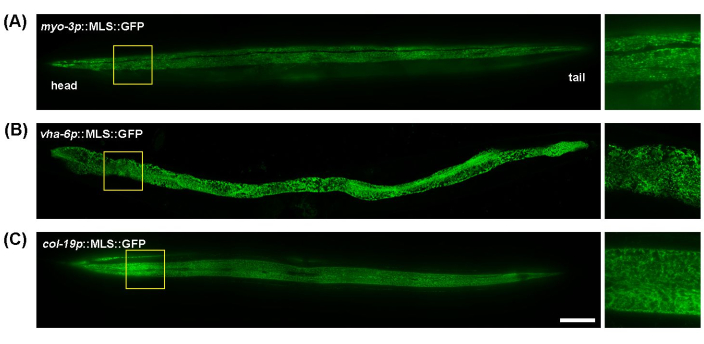
Figura 1: Immagini dei mitocondri in tutto il corpo di C. elegans. L'imaging mitocondriale è stato eseguito per l'intera durata del giorno 5 animali adulti cresciuti in EV dallo stadio L1 in animali transgenici con espressione di MLS::GFP nel muscolo (A), nell'intestino (B) e nell'ipoderma (C). Barra di scala: 100 μm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Confronto dell'imaging mitocondriale tra microscopio composto e microscopio confocale. L'imaging mitocondriale è stato eseguito in animali adulti al giorno 1 cresciuti in EV dallo stadio L1 in animali transgenici con espressione di MLS::GFP nel muscolo (A), nell'intestino (B) e nell'ipoderma (C). Barra di scala: 5 μm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Valutazione della frammentazione mitocondriale di diversi tessuti dopo il trattamento con M9, azoturo di sodio e tetramisolo. L'imaging mitocondriale è stato eseguito in animali adulti al giorno 1 con espressione di MLS::GFP nel muscolo, nell'intestino e nell'ipoderma. Gli animali sono stati allevati con EV a partire dallo stadio L1. Gli animali sono stati posizionati su vetrini contenenti M9, azoturo di sodio (1 mM, 10 mM e 100 mM) o tetramisolo (1 mM, 10 mM e 100 mM) e l'imaging è stato eseguito immediatamente dopo la preparazione del vetrino (0 min) o 15 min o 30 min dopo la preparazione del vetrino. Immagini rappresentative sono mostrate per n > 5 animali per ceppo per 2 repliche biologiche. Barre di scala: 5 μm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Imaging mitocondriale di C. elegans durante l'invecchiamento in diversi tessuti e quantificazione della morfologia mitocondriale utilizzando MitoMAPR. (A) L'imaging mitocondriale è stato eseguito in animali adulti nei giorni 1, 5, 9, 13 e 15 dell'età adulta con espressione di MLS::GFP nel muscolo, nell'intestino e nell'ipoderma. Gli animali sono stati allevati con EV dallo stadio L1 e spostati su piastre EV contenenti FUDR dallo stadio adulto del giorno 1. Le immagini sono rappresentative di n ≥ 5 animali per ceppo per ≥ 3 repliche biologiche. Barra di scala: 5 μm. (B) La quantificazione della lunghezza dell'oggetto dei mitocondri muscolari e intestinali dei vermi nei giorni 1, 5, 9, 13 e 15 e la quantificazione dei punti di giunzione dei mitocondri ipodermici dei vermi nei giorni 1, 5, 9, 13 e 15. Tutti i singoli punti dati combinati con la media ± SD. I grafici sono stati tracciati e analizzati statisticamente utilizzando un test t di Student. ns = non significativo, *p < 0,03; **p < 0,002; p < 0,0002; p < 0,0001. Clicca qui per visualizzare una versione più grande di questa figura.
| Giorni di vermi | Numero di vermi | Volume del tampone (mL) |
| 1 | 25 | 13 |
| 5 | 25 | 14 |
| 9 | 20 | 15 |
| 13 | 20 | 15 |
Tabella 1: Numero raccomandato di worm e volumi di buffer durante la preparazione dei vetrini.
Figura 1 supplementare: Flussi di lavoro dell'applicazione di una quantificazione di una singola immagine utilizzando MitoMAPR e l'elaborazione batch per più immagini. (A) Flusso di lavoro per quantificare la morfologia mitocondriale da una singola immagine utilizzando la macro MitoMAPR. (B) Proiezione Z di file di immagine 3D grezza utilizzando Fiji (proiezione massima). (C) Ritaglio della regione di interesse dall'immagine Z proiettata. (D) Immagini dello Skeletor acquisite dalla macro MitoMAPR. Barre di scala: 5 μm. (E) Flusso di lavoro per quantificare la morfologia mitocondriale da più immagini utilizzando il processo batch utilizzando la macro MitoMAPR. (F) Screenshot della macro di ritaglio. (G) Screenshot di un processo batch per la proiezione Z. (H) Screenshot della finestra di conferma della selezione della macro di ritaglio batch. Clicca qui per scaricare questo file.
Tabella supplementare 1: Analisi quantitativa delle metriche della morfologia mitocondriale nel tessuto muscolare di C. elegans . Le metriche registrate includono oggetti, reti, giunzioni per rete, punti di giunzione, lunghezza dell'oggetto, impronta mitocondriale, area dell'oggetto e copertura mitocondriale, misurate in giorni diversi. I dati forniscono una base per valutare i cambiamenti nella morfologia mitocondriale con l'invecchiamento. Clicca qui per scaricare questo file.
Tabella supplementare 2: Effetti delle sostanze chimiche immobilizzanti sulla morfologia mitocondriale in C. elegans. I dati includono misurazioni come oggetti, reti, giunzioni per rete, punti di giunzione, lunghezza dell'oggetto, impronta mitocondriale, area dell'oggetto e copertura mitocondriale in concentrazioni e punti temporali variabili dopo la preparazione del campione con tampone M9 o diverse concentrazioni di tetramsolo e sodio azide. Clicca qui per scaricare questo file.
Discussione
L'imaging fluorescente della morfologia mitocondriale è il modo più comune per determinare i cambiamenti nei mitocondri. Mentre le tecniche di microscopia avanzate come la microscopia elettronica a trasmissione (TEM), la microscopia a forza atomica e la microscopia crioelettronica offrono una risoluzione più elevata, la microscopia a fluorescenza rimane più economica e accessibile alla maggior parte dei ricercatori. Inoltre, la microscopia fluorescente può essere eseguita in cellule vive e in organismi modello trasparenti come C. elegans, l'imaging può essere eseguito in animali interi26,27. L'imaging di C. elegans transgenico è abbastanza semplice e i marcatori fluorescenti geneticamente codificati consentono un imaging più affidabile e robusto dei mitocondri poiché non richiede un'elaborazione complessa del campione né soffre di colorazione variabile tra i tipi di cellule o condizioni da coloranti mitocondriali convenzionali come MitoTracker o TMRE 28,29,30. I marcatori fluorescenti geneticamente codificati di solito comportano l'etichettatura diretta delle proteine mitocondriali con fluorofori o la coniugazione di fluorofori con una sequenza di localizzazione mitocondriale minima. Questi costrutti sono spesso guidati da promotori tessuto-specifici, che consentono la visualizzazione dei mitocondri in diversi tessuti come i muscoli, l'intestino o i tessuti dell'ipoderma31. Generalmente, queste proteine marcate in fluorescenza sono sovraespresse, il che può avere effetti fisiologici potenzialmente indesiderati se le proteine mitocondriali a lunghezza intera sono sovraespresse; pertanto, le sequenze MLS minime sono un'opzione migliore32. Tuttavia, anche con la sovraespressione di fusioni minime di proteine MLS-fluorescenti, l'elevata sovraespressione dovrebbe essere evitata, poiché l'importazione di una grande quantità di proteine nei mitocondri può far collassare il potenziale della membrana mitocondriale e avere un impatto sulla salute degli animali33. Mentre una caratterizzazione esaustiva di tutti i ceppi di C. elegans attualmente disponibili va oltre lo scopo di questo manoscritto, un'analisi comparativa dettagliata di numerosi reporter mitocondriali e i pro e i contro di ciascuno di essi può essere trovata qui31.
Per l'imaging di cellule vive dei mitocondri in C. elegans, la microscopia composta standard o a campo largo può essere un'opzione preferita a causa dell'elevata velocità e facilità di questi metodi rispetto alla microscopia confocale. In questo studio, è dimostrato che le cellule piatte come il muscolo e l'ipoderma beneficiano minimamente della microscopia confocale e la microscopia composta consente l'acquisizione con una risoluzione sufficiente per visualizzare la corretta morfologia mitocondriale. Le cellule più grandi come l'intestino rendono difficile la microscopia composta a causa della luce sfocata. Pertanto, la microscopia confocale è necessaria per un imaging affidabile della morfologia mitocondriale intestinale.
Una considerazione importante per l'imaging 3D sull'intero spessore del tessuto negli animali vivi è quella di prevenire il movimento dei vermi durante l'acquisizione dell'immagine. I ricercatori usano spesso metodi per paralizzare i vermi, come il tetramsolo o l'azide di sodio34. L'azide di sodio inibisce la citocromo c ossidasi (complesso IV), un enzima critico nella catena di trasporto degli elettroni dei mitocondri, portando alla paralisi generale a causa della mancanza di ATP necessario per le contrazioni muscolari e altre funzioni cellulari34,35. Il tetramsolo agisce imitando l'acetilcolina alle giunzioni neuromuscolari, causando una depolarizzazione persistente e una contrazione muscolare36. Tuttavia, l'esposizione a questi farmaci può indurre la frammentazione mitocondriale causando stress ossidativo23. In questo studio, è stato riscontrato che il tetramsolo induceva molto rapidamente la frammentazione mitocondriale, ma l'azide di sodio aveva un effetto molto più limitato.
C. elegans offre un modo molto semplice e facile per studiare l'impatto dell'invecchiamento sulla morfologia mitocondriale grazie alla sua breve durata di vita e alla facilità di invecchiamento degli animali. In questo caso, abbiamo optato per l'esposizione al FUDR, che è un metodo robusto per sterilizzare chimicamente gli animali impedendo la replicazione del DNA 20,37,38. Tuttavia, la FUDR può avere effetti indesiderati su specifici parametri di invecchiamento e, per coloro che sono preoccupati per gli effetti fuori bersaglio della FUDR, possono essere utilizzate altre strategie per rimuovere la progenie39. Ad esempio, ci sono mutanti sterili, tra cui il mutante germinale glp-4 sensibile alla temperatura, o mutanti carenti di spermatozoi, come CF512 40,41,42. In alternativa, gli animali possono anche essere invecchiati naturalmente raccogliendo manualmente gli adulti dalla loro progenie ogni giorno.
Anche la quantificazione dei cambiamenti nella morfologia mitocondriale è una considerazione molto importante, poiché potrebbe esserci una significativa variabilità nella morfologia mitocondriale tra gli animali. Pertanto, l'esecuzione di analisi quantitative e statistiche su un campione di dimensioni sufficienti è essenziale per trarre conclusioni significative sui cambiamenti della morfologia mitocondriale. Tuttavia, l'analisi e la quantificazione delle immagini possono soffrire notevolmente di distorsioni soggettive e sfide derivanti dall'imaging di strutture altamente complesse come i mitocondri interconnessi. A tal fine, ecco un riassunto di un metodo automatizzato per quantificare la morfologia mitocondriale utilizzando numerose metriche sviluppate dal laboratorio Mair. MitoMAPR consente la quantificazione oggettiva e automatizzata della morfologia mitocondriale, misurando vari aspetti dei mitocondri, tra cui la rete mitocondriale, la lunghezza dell'oggetto, la distribuzione, la copertura della rete e l'impronta mitocondriale. MitoMAPR è una macro gratuita per ImageJ ed è quindi disponibile per tutti i laboratori con un computer funzionante. Un aspetto importante dell'utilizzo di MitoMAPR è quello di eseguire la quantificazione su un campione di grandi dimensioni per determinare quale metrica della morfologia mitocondriale è più robusta per determinare i cambiamenti nelle condizioni sperimentali testate43,44. Qui, si è scoperto che la lunghezza dell'oggetto e i punti di giunzione sono le migliori metriche per determinare i cambiamenti durante l'invecchiamento nel muscolo, nell'intestino e nell'ipoderma. Un approccio alternativo per analizzare i cambiamenti nella morfologia mitocondriale è la generazione di rappresentazioni 3D dei mitocondri da immagini z-stack, seguite dall'analisi 3D 16,45. Ciò può essere ottenuto utilizzando software disponibili in commercio, come Image-Pro Plus con i moduli SharpStack Total Deconvolution e 3D Constructor. È stato dimostrato che la quantificazione delle rappresentazioni mitocondriali 3D fornisce informazioni più accurate sulla forma mitocondriale e sulle proprietà della rete46. Tuttavia, questo metodo è tecnicamente più complesso e costoso rispetto all'approccio semi-3D, che prevede la compressione di più sezioni di immagine da uno z-stack a campo largo o confocale in un'unica proiezione 2D e l'analisi con strumenti come mitoMAPR. Questo studio riassume l'uso dell'espressione a copia singola della GFP mirata alla matrice mitocondriale e descrive in dettaglio alcune insidie da evitare durante l'imaging.
Limitazioni e considerazioni temporali
Sebbene la microscopia confocale sia raccomandata per tessuti grandi e spessi, come il tessuto intestinale, le tecniche standard di microscopia confocale a scansione lineare possono essere troppo lente per eseguire l'imaging rapido dei mitocondri. Ciò è particolarmente vero considerando i nostri dati che mostrano che tenere i vermi sui vetrini per lunghi periodi di tempo può provocare la frammentazione dei mitocondri. Sebbene la tecnologia della microscopia confocale sia progredita in modo significativo, i microscopi in grado di eseguire immagini rapide, come il disco rotante, l'Airiscan o quello utilizzato in questo studio, possono essere proibitivi per alcuni laboratori. In questi casi, la microscopia composta può essere combinata con metodi di clearance computazionale per rimuovere la luce sfocata, come la deconvoluzione47.
Inoltre, come descritto, MitoMAPR è una potente macro automatizzata per misurare quantitativamente la lunghezza dei mitocondri e l'interconnessione della rete mitocondriale. Tuttavia, dovrebbe essere utilizzato con attenzione considerando le limitazioni qui elencate. In primo luogo, l'utilizzo di una GFP mirata alla matrice osserva solo la frammentazione mitocondriale della membrana interna, che potrebbe non ricapitolare completamente la morfologia mitocondriale della membrana esterna, poiché gli eventi di fissione della membrana interna possono verificarsi in assenza di fissione della membrana esterna. Pertanto, per un imaging più raffinato di entrambe le membrane, è necessario utilizzare sia un fluoroforo mirato alla matrice che un fluoroforo mirato alla membrana esterna mitocondriale. Poiché i marcatori della membrana mitocondriale esterna possono subire le stesse conseguenze delle proteine localizzate nella matrice se altamente sovraespressi, si raccomanda l'uso di marcatori a copia singola, in particolare quelli che utilizzano sequenze di localizzazione mitocondriale minime invece di proteine complete come quelle utilizzate qui31.
Come descritto nella Figura 3, i mitocondri possono frammentarsi rapidamente al microscopio e diversi metodi per preparare i vetrini dei campioni possono influenzare in larga misura la morfologia a seconda del tampone utilizzato. I dati rappresentativi descritti in questo manoscritto mostrano che i mitocondri dei vermi subiscono una frammentazione minima sotto M9 e basse concentrazioni di sodio azide. Il tetramisolo, un'altra sostanza chimica ampiamente utilizzata per paralizzare i vermi, frammenta rapidamente il mitocondriale, indicando che il suo uso dovrebbe essere generalmente evitato. Sebbene M9 e l'azide di sodio non abbiano mostrato una frammentazione significativa dei mitocondri, è importante notare che ceppi diversi potrebbero rispondere in modo diverso rispetto ai risultati mostrati qui. Mentre altri studi hanno dimostrato che l'azide di sodio può frammentare i mitocondri, è possibile che i nostri ceppi non mostrino una frammentazione mitocondriale significativa con l'esposizione all'azide di sodio a causa del basso livello di espressione dei nostri costrutti. Un'elevata espressione di proteine localizzate nei mitocondri può far collassare il potenziale di membrana e, quindi, diversi ceppi utilizzati in altri studi possono rendere i mitocondri più suscettibili alla frammentazione dalle stesse concentrazioni di sodio azide utilizzate in questo studio. Indipendentemente da ciò, è necessario prestare attenzione per garantire che le sostanze chimiche che paralizzano i vermi non causino la frammentazione mitocondriale in condizioni specifiche da testare prima dell'uso in tutti gli studi, poiché mutanti o condizioni specifiche possono essere più suscettibili alla frammentazione mitocondriale indotta da farmaci. Inoltre, anche mantenere i vermi in M9 può comportare cambiamenti nella loro morfologia e attività mitocondriale naturale, poiché è stato precedentemente dimostrato che la loro attività di nuoto nel tampone M9 influenza la fissione mitocondriale e le dinamiche di fusione. Inoltre, lasciare i vermi in M9 per lunghi periodi di tempo può attivare risposte all'ipossia che influenzano significativamente i mitocondri causando interruzioni della proteostasi mitocondriale48,49.
Infine, un'altra considerazione importante è che, mentre i cambiamenti nella morfologia mitocondriale sono spesso correlati con i cambiamenti nella funzione mitocondriale, non sempre c'è una correlazione diretta tra i due. Pertanto, si raccomanda un'analisi più approfondita della funzione mitocondriale. Ad esempio, il tasso di consumo di ossigeno può essere misurato utilizzando uno strumento Seahorse50, il potenziale di membrana mitocondriale può essere misurato utilizzando coloranti potenziali di membrana come JC9 o TMRE51, lo stato ossidativo mitocondriale può essere misurato utilizzando coloranti sensibili alla redox come roGFP52 e la resilienza allo stress mitocondriale può essere misurata utilizzando la sensibilità a fattori di stress come il rotenone53. Poiché l'imaging dei mitocondri può essere piuttosto rapido, offriamo i metodi qui come primo passaggio facile per determinare se le condizioni sperimentali influenzano la morfologia mitocondriale. Questi metodi sono adatti anche per lo screening su larga scala di farmaci o geni, con il follow-up di un'analisi mitocondriale più approfondita utilizzando metriche aggiuntive raccomandate. Nel complesso, qui esaminiamo quelli che si ritiene essere i metodi più semplici per visualizzare la morfologia mitocondriale in C. elegans con errori sperimentali minimi.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
J.K. è supportato dalla USC Provost Fellowship; M.A. e G.G. sono supportati da T32AG052374; M.V. è supportato da 1R25AG076400; e R.H.S. è supportato da R01AG079806 del National Institute on Aging e 2022-A-010-SUP della Larry L. Hillblom Foundation. Alcuni ceppi sono stati forniti dal CGC, che è finanziato dalla sovvenzione P40 OD010440 dell'Office of Research Infrastructure Programs del NIH. Alcune analisi genetiche sono state eseguite utilizzando Wormbase, che è finanziato da una sovvenzione U41 HG002223.
Materiali
| Name | Company | Catalog Number | Comments |
| 5-fluoro-2'-deoxyuridine (FUDR) | Spectrum Chemical | F2026-10GMBL | for proliferation inhibition |
| APEX IPTG | Genesee | 18-242 | for RNAi |
| Bacto Agar | VWR | 90000-764 | for NGM plates |
| Bacto Peptone | VWR | 97064-330 | for NGM plates |
| Calcium chloride dihydrate | VWR | 97061-904 | for NGM plates |
| Carbenicillin | VWR | 76345-522 | for RNAi |
| Cholesterol | VWR | 80057-932 | for NGM plates |
| Confocal microscope | Stellaris 5 | ||
| DMSO | VWR | BDH1115-1LP | solvent for drugs |
| HistoBond microscope slides | VWR | 16005-110 | for slides preparation |
| LB Broth | VWR | 95020-778 | for LB |
| LEICA S7E Dissecting Scope | Leica | 10450840 | Standard dissecting microscope |
| LEICA STELLARIS 5 | Leica | 158101100 | Confocal Microscope |
| LEICA THUNDER | Leica | 11525679 | Compound Microscope |
| Magnesium sulfate heptahydrate | VWR | 97062-132 | for NGM plates, M9 |
| pL4440 empty vector plasmid | addgene | plasmid #1654 | for empty vector plasmid |
| Potassium Chloride | VWR | 97061-566 | for bleach soluton |
| Potassium phosphate dibasic | VWR | EM-PX1570-2 | for NGM plates |
| Potassium phosphate monobasic | VWR | EM-PX1565-5 | for M9 |
| Sodium azide | VWR | 97064-646 | for paralyzing worms |
| Sodium Chloride | VWR | EM-SX0420-5 | for NGM plates, M9 |
| Sodium hypochlorite | VWR | RC7495.7-32 | for bleach solution |
| Sodium phosphate dibasic | VWR | 71003-472 | for M9 |
| Tetracycline hydrochloride | VWR | 97061-638 | for RNAi |
| WOB-L® Dry Vacuum Pumps, Standard-Duty, Welch® | VWR | 80077-612 | for aspiration |
Riferimenti
- Protasoni, M., Zeviani, M. Mitochondrial structure and bioenergetics in normal and disease conditions. Int J Mol Sci. 22 (2), 586 (2021).
- Wang, Y., et al. The role of mitochondrial dynamics in disease. MedComm. 4 (6), e462 (2023).
- Kamer, K. J., Mootha, V. K. The molecular era of the mitochondrial calcium uniporter. Nat Rev Mol Cell Biol. 16 (9), 545-553 (2015).
- Chen, W., Zhao, H., Li, Y. Mitochondrial dynamics in health and disease: mechanisms and potential targets. Signal Transduct Target Ther. 8 (1), 333 (2023).
- Adebayo, M., Singh, S., Singh, A. P., Dasgupta, S. Mitochondrial fusion and fission: The fine-tune balance for cellular homeostasis. FASEB J. 35 (6), e21620 (2021).
- Madan, S., Uttekar, B., Chowdhary, S., Rikhy, R. Mitochondria lead the way: mitochondrial dynamics and function in cellular movements in development and disease. Front Cell Dev Biol. 9, 781933 (2021).
- Sharma, A., Smith, H. J., Yao, P., Mair, W. B. Causal roles of mitochondrial dynamics in longevity and healthy aging. EMBO Rep. 20 (12), e48395 (2019).
- Rolland, S. G., Lu, Y., David, C. N., Conradt, B. The BCL-2-like protein CED-9 of C. elegans promotes FZO-1/Mfn1,2- and EAT-3/Opa1-dependent mitochondrial fusion. J Cell Biol. 186 (4), 525-540 (2009).
- Labrousse, A. M., Zappaterra, M. D., Rube, D. A., van der Bliek, A. M. C. elegans dynamin-related protein DRP-1 controls severing of the mitochondrial outer membrane. Mol Cell. 4 (5), 815-826 (1999).
- Dickinson, D. J., Pani, A. M., Heppert, J. K., Higgins, C. D., Goldstein, B. Streamlined genome engineering with a self-excising drug selection cassette. Genetics. 200 (4), 1035-1049 (2015).
- Bosher, J. M., Labouesse, M. RNA interference: Genetic wand and genetic watchdog. Nat Cell Biol. 2 (2), E31-E36 (2000).
- Yu, C. C. J., et al. Expansion microscopy of C. elegans. eLife. 9, e46249 (2020).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Front Endocrinol. 11, 554994 (2020).
- Dingley, S., et al. Mitochondrial respiratory chain dysfunction variably increases oxidant stress in Caenorhabditis elegans. Mitochondrion. 10 (2), 125-136 (2010).
- Presley, A. D., Fuller, K. M., Arriaga, E. A. MitoTracker Green labeling of mitochondrial proteins and their subsequent analysis by capillary electrophoresis with laser-induced fluorescence detection. J Chromatogr B Anal Technol Biomed Life Sci. 793 (1), 141-150 (2003).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Curr Protoc Cell Biol. Chapter 4, Unit 4.25.1-Unit 4.25.21 (2010).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. Novel imaging tools to study mitochondrial morphology in Caenorhabditis elegans. Life Sci Alliance. 7 (11), e202402918 (2024).
- Benedetti, C., Haynes, C. M., Yang, Y., Harding, H. P., Ron, D. Ubiquitin-like protein 5 positively regulates chaperone gene expression in the mitochondrial unfolded protein response. Genetics. 174 (1), 229-239 (2006).
- Begelman, D. V., et al. An aco-2::gfp knock-in enables the monitoring of mitochondrial morphology throughout C. elegans lifespan. microPublication Biol. 2022, (2022).
- Castro Torres, T., et al. Surveying low-cost methods to measure lifespan and healthspan in Caenorhabditis elegans. J Vis Exp. 183, e64091 (2022).
- Rasmussen, N. R., Reiner, D. J. Nuclear translocation of the tagged endogenous MAPK MPK-1 denotes a subset of activation events in C. elegans development. J Cell Sci. 134 (17), jcs258456 (2021).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Luz, A. L., et al. Mitochondrial morphology and fundamental parameters of the mitochondrial respiratory chain are altered in Caenorhabditis elegans strains deficient in mitochondrial dynamics and homeostasis processes. PLoS One. 10 (6), e0130940 (2015).
- Oorloff, M., et al. Mechanical stress through growth on stiffer substrates impacts animal health and longevity in C. elegans. bioRxiv. , (2024).
- Chu, X., Wu, S., Raju, R. NLRX1 regulation following acute mitochondrial injury. Front Immunol. 10, 2431 (2019).
- Shah, P., Bao, Z., Zaidel-Bar, R. Visualizing and quantifying molecular and cellular processes in Caenorhabditis elegans using light microscopy. Genetics. 221 (4), iyac068 (2022).
- Wang, Y., Wang, P., Li, C. Fluorescence microscopic platforms imaging mitochondrial abnormalities in neurodegenerative diseases. Adv Drug Deliv Rev. 197, 114841 (2023).
- Ding, J., et al. An expanded GCaMP reporter toolkit for functional imaging in Caenorhabditis elegans. G3 (Bethesda, Md). 13 (10), jkad183 (2023).
- Sarasija, S., Norman, K. R. Analysis of mitochondrial structure in the body wall muscle of Caenorhabditis elegans. Bio-Protocol. 8 (7), e2801 (2018).
- Chaweeborisuit, P., Suriyonplengsaeng, C., Suphamungmee, W., Sobhon, P., Meemon, K. Nematicidal effect of plumbagin on Caenorhabditis elegans: A model for testing a nematicidal drug. Z Naturforsch C J Biosci. 71 (5-6), 121-131 (2016).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. . Novel imaging tools to study mitochondrial dynamics in Caenorhabditis elegans. 7, 11 (2024).
- Bolognesi, B., Lehner, B. Reaching the limit. eLife. 7, e39804 (2018).
- Jishi, A., Qi, X. Altered mitochondrial protein homeostasis and proteinopathies. Front Mol Neurosci. 15, 867935 (2022).
- Morton, K. S., Wahl, A. K., Meyer, J. N. The effect of common paralytic agents used for fluorescence imaging on redox tone and ATP levels in Caenorhabditis elegans. PLoS One. 19 (4), e0292415 (2024).
- Noumi, T., Maeda, M., Futai, M. Mode of inhibition of sodium azide on H+-ATPase of Escherichia coli. FEBS Lett. 213 (2), 381-384 (1987).
- Siete, C., Xiong, R., Khalid, A., Hsieh, Y. -. W., Chuang, C. -. F. Immobilization of C. elegans with different concentrations of an anesthetic for time-lapse imaging of dynamic protein trafficking in neurons. microPublication Biology. 2024, (2024).
- Mitchell, D. H., Stiles, J. W., Santelli, J., Sanadi, D. R. Synchronous growth and aging of Caenorhabditis elegans in the presence of fluorodeoxyuridine. J Gerontol. 34 (1), 28-36 (1979).
- Santi, D. V., McHenry, C. S. 5-Fluoro-2'-deoxyuridylate: covalent complex with thymidylate synthetase. Proc Natl Acad Sci U S A. 69 (7), 1855-1857 (1972).
- Wang, H., Zhao, Y., Zhang, Z. Age-dependent effects of floxuridine (FUdR) on senescent pathology and mortality in the nematode Caenorhabditis elegans. Biochem Biophys Res Commun. 509 (3), 694-699 (2019).
- Beanan, M. J., Strome, S. Characterization of a germ-line proliferation mutation in C. elegans. Development (Cambridge, England). 116 (3), 755-766 (1992).
- Austin, J., Kimble, J. glp-1 is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans. Cell. 51 (4), 589-599 (1987).
- Garigan, D., et al. Genetic analysis of tissue aging in Caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Zhang, Y., et al. Neuronal TORC1 modulates longevity via AMPK and cell nonautonomous regulation of mitochondrial dynamics in C. elegans. eLife. 8, e49158 (2019).
- Dutta, N., et al. Investigating impacts of the mycothiazole chemotype as a chemical probe for the study of mitochondrial function and aging. GeroScience. 46 (6), 6009-6028 (2024).
- Tronstad, K., et al. Regulation and quantification of cellular mitochondrial morphology and content. Curr Pharm Des. 20 (35), 5634-5652 (2014).
- Nikolaisen, J., et al. Automated quantification and integrative analysis of 2D and 3D mitochondrial shape and network properties. PLoS One. 9 (7), e101365 (2014).
- Lee, J. S., Wee, T. L. E., Brown, C. M. Calibration of wide-field deconvolution microscopy for quantitative fluorescence imaging. J Biomol Tech. 25 (1), 31-40 (2014).
- Hong, M., Kwon, J. Y., Shim, J., Lee, J. Differential hypoxia response of hsp-16 genes in the nematode. J Mol Biol. 344 (2), 369-381 (2004).
- Yan, J., Sun, C. L., Shin, S., Van Gilst, M., Crowder, C. M. Effect of the mitochondrial unfolded protein response on hypoxic death and mitochondrial protein aggregation. Cell Death Dis. 12 (7), 711 (2021).
- Koopman, M., et al. A screening-based platform for the assessment of cellular respiration in Caenorhabditis elegans. Nat Protoc. 11 (10), 1798-1816 (2016).
- Shpilka, T., et al. UPRmt scales mitochondrial network expansion with protein synthesis via mitochondrial import in Caenorhabditis elegans. Nat Comm. 12 (1), 479 (2021).
- Braeckman, B. P., Smolders, A., Back, P., De Henau, S. In vivo detection of reactive oxygen species and redox status in Caenorhabditis elegans. antioxidants & redox signaling. 25 (10), 577-592 (2016).
- Bar-Ziv, R., et al. Measurements of physiological stress responses in C. elegans. J Vis Exp. 159, e61001 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon