Method Article
Imaging and Quantifying Mitochondrial Morphology in C. elegans during aging (노화 중 예쁜꼬마선충의 미토콘드리아 형태학의 이미징 및 정량화)
요약
이 프로토콜은 노화 중 예쁜꼬마선충 의 여러 조직에서 미토콘드리아 형태를 이미징하기 위한 표준화된 접근 방식을 제공합니다.
초록
대부분의 진핵 세포에서 발견되는 중요한 세포 소기관인 미토콘드리아는 호기성 호흡을 통한 에너지 생산의 주요 부위입니다. 미토콘드리아는 '세포의 발전소'로 잘 알려진 역할 외에도 세포 대사, 증식, 면역 신호 및 호르몬 신호 조절을 포함한 다른 많은 필수 세포 과정에도 관여합니다. 노화 중 또는 미토콘드리아 스트레스 하에서 미토콘드리아 기능의 저하는 종종 미토콘드리아 형태와 부피의 뚜렷한 변화를 특징으로 합니다. 선충인 C. elegans 는 투명한 몸체와 짧은 수명으로 인해 수명 동안 생체 현미경 검사를 용이하게 하기 때문에 이러한 변화를 연구하기에 이상적인 모델입니다. 그러나 예쁜꼬마선충(C. elegans ) 분야 내에서도 미토콘드리아 이미징을 위한 수많은 형질전환 구조와 방법을 사용할 수 있으며, 각각 고유한 한계가 있습니다. 여기서, 단일 복제, 매트릭스 국소화 GFP 구조는 예쁜꼬마선충에서 미토콘드리아 형태를 이미징하기 위한 강력하고 신뢰할 수 있는 방법으로 제시됩니다. 이 연구는 특히 노화 과정에서 미토콘드리아 이미징을 수행할 때 오류를 최소화하고 복제 간 및 연구 간 변동성을 줄이기 위해 실험적으로 제어 가능한 요인에 중점을 둡니다. 또한 mitoMAPR은 노화 중 조직 유형 전반에 걸쳐 미토콘드리아 형태의 변화를 정량화하는 강력한 방법으로 권장됩니다.
서문
미토콘드리아는 이중 인지질막으로 둘러싸인 세포 소기관이며 신진대사 및 에너지 생산의 핵심 부위로서 세포 생체 에너지를 유지하는 데 중요한 역할을 합니다1. 많은 수의 미토콘드리아는 세포의 요구를 충족시키기 위해 ATP의 형태로 지속적으로 에너지를 생성합니다2. 이러한 중요한 역할 외에도 미토콘드리아는 신호 전달, 자가포식, 선천성 면역, 세포 주기 및 세포 사멸 경로와 같은 복잡한 세포 과정에도 참여합니다 2,3. 미토콘드리아는 다양한 에너지 요구 사항과 미토콘드리아 건강에 따라 작은 개별 소기관에서 광범위하게 상호 연결된 관형 네트워크에 이르기까지 다양한 형태를 나타냅니다4.
미토콘드리아의 주요 특징은 미토콘드리아의 역동적인 특성으로, 긴밀하게 조정되고 연속적인 핵분열 및 핵융합 사건4을 통해 이러한 다양한 형태 사이를 번갈아 가며 볼 수 있습니다. 이러한 상반되는 과정 사이의 정확한 균형은 미토콘드리아 형태, 수, 크기 및 세포질 내 위치를 조절합니다5. 또한 이러한 융합 및 분열 과정은 미토콘드리아5의 품질 관리를 유지하는 데 중요합니다. 예를 들어, 미토콘드리아 분열은 미토파지를 통해 손상된 미토콘드리아를 제거하고 자가포식2를 통해 미토콘드리아를 선택적으로 제거하는 데 필수적인 구성 요소입니다. 미토콘드리아 역학은 또한 세포 분열, 발달, 다양한 스트레스 요인에 대한 저항, 세포 대사 유지에 중요한 역할을 합니다6.
미토콘드리아 역학의 붕괴는 신경퇴행성 질환, 대사 질환, 심혈관 질환 및 암을 포함한 수많은 질병과 관련이 있습니다4. 또한, 노화는 미토콘드리아의 극적인 리모델링과 관련이 있는데, 이는 주로 이러한 핵융합 역학의 변화7에 기인합니다. 따라서 다양한 스트레스 또는 질병 상태 하에서 그리고 노화 과정 전반에 걸쳐 미토콘드리아 형태의 변화를 시각화하고 모니터링하면 세포 기능, 질병 메커니즘 및 잠재적인 치료 전략을 이해하는 데 귀중한 통찰력을 얻을 수 있습니다.
많은 분자 경로와 마찬가지로 미토콘드리아 역학 및 기능도 선충 Caenorhabditis elegans와 같은 모델 유기체를 포함하여 진핵생물 전반에 걸쳐 고도로 보존되어 있습니다. 인간 세포와 유사하게, 예쁜꼬마선충의 미토콘드리아 융합은 외부 미토콘드리아 막(OMM)과 내부 미토콘드리아 막(IMM)의 융합을 각각 조절하는 FZO-1(포유류 Mfn1/2의 ortholog) 및 EAT-3(포유류 Opa1의 ortholog)를 포함한 다이나민 관련 구아닌 트리포스파타아제(GTPase) 단백질의 기능에 의해 달성된다8. 미토콘드리아 분열은 다이나민 관련 단백질(DRP-1, human Drp1의 ortholog)에 의해 조절되며, 이 단백질은 미토콘드리아 외막 주위에 고리 모양의 복합체를 형성하여 미토콘드리아 세포막을 수축시키고 결국 분리함으로써 미토콘드리아 분열을 촉진합니다9. 미토콘드리아 융합은 미토콘드리아 DNA, 단백질 및 지질을 포함한 미토콘드리아 내용물의 혼합을 허용함으로써 미토콘드리아 품질 관리에서 중추적인 역할을 하며, 건강한 미토콘드리아9의 함량으로 부분적으로 손상된 미토콘드리아를 보완할 수 있습니다. 반면에, 미토콘드리아 분열은 미토콘드리아가 분열할 수 있도록 하여 새로운 미토콘드리아를 생성하고, 세포질 내에서뿐만 아니라 세포 분열 중에 딸 세포로의 분포를 촉진하여 적절한 미토콘드리아 유전과 기능을 보장합니다9. 이 이벤트는 또한 손상되거나 기능 장애가 있는 미토콘드리아 분절을 분리하는 데 필수적이며, 이는 미토파지9를 통해 분해의 표적이 될 수 있습니다.
예쁜꼬마선충(C. elegans )은 완전한 게놈과 유전자 변형을 촉진하는 CRISPR/Cas910, 유전자의 과발현을 위한 수많은 방법, RNA 간섭(RNAi)11을 위한 박테리아 식품 기반 방법을 포함한 다양한 유전 도구의 가용성으로 인해 오랫동안 가장 강력한 유전자 모델 시스템 중 하나로 간주되어 왔습니다. 또한, 투명한 해부학적 구조로 인해 살아있는 유기체에서 현미경 이미징이 가능합니다12. 마지막으로, 상대적으로 짧은 수명, 낮은 유지 보수 비용, 많은 수의 연령에 맞는 동물을 생산하기 쉽기 때문에 노화 생물학을 위한 훌륭한 모델 시스템이 됩니다13. 이러한 이점은 주요 미토콘드리아 조절 경로의 알려진 보존과 결합되어 예쁜꼬마선충을 노화 중 미토콘드리아 역학을 연구하기 위한 매우 매력적인 모델 시스템으로 만듭니다.
형광 마커는 미토콘드리아를 포함한 세포 구성 요소를 시각화하고 연구하기 위해 생물학 연구에서 널리 사용됩니다. 일반적으로 사용되는 MitoTracker 및 테트라메틸로다민 에틸 에스테르(TMRE)와 같은 특정 세포 투과성 염료가 있습니다14. 전자는 전체 음전하를 띤 미토콘드리아 기질(15)을 염색하는 데 사용되는 반면, 후자는 양전하를 띤 트리페닐포스포늄 이온16을 통해 상대적인 미토콘드리아 막 전위를 평가하는 데 사용됩니다. 형질전환(transgenics)이 필요하지 않다는 장점이 있지만, 노화 과정에서 구조와 투과성이 변하는 기생충의 두꺼운 표피와 서로 다른 조직에 걸친 염료 침투의 가변성으로 인해 예쁜꼬마선충(C. elegans)에서 염료 기반 접근이 어렵다 17. 더욱이, 미토콘드리아 형태학의 평가는 염료 응집(dye aggregation)17과 같은 염료의 잠재적인 off-target 효과에 의해 혼동됩니다. 대신, 미토콘드리아 국소화 형광단을 발현하는 유전적 방법이 웜 모델에서 일반적으로 사용됩니다.
여기에서 본 연구는 세포 유형 특이적 프로모터에서 미토콘드리아 기질에 국한된 GFP(이하 MLS::GFP)를 발현하는 균주를 강조하는 데 중점을 둡니다. 중요한 것은 이러한 형질전환 라인이 알려진 게놈 자리에서 리포터의 단일 복제 발현을 보장하기 위해 MosSCI 방법을 사용하여 만들어졌으며, 이는 사용 가능한 다른 균주와의 문제를 방지한다는 것입니다. 예를 들어, 일부 미토콘드리아 표적 단백질의 고복제 발현은 염색체 외 플라스미드 어레이를 알 수 없는 복제 번호18을 가진 무작위 유전자 자리에 통합하기 때문에 발현 수준의 변동성을 유발합니다. 또한, 그들은 이전에 미토콘드리아에 손상을 유도하는 것으로 나타났는데, 이는 세포가 과발현된 미토콘드리아 단백질을 적절하게 국소화하는 데 큰 부담이 되기 때문입니다19. 따라서 정확하고 안정적이며 낮은 유전자 발현과 알려진 유전자 자리로의 교차 용이성으로 인해 이러한 MosSCI 형질전환 방법이 선호됩니다. 이 연구는 이러한 균주를 사용하여 예쁜꼬마선충의 근육, 장 및 피하부에서 미토콘드리아를 이미지화하는 방법을 표준화합니다. 또한 예쁜꼬마선충 에서 노화하는 동안 미토콘드리아에 대한 재현 가능한 실험 분석을 고려하고 보장하는 데 중요한 중요한 기술적 응용 분야와 문제 해결 방법을 강조합니다.
프로토콜
이 연구에 사용된 시약 및 장비에 대한 자세한 내용은 재료 표에 나열되어 있습니다.
1. 예쁜꼬마선충의 성장과 유지
- 선충 성장 배지(NGM) 플레이트 준비
- 예쁜꼬마선충 배양의 경우, 1mM의 CaCl2, 12.93μM(5μg/mL)의 콜레스테롤, 25mM의 KPO4(pH 6.0), 1mM의 MgSO4, 2.5mM(0.25% w/v)의 펩톤 및 51.3mM의 NaCl을 포함하는 NGM과 함께 표준 2% 한천 플레이트를 사용합니다. Castro Torres et al.20에 설명된 NGM 플레이트를 주입하는 자세한 방법을 참조하십시오.
참고: RNA 간섭(RNAi) 실험의 경우 1L NGM-한천 플레이트에 대해 1mL의 1M IPTG와 100mg/mL의 카르베니실린 1mL를 추가합니다. - NGM 플레이트가 응고된 후 실온에서 24-48시간 동안 용해성 배양액(LB)에서 OP50 배양을 성장시키거나 항생제(암피실린/카르베니실린 100μg/mL + 테트라사이클린 5μg/mL)와 함께 37°C에서 12-16시간 동안 진탕하여 LB에 pL4440 빈 벡터 플라스미드를 운반하는 HT115 배양을 배양합니다.
참고: 이 플라스미드는 상용 소스에서 구할 수 있습니다( 재료 표 참조). - 200μL의 OP50 또는 HT115 배양을 60mm 플레이트에 시딩하거나 1mL를 100mm 플레이트에 시딩합니다.
- 더 이상 젖지 않을 때까지 플레이트를 건조시키고 최대 2개월 동안 4°C의 밀봉된 용기에 플레이트를 보관하십시오.
- 선택 사항: 100μL의 10mg/mL 5-플루오로-2'-데옥시우리딘(FUDR)을 박테리아가 파종된 NGM-한천 플레이트에 직접 첨가하여 L4/성충을 화학적으로 살균하고 어린 기생충의 발생을 방지합니다.
참고: 이 연구에서 개발 및 사용된 균주는 다음과 같습니다.
(1) RHS191 - uthSi17[myo3p::MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] 나
(2) RHS192 - uthSi83[col19p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)]I
(3) RHS193 - uthSi80[vha-6p:: MLS::GFP::unc-54 3'UTR::cb-unc-119(+)] IV
- 예쁜꼬마선충 배양의 경우, 1mM의 CaCl2, 12.93μM(5μg/mL)의 콜레스테롤, 25mM의 KPO4(pH 6.0), 1mM의 MgSO4, 2.5mM(0.25% w/v)의 펩톤 및 51.3mM의 NaCl을 포함하는 NGM과 함께 표준 2% 한천 플레이트를 사용합니다. Castro Torres et al.20에 설명된 NGM 플레이트를 주입하는 자세한 방법을 참조하십시오.
- 동기화 C. elegans 표백으로
- 많은 수의 선충이 필요한 대규모 분석의 경우, 동물로 가득 찬 60mm NGM-한천 플레이트를 작은 조각으로 자르고 증식을 위해 박테리아가 파종된 100mm 플레이트에 덩어리로 만듭니다. 덩어리 후 2 ° C에서 자랄 때 3mm 플레이트에 100mm 플레이트가 벌레로 가득 찰 때까지 20-20 일이 소요됩니다. 확장 동물에 대한 자세한 권장 사항은 Castro Torres et al.20을 참조하십시오.
- 동기화를 위해 웜을 수집하려면 5-10mL의 M9 용액(22mM의 KH2PO4 monobasic, 42.3mM의 Na2HPO4, 85.6mM의 NaCl, 1mM의 MgSO4)을 웜이 있는 100mm NGM-한천 플레이트에 붓고 M9 용액을 부드럽게 소용돌이쳐 박테리아 잔디에서 웜을 느슨하게 합니다.
- 혈청학적 피펫으로 기생충을 빨아들이고 15mL 원뿔형 튜브로 옮깁니다.
참고: 플라스틱 혈청학적 피펫은 웜이 플라스틱 피펫의 내벽에 달라붙게 만드는 경향이 있으므로 유리 혈청학 피펫을 사용하는 것이 좋습니다. - 실온에서 1,100 x g 에서 30초 동안 원심분리기를 사용하여 웜을 펠릿화하고 진공 펌프를 사용하여 상층액을 흡인합니다.
- 6% 차아염소산나트륨 1.5mL, 5M NaOH 또는 KOH 0.75mL, dH2O2.75mL를 함유하는 표백 용액 5mL를 준비합니다. 표백 용액 5mL를 웜 펠렛에 붓습니다.
주의: 차아염소산나트륨과 수산화물 용액은 부식성이 있으므로 이 단계에서는 장갑과 실험복을 착용하는 것이 좋습니다. - 동물의 모든 몸이 완전히 용해되고 알만 남을 때까지 벌레 + 표백 용액 혼합물을 ~4-6분 동안 배양합니다. 용해 과정을 돕기 위해 혼합물을 세게 흔듭니다.
알림: 해부 현미경으로 매분 벌레를 확인하십시오. 성충의 시체가 얼마나 많이 용해되는지에 따라 표백 시간을 단축하거나 연장합니다. 알을 표백 용액 안에 장기간 방치하면 알이 손상되고 선충의 생존 능력에 영향을 미칩니다. - 계란/표백 용액 혼합물을 실온에서 30 x g 에서 1,100초 동안 원심분리하여 계란을 펠릿으로 만든 다음 진공 펌프를 사용하여 상층액을 조심스럽게 흡인하고 계란 펠릿을 흡인하지 않도록 합니다.
알림: 계란이 표백 용액에 장기간 노출되는 것을 방지하기 위해 신속하게 세척하는 것이 좋습니다. - 5-10mL의 M9 용액으로 계란을 4 번 씻으십시오.
- 소량의 M9 용액에 난자를 재현탁하고 최대 50μL까지 피펫팅하여 박테리아가 파종된 NGM 플레이트에 플레이트를 만듭니다. 난자 혼합물 3-5 μL를 플레이트에 피펫팅하고 이 부피의 난자 수를 세어 난자 수의 대략적인 추정치를 계산합니다. M9를 추가하여 계란 혼합물을 이 부피의 셀 수 있는 계란 수에 희석합니다.
참고: 플레이트당 100마리 미만의 웜을 도금하는 것이 좋습니다. 동물의 도금 수에 대한 자세한 권장 사항은 Castro Torres et al.20을 참조하십시오. - 또는 더 긴밀한 연령 동기화를 위해 선충을 L1-arrest합니다. L1 어레스팅을 위해 15mL 원추형 튜브의 계란 펠릿에 적절한 양의 M9 용액을 10-12mL 부피로 첨가합니다. 벌레가 20 ° C에서 16-24 시간 동안 회전자에서 회전하도록하십시오. 원심분리기(1.2.7단계 참조)하고 상층액을 흡인합니다. L1 동물의 농도를 계산하고 1.2.9단계에서 난자에 대해 설명한 대로 NGM 플레이트에 플레이트합니다.
- 예쁜꼬마선충 개체군을 원하는 나이로 숙성: L4/성체 동물을 1.1.6단계에서 설명한 대로 5-플루오로-2'-데옥시우리딘(FUDR)이 포함된 플레이트로 옮겨 자손을 방지하고 이미 생산된 자손을 제거합니다. FUDR 없이 웜을 노화시키는 대체 방법은 Castro Torres et al.20을 참조하십시오.
참고: 첫 번째 유충 단계에서 벌레를 동기화하는 방법으로 표백을 선택하기 전에 몇 가지 중요한 요소를 고려해야 합니다. 앞서 강조한 바와 같이, 표백제에 대한 과다 노출은 배아를 손상시키고, 부화율을 감소시키며, 표백제 내성 균주에 대한 선택 압력을 생성하여 잠재적으로 개체군의 유전학을 변화시킬 수 있습니다. 또한 배아를 장기간 먹이지 않고 방치하면 영양소 고갈로 인해 신진대사와 발달이 손상될 수 있습니다. 따라서 알을 낳는 방법 또는 NemaSync 방법을 표백 방법의 대안으로 사용할 수 있습니다. 알을 낳는 절차에 대한 방법은 Castro Torres et al.20에 의해 참조된 연구에서 단계적으로 설명됩니다. NemaSync는 화학 물질21을 사용하지 않고 수동 웜 싱크로나이저를 사용하여 웜을 동기화하는 새롭고 더 비싼 상용 방법입니다.
2. 예쁜꼬마선충(C. elegans)의 미토콘드리아의 이미징
- 예쁜꼬마선충(C. elegans)의 이미징을 위한 슬라이드 준비
- 10-20 μL의 M9 용액을 유리 슬라이드에 추가하고 특정 연령의 원하는 수의 성충을 슬라이드의 용액으로 옮깁니다. 약 25마리의 웜이 권장됩니다.
참고: 기생충을 마비시키기 위해 전통적인 방법(예: 테트라미솔 또는 아지드화나트륨)을 사용하면 특정 조건에서 미토콘드리아 단편화가 발생할 수 있습니다(아래 참조). 대신, 영구적인 양전하를 띤 표면이 있는 HistoBond 슬라이드를 사용하는 것이 좋으며, 이는 인공물을 유발할 수 있는 약물을 사용하지 않고도 기생충의 이동을 줄일 수 있습니다. 웜과 M9 용액의 적절한 비율을 맞추는 것이 중요한데, 부피가 적으면 슬라이드와 커버슬립 사이에 동물이 끼일 수 있고, 부피가 크면 이미지 획득 중에 동물이 움직일 수 있습니다. 권장 사항은 표 1에서 찾을 수 있습니다. - 또는, 기생충을 마비시키는 약물을 사용하는 경우, 미토콘드리아의 단편화를 제한하면서 사용할 수 있는 농도 범위와 시점에 대한 이 원고의 대표적인 결과를 참조하십시오.
참고: 건강한 야생형 동물에서는 큰 단편화가 없었지만, 특정 돌연변이 또는 질환은 이러한 약물의 동일한 농도에서 미토콘드리아 단편화를 경험할 수 있으므로 이러한 약물을 사용할 때 몇 가지 테스트가 필요하다는 점을 기억하는 것이 중요합니다. - 슬라이드의 M9 용액에 든 벌레를 커버 유리로 덮고 매니큐어를 사용하여 측면을 밀봉하고 M9 용액의 증발을 방지합니다.
- 선택 사항: 근육 이미징의 경우 커버 슬립을 부드럽게 살짝 밀어 벌레를 굴립니다. 이를 통해 웜 측면의 체벽 근육을 초점면과 더 잘 정렬하여 고품질 이미지를 얻을 수 있습니다.
- 10-20 μL의 M9 용액을 유리 슬라이드에 추가하고 특정 연령의 원하는 수의 성충을 슬라이드의 용액으로 옮깁니다. 약 25마리의 웜이 권장됩니다.
- 복합 현미경을 사용한 미토콘드리아의 이미징
- 근육 및 피하 미토콘드리아(myo-3p::MLS::GFP 및 col-19p::MLS::GFP)에 대한 표준 광시야 현미경을 사용합니다.
참고: 63x/1.4 Plan Aprochromat 대물렌즈, GFP 필터(11525314), DFC9000 GT 카메라 및 LED5 광원이 장착된 시판되는 이미저를 사용했으며 호환 가능한 현미경 소프트웨어를 사용했습니다. - 각 현미경 및 실험 설정에 대한 이미징 설정을 최적화합니다.
참고: 그러나 이 원고에 사용된 이미저를 사용한 이미징을 위한 매개변수는 시작점으로 다음과 같습니다: 치수는 피하 미토콘드리아의 경우 0.50μm의 단계 크기에서 2048 (x) * 2048(y) 픽셀이며 근육 미토콘드리아의 경우 소프트웨어에 의해 자동으로 계산되는 "시스템 최적화"입니다. 노출 시간은 근육 미토콘드리아의 경우 50ms, 피하 미토콘드리아의 경우 50ms입니다. 475nm LED는 50% 전력으로, 중성 밀도 필터는 근육 30%, 피하 미토콘드리아의 경우 17%로 설정되었습니다. Z-범위는 GFP 신호가 보이는 위치에서 끝나는 위치까지 시작합니다. 최대 투영에서 노이즈를 최소화하기 위해 최대 투영에 사용되는 z-평면의 수를 줄일 수 있습니다. - 장 미토콘드리아(vha-6p::MLS::GFP)의 이미징에는 컨포칼 현미경을 사용하십시오.
참고: 이미징은 63x/1.4 Plan ApoChromat 대물렌즈, 백색광 레이저(WLL), 음향 광학 빔 분배기, HyD S 검출기가 장착된 상용 컨포칼 현미경에서 수행되었으며 현미경 소프트웨어에서 실행되었습니다. 앞서 언급했듯이 각 개별 실험 설정을 최적화해야 하지만 시작점으로 이 연구에 사용된 Stellaris를 사용한 이미징 매개변수는 다음과 같습니다. WLL은 85.00% 최대 출력으로 설정되었으며 3.00%의 강도에서 485nm 레이저 라인으로 설정되었습니다. HyD S 검출기는 아날로그 설정에서 490의 이득으로 590-25nm로 설정되었습니다. 1,000Hz의 스캔 속도, 2.25 줌, 라인 평균 2, 1 AU(95.5 μm)의 핀홀로 0.495 μm(크기 z)의 단계 크기에서 5(z) 픽셀로 1024 (x) * 1024(y) 또는 82.01 μm (x) * 82.01 μm(y)의 영역을 단방향으로 스캔했습니다. Z 범위는 GFP 신호가 보이는 위치에서 시작하여 끝나는 위치까지 이동합니다. 최대 투영에서 노이즈를 최소화하기 위해 최대 투영에 사용되는 z-평면의 수를 줄일 수 있습니다.
- 근육 및 피하 미토콘드리아(myo-3p::MLS::GFP 및 col-19p::MLS::GFP)에 대한 표준 광시야 현미경을 사용합니다.
- 미토콘드리아 형태학의 정량화
참고: 자세한 내용은 Schindelin et al.22 및 보충 그림 1A-D를 참조하십시오.- FIJI(피지는 ImageJ, https://fiji.sc/) 소프트웨어를 다운로드하여 설치합니다. 그런 다음 FIJI를 엽니다.
- MitoMAPR 매크로를 다운로드하고 설치하려면 https://doi.org/10.7554/eLife.49158.033 에서 소스 코드 1을 다운로드하십시오. 그런 다음 "A."에 언급된 모든 코드를 복사합니다. MitoMAPR-1.0에 대한 IJM 코드." 그런 다음 피지를 열고 플러그인 > 새 > 매크로로 이동하여 이전에 복사한 코드를 매크로 창에 붙여넣습니다. File > Save as 옵션으로 이동하여 나중에 사용할 수 있도록 매크로를 저장합니다.
- 파일을 Fiji 도구 모음으로 끌어다 놓거나 파일 > 열기로 이동하여 FIJI에서 Z 스택이 있는 3D 현미경 이미지를 엽니다.
- Image > Stacks > Z Project로 이동한 다음 초점이 맞는 이미지가 있는 슬라이스 범위를 최대 투영으로 선택하고 투영 유형으로 Max Intensity를 선택하여 이미지의 최대 투영을 만듭니다.
- File (파일) > Save As > Tiff(Tiff로 저장)로 이동하여 이미지를 TIFF로 저장합니다.
- "Rectangle" 도구를 사용하여 전체 이미지에서 관심 영역(ROI)을 자르고 ROI를 그린 다음 Image > Crop으로 이동합니다.
- File (파일) > Save As > Tiff(Tiff로 저장)로 이동하여 이미지를 TIFF로 저장합니다.
- 매크로 파일을 Fiji 도구 모음으로 끌어다 놓아 이전에 저장한 MitoMAPR 매크로를 실행한 다음 실행을 클릭합니다. 2.3.6에서 Tiff 파일이 저장된 폴더를 선택하라는 새 창이 나타납니다.
- 영역을 선택하라는 새 창이 표시됩니다. 앞서 2.3.4단계에서 언급한 대로 사각형 도구를 사용하여 이미지에 사각형을 만들고 확인을 누릅니다.
참고: 2.3.6단계에서 설명한 대로 이전에 자른 경우 전체 이미지를 선택할 수 있습니다. 미토콘드리아 형태와 관련된 모든 값이 포함된 "데이터" 창을 파일 > 다른 이름으로 저장 > 저장으로 이동하여 Excel 파일로 저장합니다.
- FIJI 매크로를 사용한 시료 일괄 처리
- 분석할 이미지 영역을 지정하기 위한 매크로를 만듭니다. 피지를 열고 플러그인 > 새 > 매크로로 이동합니다. 다음 코드를 텍스트 상자에 붙여넣고 2.3.2(보충 그림 1E-H)에 설명된 대로 매크로를 저장합니다.
참고 : 사각형 만들기 (100, 100, 200, 200); run(지정..., 너비 = 100, 높이 = 100, x = 100, y = 100 배율). - 열려 있는 모든 이미지를 TIFF 파일로 저장하기 위한 매크로를 만듭니다. 매크로 창에서 File > New(새로 만들기 )로 이동하여 다음 코드를 텍스트 상자에 붙여넣습니다. 2.3.2에 설명된 대로 매크로를 저장합니다.
참고 : dir = getDirectory ( " 폴더 선택"); ids=newArray(n이미지); for (i=0; i - 배치 MitoMAPR에 대한 매크로를 만듭니다. 소스 코드 1 https://doi.org/10.7554/eLife.49158.033 이동하여 B.)에 언급된 모든 코드를 복사합니다. MitoMAPR-1.0_Batch에 대한 IJM 코드". 매크로 창 내에서 File > New(파일 새로 만들기)로 이동하여 소스 코드 1의 코드를 텍스트 상자에 붙여넣고 2.3.2단계에 설명된 대로 매크로를 저장합니다.
- 반복 자르기를 위한 매크로를 만듭니다. 소스 코드 1 https://doi.org/10.7554/eLife.49158.033 이동하여 C.) IJM code for CropR"에 언급된 모든 코드를 복사합니다. 매크로 창 내에서 File > New(파일 새로 만들기)로 이동하여 소스 코드 1의 코드를 텍스트 상자에 붙여넣고 2.3.2단계에 설명된 대로 매크로를 저장합니다.
- 일괄 처리를 시작하려면 먼저 모든 이미지를 열고 2.4.2단계에서 저장한 매크로를 실행하여 처리할 모든 이미지를 TIFF로 저장합니다. 모든 이미지는 지정된 폴더에 TIFF로 저장됩니다. 저장된 모든 이미지를 닫습니다.
- 2.4.5단계에서 저장한 이미지가 3D 이미지(Z 스택이 포함된 이미지)인 경우 Z 투영으로 만들어 2D 이미지로 변환합니다. 이미지를 Z-project> 일괄 처리하려면 FIJI 기본 창에서 Batch > Macro 처리로 이동하여 일괄 처리 창을 엽니다.
- "입력"의 경우 2.4.5에서 처리할 모든 3D 이미지가 포함된 폴더의 위치를 선택합니다. "출력"의 경우 처리 후 저장된 이미지의 원하는 위치를 선택합니다. 출력 형식을 TIFF로 선택합니다. 창의 큰 텍스트 상자에 다음 코드를 붙여넣고 프로세스 버튼을 누릅니다: run("Z Project...", "projection=[Max Intensity]").
- 2.4.4단계의 반복 자르기 매크로를 2.4.3단계의 지정 영역 매크로와 함께 사용하여 처리할 모든 2D 이미지의 동일한 치수로 ROI를 크롭합니다. 2.4.4단계에서 반복 자르기 매크로를 실행합니다. 디렉토리를 묻는 창이 나타납니다.
- 2.4.5단계의 2D 이미지 또는 2.4.6단계의 2D Z 투영 이미지만 포함된 폴더를 선택하고 선택(Select ) 버튼을 누릅니다. 매크로는 선택한 폴더에 있는 이미지 중 하나를 열고 두 개의 버튼이 있는 "선택하기"라고 표시된 창이 나타납니다.
- 2.4.3단계에서 지정한 선택 매크로를 실행합니다. 필요에 따라 해당 매크로 텍스트 상자의 너비 및 높이 값을 수정하여 선택 차원을 변경하고 Run 을 눌러 새 선택을 관찰합니다. 매크로에서 X 및 Y 값을 변경하면 선택 영역의 왼쪽 위 모서리 위치가 수정됩니다.
- 선택 치수가 만족스러우면 사각형을 원하는 영역으로 드래그한 다음 "선택" 창에서 확인 버튼을 누릅니다. 자른 영역은 2.4.7단계에서 선택한 폴더에 저장됩니다.
- 매크로는 폴더의 나머지 모든 이미지를 반복합니다. 각 이미지에 대해 차원 값을 변경하지 않고 2.4.8단계의 매크로를 실행하여 모든 이미지에 대해 동일한 차원이 잘리도록 합니다. 각 이미지에 대해 선택 사각형을 원하는 위치로 드래그하고 "선택하기" 창에서 확인 버튼을 누릅니다.
- 2.4.8-2.4.9단계에서 자른 이미지가 포함된 폴더를 열고 자른 모든 이미지를 새 폴더로 이동합니다.
- 2.4.3단계의 배치 MitoMAPR 매크로를 실행하여 잘린 모든 이미지를 분석합니다. 새 창에서 디렉토리를 선택하라는 메시지가 표시됩니다. 2.4.10단계에서 자른 모든 이미지가 있는 폴더를 선택합니다. 완료되면 "데이터"라는 창이 미토콘드리아 형태와 관련된 모든 값과 함께 나타납니다. 2.3.9단계에 설명된 대로 모든 데이터를 Excel 파일로 저장합니다.
- 분석할 이미지 영역을 지정하기 위한 매크로를 만듭니다. 피지를 열고 플러그인 > 새 > 매크로로 이동합니다. 다음 코드를 텍스트 상자에 붙여넣고 2.3.2(보충 그림 1E-H)에 설명된 대로 매크로를 저장합니다.
결과
예쁜꼬마선충(C. elegans )은 투명한 몸체로 인해 미토콘드리아 이미징을 위한 훌륭한 모델로, 과도한 샘플 준비 없이 살아있는 동물을 쉽게 이미징할 수 있습니다. 또한 미토콘드리아 표적 형광 단백질을 발현하기 위해 조직 특이적 프로모터를 사용하여 다른 조직의 미토콘드리아 형태를 쉽게 시각화할 수 있습니다. 여기에서는 myo-3p (근육 미토콘드리아), vha-6p (장 미토콘드리아) 및 col-19p (피하 미토콘드리아)를 사용하여 미토콘드리아 표적 GFP(ATP-1 단백질의 미토콘드리아 국소화 서열)의 발현을 유도했습니다. 예쁜꼬마선충(C. elegans )의 체벽 근육의 미토콘드리아는 근육 섬유(근섬유)를 따라 정렬되는 세뇨관 형태를 나타냅니다(그림 1A). 장 미토콘드리아는 고도로 상호 연결된 거미줄과 같은 구조를 보였으며 정렬이 덜 균일했습니다(그림 1B). 그리고 피하부에는 장이나 근육에 비해 더 둥글거나 타원형으로 보이는 세뇨관 모양의 미토콘드리아가 있습니다(그림 1C). 미토콘드리아 형태는 일반적으로 근육과 장에 있는 기생충의 길이에 걸쳐 일관되지만, 하피는 기생충의 근위부와 원위부 말단 사이의 미토콘드리아의 상호 연결성에 약간의 차이를 보입니다. 따라서 재현 가능한 실험 결과를 얻으려면 신체의 특정 부위에 집중하는 것이 좋습니다. 여기에서 인두와 외음부 사이의 영역(인두 아래 ~100-200 μm)이 일관되게 이미지화됩니다.
근육 및 피하 미토콘드리아의 경우 복합 현미경으로 이미징하면 세포의 평탄도와 초점이 맞지 않는 빛의 수준이 낮기 때문에 충분한 해상도를 얻을 수 있습니다. 그러나 장 미토콘드리아는 장의 다른 부분에서 오는 많은 양의 초점이 맞지 않는 빛으로 인해 해상도가 제한되기 때문에 복합 현미경으로 관찰하기 어렵습니다(그림 2). 따라서 컨포칼 현미경으로 장 미토콘드리아를 이미지화하여 초점이 맞지 않는 빛을 줄이고 적절한 미토콘드리아 형태를 시각화하는 것이 좋습니다.
미토콘드리아의 모양은 매우 역동적이며 동물의 대사 환경에 따라변화합니다 23 또는 다양한 기질 강성24에 대한 노출로 인해 변화합니다. 따라서 먹이 공급원이 없는 상태에서 뻣뻣한 유리 기판 위에 동물을 장기간 현미경 슬라이드에 방치하면 미토콘드리아 형태에 잠재적으로 영향을 미칠 수 있습니다. 여기에서 이 연구는 M9 용액의 슬라이드에서 약 30분 후에 벌레의 미토콘드리아가 단편화되며, 피하 미토콘드리아가 가장 단편화를 보인다는 것을 발견했습니다(그림 3). 따라서 미토콘드리아의 이미징은 샘플 전처리 후 신속하게 수행되어야 합니다.
아지드화나트륨(sodium azide) 및 테트라미솔(tetramisole)을 포함하여 기생충의 이동성을 제한하는 화학 물질은 3D 이미징을 위해 동물의 여러 z-절편을 캡처하기 위해 고정된 기생충이 필요하기 때문에 예쁜꼬마선충의 라이브 이미징에 일반적으로 사용됩니다. 이전 연구에서는 아지드화나트륨 또는 테트라미솔에 노출되면 미토콘드리아 단편화가 발생할 수 있다고 밝혔다25. 놀랍게도, 아지드화나트륨은 높은 농도에서도 근육이나 장의 미토콘드리아 형태에 제한된 영향을 미친다는 것이 밝혀졌습니다. 그러나 피하부는 M9 대조군보다 더 일찍 미토콘드리아 단편화를 보였습니다(그림 3). 중요한 것은 고농도의 테트라미솔(100mM)이 모든 세포 유형에서 노출 직후 상당한 미토콘드리아 단편화를 초래했다는 것입니다. 이에 비해 중간 농도(10mM)는 M9 대조군에 비해 단편화가 더 빨랐습니다. 낮은 농도(1mM)는 미토콘드리아 형태에 제한된 영향을 미쳤습니다. 이러한 데이터는 아지드화나트륨의 활용이 미토콘드리아의 신속한 이미징을 위한 실행 가능한 옵션이 될 수 있는 반면, 테트라미솔은 일반적으로 피해야 함을 시사합니다.
예상대로, 예쁜꼬마선충 의 모든 조직의 미토콘드리아는 자연적인 노화 과정에서 단편화를 보입니다(그림 4A). 단편화는 미토콘드리아로 시각화할 수 있으며, 이는 어린 동물에서 나타나는 선형의 관 모양의 미토콘드리아와 크게 다른 절단된 구형 구조로 나타납니다. 장은 나중에 구형 자가형광 구조에서 증가하므로 자가형광을 실제 미토콘드리아 구조와 혼동하지 않도록 주의해야 합니다. 웜에 따라 미토콘드리아 구조의 변화는 가변적일 수 있기 때문에 몇 마리의 웜을 이미징하는 것보다 상당한 샘플 크기에 대해 미토콘드리아 형태의 정량화를 수행하는 것이 중요합니다. 여기서는 물체, 네트워크, 네트워크당 접합부, 접합점, 물체 길이, 미토콘드리아 발자국, 미토콘드리아 범위 및 물체 면적을 포함한 다양한 메트릭 세트를 사용하여 미토콘드리아 형태를 자동으로 정량화할 수 있는 mitoMAPR이 사용되었습니다(보충 표 1 및 보충 표 2). 자동화는 사용자의 주관적인 편견을 제거합니다. 여기에서는 물체 길이 메트릭이 노화 중 근육 및 장 미토콘드리아 형태의 변화를 정량적으로 측정하는 데 최적이며, 접합점 메트릭은 노화 중 피하 미토콘드리아 형태의 변화를 측정하는 데 최적이라고 보고합니다(그림 4B).
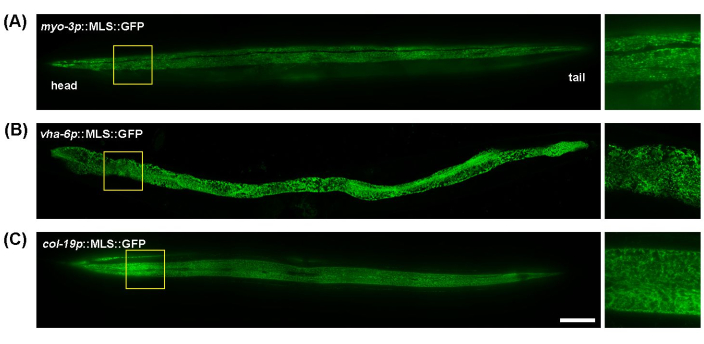
그림 1: 예쁜꼬마선충(C. elegans)의 전신에 걸친 미토콘드리아의 이미지. 미토콘드리아 영상은 근육(A), 장(B) 및 피하(C)에서 MLS::GFP 발현이 있는 형질전환 동물의 L1 단계에서 EV로 성장한 5일차 전체 기간에 걸쳐 수행되었습니다. 기준자: 100 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 복합 현미경과 컨포칼 현미경 간의 미토콘드리아 이미징 비교. 미토콘드리아 영상은 근육(A), 장(B) 및 하피(C)에서 MLS::GFP 발현이 있는 형질전환 동물의 L1 단계에서 EV에서 성장한 1일차 성체 동물에서 수행되었습니다. 눈금 막대 : 5μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: M9, 아지드화나트륨 및 테트라미솔 처리 후 다양한 조직의 미토콘드리아 단편화 평가. 미토콘드리아 영상은 근육, 장 및 피하주에서 MLS::GFP가 발현된 1일차 성체 동물에서 수행되었습니다. 동물은 L1 단계부터 EV에서 성장했습니다. 동물을 M9, 아지드화나트륨(1mM, 10mM 및 100mM) 또는 테트라미솔(1mM, 10mM 및 100mM)을 함유한 슬라이드에 놓고 슬라이드 준비(0분) 직후 또는 슬라이드 준비 후 15분 또는 30분 후에 이미징을 수행했습니다. 2개의 생물학적 복제물에 대해 균주당 n > 5마리의 동물에 대한 대표 이미지가 표시됩니다. 눈금 막대 : 5μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: 다양한 조직에서 노화 중 예쁜꼬마선충 의 미토콘드리아 이미징 및 MitoMAPR을 사용한 미토콘드리아 형태 정량화. (A) 미토콘드리아 영상은 성체의 1, 5, 9, 13, 15일에 성체 동물에서 근육, 장 및 피하주피에서 MLS::GFP의 발현과 함께 수행되었습니다. 동물은 L1 단계에서 EV에서 성장했고 1일차 성체 단계에서 FUDR을 포함하는 EV 플레이트로 이동했습니다. 이미지는 ≥ 3개의 생물학적 복제물에 대해 균주당 n≥ 5마리의 동물을 나타냅니다. 눈금 막대 : 5μm. (B) 1일, 5일, 9일, 13일, 15일에 기생충의 근육 미토콘드리아와 장 미토콘드리아의 물체 길이에 대한 정량화와 1일, 5일, 9일, 13일, 15일에 기생충의 피하 미토콘드리아의 접합점에 대한 정량화. 모든 개별 데이터 포인트는 평균 ± SD와 결합되었습니다. 그래프는 학생 t-test를 사용하여 플롯되고 통계적으로 분석되었습니다. ns = 유의하지 않음, *p < 0.03; **p < 0.002; p < 0.0002; 피 < 0.0001. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 벌레의 날 | 웜의 수 | 완충액의 부피 (mL) |
| 1 | 25 | 13 |
| 5 | 25 | 14 |
| 9 | 20 | 15 |
| 13 | 20 | 15 |
표 1: 슬라이드를 준비할 때 권장되는 웜 수와 버퍼의 양.
보충 그림 1: MitoMAPR을 사용한 단일 이미지 정량화 응용 프로그램 및 여러 이미지에 대한 일괄 처리. (A) MitoMAPR 매크로를 사용하여 단일 이미지에서 미토콘드리아 형태를 정량화하는 워크플로우. (B) 피지를 사용한 원시 3D 이미지 파일의 Z 투영(최대 투영). (C) Z 투영된 이미지에서 관심 영역을 자르는 단계. (D) MitoMAPR 매크로에서 획득한 골격 이미지. 눈금 막대 : 5μm. (E) MitoMAPR 매크로를 사용한 배치 프로세스를 사용하여 여러 이미지에서 미토콘드리아 형태를 정량화하는 워크플로우. (F) 자르기 매크로의 스크린샷. (G) Z 프로젝션을 위한 배치 프로세스의 스크린샷. (H) 일괄 자르기 매크로의 선택 확인 창 스크린샷. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 1: 예쁜꼬마선충 근육 조직의 미토콘드리아 형태 메트릭에 대한 정량 분석. 기록된 메트릭에는 개체, 네트워크, 네트워크당 접합 수, 연결 지점, 개체 길이, 미토콘드리아 발자국, 개체 면적 및 미토콘드리아 범위가 포함되며, 이는 여러 날에 걸쳐 측정됩니다. 데이터는 노화에 따른 미토콘드리아 형태의 변화를 평가하기 위한 기초를 제공합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 2: 예쁜꼬마선충의 미토콘드리아 형태에 대한 고정화 화학물질의 효과. 데이터에는 M9 완충액 또는 다양한 농도의 테트라미솔 및 아지드화나트륨으로 시료를 준비한 후 다양한 농도 및 시점에 걸쳐 물체, 네트워크, 네트워크당 접합부, 접합점, 물체 길이, 미토콘드리아 발자국, 물체 면적 및 미토콘드리아 범위와 같은 측정이 포함됩니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
미토콘드리아 형태에 대한 형광 이미징은 미토콘드리아의 변화를 결정하는 가장 일반적인 방법입니다. 투과 전자 현미경(TEM), 원자력 현미경 및 초저온 전자 현미경과 같은 고급 현미경 기술은 더 높은 해상도를 제공하지만 형광 현미경은 더 저렴하고 대부분의 연구자가 접근할 수 있습니다. 또한, 형광 현미경 검사는 살아있는 세포에서 수행 할 수 있으며, C. elegans와 같은 명확한 모델 유기체에서는 전체 동물에서 이미징을 수행 할 수 있습니다26, 27. 형질전환 예쁜꼬마선충(C. elegans)의 이미징은 매우 간단하며, 유전적으로 인코딩된 형광 마커는 복잡한 샘플 처리가 필요하지 않거나 MitoTracker 또는 TMRE 28,29,30와 같은 기존 미토콘드리아 염료의 세포 유형 또는 조건에 따라 다양한 염색이 필요하지 않기 때문에 미토콘드리아의 보다 안정적이고 강력한 이미징을 가능하게 합니다. 유전적으로 인코딩된 형광 마커는 일반적으로 미토콘드리아 단백질을 형광단으로 직접 태깅하거나 최소한의 미토콘드리아 국소화 서열을 가진 형광단의 접합을 포함합니다. 이러한 구조는 종종 조직 특이적 프로모터에 의해 구동되어 근육, 장 또는 피하 조직과 같은 다른 조직에서 미토콘드리아를 시각화할 수 있습니다31. 일반적으로 이러한 형광 표지 단백질은 과발현되며, 이는 전장 미토콘드리아 단백질이 과발현될 경우 잠재적으로 원치 않는 생리학적 효과를 가질 수 있습니다. 따라서, 최소 MLS 염기서열(minimal MLS sequences)이 더 나은 선택지이다 32. 그러나 최소한의 MLS-형광 단백질 융합체의 과발현에도 불구하고 많은 양의 단백질을 미토콘드리아로 가져오는 것은 미토콘드리아 막의 잠재력을 붕괴시키고 동물 건강에 영향을 미칠 수 있으므로 높은 과발현을 피해야 합니다33. 현재 사용 가능한 모든 예쁜꼬마선충 균주에 대한 철저한 규명은 이 원고의 범위를 벗어나지만, 수많은 미토콘드리아 리포터에 대한 상세한 비교 분석과 각각의 장단점은 여기에서 찾을 수 있습니다31.
예쁜꼬마선충(C. elegans)의 미토콘드리아에 대한 생세포 이미징의 경우, 컨포칼 현미경 검사에 비해 이러한 방법의 빠르고 용이함 때문에 표준 화합물 또는 광시야 현미경 검사가 선호되는 옵션일 수 있습니다. 이 연구에서는 근육 및 피하주사와 같은 편평한 세포가 컨포칼 현미경 검사의 이점을 최소화하고 복합 현미경 검사를 통해 적절한 미토콘드리아 형태를 시각화할 수 있는 충분한 해상도로 획득할 수 있음을 보여주었습니다. 장과 같은 더 큰 세포는 초점이 맞지 않는 빛으로 인해 복합 현미경 검사를 어렵게 만듭니다. 따라서 장 미토콘드리아 형태의 신뢰할 수 있는 이미징을 위해 컨포칼 현미경이 필요합니다.
살아있는 동물의 전체 조직 두께에 걸친 3D 이미징을 위한 중요한 고려 사항은 이미지 획득 중에 벌레의 움직임을 방지하는 것입니다. 연구자들은 종종 테트라미솔(tetramisole)이나 아지드화나트륨(sodium azide)34와 같은 기생충을 마비시키는 방법을 사용한다. 아지드화나트륨은 미토콘드리아의 전자전달계에서 중요한 효소인 시토크롬 c 산화효소(복합체 IV)를 억제하여 근육 수축 및 기타 세포 기능에 필요한 ATP의 부족으로 인해 전반적인 마비를 유발합니다34,35. 테트라미솔은 신경근 접합부에서 아세틸콜린을 모방하여 작용하여 지속적인 탈분극과 근육 수축을 유발합니다36. 그러나 이러한 약물에 노출되면 산화 스트레스를 유발하여 미토콘드리아 단편화를 유발할 수 있습니다23. 이 연구에서 테트라미솔은 매우 빠르게 미토콘드리아 단편화를 유도하는 것으로 밝혀졌지만 아지드화나트륨은 훨씬 더 제한된 효과를 보였습니다.
예쁜꼬마선충(C. elegans)은 수명이 짧고 동물의 노화가 쉽기 때문에 노화가 미토콘드리아 형태에 미치는 영향을 연구할 수 있는 매우 간단하고 쉬운 방법을 제공합니다. 여기에서는 DNA 복제를 방지하여 동물을 화학적으로 불임화하는 강력한 방법인 FUDR에 대한 노출을 선택했습니다 20,37,38. 그러나, FUDR은 특정 노화 매개변수에 원치 않는 영향을 미칠 수 있으며, FUDR의 off-target effect에 대해 우려하는 사람들을 위해 자손을 제거하는 다른 전략을 활용할 수 있다39. 예를 들어, 온도에 민감한 생식계열 돌연변이 GLP-4를 포함한 무균 돌연변이 또는 CF51240,41,42와 같은 정자 결핍 돌연변이가 있습니다. 대안으로, 동물은 매일 자손에서 성인을 수동으로 선택하여 자연적으로 노화 할 수도 있습니다.
미토콘드리아 형태학의 변화를 정량화하는 것도 매우 중요한 고려 사항인데, 이는 동물에 따라 미토콘드리아 형태에 상당한 차이가 있을 수 있기 때문입니다. 따라서 충분한 표본 크기에 걸쳐 정량적 분석 및 통계를 수행하는 것은 미토콘드리아 형태학의 변화에 대한 중요한 결론을 내리는 데 필수적입니다. 그러나 이미지 분석 및 정량화는 주관적 편향과 상호 연결된 미토콘드리아와 같은 매우 복잡한 구조를 이미징하는 데 따른 문제로 인해 큰 어려움을 겪을 수 있습니다. 이를 위해 Mair 실험실에서 개발한 수많은 지표를 사용하여 미토콘드리아 형태를 정량화하는 자동화된 방법을 요약합니다. MitoMAPR을 사용하면 미토콘드리아 네트워크, 물체 길이, 분포, 네트워크 커버리지 및 미토콘드리아 발자국을 포함하여 미토콘드리아의 다양한 측면을 측정하여 미토콘드리아 형태를 객관적이고 자동으로 정량화할 수 있습니다. MitoMAPR은 ImageJ를 위한 무료 매크로이므로 작동하는 컴퓨터가 있는 모든 실험실에서 사용할 수 있습니다. MitoMAPR 사용의 한 가지 중요한 측면은 테스트 중인 실험 조건의 변화를 결정하기 위해 가장 강력한 미토콘드리아 형태 메트릭을 결정하기 위해 큰 표본 크기에 걸쳐 정량화를 수행하는 것입니다43,44. 여기서, 물체의 길이와 접합 지점은 근육, 장 및 피하의 노화 중 변화를 결정하는 가장 좋은 지표라는 것이 밝혀졌습니다. 미토콘드리아 형태학의 변화를 분석하기 위한 대안적인 접근법은 z-스택 이미지에서 미토콘드리아의 3D 표현을 생성한 다음 3D 분석을 수행하는 것입니다16,45. 이는 SharpStack Total Deconvolution 및 3D Constructor 모듈이 있는 Image-Pro Plus와 같은 상용 소프트웨어를 사용하여 달성할 수 있습니다. 3D 미토콘드리아 표현의 정량화는 미토콘드리아 모양과 네트워크 속성에 대한 보다 정확한 통찰력을 제공하는 것으로 나타났습니다46. 그러나 이 방법은 광시야 또는 공초점 z-스택의 여러 이미지 섹션을 단일 2D 프로젝션으로 축소하고 mitoMAPR과 같은 도구로 분석하는 세미 3D 접근 방식에 비해 기술적으로 더 복잡하고 비용이 많이 듭니다. 이 연구는 미토콘드리아 기질 표적 GFP의 단일 복제 발현의 사용법을 요약하고 이미징 중에 피해야 할 몇 가지 함정을 자세히 설명합니다.
제한 사항 및 시간 고려 사항
장 조직과 같이 크고 두꺼운 조직에는 컨포칼 현미경 검사가 권장되지만, 표준 라인 스캐닝 컨포칼 현미경 검사 기법은 미토콘드리아의 신속한 이미징을 수행하기에는 너무 느릴 수 있습니다. 이는 슬라이드에 웜을 장기간 보관하면 미토콘드리아의 단편화를 초래할 수 있음을 보여주는 데이터를 고려할 때 특히 그렇습니다. 컨포칼 현미경 기술이 크게 발전했지만 스피닝 디스크, Airyscan 또는 이 연구에 사용된 것과 같이 빠른 이미징을 수행할 수 있는 현미경은 일부 실험실에서 비용이 많이 들 수 있습니다. 이러한 경우, 복합 현미경 검사는 컴퓨터 제거 방법과 결합되어 디콘볼루션47과 같이 초점이 맞지 않는 빛을 제거할 수 있습니다.
또한 앞서 설명한 바와 같이 MitoMAPR은 미토콘드리아의 길이와 미토콘드리아 네트워크의 상호 연결을 정량적으로 측정하는 강력한 자동화 매크로입니다. 그러나 여기에 나열된 제한 사항을 고려하여 신중하게 사용해야 합니다. 첫째, 매트릭스 표적 GFP를 사용하면 내막의 미토콘드리아 단편화만 관찰할 수 있으며, 이는 외막의 미토콘드리아 형태를 완전히 재현하지 못할 수 있으며, 이는 외막 분열이 없을 때 발생할 수 있기 때문입니다. 따라서 두 멤브레인의 보다 정교한 이미징을 위해서는 매트릭스 표적 형광단과 미토콘드리아 외막 표적 형광단을 모두 사용해야 합니다. 외부 미토콘드리아 막 마커는 과도하게 발현될 경우 기질 국소화 단백질과 동일한 결과를 겪을 수 있기 때문에 단일 복제 마커의 사용이 권장되며, 특히 여기에 사용된 것과 같은 완전한 단백질 대신 최소한의 미토콘드리아 국소화 서열을 사용하는 마커의 사용이 권장됩니다31.
그림 3에서 설명한 것처럼 미토콘드리아는 현미경으로 빠르게 단편화될 수 있으며, 사용하는 버퍼에 따라 샘플 슬라이드를 준비하는 다양한 방법이 형태에 큰 영향을 미칠 수 있습니다. 이 원고에 기술된 대표적인 데이터는 기생충의 미토콘드리아가 M9 하에서 최소한의 단편화와 낮은 농도의 아지드화나트륨을 겪는다는 것을 보여줍니다. 기생충을 마비시키는 데 널리 사용되는 또 다른 화학 물질인 테트라미솔은 미토콘드리아를 빠르게 조각내므로 일반적으로 사용을 피해야 함을 나타냅니다. M9와 아지드화나트륨은 미토콘드리아의 유의미한 단편화를 나타내지 않았지만, 다른 균주가 여기에 표시된 결과와 다르게 반응할 수 있다는 점에 유의하는 것이 중요합니다. 다른 연구에서는 아지드화나트륨이 미토콘드리아를 단편화할 수 있다는 것을 보여주었지만, 당사 균주의 발현 수준이 낮기 때문에 아지드화나트륨에 노출되어도 상당한 미토콘드리아 단편화를 보이지 않을 수 있습니다. 미토콘드리아 국소화 단백질의 높은 발현은 막 전위를 붕괴시킬 수 있으며, 따라서 다른 연구에서 사용된 다른 균주는 미토콘드리아를 이 연구에서 사용된 것과 동일한 농도의 아지드화나트륨으로 인한 단편화에 더 취약하게 만들 수 있습니다. 그럼에도 불구하고, 특정 돌연변이 또는 조건은 약물 유도 미토콘드리아 단편화에 더 취약할 수 있으므로 기생충을 마비시키는 화학 물질이 특정 조건에서 미토콘드리아 단편화를 일으키지 않도록 주의를 기울여야 하며, 모든 연구에서 사용하기 전에 테스트해야 합니다. 더욱이, M9 완충액에서의 수영 활동이 이전에 미토콘드리아 분열 및 융합 역학에 영향을 미치는 것으로 나타났기 때문에 M9에 지렁이를 유지하는 것만으로도 자연적인 미토콘드리아 형태와 활동에 변화가 생길 수 있습니다. 또한, M9에 기생충을 장기간 방치하면 미토콘드리아 단백질화 장애를 일으켜 미토콘드리아에 큰 영향을 미치는 저산소증 반응을 활성화할 수 있다48,49.
마지막으로, 또 다른 중요한 고려 사항은 미토콘드리아 형태의 변화가 종종 미토콘드리아 기능의 변화와 상관관계가 있지만 둘 사이에 항상 직접적인 상관관계가 있는 것은 아니라는 것입니다. 따라서 미토콘드리아 기능에 대한 보다 철저한 분석이 권장됩니다. 예를 들어, 산소 소비율은 Seahorse 기기(50)를 사용하여 측정할 수 있고, 미토콘드리아 막 전위는 JC9 또는 TMRE51과 같은 막 전위 염료를 사용하여 측정할 수 있으며, 미토콘드리아 산화 상태는 roGFP52와 같은 산화 환원 민감성 염료를 사용하여 측정할 수 있으며, 미토콘드리아 스트레스 복원력은 로테논(53)과 같은 스트레스 요인에 대한 민감도를 사용하여 측정할 수 있습니다. 미토콘드리아의 이미징은 매우 빠를 수 있기 때문에 실험 조건이 미토콘드리아 형태에 영향을 미치는지 여부를 결정하기 위한 쉬운 첫 번째 단계로 여기에서는 방법을 제공합니다. 이러한 방법은 약물 또는 유전자의 대규모 스크리닝에도 적용할 수 있으며, 추가 지표를 사용하여 보다 철저한 미토콘드리아 분석을 추적하는 것이 좋습니다. 전체적으로, 여기에서는 최소한의 실험 오류로 C . elegans 의 미토콘드리아 형태를 이미지화하는 가장 간단한 방법으로 여겨지는 방법을 검토합니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
JK는 USC Provost Fellowship의 지원을 받습니다. M.A. 및 G.G.는 T32AG052374의 지원을 받습니다. MV는 1R25AG076400에서 지원됩니다. RHS는 National Institute on Aging의 R01AG079806와 Larry L. Hillblom Foundation의 2022-A-010-SUP의 지원을 받습니다. 일부 균주는 CGC에서 제공했으며, CGC는 NIH Office of Research Infrastructure Programs 보조금 P40 OD010440에서 자금을 지원합니다. 일부 유전자 분석은 U41 보조금 HG002223으로 지원되는 Wormbase를 사용하여 수행되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 5-fluoro-2'-deoxyuridine (FUDR) | Spectrum Chemical | F2026-10GMBL | for proliferation inhibition |
| APEX IPTG | Genesee | 18-242 | for RNAi |
| Bacto Agar | VWR | 90000-764 | for NGM plates |
| Bacto Peptone | VWR | 97064-330 | for NGM plates |
| Calcium chloride dihydrate | VWR | 97061-904 | for NGM plates |
| Carbenicillin | VWR | 76345-522 | for RNAi |
| Cholesterol | VWR | 80057-932 | for NGM plates |
| Confocal microscope | Stellaris 5 | ||
| DMSO | VWR | BDH1115-1LP | solvent for drugs |
| HistoBond microscope slides | VWR | 16005-110 | for slides preparation |
| LB Broth | VWR | 95020-778 | for LB |
| LEICA S7E Dissecting Scope | Leica | 10450840 | Standard dissecting microscope |
| LEICA STELLARIS 5 | Leica | 158101100 | Confocal Microscope |
| LEICA THUNDER | Leica | 11525679 | Compound Microscope |
| Magnesium sulfate heptahydrate | VWR | 97062-132 | for NGM plates, M9 |
| pL4440 empty vector plasmid | addgene | plasmid #1654 | for empty vector plasmid |
| Potassium Chloride | VWR | 97061-566 | for bleach soluton |
| Potassium phosphate dibasic | VWR | EM-PX1570-2 | for NGM plates |
| Potassium phosphate monobasic | VWR | EM-PX1565-5 | for M9 |
| Sodium azide | VWR | 97064-646 | for paralyzing worms |
| Sodium Chloride | VWR | EM-SX0420-5 | for NGM plates, M9 |
| Sodium hypochlorite | VWR | RC7495.7-32 | for bleach solution |
| Sodium phosphate dibasic | VWR | 71003-472 | for M9 |
| Tetracycline hydrochloride | VWR | 97061-638 | for RNAi |
| WOB-L® Dry Vacuum Pumps, Standard-Duty, Welch® | VWR | 80077-612 | for aspiration |
참고문헌
- Protasoni, M., Zeviani, M. Mitochondrial structure and bioenergetics in normal and disease conditions. Int J Mol Sci. 22 (2), 586 (2021).
- Wang, Y., et al. The role of mitochondrial dynamics in disease. MedComm. 4 (6), e462 (2023).
- Kamer, K. J., Mootha, V. K. The molecular era of the mitochondrial calcium uniporter. Nat Rev Mol Cell Biol. 16 (9), 545-553 (2015).
- Chen, W., Zhao, H., Li, Y. Mitochondrial dynamics in health and disease: mechanisms and potential targets. Signal Transduct Target Ther. 8 (1), 333 (2023).
- Adebayo, M., Singh, S., Singh, A. P., Dasgupta, S. Mitochondrial fusion and fission: The fine-tune balance for cellular homeostasis. FASEB J. 35 (6), e21620 (2021).
- Madan, S., Uttekar, B., Chowdhary, S., Rikhy, R. Mitochondria lead the way: mitochondrial dynamics and function in cellular movements in development and disease. Front Cell Dev Biol. 9, 781933 (2021).
- Sharma, A., Smith, H. J., Yao, P., Mair, W. B. Causal roles of mitochondrial dynamics in longevity and healthy aging. EMBO Rep. 20 (12), e48395 (2019).
- Rolland, S. G., Lu, Y., David, C. N., Conradt, B. The BCL-2-like protein CED-9 of C. elegans promotes FZO-1/Mfn1,2- and EAT-3/Opa1-dependent mitochondrial fusion. J Cell Biol. 186 (4), 525-540 (2009).
- Labrousse, A. M., Zappaterra, M. D., Rube, D. A., van der Bliek, A. M. C. elegans dynamin-related protein DRP-1 controls severing of the mitochondrial outer membrane. Mol Cell. 4 (5), 815-826 (1999).
- Dickinson, D. J., Pani, A. M., Heppert, J. K., Higgins, C. D., Goldstein, B. Streamlined genome engineering with a self-excising drug selection cassette. Genetics. 200 (4), 1035-1049 (2015).
- Bosher, J. M., Labouesse, M. RNA interference: Genetic wand and genetic watchdog. Nat Cell Biol. 2 (2), E31-E36 (2000).
- Yu, C. C. J., et al. Expansion microscopy of C. elegans. eLife. 9, e46249 (2020).
- Zhang, S., Li, F., Zhou, T., Wang, G., Li, Z. Caenorhabditis elegans as a useful model for studying aging mutations. Front Endocrinol. 11, 554994 (2020).
- Dingley, S., et al. Mitochondrial respiratory chain dysfunction variably increases oxidant stress in Caenorhabditis elegans. Mitochondrion. 10 (2), 125-136 (2010).
- Presley, A. D., Fuller, K. M., Arriaga, E. A. MitoTracker Green labeling of mitochondrial proteins and their subsequent analysis by capillary electrophoresis with laser-induced fluorescence detection. J Chromatogr B Anal Technol Biomed Life Sci. 793 (1), 141-150 (2003).
- Mitra, K., Lippincott-Schwartz, J. Analysis of mitochondrial dynamics and functions using imaging approaches. Curr Protoc Cell Biol. Chapter 4, Unit 4.25.1-Unit 4.25.21 (2010).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. Novel imaging tools to study mitochondrial morphology in Caenorhabditis elegans. Life Sci Alliance. 7 (11), e202402918 (2024).
- Benedetti, C., Haynes, C. M., Yang, Y., Harding, H. P., Ron, D. Ubiquitin-like protein 5 positively regulates chaperone gene expression in the mitochondrial unfolded protein response. Genetics. 174 (1), 229-239 (2006).
- Begelman, D. V., et al. An aco-2::gfp knock-in enables the monitoring of mitochondrial morphology throughout C. elegans lifespan. microPublication Biol. 2022, (2022).
- Castro Torres, T., et al. Surveying low-cost methods to measure lifespan and healthspan in Caenorhabditis elegans. J Vis Exp. 183, e64091 (2022).
- Rasmussen, N. R., Reiner, D. J. Nuclear translocation of the tagged endogenous MAPK MPK-1 denotes a subset of activation events in C. elegans development. J Cell Sci. 134 (17), jcs258456 (2021).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Luz, A. L., et al. Mitochondrial morphology and fundamental parameters of the mitochondrial respiratory chain are altered in Caenorhabditis elegans strains deficient in mitochondrial dynamics and homeostasis processes. PLoS One. 10 (6), e0130940 (2015).
- Oorloff, M., et al. Mechanical stress through growth on stiffer substrates impacts animal health and longevity in C. elegans. bioRxiv. , (2024).
- Chu, X., Wu, S., Raju, R. NLRX1 regulation following acute mitochondrial injury. Front Immunol. 10, 2431 (2019).
- Shah, P., Bao, Z., Zaidel-Bar, R. Visualizing and quantifying molecular and cellular processes in Caenorhabditis elegans using light microscopy. Genetics. 221 (4), iyac068 (2022).
- Wang, Y., Wang, P., Li, C. Fluorescence microscopic platforms imaging mitochondrial abnormalities in neurodegenerative diseases. Adv Drug Deliv Rev. 197, 114841 (2023).
- Ding, J., et al. An expanded GCaMP reporter toolkit for functional imaging in Caenorhabditis elegans. G3 (Bethesda, Md). 13 (10), jkad183 (2023).
- Sarasija, S., Norman, K. R. Analysis of mitochondrial structure in the body wall muscle of Caenorhabditis elegans. Bio-Protocol. 8 (7), e2801 (2018).
- Chaweeborisuit, P., Suriyonplengsaeng, C., Suphamungmee, W., Sobhon, P., Meemon, K. Nematicidal effect of plumbagin on Caenorhabditis elegans: A model for testing a nematicidal drug. Z Naturforsch C J Biosci. 71 (5-6), 121-131 (2016).
- Valera-Alberni, M., Yao, P., Romero-Sanz, S., Lanjuin, A., Mair, W. B. . Novel imaging tools to study mitochondrial dynamics in Caenorhabditis elegans. 7, 11 (2024).
- Bolognesi, B., Lehner, B. Reaching the limit. eLife. 7, e39804 (2018).
- Jishi, A., Qi, X. Altered mitochondrial protein homeostasis and proteinopathies. Front Mol Neurosci. 15, 867935 (2022).
- Morton, K. S., Wahl, A. K., Meyer, J. N. The effect of common paralytic agents used for fluorescence imaging on redox tone and ATP levels in Caenorhabditis elegans. PLoS One. 19 (4), e0292415 (2024).
- Noumi, T., Maeda, M., Futai, M. Mode of inhibition of sodium azide on H+-ATPase of Escherichia coli. FEBS Lett. 213 (2), 381-384 (1987).
- Siete, C., Xiong, R., Khalid, A., Hsieh, Y. -. W., Chuang, C. -. F. Immobilization of C. elegans with different concentrations of an anesthetic for time-lapse imaging of dynamic protein trafficking in neurons. microPublication Biology. 2024, (2024).
- Mitchell, D. H., Stiles, J. W., Santelli, J., Sanadi, D. R. Synchronous growth and aging of Caenorhabditis elegans in the presence of fluorodeoxyuridine. J Gerontol. 34 (1), 28-36 (1979).
- Santi, D. V., McHenry, C. S. 5-Fluoro-2'-deoxyuridylate: covalent complex with thymidylate synthetase. Proc Natl Acad Sci U S A. 69 (7), 1855-1857 (1972).
- Wang, H., Zhao, Y., Zhang, Z. Age-dependent effects of floxuridine (FUdR) on senescent pathology and mortality in the nematode Caenorhabditis elegans. Biochem Biophys Res Commun. 509 (3), 694-699 (2019).
- Beanan, M. J., Strome, S. Characterization of a germ-line proliferation mutation in C. elegans. Development (Cambridge, England). 116 (3), 755-766 (1992).
- Austin, J., Kimble, J. glp-1 is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans. Cell. 51 (4), 589-599 (1987).
- Garigan, D., et al. Genetic analysis of tissue aging in Caenorhabditis elegans: A role for heat-shock factor and bacterial proliferation. Genetics. 161 (3), 1101-1112 (2002).
- Zhang, Y., et al. Neuronal TORC1 modulates longevity via AMPK and cell nonautonomous regulation of mitochondrial dynamics in C. elegans. eLife. 8, e49158 (2019).
- Dutta, N., et al. Investigating impacts of the mycothiazole chemotype as a chemical probe for the study of mitochondrial function and aging. GeroScience. 46 (6), 6009-6028 (2024).
- Tronstad, K., et al. Regulation and quantification of cellular mitochondrial morphology and content. Curr Pharm Des. 20 (35), 5634-5652 (2014).
- Nikolaisen, J., et al. Automated quantification and integrative analysis of 2D and 3D mitochondrial shape and network properties. PLoS One. 9 (7), e101365 (2014).
- Lee, J. S., Wee, T. L. E., Brown, C. M. Calibration of wide-field deconvolution microscopy for quantitative fluorescence imaging. J Biomol Tech. 25 (1), 31-40 (2014).
- Hong, M., Kwon, J. Y., Shim, J., Lee, J. Differential hypoxia response of hsp-16 genes in the nematode. J Mol Biol. 344 (2), 369-381 (2004).
- Yan, J., Sun, C. L., Shin, S., Van Gilst, M., Crowder, C. M. Effect of the mitochondrial unfolded protein response on hypoxic death and mitochondrial protein aggregation. Cell Death Dis. 12 (7), 711 (2021).
- Koopman, M., et al. A screening-based platform for the assessment of cellular respiration in Caenorhabditis elegans. Nat Protoc. 11 (10), 1798-1816 (2016).
- Shpilka, T., et al. UPRmt scales mitochondrial network expansion with protein synthesis via mitochondrial import in Caenorhabditis elegans. Nat Comm. 12 (1), 479 (2021).
- Braeckman, B. P., Smolders, A., Back, P., De Henau, S. In vivo detection of reactive oxygen species and redox status in Caenorhabditis elegans. antioxidants & redox signaling. 25 (10), 577-592 (2016).
- Bar-Ziv, R., et al. Measurements of physiological stress responses in C. elegans. J Vis Exp. 159, e61001 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유